拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用的制作方法
本发明属于基因工程技术领域,具体涉及一种在拟南芥中表达的WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用,包括核苷酸编码序列的克隆,表达载体构建,对拟南芥此基因内源的不同器官、组织的空间表达模式,长日照条件下此基因的表达模式变化进行分析鉴定所使用的方法,以及转化此基因于拟南芥(Columbia)内,进行分子鉴定和开花实验,检测其基因表达量变化及对开花调控的影响。
背景技术:
转录因子又称反式作用因子,是指能与真核基因启动子区域中的顺式作用元件发生特异性结合,激活或抑制下游基因的转录,从而保证目的基因在特定的强度、在特定的时间与空间表达的蛋白质分子(刘蕾等,2008)。
WRKY蛋白N端是一个由60个氨基酸组成的保守结构域,其中WRKYGQK是N端绝对保守的7个氨基酸残基,也就是WRKY结构域。少数WRKY蛋白的WRKY结构域可以被WRRY,WSKY,WKRY,WVKY或WKKY取代(Rushton and Somssich et al., 2010)。在蛋白C是一个锌指结构域。根据WRKY结构域和锌指结构的特点,WRKY家族可以被分成3 类,家族I 含有2 个WRKY结构域; 家族II 多数只有1 个WRKY域,家族I和II包含相同的Cys2 - His2 锌指结构序列; 家族III有1 个WRKY域,并包含着2 种不同的锌指结构序列,即Cys2 - His /Cys和Cys2 - His2。这三类成员可以特异性结合到W box (T)(T)TGAC(C/T)上(ISHIGURO and NAKAMURA, 1994; Eulgem and Rushton et al., 2000)。
WRKY家族转录因子最初是由2位日本科学家从甜薯中克隆出来(ISHIGURO and NAKAMURA, 1994)。经过二十年的发展,WRKY家族成员在不断地丰富。据统计,WRKY家族在拟南芥和水稻中分别有70、100多个(Hu and Ye et al., 2015),大豆中约有197个成员(Schmutz and Cannon et al., 2010),棉花中已发现超过200多个成员(Dou and Zhang et al., 2014)。此外,还存在于油菜、玉米等其它植物中。WRKY家族已成为植物中最大的转录因子家族之一,并广泛参与植物多项生理过程,如响应生物与非生物胁(Huang and Duman, 2002; Rizhsky and Davletova et al., 2004; Zheng and Abu Qamar et al., 2006),调控叶片衰老(Miao and Laun et al., 2004),参与种皮发育(Johnson and Kolevski et al., 2002)以及GA、ABA信号传导等(Zhang and Xie et al., 2004; Zhang and Shin et al., 2009),从而最终影响植物的新陈代谢,形态建成等。拟南芥AtWRKY44,又被称为 TRANSPARENT TESTA GLABRA2(TTG2),是WRKY家族中第一个确认参与植物形态建成的蛋白,参与拟南芥的表皮毛形成、种皮发育、响应干旱下的开花等(Johnson and Kolevski et al., 2002; Han and Zhang et al., 2013)。
本发明通过克隆AtWRKY44基因,对其在长日照条件下的开花时间下相关mRNA节律性表达水平进行鉴定,并分析AtWRKY44参与光周期开花调控途径,发现在GI过表达和gi-2突变体中表达量均下调,同时WRKY44与miRNA172靶基因TOE1蛋白能在酵母内进行互作,说明GI、WRKY44和TOE1三者存在某种平衡。将该基因转入野生型拟南芥后,观察转基因株系在长日照下的开花时间以及干旱胁迫下的开花时间。这极大地丰富了WRKY家族转录因子生物学功能,为研究拟南芥如何调控开花打下基础,为最终提高产量提供基因来源和技术支持。
技术实现要素:
本发明的目的在于丰富已有的拟南芥基因AtWRKY44的生物学功能,并提供该基因的应用。
本发明首先提供拟南芥WRKY家族转录因子基因,名为AtWRKY44,为具有特定序列的DNA分子,基因组全长2226bp,其中编码序列1290bp,具体如SEQ ID NO.1所示。
本发明还提供这种AtWRKY44的蛋白编码序列,有321个氨基酸残基,其氨基酸序列为SEQ ID NO.2所示。
本发明还提供用于调取获得拟南芥样品中基因AtWRKY44的一对核苷酸引物。该引物根据基因AtWRKY44设计,使用此对引物对拟南芥样品cDNA进行PCR扩增可获得长1290bp的基因片段。具体的引物序列为:
Forward primer 5'ATGGAGGTGAATGATGGTGAAAG3'(SEQ ID NO.3);
Reverse primer5'TCAAATTGTTTGCTTAGAAAG3' (SEQ ID NO.4)。
本发明还提供用于构建pSKM36- AtWRKY44-MYC载体的引物序列,根据所述基因AtWRKY44编码序列设计,含有AscI/SnaBI酶切位点:
Forward primer 5'GCGCGCATGGAGGTGAATGATGGTGAAAG3'(Asc I)(SEQ ID NO.5);
Reverse primer5'TACGTATCAAATTGTTTGCTTAGAAAG3'(SnaB I)(SEQ ID NO.6)。
本发明还提供构建pSKM36 -AtWRKY44表达载体的方法,其具体步骤如下:
(1)以pCRBlunt-AtWRKY44载体质粒为模板,利用引物SEQ ID NO.5和SEQ ID NO.6克隆出含有SEQ ID NO.1的序列;
(2)将上述序列构建到pSKM36载体中,酶切位点分别为5'- AscIⅠ,3' -SnaBⅠ。经转化,进行阳性克隆的PCR验证。
本发明还提供检测拟南芥基因AtWRKY44在长日照下mRNA表达量变化的方法,包括利用所述基因AtWRKY44的核苷酸序列作为设计引物的保守区段,调取其序列的引物:
Forward primer5'TTTCTTCGTTCCCTAACTCCT3'(SEQ ID NO.7);
Reverse primer5'CCACTCTTTCACCATCATTCAC3' (SEQ ID NO.8)。
本发明还提供检测拟南芥在长日照条件下,基因AtWRKY44表达含量变化的方法,具体步骤为:将拟南芥在长日照下培养至10天后,从开灯时间0小时开始,每隔4小时提取拟南芥的总RNA;利用反转录试剂盒将总RNA反转录成cDNA,利用引物SEQ ID NO.7和SEQ ID NO.8,进行定量PCR检测。
本发明还提供检测拟南芥GI过表达转基因株系中AtWRKY44基因启动子中不同区域的丰富程度所用的两对引物,即利用该基因启动子的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'AAATTGCAAGCTAACAAAGTAAC3' (SEQ ID NO.9);
Reverse primer5'AAATTCAGTCATCTTGTCCAATAC3'(SEQ ID NO.10);
Forward primer5'ATAAGTTGGTACAGTTCAGTTCAC3'(SEQ ID NO.11);
Reverse primer5'TTTAGTTGAAGACTACAGCGAC3'(SEQ ID NO.12)。
本发明还提供检测拟南芥基因FT的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'GGTGGAGAAGACCTCAGGAA3'( SEQ ID NO.13);
Reverse primer5'GGTTGCTAGGACTTGGAACATC3' (SEQ ID NO.14)。
本发明还提供检测拟南芥基因CO的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'ATTCTGCAAACCCACTTGCT3'(SEQ ID NO.15);
Reverse primer5'TTCCAGAAATTGGTAGAATTGGA3'(SEQ ID NO.16)。
本发明还提供检测拟南芥基因TOE1的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'GCGTGGAGTTAGCTTGAGGA3'(SEQ ID NO.17);
Reverse primer5'TCCAGTAAAGGCGATGATCC3'(SEQ ID NO.18)。
本发明还提供检测拟南芥基因TOE2的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'CCTCCGCATAACTCAAGGTT3'(SEQ ID NO.19);
Reverse primer5'GAATTCAGCGTCGTTTTGG3'(SEQ ID NO.20)。
本发明还提供检测拟南芥基因TOE3的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'GGAAGCAAGTGTACTTAGGTGGA3'(SEQ ID NO.21);
Reverse primer5'TAATCGCCGCTCGATCATA3'(SEQ ID NO.22)。
本发明还提供检测拟南芥基因SMZ的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'GGATGATTATAGGCATGACATCG3'(SEQ ID NO.23);
Reverse primer5'TCTCGCCTAAGTGTTTGCAC3'(SEQ ID NO.24)。
本发明还提供检测拟南芥基因SNZ的mRNA表达水平的一对引物,即利用该基因的核苷酸序列作为设计引物的保守区段,调取其序列的引物序列:
Forward primer5'GGCCGTTGGGAATCTCAT3'(SEQ ID NO.25);
Reverse primer5'GTACGCTCTTGCGGCTGT3'(SEQ ID NO.26)。
本发明还提供检测拟南芥在转入AtWRKY44基因后,对FT,CO,TOE1及其同源基因的转录水平的影响的方法,即利用引物序列SEQ ID NO.13,SEQ ID NO.14,SEQ ID NO.15,SEQ ID NO.16,SEQ ID NO.17,SEQ ID NO.18,SEQ ID NO.19,SEQ ID NO.20,SEQ ID NO.21,SEQ ID NO.22,SEQ ID NO.23,SEQ ID NO.24,SEQ ID NO.25,SEQ ID NO.26进行定量PCR检测。
本发明对拟南芥cDNA样品进行Real-timePCR, 然后检测该基因在长日照下的节律性表达;样品为拟南芥的RNA 经过逆转录后所得cDNA,其具体步骤如下:
(1)提取拟南芥器官的总RNA(Trizol,市售);
(2)利用反转录试剂盒(市售)将总RNA反转录成cDNA,根据SEQ ID NO.1,跨越两个外显子的131bp作为PCR产物,进行实时定量PCR检测。
本发明还提供检测拟南芥哥伦比亚在转入基因AtWRKY44后,拟南芥开花时间和FT基因表达量的变化的方法,具体是利用反转录试剂盒将总RNA反转录成cDNA,利用引物SEQ ID NO.13和SEQ ID NO.14,进行定量PCR检测。结果显示:转入基因AtWRKY44后,与野生型对照组相比,拟南芥延迟开花,FT表达量下调。
本发明中,可选用本领域已经知道的各种载体,如市售的载体以及质粒。
本发明研究了长日照下AtWRKY4不同转基因株系的mRNA水平和开花时间。AtWRKY44与光周期调控途径中基因GIGANTEA(简写为GI),TARGET OF EAT1(简写为TOE1)以及成花素FLOWER LOCUS T(简写为FT)的关系。长日照下,wrky44突变体开花时间提前,并且AtWRKY44在拟南芥哥伦比亚(Columbia,简写为Col)中的mRNA表达上具有节律性。Real-time PCR检测结果显示:AtWRKY44在植物的各个器官中均有表达,其中在叶和种皮表达量最高。GI蛋白可以特异地结合在AtWRKY44基因启动子中两个位点上。酵母双杂交实验显示AtWRKY44能与miRNA172的靶基因TOE1蛋白结合,并且TOE1及其同源基因的mRNA水平在wrky44突变体中有所上调。同时,FT的mRNA水平在wrky44突变体中显著高于野生型Col。FTmRNA的积累有助于拟南芥提前开花,AtWRKY44基因对开花时间的调控,可用于植物育种增产等。
可见,本发明提供的拟南芥基因AtWRKY44可用于植物品种改良,如用于改变植物的开花时间,避开不利的非生物胁迫,最后提高作物产量。
附图说明
图1拟南芥中WRKY44基因的节律性表达。
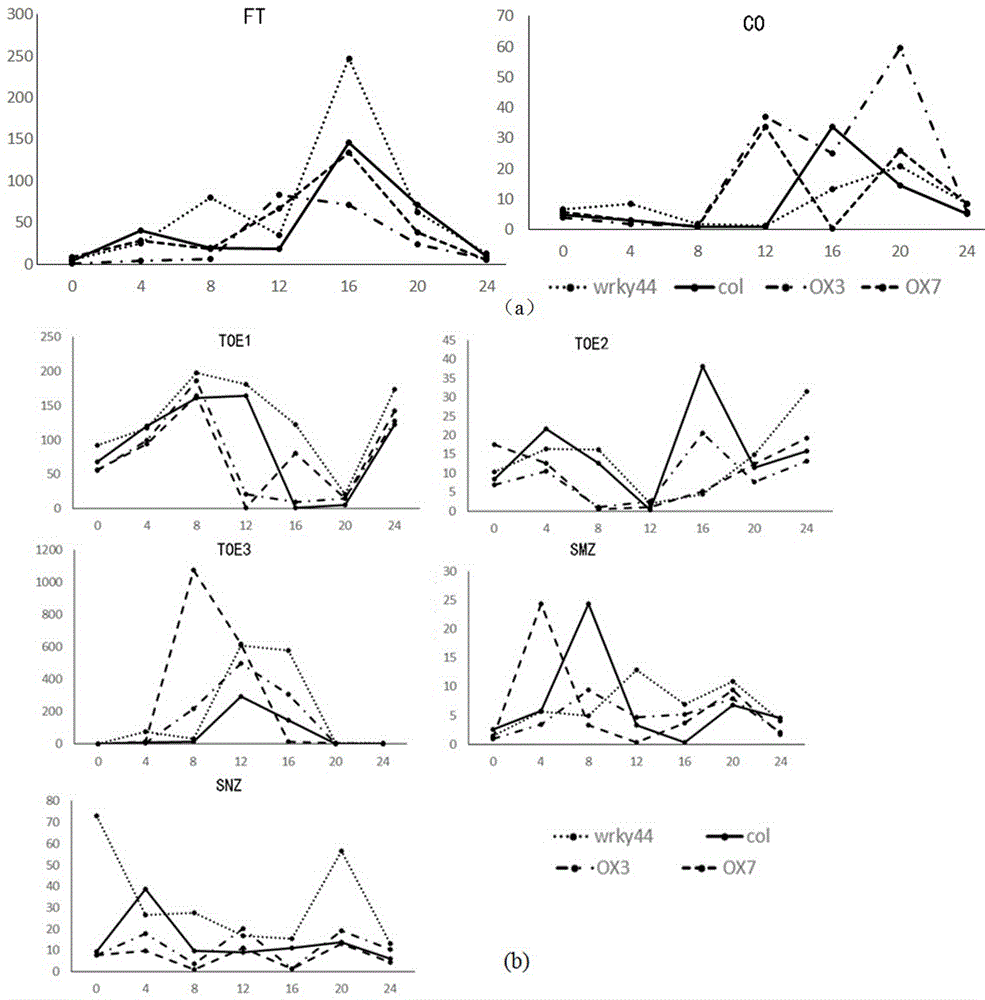
图2 WRKY44转基因株系中FT和CO的节律性表达。WRKY44转基因株系中TOE1及其同源基因TOE2,TOE3,SMZ和SNZ的节律性表达。
图3 WRKY44与TOE1蛋白的酵母双杂交分析。
图4染色质免疫共沉淀(CHIP)分析。
具体实施方式
下面结合具体实施实例进一步阐释本发明。应理解,这些实施例仅以用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体的实验方法,均可按照常规方法进行。如Sambrook等分子克隆:实验手册(New York: Cold Spring Harbor Laboratory Press, 1989)中所述条件,或按照制造生产厂商的使用说明。
实施例1,拟南芥基因AtWRKY44的克隆
1. 拟南芥品种Col在温室中培养:生长条件为光周期16h /8h (L/D),23℃;
2. RNA提取,取100毫克左右新鲜的拟南芥植物组织材料,液氮充分研磨。加1 ml Trizol试剂,涡旋15 s后室温放置5 min。加0.2 ml氯仿,去蛋白,12000rpm离心10min后上清转移至新的离心管,加等体积异丙醇,充分混匀,室温放置10 min,12000 rpm离心10min,弃上清,用DEPC处理过的水配制的75%乙醇1 ml洗涤沉淀,重复一次。室温干燥5-10 min,溶于20 μl DEPC水中,测OD值,电泳检测;
3. 基因的克隆。通过对应的拟南芥的AtWKY44基因进行生信分析,设计引物。以逆转录的拟南芥cDNA第一链为模板,利用正向引物和反向引物进行PCR,获得基因全长,具体序列信息参见SEQ ID NO.1。
实施例2,拟南芥中WRKY44基因的节律性表达分析
提取拟南芥总RNA,利用反转录试剂盒将RNA反转录出cDNA,利用引物SEQ ID NO.7和SEQ ID NO.8,进行实时荧光定量PCR检测。结果显示该基因具有光周期相关的节律性表达(图1)。
实施例3 ,WRKY44转基因株系中FT,CO和TOE1及其同源基因的节律性表达分析
分别提取各转基因株系拟南芥总RNA,利用反转录试剂盒将RNA反转录出cDNA,利用引物SEQ ID NO.13-SEQ ID NO.26,进行实时荧光定量PCR检测。结果显示这些基因的转录水平在WRKY44转基因各株系有不同程度的变化(图2)。FT基因在wrky44, OXs株系中的表达模式基本不变,但是表达水平有所变化。在ZT8、ZT16时,FT在突变体中的mRNA水平高于col。过表达OX3中FT含量低于col。CO在col中的表达量在ZT16时达到峰值,但是在WRKY44-OX中在ZT12,ZT20达到峰值。wrky44突变体中CO表达量一直处于比较低的水平。TOE1基因的mRNA水平在wrky44突变体中上调,在WRKY44过表达株系中下调,TOE2在四个株系中的表达量没有明显的变化,col中TOE3含量均低于wrky44和WRKY44-OXs,OX7和col,wrky44和OX3中具有相似的表达水平和表达模式,SNZ在wrky44突变体中表达量上调,在过表达中表达量下调,表明WRKY44不仅能与TOE1在蛋白水平上互作,在转录水平上也能调控TOE1及其同源基因的表达。
实施例4, WRKY44与TOE1蛋白的酵母双杂
1、分别在YPDA平板上复苏酵母Y187和AH109两种菌株,挑取单克隆各一个,于5mlYPDA液体培养基中,30℃摇床过夜;
2、在50ml YPDA中扩大培养,至OD值为0.4-0.6;
3、在1.5ml灭菌的离心管中收菌,1000rpm离心5min,常温;
4、去上清,用无菌水重悬,1000rpm离心5min;
5、去上清,用1.1XLiAc/TE重悬酵母,5000rpm离心30s;
6、去上清,加入50ml1.1XLiAc/TE,分别在AH109和Y187菌株中加入相应的质粒各2μl, 95℃预变性的鲑鱼精DNA 5μl,最后加入500μl 40%的PEG,30℃摇床30min;
7、42℃ 15min,每5min摇晃一次。5000rpm离心15s,去上清,加入100μl 无菌水,涂板,置于30℃培养箱;
8、约2-3d后,挑取将AH109和Y187菌株上的单克隆,用无菌水混匀后,在2XYPDA平板上点样,于30℃培养箱结合 4h;
9、将结合后的克隆再点样在含有X-gal和Aureobasidin A(AbA)的二缺平板上。然后观察酵母生长状态。携带AD-44和BD-TOE1的酵母能够在平板上生长(图3),说明WRKY44与TOE1蛋白能再酵母内互作。
实施例5,染色质免疫共沉淀(CHIP)分析
利用小鼠单克隆MYC抗体,将培养合适的拟南芥幼苗通过染色质免疫沉淀,提取符合要求的DNA。并在WRKY44启动子区域上设计18对引物,进行实时荧光定量PCR,寻找GI在WRKY44启动子上可能的结合位点(图4)。结果显示,第11和13片段具有较高的富集,说明该位点正是GI在WRKY44启动子上的结合位点。
参考文献
Dou, L. and X. Zhang, et al. (2014). "Genome-wide analysis of the WRKY gene family in cotton." MOLECULAR GENETICS AND GENOMICS 289 (6): 1103-1121.
Eulgem, T. and P. J. Rushton, et al. (2000). "The WRKY superfamily of plant transcription factors." TRENDS IN PLANT SCIENCE 5 (5): 199-206.
Han, Y. and X. Zhang, et al. (2013). "The Suppression of WRKY44 by GIGANTEA-miR172 Pathway Is Involved in Drought Response of Arabidopsis thaliana." PLOS ONE 8 (e7354111).
Hu, L. and M. Ye, et al. (2015). "The Rice Transcription Factor WRKY53 Suppresses Herbivore-Induced Defenses by Acting as a Negative Feedback Modulator of Mitogen-Activated Protein Kinase Activity." PLANT PHYSIOLOGY 169 (4): 2907-2921.
Huang, T. and J. G. Duman (2002). "Cloning and characterization of a thermal hysteresis (antifreeze) protein with DNA-binding activity from winter bittersweet nightshade, Solanumdulcamara." PLANT MOLECULAR BIOLOGY 48 (4): 339-350.
Ishiguro, S. and K. Nakamura (1994). "Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5' upstream regions of genes-coding for sporamin and beta-amylase from sweet-potato." MOLECULAR & GENERAL GENETICS 244 (6): 563-571.
Johnson, C. S. and B. Kolevski, et al. (2002). "TRANSPARENT TESTA GLABRA2, a trichome and seed coat development gene of Arabidopsis, encodes a WRKY transcription factor." PLANT CELL 14 (6): 1359-1375.
Miao, Y. and T. Laun, et al. (2004). "Targets of the WRKY53 transcription factor and its role during leaf senescence in Arabidopsis." PLANT MOLECULAR BIOLOGY 55 (6): 853-867.
Rizhsky, L. and S. Davletova, et al. (2004). "The zinc finger protein Zat12 is required for cytosolic ascorbate peroxidase 1 expression during oxidative stress in Arabidopsis." JOURNAL OF BIOLOGICAL CHEMISTRY 279 (12): 11736-11743.
Rushton, P. J. and I. E. Somssich, et al. (2010). "WRKY transcription factors." TRENDS IN PLANT SCIENCE 15 (5): 247-258.
Schmutz, J. and S. B. Cannon, et al. (2010). "Genome sequence of the palaeopolyploid soybean." NATURE 463 (7278): 178-183.
Zhang, Z. L. and Z. Xie, et al. (2004). "A rice WRKY gene encodes a transcriptional repressor of the gibberellin signaling pathway in aleurone cells." PLANT PHYSIOLOGY 134 (4): 1500-1513.
Zhang, Z. and M. Shin, et al. (2009). "A negative regulator encoded by a rice WRKY gene represses both abscisic acid and gibberellins signaling in aleurone cells." PLANT MOLECULAR BIOLOGY 70 (1-2): 139-151.
Zheng, Z. and S. Abu Qamar, et al. (2006). "Arabidopsis WRKY33 transcription factor is required for resistance to necrotrophic fungal pathogens." PLANT JOURNAL 48 (4): 592-605.。
<110> 复旦大学
<120> 拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用
<130> 001
<160> 26
<170> PatentIn version 3.3
<210> 1
<211> 1290
<212> DNA
<213>
<400> 1
atggaggtga atgatggtga aagagtggta atagcaaaac cagttgcctc aagaccttct 60
tcaagttctg gcttcaggac attcactgag cttcttactg attcagttac cgtatctccg 120
caaacgactt gccacgagat tgtagacgct gctataagac caaagacttt gagattcaat 180
cagccagtag cagcttcggt ctcatgtcca cgggctgaag taaaaggtat tggaaatgga 240
atgtcttgtg atgatgattc agatagcaga aactacgtcg tttataaacc gaaagcaaag 300
cttgtctcca aagcaaccgt ctctgcattg gctaatatgc ttcaggggaa tcgtcaacaa 360
acttggagac aaagcgaagc agtatcctat gggaagagtg tgagccaagg tacacatcga 420
gcaggtccta atctagtcca gaaagttcca tcttttacgg aatcagagac atctactggg 480
gatagatctt ctgtggatgg atacaactgg aggaaatacg gacagaagca agttaaagga 540
agtgagtgtc caaggagtta ttacaaatgc acacacccga aatgtccggt gaagaagaaa 600
gtagagagat cagtggaagg tcaagtttca gagattgtgt atcaaggtga acataatcac 660
tcaaagccat cttgtcctct tccacggcgt gcttcatcat caatctcttc agggtttcaa 720
aaaccaccaa aatcgattgc ttctgaagga tcaatgggac aagaccctaa caataatctc 780
tattctcctc tatggaacaa tcaaagcaat gactctactc aaaacagaac agagaagatg 840
agtgaaggtt gtgttattac accatttgag tttgctgttc caagatcgac aaattcaaat 900
cccggaactt cagattccgg ttgcaagagt agccaatgtg atgaaggaga gcttgatgat 960
ccaagcagaa gcaaaagaag gaagaacgag aagcaatcta gtgaagcagg agtatcgcaa 1020
ggctcagtgg aatcagacag tcttgaagat ggattcaggt ggagaaagta tggacaaaaa 1080
gtggtaggag gcaatgcgta tccaagaagt tattacagat gcacaagtgc taattgcaga 1140
gcaaggaaac atgttgaacg agcgagtgat gatccaagag ctttcattac aacctacgag 1200
ggtaaacata atcatcattt gctcttgagc cctccatctt cctctactct tccctttaac 1260
tccccacaac tttctaagca aacaatttga 1290
<210> 2
<211> 429
<212> PRT
<213>
<400> 2
Met Glu Val Asn Asp Gly Glu Arg Val Val Ile Ala Lys Pro Val Ala
1 5 10 15
Ser Arg Pro Ser Ser Ser Ser Gly Phe Arg Thr Phe Thr Glu Leu Leu
20 25 30
Thr Asp Ser Val Thr Val Ser Pro Gln Thr Thr Cys His Glu Ile Val
35 40 45
Asp Ala Ala Ile Arg Pro Lys Thr Leu Arg Phe Asn Gln Pro Val Ala
50 55 60
Ala Ser Val Ser Cys Pro Arg Ala Glu Val Lys Gly Ile Gly Asn Gly
65 70 75 80
Met Ser Cys Asp Asp Asp Ser Asp Ser Arg Asn Tyr Val Val Tyr Lys
85 90 95
Pro Lys Ala Lys Leu Val Ser Lys Ala Thr Val Ser Ala Leu Ala Asn
100 105 110
Met Leu Gln Gly Asn Arg Gln Gln Thr Trp Arg Gln Ser Glu Ala Val
115 120 125
Ser Tyr Gly Lys Ser Val Ser Gln Gly Thr His Arg Ala Gly Pro Asn
130 135 140
Leu Val Gln Lys Val Pro Ser Phe Thr Glu Ser Glu Thr Ser Thr Gly
145 150 155 160
Asp Arg Ser Ser Val Asp Gly Tyr Asn Trp Arg Lys Tyr Gly Gln Lys
165 170 175
Gln Val Lys Gly Ser Glu Cys Pro Arg Ser Tyr Tyr Lys Cys Thr His
180 185 190
Pro Lys Cys Pro Val Lys Lys Lys Val Glu Arg Ser Val Glu Gly Gln
195 200 205
Val Ser Glu Ile Val Tyr Gln Gly Glu His Asn His Ser Lys Pro Ser
210 215 220
Cys Pro Leu Pro Arg Arg Ala Ser Ser Ser Ile Ser Ser Gly Phe Gln
225 230 235 240
Lys Pro Pro Lys Ser Ile Ala Ser Glu Gly Ser Met Gly Gln Asp Pro
245 250 255
Asn Asn Asn Leu Tyr Ser Pro Leu Trp Asn Asn Gln Ser Asn Asp Ser
260 265 270
Thr Gln Asn Arg Thr Glu Lys Met Ser Glu Gly Cys Val Ile Thr Pro
275 280 285
Phe Glu Phe Ala Val Pro Arg Ser Thr Asn Ser Asn Pro Gly Thr Ser
290 295 300
Asp Ser Gly Cys Lys Ser Ser Gln Cys Asp Glu Gly Glu Leu Asp Asp
305 310 315 320
Pro Ser Arg Ser Lys Arg Arg Lys Asn Glu Lys Gln Ser Ser Glu Ala
325 330 335
Gly Val Ser Gln Gly Ser Val Glu Ser Asp Ser Leu Glu Asp Gly Phe
340 345 350
Arg Trp Arg Lys Tyr Gly Gln Lys Val Val Gly Gly Asn Ala Tyr Pro
355 360 365
Arg Ser Tyr Tyr Arg Cys Thr Ser Ala Asn Cys Arg Ala Arg Lys His
370 375 380
Val Glu Arg Ala Ser Asp Asp Pro Arg Ala Phe Ile Thr Thr Tyr Glu
385 390 395 400
Gly Lys His Asn His His Leu Leu Leu Ser Pro Pro Ser Ser Ser Thr
405 410 415
Leu Pro Phe Asn Ser Pro Gln Leu Ser Lys Gln Thr Ile
420 425
<210> 3
<211> 23
<212> DNA
<213>
<400> 3
atggaggtga atgatggtga aag 23
<210> 4
<211> 21
<212> DNA
<213>
<400> 4
tcaaattgtt tgcttagaaa g 21
<210> 5
<211> 29
<212> DNA
<213>
<400> 5
gcgcgcatgg aggtgaatga tggtgaaag 29
<210> 6
<211> 27
<212> DNA
<213>
<400> 6
tacgtatcaa attgtttgct tagaaag 27
<210> 7
<211> 21
<212> DNA
<213>
<400> 7
tttcttcgtt ccctaactcc t 21
<210> 8
<211> 22
<212> DNA
<213>
<400> 8
ccactctttc accatcattc ac 22
<210> 9
<211> 23
<212> DNA
<213>
<400> 9
aaattgcaag ctaacaaagt aac 23
<210> 10
<211> 24
<212> DNA
<213>
<400> 10
aaattcagtc atcttgtcca atac 24
<210> 11
<211> 24
<212> DNA
<213>
<400> 11
ataagttggt acagttcagt tcac 24
<210> 12
<211> 22
<212> DNA
<213>
<400> 12
tttagttgaa gactacagcg ac 22
<210> 13
<211> 20
<212> DNA
<213>
<400> 13
ggtggagaag acctcaggaa 20
<210> 14
<211> 22
<212> DNA
<213>
<400> 14
ggttgctagg acttggaaca tc 22
<210> 15
<211> 20
<212> DNA
<213>
<400> 15
attctgcaaa cccacttgct 20
<210> 16
<211> 23
<212> DNA
<213>
<400> 16
ttccagaaat tggtagaatt gga 23
<210> 17
<211> 20
<212> DNA
<213>
<400> 17
gcgtggagtt agcttgagga 20
<210> 18
<211> 20
<212> DNA
<213>
<400> 18
tccagtaaag gcgatgatcc 20
<210> 19
<211> 20
<212> DNA
<213>
<400> 19
cctccgcata actcaaggtt 20
<210> 20
<211> 19
<212> DNA
<213>
<400> 20
gaattcagcg tcgttttgg 19
<210> 21
<211> 23
<212> DNA
<213>
<400> 21
ggaagcaagt gtacttaggt gga 23
<210> 22
<211> 19
<212> DNA
<213>
<400> 22
taatcgccgc tcgatcata 19
<210> 23
<211> 23
<212> DNA
<213>
<400> 23
ggatgattat aggcatgaca tcg 23
<210> 24
<211> 20
<212> DNA
<213>
<400> 24
tctcgcctaa gtgtttgcac 20
<210> 25
<211> 18
<212> DNA
<213>
<400> 25
ggccgttggg aatctcat 18
<210> 26
<211> 18
<212> DNA
<213>
<400> 26
gtacgctctt gcggctgt 18
- 还没有人留言评论。精彩留言会获得点赞!
- 拟南芥SSCD1基因突变在调控植物体内茉莉酸合成中的应用的制作方法与工艺
- 一种基因调控网络的优化控制方法与流程
- 拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用的制作方法与工艺
- 针对免疫调控能力相关基因个体化配方食品及其制备方法与流程
- REVOLUTA基因在调控豆科植物小叶片数目和叶茎比中的应用的制作方法与工艺
- 玉米锌铁调控转运体ZmZIP3基因及其应用的制作方法与工艺
- 拟南芥基因SPOC1在调控植株开花期中的应用的制作方法与工艺
- 调控石榴花胚珠发育的PgAG基因及其用途的制作方法与工艺
- 光效调控基因HPE1及其应用的制作方法与工艺
- 菘蓝IiAP2/ERF049基因在调控木脂素类化合物合成中的应用的制作方法与工艺