光效调控基因HPE1及其应用的制作方法
本发明涉及一种新基因及其应用,特别涉及一种新的光效调控基因及其应用。
背景技术:
随着全球人口数量的不断增加,粮食短缺逐渐成为世界性问题之一,提高作物产量迫在眉睫(Food and Agriculture Organization of the United Nations, 2015)。光合作用被称为地球上最伟大的生化反应,它可以利用太阳能把无机物转换为有机物,完成物质和能量的双重转换(Vinyard et al., 2013),其效率直接决定着作物产量(Long et al., 2006a)。据统计,地球上所有的植物通过光合作用所利用的光能,整体平均不超过0.1%。太阳光能中真正被转化为农作物各类物质的能量不到转变为生物量的太阳能的5.0%。因此,光合作用效率的有效提高,是增加作物产量的重要途径,为粮食短缺问题的最终解决提供帮助。
植物对光能利用率是指太阳光中的能量被植物进行光合作用时转化成化学能而贮存于光合产物中的百分率。越来越多的研究表明,植物光合作用远未达到其最大理论效率(Long et al., 2015)。在过去的50年中,科学家通过生物化学、遗传学以及结构学等方法对于光合作用的调控机理的认识有了很大的提高,不仅了解了光合作用的中间过程,而且鉴定了光合作用调控关键蛋白(Mulo et al., 2008),并解析了光合系统复合物三维结构 (Qin et al., 2015),为提高光合作用效率提供了理论依据。另外,超级计算机的快速发展可以允许科学家对光合作用过程中的每一个阶段进行模拟,确定并改进植物生长中的瓶颈问题(Zhu et al., 2007a; Zhu et al., 2013)。由此可见,对于光合作用调控机理的深入理解,是有效提高光合作用产量的关键。
随着对于光合作用机理的不断理解,科学家通过基因工程方法改变光合作用的关键位点,不断尝试着提高植物光合作用效率(Barampuram and Zhang, 2011)。目前,提高光合作用效率的主要策略之一是优化植物对于能量和物质的有效利用(Ainsworth and Long, 2005; Long et al., 2006b; Zhu et al., 2007b)。例如,Blankenship通过扩大光系统广谱吸收范围增加了光能利用率(Blankenship and Chen, 2013);Miyagawa等通过过表达光合作用暗反应SBPase酶,有效增加了光合作用暗反应速率(Miyagawa et al., 2001); Lin等通过基因工程方法向烟草中转入外源蓝细菌Rubisco,从而使得光合作用碳固定更加高效(Lin et al., 2014)。另外,尽量降低光合作用中的能量损失也可以提光合作用效率。例如,Kebeish等通过改变光呼吸途径减少了光能损耗,有效增加了光合作用效率以及整体生物量(Kebeish et al., 2007)。尽管如此,目前对于有效提高光合作用效率的可用靶标还是较为有限,系统筛选提高光合作用效率的可用靶标仍是今后的热点问题。特别是,如何有效调整光能的吸收以及调控光能的利用效率,是重要的关注点。
技术实现要素:
本发明的目的在于提供一种新型的与光效调控相关的基因HPE1。
本发明的另一个目的在于提供上述基因的应用。
本发明所采取的技术方案是:
光效调控基因HPE1,其序列如SEQ ID NO:1所示或为其同源保守序列。进一步的,其同源保守序列来自人工规模栽培作物。
作为上述技术方案的进一步改进,人工规模栽培作物选自禾本科作物、豆科作物、茄科作物。
一种提高植物光合作用效率的方法,包括将其光效调控基因HPE1调降、敲除、干扰其正常表达,或使光效调控HPE1 蛋白失活。
作为上述技术方案的进一步改进,植物选自禾本科作物、豆科作物、茄科作物。
一种快速筛查高光合作用效率作物的方法,包括检测其光效调控基因HPE1的表达情况,光效调控基因HPE1活性降低、失活或缺失则判定作物的光合作用效率更高。
作为上述技术方案的进一步改进,作物选自禾本科作物、豆科作物、茄科作物。
一种获得高光合作用效率作物的转基因方法,包括将作物的光效调控基因HPE1敲除。
作为上述技术方案的进一步改进,作物选自禾本科作物、豆科作物、茄科作物。
本发明的有益效果是:
发明人从拟南芥突变体库中筛选到一个高光效突变体HPE1,通过分子鉴定,克隆到了一个编码含RRM结构域叶绿体蛋白的基因HPE1 (AT1G70200),并证实了HPE1参与拟南芥光效调控,是一种重要的调控植物光合效率的功能基因。利用正向遗传学方法,获得两个HPE1的纯合缺失突变体株系,发现HPE1基因的功能缺失可有效优化捕光系统,提高植物光合作用效率,增加有机物积累和植株整体生物量。
HPE1基因(蛋白)在其他作物中具有序列保守性,提示HPE1在其他作物中的功能可能存在保守性,可以预见,HPE1基因是一种重要的调控植物光合效率的功能基因,利用该基因有望获得光合作用效率更高的作物。
附图说明
图1是HPE1基因缺失提高拟南芥光合作用光反应活性图;
图2是HPE1基因缺失降低拟南芥光能损失与光抑制效率图;
图3是HPE1基因缺失增加拟南芥有机物及生物量积累图;
图4是HPE1蛋白特异性定位于叶绿体图;
图5是HPE1蛋白序列在作物中的保守性分析图。
具体实施方式
发明人通过实验发现,HPE1基因位于拟南芥1号染色体上,基因座位号为LOC_AT1G70200(TAIR登录号)。其全长基因组序列约为2104bp,包括4个外显子,3个内含子。其cDNA全长1883 bp(SEQ ID NO:2),编码538个氨基酸。
HPE1编码的蛋白质序列如SEQ ID NO:3所示,存在有叶绿体信号肽(Chloroplast Transit Peptide, CTP)结构域和RNA识别结构域(RNA Recognition Motif, RRM)。
发明人通过实验证实,HPE1参与拟南芥光效调控,是一种重要的调控植物光合效率的功能基因。利用正向遗传学方法,获得两个HPE1的纯合缺失突变体株系,发现HPE1基因的功能缺失可有效优化捕光系统,提高植物光合作用效率,增加有机物积累和植株整体生物量。
鉴于HPE1基因在植物中具有较高的保守性,可以预见在其他被子植物中同样具有相同或相近的功能。因此,通过对HPE1基因进行处理,有望获得高光效的作物,如禾本科作物、豆科作物、茄科作物、蔷薇科作物、十字花科作物等作物,包括但不限于水稻、小麦、高粱、玉米、大豆、马铃薯、菠菜等。
下面结合实验,进一步说明本发明的技术方案。
一.拟南芥HPE1突变体中HPE1基因表达分析
1.拟南芥总RNA的提取:
取三周龄拟南芥野生型(Col-0)和HPE1(hpe1-1,hpe1-2)突变体嫩叶,在1.5mL EP中管中用液氮冷冻并研磨成粉状,转入塑料离心管,并按照每0.1g材料加入1mL提取试剂的比例加入Trizol试剂(Invitrogen公司产RNA专用提取试剂),混合均匀;再按照每0.1g材料加入200uL 氯仿的比例加入氯仿,混合均匀,10 000g,4℃离心15m,弃去中间层及下层有机相,收集上层水相转到新的离心管内;加入600uL 异丙醇,混合均匀,室温静置20m;10 000g,4℃离心15m,收集沉淀,待异丙醇挥发后溶于无RNA酶的超纯水中,-80℃冻存。
2.HPE1基因的RT-PCR表达分析:
1.第一链cDNA的合成
1)取出-80℃保存的水稻愈伤组织总RNA 3 µL,加入2 µL的Oligo (dT)16 (10 mM),混匀后置于70℃中水浴5 min;
2)冰上放置5 min后,于冰上向EP管中先后加入dNTP Mixture (10 mM) 2µL、5×RT Buffer 4 µL、Rnase抑制剂 1 µL(10 U/µL)、Rnase-free ddH2O 8 µL、ReverTra Ace 1 µL;
3)将EP管置于PCR仪中,按30℃ 10 min,42℃ 60 min,99℃ 5 min,4℃ 5 min程序后瞬时离心,反转录得到第一链的单链cDNA,于-20℃冰箱保存。
2.cDNA为模板的PCR
设计如下两条引物:
HPE1-F:5'- TCTAGAATGAACGGAGCTTCGCTCT-3';(SEQ ID NO:4)
HPE1-R:5'- GGTACCTTCATCATTGTTTGAGGTTG-3'(SEQ ID NO:5)。
PCR反应体系为:cDNA模板2µL、引物HPE1-F 0.5 µL、引物HPE1-R 0.5 µL、dNTP 2 µL、10×PCR buffer 2.5 µL、H2O 18 µL、TaKaRa LA Taq酶 0.25 µL。
扩增条件为:94℃变性 5 min;94℃ 30 s, 60℃ 30 s,72℃120 s 28个循环;72℃延伸10 min。PCR产物在1%琼脂糖胶中分离并拍照,对照组野生型拟南芥可以扩增出HPE1基因,而hpe1突变体中扩增不出HPE1基因(图1B)。
二.拟南芥HPE1突变体叶绿素荧光参数分析
植株暗适应20 min 后,利用M 系列叶绿素荧光仪IMAGING-PAM MAXI版(Heinz-Walz),测定响应的叶绿素荧光参数。
(1)最小荧光(F0)、最大荧光(Fm)以及最大光合作用效率(Fv/Fm)的测定:
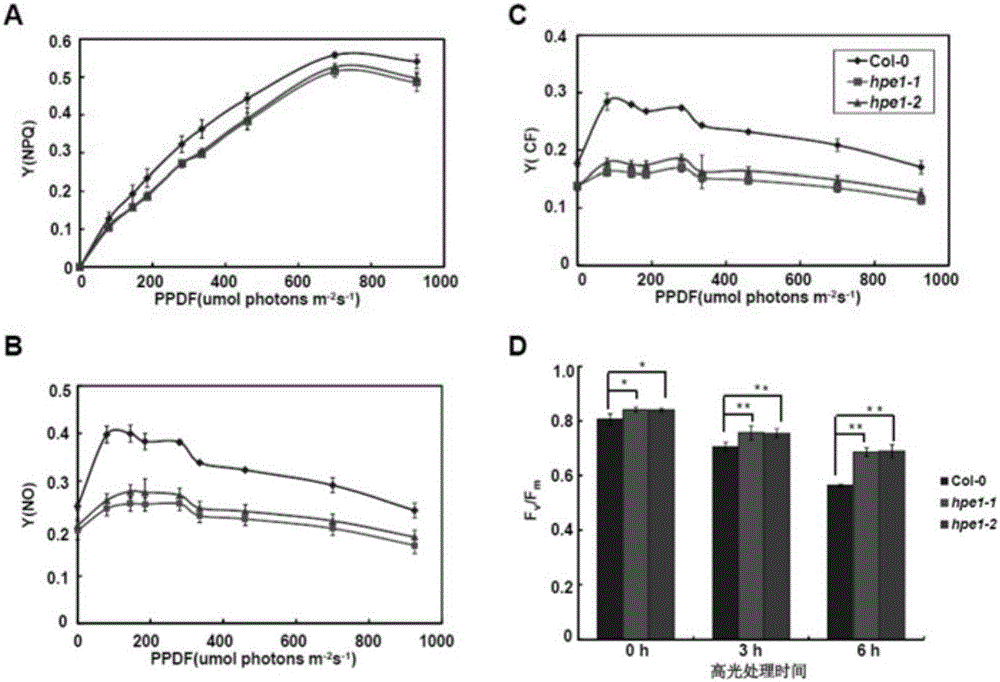
打开叶绿素荧光仪IMAGING-PAM饱和脉冲(2800 μmol photons m-2 s-1)测定F0、Fm以及Fv/Fm,突变体最大光合作用效率Fv/Fm高于野生型拟南芥植株(图1C)。
(2)光响应曲线测定:
F0和Fm测定后,设置光强参数以及时间间隔,测定实际光合作用效率(ФPSII)和电子传递速率(ETR)和1-qL的光相应曲线。光强设置为0,81,145,186,281,335,461,701,以及 926 μmol photons m-2 s-1。每隔3 min 打开饱和脉冲(2800μmol photons m-2 s-1)记录一次数值(图1D)。
(3)光抑制实验
高光(~1200 μmol photons m-2 s-1)处理植株0 h、3 h、6 h后,将植株暗适应20 min,测定Fv/Fm,比较植株的光抑制程度,结果表明hpe1突变体中光抑制程度较野生型轻(图2C)。
三.拟南芥HPE1突变体碳水化合物及生物量分析
1.拟南芥HPE1突变体碳水化合物分析
拟南芥代谢物(可溶性糖和脯氨酸)含量测定采用GC-MS检测并定量。具体步骤如下:
1)取4-6片长势一致的5周龄大小的拟南芥叶片,用液氮研磨成粉末状,称取100 mg左右的样品加入到含有1400 µL的甲醇(-20℃预冷)的2 mL离心管种;
2)加入60 µL ribitol (0.2mg/mL)作为内标,震荡10 S混匀,70℃水浴10 min,11000 g离心10 min;
3)将上清转移到10 mL的玻璃离心管中,加入750 uL氯仿(-20℃预冷)和1500 µL超纯水-4℃预冷),震荡10 S混匀,2200 g离心15 min;
4)转移200 uL上清液到新的1.5 mL离心管中,真空干燥的样品加入40 uL METHOXYAMINATION试剂(20 mg/mL,溶于吡啶),37℃震荡孵育2 h;加入70 uL硅烷化试剂MSTFA,37℃震荡孵育30 min;
5)将衍生完全的样品转移到内衬管中,并置于2 mL GC-MS进样瓶,1uL衍生后的代谢物样品通过Trace GC Untra气相色谱仪(Thermo Fisher)在DSQ II单四级杆质谱仪(Thermo Fisher)上检测并分析;
6)数据通过Xcalibur(Thermo Fisher)软件分析和定量,结果表明hpe1突变体中碳水化合物葡萄糖和果糖,以及脯氨酸含量较野生型显著提高(图3A-3C)。
2.拟南芥HPE1生物量分析
对五周龄野生型、hpe1-1和hpe1-2突变体拟南芥植株进行鲜重、干重统计(图3D-3F)。从图中可以看出,与野生型相比,hpe1-1和hpe1-2突变体拟南芥植株的鲜重、干重都有显著提高。
四.HPE1蛋白亚细胞定位分析
1.HPE1基因的克隆
设计如下两条引物:
HPE1-F:5'- TCTAGAATGAACGGAGCTTCGCTCT-3';
HPE1-R:5'- GGTACCTTCATCATTGTTTGAGGTTG-3'。
PCR反应体系为:cDNA模板2µL、引物HPE1-F 0.5 µL、引物HPE1-R 0.5 µL、dNTP 2 µL、10×PCR buffer 2.5 µL、H2O 18 µL、TaKaRa LA Taq酶 0.25 µL。
扩增条件为:94℃变性 5 min;94℃ 30 s, 60℃ 30 s,72℃120 s 30个循环;72℃延伸10 min。PCR产物在1%琼脂糖胶中分离并拍照。
2.HPE1-GFP融合蛋白表达载体的构建
以Columbia-0(Col-0)cDNA为模板,PCR得到的DNA片段先连接至pMD 18-T 载体中,送至英潍捷基(上海)贸易有限公司测序。本实验室利用pBluescript载体构建了pRiActin中间载体,在此基础上将测序正确的OsLYP4基因的编码序列连接入pRiActin载体后,将经鉴定正确的HPE1过表达目的基因片段克隆用XbaI和Kpn I双酶切;原生质体瞬时表达载体pUC-GFP分别用XbaI和Kpn I双酶切,回收并纯化。将回收得到的过表达基因片段用T4 DNA连接酶连接入原生质体瞬时表达载体pUC-GFP的相应位点中,连接产物转化大肠杆菌DH5α感受态细胞,同时将瞬时表达载体pUC-GFP酶切回收后的产物也转化大肠杆菌DH5α感受态细胞,作为对照;挑选阳性克隆提取质粒DNA,进行酶切鉴定,最后得到在35S启动子驱动下的HPE1-GFP融合蛋白表达载体。
3.利用拟南芥原生质体系统瞬时表达HPE1-GFP融合蛋白
(1)试剂配置
1.母液(所有试剂均购于sigma)
以下所有溶液均由母液配制。
2.酶液
将上述溶液混匀,55℃孵育10min;冷却到室温后加入200µl 1M CaCl2和200µl 10% BSA,并用0.45µm 滤网过滤。
5~10ml酶液能酶解10~20片叶子,最终能分离出(0.5~1)×106个原生质体,每个样品需要(1~2)×104个原生质体
3.
4.
5.
6.
(2)原生质体分离
选取3~4周龄、充分伸展的拟南芥叶片(叶位:2、3、4对)
方法:
1)将酶液用0.45µm的滤器过滤至一个15cm的平皿中;
2)用刀片将叶片切成0.5~1mm的细条(比较麻烦,注意勤换刀片),及时放入酶液,用镊子小心翻动叶片细条,使它们浸没在酶液中;
3)10mm汞柱抽真空30min;
4)黑暗静置酶解约3h;
5)轻轻晃动平皿,释放原生质体,此时,酶液应该变绿;
6)加入等体积的W5溶液,轻轻晃动平皿,充分释放原生质体;
7)用55µm的尼龙网过滤原生质体,收集至15ml圆底塑料试管中;
8)800rmp离心1~2min;
9)用预冷的W5溶液清洗一次;
10)冰上静置30min;
11)镜检原生质体(拟南芥原生质体的大小约为30~50µm),计数;
12)从冰上取出原生质体,800rmp离心1min;
13)去上清,用MMg溶液重悬原生质体,定量至(1-2)×105个/ml。
(3)PEG转化
1)在一个2ml的圆底EP管加入10 µl DNA(5 kb大小的质粒DNA 10~20µg);
2)加入100µl原生质体(2 ×104个原生质体),混匀;
3)加入110µl PEG/Ca溶液,混匀;
4)从第一个开始计时,黑暗放置15min;
5)用440µl W5溶液稀释,并轻轻混匀;
6)800rmp离心2min,去上清;
7)加入一定体积的WI溶液,分散原生质体,平铺于6/12/24孔板中培养;
8)过夜培养。
4.激光共聚焦观察HPE1-GFP融合蛋白
荧光蛋白标记的目的蛋白在原生质体细胞中表达后,利用激光扫描共聚焦显微镜进行观察。GFP使用433 nm激发,并使用500-530 nm波长收集GFP信号;叶绿素自发荧光用488或514 nm波长的激光激发,并用650-750 nm收集(图4B)。
从图中可以清楚地看出,HPE1蛋白特异性定位于叶绿体中。
五.HPE1 的生物信息学分析
利用ClustalX 进行拟南芥HPE1 蛋白在其他陆生植物中的同源性分析(www. clustal.org),包括烟草、大豆、菠菜、玉米、高粱、水稻等,发现HPE1蛋白序列在其他作物中有较高的保守性(图5),可以预见,HPE1基因在其他作物极有可能存在相同的作用,通过调控HPE1基因,可以获得相应的高光效作物。
<110> 中山大学
<120> 光效调控基因HPE1及其应用
<130> HPE1
<160> 5
<170> PatentIn version 3.5
<210> 1
<211> 2104
<212> DNA
<213> 拟南芥
<400> 1
agtttcttctccggcgaagaagaggataacgaaaagaaaacacatagctatgaacggagcttcgctctgcaactccgttttgcttcattctcattctctctctcgttttcagtcatcctcttcttcttgttctgtgtatctgatttcttctgcgcgtactacgtctaagtatatctcaatttctcatagaattcgacagagctttcgagaagttattgaaaaaaggagtaatgggttttgttcattcgctgtaaacaagcgtagaagtggcgattccgttattgtggagtcagacgatgatgatgaagaagatgatgaagattggggtgaatttgatggagaagatgaaggagaagaagaagaagaagatgaaggagagttcttaccaatggataagatgaagaaatggttagagaaaaagcctcgtggatttggtttaggtaaaaagtacgaaactttaattgaagataagttgctcgacgaaattgagcaaagttggaaagctcaagctgctaatctcaataagcttaagaatgatcctctcaagtctcaacagcttaagcgtgacgataatctcctcaaaggtttcgtcttttcttcttttttttgttaattgagttatagtttggaatcttatttagctttgttgtgttgtaaaggaactggagaaactcaaattgggtttcgtgttcgtgtgactaatcttcctaagaagaagaatgttcatagagatcttaaggttgcgtttaaagaagtaagtggcgttttgagtataacaccagctgtgtctgggaataagaagactaaggatcctgtgtgtaaaggatttgctcatgttgatttcaaaactgagattgatgcaaacaggtatgagacttgtggtgtttatagttaagtggtaatgaggcaaagttttgatttttttctcttaggtttgtgaaacagttcacgggacagagcttagcatttgggaaggttataaagcaaatcaaatgtcaggttgtggagtttacttctgatgattctgtatcgaaggaagtgtacttagataacggtttcaaggtgcagaagcttccgtattctggtcttgaaggagattctagtgctgatgttgttgaagaagaggttttgttgggttcaggggaagaatcggatgattcagaagaagaggtagatgaaagagaagtggaagacgatgagcggaatcatatctctagtagcatagaatcttctcccattgaaatgacacgtgattcgaataccaagttaaagtttgagaaacaagttgtgaagcgagagataagagaacatgaggaactagagacacctttagtttcattccaggtgaataaatctgaggaagctgcagcggaaacacacttggatgatgaacaatctgaggaagctgtagcagaaacactcttgaatgatgaacttgatggggatgatgaagaagaagttgctgaagacaatcttgaacctctgaacagttcattgtcatcctcagaagaaaatagagtagatagaatccgtaggctagagcaaaagcttctcggtaaagaaaaactcttgggtggaggagttggctttgataaaccagaagcaaaacccgctagagttgaaggaaagaagaaggagaaaaagaagaagacaaagattcttgtgaaaggccaagccaaaaaggggacaaagatagaaatccctggatcatcaaagaggtaaagagcatacagcaatttttgctaatcaatgtagaatctctactcaaattgattgatttcgacaattatgattcacaggttaaaggtgaaggagaaggctctattaactggcgtcttagtcaagtatgcggctaaagttgcttcaacctcaaacaatgatgaatgatatcattaacaagtgatcaaattttgatataatctttcatcaactgctagtagtgagccatcttcgatttgtttttataaaagtgttacatgaataggtgataacgttacgtggggaaatgctattttttctttctacatcttcatcaatttgtggagacagcaacgttcataaactacattttcagacttaaaaattgaaacttggcaggtaatac
<210> 2
<211> 1883
<212> DNA
<213> 拟南芥
<400> 2
agtttcttctccggcgaagaagaggataacgaaaagaaaacacatagctatgaacggagcttcgctctgcaactccgttttgcttcattctcattctctctctcgttttcagtcatcctcttcttcttgttctgtgtatctgatttcttctgcgcgtactacgtctaagtatatctcaatttctcatagaattcgacagagctttcgagaagttattgaaaaaaggagtaatgggttttgttcattcgctgtaaacaagcgtagaagtggcgattccgttattgtggagtcagacgatgatgatgaagaagatgatgaagattggggtgaatttgatggagaagatgaaggagaagaagaagaagaagatgaaggagagttcttaccaatggataagatgaagaaatggttagagaaaaagcctcgtggatttggtttaggtaaaaagtacgaaactttaattgaagataagttgctcgacgaaattgagcaaagttggaaagctcaagctgctaatctcaataagcttaagaatgatcctctcaagtctcaacagcttaagcgtgacgataatctcctcaaaggaactggagaaactcaaattgggtttcgtgttcgtgtgactaatcttcctaagaagaagaatgttcatagagatcttaaggttgcgtttaaagaagtaagtggcgttttgagtataacaccagctgtgtctgggaataagaagactaaggatcctgtgtgtaaaggatttgctcatgttgatttcaaaactgagattgatgcaaacaggtttgtgaaacagttcacgggacagagcttagcatttgggaaggttataaagcaaatcaaatgtcaggttgtggagtttacttctgatgattctgtatcgaaggaagtgtacttagataacggtttcaaggtgcagaagcttccgtattctggtcttgaaggagattctagtgctgatgttgttgaagaagaggttttgttgggttcaggggaagaatcggatgattcagaagaagaggtagatgaaagagaagtggaagacgatgagcggaatcatatctctagtagcatagaatcttctcccattgaaatgacacgtgattcgaataccaagttaaagtttgagaaacaagttgtgaagcgagagataagagaacatgaggaactagagacacctttagtttcattccaggtgaataaatctgaggaagctgcagcggaaacacacttggatgatgaacaatctgaggaagctgtagcagaaacactcttgaatgatgaacttgatggggatgatgaagaagaagttgctgaagacaatcttgaacctctgaacagttcattgtcatcctcagaagaaaatagagtagatagaatccgtaggctagagcaaaagcttctcggtaaagaaaaactcttgggtggaggagttggctttgataaaccagaagcaaaacccgctagagttgaaggaaagaagaaggagaaaaagaagaagacaaagattcttgtgaaaggccaagccaaaaaggggacaaagatagaaatccctggatcatcaaagaggttaaaggtgaaggagaaggctctattaactggcgtcttagtcaagtatgcggctaaagttgcttcaacctcaaacaatgatgaatgatatcattaacaagtgatcaaattttgatataatctttcatcaactgctagtagtgagccatcttcgatttgtttttataaaagtgttacatgaataggtgataacgttacgtggggaaatgctattttttctttctacatcttcatcaatttgtggagacagcaacgttcataaactacattttcagacttaaaaattgaaacttggcaggtaatac
<210> 3
<211> 538
<212> PRT
<213> 拟南芥
<400> 3
Met Asn Gly Ala Ser Leu Cys Asn Ser Val Leu Leu His Ser His Ser
1 5 10 15
Leu Ser Arg Phe Gln Ser Ser Ser Ser Ser Cys Ser Val Tyr Leu Ile
20 25 30
Ser Ser Ala Arg Thr Thr Ser Lys Tyr Ile Ser Ile Ser His Arg Ile
35 40 45
Arg Gln Ser Phe Arg Glu Val Ile Glu Lys Arg Ser Asn Gly Phe Cys
50 55 60
Ser Phe Ala Val Asn Lys Arg Arg Ser Gly Asp Ser Val Ile Val Glu
65 70 75 80
Ser Asp Asp Asp Asp Glu Glu Asp Asp Glu Asp Trp Gly Glu Phe Asp
85 90 95
Gly Glu Asp Glu Gly Glu Glu Glu Glu Glu Asp Glu Gly Glu Phe Leu
100 105 110
Pro Met Asp Lys Met Lys Lys Trp Leu Glu Lys Lys Pro Arg Gly Phe
115 120 125
Gly Leu Gly Lys Lys Tyr Glu Thr Leu Ile Glu Asp Lys Leu Leu Asp
130 135 140
Glu Ile Glu Gln Ser Trp Lys Ala Gln Ala Ala Asn Leu Asn Lys Leu
145 150 155 160
Lys Asn Asp Pro Leu Lys Ser Gln Gln Leu Lys Arg Asp Asp Asn Leu
165 170 175
Leu Lys Gly Thr Gly Glu Thr Gln Ile Gly Phe Arg Val Arg Val Thr
180 185 190
Asn Leu Pro Lys Lys Lys Asn Val His Arg Asp Leu Lys Val Ala Phe
195 200 205
Lys Glu Val Ser Gly Val Leu Ser Ile Thr Pro Ala Val Ser Gly Asn
210 215 220
Lys Lys Thr Lys Asp Pro Val Cys Lys Gly Phe Ala His Val Asp Phe
225 230 235 240
Lys Thr Glu Ile Asp Ala Asn Arg Phe Val Lys Gln Phe Thr Gly Gln
245 250 255
Ser Leu Ala Phe Gly Lys Val Ile Lys Gln Ile Lys Cys Gln Val Val
260 265 270
Glu Phe Thr Ser Asp Asp Ser Val Ser Lys Glu Val Tyr Leu Asp Asn
275 280 285
Gly Phe Lys Val Gln Lys Leu Pro Tyr Ser Gly Leu Glu Gly Asp Ser
290 295 300
Ser Ala Asp Val Val Glu Glu Glu Val Leu Leu Gly Ser Gly Glu Glu
305 310 315 320
Ser Asp Asp Ser Glu Glu Glu Val Asp Glu Arg Glu Val Glu Asp Asp
325 330 335
Glu Arg Asn His Ile Ser Ser Ser Ile Glu Ser Ser Pro Ile Glu Met
340 345 350
Thr Arg Asp Ser Asn Thr Lys Leu Lys Phe Glu Lys Gln Val Val Lys
355 360 365
Arg Glu Ile Arg Glu His Glu Glu Leu Glu Thr Pro Leu Val Ser Phe
370 375 380
Gln Val Asn Lys Ser Glu Glu Ala Ala Ala Glu Thr His Leu Asp Asp
385 390 395 400
Glu Gln Ser Glu Glu Ala Val Ala Glu Thr Leu Leu Asn Asp Glu Leu
405 410 415
Asp Gly Asp Asp Glu Glu Glu Val Ala Glu Asp Asn Leu Glu Pro Leu
420 425 430
Asn Ser Ser Leu Ser Ser Ser Glu Glu Asn Arg Val Asp Arg Ile Arg
435 440 445
Arg Leu Glu Gln Lys Leu Leu Gly Lys Glu Lys Leu Leu Gly Gly Gly
450 455 460
Val Gly Phe Asp Lys Pro Glu Ala Lys Pro Ala Arg Val Glu Gly Lys
465 470 475 480
Lys Lys Glu Lys Lys Lys Lys Thr Lys Ile Leu Val Lys Gly Gln Ala
485 490 495
Lys Lys Gly Thr Lys Ile Glu Ile Pro Gly Ser Ser Lys Arg Leu Lys
500 505 510
Val Lys Glu Lys Ala Leu Leu Thr Gly Val Leu Val Lys Tyr Ala Ala
515 520 525
Lys Val Ala Ser Thr Ser Asn Asn Asp Glu
530 535
<210> 4
<211> 25
<212> DNA
<213> 人工引物
<400> 4
tctagaatga acggagcttc gctct 25
<210> 5
<211> 26
<212> DNA
<213> 人工引物
<400> 5
ggtaccttca tcattgtttg aggttg 26
- 还没有人留言评论。精彩留言会获得点赞!
- 拟南芥SSCD1基因突变在调控植物体内茉莉酸合成中的应用的制作方法与工艺
- 一种基因调控网络的优化控制方法与流程
- 拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用的制作方法与工艺
- 针对免疫调控能力相关基因个体化配方食品及其制备方法与流程
- REVOLUTA基因在调控豆科植物小叶片数目和叶茎比中的应用的制作方法与工艺
- 玉米锌铁调控转运体ZmZIP3基因及其应用的制作方法与工艺
- 拟南芥基因SPOC1在调控植株开花期中的应用的制作方法与工艺
- 调控石榴花胚珠发育的PgAG基因及其用途的制作方法与工艺
- 光效调控基因HPE1及其应用的制作方法与工艺
- 菘蓝IiAP2/ERF049基因在调控木脂素类化合物合成中的应用的制作方法与工艺