玉米锌铁调控转运体ZmZIP2基因及其应用的制作方法
技术领域
本发明涉及从植物中分离的金属离子转运体基因,尤其涉及从玉米中分离的与锌铁的吸收、转运或储存有关的调控转运体ZmZIP2基因,本发明进一步涉及调控转运体ZmZIP2基因在调控植物吸收、转运或储存锌或铁能力,促进胚和胚乳的发育或成熟以及增加粮食作物种子锌铁含量中的应用,属于植物金属离子调控转运体基因的分离和应用领域。
背景技术:
锌和铁是生物体所必需的微量元素,在植物的生长发育过程中有着重要作用(Wintz H,Fox T,Wu YY,et al.Expression profiles of Arabidopsis thaliana in mineral deficiencies reveal novel transporters involved in metal homeostasis.The Journal of biological chemistry 2003,278(48):47644-47653.)。锌是生物体300多种酶和重要蛋白质的结构辅助因子(Haydon MJ,Cobbett CS.A novel major facilitator superfamily protein at the tonoplast influences zinc tolerance and accumulation in Arabidopsis.Plant physiology 2007,143(4):1705-1719.)。锌不仅参与机体的各种代谢,在生物膜稳定和基因表达调控等生理机能中也担负着重要的角色(Mathews WR,Wang F,Eide DJ,et al.Drosophila fear of intimacy encodes a Zrt/IRT-like protein(ZIP)family zinc transporter functionally related to mammalian ZIP proteins.The Journal of biological chemistry 2005,280(1):787-795.)。适量增加植物体内锌的含量可提高作物产量,而锌的缺乏会导致叶绿素、脂质、蛋白、质膜的氧化破坏,植物体内锌离子的过度积累又会对植物产生毒害。
铁在细胞呼吸、光合作用和金属蛋白的催化反应过程中发挥重要作用,是重要的电子传递体,因此,铁元素在原核和真核生物的生命活动中具有不可替代的功能。另外,细胞内过高的Fe3+/Fe2+氧化还原势会导致超氧化合物的产生,对细胞造成伤害(Briat JF,Lebrun M.Plant responses to metal toxicity.Comptes rendus de l'Academie des sciences Serie III,Sciences de la vie 1999,322(1):43-54.)。因此,严格控制植物体内金属离子的平衡是至关重要的。
参与锌铁吸收的蛋白主要有三类,都是以蛋白家族形式存在的,包括:ZIP,即锌调控转运体(Zinc-regulated transporter,ZRT)和铁调控转运体(Iron-regulated transporter,IRT)。酵母功能互补实验显示ZIP家族基因能够转运包括Zn2+、Fe2+、Cu2+、Cd2+在内的多种金属离子(Colangelo EP,Guerinot ML.Put the metal to the petal:metal uptake and transport throughout plants.Current opinion in plant biology 2006,9(3):322-330.)。ZIP一般由309-476个氨基酸残基组成,有8个潜在的跨膜结构域和相似的拓扑结构,第3和第4跨膜区之间有一长的可变区,可变区位于胞内,其C、N末端位于胞外,该区富含组氨酸残基,可能与金属的结合、转运有关(Guerinot ML.The ZIP family of metal transporters.Biochim Biophys Acta 2000,1465(1-2):190-198.)。
目前在拟南芥、水稻、蒺藜、苜蓿、大豆、野生型二粒小麦、葡萄等植物中鉴定出ZIP基因并对其功能进行了研究。在拟南芥中发现16个ZIP家族基因,AtIRT1是通过酵母互补实验分离得到的第一个ZIP功能基因,其主要在根部表达,且该基因的过表达可导致镍的过度积累(Eide D,Broderius M,Fett J,et al.A novel iron-regulated metal transporter from plants identified by functional expression in yeast.Proceedings of the National Academy of Sciences of the United States of America 1996,93(11):5624-5628;Henriques R,Jasik J,Klein M,et al.Knock-out of Arabidopsis metal transporter gene IRT1results in iron deficiency accompanied by cell differentiation defects.Plant molecular biology 2002,50(4-5):587-597;Varotto C,Maiwald D,Pesaresi P,et al.The metal ion transporter IRT1is necessary for iron homeostasis and efficient photosynthesis in Arabidopsis thaliana.The Plant journal:for cell and molecular biology 2002,31(5):589-599;Vert G,Grotz N,Dedaldechamp F,et al.IRT1,an Arabidopsis transporter essential for iron uptake from the soil and for plant growth.Plant Cell 2002,14(6):1223-1233;Nishida S,Tsuzuki C,Kato A,et al.AtIRT1,the primary iron uptake transporter in the root,mediates excess nickel accumulation in Arabidopsis thaliana.Plant&cell physiology 2011,52(8):1433-1442.)。AtIRT2主要在根部表达,定位在囊泡,推测具有细胞内过量金属元素的解毒功能(Vert G,Briat JF,Curie C.Arabidopsis IRT2gene encodes a root-periphery iron transporter.The Plant journal:for cell and molecular biology 2001,26(2):181-189;Vert G,Barberon M,Zelazny E,et al.Arabidopsis IRT2cooperates with the high-affinity iron uptake system to maintain iron homeostasis in root epidermal cells.Planta 2009,229(6):1171-1179.14,15)。AtIRT3能互补锌、铁转运双突变体,过表达AtIRT3会使锌在地上部以及铁在地下部积累(Lin YF,Liang HM,Yang SY,et al.Arabidopsis IRT3is a zinc-regulated and plasma membrane localized zinc/iron transporter.The New phytologist 2009,182(2):392-404.)。表达分析显示,AtZIP1、AtZIP5、AtZIP9、AtZIP12和AtIRT3受缺锌诱导,由此推测,这些基因在缺锌条件下可能增强锌的吸收能力(Kramer U,Talke IN,Hanikenne M.Transition metal transport.FEBS Lett 2007,581(12):2263-2272.)。
玉米(Zea mays)是中国重要的粮食、饲料和经济作物,增加玉米籽粒中锌、铁等微量元素的含量,对提高饮食或饲料利用效率、促进经济的发展及人体健康尤为重要。目前,已知许多转运蛋白参与了植物体内锌铁离子平衡网络系统,其中ZIP(Zinc-regulated transporters,Iron-regulated transporter-like proteins,ZIP)基因家族对锌、铁等二价金属离子的吸收、运输和储存起着重要作用,在拟南芥、水稻、大麦、大豆上,已报道了一些有关ZIP家族基因的研究,但对ZIP家族基因在植物体内具体的作用机制尚未完全了解,而关于玉米的ZIP家族基因的研究报道较少。了解Zn2+、Fe2+在玉米中的吸收、运输方式,分布规律和调节机制,将有助于改善玉米在锌、铁缺乏的环境中的生长发育,为进一步揭示玉米中ZIP家族基因的作用机理奠定基础,为玉米锌、铁高效转基因育种提供候选基因,也为人类锌铁营养提供良好的基础。
技术实现要素:
本发明的目的之一是提供从玉米(Zea mays)中分离的锌铁调控转运体基因;
本发明的目的之二是提供锌铁调控转运体基因所编码的蛋白质;
本发明的目的之三是将所述的锌铁调控转运体基因应用于调控植物对锌铁等金属离子的吸收、转运或储存,促进胚根和胚芽发育以及胚成熟或者增加粮食作物籽粒中锌铁含量。
本发明的上述目的是通过以下技术方案来实现的:
从玉米(Zea mays)中分离的锌铁调控转运体ZmZIP1基因,其cDNA序列为(a)、(b)或(c)所示:
(a)、SEQ ID No.1所示的核苷酸序列;
(b)、编码SEQ ID No.2所示氨基酸的核苷酸序列;
(c)、与SEQ ID No.1所示核苷酸的互补序列在严谨杂交条件能够进行杂交的核苷酸,该核苷酸所编码的蛋白质具有锌铁调控转运体的功能。
从玉米(Zea mays)中分离的锌铁调控转运体ZmZIP2基因,其cDNA序列为(a)、(b)或(c)所示:
(a)、SEQ ID No.3所示的核苷酸序列;
(b)、编码SEQ ID No.4所示氨基酸的核苷酸序列;
(c)、与SEQ ID No.3所示核苷酸的互补序列在严谨杂交条件能够进行杂交的核苷酸,该核苷酸所编码的蛋白质具有锌铁调控转运体的功能。
从玉米(Zea mays)中分离的锌铁调控转运体ZmZIP3基因,其cDNA序列为(a)、(b)或(c)所示:
(a)、SEQ ID No.5所示的核苷酸序列;
(b)、编码SEQ ID No.6所示氨基酸的核苷酸序列;
(c)、与SEQ ID No.5所示核苷酸的互补序列在严谨杂交条件能够进行杂交的核苷酸,该核苷酸所编码的蛋白质具有锌铁调控转运体的功能。
从玉米(Zea mays)中分离的锌铁调控转运体ZmZIP7基因,其cDNA序列为(a)、(b)或(c)所示:
(a)、SEQ ID No.7所示的核苷酸序列;
(b)、编码SEQ ID No.8所示氨基酸的核苷酸序列;
(c)、与SEQ ID No.7所示核苷酸的互补序列在严谨杂交条件能够进行杂交的核苷酸,该核苷酸所编码的蛋白质具有锌铁调控转运体的功能。
从玉米(Zea mays)中分离的锌铁调控转运体ZmZIP8基因,其cDNA序列为(a)、(b)或(c)所示:
(a)、SEQ ID No.9所示的核苷酸序列;
(b)、编码SEQ ID No.10所示氨基酸的核苷酸序列;
(c)、与SEQ ID No.9所示核苷酸的互补序列在严谨杂交条件能够进行杂交的核苷酸,该核苷酸所编码的蛋白质具有锌铁调控转运体的功能。
所述“严谨杂交条件”意指在所属领域中已知的低离子强度和高温的条件。通常,在严谨条件下,探针与其靶序列杂交的可检测程度比与其它序列杂交的可检测程度更高(例如超过本底至少2倍。严谨杂交条件是序列依赖性的,在不同的环境条件下将会不同,较长的序列在较高温度下特异性杂交。通过控制杂交的严谨性或洗涤条件可鉴定与探针100%互补的靶序列。对于核酸杂交的详尽指导可参考有关文献(Tijssen,Techniques in Biochemistry and Molecular Biology-Hybridization with Nucleic Probes,"Overview of principles of hybridization and the strategy of nucleic acid assays.1993)。更具体的,所述严谨条件通常被选择为低于特异序列在规定离子强度pH下的热熔点(Tm)约5-10℃。Tm为在平衡状态下50%与目标互补的探针杂交到目标序列时所处的温度(在指定离子强度、pH和核酸浓度下)(因为目标序列过量存在,所以在Tm下在平衡状态下50%的探针被占据)。严谨条件可为以下条件:其中在pH 7.0到8.3下盐浓度低于约1.0M钠离子浓度,通常为约0.01到1.0M钠离子浓度(或其它盐),并且温度对于短探针(包括(但不限于)10到50个核苷酸)而言为至少约30℃,而对于长探针(包括(但不限于)大于50个核苷酸)而言为至少约60℃。严谨条件也可通过加入诸如甲酰胺的去稳定剂来实现。对于选择性或特异性杂交而言,正信号可为至少两倍的背景杂交,视情况为10倍背景杂交。例示性严谨杂交条件可如下:50%甲酰胺,5×SSC和1%SDS,在42℃下培养;或5×SSC,1%SDS,在65℃下培养,在0.2×SSC中洗涤和在65℃下于0.1%SDS中洗涤。所述洗涤可进行5、15、30、60、120min或更长时间。
优选的,本发明从玉米中所分离的锌铁调控转运体ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8基因的cDNA序列分别为SEQ ID No.1、SEQ ID No.3、SEQ ID No.5、SEQ ID No.7或SEQ ID No.9所示的核苷酸序列。
本发明目的之二是提供由上述锌铁调控转运体ZmZIPs基因(ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8)所编码的能够吸收、转运或储存锌铁的蛋白质;
本发明目的之二是通过以下技术方案来实现的:
ZmZIPs基因所编码的锌铁调控转运体,其氨基酸序列为(a)或(b)所示:
(a)、SEQ ID No.2、SEQ ID No.4、SEQ ID No.6、SEQ ID No.8或SEQ ID No.10所示的氨基酸序列;
(b)、将SEQ ID No.2、SEQ ID No.4、SEQ ID No.6、SEQ ID No.8或SEQ ID No.10所示的氨基酸序列通过一个或多个氨基酸残基的替换、缺失或/和插入而衍生得到的仍具有锌铁调控转运体功能的蛋白变体。
所述的“多个”通常意味着2-8个,优选为2-4个,这取决于锌铁调控转运体三维结构中氨基酸残基的位置或氨基酸的种类;所述的“替换”是指分别用不同的氨基酸残基取代一个或多个氨基酸残基;所述的“缺失”是指氨基酸残基数量的减少,也即是分别缺少其中的一个或多个氨基酸残基;所述的“插入”是指氨基酸残基序列的改变,相对天然分子而言,所述改变导致添加一个或多个氨基酸残基。
本发明所述的蛋白变体可由遗传多态性或人为操作产生,这些操作方法通常为本领域所了解。例如,可通过DNA的突变来制备锌铁调控转运体的氨基酸序列变体或片段,其中由于诱变或改变多核苷酸的方法为本领域所习知。其中,保守的取代是将一种氨基酸残基替换成具有相似性质的另一种氨基酸。因此,本发明所述的锌铁调控转运体及其编码基因包括天然存在的序列和变体两种形式。“变体”意指基本相似的序列,对于多核苷酸,变体包含天然多核苷酸中一个或多个位点处一个或多个核苷酸的缺失、插入或/和替换。对于多核苷酸,保守的变体包括由于遗传密码的简并性而不改变编码的氨基酸序列的那些变体。诸如此类天然存在的变体可通过现有的分子生物学技术来鉴定。变体多核苷酸还包括合成来源的多核苷酸,例如,采用定点诱变所得到的仍编码SEQ ID No.2所示的氨基酸的多核苷酸变体,或者是通过重组的方法(例如DNA重排等)。本领域技术人员可通过以下分子生物技术手段来筛选或评价变体多核苷酸所编码蛋白的功能或活性:DNA结合活性,蛋白之间的相互作用,瞬时研究中基因表达的激活情况或转基因植物中表达的效应等。
从亚细胞定位结果可知,本发明分离的5个ZmZIPs基因(ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8基因)所编码的蛋白定位在质膜与细胞内膜上。为进一步确定细胞内膜的具体定位,本发明选用ER marker与5个ZmZIPs基因共定位进行拟南芥叶肉原生质体的转化,结果证明这5个ZmZIPs基因均定位在细胞质膜及内质网上。在亚细胞定位的基础上,通过real-time RT-PCR表达分析发现,正常营养条件下,5个ZmZIPs基因主要在地上部表达,其中,缺锌条件下,ZmZIP8在96h地上部表达上调,ZmZIP3在6h时地上部和地下部表达量都有所升高;在高锌条件ZmZIP7和ZmZIP8在地上部的表达量是逐渐降低的,ZmZIP3在地下部的表达量明显的降低。这些结果表明,ZmZIP3、ZmZIP7和ZmZIP8在幼苗时期对锌的浓度比较敏感。缺铁条件下,ZmZIP7和ZmZIP8在地上与地下的表达量是逐渐增高的,说明ZmZIP7和ZmZIP 8对铁的浓度比较敏感。缺铜、缺锰条件下,5个ZmZIPs基因的表达量都没有明显的变化。
酵母互补实验表明,本发明所分离的5个ZmZIPs不论在低锌还是在低铁条件下都表现出不同程度的转运锌或铁活性,说明本发明所分离的5个ZmZIPs均具有转运锌铁的功能。
目前,已知许多转运蛋白参与了植物体内锌铁离子平衡网络系统,一些蛋白也被应用到植物的转基因研究中,比如在大麦中过表达AtZIP1基因能够增加锌和铁在种子中的含量(Ramesh SA,Choimes S,Schachtman DP.Over-expression of an Arabidopsis zinc transporter in hordeum vulgare increases short-term zinc uptake after zinc deprivation and seed zinc content.Plant molecular biology 2004,54(3):373-385.),同样,过表达OsIRT1基因,水稻中锌和铁的含量在地上部、地下部和种子中都有所提高(Lee S,An G.Over-expression of OsIRT1leads to increased iron and zinc accumulations in rice.Plant,cell&environment 2009,32(4):408-416.)。然而,在水稻中过表达OsZIP4、OsZIP5、OsZIP8,OsZIP9结果导致过量的锌聚集于根部,降低了植株地上部分的锌含量(Lee S,Kim SA,Lee J,et al.Zinc deficiency-inducible OsZIP8encodes a plasma membrane-localized zinc transporter in rice.Molecules and cells 2010,29(6):551-558;Lee S,Jeong HJ,Kim SA,et al.OsZIP5is a plasma membrane zinc transporter in rice.Plant molecular biology2010,73(4-5):507-517;Ishimaru Y,Masuda H,Suzuki M,et al.Overexpression of the OsZIP4zinc transporter confers disarrangement of zinc distribution in rice plants.Journal of experimental botany2007,58(11):2909-2915.)没有达到在籽粒中增加锌含量的目的,因此,这些基因的过表达对水稻籽粒中锌的富集是不利的。这些结果表明,异位过表达对于锌铁的积累与分布可能会起到一定的作用。然而,有关锌铁转运蛋白在籽粒中的研究还很少。
因此,本发明提供了一种调控植物吸收、转运或储存锌铁的能力的方法,包括:将本发明ZmZIPs基因可操作的与表达调控元件相连接得到重组植物表达载体;将重组植物表达载体转化到植物中,在植物体中过表达ZmZIPs基因,能够有效调控或改善目标植物对锌铁的吸收、转运或储存的能力。
本发明提供了一种解除过量的锌铁对植物体毒害的方法,该方法包括:将本发明ZmZIPs基因可操作的与表达调控元件相连接得到重组植物表达载体;将重组植物表达载体转化到植物中,在植物体中过表达ZmZIPs基因。
本发明还提供了一种调控或促进植物种子胚发育的方法,该方法包括:将本发明ZmZIPs基因可操作的与表达调控元件相连接得到重组植物表达载体;将重组植物表达载体转化到植物中,在植物体中过表达ZmZIPs基因;其中,所述的表达调控元件中启动子优选为种子特异性表达的启动子。
本发明进一步提供了一种调控或促进种子胚成熟的方法,该方法包括:将本发明ZmZIPs基因可操作的与表达调控元件相连接得到重组植物表达载体;将重组植物表达载体转化到植物中,在植物体中过表达ZmZIPs基因;其中,所述的表达调控元件中的启动子优选为种子特异性表达的启动子。
本发明进一步提供了含有所述锌铁调控转运体ZmZIPs基因的重组植物表达载体以及含有该重组植物表达载体的宿主细胞。
将本发明所述锌铁调控转运体ZmZIPs基因可操作的与表达调控元件相连接,得到可以在植物中表达该锌铁调控转运体基因的重组植物表达载体。“可操作的连接”指两个或更多个元件之间功能性的连接,可操作的连接的元件可为邻接或非邻接的。例如,该重组植物表达载体可以由5′端非编码区,SEQ ID No.1(或者是SEQ ID No.3、SEQ ID No.5、SEQ ID No.7或SEQ ID No.9中的任一序列)所示的核苷酸和3′非编码区组成,其中,所述的5′端非编码区可以包括启动子序列、增强子序列或/和翻译增强序列;所述的启动子可以是组成性启动子、诱导型启动子、组织或器官特异性启动子;所述的3′非编码区可以包含终止子序列、mRNA切割序列等。合适的终止子序列可取自根癌农杆菌的Ti-质粒,例如章鱼碱合成酶或胭脂碱合成酶终止区。例如,为了使本发明的锌铁调控转运体基因在粮食作物种子进行特异性表达,可以将锌铁调控转运体基因连接在种子特异性表达启动子的下方构建得到重组植物表达载体,将该重组植物表达载体转化受体植物后,锌铁调控转运体基因可以在受体植物的种子里进行特异性表达,达到促进胚根和胚芽发育、促进胚成熟或增加种子锌铁含量或调控胚发育的效果。
另外,本领域技术人员可以将ZmZIPs的核苷酸序列进行优化以增强其在植物中的表达。例如。可采用目标植物的偏爱密码子进行优化来合成多核苷酸以增强该基因在目标植物中的表达水平,这些方法均为本领域技术人员所习知。
此外,该重组植物表达载体还可含有用于选择转化细胞的选择性标记基因。选择性标记基因用于选择经转化的细胞或组织。所述的选择性标记基因包括:编码抗生素抗性的基因以及赋予除草化合物抗性的基因等。此外,所述的标记基因还包括表型标记,例如β-半乳糖苷酶和荧光蛋白等。
所述的“转化”指将基因导入到植物细胞内部这样的方式将多核苷酸或多肽遗传转化到植物中。将所述多核苷酸或多肽引入到植物中的方法为本领域所习知,包括但不限于稳定转化法、瞬时转化法和病毒介导法等。“稳定转化”指被引入的多核苷酸构建体整合至植物细胞的基因组中并能通过其子代遗传;“瞬时转化”指多核苷酸被引入到植物中但只能在植物中暂时性表达或存在。
转化方案以及将所述多核苷酸引入植物的方案可视用于转化的植物(单子叶植物或双子叶植物)或植物细胞的类型而变化。将所述多核苷酸转化植物细胞的合适方法包括:显微注射、电穿孔、农杆菌介导的转化、直接基因转移以及高速弹道轰击等。在特定的实施方案中,可利用多种瞬时转化法将本发明的ZmZIPs基因提供给植物。在其它实施方案中,本发明的ZmZIPs基因可通过将植物与病毒或病毒核酸接触来引入到植物中,通常,这样的方法涉及将本发明的ZmZIPs基因构建体引入病毒DNA或RNA分子中。
利用常规方法可使已转化的细胞再生稳定转化植株(McCormick et al.Plant Cell Reports.1986.5:81-84)。本发明可用于转化任何植物种类,包括但不限于:单子叶植物或双子叶植物;优选的,所述的目标植物包括粮食作物、蔬菜或果树等,更优选为粮食作物,例如,可以是玉米、水稻、大麦小麦、高粱、大豆、马铃薯等粮食作物。
本发明所涉及到的术语定义
除非另外定义,否则本文所用的所有技术及科学术语都具有与本发明所属领域的普通技术人员通常所了解相同的含义。虽然在本发明的实践或测试中可使用与本文所述者类似或等效的任何方法、装置和材料,但现在描述优选方法、装置和材料。
术语“重组宿主细胞株”或“宿主细胞”意指包含本发明多核苷酸的细胞,而不管使用何种方法进行插入以产生重组宿主细胞,例如直接摄取、转导、f配对或所属领域中已知的其它方法。外源性多核苷酸可保持为例如质粒的非整合载体或者可整合入宿主基因组中。宿主细胞可为原核细胞或真核细胞,宿主细胞还可为单子叶或双子叶植物细胞。
术语“核苷酸”意指单股或双股形式的脱氧核糖核苷酸、脱氧核糖核苷、核糖核苷或核糖核苷酸及其聚合物。除非特定限制,否则所述术语涵盖含有天然核苷酸的已知类似物的核酸,所述类似物具有类似于参考核酸的结合特性并以类似于天然产生的核苷酸的方式进行代谢。除非另外特定限制,否则所述术语也意指寡核苷酸类似物,其包括PNA(肽核酸)、在反义技术中所用的DNA类似物(硫代磷酸酯、磷酰胺酸酯等等)。除非另外指定,否则特定核酸序列也隐含地涵盖其保守修饰的变异体(包括(但不限于)简并密码子取代)和互补序列以及明确指定的序列。特定而言,可通过产生其中一个或一个以上所选(或所有)密码子的第3位经混合碱基和/或脱氧肌苷残基取代的序列来实现简并密码子取代。
术语“多肽”、“肽”和“蛋白质”在本文中互换使用以意指氨基酸残基的聚合物。即,针对多肽的描述同样适用于描述肽和描述蛋白,且反之亦然。所述术语适用于天然产生氨基酸聚合物以及其中一个或一个以上氨基酸残基为非天然编码氨基酸的氨基酸聚合物。如本文中所使用,所述术语涵盖任何长度的氨基酸链,其包括全长蛋白(即抗原),其中氨基酸残基经由共价肽键连接。
附图说明
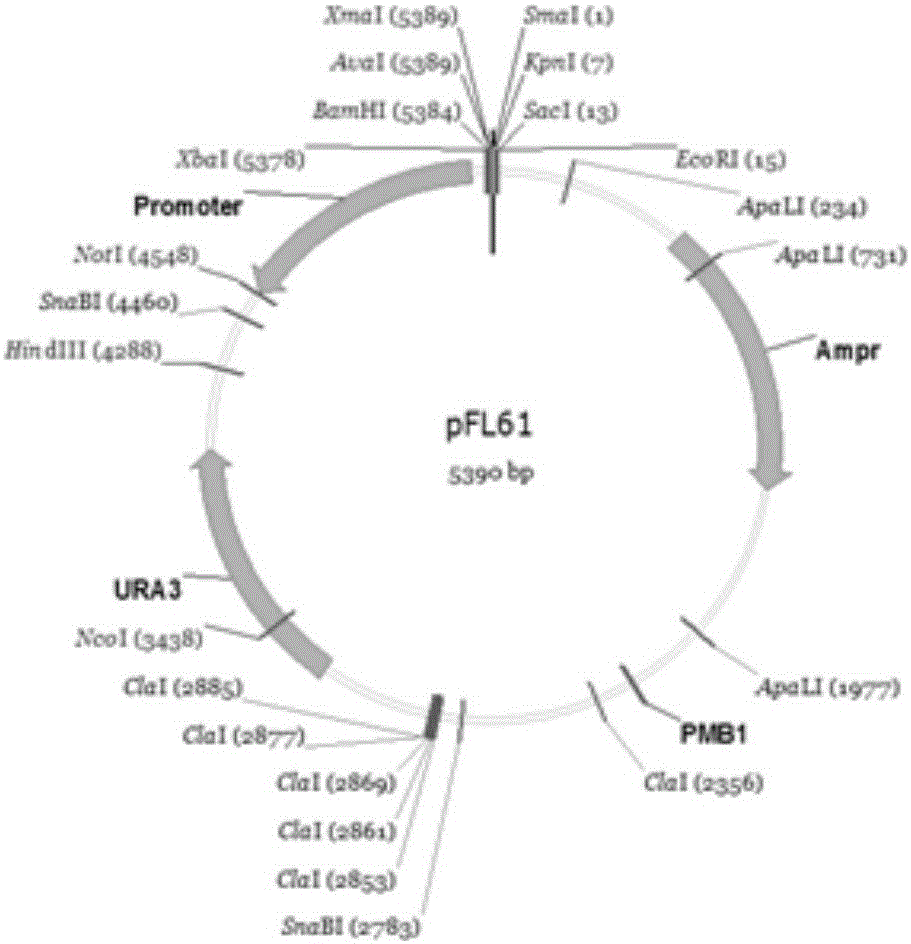
图1酵母表达载体pFL61的示意图。
图2标准Hoagland培养基条件下ZmZIPs的表达模式;S(shoot),R(root)。
图3 ZmZIPs基因在各种处理条件下的表达模式。
图4 ZmZIPs基因在玉米胚和胚乳发育过程中的表达模式。
图5 pRTL2NGFP-ZmZIPs重组载体酶切鉴定;M为1Kb的Marker;1-5分别为pRTL2NGFP-ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7、ZmZIP8双酶切的结果
图6 ZmZIPs洋葱表皮细胞中的亚细胞定位;GFP为pRTL2NGFP空载体定位情况;ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7、ZmZIP8为pRTL2NGFP-ZmZIP1、2、3、7、8的亚细胞定位情况。
图7 ZmZIPs拟南芥叶肉原生质体中的亚细胞定位;GFP为pRTL2NGFP空载体定位情况;GFP为pRTL2NGFP空载体定位情况;ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7、ZmZIP8分别为pRTL2NGFP-ZmZIP1、2、3、7、8的亚细胞定位情况。
图8 pFL61-ZmZIPs及pFL61-OsZIP5、pFL61-OsZIP8、pFL61-OsIRT1正向连接重组载体酶切鉴定;M为1Kb的Marker;1-8依次为pFL61-ZmZIP1、pFL61-ZmZIP2、pFL61-ZmZIP3、pFL61-ZmZIP7、pFL61-ZmZIP8、pFL61-OsZIP5、pFL61-OsZIP8、pFL61-OsIRT1双酶切结果。
图9 ZmZIPs酵母互补实验结果。
图10 ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7和ZmZIP8的植物表达载体示意图。
图11 ZmZIPs基因在拟南芥中过表达提高拟南芥中锌含量的实验结果;ZmZIP2为转ZmZIP2基因在拟南芥中过表达提高拟南芥中铁或锌含量的实验结果;ZmZIP3为转ZmZIP3基因在拟南芥中过表达提高拟南芥中铁或锌含量的实验结果;ZmZIP7为转ZmZIP7基因在拟南芥中过表达提高拟南芥中铁或锌含量的实验结果;ZmZIP8为转ZmZIP8基因在拟南芥中过表达提高拟南芥中铁或锌含量的实验结果;WT为野生型哥伦比亚种子中铁和锌含量测定的结果。
具体实施方式
下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但这些实施例仅是范例性的,并不对本发明的范围构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改和替换均落入本发明的保护范围内。
实验材料
1.1植物材料
玉米自交系X178由河北农业大学农学院/国家玉米改良中心河北分中心实验室提供,水稻自交系日本晴由北京师范大学生命科学院惠赠。
1.2菌株与载体
大肠杆菌(E.coli)菌株Mach1-T1和农杆菌(A.tumefacterium)菌株EHA105、GV3101均由本实验室保存。pGEM-Teasy载体购自Promega公司。酵母表达载体pFL61(示意图见图1)、酵母菌株zrt1zrt2ZHY3(MATαade6can1his3leu2trp1ura3zrt1::LEU2zrt2::HIS3),fet3fet4 DEY1453(MATa/MATa ade2/+can1/can1his3/his3leu2/leu2trp1/trp1ura3/ura3 fet3-2::HIS3/fet3-2::HIS3fet4-1::LEU2/fet4-1::LEU2),DY1455(MATa ade6 can1 his3 leu2 trp1 ura3)由南京农业大学张红生教授友情惠赠。
实施例1玉米锌铁调控转运体ZmZIPs基因的克隆
1、植物材料的处理
先把蛭石用Hoagland营养液浸透,将玉米自交系X178种子点播于育苗盘中,上面覆盖上一层干蛭石,在温室(16h光照/8h黑暗,26℃)中培养,12天幼苗长至2叶一心时移入标准Hoagland营养液中生长6天至3叶一心(每3天换一次营养液),3叶一心的玉米幼苗在标准营养液和不加锌、铁、铜、锰、高锌、铁的条件下处理0、6、12、24、48、96h后,分别收取幼苗地上部和根,液氮速冻后于-80℃保存用于总RNA提取。
2、玉米总RNA的提取
采用Trizol法提取玉米总RNA。
3、cDNA的合成
(1)去除DNA,按下述配制反应体系:总RNA(1μg/μL)1.0μL,DNAse I(10U/μL)
1.0μL,10×DNAse I buffer 1.0μL,DEPC H2O 7.0μL,总计10.0μL;37℃30min,加入1μL 25mM EDTA,65℃5min终止反应。
(2)、加入1μL oligo(dT18),65℃5min;
(3)、以上共12μL,再加入以下组分得到反转录体系:5×反应缓冲液4.0μL,Ri RT(20U/μL)
1.0μL,Re RT(200U/μL)1.0μL,10mM dNTP mix 2.0μL,总计:20.0μL;42℃60min,70℃5min,终止反应。
4、目的基因的克隆
(1)、根据目的基因的ORF框设计引物:
ZmZIP1F 5'-GCGGCCGCATGCGCCGCCAAAGCCT-3'NotI
ZmZIP1R 5'-GCGGCCGCTTATTCTACCAGAGAAATGCCTAGAGCG-3'NotI
ZmZIP2F 5'-TACGTAATGGCCCGCGCCAC-3'SnaBI
ZmZIP2R 5'-TACGTATCAGGTGTCCCATATCATGACG-3'SnaBI
ZmZIP3F 5'-CCCGGGATGGGAGCTGTGAAGCATACATTG-3'SmaI
ZmZIP3R 5'-GGTACCCTATGCCCATATAGCAAGCATGGAC-3'KpnI
ZmZIP7F 5'-TCTAGAATGGTTCTCGCCGGCCTC-3'XbaI
ZmZIP7R 5'-GAGCTCTCAAGCCCATATTGCAAGTGATGACATAG-3'SacI
ZmZIP8F 5'-CCCGGGATGGCCATGAGGCCACG-3'SmaI
ZmZIP8R 5'-GAGCTCCTAGGCCCACTTGGCCAGC-3'SacI
以上述步骤3的cDNA为模板,选用ExTaq酶,2×GCI buffer进行PCR扩增,PCR程序为:95℃预变性4min;94℃变性1min,60℃退火1min,72℃延伸1min,33个循环;72℃延伸10min;
(2)、将克隆得到的片段克隆到pGEM-T载体中,转化Mach1-T1菌株;
(3)、经酶切鉴定获得阳性的重组质粒,测序得到正确克隆,所克隆的第1个基因命名为ZmZIP1,该基因的cDNA序列为SEQ ID No.1所示,所推导的氨基酸序列为SEQ ID No.2所示;所克隆的第2个基因命名为ZmZIP2,该基因的cDNA序列为SEQ ID No.3所示,所推导的氨基酸序列为SEQ ID No.4所示;所克隆的第3个基因命名为ZmZIP3,该基因的cDNA序列为SEQ ID No.5所示,所推导的氨基酸序列为SEQ ID No.6所示;所克隆的第4个基因命名为ZmZIP7,该基因的cDNA序列为SEQ ID No.7所示,所推导的氨基酸序列为SEQ ID No.8所示;所克隆的第5个基因命名为ZmZIP8,该基因的cDNA序列为SEQ ID No.9所示,所推导的氨基酸序列为SEQ ID No.10所示。
实施例2 ZmZIPs在幼苗、胚和胚乳中的表达模式
将实施例1步骤3中反转录的cDNA稀释10倍作为PCR反应的模板,PCR反应体系如下:
cDNA 2.0μL,ExTaq 0.1μL,2×GCI缓冲液10.0μL,10mM dNTP mix 0.8μL,上游引物RTZmZIP1F/RTZmZIP2F/RTZmZIP3F/RTZmZIP7F/RTZmZIP8F(10μM/μL)1.0μL,下游引物RTZmZIP1R/RTZmZIP2R/RTZmZIP3R/RTZmZIP7R/RTZmZIP8R(10μM/μL)1.0μL,ddH2O 5.1μL,总计20.0μL;
RTZmZIP1F 5'-CCTCTCTGCGTTGGTTGCTCT-3'
RTZmZIP1R 5'-TTGATGGTTGTTTTCTGGTCGT-3'
RTZmZIP2F 5'-CCACAAATGGCACGAGGTCT-3'
RTZmZIP2R 5'-CGAAGACGGAGTGGAAGCAAA-3'
RTZmZIP3F 5'-GCCTCTTGTTGGTGCCCTTA-3'
RTZmZIP3R 5'-TCAACAATGAACGCTGTAGTGCT-3'
RTZmZIP7F 5'-ACTAGGTGGGTGCATTGCTCAG-3'
RTZmZIP7R 5'-TGCCAGCAGATACCGAGTCAA-3'
RTZmZIP8F 5'-CGTGTCATCGCTCAGGTTCTTG-3'
RTZmZIP8R 5'-CCCTCGAACATTTGGTGGAAG-3'
PCR反应条件:94℃预变性4min;30个循环,每个循环94℃变性45秒,60℃退火1min,72℃延伸1min;最后再延伸72℃10min,降温至16℃,取出PCR产物放入4℃保存。
目的基因表达量的检测:实施例1步骤3中反转录的cDNA稀释20倍作为Real-time PCR反应的模板,Actin为内参照,反应体系如下:cDNA 5.0μL,SYBR Green I 10.0μL,Rox 0.4μL,上游引物ZmActin1F(10μM/μL)0.4μL,下游引物ZmActin1R(10μM/μL)0.4μL,ddH2O3.8μL,总计10.0μL;
ZmActin1F 5'-ATGTTTCCTGGGATTGCCGAT-3'
ZmActin1R 5'-CCAGTTTCGTCATACTCTCCCTTG-3'
所用程序:95℃2min,95℃15sec,60℃34sec,40个循环,通过ΔΔCt法计算表达量。
通过real-time RT-PCR表达分析发现,正常营养条件下,5个ZmZIPs基因主要在地上部表达,其中,缺锌条件下,ZmZIP8在96h地上部表达上调,ZmZIP3在6h时地上部和地下部表达量都有所升高;在高锌条件ZmZIP7和ZmZIP8在地上部的表达量是逐渐降低的,ZmZIP3在地下部的表达量明显的降低。这些结果表明,ZmZIP3、ZmZIP7和ZmZIP8在幼苗时期对锌的浓度比较敏感。缺铁条件下,ZmZIP7和ZmZIP8在地上与地下的表达量是逐渐增高的,说明ZmZIP7和ZmZIP8对铁的浓度比较敏感。缺铜、缺锰条件下,5个ZmZIPs基因的表达量都没有明显的变化。5个ZmZIPs基因可能参与胚和胚乳的发育(图2、图3和图4)。
实施例3 ZmZIPs的生物信息学分析
ZmZIPs由367-483个氨基酸组成,含有6-9个跨膜结构域,在第3与第4跨膜区之间有一富含组氨酸的可变区,可能和金属离子的结合转运有关。进化树分析显示,ZmZIP1与AtIAR1、OsIAR1进化关系较近。另外,ZmZIP3和ZmZIP4与OsZIP3和OsZIP4形成一个基因簇,ZmZIP2与OsZIP2邻近,和锌转运体OsZIP1,AtZIP2和AtZIP11在一个分支上,ZmZIP5与ZmZIP7在一个分支上,ZmZIP8与OsZIP8,ZmZIP6与OsZIP6进化关系较近,这些结果显示,本发明所分离的5个ZmZIPs可能是锌铁转运体。
实施例4ZmZIPs的亚细胞定位
1、融合表达载体的构建
根据ZmZIPs基因的序列设计引物,引物序列如下:
ZmZIP1GF 5'-GAATTCATGCGCCGCCAAAGCCT-3'EcoRI
ZmZIP1GR 5'-TCTAGATTCTACCAGAGAAATGCCTAGAGCG-3'XbaI
ZmZIP2GF 5'-GAATTCATGGCCCGCGCCACCAA-3'EcoRI
ZmZIP2GR 5'-TCTAGAGGTGTCCCATATCATGACGACGG-3'XbaI
ZmZIP3GF5'-GAATTCATGGGAGCTGTGAAGCATAC-3'EcoRI
ZmZIP3GR5'-TCTAGATGCCCATATAGCAAGCATGGACAT-3'XbaI
ZmZIP7GF 5'-GAATTCATGGTTCTCGCCGGCCTC-3'EcoRI
ZmZIP7GR 5'-TCTAGAAGCCCATATTGCAAGTGATGACATAG-3'XbaI
ZmZIP8GF 5'-GAATTCATGGCCATGAGGCCACGC-3'EcoRI
ZmZIP8GR 5'-TCTAGAGGCCCACTTGGCCAGCAT-3'XbaI
加入合适的酶切位点,并且基因3’端去除终止密码子,以克隆基因时连接到pGEM-T载体上测序正确的质粒为模板,选用ExTaq酶与2×GCI buffer进行PCR扩增,PCR程序为:95℃预变性4min;94℃变性1min,60℃退火1min,72℃延伸1min,33个循环;72℃延伸10min。扩增片段经1%琼脂糖凝胶电泳回收后克隆到pGEM-T载体中,转化大肠杆菌菌株Mach1-T1,经LB培养基(IPTG、X-gal、Amp)得到阳性克隆,提质粒、酶切和测序验证;以克隆基因时连接到pGEM-T载体上测序正确的质粒为模板,选用ExTaq酶与2×GCI buffer进行PCR扩增,扩增片段经1%琼脂糖凝胶电泳回收后克隆到pGEM-T载体中,测序正确的质粒酶切后,将目的片段构建到pRTL2NGFP载体上,分别命名为pRTL2NGFP-ZmZIP1、pRTL2NGFP-ZmZIP2、pRTL2NGFP-ZmZIP3、pRTL2NGFP-ZmZIP7、pRTL2NGFP-ZmZIP8,图5为酶切鉴定图。
2、用相应的酶切pRTL2NGFP载体与不同的酶切后的基因片段,经T4DNA连接酶连接,转化Mach1-T1菌株,提质粒酶切鉴定筛选出正确重组体大提质粒用于基因枪转化洋葱表皮。
3、基因枪微弹的制备
4、用基因枪进行洋葱表皮转化
从定位结果可知5个ZmZIPs(ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8)均定位在质膜与细胞内膜上(图6)。为进一步确定细胞内膜的具体定位,选用ER marker与ZmZIPs共定位进行拟南芥叶肉原生质体的转化,结果证明5个ZmZIPs均定位在细胞质膜及内质网上(图7)。
实施例五酵母互补实验
1、酵母表达载体的构建
根据目的基因序列加入合适的酶切位点设计引物:
ZmZIP1YF 5'-GCGGCCGCATGCGCCGCCAAAGCCT-3'NotI
ZmZIP1YR 5'-GCGGCCGCTTATTCTACCAGAGAAATGCCTAGAGCG-3'NotI
ZmZIP2YF 5'-TACGTAATGGCCCGCGCCAC-3'SnaBI
ZmZIP2YR 5'-TACGTATCAGGTGTCCCATATCATGACG-3'SnaBI
ZmZIP3YF5'-CCCGGGATGGGAGCTGTGAAGCATACATTG-3'SmaI
ZmZIP3YR5'-GGTACCCTATGCCCATATAGCAAGCATGGAC-3'KpnI
ZmZIP7YF 5'-TACGTAATGGTTCTCGCCGGCCTC-3'SnaBI
ZmZIP7YR 5'-TACGTATCAAGCCCATATTGCAAGTGATGACATAG-3'SnaBI
ZmZIP8YF 5'-TGCCATGGCCATGAGGCCAC-3'
ZmZIP8YR 5'-CTAGGCCCACTTGGCCAGCATG-3'
OsZIP5YF5'-CCCGGGGAGCCATCGGCGATGGCGA-3'SmaI
OsZIP5YR5'-GAGCTCGTGATGGTCACTCACTCATCACGCC-3'SacI
OsZIP8YF 5'-GCGGCCGCATGAGGACGAACACCACC-3'NotI
OsZIP8YR 5'-GCGGCCGCCCTCTACATTAGTCCCTGAG-3'NotI
OsIRT1YF 5'-GCGGCCGCCCCGGGATGGCGACGCCGCGGA-3'NotI,SmaI
OsIRT1YR 5'-GCGGCCGCCCCGGGTCACGCCCACTTGGCCATG-3'NotI,SmaI
以克隆基因时连接到pGEM-T载体上测序正确的质粒为模板,选用ExTaq与2×GCI buffer进行PCR扩增,PCR程序为:95℃预变性4min;94℃变性1min,60℃退火1min,72℃延伸1min,33个循环;72℃延伸10min。扩增片段经1%琼脂糖凝胶电泳回收后克隆到pGEM-T载体中,转化大肠杆菌菌株Mach1-T1,经LB培养基(IPTG、X-gal、Amp)得到阳性克隆,提质粒、酶切和测序验证,测序正确的质粒酶切后,将目的片段构建到pFL61载体上,命名为pFL61-ZmZIP1、pFL61-ZmZIP2、pFL61-ZmZIP3、pFL61-ZmZIP7、pFL61-ZmZIP8及pFL61-OsZIP5、pFL61-OsZIP8和pFL61-OsIRT1,图8为酶切鉴定图。
用NotI酶切pFL61载体与酶切后的ZmZIPs片段经T4DNA连接酶连接,转化Mach1-T1菌株,提质粒酶切鉴定筛选出正确重组体大提质粒用于转化酿酒酵母。
2、电击转化法转化酵母
(1)、从YPD平板上挑取zrt1zrt2ZHY3、fet3fet4DEY1453和DY1455的单菌落于20mL的YPD液体培养基中,28℃摇床培养约24h;
(2)、吸取以上2%体积的菌液转接到100mL的YPD培养基中继续扩繁约4-5h,待菌液OD600为1.2-1.5时即可制备感受态;
(3)、将菌液收集到50mL的离心管中,4℃,5,000rpm,离心5min,倒掉上清;
(4)、加入等体积的去离子水,冰上重悬菌体,4℃,5,000rpm,5min离心,倒掉上清;
(5)、加入1/2体积的去离子水,冰上重悬菌体,4℃,5,000rpm,5min离心,倒掉上清;
(6)、加入10mL的1M山梨醇溶液,冰上重悬菌体,4℃,5,000rpm,5min离心,倒掉上清;
(7)、加入450-600μL的山梨醇溶液,用去头的枪头轻吸,重悬菌体;
(8)、按照每个1.5mL的离心管里加入约100μL的感受态为准,分装;
(9)、在每管感受态中加入适量的DNA(10μL左右,c≥200ng/μL),冰上放置1-2min,之后吸到预冷的电击杯中,不要有气泡;
(10)、电击转化,立即加入约800μL,1M的预冷的山梨醇溶液,重悬菌体;
(11)、从电击杯中吸出菌体,涂布SD/Ura-平板;
(12)、SD平板上28℃培养约6天可长出肉眼可见的菌斑。
3、酵母阳性克隆的鉴定
(1)、取1.5mL酵母培养物,9,000rpm离心30秒,尽可能的吸弃上清,收集酵母细胞;
(2)、加入600μL Sorbitol buffer,轻柔吹打充分重悬细胞,加入80U的Lyticase,充分颠倒混匀,37℃温育30min消化细胞壁,中间颠倒数次;
(3)、13,000rpm离心1min,尽可能吸弃上清,加入250μL溶液YP1重悬菌体沉淀,涡旋震荡至彻底悬浮;
(4)、加入250μL YP2溶液,轻柔地翻转,使菌体充分裂解,室温放置4min;
(5)、加入350μL YP3溶液,轻柔地翻转,充分混匀时会出现白色絮状沉淀,冰上静置3-5min,13,000rpm离心5min,小心吸取上清液。
(6)、将上一步所得上清液加入吸附柱AC中(吸附柱放入收集管中),12,000rpm离心30-60秒,倒掉收集管中的废液;
(7)、加入500μL去蛋白液PD,12,000rpm离心30-60秒,弃废液;
(8)、加入500μL漂洗液WB(已加无水乙醇),12,000rpm离心30-60秒,弃废液;
(9)、加入500μL漂洗液WB,12,000rpm离心30-60秒,弃废液;
(10)、将吸附柱AC放回空收集管中,13,000rpm离心2min,除去漂洗液;
(11)、取出吸附柱AC,放入一个干净的离心管中,在吸附膜的中间部位加50μL洗脱缓冲液EB(65-70℃水浴),室温放置2min,13,000rpm离心1min。
(12)、以抽提的1μL DNA为模板,基因的两端引物为PCR扩增引物,进行PCR扩增验证目的基因,验证正确的菌液,加入25%的甘油于-80℃保存。
4、酵母互补实验结果
将pFL61、pFL61-ZmZIP1、pFL61-ZmZIP2、pFL61-ZmZIP3、pFL61-ZmZIP7、pFL61-ZmZIP8、pFL61-OsZIP5、pFL61-OsZIP8、pFL61-OsIRT1质粒分别转化到酵母突变株zrt1zrt2ZHY3和fet3fet4DEY1453中,pFL61为阴性对照,OsZIP5、OsZIP8(Lee S,Kim SA,Lee J,et al.Zinc deficiency-inducible OsZIP8encodes a plasma membrane-localized zinc transporter in rice.Molecules and cells 2010,29(6):551-558;Lee S,Jeong HJ,Kim SA,et al.OsZIP5is a plasma membrane zinc transporter in rice.Plant molecular biology 2010,73(4-5):507-517;Ishimaru Y,Masuda H,Suzuki M,et al.Overexpression of the OsZIP4zinc transporter confers disarrangement of zinc distribution in rice plants.Journal of experimental botany 2007,58(11):2909-2915.)为锌转运体的阳性对照,OsIRT1(Lee S,An G.Over-expression of OsIRT1leads to increased iron and zinc accumulations in rice.Plant,cell&environment 2009,32(4):408-416.)为铁转运体的阳性对照,pFL61转化野生型菌株DY1455作为另一阳性对照,转化后鉴定为阳性的酵母菌在SD液体培养基中培养,酵母菌液分别稀释4个浓度(OD600=1、0.1、0.01、0.001),然后取5μL点在低锌、低铁和正常SD的培养基中,低锌培养基(SD培养基加入0.4mM EDTA、0.4mM EDTA和250μM ZnSO4、0.4mM EDTA和300μM ZnSO4),低铁培养基(SD培养基加入50mM MES、50mM MES和50μM FeCl3、50mM MES和100μM FeCl3)酵母互补参照Lin,Y.F的试验(Lin YF,Liang HM,Yang SY,et al.Arabidopsis IRT3is a zinc-regulated and plasma membrane localized zinc/iron transporter.The New phytologist 2009,182(2):392-404.)方法进行,28℃培养,6天观察试验结果。
实验结果显示,在低锌条件下,加有250μM ZnSO4的培养基中能明显观察到DY-pFL61(野生型)、Z-ZmZIP1、Z-ZmZIP2、Z-ZmZIP3、Z-ZmZIP7、Z-ZmZIP8、Z-OsZIP5、Z-OsZIP8、Z-OsIRT1比空载体Z-pFL61长势好,并且,Z-ZmZIP1、Z-ZmZIP2、Z-ZmZIP3、Z-ZmZIP7、Z-ZmZIP8与已经报道的水稻OsZIP长势相当。在低铁条件下,D-ZmZIP1、D-ZmZIP2、D-ZmZIP3、D-ZmZIP7、D-ZmZIP8比空载体D-pFL61长势好,但是没有已经报道的D-OsIRT1的转运活性强(图9);ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8不论在低锌还是在低铁条件下都表现出不同程度的转运活性,说明ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8具有转运锌铁的功能。
实验例1 ZmZIPs基因在拟南芥中过表达提高拟南芥种子中铁和锌含量的实验
将ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7以及ZmZIP8基因分别与组成型35S启动子控制的植物表达载体pBI121相连接构建得到ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7和ZmZIP8基因重组植物表达载体(图10);将构建的重组植物表达载体分别转化到拟南芥中,鉴定获得阳性的转ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7和ZmZIP8基因拟南芥;将阳性的转ZmZIPs基因拟南芥与野生型哥伦比亚在相同的载培条件下培养,收获转ZmZIPs基因拟南芥种子和野生型种子,分别测定转ZmZIPs基因拟南芥种子和野生型拟南芥种子中铁或锌的含量;称取一定量的种子材料经微波消解,定容,用ICP-MS方法进行锌铁含量的测定;每批测定200mg种子,测定三批,取三批数据的平均值。测定结果见图11。从图11的结果可见,在拟南芥中过表达ZmZIP1、ZmZIP2、ZmZIP3、ZmZIP7或ZmZIP8基因均能够不同程度的提高种子中铁或锌含量。
<110> 河北农业大学
<120> 玉米锌铁调控转运体ZmZIP2基因及其应用
<130> XLB--0030
<160> 10
<170> PatentIn version 3.5
<210> 1
<211> 1473
<212> DNA
<213> Zea mays
<400> 1
atgcgccgcc aaagcctcgc caccgtactg ctgctcctgg tggccgccgc ggccctggcc 60
gcccccgccg ctgggcactc tgagtcctcc tgccccttct acgaccacgg cggccacggc 120
gaaccacacg aacgccacga ccatggccac agctgcggcg gcggcgcgga ccatgagcac 180
caccatcacc atcaccacgg ccatggacac ggcgagatcc agcggctgct cccggaggag 240
atggcggagg aggcggatct cgagctcgag tccttcggtt acgaagacca tgaccatgac 300
cacggccacc accaccacca ccaccatcac cacagccacg gcgacatgga gacatcgccc 360
atgggcgtgt ggctgagcgc gatggggtgc tcgctgctgg tcagtatggc gtcccttgtc 420
tgcctcgtcc tcttgccggt catcttcttt aaggggaaac cgtctaaggc catagtggat 480
tcgcttgcag tgtttggggc aggagctatg cttggagatt catttcttca tcagctgcca 540
catgcttttg gtggaggaca ttctcactcg catgatcatg agggtcatga tcatgctcat 600
gctcatgagc atgcacatgc acactcactg caagatctat ctgtgggttt gtctgtacta 660
tttggcattg tactgttttt tattgtcgag aagattgtga ggtatgttga agacaattct 720
caaaatgggg ctcatagcat gggtcatggg caccatcatc ataatcataa acggcacgat 780
tctagcgata aagccaaatt gaattaccaa aagagtgata ctgatggtaa agacattgat 840
catgctgaag aggaaccttc ggttaatgat accactggaa aaataagtga tggccatgaa 900
tcagaagcta ctatacgcaa gaggagctca tccaaagcca ctgatggaga agccaccaat 960
tctggaaggg atcctgcccc tgaaaaagca ccatcaaatg aaggttcatc aatttcaaac 1020
tctaacttag tgtttggcta cctcaacctt ttctcagatg gtgttcataa cttcactgat 1080
gggatggctc ttgggagtgc ctttctgctg catggtcctg ttggtggctg gtctaggact 1140
ttatttctgc ttgcacatga acttccccaa gaggtaggag attttggtat ccttgtgcgg 1200
tcaggcttca cggtatctaa ggccttattc ttcaatttcc tctctgcgtt ggttgctctt 1260
gctggaacag cactagcatt gtctttgggt aaagatccgg gacattcctc cctgattgag 1320
ggtttcaccg ccggtggttt catttacatt gctgtcgcgg gagtcctacc acagatgaac 1380
gaccagaaaa caaccatcaa gagctcagta gctcagctga tctccctggc aatggggatg 1440
ctggtcgctc taggcatttc tctggtagaa taa 1473
<210> 2
<211> 490
<212> PRT
<213> Zea mays
<400> 2
Met Arg Arg Gln Ser Leu Ala Thr Val Leu Leu Leu Leu Val Ala Ala
1 5 10 15
Ala Ala Leu Ala Ala Pro Ala Ala Gly His Ser Glu Ser Ser Cys Pro
20 25 30
Phe Tyr Asp His Gly Gly His Gly Glu Pro His Glu Arg His Asp His
35 40 45
Gly His Ser Cys Gly Gly Gly Ala Asp His Glu His His His His His
50 55 60
His His Gly His Gly His Gly Glu Ile Gln Arg Leu Leu Pro Glu Glu
65 70 75 80
Met Ala Glu Glu Ala Asp Leu Glu Leu Glu Ser Phe Gly Tyr Glu Asp
85 90 95
His Asp His Asp His Gly His His His His His His His His His Ser
100 105 110
His Gly Asp Met Glu Thr Ser Pro Met Gly Val Trp Leu Ser Ala Met
115 120 125
Gly Cys Ser Leu Leu Val Ser Met Ala Ser Leu Val Cys Leu Val Leu
130 135 140
Leu Pro Val Ile Phe Phe Lys Gly Lys Pro Ser Lys Ala Ile Val Asp
145 150 155 160
Ser Leu Ala Val Phe Gly Ala Gly Ala Met Leu Gly Asp Ser Phe Leu
165 170 175
His Gln Leu Pro His Ala Phe Gly Gly Gly His Ser His Ser His Asp
180 185 190
His Glu Gly His Asp His Ala His Ala His Glu His Ala His Ala His
195 200 205
Ser Leu Gln Asp Leu Ser Val Gly Leu Ser Val Leu Phe Gly Ile Val
210 215 220
Leu Phe Phe Ile Val Glu Lys Ile Val Arg Tyr Val Glu Asp Asn Ser
225 230 235 240
Gln Asn Gly Ala His Ser Met Gly His Gly His His His His Asn His
245 250 255
Lys Arg His Asp Ser Ser Asp Lys Ala Lys Leu Asn Tyr Gln Lys Ser
260 265 270
Asp Thr Asp Gly Lys Asp Ile Asp His Ala Glu Glu Glu Pro Ser Val
275 280 285
Asn Asp Thr Thr Gly Lys Ile Ser Asp Gly His Glu Ser Glu Ala Thr
290 295 300
Ile Arg Lys Arg Ser Ser Ser Lys Ala Thr Asp Gly Glu Ala Thr Asn
305 310 315 320
Ser Gly Arg Asp Pro Ala Pro Glu Lys Ala Pro Ser Asn Glu Gly Ser
325 330 335
Ser Ile Ser Asn Ser Asn Leu Val Phe Gly Tyr Leu Asn Leu Phe Ser
340 345 350
Asp Gly Val His Asn Phe Thr Asp Gly Met Ala Leu Gly Ser Ala Phe
355 360 365
Leu Leu His Gly Pro Val Gly Gly Trp Ser Arg Thr Leu Phe Leu Leu
370 375 380
Ala His Glu Leu Pro Gln Glu Val Gly Asp Phe Gly Ile Leu Val Arg
385 390 395 400
Ser Gly Phe Thr Val Ser Lys Ala Leu Phe Phe Asn Phe Leu Ser Ala
405 410 415
Leu Val Ala Leu Ala Gly Thr Ala Leu Ala Leu Ser Leu Gly Lys Asp
420 425 430
Pro Gly His Ser Ser Leu Ile Glu Gly Phe Thr Ala Gly Gly Phe Ile
435 440 445
Tyr Ile Ala Val Ala Gly Val Leu Pro Gln Met Asn Asp Gln Lys Thr
450 455 460
Thr Ile Lys Ser Ser Val Ala Gln Leu Ile Ser Leu Ala Met Gly Met
465 470 475 480
Leu Val Ala Leu Gly Ile Ser Leu Val Glu
485 490
<210> 3
<211> 1080
<212> DNA
<213> Zea mays
<400> 3
atggcccgcg ccaccaacgc ccaccaccac cgcctccgcc tcctcctctg cctctccctc 60
gcggccgccg cgtgggcgca cggcggaggc ggggactccg acgccgacgc cgacgcggat 120
gggggcgccg ccgccaggcc ggacctgcgc gcgcgcagcc tggtggaggc caagctgtgg 180
tgcctggcgg tggtgttcgt cggcacgctg ctgggcgggg tgtcccccta cttcatgcgc 240
tggaacgagg cgttcctcgc gctgggcacg cagttcgcgg gcggcgtctt cctcggcacg 300
gcgctcatgc acttcctcag cgacgccaac gagacgttcg gggacctgct ccccgacagc 360
gggtacccct gggcgttcat gctcgcctgc gccggttacg tcgtcaccat gctcgccgac 420
gtcgccatct cctacgtcgt ctcacggtca caggggcgca gcaccggcac cgccgctacc 480
ggcggttctg atgcagggct ggaggagggc aagatgagaa ccacaaatgg cacgaggtct 540
gagcccacac cagctgatgc acacggatct gatcactcgg ccgcatccat tctgcgcaac 600
gcgagcacga tcggtgacag cgtgctgctc atagtagccc tttgcttcca ctccgtcttc 660
gagggcatcg ccatcgggat cgccgagacc aaggccgacg catggaaggc gctgtggacc 720
ataagcctgc acaagatctt cgcggccatc gccatgggca tcgcgctgct ccggatgctg 780
cccaaccggc ccctcctctc ctgcttcgcc tacgccttcg cgttcgccat ctccagcccc 840
gtcggcgtcg gcatcggcat catcatcgat gccaccacgc agggccgggt ggccgactgg 900
atcttcgccg tctccatggg cctcgccacg ggcatcttcg tctacgtctc catcaaccac 960
ctcctctcca aagggtaccg gccccagagg cccgtcgccg tcgacacgcc ggtcgggagg 1020
tggctcgccg tcgtgttcgg cgtggctgtc atcgccgtcg tcatgatatg ggacacctga 1080
<210> 4
<211> 359
<212> PRT
<213> Zea mays
<400> 4
Met Ala Arg Ala Thr Asn Ala His His His Arg Leu Arg Leu Leu Leu
1 5 10 15
Cys Leu Ser Leu Ala Ala Ala Ala Trp Ala His Gly Gly Gly Gly Asp
20 25 30
Ser Asp Ala Asp Ala Asp Ala Asp Gly Gly Ala Ala Ala Arg Pro Asp
35 40 45
Leu Arg Ala Arg Ser Leu Val Glu Ala Lys Leu Trp Cys Leu Ala Val
50 55 60
Val Phe Val Gly Thr Leu Leu Gly Gly Val Ser Pro Tyr Phe Met Arg
65 70 75 80
Trp Asn Glu Ala Phe Leu Ala Leu Gly Thr Gln Phe Ala Gly Gly Val
85 90 95
Phe Leu Gly Thr Ala Leu Met His Phe Leu Ser Asp Ala Asn Glu Thr
100 105 110
Phe Gly Asp Leu Leu Pro Asp Ser Gly Tyr Pro Trp Ala Phe Met Leu
115 120 125
Ala Cys Ala Gly Tyr Val Val Thr Met Leu Ala Asp Val Ala Ile Ser
130 135 140
Tyr Val Val Ser Arg Ser Gln Gly Arg Ser Thr Gly Thr Ala Ala Thr
145 150 155 160
Gly Gly Ser Asp Ala Gly Leu Glu Glu Gly Lys Met Arg Thr Thr Asn
165 170 175
Gly Thr Arg Ser Glu Pro Thr Pro Ala Asp Ala His Gly Ser Asp His
180 185 190
Ser Ala Ala Ser Ile Leu Arg Asn Ala Ser Thr Ile Gly Asp Ser Val
195 200 205
Leu Leu Ile Val Ala Leu Cys Phe His Ser Val Phe Glu Gly Ile Ala
210 215 220
Ile Gly Ile Ala Glu Thr Lys Ala Asp Ala Trp Lys Ala Leu Trp Thr
225 230 235 240
Ile Ser Leu His Lys Ile Phe Ala Ala Ile Ala Met Gly Ile Ala Leu
245 250 255
Leu Arg Met Leu Pro Asn Arg Pro Leu Leu Ser Cys Phe Ala Tyr Ala
260 265 270
Phe Ala Phe Ala Ile Ser Ser Pro Val Gly Val Gly Ile Gly Ile Ile
275 280 285
Ile Asp Ala Thr Thr Gln Gly Arg Val Ala Asp Trp Ile Phe Ala Val
290 295 300
Ser Met Gly Leu Ala Thr Gly Ile Phe Val Tyr Val Ser Ile Asn His
305 310 315 320
Leu Leu Ser Lys Gly Tyr Arg Pro Gln Arg Pro Val Ala Val Asp Thr
325 330 335
Pro Val Gly Arg Trp Leu Ala Val Val Phe Gly Val Ala Val Ile Ala
340 345 350
Val Val Met Ile Trp Asp Thr
355
<210> 5
<211> 1104
<212> DNA
<213> Zea mays
<400> 5
atgggagctg tgaagcatac attgaaaatg ctttcatggc tcctactctt cgcacaattg 60
gctgcggcca ccacctccaa gtgcacaaat gccacaaatg ggactgagac tgacagcctg 120
ggtgcaatga aactgaagct gattgccatt gcatccatcc tcacagctgg agcagctggt 180
gtgctagtgc cagtacttgg ccgctccatg gccacgctac atcctgatgg tgacatcttt 240
ttcgctgtca aggcatttgc agccggtgtc atcctggcca cgggcatggt ccacattctg 300
ccagcagcat ttgatgggct gacatcccca tgcctctaca aaggtggcag tggtgggaac 360
attttcccct ttgcaggact cattgcaatg tctgctgcaa tggccactat ggtgatagac 420
tcactagctg ctgggtacta ccgccggtct cacttcaaga aggcacggcc aatcgacatc 480
ctagagatcc atgaacaacc aggagatgag gaaaggtccg ggcatgcaca acatgtgcat 540
gtgcacaccc atgcaacaca tgggcattca cacggagagg tggatgtcat cagttcaccg 600
gaggaggctt caatagctga cacgatccgg cacagggtgg tatctcaggt ccttgagctc 660
ggaattttgg tgcattcagt gataattggg gtgtccttag gtgcatctgt gaggtcatcg 720
accataaggc ctcttgttgg tgcccttagc ttccatcaat tctttgaagg cattggcctg 780
ggtggttgca ttgtgcaggc aaatttcaag ttgagggcta ccgtcatgat ggcaattttt 840
ttctccctga ctgcacccat tggcattgca ttagggattg gaatttcatc aagctacaat 900
ggccatagca ctacagcgtt cattgttgaa ggagtgttca actcagcctc agcaggaatt 960
ttgatctaca tgtcattagt cgatcttctg gccacagatt tcaataagcc caaactgcag 1020
acgaatacaa agcttcagct gatgacatat cttgcacttt tcttaggtgc agggatgatg 1080
tccatgcttg ctatatgggc atag 1104
<210> 6
<211> 367
<212> PRT
<213> Zea mays
<400> 6
Met Gly Ala Val Lys His Thr Leu Lys Met Leu Ser Trp Leu Leu Leu
1 5 10 15
Phe Ala Gln Leu Ala Ala Ala Thr Thr Ser Lys Cys Thr Asn Ala Thr
20 25 30
Asn Gly Thr Glu Thr Asp Ser Leu Gly Ala Met Lys Leu Lys Leu Ile
35 40 45
Ala Ile Ala Ser Ile Leu Thr Ala Gly Ala Ala Gly Val Leu Val Pro
50 55 60
Val Leu Gly Arg Ser Met Ala Thr Leu His Pro Asp Gly Asp Ile Phe
65 70 75 80
Phe Ala Val Lys Ala Phe Ala Ala Gly Val Ile Leu Ala Thr Gly Met
85 90 95
Val His Ile Leu Pro Ala Ala Phe Asp Gly Leu Thr Ser Pro Cys Leu
100 105 110
Tyr Lys Gly Gly Ser Gly Gly Asn Ile Phe Pro Phe Ala Gly Leu Ile
115 120 125
Ala Met Ser Ala Ala Met Ala Thr Met Val Ile Asp Ser Leu Ala Ala
130 135 140
Gly Tyr Tyr Arg Arg Ser His Phe Lys Lys Ala Arg Pro Ile Asp Ile
145 150 155 160
Leu Glu Ile His Glu Gln Pro Gly Asp Glu Glu Arg Ser Gly His Ala
165 170 175
Gln His Val His Val His Thr His Ala Thr His Gly His Ser His Gly
180 185 190
Glu Val Asp Val Ile Ser Ser Pro Glu Glu Ala Ser Ile Ala Asp Thr
195 200 205
Ile Arg His Arg Val Val Ser Gln Val Leu Glu Leu Gly Ile Leu Val
210 215 220
His Ser Val Ile Ile Gly Val Ser Leu Gly Ala Ser Val Arg Ser Ser
225 230 235 240
Thr Ile Arg Pro Leu Val Gly Ala Leu Ser Phe His Gln Phe Phe Glu
245 250 255
Gly Ile Gly Leu Gly Gly Cys Ile Val Gln Ala Asn Phe Lys Leu Arg
260 265 270
Ala Thr Val Met Met Ala Ile Phe Phe Ser Leu Thr Ala Pro Ile Gly
275 280 285
Ile Ala Leu Gly Ile Gly Ile Ser Ser Ser Tyr Asn Gly His Ser Thr
290 295 300
Thr Ala Phe Ile Val Glu Gly Val Phe Asn Ser Ala Ser Ala Gly Ile
305 310 315 320
Leu Ile Tyr Met Ser Leu Val Asp Leu Leu Ala Thr Asp Phe Asn Lys
325 330 335
Pro Lys Leu Gln Thr Asn Thr Lys Leu Gln Leu Met Thr Tyr Leu Ala
340 345 350
Leu Phe Leu Gly Ala Gly Met Met Ser Met Leu Ala Ile Trp Ala
355 360 365
<210> 7
<211> 1164
<212> DNA
<213> Zea mays
<400> 7
atggttctcg ccggcctccg ccggcacgtc ggccagttct tgaccagcag taatgagctt 60
atggcggcgt cgctctcggc cgtgagctgc gcggacgagg tgcagaaggc ggagggagcg 120
gggtgccgtg acgacgccgc ggcgctgcgg ctcaagaagg tggccatggc ggcgatactg 180
gtggccgggg tgctcggggt ggtcctgccg ctcgcggggc ggaagcggcg cgcgctgcgc 240
acggacagcg ccgcgttcct ggcggccaag gcgttcgccg ccggggtcat cctggccacg 300
gggttcgtcc acatgcttca cgacgccgag cacgcgctgt ccagcccgtg cctccccgcc 360
gcgccgtggc gacggttccc agtccccggg ttcgtcgcca tggccgccgc gctagccacg 420
ctggtgctcg acttccttgc caccaggttc tacgaggcca agcatcgcga cgaggccgcg 480
cgcgtcaagg cggccgccgc cgccgcactg gttgcgacct cctctgggag cgacgaggac 540
atcaccgtgg tcaccgtcga tgaggacgag cgcaaggccc cgctgttgca aacgcactgc 600
catggacacg gtcacagcca cagccacagc catgtgcatg agccggtgca ggtagagggt 660
agcgaggcgg aggtgtcggc acatgtgcgc tccattgtcg tgtcacagat actggagatg 720
gggattgtgt cacactctgt gatcatcgga ttgtcactgg gagtgtcgcg gagcccatgc 780
acaatcaggc cacttgttgc agcacttgca ttccaccagt tcttcgaggg gtttgcacta 840
ggtgggtgca ttgctcaggc acaattcaag aacctttcag cagttctaat ggcatctttc 900
ttcgccatca caacaccggc aggaatagct gctggggcag gcatgaccac attctacaat 960
cccaatagcc caagggccct tgtggtcgag ggcattcttg actcggtatc tgctggcatt 1020
ctcatataca tgtcactagt ggatctcatt gctgtagatt tcttgggtgg aaagatgaca 1080
gggaccctgc gacaacaagt gatggcatac atcgcattgt tcctcggtgc gctctctatg 1140
tcatcacttg caatatgggc ttga 1164
<210> 8
<211> 387
<212> PRT
<213> Zea mays
<400> 8
Met Val Leu Ala Gly Leu Arg Arg His Val Gly Gln Phe Leu Thr Ser
1 5 10 15
Ser Asn Glu Leu Met Ala Ala Ser Leu Ser Ala Val Ser Cys Ala Asp
20 25 30
Glu Val Gln Lys Ala Glu Gly Ala Gly Cys Arg Asp Asp Ala Ala Ala
35 40 45
Leu Arg Leu Lys Lys Val Ala Met Ala Ala Ile Leu Val Ala Gly Val
50 55 60
Leu Gly Val Val Leu Pro Leu Ala Gly Arg Lys Arg Arg Ala Leu Arg
65 70 75 80
Thr Asp Ser Ala Ala Phe Leu Ala Ala Lys Ala Phe Ala Ala Gly Val
85 90 95
Ile Leu Ala Thr Gly Phe Val His Met Leu His Asp Ala Glu His Ala
100 105 110
Leu Ser Ser Pro Cys Leu Pro Ala Ala Pro Trp Arg Arg Phe Pro Val
115 120 125
Pro Gly Phe Val Ala Met Ala Ala Ala Leu Ala Thr Leu Val Leu Asp
130 135 140
Phe Leu Ala Thr Arg Phe Tyr Glu Ala Lys His Arg Asp Glu Ala Ala
145 150 155 160
Arg Val Lys Ala Ala Ala Ala Ala Ala Leu Val Ala Thr Ser Ser Gly
165 170 175
Ser Asp Glu Asp Ile Thr Val Val Thr Val Asp Glu Asp Glu Arg Lys
180 185 190
Ala Pro Leu Leu Gln Thr His Cys His Gly His Gly His Ser His Ser
195 200 205
His Ser His Val His Glu Pro Val Gln Val Glu Gly Ser Glu Ala Glu
210 215 220
Val Ser Ala His Val Arg Ser Ile Val Val Ser Gln Ile Leu Glu Met
225 230 235 240
Gly Ile Val Ser His Ser Val Ile Ile Gly Leu Ser Leu Gly Val Ser
245 250 255
Arg Ser Pro Cys Thr Ile Arg Pro Leu Val Ala Ala Leu Ala Phe His
260 265 270
Gln Phe Phe Glu Gly Phe Ala Leu Gly Gly Cys Ile Ala Gln Ala Gln
275 280 285
Phe Lys Asn Leu Ser Ala Val Leu Met Ala Ser Phe Phe Ala Ile Thr
290 295 300
Thr Pro Ala Gly Ile Ala Ala Gly Ala Gly Met Thr Thr Phe Tyr Asn
305 310 315 320
Pro Asn Ser Pro Arg Ala Leu Val Val Glu Gly Ile Leu Asp Ser Val
325 330 335
Ser Ala Gly Ile Leu Ile Tyr Met Ser Leu Val Asp Leu Ile Ala Val
340 345 350
Asp Phe Leu Gly Gly Lys Met Thr Gly Thr Leu Arg Gln Gln Val Met
355 360 365
Ala Tyr Ile Ala Leu Phe Leu Gly Ala Leu Ser Met Ser Ser Leu Ala
370 375 380
Ile Trp Ala
385
<210> 9
<211> 1191
<212> DNA
<213> Zea mays
<400> 9
atggccatga ggccacgcgc cgcgctcgcc ctcgccctgg cagccggcgt cccgctggtc 60
ctcctcctcc tcctcgcgcc cggcgcgcgc gccgacgacg gcagcggcgg gtgcggggcg 120
gcgggcggcg gcgaggcggc gccgggcgac cgcgcgcggg ccagggcgct gaagatcgcg 180
gccttcttct ccatcctcgt gtgcggggcg ctgggctgct gcctgcccgt gctggggcgg 240
cgcgtgccgg cgctgcgccc cgacagggac gtgttcttcc tgatcaaggc gttcgcggcg 300
ggggtcatcc tggccacggg gttcatccac atcctccccg acgcgttcga gaagctcacg 360
tccgattgcc tctccgacgg gccgtggcag gacttcccct tcgcggggct cggcgccatg 420
gtcggcgcca tcggcacgct cgtcgtcgac accgtcgcca cgggctactt cacgcgcgtc 480
cacttcaagg acagcgccgc cgccgccgtg ggcgccgccg ccgtcggcga cgaggagaag 540
cagcagcagc aggcggcgtc ggcgccgcac gtcgacgacg gagcagacgg cgacggccac 600
ggccacggcg ggcacgtgca catgcacacg cacgcgacgc acgggcactc gcacggcgcc 660
tcggcgctcg tggccgccgt cggcggcgcc gagggcgaca aggagcacgc gctgcgccac 720
cgtgtcatcg ctcaggttct tgagcttggg attgtggtgc actcggtgat catcggcatc 780
tccctcggcg cgtctcaaga ccccagcacc atcaagcctc ttgtggtcgc cctcagcttc 840
caccaaatgt tcgagggcat gggtcttggc ggctgcatcg ttcaggccaa gttcaagctg 900
cggtcgatcg tgacgatggt gctcttcttc tgcctgacga cgccggtggg catcgtggtg 960
ggcgtcggga tctcgtcggt gtacgacgag gacagcccca cggcgctggt cgtggagggc 1020
gtgctcaact cggtggcggc ggggatcctg gtgtacatgg cgctggtgga cctgctcgcc 1080
gaggacttca tgaaccctag ggtgcagagc cggggcaagc tgcagctcgg catcaacgcc 1140
tccatgctcg tcggcgccgg cctcatgtcc atgctggcca agtgggccta g 1191
<210> 10
<211> 396
<212> PRT
<213> Zea mays
<400> 10
Met Ala Met Arg Pro Arg Ala Ala Leu Ala Leu Ala Leu Ala Ala Gly
1 5 10 15
Val Pro Leu Val Leu Leu Leu Leu Leu Ala Pro Gly Ala Arg Ala Asp
20 25 30
Asp Gly Ser Gly Gly Cys Gly Ala Ala Gly Gly Gly Glu Ala Ala Pro
35 40 45
Gly Asp Arg Ala Arg Ala Arg Ala Leu Lys Ile Ala Ala Phe Phe Ser
50 55 60
Ile Leu Val Cys Gly Ala Leu Gly Cys Cys Leu Pro Val Leu Gly Arg
65 70 75 80
Arg Val Pro Ala Leu Arg Pro Asp Arg Asp Val Phe Phe Leu Ile Lys
85 90 95
Ala Phe Ala Ala Gly Val Ile Leu Ala Thr Gly Phe Ile His Ile Leu
100 105 110
Pro Asp Ala Phe Glu Lys Leu Thr Ser Asp Cys Leu Ser Asp Gly Pro
115 120 125
Trp Gln Asp Phe Pro Phe Ala Gly Leu Gly Ala Met Val Gly Ala Ile
130 135 140
Gly Thr Leu Val Val Asp Thr Val Ala Thr Gly Tyr Phe Thr Arg Val
145 150 155 160
His Phe Lys Asp Ser Ala Ala Ala Ala Val Gly Ala Ala Ala Val Gly
165 170 175
Asp Glu Glu Lys Gln Gln Gln Gln Ala Ala Ser Ala Pro His Val Asp
180 185 190
Asp Gly Ala Asp Gly Asp Gly His Gly His Gly Gly His Val His Met
195 200 205
His Thr His Ala Thr His Gly His Ser His Gly Ala Ser Ala Leu Val
210 215 220
Ala Ala Val Gly Gly Ala Glu Gly Asp Lys Glu His Ala Leu Arg His
225 230 235 240
Arg Val Ile Ala Gln Val Leu Glu Leu Gly Ile Val Val His Ser Val
245 250 255
Ile Ile Gly Ile Ser Leu Gly Ala Ser Gln Asp Pro Ser Thr Ile Lys
260 265 270
Pro Leu Val Val Ala Leu Ser Phe His Gln Met Phe Glu Gly Met Gly
275 280 285
Leu Gly Gly Cys Ile Val Gln Ala Lys Phe Lys Leu Arg Ser Ile Val
290 295 300
Thr Met Val Leu Phe Phe Cys Leu Thr Thr Pro Val Gly Ile Val Val
305 310 315 320
Gly Val Gly Ile Ser Ser Val Tyr Asp Glu Asp Ser Pro Thr Ala Leu
325 330 335
Val Val Glu Gly Val Leu Asn Ser Val Ala Ala Gly Ile Leu Val Tyr
340 345 350
Met Ala Leu Val Asp Leu Leu Ala Glu Asp Phe Met Asn Pro Arg Val
355 360 365
Gln Ser Arg Gly Lys Leu Gln Leu Gly Ile Asn Ala Ser Met Leu Val
370 375 380
Gly Ala Gly Leu Met Ser Met Leu Ala Lys Trp Ala
385 390 395
- 还没有人留言评论。精彩留言会获得点赞!
- 拟南芥SSCD1基因突变在调控植物体内茉莉酸合成中的应用的制作方法与工艺
- 一种基因调控网络的优化控制方法与流程
- 拟南芥WRKY家族转录因子AtWRKY44基因,蛋白编码序列及其应用的制作方法与工艺
- 针对免疫调控能力相关基因个体化配方食品及其制备方法与流程
- REVOLUTA基因在调控豆科植物小叶片数目和叶茎比中的应用的制作方法与工艺
- 玉米锌铁调控转运体ZmZIP3基因及其应用的制作方法与工艺
- 拟南芥基因SPOC1在调控植株开花期中的应用的制作方法与工艺
- 调控石榴花胚珠发育的PgAG基因及其用途的制作方法与工艺
- 光效调控基因HPE1及其应用的制作方法与工艺
- 菘蓝IiAP2/ERF049基因在调控木脂素类化合物合成中的应用的制作方法与工艺