水稻FAH基因在水稻育性控制中的应用的制作方法
本发明属于基因工程应用技术领域,涉及水稻FAH基因在控制水稻育性方面的应用,具体是通过抑制水稻FAH基因的表达创建水稻雄性不育材料。
背景技术:
水稻是重要的粮食作物,其生产对我国粮食的供给具有重要的意义。杂交水稻的推广,使水稻产量得到大幅度提高,一定程度上缓解了我国人口迅速增长对粮食需求急剧增长的压力。杂交水稻是选用遗传上具有一定差异的优良水稻品种为亲本,采用“三系法”或“二系法”等生产具有杂种优势的杂交种。杂交种在产量、生长势、生活力、繁殖力等方面都优于双亲。“三系法”是利用不育系、保持系和恢复系进行杂交育种,是水稻育种和推广的一个巨大成就。其中,雄性不育系雌蕊发育正常,而雄蕊的发育退化或败育,不能自花授粉结实;保持系雌雄蕊发育正常,将其花粉授予雄性不育系的雌蕊,不仅可结种子,而且播种后仍可获得雄性不育植株;恢复系花粉授予不育系的雌蕊,所产生的种子播种后,长成的植株又恢复了育性。雄性不育性的利用是制备杂交水稻的重要环节。但是从自然界中寻找的不育系并不多。利用基因工程的方法获得不育系是一条有效的途径。如通过干扰某些基因的表达使花粉败育获得不育系(de Groot et al,2004;Chen et al,2007;Shi et al,2015)。
FAH基因编码的延胡索酰乙酰乙酸水解酶参与酪氨酸降解途径。在拟南芥中编码FAH的基因突变后会造成拟南芥在短日照条件下的细胞死亡(Han et al,2013)。但是,目前尚未有关于水稻FAH基因在控制育性方面的研究报道。
技术实现要素:
本发明的目的在于,针对上述现有技术的不足,提供水稻FAH基因在水稻育性控制中的应用,为培育水稻不育系提供新的方法。
为达上述目的,本发明所采用的技术方案是:水稻FAH基因在控制水稻育性中的应用,该FAH基因的核苷酸序列如SEQ ID No.3所示。其中,该水稻FAH基因在育性控制方面的应用是指干扰该基因的表达降低水稻植物的育性,即该基因的表达降低使水稻植物的育性降低。
本发明通过基因工程手段将该FAH基因与干扰表达载体连接后,转化至其它植物如水稻中,抑制该基因在转化后的植物体内的表达,可以使该植物的育性降低。
本发明同时提供上述水稻FAH基因在培育水稻雄性不育植株中的应用,该FAH基因的核苷酸序列如SEQ ID No.3所示。
本发明还提供一种培育水稻雄性不育植株的方法,该方法是将含权利要求1所述FAH基因的干扰表达载体导入水稻植株中,能抑制水稻中FAH基因的表达,使水稻育性降低,该水稻植株经培育即得水稻雄性不育植株。
本发明克隆了水稻FAH基因,分析该基因在水稻不同组织中的表达,发现该基因在水稻幼穗中的表达水平最高。进一步将该基因与植物干扰表达载体连接重组后,转化至水稻日本晴中,发现转基因水稻中该基因的表达水平降低,并且转基因水稻的花粉活力和育性都降低。通过对水稻中该基因的表达干扰,降低其在水稻中的表达水平,可以获得雄性不育的转基因水稻,这些不育系能用于杂交水稻的生产。另外可以利用该基因抑制其它植物中其同源基因的表达,而控制其它植物的育性。
附图说明
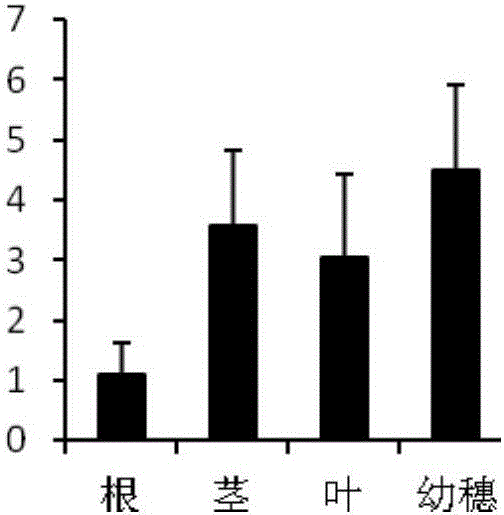
图1为水稻FAH基因在水稻不同组织中的表达分析。
图2为FAH基因干扰表达转基因水稻的鉴定。
其中,A:FAH基因干扰表达转基因水稻的PCR鉴定;WT表示非转基因对照水稻;1-4表示FAH干扰表达转基因水稻;
B:FAH基因干扰表达转基因水稻中FAH基因的表达分析;WT表示非转基因对照水稻;1,3,4表示FAH干扰表达转基因水稻。
图3是FAH基因干扰表达转基因水稻的育性观察。
其中,A:FAH基因干扰表达转基因水稻植株的观察;B:FAH基因干扰表达转基因水稻种子的观察;C:FAH基因干扰表达转基因水稻花粉活力的观察。A、B、C中的WT表示非转基因对照水稻,RNAi表示FAH干扰表达转基因水稻。
具体实施方式
实施例1水稻FAH基因cDNA的克隆及其序列分析
将水稻日本晴种子于37℃催芽1天后,播种于温室中,取三叶期水稻叶片提取RNA。RNA的提取采用大连TAKARA公司的RNAiso Plus试剂进行,具体操作步骤如下:①取100mg水稻叶片,液氮研磨成粉末后,加入1ml RNAiso Plus溶液,充分混匀后,室温放置5min,12000rpm/min离心5min,取上清液;②加入200ul氯仿于上清液中,充分混匀后,室温静置5min,12000rpm/min离心10min,取上清液;③加入500ul异丙醇,充分混匀后,室温静置10min,12000rpm/min离心15min,取沉淀;④沉淀用500ul 75%乙醇洗涤两次;⑤去乙醇,沉淀自然干燥后,用20ul双蒸水溶解后备用。RNA反转录成cDNA,采用YOYOBO公司的ReverTra Ace qPCR RT Master Mix with gDNA Remover试剂盒进行。具体操作如下:取1ug RNA和适量双蒸水混匀,总体积为12ul,65℃处理5min,冰上迅速冷却后,加入4ⅹDNase Mix 4ul,37℃处理5min。加入5ⅹRT Master Mix 4ul,37℃处理15min后,再于50℃处理5min,98℃处理5min,终止反应。
根据Genbank中水稻日本晴FAH(序列号AK070521.1)基因的序列,设计引物FAHF(5′-ATCTGCGCCTATTTATTGCGGCCTCT-3′,见SEQ ID No.1)和FAHR(5′-GACTTCAAATAAGTTCTTATTGTAACTGGAG-3′,见SEQ ID No.2)。以反转录获得的cDNA为模板,采用RT-PCR法克隆水稻日本晴的FAH基因。PCR反应体系为25ul,其中5×TransStart FastPfu Buffer 5ul、2.5mM dNTP 2.5ul、10mM引物FAHF 1ul、10mM引物FAHR 1ul、cDNA 10ng、TransStart FastPfu DNA Polymerase 0.5ul,补ddH2O至25ul;反应程序为:95℃预变性2min,95℃变性20S,62℃退火20S,72℃延伸1min20S,循环35次,72℃最后延伸10min。获得的FAH基因由华大基因公司测序,序列分析表明,克隆的FAH基因序列与Genbank中日本晴的FAH基因序列完全一致。说明克隆的基因为水稻日本晴的FAH基因。该水稻FAH基因的cDNA序列见SEQ ID No.3,其编码区包含1290个核苷酸(见SEQ ID No.4),编码一个长度为430个氨基酸的蛋白质。
实施例2 FAH基因在水稻不同组织中的表达分析
以孕穗期的水稻日本晴的根、茎、叶和幼穗为材料,分析FAH基因在水稻不同组织中的表达水平。
该孕穗期的水稻日本晴的根、茎、叶和幼穗的RNA的提取及反转录方法同实施例1。采用荧光定量PCR法分析FAH基因在不同组织中的表达水平。根据克隆的FAH基因序列,设计荧光定量PCR引物OSFAH1(5′-CGCCAGGAAACGCTCAAC-3′,见SEQ ID No.5)和OSFAH2(5′-GTCGCTCATGGGAACAAGG-3′,见SEQ ID No.6)。以OSFAH1和OSFAH2为引物,水稻各个组织的RNA反转录产物为模板,反应体系为20ul(模板400ng,OSFAH1和OSFAH2各0.8ul,美国ROCH公司的荧光定量试剂FastStart Universal SYBR Green Master(ROX)10ul,补双蒸水至总体积为20ul),以水稻OsUBQ5基因(Genbank序列号:AK061988)为内参,OsUBQ5基因的荧光定量PCR引物为:OsUBQ51(5′-ACCACTTCGACCGCCACTACT-3,见SEQ ID No.7);OsUBQ52(5′-ACGCCTAAGCCTGCTGGTT-3′,见SEQ ID No.8)。采用美国伯乐公司的CFX Connect Real-Time PCR System荧光定量PCR仪进行荧光定量PCR反应。反应程序为:95℃预变性10min;95℃变性15sec,60℃延伸1min,共40个循环。FAH在水稻不同组织中的表达水平见图1,实验结果表明,FAH基因在水稻幼穗中的表达水平最高,说明该基因在水稻幼穗的发育中发挥着重要的功能。
实施例3 FAH基因干扰表达转基因水稻的鉴定
为进一步鉴定FAH基因在水稻幼穗发育中的功能,本发明构建了FAH基因的干扰表达载体,并转化水稻日本晴,观察FAH干扰表达转基因水稻的育性。具体步骤如下:
A.根据已克隆的水稻FAH基因的序列(如SEQ ID No.3所示),采用Premier 5软件设计特异性引物P1(CAGCTTGTTGCAAGGGT,见SEQ ID No.9)和P2(GACTTCAAATAAGTTCTTATTGTAAC,见SEQ ID No.10),以已克隆的FAH基因为模板,采用高保真酶TransStart FastPfu DNA Polymerase(TRNAsGen)PCR扩增FAH基因序列,PCR反应体系为25ul,其中5×TransStart FastPfu Buffer 5ul,2.5mM dNTP 2.5ul,10mM引物P1 1ul,10mM引物P2 1ul,水稻FAH基因cDNA 10ng,TransStart FastPfu DNA Polymerase 0.5ul,补ddH2O至25ul;反应程序为:95℃预变性2min,95℃变性20S,54℃退火20S,72℃延伸20S,循环35次,72℃最后延伸10min。纯化PCR扩增产物。载体ptENTRA经XcmI(大连宝生物)酶切,酶切体系为20μl,其中10x buffer M 2ul、ptENTRA 0.5μg、XcmI 1ul(1U/1ul),补ddH2O至20ul,37℃酶切过夜。酶切产物纯化后,采用T4 DNA Ligase(Fermentas)连接扩增的水稻FAH基因和载体ptENTRA。FAH基因与ptENTRA载体按照3:1的分子比例混合,连接体系20ul,其中10xT4 DNA Ligase Buffer 2ul、T4 DNA Ligase 1ul,补ddH2O至20ul,16℃连接过夜。采用热激法转化大肠杆菌DH5α,在含Kan 50ug/ml的LB的培养基上筛选转化子,通过菌落PCR和测序进一步验证转化子,从而获得FAH基因与载体ptENTRA重组的载体。将该载体用Invitrogen(上海)公司的LR CLONASE ENZYME MIX同源重组酶和植物干扰表达载体pANDA进行同源重组。反应体系为:含FAH基因的ptENTRA载体150ng、干扰表达载体pANDA150ng,加入TE缓冲液至总体积为8ul,加入2ul LR CLONASE ENZYME MIX后,25℃处理过夜。再加入1ul蛋白酶K于反应体系中,37℃处理10min,终止反应。取该反应产物1ul转化大肠杆菌DH5α,在含Kan 50ug/ml和潮霉素50ug/ml的LB培养基上筛选转化子,FAH基因干扰表达载体经酶切和测序进行进一步鉴定。
B.将构建好的FAH基因干扰表达载体采用电激转化法转化农杆菌GV3101,在含50ug/ml Kan、50ug/ml庆大霉素、20ug/ml利福平和50ug/ml潮霉素的YEB培养基上筛选耐性转化子。
C.FAH干扰表达载体转化水稻日本晴:将含FAH基因干扰表达载体的农杆菌送武汉伯远生物科技有限公司进行水稻的转化,转化的水稻品种为日本晴。
D.FAH基因干扰表达转基因水稻的鉴定:
取转基因水稻幼叶,采用CTAB法提取DNA。具体操作如下:取100mg水稻叶片,液氮研磨成粉末后,加入600ul 2%CTAB溶液(由100mmol/l pH 8.0 Tris-HCl、20mmol/l EDTA、1.4mol/l NaCl及2%CTAB组成),充分混匀后,65℃水浴30min;待材料冷却至室温后,加入600ul氯仿,充分混匀后,10000rpm/min离心10min;取上清液加入600ul预冷的异丙醇,混匀后,10000rpm/min离心10min,取沉淀。沉淀用600ul 75%的乙醇洗涤2次,去乙醇,自然干燥备用。用30ul双蒸水溶解提取的DNA,加入1ul RNA酶,37℃处理30min,处理的DNA溶液用于后续的PCR实验。
根据干扰表达载体pANDA的序列以及FAH基因的序列,设计引物GUSlinker(CCGAATACGGCGTGGAT,见SEQ ID No.11)和P2(GACTTCAAATAAGTTCTTATTGTAAC,见SEQ ID No.10)。以GUSlinker和P2为引物,前述的DNA溶液为模板,PCR鉴定转基因植株。PCR反应体系为25ul,其中5×TransStart FastPfu Buffer 5ul、2.5mM dNTP 2.5ul、10mM引物GUSlinker 1ul、10mM引物P2 1ul、转基因水稻DNA 10ng、TransStart FastPfu DNA Polymerase 0.5ul,补ddH2O至25ul。反应程序为:95℃预变性2min,95℃变性20S,54℃退火20S,72℃延伸1min,循环35次,72℃最后延伸10min。PCR结果见图2A。图2A显示,非转基因对照材料中未扩增到任何带,而转FAH基因干扰表达载体的水稻植株中扩增出预期大小的目的带,表明FAH基因的干扰载体已经转入水稻中。同时提取转基因材料叶片中的RNA,采用荧光定量PCR检测转基因水稻中FAH基因的表达水平。RNA提取及其反转录以及荧光定量PCR的操作同实施例1。实验结果表明,转FAH基因干扰表达载体的水稻中FAH基因的表达低于非转基因材料中FAH的表达水平,说明FAH干扰表达载体的导入干扰了日本晴中FAH基因的表达,使该基因的表达水平降低,见图2B。
实施例4 FAH干扰表达转基因水稻的育性观察
水稻花粉活力实验采用碘-碘化钾染色法。具体操作如下,取同时期的转基因水稻材料及其对照的成熟花药置于载玻片上,用镊子将其捣碎,使花粉粒释放出来,再加入1滴1%的碘-碘化钾溶液进行染色,染色后置于显微镜下观察。染成蓝色的花粉粒是具有活力的花粉粒,染成黄褐色的花粉粒为发育不良活性低的花粉粒。观察转基因材料及其对照的花粉活力以及育性,结果见图3,显示,非转基因对照水稻籽粒饱满,而转FAH基因干扰表达载体的水稻籽粒为秕谷(图3B)。进一步鉴定转基因水稻植株的花粉粒的活力,发现其花粉粒被染成黄褐色,而非转基因对照水稻的花粉粒除少数几个外,其余的被染成蓝色(图3C)。这些结果表明转基因水稻材料的花粉活力和育性都下降,而花粉无活力则造成转基因水稻材料的不育。因此,通过干扰水稻中FAH的表达,可以影响水稻花粉的活力,从而获得水稻雄性不育植株。
综上所述,经过这些实验,发明人证明了水稻FAH基因在植物育性方面有着重要的功能。通过将该基因与植物干扰表达载体连接后,转化植物,干扰转化植物内FAH同源基因的表达,能获得育性降低的植物,为培育植物不育系奠定了理论和生产实践基础。
参考文献:
1.de Groot P,Weterings K,de Been M,Wittink F,Hulzink R,Custers J,van Herpen M,Wullems G(2004)Silencing of the pollen-specific gene NTP303and its family members in tobacco affects in vivo pollen tube growth and results in male plants.Plant Mol Biol 55:715-726。
2.Chen RZ,Zhao X,Shao Z,Wei Z,Wang YY,Zhu LL,Zhao J,Sun MX,RF He,GC He(2007)Rice UDP-Glucose pyrophosphorylase1is essential for pollen callose deposition and its cosuppression results in a new type of thermosensitive genic male sterility.Plant Cell 19:847-861。
3.Shi X,Sun X,Zhang Z,Feng D,Zhang Q,Han L,Wu J,Lu T(2015)GLUCAN SYNTHASE-LIKE 5(GSL5)plays an essential role in male fertility by regulating callose metabolism during microsporogenesis in rice.Plant Cell Physiol 56:497-509。
4.Han CY,Ren CM,Zhi TT,Zhou Z,Liu Y,Chen F,Peng W,Xie DX(2013)Disruption of fumarylacetoacetate hydrolase causes spontaneous cell death under short-day conditions in Arabidopsis.Plant Physiol 162:1956-1964。
SEQUENCE LISTING
<110> 湖南农业大学
<120> 水稻FAH基因在水稻育性控制中的应用
<130> 011
<160> 11
<170> PatentIn version 3.3
<210> 1
<211> 26
<212> DNA
<213> 合成
<400> 1
atctgcgcct atttattgcg gcctct 26
<210> 2
<211> 31
<212> DNA
<213> 合成
<400> 2
gacttcaaat aagttcttat tgtaactgga g 31
<210> 3
<211> 1618
<212> DNA
<213> Oryza sativa
<400> 3
atctgcgcct atttattgcg gcctcttctt cctcctctcc tcgcctcgcc tcgctcttcc 60
actgttcatc tgcgccacga cacgacacga cacgacacga gacgagagag gagtcggcaa 120
tggcggaggg gtcgcggtcg ccgctgaggt cgttcgtgga ggtggcgccg gggtcgcact 180
tccccatcca gaacctcccc ttcggggtct tccgccgcag gggctcgccg gagccggagc 240
cgccgcgccc ggccgtcgcc atcggggact tcgcgctcga cctcgccgcc gtctccgacg 300
cgggcctctt ccacgggccc ctcctctccg cctccccctg cttccgccag gaaacgctca 360
acatgttcct ggggatgggg cgtccggcct ggaaggaggc gcgcgccacg ctgcagaaga 420
tcctctcagc tgatgagccg gttttgcgtg acaacgaggc cttgaagaag aagtgccttg 480
ttcccatgag cgacacagag atgcttctgc caatcacggt aggagactac accgacttct 540
tttgttctgt gcaccatgca aggaattgtg ggttcatctt ccgtgggcca cagaccccag 600
tcaacccaaa ttggtttcag cttccagtag gttatcatgg tcgtgcatcc tctgtgattg 660
tatctggaac agacatcatt cgacccaaag ggcaaggcca tccaacagga gattctcgac 720
cttattttgg tccttctaag aagcttgatt ttgaacttga gatggcagcc attgttggac 780
cagggaatga attgggaaaa cctatcgata ttaatgatgc tgaagaacat atctttggcc 840
taatgataat gaatgattgg agtgccagag atatccaagc ttgggaaact atacctcttg 900
gacctttcct tgggaaaagt ttcagtacca cagtatcacc ctggatcgtt actatggatg 960
ccctaaagcc tttcacctgt gaagctccta agcaggaacc tgaacctttg ccttacttag 1020
ctgaaaagaa ccacgtaaac tacgatattc ctcttgaggt ctggattaag cccaaggagc 1080
aaagtgaacc atcaatggtt gcaaagagta acttcaagca tctgtattgg actttaacac 1140
agcaactagc acaccacact gttaatggat gcaatctgag accaggggat atgtttgcaa 1200
ctggcacact aagcggacct gagacagaat ctttgggatg tttgctggag ctaacatgga 1260
atgggcagaa ggagatatca gttggaaact cgacccgcaa gttcctagaa gatggggatg 1320
aggtcatctt gacagcttgt tgcaagggtg aaggctacaa tgtcggattc ggaacctgca 1380
ctgggaaggt tctgcctgca cttccatgat ccaacatata gaggatagcc tgctttcatc 1440
tcatcagtgt gtcaataatc tctgaagaaa gcatgcaatg aggcaatgta tctagctagt 1500
tacatctata ttttagtgct tagtgatggt aaaaatacta ataaggtatg cttccctgag 1560
ctttacagaa gggatgtaat taatgttctc cagttacaat aagaacttat ttgaagtc 1618
<210> 4
<211> 1290
<212> DNA
<213> Oryza sativa
<400> 4
atggcggagg ggtcgcggtc gccgctgagg tcgttcgtgg aggtggcgcc ggggtcgcac 60
ttccccatcc agaacctccc cttcggggtc ttccgccgca ggggctcgcc ggagccggag 120
ccgccgcgcc cggccgtcgc catcggggac ttcgcgctcg acctcgccgc cgtctccgac 180
gcgggcctct tccacgggcc cctcctctcc gcctccccct gcttccgcca ggaaacgctc 240
aacatgttcc tggggatggg gcgtccggcc tggaaggagg cgcgcgccac gctgcagaag 300
atcctctcag ctgatgagcc ggttttgcgt gacaacgagg ccttgaagaa gaagtgcctt 360
gttcccatga gcgacacaga gatgcttctg ccaatcacgg taggagacta caccgacttc 420
ttttgttctg tgcaccatgc aaggaattgt gggttcatct tccgtgggcc acagacccca 480
gtcaacccaa attggtttca gcttccagta ggttatcatg gtcgtgcatc ctctgtgatt 540
gtatctggaa cagacatcat tcgacccaaa gggcaaggcc atccaacagg agattctcga 600
ccttattttg gtccttctaa gaagcttgat tttgaacttg agatggcagc cattgttgga 660
ccagggaatg aattgggaaa acctatcgat attaatgatg ctgaagaaca tatctttggc 720
ctaatgataa tgaatgattg gagtgccaga gatatccaag cttgggaaac tatacctctt 780
ggacctttcc ttgggaaaag tttcagtacc acagtatcac cctggatcgt tactatggat 840
gccctaaagc ctttcacctg tgaagctcct aagcaggaac ctgaaccttt gccttactta 900
gctgaaaaga accacgtaaa ctacgatatt cctcttgagg tctggattaa gcccaaggag 960
caaagtgaac catcaatggt tgcaaagagt aacttcaagc atctgtattg gactttaaca 1020
cagcaactag cacaccacac tgttaatgga tgcaatctga gaccagggga tatgtttgca 1080
actggcacac taagcggacc tgagacagaa tctttgggat gtttgctgga gctaacatgg 1140
aatgggcaga aggagatatc agttggaaac tcgacccgca agttcctaga agatggggat 1200
gaggtcatct tgacagcttg ttgcaagggt gaaggctaca atgtcggatt cggaacctgc 1260
actgggaagg ttctgcctgc acttccatga 1290
<210> 5
<211> 18
<212> DNA
<213> 合成
<400> 5
cgccaggaaa cgctcaac 18
<210> 6
<211> 19
<212> DNA
<213> 合成
<400> 6
gtcgctcatg ggaacaagg 19
<210> 7
<211> 21
<212> DNA
<213> 合成
<400> 7
accacttcga ccgccactac t 21
<210> 8
<211> 19
<212> DNA
<213> 合成
<400> 8
acgcctaagc ctgctggtt 19
<210> 9
<211> 17
<212> DNA
<213> 合成
<400> 9
cagcttgttg caagggt 17
<210> 10
<211> 26
<212> DNA
<213> 合成
<400> 10
gacttcaaat aagttcttat tgtaac 26
<210> 11
<211> 17
<212> DNA
<213> 合成
<400> 11
ccgaatacgg cgtggat 17
- 还没有人留言评论。精彩留言会获得点赞!