一种比较断奶仔猪抗应激生理反应能力的标记引物和方法与流程
本发明属于分子生物技术领域,涉及一种断奶仔猪抗应激生理反应能力的标记引物和方法。
背景技术:
仔猪早期断奶导致早期断奶综合症,采食量下降,体重减轻甚至停滞不长,免疫力下降,肠道结构和功能遭到破坏,容易发生腹泻和水肿,重度的会致死。早期断奶综合征导致的仔猪生长受阻,给养猪业带来巨大的经济损失。仔猪断奶相关研究一直是热点,Dunsford等(1989)研究发现,21日龄断奶仔猪在断奶后第3天肠绒毛高度显著下降,这种显著变化持续到断奶后第12天。顾宪红等(2001)研究结果表明断奶导致仔猪肠绒毛高度缩短,隐窝加深。断奶导致的仔猪肠道结构这种变化,造成肠道消化吸收面积减小,导致消化能力下降、养分吸收减少。因此,在断奶后具有更高的肠道发育状况的断奶仔猪生长状况会更加优良,也就是说具有更强的抗应激生理反应能力。通过在断奶仔猪日粮中添加仔猪所需的营养物质从而减轻断奶应激的相关研究已有很多。李垚等(2005)的研究表明,在21日龄断奶仔猪日粮中添加表皮生长因子可以提高促肾上腺皮质激素、皮质醇、胰岛素水平,促进肌肉蛋白质的合成与周转,增加蛋白质、脂肪、糖原的合成,刺激细胞的增殖与分化,有效地缓解断奶应激。杨彩梅等(2005)的研究表明,日粮中添加谷氨酰胺能促进断奶初期仔猪的采食和生长,能提高断奶仔猪小肠食糜中脂肪酶、淀粉酶和糜蛋白酶的活力。从现有研究中可以了解到,当断奶仔猪猪血清谷氨酰胺酶GA、皮质醇Cor含量越高、小肠绒毛高度约高对营养物质的吸收利用能力就越强,断奶仔猪的抗应激生理反应能力也就越强。GA是谷氨酰胺分解的关键酶和限速酶,仔猪断奶后,母源谷氨酰胺消失,小肠营养物质缺乏,体内谷氨酰胺水解代谢加强以缓断奶应激。仔猪断奶后血清Cor浓度会升高,促进仔猪体内合成代谢,使仔猪抗应激能力加强。Cor是介导动物体内应激反过程的一种重要中间介质,血清Cor浓度的高低能灵敏地反映机体遭受创伤程的强弱,因此可以把GA、Cor看作是判定动物遭受应激程度的一个敏感而有效指标。寻找到高血清GA、Cor含量和小肠绒毛高度的断奶仔猪,对提高断奶仔猪抗应激生理反应能力,减少断奶仔猪因断奶而引起的生长状况下降,提高效益减少养殖成本具有重要的意义。
已有研究表明不同猪种抗应激能力存在一定差异,且中国地方猪种抗应激能力普遍较外来猪种强。现有的研究表明,血清生化指标、肠黏膜形态是研究断奶仔猪断奶应激的重要指标,而现有的研究对地方猪种和外来猪种断奶应激差异性的报道较少。尤其是奶应激期,中外猪种仔猪的抗应激能力及其遗传机理相关研究目前未见报道。
技术实现要素:
本发明的目的在于以安徽地方优良品种安庆六白猪和国外引进品种长白猪为研究对象,研究两个猪种在断奶应激期血清生化指标变化、肠黏膜形态变化。探讨品种间断奶应激期仔猪血清指标的差异、肠道结构发育和功能的差异、相关候选基因表达的差异以及基因表达与各抗应激生化指标的相关性,旨在找出安徽地方优良品种安庆六白猪候选基因与肠道发育的内在联系,筛选影响仔猪抗断奶应激能力及肠道发育的相关候选基因,为今后品种选育和新品系的建立提供参考。
1.本发明提供一种比较断奶仔猪血清GA、Cor含量以及小肠绒毛高度差异的标记引物,该引物对包括GLS、EGFR、IGF-1、IGF-1R;其中,
引物对GLS,正向引物序列如SEQ ID NO.1所示,反向引物序列如SEQ ID NO.2所示;
引物对EGFR,正向引物序列如SEQ ID NO.3所示,反向引物序列如SEQ ID NO.4所示;
引物对IGF-1,正向引物序列如SEQ ID NO.5所示,反向引物序列如SEQ ID NO.6所示;
引物对IGF-1R,正向引物序列如SEQ ID NO.7所示,反向引物序列如SEQ ID NO.8所示;
2.本发明还提供一种比较断奶仔猪血清GA、Cor含量以及小肠绒毛高度差异的方法,该方法分别以与血清GA、Cor含量呈正相关的候选基因GLS和EGFR的表达水平比较不同断奶仔猪个体血清GA、Cor含量差异,与小肠绒毛高度呈正相关的候选基因IGF-1、IGF-R的表达水平比较不同断奶仔猪个体肠绒毛高度差异。
3.本发明还提供一种比较断奶仔猪肠道发育状况的方法,该方法以与断奶仔猪肠道生长发育相关联的血清GA、Cor含量、仔猪小肠绒毛高度为对象,通过分析与血清GA、Cor含量呈正相关的候选基因GLS和EGFR的表达水平,与小肠绒毛高度呈正相关的候选基因IGF-1、IGF-R的表达水平,判定断奶仔猪肠道发育状况,标记基因表达水平高的断奶仔猪比标记基因表达水平低的断奶仔猪肠道发育更良好。
4.上述3提供的比较断奶仔猪肠道发育状况的方法,具体包括如下步骤:(1)断奶仔猪小肠总RNA提取;(2)引物设计:分别设计候选基因GLS的反转录引物对GLS、候选基因EGFR的反转录引物对EGFR、候选基因IGF-1的反转录引物对IGF-1、候选基因IGF-1R的反转录引物对IGF-1R;(3)反转录合成cDNA:使用反转录试剂盒以步骤(1)得到的总RNA进行反转录合成cDNA;(4)采用步骤(2)通过q-RT-PCR方法测定候选基因GLS、EGFR、IGF-1、IGF-1R在断奶仔猪个体小肠中的表达量;(5)根据各候选基因在不同断奶仔猪个体的表达量的差异确定不同断奶仔猪肠道发育状况;
其中,引物对GLS,正向引物序列如SEQ ID NO.1所示,反向引物序列如SEQ ID NO.2所示;
引物对EGFR,正向引物序列如SEQ ID NO.3所示,反向引物序列如SEQ ID NO.4所示;
引物对IGF-1,正向引物序列如SEQ ID NO.5所示,反向引物序列如SEQ ID NO.6所示;
引物对IGF-1R,正向引物序列如SEQ ID NO.7所示,反向引物序列如SEQ ID NO.8所示。
5.上述4提供的比较断奶仔猪肠道发育状况的方法,其中,步骤(4)所述q-RT-PCR方法具体包括如下步骤:
(1)标准品制备及条件优化:每个待测RNA的反转录产物取等量混合,将cDNA混合样按梯度稀释,用RT-PCR的反应体系,对每个目的基因作标准曲线来优化整个反应条件;
(2)q-RT-PCR标准曲线的建立和扩增曲线:采用RT-PCR方法作目的基因GLS、EGFR、IGF-1、IGF-1R的标准曲线,得到各个目的基因及内参β-Actin基因的标准曲线,对结果相关数据进行分析,当标准曲线的相关系数及扩增效率均等于或接近于1.000时,表明实验数据误差较小,可信度较高,实验所得的Ct值可准确地测定起始cDNA的拷贝数;目的基因和看家基因标准曲线的斜率小于0.1,表明两个基因的标准曲线的斜率基本一致,在后续实验中可以使用分析Comparative Delta-delta Ct法进行相对定量分析;
(3)相关候选基因的q-RT-PCR反应:采用RT-PCR法检测猪GLS、EGFR、IGF-1、IGF-1R、FUT1、LYZ、TAP1、SLA-DQB、TLR4基因及内参基因β-Actin在断奶仔猪小肠各段的表达量;
(4)数据分析:目的基因mRNA表达丰度采用2-ΔΔCT法对有效数据进行统计分析,计算出每个样本目的基因的相对表达水平,通过内参基因β-Actin校正;
其中,内参基因β-Actin的扩增引物对β-antin上游序列如SEQ ID NO.9所示,下游引物序列如SEQ ID NO.10所示。
6.上述5提供的比较断奶仔猪肠道发育状况的方法,其中,步骤(4)q-RT-PCR反应体系为20μL,包括:SYBR绿色荧光染料10μL,上游引物1μL,下游引物1μL,cDNA1μL,超纯水7μL;反应条件为:95℃预变性30s;95℃变性10s,退火温度下退火30s,40个循环,于65℃5s至95℃5s时搜集荧光值;其中,引物对GLS的退火温度为58.8℃;引物对EGFR的退火温度为60℃;引物对IGF-1和IGF-1R的退火温度为61℃。
7.上述4提供的比较断奶仔猪肠道发育状况的方法,其中,步骤(3)反转录合成cDNA具体包括如下步骤:(1)基因组DNA除去反应:体系包括:5×gDNA Eraser缓冲液2μL,gDNA Eraser1μL,总RNA1μg,加超纯水至10μL;反应条件为:42℃反应2min,4℃保存,得反应液I;(2)反转录反应:体系包括:反应液I 10μL,5×PrimeBuffer2 4.0μL,PrimeRT酶混合液I 1.0μL,RT Primer Mix 1.0μL,加超纯水至20μL;反应条件为:使用普通PCR仪设置程序37℃反应15min,85℃反应5s,4℃保存,得反转录样品cDNA,检测后置于-20℃条件下备用。
8.上述3-7任一项提供的比较断奶仔猪肠道发育状况的方法,其中,所述断奶仔猪品种为安徽地方品种安庆六白猪。
9.上述3-7任一项提供的方法在断奶仔猪抗应激生理反应能力比较中的应用,其中,该方法比较GLS、EGFR和IGF-1、IGF-R的表达水平,基因表达水平越高,说明与基因表达正相关的GA、Cor和仔猪小肠绒毛高度水平越高,仔猪断奶抗应激能力越强。
10.上述9提供的方法在断奶仔猪抗应激生理反应能力比较中的应用,其中,所述断奶仔猪品种为安徽地方品种安庆六白猪
本申请提供的“比较”概念是断奶仔猪个体间的比较,“高低”、“强弱”均是指个体间的比较,根据GLS、EGFR和IGF-1、IGF-R的表达水平,确定不同个体间生化指标血清GA、Cor含量的高低以及小肠绒毛高度的大小,从而比较断奶仔猪不同个体间肠道发育以及抗应激生理反应能力大小。
本发明具有如下优点:
1.本申请获得了断奶仔猪尤其是安徽地方品种安庆六白猪断奶仔猪肠道发育与候选基因表达差异之间的关系,填补了现有技术的空白,由此得出断奶仔猪抗应激生理反应能力与候选基因表达差异之间的关系,为今后品种选育和新品系的建立提供参考。
2.本发明以仔猪抗应激生化指标血清GA、Cor含量以及小肠绒毛高度为参照,在大量基因中寻找到与血清GA、Cor含量以及小肠绒毛高度成正相关的候选基因,从而根据候选基因表达情况可以快速获得断奶仔猪尤其是安徽地方品种安庆六白猪断奶仔猪的肠道发育以及对断奶抗应激能力的高低,减少选育过程中的复杂性,避免断奶仔猪因为受到刺激等因素导致生化指标急剧变化而与实际不符,保证了结果的准确性。
附图说明
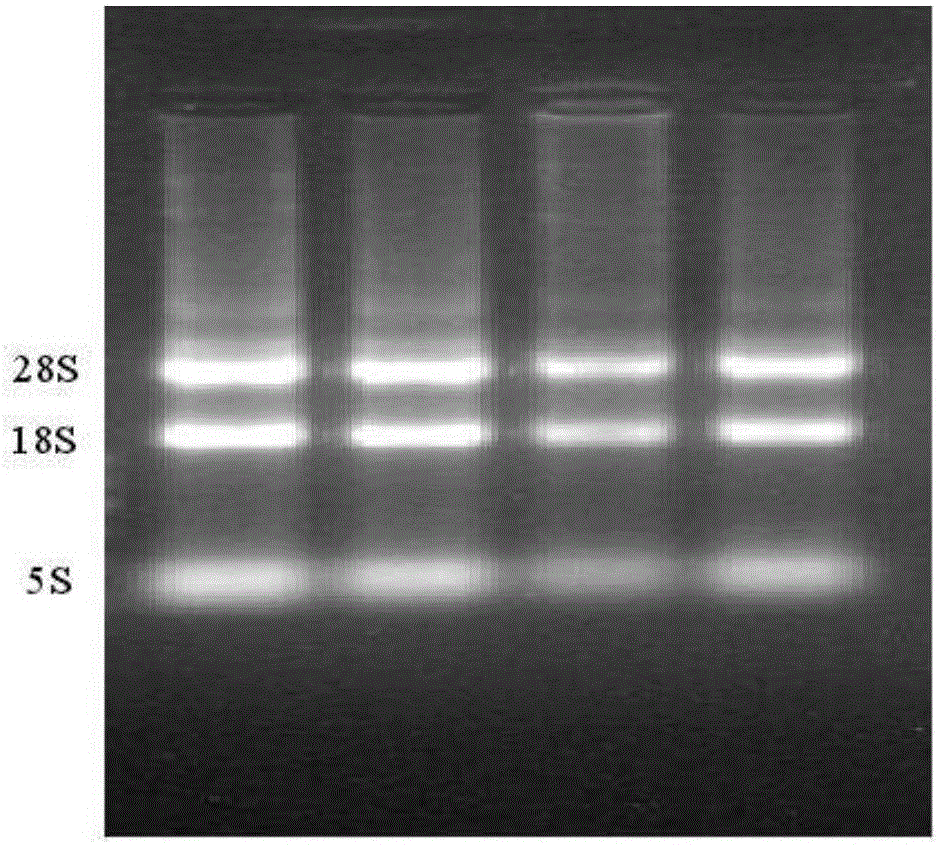
图1部分RNA电泳图
图2部分cDNA电泳图;其中,β-Actin条带114bp
图3 GLS基因RT-PCR曲线;其中,A图为标准曲线;B图为扩增曲线;C图为溶解曲线
图4 IGF-1基因RT-PCR曲线;其中,A图为标准曲线;B图为扩增曲线;C图为溶解曲线
图5 IGF-1R基因RT-PCR曲线;其中,A图为标准曲线;B图为扩增曲线;C图为溶解曲线
图6 EGFR基因RT-PCR曲线;其中,A图为标准曲线;B图为扩增曲线;C图为溶解曲线
图7 33日龄和39日龄安庆六白猪和长白猪各肠段黏膜GLS基因表达情况
图8 33日龄和39日龄安庆六白猪和长白猪各肠段黏膜IGF-1基因表达情况
图9 33日龄和39日龄安庆六白猪和长白猪各肠段黏膜IGF-1R基因表达情况
图10 33日龄和39日龄安庆六白猪和长白猪各肠段黏膜EGFR基因表达情况
其中,图7-10中Δ表示不同品种同一日龄仔猪同一肠段差异显著(P<0.05),ΔΔ表示不同品种同一日龄仔猪同一肠段差异极显著(P<0.01);*表示同一品种不同日龄仔猪同一肠段差异显著(P<0.05),**表示同一品种不同日龄仔猪同一肠段差异极显著(P<0.01);不同小写字母表示单一品种单一日龄仔猪各肠段间差异显著(P<0.05),不同大写字母表示单一
品种单一日龄仔猪各肠段间差异极显著(P<0.01)。A:安庆六白猪,L:长白猪。
具体实施方式
下面结合具体实施例对本发明进行具体阐述。
实施例1:
一、试验方法
1.1试验动物及样品采集
从安徽农业大学试验基地选择健康状态良好的长白猪和安庆六白猪28日龄断奶仔猪各20头,分别于33日龄、39日龄各取10头屠宰。前腔静脉采血,制备血清,置-20℃冰箱保存,用于血清生化指标检测。取小肠十二指肠、空肠、回肠各肠段8cm,迅速放入备有4%多聚甲醛的固定液中,待做组织切片;另从小肠各段截取8cm,用1%PBS溶液将肠段彻底冲洗干净,刮取肠黏膜无菌的冻存管中,并迅速放入液氮中保存,样品放入-80℃条件下保存至使用;另从小肠各段截取8cm,用1%PBS溶液将肠段彻底冲洗干净,并迅速放入液氮中保存,样品放入-80℃条件下保存至使用,待做荧光定量检测。
试验所需仪器和试剂如果没有特别说明,均属于本领域普通技术人员通过市场可以购买获得的。
1.2血清指标测定
从仔猪前腔静脉采血,4度冰箱静置一夜后,3000r/min离心15min分离制备血清。采用武汉新启迪公司的相应ELISA试剂盒,采用双抗体夹心酶联免疫法测定血清GH、GA、Cor、CD3、CD4、T4、T3。
1.3肠黏膜二糖酶、ATP酶活性含量测定
取0.2g各肠段黏膜放于1mL 1%的PBS缓冲液中研磨,制备匀浆液。采用南京建成公司的相应试剂盒测定乳糖酶、麦芽糖酶、蔗糖酶、ATP酶活性,方法和操作按说明书进行,检测每毫克蛋白质中的二糖酶及ATP酶活力。
1.4切片制作及观察
1.4.1切片制作
小肠各段固定16小时到24小时后,取出组织,经水洗、透明、浸蜡、包埋等处理后,制备连续横断切片,厚度为5μm,每隔10张切片取1张切片。
1.4.2HE染色
取5张切片,切片脱蜡、苏木精-伊红、分色、脱水、透明、封片。每张切片观察5个典型视野,用目镜测量测微尺测量5根最长的绒毛长度(以肠腺绒毛连接处到绒毛顶端为准,最深的隐窝深度(以肠腺绒毛连接处到肠腺基部为准)。每项指标均测定25组数据。
1.5实时荧光定量RT-PCR测定各个基因的表达水平
1.5.1十二指肠、空肠、回肠中总RNA提取
按Total RNA Kit II(50)(Omega)说明书提取总RNA,步骤如下:
(1)1.5mL无RNA酶离心管中加入1mL RNA-SOW Reagent裂解液,取约100mg的十二指肠组织(-80℃冰箱保存)于研钵中,边加液氮边研磨至粉末状,迅速转入含裂解液的1.5mL无RNA酶离心管中,充分震荡摇匀于室温下静置2~3min。
(2)静置后向里加入0.2mL氯仿,摇匀,剧烈振荡15-20s,随后放在冰上孵化10min。
(3)4℃,12000rmp离心15min后,混合物分离成3层,中间层和上层为水相RNA保存在上层水相中,下层为苯酚-氯仿相。
(4)将80%的水相转入新的EP管中,并向里加入三分之一体积的无水乙醇,最大速度漩涡震荡15s,接下来的步骤的离心操作均在室温条件下进行。
(5)将离心柱置于2mL收集管中,将步骤5中的液体转入离心柱中,10000rmp离心30~60s,弃流出液。
(6)将离心柱放入新的2mL收集管中,并加入300μl RNA Wash Buffer I,10000rmp离心30~60s,弃流出液。
(7)向离心柱中加入400μL RNA Wash Buffer II,10000rmp离心30~60s,弃流出液。
(8)将离心柱放入新的2mL收集管中,向里加入500μLRNA Wash Buffer II,10000rmp离心30~60s,弃流出液。
(9)重复步骤8,13000rmp离心2min,丢掉流出液,超净台内干燥离心柱5min。
(10)洗脱RNA,将离心柱转入1.5mL无RNA酶离心管中,向离心柱中加入300μLDEPC水,室温孵化2min,13000rmp离心1min,弃离心柱,保留流出液,即RNA,置于-80℃冰箱中保存。空肠、回肠中的总RNA提取同上述方法。
1.5.2RNA提取质量的检测
(1)使用紫外分光光度计测定RNA的OD260值、OD280值及浓度。OD260/OD280值在1.8-2.0之间的RNA为合格品。
(2)甲醛变性胶检测RNA质量。取RNA样5μL与1μL的5×Loading buffer混匀,置于70℃水浴锅内加热10min,后置于冰上骤冷以消除RNA的二级结构。用1.2%甲醛变性凝胶电泳检测RNA质量,电压为100v。凝胶成像系统下观察条带,通常可以看到3条带,一般为28s、18s、5s,其中28s、18s较亮,5s较暗,且28s的亮度是18s的两倍的RNA质量比较好(见图1),留下符合要求的RNA样。
(3)选择符合上述两个条件的RNA样稀释至1μg/μL分装于无RNA酶离心管内,于-80℃条件下保存以供反转录。
1.5.3组织提取的总RNA质量检测结果
提取安庆六白猪和长白猪十二指肠、空肠及回肠中的RNA并用甲醛变性胶进行电泳检测,电泳图中的18S、5S及28S三条条带清晰可见。因此,总RNA的质量符合实验要求,可进行反转录合成cDNA(图1)。
1.5.4引物设计与合成
根据GenBank中猪GLS、EGFR、IGF-1、IGF-1R的序列,设计引物序列,引物由上海生工生物公司合成。引物序列和PCR参数如表1所示。
表1.用于Real-time quantitative PCR的引物序列及PCR参数
1.5.5反转录合成cDNA
使用反转录试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser(TAKARA),反应体系及反应条件如下,见表2、3。
表2.反转录第一步基因组DNA除去反应
反应条件:使用普通PCR仪设置程序42℃反应2min,4℃保存,得反应液I。
表3.反转录反应
反应条件:使用普通PCR仪设置程序37℃反应15min,85℃反应5s,4℃保存。得反转录样品cDNA,检测后置于-20℃条件下备用。
1.5.6cDNA纯度检测
反应体系及反应条件如下,反应完成后取6μL PCR产物在1%琼脂糖凝胶上以120V电压电泳15min,凝胶成像系统下观察条带以鉴定cDNA纯度(β-Actin为例)。
表4.常规PCR反应
1.5.7cDNA纯度鉴定结果
将提取的总RNA按照相关反转录试剂盒说明合成cDNA。按照常规PCR反应体系添加反应物,引物为β-Actin基因,质粒为cDNA。采用琼脂糖凝胶电泳实验检测扩增的产物,在电泳图上可看到样品得到很好的β-Actin电泳条带。从电泳图片结果看,PCR扩增的产物条带清晰,且无杂带,说明该引物可特异性地扩增出目的产物。
1.5.8q-RT-PCR
(1)标准品制备及条件优化
每个待测RNA的反转录产物取等量混合,将cDNA混合样按梯度稀释,用RT-PCR的反应体系,对每个目的基因作标准曲线来优化整个反应条件。用标准曲线验证目的基因和内参基因的引物有效性、最佳退火温度、使用浓度及RT-PCR反应中的试剂,以评估每个目的基因和内参基因RT-PCR反应的最佳条件。RT-PCR反应体系及条件如下(表5),每个样品重复3次,并设阴性对照。
(2)q-RT-PCR标准曲线的建立和扩增曲线
采用q-RT-PCR方法作目的基因GLS、EGFR、IGF-1、IGF-1R的标准曲线,得到各个目的基因及β-Actin基因的标准曲线,对结果相关数据进行分析,当标准曲线的相关系数及扩增效率均等于或接近于1.000时,表明实验数据误差较小,可信度较高,实验所得的Ct值可准确地测定起始cDNA的拷贝数。目的基因和看家基因标准曲线的斜率小于0.1,表明两个基因的标准曲线的斜率基本一致,在后续实验中可以使用分析Comparative Delta-delta Ct法进行相对定量分析。根据熔解曲线确认扩增产物的特异性需要,当熔解曲线的特异性扩增产物只有一个峰值,且无非特异性峰存在,说明引物的特异性良好,可以在后续实验中使用。根据上述要求进行试验,从而得到所有样品扩增曲线,见图3-6。
(3)相关候选基因的q-RT-PCR反应
采用RT-PCR法检测猪GLS、EGFR、IGF-1、IGF-1R基因及内参基因β-Actin在断奶仔猪小肠各段的表达量。反应体系及反应条件如下(表5),反应体系为20μL。基因表达量分析:目的基因mRNA表达丰度采用2-ΔΔCT法对有效数据进行统计分析,计算出每个样本目的基因的相对表达水平,通过内参基因β-Actin校正。
表5.q-RT-PCR反应体系及反应条件
1.6数据分析
试验数据用平均数±标准差(mean±SD)表示,采用SPSS20.0软件对结果中的数据进行分析,品种间同一日龄的均值差异、品种内不同日龄的均值差异、品种内种不同日龄同一肠段的均值差异均采用独立样本T检验统计分析,单一品种单一日龄小肠各段的均值差异采用One-Way ANOVA统计分析,显著性差异水平为P<0.05,极显著性差异水平为P<0.01。采用SPSS20.0软件Bivariate correlation程序进行数据相关性分析。
(二)数据分析
2.1小肠黏膜GLS、IGF-1、IGF-1R、EGFRmRNA的相对表达水平
2.1.1GLS mRNA的相对表达水平
33日龄时,安庆六白猪空肠GLS mRNA表达水平显著高于长白猪(P<0.05),十二指肠和回肠GLS mRNA表达水平与长白猪差异不显著。39日龄时,安庆六白猪小肠各段GLS mRNA表达水平与长白猪差异不显著。同一猪种39日龄小肠各段GLS mRNA表达水平与33日龄比较发现,安庆六白猪十二指肠和回肠GLS mRNA表达水平均显著提高了(P<0.05);长白猪十二指肠和回肠GLS mRNA表达水平均极显著提高了(P<0.01),空肠GLS mRNA表达水平显著提高(P<0.05)。
33日龄和39日龄时,长白猪和安庆六白猪小肠各段GLS mRNA的表达水平依次均是空肠最高,十二指肠次之,回肠最低。33日龄时,安庆六白猪空肠GLS mRNA的表达水平极显著高于十二指肠和回肠(P<0.01),而十二指肠和回肠之间无显著差异;长白猪空肠GLS mRNA的表达水平显著高于回肠(P<0.05),空肠及回肠与十二指肠无显著差异。39日龄时,安庆六白猪空肠GLS mRNA的表达水平极显著高于十二指肠(P<0.01),显著高于回肠(P<0.05),而十二指肠和回肠之间无显著差异;长白猪小肠各段GLS mRNA的表达水平无显著差异(见图4)。
2.1.2IGF-1mRNA的相对表达水平
33日龄时,长白猪十二指肠、空肠IGF-1mRNA表达水平极显著、显著高于安庆六白猪(P<0.01,P<0.05),回肠IGF-1mRNA表达水平与安庆六白猪差异不显著。39日龄时,安庆六白猪小肠各段IGF-1mRNA表达水平与长白猪差异不显著。同一猪种39日龄小肠各段IGF-1mRNA表达水平与33日龄比较发现,安庆六白猪十二指肠IGF-1mRNA表达水平极显著提高了(P<0.05),空肠与回肠IGF-1mRNA表达水平趋势是升高了;长白猪小肠各段IGF-1mRNA表达水平变化不显著。
33日龄和39日龄时,两猪种小肠各段IGF-1 mRNA的表达水平依次均是空肠最高,十二指肠次之,回肠最低。33日龄时,安庆六白猪空肠IGF-1 mRNA的表达水平极显著高于十二指肠和回肠(P<0.01),十二指肠IGF-1 mRNA的表达水平显著高于回肠(P<0.05);长白猪十二指肠及空肠IGF-1mRNA的表达水平均极显著高于(P<0.01),空肠与十二指肠无显著差异。39日龄时,安庆六白猪和长白猪小肠各段IGF-1 mRNA的表达水平均无显著差异(见图5)。
2.1.3IGF-1R mRNA的相对表达水平
33日龄和39日龄时,长白猪小肠各段IGF-1R mRNA表达水平均表现出高于安庆六白猪的趋势,品种之间无显著差异。同一猪种39日龄断奶仔猪小肠各段IGF-1R mRNA表达水平与33日龄断奶仔猪比较发现,两猪种小肠各段IGF-1R mRNA表达水平均有所提高,变化不明显。
33日龄和39日龄时,两猪种小肠各段IGF-1R mRNA的表达水平依次均是空肠最高,十二指肠次之,回肠最低。33日龄时,安庆六白猪空肠IGF-1R mRNA的表达水平极显著高于回肠(P<0.01),十二指肠与空肠及回肠无显著差异;39日龄时,安庆六白猪小肠各段IGF-1R mRNA的表达水平差异不显著。33日龄和39日龄时,长白猪空肠IGF-1R mRNA的表达水平显著高于回肠(P<0.05),十二指肠与空肠及回肠无显著差异(见图6)。
2.1.4EGFR mRNA的相对表达水平
33日龄时,安庆六白猪十二指肠和空肠EGFR mRNA表达水平均显著高于长白猪(P<0.05),回肠EGFR mRNA表达水平与长白猪差异不显著。39日龄时,安庆六白猪小肠各段EGFR mRNA表达水平与长白猪差异不显著。同一猪种39日龄小肠各段EGFR mRNA表达水平与33日比较发现,安庆六白猪小肠各段EGFR mRNA表达水平变化不显著;长白猪十二指肠和空肠EGFR mRNA表达水平有所提高,回肠EGFR mRNA表达水平有所下降,变化均不显著。
33日龄和39日龄时,长白猪和安庆六白猪小肠各段EGFR mRNA的表达水平依次均是空肠最高,十二指肠次之,回肠最低。33日龄时,安庆六白猪回肠EGFR mRNA的表达水平极显著低于空肠(P<0.01),显著低于于十二指肠(P<0.05),十二指肠与空肠差异不显著,39日龄时,安庆六白猪十二指肠及空肠EGFR mRNA的表达水平显著高于回肠(P<0.05),而十二指肠和空肠之间无显著差异;33日龄和39日龄时,长白猪空肠EGFR mRNA的表达水平显著高于回肠(P<0.05),空肠及十二指肠与回肠无显著差异(见图7)。
2.2相关性分析结果
2.2.1小肠GLS及EGFR基因表达量与血清GA及Cor含量的相关性分析
GLS基因在33日龄和39日龄安庆六白猪空肠段表达量与血清GA含量显著正相关(P<0.05),在39日龄长白猪十二指肠段表达量与血清GA含量显著正相关(P<0.05)。EGFR基因在33日龄安庆六白猪十二指肠段表达量与血清GA含量显著正相关(P<0.05)(见表6)。
表6.GLS和EGFR mRNA表达量与血清GA含量的相关性
注:**表示极显著相关,*表示显著相关(表12、13、17、18同)
GLS基因在33日龄安庆六白猪十二指肠段及长白猪空肠段表达量与血清Cor含量均显著正相关(P<0.05)。EGFR基因在33日龄两猪种空肠段及39日龄安庆六白猪十二指段表达量与血清Cor含量均显著正相关(P<0.05)(见表7)。
表7.GLS和EGFR mRNA表达量与血清Cor含量的相关性
2.2.2小肠IGF-1及IGF-1R基因表达量与小肠绒毛高度的相关性分析
33日龄安庆六白猪空肠段和长白猪十二指肠段IGF-1基因表达量与肠绒毛高度显著正相关(P<0.05),39日龄安庆六白猪十二指肠IGF-1、IGF-1R基因表达量与肠绒毛高度显著正相关(P<0.05)。
表8.IGF-1和IGF-1R基因表达量与小肠绒毛高度的相关性
2.3结论
小肠生长发育相关基因GLS及EGFR基因在小肠各段的表达与仔猪抗应激生化指标GA、Cor达高度正相关;IGF-1及IGF-1R基因在小肠各段的表达与小肠绒毛高度显著正相关。在不同断奶仔猪个体间,GLS及EGFR基因表达水平高低可以用来比较血清GA、Cor含量的多少,IGF-1及IGF-1R基因表达水平可以用来比较小肠绒毛高度差异,从而推测出断奶仔猪小肠生长发育以及抗断奶应激生理反应的能力。
可以知道,上述实施例仅为了说明发明原理而采用的示例性实施方式,然而本发明不仅限于此,本领域技术人员在不脱离本发明实质情况下,可以做出各种改进和变更,这些改进和变更也属于本发明的保护范围。
<110>安徽农业大学
<120>一种比较断奶仔猪抗应激生理反应能力的标记引物和方法
<160>10
<210>1
<211>19
<212> DNA
<213>人工序列
<220>
<223>上游引物序列GLS -F
<400>1
AGGGCAGTTT GCTTTCCAT 19
<210>2
<211>20
<212> DNA
<213>人工序列
<220>
<223>下游引物序列GLS-R
<400>2
CTTGTCCAGA GGAGGAGACC 20
<210>3
<211> 20
<212> DNA
<213>人工序列
<220>
<223>上游引物序列EGFR -F
<400>3
GGCCTCCATG CTTTTGAGAA 20
<210>4
<211>19
<212> DNA
<213>人工序列
<220>
<223>下游引物序列EGFR -R
<400>4
GACGCTATGT CCAGGCCAA 19
<210>5
<211>19
<212> DNA
<213>人工序列
<220>
<223>上游引物序列IGF-1 -F
<400>5
CCTCAAGCCT GCCAAGTCG 19
<210>6
<211>19
<212> DNA
<213>人工序列
<220>
<223>下游引物序列IGF-1 -R
<400>6
GGTAACTCGT GCAGAGCAAA GG 22
<210>7
<211>22
<212> DNA
<213>人工序列
<220>
<223>上游引物序列IGF-1R -F
<400>7
CCACCCAACA CCTACCGCTT CG 22
<210>8
<211>22
<212> DNA
<213>人工序列
<220>
<223>下游引物序列IGF-1R -R
<400>8
GCACTCGCCA TCATGGATCA CA 22
<210>9
<211> 20
<212> DNA
<213>人工序列
<220>
<223>下游引物序列β-antin -F
<400>9
CTCGATCATG AAGTGCGACG 20
<210>10
<211> 23
<212> DNA
<213>人工序列
<220>
<223>下游引物序列β-antin -R
<400>10
GTGATCTCCT TCTGCATCCT GTC 23
- 还没有人留言评论。精彩留言会获得点赞!