利用piggyBac转座子在CHO细胞中获得表达目标蛋白的稳定细胞群的方法与流程
本发明涉及生物制药领域,特别是涉及一种在CHO(Chinese Hamster Ovary,中国仓鼠卵巢细胞)细胞中利用piggyBac转座子快速高效表达蛋白的方法。
背景技术:
DNA转座子是天然存在于动物细胞基因组中的重复序列。PiggyBac转座子最初发现于昆虫细胞中,核心原件包括转座酶和末端反向重复序列。PiggyBac转座子的基因序列以及末端反向重复序列已经公开,其在GENBANK中的编号为GU937109.1和ABS12111.1。PiggyBac转座系统能够通过“剪切和粘贴”的方式在基因组和质粒间转移DNA序列。与其他转座系统相比,piggyBac转座酶可以将两个末端反向重复序列以及二者之间的序列精确插入基因组核酸序列为TTAA的位置,且不造成原位置的碱基增加或缺失。随着对转座子机理研究的不断深入以及在哺乳动物细胞中应用的逐渐成熟,转座子被越来越多的用于干细胞诱导,基因转移等动物细胞工程领域中。
快速高效的蛋白表达在科研和工业领域有着广泛的需求。瞬时转染技术可以在两周内表达近克级别蛋白,然而其表达量通常远低于稳定细胞群,而且其质粒游离于基因组外的特点又限制了工作体积的放大,所以瞬时转染较难满足较大规模的产品需求。传统的稳定细胞群构建过程中,含有目标蛋白的DNA序列随机插入基因组中。此整合概率通常低于万分之一,因此造成转染后细胞群在抗生素筛选阶段恢复缓慢。虽然传统的稳定细胞群表达量较高,然而其长达数月之久的构建时间也很难适应如今行业发展的需求。
现有技术中,也有报道利用其他转座子构建稳定细胞表达蛋白的方法,但从实验结果看来,现有技术中采用Tol2转座子的方法一般都需要一个月以上的时间才能构建获得稳定细胞群,将该方法用于生产目标蛋白仍然存在生产效率低下的缺陷。其他转座子,如Sleeping Beauty,Tol2和Mos1在CHO细胞中的转座效率均不及piggyBac。因此,本领域亟需一种利用转座子系统快速高效表达蛋白的方法。
技术实现要素:
在生物技术领域,尤其是制药研发早起,对克级别蛋白的需求与日俱增。瞬时转染技术虽然表达周期短,但较低的表达量导致量产蛋白的难度较大。一般的稳定细胞群虽然表达量较高,然而其长达数月的构建周期难以跟上快速发展的生物制药领域的需求。
本发明所要解决的技术问题在于,提供一种数周内完成构建高表达目标蛋白的哺乳动物细胞群,并可直接用于放大生产目标蛋白的方法,该方法是利用piggyBac转座子在CHO细胞中快速高效表达蛋白。
为解决上述技术问题,本发明的利用piggyBac转座子在CHO细胞中获得快速高效表达目标蛋白的稳定细胞群的方法,包括以下步骤:
1)构建含有编码目标蛋白基因和抗生素筛选基因的基因片段的质粒,构建含有piggyBac转座子序列的质粒;
2)将步骤1)中构建的质粒通过转染同时引入CHO细胞中;
3)对步骤2)中转染后细胞直接在摇瓶中进行抗生素筛选;
4)将步骤3)筛选后的细胞每隔2~4天通过批次补料的方法生产目标蛋白,整个稳定细胞群构建时间由原来的数月缩短至数天,优选为一到两周,一般一周的时间就可满足大多数蛋白表达。
上述步骤1),可将目标蛋白序列构建至含有抗生素基因的质粒上,转座酶序列所在质粒上无须含有抗生素基因。
具体的,目标蛋白可以是抗体,融合蛋白,重组蛋白或者酶中任意一种。
具体的,抗生素筛选基因可以是嘌呤霉素,杀稻瘟菌素,博莱霉素或潮霉素等真核动物抗生素中任意一种或者几种。
具体的,含有编码目标蛋白基因和抗生素筛选基因的基因片段的上游和下游分别有一个piggyBac转座酶识别的末端反向重复序列。
较佳的,若目标蛋白包含多个转录单元(如抗体的重链和轻链),可将不同转录单元克隆至含有不同抗生素基因的载体中。
较佳的,待转染宿主CHO细胞已经过驯化,可在无血清条件下悬浮培养。
较佳的,步骤1),使用去内毒素的试剂盒提取质粒DNA。
较佳的,步骤2),含有编码目标蛋白基因的质粒与含有转座子序列的质粒的推荐质量比为10:1。
较佳的,步骤3),转染后24小时内,向转染后细胞中加入等体积含有两倍正常浓度筛选抗生素的传代培养基。
较佳的,步骤3),每隔2~4天使用含有筛选抗生素的传代培养基传代。
较佳的,步骤4),待细胞活率恢复到90%左右,将步骤3)筛选后的细胞稀释进入生产使用的基础培养基中,通过批次补料的工艺表达目标蛋白。
本发明利用piggyBac转座系统能够整合质粒DNA片段进入基因组中的特性,实现了在CHO细胞中快速高效的表达蛋白。本发明的优势在于:1)采用本方法可以快速构建表达目标蛋白的稳定的细胞群,经过抗生素筛选在一周时间即可构建稳定的细胞群,可以满足绝大多数蛋白的表达。2)采用本方法获得的稳定细胞群可以高效的表达蛋白,在抗生素筛选一周后,最高可达近1克/升以上(如图3所示),而现有技术中对于相同目标蛋白,瞬时转染的表达量均仅为200毫克/升左右,本方法的蛋白表达量显著提高。3)本方法的整个筛选周期在一到两周时间达到最优的效果,一般在两周时间即可实现较好的效果,从转染到批次补料结束短至两周半的方法。4)此方法操作简便,整个周期和现有瞬时转染工艺相当但表达量远高于瞬时转染,可作为大体积瞬时转染的替代方案。由piggyBac转座子介导的细胞群筛选和体积放大可同步进行,因此也无须像传统稳定细胞群一样保存已构建的细胞,省去了大量液氮存储和管理的成本。另外,由于目标蛋白稳定整合进入宿主细胞基因组,细胞表达量在数周内稳定,因此对于需要培养体积高达200L的蛋白生产,从转染到批次补料结束也可以在一个月内完成。
附图说明
图1是不同抗生素筛选时间对稳定细胞群最终表达量的影响;
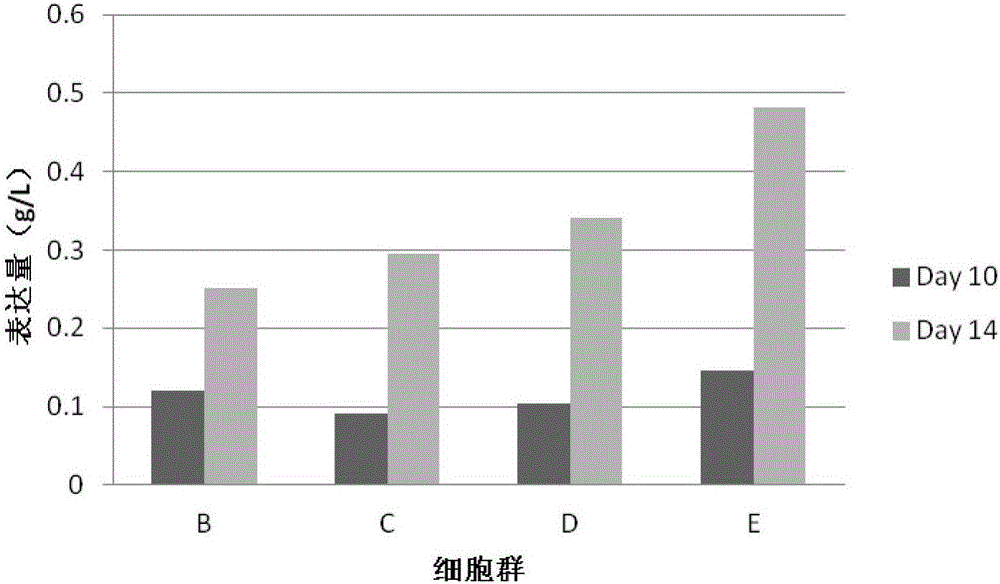
图2是不同稳定细胞群在抗生素筛选一周后的表达量;
图3是细胞群表达量的稳定性评估。
具体实施方式
下面将结合附图对本发明的技术方案进行清楚、完整的描述,显然,所描述的实施例是本发明的一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动的前提下所获得的所有其他实施例,都属于本发明保护的范围。
为对本发明的技术内容、特点与功效有更具体的了解,现结合具体的实施例,对本发明的技术方案的进一步详细描述如下:
实施例1
本实例利用piggyBac转座子构建不同的稳定细胞群,利用抗生素筛选2天、5天或7天后,进行批次补料实验表达蛋白,从而获得合适的抗生素筛选时间。具体地说,包括以下步骤:
步骤1,将目标蛋白的基因和抗生素基因克隆至含有一对piggyBac转座酶识别的末端反向重复序列之间。对于抗体或Fc融合蛋白等含有多个表达单元的目标蛋白,可将不同表达单元克隆至含有不同抗生素基因的载体上。
步骤2,使用去内毒素的质粒抽提试剂盒分别提取含有转座酶和目标蛋白的质粒。所有质粒不需要进行酶切线性化。
步骤3,宿主细胞为已经经过悬浮驯化的CHO细胞。转染当天宿主细胞期望密度在3.5至4.5E6/毫升之间。
步骤4,对于编码人IgG1抗体的细胞群A,将20微克含有抗体的质粒以及转座酶的质粒(二者质量比为10:1)和60微克聚醚酰亚胺(Polyetherimide,缩写为PEI)依次加入10毫升宿主细胞中,混匀后将摇管放回摇床中。
步骤5,转染后24小时内,向每管中加入10毫升含有筛选抗生素的传代培养基。抗生素种类根据步骤1中质粒含有的抗生素基因选择,抗生素浓度根据未转染的宿主细胞的杀伤曲线确定。此步骤加入的抗生素浓度为正常筛选培养基的抗生素浓度的二倍,使得加入筛选培养基后,最终抗生素浓度仍为正常筛选抗生素浓度。
步骤6,加压后每隔2~4天使用含有筛选抗生素的传代培养基传代,接种密度需要根据细胞活率和传代时间做出相应的调整。
步骤7,一周后,全部4组细胞活率可恢复至90%左右,将细胞按照统一接种密度稀释进入生产使用的基础培养基中。充分混匀后将细胞放入摇床中。
步骤8,根据细胞生长状况,根据不同的细胞选择适宜的补料培养基进行批次补料实验。
对比例:瞬时转染组,通过瞬时转染技术(为现有技术,在此不赘述),细胞群A的蛋白表达量为150mg/L。
实验结果如图1所示。利用piggyBac转座子构建细胞群的方法,在抗生素筛仅2天、5天或7天后,分别经过批次补料实验,14天后可获得蛋白表达量分别为112mg/L,207mg/L和285mg/L。其中,抗生素筛2天组可获得的蛋白表达量就基本能够达到瞬时转染组的蛋白表达量水平,抗生素筛5天组或7天组可获得的蛋白表达量明显高于瞬时转染组。相比之下,本方法在一周之内可构建稳定细胞群,随着筛选时间的增加,批次补料后的表达量也显著增加。综合权衡细胞群的构建时间和表达量,筛选周期一周就可以满足绝大多数蛋白表达。
实施例2
本实例利用piggyBac转座子构建编码人IgG1抗体的B、C、D、E四组不同的稳定细胞群,利用抗生素筛选一周后,进行批次补料实验表达蛋白,记录补料天数为10天和14天的蛋白表达量数据。具体地说,包括以下步骤:
步骤1,将目标蛋白的基因和抗生素基因克隆至含有一对piggyBac转座酶识别的末端反向重复序列之间。对于抗体或Fc融合蛋白等含有多个表达单元的目标蛋白,可将不同表达单元克隆至含有不同抗生素基因的载体上。
步骤2,使用去内毒素的质粒抽提试剂盒分别提取含有转座酶和目标蛋白的质粒。所有质粒不需要进行酶切线性化。
步骤3,宿主细胞为已经经过悬浮驯化的CHO细胞。转染当天宿主细胞期望密度在3.5至4.5E6/毫升之间。
步骤4,对于每个细胞群B~E,将20微克含有目标蛋白的质粒以及转座酶的质粒(二者质量比为10:1)和60微克聚醚酰亚胺(Polyetherimide,缩写为PEI)依次加入10毫升宿主细胞中,混匀后将摇管放回摇床中。
步骤5,转染后24小时内,向每管中加入10毫升含有筛选抗生素的传代培养基。抗生素种类根据步骤1中质粒含有的抗生素基因选择,抗生素浓度根据未转染的宿主细胞的杀伤曲线确定。此步骤加入的抗生素浓度为正常筛选培养基的抗生素浓度的二倍,使得加入筛选培养基后,最终抗生素浓度仍为正常筛选抗生素浓度。
步骤6,加压后每隔2~4天使用含有筛选抗生素的传代培养基传代,接种密度需要根据细胞活率和传代时间做出相应的调整。
步骤7,一周后,全部4组细胞活率可恢复至90%左右,将细胞按照统一接种密度稀释进入生产使用的基础培养基中。充分混匀后将细胞放入摇床中。
步骤8,根据细胞生长状况,根据不同的细胞选择适宜的补料培养基进行批次补料实验。
对比例:瞬时转染组,通过瞬时转染技术(为现有技术,在此不赘述),细胞群B、C、D、E的蛋白表达量均在150~200mg/L之间。
实验结果如图2所示。四组不同的细胞群,在抗生素筛选一周后,分别经批次补料实验,记录补料10天和补料14天后的蛋白表达量。其中,补料10天时,四组细胞群的蛋白表达量分别为121mg/L,91mg/L,104mg/L和146mg/L;补料14天时,四组细胞群的蛋白表达量分别为251mg/L,295mg/L,342mg/L和482mg/L,细胞群E的产量最高可达近500毫克/升。而对于相同目标蛋白,瞬时转染的表达量最高仅达到200毫克/升。可见采用本发明的方法,其蛋白表达量明显高于瞬时转染。与此同时,对于可能进行抗体药物开发的候选蛋白来讲,在产品质量方面,利用piggyBac转座系统构建稳定细胞群生产的蛋白和瞬时转染产品相比与日后产品更接近。和传统的细胞群生产,此种方法在产品产量和质量相似的同时大幅缩短的周期。因此,利用piggyBac转座系统构建稳定细胞株在许多药物开发早期样品制备方面较传统的瞬时转染和传统细胞群均有较大的优势。
实施例3
本实例通过评估利用piggyBac转座子构建的稳定细胞群表达量的稳定性,来预估这种方法的适用生产体积。具体地说,包括以下步骤:
步骤1,将目标蛋白的基因和抗生素基因克隆至含有一对piggyBac转座酶识别的末端反向重复序列之间。对于抗体或Fc融合蛋白等含有多个表达单元的目标蛋白,可将不同表达单元克隆至含有不同抗生素基因的载体上。
步骤2,使用去内毒素的质粒抽提试剂盒分别提取含有转座酶和目标蛋白的质粒。所有质粒不需要进行酶切线性化。
步骤3,宿主细胞为已经经过悬浮驯化的CHO细胞。转染当天宿主细胞期望密度在1至2E6/毫升之间。
步骤4,对于编码人IgG1抗体的细胞群F,将60微克含有目标蛋白的质粒以及转座酶的质粒(二者质量比为10:1)转染进入5毫升宿主细胞中,混匀后将摇管放回摇床中。
步骤5,转染后24小时内,向每管中加入5毫升含有筛选抗生素的传代培养基。抗生素种类根据步骤1中质粒含有的抗生素基因选择,抗生素浓度根据未转染的宿主细胞的杀伤曲线确定。此步骤加入的抗生素浓度为正常筛选培养基的抗生素浓度的二倍,使得加入筛选培养基后,最终抗生素浓度仍为正常筛选抗生素浓度。
步骤6,加压后每隔2~4天使用含有筛选抗生素的传代培养基传代,接种密度需要根据细胞活率和传代时间做出相应的调整。
步骤7,抗生素筛选一周,两周和三周后,分别将细胞按照特定的密度稀释进入生产使用的基础培养基中。充分混匀后将细胞放入摇床中。
步骤8,根据细胞生长状况,根据不同的细胞选择适宜的补料培养基进行批次补料实验。
实验结果如图3所示。在抗生素筛选一周,两周和三周后,细胞群F均通过批次补料放大生产14天,生产目标蛋白的表达量依次为1.1克/升,1.9克/升和1.8克/升。结合传代时体积的放大和表达量的稳定性,抗生素筛选两周后可满足50升以上的批次补料生产。通过批次补料放大生产,对于需要培养体积高达200L的蛋白生产,从转染到批次补料结束也可以在一个月内完成。
综上所述,上述各实施例及附图仅为本发明的较佳实施例而已,并不用以限定本发明的保护范围,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,皆应包含在本发明的保护范围内。
- 还没有人留言评论。精彩留言会获得点赞!