用于诊断膀胱癌的方法与流程
发明领域
本发明涉及用于通过分析源自受试者的生物样品鉴定所述受试者中的膀胱癌的方法。更具体地,本发明涉及通过分析来自尿样品的dna鉴定膀胱癌。该方法基于正常和膀胱癌dna之间dna甲基化的差异。
发明背景
膀胱癌是由膀胱中的细胞引起的癌症。大多数膀胱癌是由膀胱内衬中的上皮细胞(尿路上皮)引起的移行细胞癌。其他类型的膀胱癌包括鳞状细胞癌、腺癌、肉瘤和小细胞癌。
膀胱癌的诊断通常通过膀胱镜检查进行,在膀胱镜检查中使用内窥镜(膀胱镜)对膀胱可视地检查。活组织检查物通常取自疑似异常的区域,来检查任何癌性细胞的存在。
膀胱癌的诊断中采用的另一种方法是尿细胞学,其中在显微镜下分析尿样品以检测癌细胞的存在。多种成像方法诸如x-射线和计算机断层扫描(ct)已经被采用,用于诊断膀胱癌,该多种成像方法中的一些涉及静脉注射染料,以使得检查的组织能够可视化。
在最近几年,用于膀胱癌的另外的诊断手段已变得可用,诸如尿抗原测试和用于检测尿样品中的染色体异常的测定(urovysiontm)。
目前可用的方法具有很多缺点,因为它们中的一些是侵入性的、费力且耗时的,并且一些方法不够灵敏和特异。另外,通过病理学检查的诊断人工地进行并且因此是主观的。主观结果是有问题的,因为在由不同病理学家并且甚至由相同病理学家在不同时间分析的相同样品的结果之间可以存在差异。
属于本发明的申请人的wo2011/001274和wo2011/070441,分别公开了基于特定基因组基因座之间的信号比率的鉴定来自天然来源的dna的方法,以及将dna样品分类成不同类型的组织的方法。
对于用于以高特异性和高灵敏度诊断有相应需要的受试者中的膀胱癌而不要求良好训练的病理学家的改进的方法和试剂盒存在未被满足的需要。对客观、不依赖使用者的诊断方法也存在需要。
发明概述
根据一些方面,本发明提供了用于通过分析来自尿样品的dna鉴定有相应需要的受试者中的膀胱癌的方法和试剂盒。本发明的方法和试剂盒基于在正常dna和膀胱癌dna之间在选择的基因组基因座处的甲基化差异。
更具体地,本发明的方法和试剂盒利用一组基因组基因座,该组基因组基因座被出乎意料地发现在源自膀胱癌的dna中被高度甲基化并且在源自健康膀胱组织的dna中未被高度甲基化,并且从而可以用于确定给定的dna样品是来自膀胱癌患者还是来自健康受试者。
出乎意料地,本发明的方法和试剂盒显示出与细胞学相比具有较高的灵敏度,具有至少90%的总灵敏度(不区分膀胱癌级别或阶段),在侵袭性肿瘤中100%的灵敏度以及在非侵袭性肿瘤中至少89%的灵敏度。
根据本发明的方法和试剂盒,用至少一种甲基化敏感性限制性酶消化来自测试的受试者的尿样品的dna,只有在dna未被甲基化时该甲基化敏感性限制性酶才裂解其识别序列。本文公开的被称为“限制性基因座”的基因组基因座的组包括至少一种甲基化敏感性限制性酶的识别序列,并且因此根据其甲基化水平而被切割(消化),其中较高程度的甲基化导致较少的酶消化。出乎意料地,来自健康个体的dna样品在本文公开的基因组基因座的组处比来自癌症患者的dna样品更彻底地被切割。因此,在其中本发明的限制性基因座被高度甲基化的dna样品和其中这些基因座具有低甲基化水平的dna样品之间消化效率的差异,在随后的扩增步骤和定量步骤中确立了不同的扩增模式。出乎意料地,扩增模式的差异允许区分来自膀胱癌组织的dna和来自正常膀胱组织的dna。
根据本发明的方法和试剂盒的扩增和定量步骤包括共扩增来自被消化的dna的至少一种限制性基因座和对照基因座。对照基因座可以是不被消化步骤中使用的甲基化敏感性限制性酶切割的基因座。然后确定扩增的基因座的信号强度并计算每个限制性基因座和对照基因座的信号强度之间的比率。现在第一次公开了针对膀胱癌dna和正常膀胱dna产生区别性信号比率,因此使得能够鉴定膀胱癌。
通过将计算的来自测试的受试者的dna的信号比率与确定的已知来源即正常个体和/或癌症患者中的相同限制性基因座的一种或更多种参考比率比较,来进行膀胱癌的鉴定。基于该比较,将测试的样品鉴定为源自癌症患者或源自健康受试者。应注意的是,在任何时候,本发明的试剂盒和方法需要确定个体基因座本身的甲基化水平。
本发明的方法和试剂盒使得能够通过测试尿样品鉴定膀胱癌,从而有利地提供了疾病的非侵入性、基于分子的诊断。请求保护的方法和试剂盒的另一种益处是,可以在尿样品中进行鉴定,尽管尿样品可包含的dna的量相对低。通过本文公开的方法和试剂盒诊断膀胱癌所赋予的另外的益处是诊断的高灵敏度和特异性。另外,本文描述的方法产生了不依赖使用者的客观结果。
根据一个方面,本发明提供了一种用于鉴定人受试者中的膀胱癌的方法,该方法包括:
(a)将来自从受试者获得的尿样品的dna应用于通过至少一种甲基化敏感性限制性内切核酸酶的消化,以获得限制性内切核酸酶处理的dna;
(b)从限制性内切核酸酶处理的dna共扩增包含seqidno:1中列出的基因座的至少一种限制性基因座,以及对照基因座,从而生成每种基因座的扩增产物;
(c)确定每种生成的扩增产物的信号强度;
(d)计算所述至少一种限制性基因座的每一种和对照基因座的扩增产物的信号强度之间的比率;以及
(e)检测所述比率相对于相应的膀胱癌参考比率的高概率评分,从而鉴定人受试者中的膀胱癌。
在一些实施方案中,检测所述比率相对于相应的膀胱癌参考比率的高概率评分包括检测高于预先定义的阈值的相对于膀胱癌参考比率的概率评分。
在一些实施方案中,至少一种限制性基因座还包括seqidno:5中列出的基因座,并且所述概率评分为seqidno:1的比率和seqidno:5的比率相对于它们相应的膀胱癌参考比率的组合概率评分。
在一些实施方案中,至少一种限制性基因座还包括seqidno:7中列出的基因座和seqidno:11中列出的基因座,并且所述概率评分为所述seqidno:1的比率、所述seqidno:5的比率、seqidno:7的比率和seqidno:11的比率相对于它们相应的膀胱癌参考比率的组合概率评分。
在一些实施方案中,至少一种限制性基因座还包括选自seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:9、seqidno:10、seqidno:12、seqidno:13、seqidno:14和seqidno:15中列出的基因座的组的至少一种另外的限制性基因座,并且所述概率评分为所述seqidno:1的比率、seqidno:5的比率、seqidno:7的比率和seqidno:11的比率以及所述至少一种另外的限制性基因座的至少一种另外的比率相对于它们相应的膀胱癌参考比率的组合概率评分。
在一些实施方案中,至少一种另外的限制性基因座包括seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:9、seqidno:10、seqidno:12、seqidno:13、seqidno:14和seqidno:15中列出的所有基因座。因此,在一些实施方案中,至少一种限制性基因座包括seqidno:1-15中列出的所有基因座。每种可能性代表本发明的单独实施方案。
在一些实施方案中,该方法还包括:提供包含非人dna序列的第一对照构建体和第二对照构建体,其中第一对照构建体包含缺乏甲基化敏感性限制性内切核酸酶的识别序列的dna序列,并且第二对照构建体包含含有甲基化敏感性限制性内切核酸酶的识别位点并且完全未被甲基化的dna序列;用甲基化敏感性限制性内切核酸酶消化第一对照构建体和第二对照构建体;以及扩增第一对照构建体和第二对照构建体;其中第一构建体的充分扩增伴随第二构建体的低扩增或不扩增的检测结果指示适当的dna消化。
在一些实施方案中,至少一种甲基化敏感性限制性内切核酸酶选自由以下组成的组:aatii、acc65i、acci、acil、acii、afel、agel、apal、apali、asci、asisi、aval、avaii、baei、bani、bbei、bceai、bcgi、bfuci、bgli、bmgbi、bsaai、bsabi、bsahi、bsai、bseyi、bsiei、bsiwi、bsli、bsmai、bsmbi、bsmfi、bspdi、bsrbi、bsrfi、bsshii、bsski、bstapi、bstbi、bstui、bstzl7i、cac8i、clai、dpni、drdi、eaei、eagi、eagl-hf、ecii、ecori、ecori-hf、faui、fnu4hi、fsei、fspi、haeii、hgai、hhai、hincii、hincii、hinfl、hinpli、hpai、hpaii、hpyl66ii、hpyl88iii、hpy99i、hpych4iv、kasi、mlui、mmei、mspali、mwoi、naei、naci、ngoniv、nhe-hfi、nhei、nlaiv、noti、noti-hf、nrui、nt.bbvci、nt.bsmai、nt.cvipii、paer7i、plei、pmei、pmli、pshai、pspomi、pvui、rsai、rsrii、sacii、sall、sali-hf、sau3ai、sau96i、scrfi、sfii、sfoi、sgrai、smai、snabi、tfii、tsci、tsei、tspmi和zrai。每种可能性代表本发明的单独实施方案。
在一些实施方案中,步骤(a)使用一种甲基化敏感性限制性内切核酸酶进行。在一些实施方案中,甲基化敏感性限制性内切核酸酶是hhai。
在一些实施方案中,步骤(a)使用一种甲基化敏感性限制性内切核酸酶进行。在一些实施方案中,甲基化敏感性限制性内切核酸酶是hinpli。
在一些实施方案中,对照基因座是缺乏甲基化敏感性限制性内切核酸酶的识别序列的基因座。
在一些实施方案中,对照基因座是seqidno:16中列出的基因座。
在一些实施方案中,步骤(b)利用实时pcr进行。
在一些实施方案中,步骤(b)利用实时pcr进行,并且该方法还包括添加荧光探针用于特异性地检测至少一种限制性基因座和至少一种对照基因座的扩增产物。
在一些实施方案中,步骤(b)利用实时pcr进行,并且所述计算所述至少一种限制性基因座的每一种和对照基因座的扩增产物的信号强度之间的比率包括确定每种基因座的定量循环(cq)和计算2(cq对照基因座-cq限制性基因座)。在一些实施方案中,相应的膀胱癌参考比率为2(cq参考dna中的对照基因座-cq参考dna中的限制性基因座),其中参考dna是膀胱癌dna。
在一些实施方案中,该方法还包括提供源自健康受试者的dna中的至少一种限制性基因座和对照基因座的扩增产物的信号强度之间的健康参考比率。在一些实施方案中,该方法还包括检测所述比率相对于相应的健康参考比率的低概率评分。
在一些实施方案中,所述膀胱癌选自由以下组成的组:移行细胞癌、鳞状细胞癌、腺癌、肉瘤和小细胞癌。每种可能性代表本发明的单独实施方案。
在一些具体的实施方案中,所述膀胱癌为移行细胞癌。
根据另一方面,本发明提供了一种治疗有相应需要的受试者中的膀胱癌的方法,该方法包括根据本发明的方法鉴定膀胱癌,以及向所述受试者施用抗癌疗法。
在一些实施方案中,所述抗癌疗法包括外科手术、放射疗法、化学疗法和免疫疗法中的一种或更多种。
根据又另一方面,本发明提供了一种用于测量疑似患有膀胱癌的人受试者中的dna的甲基化比率的方法,该方法包括:(a)从受试者的尿样品获得dna样品,并使dna样品经历用至少一种甲基化敏感性限制性内切核酸酶的消化以获得限制性内切核酸酶处理的dna;(b)从限制性内切核酸酶处理的dna共扩增包含seqidno:1中列出的基因座的至少一种限制性基因座和对照基因座,从而生成每种基因座的扩增产物;以及(c)利用计算机将所述至少一种限制性基因座和所述对照基因座的信号强度的比率与膀胱癌dna中的所述基因座的参考比率比较,从而评价所述比率是否指示膀胱癌。
本发明的这些和另外的方面和特征从以下的详细描述、实施例和权利要求将变的明显。
附图简述
图1a展示了来自健康受试者的样品中的对照基因座和两种限制性基因座的示例性扩增曲线。
图1b展示了来自癌症患者的样品中的对照基因座和两种限制性基因座的示例性扩增曲线。
图2是描述根据本发明的一些实施方案的用于鉴定受试者中的膀胱癌的方法的流程图。
图3a是展示在代表性研究中90%的总灵敏度的饼状图。
图3b是展示在代表性研究中83%的总特异性的饼状图。
图4a展示了在代表性研究中就膀胱癌阶段而言方法的灵敏度。
图4b展示了在代表性研究中就膀胱癌级别而言方法的灵敏度。
发明详述
本发明涉及使用来自尿样品的dna,基于检测预先确定的基因座处的dna甲基化的改变鉴定膀胱癌。
本发明包括计算在用至少一种甲基化敏感性限制性酶消化测试的dna样品的dna后从该dna样品共扩增的选择的基因座之间的信号强度比率,并将这些比率与一种或更多种参考比率比较,该一种或更多种参考比率从源自已知来源的健康或膀胱癌的多于一个dna样品获得。基于该比较,将测试的样品鉴定为源自癌症患者或源自健康受试者。
如本文使用的,术语“多于一个/种”指‘至少两个/种’或‘两个/种或更多个/种’。
本发明的方法是特别地有益的,因为它们提供了用于诊断膀胱癌的非侵入性、不依赖使用者并且甚至可以在含有相对低量dna的尿样品中进行的高度灵敏且特异的手段。
此外,与利用甲基化分析来区分癌性组织和正常组织的常规方法(其要求确定特定基因组基因座处的实际甲基化水平)相比,本文描述的方法不要求评价绝对甲基化水平。因此本文公开的方法消除了对在确定甲基化水平本身中涉及的标准曲线和/或另外的费力步骤的需要,从而提供了简单且有成本效益的程序。通过本发明的方法获得的信号比率赋予了超过用于分析甲基化的方法的另外的优势,该信号比率在相同的反应混合物中(即,在相同的反应条件下)从相同的dna模板扩增的基因座之间计算。这致使该方法对多种“噪音”因素诸如模板dna浓度、pcr条件的改变和抑制剂的存在不敏感。此类噪音则是基于通过比较来自不同扩增反应的信号来定量基因座的甲基化水平的现有方法固有的。
在一些实施方案中,提供了一种用于鉴定受试者中的膀胱癌的方法,该方法包括:(a)用至少一种甲基化敏感性限制性内切核酸酶消化来自从受试者获得的尿样品的dna;(b)至少扩增消化的dna样品中的seqidno:1中列出的限制性基因座和对照基因座,其中对照基因座为在膀胱癌dna中和在健康dna中表现出相同的消化和扩增图谱的基因座,从而生成所述dna样品中的每种基因座的扩增产物;(c)确定每种生成的扩增产物的信号强度;(d)计算所述dna样品中的至少一种限制性基因座和对照基因座的扩增产物的信号强度之间的比率;以及(e)检测所述比率相对于相应的膀胱癌参考比率的高概率评分,从而鉴定人受试者中的膀胱癌。
在一些实施方案中,检测所述比率相对于相应的膀胱癌参考比率的高概率评分包括检测高于预先定义的阈值的概率评分。
在一些实施方案中,检测相对于膀胱癌参考比率的高概率评分包括将该比率与相应的膀胱癌参考比率以及任选地与健康参考比率比较,以及基于该比较分配概率评分,其中高概率评分指示癌症。
因此,在一些实施方案中,本发明的方法包括提供膀胱癌参考比率以及任选地健康参考比率。
在一些实施方案中,步骤(e)包括将该比率与参考标度比较(areferencescale),该参考标度包括在膀胱癌患者中以及在健康个体中确定的比率,其中所述概率评分为基于所述比率在参考标度内的相对位置分配给所述比率的评分。在一些实施方案中,比率的值越高,分配至其的评分越高。
在一些实施方案中,该方法还包括在步骤(b)中扩增至少一种另外的限制性基因座,从而生成该至少一种另外的限制性基因座的扩增产物;以及针对每种另外的限制性基因座重复步骤(c)-(d)。在一些实施方案中,在步骤(e)中,获得多于一个比率相对于其相应的膀胱癌参考比率的多于一个个体概率评分,其中每个比率为在不同的限制性基因座和对照基因座之间。组合概率评分基于该多于一个概率评分计算。
在一些实施方案中,组合评分可以是平均评分。在其他实施方案中,组合评分可以是个体概率评分的加权平均值。在一些实施方案中,组合评分可以是所有个体概率评分的和。通常,组合评分可以包括代表所有个体概率评分的任何数学或统计值。
在一些实施方案中,检测到高组合概率评分鉴定出人受试者中的膀胱癌。在一些实施方案中,检测到高组合概率评分为检测到高于预先定义的阈值的组合概率评分。
术语“来自……的dna”、“源自……的dna”、“……内的dna”等可互换,并且指从尿样品获得的dna、从尿样品分离的dna或尿样品原样,即含有dna的尿样品或其带有的dna样品。因此,这些术语包括但不限于,从尿样品分离的dna、富含dna的尿样品、和未被操作的尿样品中的dna。
人基因组中的甲基化以5-甲基胞嘧啶的形式存在,并且局限于为也被称为cpg二核苷酸的序列cg的一部分的胞嘧啶残基(为其他序列的一部分的胞嘧啶残基不被甲基化)。人基因组中的一些cg二核苷酸被甲基化,并且其他的不被甲基化。另外,甲基化是细胞和组织特异性的,以使得特定的cg二核苷酸在某些细胞中可被甲基化并且同时在不同的细胞中未被甲基化,或者在某些组织中被甲基化并且同时在不同的组织中未被甲基化。dna甲基化是基因转录的重要调节器。与正常组织相比异常的dna甲基化模式(超甲基化和低甲基化两者),已经与大量的人恶性肿瘤相关。
术语“膀胱癌”指由膀胱中的细胞引起的癌症。在本文中使用膀胱癌来包括移行细胞癌(tcc,也称为尿路上皮细胞癌)、鳞状细胞癌、腺癌、小细胞癌和肉瘤。每种可能性代表本发明的单独实施方案。
由膀胱的内衬中的上皮细胞(尿路上皮)引起的tcc是最常见的膀胱癌类型,占病例的多于90%。tcc通常包括两个亚型:乳头状癌,其中肿瘤从膀胱的内表面向中空部分(hollowcenter)以细长的、指样凸起形式生长;和扁平癌,其中肿瘤不朝向膀胱的中空部分生长。鳞状细胞癌和腺癌是较不常见的膀胱癌类型,并且小细胞癌和肉瘤相对罕见。
膀胱癌通常可以被进一步分类为:非侵袭性的,其中癌细胞局限于移行上皮的内层;或侵袭性的,其中癌细胞生长进入膀胱的固有层或甚至更加深入到肌肉层。膀胱癌还可以被描述为表面侵袭性的或非肌肉侵袭性的。这些术语包括非侵袭性肿瘤以及尚未生长进入到膀胱的主要肌肉层的任何侵袭性肿瘤两者。
膀胱癌的常用分类之一是t类别,t类别描述主要肿瘤已经生长进入膀胱壁(或超过)多远。通常,膀胱癌始于尿路上皮,并且随着癌生长可以侵袭进入膀胱的其他层,从而变成更为晚期。根据t类别的分类包括以下术语:
-t0:无原发性肿瘤的迹象
-ta:非侵袭性乳头状癌
-tis或cis:非侵袭性扁平癌(原位扁平癌)
-t1:肿瘤已经从膀胱内衬的细胞层生长进入下面的结缔组织。它尚未生长进入膀胱的肌肉层。
-t2:肿瘤已经生长进入肌肉层。
-t2a:肿瘤已经生长仅进入肌肉层的半层内。
-t2b:肿瘤已经生长进入肌肉层的半层外。
-t3:肿瘤已经生长穿过膀胱的肌肉层并进入其周围的脂肪组织层。
-t3a:向脂肪组织的扩散仅通过使用显微镜才能观察到。
-t3b:向脂肪组织的扩散大到足以在成像测试上观察到或足以由外科医生观察或感觉到。
-t4:肿瘤已经扩散超出脂肪组织并进入附近的器官或结构。它可以生长进入以下的任一种中:前列腺的基质(主要组织)、精囊、子宫、阴道、盆壁或腹壁。
-t4a:肿瘤已扩散至前列腺的基质(在男性中)或扩散至子宫和/或阴道(在女性中)。
-t4b:肿瘤已经扩散至盆壁或腹壁。
通过膀胱癌的级别(g)来描述它也是常见的,膀胱癌的级别表示当在显微镜下观察时,癌细胞与健康细胞的相似度。健康组织通常包含群聚在一起的多种类型的细胞。如果癌症看起来与健康组织相似并且含有不同的细胞组群,其是所谓的分化肿瘤或低级别肿瘤。如果癌组织看起来与健康组织很不同,其是所谓的低分化肿瘤或高级别肿瘤。
泌尿外科医师还可基于肿瘤复发或进展(生长或扩散)的可能性,利用以下类别对肿瘤的级别进行分类,以基于该级别计划治疗:
-乳头状瘤(或低度恶性潜能的良性乳头状尿路上皮肿瘤;
punlmp)-可复发但具有低进展风险。
-低级别:与punlmp相比可能复发和进展。
-高级别:最可能复发和进展。
膀胱癌有时可以同时影响膀胱的许多区域。如果发现多于一个肿瘤,将字母m添加到合适的t类别。
如本文使用的,术语“膀胱癌的鉴定”、“鉴定受试者中的膀胱癌”和“将受试者鉴定为患有膀胱癌”可互换,并且包括以下的任一种或更多种:筛选膀胱癌、检测膀胱癌的存在、检测膀胱癌的复发、检测对膀胱癌的易感性、检测对膀胱癌治疗的耐受性、检测对膀胱癌的治疗有效性、确定膀胱癌的阶段(严重度)、确定膀胱癌的预后以及受试者中的膀胱癌的早期诊断。每种可能性代表本发明的单独实施方案。
如本文使用的,术语“受试者”与“个体”可互换并且指人受试者。受试者可以疑似患有膀胱癌。在一些实施方案中,受试者可以例如基于疾病的既往史、遗传素质和/或家族史而处于发展膀胱癌的风险,可以是已经暴露至致癌物、职业危害、环境危害的任一种或更多种的受试者和/或表现出疑似临床癌症征象的受试者。在一些实施方案中,受试者可以显示出膀胱癌的至少一种症状或特征,包括例如血液在尿中的存在、排尿频率的改变、贫血、全身无力或刺激症状。在其他实施方案中,受试者可以是无症状的。
尿样品收集和处理
在一些实施方案中,可以利用常规收集容器或管从受试者收集尿样品。
在一些实施方案中,可以根据本领域已知方法从尿样品提取基因组dna。示例性程序被描述于例如sambrook等,molecularcloning:alaboratorymanual,第4版,coldspringharborlaboratorypress,coldspringharbor,ny,2012中。
dna消化
根据本发明的方法,使来自尿样品的dna经历用至少一种甲基化敏感性限制性内切核酸酶的消化,例如用一种、两种、三种甲基化敏感性限制性内切核酸酶的消化。每种可能性代表本发明的单独实施方案。
在一些实施方案中,从尿样品提取的全部dna被用于消化步骤。在一些实施方案中,在经历消化之前,未对dna定量。在其他实施方案中,在dna的消化之前,可以对其定量。
本文使用的“限制性内切核酸酶”与“限制性酶”可互换使用,指在或靠近被称为限制性位点的特异性识别核苷酸序列处切割dna的酶。
“甲基化敏感性”限制性内切核酸酶是只有在其识别序列未被甲基化时才裂解其识别序列(而被甲基化的位点则保持完整)的限制性内切核酸酶。因此,dna样品被甲基化敏感性限制性内切核酸酶消化的程度取决于甲基化水平,其中较高的甲基化水平保护免于裂解并且相应地导致较少的消化。
在一些实施方案中,至少一种甲基化敏感性限制性内切核酸酶可以选自由以下组成的组:aatii、acc65i、acci、acil、acii、afel、agel、apal、apali、asci、asisi、aval、avaii、baei、bani、bbei、bceai、bcgi、bfuci、bgli、bmgbi、bsaai、bsabi、bsahi、bsai、bseyi、bsiei、bsiwi、bsli、bsmai、bsmbi、bsmfi、bspdi、bsrbi、bsrfi、bsshii、bsski、bstapi、bstbi、bstui、bstzl7i、cac8i、clai、dpni、drdi、eaei、eagi、eagl-hf、ecii、ecori、ecori-hf、faui、fnu4hi、fsei、fspi、haeii、hgai、hhai、hincii、hincii、hinfl、hinpli、hpai、hpaii、hpyl66ii、hpyl88iii、hpy99i、hpych4iv、kasi、mlui、mmei、mspali、mwoi、naei、naci、ngoniv、nhe-hfi、nhei、nlaiv、noti、noti-hf、nrui、nt.bbvci、nt.bsmai、nt.cvipii、paer7i、plei、pmei、pmli、pshai、pspomi、pvui、rsai、rsrii、sacii、sall、sali-hf、sau3ai、sau96i、scrfi、sfii、sfoi、sgrai、smai、snabi、tfii、tsci、tsei、tspmi和zrai。每种可能性代表本发明的单独实施方案。
在一些实施方案中,尿样品可以经历用一种甲基化敏感性限制性内切核酸酶的消化。在一些具体实施方案中,甲基化敏感性限制性内切核酸酶可以是hhai。
在一些实施方案中,尿样品可以经历用一种甲基化敏感性限制性内切核酸酶的消化。在一些具体实施方案中,甲基化敏感性限制性内切核酸酶可以是hinp1i。
在一些实施方案中,可以进行dna消化至完全消化。在一些实施方案中,甲基化敏感性限制性内切核酸酶可以是hhai,并且完全消化可以在与酶在37℃孵育1至2个小时之后来完成。
在一些实施方案中,可以进行dna消化至完全消化。在一些实施方案中,甲基化敏感性限制性内切核酸酶可以是hinp1i,并且完全消化可以在与酶在37℃孵育1至2个小时之后来完成。
基因组基因座的扩增
如本文使用的术语“基因组基因座”或“基因座”可互换并且指染色体上特定位置处的dna序列。特定位置可以通过染色体上的分子位置,即通过起始碱基对和末端碱基对的编号来标识。给定基因组位置处的dna序列的变体被称为等位基因。基因座的等位基因位于同源染色体上的相同位点处。基因座包括基因序列以及其他遗传元件(例如,基因间序列)。
在本文“限制性基因座”被用于描述包含方法中使用的甲基化敏感性限制性酶的识别序列的基因座。
“对照基因座”和“内部参考基因座”可互换并且在本文用于描述这样的基因座,即所述基因座通过消化步骤中应用的限制性酶的消化取决于癌症的存在或不存在。在一些实施方案中,对照基因座是在膀胱癌中和在健康组织中表现出相同的消化和扩增图谱的基因座。在一些实施方案中,对照基因座是缺乏消化步骤中应用的限制性酶的识别序列的基因座。有利地,对照基因座是内部基因座,即分析的dna样品内的基因座,从而消除了对外部/另外的对照样品的需要。
在一些实施方案中,限制性基因座(therestrictionlocusorrestrictionloci)包括如下的seqidno:1-15中列出的基因座中的任一个或更多个:
seqidno:1,对应于第17号染色体上的位置65676359-65676418,(kcnj2基因);
seqidno:2,对应于第9号染色体上的位置21958446-21958585,(cdkn2a基因);
seqidno:3,对应于第6号染色体上的位置336844-336903,(irf4基因);
seqidno:4,对应于第21号染色体上的位置33319507-33319636,(olig2基因);
seqidno:5,对应于第6号染色体上的位置166502151-166502220,基因间区域;
seqidno:6,对应于第18号染色体上的位置896902-897031,(adcyap1基因);
seqidno:7,对应于第5号染色体上的位置32747873-32748022,(npr3基因);
seqidno:8,对应于第6号染色体上的位置27949195-27949264,基因间区域;
seqidno:9,对应于第7号染色体上的位置27191603-27191672,(hoxa9基因);
seqidno:10,对应于第16号染色体上的位置170170302-170170361,基因间区域;
seqidno:11,对应于第15号染色体上的位置30797737-30797876,基因间区域;
seqidno:12,对应于第1号染色体上的位置7936767-7936866,基因间区域;
seqidno:13,对应于第1号染色体上的位置170077565-170077634,(dnm3基因);
seqidno:14,对应于第2号染色体上的位置1727592-1727661,(pxdn基因);以及
seqidno:15,对应于第8号染色体上的位置72919092-72919231,(msc基因);
出乎意料地,本发明的发明人鉴定出seqidno:1-15中列出的限制性基因座在癌性膀胱组织和正常膀胱组织之间被不同地甲基化。更具体地,与正常组织相比,在膀胱癌组织中这些基因座具有增加的甲基化。
与来自正常非癌性膀胱组织的dna相比,在来自癌性膀胱组织的dna中这些基因座中的每一种包含更多地被甲基化的cg二核苷酸。有利地,被不同地甲基化的cg二核苷酸位于甲基化敏感性限制性酶的识别位点内。
在一些实施方案中,这些基因座的每一种可以包含至少一个甲基化敏感性限制性酶的限制性位点,其中cg二核苷酸在膀胱癌细胞中比在正常细胞中被更多地甲基化,意味着与正常组织相比,在癌性组织中更大量的细胞在该位置处包含甲基化。在一些实施方案中,这些基因座的每一种可以包含至少一个hhai限制性位点(gcgc)。在一些实施方案中,这些基因座的每一种可以包含至少一个hinp1i限制性位点。甲基化敏感性限制性酶只有在其识别序列未被甲基化时才裂解其识别序列。因此,包含较高百分比的其中限制性位点中的cg二核苷酸被甲基化的dna分子的dna样品与包含较高百分比的其中cg二核苷酸未被甲基化的dna分子的dna样品相比,将被消化至较低的程度。基于本文公开的方法,与来自正常(健康)个体的dna相比,来自膀胱癌患者的尿样品的dna通过甲基化敏感性限制性酶的dna消化较不彻底。出乎意料地发现,消化效率的差异建立了在随后的扩增和定量步骤中的不同扩增模式,该不同扩增模式使得能够区分来自癌性膀胱组织的dna和来自正常膀胱组织的dna。
在一些实施方案中,seqidno:1-15中列出的基因座中的每一种可以包含另外的cg二核苷酸,该另外的cg二核苷酸的甲基化状态与测定无关或对测定无影响-只有在限制性酶(例如hhai或hinp1i)的识别序列处的甲基化是相关的。
在一些实施方案中,对照基因座是如seqidno:16中列出的,其对应于第7号染色体上的位置121380854-121380913(基因间区域)。在一些实施方案中,也被称为内部参考基因座的对照基因座不包含限制性酶的识别序列。在一些实施方案中,当用甲基化敏感性限制性酶消化dna样品时,对照基因座的序列保持完整(不管其甲基化状态如何)。
在一些实施方案中,对照基因座的序列在膀胱癌组织中和在健康膀胱组织中表现出相同的消化和扩增图谱。
在一些实施方案中,对照基因座包含seqidno:16中列出的基因座,并且在用甲基化敏感性限制性酶消化之后的对照基因座的扩增模式不受甲基化影响。
在下文中使用甲基化敏感性限制性酶hhai和hinp1i示例了使用seqidno:1-15中列出的限制性基因座区分膀胱癌患者的尿样品和健康个体的尿样品的优势。这些限制性基因座的每一种和seqidno:16中列出的对照基因座在用hhai或用hinp1i消化后的扩增在大的癌症患者和健康个体组中进行。与对照组相比,在癌症组中,每种限制性基因座和对照基因座的扩增产物之间的信号比率的计算结果显示出显著较高的平均信号比率(高至少一个数量级)。
在一些实施方案中,方法包括在dna样品的消化后扩增至少一种限制性基因座和至少一种基因组基因座。
如本文使用的,“至少一种(限制性/对照)基因座”可以包括单种基因座或多于一种单独的基因座,以使得措辞如“包含seqidno:1中列出的基因座和seqidno:2中列出的基因座的至少一种限制性基因座”表示至少这两种单独的基因组基因座被扩增。
在一些实施方案中,方法包括扩增seqidno:1中列出的限制性基因座和对照基因座以及任选地至少一种另外的限制性基因座。
在一些实施方案中,方法包括扩增如seqidno:1中列出的限制性基因座和如seqidno:16中列出的对照基因座。
在一些实施方案中,方法包括扩增多于一种限制性基因座(即,至少两种限制性基因座)和对照基因座。
在一些实施方案中,多于一种限制性基因座包括如seqidno:1中列出的限制性基因座和如seqidno:5中列出的限制性基因座。在一些实施方案中,方法包括扩增如seqidno:1中列出的第一限制性基因座并且还包括扩增如seqidno:5中列出的第二限制性基因座。
在一些实施方案中,多于一种限制性基因座还包括选自seqidno:7和seqidno:11中列出的基因座的至少一种限制性基因座。在一些实施方案中,多于一种限制性基因座还包括分别如seqidno:7和11中列出的第三和第四限制性基因座。
在一些实施方案中,多于一种限制性基因座还包括选自由以下组成的组的至少一种另外的限制性基因座:seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:9、seqidno:10、seqidno:12、seqidno:13、seqidno:14和seqidno:15。每种可能性代表本发明的单独实施方案。
在一些实施方案中,多于一种限制性基因座包括如seqidno:1列出的基因座并且还包括选自由seqidno:2-15组成的组的1、2、3、4、5、6、7、8、9、10、11、12、13或14种另外的限制性基因座。每种可能性代表本发明的单独实施方案。
在一些实施方案中,多于一种限制性基因座包括如seqidno:1和5列出的基因座并且还包括选自由以下组成的组的1、2、3、4、5、6、7、8、9、10、11、12或13种另外的限制性基因座:seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:7、seqidno:8、seqidno:9、seqidno:10、seqidno:11、seqidno:12、seqidno:13、seqidno:14和seqidno:15。每种可能性代表本发明的单独实施方案。
在一些实施方案中,多于一种限制性基因座包括如seqidno:1、5和7列出的基因座并且还包括选自由以下组成的组的1、2、3、4、5、6、7、8、9、10、11或12种另外的限制性基因座:seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:9、seqidno:10、seqidno:11、seqidno:12、seqidno:13、seqidno:14和seqidno:15。每种可能性代表本发明的单独实施方案。
在一些实施方案中,多于一种限制性基因座包括如seqidno:1、5、7和11列出的基因座并且还包括选自由以下组成的组的1、2、3、4、5、6、7、8、9、10或11种另外的限制性基因座:seqidno:2、seqidno:3、seqidno:4、seqidno:6、seqidno:8、seqidno:9、seqidno:10、seqidno:12、seqidno:13、seqidno:14和seqidno:15。每种可能性代表本发明的单独实施方案。
在一些实施方案中,多于一种限制性基因座包括seqidno:1-15中列出的限制性基因座。在一些实施方案中,多于一种限制性基因座由seqidno:1-15中列出的限制性基因座组成。在一些实施方案中,方法包括扩增如seqidno:1-15中列出的多于一种限制性基因座。
如本文使用的,“扩增”指一种或更多种感兴趣的特定核酸靶的拷贝数的增加。扩增通常在pcr反应混合物的存在下通过聚合酶链式反应(pcr)进行,该pcr反应混合物可以包括补充有dna模板、聚合酶(通常地taq聚合酶)、dntp、引物和探针(视情况而定)的合适的缓冲液,如本领域已知的。
如本文使用的术语“多核苷酸”包括任何长度的聚合形式的核苷酸(脱氧核糖核苷酸或核糖核苷酸)或其类似物。本文还使用术语“寡核苷酸”以包括通常长度多达100个碱基的聚合形式的核苷酸。
“扩增产物”总体地指在扩增反应中生成并积累的特定靶序列的核酸分子。该术语通常指使用给定的扩增引物组通过pcr生成的核酸分子。
如本文使用的,“引物”定义了能够与靶序列退火(杂交),从而产生可在适合的条件下作为dna合成的起始点的双链区域的寡核苷酸。术语“引物对”在本文中指一对寡核苷酸,其被选择一起用于通过许多类型的扩增方法中的一种、优选地pcr扩增选择的核酸序列。如本领域通常已知的,引物可以被设计为在选择的条件下与互补序列结合。
取决于特定的测定形式和特定需要,引物可以是任何合适的长度。在一些实施方案中,引物可以包括至少15个核苷酸的长度,优选地19-25个核苷酸之间的长度。可以改造引物以特别地适于选择的核酸扩增系统。如本领域通常已知的,可以通过考虑寡核苷酸引物与其靶向的序列的杂交的解链点设计该寡核苷酸引物(sambrook等,同上)。
在一些实施方案中,可以使用反向和正向引物对从相同的dna样品(消化的样品)扩增限制性基因座和对照基因座,所述反向和正向引物对如本领域已知被设计为特异性地扩增每种基因座。在一些实施方案中,引物可以被设计为扩增基因座连同其5’和3’侧翼序列。
在一些实施方案中,5’侧翼序列可以包含1-60个之间的碱基。在另外的实施方案中,5’侧翼序列为在10-50个碱基之间。例如,5’侧翼序列可以包含紧邻基因座上游的10个碱基、15个碱基、20个碱基、25个碱基、30个碱基、35个碱基、40个碱基、45个碱基或50个碱基。每种可能性代表本发明的单独实施方案。
在一些实施方案中,3’侧翼序列可以包含1-90个之间的碱基。在一些实施方案中,3’侧翼序列可以包含5-80个之间的碱基。在一些实施方案中,3’侧翼序列可以包含紧邻基因座下游的5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79或80个碱基。每种可能性代表本发明的单独实施方案。
在一些实施方案中,引物可以被设计为生成长度为75-225个碱基之间的扩增产物。
在一些实施方案中,方法包括在相同的反应混合物中同时扩增多于一种靶序列(至少一种限制性基因座和一种对照基因座),这是一种被称为多重扩增或共扩增的过程。该过程要求同时使用多个引物对。如本领域已知的,可以设计引物以使得它们可以在扩增期间在相同的退火温度工作。在一些实施方案中,在本文公开的方法中使用具有相似解链温度(tm)的引物。对于以混合物(pool)使用的引物,在约3℃-5℃之间的tm变化被认为是可接受的。
在一些实施方案中,所有的限制性基因座和对照基因座可以在单个反应混合物中扩增。在其他实施方案中,例如由于特定仪器的技术限制,可以将消化的dna样品分成若干等份,将每一等份补充有用于扩增一种或更多种限制性基因座和对照基因座的引物对。因此,即使dna样品被分成若干等份,在每个等份中扩增对照基因座,并进行同时扩增(即,来自同一等份)的对照基因座和限制性基因座的信号比率的计算。
在一些实施方案中,方法可以在消化和扩增步骤中使用包含非人dna序列的对照构建体。在一些实施方案中,对照构建体包含人工(合成)dna序列。对照构建体被用于作为消化和扩增过程的对照,例如监测消化和扩增步骤的有效性和质量。在一些实施方案中,对照构建体和dna样品同时且任选地在同一容器(例如测试管、小瓶等)内通过至少一种甲基化敏感性限制性酶消化。在一些实施方案中,方法可以包括使对照构建体连同dna样品一起经历用至少一种甲基化敏感性限制性酶的消化的步骤。在一些实施方案中,方法可以包括向dna样品添加对照构建体,并使两者经历用至少一种甲基化敏感性限制性酶的消化。
在一些实施方案中,甲基化敏感性限制性内切核酸酶可以连同以下对照构建体中的一种或更多种一起用于消化步骤:第一对照构建体,该第一对照构建体包含缺乏甲基化敏感性限制性内切核酸酶的识别序列的dna序列;和第二对照构建体,该第二对照构建体包含含有甲基化敏感性限制性内切核酸酶的识别序列并完全未被甲基化的dna序列,以使得第一对照构建体保持完整,而第二对照构建体被完全消化或至少部分消化。在随后的扩增步骤中,可以添加对对照构建体特异性的引物(和任选地探针)。第一构建体的充分扩增伴随第二构建体的足够较低的扩增的检测结果指示适当的dna消化。
在一些实施方案中,检测第一构建体的充分扩增伴随第二构建体的足够较低的扩增可以包括在实时pcr中检测第一对照构建体和第二对照构建体的定量循环(cq)之间至少5个循环的差异。在一些实施方案中,差异可以是至少6个循环、至少7个循环或至少8个循环。每种可能性代表本发明的单独实施方案。
在一些实施方案中,第一对照构建体和第二对照构建体可以分别包含seqidno:17和18。在一些实施方案中,用于扩增和检测第一对照构建体的引物和探针可以包括seqidno:19(正向)、seqidno:20(反向)和seqidno:21(探针)。在一些实施方案中,用于扩增和检测第二对照构建体的引物和探针可以包括seqidno:22(正向)、seqidno:23(反向)和seqidno:24(探针)。
在一些实施方案中,基因组基因座的扩增可以利用实时pcr(rt-pcr)(也被称为定量pcr(qpcr))进行,其中同时进行扩增和扩增产物的检测。
在一些实施方案中,在rt-pcr中检测扩增产物可以使用多核苷酸探针,通常地荧光标记的多核苷酸探针完成。
如本文使用的,“多核苷酸探针”或“寡核苷酸探针”可互换并且指与感兴趣的基因座的核酸序列内,例如限制性基因座或对照基因座的序列内的特定子序列互补的标记的多核苷酸。在一些实施方案中,检测基于组合的报告物和猝灭物分子(rochemolecularsystemsinc.)通过利用taqman测定完成。在此类测定中,多核苷酸探针具有附连至其5’末端的荧光部分(荧光团)和附连至3’末端的猝灭物。在pcr扩增期间,多核苷酸探针与模板上的其靶序列选择性地杂交,并且随着聚合酶复制模板,由于聚合酶的5’核酸酶活性,它还裂解多核苷酸探针。当多核苷酸探针是完整的时,猝灭物和荧光部分之间紧密靠近,往往导致低水平的背景荧光。当多核苷酸探针被裂解时,猝灭物与荧光部分解偶联,导致荧光强度的增强。荧光信号与扩增产物的量相关联,即,信号随着扩增产物积累而增强。
如本文使用的,“与……选择性地杂交”(以及“选择杂交”、“与……特异性地杂交”和“特异性杂交”)指核酸分子(诸如引物或探针)在严格条件下优先与特定的互补核苷酸序列结合、双链化或杂交。术语“严格条件”指这样的条件:核酸分子在所述条件下将优先与其靶序列杂交,并且在较小程度上与其他非靶序列杂交或根本不与其他非靶序列杂交。在核酸杂交的上下文中的“严格杂交”是序列依赖性的,并且在不同的条件下不同,如本领域已知的。
多核苷酸探针的长度可以变化。在一些实施方案中,多核苷酸探针可以包含15-30个之间的碱基。在另外的实施方案中,多核苷酸探针可以包含25-30个之间的碱基。在一些实施方案中,多核苷酸探针可以包含20-30个之间的碱基,例如20个碱基、21个碱基、22个碱基、23个碱基、24个碱基、25个碱基、26个碱基、27个碱基、28个碱基、29个碱基、30个碱基。每种可能性代表本发明的单独实施方案。
多核苷酸探针可以被设计为与模板的任一链结合。另外的考虑因素包括多核苷酸探针的tm,其应优选地与引物的tm相兼容。可以使用计算机软件来设计引物和探针。
如以上所述的,本文公开的方法可以包括在相同的反应混合物中同时扩增多于一种靶序列(至少一种限制性基因座和一种对照基因座)。为了区分平行扩增的多个靶序列,可以使用用不同荧光颜色标记的多核苷酸探针。
在一些实施方案中,多核苷酸探针形成荧光团/猝灭物对,如本领域已知的,并且包括例如fam-tamra、fam-bhq1、yakimayellow-bhq1、atto550-bhq2和rox-bhq2。
在一些实施方案中,染料组合可以与选择的rt-pcr热循环仪相兼容。
在一些实施方案中,可以在每个pcr循环期间监测荧光,提供扩增曲线,该扩增曲线将来自探针的荧光信号的变化显示为循环数的函数。
在rt-pcr的上下文中,使用以下术语:
“定量循环”(“cq”)指其中荧光增强高于阈值的循环数,该阈值通过软件自动地设置或由使用者手动地设置。在一些实施方案中,阈值可以是对于所有基因座恒定的,并且可以提前设置,然后进行扩增和检测。在其他实施方案中,可以在运行后基于在扩增循环期间检测的每个基因座的最大荧光水平,分别定义该基因座的阈值。
“阈值”指用于cq确定的荧光值。在一些实施方案中,阈值可以是高于基线荧光和/或高于背景噪音并且在扩增曲线的指数生长阶段内的值。
“基线”指其中存在很小或不存在荧光变化的pcr的初始循环。
可以使用计算机软件来分析扩增曲线并确定基线、阈值和cq。
在用至少一种甲基化敏感性限制性酶消化之后,其中酶的识别位点中的cg二核苷酸被甲基化的基因座以高效率扩增,因为dna分子被保护免于消化。结果是相对低的cq值,因为可检测的扩增产物在相对少(低)的扩增循环数之后显示。相反,其中酶的识别位点中的cg二核苷酸未被甲基化的基因座在消化步骤期间被更彻底地切割,并且因此在扩增和定量步骤中导致较高的cq值(即,在相对高的扩增循环数之后显示可检测的扩增产物)。
在可选的实施方案中,可以通过使用荧光标记的引物的常规pcr、随后进行扩增产物的毛细管电泳来进行扩增产物的扩增和检测。在一些实施方案中,在扩增后,通过毛细管电泳分离扩增产物,并定量荧光信号。在一些实施方案中,可以生成根据尺寸(bp)或距注射的时间绘制荧光信号的变化的电泳图,其中电泳图中的每个峰对应于单个基因座的扩增产物。峰高(例如使用“相对荧光单位”,rfu提供)可以代表来自扩增的基因座的信号的强度。计算机软件可以被用来检测峰,并计算其扩增产物在毛细管电泳仪器上运行的一组基因座的荧光强度(峰高)以及随后信号强度之间的比率。
对于用甲基化敏感性限制性酶例如hhai或hinp1i消化的dna样品,其中酶的识别位点中的cg二核苷酸被甲基化的基因座在电泳图中产生相对强的信号(较高的峰)。相反,其中酶的识别位点中的cg二核苷酸未被甲基化的基因座在电泳图中产生相对弱的信号(较低的峰)。
在一些实施方案中,引物的荧光标记物包括以下的任一种:荧光素、fam、丽丝胺(lissamine)、藻红蛋白、罗丹明、cy2、cy3、cy3.5、cy5、cy5.5、cy7、fluorx、joe、hex、ned、vic和rox。
信号比率
如本文使用的术语“比率”或“信号比率”指从单个dna样品中(相同的反应混合物中)的一对基因组基因座的共扩增、特别地限制性基因座和对照基因座的共扩增获得的信号的强度之间的比率。
如本文使用的,“seqidno:x的比率”指在如本文详述的用限制性酶消化的dna样品中的seqidno:x中列出的基因座和对照基因座的共扩增之后,seqidno:x中列出的基因座的信号强度和对照基因座的信号强度之间的比率。
如本文使用的术语“信号强度”指反映对应于基因座的完整拷贝的初始量的基因座特异性扩增产物的量的量度。然而,信号强度可以不指示扩增产物/完整基因座的实际量,并且可以不涉及扩增产物/完整基因座的任何绝对量的计算。因此,为了计算扩增子信号的比率,可以不需要标准曲线或参考dna,因为不需要计算实际dna浓度或dna甲基化水平本身。
在一些示例性实施方案中,扩增以及扩增产物的检测通过rt-pcr进行,其中特定基因座的信号强度可以通过计算的该基因座的cq表示。在该情况中,信号比率可以通过以下计算结果表示:2(对照基因座的cq–限制性基因座的cq)。
在另外的示例性实施方案中,扩增产物的检测通过毛细管电泳进行,其中特定基因座的信号强度为其对应峰的相对荧光单位(rfu)数。信号比率可以通过将每个限制性基因座的峰高除以对照基因座的峰高来计算。
在一些实施方案中,计算dna样品中的限制性基因座和对照基因座的扩增产物的信号强度之间的比率包括:(i)确定限制性基因座的扩增产物的信号强度;(ii)确定对照基因座的扩增产物的信号强度;以及(iii)计算两种信号强度之间的比率。
在一些实施方案中,计算dna样品中的限制性基因座和对照基因座的扩增产物的信号强度之间的比率包括:确定每种基因座的cq,以及计算对照基因座的cq和限制性基因座的cq之间的差异。在一些实施方案中,该计算还包括应用下式:2^(对照基因座的cq–限制性基因座的cq)。
在一些实施方案中,计算信号比率可以是计算每种限制性基因座和对照基因座之间的多于一个信号比率。
在一些实施方案中,计算信号比率可以是计算seqidno:1中列出的限制性基因座和对照基因座之间的比率。
在一些实施方案中,扩增seqidno:1-15中列出的基因座中的多于一种基因座,其中方法包括计算seqidno:1-15中列出的基因座的每一种和对照基因座之间的比率,例如seqidno:1中列出的基因座和对照基因座之间的比率、seqidno:2中列出的基因座和对照基因座之间的比率等。
在一些实施方案中,可以使用计算机软件来计算扩增产物的信号强度之间的比率。
参考比率
术语“参考比率”或“参考信号比率”可互换使用,并且指在来自已知来源的dna中确定的信号强度比率。给定的一对限制性基因座和对照基因座的参考比率可以以很多种方式表示。在一些实施方案中,给定的一对基因座的参考比率可以是信号比率。在一些实施方案中,给定的一对基因座的参考比率可以是从来自已知来源的大的dna样品组获得的统计值,诸如大的参考比率组的平均值,例如,在大的癌症患者组中确定的平均值或在大的健康个体组中确定的平均值。
在其他实施方案中,给定的一对基因座的参考比率可以是多于一种比率,诸如在来自已知来源的大的dna样品组中确定的该对基因座的比率分布。在一些实施方案中,参考比率可以是参考标度。
在一些实施方案中,给定的一对基因座的参考标度可以包括在来自相同参考来源的多于一个dna样品中测量的该对基因座的信号比率。例如,参考膀胱癌患者的参考标度或参考健康个体的参考标度。在其他实施方案中,给定的一对基因座的参考标度可以包括来自健康个体和患病个体两者的信号比率,即组合了来自两种来源的参考比率的信号标度。通常,当使用信号标度时,分布值以使得来自健康个体的值在标度的一端,例如低于截断(cutoff),而来自癌症患者的值在标度的另一端,例如高于截断。在一些实施方案中,可以将计算的来自已知来源的测试dna样品的信号比率与健康和癌症参考比率的参考标度相比较,并且概率评分可以是基于计算的信号比率在标度内的相对位置分配至该计算的信号比率的评分。在一些实施方案中,计算的信号比率越高,分配至其的评分越高,并且相应地,关于膀胱癌的概率高。
术语“膀胱癌参考比率”或“膀胱癌dna中的参考比率”可互换地指在来自膀胱癌患者的尿样品的dna中测量的给定的限制性基因座和给定的对照基因座之间的信号强度比率。膀胱癌参考比率代表膀胱癌dna,即来自癌性膀胱组织的dna中的信号强度比率。膀胱癌参考比率可以是单个比率、统计值或多于一个比率(例如,分布),如以上详述的。
术语“健康参考比率”、“正常参考比率”或“健康dna中的参考比率”可互换地指在来自正常个体的尿样品中测量的给定的限制性基因座和给定的对照基因座之间的信号强度比率。在本文中,“健康”或“正常”个体被定义为无可检测的膀胱疾病或症状、包括通过常规诊断方法确定的膀胱癌在内的膀胱相关疾病的个体。健康参考比率代表正常膀胱dna,即来自正常、非癌性膀胱组织的dna中的信号强度比率。健康参考比率可以是单个比率、统计值或多于一个比率(例如,分布),如以上详述的。
在一些实施方案中,本文公开的方法包括预先确定来自癌性膀胱dna的参考比率。在一些实施方案中,本发明的方法包括预先确定来自正常膀胱dna的参考比率。
如以上所述的,信号比率可以通过多种方法确定,包括例如在毛细管电泳后测量峰或在rt-pcr后计算cq。要理解的是,利用本文公开的方法获得参考比率以及测量的未知来源的测试样品的比率以确定膀胱癌。
确定膀胱癌
在一些实施方案中,本文公开的方法基于与参考比率相比评价计算的来自未知来源的尿样品的dna的信号比率,以鉴定膀胱癌。
在一些实施方案中,计算的信号比率指示dna是膀胱癌dna。
本领域技术人员将领会到,计算的测试样品的信号比率与相应的参考信号比率的比较可以利用多种统计手段以很多种方式进行。
在一些实施方案中,将计算的给定的一对基因座的测试信号比率与参考信号比率进行比较包括将测试信号比率与单个参考值比较。单个参考值可以对应于获得的来自大的癌症患者或健康个体群体的参考信号比率的平均值。在其他实施方案中,将计算的给定的一对基因座的测试信号比率与参考信号比率进行比较包括将测试信号比率与多于一个参考信号比率的分布或标度进行比较。
可以采用已知的统计手段,以确定计算的给定的限制性基因座和对照基因座之间的信号比率是对应于膀胱癌参考比率还是对应于正常参考比率。在一些实施方案中,检测到计算的比率与膀胱癌参考比率紧密接近将受试者鉴定为患有膀胱癌的受试者。相反,在一些实施方案中,检测到计算的比率与正常参考比率紧密接近则将受试者鉴定为未患有膀胱癌的受试者。
在一些实施方案中,方法包括将计算的信号比率与其相应的膀胱癌参考比率(即,与确定的膀胱癌中的同一对基因座的信号比率)进行比较,以获得反映计算的信号比率是膀胱癌比率的可能性的概率评分。计算的信号比率与参考比率越接近,概率评分并且相应地计算的信号比率是膀胱癌比率的可能性越高。在一些实施方案中,概率评分基于计算的信号比率在膀胱癌参考比率的分布内的相对位置。
在一些实施方案中,方法包括将计算的多于一种限制性基因座相对于对照基因座的多于一种信号比率与其相应的膀胱癌参考比率比较。
在一些实施方案中,信号比率的模式可以利用统计手段和计算机化的算法来分析,以确定其代表膀胱癌的模式还是正常、健康模式。例如在属于本发明的申请人的wo2011/070441中公开了示例性算法。算法可以包括但不限于机器学习和模式识别算法。
在一些示例性实施方案中,可以将每个计算的比率(针对每对限制性基因座和对照基因座),与从来自癌症患者和未罹患癌症的个体两者的大的尿样品组生成的该对的参考比率的标度进行比较。标度可以代表在来自癌症患者和正常个体的大量样品中计算的限制性基因座和对照基因座对之间的信号比率。标度可以呈现出阈值(在后文也被称为‘截断’或‘预先定义的阈值’),高于阈值的部分为对应于膀胱癌的参考比率并且低于阈值的部分为对应于健康个体的参考比率,或颠倒过来。
在一些实施方案中,在标度底部和/或低于截断的较低的比率可以来自正常个体(健康,即未罹患膀胱癌)的样品,而在标度上部和/或高于预先确定的截断的较高的比率可以来自癌症患者。对于每个比率(每种限制性基因座和对照基因座之间的比率),可以基于其在标度上的相对位置给出评分,并且将每种基因座的个体评分组合来给出单个评分。在一些实施方案中,可以对个体评分求和来给出单个评分。在其他实施方案中,可以对个体评分求平均值来给出单个评分。在一些实施方案中,可以使用单个评分来确定受试者是否患有癌症,其中高于预先定义的阈值的评分指示膀胱癌。
在一些实施方案中,评分是0-100之间的数,其反映计算的信号比率是膀胱癌比率的概率,其中0为最低的概率并且100是最高的概率。在一些实施方案中,阈值评分被确定,其中等于或高于该阈值评分的评分指示膀胱癌。阈值可以是例如40、50、60或70。每种可能性代表本发明的单独实施方案。
在另外的示例性实施方案中,对于每个计算的比率(每种限制性基因座和对照基因座之间的比率),其代表膀胱癌dna的概率可以基于与相应的膀胱癌参考比率和正常参考比率的比较来确定,并且可以指定评分(概率评分)。因此,组合计算的每个比率(针对每种基因座)的个体概率评分(例如对其求和或求平均值),以给出组合评分。组合评分可以用于确定受试者是否患有癌症,其中高于预先定义的阈值的组合评分指示膀胱癌。
因此,在一些实施方案中,确定阈值或截断评分,高于(或低于)该阈值或截断评分,受试者被鉴定为患有膀胱癌。阈值评分将健康受试者群体与非健康受试者群体区分开来。
在一些实施方案中,本发明的方法包括提供阈值评分。
在一些实施方案中,确定阈值评分包括在健康或患有膀胱癌的大的受试者群体中测量信号比率。
在一些实施方案中,阈值为在统计学上显著的值。统计学显著性通常通过比较两个或更多个群体,并确定置信区间(ci)和/或p值来确定。在一些实施方案中,统计学上显著的值指置信区间(ci)为约90%、95%、97.5%、98%、99%、99.5%、99.9%及99.99%,同时优选的p值为小于约0.1、0.05、0.025、0.02、0.01、0.005、0.001或小于0.0001。每种可能性代表本发明的单独实施方案。根据一些实施方案,阈值评分的p值为至多0.05。
如本文使用的,当提及可测量的值时术语“约”意指包括从指定的值的+/-10%、更优选地+/-5%、甚至更优选地+/-1%、并且仍然更优选地+/-0.1%的变化。
在一些实施方案中,方法还包括将计算的给定的限制性基因座和对照基因座之间的信号比率与其相应的正常膀胱参考比率进行比较,以获得概率评分,其中检测到所述比率相对于相应的健康参考比率的低概率评分指示受试者患有膀胱癌。
在一些实施方案中,本文公开的方法的灵敏度可以是至少约75%。在一些实施方案中,方法的灵敏度可以是至少约80%。在一些实施方案中,方法的灵敏度可以是至少约85%。在一些实施方案中,方法的灵敏度可以是至少约90%。
在一些实施方案中,如本文使用的诊断测定的“灵敏度”指测试为阳性的患病个体的百分比(“真阳性”的百分比)。相应地,未被测定检测出的患病个体为“假阴性”。未患病并且在测定中测试为阴性的受试者被称为“真阴性”。诊断测定的“特异性”为一(1)减去假阳性率,其中“假阳性”率被定义为无疾病、测试为阳性的那些受试者的比例。尽管特定的诊断方法可能不提供状况的最终诊断,但是如果该方法提供了有助于诊断的阳性指示,这就足够了。
在一些实施方案中,本文公开的方法的特异性可以是至少约65%。在一些实施方案中,方法的特异性可以是至少约70%。在一些实施方案中,方法的特异性可以是至少约75%。在一些实施方案中,方法的特异性可以是至少约80%。
膀胱癌治疗
在一些实施方案中,提供了用于治疗膀胱癌的方法,所述方法包括根据本发明的方法鉴定膀胱癌,以及向所述受试者施用抗癌疗法。
用于治疗膀胱癌的方法可以包括以下的一种或更多种:外科手术,以去除肿瘤、膀胱的部分或在更严重的情况中整个膀胱;放射疗法(外部或内部);化学疗法(全身的或局部的);以及免疫疗法,如本领域已知的。每种可能性代表本发明的单独实施方案。
试剂盒
在一些实施方案中,提供了一种用于鉴定人受试者中的膀胱癌的试剂盒。在一些实施方案中,试剂盒用于根据本发明的方法鉴定膀胱癌。在一些实施方案中,试剂盒包括:至少一种甲基化敏感性限制性酶;用于扩增至少一种限制性基因座和至少一种对照基因座的引物对;用于检测至少一种限制性基因座和至少一种对照基因座的扩增产物的工具;以及用于进行膀胱癌的鉴定的说明手册。在一些实施方案中,说明手册可以是电子说明手册。
在一些实施方案中,说明手册可以提供膀胱癌参考比率以及任选地健康参考比率。
在一些实施方案中,说明手册可以包括用于进行以上描述的方法步骤的说明。
在一些实施方案中,说明手册可以包括指导信号比率和膀胱癌参考比率以及任选地与健康参考比率之间的关联性的说明。
在一些实施方案中,说明手册可以提供用于计算标志物评分,即给定的限制性基因座的评分的说明。在一些实施方案中,说明手册可以提供用于计算多于一个标志物评分的总评分的说明。在一些实施方案中,说明手册可以提供用于确定膀胱癌的阈值评分,高于该阈值评分,受试者被鉴定为患有癌症。在其他实施方案中,说明手册可以提供用于确定膀胱癌的阈值评分,低于该阈值评分,受试者被鉴定为患有癌症。
在一些实施方案中,试剂盒包括单种甲基化敏感性内切核酸酶。在一些实施方案中,甲基化敏感性内切核酸酶是hhai。
在一些实施方案中,试剂盒包括单种甲基化敏感性内切核酸酶。在一些实施方案中,甲基化敏感性内切核酸酶是hinp1i。
在一些实施方案中,试剂盒还可以包括计算机软件。在一些实施方案中,计算机软件可以是计算信号强度、信号比率和标志物评分中的至少一种的计算机软件。
在一些实施方案中,试剂盒包括:hhai;与如本文描述的至少一种限制性基因座和至少一种对照基因座互补的引物对;以及与至少一种限制性基因座和至少一种对照基因座互补的荧光多核苷酸探针。
在一些实施方案中,试剂盒包括:hinp1i;与如本文描述的至少一种限制性基因座和至少一种对照基因座互补的引物对;以及与至少一种限制性基因座和至少一种对照基因座互补的荧光多核苷酸探针。
用于扩增seqidno:1-15中列出的限制性基因座的示例性引物在如下的seqidno:25-155中列出(正向(for)=正向(forward),反向(rev)=反向(reverse)):
基因座1正向:seqidno:25-29;基因座1反向:seqidno:30-35
基因座2正向:seqidno:36-38;基因座2反向:seqidno:39-42
基因座3正向:seqidno:43-51;基因座3反向:seqidno:52-61
基因座4正向:seqidno:62-64;基因座4反向:seqidno:65-67
基因座5正向:seqidno:68-70;基因座5反向:seqidno:71-73
基因座6正向:seqidno:74-76;基因座6反向:seqidno:77-79
基因座7正向:seqidno:80-85;基因座7反向:seqidno:86-89
基因座8正向:seqidno:90-92;基因座8反向:seqidno:93-95
基因座9正向:seqidno:96-99;基因座9反向:seqidno:100-102
基因座10正向:seqidno:103-106;基因座10反向:seqidno:107-112
基因座11正向:seqidno:113-116;基因座11反向:seqidno:117-120
基因座12正向:seqidno:121-125;基因座12反向:seqidno:126-130
基因座13正向:seqidno:131-135;基因座13反向:seqidno:136-140
基因座14正向:seqidno:141-145;基因座14反向:seqidno:146-151
基因座15正向:seqidno:152-153;基因座15反向:seqidno:154-155。
用于扩增seqidno:16中列出的对照基因座的示例性引物在如下seqidno:156-167中列出:
对照正向:seqidno:156-161;
对照反向:seqidno:162-167。
在一些实施方案中,试剂盒包括第一对照构建体和第二对照构建体中的至少一种,如以上描述的,每种对照构建体包含非人/人工dna序列。在一些实施方案中,试剂盒包括如以上描述的第一对照构建体和第二对照构建体两者。
在一些实施方案中,试剂盒包括填充有至少一种核苷酸引物对的一个或更多个容器。在一些实施方案中,本发明的试剂盒中包括的每种核苷酸引物对可以包括与选自seqidno:1-15中列出的限制性基因座的限制性基因座内的子序列或与其侧翼序列互补的引物,其中所述每种核苷酸引物对被设计为选择性地扩增基因组的包含该基因座的片段。
在一些实施方案中,试剂盒可以包括用于选择性地扩增以上描述的基因座的组合的引物对。
在一些实施方案中,试剂盒还可以包括用于选择性地扩增第一人工对照构建体和第二人工对照构建体的核苷酸引物对。
在一些实施方案中,试剂盒还可以包括寡核苷酸探针,该寡核苷酸探针用于检测使用试剂盒中的引物扩增的基因座的扩增产物。每种寡核苷酸探针可以与基因座内的子序列互补,并且可以能够与其杂交。在一些实施方案中,寡核苷酸探针可以是荧光标记的。
在一些实施方案中,试剂盒还可以包括dna消化、基因座扩增和扩增产物的检测所需要的至少一种另外的成分,诸如dna聚合酶和核苷酸混合物。
在一些实施方案中,试剂盒还可以包括用于消化和扩增的合适的反应缓冲液,以及用于进行膀胱癌鉴定的书面方案。书面方案可以包括用于进行本文公开的任一步骤的说明,包括但不限于dna消化参数、pcr循环参数、信号比率分析以及与参考比率的比较。
以下实施例被展示以更充分地说明本发明的某些实施方案。但是,它们绝不应以任何方式被解释为限制本发明的广泛的范围。本领域技术人员可以容易地设想本文公开的原理的许多变化和修改,而不背离本发明的范围。
实施例
实施例1-用于检测膀胱癌的基因组基因座
十五(15)个人的基因组基因座被鉴定为与正常组织相比在膀胱癌组织中具有增加的甲基化(下文表1,seqidno:1-15)。发现这些基因座使得能够区分来自膀胱癌患者的dna和来自正常个体(未罹患膀胱癌)的dna。
表1-基因组基因座
*描述指在hg18基因组构造(build)上的位置
实施例2-测试基因组基因座的组
从69名膀胱癌患者和未罹患膀胱癌的40名健康受试者获取尿样品。膀胱癌患者群体包括55名男性和14名女性,年龄在从41-92岁的范围(平均72岁)。所有69名癌症患者为tcc(移行细胞癌),阶段分布为:ta(非侵袭性乳头状癌)-42名患者,t1-10名患者,t2-10名患者,cis(原位癌)-8名患者。健康群体包括33名男性和7名女性,年龄在从55-88岁的范围(平均70岁)。使用qiaamp血液迷你试剂盒(qiaampbloodminikit;qiagen,hilden,germany)从所有尿样品提取dna。
如图2中概述的处理每个样品中的dna。简而言之,使从每个样品提取的dna经历用甲基化敏感性限制性内切核酸酶(hhai或hinp1i)消化。消化反应物(总体积120微升)在消化缓冲液中包括所有的提取的dna(未定量)和hhai或hinp1i。在37℃进行消化持续2小时。
接下来,对消化的dna样品进行定量实时(rt)pcr,以在每个dna样品中扩增以上表1中详述的15种限制性基因座和如seqidno:16中列出的对照基因座(参见表1)。对照基因座是不含hhai或hinp1i的识别序列并且不管其甲基化状态如何当用限制性酶消化dna样品时保持完整的基因座。该对照基因座(也被称为内部参考基因座)具有在用hhai或用hinp1i消化后不受甲基化影响的扩增模式。
具体地,将每个消化的dna样品分成八(8)个包含10微升消化的dna的等份。将7个等份补充有用于扩增15个限制性基因座中的2个限制性基因座和对照基因座(对照基因座将在每个等份中都被扩增)的引物对。将第8个等份补充有用于扩增剩余的1个限制性基因座和对照基因座的引物对。为在75至225个碱基之间的扩增子被扩增,每个扩增子包含16个基因座中的一个连同为5-80个碱基的5’和3’侧翼序列。
每个扩增反应物(总体积25微升)还包含dntp和反应缓冲液。为了使得能够在扩增期间检测扩增产物,向反应物添加荧光标记的多核苷酸探针(每种基因座一种)。使用以下荧光标记物:fam、joe、rox和cy5。在abi7500fastdx仪器中用以下pcr程序进行rt-pcr反应:95℃持续10分钟的酶的初始活化,然后是以下的45个循环:在95℃,15秒;然后在60℃,1分钟。
rt-pcr后,对将来自探针的荧光信号的变化显示为循环数的函数的定量pcr曲线进行分析,以计算每种基因座的定量循环(cq)。
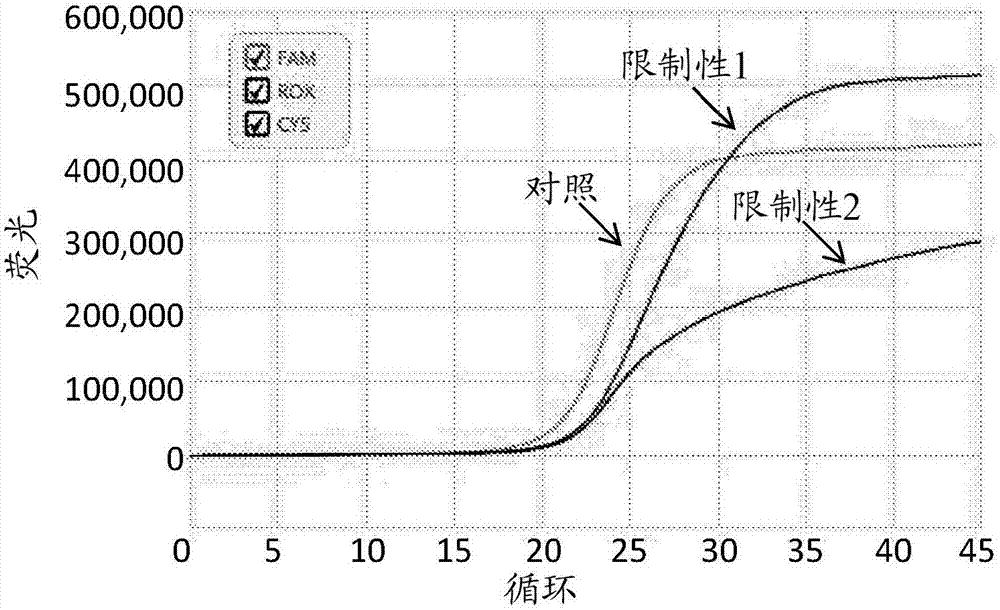
图1a和1b显示了来自癌症患者(图1b)和健康受试者(图1a)的样品中的对照基因座和两种限制性基因座的示例性定量pcr曲线。在来自癌症患者的样品中,其中限制性基因座具有高甲基化水平并因此在用hhai消化时几乎保持完整,两种限制性基因座以高效率被扩增,即它们比对照基因座(其根本未被切割)晚大约1个循环上升。在健康样品中,其中限制性基因座的特征为低甲基化水平并因此当用hhai消化时被彻底切割,扩增效率比对照基因座的扩增效率低的多。如可从图获知的,在癌症患者和健康受试者之间限制性基因座的cq值不同。
对于每个样品,如下计算限制性基因座1-15(seqidno:1-15)的每一种的信号强度和对照基因座(seqidno:16)的信号强度之间的比率:确定每种限制性基因座和对照基因座的cq。cq值被用于下式:
2(对照基因座的cq–限制性基因座的cq)。
要理解的是,计算是针对在同一等份中共扩增的限制性基因座和对照基因座进行的。
获得的每种限制性基因座相对于对照基因座的数值代表该限制性基因座和对照基因座之间的信号比率。
总而言之,计算每个样品的15个比率(即,根据上式的15个计算结果)。表2总结了在癌症组中以及在对照组中每种基因座相对于对照基因座的平均比率和标准偏差(std)。如可从表中看出的,获得的从膀胱癌样品提取的dna的比率与获得的从健康(无膀胱癌)志愿者的样品提取的dna的比率显著不同。具体地,与对照组相比,在癌症组中观察到每个基因座的显著较高的比率(高至少一个数量级),突显了使用这些基因座区分膀胱癌患者的尿样品和正常/健康个体的那些尿样品的优势。
表2-信号比率
接下来,计算seqidno:1中列出的基因座以及基因座的若干组合或亚组关于膀胱癌的鉴定的灵敏度、特异性和roc曲线下面积(auc)。数据被总结于表3。
表3-灵敏度、特异性和auc
实施例3-具有膀胱癌史的患者的诊断
从经历常规监测的具有膀胱癌史的222名患者获得排泄的尿样品。40名患者(18%)的亚组具有活组织检查证实的复发性膀胱癌。阶段的分布为:16-ta、13-t1、3-t2和12-cis并且级别分布为19-lg和26-hg。要注意的是,分阶段的样品数目以及分级别的样品数目相加超过40,因为一些样品表现出多于一个阶段/级别(例如在同一患者中,t1阶段的肿瘤伴随cis)。
如以上实施例2中描述的提取、消化并扩增dna。
对于每个样品,计算限制性基因座1-15(seqidno:1-15)的每一种的信号强度和对照基因座(seqidno:16)的信号强度之间的比率,如以上描述的。
对于患有lg和hg肿瘤的患者,相对于甲基化比率的参考组(最高为100并且最低为0)归一化的平均甲基化比率,也被称为“epi评分”,分别为69和85,因此与肿瘤级别显著地相关联(p=0.006)。
在表4中列出了每个阶段的平均epi评分。使用kolmogorov-smirnov检验的统计分析显示出了ta和t2epi评分之间(p=0.0077)以及ta和cis之间(p=0.0155)的显著统计学差异。
表4-epi评分分析
总灵敏度和特异性分别为90%和83%(图3a和3b)。在图4a和4b中分别显示了每个阶段和级别的灵敏度。该群组研究的阴性预测值为97.4%。
在这些病例中的173例中,细胞学结果是可用的。细胞学表现导致40%的灵敏度和96%的特异性。
173个样品中的15个来自患有肿瘤的患者。通过细胞学检测出的所有六(6)个肿瘤通过本文公开的方法也被检测出,并且被该方法漏掉的两(2)个肿瘤也被细胞学漏掉。出乎意料地,七(7)个肿瘤仅由本发明的方法检测出。
该结果表明,本文公开的方法是用于在排泄的尿样品中检测膀胱癌的灵敏且特异的不依赖操作者的测定。此外,与细胞学相比,本文公开的方法具有显著较高的灵敏度和略微较低的特异性。因此,本发明的方法可以在膀胱癌患者的监测中良好地替代尿细胞学,而没有漏掉复发例的风险。
以上具体实施方案的描述将如此完全地揭示本发明的一般性质,使得其他人可以通过应用现有的知识,容易地为各种应用修改和/或调整此类具体实施方案而不需要过度实验且不背离一般概念,且因此,此类调整和修改应当并且预期被包含在本公开的实施方案的等效方式的含义和范围内。应理解的是,本文采用的措辞或术语是为了描述而非限制的目的。用于进行多种公开的化学结构和功能的手段、材料和步骤可采取多种替代形式而不背离本发明。
序列表
<110>纽克莱克斯有限公司
<120>用于诊断膀胱癌的方法
<130>nclx/006us
<150>14/792270
<151>2015-07-06
<160>167
<170>patentinversion3.5
<210>1
<211>60
<212>dna
<213>智人(homosapiens)
<400>1
tttttaggggtctgcgcgctctcgcctggctgctgggaaggagggctgggaggcgctgaa60
<210>2
<211>140
<212>dna
<213>智人(homosapiens)
<400>2
tccaggcgcgccgaccgctcaagcgctccaggtccacccggcggagggcagagaaagcgc60
gaccgcgcggcccgcagggttgcaagaagaaaacgagtgttatataatgagtctcagtgg120
ttgctcacaatgccaggcgc140
<210>3
<211>60
<212>dna
<213>智人(homosapiens)
<400>3
ggctgataccagagaggacccggagcgcgaaccagaggttcgacctccagggcagcgcag60
<210>4
<211>130
<212>dna
<213>智人(homosapiens)
<400>4
ggggcgccaaatgcccacgtgttgatgaaaccagtgagatgggaacaggcggcgggaaac60
cagacagaggaagagctagggaggagaccccagccccggatcctgggtcgccagggtttt120
ccgcgcgcat130
<210>5
<211>70
<212>dna
<213>智人(homosapiens)
<400>5
tctgagaagtgtcctcctcgctctcttataaaaacaggacttgttgccgaggtcagcgcg60
cgcatcgagt70
<210>6
<211>130
<212>dna
<213>智人(homosapiens)
<400>6
atcagcgcgaactattcgtttagtggccttaaaacaccctggtttcaccctcagctattt60
tcaagttcccgtgtgcctggcactttctccgtgcgagaagcaccggagggtgcggacgcg120
ccacagtctg130
<210>7
<211>150
<212>dna
<213>智人(homosapiens)
<400>7
tgctctgcgcagcgtggagggcaacgggactgggaggcggcttctgccgccgggcactcg60
cttccaggtggcttacgaggattcagactgtgggaaccgtgcgctcttcagcttggtgga120
ccgcgtggcggcggcgcggggcgccaagcc150
<210>8
<211>70
<212>dna
<213>智人(homosapiens)
<400>8
cgcgcagaactttgcggtggcgcttagcgcctcctttgcccagacccttcccgcctttgc60
cgcgcccaga70
<210>9
<211>70
<212>dna
<213>智人(homosapiens)
<400>9
cgtaatcgccggtgtaactcatgttggctggggggcctcccggcgcgcgcggagaggctg60
gggtgcgccc70
<210>10
<211>60
<212>dna
<213>智人(homosapiens)
<400>10
cgattcagagggccccggtcggagctgtcggagattgagcgcgcgcggtcccgggatctc60
<210>11
<211>140
<212>dna
<213>智人(homosapiens)
<400>11
cgcgcggcagccctccgtgcgcgcaggctcgggtgcgttgttcgcgggggtgaattgtga60
agaaccatcgcggggtccttcctgctgaggccgcggacaccgtgacctcgctgctctggg120
tctgcagggaaacgtaggaa140
<210>12
<211>100
<212>dna
<213>智人(homosapiens)
<400>12
tgacctttaggggctgttactctcagaccaggcccagcagcacccggcgcatttacgtcg60
gatctgacccctgcaagcaccggcgcgaccgcgctagcgg100
<210>13
<211>70
<212>dna
<213>智人(homosapiens)
<400>13
agaacttcgtgggcaggtaagcgcgcagggcgcggagtaaggatgcggcagtggggcgac60
cccgctgcgg70
<210>14
<211>70
<212>dna
<213>智人(homosapiens)
<400>14
ctgcctgcgctcactacgggttttttagttggcgccttaatgtttgtaacacttttagag60
cgcttgctct70
<210>15
<211>140
<212>dna
<213>智人(homosapiens)
<400>15
ggccgggaagcgcggggtgagaaagcgaggtgggtggcgagagcgtgagcgcccctctgc60
tgaccccggggagcgtggactacgagttggcgcccaagtccagaatccgcgcgcaccgcg120
gtaagctgcgccttttgaaa140
<210>16
<211>60
<212>dna
<213>智人(homosapiens)
<400>16
agactaacttttctcttgtacagaatcatcaggctaaatttttggcattatttcagtcct60
<210>17
<211>80
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>对照构建体
<400>17
aagtctcgagtcgatgtcactaaccatgagtattccatgtgccgtcatctaacaagtact60
gcataccctggataatggct80
<210>18
<211>100
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>对照构建体
<400>18
taccaggtctacaaagctcgactctgcctgcgctcactacgggttttttagttggcgcct60
taatgtttgtaacacttttagagcgcttgctctgatcctc100
<210>19
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>19
aagtctcgagtcgatgtcac20
<210>20
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>20
agccattatccagggtatgc20
<210>21
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>探针
<400>21
accatgagtattccatgtgccgt23
<210>22
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>22
taccaggtctacaaagctcgac22
<210>23
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>23
acgcctactaatacacatcggag23
<210>24
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>探针
<400>24
cgctcactacgggttttttagttgg25
<210>25
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>25
cagacccggttaacactgaag21
<210>26
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>26
gacccggttaacactgaagttgc23
<210>27
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>27
ccagacccggttaacactga20
<210>28
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>28
ccggttaacactgaagttgctt22
<210>29
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>29
tccagacccggttaacactga21
<210>30
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>30
ctcagatctgccctcgcttc20
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>31
ctcagatctgccctcgcttc20
<210>32
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>32
gcaactcctacagacactcagat23
<210>33
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>33
gacactcagatctgccctcg20
<210>34
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>34
atcagaacaattagagagtaatgat25
<210>35
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>35
cagatctgccctcgcttca19
<210>36
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>36
ggtccagcagctagtccact20
<210>37
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>37
ctccctggtccagcagctag20
<210>38
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>38
cccgcctggctgctccag18
<210>39
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>39
caccaggtgcaaaagatgcct21
<210>40
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>40
cgggaagggaaaggccacat20
<210>41
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>41
aaggccacatcttcacgcctt21
<210>42
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>42
tgagcaaccactgagactcatt22
<210>43
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>43
cgtcctggagctccgactc19
<210>44
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>44
ccgactcccaccccatctg19
<210>45
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>45
ataccagagaggacccggag20
<210>46
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>46
gaaccagaggttcgacctcca21
<210>47
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>47
cccggcttcggagcgggaa19
<210>48
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>48
cagtttcaccgctcgatcttg21
<210>49
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>49
cgagtccagggcgaggtaag20
<210>50
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>50
gaggctgataccagagaggac21
<210>51
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>51
cacaactgcctgcgagaaaca21
<210>52
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>52
agcttgtggcgaccccgtc19
<210>53
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>53
tagctcatcccgtccagcttg21
<210>54
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>54
cgcctcagccactcctggg19
<210>55
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>55
ggccttggagcccaaaccag20
<210>56
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>56
gctcgggcagagccaagga19
<210>57
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>57
tcttgtgtgggtgccttgga20
<210>58
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>58
ctggaggtcgaacctctggttc22
<210>59
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>59
cgctccgaagccggggtac19
<210>60
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>60
ggtgggagtcggagctccag20
<210>61
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>61
ggagcgacccaaatgtggag20
<210>62
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>62
cctgggcatcgtttacatttgg22
<210>63
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>63
gcatcgtttacatttggggc20
<210>64
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>64
gagtagagcaccaagatagtg21
<210>65
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>65
gcagccgcaccttttgggat20
<210>66
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>66
gggatgggatggtttggaga20
<210>67
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>67
gagtctaacaaactaacctgatg23
<210>68
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>68
acaggacttgttgccgaggtc21
<210>69
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>69
atggacggaaataagcaaaagca23
<210>70
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>70
gcaaaagcaaaacaaccccttc22
<210>71
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>71
ttccgtgaaagcaatgacacag22
<210>72
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>72
aataaaacaaccgctgccgaca22
<210>73
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>73
caatgacacagcagaaaccac21
<210>74
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>74
gccaggcactcgctggatct20
<210>75
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>75
ccgccatcgataacagatcag21
<210>76
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>76
ggcaccgccatcgataacag20
<210>77
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>77
cccctaaacttagccagttcg21
<210>78
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>78
tggcaggaacatgaataataaatg24
<210>79
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>79
ggcggcggctcagactgtg19
<210>80
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>80
ggcttacgaggattcagactg21
<210>81
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>81
ggactgggaggcggcttct19
<210>82
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>82
cgtggagggcaacgggact19
<210>83
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>83
cactcgcttccaggtggctt20
<210>84
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>84
cggccatcgagtatgctctg20
<210>85
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>85
gatgactcgtacttgttttcact23
<210>86
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>86
cccaggataaggtctggcttg21
<210>87
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>87
ctgctgcatactcgcacactg21
<210>88
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>88
cacactggccccaggataag20
<210>89
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>89
gatgcaagccgggccactg19
<210>90
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>90
ggtgatgccctgaatgttgtc21
<210>91
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>91
cttaacgcctccacgccgt19
<210>92
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>92
ccgtgccaggcgtcggat18
<210>93
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>93
tttgtttagtacaagacatgtctg24
<210>94
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>94
cgtgttgcattttgtttagtaca23
<210>95
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>95
gctgcgaggcttgccgtgt19
<210>96
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>96
actcggtgttctcaccgaaag21
<210>97
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>97
acgtaatcgccggtgtaactc21
<210>98
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>98
cctttctttgtagccacctcag22
<210>99
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>99
ggaagcaacagatcgtcactc21
<210>100
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>100
ttgagcacaagcatgctgcatg22
<210>101
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>101
ccctctgcacactcgagaac20
<210>102
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>102
ggacgagagttgagctctcac21
<210>103
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>103
aggttgcagtgagccggaatg21
<210>104
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>104
aataccatatagtgaacacctaaga25
<210>105
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>105
ccaatttttgtgtttttagtagag24
<210>106
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>106
ggatccagggcgattcagag20
<210>107
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>107
cagggcctcgtcggagatc19
<210>108
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>108
tcgtcggagatcccgggac19
<210>109
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>109
tgctgcccagcttcttccaca21
<210>110
<211>17
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>110
ggtcccgcggcctccag17
<210>111
<211>17
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>111
gcagcttcgccgcccgg17
<210>112
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>112
cggaccggccaggggtcc18
<210>113
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>113
caggacccgctcagttccac20
<210>114
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>114
cagggatgtgagtgggcgg19
<210>115
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>115
gaagagggccgcaaaccaac20
<210>116
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>116
acccgccgcactgacaggt19
<210>117
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>117
tttcctacgtttccctgcagac22
<210>118
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>118
atcctgcccgctcctgacaa20
<210>119
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>119
ccagcaccaggagcgtgttc20
<210>120
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>120
cgcgatggttcttcacaattca22
<210>121
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>121
acgcgacgctctaaaggaagt21
<210>122
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>122
ctctcagaccaggcccagca20
<210>123
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>123
gctgaggcaaagcgactcca20
<210>124
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>124
ctctaaaggaagtgacctttag22
<210>125
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>125
ggctgttactctcagaccag20
<210>126
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>126
aacctaccgactgacctgcat21
<210>127
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>127
cctgcatgacgtaggtccact21
<210>128
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>128
gggtcagatccgacgtaaag20
<210>129
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>129
gcgtgttcccagccgctag19
<210>130
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>130
gggattggccgagtcaggtc20
<210>131
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>131
acagagctgcctgctggagc20
<210>132
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>132
cggcaagagctcggtgctc19
<210>133
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>133
agaacttcgtgggcaggtaag21
<210>134
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>134
cgtctgcaggaccgcgttttc21
<210>135
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>135
agctgatcccgctggtgaa19
<210>136
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>136
aatggctttgtatcgcacgatg22
<210>137
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>137
ccactgccgcatccttactc20
<210>138
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>138
cctggtccatccaccctctg20
<210>139
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>139
tgcccgtccacgttccaac19
<210>140
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>140
cacggacaccgccactctg19
<210>141
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>141
gagcattggcacccctgattc21
<210>142
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>142
cttacccgtggtttctgcctg21
<210>143
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>143
agagcattggcacccctgatt21
<210>144
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>144
acttctcttacccgtggtttct22
<210>145
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>145
ctggggcctacgcgaagct19
<210>146
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>146
ctctaaaagtgttacaaacattaag25
<210>147
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>147
tggaatcgtgcccggatcag20
<210>148
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>148
tttttggaacgttaaataatttccta26
<210>149
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>149
cctcagtttcctcaaaaggaat22
<210>150
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>150
cgtgcccggatcagagcaag20
<210>151
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>151
gcctggaatcgtgcccggat20
<210>152
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>152
tccaaggaagactaaaaacccag23
<210>153
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>153
taaaaacccaggccgggaagc21
<210>154
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>154
tggtttgttccaaggagtacag22
<210>155
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>155
agtacagatagccttttcaaaag23
<210>156
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>156
agcaaggtgaagactaacttttc23
<210>157
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>157
cttgtacagaatcatcaggctaa23
<210>158
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>158
gtccttggagacatctgagagattc25
<210>159
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>159
gacatctgagagattccggg20
<210>160
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>160
caaagaaagcaaggtgaagact22
<210>161
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>161
cagtccttggagacatctgagaga24
<210>162
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>162
tgaagtgaactattccttaggtg23
<210>163
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>163
gtctccaaggactgaaataatgc23
<210>164
<211>28
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>164
gagtagcaaaaaatagctgaagtgaact28
<210>165
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>165
tgacaaccaatgagtagcaaaaa23
<210>166
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>166
gttgtcagtgtggccagaga20
<210>167
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<223>引物
<400>167
tgttgtcagtgtggccagaga21
- 还没有人留言评论。精彩留言会获得点赞!