一种D-乳酸脱氢酶、含有该酶的工程菌株及其构建和应用的制作方法
本发明涉及基因工程领域,具体涉及一种d-乳酸脱氢酶,一种含有该酶的芽孢杆菌基因工程菌的构建以及在生产d-乳酸中的应用。
背景技术:
乳酸(lacticacid)是一种重要的工业原料,其可应用于医药、食品、化妆品和石化工业等;近年来,乳酸作为单体还被用来合成高强度生物可降解塑料聚乳酸(polylacticacid,pla)。传统来说,pla是用高光学纯的l-乳酸聚合成的,而使用混合型l和d-乳酸,可以明显提高以聚乳酸为材料产品的机械性能、热稳定性和抗水解性,由此极大激发了d-乳酸的市场需求。虽然乳酸能以石油为原料合成,但所合成的乳酸是两种同分异构体的混合物,不适合用于pla的生产。生产pla所需的高光学纯l和d-乳酸只能通过微生物发酵而产生,许多学者们也因此研究了利用微生物发酵法生产d-乳酸。
高温发酵可以最小化污染的风险,提高原料转化速率,并且能够降低供热成本等优点。到目前为止,高温生产光学纯l-乳酸已被广泛研究,这对于l-乳酸的商业化生产有着巨大的贡献。至于d-乳酸,人们一直渴望实现其高温发酵生产,但少有关于嗜热生产d-乳酸的报道,且报道的d-乳酸的浓度和生产速率及其丰富的培养基成分不适合工业化规模的生产。因此,很有必要开发一种低成本的、强健的微生物平台来高温高产d-乳酸。
烟酰胺腺嘌呤二核苷酸(nicotinamideadeninedinucleotide,nadh)依赖的d-乳酸脱氢酶(d-lactatedehydrogenase)是微生物合成d-乳酸的关键酶。然而,大多数天然存在的d-乳酸脱氢酶对热不稳定,这也是微生物高温生产d-乳酸的主要瓶颈之一。有报道在体外测定植物乳杆菌(lactobacillusplantarum)中d-乳酸脱氢酶酶活表明,该酶在42℃具有很高的酶活,但在50℃中孵育3分钟后就会完全失活,而该菌体内的l-乳酸脱氢酶用相同的方法处理后仍然保留94%的酶活。其他一些体内实验也表明d-乳酸脱氢酶在高温条件下也容易失活。总之,上述报道菌株d-乳酸脱氢酶的低活性,或者说极差的热稳定性阻碍了它们在高温条件下生产d-乳酸。为实现高温高效产d-乳酸,寻找一个热稳定性高的d-乳酸脱氢酶是一个重要的任务。
耐热地衣芽孢杆菌(bacilluslicheniformis)atcc14580是一株兼性厌氧、革兰氏阳性、内生孢子生成细菌。它具有许多作为微生物发酵平台的潜在优势:1)它能够利用各种五碳糖和六碳糖;2)它具有较快的细胞生长速率,从而可以缩短发酵周期;3)它可以进行遗传操作;4)它是食品和药物管理局公认的“大体上认为是安全的”菌株,这些优点表明菌株atcc14580可作为一株理想的平台菌株。目前,该菌株已经被证实可用于2,3-丁二醇的高温发酵生产。
因此,本领域的技术人员致力于以耐热地衣芽孢杆菌atcc14580为出发菌株,开发一种d-乳酸高温高产工程菌株及其制备和应用。
技术实现要素:
针对现有技术中大多数天然存在的d-乳酸脱氢酶热稳定性差,现有的生产d-乳酸的嗜热生产成本高、但d-乳酸浓度和生产速率不适合工业化规模生产的缺陷,本发明提供一种d-乳酸高温高产工程菌株,及该菌株的的制备和应用。本发明公开了一个具有独特性质的来自热脱硫杆菌属(thermodesulfatatorindicus)的d-乳酸脱氢酶,该酶具有非常好的嗜热性及热稳定性。对该酶进行大肠杆菌密码子使用优化后,利用优化后的酶和基因工程改造的方法,使得改造后的地衣芽孢杆菌的发酵产物从天然的产2,3-丁二醇重定向为高产光学纯d-乳酸。
本发明的一个方面提供了一种d-乳酸脱氢酶。在一个具体实施方式中,该d-乳酸脱氢酶具有下列氨基酸序列之一:
1)seqidno.1所示的氨基酸序列;
2)seqidno.1所示的氨基酸序列通过一个或几个氨基酸残基的取代、缺失、插入或添加衍生产生的氨基酸序列,该氨基酸序列产生的蛋白质具有d-乳酸脱氢酶活性;
3)seqidno.1所示的氨基酸序列被保守性替换的氨基酸序列;
4)包含与seqidno.1所示的氨基酸序列具有至少80%同源性的氨基酸序列。
进一步地,该氨基酸残基的取代、缺失、插入或添加发生于功能域之外,功能域包括d-乳酸脱氢酶的酶催化位点、配体结合位点和nad结合位点。
进一步地,按照相对位置计算,酶催化位点的关键氨基酸为第229位的arg,第258位的glu,第290位his;配体结合位点的关键氨基酸为第75-77位的ser、ala、gly,第99位的tyr,第229位的arg,第290位的his,第293位的phe;nad结合位点的关键氨基酸为第99位tyr,第104位ile,第150位gly,第152-155位的gly、lys、ile、gly,第172-174位的tyr、asp、pro,第199-202位的his、cys、pro、leu,第206位的asn,第209位的met,第227-229的thr、ala、arg,第253-254位的asp、val,第290位的his,第292-293位的ala、phe。
本发明的另一份方面提供了一种核苷酸序列。在一个具体实施方式中,该核苷酸序列编码如权利要求1所述的d-乳酸脱氢酶。
进一步地,该核苷酸序列具有下列核苷酸序列之一:
1)seqidno.2所示的核苷酸序列;
2)seqidno.3所示的核苷酸序列;
3)与seqidno.2或seqidno.3所示的核苷酸序列具有80%以上的同源性的核苷酸序列;
4)在高严谨条件与seqidno.2或seqidno.3所示的核苷酸序列的互补链杂交的核苷酸序列。
本发明的又一个方面提供了如上所述的d-乳酸脱氢酶或如上所述的核苷酸序列在d-乳酸生产中的应用。
本发明的再一个方面提供了一种基因工程菌株。在一个具体实施方式中,该基因工程菌株具有如上所述的d-乳酸脱氢酶或如上所述的核苷酸序列编码的d-乳酸脱氢酶。
进一步地,该基因工程菌中丙酮酸合成l-乳酸的途径被阻断,丙酮酸合成2,3-丁二醇的途径被阻断。
优选地,丙酮酸合成l-乳酸的途径,通过l-乳酸脱氢酶基因的失活或缺失而被阻断;丙酮酸合成2,3-丁二醇的途径,通过乙酰乳酸合成酶基因和乙酰乳酸脱羧酶基因中的一种或两种的失活或缺失而被阻断。
优选地,该基因工程菌中丙酮酸合成甲酸、丙酮酸合成乙酸和丙酮酸合成乙醇的途径中的一种或多种被阻断。
优选地,丙酮酸合成甲酸的途径,通过丙酮酸甲酸裂解酶基因和丙酮酸甲酸裂解酶活化酶基因中的一种或两种的失活或缺失而被阻断;丙酮酸合成乙酸的途径,通过丙酮酸脱氢酶基因和乙酸激酶中的一种或两种的失活或缺失而被阻断;丙酮酸合成乙醇的途径,通过丙酮酸脱氢酶基因和乙醇脱氢酶基因中的一种或两种的失活或缺失而被阻断。
优选地,该基因工程菌株中原始的d-乳酸脱氢酶基因失活或缺失。
进一步地,该基因工程菌株的起始菌为嗜热菌。
进一步地,该基因工程菌株的起始菌为芽孢杆菌。
优选地,该芽孢杆菌选自地衣芽孢杆菌、凝结芽孢杆菌、枯草芽孢杆菌、液化淀粉芽孢杆菌、短小芽孢杆菌、环状芽孢杆菌和解硫胺素芽孢杆菌。
优选地,起始菌中的限制性修饰系统失活或被敲除。
优选地,起始菌为地衣芽孢杆菌(bacilluslicheniformis)atcc14580的δhsdr1δhsdr2双突变菌株mw3。
进一步地,上述d-乳酸脱氢酶的编码基因的启动子改造为组成型启动子。
优选地,该组成型启动子为地衣芽孢杆菌atcc14580的α-乙酰乳酸合成酶的启动子pals、质粒pmmpc中的pc启动子、枯草芽孢杆菌(b.subtilis)中的p43启动子或地衣芽孢杆菌atcc14580的l-乳酸脱氢酶的启动子pldh。
进一步优选地,该组成型启动子为地衣芽孢杆菌atcc14580的α-乙酰乳酸合成酶的启动子pals。
进一步地,该基因工程菌株是地衣芽孢杆菌(bacilluslicheniformis)bn11,保藏号为cctccno:m2016026,于2016年1月8日保藏于中国典型培养物保藏中心,保藏地址为中国武汉武汉大学。
本发明的又一个方面提供了一种如上所述的基因工程菌株的制备方法。在一个具体实施方式中,该方法包括,使起始菌中的l-乳酸脱氢酶基因失活或缺失;引入上述的d-乳酸脱氢酶的核苷酸序列。
进一步地,该方法还包括,使起始菌中的原始的d-乳酸脱氢酶基因失活或缺失;阻断丙酮酸合成2,3-丁二醇的途径。
进一步地,该方法还包括,将引入的所述核苷酸序列的启动子改造为组成型启动子。
进一步地,起始菌为地衣芽孢杆菌atcc14580的δhsdr1δhsdr2双突变菌株mw3;将α-乙酰乳酸脱羧酶(alsd)和乙酰乳酸合成酶(alss)基因替换为如上所述的核苷酸序列。
本发明的又一个方面还提供了上述基因工程菌株的应用,尤其是在生产d-乳酸中的应用。
在一个具体实施方式中,该生产采用的碳源选自葡萄糖、木糖、麦芽糖、乳糖和蔗糖中的一种或多种。
优选地,该生产采使用含有花生粕或玉米浆干粉的廉价培养基进行发酵。
优选地,该生产的发酵温度为45℃~55℃。
优选地,该生产的发酵培养基ph为6.0~8.0。
优选地,该生产的发酵工艺为连续发酵或补料流加工艺。
本发明的还有一方面提供了一种d-乳酸脱氢酶。在一个具体实施方式中,该d-乳酸脱氢酶具有下列氨基酸序列之一:
1)具有seqidno.1所示的氨基酸序列;
2)将seqidno.1的氨基酸序列通过一个或几个氨基酸残基的取代、缺失或插入产生的衍生蛋白质的氨基酸序列,该衍生蛋白质具有d-乳酸脱氢酶活性;
3)包含与seqidno.1具有至少大约80%同源性的氨基酸序列。
该蛋白来来源于热脱硫杆菌属(t.indicus)。上述蛋白可人工合成,也可先合成其编码基因,再进行生物表达得到。
本发明的还提供了编码上述d-乳酸脱氢酶的核酸分子,这些核酸分子可以是dna,如cdna、基因组dna或重组dna;也可以是rna,如mrna、hnrna或trna等。
进一步地,本发明提供了一种编码上述d-乳酸脱氢酶的核苷酸序列,该核苷酸序列具有下列核苷酸序列之一:
1)具有seqidno.2所示的核苷酸序列;
2)具有seqidno.3所示的核苷酸序列;
3)与seqidno.2或seqidno.3具有80%以上的同源性的核苷酸序列。
本发明的另一方面提供了上述d-乳酸脱氢酶或上述核苷酸序列在d-乳酸生产中的应用。
本发明的又一方面提供了一种基因工程菌株,其特征在于,所述基因工程菌株具有如权利要求1所述的d-乳酸脱氢酶或如权利要求2所述的核苷酸序列编码的d-乳酸脱氢酶。
进一步地,上述基因工程菌株中l-乳酸脱氢酶基因的失活或缺失,乙酰乳酸合成酶和/或乙酰乳酸脱羧酶基因的失活或缺失。
进一步地,上述基因工程菌株还具有一个或多个基因的失活或缺失,所述一个或多个基因选自:丙酮酸甲酸裂解酶基因、丙酮酸甲酸裂解酶活化酶基因、丙酮酸脱氢酶基因、乙酸激酶和乙醇脱氢酶基因。
进一步地,上述基因工程菌株的起始菌为芽孢杆菌。进一步地,该芽孢杆菌选自地衣芽孢杆菌、凝结芽孢杆菌、枯草芽孢杆菌、液化淀粉芽孢杆菌、短小芽孢杆菌、环状芽孢杆菌和解硫胺素芽孢杆菌。优选地,起始菌为地衣芽胞杆菌。优选地,起始菌为地衣芽孢杆菌(bacilluslicheniformis)atcc14580。优选地,起始菌为地衣芽孢杆菌atcc14580的δhsdr1δhsdr2双突变菌株mw3。
进一步地,上述d-乳酸脱氢酶基因的启动子为地衣芽孢杆菌atcc14580的α-乙酰乳酸合成酶的启动子pals、质粒pmmpc中的pc启动子、枯草芽孢杆菌(b.subtilis)中的p43启动子或地衣芽孢杆菌atcc14580的l-乳酸脱氢酶的启动子pldh。优选地,上述启动子与ldhti基因的起始密码子“atg”直接相连。
进一步地,上述基因工程菌株是保藏号为地衣芽孢杆菌(bacilluslicheniformis)bn11,保藏号为cctccno:m2016026,于2016年1月8日保藏于中国典型培养物保藏中心,保藏地址为中国武汉武汉大学。
本发明的再一方面提供了一种制备上述的基因工程菌株的方法,包括以下步骤:
1)敲除起始菌株中的l-乳酸脱氢酶基因;
2)引入上述编码d-乳酸脱氢酶的核苷酸序列;
3)将乙酰乳酸合成酶和/或乙酰乳酸脱羧酶基因进行失活或缺失;
4)在进行步骤3)的同时或之后,在步骤2)中引入的所述核苷酸序列之前加上启动子序列。
具体地,以敲除了l-乳酸脱氢酶基因的地衣芽孢杆菌atcc14580的突变菌株mw3作为宿主,并将α-乙酰乳酸脱羧酶(alsd)和乙酰乳酸合成酶(alss)替换为上述d-乳酸脱氢酶,获得能够利用葡萄糖在30℃~55℃温度下发酵产生d-乳酸的耐热工程芽孢菌株cctccno:m2016026。
本发明的还有一方面提供了一种上述基因工程菌株的应用,尤其是在生产d-乳酸中的应用。进一步地,所述菌株在使用葡萄糖、木糖、麦芽糖、乳糖、蔗糖或其组合为碳源进行d-乳酸生产中的应用。进一步地,使用含有花生粕、玉米浆干粉的廉价培养基进行发酵。进一步地,在介于厌氧和微氧的环境下,ph为6.0~8.0,温度条件为45℃~55℃的条件下进行发酵生产d-乳酸。进一步地,所述发酵工艺为连续发酵或补料流加工艺。
进一步地,首先对上述基因工程菌株进行种子培养获得种子培养液,然后以葡萄糖、木糖、麦芽糖、乳糖、蔗糖中的一种作为碳源,酵母粉、蛋白胨、花生粕、玉米浆干粉作为氮源的发酵培养基进行发酵培养,得到d-乳酸。具体包括如下步骤:
1)斜面培养:将工程芽胞杆菌菌种接种于含有20g/l琼脂的固体斜面培养基上,45~55℃条件下,培养24~48h;
2)种子培养:将经过斜面培养的工程芽胞杆菌在无菌条件下接种到种子培养基中,45~55℃条件下,静止培养24~36h,加入中和剂控制发酵液ph,制得种子培养液;
3)发酵培养:按5~20%体积比的接种量接入到发酵培养基中,在45℃~55℃环境下培养48~90h,温度优选50℃。
优选地,步骤2)所述的种子培养基每升中含有:葡萄糖60~120g或木糖40~70g,酵母粉8~12g,蛋白胨3~8g,碳酸钙50g,余量为水,优选含有:葡萄糖90g,酵母粉10g,蛋白胨5g,碳酸钙50g,余量为水;该种子培养基的ph为6.0。115℃灭菌15min。中和剂包括naoh、nh4oh和ca(oh)2中的一种或几种。
优选地,步骤3)中所述的发酵培养基的组分及其含量为:碳源40~180g/l、氮源添加量5~20g/l。优选地,步骤3)中所述的发酵工艺为补料流加工艺,该补料流加工艺是指:当发酵液中总还原糖含量低于20g/l时补加碳源,使总还原糖含量维持在30~70g/l,或达到50~70g/l。优选地,所述的发酵培养基的ph为6.0~8.0。
本发明提供的改造的基因工程菌株,可以利用低成本的原料和较高的发酵温度发酵生产高浓度、高光学纯度的d-乳酸,在节约成本的同时提高生产效率,降低生产成本,适合于工业生产中推广应用。其中d-乳酸产量最高可达226g/l,光学纯度为99.9%,糖酸转化率最高可达93.6%,发酵生产能力3.2g/[l·h]。因此,利用本发明方法生产d-乳酸,能节约成本、简化操作流程,具有广阔的工业应用前景。
以下将结合附图对本发明的构思、具体步骤及产生的技术效果作进一步说明,以充分地了解本发明的目的、特征和效果。
附图说明
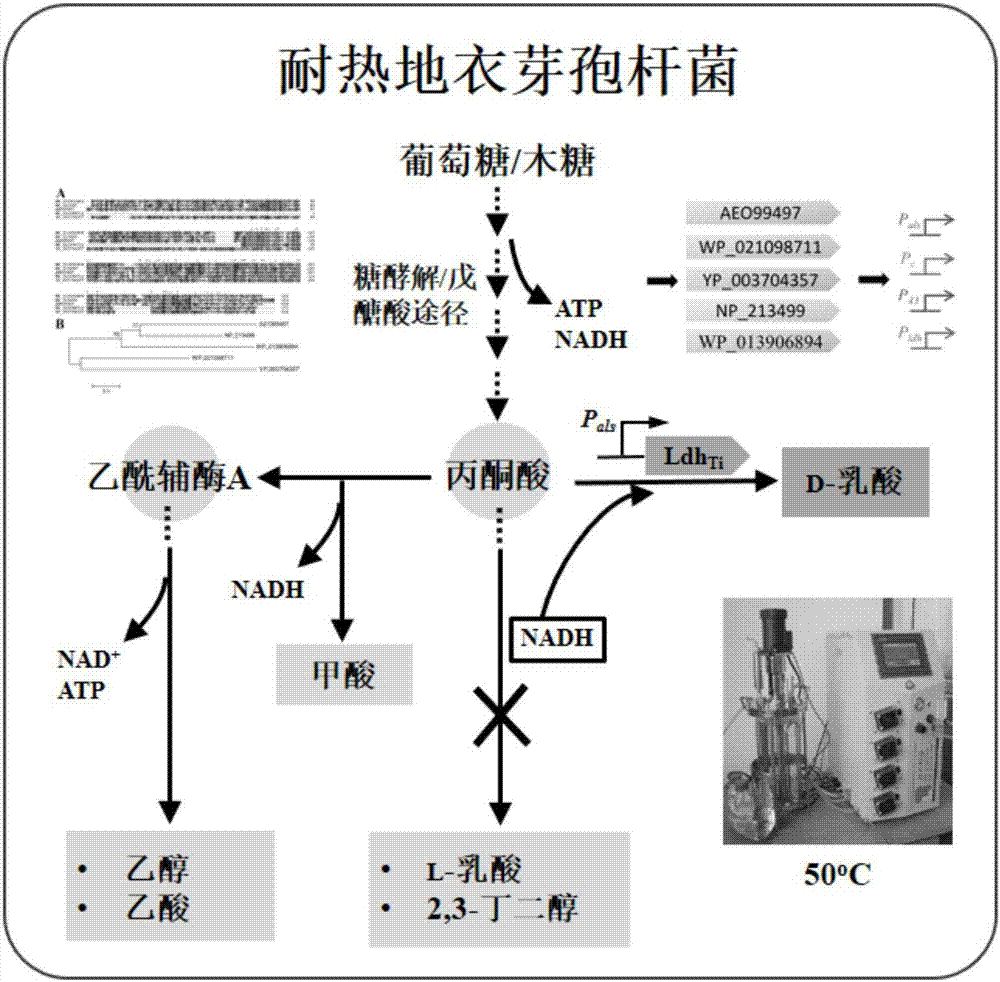
图1是本发明的一个具体实施方式的耐热地衣芽孢杆菌的构建及产d-乳酸途径示意图。
图2是sds-page检测纯化d-乳酸脱氢酶蛋白的分子量大小。其中,泳道m为蛋白分子量标准;泳道1为含有petduet-1空载体的大肠杆菌e.colibl21(de3)诱导后获得的粗酶液;泳道2为含有原核表达载体petduet-ldhti的大肠杆菌e.colibl21(de3)诱导后获得的粗酶液;泳道3为纯化后d-乳酸脱氢酶。
图3是d-乳酸脱氢酶的最适反应ph值测定结果。
图4是d-乳酸脱氢酶的最适反应温度测定结果。
图5是各种质粒的构建:a.pkvmδldh;b.pkvmn1;c.pkvmn2;d.pkvmn4;e.pkvmn6;f.pkvma1。
图6是菌株bn11产d-乳酸的hplc光学纯度分析。
图7是菌株bn11利用不同葡萄糖糖浓度发酵生产d-乳酸的情况:a.不同浓度糖的消耗情况;b.不同浓度的糖产d-乳酸的情况。初始糖浓度:
图8是菌株bn11进行分批发酵和补料(葡萄糖)流加发酵生产d-乳酸的情况。a.分批发酵结果;b.补料(葡萄糖)流加发酵结果。■,细胞密度;●,葡萄糖;▲,d-乳酸;◆,甲酸;▼,乙酸;★,乙醇。
图9是菌株ba11进行分批发酵和补料(葡萄糖)流加发酵生产d-乳酸的情况。a.分批发酵结果;b.补料(葡萄糖)流加发酵结果。■,细胞密度;●,葡萄糖;▲,d-乳酸;◆,甲酸;▼,乙酸;★,乙醇。
图10是菌株bn11利用木糖进行补料流加发酵生产d-乳酸的情况。■,细胞密度;●,木糖;▲,d-乳酸;◆,甲酸;▼,乙酸;★,乙醇。
图11是菌株bn11进行利用廉价培养基进行补料(葡萄糖)流加发酵生产d-乳酸的情况。■,细胞密度;●,葡萄糖;▲,d-乳酸;◆,甲酸;▼,乙酸;★,乙醇。
图12是部分典型嗜热菌中所预测的d-乳酸脱氢酶的发育树。
图13是seqidno.1所示的氨基酸序列(query)与其它d-乳酸脱氢酶进行氨基酸序列比对,获得的酶催化位点的分析结果。
图14是seqidno.1所示的氨基酸序列(query)与其它d-乳酸脱氢酶进行氨基酸序列比对,获得的配体结合位点的分析结果。
图15是seqidno.1所示的氨基酸序列(query)与其它d-乳酸脱氢酶进行氨基酸序列比对,获得的nad结合位点的分析结果。
具体实施方式
本发明的一个方面提供了一种d-乳酸脱氢酶,其能催化丙酮酸合成d-乳酸。
在一个优选的具体实施方式中,该d-乳酸脱氢酶具有下列氨基酸序列之一:
1)seqidno.1所示的氨基酸序列;
2)seqidno.1所示的氨基酸序列通过一个或几个氨基酸残基的取代、缺失、插入或添加衍生产生的氨基酸序列;
3)seqidno.1所示的氨基酸序列被保守性替换的氨基酸序列;
4)包含与seqidno.1所示的氨基酸序列具有至少80%同源性的氨基酸序列。
本文所说的“保守性替换”应当被理解为:用另一种特征相似的氨基酸取代多肽中的给定氨基酸的那些取代。典型地,把以下替换视作保守取代:将如ala、val、leu和ile的脂族氨基酸替换为另一种脂族氨基酸;用thr替换ser,反之亦然;将如asp或glu的酸性残基替换为另一种酸性残基;将如asn或gln的含有酰胺基的残基替换为另一种含有酰胺基的残基;将如lys或arg的碱性残基替换为另一种碱性残基;将如phe或tyr的芳族残基替换为另一种芳族残基。
功能等效的氨基酸总体上是与它们替代的这些氨基酸在大小和/或特征(例如,电荷或疏水性)方面是类似的。类似特性的氨基酸可以分组如下:
(1)疏水性:his、trp、trp、tyr、phe、met、leu、ile、val、ala;
(2)中性疏水性:cys、ser、thr;
(3)极性:ser、thr、asn、gln;
(4)酸性/带负电荷的:asp、glu;
(5)带电荷的:asp、glu、asn、lys、his;
(6)碱性/带正电荷的:asn、lys、his;
(7)碱性的:asn、gln、his、lys、arg;
(8)影响链取向的残基:gly、pro;以及
(9)芳香族:trp、tyr、phe、his。
部分典型嗜热菌中所预测的d-乳酸脱氢酶的氨基酸序列,利用mega5软件,通过邻接法绘制成的发育树如图12所示。其中,加粗字体的菌株为seqidno.1所示的d-乳酸脱氢酶的供体菌,与它同源性最低的是trueperaradiovictrix中的d-乳酸脱氢酶,两者氨基酸相似性为28%;与它相似性最高的则是treponemacaldariumdsm7334中的d-乳酸脱氢酶,两者氨基酸相似性为45%。
优选地,氨基酸残基的取代、缺失、插入或添加发生于功能域之外,该功能域包括d-乳酸脱氢酶的酶催化位点、配体结合位点和nad结合位点。以seqidno.1所示的氨基酸序列与其它d-乳酸脱氢酶进行氨基酸序列比对,得到如图13、14和15所示的催化位点、配体结合位点和nad结合位点的比对分析结果。
更优选地,氨基酸残基的取代、缺失、插入或添加发生于酶催化位点、配体结合位点和nad结合位点的关键氨基酸之外。其中,酶催化位点的关键氨基酸为第229位的arg,第258位的glu,第290位his;配体结合位点的关键氨基酸为第75-77位的ser、ala、gly,第99位的tyr,第229位的arg,第290位的his,第293位的phe;nad结合位点的关键氨基酸为第99位tyr,第104位ile,第150位gly,第152-155位的gly、lys、ile、gly,第172-174位的tyr、asp、pro,第199-202位的his、cys、pro、leu,第206位的asn,第209位的met,第227-229的thr、ala、arg,第253-254位的asp、val,第290位的his,第292-293位的ala、phe。
本发明的另一个方面提供了一种核苷酸序列,其编码上述d-乳酸脱氢酶。
在一个优选的具体实施方式中,该核苷酸序列具有下列核苷酸序列之一:
1)seqidno.2所示的核苷酸序列;
2)seqidno.3所示的核苷酸序列;
3)与seqidno.2或seqidno.3所示的核苷酸序列具有80%以上的同源性的核苷酸序列;
4)在高严谨条件与seqidno.2或seqidno.3所示的核苷酸序列的互补链杂交的核苷酸序列。
本文所说的“在低严谨度、中严谨度、高严谨度、或极高严谨度条件杂交”描述了杂交和洗涤的条件。进行杂交反应的指导可见于currentprotocolsinmolecularbiology,johnwiley&sons,n.y.(1989),6.3.1-6.3.6。此文献中描述了水相和非水相方法,任一种都可用。本文的具体杂交条件如下:1)低严谨度杂交条件是在6x氯化钠/柠檬酸钠(ssc)中在约45℃杂交,之后在0.2xssc,0.1%sds中至少在50℃(对于低严谨度条件,洗涤的温度应升高到55℃)洗涤两次;2)中严谨度杂交条件是在6xssc中在约45℃杂交,之后在0.2xssc,0.1%sds中在60℃洗涤一或多次;3)高严谨度杂交条件是在6xssc中在约45℃杂交,之后在0.2xssc,0.1%sds中在65℃洗涤一或多次;4)极高严谨度杂交条件是在0.5m磷酸钠,7%sds中在65℃杂交,之后在0.2xssc,1%sds中在65℃洗涤一或多次。高严谨度条件(3)是优选的,除特别指出之外应使用该条件。
本发明的再一个方面提供了一种基因工程菌株,其具有如上所述的d-乳酸脱氢酶或如上所述的核苷酸序列编码的d-乳酸脱氢酶。
在一个具体实施方式中,该基因工程菌株的起始菌株的丙酮酸合成l-乳酸的途径和丙酮酸合成2,3-丁二醇的途径被阻断。在一个实施例中,通过l-乳酸脱氢酶基因的失活或缺失阻断丙酮酸合成l-乳酸的途径;通过乙酰乳酸合成酶基因和乙酰乳酸脱羧酶基因中的一种或两种的失活或缺失阻断丙酮酸合成2,3-丁二醇的途径。
在另一个具体实施方式中,除了上述途径被阻断外,起始菌株的丙酮酸合成甲酸、丙酮酸合成乙酸和丙酮酸合成乙醇的途径中的一种或多种也被阻断。通过丙酮酸甲酸裂解酶基因和丙酮酸甲酸裂解酶活化酶基因中的一种或两种的失活或缺失阻断丙酮酸合成甲酸的途径;通过丙酮酸脱氢酶基因和乙酸激酶中的一种或两种的失活或缺失阻断丙酮酸合成乙酸的途径;通过丙酮酸脱氢酶基因和乙醇脱氢酶基因中的一种或两种的失活或缺失阻断丙酮酸合成乙醇的途径。
优选地,起始菌株为嗜热菌。且更优选的,该起始菌株中的限制性修饰系统失活或被敲除。
优选地,上述d-乳酸脱氢酶的基因的启动子被改造为组成型启动子,即不需要诱导物;组成型启动子的调控不受外界条件的影响,所启动基因的表达具有持续性。
图1是一个实施例中d-乳酸工程菌株的构建及产d-乳酸途径的示意图。
本文中“阻断”是指,通过各种基因工程的方法,将某一途径阻断。包括将该途径中的一种或多种催化酶的基因失活或缺失,从而使该途径不能进行。
本文中“原始的d-乳酸脱氢酶基因”是指起始菌本身携带的d-乳酸脱氢酶基因。
本领域技术人员可知,起始菌株也可以不含上述被缺失或失活的酶或被阻断的途径,如l-乳酸脱氢酶基因、d-乳酸脱氢酶基因等,那么在构建基因工程菌株时,这些基因或途径自然缺失,不需要额外进行基因工程操作使其失活或缺失或被阻断。
本发明的又一个方面提供了上述基因工程菌株的应用,尤其是在d-乳酸生产中的应用。
在一个具体实施方式中,上述基因工程菌株可以利用选自葡萄糖、木糖、麦芽糖、乳糖和蔗糖中的一种或多种的碳源进行发酵生产d-乳酸;也可以利用含有花生粕或玉米浆干粉的廉价培养基进行发酵生产d-乳酸。由于该基因工程菌株的起始菌株为嗜热菌,并且引入的d-乳酸脱氢酶的的原始来源也是嗜热菌,因此,该基因工程菌株能在较高的发酵温度下生产d-乳酸。
以下结合具体实施例进一步详述本发明。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂、菌株等,如无特殊说明,均可从商业途径得到。
试剂和菌株:本发明中的所有试剂均是市场购买的试剂级以上的试剂。其中fastpfudna聚合酶购自北京全式金生物技术有限公司。所有的限制性内切酶和t4dna连接酶购自neb(newenglandbiolabs)。异丙基-β-d-硫代吡喃半乳糖苷(iptg)、二硫苏糖醇(dtt)、苯甲基磺酰氟(pmsf)购自德国默克公司。l-乳酸和d-乳酸标品购自西格玛奥德里奇。pmd18-t和所有的引物合成于大连宝生物公司。地衣芽孢杆菌atcc14580可以从atcc网站直接购买获得,地衣芽孢杆菌atcc14580的双突变菌株mw3(δhsdr1,δhsdr2)可以根据文献:generationofreadilytransformablebacilluslicheniformismutants(biancawaschkawetal.,applmicrobiolbiotechnol,(2008)78:181-188)中的方法构建获得,双突变菌株mw3作为dna操作时使用的宿主菌。大肠杆菌e.colidh5α和bl21(de3)分别用作克隆宿主菌和表达宿主菌。大肠杆菌e.colis17-1用作接合转移的供体菌。petduet-1用作表达载体。穿梭质粒pkvm1带有氨苄青霉素和红霉素抗性,用于地衣芽孢杆菌mw3的基因敲除。luria-bertani(lb)培养基用作培养大肠杆菌和芽孢杆菌。氨苄青霉素(100g/ml),红霉素(5g/ml),多粘菌素b(40g/ml)用于大肠杆菌和芽孢杆菌的筛选。x-gal(40g/ml)用于蓝白斑筛选。
用于菌株产d-乳酸的hplc分析使用的是手性柱:mcigelcrs10w。
实施例1:d-乳酸脱氢酶基因及其蛋白的获得
1.d-乳酸脱氢酶ldhti基因的获得
在ncbi中以超嗜热菌aquifexaeolicusvf5中的d-乳酸脱氢酶np_213499为模板,经过比对得到一个与np_213499相似度为38%的可能是d-乳酸脱氢酶的蛋白wp_013906894,其序列如seqidno.1所示。进一步查找发现该蛋白存在于热脱硫杆菌(thermodesulfatatorindicus)dsm15286中,其对应的核苷酸序列为一个完整的开放阅读框,如seqidno.2所示,长度为978bp,编码如seqidno.1所示的氨基酸残基组成的蛋白质,该蛋白命名为ldhti。依照大肠杆菌e.colik12的密码子使用优化后,用pcr合成方法,采用如表1所示的引物,引物之间互为模版,从而合成密码子优化的核苷酸序列,其序列如seqidno.3所示。将得到的如seqidno.3所示的基因插入pmd18-t载体中,获得pmd18-t-ldhti质粒,并进行核苷酸序列测定。本领域技术人员可知,seqidno.3所示的基因,也可以直接通过基因合成的方式直接合成。
表1用于pcr合成d-乳酸脱氢酶ldhti的引物序列
下划线代表酶切位点。
2.重组原核表达载体的构建
将上述获得的pmd18-t-ldhti质粒用如表1所示的引物1和40进行pcr扩增,获得的目的基因片段用bamhi和hindiii双酶切后,连接到经相同的酶双酶切处理的表达载体petduet-1(novagen公司)中,将含有ldhti基因的连接产物转化大肠杆菌转化大肠杆菌e.colibl21(de3)(novagen公司),经pcr和dna测序鉴定后将含有正确ldhti基因的阳性克隆中的质粒命名为petduet-ldhti,可用其进行d-乳酸脱氢酶的表达。
3.d-乳酸脱氢酶的原核表达及纯化
1)d-乳酸脱氢酶ldhti的原核表达
将上述获得的含有原核表达载体petduet-ldhti的大肠杆菌e.colibl21(de3)在37℃下摇菌至od600nm为0.6~0.8后,加入终浓度为1mm的iptg诱导,16℃下摇床培养16h或30℃下摇床培养8h后离心收集菌体,重悬于50mm磷酸盐缓冲液(pbs;ph7.4)中,超声破碎细胞,收集上清,进行sds-page电泳分析。
结果如图2所示,从图中可以看出携带目的基因的原核表达载体petduet-ldhti在大肠杆菌e.colibl21(de3)中获得大量表达,表达的重组蛋白的单亚基分子量约为37kda,与预期结果相符。
2)d-乳酸脱氢酶ldhti的纯化
通过his-tag标签对步骤1)表达的蛋白进行纯化。具体如下:
将步骤1)获得的经过sds-page检测的原核表达产物进行亲和层析纯化,即将超声后的上清过填充有ni-nta凝胶的层析柱,含有his-tag标签的蛋白将结合到ni-nta凝胶上,然后用清洗缓冲液(25mmtris盐酸缓冲液,500mmnacl,50mm咪唑,以上浓度均为在溶液中的终浓度,ph8.0)洗涤非特异性结合的杂蛋白,最后用洗脱缓冲液(25mmtris盐酸缓冲液,500mmnacl,220mm咪唑,以上浓度均为在溶液中的终浓度,ph8.0)将目的蛋白洗脱,即得到纯化后的目的蛋白。然后使用分子筛凝胶柱更换缓冲液为50mm磷酸盐缓冲液(ph7.0),获得最终纯化后目的蛋白d-乳酸脱氢酶ldhti。
对最终纯化后目的蛋白d-乳酸脱氢酶ldhti进行sds-page电泳,结果如图2泳道3所示,目的条带单一,大小约为37kda,表明所得目的蛋白较纯。
实施例2:实施例1所得目的蛋白d-乳酸脱氢酶ldhti的酶学特性鉴定
用下列底物来鉴定实施例1所得目的蛋白的酶学特性:丙酮酸、d-乳酸,l-乳酸、甘油酸、苯丙酮酸、乙醛酸和草酰乙酸。反应体系:50mm磷酸盐缓冲液(ph7.0)、0.2mm的nadh或nad+为辅酶、适量的d-乳酸脱氢酶ldhti以及不同浓度的底物,于37℃反应。酶活的测定方法是在340nm紫外波长下测定nadh的氧化或nad+的还原,一个单位酶活定义为每分钟氧化或还原1μmol的nadh或nad+所需的酶量。
结果表明:实施例1所得目的蛋白既可以催化丙酮酸、乙醛酸、草酰乙酸、d-乳酸和苯丙酮酸,而对l-乳酸和甘油酸没有活性(表2)。尤其,d-乳酸脱氢酶ldhti对丙酮酸具有最高的催化效率,且可以专一性的催化d-乳酸。因此,上述结果表明实施例1所得目的蛋白确实为d-乳酸脱氢酶ldhti。
表2d-乳酸脱氢酶ldhti催化的底物谱范围。
–:表明酶对该底物没有活性。
其中,实施例1获得的d-乳酸脱氢酶ldhti的最适反应ph值和最适反应温度是通过如下两个试验确定的:
1)d-乳酸脱氢酶ldhti的最适反应ph值确定
酶活的测定方法是在340nm紫外波长下测定nadh的变化,一个单位酶活定义为每分钟氧化1μmol的nadh所需的酶量。在37℃条件下,以20mm的丙酮酸为底物,以0.2mm的nadh为辅酶,测定了所述d-乳酸脱氢酶从ph3.0到11.0的范围内,酶活性的变化情况。缓冲体系:ph3.0到7.0的50mm柠檬酸-柠檬酸钠缓冲液;ph7.0到9.0的50mm磷酸盐缓冲液;ph9.0到11.0的50mm碳酸钠-碳酸氢钠缓冲液。实验重复三次。结果如图3所示,该酶的最适反应ph值为6.0。图3中酶相对活力是测定不同ph值条件下酶的活力,依据在ph6.0的最高值为100%,其他ph时的酶活力数值相应折算得到酶相对活力。
2)d-乳酸脱氢酶ldhti的最适反应温度确定
酶活的测定方法是在340nm紫外波长下测定nadh的变化,一个单位酶活定义为每分钟氧化1μmol的nadh所需的酶量。d-乳酸脱氢酶最适反应温度的测定条件如下:20mm的丙酮酸为底物,0.2mm的nadh为辅酶,50mm的磷酸盐缓冲液(ph7.0)体系;分别测定了从30℃到100℃的酶活力,实验重复三次。结果如图4所示,该酶的最适反应温度是70℃。图4中酶相对活力是测定不同温度条件下酶的活力,依据在70℃的最高值为100%,其他温度时的酶活力数值相应折算得到酶相对活力。
实施例3:耐热地衣芽孢杆菌菌株的制备
1.构建本发明中的各种敲除质粒
1)l-乳酸脱氢酶基因敲除质粒的构建:以atcc14580基因组dna为模板,用引物对ldh-up-f/ldh-up-r和ldh-dn-f/ldh-dn-r分别pcr扩增l-乳酸脱氢酶基因的上下游同源臂,然后用bamhi/xhoi和xhoi/ncoi分别双酶切上下游同源臂,同时用bamhi/ncoi双酶切质粒pkvm1,上述酶切后的载体和片段用t4dna连接酶连接后,转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmδldh,质粒构建如图5a所示。其中,pkvm1质粒可以通过文献sizeunlimitedmarkerlessdeletionsbyatransconjugativeplasmid-systeminbacilluslicheniformis(rachinger,m.etal.,journalofbiotechnology,2013,167(4),365-369)构建而成。
2)2,3-丁二醇代谢途径乙酰乳酸合成酶(alss)和乙酰乳酸脱羧酶(alsd)基因敲除质粒的构建:
其中,使用到的引物序列如表3所示。
(1)以atcc14580基因组dna为模板,用引物对als1-up-f/als1-up-r和als-dn-f/als1-dn-r分别pcr扩增上下游同源臂,同时用引物对als-ldh-f/als-ldh-rpcr扩增d-乳酸脱氢酶ldhti基因,然后用引物als1-up-f和als1-dn-r以重组pcr的方法将以上三段基因进行融合。将上述重组pcr产物和pkvm1分别用酶bamhi/ncoi进行双酶切,将t4dna连接酶连接后的产物转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmn1(图5b)。此质粒上游同源臂末端完整保留了alss的启动子区域(pals),且与ldhti的起始密码子“atg”直接相连。其中,atcc14580的基因组序列可从ncbi上获得。其中,pals的序列如seqidno.69所示。
(2)用pc启动子起始ldhti表达的敲除质粒构建:以质粒pmmpc为模板,用引物als2-pc-f和als2-pc-r进行pcr扩增,获得pc启动子的基因。其中,pmmpc质粒可以根据文献newconstitutivevectors:usefulgeneticengineeringtoolsforbiocatalysis(xu,y.,tao,f.,ma,c.,&xu,p.,appliedandenvironmentalmicrobiology,2013,79(8),2836-2840)中的方法,从质粒pmmb66eh构建而成;以atcc14580基因组dna为模板,用引物对als2-up-f/als2-up-r和als-dn-f/als2-dn-r分别pcr扩增上下游同源臂,同时用引物对als2-ldh-f/als-ldh-rpcr扩增d-乳酸脱氢酶ldhti基因,然后用引物als2-up-f和als2-dn-r以重组pcr的方法将以上四段基因进行融合。将上述重组pcr产物和pkvm1分别用酶bamhi/xmai进行双酶切,将t4dna连接酶连接后的产物转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmn2(图5c)。其中,pc的序列如seqidno.70所示。
(3)用p43启动子起始ldhti表达的敲除质粒构建:以枯草芽孢杆菌(b.subtilis)基因组dna为模板,用引物als4-p43-f和als4-p43-r进行pcr扩增,获得p43启动子的基因,其中,p43启动子的信息,可以从文献isobutanolproductionatelevatedtemperaturesinthermophilicgeobacillusthermoglucosidasius(lin,p.p.,rabe,k.s.,takasumi,j.l.,kadisch,m.,arnold,f.h.,&liao,j.c.,metabolicengineering,2014,24,1-8)中获知;以atcc14580基因组dna为模板,用引物对als2-up-f/als4-up-r和als-dn-f/als2-dn-r分别pcr扩增上下游同源臂,同时用引物对als2-ldh-f/als-ldh-rpcr扩增d-乳酸脱氢酶ldhti基因,然后用引物als2-up-f和als2-dn-r以重组pcr的方法将以上四段基因进行融合。将上述重组pcr产物和pkvm1分别用酶bamhi/xmai进行双酶切,将t4dna连接酶连接后的产物转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmn4(图5d)。其中,p43的序列如seqidno.71所示。
(4)用pldh启动子起始ldhti表达的敲除质粒构建:pldh为地衣芽孢杆菌atcc14580l-乳酸脱氢酶的启动子。以atcc14580基因组dna为模板,用引物als6-pldh-f和als6-pldh-r进行pcr扩增,获得pldh启动子的基因;用引物对als2-up-f/als6-up-r和als-dn-f/als2-dn-r分别pcr扩增上下游同源臂,同时用引物对als2-ldh-f/als-ldh-rpcr扩增d-乳酸脱氢酶ldhti基因,然后用引物als2-up-f和als2-dn-r以重组pcr的方法将以上四段基因进行融合。将上述重组pcr产物和pkvm1分别用酶bamhi/xmai进行双酶切,将t4dna连接酶连接后的产物转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmn6(图5e)。其中,pldh的序列如seqidno.72所示。
(5)替换ldhti为适中温d-乳酸脱氢酶的敲除质粒构建:以atcc14580基因组dna为模板,用引物对als1-up-f/alsa-up-r和als-dn-f/als1-dn-r分别pcr扩增上下游同源臂;以大肠杆菌e.colik12基因组dna为模板,用引物alsa-ldha-f/alsa-ldha-rpcr扩增d-乳酸脱氢酶ldha基因,然后用引物als1-up-f和als1-dn-r以重组pcr的方法将以上三段基因进行融合。将上述重组pcr产物和pkvm1分别用酶bamhi/ncoi进行双酶切,将t4dna连接酶连接后的产物转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvma1(图5f)。
表3载体构建用到的引物
注:“-f”代表正向引物;“-r”代表反向引物;下划线表示酶切位点。
3)甲酸途径丙酮酸甲酸裂解酶(pfla)基因的敲除质粒的构建
以atcc14580基因组dna为模板,用引物对pfla1-f/pfla1-r和pfla2-f/pfla2-r分别pcr扩增丙酮酸甲酸裂解酶基因的上下游同源臂,然后用ecori/xhoi和xhoi/bamhi分别双酶切上下游同源臂,同时用bamhi/ncoi双酶切质粒pkvm1,上述酶切后的载体和片段用t4dna连接酶连接后,转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmδpfla。
其中,引物序列如下(5'-3'):
pfla1-f:ccggaattcatggaacaatggaaaggt
pfla1-r:tcgctcgagtggaaatgtcaaacccatag
pfla2-f:ccgctcgaggacgatggccactgggatc
pfla2-r:atcggatccctacatcgattcatggaagg
4)乙醇途径乙醇脱氢酶(adhb)基因的敲除质粒的构建
以atcc14580基因组dna为模板,用引物对adhb-up-f/adhb-up-r和adhb-dn-f/adhb-dn-r分别pcr扩增丙酮酸甲酸裂解酶基因的上下游同源臂,然后用bamhi/xhoi和xhoi/ncoi分别双酶切上下游同源臂,同时用bamhi/ncoi双酶切质粒pkvm1,上述酶切后的载体和片段用t4dna连接酶连接后,转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmδadhb。
其中,引物序列如下(5'-3'):
adhb-up-f:tacgggatccgaacgggaatcggcaaagggatt
adhb-up-r:tccgctcgagcgatgataaaggctgccgagcta
adhb-dn-f:accgctcgagtcgtcacactcccattatcg
adhb-dn-r:catgccatggcgtcgtatttgccgtcagct
5)乙酸途径乙酸激酶(ack)基因的敲除质粒的构建
以atcc14580基因组dna为模板,用引物对ack-up-f/ack-up-r和ack-dn-f/ack-dn-r分别pcr扩增丙酮酸甲酸裂解酶基因的上下游同源臂,然后用bamhi/xhoi和xhoi/ncoi分别双酶切上下游同源臂,同时用bamhi/ncoi双酶切质粒pkvm1,上述酶切后的载体和片段用t4dna连接酶连接后,转化大肠杆菌e.colis17-1,经dna测序验证正确后,将阳性克隆质粒命名为pkvmδack。
其中,引物序列如下(5'-3'):
ack-up-f:tacgggatccgaaggctttccggccttact
ack-up-r:tccgctcgagcatcgtcattccgacgaatg
ack-dn-f:accgctcgaggagcttccagcattgattgc
ack-dn-r:catgccatggatgcggtcatctgcgatctt
2.将构建的载体用于改造耐热地衣芽孢杆菌
1)耐热地衣芽孢杆菌基因敲除步骤:
(1)将含有敲除质粒的大肠杆菌e.colis17-1和地衣芽孢杆菌mw3在lb培养基中分别培养至od600nm≈1.2,6000rpm离心5min,用0.9%生理盐水洗两遍,将两个菌混合后重悬并离心,然后用lb培养基重悬并滴在lb平板上,在30℃过夜培养,用预热(30℃)的lb收集细胞后,涂布于lb固体平板(红霉素和多粘菌素b),30℃培养;
(2)挑转化子至lb培养基(红霉素),30℃培养,然后系列稀释至lb平板(红霉素和x-gal),42℃过夜培养,蓝色菌落为正确的转化子;
(3)挑转化子至lb培养基,30℃无抗性培养并转接两代,稀释涂平板至lb平板(x-gal),挑白色转化子,并进行分子验证。
2)耐热地衣芽孢杆菌敲除菌株的制备过程
遗传操作选用的宿主菌为敲除了限制性修饰系统(δhsdr1,δhsdr2)的突变株mw3,该突变菌株可以更高效的进行外源dna的转化,同时其生长和分泌蛋白的能力与野生型atcc14580相同,不受敲除的影响。耐热地衣芽孢杆菌mw3敲除菌株的制备:
(1)敲除了l-乳酸脱氢酶(ldh)基因的宿主菌株bl2的制备:利用上述遗传操作方法将菌株mw3和含有敲除质粒pkvmδldh的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(2)敲除了l-乳酸脱氢酶(ldh)基因和2,3-丁二醇途径乙酰乳酸合成酶(alss)及乙酰乳酸脱羧酶(alsd)基因,并保留启动子pals的宿主菌株bn11的制备:利用上述遗传操作方法将菌株bl2和含有敲除质粒pkvmn1的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(3)敲除了l-乳酸脱氢酶(ldh)基因和2,3-丁二醇途径乙酰乳酸合成酶(alss)及乙酰乳酸脱羧酶(alsd)基因,并替换pals为pc启动子的宿主菌株bn22的制备:利用上述遗传操作方法将菌株bl2和含有敲除质粒pkvmn2的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(4)敲除了l-乳酸脱氢酶(ldh)基因和2,3-丁二醇途径乙酰乳酸合成酶(alss)及乙酰乳酸脱羧酶(alsd)基因,并替换pals为p43启动子的宿主菌株bn44的制备:利用上述遗传操作方法将菌株bl2和含有敲除质粒pkvmn4的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(5)敲除了l-乳酸脱氢酶(ldh)基因和2,3-丁二醇途径乙酰乳酸合成酶(alss)及乙酰乳酸脱羧酶(alsd)基因,并替换pals为pldh启动子的宿主菌株bn66的制备:利用上述遗传操作方法将菌株bl2和含有敲除质粒pkvmn6的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(6)替换bn11中嗜热d-乳酸脱氢酶ldhti为适中温型的d-乳酸脱氢酶ldha宿主菌株ba11的制备:利用上述遗传操作方法将菌株bn11和含有敲除质粒pkvma1的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(7)从菌株bn11出发,敲除了丙酮酸甲酸裂解酶(pfla)基因的宿主菌株bn12的制备:利用上述遗传操作方法将菌株bn11和含有敲除质粒pkvmδpfla的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(8)从菌株bn12出发,敲除了乙醇脱氢酶(adhb)基因的宿主菌株bn13的制备:利用上述遗传操作方法将菌株bn11和含有敲除质粒pkvmδadhb的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
(9)从菌株bn13出发,敲除了乙酸激酶(ack)基因的宿主菌株bn14的制备:利用上述遗传操作方法将菌株bn11和含有敲除质粒pkvmδack的大肠杆菌e.colis17-1进行双亲结合,然后分别通过单交换和双交换得到转化子,再提取基因组,通过pcr鉴定,挑取阳性菌株,并进行下一步实验。
实施例4:原始菌株、ldh敲除菌株以及含有不同启动子和ldhti基因的菌株的发酵情况
本实施例在5l全自动发酵罐中进行,所使用的各培养基的组成如下:
斜面培养基每升中含有:葡萄糖30~50g,酵母粉5~10g,蛋白胨2~8g,碳酸钙30g,琼脂粉15~25g,余量为水。所述斜面培养基的ph为7.0。115℃灭菌15min。
种子培养基每升中含有:葡萄糖40~120g,酵母粉5~10g,蛋白胨2~8g,碳酸钙50g,余量为水。所述种子培养基的ph为6.0~8.0。115℃灭菌15min。
发酵培养基每升中含有:葡萄糖120g,酵母粉5~10g,蛋白胨2~8g,余量为水;所述发酵培养基的ph为6.5~7.5。115℃条件下灭菌15min。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:将菌株接种于斜面培养基上,50℃培养24h;
(2)种子培养:将步骤(1)培养的菌株在无菌条件下用接种环接2环于装有40ml种子培养基的40ml三角瓶中,50℃静止培养24h,制得种子培养液1;将10ml种子培养液1在无菌条件下接入装有100ml种子培养基的500ml三角瓶中,50℃静止培养24h,制得种子培养液2;
(3)发酵培养:将300ml步骤(2)制得的种子培养液2在无菌条件下接入装有2.7l发酵培养基的发酵罐中,50℃、70rpm搅拌培养,每3h取样一次,12h后结束发酵。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率(表4)。其中,转化率(%)=乳酸的产量(g/l)/葡萄糖消耗量(g/l)×100%。其中,通过检测d-乳酸和l-乳酸的hplc检测,可以获知,bn11产的d-乳酸的光学纯度可达到99.9%(图6)。
表4不同菌株的葡萄糖消耗和产物生成情况。
–:表明产物的量小于0.01g/l.
由于本实施例采用的起始菌株地衣芽孢杆菌atcc14580天然高产2,3-丁二醇,即表明该途径的活性很强,而pals则是2,3-丁二醇合成基因簇中第一个基因乙酰乳酸合成酶(alss)的启动子,因此,这可能是基因工程菌株bn11(采用该启动子)表现出良好的转化率的原因。
实施例5:实施例4中最优菌株阻断副产物生成后的发酵情况
本实施例在5l全自动发酵罐中进行,使用菌株为bn11、bn12(阻断副产物甲酸)、bn13(阻断副产物甲酸和乙醇)、bn14(阻断副产物甲酸、乙醇和乙酸),所使用的各培养基的组成如下:
斜面培养基、种子培养基和发酵培养基同实施例4。
发酵生产d-乳酸的方法同实施例4。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率(表5)。其中,转化率(%)=乳酸的产量(g/l)/葡萄糖消耗量(g/l)×100%。其中,通过检测d-乳酸和l-乳酸的hplc检测,可以获知,bn11产的d-乳酸的光学纯度可达到99.9%。
表5阻断副产物生成的不同菌株的葡萄糖消耗和产物生成情况。
–:表明产物的量小于0.01g/l.
结果表明,在阻断2,3-丁二醇和l-乳酸的生成后,进一步阻断其他副产物,能够提高葡萄糖到d-乳酸的转化率。而且,阻断的副产物越多,d-乳酸的转化率越高。
实施例6:实施例4中最优菌株的发酵条件的优化
本实施例在5l全自动发酵罐中进行,所选用菌株为bn11,所使用的各培养基的组成如下:
斜面培养基和种子培养基同实施例4。
发酵培养基每升中含有:1)确定最适发酵ph时,培养基组成为:葡萄糖90g,酵母粉5~10g,蛋白胨2~8g,余量为水,ph分别调至6.0、6.5、7.0、7.5和8.0;2)为确定菌株在不同葡萄糖浓度的培养基中生产d-乳酸的能力,培养基组成为:酵母粉5~10g,蛋白胨2~8g,葡萄糖的量为60g、87g、122g、148g、180g和202g,余量为水。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:同实施例4;
(2)种子培养:同实施例4;
(3)发酵培养:将300ml步骤(2)制得的种子培养液2在无菌条件下接入装有2.7l不同条件的发酵培养基的发酵罐中,50℃、70rpm搅拌培养,每3h取样一次,12h后结束发酵。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率。
实验结果表明,实验菌株bn11在50℃条件下,最适发酵ph是7.0,在该ph值下,消耗葡萄糖速率最快,糖酸转化率最高,同时副产物也最少(表6)。从图7可知,当葡萄糖浓度不超过180g/l时,菌株bn11消耗葡萄糖和生产d-乳酸的速率没有明显的被抑制;当葡萄糖浓度高于此值,如202g/l时,葡萄糖的消耗明显受到抑制,d-乳酸的生产速率也随之降低。
表6ph对菌株bn11产d-乳酸的影响。
–:表明产物的量小于0.01g/l.
实施例7:利用实施例4中最优菌株进行分批发酵和补料(糖)流加发酵生产d-乳酸
本实施例在5l全自动发酵罐中进行,所选用菌株为bn11,所使用的各培养基的组成如下:
斜面培养基和种子培养基同实施例4。
发酵培养基每升中含有:1)分批发酵生产d-乳酸时,培养基组成为:葡萄糖180g,酵母粉5~10g,蛋白胨2~8g,余量为水;2)补料(糖)流加发酵生产d-乳酸,培养基组成为:葡萄糖40~70g,酵母粉5~10g,蛋白胨2~8g,余量为水。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:同实施例4;
(2)种子培养:同实施例4;
(3)发酵培养:将300ml步骤(2)制得的种子培养液2在无菌条件下接入装有2.7l分别含有分批发酵和补料(糖)流加发酵培养基的发酵罐中,50℃、70rpm搅拌培养,每2~5个小时取样一次,测定发酵液中的残糖量。分批发酵时,当发酵过程中葡萄糖消耗完或消耗速率趋于0时,结束发酵;补料(糖)流加发酵时,当葡萄糖浓度降到10~20g/l时,流加葡萄糖,使葡萄糖浓度达到50~70g/l,共计补糖2~5次。当发酵过程中葡萄糖消耗速率趋于0时,结束发酵。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率及生产速率。
从图8a可知,实验菌株bn11在50℃条件下进行分批发酵时,40h后葡萄糖消耗完毕,发酵结束。生产出的d-乳酸浓度为167.7g/l,副产物乙醇浓度为5.8g/l,甲酸浓度为1.1g/l,乙酸浓度为3.6g/l。糖酸转化率和生产速率分别为93.0%和4.2g/[l·h]。
从图8b可知,菌株bn11在50℃条件下进行补料(糖)流加发酵时,共流加葡萄糖补料4次,70h后结束发酵。其中前38h生产了173.2g/ld-乳酸,生产速率为4.6g/[l·h];从38h到70h,d-乳酸浓度从173.2g/l增加到226.6g/l,生产速率为1.7g/[l·h]。发酵完毕后,共消耗葡萄糖242.1g/l,平均生产速率为3.2g/[l·h],糖酸转化率为93.6%。副产物甲酸最终的浓度为0.56g/l,乙酸和乙醇的终浓度分别为5.5g/l和4.3g/l。
实施例8:替换嗜热ldhti为适中温d-乳酸脱氢酶(ldha)的菌株进行分批发酵和补料(糖)流加发酵生产d-乳酸
本实施例在5l全自动发酵罐中进行,所选用菌株为ba11,所使用的各培养基的组成如下:
斜面培养基和种子培养基同实施例4。
发酵培养基每升中含有:同实施例7。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:同实施例4;
(2)种子培养:同实施例4;
(3)发酵培养:同实施例7。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率及生产速率。
从图9a可知,实验菌株ba11在50℃条件下进行分批发酵时,40h后结束发酵,剩余的残糖为57.1g/l。最终生产出的d-乳酸浓度为100.8g/l,副产物乙醇浓度为9.5g/l,甲酸浓度为3.9g/l,乙酸浓度为4.5g/l。糖酸转化率和生产速率分别为82.4%和2.5g/[l·h]。
从图9b可知,菌株ba11在50℃条件下进行补料(糖)流加发酵时,共流加葡萄糖补料3次,70h后结束发酵。最终共消耗葡萄糖196.9g/l,生产d-乳酸168.6g/l,平均生产速率为2.4g/[l·h],糖酸转化率为85.6%。副产物甲酸和乙醇最终的浓度为1.0g/l,乙酸的终浓度为5.5g/l。
实施例9:实施例4中最优菌株利用木糖进行补料流加发酵生产d-乳酸
本实施例在5l全自动发酵罐中进行,所选用菌株为bn11,所使用的各培养基的组成如下:
斜面培养基每升中含有:木糖30~50g,酵母粉5~10g,蛋白胨2~8g,碳酸钙30g,琼脂粉15~25g,余量为水。所述斜面培养基的ph为7.0。115℃灭菌15min。
种子培养基每升中含有:木糖40~70g,酵母粉5~10g,蛋白胨2~8g,碳酸钙50g,余量为水。所述种子培养基的ph为6.0~8.0。115℃灭菌15min。
发酵培养基每升中含有:木糖40~60g,酵母粉5~10g,蛋白胨2~8g,余量为水;所述发酵培养基的ph为6.5~7.5。115℃条件下灭菌15min。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:同实施例4;
(2)种子培养:同实施例4;
(3)发酵培养:将300ml步骤(2)制得的种子培养液在无菌条件下接入装有2.7l发酵培养中,50℃、70rpm搅拌培养,每2~5个小时取样一次,测定发酵液中的残糖量。当木糖浓度降到10~20g/l时,流加木糖,使木糖达到40~60g/l,共计补糖2~5次。当发酵过程中木糖消耗速率趋于0时,结束发酵。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率及生产速率。
从图10可知,菌株bn11以木糖为碳源,在50℃条件下进行补料流加发酵时,共流加木糖补料1次,70h后结束发酵。最终共消耗木糖114.7g/l,生产d-乳酸72.1g/l,平均生产速率约为1.0g/[l·h]。副产物甲酸终浓度为23.0g/l,乙酸和乙醇的终浓度分别为7.2g/l和8g/l。
实施例10:实施例4中最优菌株利用廉价培养基进行补料(糖)流加发酵生产d-乳酸
本实施例在5l全自动发酵罐中进行,所选用菌株为bn11,所使用的各培养基的组成如下:
斜面培养基和种子培养基同实施例4。
发酵培养基每升中含有:葡萄糖40~70g,酵母粉1~5g,磷酸二氢钾0.25~1.0g/l,磷酸氢二钾0.25~1.0g/l,硫酸铵1.5~10.0g/l,磷酸氢二铵1.2~5.0g/l,硫酸锌0.15~0.8g/l,玉米浆干粉1~10g/l,余量为水。115℃条件下灭菌15min。
本实施例所述发酵生产d-乳酸的方法包括以下步骤:
(1)斜面培养:同实施例4;
(2)种子培养:同实施例4;
(3)发酵培养:将600ml步骤(2)制得的种子培养液2在无菌条件下接入装有2.4l廉价发酵培养基中,50℃、70rpm搅拌培养,每2~5个小时取样一次,测定发酵液中的残糖量。当葡萄糖浓度降到10~20g/l时,流加葡萄糖,使葡萄糖浓度达到50~70g/l,共计补糖2~5次。当发酵过程中葡萄糖消耗速率趋于0时,结束发酵。
发酵结束后,取发酵液上清通过hplc检测分析d-乳酸、副产物和总还原糖的浓度,计算糖酸转化率及生产速率。
从图11可知,菌株bn11利用廉价培养基在50℃条件下进行补料(糖)流加发酵时,共流加葡萄糖补料4次,90h后结束发酵。最终共消耗葡萄糖190.9g/l,生产d-乳酸175.7g/l,平均生产速率为2.0g/[l·h],糖酸转化率为92.0%。副产物乙酸终浓度为7.5g/l,甲酸和乙醇的终浓度分别为0.62g/l和1.6g/l。
以上详细描述了本发明的较佳具体实施例。应当理解,本领域的普通技术人员无需创造性劳动就可以根据本发明的构思作出诸多修改和变化。因此,凡本技术领域中技术人员依本发明的构思在现有技术的基础上通过逻辑分析、推理或者有限的实验可以得到的技术方案,皆应在由权利要求书所确定的保护范围内。
序列表
<110>上海交通大学
<120>一种d-乳酸脱氢酶、含有该酶的工程菌株及其构建和应用
<160>84
<170>patentinversion3.5
<210>1
<211>325
<212>prt
<213>热脱硫杆菌属dsm15286(thermodesulfatatorindicusdsm15286)
<400>1
metlysvalilephephesermethisprotyrglugluglupheleu
151015
glyproileleuproserasptrpaspvalglumetthrproaspphe
202530
leuaspgluthrthrvalglulysalalysglyalaglnvalvalser
354045
leuphevalserasplysalaaspglyprovalleuglualaleuhis
505560
sertyrglyvalglyleuleualaleuargseralaglytyrasphis
65707580
ileaspilegluthralalysargleuglyilelysvalvalasnval
859095
proalatyrserprohisalailealaasphisthrleualailemet
100105110
leualaleuileargargleuhisargalahisasplysvalargleu
115120125
glyasppheaspleuaspglyleumetglypheaspleuasnglylys
130135140
valalaglyvalileglyleuglylysileglyargleuvalalathr
145150155160
argleulysalapheglycyslysvalleuglytyraspprotyrile
165170175
glnprogluilevalgluasnvalaspleuaspthrleuilethrgln
180185190
alaaspileileserilehiscysproleuthrarggluasnphehis
195200205
metpheasnglugluthrphelysargmetlysproglyalaileleu
210215220
valasnthralaargglyglyleuileaspthrlysalaleuleuglu
225230235240
alaleulysserglylysleuglyglyalaalaleuaspvaltyrglu
245250255
tyrgluargglyleuphephelysasnhisglnlysgluglyilelys
260265270
aspprotyrleualaglnleuleuglyleualaasnvalvalleuthr
275280285
glyhisglnalapheleuthrargglualavallysasnilegluglu
290295300
thrthrvalgluasnileleuglutrpglnlysasnproglnalalys
305310315320
leulysasngluile
325
<210>2
<211>978
<212>dna
<213>热脱硫杆菌属dsm15286(thermodesulfatatorindicusdsm15286)
<400>2
atgaaagtaattttttttagtatgcacccgtatgaagaggaatttttagggcctatttta60
ccttctgactgggacgtagaaatgacgcctgactttttagacgaaaccacggtggaaaag120
gctaaaggagcccaggtagtaagcttgtttgtttcggacaaagctgatggtcccgtactt180
gaagcgctacattcttacggagtgggccttttggcccttcgtagtgctggctatgatcac240
atagatattgagaccgcaaaacgcctgggtataaaagtagttaatgtgccagcctattct300
ccccacgctatcgctgaccatactttagctataatgcttgctcttattcgtcgtcttcac360
cgggcccatgataaagtgcgcctgggagattttgatctcgatggtcttatgggctttgat420
ttaaatggcaaagttgctggtgtaattgggctagggaaaataggtcgcctggtagctacc480
cgcttaaaagcgtttggttgcaaagttttaggctatgatccatacattcagccggaaata540
gtagaaaatgttgatttggatacccttatcactcaggctgatatcatttctattcattgt600
cctcttacgcgagaaaattttcatatgtttaatgaagagacttttaagcggatgaaaccc660
ggggctattttggttaacacggcgcgaggaggtcttatagataccaaggccttgcttgag720
gccttaaagtctggaaaacttggcggcgcagcccttgatgtgtatgaatatgaacggggc780
ctcttttttaaaaaccaccaaaaagaaggtataaaagacccttatcttgcccagcttttg840
gggttggccaatgtagtgttaaccgggcatcaggcctttcttacgcgagaggctgtaaaa900
aacatagaagaaactaccgtagaaaatattttagaatggcaaaagaatccccaggcaaag960
cttaaaaatgaaatctga978
<210>3
<211>978
<212>dna
<213>人工序列
<220>
<223>d-乳酸脱氢酶基因密码子优化
<400>3
atgaaagtaatttttttttctatgcacccgtatgaagaggaatttctgggtccgattctg60
ccgtctgactgggacgtagaaatgaccccggactttctggacgaaaccaccgtggaaaag120
gctaaaggtgcccaggtagtaagcctgtttgtttctgacaaagctgatggtccggtactg180
gaagcgctgcattcttacggtgtgggcctgctggccctgcgttctgctggctatgatcac240
atcgatattgagaccgcaaaacgcctgggtatcaaagtagttaacgtgccagcctattct300
ccgcacgctatcgctgaccatactctggctatcatgctggctctgattcgtcgtctgcac360
cgtgcccatgataaagtgcgcctgggtgattttgatctggatggtctgatgggctttgat420
ctgaacggcaaagttgctggtgtaattggtctgggtaaaatcggtcgcctggtagctacc480
cgcctgaaagcgtttggttgcaaagttctgggctatgatccatacattcagccggaaatc540
gtagaaaacgttgatctggataccctgatcactcaggctgatatcatttctattcattgt600
ccgctgacccgtgaaaactttcatatgtttaacgaagagacttttaagcgtatgaaaccg660
ggtgctattctggttaacaccgcgcgtggtggtctgatcgataccaaggccctgctggag720
gccctgaagtctggtaaactgggcggcgcagccctggatgtgtatgaatatgaacgtggc780
ctgttttttaaaaaccaccaaaaagaaggtatcaaagacccgtatctggcccagctgctg840
ggtctggccaacgtagtgctgaccggtcatcaggcctttctgacccgtgaggctgtaaaa900
aacatcgaagaaactaccgtagaaaacattctggaatggcaaaagaacccgcaggcaaag960
ctgaaaaacgaaatctaa978
<210>4
<211>28
<212>dna
<213>人工序列
<220>
<223>p1
<400>4
ccgcggatccgatgaaagtaattttttt28
<210>5
<211>46
<212>dna
<213>人工序列
<220>
<223>p2
<400>5
tcttcatacgggtgcatagaaaaaaaaattactttcatcggatccg46
<210>6
<211>45
<212>dna
<213>人工序列
<220>
<223>p3
<400>6
ttctatgcacccgtatgaagaggaatttctgggtccgattctgcc45
<210>7
<211>45
<212>dna
<213>人工序列
<220>
<223>p4
<400>7
ggggtcatttctacgtcccagtcagacggcagaatcggacccaga45
<210>8
<211>45
<212>dna
<213>人工序列
<220>
<223>p5
<400>8
gggacgtagaaatgaccccggactttctggacgaaaccaccgtgg45
<210>9
<211>45
<212>dna
<213>人工序列
<220>
<223>p6
<400>9
cttactacctgggcacctttagccttttccacggtggtttcgtcc45
<210>10
<211>45
<212>dna
<213>人工序列
<220>
<223>p7
<400>10
taaaggtgcccaggtagtaagcctgtttgtttctgacaaagctga45
<210>11
<211>45
<212>dna
<213>人工序列
<220>
<223>p8
<400>11
gcagcgcttccagtaccggaccatcagctttgtcagaaacaaaca45
<210>12
<211>45
<212>dna
<213>人工序列
<220>
<223>p9
<400>12
ggtactggaagcgctgcattcttacggtgtgggcctgctggccct45
<210>13
<211>45
<212>dna
<213>人工序列
<220>
<223>p10
<400>13
aatatcgatgtgatcatagccagcagaacgcagggccagcaggcc45
<210>14
<211>45
<212>dna
<213>人工序列
<220>
<223>p11
<400>14
ctggctatgatcacatcgatattgagaccgcaaaacgcctgggta45
<210>15
<211>45
<212>dna
<213>人工序列
<220>
<223>p12
<400>15
gaataggctggcacgttaactactttgatacccaggcgttttgcg45
<210>16
<211>45
<212>dna
<213>人工序列
<220>
<223>p13
<400>16
agttaacgtgccagcctattctccgcacgctatcgctgaccatac45
<210>17
<211>45
<212>dna
<213>人工序列
<220>
<223>p14
<400>17
aatcagagccagcatgatagccagagtatggtcagcgatagcgtg45
<210>18
<211>45
<212>dna
<213>人工序列
<220>
<223>p15
<400>18
gctatcatgctggctctgattcgtcgtctgcaccgtgcccatgat45
<210>19
<211>45
<212>dna
<213>人工序列
<220>
<223>p16
<400>19
ccagatcaaaatcacccaggcgcactttatcatgggcacggtgca45
<210>20
<211>45
<212>dna
<213>人工序列
<220>
<223>p17
<400>20
cctgggtgattttgatctggatggtctgatgggctttgatctgaa45
<210>21
<211>45
<212>dna
<213>人工序列
<220>
<223>p18
<400>21
ccaattacaccagcaactttgccgttcagatcaaagcccatcaga45
<210>22
<211>45
<212>dna
<213>人工序列
<220>
<223>p19
<400>22
gcaaagttgctggtgtaattggtctgggtaaaatcggtcgcctgg45
<210>23
<211>45
<212>dna
<213>人工序列
<220>
<223>p20
<400>23
accaaacgctttcaggcgggtagctaccaggcgaccgattttacc45
<210>24
<211>45
<212>dna
<213>人工序列
<220>
<223>p21
<400>24
cgcctgaaagcgtttggttgcaaagttctgggctatgatccatac45
<210>25
<211>45
<212>dna
<213>人工序列
<220>
<223>p22
<400>25
ttttctacgatttccggctgaatgtatggatcatagcccagaact45
<210>26
<211>45
<212>dna
<213>人工序列
<220>
<223>p23
<400>26
tcagccggaaatcgtagaaaacgttgatctggataccctgatcac45
<210>27
<211>47
<212>dna
<213>人工序列
<220>
<223>p24
<400>27
atgaatagaaatgatatcagcctgagtgatcagggtatccagatcaa47
<210>28
<211>45
<212>dna
<213>人工序列
<220>
<223>p25
<400>28
tcaggctgatatcatttctattcattgtccgctgacccgtgaaaa45
<210>29
<211>45
<212>dna
<213>人工序列
<220>
<223>p26
<400>29
aaagtctcttcgttaaacatatgaaagttttcacgggtcagcgga45
<210>30
<211>47
<212>dna
<213>人工序列
<220>
<223>p27
<400>30
ctttcatatgtttaacgaagagacttttaagcgtatgaaaccgggtg47
<210>31
<211>45
<212>dna
<213>人工序列
<220>
<223>p28
<400>31
ccacgcgcggtgttaaccagaatagcacccggtttcatacgctta45
<210>32
<211>45
<212>dna
<213>人工序列
<220>
<223>p29
<400>32
gttaacaccgcgcgtggtggtctgatcgataccaaggccctgctg45
<210>33
<211>45
<212>dna
<213>人工序列
<220>
<223>p30
<400>33
gcccagtttaccagacttcagggcctccagcagggccttggtatc45
<210>34
<211>45
<212>dna
<213>人工序列
<220>
<223>p31
<400>34
ctgaagtctggtaaactgggcggcgcagccctggatgtgtatgaa45
<210>35
<211>45
<212>dna
<213>人工序列
<220>
<223>p32
<400>35
ttttaaaaaacaggccacgttcatattcatacacatccagggctg45
<210>36
<211>45
<212>dna
<213>人工序列
<220>
<223>p33
<400>36
gaacgtggcctgttttttaaaaaccaccaaaaagaaggtatcaaa45
<210>37
<211>45
<212>dna
<213>人工序列
<220>
<223>p34
<400>37
gcagctgggccagatacgggtctttgataccttctttttggtggt45
<210>38
<211>45
<212>dna
<213>人工序列
<220>
<223>p35
<400>38
cgtatctggcccagctgctgggtctggccaacgtagtgctgaccg45
<210>39
<211>45
<212>dna
<213>人工序列
<220>
<223>p36
<400>39
cctcacgggtcagaaaggcctgatgaccggtcagcactacgttgg45
<210>40
<211>45
<212>dna
<213>人工序列
<220>
<223>p37
<400>40
cctttctgacccgtgaggctgtaaaaaacatcgaagaaactaccg45
<210>41
<211>48
<212>dna
<213>人工序列
<220>
<223>p38
<400>41
ttgccattccagaatgttttctacggtagtttcttcgatgttttttac48
<210>42
<211>45
<212>dna
<213>人工序列
<220>
<223>p39
<400>42
tagaaaacattctggaatggcaaaagaacccgcaggcaaagctga45
<210>43
<211>40
<212>dna
<213>人工序列
<220>
<223>p40
<400>43
gcccaagcttttagatttcgtttttcagctttgcctgcgg40
<210>44
<211>32
<212>dna
<213>人工序列
<220>
<223>ldh-up-f
<400>44
tacgggatcctcggcgaccgatgaaccgaact32
<210>45
<211>30
<212>dna
<213>人工序列
<220>
<223>ldh-up-r
<400>45
tccgctcgaggactcatcattcctttgccg30
<210>46
<211>29
<212>dna
<213>人工序列
<220>
<223>ldh-dn-f
<400>46
ccgctcgagggcgtaactgaacaccatga29
<210>47
<211>31
<212>dna
<213>人工序列
<220>
<223>ldh-dn-r
<400>47
catgccatggcaaagaaagcgatgaccggca31
<210>48
<211>32
<212>dna
<213>人工序列
<220>
<223>als1-up-f
<400>48
acgcggatccctttggcaatgacgatcagcga32
<210>49
<211>48
<212>dna
<213>人工序列
<220>
<223>als1-up-r
<400>49
tgcatagaaaaaaaaattactttcatagccctcactcctccattttca48
<210>50
<211>28
<212>dna
<213>人工序列
<220>
<223>als-dn-f
<400>50
atgaaaaagccctctttgaaaagggggc28
<210>51
<211>32
<212>dna
<213>人工序列
<220>
<223>als1-dn-r
<400>51
catgccatggggtttcataagaccgctgatga32
<210>52
<211>20
<212>dna
<213>人工序列
<220>
<223>als1-ldh-f
<400>52
atgaaagtaatttttttttc20
<210>53
<211>46
<212>dna
<213>人工序列
<220>
<223>als-ldh-r
<400>53
ccccttttcaaagagggctttttcatttagatttcgtttttcagct46
<210>54
<211>32
<212>dna
<213>人工序列
<220>
<223>als2-up-f
<400>54
acgcggatccgcaaacagctgttcatgaaccg32
<210>55
<211>48
<212>dna
<213>人工序列
<220>
<223>als2-up-r
<400>55
aacgcgctgttgttcttgtatcggcacgggtacatttgaaggatcttg48
<210>56
<211>32
<212>dna
<213>人工序列
<220>
<223>als2-dn-r
<400>56
tccccccgggggtttcataagaccgctgatga32
<210>57
<211>21
<212>dna
<213>人工序列
<220>
<223>als2-pc-f
<400>57
tgccgatacaagaacaacagc21
<210>58
<211>47
<212>dna
<213>人工序列
<220>
<223>als2-pc-r
<400>58
attactttcatagccctcactcctccacgggttcgctacctgcatta47
<210>59
<211>35
<212>dna
<213>人工序列
<220>
<223>als2-ldh-f
<400>59
ggaggagtgagggctatgaaagtaatttttttttc35
<210>60
<211>48
<212>dna
<213>人工序列
<220>
<223>als4-up-r
<400>60
tgccccggcctgcatgcacgtcgacacgggtacatttgaaggatcttg48
<210>61
<211>20
<212>dna
<213>人工序列
<220>
<223>als4-p43-f
<400>61
tgtcgacgtgcatgcaggcc20
<210>62
<211>46
<212>dna
<213>人工序列
<220>
<223>als4-p43-r
<400>62
attactttcatagccctcactcctcctataatggtaccgctatcac46
<210>63
<211>48
<212>dna
<213>人工序列
<220>
<223>als6-up-r
<400>63
tttcttccttaaatttgtacattttccgggtacatttgaaggatcttg48
<210>64
<211>20
<212>dna
<213>人工序列
<220>
<223>als6-pldh-f
<400>64
gaaaatgtacaaatttaagg20
<210>65
<211>46
<212>dna
<213>人工序列
<220>
<223>als6-pldh-r
<400>65
attactttcatagccctcactcctccgactcatcattcctttgccg46
<210>66
<211>48
<212>dna
<213>人工序列
<220>
<223>alsa-up-r
<400>66
tttgtgctataaacggcgagtttcatagccctcactcctccattttca48
<210>67
<211>21
<212>dna
<213>人工序列
<220>
<223>alsa-ldha-f
<400>67
atgaaactcgccgtttatagc21
<210>68
<211>47
<212>dna
<213>人工序列
<220>
<223>alsa-ldha-r
<400>68
ccccttttcaaagagggctttttcatttaaaccagttcgttcgggca47
<210>69
<211>300
<212>dna
<213>地衣芽孢杆菌(bacilluslicheniformis)atcc14580
<400>69
aaggtgacgcctatttcactttctagctgtttaatctgctggctgagcggaggctgagtc60
atgttcagccgaagagctgcttttccgaaatgcagttcttcggcaacaaccataaaataa120
cgaagatggcgcagctccattaatcactcattcctttctgaatgcgatttcagtcgtttt180
acatattaattgtaagacaaagaagtattggaaaacaatttccacaagatgtatatttaa240
taatacaataattttattaaaaattcattgtaaatgaatgaaaatggaggagtgagggct300
<210>70
<211>225
<212>dna
<213>食酸代尔夫特菌(delftiaacidovorans)c17
<400>70
tgccgatacaagaacaacagcgcgttgagcgcctgccggtgggtggccggcgccacttgc60
ttctcggtggcgagcatggtcagaaaaccctcgacttcagcttgccccatttcgcgcgga120
tgtcgaaacccaccatggctgcgggccgtccacaacacaaatgccttggcccagtagaca180
taagccttctcggtctgtaggctgtaatgcaggtagcgaacccgt225
<210>71
<211>426
<212>dna
<213>枯草芽孢杆菌(b.subtilis)
<400>71
tgtcgacgtgcatgcaggccggggcatatgggaaacagcgcggacggagcggaatttcca60
atttcatgccgcagccgcctgcgctgttctcatttgcggcttccttgtagagctcagcat120
tattgagtggatgattatattccttttgataggtggtatgttttcgcttgaacttttaaa180
tacagccattgaacatacggttgatttaataactgacaaacatcaccctcttgctaaagc240
ggccaaggacgctgccgccggggctgtttgcgtttttaccgtgatttcgtgtatcattgg300
tttacttatttttttgccaaagctgtaatggctgaaaattcttacatttattttacattt360
ttagaaatgggcgtgaaaaaaagcgcgcgattatgtaaaatataaagtgatagcggtacc420
attata426
<210>72
<211>301
<212>dna
<213>地衣芽孢杆菌(bacilluslicheniformis)atcc14580
<400>72
gcgataattgaatcatgctgaagaatagtggtgttatgagcgctgaaaatgggaaacctt60
atattgatataggcccgatgcacttgcagataaaaccgtcttttgcaacgcgtttatcaa120
tttttctttagacataaggcatatttttaccagaaaatgtacaaatttaaggaagaaata180
cgtggagacacaaaatagacataaataaaattgaaaaagtatgtgaagtattgcacaata240
tgaatgtgaagaatttcacaaacagcttagtttaaaacaaacggcaaaggaatgatgagt300
c301
<210>73
<211>27
<212>dna
<213>人工序列
<220>
<223>pfla1-f
<400>73
ccggaattcatggaacaatggaaaggt27
<210>74
<211>29
<212>dna
<213>人工序列
<220>
<223>pfla1-r
<400>74
tcgctcgagtggaaatgtcaaacccatag29
<210>75
<211>28
<212>dna
<213>人工序列
<220>
<223>pfla2-f
<400>75
ccgctcgaggacgatggccactgggatc28
<210>76
<211>29
<212>dna
<213>人工序列
<220>
<223>pfla2-r
<400>76
atcggatccctacatcgattcatggaagg29
<210>77
<211>33
<212>dna
<213>人工序列
<220>
<223>adhb-up-f
<400>77
tacgggatccgaacgggaatcggcaaagggatt33
<210>78
<211>33
<212>dna
<213>人工序列
<220>
<223>adhb-up-r
<400>78
tccgctcgagcgatgataaaggctgccgagcta33
<210>79
<211>30
<212>dna
<213>人工序列
<220>
<223>adhb-dn-f
<400>79
accgctcgagtcgtcacactcccattatcg30
<210>80
<211>30
<212>dna
<213>人工序列
<220>
<223>adhb-dn-r
<400>80
catgccatggcgtcgtatttgccgtcagct30
<210>81
<211>30
<212>dna
<213>人工序列
<220>
<223>ack-up-f
<400>81
tacgggatccgaaggctttccggccttact30
<210>82
<211>30
<212>dna
<213>人工序列
<220>
<223>ack-up-r
<400>82
tccgctcgagcatcgtcattccgacgaatg30
<210>83
<211>30
<212>dna
<213>人工序列
<220>
<223>ack-dn-f
<400>83
accgctcgaggagcttccagcattgattgc30
<210>84
<211>30
<212>dna
<213>人工序列
<220>
<223>ack-dn-r
<400>84
catgccatggatgcggtcatctgcgatctt30
- 还没有人留言评论。精彩留言会获得点赞!