一种共表达热激蛋白提高外源蛋白表达量的方法与流程
本发明涉及一种共表达热激蛋白提高外源蛋白表达量的方法,属于酶工程技术领域。
背景技术:
毕赤酵母(komagataellaphaffii,k.phaffii,原命名为pichiapastoris)是重要的蛋白表达平台微生物,能以甲醇或者甘油作为单一的碳源和能源,其中甲醇可作为诱导剂,诱导醇氧化酶(aox1)启动子。毕赤酵母表达系统和其它蛋白表达系统相比较,具备如下优势:具有强诱导性和强启动性启动子,利于外源基因高效表达;蛋白质加工折叠和翻译后修饰的作用,使外源蛋白的结构与天然蛋白相似,从而表现出一定的生物活性;上清液中内源蛋白分泌量非常少,有利于表达产物的分离纯化;外源基因与毕赤酵母染色体通过基因重组的方式整合,避免了外源基因的丢失,为外源蛋白插入酵母并稳定传代提供了可能。
提高毕赤酵母外源蛋白的表达量是当前微生物研究的重要课题之一,在工业中毕赤酵母中高效表达外源蛋白具有相当高的应用价值。毕赤酵母基因组序列的解读,有利于根据高产蛋白的特定属性来改良菌株品系。目前已有多种外源蛋白在毕赤酵母中取得较好的表达效果。
迄今大多数研究都集中在优化发酵条件,建立发酵条件与外源蛋白产量之间关系。但是这种简单的对应关系,未能明确发酵菌株与蛋白产量之间联系的依据。
此外,利用毕赤酵母生产具有较高酶活性的蛋白还面临一些难题,比如,虽高蛋白表达量,酶活性较低。有研究报道利用伴侣蛋白来促进外源蛋白的折叠,从而提高蛋白的表达水平,以及比活力。如smit等通过共表达kar2的方式提高了酿酒酵母中葡萄糖苷酶的表达量;卢鑫等在毕赤酵母中共表达伴侣蛋白(pdi和ero1)提高了米根霉脂肪酶的产量;沙冲等进一步研究发现同时共表达伴侣蛋白pdi和ero1,多种伴侣蛋白通过协同作用,可较大程度提高外源蛋白的活力。另有研究报道,共表达伴侣蛋白并不能提高外源蛋白的表达水平,在有些情况下反而使得外源蛋白的表达水平降低,如当在酿酒酵母中共表达scj1时,外源蛋白人白蛋白的表达水平降低了60%(antioxidants&redoxsignaling,2014,21:414-437)。
目前仅仅局限在利用少数几种伴侣蛋白进行共表达来提高外源蛋白的表达量,急需在研究毕赤酵母高效表达机制的基础上开发其他新型的能够促进外源蛋白表达的因子。
技术实现要素:
本发明提供了一种提高毕赤酵母中外源蛋白表达量的方法,所述方法是在表达外源目的蛋白的同时过表达热激蛋白sti1、fes1、cpr6中的任意一种或者多种。
在一种实施方式中,所述热激蛋白sti1、fes1、cpr6是来源于毕赤酵母的热激蛋白。
在一种实施方式中,所述热激蛋白sti1、fes1、cpr6的氨基酸序列分别如seqidno:1、seqidno:2、seqidno:3所示。
在一种实施方式中,所述外源目的蛋白可以是磷脂酶、脯氨酰内肽酶、脂肪酶、蛋白酶、糖化酶等。
在一种实施方式中,所述方法是在同一宿主中表达外源蛋白和所述热激蛋白。
在一种实施方式中,所述宿主为毕赤酵母。
本发明的第二个目的是提供一种外源蛋白表达量提高的毕赤酵母重组菌,所述毕赤酵母重组菌在表达外源目的蛋白的基础上,还同时过表达了热激蛋白sti1、fes1、cpr6中的任意一种或者多种。
在一种实施方式中,所述表达热激蛋白sti1、fes1、cpr6的氨基酸序列分别如seqidno:1、seqidno:2、seqidno:3所示。
在一种实施方式中,所述外源目的蛋白可以是磷脂酶、脯氨酰内肽酶、脂肪酶、蛋白酶、糖化酶等。
在一种实施方式中,所述外源目的蛋白为紫红链霉菌磷脂酶a2。
在一种实施方式中,所述毕赤酵母重组菌是以毕赤酵母gs115、x33、km71、km71h、smd1168、smd1163等中的任意一种为宿主菌构建得到的。
在一种实施方式中,所述毕赤酵母重组菌是以ppic9k、ppicza/b/c、ppic9k-his、ppic3.5k、ppiczalphaa/b/c、pgapzalpha、pao815、phil-s1、ppinkhc等中的任意一种为质粒载体构建得到的。
本发明的第三个目的是提供所述毕赤酵母重组菌的应用。
在一种实施方式中,所述应用是应用于食品、酿造、造纸、制革、制糖、化工等领域,更具体地,是应用于食品与酿造领域。
本发明的优点和效果:
本发明通过在表达外源目的蛋白的同时过表达热激蛋白sti1、fes1、cpr6,显著提高了外源目的蛋白的表达量。
附图说明
图1为共表达热激蛋白和磷脂酶的重组菌株的摇瓶发酵菌体浓度生长曲线;
图2为共表达热激蛋白和磷脂酶的重组菌株的摇瓶发酵菌体浓度生长曲线;
图3为共表达热激蛋白相关基因菌株的摇瓶发酵蛋白浓度曲线;
图4为共表达热激蛋白相关基因菌株的摇瓶发酵蛋白浓度曲线;
图5为共表达热激蛋白相关基因菌株的摇瓶发酵上清液酶活曲线;
图6为共表达热激蛋白相关基因菌株的摇瓶发酵上清液酶活曲线;
图7为共表达热激蛋白产脯氨酰内肽酶的重组菌菌体生长曲线;
图8为共表达热激蛋白产脯氨酰内肽酶的重组菌产蛋白浓度曲线;
图9为共表达热激蛋白产脯氨酰内肽酶的重组菌的脯氨酰内肽酶酶活曲线。
具体实施方案
(1)生物量测定
采用细胞干重(dcw)表示。取样后置于离心机中离心(12000r·min-1,6min),将菌体保留,加生理盐水悬浮菌体,重复离心去上清,然后放置在90℃烘箱中烘干得细胞干重。
(2)蛋白含量测定
利用考马斯亮蓝法测定发酵液上清中总蛋白浓度。取上述发酵上清液,测定595nm波长下测吸光度,每组设置三个平行,计算上清液中总蛋白浓度。
(3)磷脂酶活力测定
使用spla2assaykit测定磷脂酶a2酶活力,方法步骤见说明书。
(4)脯氨酰内肽酶酶活力测定方法
具体操作步骤见文献(journalofmolecularcatalysisb:enzymatic,2016,125:81-87)。酶活计算公式为:
其中,u·ml-1:酶活力单位;δa:反应体系吸光度变化;v:总反应体系(ml);v:发酵液体积(ml);γ:摩尔消光系数(cm2·mol-1);β:比色杯光程(cm)。
下面是对本发明进行具体描述。
实施例1:共表达热激蛋白相关基因菌株的构建
以小分子量蛋白磷脂酶(21kd)以及大分子量蛋白脯氨酰内肽酶(66kd)两种蛋白作为外源目的蛋白表达对象,构建共表达热激蛋白相关基因的重组菌株。
具体是:
(1)利用pcr方法获取毕赤酵母中热激蛋白(sti1,fes1,cpr6和ssa1)的基因序列(核苷酸序列分别如seqidno:4、seqidno:5、seqidno:6、seqidno:7所示),并在序列两端添加酶切位点。以ppicza为载体,使用限制性内切酶酶asuii和noti,酶切上述基因和载体。利用t4dna连接酶链接目的基因和载体,转化至dh5α感受态细胞,筛选验证获得重组质粒。
(2)将上述构建的重组质粒用限制性内切酶saci或pmei酶切线性化,胶回收目的片段,分别电转感受态细胞(毕赤酵母k.phaffiigs115/ppic9k-mlmh、gs115/ppic9k-pla2-1和gs115/ppic9k-pla2-12感受态细胞),培养并验证阳性菌株,得到了如表1所示的毕赤酵母重组菌。
其中,表达紫红链霉菌磷脂酶a2的毕赤酵母菌株k.phaffiigs115/ppic9k-pla2-1(含1拷贝磷脂酶基因)、k.phaffiigs115/ppic9k-pla2-12(含12拷贝磷脂酶基因)为本实验室之前构建的菌株(参见:刘爱霞,硕士毕业论文,江南大学,2015年)。gs115/ppic9k-mlmh代表以k.phaffiigs115为宿主、ppic9k为载体,重组表达了脯氨酰内肽酶融合蛋白mlmh的重组菌,具体是从质粒ppicza-mlmh(journalofmolecularcatalysisb:enzymatic,2016,125:81-87)中pcr克隆得到mlmh基因,通过酶切连接构建质粒ppic9k-mlmh,然后转化到k.phaffiigs115宿主中得到gs115/ppic9k-mlmh。
表1共表达热激蛋白和外源目的蛋白的毕赤酵母重组菌列表
实施例2:
以小分子量蛋白磷脂酶(21kd)以及大分子量蛋白脯氨酰内肽酶(66kd)两种蛋白作为外源目的蛋白表达对象,分析含有热激蛋白相关基因的重组菌株对外源目的蛋白表达的影响。
其中毕赤酵母摇瓶发酵方法如下:
将甘油管保藏菌株用平板划线法培养,30℃培养3–5d。挑取单克隆至bmgy培养基中,在30℃200r·min-1条件下培养16h–18h至od600达到2–6。将菌液在无菌条件下转移至无菌的离心管中,在6000r·min-1条件下离心10min,弃掉上清收集菌体,用bmmy培养基重悬菌体,28℃250r·min-1条件下培养,每24h添加1%的甲醇诱导表达,定时取样测定发酵液的菌体浓度和发酵上清液的蛋白浓度。
(1)产磷脂酶重组菌株共表达热激蛋白相关基因的摇瓶发酵结果
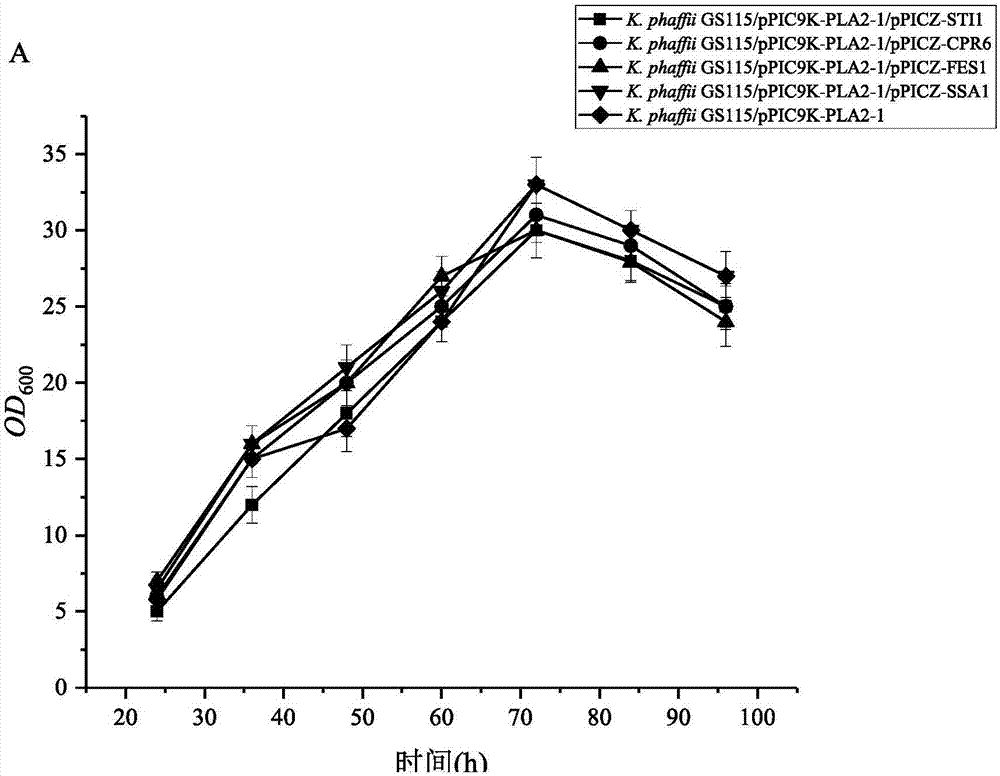
将构建的共表达热激蛋白菌株(表1)与出发菌株(gs115/ppic9k-pla-1和gs115/ppic9k-pla-12)进行摇瓶发酵。结果显示,在96h发酵周期内,菌株的菌体浓度(od600)几乎完全一致,各菌株的生长趋势基本一致,不同共表达菌株间没有明显差异,od600都在72h达到最大值,然后开始下降。由此可见,共表达蛋白对菌株生长没有明显影响(图1~图2)。
摇瓶发酵结果表明,在84h时共表达热激蛋白菌株的胞外总蛋白浓度都达到最大值。共表达同一热激蛋白相关基因,含有高拷贝磷脂酶基因的菌株发酵胞外总蛋白浓度及胞外磷脂酶活力高于低拷贝菌株(图3~图6)。共表达热激蛋白对含有不同拷贝数磷脂酶基因菌株的影响趋势一致,其中提高胞外磷脂酶活力倍数最大的是sti1,达到1.51倍,然后依次是fes1,ssa1和cpr6。
从图5中可以看出,共表达sti1、fes1、ssa1、cpr6和1拷贝磷脂酶基因的重组菌在发酵84h下达到的酶活分别为0.63u·l-1、0.58u·l-1、0.52u·l-1、0.45u·l-1,相比对照菌株gs115/ppic9k-pla-1,分别提高了1.51倍、1.39倍、1.24倍、1.08倍。
从图6中可以看出,共表达sti1、fes1、ssa1、cpr6和12拷贝磷脂酶基因的重组菌在发酵84h下达到的酶活分别为0.67u·l-1、0.64u·l-1、0.57u·l-1、0.56u·l-1,相比对照菌株gs115/ppic9k-pla-12,分别提高了1.24倍、1.19倍、1.05倍、1.04倍。
(2)产脯氨酰内肽酶重组菌株共表达热激蛋白相关基因摇瓶发酵
以表达脯氨酰内肽酶基因的毕赤酵母作为对照菌株,分析共表达热激蛋白对其的影响,结果如图7、图8、图9所示。
如图7、图8所示,共表达菌株与对照菌株(k.phaffiigs115/ppic9k-mlmh)菌体浓度(od600)生长趋势相似。当发酵96h时,共表达fes1使得胞外总蛋白浓度由0.20g·l-1提升至0.26g·l-1,而ssa1、cpr6、sti1菌株,胞外总蛋白浓度依次是0.25g·l-1、0.24g·l-1、0.23g·l-1。上述共表达基因fes1、ssa1、cpr6、ti1的重组菌,与对照相比,胞外总蛋白浓度分别提高了1.30倍、1.25倍、1.20倍、1.15倍。
如图9所示,共表达热激蛋白也同样能够提高脯氨酰内肽酶的表达水平,提高程度依次是fes1,ssa1,cpr6和sti1。当发酵96h时,共表达菌株的酶活达到了最高。共表达菌株fes1、cpr6、ssa1、sti1的酶活分别为210u·l-1、195u·l-1、190u·l-1、185u·l-1,与对照菌gs115/ppic9k-mlmh的酶175u·l-1相比,分别提高了1.20倍、1.11倍、1.09倍、1.06倍。
虽然本发明已以较佳实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可做各种的改动与修饰,因此本发明的保护范围应该以权利要求书所界定的为准。
序列表
<110>江南大学
<120>一种共表达热激蛋白提高外源蛋白表达量的方法
<160>7
<170>patentinversion3.3
<210>1
<211>572
<212>prt
<213>人工序列
<400>1
metserserglugluphelysalaglnglyasnglnalapheglnala
151015
lysasptyrglulysalavalserphephethrglnalailegluala
202530
serprothrproasnhisileleupheserasnargseralaalatyr
354045
alaserleuglyglntyrglnaspalaleuaspaspalaasnlyscys
505560
valgluileasnglysertrpalalysglytyrasnargvalglyala
65707580
alahistyrglyargglyglutrpaspglualahislysalatyrser
859095
lysalaleugluleuaspproalaasnlysmetalalysgluglyleu
100105110
asngluthrgluilealaargaspalaglyasnaspvallysasnile
115120125
pheseraspalaglymetvalglulysleulyslysasnprolysthr
130135140
alagluleumetlysaspprogluleuvalalalysvalglnlysleu
145150155160
glnthraspprolyssermetserglngluleupheseraspproarg
165170175
leumetthrvalmetglyalametleuglyvalaspleuglyvalgln
180185190
proserglnglnseralaproglngluaspthrprovalproaspala
195200205
tyrprogluproserserlysprogluthrasnthrthrseralalys
210215220
asnalaalaalaprogluproglulysglualathrprogluproval
225230235240
aspasnserlysgluglualaaspasnleulysglnglnalaasngln
245250255
leutyrlyslysargglnpheaspglualailegluleutyrasnlys
260265270
alatrpgluthrpheglnaspilethrtyrleuasnasnargalaala
275280285
alaglupheglulysglyasptyraspalathrilegluthrcysglu
290295300
asnalavalglulysglyarggluleuargalaasptyrlysleuval
305310315320
alalysserphealaargleuglyseralatyrleulyslysaspasp
325330335
leuproasnalailelysphepheglulysserleuthrgluhisarg
340345350
serproaspvalleuserlysleuargalaalaglualaaspleulys
355360365
lyslysglualagluglutyrileaspproglulysalaglugluala
370375380
argleuglnglylysaspphephethrlysglyasptrpproalaala
385390395400
vallysalatyrthrglumetileasnargalaprolysaspalaarg
405410415
glytyrserasnargalaalaalaleualalysleumetserphepro
420425430
aspalavallysaspcysasplysalailegluleuaspproserphe
435440445
valargalatyrilearglysalathralaleuilealametlysasp
450455460
pheasnlysalametthrthrleugluglualaargthrvalaspala
465470475480
aspthrasngluglylysalaalaasngluileasnglyleutyrtyr
485490495
lysalaserserglnargphealaalaileaspglygluthrproglu
500505510
glnthrphegluargalaserlysaspprogluvalseralaileleu
515520525
glnaspprovalmetasnserileleuglnglnalaarggluasnpro
530535540
alaalaleuglngluhismetlysasnprogluvalalalyslysile
545550555560
asnileleuilealaalaglyvalileargthrarg
565570
<210>2
<211>287
<212>prt
<213>人工序列
<400>2
metglulysleuleulystrpserileseralaasnserglnaspglu
151015
gluserlyslysargalaglyglnproaspprogluleuleualagln
202530
leupheglyglyproaspgluprothrleumetasnglusermetlys
354045
valileasnasnprogluthraspleugluasnlysgluvalalaphe
505560
aspasnpheglumetleuilegluasnmetaspasnalaasnasnile
65707580
gluasnmethisleutrpproproleuleuglnasnleuaspserglu
859095
tyrileserleuargargphealacyssercysileglythralaval
100105110
glnasnasnprolyscysglngluhispheleulyshisseraspgly
115120125
ilelyslysleuilealaileserserasnserglugluaspaspser
130135140
vallysleulysalaleutyralaleuserasnvalleuarghisasn
145150155160
lysproalatyrgluglupheserasnglnglyglytrpasngluile
165170175
serproleuleuthrserleuaspasnserasnglulysilelysleu
180185190
argthrleuserleuleuserserileilethrasnglyleuserglu
195200205
gluvalilegluhisleuhisasnasnlysvalvalilesermetleu
210215220
lysvalleulysalagluglyhisilethrserileasplysvalleu
225230235240
serleuleuthrthrleuvalglnasnlyspheargpheseraspglu
245250255
gluileasnleuileargglnserleuthrthrilealagluleuglu
260265270
aspserleuasnasnaspaspleuasnthrilelysglnvalleu
275280285
<210>3
<211>361
<212>prt
<213>人工序列
<400>3
metthrproargserhisilephepheaspileserileasnasngln
151015
proalaglyargileilephegluleupheasnaspilevalprolys
202530
thralagluasnpheargalaleuserthrglyglulysglyilegly
354045
lysserglylysproleuhistyrlysglyserthrphehisargile
505560
ilelysaspphemetvalglnglyglyaspphethrasnglyasngly
65707580
thrglyglygluseriletyrglyglulysphegluaspgluasnphe
859095
glnleuthrhisasplyspropheleuleusermetalaasnalagly
100105110
proglythrasnglyserglnphepheilethrthrvalprothrpro
115120125
hisleuaspasnlyshisvalvalpheglylysvalilealaglylys
130135140
alathrvalarglysilegluargasnsergluglyglualaproile
145150155160
gluprovalvalilegluaspcysglygluleuprogluaspalaasp
165170175
leuthrileseraspgluthrglyasplystyrglugluvalleulys
180185190
aspasngluasnileaspileaspaspphegluglnvaltyrglnala
195200205
ilethrgluilelysgluleuglythrlystyrphelysasnglyasp
210215220
thrlysilealapheglulystyrglnlysalaalaasntyrleuleu
225230235240
glutyrileproseraspleuserglugluglnserserlysleuglu
245250255
leuleulysthrservalpheserasnvalalaleualaglyleulys
260265270
valserlysphelysaspthrilelystyralathrleuvalileglu
275280285
aspgluseralaaspalalysalalysserlysglytyrtyrargarg
290295300
glyseralatyrserserleulysaspgluaspseralaileserasp
305310315320
pheglnlysalaleugluleuserproglyaspproalailesergln
325330335
serleuglnargthrthrlysalaarglysaspargleualalysglu
340345350
lysalaalaleuserlysphepheglu
355360
<210>4
<211>1719
<212>dna
<213>人工序列
<400>4
atgtcatctgaagaattcaaagcccagggaaaccaggctttccaagccaaggattacgaa60
aaggcggtgtcatttttcacccaggcaattgaggcctctcccacccctaatcacatttta120
ttctcaaatcgttcagccgcctacgcttccttaggacagtaccaagatgcattggatgac180
gcaaataagtgtgttgaaatcaatggttcttgggccaaaggttacaacagagtcggtgct240
gctcactacggaagaggtgagtgggacgaagctcacaaggcctactccaaagctctagaa300
ttagaccctgctaacaagatggccaaggagggactcaatgagacagagattgcaagagat360
gctggtaatgatgtgaagaacatcttttccgatgccggcatggtggaaaaattgaaaaag420
aacccaaagactgcagaactgatgaaagacccagagttggtggcaaaagtgcaaaagttg480
caaactgatcccaagtcaatgtctcaggaattgttttccgatccaagattaatgaccgta540
atgggtgccatgcttggtgttgatttgggtgtccagccatctcaacagtctgccccacaa600
gaggacactcctgtgcctgacgcatacccagagccaagttcgaagccagaaactaatact660
acttctgcaaaaaacgcagctgctccagaacctgaaaaagaggccacacctgagccagtt720
gacaattctaaagaggaggccgacaatttgaaacaacaggcaaaccaattgtacaagaag780
aggcagttcgatgaggctattgagctttacaacaaggcctgggaaactttccaggatatt840
acttatttaaacaaccgtgctgctgcagagtttgaaaagggagactatgatgctaccatt900
gaaacctgtgaaaatgctgtcgagaaaggtagagaattaagagctgactacaagctggtt960
gctaaatcctttgcacgtcttggatctgcctacttgaagaaagatgacttgccaaacgca1020
atcaaattctttgaaaaatctttgacagaacaccgtagccctgatgtcctcagcaaacta1080
agagctgcagaggcagacctaaagaaaaaagaggccgaggaatatatagatcctgagaag1140
gcagaggaggcccgtttgcagggaaaggattttttcaccaaaggtgactggcctgccgca1200
gtgaaagcatacacagagatgattaacagagctcctaaagatgccagaggttactccaac1260
cgtgctgcagctttagccaaactgatgtccttccccgacgccgtgaaagactgtgacaag1320
gcaatagagctggacccaagctttgtcagggcttatatccgtaaggccactgccttgatt1380
gccatgaaggacttcaacaaggctatgaccactttggaagaggcaagaacggtcgacgct1440
gacactaatgagggaaaagctgccaacgaaatcaacggcttatactacaaggcctcgtcc1500
cagagatttgctgcaattgatggggaaactcccgaacagacttttgaacgtgctagcaag1560
gaccctgaagtttcggctattctccaagatcccgtgatgaactctattctacagcaagca1620
agagagaaccctgctgctttgcaagagcacatgaagaaccccgaagttgcaaagaagatc1680
aacatactcattgctgccggtgtcattcgtactcgttaa1719
<210>5
<211>864
<212>dna
<213>人工序列
<400>5
atggagaagctactcaaatggtcgatatcagcaaactctcaggatgaggaatccaagaaa60
agagctgggcagccagacccagaactgctggctcagttgtttggaggccccgacgaaccc120
acattgatgaacgaatccatgaaggtgattaacaaccccgaaacagatttagaaaacaaa180
gaggttgcctttgacaattttgaaatgctcattgaaaatatggacaatgctaataacatt240
gaaaatatgcatctttggcctcctttgcttcaaaacctcgatagcgagtatatctctcta300
aggagattcgcctgttcttgtataggcactgcagttcagaacaaccccaaatgtcaggaa360
cattttctgaagcactctgatggtataaagaagttgattgcaatctcttctaattcggag420
gaggatgattcggtcaaactaaaggccctttatgcattgagcaatgttctaaggcataat480
aaaccagcttacgaagagttcagcaaccaaggtggttggaatgagataagcccgctgttg540
acatctttggataactctaatgagaaaataaagcttagaacgctgtctttgctttcctcc600
atcattaccaatggactatcagaagaagttatcgaacatttgcacaacaacaaggtggtt660
attagtatgctcaaggttttgaaagcagaagggcacattacctctatagacaaagttttg720
agtcttctgaccactctagtgcagaataagttcagattttcagatgaagagattaacttg780
attcgccaatcgttgacaactatagcagaactagaggacagtctgaacaacgatgatttg840
aataccataaagcaagtactttaa864
<210>6
<211>1086
<212>dna
<213>人工序列
<400>6
atgactccccgttctcatattttctttgacatctccatcaacaaccagccagctggcaga60
ataatctttgagctcttcaatgacattgttcctaagacagcagagaattttagagcttta120
tctactggtgagaaaggtataggtaagtctgggaaaccattgcactacaagggttctact180
ttccataggatcattaaggattttatggtacaaggtggtgactttaccaacggtaacggt240
actggaggcgaatccatatatggagaaaaatttgaagatgaaaatttccaattgactcat300
gacaaaccgttccttctctctatggcaaacgctggaccaggaactaatggatcccagttt360
tttatcaccaccgttcctactcctcatctggataacaagcatgtagtgtttggaaaagta420
attgctggtaaagccacagttagaaagattgaaagaaactccgaaggtgaagctccaatt480
gaacccgttgtcattgaggactgtggtgaacttccagaagacgcagatttgaccatctcc540
gacgagactggagacaagtatgaggaagttctgaaagataatgagaacatagacatcgat600
gactttgaacaggtctaccaggccatcactgaaatcaaagaattgggtacaaagtatttc660
aaaaatggtgacaccaaaatcgccttcgaaaagtatcaaaaggctgctaattatttgctg720
gaatacataccatcagatttatcagaggaacagagctctaagttggagctgctaaaaaca780
tctgtcttctccaacgtggcattggctggactgaaagtttccaagttcaaagacacgatt840
aagtatgccacattggtcattgaggatgaatctgcggatgcaaaggccaagtccaaaggc900
tactaccgtagaggaagtgcttacagctcactgaaagacgaagattcagccatctcagat960
ttccagaaagcacttgaattatccccaggtgatcctgcaattagccaatctctacaaaga1020
accacgaaggccagaaaagaccgtcttgccaaagagaaagctgctttgtctaagttcttt1080
gagtag1086
<210>7
<211>1740
<212>dna
<213>人工序列
<400>7
atgccagctgtcggtattgatttaggaactacatactcatgtgtggcacatttcgccaat60
gatcgtgttgagatcattgcaaatgaccagggtaacagaactaccccatcattcgtcgct120
ttcactgacaccgagagactgattggtgacgctgctaagaaccaagctgctatgaaccca180
gccaacaccgttttcgacgccaagcgtttgatcggtagaaagttctcagatgctgagacc240
caagctgatatcaagcacttcccattcaaggtcgttgacaagggtggaaagccaaacatt300
caagttgaattcaaaggtgagactaaggtcttcactccagaagagatctcttccatggtt360
ttgaccaagatgaaggacactgctgagcaattccttggtgacaaggttaacgatgccgtt420
gtcactgtcccagcttacttcaacgactctcagagacaggccactaaggatgctggtctc480
attgccggtttgaacgttatgagaatcatcaacgagccaaccgctgctgccattgcctac540
ggtttggacaaaaaagctgagggtgagaagaacgttctgatcttcgacttgggtggagga600
actttcgatgtttctttgctgtccattgaagacggtattttcgaggtcaaggctactgct660
ggtgacactcacttaggtggtgaggactttgacaacagattggtcaaccacttcatcgct720
gaattcaagagaaagaacaagaaggacttgtcttcaaaccaaagagctttgagaagattg780
agaaccgcctgtgagcgtgctaagagaactttgtcttcttctgctcaaacttccatcgag840
atcgactctttgtttgaaggtgttgacttttacacttctttgaccagagccagattcgag900
gaactgtgtggtgatctattcagatctaccattgagcctgtggagaaggttttgaaggac960
gctaagttggacaagtcccaagtcaacgagattgtcttggttggtggttccaccagaatt1020
ccaaaggtccaaaagttggtgtctgacttcttcaacggtaaggaaccaaacagatctatc1080
aacccagatgaggctgttgcctacggtgccgctgtgcaagctgctattctttccggtgac1140
acttcctccaagacccaagatttgttgctgttggacgttgctcctctgtccctgggtatt1200
gagactgctggtggtatcatgaccaagctgatcccaagaaactctaccatcccaaccaag1260
aagtccgagactttctccacttacgctgacaaccaaccaggtgtgttgattcaggtatac1320
gagggtgagagagccaagactgccgacaacaacttattgggtaagttcgagctgtctggt1380
attcctccagctccaagaggtgttcctcagatcgaggtcactttcgacatggatgccaat1440
ggtatcttgaacgtctctgccgttgagaagggtaccggtaaggctcaacagatcactatc1500
accaacgacaagggtagattgtccaaggaagacattgaggccatgatctctgaggctgag1560
aagtacaaggatgaagatgagaaggaggctgccagaatccaagctagaaacgctctggag1620
tcttactcattctctctgaagaacaccttgaacgagaaggaagtcggtgagaaattggat1680
gctgccgacaaggagtctttgaccaaggctattgacgagactacctcatggatcgacgag1740
- 还没有人留言评论。精彩留言会获得点赞!