一种组氨酸激酶基因Hisk2301的用途的制作方法
本发明属于生物技术领域和遗传工程领域,涉及一种组氨酸激酶基因hisk2301的用途。
背景技术:
产油酵母细胞中富含多不饱和脂肪酸(polyunsaturatedfattyacids,pufas),pufas在人体内具有广泛的生理功能,其合成变化会导致多种疾病,比如心血管疾病、肥胖、非胰岛素依赖性糖尿病、高血压、神经性疾病和癌症等。pufas在细胞膜生物学方面也具有重要的功能,细胞膜膜脂中pufas相对含量对大多数生物膜的物理性质具有重要的影响,比如细胞膜的流动性,稳定的流动性对于维持细胞膜正常功能具有十分重要的作用。传统的pufas来源在产量、质量、生产成本以及安全性等方细胞中油脂方面的问题无法满足市场的大量需求,研究开发新的pufas资源已成为热点。产油酵母成分和常见的植物油相类似,而且产油酵母中油脂具有独特与明确的生物化学和物理化学性质,作为生活中植物食用油的替代品以及生产生物柴油的原料,在工业上具有重要的潜在应用价值。除了通过发酵工艺学研究获得高产量油脂外,研究不同条件下提高脂肪酸脱氢酶活性或者基因表达水平来提高细胞中pufas的组成和含量,也已成为该领域的研究热点课题。
长期以来人们都在研究如何从动植物油脂中提取pufas,但因为动植物的生长随着季节、地理位置等的影响而不断发生变化,所以pufas含量和构成也随之而变,况且从动植物中提取pufas的成本高,周期长,不能适应市场的需要。另外动植物油脂资源含油量及不饱和脂肪酸类型、比例均受到一定的限制。因此,近年来人们一直在探索利用pufas的新来源---即利用微生物技术来生产pufas;微生物具有发酵周期短、油脂含量高、培养成本低、不受原料控制、对环境的适应性强、生物转化率高等优点。
组氨酸激酶(histidinekinases,hk)是一种跨膜转移酶类蛋白质,在信号转导过程中可作为信号分子的细胞受体。hk一般作为双组份信号转导系统(two-componentsignaltransductionsystems,tcss)成分来传递信号,双组份信号转导系统普遍存在于原核和真核生物中,主要由一个hk和一个应答调控蛋白(responseregulatorprotein,rr)组成。双组份信号转导系统参与细胞生长、胁迫抗性、次级代谢产物调控、维持细胞毒力等多种机制的调控。因此,hk在很多细胞生物学过程中起到重要的调节作用,但是目前没有关于hk促进多不饱和脂肪酸合成的研究报道。
技术实现要素:
本发明的目的是提供一种组氨酸激酶基因hisk2301的新用途,即组氨酸激酶基因hisk2301在低温下生产多不饱和脂肪酸中的应用。
组氨酸激酶基因hisk2301从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235中分离得到,该基因核苷酸序列如seqidno:1所示或该核苷酸序列的片段,或与seqidno:1互补的核苷酸序列,该基因序列长为3888bp(碱基),该基因编码的氨基酸序列如seqidno:2所示的多肽或其片段。
本发明将组氨酸激酶基因hisk2301直接与不同表达载体(质粒、病毒或运载体)连接构建重组载体,并在低温下促进红冬孢酵母ym25235生产多不饱和脂肪酸。
具体涉及从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235中克隆的组氨酸激酶基因hisk2301插入prh2034质粒,构建了重组表达质粒prhhisk2301,将其转入红冬孢酵母ym25235中进行过表达,获得转基因菌株ym25235/prhhisk2301,并对其表达特性进行研究,为阐明hisk2301基因与其低温适应性机制和低温下多不饱和脂肪酸的合成机制奠定基础。
本发明的有益效果:本发明从红冬孢酵母(rhodosporidiumkratochvilovae)ym25235的染色体基因组中分离得到双组份系统的核心分子组氨酸激酶的基因hisk2301。该基因全长3888bp,此前的研究认为hisk2301主要参与渗透压胁迫的响应。本研究表明基因hisk2301不但有抗渗透胁迫的功能,且与盐压抗性和红冬孢酵母中pufas的低温合成有关,该基因在低温下能提高红冬孢酵母ym25235中催化油酸转化为亚油酸和亚麻酸的δ12/15-脂肪酸脱氢酶基因rkd12基因的mrna转录水平的提高,从而引起细胞膜脂中pufas的合成量显著增加。本研究将有助于阐明红冬孢酵母ym25235中亚油酸和α-亚麻酸低温合成调节的信号转导途径,揭示低温条件下ym25235菌株中亚油酸和α-亚麻酸合成的调节机制,而且也将有助于进一步揭示真菌中多不饱和脂肪酸合成的调节机制,并为阐明真菌低温适应机制的研究提供参考,而且探索低温条件下多不饱和脂肪酸合成的调节机制有助于利用相关的合成调节过程,为今后大规模生产多不饱和脂肪酸的研究与应用奠定基础。
附图说明
图1为本发明的红冬孢酵母ym25235的hisk2301基因pcr扩增图;
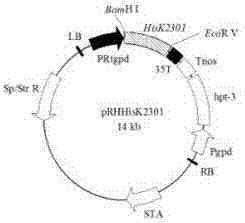
图2为重组质粒prhhisk2301的质粒图谱;
图3为重组质粒prhhisk2301限制性酶切分析;其中:1、空质粒prh2304的bamhⅰ、ecorⅴ双酶切;2、重组质粒prhhisk2301的bamhⅰ、ecorⅴ双酶切;3、hisk2301基因的pcr产物;4、dna分子量标记dl15000;
图4为低温条件下hisk2301基因转化的红冬孢酵母ym25235细胞脂肪酸气相色谱分析a:ym25235/prh2304at15℃;b:ym25235/prhrkhisk2301at15℃。
具体实施方式
下面结合附图和实施例对本发明作进一步详细说明,但本发明保护范围不局限于所述内容,实施例中使用的试剂和方法,如无特殊说明,均采用常规试剂和使用常规方法。
实施例1:红冬孢酵母(rhodosporidiumkratochvilovae)ym25235组氨酸激酶基因hisk2301基因克隆
采用omega试剂盒e.z.n.afungalrnakit提取红冬孢酵母ym25235总rna,反转录试剂盒takarafirststrandcdnasynthesiskit合成cdna。根据红冬孢酵母ym25235的转录组序列设计特异性引物(引物1和引物2)进行pcr扩增;反应所用引物、组份和扩增条件如下:
引物1:hisk2301-f:5’-cacaccgcggatccatgccgcagacgcc-3’(seqidno:3)
引物2:hisk2301-r:5’-ctgaccgcgatatctcactgcgggtcgtt-3’(seqidno:4)
(ggatcc为bamhⅲ酶切位点,gatatc为ecorv酶切位点);
pcr扩增体系如下(50μl):
pcr扩增条件:95℃预变性5min,再用95℃变性30s,62℃退火15s,72℃延伸2min30s进行30个循环,最后72℃彻底延伸10min。反应完后取产物2μl,然后在浓度为1%的琼脂糖凝胶中,进行电泳分析,结果如图1所示。经凝胶成像系统成像确认片段大小正确后,用百泰克生物技术有限公司多动能dna纯化回收试剂盒回收目的片段,然后将pcr扩增得到的目的基因连接到pmd-18t上,连接产物转化至大肠杆菌dh5α感受态细胞中,用含有氨苄青霉素(amp+)的lb固体平板进行筛选,挑取平板上的转化子进行菌落pcr筛选阳性克隆,然后送去上海生工测序。测序结果显示,获得一段3888bp长的序列,命名为hisk2301,序列组成如seqidno:1所示的核苷酸序列。
实施例2:hisk2301过表达载体prhhisk2301的构建
以反转录的ym25235cdna为模板,用hisk2301-f和hisk2301-r作为引物扩增hisk2301编码序列,获得的hisk2301片段大小约3888bp,将扩增获得的hisk2301片段经bamhⅰ、ecorⅴ两个限制性内切酶进行酶切后,连接到表达载体prh2304上获得重组质粒prhhisk2301(图2)。将获得的重组质粒转入大肠杆菌dh5α中扩增,再经菌落pcr验证后提取重组质粒,并用bamhⅰ、ecorⅴ对prhhisk2301进行双酶切验证。结果表明,重组质粒prhhisk2301经双酶切后产生了3.8kb和10kb左右的两个条带(图3第2泳道),这两个条带分别与hisk2301片段和prh2304载体经双酶切后的片段大小一致,初步表明重组质粒prhhisk2301构建成功。用测序引物进行测序,将酶切验证正确的质粒送出测序进一步进行验证。测序结果表明,测序所获得的序列与目的序列完全一致,没有出现任何碱基突变和缺失等。
实施例3:hisk2301基因与低温合成多不饱和脂肪酸关系的分析
1、重组质粒prh2034-hisk2301转化红冬孢酵母ym25235菌株
采用醋酸锂转化法将重组质粒prh2034-hisk2301转化至红冬孢酵母ym25235菌株,以含潮霉素b(hygromycinb)终浓度为150µg/ml的ypd培养基筛选转化子;提取基因组dnapcr验证转化子。
2、hisk2301基因与红冬孢酵母脂肪酸含量变化的分析
将转入空质粒prh2304和重组质粒prhhisk2301的两株转基因红冬孢酵母ym25235分别在30℃培养24h后,立即将培养物置于15℃继续培养24h进行冷处理,冷处理后分别提取两株转基因酵母的脂肪酸并进行甲酯化,以转化空质粒prh2304的红冬孢酵母菌株作为对照,进行气相色谱分析相同处理条件下两个质粒转化菌株的脂肪酸含量变化。图4中的a图和b图分别代表空质粒prh2304转化的对照菌株和重组质粒prhhisk2301转化的过表达菌株的脂肪酸色谱图,由图可以看出,重组质粒prhhisk2301转化菌株在13.3min处的亚油酸(c18:2la)(灰色箭头所示)值明显增加,其含量占总脂肪酸含量的29.53%(表1),而转化空质粒prh2304的对照菌株中亚油酸含量占总脂肪酸含量的23.47%。因此在相同的处理条件下增加了25.82%。在亚油酸含量增加的同时,重组质粒prhhisk2301转化菌株在15.5min处的α-亚麻酸(c18:3ala)峰值(图4黑色箭头所示)也明显增加,其脂肪酸含量占总脂肪酸含量的14.17%(表1),而对照菌株中α-亚麻酸含量占总脂肪酸含量的11.04%,增加了28.35%。这些结果表明,组氨酸激酶hisk2301的过表达引起红冬孢酵母菌株中的亚油酸和亚麻酸两种多不饱和脂肪酸的含量增加。
表1:15℃条件下hisk2301转基因红冬孢酵母ym25235细胞中脂肪酸含量
序列表
<110>昆明理工大学
<120>一种组氨酸激酶基因hisk2301的用途
<160>4
<170>patentinversion3.3
<210>1
<211>3888
<212>dna
<213>rhodosporidiumkratochvilovae
<400>1
atgccgcagacgcccacaccgagcaccgagccagaccccttcgccgcctcgttcgcctcg60
tccccctcgccagctccgcccccgccgccgcgcccgccgctcgccgcacacctcttctcg120
tccggctcgagcgcgtccagcgccgccgtctcgactcccgcgacctccgtcttcagctac180
tctaccagccccgccagcagcttcgtcgacatgaccgcctgccctacccccgccccggcc240
ctcgccctcgcgcaggaagccggcgcagtgggcctcagccccgagcccctctcgcccgac300
aacatgagcaggccgctcgcgtccgcccagcccctccacaccttcccgaccttcctcctc360
tcgctcctcaccgccctcgagagcgaccccgcgagcgtccgctcccacctcgccaacctc420
gcagcgtcccacgccgcgcccgcgaccttctcccccacgcccgagggcaagcccgtcgag480
acggacgccatcatcgcgtgcgcgtcccgcatcgccgaccgcctccaggccgccgagcgc540
ccgcaggacgaacccgtccagccggcgagcgaacccgcagccgaggaggaggccgttccg600
ccgccggtggtagcgcccgccgtgtcgcggacgacgagcgacgactcggtgccgccgaag660
aagcggcccgtcgtccccggcgacacggtcgaggaagtgcgccgcaactgcgaggaccag720
atccaggcgctcaaggtcctccacgccgaggagctccaccgcgcgcagctctcgcacgac780
aacgaggtccgttccatcgccgtcgtcacgtctgccattgcgcgcggagacctcacgaag840
acgatcgacgccgaggtcgagggcgagatggcgacgctcaaggagacgatcaacgacatg900
gtgtacaagctccgcctcttctcgagcgaggtcacgcgcgtgtcgctcgacgtcggcacg960
atgggccagctcggcggacaggccgtcgttaccggcgtcgaggggacgtggaagaccttg1020
acggagacggtcaacacgatggcgacgcagctcacggttcagatccgctccatcgccatc1080
gtcacgtccgccatcgcgcggggcgatctctcgaagacgatcgacgccgaggtgcagggc1140
gagatggcgaccctcaaggagacggtcaactcgatggtgacgcgcctccgcgtcttctcg1200
agcgaggtgacgcgcgttgcgcgcgaggtcggcactgatgggcagctcggagggcaggcg1260
gtcgtgcatggcgtggaggggacgtggaaggagcttaccgactcggtcaaccgcatggcg1320
cagaacctcacgctgcaggtgcgcgagatcgcgagcgtcaccaaggcggtcgcgaacggc1380
gacctgagcaagacggtcgacattggcgcgtcgggcgagatccgggagctcaagacgacc1440
gtcaactcgatggtcgcgcaactccgccgcttcgccgccgaggtcacacgcgtcgcgctc1500
gaagtcggcaccgaagggcagctaggcggcacggcgaacgtcgagggcgtgcagggcgag1560
tggaagtcgctcgtgtcctcggtcaacacgatggcgatgaacctcacgcagcaagtgcgc1620
tccatcgccgacgtcacgcgcgctgtcgccgagggagacctttcgaggaagatcgatgtc1680
aaggtcaagggtgagatgctcgacctcaagctcacggtcaactcgatggtcgattcgctc1740
cggctgttcgcagccgaagtgacgcgcgtcgccaaggaggtcggcacggacgggcaactc1800
ggtggccaggcgtacgtgatcaacgtctcgggcgagtggaagtctctcgtcgactcggtc1860
aaccagatgtgcggcaacctcaccgaccaggtccgctccatcgccaaggccacgaccgcc1920
gtcgcgaggggcgacctcacgcagaaggtcatgatcgaggcgaacggcgaggtcctccag1980
ctcgtcgttacgatcaacgagatggtcgaccgcctcgcaacattcgcgtccgaggtcacg2040
cgcgtcgcgcacgaggtcgggaccaagggcaacctcggcgtgacggccaaggtcgacaac2100
atcgaggggacgtggcaggagatcacgaacaacgtcaacacgatggcgacgaacctcacg2160
tcgcaggtgcgcgcgtttgcgcagatctcggcggccgcgaccgagggcgacttcagctcg2221
tttgtcacggtcgaggcgagcggcgagatggactcgctcaaaaccaagatcaacaagatg2281
gtcttctcgctccgcgactcgctgcagaagaacacgatggcgcgcgaggcggccgagctc2341
gcgaaccgctccaagagcgagttcctcgccaacatgtcgcacgagatccggacgccgatg2400
aacggcatcatcggcctgacgggcgtcacgctcgagacggacctcacgcggcagcagcgc2460
gagaacctcatgatcgtctcgaacctcgccaactcgctcctcctcatcatcgacgacatc2520
ctcgacatctccaagatcgaggccggccgcatgacggtcgagcagatcccgttcagcgtc2580
cgctcggccgtgttcggcatcctcaagacgctcgccgtcaaggcgacgcagagccggctc2640
gacctcatgtactcggtcgagagcgacatccccgacctcctcgtcggcgacccgttccgc2700
ctccggcaggtcatcaccaacctcgtcggcaacgcgatcaagttcacgcagcgtggccag2760
gtcgccctctcgtgccgactcgccgccgccgacatggaggagaagacgtaccagctcgag2820
ttctgcatcagcgacacggggatcgggatcaagcccgacaagctcaacctcatcttcgac2880
acgtttgcgcaggcggacggctccacgacgcgcaagtacggcgggaccggcctcgggctc2940
accatttcgaagcgtctcgtgcagctcatgggcggcgagctctgggtcacgtcgcagttt3000
gggcgcgggtcgcagttctactttacgatccagtgccgcatcggcgagtggaacctcgac3060
caggtgcggcagaagacgctcatcccgcacccggggcgctgcatcctcttcatcgacacg3120
ctccaccacgacccgagcgtgatcgacagcgtcgagcagctcgggctcgagatcaccgtc3180
gtcgactcgctcgaggaggcgtgcatgctcgaccactcgcagaacggctatttcgacacc3240
gtcctcgtcgaccagctctcgatcgtcgagcgtctccgcgacgtcgagcacctccgctac3300
atccccctcgtcctgattacgccgcagatcccgcagctcaacctcaagtactgcctcgac3360
tttgggatcgccaactgcgtcgagtcgccgacgaacgcgcaggacatgtgcaacgcgctc3420
ctccctgcgctcgaggcgagcaaccggataccctcggagcgcggcggcgacgcgtctttc3480
aaggttctcctcgccgaggacaacatcgtcaaccagaaagtcgcgctcaaattcctcgag3540
agcgcggggcaccagagcgaggtcgtcgagaacggcgcgctcgcgctcgaggcggtccgc3600
aagaactactacgacatcgtcctgatggacttgagcatgccgatcatgggcgggcaggag3660
gcgacgcagatcatccgccagttcgagcaggacaacggcctcgagcggctgcccatcgtc3720
gccctcacggcgcacgcgatgctcggcgaccgcgagaagtgcatccaggccggcatggac3780
gactacctcaccaagccgctccgcaagccggacctgctcgccaccatcaacaagattgtc3840
ttggcgaggcgagcgggttcgtctcccaccgcgttgcgcttcttctga3888
<210>2
<211>1295
<212>prt
<213>rhodosporidiumkratochvilovae
<400>2
metproglnthrprothrproserthrgluproaspprophealaalaserphealaser
110
serproserproalaproproproproproargproproleualaalahisleupheser
30
serglyserseralaserseralaalavalserthrproalathrservalphesertyr
50
serthrserproalaserserphevalaspmetthralacysprothrproalaproala
70
leualaleualaglnglualaglyalavalglyleuserprogluproleuserproasp
90
asnmetserargproleualaseralaglnproleuhisthrpheprothrpheleuleu
110
serleuleuthralaleugluseraspproalaservalargserhisleualaasnleu
130
alaalaserhisalaalaproalathrpheserprothrprogluglylysprovalglu
150
thraspalaileilealacysalaserargilealaaspargleuglnalaalagluarg
170
proglnaspgluprovalglnproalasergluproalaalaglugluglualavalpro
190
proprovalvalalaproalavalserargthrthrseraspaspservalproprolys
210
lysargprovalvalproglyaspthrvalglugluvalargargasncysgluaspgln
230
ileglnalaleulysvalleuhisalaglugluleuhisargalaglnleuserhisasp
250
asngluvalargserilealavalvalthrseralailealaargglyaspleuthrlys
270
thrileaspalagluvalgluglyglumetalathrleulysgluthrileasnaspmet
290
valtyrlysleuargleuphesersergluvalthrargvalserleuaspvalglythr
310
metglyglnleuglyglyglnalavalvalthrglyvalgluglythrtrplysthrleu
330
thrgluthrvalasnthrmetalathrglnleuthrvalglnileargserilealaile
350
valthrseralailealaargglyaspleuserlysthrileaspalagluvalglngly
370
glumetalathrleulysgluthrvalasnsermetvalthrargleuargvalpheser
390
sergluvalthrargvalalaarggluvalglythraspglyglnleuglyglyglnala
410
valvalhisglyvalgluglythrtrplysgluleuthraspservalasnargmetala
430
glnasnleuthrleuglnvalarggluilealaservalthrlysalavalalaasngly
450
aspleuserlysthrvalaspileglyalaserglygluilearggluleulysthrthr
470
valasnsermetvalalaglnleuargargphealaalagluvalthrargvalalaleu
490
gluvalglythrgluglyglnleuglyglythralaasnvalgluglyvalglnglyglu
510
trplysserleuvalserservalasnthrmetalametasnleuthrglnglnvalarg
530
serilealaaspvalthrargalavalalagluglyaspleuserarglysileaspval
550
lysvallysglyglumetleuaspleulysleuthrvalasnsermetvalaspserleu
570
argleuphealaalagluvalthrargvalalalysgluvalglythraspglyglnleu
590
glyglyglnalatyrvalileasnvalserglyglutrplysserleuvalaspserval
610
asnglnmetcysglyasnleuthraspglnvalargserilealalysalathrthrala
630
valalaargglyaspleuthrglnlysvalmetileglualaasnglygluvalleugln
650
leuvalvalthrileasnglumetvalaspargleualathrphealasergluvalthr
670
argvalalahisgluvalglythrlysglyasnleuglyvalthralalysvalaspasn
690
ilegluglythrtrpglngluilethrasnasnvalasnthrmetalathrasnleuthr
710
serglnvalargalaphealaglnileseralaalaalathrgluglyasppheserser
730
phevalthrvalglualaserglyglumetaspserleulysthrlysileasnlysmet
750
valpheserleuargaspserleuglnlysasnthrmetalaargglualaalagluleu
770
alaasnargserlysserglupheleualaasnmetserhisgluileargthrpromet
790
asnglyileileglyleuthrglyvalthrleugluthraspleuthrargglnglnarg
810
gluasnleumetilevalserasnleualaasnserleuleuleuileileaspaspile
830
leuaspileserlysileglualaglyargmetthrvalgluglnilepropheserval
850
argseralavalpheglyileleulysthrleualavallysalathrglnserargleu
870
aspleumettyrservalgluseraspileproaspleuleuvalglyaspprophearg
890
leuargglnvalilethrasnleuvalglyasnalailelysphethrglnargglygln
910
valalaleusercysargleualaalaalaaspmetgluglulysthrtyrglnleuglu
930
phecysileseraspthrglyileglyilelysproasplysleuasnleuilepheasp
950
thrphealaglnalaaspglyserthrthrarglystyrglyglythrglyleuglyleu
970
thrileserlysargleuvalglnleumetglyglygluleutrpvalthrserglnphe
990
glyargglyserglnphetyrphethrileglncysargileglyglutrpasnleuasp
1010
glnvalargglnlysthrleuileprohisproglyargcysileleupheileaspthr
1030
leuhishisaspproservalileaspservalgluglnleuglyleugluilethrval
1050
valaspserleugluglualacysmetleuasphisserglnasnglytyrpheaspthr
1070
valleuvalaspglnleuserilevalgluargleuargaspvalgluhisleuargtyr
1090
ileproleuvalleuilethrproglnileproglnleuasnleulystyrcysleuasp
1110
pheglyilealaasncysvalgluserprothrasnalaglnaspmetcysasnalaleu
1130
leuproalaleuglualaserasnargileprosergluargglyglyaspalaserphe
1150
lysvalleuleualagluaspasnilevalasnglnlysvalalaleulyspheleuglu
1170
seralaglyhisglnsergluvalvalgluasnglyalaleualaleuglualavalarg
1190
lysasntyrtyraspilevalleumetaspleusermetproilemetglyglyglnglu
1210
alathrglnileileargglnphegluglnaspasnglyleugluargleuproileval
1230
alaleuthralahisalametleuglyaspargglulyscysileglnalaglymetasp
1250
asptyrleuthrlysproleuarglysproaspleuleualathrileasnlysileval
1270
leualaargargalaglyserserprothralaleuargphephe***
12901295
<210>3
<211>28
<212>dna
<213>人工序列
<400>3
cacaccgcggatccatgccgcagacgcc28
<210>4
<211>29
<212>dna
<213>人工序列
<400>4
ctgaccgcgatatctcactgcgggtcgtt29
- 还没有人留言评论。精彩留言会获得点赞!