萝卜细胞核雄性不育突变基因Rms1及其应用的制作方法
本发明属于植物基因工程技术领域。具体涉及一种萝卜雄性不育突变基因rms1,同时还涉及该基因的用途。
背景技术:
十字花科植物大多为异花受粉作物,存在着明显的杂种优势,但由于它们的花器较小,人工去雄操作费时费力,且一次授粉获得的杂交种子很少,为解决这一问题,育种工作者选用自交不亲和系育种,但自交不亲和系易受环境等因素的影响,亲和性不稳定,经常出现制种纯度不高的现象。利用植物雄性不育系进行杂交制种,较好地解决了自交不亲和系制种的问题,并可提高杂交种子的纯度,增加农作物的产量,己成为国际种业的主要发展方向。萝卜雄性不育系育种是开展研究较早,并且进行商业化应用较为成熟的一项技术。
目前在生产上使用的萝卜雄性不育系,通常是由细胞质基因和细胞核基因共同互作形成的一种雄性不育现象,萝卜的不育细胞质一般有ogura-cms(orf138)、kos-cms(orf125)、dcgms-cms和nwb-cms等类型,但最常见的还是ogura不育类型;由线粒体基因orf138和细胞核中的两对隐性基因控制,不育系基因型为sms1ms1ms2ms2,保持系基因型为nms1ms1ms2ms2,(s为不育型细胞质,n为正常细胞质)。萝卜雄性不育表现为花药退化,或不能形成具有正常功能的花粉,而雌蕊发育正常。保持系则具有正常的雄性和雌性器官,自交能正常结实,当与不育系杂交时,能够保持不育系的不育性。萝卜细胞质雄性不育基因存在于线粒体dna上,主要是由于线粒体基因组分子内或分子间频繁重组所形成的异常嵌合基因造成的,目前研究认为,ogura不育类型雄性不育的主控基因为orf138,orf138与线粒体基因组中具有正常功能的基因共转录,产生异常转录本和翻译产物,从而在花药发育的某个时期干扰线粒体的正常功能发挥,最终导致雄性不育现象的发生。当用恢复系与不育系进行杂交时,恢复系中的恢复基因能够产生一种ppr蛋白,该蛋白抑制orf138的表达,从而达到育性恢复的目的。
到目前为止所知的萝卜雄性不育类型中,ogura雄性不育是导入率和稳定性方面最优秀的不育材料,同时ogura雄性不育也是目前研究最为深入的一种雄性不育材料,日本学者yamagishi通过对野生种、栽培种等不育材料进行测序,将ogura雄性不育基因orf138分为九个类型。在日本发现ogura后,我国从上世纪70年代初先后发现萝卜雄性不育株,开始了萝卜雄性不育系的育种工作,到80年代中期已先后育出了多个品种在生产上推广,如山东的鲁萝卜三号、辽宁的红丰一号、山西的国光、丰光、河南的郑研等品种。随着生物技术的不断发展,科技工作者将萝卜ogura雄性不育基因先后引入到油菜、甘蓝、大白菜、花椰菜等多种十字花科作物上。我国目前发现和使用的萝卜雄性不育系同样为ogura细胞质雄性不育,目前全世界范围内使用的萝卜雄性不育系几乎全部为ogura细胞质类型,随着雄性不育材料使用的越来越普及,萝卜及其它十字花科作物的细胞质类型将严重单一化。
在十字花科作物中,目前已在大白菜、甘蓝、油菜、小白菜等作物的自然群体中发现了显性或隐性核雄性不育材料。如南京农业大学上世纪80年代在小白菜中发现细胞核雄性不育材料,并利用“两用系”进行杂交制种;福建省农科院在当地菜心品种“七叶心”中发现核雄性不育材料,并获得了雄性不育两用系;沈阳农业大学在小青口、二青帮大白菜品种中获得了隐性核基因雄性不育材料;沈阳市农科所在“万泉青帮”地方大白菜品种中发现了显性核基因雄性不育材料,并提出了“显性核基因雄性不育与显性上位基因互作”雄性不育遗传模式,最终获得了具有100%不育率的大白菜雄性不育系;中国农科院在甘蓝的自然群体中发现了显性核基因控制的雄性不育材料。在萝卜的研究上,在自然群体中发现由核基因控制的雄性不育材料的极少。
技术实现要素:
本发明克服了现有技术的不足,从萝卜核雄性不育材料中分离克隆到一个萝卜雄性不育基因突变体基因rms1,该基因通过抑制萝卜花粉壁的发育,使其不能形成具有正常功能的花粉导致萝卜雄性不育。
本发明一方面,提供了一种萝卜细胞核雄性不育突变基因rms1,其cdna序列如seqidno.1所示,所编码的蛋白质序列如seqidno.2所示。
本发明萝卜细胞核雄性不育基因rms1,通过下述方式获得:本发明利用核雄性不育系和保持系材料,从不育系136a(该材料记载在,萝卜隐性核雄性不育系136a的选育及其育性研究,《山东农业科学》,2016年07期)中克隆到了自然突变不育基因rms1,并在保持系中克隆到了野生型不育基因rms1,利用geteway系统将rms1和rms1基因进行bp反应连接到pdonr221,然后通过转化的方法在大肠杆菌dh5a中得到大量克隆,接着进行lr反应,把两个基因连接到表达载体pbi121上,并在大肠杆菌dh5a中表达;筛选转基因大肠杆菌,并提取转化质粒,最后将转化质粒转入农杆菌株gv3101中。将含有rms1基因的转化农杆菌菌株转入到萝卜雄性不育系136a中,从而使不育系136a育性恢复,验证rms1突变导致了萝卜败育,为其在杂交育种中的应用提供理论支持。
该萝卜雄性不育基因rms1可通过干扰花粉细胞壁的发育,导致花粉败育产生萝卜雄性不育,该基因产生的萝卜雄性不育系败育稳定,不受环境等因素影响,同时不影响其它器官的发育。可作为萝卜育种中的雄性不育系使用。
本发明所述的基因可用于萝卜及其它十字花科作物的育种,为萝卜杂交育种提供了一个新的不育源,对萝卜及其它十字花科作物的杂交育种都具有十分重要的意义。
因此,本发明另一方面,提供萝卜雄性不育基因rms1在十字花科作物杂交育种中的应用。
优选地,所述十字花科作物是萝卜。
优选地,所述萝卜雄性不育基因rms1在萝卜三系育种中的应用。
本发明再一方面,还提供一种萝卜雄性不育株恢复育性的方法,该方法是将rms1基因转入雄性不育株中。所述雄性不育株中含有纯合的rms1基因。
具体方法是,建立萝卜带柄子叶离体再生体系,利用农杆菌介导rms1基因转化,将rms1基因转入萝卜雄性不育株中。提取转基因植株dna,对转基因植株进行检测,将检测出的雄性不育系转基因阳性植株移于大田,收获后来年春种植,观察其育性情况。经种植观察雄性不育阳性株全部恢复育性。说明雄性不育株中转入rms1基因后,可恢复育性。由于rms1基因的突变可以导致萝卜出现雄性不育现象,并且萝卜rms1与rms1是等位基因,rms1相对于rms1来说是隐性基因,当含有纯合的rms1基因的不育系转入rms1基因后,其育性恢复。
本发明将分离克隆到的rms1基因转育到萝卜自交系中,就可产生该自交系的不育系,可用于萝卜的三系制种。该基因除对花粉细胞壁的发育影响外,对其它器官影响不明显。
本发明与其他技术相比,具有以下特点和效果:
1、本发明在国内外未见报道,系首次克隆得到该rms1基因,并对其功能进行研究。
2、本发明发现的萝卜雄性不育基因除抑制花粉细胞壁的发育外,对其它器官影响不明显。
3、本发明发现的萝卜雄性不育基因解决萝卜细胞质雄性不育容易造成细胞质单一的问题,并且保持系选育简便。
4、本发明发现的rms1基因突变体rms1是来自于自然变异,用该基因产生的雄性不育系不存在转基因的问题。
5、本发明所产生的雄性不育材料能够直接应用于萝卜的三系育种。
附图说明
图1为rms1与rms1基因的cdna序列差异比对情况。
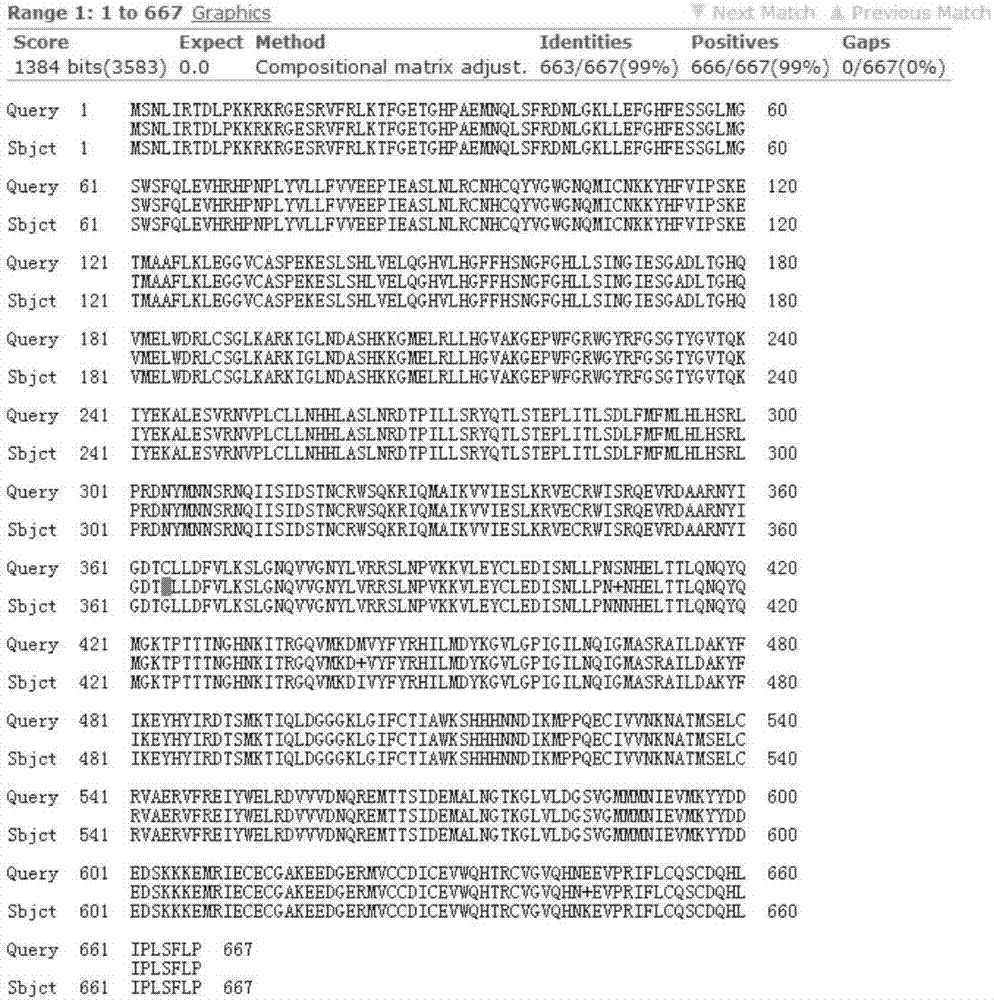
图2为rms1与rms1基因的氨基酸序列差异比对情况;query为rms1;sbjct为rms1。
图3为转化后萝卜带柄子叶生成的愈伤组织。
图4为由萝卜愈伤组织分化出的丛生芽移植到生根培养基上。
图5为转化苗在生根培养基上分化出根系,处在移栽前的转化苗。
图6为转基因阳性植株检测结果,有条带的为阳性转化植株。
图7为纯合雄性不育系的pcr检测结果,具有两条条带的为保持系,是杂合体;具有单条带的为纯合体,是不育系。
图8为通过对阳性转化苗和纯合不育系的检测后,明确为不育系后代的转化苗栽于大田,转rms1基因雄性不育株的育性已经恢复。
具体实施方式
本发明通过以下详细的实施例给出。根据以下的描述和这些实施例,本领域技术人员可以确定本发明的基本特征,并且在不偏离本发明精神和范围的情况下,容许对本发明做出各种改变和修改,以使其适用各种用途和条件。
本发明实施例中的引物均由华大基因合成,序列测序由华大基因完成,dna抽提,rna抽提,pcr及试剂配方参照j.萨姆布鲁克等,分子克隆实验指南,第三版,金冬雁等(译),科学出版社,2002。
实施例1:萝卜rms1基因的克隆
1.1萝卜雄性不育系花蕾总rna的提取
(1)提取萝卜雄性不育系136a花蕾材料放入研钵中,利用液氮将其研磨成粉末(可直接应用于下面的实验或者放入-80℃超低温冰箱保存备用);
(2)等液氮挥发后,立即将100-200mg样品粉末转入到1.5ml离心管中,然后迅速加入1mltrizol提取液,涡旋震荡使样品充分溶入提取液中,室温放置5min;
(3)4℃,12000rpm离心10min,将0.9ml上清液转移到新的1.5ml离心管中,再加入0.2ml氯仿剧烈振荡15sec,混匀后室温放置2-5min;
(4)4℃,12000rpm离心10min,将0.4ml上清液转移到新的1.5ml离心管中,再加入0.4ml异丙醇,上下翻转15次混匀溶液,室温放置15min;
(5)4℃,12000rpm离心10min,弃上清,用1ml75%的乙醇洗涤沉淀两次,4℃,8000rpm离心5min;
(6)弃上清,在超净工作台中开盖,干燥rna约2-5min,加入40μlrnase-free水,在60℃中充分溶解rna10min;
(7)用紫外分光光度计测rna样品的od值和浓度,a260/a280达到1.7-2.0为最佳;琼脂糖凝胶电泳检测其质量。
1.2rna的反转录
(1)向离心管中依次加入下列物质(40μl反应体系):
(2)轻轻混匀后,65℃变性5min,立即插到冰上,冰浴到1min以上;
(3)向离心管中依次加入下列物质
5χbuffer8μl
20mmdtt4μl
mmlv1μl
(4)轻轻混匀后,42℃恒温水浴1h,65℃变性10min,-20℃保存备用。
1.3rms1基因克隆
f5’-atgtcgaatctgattcgaacag-3’(seqidno.5)
r5’-ttaaggcaaaaaagagagagggat-3’(seqidno.6)
(1)高保真酶primerstar进行gateway系统的第一步扩增反应体系如下(50μl体系):
扩增条件如下:
反应结束后,反应液于0.8%tae琼脂糖凝胶电泳检测。
(2)克隆基因片段的纯化回收(天根试剂盒)
1)将切下带有目的片段的凝胶放入1.5ml离心管并称凝胶重量,加入3倍体积的溶胶液,60℃溶胶10min,溶胶期间不断翻转;
2)凝胶完全融化后,全部吸取到回收柱中,放置片刻;
3)室温,12000rpm离心30s,弃溶液;
4)在柱中加入700μl的漂洗液,12000rpm离心1min,弃漂洗液;
5)在柱中加入500μl的漂洗液,12000rpm离心1min,弃漂洗液;
6)空柱,12000rpm离心2min;
7)回收柱开盖晾1-2min,放入新的干净的1.5ml离心管,加入60℃预热的40μl灭菌水或eb缓冲液,放置2min;
8)12000rpm离心1min,所得溶液即为回收片段。
(3)高保真酶primerstar进行gateway系统的第二步扩增。
attb引物序列
attb:f:5’-ggggacaagtttgtacaaaaagcaggct-3’(seqidno.7)
attb-r:5’-ggggaccactttgtacaagaaagctgggt-3’(seqidno.8)
反应体系如下(100μl体系):
扩增条件如下:
反应结束后,反应液于0.8%tae琼脂糖凝胶电泳检测。
1.4克隆基因片段的bp反应
bp反应(gateway系统)体系如下:
bpclonase1μl
dna回收片段(bp)3μl
入门载体pdonr2211μl
25℃反应8小时、过夜。
1.5大肠杆菌感受态的制备(无菌操作)
(1)取dh5a菌种接种于20mllb液体培养基中,37℃摇床培养过夜;
(2)按1:100接种于50mllb液体培养基中,37℃,200rpm培养1小时,od600值为0.4-0.6;
(3)将菌液放置在冰上30min;
(4)4℃下4200rpm离心10min,弃上清,加10ml预冷的0.1m的cacl2悬浮菌体;
(5)将菌液放置在冰上10min;
(6)4℃下4200rpm离心10min,弃上清,加2ml预冷的0.1m的cacl2悬浮菌体,分装于1.5ml离心管中,现用或加入终体积30%甘油经液氮速冻后-80℃保存备用。
1.6大肠杆菌的质粒转化(无菌操作)
(1)将1-5μl质粒dna或者连接产物加入50μl的感受态细胞中,轻弹离心管混匀,冰浴30min;
(2)42℃温水浴热激90s,立即冰浴2-3min;
(3)加入1mllb培养基,37℃培养40-50min;
(4)室温下4000rpm离心3min,收集菌体;
(5)将菌涂布于含有相应抗生素的培养平板上,37℃倒置培养过夜。
1.7大肠杆菌pcr验证
反应体系如下(20μl体系)
扩增条件如下:
反应结束后,反应液于0.8%tae琼脂糖凝胶电泳检测。
我们用同样的方法还获得了rms1基因的dna克隆和rms1基因的cdna克隆。
1.8dna测序
挑取3株含有重组质粒的阳性单菌落用含有kan(25mg/l)的液体lb摇过夜,然后送华大基因公司进行测序,得到3组结果一致的rms1基因cdna序列如表seqidno.1所示,其编码蛋白序列如seqidno.2所示。
同样测得rms1基因的cdna序列和编码蛋白序列如seqidno.3和seqidno.4所示。
经过序列分析,rsm1和rms1基因的cdna存在10碱基的差异,在氨基酸上有一个变异,和氨基酸的同性变异。如图1,图2所示。
1.9大肠杆菌质粒dna的提取
(1)挑取单克隆接种于10ml含有相应抗生素的lb液体培养基中,37℃培养8小时并过夜;
(2)室温12000rpm离心1min,收集菌体;
(3)弃上清,加入100μl低温预冷的溶液ⅰ,振荡悬浮菌体;
(4)加200μl新鲜配制的溶液ⅱ,快速翻转混匀,冰浴5min;
(5)溶液澄清后加入150μl低温预冷的溶液ⅲ,轻轻混匀后冰浴5min;
(6)4℃下12000rpm离心10min,吸取上清至新的1.5ml离心管中;
(7)加入等体积的苯酚/氯仿/异戊醇(24/25/1),振荡混匀;
(8)室温12000rpm离心10min,转移上层水相于另一新的1.5ml离心管中,加入等体积的氯仿/异戊醇(24/1),再抽提一次,振荡混匀;
(9)室温12000rpm离心10min,转移上层水相于另一新的1.5ml离心管中,加入等体积的异戊醇,混匀并于-20℃放置30min;
(10)室温12000rpm离心10min,弃上清保留沉淀;
(11)用75%乙醇洗涤沉淀两次,真空干燥5min,溶于40μl灭菌水,放至-20℃保存备用。
1.10克隆基因片段的lr反应
lr反应(gateway系统)体系如下:
lrclonase1μl
大肠杆菌质粒dna3μl
表达载体pbi121s1μl
25℃反应8小时,过夜。
1.11大肠杆菌感受态的制备和质粒转化(同1.5、1.6)
1.12大肠杆菌pcr验证(同1.7)
1.13大肠杆菌质粒dna的提取(同1.9)
验证转入pbi121s载体的阳性pbi121s::rms1质粒的克隆区域核苷酸序列为目标片段。
1.14农杆菌感受态的制备(无菌操作)
(1)取农杆菌gv3101菌种接种于10mlyep液体培养基中,28℃摇床培养过夜;
(2)按1:50接种于50mlyep液体培养基中,28℃振荡培养3-4小时,到od600值为0.4-0.6;
(3)4℃下4200rpm离心10min,收集菌体;
(4)弃上清,加10ml预冷的0.15m的nacl悬浮菌体;
(5)重复步骤3;
(6)弃上清,加入2ml预冷的20mm的cacl2悬浮菌体,分装于1.5ml离心管路,现用或加入终体积7%dmso经液氮速冻后-80℃保存备用。
1.15农杆菌的质粒转化(无菌操作)
(1)将1μl质粒dna加入50μl的感受态细胞中,轻弹离心管混匀,冰浴30min;
(2)液氮速冻1min;然后37℃水浴5min,立即冰浴2-3min;
(3)加入1mlyep培养基,28℃培养2-4小时;
(4)室温4000rpm,离心3min,收集菌体;
(5)将菌涂布于含有相应抗生素的yep培养平板上,28℃倒置培养48小时。
1.16转化农杆菌的pcr验证
反应体系如下(20μl体系)
扩增条件如下:
反应结束后,反应液于0.8%tae琼脂糖凝胶电泳检测。
实施例2农杆菌介导的萝卜rms1基因转化
2.1萝卜带柄子叶离体再生体系的建立及外源基因转化
(1)种子消毒及预培养
将萝卜种子用70%的乙醇消毒5min,去除乙醇后用0.1%的hgcl2消毒10min,用无菌水冲洗4-5遍,在25℃下无菌水浸种6小时。
将萌动的萝卜种子放在ms培养基上,培养温度25℃,光照16小时,黑暗8小时。
(2)萝卜出苗5天时,取萝卜带柄子叶放入预培养基上(ms+6-ba6mg/l+naa0.05mg/l),暗培养2天。
(3)农杆菌浸染
将4℃保存的带有表达载体的农杆菌在添加了50mg/l利福平的yep固体培养基上划线,28℃黑暗培养3天后挑取单菌落,接入含有50mg/l利福平的yep液体培养基,黑暗下28℃,200r/min振荡培养24小时后再次转接,相同条件下培养16小时。将菌液置于灭菌的离心管中,5000rpm离心5min收集菌体,将浸染液悬菌体调整到od600为0.4~0.6,加入30mg/l乙酰丁香酮备用。
将预培养的子叶放入调整好的农杆菌悬浮液中,浸5min,取出,用灭菌滤纸吸干多余的菌液,放入共培养培养基上。
(4)共培养
共培养培养基为ms+6-ba6mg/l+naa0.05mg/l,在黑暗条件下,30℃共培养4天。
(5)筛选及芽诱导
将共培养后的子叶用无菌水清洗3次,再用无菌滤纸吸干,转入到筛选及芽诱导培养基(ms+6-ba6mg/·l+naa0.05mg/l+cef500mg/l+kan25mg/l)上,在25℃下,光照16小时进行培养,每两周更换一次培养基,直到分化出丛生芽(图3、4)。
(6)生根培养
筛选后长至2cm以上的丛生芽单独剪下,转移到生根筛选培养基上,在25℃下,光照16小时进行培养,连续培养2周(图5)。生根培养基为ms+6-ba2mg/·l+naa0.08mg/l+cef500mg/l+kan25mg/l。
(7)炼苗及移栽
根系生长良好的小苗转移到炼苗培养基上,在25℃下,光照16小时进行培养,培养1周左右,当将小苗取出,除去残存培养基移入土中,用薄膜覆盖,以提高湿度。炼苗培养基为ms+naa0.08mg/l+蔗糖7g/l。
2.2提取转基因植株dna
称取0.5g的t1代再生植株叶片,剪碎后用液氮研磨,迅速将粉末转移至1.5ml离心管中,然后加入700μl预热至65℃的2xctab提取缓冲液及0.1%的疏基乙醇,轻轻颠倒离心管混匀,然后置于65℃水浴保温2小时,不时颠倒摇动。混合物冷却至室温后加入等体积的酚:氯仿:异戊醇(25:24:1),4℃下10000g离心10min。将水相转移至一干净离心管中,加入两倍体积的无水乙醇,摇匀,-20℃放置30min沉淀dna。4℃下10000g离心10min,弃去液体,并用70%乙醇洗涤两次,室温干燥dna后,加100μl含无dna酶的rna酶的te液溶解,37℃水浴30min。加入200μl无水乙醇,温和混匀,-20℃放置30min沉淀dna。4℃下10000g离心10min。弃去液体,并用70%乙醇洗涤两次,室温干燥dna后,加40μl无菌水溶解dna,4℃保存选用。
2.3pcr检测阳性转基因植株
抗性植株总dna用35s启动子的正向引物和rms1基因引物的反向引物进行pcr检测。
35s启动子引物序列:
35s-f:5’-gcagaggcatcttcaacg-3’(seqidno.9)
35s-r:5’-cacgatctacccgagcaa-3’(seqidno.10)
rms1基因引物序列:
rms1-f:5’-atgtcgaatctgattcgaacag-3’(seqidno.5)
rms1-r:5’-ttaaggcaaaaaagagagaggga-3’(seqidno.6)
35s启动子f引物与rms1基因r引物序列:
35s-f:5’-gcagaggcatcttcaacg-3’(seqidno.9)
rms1-r:5’-ttaaggcaaaaaagagagaggga-3’(seqidno.6)
反应体系如下(20μl)
扩增条件如下:
反应结束后,反应液于0.8%tae琼脂糖凝胶电泳检测,见图6。有条带的为阳性转基因植株。
2.4植株基因型检测
利用我们在基因定位过程中找到的与rms1基因连锁的特异片段引物:
f:5’-ccacaagaccacaagtctgc-3’(seqidno.11)
r:5’-atgcggaactccgaactatg-3’(seqidno.12)
实验操作同2.3。
实验结果如图7。图中有两条条带的为保持系(原来即是可育株),有一条条带的为不育系。
2.5育性结果分析
将2.3和2.4两步骤检测出的雄性不育系阳性植株移于大田,待萝卜根长至直径3cm时收获,放在4℃将冰箱中保存。来年春种植于网棚中,观察其育性情况。经种植观察所选出的雄性不育阳性株全部恢复育性。说明由于rms1基因的突变可以导致萝卜出现雄性不育现象,并且萝卜rms1与rms1是等位基因,rms1相对于rms1来说是隐性基因,当含有纯合的rms1基因的不育系转入rms1基因后,其育性恢复。如图8。
sequencelisting
<110>山东省农作物种质资源中心
<120>萝卜细胞核雄性不育突变基因rms1及其应用
<130>
<160>12
<170>patentinversion3.3
<210>1
<211>2004
<212>dna
<213>raphanussativusl.
<400>1
atgtcgaatctgattcgaacagatcttcccaagaagaggaagagaggagaaagtagggtt60
tttaggctgaagactttcggagagacaggacatccagctgagatgaaccagttgtctttt120
agagacaaccttgggaagcttcttgagtttggtcacttcgagagctccggtctcatggga180
agttggtcctttcagctcgaggttcatcgtcacccaaatcctctctatgttcttctcttt240
gtcgtcgaagagcccattgaagcctctcttaacctacgttgcaaccattgccaatatgtt300
ggttggggaaaccaaatgatatgcaacaagaagtaccattttgtgatcccctcaaaggaa360
acaatggctgcctttctaaaactagaaggcggagtctgcgcttctccggaaaaagaaagt420
ctatctcatctcgtggagcttcagggccatgtccttcacggctttttccactccaacgga480
tttggccacttgctctctatcaatggcatcgagtccggtgccgacctgaccggtcatcaa540
gtcatggagttgtgggatcgcctctgctctggtttaaaggccaggaaaataggcttgaac600
gatgcatcgcacaaaaaagggatggaactaaggctgctgcatggagtggcaaaaggagag660
ccgtggttcggtcgttggggttaccggttcgggtcagggacatacggagtaactcaaaag720
atctacgagaaggcacttgagtcggtccgcaacgtacccttgtgtttgcttaaccatcac780
ctagccagccttaaccgtgacactccaatcctcttgtcaaggtaccaaacgttatccacc840
gagccattaatcactctcagtgatctcttcatgttcatgcttcatctccattcgcgtctt900
ccaagagataactatatgaacaactcccgaaaccaaatcatctccattgatagtaccaac960
tgtagatggtctcagaaacggattcaaatggctatcaaagtggttatagagtcgttgaaa1020
agggtcgaatgccggtggatatcgagacaagaagtgagggacgcagctagaaactacatc1080
ggggacacatgtttgcttgacttcgtgttgaagtcgctcggtaaccaagtggtgggaaac1140
tatttggttcgacgtagtttaaatccggtgaagaaagtgctagagtattgcttggaggac1200
atatcaaatttattaccaaatagtaaccatgaactcacaaccctccaaaatcaatatcaa1260
atggggaagacgcctacgacaacgaacggacataacaagatcacaagaggtcaagtgatg1320
aaagacatggtttacttttacagacacattcttatggactacaagggagtgttaggcccc1380
attggcatactgaaccaaatcggaatggcttcaagggcaatcctcgacgctaaatacttc1440
atcaaagagtatcactacatcagagatacgtcgatgaaaacaattcagttagatggaggt1500
ggcaaattaggaatattctgcacaatcgcgtggaaatctcatcatcataacaatgacata1560
aagatgcctccacaagaatgcatagtggtaaacaaaaatgcaacaatgagtgaactatgt1620
agagtggcagagagagtgtttagagagatctactgggaactaagagacgtcgtggtggat1680
aatcaaagggagatgactacaagtatcgatgaaatggcattgaatgggactaaaggattg1740
gtgttagatgggagtgtaggaatgatgatgaacattgaagtgatgaagtattatgatgat1800
gaagatagtaagaagaaggagatgaggatcgagtgtgagtgtggagcaaaggaggaagat1860
ggagagaggatggtgtgttgtgatatttgtgaagtatggcaacacacaaggtgtgttggt1920
gttcaacacaatgaggaagttcctcgcatttttctttgtcaaagctgtgaccaacatctt1980
atccctctctcttttttgccttaa2004
<210>2
<211>667
<212>prt
<213>raphanussativusl.
<400>2
metserasnleuileargthraspleuprolyslysarglysarggly
151015
gluserargvalpheargleulysthrpheglygluthrglyhispro
202530
alaglumetasnglnleuserpheargaspasnleuglylysleuleu
354045
glupheglyhisphegluserserglyleumetglysertrpserphe
505560
glnleugluvalhisarghisproasnproleutyrvalleuleuphe
65707580
valvalglugluproileglualaserleuasnleuargcysasnhis
859095
cysglntyrvalglytrpglyasnglnmetilecysasnlyslystyr
100105110
hisphevalileproserlysgluthrmetalaalapheleulysleu
115120125
gluglyglyvalcysalaserproglulysgluserleuserhisleu
130135140
valgluleuglnglyhisvalleuhisglyphephehisserasngly
145150155160
pheglyhisleuleuserileasnglyilegluserglyalaaspleu
165170175
thrglyhisglnvalmetgluleutrpaspargleucysserglyleu
180185190
lysalaarglysileglyleuasnaspalaserhislyslysglymet
195200205
gluleuargleuleuhisglyvalalalysglygluprotrpphegly
210215220
argtrpglytyrargpheglyserglythrtyrglyvalthrglnlys
225230235240
iletyrglulysalaleugluservalargasnvalproleucysleu
245250255
leuasnhishisleualaserleuasnargaspthrproileleuleu
260265270
serargtyrglnthrleuserthrgluproleuilethrleuserasp
275280285
leuphemetphemetleuhisleuhisserargleuproargaspasn
290295300
tyrmetasnasnserargasnglnileileserileaspserthrasn
305310315320
cysargtrpserglnlysargileglnmetalailelysvalvalile
325330335
gluserleulysargvalglucysargtrpileserargglngluval
340345350
argaspalaalaargasntyrileglyaspthrcysleuleuaspphe
355360365
valleulysserleuglyasnglnvalvalglyasntyrleuvalarg
370375380
argserleuasnprovallyslysvalleuglutyrcysleugluasp
385390395400
ileserasnleuleuproasnserasnhisgluleuthrthrleugln
405410415
asnglntyrglnmetglylysthrprothrthrthrasnglyhisasn
420425430
lysilethrargglyglnvalmetlysaspmetvaltyrphetyrarg
435440445
hisileleumetasptyrlysglyvalleuglyproileglyileleu
450455460
asnglnileglymetalaserargalaileleuaspalalystyrphe
465470475480
ilelysglutyrhistyrileargaspthrsermetlysthrilegln
485490495
leuaspglyglyglylysleuglyilephecysthrilealatrplys
500505510
serhishishisasnasnaspilelysmetproproglnglucysile
515520525
valvalasnlysasnalathrmetsergluleucysargvalalaglu
530535540
argvalphearggluiletyrtrpgluleuargaspvalvalvalasp
545550555560
asnglnargglumetthrthrserileaspglumetalaleuasngly
565570575
thrlysglyleuvalleuaspglyservalglymetmetmetasnile
580585590
gluvalmetlystyrtyraspaspgluaspserlyslyslysglumet
595600605
argileglucysglucysglyalalysglugluaspglygluargmet
610615620
valcyscysaspilecysgluvaltrpglnhisthrargcysvalgly
625630635640
valglnhisasnglugluvalproargilepheleucysglnsercys
645650655
aspglnhisleuileproleuserpheleupro
660665
<210>3
<211>2004
<212>dna
<213>raphanussativusl.
<400>3
atgtcgaatctgattcgaacagatcttcccaagaagaggaagagaggagaaagtagggtt60
tttaggctgaagactttcggagagacaggacatccagctgagatgaaccagttgtctttt120
agagacaaccttgggaagcttcttgagtttggtcacttcgagagctccggtctcatggga180
agttggtcctttcagctcgaggttcatcgtcacccaaatcctctctatgttcttctcttt240
gtcgtcgaagagcccattgaagcctctcttaacctacgttgcaaccattgccaatatgtt300
ggttggggaaaccaaatgatatgcaacaagaagtaccattttgtgatcccctcaaaggaa360
acaatggctgcctttctaaaactagaaggcggagtctgcgcttctccggaaaaagaaagt420
ctatctcatctcgtggagcttcagggccatgtccttcacggctttttccactccaacgga480
tttggccacttgctctctatcaatggcatcgagtccggtgccgacctgaccggtcatcaa540
gtcatggagttgtgggatcgcctctgctctggtttaaaggccaggaaaataggcttgaat600
gatgcatcgcacaagaaagggatggaactaaggctgctgcatggagtggcaaaaggagag660
ccgtggttcggtcgttggggttaccggttcgggtcagggacatacggagtaactcaaaag720
atctacgagaaggcacttgagtcggtccgcaacgtacccttgtgtttgcttaaccatcac780
ctagccagccttaaccgtgacactccaatcctcttgtcaaggtaccaaactttatccacc840
gagccattaatcactctcagtgatctcttcatgttcatgcttcatctccattcgcgtctt900
ccaagagataactatatgaacaactcccgaaaccaaatcatctccattgatagtaccaac960
tgtagatggtctcagaaacggattcaaatggctatcaaagtggttatagagtcgttgaaa1020
agggtcgaatgccggtggatatcgagacaagaagtgagggacgcagctagaaactacatc1080
ggggacacaggtttgcttgacttcgtgttgaagtcgctcggtaaccaagtggtgggaaac1140
tatttggttcgacgtagtttaaatccggtgaagaaagtgctagagtattgcttggaggac1200
atatcaaatttattaccaaataataaccatgaactcacaaccctccaaaatcaatatcaa1260
atggggaagacgcctacgacaacgaacggacataacaagatcacaagaggtcaagtgatg1320
aaagacatagtttacttttacagacacattcttatggactacaagggagtgttaggcccc1380
ataggcatactgaaccaaatcggaatggcttcaagggcaatcctcgacgctaaatacttc1440
atcaaagagtatcactacatcagagatacgtcgatgaaaacaattcagttagatggaggt1500
ggcaaattaggaatattctgcacaatcgcgtggaaatctcatcatcataacaatgacata1560
aagatgcctccacaagaatgcatagtggtaaacaaaaatgcaacaatgagtgaactatgt1620
agagtggcagagagagtgtttagagagatctactgggaactaagagacgtcgtggtggat1680
aatcaaagggagatgactacaagtatcgatgaaatggcattgaatgggactaaaggattg1740
gtgttagatgggagtgtaggaatgatgatgaacattgaagtgatgaagtattatgatgat1800
gaagatagtaagaagaaggagatgaggatcgagtgtgagtgtggagcaaaggaggaagat1860
ggagagaggatggtttgttgtgatatttgcgaagtatggcaacacacaaggtgtgttggt1920
gttcaacataatgaggaagttcctcgcatttttctttgtcaaagctgtgaccaacatctt1980
atccctctctcttttttgccttaa2004
<210>4
<211>667
<212>prt
<213>raphanussativusl.
<400>4
metserasnleuileargthraspleuprolyslysarglysarggly
151015
gluserargvalpheargleulysthrpheglygluthrglyhispro
202530
alaglumetasnglnleuserpheargaspasnleuglylysleuleu
354045
glupheglyhisphegluserserglyleumetglysertrpserphe
505560
glnleugluvalhisarghisproasnproleutyrvalleuleuphe
65707580
valvalglugluproileglualaserleuasnleuargcysasnhis
859095
cysglntyrvalglytrpglyasnglnmetilecysasnlyslystyr
100105110
hisphevalileproserlysgluthrmetalaalapheleulysleu
115120125
gluglyglyvalcysalaserproglulysgluserleuserhisleu
130135140
valgluleuglnglyhisvalleuhisglyphephehisserasngly
145150155160
pheglyhisleuleuserileasnglyilegluserglyalaaspleu
165170175
thrglyhisglnvalmetgluleutrpaspargleucysserglyleu
180185190
lysalaarglysileglyleuasnaspalaserhislyslysglymet
195200205
gluleuargleuleuhisglyvalalalysglygluprotrpphegly
210215220
argtrpglytyrargpheglyserglythrtyrglyvalthrglnlys
225230235240
iletyrglulysalaleugluservalargasnvalproleucysleu
245250255
leuasnhishisleualaserleuasnargaspthrproileleuleu
260265270
serargtyrglnthrleuserthrgluproleuilethrleuserasp
275280285
leuphemetphemetleuhisleuhisserargleuproargaspasn
290295300
tyrmetasnasnserargasnglnileileserileaspserthrasn
305310315320
cysargtrpserglnlysargileglnmetalailelysvalvalile
325330335
gluserleulysargvalglucysargtrpileserargglngluval
340345350
argaspalaalaargasntyrileglyaspthrglyleuleuaspphe
355360365
valleulysserleuglyasnglnvalvalglyasntyrleuvalarg
370375380
argserleuasnprovallyslysvalleuglutyrcysleugluasp
385390395400
ileserasnleuleuproasnasnasnhisgluleuthrthrleugln
405410415
asnglntyrglnmetglylysthrprothrthrthrasnglyhisasn
420425430
lysilethrargglyglnvalmetlysaspilevaltyrphetyrarg
435440445
hisileleumetasptyrlysglyvalleuglyproileglyileleu
450455460
asnglnileglymetalaserargalaileleuaspalalystyrphe
465470475480
ilelysglutyrhistyrileargaspthrsermetlysthrilegln
485490495
leuaspglyglyglylysleuglyilephecysthrilealatrplys
500505510
serhishishisasnasnaspilelysmetproproglnglucysile
515520525
valvalasnlysasnalathrmetsergluleucysargvalalaglu
530535540
argvalphearggluiletyrtrpgluleuargaspvalvalvalasp
545550555560
asnglnargglumetthrthrserileaspglumetalaleuasngly
565570575
thrlysglyleuvalleuaspglyservalglymetmetmetasnile
580585590
gluvalmetlystyrtyraspaspgluaspserlyslyslysglumet
595600605
argileglucysglucysglyalalysglugluaspglygluargmet
610615620
valcyscysaspilecysgluvaltrpglnhisthrargcysvalgly
625630635640
valglnhisasnlysgluvalproargilepheleucysglnsercys
645650655
aspglnhisleuileproleuserpheleupro
660665
<210>5
<211>22
<212>dna
<213>人工序列
<400>5
atgtcgaatctgattcgaacag22
<210>6
<211>24
<212>dna
<213>人工序列
<400>6
ttaaggcaaaaaagagagagggat24
<210>7
<211>28
<212>dna
<213>人工序列
<400>7
ggggacaagtttgtacaaaaagcaggct28
<210>8
<211>29
<212>dna
<213>人工序列
<400>8
ggggaccactttgtacaagaaagctgggt29
<210>9
<211>18
<212>dna
<213>人工序列
<400>9
gcagaggcatcttcaacg18
<210>10
<211>18
<212>dna
<213>人工序列
<400>10
cacgatctacccgagcaa18
<210>11
<211>20
<212>dna
<213>人工序列
<400>11
ccacaagaccacaagtctgc20
<210>12
<211>20
<212>dna
<213>人工序列
<400>12
atgcggaactccgaactatg20
- 还没有人留言评论。精彩留言会获得点赞!