一种检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因及其应用的制作方法
本发明属于分子生物学
技术领域:
,具体涉及一种检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因及其应用。
背景技术:
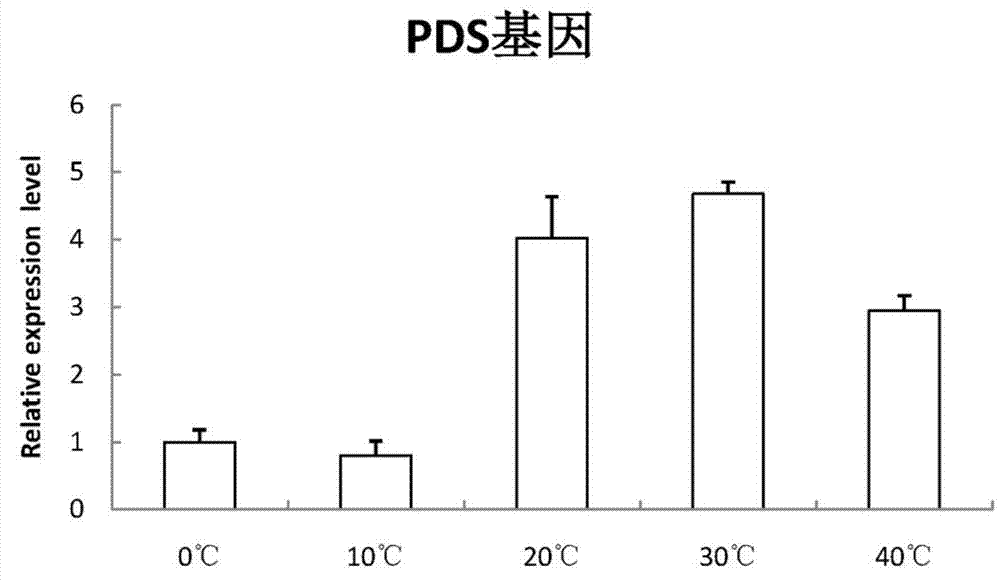
:随着现代人类工业的发展的影响,全球气候正在逐渐变暖。昆虫是变温动物,温度对昆虫的影响至关重要。过高的环境温度将直接使昆虫的生长发育、繁殖、种群发展及生存等受到严重的改变。以往,人们根据环境气候条件、虫群基数及食物等来预测预报害虫的发生,但是高温条件改变了昆虫的生存状况,导致在昆虫预测报中信息偏差而不准确。不同种类的昆虫对高温胁迫的耐受性表现出高低不同,有些昆虫在高于正常生长温度较多的条件下仍能正常生存,而有些昆虫则对温度的变化极其敏感,较小范围的温度波动就可能对其产生较大的影响甚至死亡。麦红吸浆虫((sitodiplosismosellana,diptera:cecidomyiidae)属于双翅目瘿蚊科,是广泛分布在北半球小麦产区的一种主要害虫,以幼虫潜伏在颖壳内吸食正在灌浆的汁液,造成麦粒瘪疮、空壳或霉烂而减产,全球每年因小麦吸浆虫的为害而造成的经济损失高达数亿美元。在中国,主要为害冬小麦,也为害一些其他禾本科植物如纤毛鹅观草、节节麦、大麦和雀麦。麦红吸浆虫通常一年或多年发生一代,春季成虫羽化后产卵在新抽出的穗上,幼虫孵化后进入颖壳吸食麦粒。两周后遇到降雨或重露水落入土壤中越夏和越冬。若春季干旱年份越冬幼虫会继续滞育直至下年春天。随着气候变暖,春季温度的增加,小麦吸浆虫发生区不断向北移动,新发生区发生程度呈现加重的趋势。在整个长江中下游—华北平原冬麦区,小麦吸浆虫发生面积、防治成本和防治后的小麦产量损失的增加,主要来自华北的新发生区。从1985年到2014年期间,新发生区累计发生面积16.66百万公顷,防治成本累计达到390.26百万美元,防治后小麦产量损失累计897.16千吨。气候变暖导致吸浆虫在华北地区发生,已经给小麦生产造成了严重后果。冬季低温及夏季的高温对吸浆虫的扩散、爆发有重要推动作用,因此,研究麦红吸浆虫在不同温度条件下的耐受性及其作用机理对于吸浆虫在农业生产上发生的预测预报、综合防控及吸浆虫发生北界的研究具有重要的意义。研究麦红吸浆虫不同温度影响下基因的表达情况并进一步对功能有一个初步了解,实时pcr(real-timequantitativepcr)作为一种在转录水平上对mrna表达量进行定量的方法,具有快速、可重复性好和高灵敏度等优点,是监测不同温度影响条件下基因表达情况的有力的工具。实时荧光定量pcr是一种强有力的检测基因表达谱的方法,可以分析基因在不同时期的表达差异以及经过不同处理的样本间基因的表达差异等等。在分析中选用一种合适的内参基因对目的基因的表达量进行校正可以提高该方法的灵敏度和重复性。由于目前许多目的基因的实时荧光定量研究是在多个不同的组织中进行比较的,因此需要尽量一致的实验条件,但由于样品在rna的产量、质量以及逆转录效率上可能存在的差别,因此实验中的表达差异比较大。为了降低实验中的差异,通常在研究中会选择看家基因作为内参基因对目的基因进行校正和标准化,以校正转录效率和cdna用量,弥补制备过程中样本纯度和浓度的差别,从而避免mrna转录的高动态性、样品操作及下游处理步骤的多变性造成的误差,因此选择合适的内参基因成为实验结果准确的关键。但是没有一种内参基因mrna表达水平在所有的条件下是一成不变的。在各种因素影响下,如在实验条件下,细胞发育的不同阶段,内参基因的表达水平是不同的。因此,研究不同的环境条件下,某一有机体内基因的表达情况,内参基因的选择至关重要。技术实现要素:本发明的目的是提供一种检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因及其应用。本发明采用的技术方案为:本发明提供了一种检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因,所述内参基因为rpl13(ribosomalproteinl13)基因,其核苷酸序列为:aacttagccttacgtttacgttccaaatttttgacaacgtcttggtattgccatccgacttcatgtgacaaacggccaattttgcagtatttgcgatctgatcgcaagcacaacactctaagggcagttgggacgcagacacgacgtttcttttcgtatgctggtgggataccgtcaaacacacgcaaacgtttcaatgcggcttgaccacggagagtcttgtgtgggatcattcctcggacagccttgtagaaaatacggcatggagctctaaagtggaatggtccgcgagctgggttgacattacagcgcttgcgcaagtaattcaagtacttgactttgtttctatagaaatgacctgaaagggtcaattcttcgcatcgtacaacgacgactttgccaccttgcaaaaggtttttggctacaactgaagcaagtcgaccgaccaagtggccacggccgtcgatgattactgccttattgtttaaacctggcatgtttacgcagcaaatacaaaaagaatggccgaaagg(seqidno.1)。根据上述的内参基因,优选地,所述不同温度胁迫为:将麦红吸浆虫幼虫分别在0℃、10℃、20℃、30℃和40℃下各处理0.5h。本发明的rpl13基因能够作为检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因应用。根据上述的应用,所述不同温度胁迫为:将麦红吸浆虫幼虫分别在0℃、10℃、20℃、30℃和40℃下各处理0.5h。本发明用于检测rpl13基因表达所用的引物为:上游引物f1:cttggtattgccatccgact(seqidno.7);下游引物r1:ttgcgtgtgtttgacggtat(seqidno.8)。本发明的积极有益效果:本发明首次从6种候选内参基因中筛选出麦红吸浆虫不同温度胁迫下能最稳定表达的内参基因rpl13基因,所述rpl13基因可作为内参基因用于研究分析麦红吸浆虫不同温度胁迫下功能基因的表达,提高了荧光定量pcr方法检麦红吸浆虫基因转录表达水平的准确性;避免了由内参基因mrna表达稳定性差造成的麦红吸浆虫基因mrna表达定量检测精确度低的问题;同时,本发明为后续不同温度条件下麦红吸浆虫基因的定量提供了参考,对定量实验的结果准确性提供了保证。此外,本发明设计的检测rpl13基因的pcr引物具有特异性,而且其扩增效率达到了96%以上,标准曲线回归系数均大于0.92,因此该引物能够为准确定量检测rpl13基因提供坚实基础。附图说明图1为本发明6个候选内参基因的实时荧光定量pcr溶解曲线图。图2为本发明6个候选内参基因的标准曲线图。图3为genorm分析6个候选内参基因的表达稳定度。图4为麦红吸浆虫pds基因在0℃、10℃、20℃、30℃和40℃的相对表达量。具体实施方式以下通过具体的实施例对本发明作进一步说明,但并不限制本发明的范围。在不偏离本发明的精神和范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改或替换均落入本发明的保护范围。若未特别指明,实施例中所用的原料、化学试剂均为常规市售商品,所用的技术手段为本领域技术人员所公知的常规手段。实施例1:样品制备取3月初带有麦红吸浆虫的虫土,经过拣选,挑取麦红吸浆虫越冬幼虫,将挑取的越冬幼虫置于培养箱中饲养,饲养温度为20±1℃,湿度为70±5%,光周期为14h:10h(光照:黑暗)。不同温度胁迫处理时,每个处理温度选取的30头幼虫置于1.5ml离心管中,每个管中加入0.4mlddh2o中,然后分别放置到温度为0℃、10℃、20℃、30℃和40℃的水浴锅中30min,期间每隔5min摇晃一次,以保证温度的准确性。30min后迅速取出,然后将温度胁迫处理后的麦红吸浆虫样品快速转移到新的1.5ml离心管中,进行液氮速冻,速冻后放置于-80℃中备用。实施例2:样品rna提取取实施例2中不同温度处理后的麦红吸浆虫幼虫样品50-100mg,各加入0.5-1ml液氮,然后各加入1000μltrizol进行研磨(首先加入200μl的trizol于样品组织中,采用与离心管配套的研磨棒进行研磨,直至看不到明显的组织后,再加800μl的trizol摇匀),研磨后在室温下放置5min;然后各加入200μl的氯仿摇匀,摇匀后在室温下放置2-3min;放置后分别置于冷冻离心机中,在4℃、12000g的条件下离心15min;分别去除上清液,然后各加入500μl异丙醇摇匀,摇匀后置于室温下放置10min,放置后分别在4℃12000g的条件下离心10min,再次分别去除上清液,然后各加入1ml75%的乙醇,分别在4℃7600g的条件下离心5min;离心后分别用移液枪吸除上清液后,将装有各样品的离心管于空气中干燥5min,再各加入40μl的depc水溶解rna,然后分别置于冷冻离心机中4℃离心15sec,置于-80℃下保存备用。实施例3:反转录(1)dna去除:取5μl实施例2提取的rna(200ng/μl)置于冰上,然后向其中加入5×gdnaeraserbuffer2.0μl、gdnaeraser1.0μl和rnasefreedh2o2μl,混合均匀后在42℃反应2min,得到去除dna的rna溶液,放置在冰上备用;(2)反转录反应:所述反转录的反应体系为(20μl):5×primescriptbuffer2(forrealtime)4μl、primescriptrtenzymemixi1μl、rtprimermix1μl、去除dna的rna溶液10μl、rnasefreedh2o4μl,共计20μl。所述反转录的反应条件为:37℃15min,85℃5sec,4℃10min。反转录后得到cdna样品,向cdna样品中加入180μl的dh2o,保存于-20℃冰箱备用。实施例4:实时荧光定量pcr内参基因的获取及引物设计获取内参基因序列:选取麦红吸浆虫ribosomalproteinl13(rpl13)、glyceraldehyde-3-phosphatedehydrogenase(gapdh)、rnapolymerasesubunitⅱ(rpⅱ)、succinatedehydrogenasecomplex,subunita(sdha)、tata-boxbindingproteinii(tbpⅱ)和18s-ribosomalrna(18srrna)等6个基因为候选内参基因,基因序列由转录组测序获得,所述获得的具体过程为以麦红吸浆虫圆茧、幼虫、蛹、成虫样品为材料,利用illuminahiseq2000测序平台对麦红吸浆虫转录组进行测序,对测序结果进行系统的生物学分析和注释,利用生物信息学方法从上述转录组中挖掘出6个内参基因的候选基因。并通过与数据库blast比对确定rpl13、gapdh、rpⅱ、sdha、tbpⅱ和18srrna的核苷酸序列,见seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5、seqidno.6。设计特异性引物:基于麦红吸浆虫内参基因序列,根据定量pcr引物设计原则,利用primer3(http://bioinfo.ut.ee/primer3-0.4.0/primer3//)软件分别设计6个内参基因的定量pcr引物(见表1),引物设计条件为:退火温度60℃,gc含量42-55%,引物长度为19-21bp。表1本发明6个候选内参基因的引物内参基因上游引物下游引物rpl13cttggtattgccatccgactttgcgtgtgtttgacggtatgapdhgacgcaccgatgtttgtatgctttagccaatggagccaagrpⅱcactgtgttcgaccatgaccccattcaacacatccagacgsdhatggtatgccattctcacgaatctatgcgcttgtcctcctttbpⅱgggtgcgaagagtgaagaaggcaactgccgaccatatttt18srrnagcatttgccaaaggtgttttccaattgctagctgacatcg实施例5:内参基因实时荧光定量pcr扩增溶解曲线及引物扩增效率的测定用于测定扩增效率的dna样品的制备:取出上述实施例3得到的保存于-20℃于冰箱中的cdna样品,0℃、10℃、20℃、30℃和40℃的cdna样本各取5μl,置于同一离心管中,得到总量为25μl的cdna混合样品。取5μlcdna混合样品进行10倍的倍比稀释,得到浓度为10-2、10-3、10-4、10-5和10-6的cdna混合样品稀释液。分别以浓度为10-2、10-3、10-4、10-5和10-6的cdna混合样品稀释液作为反应模板,进行荧光定量pcr反应,以判断6个候选内参基因扩增引物的特异性及扩增效率。为保证实验结果的可靠性,设置两组平行实验,同时,每个内参基因的荧光定量pcr包括两个平行的单一阴性对照,阴性对照以去离子水作为反应模板,以验证基因引物有无二聚体。实时荧光定量pcr的体系为(20μl):sybrpremixextaqii(2×)10μl,上游引物(10μl/mol)0.8μl,下游引物(10μl/mol)0.8μl,roxreferencedye(50×)0.4μl,dna模板2μl,dh2o6μl,共20μl;其实时荧光定量pcr扩增反应条件为:95℃预变性10min;然后95℃、15sec,60℃、30sec共进行40个循环。实时荧光定量pcr扩增循环结束后分析、绘制溶解曲线,并进行熔解曲线分析以确定扩增产物的特异性。温度从60℃缓慢递增到95℃,连续测定样品的荧光强度以获取溶解曲线。溶解曲线分析步骤为:95℃、15sec,60℃、1min,然后开始每个循环增加0.3℃,从60℃缓慢递增到95℃结束。所述实时荧光定量pcr扩增采用的麦红吸浆虫体内候选内参基因为rpl13、gapdh、rpii、sdha、tbpii和18srrna。本发明6个候选内参基因的实时荧光定量pcr扩增溶解曲线见图1,由图1可知,本发明6个候选内参基因的实时荧光定量pcr扩增溶解曲线均为单峰,因此,说明本发明设计的6个候选内参基因的扩增引物均具有特异性。通过steponesoftwarev2.1系统软件的分析,分别计算以浓度为10-2、10-3、10-4、10-5和10-6的cdna混合样品稀释液作为反应模板的实时荧光定量pcr反应中各内参基因的循环阈值(ct值),每个稀释梯度的起始模板浓度与ct值之间呈线性关系,起始模板中所含原始拷贝数越大,ct值越小。根据ct值在excel中经对数拟合做图,得到每一种内参基因引物扩增标准曲线(见图2)。根据标准曲线斜率确定每对引物扩增效率y=[10∧(-1/slope)-1]×100,其中y为扩增效率,slope为曲线斜率。本发明6个候选内参基因的扩增效率见表2。表2本发明6个候选内参基因的标准曲线方程、回归系数r2和扩增效率候选内参基因标准曲线方程扩增效率斜率回归系数rpl13y=-2.854x+34.446124.3%-2.850.985gapdhy=-2.7154x+33.928133.2%-2.720.924rpⅱy=-2.3756x+37.416163.1%-2.380.937sdhay=-2.7029x+36.985134.6%-2.700.967tbpⅱy=-3.0562x+40.472112.2%-3.060.99418srrnay=-3.189x+30.405105.8%-3.190.982实施例6:候选内参基因的实时荧光定量pcr反应分别提取在0℃、10℃、20℃、30℃、40℃处理30min后的麦红吸浆虫幼虫样本的总rna(rna提取方法同实施例2),分别反转录合成cdna(反转录合成cdna的方法同实施例3),分别以各温度处理后的麦红吸浆虫样本cdna为模板对各候选内参基因(rpl13、gapdh、rpⅱ、sdha、tbp和18srna)进行实时荧光定量pcr扩增,各设置三组平行试验。所述实时荧光定量pcr的反应体系为(20μl):sybrpremixextaqii(2×)10μl,上游引物(10μl/mol)0.8μl,下游引物(10μl/mol)0.8μl,roxreferencedye(50×)0.4μl,dna模板2μl,dh2o6μl,共20μl;所述实时荧光定量pcr的反应条件为:95℃预变性10min;然后95℃、15sec,60℃、30sec共进行40个循环。通过steponesoftwarev2.1系统软件的分析,获得各个候选内参基因在不同温度处理的麦红吸浆虫样本中的荧光定量pcr循环ct值。根据标准曲线将每个候选内参基因实施荧光定量pcr扩增反应所得到的ct值转化为相对定量数据,根据得到的各个候选内参基因在不同温度处理的麦红吸浆虫样本中的荧光定量pcr循环ct值,利用genorm软件程序计算出各个候选内参基因表达稳定度的平均值m;对各个内参基因的表达稳定度进行排序(m值越小,表达越稳定)。genorm软件分析结果显示(图3),不同温度处理条件下的麦红吸浆虫中的gapdh、rpⅱ、rpl13、sdha、tbpⅱ、18srrna表达稳定度的平均值m依次为:0.126196、0.543037、0.073868、0.459417、0.363932、0.278271。表达稳定度由高到低排序依次为:rpl13>gapdh>18srrna>tbpⅱ>sdha>rpⅱ,因此,可以选用稳定性最高的rpl13基因作为内参基因,以检测不同目的基因在不同温度胁迫下麦红吸浆虫体内的转录表达,以提高麦红吸浆虫不同目的基因mrna表达定量检测精确度。实施例7:内参基因的应用分别以0℃、10℃、20℃、30℃和40℃五个温度梯度处理的麦红吸浆虫幼虫为实验样本,以rpl13为内参基因,利用实时荧光定量pcr检测麦红吸浆虫番茄八氢番茄红素脱饱和酶基因(phytoenedesaturase,pds)在不同温度胁迫下的表达水平。所述实时荧光定量pcr的反应体系为(20μl):sybrpremixextaqii(2×)10μl,上游引物(10μl/mol)0.8μl,下游引物(10μl/mol)0.8μl,roxreferencedye(50×)0.4μl,dna模板2μl,dh2o6μl,共20μl;所述实时荧光定量pcr的反应条件为:95℃预变性10min;然后95℃、15sec,60℃、30sec共40个循环。利用公式2^-△△ct得到基因的相对表达量。其中△ct=目的基因ct-内参基因ct,△△ct=目的基因△ct-对照△ct(其中,对照△ct=0℃ct-内参基因ct)。统计分析结果显示(图4):在0℃、10℃、20℃、30℃和40℃五个不同温度处理情况下,pds基因的表达量差异较大,相对于0℃的表达量,pds基因在10℃有少量下调,其表达量为0℃的表达量的0.8倍,在20℃、30℃有一个大的表达量的升高,分别达到了0℃的表达量的4.0倍和4.7倍,到40℃后pds基因的表达量又有所降低,为0℃的表达量的为2.9倍;而内参基因rpl13的表达在不同温度条件下则能稳定表达,因此利用rpl13作为内参基因来标准化目的基因的表达是可行的,而且能够准确反应目的功能基因在不同温度胁迫下基因表达的变化,可作为定量麦红吸浆虫不同温度胁迫的内参基因。sequencelisting<110>河南省农业科学院植物保护研究所<120>一种检测麦红吸浆虫在不同温度胁迫下基因转录表达的内参基因及其应用<130>1<160>8<170>patentinversion3.5<210>1<211>533<212>dna<213>artificialsequence<220><223>1<400>1aacttagccttacgtttacgttccaaatttttgacaacgtcttggtattgccatccgact60tcatgtgacaaacggccaattttgcagtatttgcgatctgatcgcaagcacaacactcta120agggcagttgggacgcagacacgacgtttcttttcgtatgctggtgggataccgtcaaac180acacgcaaacgtttcaatgcggcttgaccacggagagtcttgtgtgggatcattcctcgg240acagccttgtagaaaatacggcatggagctctaaagtggaatggtccgcgagctgggttg300acattacagcgcttgcgcaagtaattcaagtacttgactttgtttctatagaaatgacct360gaaagggtcaattcttcgcatcgtacaacgacgactttgccaccttgcaaaaggtttttg420gctacaactgaagcaagtcgaccgaccaagtggccacggccgtcgatgattactgcctta480ttgtttaaacctggcatgtttacgcagcaaatacaaaaagaatggccgaaagg533<210>2<211>1246<212>dna<213>artificialsequence<220><223>1<400>2ccttgatgtgacactcgtcttctgaaagcggttgtgttaagtcatatcccatcaaagcaa60gcaagaaggtaaaaaaatgtcgaaaattggaatcaacggttttgggcgtatcggtcgtct120tgtgctccgtgctgctgttgagaagggagctcaagttgttgccgtgaatgacccatttat180tggagtcgattacatggtgtatttattcaagtatgattccacccatggtcgtttcaaagg240aactgtcagtgctgaaggtggttttttggttgtcaatgggcaaaagataactgttttttc300ggaacgcgatccaaaggacatcaaatgggcatcagctggagctgaatacatcgtcgaatc360gactggtgtatttacaactattgaaaaggctagcactcacttggctggtggtgcaaagaa420agtaatcataagtgctccatcagctgacgcaccgatgtttgtatgcggagtaaatttgga480tgcctatgatccaaaatatcaagttatttcgaatgcttcatgtacaaccaattgcttggc540tccattggctaaagtaattcatgacaattttgagattgtcgaaggtttgatgacaacggt600gcatgctacaaccgcaacccaaaaaactgttgatggtccatctggcaaattgtggcgcga660cggtcgcggggctgctcagaatataattccagccgcaactggcgctgctaaagccgtcgg720gaaggttattccgtctttgaacggtaaattgactggtatggctttccgtgtaccaactgc780aaatgtttctgttgttgatctcacttgccgccttggcaaaccagccaagtacgatgaaat840caagcagaaaatcaaggaggctgccgaaggcccattgaaaggaattctcgattacaccga900agatgaagtggtttcatcagacttcattggttctactcattcgtcagtctttgacgcaaa960ggccggaatccaactttcggacacctttgtcaagctcatttcttggtacgacaatgagta1020cggttatagcaaccgtgtcattgacctcatcaaatacgttcaaaccaaagattaaacgtt1080ttgttttgaattcagtcatataagtcacattgaatgtaactaaatactaaattcacaata1140aaatattatatcccagaacaaaaatttgtccacgcactacatgtagaaataattttgcag1200taatgtatcattgaaatgcgtcttttttaataacactgcacaacac1246<210>3<211>4320<212>dna<213>artificialsequence<220><223>1<400>3tcagcgtgtgacgcggcatccatgagcacatcaaccgtttcttcgaatgagcatctcatc60agtgcaccagtatcttgtcgattgataccgtgacgtgtgatggccatcaaatggccctta120gccgtcatcacatcgcacaacaaggctaaatgccgatagttgacgtacaatccgtagaat180tgcaacacggcattcatttctttttcaacggattttcgcaccgcttcgattccaagcact240tggaaaatctcacaaatatcgttactgtacgtacgaaccggatccacatcacgttcgctc300aatactttcatcattgaagtaccgtccgtttccaatagccattcgccaatggccttgaat360tcaccggtgtcagtgattgaaatccgtttcttactgtctgtttgtggcaaatgcatgtac420actttgctaatggcttcgataccttgcagtgtcatatcggacaacatgttcgcttcaatg480caacgcaagaacatatcgtcttccattttatcaacggtgtcctcttcattgtcttggaat540ttactgtcttcactgttcatgatacgaatacgtagtaccaatttgtcagcgttatcatcg600ttgaagatacaattcaaatcctcaccaaaaccggcattaatcttctccgcaatctgttcc660atggtcaatttcttatcagtcatacgtttgcgatccaattcaatacgcaacagccatggt720gaaattcgtgttggatcgaaatccggcatttcatagtaaacgttaacgaattcttgatct780tcggcgataacagttcgttgtggatcgggatcatagtagatggccgtgtttgcggtgacc840ttacgcaaagtggtgtgttccaagcgacacagtacatttttggctttttcagcatcacgt900gcagcaccgccggtcaagaaaacggtcaacgatggagctttcggtttctttgaaatgttg960ataatttcctttaaacgaggcacacccagcgttacgttcttcgatgacacaccagcaaaa1020tggaaagtgttcagtgtcatctgagtggccggttcaccgagtgactgagcggccaaagcg1080ccaaccatttcacctgggttggcttgagcctgttggaaacgtgattcaatttcacccacc1140aaccattcaaacgcttcgctggtcaaacgatgatcttcggccacatatttggtgcacagt1200gttgaacgcaccaaacactggaacagcaatgttgcattttcattggcttgcttcgaaatt1260cgatcgttacccgctacaattacacatttttgtagcaattctttgacaccattgatcact1320ttgattggactcaaatcggttggcacacgtttgttaatgtggaaaatcttctgtacattc1380caaatcatacgctgtaaattgcatggcaatacgacttttgattcaccacttgggaaaatc1440tgacgcaatgcctcacgatcacggcacaattgatcccattcaccttccagttcttggatt1500acgaaacccgattccgtcaattcttttacaacatcttcattgaaaatgcgtcgcaagtaa1560cgttcattggacatatcgaatttgaaacgtttctcaaatgttttgtgcgacaatttaatc1620gtcggcaaactttggaattcgaccgtttcaccgcacagaccatcctcaccgtaccgcaat1680tgaatcaattgtccaaccgaattacgaacggttccatcgtagttcaccatcaccgactcc1740atagctttgatcaaacgacgttggatataaccagtttcggcggtttttacagcagtatcg1800ataagaccttcacgacctcccatagcgtggaaatagaattcagacggtgtcaaaccggcc1860agatatgaattttcaacgaaacctcttgaatccggaccgtaatcgtctttaatgaaatgt1920ggcaacgttcgcttacggaacccgaacggaatacgtttaccttcaacgttttgttgacct1980acacaagcaataacttgagaaatgttgatgttggaaccttttgaacccgaaacgaccata2040gcctttagattattgtattcagtcaatgatttcttcgcagagccaccagttttatcacga2100gcatcgttcaaaatacgatttacttgattttcgaaagtctgacgcaacgtattaccgggt2160gtgggttccaattccatgttgtgagccttttgaataacgccaatgacatcttctttggct2220ttcctaatggactgttggatttcgtgatatgtctgcggatcagcaattgtgtcaccaata2280cccatactgtgaccttccaacagcaaccaattgttaaccgttgtctgaatattaccgtag2340aatcgaccggctatctcgtggcccaattcaaggaacacaatgtgcaacagtgaaccagcc2400gatgttcccagtgttttcttacataaaatgcccataatcaattcactgtgttcgaccatg2460acctttgtgtcaccgggcgagatccatttgtatggcccgtcatcctcttcgtctggatgt2520gttgaatgggttcgaatcaggttcacattgccgggaataatgagggtgaacaattgcttt2580ccggtccatagtggctttggttttaaaatacatggttgtggcattttaccgtcccaagtt2640ggcaaaaacatgagcaaattcatcatttgctccttctcaataaaaacatcacgtttggtc2700atttttcgtactgcggtcaatgtatcttgcacaatacccataaccggtttgtttgcttgc2760ggtgtgataatttggcgcggtgttatgtgaatattttccacttcggcacgcgtttccatc2820gattgtggcacgtgcaagttcatttcgtcaccgtcgaaatcggcattgtaaggcgatgta2880caactgaggttcattcgaaaggttgaccaaggcaacactttgactctgtgacccatcata2940ctcatcttgtgcagcgttggttgtcgattgaaaatgaccaaatcatcgtcgcgcaaatgt3000cgttcaactttgtagccccattgcaaatgcaaatcactgggtttcggatggaatcttaaa3060tcgatacgttcaccattgtcacgaacgatatatttggcgccgggatattgtgaatgtcca3120cgcttaaccagctcttgcatgcgatcgatgttgaatggtgtgacgagttcggggaacgtt3180aaattttgtgcaatcgaacgtggaacgccgacttgatcgatacgcaaatttggatcaggt3240gtgatgaccgtacgtgcggagaaatcgacacgttttcccatcaaattaccacgaatacga3300ccttctttacctttgagccgagccttgaccgcttttaatggtttgccggatttttgtgtt3360gcacgcggcataccgggcatatcattgtctgtgaatgtggccacatgaaattgtagcatt3420ttaatattttcggcgataacatgggcggccgcaccaatcgcttcgttctttttcagttca3480ttgtttgctttgataatgtcggccagtttgtgcgttaaatcgtcttgatttttagccgaa3540ccgaacataacaacggctggtcgtaccgacaacggtggcaccggcatcacggtgacaatc3600atccaatcgggccgcgcatatttcggatccataccaagtatgtaacattcttcgtcggtg3660atatgcttgaaaatttcccacacccgttcggctgtcaccacaatctttttctcctgtgaa3720ttttcattgacatttttccattcggcggttaattccaatccgcttcgtttaattgacggt3780tgataatgaccgcaaccgccatgaccttgtttctttgtcggatcagcttgggcagcgtct3840tttgccaaatccatatcttcaccaccctcacaaatctttttacctttacacaaatcatac3900acatatgtcaaacgtttacgtggttgtccttttgttttcataacaacttctttgattttc3960ggattcgttgagctaaccagcattttcgaacagtagaagcacacgcagcgcagaattttc4020atggtttttgtcaagaaaccgacgtggtaaacgggtttgcataattcaatgtgaccaaag4080tgaccgggacattcagtcatattgccggcacaagtttgacatcgggatgtacgttcgatc4140acaccttgacgcggatccattaaaccgccaagctttggtcgaccaccttccatcgtctcg4200ggatactgaattccaccttccgtaactgacatacgacggatttcatcgggtgatagaata4260ccgaattgcacacgcttcaccgtgcgcaacgttgccttggagtcactcatagccattttg4320<210>4<211>2299<212>dna<213>artificialsequence<220><223>1<400>4tgcttgtcagtttgcctgttctcaaagcattttttacccaattgtcgatgtattctagat60gtttctacgtgtatgcatcgtaaactacacgaaagcgacagacacacagatccatactaa120agcagatgaacagttttatgtgcgtgataaatacgagcgaaacgaaatcgcatgaaataa180gaaaaacacggaaactttatttttcccacattgctgaaatagtgaatattttgaataaga240actcttggtgcggattttaggtgattccatcatgagtgcactgatgaggatttctcaagt300tcttagcaaaaatgctaagtcaatgatcaatgcaagttcggtcggtgccaagagtttaca360ttattcctctggtcaacaaaatgcaaaagtctcttctgatgccatttccaatgagtacaa420agtcgttgaccatgctttcgacgccattgtcgttggtgctggtggtgcaggtttgcgagc480tgcattcggattagtcgctgaaggcttcaaaactgccgtcatcacaaaattgttcccaac540tcgttcacataccattgctgctcaaggcggtatcaatgccgcattgggaaacatggaacc600agacgattggaaattccattgttacgacacagttaaaggttcagattggctcggtgatca660ggatgccattcattatatgacacgtgaagcgccaaaggctgtcattgagttggagaacta720tggtatgccattctcacgaacaaaagaaggtaaaatctatcaacgtgccttcggtggtca780gagttataactatggcaaaggaggacaagcgcatagatgttgtgccgtcgctgacagaac840tggacattcactgttgcacacattgtatggccagtcattgaactatgactgccactattt900catcgaatatttcgcacttgatttgcttatggaaaatggaaaatgcgtcggtgtcattgc960catttgccttgaagacggctctattcatcgattccgtgcaaacagcacagttttggcgac1020gggtggttacggtcgtgcatacttttcatgtacatcggctcacacttgcaccggtgacgg1080aacagcaatggttgcccgacaaaatttaccatgcgaagatcttgaattcgtgcaattcca1140tccgacgggtatttatggtgctggatgtttgattacagaaggttgtcgcggtgaaggtgg1200ttacttggtcaatgctaaaggtgaacgttttatggagcgatacgccccggttgccaaaga1260tttggcatctcgtgatgttgtctcacgttcgatgacaatggaaatccgagagggacgtgg1320tgttggaccactcgaagatcacgttttcttgcaattgcatcatttaccacccgagcaatt1380ggcaatgcgtttgccaggtatttcggaaacggccatgatctttgccggcgttgatgtgac1440ccgtgaaccaatccccgttatacccaccgtacattataatatgggaggcgttccaacgaa1500ttggcgaggcgaatgcttaaccatcgatcaaaatggtaacgataacgttgttgagggatt1560gtatgcagctggagaagcaggttgttcatcggtgcatggtgccaatcgtttgggtgctaa1620ttcattgttggatttggttgtttttggccgtggatgcgctaaaacaattgcagctaaaca1680taagccaggagaaaaagtacctgagttaccggatagtgctggtgaatcgtccgtagccaa1740tttagattatttgttgcacaaaaatggcacagtttcaacggctgatcttcgcttgagaat1800gcaacgcacgatgcaaaatcatgcagctgtattccgtgatggccctgtacttaaagaagg1860ttgcgagaaaatgtccgaaatcttcaaagaattcgccgatattaaagttaccgataaatc1920tcttgtttggaattcagatttgattgaaacattagagttacaaaatctgttgcaaaatgc1980tctgatgaccatttatggtgccgaaaatcgtaaggaatctcgtggtgctcacgctcgcga2040agactacaaaactcgtatcgatgaatacgattattcgaaaccaattgaaggacaacagaa2100gaaatcaatcgacgaacactggcgcaaacacactctgctttggatcaacgaatctggaga2160tgttagcattaaatatagaccagttatcgataagacactcgacgaagaaatgcacacggt2220gccgccagccattcgttcctactaaagcaattttttgtttttatttatcaacaataacaa2280caacaacaacagcaacaac2299<210>5<211>846<212>dna<213>artificialsequence<220><223>1<400>5caatgacaccgatgacacccggctcacaagatcccgctattttgcctcaattacaaaaca60ttgtgtcaacggtgaatttaaattgtaagcttgatcttaagaaaattgcattacatgccc120gtaacgctgagtacaatccaaaacgttttgctgccgtaattatgcgaatacgtgaaccga180ggacgactgccttgatcttttcatcggggaaaatggtgtgtacgggtgcgaagagtgaag240aagattcccgtttggccgcccgcaaatatgctcgaattatacaaaagctcggttttacgg300cgaaatttttagattttaaaatacaaaatatggtcggcagttgcgacgttaaatttccaa360ttcgattggagggtttagttttaacacactctaagttcagttcgtatgagccagaattgt420ttccgggtcttatttatcgtatggtcaaaccaagaattgtgctgttgatttttgtgtcgg480gcaaagtcgtacttaccggtgccaaagttcgtcaagaaatctacgatgcattcgacaaca540tttatccaattttgaagagcttcaagaaacaataggaaaagtaaaaatgacatcggtgat600ttggtgcggctaacatatgacacacacgcaaacaaaacaaaaccaaacacatcaacattt660cgattagactaaataaggagttcccttagcagtacacacacacatcggtcttttttgact720taatcatcattaatttttatctcgtaatgtcgtttagtttgattattaaaattacaaaaa780caacaaaaaatggaataaatgattatttcacgaagaaaattcccatgcaagaaaaaaaaa840acgaac846<210>6<211>473<212>dna<213>artificialsequence<220><223>1<400>6tgttcacttaagctcatttcagtgctcttcatcgagtgttgttgtgggccgatacaaatt60actttgaacaaattagagtgcttaaagcaggcttgtaatgcctgaatattttgtgcatgg120aataatgtaatgggacatctgttctactttcattggttttttagatcaagatgtaatgat180taatggaggcaattgggggcattagtattacaacgcgagaggtgaaattcttagaccgta240gtaagactaactaaagcgaaagcatttgccaaaggtgttttcatttaatcaagaacgaaa300gttagaggttcgaaggcgatcagataccgccctagttctaaccataaacgatgtcagcta360gcaattgggtggagctactactatggctctctcaggcgcttctcgggaaaccaaagcttt420tgggctccgggggaagtatggttgcaaagctgaaacttaaaggaattgacgga473<210>7<211>20<212>dna<213>artificialsequence<220><223>1<400>7cttggtattgccatccgact20<210>8<211>20<212>dna<213>artificialsequence<220><223>1<400>8ttgcgtgtgtttgacggtat20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1