一种miRNA预测方法、miRNA系统及应用与流程
本申请涉及核酸分析检测领域,特别是涉及一种mirna预测方法,mirna预测方法得到的mirna系统,以及mirna预测方法的应用。
背景技术:
mirna是一类由内源基因编码的长度约为22个核苷酸的非编码单链rna分子,通过与基因完全或非完全互补结合,进行基因表达调控,进而引起形状、表型的差异。目前mirna相关的数据库很多,诸如mirbase、tarbase等,有利于对mirna进行下游分析。但是,除了少部分模式生物外,大部分物种的已知mirna数量非常有限,例如文昌鱼在mirbase中收录的mirna只有173条,这在某种程度上局限了对mirna的进一步研究,因此,对于潜在的新的mirna的鉴定是至关重要的。
目前也有很多mirna预测方法,如mirdeep2、mira、mireap等;它们都是基于测序数据,再结合基因组上的比对结果,通过判断测序片段在基因上的侧翼序列是否能形成发夹结构,来推测潜在的mirna分子。这些mirna预测方法,都是基于单次测序结果进行预测,对于不同批次或不同实验室的数据平行分析,需要全部放在一起进行,不仅影响项目周期,也增加计算资源消耗;并且,现有的mirna预测方法整体运行时间较长。
技术实现要素:
本申请的目的是提供一种新的mirna预测方法,该mirna预测方法的应用,以及该mirna预测方法获得的mirna系统。
本申请采用了以下技术方案:
本申请的一方面公开了一种mirna预测方法,包括以下步骤,
(1)设定种子序列长度;其中,种子序列长度是根据已知mirna的前体序列发夹结构特征设计的,优选的,在对待测物种的mirna进行预测时,是根据待测物种目前已经公开的mirna前体序列发夹结构特征设计的;
需要说明的是,mirna前体序列的发夹结构是目前预测mirna的主要依赖特征,一般认为符合类似结构的序列,是潜在的mirna分子;本申请根据mirna前体序列的发夹结构特征设定种子序列长度,具体来说,就是根据待测物种已知mirna前体序列的发夹结构长度或平均长度或最具代表性的长度,来定义种子序列的长度,而本申请的预测方法就是要找出和待测物种已知mirna前体序列的发夹结构最接近的前体序列,将其作为潜在的mirna分子,放入mirna集中,实现mirna预测;
(2)对a、t、c、g四种碱基进行步骤(1)设定的序列长度的全排列,形成种子序列集;
需要说明的是,四种碱基进行设定的序列长度的全排列,即每个碱基分别为a、t、c、g,如此排列形成种子序列集;例如,设定长度为7bp,即设定种子序列长度为7bp,则其中每个碱基分别为a、t、c、g进行排列,形成47条序列,所有序列构成种子序列集;
(3)去除种子序列集中的冗余种子序列,具体包括,如果一条种子序列与另一条种子序列的反向互补序列相同,则只保留其中一条;
(4)在步骤(3)剩余的种子序列中,搜索每一条种子序列和其反向互补序列在待测物种的基因组上的位置,得到种子序列位置集;
需要说明的是,如果种子序列或其反向互补序列在待测物种的基因组上没有相应的位置,则直接滤除该种子序列;
(5)将步骤(4)获得的种子序列位置集,在基因组上,按从小到大的顺序排列;
(6)按位置集的位置遍历,如果基因组上相邻的前后两个位置正好是互补序列,且两者的距离小于或等于设定前体序列长度,则输出前面位置对应的种子序列前50bp至后面位置对应的种子序列后50bp的一段核酸序列,作为初选前体序列;
需要说明的是,其中初选前体序列实际上就是包括两个种子序列、两个种子序列之间的序列,以及两个种子序列前后各50bp的一段基因组序列;其中,“设定前体序列长度”默认为500bp,这是参考目前大多数物种的前体序列长度而确定的,当然,根据具体分析的对象物种,统计其前体序列长度,可以对“设定前体序列长度”进行相应的调整,在此不做具体限定;
(7)采用二级结构预测软件对步骤(6)获得的初选前体序列进行二级结构预测,如果不能形成发夹结构,则弃用该初选前体序列;如果能形成发夹结构,则从发夹结构的起始点开始,逐个碱基向后推移的取20-25bp的片段及其互补序列片段作为预成熟体,对各个预成熟体进行打分,取其中分数最高者及其互补序列,并给出两者对应的二级结构;其中,对各成熟体进行打分的依据主要是碱基互补情况,即互补匹配数和空位数,匹配数越多,空位数越少,分数越高。
需要说明的是,虽然在筛选时初选前体序列的前后两个种子序列是互补的,但是,因为种子序列长度较短,所以筛选到的初选前体序列可能只有两个种子序列那段是互补的,而整条初选前体序列本身折叠不出二级结构;
需要说明的是,取其中分数最高者及其互补序列是因为,本申请的预测方法不能确认最终的mirna序列是预测得到的成熟体序列还是其互补序列,因此,将分数最高的预成熟体序列及其对应的互补序列一起放入mirna集中;
(8)合并步骤(7)获得的分数最高的成熟体序列及其对应的互补序列,以及各成熟体序列和其互补序列的二级结构,组成待测物种的mirna集。
需要说明的是,本申请的mirna预测方法,直接在待测物种的基因组序列中进行搜索,所获得的mirna集,实际上包含了该待测物种所有可能的mirna;而现有的mirna分析方法通常只能得到待测物种某个组织特定时期的mirna;相比之下,本申请的预测方法及其获得的mirna集更加完整。可以理解,本申请的mirna集,是包含了物种在各个时期所有可能存在的mirna,一方面,这些mirna可能只是理论预测存在的,并非全部都是真实存在的mirna,本申请的预测方法提供所有可能存在的mirna方便了后续研究;另一方面,本申请的预测方法只是能够相对其它方法,更准确的预测mirna,但是,对于具体的真实的mirna成熟体本身而言,差一个碱基就会导致完全不同的成熟体,因此,本申请预测的mirna在具体位置上会与真实的mirna存在一些差异,但是,其必然都包含在本申请所预测的前体序列内。因此,在步骤(7)中通过二级结构预测软件筛选获得能够形成发夹结构的前体序列,根据不同的使用需求,至此已经能够满足后续科研的使用需求,则无需进一步的列举所有可能的预成熟体并对其进行打分、筛选分数最高者。
还需要说明的是,现有的mirna分析方法通常是单线程运行,即每个步骤串行运行,很少把中间某一部分拆开并行,因此整体运行时间较长;而本申请的mirna预测方法,在将每条种子序列和其反向互补序列定位到基因组上后,可以统一输出所有可能的初选序列,然后各初选序列可以并行,同时进行二级结构预测,最终合并组成mirna集,即可以采用多线程执行,能有效提升运行速度,缩短预测时间。
优选的,本申请的mirna预测方法还包括步骤(9),将待测物种所有已知mirna与步骤(8)生成的mirna集比对,将比对上的已知mirna在mirna集中进行标注,将未比对上的已知mirna加入mirna集中形成新的mirna集,并对新加入的已知mirna进行标注。
优选的,步骤(1)中,设定种子序列长度为2-8bp。
更优选的,步骤(1)中,设定种子序列长度为7bp。
需要说明的是,理论上种子序列的长度只要小于或等于mirna前体序列的发夹结构长度即可,这个长度通常为2-8bp,但考虑到运算效率,如果长度过小的话会有很多组合,运行比较慢;而种子序列太长的话,不一定能找到互补对,因为mirna和对应的star序列并不是完全互补存在。因此,优选的默认设定种子序列长度为7bp,特别是待测物种可供参考的mirna较少的情况下,默认设定为7bp,既保证一定能找到互补对,又能较快完成检索。
优选的,步骤(7)中,采用的二级结构预测软件为rnafold。
本申请的另一面公开了本申请的mirna预测方法在制备mirna预测装置或mirna预测系统中的应用。
可以理解,基于本申请的mirna预测方法,完全可以制备一个独立的用于mirna预测的装置或者系统,以方便mirna的研究。
本申请的再一面公开了一种用于mirna预测的装置,该装置按照本申请的mirna预测方法进行mirna预测。
本申请的再一面公开了一种用于mirna预测的系统,该系统按照本申请的mirna预测方法进行mirna预测。
本申请的再一面公开了一种mirna系统,该mirna系统中包含由本申请的mirna预测方法获得的mirna集。
需要说明的是,本申请的mirna系统中包含本申请的mirna预测方法获得的mirna集,由于该mirna集能够涵盖待测物种更为完整的所有的mirna信息,因此,更方便待测物种的mirna研究。其中,本申请的mirna系统可以包括一个或多个待测物种的mirna集,或者本申请的mirna系统也可以作为一个mirna研究平台,包括某类物种或所有物种的mirna集。
还需要说明的是,本申请中,mirna预测系统跟mirna系统可以是两个独立的运行系统,mirna预测系统是基于本申请的mirna预测方法构建的用于预测某个待测物种的mirna集的系统,mirna系统则是涵盖一个或多个物种的mirna集的系统。
本申请的有益效果在于:
本申请的mirna预测方法,直接在基因组序列中搜索mirna,能够获得待测物种所有可能的mirna,与现有的mirna分析方法相比,本申请的mirna预测方法所得到的mirna集更完整。并且,本申请的mirna预测方法效率高、更便捷,为后续的mirna深入研究奠定了基础。
附图说明
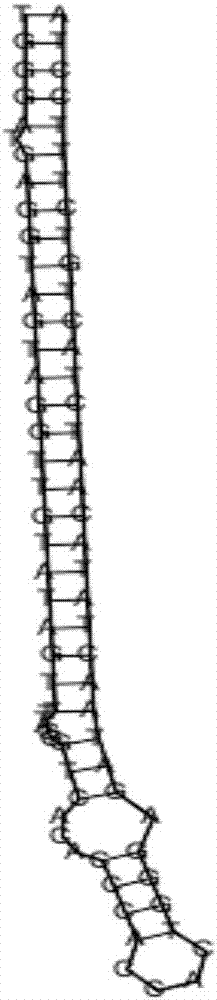
图1是本申请实施例预测的mirna二级结构。
具体实施方式
现有的mirna预测方法都是基于单次测序结果进行的,或者将多次测序结果放在一起进行mirna预测;一方面,基于测序结果的mirna预测,周期较长,成本高;另一方面,基于单次或多次测序结果的mirna预测,只能反映组织在某个或某几个特定时期的mirna表达信息,该特定时期即采集测序样品时的组织所处的时期,因此,不能更全面的反映待测物种所有的mirna表达信息。
基于以上问题,本申请提出了一种新的mirna预测方法,本申请的预测方法,不需要依赖于测序,直接在待测物种的基因组序列中进行搜索,获得的mirna集不是待测物种某个特定时期表达的mirna,而是待测物种所有的可能的mirna,因此,本申请的mirna预测方法能够得到更完整的mirna。并且,本申请的mirna预测方法,不依赖于测序,可多线程执行,效率更高、更便捷,所获得的完整的mirna集,更方便后续研究。
下面通过具体实施例对本申请作进一步详细说明。以下实施例仅对本申请进行进一步说明,不应理解为对本申请的限制。
实施例
本例将人的20条mirna的前体用n连接起来作为模拟的基因组序列,对其进行mirna预测,拼接获得的模拟基因组序列为seqidno.1所示序列,其长度为2566bp。
seqidno.1:
tgggatgaggtagtaggttgtatagttttagggtcacacccaccactgggagataactatacaatctactgtctttcctannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnaggttgaggtagtaggttgtatagtttagaattacatcaagggagataactgtacagcctcctagctttcctnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncggggtgaggtagtaggttgtgtggtttcagggcagtgatgttgcccctcggaagataactatacaacctactgccttccctgnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngcatccgggttgaggtagtaggttgtatggtttagagttacaccctgggagttaactgtacaaccttctagctttccttggagcnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncctaggaagaggtagtaggttgcatagttttagggcagggattttgcccacaaggaggtaactatacgacctgctgcctttcttaggnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncccgggctgaggtaggaggttgtatagttgaggaggacacccaaggagatcactatacggcctcctagctttccccaggnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnntcagagtgaggtagtagattgtatagttgtggggtagtgattttaccctgttcaggagataactatacaatctattgccttccctgannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnntgtgggatgaggtagtagattgtatagttttagggtcataccccatcttggagataactatacagtctactgtctttcccacgnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnccttggagtaaagtagcagcacataatggtttgtggattttgaaaaggtgcaggccatattgtgctgcctcaaaaatacaaggnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngtcagcagtgccttagcagcacgtaaatattggcgttaagattctaaaattatctccagtattaactgtgctgctgaagtaaggttgacnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngtcagaataatgtcaaagtgcttacagtgcaggtagtgatatgtgcatctactgcagtgaaggcacttgtagcattatggtgacnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnntgttctaaggtgcatctagtgcagatagtgaagtagattagcatctactgccctaagtgctccttctggcannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngcagtcctctgttagttttgcatagttgcactacaagaagaatgtagttgtgcaaatctatgcaaaactgatggtggcctgcnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncactgttctatggttagttttgcaggtttgcatccagctgtgtgatattctgctgtgcaaatccatgcaaaactgactgtggtagtgnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnacattgctacttacaattagttttgcaggtttgcatttcagcgtatatatgtatatgtggctgtgcaaatccatgcaaaactgattgtgataatgtnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngtagcactaaagtgcttatagtgcaggtagtgtttagttatctactgcattatgagcacttaaagtactgcnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnntgtcgggtagcttatcagactgatgttgactgttgaatctcatggcaacaccagtcgatgggctgtctgacannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnggctgagccgcagtagttcttcagtggcaagctttatgtcctgacccagctaaagctgccagttgaagaactgttgccctctgccnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnggccggctggggttcctggggatgggatttgcttcctgtcacaaatcacattgccagggatttccaaccgaccnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnctccggtgcctactgagctgatatcagttctcattttacacactggctcagttcagcaggaacaggag
seqidno.1所示序列中“n”为随机序列。
本例的20条mirna分别为:hsa-let-7a-5p、hsa-let-7a-2-3p、hsa-let-7b-5p、hsa-let-7c-5p、hsa-let-7d-5p、hsa-let-7e-5p、hsa-let-7f-5p、hsa-let-7f-2-3p、hsa-mir-15a-5p、hsa-mir-16-5p、hsa-mir-17-5p、hsa-mir-18a-5p、hsa-mir-19a-5p、hsa-mir-19b-1-5p、hsa-mir-19b-2-5p、hsa-mir-20a-5p、hsa-mir-21-5p、hsa-mir-22-5p、hsa-mir-23a-5p、hsa-mir-24-1-5p。
本例的mirna预测方法具体如下:
(1)设定种子序列长度为7bp;
(2)对a、t、c、g四种碱基进行设定序列长度的全排列,获得4的七次方条种子序列,形成种子序列集;
(3)去除种子序列集中反向互补的种子序列,即如果一条种子序列与另一条种子序列的反向互补序列相同,则只保留其中一条;
(4)在步骤(3)剩余的种子序列中,搜索每一条种子序列和其反向互补序列在模拟序列上的位置,得到种子序列位置集;
(5)将步骤(4)获得的种子序列位置集,在模拟序列上,按从小到大的顺序排列;
(6)按位置集的位置遍历,如果模拟序列上相邻的前后两个位置正好是互补序列,且他们的距离小于或等于500时,输出前面位置对应的种子序列前50bp至后面位置对应的种子序列后50bp的一段该段核酸序列,作为初选前体序列;
(7)采用二级结构预测软件对步骤(6)获得的初选前体序列的输出序列进行二级结构预测,如果不能形成发夹结构,则弃用该输出初选前体序列;如果能形成发夹结构,则采用二级结构预测软件对该初选前体序列的每个可能的二级结构进行打分,最终给出最可能的成熟体序列和对应的互补序列,同时给出对应的二级结构图;
(8)合并步骤(7)获得的所有成熟体序列及其对应的互补序列,以及各成熟体序列和其互补序列的二级结构,组成mirna集。
结果显示,本例的预测方法最终得到的mirna集中,预测存在23条mirna,本例待测的20条mirna都包含在其中。可见,本例的mirna预测方法能够有效的预测出所有存在的mirna。本例预测的23条mirna中,其中一条预测的mirna,序列seqidno.2所示序列,二级结构如图1所示,该预测mirna与hsa-let-7a-5p的折叠结构一致。
seqidno.2:
5’-tgggatgaggtagtaggttgtatagttttagggtcacacccaccactgggagataactatacaatctactgtctttccta-3’
需要说明的是,本申请的mirna预测方法能够预测出所有可能存在的mirna,即把所有可能都找出来,其中,当然会有假阳性的序列,即预测为mirna但实际上并非mirna,但这不影响后续的下游分析和研究。
另外,按照本申请的mirna预测方法虽然能够预测出待测的20条mirna,但是,对于部分成熟体的具体预测位置与mirna的真实位置存在差异;在能够准确预测出所存在的mirna的情况下,具体位置的差异可以根据后续试验进行调整,这并不影响后续的下游分析和研究。
以上内容是结合具体的实施方式对本申请所作的进一步详细说明,不能认定本申请的具体实施只局限于这些说明。对于本申请所属技术领域的普通技术人员来说,在不脱离本申请构思的前提下,还可以做出若干简单推演或替换。
sequencelisting
<110>深圳华大基因科技服务有限公司
<120>一种mirna预测方法、mirna系统及应用
<130>17i24440
<160>2
<170>patentinversion3.3
<210>1
<211>2566
<212>dna
<213>人工序列
<220>
<221>misc_feature
<222>(81)..(130)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(203)..(252)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(336)..(385)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(470)..(519)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(607)..(656)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(736)..(785)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(873)..(922)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1006)..(1055)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1139)..(1188)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1278)..(1327)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1412)..(1461)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1533)..(1582)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1665)..(1714)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1802)..(1851)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(1948)..(1997)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(2069)..(2118)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(2191)..(2240)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(2326)..(2375)
<223>nisa,c,g,ort
<220>
<221>misc_feature
<222>(2449)..(2498)
<223>nisa,c,g,ort
<400>1
tgggatgaggtagtaggttgtatagttttagggtcacacccaccactgggagataactat60
acaatctactgtctttcctannnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn120
nnnnnnnnnnaggttgaggtagtaggttgtatagtttagaattacatcaagggagataac180
tgtacagcctcctagctttcctnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn240
nnnnnnnnnnnncggggtgaggtagtaggttgtgtggtttcagggcagtgatgttgcccc300
tcggaagataactatacaacctactgccttccctgnnnnnnnnnnnnnnnnnnnnnnnnn360
nnnnnnnnnnnnnnnnnnnnnnnnngcatccgggttgaggtagtaggttgtatggtttag420
agttacaccctgggagttaactgtacaaccttctagctttccttggagcnnnnnnnnnnn480
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncctaggaagaggtagtaggtt540
gcatagttttagggcagggattttgcccacaaggaggtaactatacgacctgctgccttt600
cttaggnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncccg660
ggctgaggtaggaggttgtatagttgaggaggacacccaaggagatcactatacggcctc720
ctagctttccccaggnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn780
nnnnntcagagtgaggtagtagattgtatagttgtggggtagtgattttaccctgttcag840
gagataactatacaatctattgccttccctgannnnnnnnnnnnnnnnnnnnnnnnnnnn900
nnnnnnnnnnnnnnnnnnnnnntgtgggatgaggtagtagattgtatagttttagggtca960
taccccatcttggagataactatacagtctactgtctttcccacgnnnnnnnnnnnnnnn1020
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnccttggagtaaagtagcagcacata1080
atggtttgtggattttgaaaaggtgcaggccatattgtgctgcctcaaaaatacaaggnn1140
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnngtcagcagtgcc1200
ttagcagcacgtaaatattggcgttaagattctaaaattatctccagtattaactgtgct1260
gctgaagtaaggttgacnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn1320
nnnnnnngtcagaataatgtcaaagtgcttacagtgcaggtagtgatatgtgcatctact1380
gcagtgaaggcacttgtagcattatggtgacnnnnnnnnnnnnnnnnnnnnnnnnnnnnn1440
nnnnnnnnnnnnnnnnnnnnntgttctaaggtgcatctagtgcagatagtgaagtagatt1500
agcatctactgccctaagtgctccttctggcannnnnnnnnnnnnnnnnnnnnnnnnnnn1560
nnnnnnnnnnnnnnnnnnnnnngcagtcctctgttagttttgcatagttgcactacaaga1620
agaatgtagttgtgcaaatctatgcaaaactgatggtggcctgcnnnnnnnnnnnnnnnn1680
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnncactgttctatggttagttttgcagg1740
tttgcatccagctgtgtgatattctgctgtgcaaatccatgcaaaactgactgtggtagt1800
gnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnacattgcta1860
cttacaattagttttgcaggtttgcatttcagcgtatatatgtatatgtggctgtgcaaa1920
tccatgcaaaactgattgtgataatgtnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn1980
nnnnnnnnnnnnnnnnngtagcactaaagtgcttatagtgcaggtagtgtttagttatct2040
actgcattatgagcacttaaagtactgcnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn2100
nnnnnnnnnnnnnnnnnntgtcgggtagcttatcagactgatgttgactgttgaatctca2160
tggcaacaccagtcgatgggctgtctgacannnnnnnnnnnnnnnnnnnnnnnnnnnnnn2220
nnnnnnnnnnnnnnnnnnnnggctgagccgcagtagttcttcagtggcaagctttatgtc2280
ctgacccagctaaagctgccagttgaagaactgttgccctctgccnnnnnnnnnnnnnnn2340
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnggccggctggggttcctggggatgg2400
gatttgcttcctgtcacaaatcacattgccagggatttccaaccgaccnnnnnnnnnnnn2460
nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnctccggtgcctactgagctgat2520
atcagttctcattttacacactggctcagttcagcaggaacaggag2566
<210>2
<211>80
<212>dna
<213>人工预测mirna
<400>2
tgggatgaggtagtaggttgtatagttttagggtcacacccaccactgggagataactat60
acaatctactgtctttccta80
- 还没有人留言评论。精彩留言会获得点赞!