高丝氨酸乙酰基转移酶突变体及其宿主细胞和应用的制作方法
本发明涉及生物
技术领域:
。具体地说,本发明涉及高丝氨酸乙酰基转移酶的突变体及其应用。
背景技术:
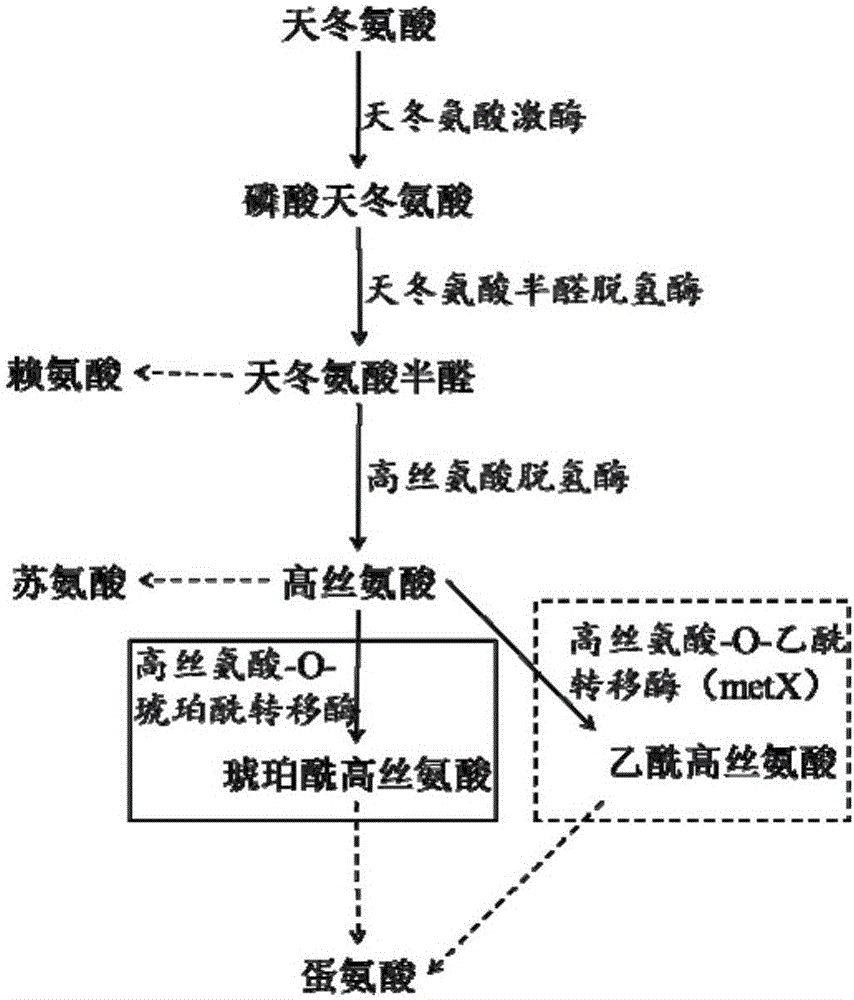
:o-乙酰高丝氨酸(o-acetyl-homoserine,oah)或o-琥珀酰高丝氨酸(o-succinyl-homoserine,osh)经水解可以产生高丝氨酸,而高丝氨酸可以转化为l-高丝氨酸内酯、γ-丁内酯、1,4-丁二醇等平台化合物(journalofindustrialmicrobiology&biotechnology,2014,41(10):1517-1524)。蛋氨酸是体内的必需氨基酸之一,广泛用于动物饲料、食品和医药等多种领域,年需求量达160万吨。蛋氨酸还能作为卵磷脂和肌酸合成的前体,并且也用作半胱氨酸和牛磺酸合成的原料。目前蛋氨酸作为饲料添加剂,在一些家畜如肉鸡、猪、肉牛、奶牛饲料方面已广泛使用。蛋氨酸衍生物-s-腺苷蛋氨酸(sam)作为甲基供体参与到代谢中间物多胺、生物素、类脂等的合成。sam可用于预防肝脏和动脉中的脂质积累,以及治疗抑郁症、炎症、肝脏疾病和肌肉痛。蛋氨酸也参与谷胱甘肽的代谢途径,谷胱甘肽是人体细胞内的主要抗氧化剂。人体细胞缺乏蛋氨酸会导致尿毒症、肌肉瘫痪等疾病。在医药制剂中,l-蛋氨酸被加入到治疗肝脏损伤的药物中用于肝脏疾病治疗。蛋氨酸缺乏会显著上调促炎性细胞因子和纤维化基因,蛋氨酸的存在会改善以上情况。如图1所示,在微生物体内,天冬氨酸经天冬氨酸激酶的作用生成天冬氨酸磷酸,然后经过天冬氨酸半醛脱氢酶和高丝氨酸脱氢酶的作用生产高丝氨酸,高丝氨酸是微生物蛋氨酸合成途径中的重要中间代谢物,在埃希氏菌(escherichia)属、沙门氏菌(salmonella)属和芽孢杆菌(bacillus)属的微生物中,高丝氨酸经过l-高丝氨酸-o-琥珀酰转移酶的作用转化为osh(biochemistry.1999oct26;38(43):14416-23.),在棒状杆菌(corynebacterium)属、钩端螺旋体(leptospira)属、奇异球菌(deinococcus)属、假单胞菌(pseudomonas)属和分枝杆菌(mycobacterium)属的微生物中,高丝氨酸经过l-高丝氨酸-o-乙酰转移酶(metx,ec2.3.1.31)的作用转化为oah(journalofbacteriology,mar.2002,p.1277-1286)。以半胱氨酸作为硫供体,osh或oah在γ-胱硫醚合酶(cystathionineγsynthase,cgs)和β-胱硫醚裂解酶(cystathionineβlyase,cbl)作用下生成高半胱氨酸,高半胱氨酸经过甲基化生成蛋氨酸。另外,由于osh或oah也可以在硫化氢解酶的作用下直接与甲硫醚反应生成蛋氨酸(journalofmicrobiologyandbiotechnology,20(8):1196-1203),oah也可用作生产蛋氨酸的前体,希杰公司开发了利用微生物生产oah,然后经过一步酶催化生产蛋氨酸的方法(cn103397057b)。在微生物体内,metx是合成蛋氨酸分支途径的第一个酶,其底物高丝氨酸同时也是苏氨酸合成途径的中间代谢物。据文献报道,大部分不同来源的metx受到蛋氨酸和sam的反馈抑制,10mmol/l蛋氨酸存在情况下,谷氨酸棒杆菌来源的metx仅保留25%的相对酶活(journalofmicrobiologyandbiotechnology,2004,14(2):373-378)。metx是oah和蛋氨酸合成的限速步骤之一。韩国希杰公司公布了一种将高丝氨酸o-琥珀酰转移酶突变为metx酶活性的方法并将其应用在oah生产中(cn103797027a),另外,希杰公司还将不同来源的metx过表达来生产oah(cn101629160b)。鉴于metx在oah和蛋氨酸合成中的重要作用,以及现有技术中的metx在活性和解除反馈抑制方面仍不尽人意,本领域急需高活性并且耐受反馈抑制的高丝氨酸乙酰基转移酶。技术实现要素:本发明的目的在于提供一种活性高并且耐受反馈抑制的高丝氨酸乙酰基转移酶。在第一方面,本发明提供一种高丝氨酸乙酰基转移酶,所述高丝氨酸乙酰基转移酶的氨基酸序列在对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基为非脯氨酸,和/或第254位的氨基酸残基为非异亮氨酸。在优选的实施方式中,所述高丝氨酸乙酰基转移酶的氨基酸序列在对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基为非脯氨酸,并且第254位的氨基酸残基为非异亮氨酸。在优选的实施方式中,所述高丝氨酸乙酰基转移酶的氨基酸序列与seqidno:3所示氨基酸序列具有90%以上,优选95%以上,更优选96%、97%、98%、99%的同源性。在优选的实施方式中,所述高丝氨酸乙酰基转移酶来源于迈氏钩端螺旋体。在优选的实施方式中,所述高丝氨酸乙酰基转移酶:a.具有如seqidno:3所示氨基酸序列,且第250位的氨基酸残基为非脯氨酸,和/或第254位的氨基酸残基为非异亮氨酸;b.具有a)所限定的序列经过一个或几个氨基酸残基,优选1-20个、更优选1-15个、更优选1-10个、更优选1-3个、最优选1个氨基酸残基的取代、缺失或添加而形成的序列,且基本具有a)所限定的高丝氨酸乙酰基转移酶功能的由a)衍生的高丝氨酸乙酰基转移酶;或c.具有a)所限定的序列,经过在a)所限定的序列的任一端的一个或几个氨基酸残基,优选1-20个、更优选1-15个、更优选1-10个、更优选1-3个、最优选1个氨基酸残基的缺失或添加,优选添加而形成的序列,且基本具有a)所限定的高丝氨酸乙酰基转移酶功能的由a)衍生的高丝氨酸乙酰基转移酶。在优选的实施方式中,所述高丝氨酸乙酰基转移酶在对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基选自以下氨基酸的一种:ser、thr、lys、arg、gln、asn和glu,优选ser或lys;在对应于seqidno:3所示氨基酸序列的第254位的氨基酸残基选自以下氨基酸的一种:asn、gln、his、arg、ala、val、leu、asp、glu、lys,优选asn或ala或asp或lys。在优选的实施方式中,所述高丝氨酸乙酰基转移酶:a.具有seqidno:3所示的氨基酸序列,并且在第250位的氨基酸残基选自以下氨基酸的一种:ser、thr、lys、arg、gln、asn和glu,优选ser或lys;和/或在第254位的氨基酸残基选自以下氨基酸的一种:asn、gln、his、arg、ala、val、leu、asp、glu、lys,优选asn或ala或asp或lys;或b.包含(a)所限定的序列经过一个或几个氨基酸残基,优选1-20个、更优选1-15个、更优选1-10个、更优选1-3个、最优选1个氨基酸残基的取代、缺失或添加而形成的序列,且基本具有a所限定的高丝氨酸乙酰基转移酶功能的由a衍生的高丝氨酸乙酰基转移酶;或c.具有a)所限定的序列,经过在a)所限定的序列的任一端的一个或几个氨基酸残基,优选1-20个、更优选1-15个、更优选1-10个、更优选1-3个、最优选1个氨基酸残基的缺失或添加,优选添加而形成的序列,且基本具有a)所限定的高丝氨酸乙酰基转移酶功能的由a)衍生的高丝氨酸乙酰基转移酶。在优选的实施方式中,所述高丝氨酸乙酰基转移酶的氨基酸序列如seqidno:5、7、27、28、29、30或31所示。在优选的实施方式中,所述高丝氨酸乙酰基转移酶耐受蛋氨酸或sam的反馈抑制。在优选的实施方式中,在10mm/l浓度的蛋氨酸存在下,所述的高丝氨酸乙酰基转移酶至少保留80%以上的相对酶活;或者在10mm/l浓度的sam存在下,所述的高丝氨酸乙酰基转移酶至少保留60%以上的相对酶活。在第二方面,本发明提供编码第一方面所述的高丝氨酸乙酰基转移酶的基因。在第三方面,本发明提供包含第二方面所述编码基因的表达载体。在第四方面,本发明提供一种宿主细胞,所述宿主细胞含有第一方面所述的高丝氨酸乙酰基转移酶。在优选的实施方式中,所述宿主细胞包含第三方面所述的表达载体或在其基因组中整合有第二方面所述的编码基因。在优选的实施方式中,所述宿主细胞来自埃希氏菌属(escherichia)、棒状杆菌属(corynebacterium)、短杆菌属(brevibacteriumsp.)、芽孢杆菌属(bacillus)、沙雷氏菌属(serratia)或弧菌属(vibrio)。在进一步优选的实施方式中,所述宿主细胞是大肠杆菌(e.coli)或谷氨酸棒状杆菌(corynebacteriumglutamicum)。在第五方面,本发明提供第一方面所述的高丝氨酸乙酰基转移酶、或第二方面所述的编码基因、或第三方面所述的表达载体、或第四方面所述的宿主细胞在生产o-乙酰高丝氨酸、蛋氨酸或sam中的应用。在第六方面,本发明提供一种制备o-乙酰高丝氨酸、蛋氨酸或sam的方法,所述方法包括以下步骤:a.培养第四方面所述的宿主细胞,使之产生o-乙酰高丝氨酸、蛋氨酸或sam;和b.任选从培养液中分离o-乙酰高丝氨酸、蛋氨酸或sam。在优选的实施方式中,所述方法在28℃至40℃,更优选在30℃或37℃实施。在第七方面,本发明提供一种制备o-乙酰高丝氨酸、蛋氨酸或sam的方法,所述方法包括以下步骤:a.利用第一方面所述的高丝氨酸乙酰基转移酶,催化从高丝氨酸生成o-乙酰高丝氨酸、蛋氨酸或sam;和b.任选从以上反应体系中分离o-乙酰高丝氨酸、蛋氨酸或sam。在第八方面,本发明提供第一方面所述高丝氨酸乙酰基转移酶的制备方法,所述方法包括以下步骤:a.改造seqidno:3所示氨基酸序列的编码序列,使得编码的氨基酸序列中对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基为非脯氨酸,和/或第254位的氨基酸残基为非异亮氨酸;b.将a得到的编码序列直接转染合适的宿主细胞或经载体引入合适的宿主细胞;c.培养b得到的宿主细胞;d.从步骤c得到的培养体系中分离所述宿主细胞产生的高丝氨酸乙酰基转移酶。在优选的实施方式中,所述对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基选自以下氨基酸的一种:ser、thr、lys、arg、gln、asn和glu,优选ser或lys;对应于seqidno:3所示氨基酸序列的第254位的氨基酸残基选自以下氨基酸的一种:asn、gln、his、arg、ala、val、leu、asp、glu、lys,优选asn或ala或asp或lys。在优选的实施方式中,所述方法还包括测定得到的高丝氨酸乙酰基转移酶的活性以及解除蛋氨酸或sam反馈抑制的能力。在第九方面,本发明提供改造野生型高丝氨酸乙酰基转移酶以便提高其活性并解除蛋氨酸或sam反馈抑制的方法,所述方法包括以下步骤:a.将野生型高丝氨酸乙酰基转移酶的氨基酸序列与seqidno:3所示氨基酸序列作比对;和b.改造所述野生型高丝氨酸乙酰基转移酶的编码序列,使得编码的氨基酸序列中对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基为非脯氨酸,和/或第254位的氨基酸残基为非异亮氨酸;c.将b得到的编码序列直接转染合适的宿主细胞或经载体引入合适的宿主细胞;d.培养c得到的宿主细胞;e.从步骤d得到的培养体系中分离所述宿主细胞产生的高丝氨酸乙酰基转移酶。在优选的实施方式中,所述对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基选自以下氨基酸的一种:ser、thr、lys、arg、gln、asn和glu,优选ser或lys;对应于seqidno:3所示氨基酸序列的第254位的氨基酸残基选自以下氨基酸的一种:asn、gln、his、arg、ala、val、leu、asp、glu、lys,优选asn或ala或asp或lys。在优选的实施方式中,所述方法还包括测定所述高丝氨酸乙酰基转移酶解除蛋氨酸或sam反馈抑制的能力。应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。附图说明图1显示了微生物体内的蛋氨酸代谢途径;其中,实线箭头代表一步反应,虚线箭头代表多步反应,实线框代表经过osh的微生物蛋氨酸代谢途径,代表微生物如埃希氏菌属、沙门氏菌属和芽孢杆菌属;虚线框代表经过oah的微生物蛋氨酸代谢途径,代表微生物如棒状杆菌属、钩端螺旋体属、奇异球菌属、假单胞菌属和分枝杆菌属。图2显示质粒pet-lmetx示意图。图3显示不同浓度蛋氨酸和sam对lmetx及其突变体的酶活抑制;其中a显示蛋氨酸的抑制作用;b显示sam的抑制作用。图4显示通过lmetx点突变所获不同突变体及其相对酶活性;其中横坐标中的横线下方代表突变发生的位点,横线上方代表突变后的氨基酸,none代表没有发生突变,相当于野生型lmetx。具体实施方式发明人经过广泛而深入的研究,出乎意料地发现对迈氏钩端螺旋菌来源的高丝氨酸乙酰基转移酶的第250位和/或254位置进行遗传改造,获得的高丝氨酸乙酰基转移酶突变体不仅显著提高了酶活性,其对蛋氨酸和sam反馈抑制的耐受性也显著提高,从而能够用于高效地生产o-乙酰高丝氨酸、蛋氨酸、sam等产物。在此基础上完成了本发明。本发明的高丝氨酸乙酰基转移酶本文所用的术语“本发明的高丝氨酸乙酰基转移酶”和“本发明的多肽”可互换使用,并具有本领域普通技术人员通常理解的含义。本发明的高丝氨酸乙酰基转移酶具有催化高丝氨酸产生o-乙酰高丝氨酸的活性。本发明的高丝氨酸乙酰基转移酶是通过对seqidno:3所示氨基酸序列进行突变得到,得到的突变体不仅具备优异的活性,还能显著解除蛋氨酸或sam的反馈抑制。具体地说,本发明的高丝氨酸乙酰基转移酶的氨基酸序列在对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基突变为非脯氨酸,和/或在第254位的氨基酸残基突变为非异亮氨酸。本领域技术人员知晓,对于野生型多肽的突变,找到能实现所需目的的位点更为重要。因此,基于本发明的教导,本领域技术人员会对seqidno:3所示氨基酸序列的第250位和/或254位的氨基酸残基或某氨基酸序列中对应于seqidno:3所示氨基酸序列的第250位和/或254位的氨基酸残基进行突变,并检测突变体的相关活性。在具体的实施方式中,本发明的高丝氨酸乙酰基转移酶在对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基可以突变为(但不限于):ser、thr、lys、arg、gln、asn和glu,优选ser或lys;在对应于seqidno:3所示氨基酸序列的第254位的氨基酸残基可以突变为(但不限于):asn、gln、his、arg、ala、val、leu、asp、glu、lys,优选asn或ala或asp或lys。此外,本领域普通技术人员也不难知晓,在多肽的某些区域,例如非重要区域改变少数氨基酸残基基本上不会改变生物活性,例如,适当替换某些氨基酸得到的序列并不会影响其活性(可参见watson等,molecularbiologyofthegene,第四版,1987,thebenjamin/cummingspub.co.p224)。因此,本领域普通技术人员能够实施这种替换并且确保所得分子仍具有所需生物活性。因此,对本发明的高丝氨酸乙酰基转移酶作进一步突变而得到仍具备高丝氨酸乙酰基转移酶的功能和活性的进一步突变体是显而易见的。例如,本领域技术人员公知在多肽的任一端增加或减少数个氨基酸残基,例如优选1-20个、更优选1-15个、更优选1-10个、更优选1-3个、最优选1个氨基酸残基不会影响得到的突变体的功能。例如,为便于纯化,技术人员往往在得到的蛋白的任一端带上6×his标签,而这种蛋白与不具备6×his标签的蛋白具有相同的功能。因此,本发明应包括本发明的高丝氨酸乙酰基转移酶的保守性突变体。这些保守性突变体可以根据,例如下表所示进行氨基酸替换而产生。本发明还提供了编码本发明多肽的多核苷酸。术语“编码多肽的多核苷酸”可以是包括编码此多肽的多核苷酸,也可以是还包括附加编码和/或非编码序列的多核苷酸。因此,本文所用的“含有”,“具有”或“包括”包括了“包含”、“主要由……构成”、“基本上由……构成”、和“由……构成”;“主要由……构成”、“基本上由……构成”和“由……构成”属于“含有”、“具有”或“包括”的下位概念。“对应于”本文所用的术语“对应于”具有本领域普通技术人员通常理解的意义。具体地说,“对应于”表示两条序列经同源性或序列相同性比对后,一条序列与另一条序列中的指定位置相对应的位置。因此,就“对应于seqidno:3所示氨基酸序列的第250位的氨基酸残基”而言,如果在seqidno:3所示氨基酸序列的一端加上6×his标签,那么所得突变体中对应于seqidno:3所示氨基酸序列的第250位就可能是第256位。在具体的实施方式中,所述同源性或序列相同性可以是90%以上,优选95%以上,更优选96%、97%、98%、99%的同源性。本领域普通技术人员公知的测定序列同源性或相同性的方法包括但不限于:计算机分子生物学(computationalmolecularbiology),lesk,a.m.编,牛津大学出版社,纽约,1988;生物计算:信息学和基因组项目(biocomputing:informaticsandgenomeprojects),smith,d.w.编,学术出版社,纽约,1993;序列数据的计算机分析(computeranalysisofsequencedata),第一部分,griffin,a.m.和griffin,h.g.编,humanapress,新泽西,1994;分子生物学中的序列分析(sequenceanalysisinmolecularbiology),vonheinje,g.,学术出版社,1987和序列分析引物(sequenceanalysisprimer),gribskov,m.与devereux,j.编mstocktonpress,纽约,1991和carillo,h.与lipman,d.,siamj.appliedmath.,48:1073(1988)。测定相同性的优选方法要在测试的序列之间得到最大的匹配。测定相同性的方法编译在公众可获得的计算机程序中。优选的测定两条序列之间相同性的计算机程序方法包括但不限于:gcg程序包(devereux,j.等,1984)、blastp、blastn和fasta(altschul,s,f.等,1990)。公众可从ncbi和其它来源得到blastx程序(blast手册,altschul,s.等,ncbinlmnihbethesda,md.20894;altschul,s.等,1990)。熟知的smithwaterman算法也可用于测定相同性。宿主细胞本文所用的术语“宿主细胞”具有本领域普通技术人员通常理解的含义,即,能够产生本发明高丝氨酸乙酰基转移酶的宿主细胞。换言之,本发明可以利用任何宿主细胞,只要本发明的高丝氨酸乙酰基转移酶能在该宿主细胞中表达。例如,适用于本发明的宿主细胞来自(但不限于)埃希氏菌属(escherichia)、棒状杆菌属(corynebacterium)、短杆菌属(brevibacteriumsp.)、芽孢杆菌属(bacillus)、沙雷氏菌属(serratia)或弧菌属(vibrio)。在进一步优选的实施方式中,所述宿主细胞是大肠杆菌(e.coli)或谷氨酸棒状杆菌(corynebacteriumglutamicum)。解除蛋氨酸或sam反馈抑制本领域技术人员应理解,本文所用的术语“解除蛋氨酸或sam反馈抑制”是指一种原本受到蛋氨酸或sam反馈抑制的酶,在经过改造后令其受蛋氨酸或sam抑制程度降低。这种降低是通过两种酶在相同蛋氨酸或sam浓度下的抑制程度比较获得的。“解除蛋氨酸或sam反馈抑制”包含了反馈抑制的部分解除到全部解除。而抑制程度是指在一定浓度的蛋氨酸或sam存在下,与不存在蛋氨酸或sam时相比,高丝氨酸乙酰基转移酶活性损失(即受到抑制)的比例。在这种条件下,高丝氨酸乙酰基转移酶活性保留下来的比例,称为酶活残存比例或酶活保留比例或相对酶活,由于:酶活损失比例+酶活残存比例=100%,所以经常用酶活残存比例表示抑制程度。酶活残存比例越高,抑制程度越低。相应的,“解除蛋氨酸或sam反馈抑制”也通常用改造前后两个酶残存酶活比例的比较来刻画。在具体的实施方式中,在10mm/l浓度的蛋氨酸存在下,所述的高丝氨酸乙酰基转移酶至少保留80%以上的相对酶活;或者,在10mm/l浓度的sam存在下,所述的高丝氨酸乙酰基转移酶至少保留60%以上的相对酶活。固定化酶本文所用的术语“固定化酶”具有本领域普通技术人员常规理解的含义。具体地说,该术语表示水溶性酶经物理或化学方法处理后,使酶与水不溶性大分子载体结合或把酶包埋在其中,使得酶在水中形成溶性凝胶或半透膜的微囊体从而导致流动性降低。固定化的酶仍具有酶活性,在催化反应中以固相状态作用于底物。酶经固定化后一般稳定性增加,易从反应系统中分离,且易于控制,能反复多次使用。便于运输和贮存,有利于自动化生产。固定化酶是近十余年发展起来的酶应用技术,在工业生产、化学分析和医药等方面有诱人的应用前景。本领域普通技术人员鉴于本文的教导,不难将本发明的高丝氨酸乙酰基转移酶处理成固定化酶,从而用于催化从高丝氨酸产生o-乙酰高丝氨酸,进而高效地生产蛋氨酸、sam等产物。本发明的应用与优点1.本发明的高丝氨酸乙酰基转移酶可以在工业上应用以产生o-乙酰高丝氨酸以及蛋氨酸、sam等产物;2.本发明的高丝氨酸乙酰基转移酶不仅具备高活性,更能有效解除蛋氨酸和sam反馈抑制,在工业上的应用前景广阔;3.本发明提供了一种待改造高丝氨酸乙酰基转移酶的点突变位点,该突变不在酶的活性中心,该位点的突变可以有效提高待改造酶的高丝氨酸乙酰基转移酶酶活,解除蛋氨酸和sam反馈抑制。下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。实施例实施例1.lmetx及其突变体表达载体和菌株的构建根据迈氏钩端螺旋菌lmetx的基因序列(seqidno:1)对基因进行全合成,根据大肠杆菌的密码子偏好性对其进行密码子优化,优化后合成的基因序列如seqidno:2所示(基因合成由中国苏州金唯智生物科技有限公司完成)。lmetx蛋白序列如seqidno:3所示。本发明人在利用优化后的lmetx基因(seqidno:2)进行进化的过程中,获得两个突变体,第一个突变体lmetx-t1的基因序列如seqidno:4所示,其蛋白质序列如seqidno:5所示,seqidno:5与seqidno:3相比,第250位脯氨酸突变为丝氨酸(p250s),第二个突变体lmetx-t2的基因序列如seqidno:6所示,其蛋白质序列如seqidno:7所示,seqidno:7与seqidno:3相比,第254位异亮氨酸突变为天冬酰胺(i254n)。以大肠杆菌常用的表达载体pet28a为模板,利用表1中所示引物pet-f1和pet-r1扩增质粒骨架;以序列seqidno:2、seqidno:4、seqidno:6所示基因为模板,利用表1中所示引物lmetx-f1和lmetx-r1分别扩增lmetx、lmetx-t1、lmetx-t1基因,扩增后的质粒骨架和lmetx及其突变体基因片段之间带有同源片段。因此,利用一步法无缝克隆试剂盒(clonexpresstmiionestepcloningkit,vazymebiotechco.,ltd)将lmetx及其突变体基因片段分别连接到pet28a载体上,获得质粒pet-lmetx(如图2所示),pet-lmetx-t1,以及pet-lmetx-t2。将质粒分别转化到大肠杆菌bl21(de3)中,获得lmetx及其突变体表达菌株bl21(pet-lmetx),bl21(pet-lmetx-t1)以及bl21(pet-lmetx-t2),利用此菌株表达的lmetx蛋白n端带有组氨酸标签,可以方便地使用镍柱进行纯化。表1.lmetx及其突变体表达载体构建所用引物引物序列pet-f1tgactggtggacagcaaatgggtcgcggatc(seqidno:8)pet-r1cggcaccaggccgctgctgtgatgatgatg(seqidno:9)lmetx-f1agcggcctggtgccgatgcctacctctgagcagaac(seqidno:10)lmetx-r1gctgtccaccagtcattacagaaaaacgccttcgtcgg(seqidno:11)实施例2.lmetx及其突变体酶活表征1.酶活测定菌株bl21(pet-lmetx),bl21(pet-lmetx-t1)以及bl21(pet-lmetx-t2)分别利用lb培养基(胰蛋白胨10g/l,酵母提取物5g/l,nacl10g/l)培养,加入终浓度为50mg/l的卡那霉素,37摄氏度培养10h后,以1%的接种量转接到新鲜的lb加卡那霉素培养基,37摄氏度培养2h后,加入终浓度0.5mm的iptg,继续20摄氏度培养15小时。使用ph为7.5,浓度为100mm的磷酸盐缓冲液洗涤菌体一次,并重悬,进行超声波破碎,破碎液在7500rpm离心5min,取上清获得粗蛋白液,蛋白纯化采用hisspintrapcolumns(购自ge公司,产品货号28-4013-53),具体方法参照说明书。利用thermofisher公司的bca蛋白定量试剂盒进行蛋白定量。利用5,5'-二硫代双(2-硝基苯甲酸)(dtnb显色剂)显色法进行酶活性测定,测定方法为:将不同浓度的高丝氨酸、乙酰-coa,以及酶液加入到100mm的磷酸盐缓冲液中,终体积为150ul,在30摄氏度、ph为7.5条件下反应不同时间后进行检测,具体可参考以往文献(journalofbacteriology1987.169(8):3458-3463.)。酶活单位u定义为:在标准酶反应条件下,每分钟催化产生1mmol的coash所需的酶量定义为一个酶活单位。lmetx及其突变体对底物高丝氨酸的km值,反应的最大速度vmax,以及kcat/km值如表2所示,可以看出,突变体lmetx-t1(p250s)和lmetx-t2(i254n)与野生酶相比,最大酶活(vmax)分别提高了大于43倍和13倍,kcat/km分别提高了大于37倍和16倍。表2.lmetx及其突变体的酶动力学参数2.抑制物对酶活的影响利用实施例2所述酶活性测定方法检测蛋氨酸和sam对lmetx及其突变体的抑制作用,其中高丝氨酸和乙酰-coa的终浓度分别为5mmol/l和1mmol/l,反应体系中加入不同浓度的蛋氨酸或sam。结果如图3所示,在低浓度的蛋氨酸或sam存在情况下,酶的活性下降显著,但是野生型酶的酶活降低幅度更大,随着蛋氨酸或sam浓度升高,酶活性趋于稳定,但是总体上,突变体受抑制程度低。因此,突变体在一定程度上解除了蛋氨酸和sam对酶活的抑制作用,特别是在低浓度情况下,抑制解除效果更明显。实施例3.lmetx及其突变体对菌株产oah的影响以带有大肠杆菌来源的thra酶基因以及谷氨酸棒杆菌来源的metx酶基因的质粒psym-4(改自ptrc99a:genbankaccessionno.u13872,详细信息见参考文献:biotechnologybulletin2017,33(9):1-6;其中,thra和metx酶基因的启动子序列都是j23110:tttacggctagctcagtcctaggtacaatgctagc(seqidno:12),启动子具体信息可参考网站http://parts.igem.org/part:bba_j23110)为模板,利用表3中的引物ptrc-f1和ptrc-r1进行pcr扩增,同时,分别以seqidno:2、seqidno:4、seqidno:6对应的lmetx及其突变体基因为模板,利用引物lmetx-f2和lmetx-r2进行pcr扩增,获得lmetx及其突变体基因。利用一步无缝克隆试剂盒将上述不同引物扩增片段进行同源重组,获得带有thra和lmetx基因的质粒ptrcax,带有thra和lmetx-t1基因的质粒ptrcax-t1,以及带有thra和lmetx-t2基因的质粒ptrcax-t2。将ptrcax、ptrcax-t1、ptrcax-t2分别通过电转化导入大肠杆菌mg1655中,获得菌株mg1655(ptrcax),mg1655(ptrcax-t1)以及mg1655(ptrcax-t2)。表3.含有thra和lmetx或其突变体基因的质粒构建引物引物序列ptrc-f1gttctgctcagaggtaggcatatgtatattctccttcttaa(seqidno:13)ptrc-r1gacgaaggcgtttttctgtaaccgctgcaggcatgcaagctt(seqidno:14)lmetx-f2ttaagaaggagaatatacatatgcctacctctgagcagaac(seqidno:15)lmetx-r2aagcttgcatgcctgcagcggttacagaaaaacgccttcgtc(seqidno:16)将菌株mg1655(ptrcax),mg1655(ptrcax-t1)以及mg1655(ptrcax-t2)分别利用lb培养基过夜培养后,按照1%的接种量转接到含有20ml发酵培养基(葡萄糖30g/l;酵母粉6g/l,(nh4)2so410g/l;kh2po42g/l;3-(n-吗啡啉)丙磺酸80g/l;mgso4·7h2o0.5g/l;feso4·5h2o0.1g/l,mnso4·5h2o0.1g/l)的500ml锥形瓶中,在震荡摇床中37℃,220r/min培养40h后取出,利用液相色谱仪测定发酵液中oah含量,结果如表4所示。可以看出,lmetx突变体lmetx-t1(p250s)和lmetx-t2(i254n)能够显著提高菌株产量。表4.过表达lmetx及其突变体对菌株生产oah的影响菌株mg1655(ptrcax)mg1655(ptrcax-t1)mg1655(ptrcax-t2)oah产量(g/l)0.01±0.000.29±0.020.18±0.01实施例4.lmetx及其突变体对菌株产蛋氨酸的影响分别以质粒ptrcax、ptrcax-t1、ptrcax-t2为模板,以表5中的ptrc-f2和ptrc-r2序列为引物进行pcr扩增,获得带有ptrc99a载体片段、thra基因、以及lmetx或其突变体p250s或i254n基因的片段。以表5中的mety-f1和mety-r1为引物,谷氨酸棒杆菌13032的基因组为模版,扩增mety蛋白基因。利用一步无缝克隆试剂盒,将mety蛋白基因分别与上述ptrc-f2和ptrc-r2引物扩增的片段相连,分别获得质粒ptrcayx、ptrcayx-t1、ptrcayx-t2,将质粒分别通过电转化导入大肠杆菌mg1655中,获得菌株mg1655(ptrcayx),mg1655(ptrcayx-t1)以及mg1655(ptrcayx-t2)。分别利用实施例3中的方法,对mg1655(ptrcayx),mg1655(ptrcayx-t1)以及mg1655(ptrcayx-t2)进行发酵培养,发酵结束后,利用hplc检测发酵液中蛋氨酸的含量,结果如表6所示。表5.含有thra、lmetx或其突变体、及其mety基因的质粒构建引物表6.过表达lmetx及其突变体对菌株生产蛋氨酸的影响菌株mg1655(ptrcayx)mg1655(ptrcayx-t1)mg1655(ptrcayx-t2)蛋氨酸产量(mg/l)12±370±1453±9实施例5.lmetx基因的定点突变及其酶活测定利用质粒pet-lmetx为模板,利用表7中的引物pet-r2为反向引物,分别利用表7中的引物pp250s/i254n、pp250k、pi254d、pi254k、pi254a为上游引物,利用pet-r2为下游引物进行pcr扩增,扩增产物分别利用一步无缝克隆试剂盒进行自我连接,获得连接到质粒pet28a上的不同lmetx突变体基因,突变体名称与对应的蛋白序列号如表7所示。利用实施例2中的酶活测定方法对突变体的vmax进行测定,结果如图4所示。与lmetx相比,突变体p250k(seqidno:27)、i254a(seqidno:28)、i254d(seqidno:29)、i254k(seqidno:30),以及双突变体p250s/i254n(seqidno:31)酶活都有不同程度提高,其中双突变体p250s/i254n提高大于88倍。证明250和254位点突变及其双突变对于提高lmetx的酶活具有显著作用。表7.lmetx定点突变引物及其突变体蛋白序列本发明提供的对应于seqidno:3的250或254位点是seqidno:3的非活性中心,而其可以显著提高酶活并解除蛋氨酸、sam的反馈抑制,这是本领域技术人员所不能预见的。但在本发明公开之后,本领域的技术人员可以从本发明中得到启示,即在对应于seqidno:3的250或254位点,和/或其临近位点进行突变,有可能得到酶活提高且解除蛋氨酸、sam反馈抑制的高丝氨酸乙酰基转移酶。在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。序列表<110>中国科学院天津工业生物技术研究所<120>高丝氨酸乙酰基转移酶突变体及其宿主细胞和应用<130>p2018-0161<160>31<170>patentinversion3.5<210>1<211>1140<212>dna<213>迈氏钩端螺旋菌(leptosiraobovata)<400>1atgcctacctccgaacagaacgagttttcccacggatccgtaggtgtcgtatatactcag60agcattcgatttgagtctttgactctagaggggggtgaaaccatcactcctcttgaaatt120gcctacgaaacgtatggcactctcaatgaaaaaaaagacaatgccattctagtttgccat180gcgctttcgggagatgctcatgcagcaggtttccatgaaggagacaaacgtcctggctgg240tgggattattatattggaccgggcaaatcctttgataccaatcgttactttatcatttct300tccaacgtaattggtggttgtaagggttccagtggaccacttaccatcaatgggaaaaat360ggaaaaccattccaatccacttttccctttgtctccataggagatatggtgaatgctcaa420gaaaaattaatcagccattttggaattcataaactatttgctgttgccggtggttcgatg480ggtggaatgcaagccttacaatggtcagtcgcatacccagatcggctcaaaaattgtatc540gtgatggcatcttcttccgaacattctgcacaacaaattgcctttaatgaagtgggaaga600caagccattctttctgatcccaattggaaccaaggtttgtacacccaggaaaacagaccg660tcaaagggacttgctcttgctcgaatgatgggtcatatcacttacttaagcgatgaaatg720atgagagaaaaatttggtcgtaaaccacccaaaggaaatatccaatccacagactttgcg780gtaggaagttatctaatctaccaaggcgaatcctttgtcgatcggtttgatgcaaactca840tatatttatgttacaaaagcattggatcattttagtttaggtacaggaaaagaacttaca900aaggtattggcaaaagtgagatgccggtttttggtagtggcttatacttccgattggttg960tatccaccgtatcaatctgaagaaattgtgaaatctttggaagtgaatgctgttcccgtt1020agttttgtagaactcaacaatccagcaggacgacatgatagttttttgttaccaagtgag1080caacaagactcgatcctaagagattttttaagttctacggacgaaggagttttcctttaa1140<210>2<211>1140<212>dna<213>人工序列(artificialsequence)<400>2atgcctacctctgagcagaacgaattctcccacggttctgtcggtgtcgtatacacgcaa60tctatccgtttcgaatccctgacgctggaaggtggcgaaactatcacccctctggaaatc120gcatacgagacgtacggtaccctgaacgagaagaaggataacgcaatcctggtctgccac180gcactgtctggcgatgctcatgctgcaggttttcatgaaggtgacaagcgtccaggttgg240tgggattattacatcggtccaggtaaatccttcgacaccaaccgttactttatcatctcc300tccaacgtgattggcggttgcaaaggttcctccggcccgctgactatcaacggtaaaaac360ggcaaaccgttccagagcaccttcccattcgtgtccatcggcgacatggtgaacgctcaa420gagaaactgatcagccacttcggtatccacaaactgttcgcagtggcaggtggtagcatg480ggtggtatgcaggctctgcaatggtctgtagcttacccggatcgtctgaagaactgtatt540gtgatggcgtctagctctgaacacagcgctcagcagattgctttcaatgaagtaggccgt600caggcgatcctgtctgacccgaactggaaccagggtctgtatacccaggagaatcgtccg660tctaaaggtctggccctggcccgtatgatgggccatattacttatctgtctgatgaaatg720atgcgcgagaaattcggccgtaaaccgccgaaaggcaacattcagtccactgacttcgcg780gtaggctcttacctgatctaccagggcgaatctttcgtggaccgttttgacgccaactct840tacatctacgttaccaaagcgctggaccacttctccctgggcactggcaaagaactgacc900aaagtactggcgaaagttcgctgtcgcttcctggttgttgcgtatacctccgactggctg960tatccgccgtatcagagcgaagaaattgttaaatccctggaagttaacgccgttccggtt1020agcttcgttgaactgaacaatccggcgggccgccacgatagctttctgctgccgagcgaa1080cagcaggatagcattctgcgcgattttctgtctagcaccgacgaaggcgtttttctgtaa1140<210>3<211>379<212>prt<213>人工序列(artificialsequence)<400>3metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproprolysglyasnileglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>4<211>1140<212>dna<213>人工序列(artificialsequence)<400>4atgcctacctctgagcagaacgaattctcccacggttctgtcggtgtcgtatacacgcaa60tctatccgtttcgaatccctgacgctggaaggtggcgaaactatcacccctctggaaatc120gcatacgagacgtacggtaccctgaacgagaagaaggataacgcaatcctggtctgccac180gcactgtctggcgatgctcatgctgcaggttttcatgaaggtgacaagcgtccaggttgg240tgggattattacatcggtccaggtaaatccttcgacaccaaccgttactttatcatctcc300tccaacgtgattggcggttgcaaaggttcctccggcccgctgactatcaacggtaaaaac360ggcaaaccgttccagagcaccttcccattcgtgtccatcggcgacatggtgaacgctcaa420gagaaactgatcagccacttcggtatccacaaactgttcgcagtggcaggtggtagcatg480ggtggtatgcaggctctgcaatggtctgtagcttacccggatcgtctgaagaactgtatt540gtgatggcgtctagctctgaacacagcgctcagcagattgctttcaatgaagtaggccgt600caggcgatcctgtctgacccgaactggaaccagggtctgtatacccaggagaatcgtccg660tctaaaggtctggccctggcccgtatgatgggccatattacttatctgtctgatgaaatg720atgcgcgagaaattcggccgtaaaccgtcgaaaggcaacattcagtccactgacttcgcg780gtaggctcttacctgatctaccagggcgaatctttcgtggaccgttttgacgccaactct840tacatctacgttaccaaagcgctggaccacttctccctgggcactggcaaagaactgacc900aaagtactggcgaaagttcgctgtcgcttcctggttgttgcgtatacctccgactggctg960tatccgccgtatcagagcgaagaaattgttaaatccctggaagttaacgccgttccggtt1020agcttcgttgaactgaacaatccggcgggccgccacgatagctttctgctgccgagcgaa1080cagcaggatagcattctgcgcgattttctgtctagcaccgacgaaggcgtttttctgtaa1140<210>5<211>379<212>prt<213>人工序列(artificialsequence)<400>5metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproserlysglyasnileglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>6<211>1140<212>dna<213>人工序列(artificialsequence)<400>6atgcctacctctgagcagaacgaattctcccacggttctgtcggtgtcgtatacacgcaa60tctatccgtttcgaatccctgacgctggaaggtggcgaaactatcacccctctggaaatc120gcatacgagacgtacggtaccctgaacgagaagaaggataacgcaatcctggtctgccac180gcactgtctggcgatgctcatgctgcaggttttcatgaaggtgacaagcgtccaggttgg240tgggattattacatcggtccaggtaaatccttcgacaccaaccgttactttatcatctcc300tccaacgtgattggcggttgcaaaggttcctccggcccgctgactatcaacggtaaaaac360ggcaaaccgttccagagcaccttcccattcgtgtccatcggcgacatggtgaacgctcaa420gagaaactgatcagccacttcggtatccacaaactgttcgcagtggcaggtggtagcatg480ggtggtatgcaggctctgcaatggtctgtagcttacccggatcgtctgaagaactgtatt540gtgatggcgtctagctctgaacacagcgctcagcagattgctttcaatgaagtaggccgt600caggcgatcctgtctgacccgaactggaaccagggtctgtatacccaggagaatcgtccg660tctaaaggtctggccctggcccgtatgatgggccatattacttatctgtctgatgaaatg720atgcgcgagaaattcggccgtaaaccgccgaaaggcaacaaccagtccactgacttcgcg780gtaggctcttacctgatctaccagggcgaatctttcgtggaccgttttgacgccaactct840tacatctacgttaccaaagcgctggaccacttctccctgggcactggcaaagaactgacc900aaagtactggcgaaagttcgctgtcgcttcctggttgttgcgtatacctccgactggctg960tatccgccgtatcagagcgaagaaattgttaaatccctggaagttaacgccgttccggtt1020agcttcgttgaactgaacaatccggcgggccgccacgatagctttctgctgccgagcgaa1080cagcaggatagcattctgcgcgattttctgtctagcaccgacgaaggcgtttttctgtaa1140<210>7<211>379<212>prt<213>人工序列(artificialsequence)<400>7metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproprolysglyasnasnglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>8<211>31<212>dna<213>人工序列(artificialsequence)<400>8tgactggtggacagcaaatgggtcgcggatc31<210>9<211>30<212>dna<213>人工序列(artificialsequence)<400>9cggcaccaggccgctgctgtgatgatgatg30<210>10<211>36<212>dna<213>人工序列(artificialsequence)<400>10agcggcctggtgccgatgcctacctctgagcagaac36<210>11<211>38<212>dna<213>人工序列(artificialsequence)<400>11gctgtccaccagtcattacagaaaaacgccttcgtcgg38<210>12<211>35<212>dna<213>人工序列(artificialsequence)<400>12tttacggctagctcagtcctaggtacaatgctagc35<210>13<211>41<212>dna<213>人工序列(artificialsequence)<400>13gttctgctcagaggtaggcatatgtatattctccttcttaa41<210>14<211>42<212>dna<213>人工序列(artificialsequence)<400>14gacgaaggcgtttttctgtaaccgctgcaggcatgcaagctt42<210>15<211>41<212>dna<213>人工序列(artificialsequence)<400>15ttaagaaggagaatatacatatgcctacctctgagcagaac41<210>16<211>42<212>dna<213>人工序列(artificialsequence)<400>16aagcttgcatgcctgcagcggttacagaaaaacgccttcgtc42<210>17<211>59<212>dna<213>人工序列(artificialsequence)<400>17gaaagctttttacggctagctcagtcctaggtacaatgctagcctttaagaaggagaat59<210>18<211>89<212>dna<213>人工序列(artificialsequence)<400>18cagagtaaacattgtgttaatggacgtcaatacatctggacatctaaacttctttgcgta60tagattgatacgcgaacgagccatgacat89<210>19<211>90<212>dna<213>人工序列(artificialsequence)<400>19ttaacacaatgtttactctggtgcctgacatttcaccgacaaagcccagggaacttcatc60acatgccaaagtacgacaattccaatgctg90<210>20<211>55<212>dna<213>人工序列(artificialsequence)<400>20gctagccgtaaaaagctttctagattgcagcaaagccgccttcgaggtcagcgat55<210>21<211>56<212>dna<213>人工序列(artificialsequence)<400>21gaaattcggccgtaaaccgtcgaaaggcaacaatcagtccactgacttcgcggtag56<210>22<211>56<212>dna<213>人工序列(artificialsequence)<400>22gaaattcggccgtaaaccgaaaaaaggcaacattcagtccactgacttcgcggtag56<210>23<211>56<212>dna<213>人工序列(artificialsequence)<400>23gaaattcggccgtaaaccgccgaaaggcaacgatcagtccactgacttcgcggtag56<210>24<211>56<212>dna<213>人工序列(artificialsequence)<400>24gaaattcggccgtaaaccgccgaaaggcaacaaacagtccactgacttcgcggtag56<210>25<211>56<212>dna<213>人工序列(artificialsequence)<400>25gaaattcggccgtaaaccgccgaaaggcaacgcccagtccactgacttcgcggtag56<210>26<211>55<212>dna<213>人工序列(artificialsequence)<400>26cggtttacggccgaatttctcgcgcatcatttcatcagacagataagtaatatgg55<210>27<211>379<212>prt<213>人工序列(artificialsequence)<400>27metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysprolyslysglyasnileglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>28<211>379<212>prt<213>人工序列(artificialsequence)<400>28metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproprolysglyasnalaglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>29<211>379<212>prt<213>人工序列(artificialsequence)<400>29metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproprolysglyasnaspglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>30<211>379<212>prt<213>人工序列(artificialsequence)<400>30metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproprolysglyasnlysglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375<210>31<211>379<212>prt<213>人工序列(artificialsequence)<400>31metprothrsergluglnasnglupheserhisglyservalglyval151015valtyrthrglnserileargphegluserleuthrleugluglygly202530gluthrilethrproleugluilealatyrgluthrtyrglythrleu354045asnglulyslysaspasnalaileleuvalcyshisalaleusergly505560aspalahisalaalaglyphehisgluglyasplysargproglytrp65707580trpasptyrtyrileglyproglylysserpheaspthrasnargtyr859095pheileileserserasnvalileglyglycyslysglysersergly100105110proleuthrileasnglylysasnglylyspropheglnserthrphe115120125prophevalserileglyaspmetvalasnalaglnglulysleuile130135140serhispheglyilehislysleuphealavalalaglyglysermet145150155160glyglymetglnalaleuglntrpservalalatyrproaspargleu165170175lysasncysilevalmetalasersersergluhisseralaglngln180185190ilealapheasngluvalglyargglnalaileleuseraspproasn195200205trpasnglnglyleutyrthrglngluasnargproserlysglyleu210215220alaleualaargmetmetglyhisilethrtyrleuseraspglumet225230235240metargglulyspheglyarglysproserlysglyasnasnglnser245250255thraspphealavalglysertyrleuiletyrglnglygluserphe260265270valaspargpheaspalaasnsertyriletyrvalthrlysalaleu275280285asphispheserleuglythrglylysgluleuthrlysvalleuala290295300lysvalargcysargpheleuvalvalalatyrthrserasptrpleu305310315320tyrproprotyrglnserglugluilevallysserleugluvalasn325330335alavalprovalserphevalgluleuasnasnproalaglyarghis340345350aspserpheleuleuprosergluglnglnaspserileleuargasp355360365pheleuserserthraspgluglyvalpheleu370375当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1