生产L-组氨酸的重组菌、其构建方法以及L-组氨酸的生产方法与流程
本发明总地涉及微生物发酵领域,具体涉及生产l-组氨酸的重组菌、其构建方法以及l-组氨酸的生产方法。
背景技术:
l-组氨酸是人和动物的第九种必需氨基酸,参与机体生长发育、抗氧化和免疫调节等重要的生理过程,是重要的药用氨基酸,可用于心脏病、贫血、胃肠溃疡治疗的输液制剂。目前,l-组氨酸生产主要采用以猪(牛)血粉为原料的蛋白质水解提取法,然而,蛋白质水解提取法存在原料成本高且利用率低、提取工艺复杂和环境污染大等缺点,使得l-组氨酸的生产成本高,价格昂贵。微生物发酵法生产l-组氨酸尚处于实验室研究阶段未得到大规模的工业化应用。目前针对l-组氨酸生产菌株选育的研究主要采用多轮传统诱变筛选和在诱变菌株的基础上进行基因工程改造的方法。通过诱变筛选获得的菌株会积累大量的负效应突变,导致菌株生长缓慢、环境耐受性降低以及营养需求增高等问题。这些缺陷限制了菌株的工业化应用。通过系统代谢工程改造构建l-组氨酸工程菌的研究报道较少,且菌株的l-组氨酸产量较低,与实现工业化应用存在较大差距。因此,构建高效合成l-组氨酸的工程菌株具有重要意义
l-组氨酸生物合成的主要载流途径是戊糖磷酸途径,当以葡萄糖为碳源时,经由戊糖磷酸途径生成l-组氨酸合成前体磷酸核糖焦磷酸(prpp),prpp和三磷酸腺苷(atp)在atp-磷酸核糖基转移酶的催化下,再经过开环、异构化及酰胺基转移等在内的一系列反应后生成l-组氨酸。l-组氨酸的生物合成具有与核苷酸合成竞争前体物质、复杂的代谢调控机制以及合成过程中高能量需求等特点,导致其工程菌的产酸水平和转化率相对较低。在前期研究工作(申请号:pct/cn2015/072220)中,发明人通过弱化6-磷酸葡萄糖异构酶编码基因(pgi)阻断上游糖酵解途径,过表达6-磷酸葡萄糖脱氢酶编码基因(zwf-opca)以增强戊糖磷酸途径的代谢能力,增强l-组氨酸合成的终端合成途径和前体合成基因的表达,以及偶联l-组氨酸合成途径和核苷酸合成途径,以谷氨酸棒杆菌野生型菌株为出发菌株,构建了携带不同基因改造靶点组合的产l-组氨酸的工程菌。然而,为了使微生物发酵生产l-组氨酸实现工业化应用,还需进一步提高工程菌的l-组氨酸产量以及缩短工程菌发酵周期。
如前述提及,atp不仅作为前体物质参与l-组氨酸分子骨架的合成,还为l-组氨酸的合成提供能量。作为三大高耗能氨基酸之一(色氨酸、精氨酸和l-组氨酸),合成1分子l-组氨酸需要消耗9.4分子atp作为能量供体,因此细胞内atp的合成和供给是限制l-组氨酸高效合成的关键因素。
在微生物细胞中,atp是重要的能量载体,为细胞的生理代谢提供能量。atp的合成以二磷酸腺苷(adp)为前体,经由糖酵解、三羧酸循环(tca循环)及氧化呼吸链,通过底物水平磷酸化或氧化磷酸化,在adp分子中引入高能磷酸基团生成atp。atp的合成前体adp的合成来源于核苷酸合成途径。葡萄糖经由戊糖磷酸途径生成磷酸核糖焦磷酸(prpp)后进入核苷酸合成途径,生成次黄嘌呤核苷酸(imp),再经腺苷琥珀酸合成酶(pura)和腺苷琥珀酸裂解酶(purb)的催化生成5’-单磷酸腺嘌呤核苷(amp),后磷酸化生成adp。amp也可通过腺嘌呤核苷酸代谢途径,经5’-核苷酸酶(usha)催化分解生成腺嘌呤核苷,腺嘌呤核苷进一步生成腺嘌呤。目前报道的提高细胞atp合成的方法,多为通过增强adp磷酸化生成atp的反应通量(如减少nadh的分解等)的方式,其对于atp合成的促进作用十分有限。然而,对于atp合成的关键限制因素——前体adp合成的改造和优化的报道极为少见。
技术实现要素:
本发明提供了一种生产l-组氨酸的重组菌,其中,所述重组菌与出发菌相比具有提高的腺苷琥珀酸合成酶pura的表达和/或活性,所述出发菌为能够积累l-组氨酸的菌株。
优选地,根据前述的重组菌,其中,所述重组菌具有至少两个拷贝的腺苷琥珀酸合成酶编码基因pura,和/或所述重组菌pura的表达由高转录或高表达活性的调控元件介导。优选地,所述调控元件为强启动子和/或优化的rbs序列。其中,所述优化的rbs序列为具有较高翻译起始效率的rbs序列。更优选地,所述强启动子为ptuf启动子,所述优化的rbs序列为ttatcggtatagggaaagattaggaaggaggttattac。
优选地,根据前述的重组菌,其中,所述重组菌与所述出发菌相比具有减弱的5’-核苷酸酶usha的表达和/或活性。优选地,所述重组菌的5’-核苷酸酶编码基因usha失活,或其调控元件为低转录或低表达活性的调控元件。
更优选地,根据前述的重组菌,其中,所述出发菌相对于原始菌具有增强的atp-磷酸核糖转移酶hisg的表达和/或活性。优选地,所述出发菌中具有两个或更多个拷贝的atp-磷酸核糖转移酶编码基因hisg,和/或所述hisg为突变的hisg,所述突变的hisg为第215位天冬酰胺突变为赖氨酸、第231位亮氨酸突变为苯丙氨酸和第235位苏氨酸突变为丙氨酸。
更优选地,根据前述的重组菌,其中,所述出发菌相比于所述原始菌具有增强的prpp合成酶prsa的表达。优选地,所述出发菌中具有两个或更多个拷贝的prpp合成酶编码基因prsa,和/或以强启动子代替prsa的启动子,所述强启动子优选为所述原始菌的psod启动子。
更优选地,根据前述的重组菌,其中,所述出发菌相对于所述原始菌具有增强的l-组氨酸合成操纵子hiseg基因和hisdcb基因的表达。优选地,以强启动子代替hiseg基因和hisdcb基因的启动子,所述强启动子优选为pglya启动子。
更优选地,根据前述的重组菌,其中,所述出发菌相比于所述原始菌具有降低的6-磷酸葡萄糖异构酶pgi的表达,和提高的6-磷酸葡萄糖脱氢酶zwf-opca的表达。优选地,所述出发菌的染色体上的6-磷酸葡萄糖异构酶编码基因pgi失活,和/或pgi的调控元件替换为低转录或低表达活性的调控元件。所述出发菌中具有两个或更多个拷贝的6-磷酸葡萄糖脱氢酶编码基因zwf-opca,和/或以强启动子代替所述原始菌染色体上tkt-tal-zwf-opca-devb操纵子的启动子,所述强启动子优选为所述原始菌的peftu启动子。
更优选地,根据前述的重组菌,其中,所述出发菌相比于所述原始菌具有增强的aicar甲基转移酶/imp环水化酶purh的表达。优选地,所述出发菌中具有两个或更多个拷贝的aicar甲基转移酶/imp环水化酶编码基因purh,和/或以强启动子替换purh的启动子。所述强启动子优选为所述原始菌的peftu启动子。
更优选地,根据前述的重组菌,其中,所述出发菌相比于所述原始菌具有减弱的磷酸核糖酰胺转移酶purf的表达。优选地,以弱启动子代替purf编码基因的启动子。更优选地,所述弱启动子为所述原始菌中的phom启动子。
还优选地,根据前述的重组菌,其中所述原始菌为选自棒杆菌属、小杆菌属、短杆菌属中的一株细菌。优选地,所述棒杆菌属的细菌选自谷氨酸棒杆菌corynebacteriumglutamicum、北京棒杆菌corynebacteriumpekinense、有效棒杆菌corynebacteriumefficiens、钝齿棒杆菌corynebacteriumcrenatum、嗜热产氨棒杆菌corynebacteriumthermoaminogenes、产氨棒杆菌corynebacteriumaminogenes、百合棒杆菌corynebacteriumlilium、美棒杆菌corynebacteriumcallunae和力士棒杆菌corynebacteriumherculis中的一株细菌;所述小杆菌属的细菌选自嗜氨小杆菌microbacteriumammoniaphilum中的一株细菌;和所述短杆菌属的细菌选自黄色短杆菌brevibacteriaceaeflvum、乳酸发酵短杆菌brevibacteriaceaelactofermentum和产氨短杆菌brevibacteriaceaeammoniagenes中的一株细菌。
本发明还提供了一种上述重组菌的构建方法,其中,包括:提高所述出发菌中腺苷琥珀酸合成酶的表达和/或活性。
优选地,根据前述的构建方法,其中,提高所述出发菌中腺苷琥珀酸合成酶的表达和/或活性通过如下至少一种方式实现:
(a)增加所述出发菌中腺苷琥珀酸合成酶编码基因的拷贝数;
(b)将所述出发菌中的腺苷琥珀酸合成酶编码基因的调控元件替换为高转录或高表达活性的调控元件,优选地,所述高转录或高表达活性的调控元件为强启动子和/或优化的rbs序列,所述强启动子优选为ptuf启动子,所述rbs序列优选为ttatcggtatagggaaagattaggaaggaggttattac。
优选地,根据前述的构建方法,其中,还包括:减弱所述出发菌中5’-核苷酸酶的表达和/或活性。优选地,减弱所述出发菌中5’-核苷酸酶的表达和/或活性通过如下至少一种方式实现:(c)使所述出发菌的5’-核苷酸酶编码基因失活,(d)将5’-核苷酸酶编码基因的调控元件替换为低转录或低表达活性的调控元件。
更优选地,根据前述的构建方法,其中,还包括:增强所述原始菌hisg的表达和/或活性。优选地,增加所述原始菌中hisg的拷贝数;和/或所述hisg为突变的hisg,所述突变的hisg为第215位天冬酰胺突变为赖氨酸、第231位亮氨酸突变为苯丙氨酸和第235位苏氨酸突变为丙氨酸。
更优选地,根据前述的构建方法,其中,还包括:增强所述原始菌prsa的表达。优选地,增加所述原始菌中prsa的拷贝数;和/或以强启动子代替prsa的启动子。所述强启动子优选为所述原始菌的psod启动子。
更优选地,根据前述的构建方法,其中,还包括:增强所述原始菌的l-组氨酸合成操纵子hiseg基因和hisdcb基因的表达。优选地,以强启动子代替hiseg基因和hisdcb基因的启动子。所述强启动子优选为pglya启动子。
更优选地,根据前述的构建方法,其中,还包括:降低所述原始菌pgi的表达,并提高zwf-opca的表达。优选地,使所述原始菌的染色体上的pgi失活,和/或pgi的调控元件替换为低转录或低表达活性的调控元件;增加所述原始菌中zwf-opca的拷贝数,和/或以强启动子代替所述原始菌染色体上tkt-tal-zwf-opca-devb操纵子的启动子。所述强启动子优选为所述原始菌的peftu启动子。
更优选地,根据前述的构建方法,其中,还包括:增强所述原始菌purh的表达。优选地,增加所述原始菌中purh的拷贝数,和/或以强启动子替换purh的启动子。所述强启动子优选为所述原始菌的peftu启动子。
更优选地,根据前述的构建方法,其中,还包括:减弱所述原始菌purf的表达。优选地,以弱启动子代替purf编码基因的启动子。更优选地,所述弱启动子为所述原始菌中的phom启动子。
本发明还提供了一种l-组氨酸的生产方法,其中,包括:发酵培养上述任一重组菌或采用上述任一构建方法构建的重组菌。
本发明所述的l-组氨酸重组菌,发酵60小时的l-组氨酸生产强度为0.01-1g/l/h,发酵结束时的l-组氨酸产量为1-60g/l,一般地,发酵产量可达2g/l以上。
本发明所提供的重组菌及其构建方法,开拓性地通过增强核苷酸合成途径中amp的合成,提高菌株atp的合成能力,为高效的组氨酸的合成提供充足的前体物质和能量载体atp,显著提高了菌株的组氨酸合成能力。
本发明开辟并且实践证明了新的提高组氨酸的发酵产量的方法并构建了相应的工程菌,观察到了可以叠加提高产量的效果,从而在实践上可用于细菌发酵l-组基酸的工业化生产。
本发明所提供的重组菌具有发酵周期短的特点,在发酵罐放大实验中约60-80小时可达到最大积累量,而目前报道的最高产量的组氨酸生产菌株的发酵时间长达120小时(mizukami,t.,hamu,a.,ikeda,m.,oka,t.,katsumata,r.,1994.cloningoftheatpphosphoribosyltransferasegeneofcorynebacteriumglutamicumandapplicationofthegenetol-histidineproduction.biosci.biotechnol.biochem.58,635-638.),易于过程和成本控制。
附图说明
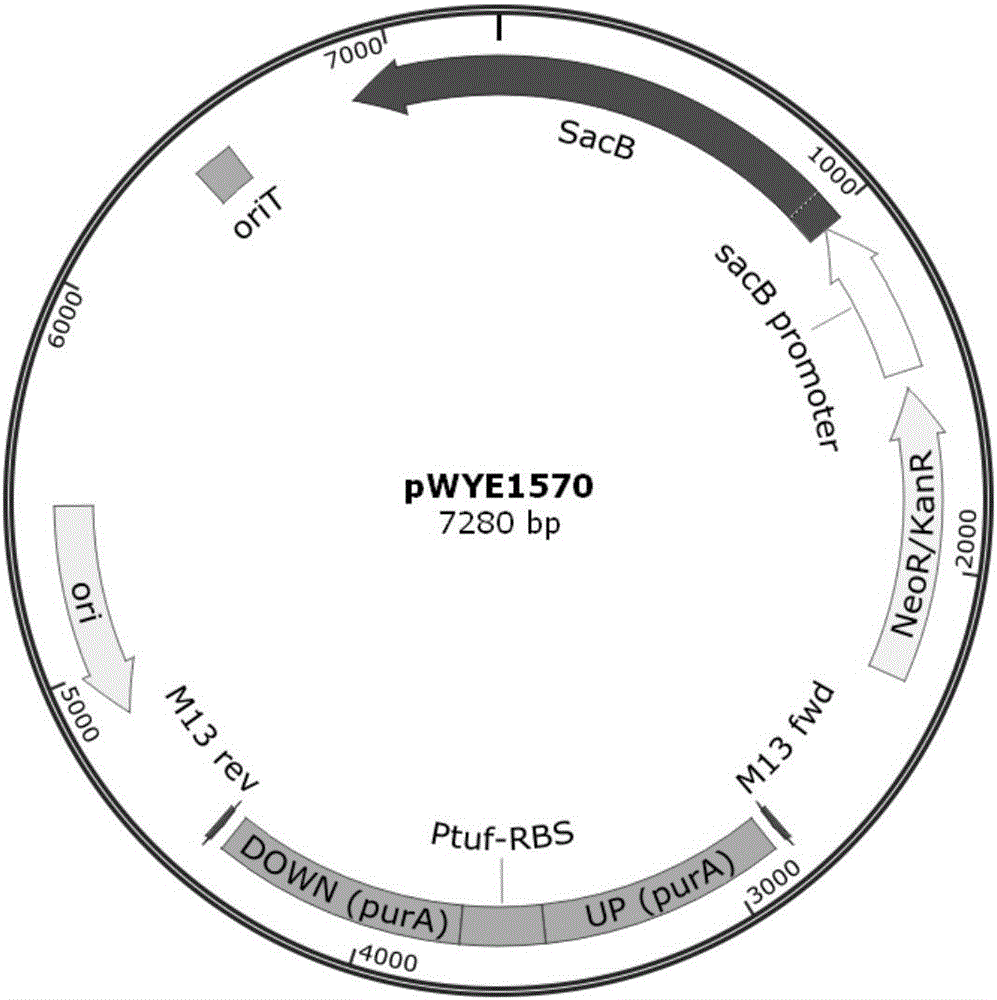
图1为质粒pwye1570的示意图;以及
图2为质粒pwye1569的示意图。
具体实施方式
以下结合附图和实施例,对本发明的具体实施方式进行更加详细的说明,以便能够更好地理解本发明的方案以及其各个方面的优点。然而,以下描述的具体实施方式和实施例仅是说明的目的,而不是对本发明的限制。具体来说,以下描述均以(野生型)谷氨酸棒杆菌为原始菌对重组工程菌的构建以及l-组氨酸的生产进行说明和实验,然而本领域技术人员应理解,本发明对氨基酸代谢途径的改造策略可用于其他合适的菌株以构建用于提高l-组氨酸的产量的工程菌。
此外,本发明引用了公开文献,这些文献是为了更清楚地描述本发明,它们的全文内容均纳入本文进行参考,就好像它们的全文已经在本文中重复叙述过一样。
在本发明的方法中,增加某基因的拷贝数可通过构建包含该基因的重组质粒,再将重组质粒导入出发菌/原始菌中实现;也可通过将该基因插入出发菌/原始菌中的染色体实现。这些方法都是本领域常用的,因此不再赘述。用于构建重组质粒的载体没有限制,可以是任何适宜的质粒,例如pxmj19。
本发明所述的失活细菌的pgi基因或usha基因,“失活”指的是相应被改造的对象发生变化,从而达到一定的效果,包括但是不限于,定点突变、插入失活和/或敲除。
本发明所用的染色体基因敲除、插入失活、基因插入、启动子替换和定点突变的方法可通过自杀性载体pk18mobsacb携带改造靶基因的同源臂发生同源重组实现的。
本发明提及的“出发菌(startingbacteria)”是指用于本发明基因改造策略的初始菌株。该菌株可以是天然存在的菌株,也可以是通过诱变或遗传工程改造等方式选育的菌株。为构建用于生产l-组氨酸的工程菌,所述出发菌优选为可积累l-组氨酸的菌株。具体可为专利pct/cn2015/072220所述l-组氨酸工程菌cg327和cg353。
本发明提及的“原始菌(originalbacteria)”是指未经任何遗传工程改造的菌株,可以是自然界存在的菌株,也可以是经人工诱变培育的菌株。具体可为野生型谷氨酸棒杆菌atcc13032。
本发明提及的方法中各步骤的执行顺序,除特别说明外,并不限于本文的文字所体现出来的顺序,也就是说,各个步骤的执行顺序是可以改变的,而且两个步骤之间根据需要可以插入其他步骤。
本发明提及的启动子peftu和ptuf为同一个启动子,序列完全一致。
根据本发明,通过改造腺嘌呤核苷酸代谢途径,可以提高菌株atp的合成能力,为l-组氨酸的合成提供更多的前体物质和能量载体,显著提高了菌株的组基酸产量。
在一种实施方案中,本发明对腺嘌呤核苷酸代谢途径的改造具体可为增强腺苷琥珀酸合成酶(pura)的表达和/或活性。pura催化imp和天冬氨酸转化生成中间产物腺苷酸代琥铂酸,腺苷酸代琥铂酸在腺苷酸代琥铂酸裂解酶的作用下,脱去延胡索酸,生成amp。增强pura的表达和/或活性可以促进imp生成amp,增加菌株amp的供应。
在一种实施方案中,本发明对腺嘌呤代谢途径的改造具体可为减弱usha的表达和/或活性。amp一方面可以通过usha催化分解去除磷酸基团生成腺嘌呤核苷,另一方面也可结合磷酸基团形成atp。弱化usha可以促使amp向atp转变。同时,腺嘌呤核苷为腺嘌呤的直接前体,腺嘌呤能够抑制pura基因表达。弱化usha的表达降低了腺嘌呤合成,也间接影响了pura的活性。
由上述描述可知,本发明通过对腺嘌呤核苷酸代谢途径进行改造,有效地提高了菌株atp的合成能力,进而实现了l-组氨酸产量的提高。
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。如未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段,可参见《分子克隆实验指南(第3版)》(科学出版社)、《微生物学实验(第4版)》(高等教育出版社)以及相应仪器和试剂的厂商说明书等。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
虽然在下述实施例中给出了强/弱启动子的示例,但对于强/弱启动子,在本发明中均没有特别限制,只要能够起到增强/减弱所启动基因的表达即可。可以列举的可用于本发明的强启动子有原始菌的peftu、psod、pglya、ppck、ppgk启动子等,弱启动子有原始菌的phom启动子等,但不限于此。
根据专利pct/cn2015/072220所述材料和方法构建质粒pxmj19-zwf-opca-prsa-hisgfbr-purh,l-组氨酸工程菌cg327,cg328以及cg353。
pxmj19-zwf-opca-prsa-hisgfbr-purh为将zwf-opca-prsa-hisgfbr片段插入质粒pxmj19的xbaⅰ和smaⅰ酶切位点间,同时将purh插入质粒pxmj19的smaⅰ和ecorⅰ酶切位点得到的载体。
cg327是将野生型谷氨酸棒杆菌atcc13032中的hiseg和hisdcb的启动子替换为谷氨酸棒杆菌内源强启动子pglya,hise和hisd基因的核糖体结合位点(rbs)替换为谷氨酸棒杆菌高表达基因的保守rbs序列(aaaggagga),hise基因的起始密码子gtg替换为atg,hisg基因替换为含三个氨基酸位点突变的hisgfbr,敲除pgi基因,并将purf的启动子替换为phom所获得的工程菌。
cg328是在cg327的基础上插入质粒pxmj19-zwf-opca-prsa-hisgfbr-purh。
cg353是将野生型谷氨酸棒杆菌atcc13032中的hiseg和hisdcb的启动子替换为谷氨酸棒杆菌内源强启动子pglya,hise和hisd基因的核糖体结合位点(rbs)替换为谷氨酸棒杆菌高表达基因的保守rbs序列(aaaggagga),hise基因的起始密码子gtg替换为atg,hisg基因替换为含三个氨基酸位点突变的hisgfbr,敲除pgi基因,prsa的启动子替换为psod,tkt-tal-zwf-opca-devb操纵子的启动子替换为peftu,purh启动子替换为peftu,purf的启动子替换为phom所获得的工程菌。
实施例1腺苷琥珀酸合成酶编码基因pura的启动子和rbs优化
为增强腺苷琥珀酸合成酶编码基因pura的表达,将pura基因的启动子替换为强启动子并优化其rbs。
根据genbank中谷氨酸棒杆菌atcc13032的pura基因及其上下游序列和ptuf启动子序列分别设计引物。在起始密码子atg前加入优化的rbs序列(ttatcggtatagggaaagattaggaaggaggttattac)。以atcc13032基因组为模板,以p1和p2为引物pcr扩增,得到225bp的pcr产物为携带ptuf启动子和优化的rbs序列的dna片段(seqidno:1),其中,从5’末端第1-187位为ptuf启动子序列,第188-225位为优化的rbs序列;以p3和p4为引物进行pcr扩增,得到660bp的pcr产物为用于同源重组的上游同源臂片段(seqidno:2);以p5和p6为引物进行pcr扩增,得到680bp的pcr产物为用于同源重组的下游同源臂片段(seqidno:3)。
采用gibson组装的方法,将纯化的三个上述pcr产物与经xbai和smai双酶切处理的同源重组载体pk18mobsacb(购自美国典型微生物保藏中心atcc,货号87097)连接。连接产物采用化学转化法转化至大肠杆菌ec135,在含卡那霉素(50μg/ml)的lb平板上筛选转化子,转化子传代培养三代后,以p7和p8为引物,采用菌落pcr鉴定转化子,得到1777bp的为阳性转化子,对鉴定正确的转化子提取质粒,经进一步序列测定验证,重组质粒pwye1570(pk18mobsacb-ptuf::ppura)构建成功。
上述所用的引物序列如下(5’→3’):
p1:aagcttgcatgcctgcaggtcgactctagatgatgaccgcagcgatg(seqidno.9)
p2:agggtaacggccagcgatactaagcttgcgg(seqidno.10)
p3:agcttagtatcgctggccgttaccctgcgaa(seqidno.11)
p4:cgattgcagccattctttccctataccgataaggacttcgtggtggctac(seqidno.12)
p5:aggaggacatacattaggaaggaggttattacatggctgcaatcgttattgt(seqidno.13)
p6:gattacgaattcgagctcggtacccgggagcatggttgcctggcca(seqidno.14)
p7:atgtgctgcaaggcgattaa(seqidno.15)
p8:tatgcttccggctcgtatgt(seqidno.16)
将序列测定正确的重组质粒pwye1570(pk18mobsacb-ptuf::ppura)电转化至l-组氨酸工程菌cg327(wt-pglya::phiseg-hisgfbr-pglya::phisdcb-δpgi::phom::ppurf),通过卡那霉素抗性正向筛选得到重组质粒整合至染色体上的菌落。通过蔗糖反向筛选,得到发生两次同源重组的阳性菌落。将阳性菌落以p9和p10为引物进行pcr扩增鉴定,得到935bp的为阳性重组子。对阳性重组子提取基因组,采用p11和p10引物进行pcr扩增并进行测序分析,结果为证实已成功将谷氨酸棒杆菌cg327中的pura的启动子替换为谷氨酸棒杆菌内源强启动子ptuf,并将其rbs序列替换为优化的rbs序列(ttatcggtatagggaaagattaggaaggaggttattac),谷氨酸棒杆菌cg584(cg327-ptuf::ppura)构建成功。
将质粒pxmj19-zwf-opca-prsa-hisgfbr-purh(pct/cn2015/072220)转化至工程菌cg584中,以p12和p13为引物,采用菌落pcr鉴定转化子,得到6164bp的为阳性转化子,对鉴定正确的转化子提取质粒鉴定进一步确定过表达质粒成功转化至工程菌中,l-组氨酸工程菌cg587(cg584/pxmj19-zwf-opca-prsa-hisgfbr-purh)构建成功。
上述所用的引物序列如下(5’→3’):
p9:tggccgttaccctgcgaatg(seqidno.17)
p10:acgaatgggtaggtgccg(seqidno.18)
p11:gagcccgaggggcgagggaa(seqidno.19)
p12:caattaatcatcggctcgta(seqidno.20)
p13:accgcttctgcgttctgatt(seqidno.21)
实施例25’-核苷酸酶编码基因usha的敲除
首先,根据genbank中谷氨酸棒杆菌atcc13032的usha基因及其上下游序列分别设计引物。以谷氨酸棒杆菌atcc13032基因组dna为模板,以p14和p15为引物pcr扩增usha基因上游同源臂(640bp,seqidno.4);以p16和p17为引物扩增usha基因下游同源臂(655bp,seqidno.5)。采用gibson组装的方法,将纯化的上述pcr产物与经xbai和smai双酶切处理的同源重组载体pk18mobsacb(购自美国典型微生物保藏中心atcc,货号87097)连接。连接产物采用化学转化法转化至大肠杆菌ec135,在含卡那霉素(50μg/ml)的lb平板上筛选转化子,转化子传代培养三代后,以p7和p8为引物,采用菌落pcr鉴定转化子,得到1507bp的为阳性转化子,对鉴定正确的转化子提取质粒,经进一步序列测定验证,重组质粒pwye1569构建成功(pk18mobsacb-δusha)。
将序列测定正确的重组质粒pwye1569(pk18mobsacb-δusha)电转化至l-组氨酸工程菌cg327和cg584中,通过卡那霉素抗性正向筛选得到重组质粒整合至染色体上的菌落。通过蔗糖反向筛选,得到发生两次同源重组的阳性菌落。将阳性菌落以p18和p19为引物进行pcr扩增鉴定,得到1455bp的为阳性重组子,对pcr产物进行测序分析,结果为证实已成功将谷氨酸棒杆菌cg584中的usha敲除,谷氨酸棒杆菌cg583(cg327δusha)和cg585(cg584δusha)构建成功。
上述所用的引物序列如下(5’→3’):
p14:aagcttgcatgcctgcaggtcgactctagatccgcccgcttgctgctg(seqidno.22)
p15:ttactacaagttataaattctcctggaaaaataccggcaatgcg(seqidno.23)
p16:ccaggagaatttataacttgtagtaaataaatcggg(seqidno.24)
p17:gattacgaattcgagctcggtacccgggtttacatcatcgcagtcg(seqidno.25)
p18:tgccgtgcgcgtggact(seqidno.26)
p19:aacccccatcgatccccac(seqidno.27)
将质粒pxmj19-zwf-opca-prsa-hisgfbr-purh分别转化至工程菌cg583和cg585中,以p12和p13为引物,采用菌落pcr鉴定转化子,得到6164bp的为阳性转化子,对鉴定正确的转化子提取质粒鉴定进一步确定过表达质粒成功转化至工程菌中,l-组氨酸工程菌cg586(cg583/pxmj19-zwf-opca-prsa-hisgfbr-purh)和cg588(cg585/pxmj19-zwf-opca-prsa-hisgfbr-purh)构建成功。
实施例3腺苷琥珀酸合成酶编码基因pura的拷贝数增加
为增强腺苷琥珀酸合成酶编码基因pura的表达,采用在染色体上增加pura基因的拷贝数的方式。增加pura基因的拷贝是在染色体的目标位置插入包括ptuf启动子和实施例1所述的优化的rbs及pura基因编码区的表达盒。
首先构建在染色体上增加pura基因的拷贝数的同源重组质粒。
根据谷氨酸棒杆菌atcc13032的目标插入位点的上下游序列、菌株cg327的ptuf-pura序列设计引物。以菌株cg327的基因组dna为模板,以p20和p21为引物扩增目标插入位点的上游同源臂(1000bp,seqidno.6);以p22和p23为引物扩增含ptuf、优化的rbs序列和pura基因的表达盒(1518bp,seqidno.7);以p24和p25为引物扩增目标插入位点的下游同源臂(1000bp,seqidno.8)。再将纯化的上述pcr产物与经xbai和smai双酶切处理的pk18mobsacb采用gibson组装的方式一步连接。连接产物采用化学转化法转化至大肠杆菌ec135,在含卡那霉素(50μg/ml)的lb平板上筛选转化子,转化子传代培养三代后,以p7和p8为引物,采用菌落pcr鉴定转化子,得到3730bp的为阳性转化子,对鉴定正确的转化子提取质粒,经进一步序列测定验证,结果该质粒为将含有目标插入位点上下游同源臂及含ptuf、优化的rbs序列和pura基因的表达盒插入载体pk18mobsacb中得到的重组质粒重组质粒,重组质粒pwye1571(pk18mobsacb-ncgl1021::pura)构建成功。
将序列测定正确的同源重组质粒pwye1571(pk18mobsacb-ncgl1021::pura)电转化至工程菌cg583中,通过卡那霉素抗性正向筛选得到重组质粒整合至染色体上的菌落,通过蔗糖致死反向筛选,得到发生两次同源重组的阳性菌落。将阳性菌落分别以p26和p27为引物进行pcr扩增,得到3929bp的为阳性重组子。将阳性重组子提取基因组dna进行测序分析,结果为将含ptuf、优化的rbs序列和pura基因的表达盒插入至染色体目标位点,得到工程菌cg589(cg583-ncgl1021::pura)。
将质粒pxmj19-zwf-opca-prsa-hisgfbr-purh转化至工程菌cg589中,以p12和p13为引物,采用菌落pcr鉴定转化子,得到6164bp的为阳性转化子,对鉴定正确的转化子提取质粒鉴定进一步确定过表达质粒成功转化至工程菌中,l-组氨酸工程菌cg590(cg589/pxmj19-zwf-opca-prsa-hisgfbr-purh)构建成功。
上述所用的引物序列如下(5’→3’):
p20:tgcatgcctgcaggtcgactctagactcattccagcgtcacgac(seqidno.28)
p21:gtaacggccagggtagagccttttgttgg(seqidno.29)
p22:ggctctaccctggccgttaccctgcgaa(seqidno.30)
p23:tcttcctgttctagttgtcagctagtacgtcatgc(seqidno.31)
p24:tgacaactagaacaggaagagcccgtaaac(seqidno.32)
p25:cgaattcgagctcggtacccgggtgggaagactcgcctctg(seqidno.33)
p26:cattgctatacgcaaacag(seqidno.34)
p27:atattttctaatgctctac(seqidno.35)
实施例4、无质粒l-组氨酸工程菌的构建
为构建无质粒l-组氨酸工程菌,将上述构建的质粒pwye1570(pk18mobsacb-ptuf::ppura)电转化至无质粒l-组氨酸工程菌cg353(wt-pglya::phiseg-hisgfbr-pglya::phisdcb-peftu::ptkt-psod::pprsa-δpgi-peftu::ppurh-phom::ppurf),通过卡那霉素抗性正向筛选得到重组质粒整合至染色体上的菌落。通过蔗糖反向筛选,得到发生两次同源重组的阳性菌落。将阳性菌落以p9和p10为引物进行pcr扩增鉴定,得到935bp的为阳性重组子。对阳性重组子提取基因组,采用p11和p10引物进行pcr扩增并进行测序分析,结果为证实已成功将谷氨酸棒杆菌cg353中的pura的启动子替换为谷氨酸棒杆菌内源强启动子ptuf,并将其rbs序列替换为优化的rbs序列(ttatcggtatagggaaagattaggaaggaggttattac),谷氨酸棒杆菌cg591(cg353-ptuf::ppura)构建成功。
将序列测定正确的重组质粒pwye1569(pk18mobsacb-δusha)电转化至l-组氨酸工程菌cg591中,通过卡那霉素抗性正向筛选得到重组质粒整合至染色体上的菌落。通过蔗糖反向筛选,得到发生两次同源重组的阳性菌落。将阳性菌落以p18和p19为引物进行pcr扩增鉴定,得到1455bp的为阳性重组子,对pcr产物进行测序分析,结果为证实已成功将谷氨酸棒杆菌cg591中的usha基因敲除,谷氨酸棒杆菌cg592(cg591δusha)构建成功。
实施例5l-组氨酸工程菌在l-组氨酸发酵生产中的应用
1、工程菌摇瓶发酵生产l-组氨酸
摇瓶发酵采用的发酵培养基具体如下:葡萄糖40g/l,(nh4)2so420g/l,kh2po40.5g/l,k2hpo4·3h2o0.5g/l,mgso4·7h2o0.25g/l,feso4·7h2o0.01g/l,mnso4·h2o0.01g/l,znso4·7h2o0.001g/l,cuso40.0002g/l,nicl2·6h2o0.00002g/l,生物素0.0002g/l,ph7.0-7.2,caco320g/l。葡萄糖单独灭菌,115℃高压灭菌15min。mgso4·7h2o以及无机盐离子单独灭菌,121℃高压灭菌20min。维生素采用0.22μm无菌滤膜过滤除菌。其余组分在121℃高压灭菌20min。
种子培养基具体如下:葡萄糖20g/l,硫酸铵5g/l,k2hpo4·3h2o1g/l,mgso4·7h2o0.4g/l,生物素50μg,维生素b11mg,安琪酵母粉(fm802)10g/l,安琪蛋白胨(fp318)10g/l。
1)、种子液的获得
将上述实施例制备的携带质粒的l-组氨酸工程菌cg586,cg587,cg588,cg590;无质粒l-组氨酸工程菌cg591和cg592;对照菌cg328和cg353;以及野生型菌株c.glutamicumatcc13032分别接种到种子培养基中,种子液培养条件为培养温度32℃,摇床转速为220r/min,培养时间8h,得到种子液,od600为20。
2)、发酵
将种子液按照体积百分含量为3%接种到含有终浓度为10μg/ml氯霉素的发酵培养基(500ml挡板三角瓶装液量为30ml)中,32℃,220r/min,培养60h,携带质粒的l-组氨酸工程菌于发酵培养6h时加入终浓度为1mmol/l的异丙基-β-d-硫代吡喃半乳糖苷(iptg)进行目的基因的诱导表达。间歇补加浓氨水控制发酵液的ph在7.0-7.2之间,根据残糖情况,补加浓度为400g/l的葡萄糖母液,控制发酵液残糖在5-10g/l。
收集发酵产物12000×g,离心5min,收集上清液。
3)、检测l-组氨酸含量
采用高效液相法,具体方法如下(2,4-二硝基氟苯柱前衍生高效液相法):取50μl上述上清液于2ml离心管中,加入200μlnahco3水溶液(0.5mol/l,ph9.0)和100μl1%的2,4-二硝基氟苯-乙腈溶液(体积比),于60℃水浴中暗处恒温加热60min,然后冷却至25℃,加入650μlkh2po4水溶液(0.01mol/l,ph7.2±0.05,用naoh水溶液调整ph),放置15min过滤后可进样,进样量为15μl。
所用色谱柱为c18柱(zorbaxeclipsexdb-c18,4.6*150mm,agilent,usa);柱温:40℃;紫外检测波长:360nm;流动相a为0.04mol/lkh2po4水溶液(ph7.2±0.05,用40g/lkoh水溶液调整ph),流动相b为55%乙腈水溶液(体积比),流动相流速为1ml/min,洗脱过程如下表1所示:
表1
以野生型菌株c.glutamicumatcc13032为参照,测定发酵过程中的葡萄糖消耗、od600以及最终的l-组氨酸产量。结果如表2所示。
表2为摇瓶发酵实验中携带质粒的l-组氨酸工程菌cg586,cg587,cg588,cg590;无质粒l-组氨酸工程菌cg591和cg592;以及对照菌cg328和cg353的l-组氨酸产量。
表2
摇瓶发酵实验中,发酵60h,野生型菌株c.glutamicumatcc13032未检测到l-组氨酸的积累。对照菌cg328的l-组氨酸产量为1.81g/l,缺失5’-核苷酸酶编码基因usha的工程菌cg586的l-组氨酸产量为1.96g/l,较对照菌株(cg328)提高了8.3%。通过启动子和rbs优化增强pura基因表达的工程菌cg587的l-组氨酸产量为2.34g/l,较对照菌株(cg328)提高了29%。同时缺失usha和分别以启动子和rbs优化(cg588)以及增加拷贝数(cg590)两种方式增强pura表达的工程菌的l-组氨酸产量分别为2.53g/l和2.58g/l,较对照菌株(cg328)分别提高了39%和42%。通过启动子和rbs优化增强pura基因表达的无质粒工程菌cg591的l-组氨酸产量为1.77g/l,较对照菌株(cg353)提高了17%。同时缺失usha以及启动子和rbs优化增强pura表达的工程菌cg592的l-组氨酸产量为1.86g/l,较对照菌株(cg353)提高了23%。
结果表明,通过增强腺苷琥珀酸合成酶编码基因pura的表达和/或缺失5’-核苷酸酶编码基因usha,工程菌的l-组氨酸产量显著提高。
2、工程菌cg588发酵罐发酵生产l-组氨酸
种子培养基具体如下:葡萄糖20g/l,硫酸铵5g/l,k2hpo4·3h2o1g/l,mgso4·7h2o0.9g/l,生物素50μg,维生素b11mg,酵母粉2g/l,蛋白胨2g/l。
发酵采用的发酵培养基具体如下:葡萄糖20g/l,硫酸铵5g/l,kh2po40.5g/l,k2hpo4·3h2o0.5g/l,mgso4·7h2o0.25g/l,feso4·7h2o10mg/l,mnso4·h2o10mg/l,维生素b10.5mg/l,酵母粉5g/l。
1)、种子液的获得
将工程菌cg588接种到种子培养基中,种子液培养条件为培养温度32℃,摇床转速为220r/min,培养时间8h,得到种子液,od600为20。
2)、发酵
将种子液按照体积百分含量为10%接种到含有终浓度10μg/ml氯霉素的发酵培养基中。
采用的发酵罐为7.5l发酵罐(bioflo115,nbs):内置定速可编程控泵,可以实现恒速补料。发酵过程中通过蠕动泵补加600g/l的葡萄糖,控制发酵体系中葡糖糖的浓度为5~10g/l,同时流加10g/l的酵母粉。通过加热套和冷却水控制发酵温度维持在32℃;通入空气提供溶氧,转速与溶氧信号级联控制溶氧维持在30%;补加浓氨水调控ph,维持在6.9左右。发酵连续进行52h。当od600=4~5时,加入iptg(异丙基硫代半乳糖苷,终浓度为0.5mmol/l)诱导重组质粒携带的基因表达。
收集发酵产物12000×g离心5min,收集上清液。
3)、检测l-组氨酸含量
按照上述“1、工程菌摇瓶发酵生产l-组氨酸”中3)的方法检上清液中的l-组氨酸含量,结果显示,工程菌cg588的l-组氨酸最高产量为22.53g/l,生产强度为0.31g/l/h。结果表明,通过增强腺苷琥珀酸合成酶编码基因pura的表达和/或缺失5’-核苷酸酶编码基因usha,增强菌株atp的合成能力,为l-组氨酸的合成提供更多的前体物质和能量载体,能够显著提高菌株l-组氨酸的合成效率。
最后应说明的是:显然,上述实施例仅仅是为清楚地说明本发明所作的举例,而并非对实施方式的限定。对于所属领域的普通技术人员来说,在上述说明的基础上还可以做出其它不同形式的变化或变动。这里无需也无法对所有的实施方式予以穷举。而由此所引申出的显而易见的变化或变动仍处于本发明的保护范围之中。
序列表
<110>中国科学院微生物研究所
<120>生产l-组氨酸的重组菌、其构建方法以及l-组氨酸的生产方法
<160>35
<170>siposequencelisting1.0
<210>1
<211>225
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>promoter
<222>(1)..(187)
<220>
<221>rbs
<222>(188)..(225)
<400>1
tggccgttaccctgcgaatgtccacagggtagctggtagtttgaaaatcaacgccgttgc60
ccttaggattcagtaactggcacattttgtaatgcgctagatctgtgtgctcagtcttcc120
aggctgcttatcacagtgaaagcaaaaccaattcgtggctgcgaaagtcgtagccaccac180
gaagtccttatcggtatagggaaagattaggaaggaggttattac225
<210>2
<211>660
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>2
atgatgaccgcagcgatgcctggggcgtcaagcatcggcaccaagcgggcaccgtagacg60
ccacggcgtggggccttcgaggcagcctcgtggcggatgagaacatgatcatcgacgatg120
acttcgccggtgatctcacccggctcccagttggtgatggtggctgaaccggcgacggcg180
accgcaacgtcatcgcggatgagcggggcaggattgaccaatccggtcagtgcgagctca240
agcgcggcagcctcatcggcgggcagaccccagttcttcgcagccgttgaaggaccaacg300
ggcacgaatccaatctccgcccacaaattatcggcgcgcatgagacgagtcaggaccgca360
gacagcgctgcatccgaaccgatcaccacgatgcgcaaacgggtttcaggctgttgtggc420
gcaaacttagggctgctcaaatgctcgacatcaggctgcttggcgatctcatcgagagaa480
ggcgtgggatcctgagggagcacatcaaaggccacatcatcaagaattttgaggtctttt540
cgggtgggcaccgcaggcaaatcgtggatctccacagaggtggaaagggcagggaattcg600
ttgatgtgtggggcatcgcaccgcaaaaccagaagacgcataccgcaagcttagtatcgc660
<210>3
<211>680
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>3
atggctgcaatcgttattgtcggcgctcaatggggcgatgaaggcaaaggtaaggccacg60
gatattctcggcggactcgtcgattacgtggttaagcccaatggcggtaacaacgctgga120
cacactgttgtggtcggcggcgagaagtacgagctaaagctccttcctgccggcgtcctc180
tccgaaacggccaccccaattttgggcaacggcgttgtgatcaaccttgaggcactgttc240
gaagaaatcgacggccttgaggctcgcggtgcggatgcatcccgcctgcgcatctctgca300
aacgctcacctggttgctccataccaccaggtgatggaccgtgttcaggaacgcttcctg360
ggcaagcgcgcaatcggcaccaccggccgtggcatcggcccaacctacgcggacaaagta420
tcccgcgtgggaatccgtgttcaagacattttcgacgaatccatccttcgtcaaaaagtc480
gaatccgccctggattacaaaaaccaggtgctggtgaagatgtacaaccgcaaggccatc540
gtcgctgaggaaatcgtgcagtacttcctctcctacgctgatcgtctgcgccccatggtc600
atcgatgccaccttggtgctcaacgaggcacttgatcagggcaagcacgttcttatggaa660
ggtggccaggcaaccatgct680
<210>4
<211>640
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>4
tccgcccgcttgctgctggccggcggtcgaattggtctcctgcattccgcgtatgtcacc60
caccattacgactacgccaagggtgactacaagtggatttacatcgaacgaaaccgacac120
gttttgctgctcagcgtgctgccgcttccattgctgttcgtgctgatcccgcagatcctc180
ggtgtgaacctgggactgtgggcgattgccgcaaaggaaaagagggtcggactcaaggtg240
aagtcccttcgcctcctgatccgcgatctaccagcgattttcaaactgcgtaggagcacg300
caggagcttgccgaactcacaccatcgcaatatctggcaaaaatggaatggcgcctagac360
aatcccaacctaggcaacattggatccaacaagattgttgcgactggatataagacctat420
tacaagttgtgtatgagtatcctgaaattgctcgcttaacaccccataaagagggtgaag480
atttaagttcaggtgcgatctgggtgaacagtacataaatatcatctttcgctaatggaa540
agccccagctcaccgaattctccattcgttttaattgcttcgttaattaaaacgccatat600
aaaaaccggcgcattgccggtatttttccaggagaattta640
<210>5
<211>655
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>5
taacttgtagtaaataaatcgggccttcccaaaagattctttcttgggaaaggcccgatt60
ttggtatttgaggtcttttagtggagtgtcttagacatttgtcgcaacactgactaactg120
gcggtcgtatttgtcagttgtcgcactttcaagcatgacccacgcagcatcttcatatga180
ggtgtacattccggtttctcgatttactcgaacaacacgattctcaagcttactgcgtgc240
cccttgagcatccataagggaagtgacgggaaactgtctttaagccctgaacgtaagaga300
tcaacgatgctagtggacagtctttagatgcctttcatcgcatagattattaaaatgcaa360
ttaccagccagatttggttgatgaagtcactgttgttcggggaatatttcagcgataagt420
ctctgttgttcttcttgttgtttttgtaaggctttccaagcggtactattttgggctgtt480
ttaatgtccgcagtgagacggttggatttgtctactattttcatgaattctttaaatttt540
atacctcctgcaatgaaagctgcgactacaagtatgagtccaagcacagctcgtgtgccg600
tcgtcaaagataaagctgattgcgtaggctagtagggcgactgcgatgatgtaaa655
<210>6
<211>1000
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>6
ctcattccagcgtcacgacgttccgaaggtactggttacctggcattgggcactaccgtt60
tctgcagcacttggaccagccctagcactttttgtcctaggaacatttgattacgacatg120
ctgtttatcgtggtcttggcaacctcggtcatctctttgatcgccgtcgtgttcatgtac180
tttaagaccagcgaccctgagccttctggggaaccagccaagttcagcttcaaatctatt240
atgaacccaaagatcatccccatcggcatctttatcttgcttatttgctttgcttactct300
ggcgtcattgcctacatcaacgcatttgctgaagaacgcgatctgattacgggtgctgga360
ttgttcttcattgcctacgcagtatcaatgtttgtgatgcgcagcttccttggcaaactg420
caggaccgtcgcggagacaacgtcgttatttactttggattgttcttcttcgttatttcc480
ttgacgattttgtcctttgccacttccaactggcacgttgtgttgtccggagtcattgca540
ggtctgggatacggcactttgatgccagcagtgcagtccatcgctgttggtgtagtagac600
aaaaccgaattcggtacggccttctccactttgttcctgtttgtggacttaggttttggc660
tttggacctattatcctgggagcagtttctgcggcaattggtttcggacctatgtatgca720
gcactggcaggtgtgggtgtgattgccggaatcttctacctgttcacacacgctcgcacc780
gatcgagctaagaatggctttgttaaacacccagagcctgtcgctttagttagctagttc840
tttcagctttccctcccgatcagcgtaaaccggcccttccggttttggggtacatcacag900
aacctgggctagcggtgtagacccgaaaataaacgagccttttgtcagggttaaggttta960
ggtatctaagctaaccaaacaccaacaaaaggctctaccc1000
<210>7
<211>1518
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>promoter
<222>(1)..(187)
<220>
<221>rbs
<222>(188)..(225)
<220>
<221>gene
<222>(226)..(1518)
<400>7
tggccgttaccctgcgaatgtccacagggtagctggtagtttgaaaatcaacgccgttgc60
ccttaggattcagtaactggcacattttgtaatgcgctagatctgtgtgctcagtcttcc120
aggctgcttatcacagtgaaagcaaaaccaattcgtggctgcgaaagtcgtagccaccac180
gaagtccttatcggtatagggaaagattaggaaggaggttattacatggctgcaatcgtt240
attgtcggcgctcaatggggcgatgaaggcaaaggtaaggccacggatattctcggcgga300
ctcgtcgattacgtggttaagcccaatggcggtaacaacgctggacacactgttgtggtc360
ggcggcgagaagtacgagctaaagctccttcctgccggcgtcctctccgaaacggccacc420
ccaattttgggcaacggcgttgtgatcaaccttgaggcactgttcgaagaaatcgacggc480
cttgaggctcgcggtgcggatgcatcccgcctgcgcatctctgcaaacgctcacctggtt540
gctccataccaccaggtgatggaccgtgttcaggaacgcttcctgggcaagcgcgcaatc600
ggcaccaccggccgtggcatcggcccaacctacgcggacaaagtatcccgcgtgggaatc660
cgtgttcaagacattttcgacgaatccatccttcgtcaaaaagtcgaatccgccctggat720
tacaaaaaccaggtgctggtgaagatgtacaaccgcaaggccatcgtcgctgaggaaatc780
gtgcagtacttcctctcctacgctgatcgtctgcgccccatggtcatcgatgccaccttg840
gtgctcaacgaggcacttgatcagggcaagcacgttcttatggaaggtggccaggcaacc900
atgctcgacgtggaccacggcacctacccattcgtcacctcctccaacccaaccgccggt960
ggcgcaagtgttggttcaggtatcggcccaaccaagatcaccagctccttgggtatcatc1020
aaggcctacaccactcgtgttggtgccggcccattcccaactgagctgtttgataagtgg1080
ggcgagtacctgcagaccgtcggtggcgaggtcggcgtgaacaccggccgtaagcgtcgc1140
tgtggctggtacgactccgtgattgctcgttacgcatcccgcgtcaacggattcaccgac1200
tacttcctgaccaagctagacgtgctcaccggcatcggtgaaatcccaatctgcgtagct1260
tacgacgttgatggtgttcgccacgatgaaatgccactgacccagtcagagttccaccac1320
gcaaccccaatctttgaaaccatgcctgcatgggacgaagacatcaccgactgcaagacc1380
ttcgaggatcttccacaaaaggcccaggactacgtccgacgtctggaagaactctctggt1440
gctcgcttctcctacatcggtgttggacctggtcgcgatcagaccatcgtcctgcatgac1500
gtactagctgacaactag1518
<210>8
<211>1000
<212>dna
<213>谷氨酸棒杆菌(corynebacteriumglutamicum)
<400>8
aacaggaagagcccgtaaacctctgactagcgtcaccctctgattaaggcgaccgcggat60
ttaagagcagaggctgccacgagcgcatcttcacggctgtgtgttgtactaaaagtacag120
cgcacagccgttcgtgcttgatcctcctcaagccccaacgccagcaacacatgggatacc180
tctccggaaccacaggcagaaccaggggagcacacaatgccttggcgttccaattccaga240
agaacagtttcagatcctatgctgtcgaagagaaaagatgcgtgtccatcaatgcgcatc300
ctaggatgtccagtcaggtgtgctcccgggatagtgagaacttcctcgatgaattcgcca360
agatctggataggattccgccctggccaattccaaggcagtggcaaaggcgatagccccc420
gcaacgttttccgtgccactacgccgccctttttcctggccgccgccatggattaccggc480
tccaggggaagctttgaccataacactccaatccctttaggcgcaccgaatttatgaccc540
gacaaacttaacgcgtcaactcccaagtcaaaggttaaatgtgcagcttgcactgcatcg600
gtgtgaaaaggcgtactgcttaccgccgccaactcagctatcggctgaatggttcccacc660
tcattgttggcataaccaatgctgatcaatgtggtgtccggcctgactgctttgcggaga720
ccctccggggagatcagcccagtgtgatcgggggataggtaggtgatctcgaaatcatga780
aacctttcaagataagcagcagtttctaggacactgtcatgctcgatcggggtggtgatg840
aggtgccggccacgaggattagctaagcacgctcctttgatagcgaggttgttggcttct900
gatccacccgacgtaaacgtcacctgtgtggggcgtcctccgataatgcgggccacccga960
gttcgagcatcctccagccccgcagaggcgagtcttccca1000
<210>9
<211>47
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(47)
<400>9
aagcttgcatgcctgcaggtcgactctagatgatgaccgcagcgatg47
<210>10
<211>31
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(31)
<400>10
agggtaacggccagcgatactaagcttgcgg31
<210>11
<211>31
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(31)
<400>11
agcttagtatcgctggccgttaccctgcgaa31
<210>12
<211>50
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(50)
<400>12
cgattgcagccattctttccctataccgataaggacttcgtggtggctac50
<210>13
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(52)
<400>13
aggaggacatacattaggaaggaggttattacatggctgcaatcgttattgt52
<210>14
<211>46
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(46)
<400>14
gattacgaattcgagctcggtacccgggagcatggttgcctggcca46
<210>15
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>15
atgtgctgcaaggcgattaa20
<210>16
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>16
tatgcttccggctcgtatgt20
<210>17
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>17
tggccgttaccctgcgaatg20
<210>18
<211>18
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(18)
<400>18
acgaatgggtaggtgccg18
<210>19
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>19
gagcccgaggggcgagggaa20
<210>20
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>20
caattaatcatcggctcgta20
<210>21
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(20)
<400>21
accgcttctgcgttctgatt20
<210>22
<211>48
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(48)
<400>22
aagcttgcatgcctgcaggtcgactctagatccgcccgcttgctgctg48
<210>23
<211>44
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(44)
<400>23
ttactacaagttataaattctcctggaaaaataccggcaatgcg44
<210>24
<211>36
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(36)
<400>24
ccaggagaatttataacttgtagtaaataaatcggg36
<210>25
<211>46
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(46)
<400>25
gattacgaattcgagctcggtacccgggtttacatcatcgcagtcg46
<210>26
<211>17
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_binding
<222>(1)..(17)
<400>26
tgccgtgcgcgtggact17
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_binding
<222>(1)..(19)
<400>27
aacccccatcgatccccac19
<210>28
<211>44
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_binding
<222>(1)..(44)
<400>28
tgcatgcctgcaggtcgactctagactcattccagcgtcacgac44
<210>29
<211>29
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(29)
<400>29
gtaacggccagggtagagccttttgttgg29
<210>30
<211>28
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(28)
<400>30
ggctctaccctggccgttaccctgcgaa28
<210>31
<211>35
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(35)
<400>31
tcttcctgttctagttgtcagctagtacgtcatgc35
<210>32
<211>30
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(30)
<400>32
tgacaactagaacaggaagagcccgtaaac30
<210>33
<211>41
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(41)
<400>33
cgaattcgagctcggtacccgggtgggaagactcgcctctg41
<210>34
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(19)
<400>34
cattgctatacgcaaacag19
<210>35
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<222>(1)..(19)
<400>35
atattttctaatgctctac19
- 还没有人留言评论。精彩留言会获得点赞!