一种木糖醇基因工程生产菌及其构建方法和应用与流程
本发明涉及基因工程技术领域,具体涉及一种木糖醇基因工程生产菌及其构建方法和应用。
背景技术
木糖醇是一种五碳糖醇,其甜度与蔗糖相当,热量值却只有其60%左右,木糖醇具有抗龋齿及代谢不依赖胰岛素、改善肝功能等特点,广泛应用于食品、医药和化工行业。
工业上生产木糖醇主要是利用半纤维素酸水解获得木糖,经分离纯化后得到纯度在95%以上的木糖在高温高压条件下用镍催化加氢制得,这种工艺条件苛刻,且容易造成污染,生产成本较高。生物法生产木糖醇不需要高温高压条件、易燃易爆氢气、污染环境的镍催化剂和高纯度的木糖等,反应条件温和、安全节能且环境友好,所以生物法转化生产木糖醇越来越受到人们的重视。
目前用于发酵法制备木糖醇的微生物几乎都是酵母菌,既有自然菌种,也有基因工程菌。酵母菌作为木糖醇生产菌株有着自己的优势,比如能耐受较高的糖浓度,对半纤维素水解液中的抑制因子抵抗性较强,等等。但是也存在不可回避的问题,如具有潜在的致病性、酵母本身所含有的木糖还原酶专一性较差等。
大肠杆菌作为生产各种高附加值化学品的理想宿主,当前研究最为透彻,基因背景清楚,利用其构建基因工程菌有着得天独厚的条件。以大肠杆菌作为宿主生产木糖醇已有报道,如苏卜利等基于质粒载体系统构建了第一代高产木糖醇菌株,利用质粒载体进行蛋白的表达,通过mrna二级结构的调节,构建了一个能在较高温度下高效可溶表达的质粒;在30℃条件下,酶活是出发菌株的5.68倍。通过敲除葡萄糖磷酸转移酶系统中酶ⅱ组分的ptsg基因,消除菌株的代谢物阻遏效应,使菌株能同时转运葡萄糖和木糖;敲除菌株自身代谢木糖的基因xyla和xylb,阻断木糖的代谢利用;敲除可转运木糖醇的果糖磷酸转移酶系统中酶ⅱ组分的ptsf基因,减少木糖醇的磷酸化;通过一系列的优化,木糖醇生产效率是出发菌株的8.71倍(“大肠杆菌基因工程菌转化半纤维素水解液生产木糖醇的研究”,苏卜利,《浙江大学》,2016年)。
辅酶是一大类有机辅因子的总称,是酶催化氧化还原反应等所必须的辅助因子。它们在反应中充当传递电子、原子或基团的作用,辅酶在一定程度上可以看作是酶反应中的第二底物。作为微生物代谢网络中的一种关键辅因子,通过调节细胞内的辅酶含量和还原型氧化型的比例可定向改变和优化微生物的细胞代谢功能,从而实现代谢流的最大化,这也是增加目标产物的重要方法之一。
辅酶nad(p)h在各类酶催化反应中都起着重要作用,尤其是氧化还原反应中,都需要辅酶nad(p)h作为电子传递参与反应。在产物合成的过程中,会消耗一定量的辅酶。因此随着反应的进行胞内的辅酶含量减少,导致催化效率降低。由于辅酶价格昂贵,通过外源添加的方式进行补充是不现实的。因此通过代谢工程调控微生物的代谢过程,提高胞内nadph的浓度,不仅能有效的提高生产效率降低成本,也能更好地保证生物转化过程的正常进行。从技术经济的角度来看,强化辅酶再生循环意义重大。
目前,基于辅酶nadph的代谢工程改造主要方法为增强ppp的代谢通量。在大肠杆菌中,葡萄糖主要存在两条代谢途径,包括糖酵解途径(emp途径)和磷酸戊糖途径(ppp途径)。其中ppp途径中有2步反应能实现nadph的再生,现有的研究中通过过表达这两步反应中的zwf和gnd基因来提高内源性辅酶nadph,该方法能在一定程度上强化胞内nadph的再生。
但在整合型表达木糖还原酶基因的大肠杆菌中,要实现木糖还原酶酶活与辅酶再生速率的匹配,需要进一步降低葡萄糖的消耗速率。目前未见利用阻断或减少emp途径葡萄糖代谢通量的方法实现辅酶nadph再生强化和减慢葡萄糖利用速率方法的报道。
技术实现要素:
本发明的目的在于提供一种可以大幅度提升木糖醇生产效率的木糖醇基因工程生产菌。
为实现上述目的,本发明采用如下技术方案:
本发明以大肠杆菌w3110为出发菌株,利用基因替换技术对原始菌株基因组中的影响木糖代谢通路及葡萄糖代谢通路中的基因进行替换,以实现木糖还原酶活与辅酶再生速率的匹配,进而提升基因工程菌转化木糖生产木糖醇的效率。
因此,本发明提供了一种木糖醇基因工程生产菌,由大肠杆菌w3110经基因组改造得到,原始大肠杆菌w3110基因组中的ptsg、xylab和ptsf均替换成木糖还原酶基因xr;还包括基因组中pfka、pfkb、pgi、stha中的至少一个替换成木糖还原酶基因xr。
所述的大肠杆菌w3110购自德国微生物菌种保藏中心dsmz,编号为dsm-5911。
本发明对大肠杆菌w3110的改造包括:
(1)在大肠杆菌细胞内存在代谢木糖的途径:木糖经过木糖异构酶(xyla)生成木酮糖,木酮糖在木酮糖激酶(xylb)的作用下生成5-磷酸木酮糖,5-磷酸木酮糖进入磷酸戊糖途径被代谢掉。同时果糖的磷酸转移酶(ptsf)途径可能也参与了木糖醇的转运,使木糖醇进入细胞的同时被磷酸化,而磷酸化的木糖醇对细胞有毒害作用,因此将基因组中xylab和ptsf替换成木糖还原酶基因xr,阻断木糖的代谢和木糖醇的磷酸化的同时增加木糖还原酶的表达,有利于工程菌高效转化木糖生产木糖醇。
(2)大肠杆菌利用糖时存在葡萄糖效应,即在有葡萄糖存在时,大肠杆菌对其他糖如木糖、阿拉伯糖等的利用都会受到严重抑制,因此,本发明将葡萄糖磷酸转移酶基因ptsg替换成木糖还原酶基因xr,降低基因工程菌对葡萄糖的利用速率,消除葡萄糖效应。
但是木糖还原酶还原木糖生成木糖醇的过程中,需要葡萄糖作为辅底物提供反应必需的辅酶nadph。因此,本发明将参与糖酵解途径(emp)的基因pfka、pfkb、pgi或stha进行替换,以阻断或减少emp途径葡萄糖代谢通量,实现ppp途径辅酶nadph再生强化和减慢葡萄糖利用速率。
作为优选,原始大肠杆菌w3110基因组中的ptsg、xylab、ptsf、pfka和pfkb均替换成木糖还原酶基因xr。研究表明,上述5个基因均被替换成木糖还原酶基因xr后,基因工程菌转化木糖生产木糖醇的生产效率达到1.92g/l。
所述木糖还原酶基因xr来源于粗糙脉孢菌,基因序列参见ncbi登录号ncu08384.1。
本发明还提供了一种构建上述木糖醇基因工程生产菌的构建方法,包括以下步骤:
(1)将大肠杆菌w3110基因组中的ptsg、xylab和ptsf均替换成木糖还原酶基因xr,得到第一代基因工程菌;
(2)将第一代基因工程菌基因组中的pfka,pfkb,pgi,stha中的至少一个替换成木糖还原酶基因xr。
步骤(1)和(2)中,利用同源重组技术进行基因替换。
具体地,步骤(1)中,包括:
a.利用特异性引物分别构建替换ptsg、xylab和ptsf基因的ptargetf质粒及对应的含有xr表达模块的修复模板;
b.将替换ptsg基因的ptargetf质粒和修复模板转化入含有pcas质粒的大肠杆菌w3110中,经同源重组,筛选获得基因组中ptsg基因替换成xr表达模块的菌株w3110△ptsg::xr;
再将替换xylab基因的ptargetf质粒和修复模板转化入菌株w3110△ptsg::xr中,经同源重组,筛选获得基因组中xylab基因替换成xr表达模块的菌株w3110△ptsg::xr,△xylab::xr;
然后将替换ptsf基因的ptargetf质粒和修复模板转化入菌株w3110△ptsg::xr,△xylab::xr,经同源重组,筛选获得基因组中ptsf基因替换成xr表达模块的菌株w3110△ptsg::xr,△xylab::xr,δptsf::xr,即第一代基因工程菌;
步骤(2)中,包括:
d.分别构建替换pfka、pfkb基因的ptargetf质粒及对应的含有xr表达模块的修复模板;
e.先将替换pfka基因的ptargetf质粒及对应的修复模板转化入步骤(1)制得的第一代基因工程菌中,经同源重组,筛选获得基因组中pfka基因替换成xr表达模块的菌株wz04△pfka::xr,再将替换pfkb基因的ptargetf质粒及对应的修复模板转化入菌株wz04△pfka::xr中,经同源重组,筛选获得基因组中pfkb基因替换成xr表达模块的菌株,即为所述的木糖醇基因工程生产菌。
所述的xr表达模块中包含启动子p43。研究表明启动子p43有助于木糖还原酶的表达,且不需要诱导剂。以专利文献cn104789586a提供的prc43m质粒为模板,利用特异性引物扩增出以p43为启动子的xr表达模块。
本发明还提供了所述的木糖醇基因工程生产菌在生产木糖醇中的应用。
所述的应用,包括:将所述的木糖醇基因工程生产菌接种于发酵培养基中,30-37℃发酵培养80-90h,发酵过程中保持菌液浓度od600小于20。
所述的发酵培养基可以利用人工配置的以木糖为主要成分的培养液,也可以利用半纤维素水解液为主要原料。
作为优选,发酵初始条件:温度37℃、转速400rpm、通气量0.6-0.8vvm、发酵培养基初始ph为6.5左右,培养过程溶氧控制在30-35%;当菌液浓度od600≥20时,进行补料,补料后发酵条件为:温度30℃,溶氧控制在20-25%。
采用分批补料方式,具体地,od600>20(37℃培养7h左右),进行第一次补料;待葡萄糖完全消耗完,进行第二次补料。
补料液成分,包括:半纤维素水解液或木糖母液(木糖终浓度60g/l)、葡萄糖母液(葡萄糖终浓度为木糖摩尔浓度的1/2)、工业级玉米浆干粉(木糖质量浓度的1/3)。
本发明具备的有益效果:
本发明提供的木糖醇基因工程生产菌,以大肠杆菌w3110为出发菌株,将基因组中的xylab和ptsf均替换成木糖还原酶基因xr,阻断木糖的代谢和木糖醇的磷酸化,提高基因工程菌对木糖的利用速率;将基因组中ptsg以及pfka,pfkb,pgi,stha中的至少一个替换成木糖还原酶基因xr,消除葡萄糖效应的同时通过阻断或减少emp途径葡萄糖代谢通量达到强化ppp途径nadph再生,为木糖还原酶还原木糖生成木糖醇提供必需的辅酶nadph;基因组中的相应基因均替换成木糖还原酶基因xr,木糖还原酶得以高效表达,极大提高了基因工程菌转化木糖生产木糖醇的效率。
附图说明
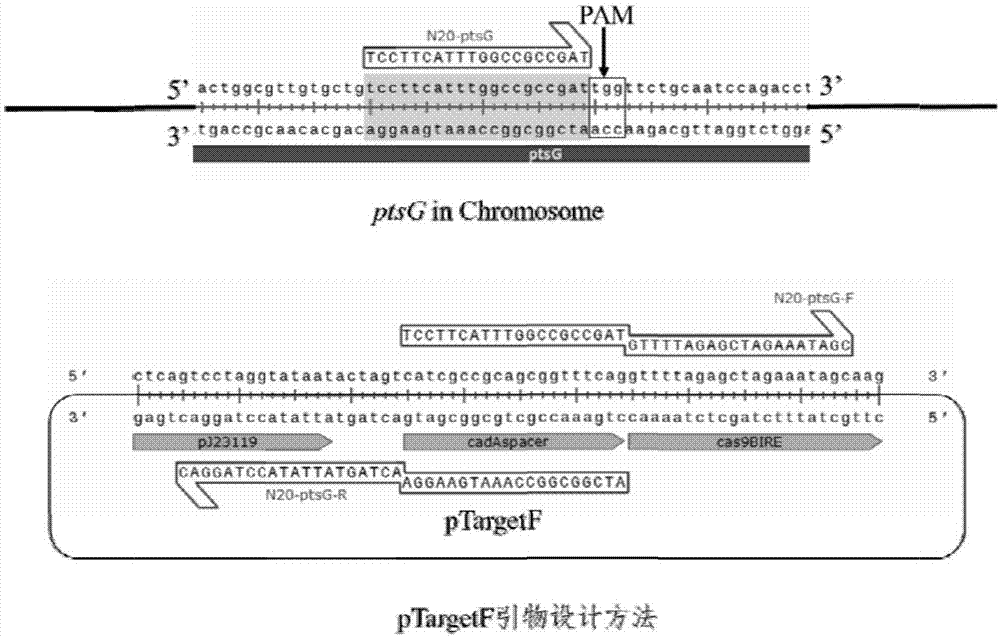
图1为ptsg中靶位点的确定和ptargetf质粒突变引物设计策略。
图2为修复模板的构建方法。
图3为使用crispr技术改造底盘细胞核酸电泳图,其中m:250bpmarker,g和g:xr为使用引物ptsg-u-f和ptsg-d-r扩增得到的片段,g:原始ptsg基因,g:xr:原始ptsg基因替换为xr;ab和ab:xr为使用引物xylab-u-f和xylab-d-r扩增得到的片段,ab:原始xylab基因,ab:xr:原始xylab基因替换为xr;f和f:xr为使用引物ptsf-u-f和ptsf-d-r扩增得到的片段,f:原始ptsf基因,f:xr:原始ptsf基因替换为xr。
图4为葡萄糖主要代谢通路分析。
图5为24h摇瓶发酵后发酵液中糖及糖醇的浓度。
图6为24h发酵后菌浓度od600测定结果。
图7为菌株wz31利用纯糖发酵产木糖醇,其中glucose为葡糖糖,xylose为木糖,xylitol为木糖醇。
图8为利用wz31使用半纤维素水解液和玉米浆干粉进行分批补料发酵,其中glucose为葡糖糖,xylose为木糖,arabinose表示阿拉伯糖,arabitol表示阿拉伯糖醇,xylitol为木糖醇。
具体实施方式
下面结合具体实施例对本发明作进一步说明。
下列实施例中采用的e.coliw3110购自德国微生物菌种保藏中心dsmz,编号为dsm-5911;ptargetf质粒addgene:#62226;pcas质粒addgene:#62225;prc43m质粒来自申请号为201510196843.8,名称为“大肠杆菌基因组整合载体、基因工程菌以及在生产木糖醇中的应用”的专利文献。
实施例1
本实施例提供一种木糖醇基因工程生产菌,实现在整合型木糖还原酶表达菌株中增强胞内nadph再生,提高木糖醇的生产效率。
所述木糖醇基因工程生产菌的构建方法,包括以下步骤:
1、构建替换或敲除目的基因的ptargetf质粒
首先需要在目标基因上找到一个pam位点,即ngg序列,确定对应的n20序列,并将ptargetf质粒上的cadaspacer替换为目的基因的n20序列。以ptsg为例,构建ptargetf-ptsg的质粒构建为例,设计并使用引物n20-ptsg-f和n20-ptsg-r对ptargetf进行全质粒突变pcr,引物设计方法如图1所示。
pcr配方和程序设置如下:
表1全质粒突变pcr体系
由ptargetf质粒图谱可知,质粒总长度为2118bp,为了保证pcr过程中延伸的完整性,本研究中采用长延伸时间的设置。
表2全质粒pcr程序设置
将得到的pcr产物进行dna核酸电泳验证,通过观察在2000bp左右处是否有亮条带,来检验pcr是否成功。
验证之后使用dpni酶进行pcr模板的消化,提高转化子的阳性率。酶消化体系如下:
enzymedpni0.5μl
10*buffer1μl
pcr产物8.5μl。
按照体系加入相应物质后水平震荡混匀,短时快速离心20s,置于37℃水浴或金属浴中60min。然后使用dh5α感受态进行转化。
挑取得到的转化子进行液体培养,使用质粒少量提取试剂盒提取质粒。使用target-check进行测序验证,最终得到突变成功的质粒ptargetf-ptsg。
2、ptsg替换为xr基因修复模板的构建
构建策略如图2所示。
使用e.coliw3110基因组作为模板,以ptsg-u-f和ptsg-u-r,ptsg-d-f和ptsg-d-r为pcr引物,进行常规pcr获得ptsg基因上下游各500bp的同源臂片段;
以prc43m质粒为模板,ptsg-xr-f和ptsg-xr-r为引物pcr得到以p43为启动子的xr表达模块。将得到的pcr产物进行dna核酸电泳并胶回收。
最后使用ptsg-u-f和ptsg-d-r为引物,ptsg上下游同源臂和xr表达模块的等比例混合物为模板,进行重叠延伸pcr,切胶回收对应长度的片段,获得替换ptsg基因的修复模板。
3、基因组基因替换方法操作
1)热激法转入pcas质粒
a.将保存在-80℃的野生型e.coliw3110划线在无抗的固体培养基平板上,过夜37℃培养。挑取单菌落于液体lb培养基中37℃,200rpm培养约10h,转接1ml菌液到装有50ml液体lb培养基的250ml三角瓶中,生长至od600到0.6~0.8,将菌液冰浴10min,按照takara大肠杆菌感受态试剂盒制备化转感受态。
b.将制备好的e.coliw3110感受态置于冰上,融化后在无菌条件下加入10μlpcas质粒,混匀后置于冰浴放置30min。
c.42℃下水浴或金属浴热激90s,立即冰浴2min。
d.加入890μl液体lb或复苏培养基,于30℃,200rpm复苏45min。
e.吸取100μl复苏后的菌液,涂布于含有50mg/l的kanr的固体lb平板上,30℃培养过夜。
f.挑取单克隆的菌落进行pcr或提取质粒验证,获得转化成功的e.coliw3110pcas。
2)电转化相应替换基因的ptargetf和修复模板donordna
a.将1)中得到的大肠杆菌为出发菌株,划线于50mg/l的硫酸卡那霉素抗性的平板上,30℃培养过夜后(后续培养均需加入相同浓度的同种抗生素),挑取单菌落至液体lb中,30℃,200rpm培养10-12h,转接1ml至50ml中lb中,30℃,200rpm培养1h后,加入工作浓度为0.5%灭菌的l-阿拉伯糖进行诱导表达red重组蛋白,继续培养至od600至0.6~0.8(约3h左右),冰浴10min,进行电转感受态的制备。
b.使用灭菌好的10mlep管,将菌液分装后,4℃,4000rpm下离心5min,弃上清。
c.用1ml预冷过的灭菌的10%的甘油重悬,4℃,4000rpm下离心10min,小心的弃去上清。
d.重复c步骤2次。
e.用100μl10%的甘油重悬,转移至灭菌的1.5mlep管中,立即使用或置于-80℃冰箱中备用。
f.使用制备好的感受态或从-80℃中取出预先制备的感受态,冰上放置5min,加入400ng构建好替换相应基因的ptargetf质粒和800ng修复模板。混匀后转移至无菌的2mm电转杯中,冰上放置10min,进行电转化。
g.电转条件:2.5kv,25μf,200ω,电转时间5ms,电转前电转杯杯壁和底座一定要用纸巾擦干,不然极易引起爆杯。同时提取的质粒和回收的修复模板中盐离子残余过高也会引起爆杯。
h.电转结束之后,立即加入1ml液体lb,用移液枪来回吹打混匀后,转移至2ml灭菌的ep管中。在30℃,150rpm下复苏约3h。
i.将复苏后的菌液离心浓缩后全部涂布于含有50mg/l的kanr和50mg/l的specr平板,30℃过夜培养。
j.通常情况下,培养12小时就能有肉眼可见的转化子。如果菌株经过了多次编辑,可能生长时间会有所延长。
4、基因替换验证
对于替换成功的阳性转化子,在替换前后序列长度相差较大的情况下,可采用菌落或菌液pcr扩增目标条带,进行dna核酸电泳,比较基因替换前后的扩增条带大小的方式来进行鉴定。如果替换前后基因大小相差在200bp以内,可采用pcr产物测序的方式来鉴定。
5、同理使用对应引物构建其他替换基因的ptargetf和修复模板,经过3轮的基因组编辑,成功的将木糖还原酶插入到基因组ptsg,xylab,ptsf区域。
经过设计得到改造后的整合型表达木糖还原酶的菌株wz01,wz02,wz03。其中,wz03(e.coliw3110,△ptsg:xr,△xylab:xr,△ptsf:xr)的基因组示意图如图3所示。
插入的木糖还原酶表达模块大小为1449bp。ptsg基因大小为1434bp,因此替换后大小变化并不明显,通过核酸胶电泳图无法断定其结果正确性,使用target-check引物进行pcr产物测序验证,成功将ptsg基因替换为木糖还原酶表达模块。xylab基因共2849bp,替换为xr后减小1400bp,电泳图可判断替换后为阳性转化子;ptsf基因为1692bp,替换后减少243bp,电泳图显示为阳性转化。最终得到的wz03菌株为ptsg、xylab和ptsf全部替换为木糖还原酶基因的表达模块的工程菌。
6、通过葡萄糖主要代谢通路(图4)分析,葡萄糖代谢中ppp途径能产生更多的nadph,因此涉及实验对emp途径的pfka、pfkb、pgi基因和转氢酶stha基因,使用木糖还原酶表达模块进行替换。
以wz03菌株为出发菌株,对可能增强辅酶nadph再生能力的基因pfka,pfkb,pgi,stha进行单个基因的替换,得到菌株wz21,wz22,wz23,wz24。并选择其中效果较优的工程菌进行双替换,得到菌株wz31和wz32。
上述实验中涉及的引物(seqidno.1-57)如表3所示。详细的菌株及其基因组类型如表4所示。
表3引物序列
表4菌株及相关基因型类型
7、对以上菌株进行摇瓶发酵实验,发酵条件如下:
(1)种子液制备
种子液培养:将划线分离的单菌落挑取至灭菌的新鲜液体lb培养基中,于37℃,200rpm培养过夜至生长稳定期。
(2)摇瓶发酵
配制摇瓶发酵培养基,250ml三角瓶中装液量45ml,接种1ml的种子液,37℃,200rpm培养4h,添加5ml灭菌后的混合糖液(含有200g/l木糖和100g/l的葡萄糖),30℃,200rpm下进行摇瓶发酵,定时取样并检测相关参数变化情况。
(3)糖和糖醇的液相检测方法
将所取的样品进行适当浓度的稀释后,使用0.22μm的过滤头进行过滤。利用dionexultimate3000高效液相系统对木糖、葡萄糖、阿拉伯糖、木糖醇和阿拉伯糖醇进行定量检测。检测器:coronachargedaerosoldetector(cad),分析柱:aminexhpx-87c(φ7.8mm×300mm),流动相:超纯水,流速:0.6ml/min,柱温设定:76℃。
取补加糖液24h后的菌液,提取胞内的氧化型和还原型辅酶,并进行浓度测定,方法如下:
1)将菌液置于冰浴中10min,4℃,4000rpm离心15min,浓缩至1ml菌液的od600为30,分离胞内氧化形式和还原形式的辅酶。
2)分别取1ml的菌液,加入:
分离氧化形式:0.5ml的0.3mhcl和50mm的tricine-naoh(ph8.0);
分离还原形式:0.5ml的0.3mnaoh;
3)所有样品60℃保温7min,氧化形式的用0.5ml的0.3mnaoh中和,还原形式的用0.5ml0.3mhcl中和,中和之后每个样品中都需加入0.1ml的1.0mtricine-naoh来维持ph的稳定。
4)4℃,13000rpm离心60min。吸取300μl上清转移至新的ep管中。
5)采用反应液在特定波长下的吸光值来测定辅酶的含量。可采用96孔板和酶标仪进行定量。测定体系为:
氧化形式:40μl样品+40μl0.1mnacl;
还原形式为:80μl样品;
6)加入等体积(80μl)的2*反应原液(1.0mtricine-naoh(ph8.0),4.2mmthiazolylbluetetrazoliumbromide(mtt),40mmedta,1.67mmphenazineethosulfate(pes),和底物(5m乙醇(用于测定nad)或25mm葡萄糖-6-磷酸(用于测定nadp)。
7)混匀之后37℃恒温5min,分别加入工作浓度为10u/ml的醇脱氢酶和0.27u/ml的葡萄糖-6-磷酸脱氢酶(母液以10*浓度进行配制)。
8)37℃下mtt的减少量可用570nm的酶标仪来检测。数据对照标准曲线。
测得的辅酶还原型和氧化型的比例如下表5所示。
表5不同基因替换后还原型与氧化型辅酶的比例
相比于对照组wz03,替换相关基因后胞内nadph/nadp+的含量均有一定量的提高,对发酵液中的糖及糖醇进行测定,结果如下图5所示。
由表5和图5分析可知,经过改造后,胞内nadph的再生能力得到了强化,消耗相同摩尔葡萄糖能产生更多的nadph,同时提高了胞内nadph/nadp+的比例,维持较高的辅酶浓度为木糖还原酶提供充足的还原力。
对各株菌株的生长od600进行测定,结果如图6所示。经改造后的菌株,4个木糖还原酶拷贝数下,辅酶nadph/nadp+比例越高,菌株的木糖醇产量也越高。5个木糖还原酶拷贝数下,wz31与wz32的辅酶nadph/nadp+比例相当,但wz31有更高的木糖醇产量。结合图6,wz32由于改造后菌株的生长能力受到破坏,24h后菌浓度仅有出发菌株wz03的40%左右,这是其木糖醇产量较低的主要原因。
将菌株wz31进行纯糖摇瓶发酵实验,分别对补加糖后10h,20h及30h进行取样,测定发酵液中的糖和糖醇浓度及od600的测定,结果如图7所示。
8、使用半纤维素水解液和玉米浆干粉进行发酵,采用wz31菌株,在不添加无抗生素及诱导剂的条件下,使用15l发酵罐进行木糖醇发酵,方法如下:
(1)一级种子液培养
将保藏的菌种划线于固体培养基平板上,37℃恒温下过夜培养。挑取单菌落于液体lb中,37℃,200rpm培养约12h。
一级种子液培养基配方(l-1):蛋白胨10g,酵母粉5g,氯化钠10g,固体培养基另加2%的琼脂粉。
(2)二级种子液培养
按体积比0.5~1%的比例转接一级种子液至二级种子液培养基中,37℃,200rpm培养7h。
二级种子液培养基配方(l-1):酵母粉7.5g,蛋白胨7.5g,氯化钠10g,葡萄糖20g。
(3)发酵过程控制
接种前先用氨水将发酵培养基的ph调节至6.5左右,按发酵培养基体积的10~15%接种培养好的二级种子液,培养温度:37℃,通气量控制在0.6vvm,初始转速控制在400rpm。调节转速和溶氧联控,保持发酵罐溶氧为30-35%,间隔一定时间取样测定菌浓od600,并在od600≥20时进行补料操作,接种和补料均采用火焰法。
补料方式:分批补料
od600>20(37℃培养7h左右),进行第一次补料。第一次补料后,葡萄糖完全消耗完,进行第二次补料。
补料液成分及终浓度:半纤维素水解液或木糖母液(木糖终浓度60g/l)、葡萄糖母液(葡萄糖终浓度为木糖摩尔浓度的1/2)、工业级玉米浆干粉(木糖质量浓度的1/3)。
补料后条件控制:溶氧控制在20-25%,温度控制:30℃。
补料后定时取样并检测发酵过程中葡萄糖、木糖、阿拉伯糖以及阿拉伯糖醇和木糖醇的浓度,监测整个工程中菌株的生长情况。最终发酵结果如图8所示。
由图8可知,使用半纤维素水解液作为底物,玉米浆干粉作为碳源,最终经过84h分批补料发酵,最终获得161.03g/l的木糖醇,木糖、阿拉伯糖及葡萄糖基本完全消耗,副产物阿拉伯糖醇仅为1.63g/l。木糖醇的生产效率为1.92g/l/h。
以上所述的实施例对本发明的技术方案和有益效果进行了详细说明,应理解的是以上所述仅为本发明的具体实施例,并不用于限制本发明,凡在本发明的原则范围内所做的任何修改、补充和等同替换等,均应包含在本发明的保护范围之内。
序列表
<110>浙江大学
<120>一种木糖醇基因工程生产菌及其构建方法和应用
<160>57
<170>siposequencelisting1.0
<210>1
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>1
tccttcatttggccgccgatgttttagagctagaaatagc40
<210>2
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>2
atcggcggccaaatgaaggaactagtattatacctaggac40
<210>3
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>3
gatcggttactggtggaaactgactc26
<210>4
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>4
cactcaataaagctgagctcaattgagagtgctcctgagtatggg45
<210>5
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>5
actcaggagcactctcaattgagctcagctttattgagtggatga45
<210>6
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctccccaacgtcttacggattaccggtttattgactaccggaag44
<210>7
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>7
ggtagtcaataaaccggtaatccgtaagacgttggggagactaa44
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>8
ggtggatgggacagtcagtaaagg24
<210>9
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>9
tgcgcaagagtgcactttgcgttttagagctagaaatagc40
<210>10
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>10
gcaaagtgcactcttgcgcaactagtattatacctaggac40
<210>11
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>11
gatatctcgatctttttgccagcgttc27
<210>12
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>12
cactcaataaagctgagctcattgaactccataatcaggtaatgc45
<210>13
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>13
acctgattatggagttcaatgagctcagctttattgagtggatga45
<210>14
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>14
ggtcaggcaggggataacgttaccggtttattgactaccggaag44
<210>15
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggtagtcaataaaccggtaacgttatcccctgcctgaccgggtg44
<210>16
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>16
gagcgaaacaaacgcatttgaccaaac27
<210>17
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>17
agcaccggttctggcttcatgttttagagctagaaatagc40
<210>18
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>18
atgaagccagaaccggtgctactagtattatacctaggac40
<210>19
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>19
gactggatgactcgcctgcgtagtcag27
<210>20
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>20
cactcaataaagctgagctctatgcctctcctgctgtcagttaaa45
<210>21
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>21
ctgacagcaggagaggcatagagctcagctttattgagtggatga45
<210>22
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>22
gccctgtaacacaccttttataccggtttattgactaccggaag44
<210>23
<211>44
<212>dna
<213>人工序列(artificialsequence)
<400>23
ggtagtcaataaaccggtataaaaggtgtgttacagggcagaaa44
<210>24
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>24
tgcgccctactccaccaggcgtaaa25
<210>25
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>25
cacaacgaattcacagccacgttttagagctagaaatagc40
<210>26
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>26
gtggctgtgaattcgttgtgactagtattatacctaggac40
<210>27
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>27
gtgactgacgaatcaccacg20
<210>28
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>28
cactcaataaagctgagctctgatttcggaaaaaggcagattcct45
<210>29
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>29
tctgcctttttccgaaatcagagctcagctttattgagtggatga45
<210>30
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>30
tccaaagttcagaggtagtcccggtttattgactaccggaagcag45
<210>31
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>31
tccggtagtcaataaaccgggactacctctgaactttggaatgca45
<210>32
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>32
gtttgcattgggaatcggcatc22
<210>33
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>33
gtcctaggtataatactagtccaaaaagaactcagtgcgc40
<210>34
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>34
gctatttctagctctaaaacgcgcactgagttctttttgg40
<210>35
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>35
gaagaagtattactggcgttccc23
<210>36
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>36
cactcaataaagctgagctccatttcctcctataggctgatttca45
<210>37
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>37
tcagcctataggaggaaatggagctcagctttattgagtggatga45
<210>38
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>38
atgctgggggaatgtttttgccggtttattgactaccggaagcag45
<210>39
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>39
tccggtagtcaataaaccggcaaaaacattcccccagcattgggg45
<210>40
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>40
gactgattgcctgccacatgatg23
<210>41
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>41
ctgccaacatgttcgagttcgttttagagctagaaatagc40
<210>42
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>42
gaactcgaacatgttggcagactagtattatacctaggac40
<210>43
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>43
gatccattttcagccttggcac22
<210>44
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>44
cactcaataaagctgagctctagcaatactcttctgattttgaga45
<210>45
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>45
aaatcagaagagtattgctagagctcagctttattgagtggatga45
<210>46
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>46
ggcctacatatcgacgatgaccggtttattgactaccggaagcag45
<210>47
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>47
tccggtagtcaataaaccggtcatcgtcgatatgtaggccggata45
<210>48
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>48
cagaagtcgccgcaagcgcagatatg26
<210>49
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>49
gaatatgcgtcgatcttccggttttagagctagaaatagc40
<210>50
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>50
cggaagatcgacgcatattcactagtattatacctaggac40
<210>51
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>51
gttcgtctacgtcgcggaaatg22
<210>52
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>52
cactcaataaagctgagctcggtagggcttacctgttcttataca45
<210>53
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>53
aagaacaggtaagccctaccgagctcagctttattgagtggatga45
<210>54
<211>45
<212>dna
<213>人工序列(artificialsequence)
<400>54
atggccatttcgataaagttccggtttattgactaccggaagcag45
<210>55
<211>46
<212>dna
<213>人工序列(artificialsequence)
<400>55
tccggtagtcaataaaccggaactttatcgaaatggccatccattc46
<210>56
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>56
gaagcacagacccaccagttactg24
<210>57
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>57
gaagggagaaaggcggacag20
- 还没有人留言评论。精彩留言会获得点赞!