检测低频变异用的接头、接头混合物及相应方法与流程
本发明涉及生物信息领域,尤其涉及基因检测,具体是指一种适用于检测低频体细胞变异和单链损伤变异的接头及其应用方法。
背景技术:
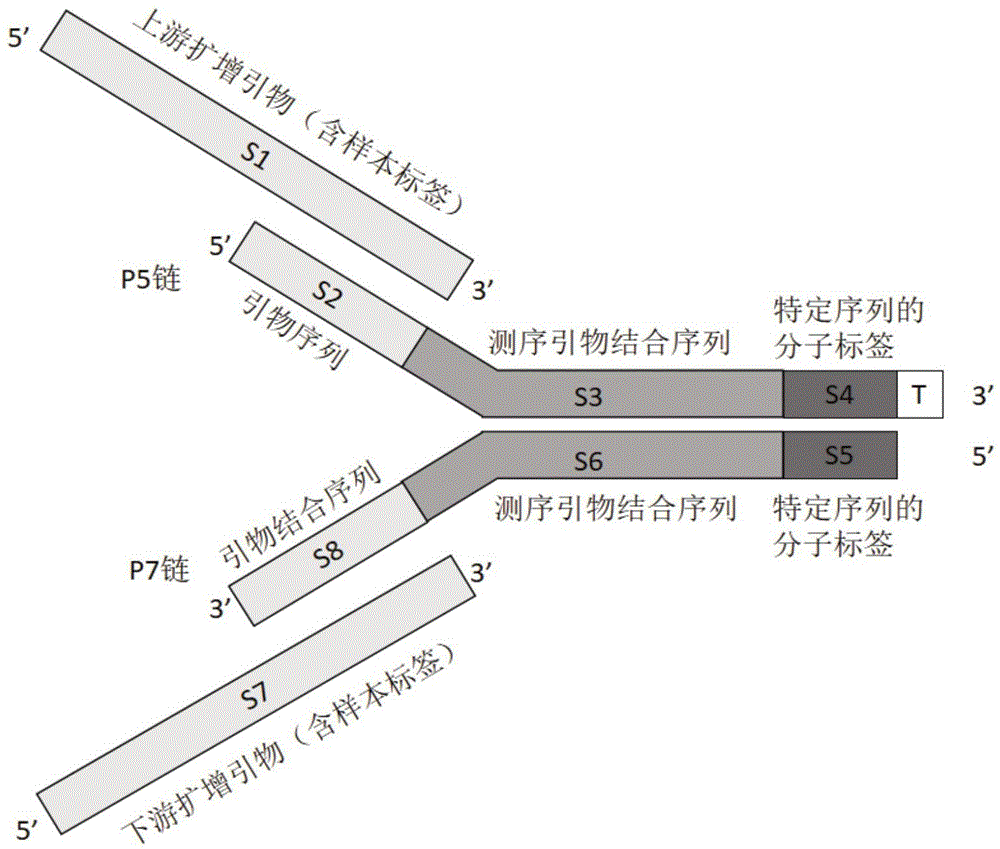
:相比较一代测序技术,二代测序技术凭借着同时并行对数百万乃至上百亿条序列进行测序的优异性能,大幅降低了测序成本,迅速推动了其在科研、法医和临床等各领域的应用。例如,孕妇外周血浆中游离dna中含有胎儿遗传信息,通过对孕妇的血浆游离dna(cell-freedna,cfdna)进行低深度全基因组测序,可以检测胎儿染色体异常,无创产前筛查迅速推动了基因检测行业的发展。而随着美国“精准医学计划”的提出,国内的肿瘤基因检测服务行业也在快速发展。近年来有研究表明凋亡或坏死的肿瘤细胞会将胞内的小片段dna释放进入血液循环系统,这些dna即是循环肿瘤dna(circulatingtumordna,ctdna)。相较于通过手术、组织活检获取肿瘤标本的传统方式,ctdna检测技术能很好地克服肿瘤时空异质性,重复检测方便,是目前液体活检技术的主流方向。但是相比较于胎儿dna占孕妇血浆cfdna中的比例(孕12周时可达4%以上),肿瘤患者血浆中的ctdna占cfdna总量的比例非常低,根据癌种和病程的不同,大部分ctdna的比例仅占0.1%~1%,因此,ctdna的检测需要更高的灵敏度和特异性。而在目前的二代测序实验流程中,在预文库制备、杂交捕获及测序过程中不可避免的会引入一些扩增和测序的错误或杂交时间过久导致的损伤,导致低频突变与背景噪音无法区分而造成假阳性或假阴性。为了提高测序纠错能力,科学家提出了一些新的方法,目前主要有两类:单链环化法和分子标签法。1、单链环化法该方法为安可济生物提出,命名为firefly技术,主要原理是先将片段大小约170bp的双链cfdna变性为单链dna,然后将单链dna连接成环状,用单侧的目的基因特异性引物进行单向rca滚环扩增,保证每个dna片段都被串联复制多次,然后引入p5/p7接头进行双端pe150bp测序,保证每个插入片段至少被重复测序两次及以上,通过重复测序来确认检测到的变异是否是真的变异。该方法的优势在于滚环扩增分子始终复制于原始分子,不会积累错误;通过多重pcr来富集靶向区域,不需要合成探针进行捕获,简化了操作;相比分子标签法,测序成本降低。该方法劣势在于单链成环的效率,多重pcr的效率都有影响,引物对数量有限制,控制产物大小比较困难,而且无法识别双链模板中的单链损伤。2、分子标签法分子标签(uniquemolecularidentifier,umi)是目前广泛使用的方法,其原理是给每一条原始dna模板加上一段特有的标签序列,文库经过pcr扩增后上机测序,在数据分析时,可以根据标签序列识别同一dna模板扩增出的多个片段,根据检出的变异在这多个片段上的分布情况,可以分辨哪些是pcr扩增、杂交捕获及测序过程中的随机错误造成的假阳性变异,哪些是患者真正携带的变异,从而提高检测灵敏度和特异性。根据分子标签标记位置的不同,可分为单链分子标签和双链分子标签。单链分子标签只能标记单链dna分子,或分别标记双链dna的两条单链,不能同时标记双链,一般适用于单链dna建库或当分子标签位于y型接头的其中一条突出端时,其优势是可以用相对较少的测序量显著减少假阳性结果,而劣势在于无法利用原始dna双链模板的另一条互补链信息进一步纠错,如果pcr扩增错误发生在指数扩增的早期循环,或者石蜡包埋样本dna(ffpedna)中含有单链损伤,则仅靠单链分子标签无法检出,需要借助双链分子标签技术才能检出。双链分子标签技术由michaelw等人于2012年发表文章提出,其特征是双链y型接头末端有12个随机核苷酸n作为分子标签,分子标签后面是4个已知序列的核苷酸作为分子标签的识别标签,识别标签之后有个突出碱基a,该接头与末端加上t碱基的双链dna分子进行ta连接,于是每个双链dna分子两端各加上了一个独特的分子标签,从而可区分不同来源的原始模板,并且可以利用正义链和反义链的配对原则进行进一步的纠错。michaelw等人于2014年对该方法进行改进,使接头的突出碱基为t,可适应目前主流的建库方法。但该方法涉及多步酶促反应和多步纯化步骤,接头制备过程较为繁琐,最终接头浓度很难精确定量,质控步骤对实验条件要求较高,接头制备的成功率不高,影响了双链分子标签技术的应用和推广。技术实现要素:本发明的目的是克服了上述现有技术的缺点,提供了一种带有新型双链分子标签的y型接头,该双链分子标签序列为特定核苷酸序列的组合,含有该双链分子标签的接头制备方法十分简便,只需要将多对含有特定核苷酸序列分子标签的接头分别退火后再等比例混合即可。使用该接头混合物对含有低频变异和单链损伤的样本进行文库制备和高通量测序,再结合本发明公开的生物信息分析流程和算法,可有效提高变异检测的准确性。为了实现上述目的,本发明一方面提供了一种检测低频变异用的接头,其具有如下构成:所述的接头包括两条互补dna单链,其中一条链p5链从5’端到3’端依次包括:与上游扩增引物部分重合的序列;与上游测序引物结合的序列;特定核苷酸序列组合的分子标签;1个突出碱基t;另一条链p7链从5’端到3’端依次包括三部分:与p5链中分子标签反向互补的分子标签;与下游测序引物结合的序列;与下游扩增引物结合的序列。较佳地,在p5链中,与上游扩增引物部分重合的序列、与上游测序引物结合的序列可以有部分序列重合,并且3’端硫代修饰;在p7链中,与下游测序引物结合的序列、与下游扩增引物结合的序列可以有部分序列重合甚至完全重合该链,并且5’端磷酸化修饰。较佳地,所述的上游扩增引物和下游扩增引物包括样本标签。较佳地,所述的接头为y型截短型接头;所述的分子标签的长度为3~12bp。较佳地,p5链为如seqidno:3所示的核苷酸序列,p7链为如seqidno:4所示,所述的上游扩增引物为如seqidno:1所示的核苷酸序列,所述的下游扩增引物为如seqidno:2所示的核苷酸序列。本发明提供了一种接头混合物,所述的接头混合物包括按比例混合的至少八种所述的接头。较佳地,所述的接头混合物中的双链分子标签组合的纵向同个位置上,四种碱基同时存在,优选地,从纵向上,分子标签组合中四种碱基a:t:g:c的比例接近1:1:1:1;从横向上,每个分子标签中避免出现连续4个及以上相同碱基的出现,优选地,在分子标签起始位置要避免连续2个及以上碱基g的出现。较佳地,所述的接头混合物中各接头的双链分子标签互相之间至少有3个及3个以上核苷酸序列的差异;优选地,所述的接头混合物中各接头的双链分子标签的长度不能完全相同;优选地,所述的接头混合物中各接头按等比例混合,或者,根据测序数据中实际测得的比例再调整各接头混合的比例。本发明提供了一种用于检测低频体细胞变异的方法,包括以下步骤:(1)分别合成所述的接头混合物中每对接头的p5链和p7链,退火形成y型接头,并按比例混合形成接头混合物;(2)将带有双链分子标签的接头混合物与游离dna片段样本进行连接反应,得到连接产物,并用带样本标签的上下游扩增引物对连接产物进行pcr扩增,得到扩增产物;(3)对扩增产物中的目的片段进行杂交捕获,得到靶向捕获文库,对靶向捕获文库进行高深度的双端测序,根据双端的样本标签对不同样本进行数据拆分;(4)对测序数据进行质控处理,去除低质量碱基、低质量读段和污染的接头序列,同时对readpair的重叠部分根据碱基质量进行纠错处理,得到干净的数据;(5)将reads比对到参考基因组上,在每一个的比对位置上,将具有相同分子标签序列、相同cigar标签和相同比对方向的readpairs归类为一个readpairsfamily。(6)对于每个readpairsfamily,根据贝叶斯定理精确计算并确定单链一致性序列sscs,重新计算碱基质量值,减少测序错误;(7)将生成的sscs寻找分子标签序列能互补的sscs,进一步生成双链一致性序列dscs,同时保留不能形成dscs的sscs,移动比对位置到下一个碱基,重复步骤(5)~(7);(8)将最终一致性序列与参考基因组比对,进行变异检测,得到初始变异集合,对上述变异集合进行注释,过滤比对错误、人群数据库、编码区域,获得最终低频真实可靠的体细胞突变。较佳地,在步骤(1)中,退火反应的条件为:95℃5min后,以0.02℃/sec的降温速度缓慢降温至25℃,或者95℃5min后,关闭pcr仪,静置直至温度降至室温;优选地,在步骤(2)中,游离dna片段样本的投入总量为20~33ng;优选地,在步骤(3)中,测序深度为10,000-30,000x;双端的样本标签为不同样本间的双端样本标签序列均不相同的udi;优选地,在步骤(5)中,降低readpairfamilysize读段簇大小的阈值为2,生成更多readpairfamilies,同时利用能与其他sscs形成dscs的只包含一条read的readpairfamily用于生成dscs,在最终输出的fastq文件中,同时保留sscs和dscs序列以及对应的碱基质量值;优选地,在步骤(6)中,根据贝叶斯定理,确定先验概率的方法为,如果观测到的碱基与可能的真实碱基一致,则先验概率为1-10-q/10,否则为10-q/10/3,q为碱基质量值,此分布用p(b,bi,qi)描述;对于4种可能的碱基,根据下述公式一计算后验概率,对于sscs上的每个碱基位置,使用readpairsfamily对应的碱基/质量值对(bi,qi),计算一致性碱基i为b时的概率(b∈{a,c,g,t}),概率值最大的碱基类型即为真实碱基,由此确定每个位置的真实碱基;同时根据下述公式二,使用真实碱基的后验概率值重新计算碱基质量,得到纠错之后的一致性序列reads。qc=-10log10(1-p[i=bc|{(bi,qi)}])公式二;优选的,在步骤(7)中,两条sscs生成dscs时,如果对应位置碱基相同,则保留此碱基,否则将此位置碱基改为n。附图说明图1显示了本发明中接头序列和扩增引物的结构示意图。图2显示了duplexadapter#10退火产物的agilent2100bioanalyzer的质检图。图3显示了接头混合物的agilent2100bioanalyzer的质检图。图4显示了生成一致性序列过程的流程图。图5显示了不同测序深度和检测下限条件下的检测灵敏度。图6显示了不同测序深度和检测下限条件下的检测特异性。具体实施方式为了能够更清楚地描述本发明的技术内容,下面结合具体实施例来进一步的描述。本发明的提供了一种带有双链分子标签的y型接头,该双链分子标签序列为特定核苷酸序列的组合,含有该双链分子标签的接头制备方法十分简便,只需要将多对含有特定核苷酸序列分子标签的接头分别退火后再等比例混合即可。使用该接头混合物对含有低频变异和单链损伤的样本进行文库制备和高通量测序,再结合本发明公开的生物信息分析流程和算法,可有效提高变异检测的准确性。本发明提供的带有双链分子标签的y型接头混合物,该接头混合物为一组接头的等比例混合物,接头混合物中的每种接头由两条dna单链退火而成,其中一条链命名为p5链,另一条命名为p7链。如图1所示,p5链从5’端到3’端按功能性依次包括四部分:与上游扩增引物s1有部分重合的序列s2,与上游测序引物结合的序列s3,以及特定核苷酸序列组合的分子标签s4,还有1个突出碱基t,该链3’端有硫代修饰,其中序列s2和序列s3可以有部分序列重合甚至完全重合。如图1所示,p7链从5’端到3’端按功能性依次包括三部分:与分子标签组合s4反向互补的序列s5(5’端磷酸化修饰),与下游测序引物结合的序列s6,以及与下游扩增引物s7部分结合的序列s8,其中序列s6和序列s8可以有部分序列重合甚至完全重合。其中,p5链的s3+s4序列和p7链的s5+s6序列有部分序列反向互补,退火后可形成y型接头。p5链s9和p7链s10分别合成后进行退火处理,然后将退火后的各种接头按等比例混合,形成接头混合物。接头混合物中,各种接头间除了双链分子标签即s4和s5不同外,其他序列皆相同。接头混合物中,双链分子标签组合的序列是特定核苷酸序列,而不是随机核苷酸序列,因此和现有技术不同的是,在分子标签序列附近不需要再加入对分子标签的识别序列。需要注意的是,出于成本考虑,本发明的接头不包含用来区分不同样本的样本标签,为截短型接头。样本标签是通过上游扩增引物s1和下游扩增引物s7在pcr扩增过程中引入,上、下游扩增引物上还包含了与测序流动槽内序列互补的序列用来进行成簇反应,因此本发明的截短型接头和上下游扩增引物配合使用。上游扩增引物s1的序列如seqidno:1所示:5’-aatgatacggcgaccaccgagatctacacnnnnnnnnacactctttccctacacgac-3’;下游扩增引物s7的序列如seqidno:2所示:5’-caagcagaagacggcatacgagatnnnnnnnngtgactggagttcagacgtgt-3’。其中,“nnnnnnnn”序列分别是文库p5端和p7端的样本标签,都是8个核苷酸长度的组合,p5端和p7端的样本标签序列各不相同,并且不同样本间的样本标签也各不相同。样本标签用来区分一同上机测序的不同样本,因为部分型号的测序仪容易发生样本间串扰,因此有必要对样本加上双样本标签。p5链全长s9序列如seqidno:3所示:5’-acactctttccctacacgacgctcttccgatctnnnnnnt-3’;p7链全长s10序列如seqidno:4所示:5’-nnnnnngatcggaagagcacacgtctgaactccagtcac-3’。其中,“nnnnnn”为分子标签序列,长度为3~12个核苷酸,对每对接头来说是特定的序列而不是随机序列。对于接头混合物中的同一对接头,分子标签为特定序列,p5链和p7链的分子标签序列为反向互补序列,退火成双链分子标签时可完全配对。p5链3’端有硫代修饰,p7链5’端有磷酸化修饰。双链分子标签的长度为3~12bp,理论上可以产生43+44+45+…+411+412种不同的双链分子标签。优选的,双链分子标签组合是色平衡的,即测序的每个循环中每个通道都能够检测到信号,也就是说,在接头混合物中,双链分子标签组合的同个位置上,四种碱基同时存在。高通量测序仪多数是双色激光的,有些是四通道的,用四种不同的光学通道检测四种核苷酸;有些是双通道的,a和c各为一个荧光,t有两个荧光,g都没有荧光。优选的,双链分子标签组合中碱基复杂度是平衡的,从纵向来看,每组分子标签组合中四种碱基a:t:g:c的比例接近1:1:1:1;从横向来看,每个分子标签中避免出现连续4个及以上相同碱基的出现;尤其对于双通道测序仪,在分子标签起始位置要避免连续2个及以上碱基g的出现,以避免碱基组成不平衡导致测序进行到这些碱基时,软件对测序信号的处理出现障碍,不能准确地识别这些碱基。优选的,接头混合物中双链分子标签间的编辑距离(editdistance)不小于3,即双链分子标签互相之间至少有3个及3个以上核苷酸序列的差异,即至少要发生3次及以上的测序错误才会导致分子标签的串扰。优选的,接头混合物中双链分子标签的数目不少于8个,对于双端测序来讲,至少产生8x8=64种组合,由于基因组dna片段中断裂于同一参考基因组起始和终止位置的概率很低,用较少的组合数已经能够区分是否来自同一原始模板分子。优选的,尽管接头混合物中各接头按等比例混合,但接头的连接反应有序列偏好性,因此实际测得的各分子标签比例并不相等,需要根据测序数据中实际测得的比例再返回来对接头混合的比例进行调整。优选的,接头混合物中各个双链分子标签的长度不能完全相同,分子标签末端都是1个突出碱基t,如果分子标签长度都一致,则在测序的之后一个循环中测得的碱基全部都是t,碱基严重不平衡,会降低测序数据质量。本发明提供了一种使用该双链分子标签编码技术检测肿瘤血液样本中低频体细胞变异的方法,包括以下步骤:(1)按上述接头的表述特征,分别合成接头混合物中每对接头的p5链和p7链,用退火缓冲液稀释至特定浓度;将p5链和p7链按摩尔数1:1比例混合,进行退火反应,形成y型双链接头;(2)将已经退火的各对接头按等摩尔数混合,形成接头混合物,稀释至工作液浓度;(3)取一定数量的从肿瘤血液样本中提取的cfdna,与带有双链分子标签的接头按一定比例进行连接反应,得到连接产物;(4)用带样本标签的上下游扩增引物对连接产物进行pcr扩增,得到扩增产物;(5)用探针对扩增产物中的目的片段进行杂交捕获,得到靶向捕获文库;(6)对靶向捕获文库进行高深度的双端测序,根据双端的样本标签对不同样本进行数据拆分;(7)对拆分得到的样本测序数据首先进行质控处理,去除低质量碱基、低质量读段以及污染的接头序列,同时对readpair的重叠部分根据碱基质量进行纠错处理,得到干净的数据;(8)将上述reads比对到参考基因组上,在每一个比对位置上,将具有相同分子标签序列,相同cigar标签和相同比对方向的readpairs归类为一个readpairsfamily;(9)对于每一个readpairsfamily,使用贝叶斯定理确定碱基序列,生成一条sscs。确定先验概率的方法为,如果观测到的碱基与可能的真实碱基一致,则先验概率为1-10-q/10,否则为10-q/10/3,q为碱基质量值,此分布用p(b,bi,qi)描述;对于4种可能的碱基,根据下述公式一计算后验概率,对于sscs上的每个碱基位置,使用readpairsfamily对应的碱基/质量值对(bi,qi),计算一致性碱基i为b时的概率(b∈{a,c,g,t}),概率值最大的碱基类型即为真实碱基,由此确定每个位置的真实碱基。同时根据公式二,使用真实碱基的后验概率值重新计算碱基质量,得到纠错之后的一致性序列readpair。qc=-10log10(1-p[i=bc|{(bi,qi)}])公式二(10)将所述sscs寻找分子标签序列能互补的sscs,进一步生成dscs。同时保留不能形成dscs的sscs。移动比对位置到下一个碱基,重复步骤(8)~(10);(11)将所述最终一致性序列与参考基因组比对,进行变异检测,得到初始变异集合;(12)对上述变异集合进行注释,过滤比对错误、人群数据库、编码区域,获得最终低频真实可靠的体细胞突变。在步骤(1)中,退火缓冲液的成分含有tris、edta、nacl等;退火的反应条件为95℃5min,然后以0.02℃/sec的降温速度缓慢降温至25℃;退火的反应条件为95℃5min,然后关闭pcr仪,静置直至温度降至室温。在步骤(3)中,ctdna的抽提试剂盒为qiaampcirculatingnucleicacidkit(qiagen);ctdna的总量为20ng~33ng,即6,000~10,000个基因组单倍体拷贝;接头与cfdna片段的比例为100:1~200:1;连接反应后进行纯化反应,使用的纯化磁珠为agencourtampurexp(beckmancoulter)。在步骤(4)中,上游扩增产物的核苷酸序列如seqidno:1所示,下游扩增产物的核苷酸序列如seqidno:2所示;pcr扩增循环数在5~10个循环,在保证足量扩增产物前提下尽量减少循环数。在步骤(5)中,探针是生物素标记的;探针可以是dna探针,也可以是rna探针;探针的长度在50~120nt;投入杂交捕获的扩增产物总量为500~750ng。在步骤(6)中,双端测序读长为2x75bp或者2x150bp;测序深度为10,000x-30,000x;双端的样本标签为uniquedualindex(udi),即不同样本间的双端样本标签序列都不相同。在步骤(8)中,常规方法需要每个readpairsfamily包含至少3对readpairs才是有效的readpairsfamily,才被用于生成sscs,两个分子标签序列互补的sscs形成的dscs序列才会被保留进一步用于变异检测,数据利用率较低,而本发明中含有2对readpair的readpairsfamily即为有效的readpairsfamily,同时如果一个readpairsfamily只含有1对readpair,但是能够与另外的sscs序列互补形成dscs,此种readpairfamily也作为有效数据被保留,从而大大提高数据利用率。在步骤(9)中,对于每一个readparisfamily,生成一条sscs的过程中,每个位置上碱基的确定,一般方法采用大多数规则,即计算此位置上每种碱基(a,t,g,c)的比例,如果某种碱基比例大于70%,认为此位置的真实碱基即为该碱基,同时使用其中较高的碱基质量值作为最终碱基质量值,此方法比较简单,而本发明根据贝叶斯定理计算每种碱基是真实碱基的概率,概率最大即为真实碱基,根据此概率计算碱基质量值,使一致性序列的碱基更加准确可靠。本发明的主要优点包括:本发明的接头含有双链分子标签,因此应用本发明技术进行低频突变检测时,相比较环化串联重复确认法和单链分子标签法,可以利用原始模板的正义链和反义链进一步纠正扩增早期错误和杂交捕获引起的单链损伤错误;本发明接头中的双链分子标签为一组特定的核苷酸序列组合,而不是随机的核苷酸序列,因此本接头的制备方法十分简便和经济,只需要简单退火和混合即可,不需要像现有技术一般要进行多步酶促反应和纯化反应;本发明接头中的双链分子标签为一组特定的核苷酸序列组合,接头混合物中最少有8种分子标签构成双端8x8=64种组合,即可有效区分在参考基因组序列上具有相同起始和终止位置的测序序列是否来自同个原始模板分子,而不需要像现有技术一般有412x412=2.8e14种组合;本发明的接头不需要分子标签的识别序列,同时分子标签的长度小于现有技术的12个核苷酸序列,因此应用本发明技术进行测序时,增加了有效读长,降低了测序成本;本发明合理地降低readpairfamily阈值为2,并且巧妙地利用了能与其他sscs形成dscs序列的只含有一条read的readpairfamily,大大提高了原始测序数据的利用率;本发明使用贝叶斯定理,准确地计算每个位置上每种碱基的概率,选取概率值最大的碱基为一致性碱基,并且根据此概率值重新计算碱基质量值,可以非常有效地降低测序仪器的随机测序错误和pcr扩增过程带入的错误。应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。本发明用特定核苷酸序列代替了现有的随机核苷酸序列,并对特定核苷酸序列进行了优化,应用该技术和自主开发的生物信息学分析算法,可以更有效检测肿瘤血液样本中的低频体细胞变异。下面结合附图和实施例,对本发明的具体实施方式作进一步的详细阐述。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。实施例1含有7bp+8bp双链分子标签接头的制备设计带有双链分子标签的接头p5链和p7链的序列:该接头混合物中设计包含16种分子标签,如下表1所示,16种分子标签中有7对为7个核苷酸长度,其余9对为8个核苷酸长度,两者长度错开1位。分子标签组合的前7个核苷酸序列中,纵向相同位置的碱基比例a:g:t:c=1:1:1:1,且分子标签之间至少有3个核苷酸序列的差异。表1合成的管子12,000g离心1分钟使干粉甩到底部,小心打开管盖,用lowtebuffer(10mmtris-hcl(ph8.0),0.1mmedta)将干粉稀释至250μm,振荡混匀并置于4℃冰箱过夜。按表2配置5xannealingbuffer。表2将每对接头按以下表3的体系进行混合,接头的终浓度为100μm。表3将pcr管放置于geneamp9700pcr仪(appliedbiosystems)上,95℃孵育5分钟,然后直接关闭pcr仪,静置1小时后取出。每管退火产物取1μl,稀释后用agilent2100bioanalyzer进行质检。以其中#10接头为例,退火产物的峰型如图2所示。因为是y型接头,所以峰型会比预期片段大小偏大一些。16管退火产物,每管取相同体积进行混合,取1μl混合物稀释后用agilent2100bioanalyzer进行质检,退火产物的峰型如图3所示。将制备好的双链分子标签接头稀释至工作液浓度,小体积分装后-20℃冻存备用,避免反复冻融。实施例2检测标准品和肿瘤血液样本中低频体细胞变异准备标准品dna:用正常的细胞系dnana18536对horizondiscovery的标准品hd701和hd753进行不同倍数的稀释,不同稀释倍数的混合物和预期的变异频率见表4和表5。dna混合物用covariss220超声打断至主峰在170bp左右,与cfdna的主峰大小相似。表4表5准备cfdna:用qiaampcirculatingnucleicacidkit(qiagen)抽提从患者全血分离后得到的血浆。预文库制备:以kapahyperprepkit(roche)为例,血浆cfdna和打断后的标准品dna取33ng,用kapahyperprepkit(roche)进行预文库制备,补平加尾后连接上实施例1中制备的接头,磁珠纯化后进行pcr扩增,通过上下游引物引入双端的样本标签,上下游引物序列为seqidno:1和seqidno:2所示。靶向捕获文库制备:以idt(integrateddnatechnologies)公司合成的dna探针大panel为例,该panel覆盖了表5和表6中所有的变异位点。具体操作如下:500ng预文库中加入humancot-1dna和adapterblocker,与dna探针65℃杂交孵育4-16小时,再加入到m270磁珠中65℃孵育45分钟,使带有链霉亲和素的m270磁珠与带有生物素标记的探针充分结合,然后用不同离子浓度和温度的清洗缓冲液清洗数次,洗去未与探针结合的非目的片段。dna探针抓下来的目的片段经过pcr扩增后,用磁珠进行纯化,即得到制备好的靶向捕获文库。靶向捕获文库经过质检合格后,在illumina平台的测序仪上进行2x75bp或2x150bp测序,原始数据的测序深度为10,000x-30,000x,用双端的样本标签进行数据的拆分。对于测序数据,首先使用fastp去除低质量碱基、污染的接头序列和低质量reads。对于r1和r2,如果有重叠,会对重叠部分根据碱基质量进行纠错处理。使用c++程序统计总数据量、比对率、on-target率、覆盖深度等质控指标。使用bwa进行初次比对,根据比对位置、分子标签序列、cigar标签和比对方向确定readpairsfamily。根据贝叶斯定理计算一致性序列中每个位置上每种碱基的概率,确定真实序列,进一步根据分子标签序列互补的两条sscs生成dscs。对上述纠错后的序列重新进行比对,检测变异,注释结果,筛选之后获得最终变异集合。分别在lod(limitofdetection)为0.001、0.002、0.005时使用hd701mix1和mix2计算检测灵敏度(ppa)和特异性(ppv),结果如表6。m1表示mix1,m2表示mix2。样本名后的数字代表检测下限,如hd701m1_0.001,表示lod=0.001,tp为truepositive的缩写,ignore指变异频率小于检测下限的位点数目,fp为falsepositive的缩写,fn为falsenegative的缩写。本方法表现出良好的灵敏度和特异性。表6sampletotalsnptpignorefpfnppappvhd701m1_0.001251247014498.41%94.64%hd701m2_0.00125124709498.41%96.48%hd701m1_0.00225124709498.41%96.48%hd701m2_0.00225124615498.40%98.01%hd701m1_0.00525124446398.79%97.60%hd701m2_0.00525123995398.76%97.95%本方法通过降低readpairsfamilysize阈值,保留能与其他sscs形成dscs的含有单个read的readpairfamily等方法大大提高了数据利用率。如表7所示,与原始的duplex方法相比(需要至少3个readpairs形成readpairfamily,只保留dscs序列),本方法提高了用于检测变异的有效数据量(6.526g),覆盖深度(1954.61)和灵敏度有所提升。相比于只使用单端umi(70.06%),本方法的特异性有大幅提升(94.64%),同时也能达到良好的检测灵敏度(98.41%),证明了本方法的检测优势。表7data_size(g)readsontargetmean_covppappvrawdata52.579350,531,218----originalduplex1.1048,095,81694.3239383.8895.62%98.36%sinotools6.52648,896,44484.51061954.6198.41%94.64%single7.97459,591,15285.86952448.3198.80%70.06%对检测到突变挑选6个低频位点进行ddpcr验证,如表8所示,其中5个为阳性结果,并且频率一致性较高。另有一个位点的变异频率在ddpcr检测下限,ddpcr检测到较低阳性信号,但是不能判定,给出阴性结果。以上验证证明本方法与ddpcr方法有高度一致性,可以准确检测低频变异。表8geneaminoacid本方法ddpcregfrp.t790m0.3%0.33%egfrp.t790m2.3%1.82%egfrp.t790m0.1%-egfrp.l858r0.3%0.89%egfrp.l858r2.3%4.60%krasp.g12d2%1.80%进行downsample实验,模拟不同测序深度下检测灵敏度和特异性的变化。检测下限lod=0.005时,当测序深度达到1300x时,检测灵敏度既能达到最优水平。lod=0.001或lod=0.002时,测序深度达到1800x时,检测灵敏度达到最优水平。在此说明书中,本发明已经参照其特定的实施例作了描述。但是,很显然仍可以作出各种修改和变换而不背离本发明的精神和范围。因此,说明书和附图应被认为是说明性的而非限制性的。序列表<110>上海鲸舟基因科技有限公司<120>检测低频变异用的接头、接头混合物及相应方法<141>2018-12-27<160>4<170>siposequencelisting1.0<210>5<211>57<212>dna<213>人工序列()<400>5aatgatacggcgaccaccgagatctacacnnnnnnnnacactctttccctacacgac57<210>5<211>53<212>dna<213>人工序列()<400>5caagcagaagacggcatacgagatnnnnnnnngtgactggagttcagacgtgt53<210>5<211>40<212>dna<213>人工序列()<400>5acactctttccctacacgacgctcttccgatctnnnnnnt40<210>5<211>39<212>dna<213>人工序列()<400>5nnnnnngatcggaagagcacacgtctgaactccagtcac39当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1