幽门螺杆菌检测体系及检测试剂盒和应用的制作方法
本发明属于生物技术领域,涉及一种复合扩增体系及检测试剂盒和应用,更具体的说,它涉及幽门螺杆菌的鉴定、分型及抗生素耐药性检测体系及试剂盒,即幽门螺杆菌检测体系及试剂盒和应用。
背景技术:
幽门螺杆菌是一种以人体为主要宿主的呈螺旋形弯曲的革兰氏阴性杆菌,因其可通过手、不洁食物、粪便等途径传染,在人群中感染率很高,全球幽门螺杆菌的感染率超过50%。我国幽门螺杆菌的感染率约为56%。幽门螺杆菌是一种具有高感染性的致病菌。幽门螺杆菌与慢性活动性胃炎、消化性溃疡、胃黏膜相关淋巴组织淋巴瘤、胃癌发生密切相关。早在1994年,who下属国际癌症研究机构已将幽门螺杆菌感染定为胃癌的i类致癌因子。全国幽门螺杆菌感染处理共识报告显示:幽门螺杆菌感染是预防胃癌最重要的可控危险因素,根除幽门螺杆菌感染可有效降低胃癌的发生率。
流行病学资料显示,幽门螺杆菌感染者几乎都存在慢性活动性胃炎,其中15%~20%发生消化性溃疡,5%~10%发生幽门螺杆菌相关消化不良,约1%发生胃恶性肿瘤(胃癌、malt淋巴瘤),幽门螺杆菌感染导致不同严重程度的疾病,可能与幽门螺杆菌菌株毒力不同以及人群易感性差异有关。caga蛋白是一种来源于幽门螺杆菌的致癌性蛋白。caga蛋白依赖iv分泌系统(t4ss)转位至上皮细胞内,并被src和abl蛋白激酶磷酸化。磷酸化的caga蛋白再与sh2磷酸酶相互作用,最终导致胃黏膜上皮细胞发生改变,该改变可能与胃黏膜组织的癌变密切相关。幽门螺杆菌中存在两种caga等位基因类型,即东亚型与西方型。一项meta分析显示,相较于西方型菌株,caga东亚型(epiya-d)菌株会显著增加胃癌发病风险(or=1.91,95%ci=1.19-3.07,p=.008)。caga东亚型的高致癌性可能与其和sh2结构域的高亲和力有关。
由于抗生素的耐药性,导致幽门螺杆菌的根除率逐年降低。幽门螺杆菌耐药率上升是根除率下降最主要的原因。目前我国推荐用于根除治疗的6种抗菌药物中,克拉霉素(耐药率20%~50%)、甲硝唑(40%~70%)和左氧氟沙星(20%~50%)均已有相当高的耐药率。常规药敏试验是通过细菌培养法来进行,因其需要在胃镜下对胃黏膜进行活检,并且培养周期较长,培养技术要求较高,可及性较低,难以大规模开展。现已证实这些抗生素的耐药机制与幽门螺杆菌的基因突变有关。快速检测幽门螺杆菌的耐药相关基因突变有助于幽门螺杆菌根除治疗方案的选择。幽门螺杆菌对克拉霉素耐药受多种因素影响,研究表明,幽门螺杆菌的23srrna突变a2142g和a2143g是目前已明确的幽门螺杆菌耐克拉霉素的原因。有研究表明,绝大部分的幽门螺杆菌对左氧氟沙星耐药,是由于幽门螺杆菌上的gyra基因asn87或asp91位点上的点突变所导致。
幽门螺杆菌的筛查方法有多种,不同方法的灵敏度和实用性均存在一定不足。比如常见的幽门螺杆菌呼气检测操作简单方便,但不能进行幽门螺杆菌分型和耐药性检测,而血清蛋白试验、活组织检查、胃黏膜常规染色、药物敏感性试验等医学检验手段操作繁琐、技术难度高,而且有创、具有一定的侵入性,取样环节需要进行严格的质量控制,难以快速普及。
鉴于上述情况,需要一种无创、快速、便捷、准确、全面的用于幽门螺杆菌鉴定、分型及抗生素耐药性检测体系及方法。本项目可通过粪便、血液、胃部灌洗液等多种样本进行检测,具有无创、非侵入、可居家取样等特点;采用实时荧光定量pcr技术(quantitativereal-timepcr,本文中简称qpcr),可一次性同时进行感染、基因分型和耐药基因的快速准确检测。
技术实现要素:
本发明提供一种幽门螺杆菌检测体系,可以无创、快速、便捷、准确、全面的用于幽门螺杆菌鉴定、分型及抗生素耐药性检测体系。
一种幽门螺杆菌检测体系,包括4种分别用于幽门螺杆菌的鉴定、分型及抗生素耐药性的扩增体系的一种或多种的组合:
1)对菌种鉴定基因16srrna和23srrna进行检测的引物及探针的幽门螺杆菌鉴定复合扩增体系;其中,引物为:
16srrna引物:
16-f:gagaagcaatactgcgaagtggag
16-r:gcgattccagcttcatgca
23srrna引物:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
16s-p:x-ctcagttcggattgtaggctgcaac-(mgb)
23s-p:x-caagacggaaagacc-(mgb);
2)对菌株分型基因caga进行检测的引物及探针的幽门螺杆菌分型鉴定扩增体系;
其中,引物为:
caga-f:agcaggtaaaggagtgggcg
caga-r:tttgcctcatcaaaatcaattgtag
探针为:
caga-p:x-gctagccctgaacccatttacgctac-(mgb);
3)包括对菌株克拉霉素耐药基因23srrna的2个突变位点进行检测的引物及探针的幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系;
其中,引物为:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
23s-p2142m:x-caagacgggaagacc-(mgb)
23s-p2143m:x-caagacggagagacc-(mgb);
4)包括对菌株左氧氟沙星耐药基因gyra的3个突变位点进行检测的引物及探针的幽门螺杆菌左氧氟沙星耐药性突变检测复合扩增体系;
其中,引物为:
gyra-f:atcgtgggtgatgtgattggt
gyra-r:catagaaaaatcttgcgccattc
体系中涉及的探针为:
gyra-p261m:x-atggcgacacrgcagt-(mgb)
gyra-p271m:x-cagtttatwatgcgttagtg-(mgb)
gyra-p272m:x-cagtttatggtgcgttagtg-(mgb)
gyra-p271+272m:x-cagtttatagtgcgttagtg-(mgb)
其中,x为5’端连接的荧光物质,为fam、ned、vic、cy5、cy3或rox。
优选的,幽门螺杆菌检测体系,包括以下4种扩增体系的一种或多种的组合:
1)对菌种鉴定基因16srrna和23srrna进行检测的引物及探针的幽门螺杆菌鉴定复合扩增体系;
其中,引物为:
16srrna引物:
16-f:gagaagcaatactgcgaagtggag
16-r:gcgattccagcttcatgca
23srrna引物:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
16s-p:(ned)-ctcagttcggattgtaggctgcaac-(mgb)
23s-p:(vic)-caagacggaaagacc-(mgb);
2)对菌株分型基因caga进行检测的引物及探针的幽门螺杆菌分型鉴定扩增体系;
其中,引物为:
caga-f:agcaggtaaaggagtgggcg
caga-r:tttgcctcatcaaaatcaattgtag
探针为:
caga-p:(fam)-gctagccctgaacccatttacgctac-(mgb);
3)包括对菌株克拉霉素耐药基因23srrna的2个突变位点进行检测的引物及探针的幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系;
其中,引物为:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
23s-p2142m:(fam)-caagacgggaagacc-(mgb)
23s-p2143m:(fam)-caagacggagagacc-(mgb);
4)包括对菌株左氧氟沙星耐药基因gyra的3个突变位点进行检测的引物及探针的幽门螺杆菌左氧氟沙星耐药性突变检测复合扩增体系;
其中,引物为:
gyra-f:atcgtgggtgatgtgattggt
gyra-r:catagaaaaatcttgcgccattc
体系中涉及的探针为:
gyra-p261m:(fam)-atggcgacacrgcagt-(mgb)
gyra-p271m:(fam)-cagtttatwatgcgttagtg-(mgb)
gyra-p272m:(fam)-cagtttatggtgcgttagtg-(mgb)
gyra-p271+272m:(fam)-cagtttatagtgcgttagtg-(mgb)。
其中,
所述幽门螺杆菌鉴定复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物16-f、16-r、23-f、23-r各1μl,所述探针16s-p、23s-p各0.6μl,模板dna体积4μl,ddh2o体积0.8μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌分型鉴定扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物caga-f、caga-r各1μl,所述探针caga-p0.6μl,模板dna体积4μl,ddh2o体积3.4μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物23-f、23-r各1μl,所述探针23s-p2142m、23s-p2143m各0.6μl,模板dna体积4μl,ddh2o体积2.8μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌的左氧氟沙星耐药性突变检测复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物gyra-f、gyra-r各1μl,所述探针gyra-p261m、gyra-p271m、gyra-p272m、gyra-p271+272m各0.6μl,模板dna体积4μl,ddh2o体积1.6μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述以上四种扩增体系,其扩增程序为:(1)95℃变性10min;(2)95℃变性15s,64℃退火15s,72℃延伸30s,延伸时收集荧光信号,共40个cycle;(3)25℃保温10s,结束反应。
另外,本发明提供了一种检测试剂盒,包括以下4种扩增体系的一种或多种的组合:
1)对菌种鉴定基因16srrna和23srrna进行检测的引物及探针的幽门螺杆菌鉴定复合扩增体系;其中,引物为:
16srrna引物:
16-f:gagaagcaatactgcgaagtggag
16-r:gcgattccagcttcatgca
23srrna引物:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
16s-p:(ned)-ctcagttcggattgtaggctgcaac-(mgb)
23s-p:(vic)-caagacggaaagacc-(mgb);
2)对菌株分型基因caga进行检测的引物及探针的幽门螺杆菌分型鉴定扩增体系;
其中,引物为:
caga-f:agcaggtaaaggagtgggcg
caga-r:tttgcctcatcaaaatcaattgtag
探针为:
caga-p:(fam)-gctagccctgaacccatttacgctac-(mgb);
3)包括对菌株克拉霉素耐药基因23srrna的2个突变位点进行检测的引物及探针的幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系;
其中,引物为:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
23s-p2142m:(fam)-caagacgggaagacc-(mgb)
23s-p2143m:(fam)-caagacggagagacc-(mgb);
4)包括对菌株左氧氟沙星耐药基因gyra的3个突变位点进行检测的引物及探针的幽门螺杆菌左氧氟沙星耐药性突变检测复合扩增体系;
其中,引物为:
gyra-f:atcgtgggtgatgtgattggt
gyra-r:catagaaaaatcttgcgccattc
体系中涉及的探针为:
gyra-p261m:(fam)-atggcgacacrgcagt-(mgb)
gyra-p271m:(fam)-cagtttatwatgcgttagtg-(mgb)
gyra-p272m:(fam)-cagtttatggtgcgttagtg-(mgb)
gyra-p271+272m:(fam)-cagtttatagtgcgttagtg-(mgb)。
以上4种扩增体系的探针5’端替换为fam、ned、vic、cy5、cy3或rox等其他荧光物质也属于本发明范畴。
其中,
所述幽门螺杆菌鉴定复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物16-f、16-r、23-f、23-r各1μl,所述探针16s-p、23s-p各0.6μl,模板dna体积4μl,ddh2o体积0.8μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌分型鉴定扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物caga-f、caga-r各1μl,所述探针caga-p0.6μl,模板dna体积4μl,ddh2o体积3.4μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物23-f、23-r各1μl,所述探针23s-p2142m、23s-p2143m各0.6μl,模板dna体积4μl,ddh2o体积2.8μl;或此体系中各组分等比扩大或缩小的新扩增体系。
所述幽门螺杆菌的左氧氟沙星耐药性突变检测复合扩增体系总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物gyra-f、gyra-r各1μl,所述探针gyra-p261m、gyra-p271m、gyra-p272m、gyra-p271+272m各0.6μl,模板dna体积4μl,ddh2o体积1.6μl;或此体系中各组分等比扩大或缩小的新扩增体系。
另外本发明也提供了检测试剂盒的用于幽门螺杆菌的鉴定、幽门螺杆菌的菌株分型、幽门螺杆菌的克拉霉素耐药性突变检测和/或幽门螺杆菌的左氧氟沙星耐药性突变检测的应用。
优选的,一种检测试剂盒,包括所述四种扩增体系和所述扩增程序,分别适用于幽门螺杆菌鉴定、幽门螺杆菌菌株分型、幽门螺杆菌克拉霉素耐药性突变检测和幽门螺杆菌左氧氟沙星耐药性突变检测。
本发明的整个方法过程
1、引物及探针的设计
首先,我们选择相关基因的特异性位点和区域,所选基因包括幽门螺杆菌的16srrna、23srrna、caga、gyra,根据候选的位点所在基因的genebank序列号从ncbi核酸数据库下载基因序列,各位点所在基因的部分序列见序列表。
用于扩增16srrna的引物对是由位于序列1(seqidno.1)中1-200位18-30个连续碱基构成的引物,和与序列1中300-500位的互补序列的18-30个连续碱基构成的引物的组合。
用于检测16srrna的探针16s-p是由位于序列1中200-250位18-30个连续碱基构成的序列,5’端使用ned修饰,3’端使用mgb修饰。
用于扩增23srrna的引物对是由位于序列2中1-200位18-30个连续碱基构成的引物,和与序列2(seqidno.2)中300-500位的互补序列的18-30个连续碱基构成的引物的组合。
用于检测23srrna的探针23s-p是由位于序列2中200-250位18-30个连续碱基构成的序列,5’端使用vic修饰,3’端使用mgb修饰。
用于检测23srrna的2142位点突变的探针23s-p2142m是由位于序列2中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于检测23srrna的2143位点突变的探针23s-p2143m是由位于序列2中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于扩增caga的引物对是由位于序列3(seqidno.3)中1-200位18-30个连续碱基构成的引物,和与序列3中300-500位的互补序列的18-30个连续碱基构成的引物的组合。
用于检测caga的探针caga-p是由位于序列3中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于扩增gyra的引物对是由位于序列4(seqidno.4)中1-200位18-30个连续碱基构成的引物,和与序列4中300-500位的互补序列的18-30个连续碱基构成的引物的组合。
用于检测gyra突变的探针gyra-p261m是由位于序列1中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于检测gyra突变的探针gyra-p271m是由位于序列1中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于检测gyra突变的探针gyra-p272m是由位于序列1中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
用于检测gyra突变的探针gyra-p271+272m是由位于序列1中200-250位18-30个连续碱基构成的序列,5’端使用fam修饰,3’端使用mgb修饰。
以上引物和探针由primer3,primerpremier5和ncbiblast等软件设计而成。设计引物时应尽量确保各引物的tm值在(60+2)℃的范围内,扩增效率类似并确保各对引物的扩增产物大小相近,最好在200-300bp范围;设计探针时应尽量确保各探针的tm值在(70+2)℃的范围内,序列特异且尽量靠近产物的5’端。设计完成后,用autodimer等软件分析引物二聚体和不同引物之间的相互作用,如果有相互作用可产生非特异产物或者二聚体的需要重新设计,直至得到符合要求的引物序列。
选用从医院得到的幽门螺杆菌阳性的胃癌患者肿瘤组织样本dna,分别用4对引物进行单重扩增,将扩增产物置于1.5%-2.0%的琼脂糖凝胶上电泳,根据电泳结果来调整pcr体系及扩增条件以得到4对引物共同的扩增条件。最终达成的效果是:在相同体系及扩增条件下,所有的引物对均能出现明亮且较单一的目的条带。
然后,将扩增所得pcr产物进行一代测序验证,确定所得产物是我们的目的产物(如附图1-5)。同时,pcr产物中也检测到了突变位点。
2.扩增体系及条件的建立
2.1qpcr酶的选择
qpcr酶是扩增体系的重要组分,多种qpcr酶都满足本发明的需要;采用热启动taq酶相比非热启动的酶,其扩增效率和特异性均较高;经过多次反复试验,多种商品化qpcr酶如tianexactgenotypingqpcrpremix(probe)(fp211),taqmanuniversalmastermixiiwithung(life),premixextaq(takara)等均可满足要求。
2.2反应体积的选择
分别采用20μl、30μl、40μl和50μl体系进行了复合扩增,结果显示:4种反应体积的扩增效果基本相当,均可满足要求。
2.3反应程序的优化
退火及延伸温度:探索退火温度从58℃到68℃之间各温度下的扩增情况,结果显示经过10个cycle的降落pcr之后,62-66℃延伸均可得到理想的效果。
2.4扩增模板的选择
初期采用胃癌患者肿瘤组织样本dna进行试验,完成反应体系和反应程序的优化后,继续选用胃癌患者粪便样本dna和血液样本等进行检测,可获得相同的试验结果。
2.5cutoff值的确定
分析荧光读值,比较幽门螺杆菌阳性样本和阴性样本的ct值,最终确定cutoff值设定为35。
2.6扩增体系结果导出
见附图6-9
3.测试本检测的灵敏度和特异性
3.1收集检测样本
本试验共收集到正常人粪便样本160份,采用商品化的粪便基因组dna提取试剂盒提取粪便样本dna。同时留取部分样本进行幽门螺杆菌试纸条检测。
3.2粪便样本的幽门螺杆菌试纸条检测
本试验对收集到的160份样本均进行了幽门螺杆菌试纸条检测,共获得阳性样本57例,阴性样本103例。后续以此结果作为样本阴阳性判定标准,便于后续灵敏度和特异性的确定。
3.3本检测试剂盒灵敏度和特异性检测
使用已提取的样本dna完成16s,23s,caga,23s-mutant,gyra-mutant的qpcr检测,读取检测样本ct值,按照cutoff值设定为35进行阴阳性分类。最终检测到阳性样本68例,阴性样本92例。若以幽门螺杆菌试纸条检测结果为标准分析,本检测结果正确样本为139例,其中阳性样本为52例,阴性样本为87例。检测结果灵敏度为91.23%,特异性为84.47%。值得一提的是,以幽门螺杆菌试纸条检测结果为标准所产生的假阳性样本,极有可能是因为本检测的灵敏度更高,可更有效检测出样本中幽门螺杆菌而导致的。此可能性还需更多试验来佐证。
本发明的优势在于:
本发明提供了用于幽门螺杆菌鉴定、分型及抗生素耐药性检测的引物及探针,分别针对16srrna、23srrna、caga、gyra基因。
本发明提供的检测体系及试剂盒可分别或同时对样本直接进行幽门螺杆菌感染检测、幽门螺杆菌东亚型分型以及克拉霉素/左氧氟沙星耐药基因突变检测。
本发明可适用于包括胃部组织,胃部灌洗液,粪便,血液,唾液,牙菌斑等多种类型的dna模板,尤其粪便样本和血液样本,无需侵入取样培养等步骤。本发明具有无创、快速、便捷、准确、全面等优势,可用于幽门螺杆菌的精准治疗指导。
附图说明
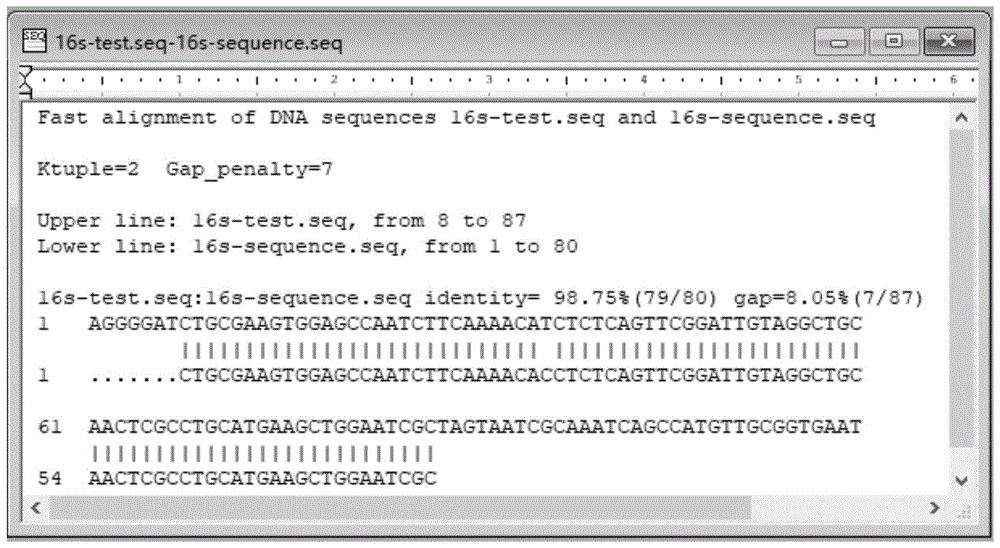
图1使用dnaman软件比对16s的pcr产物测序结果与目标序列一致性,结果显示我们获得了正确的pcr产物。
图2使用dnaman软件比对23s的pcr产物测序结果与目标序列一致性,结果显示我们获得了正确的pcr产物。
图3使用dnaman软件比对caga的pcr产物测序结果与目标序列一致性,结果显示我们获得了正确的pcr产物。
图4使用dnaman软件比对23s的pcr产物测序结果与目标序列一致性,结果显示我们检测到了预期的突变位点。
图5使用dnaman软件比对gyra的pcr产物测序结果与目标序列一致性,结果显示我们检测到了预期的突变位点。
图6幽门螺杆菌的鉴定复合扩增体系,16srrna和23srrna扩增曲线。
图7幽门螺杆菌的分型鉴定扩增体系,caga扩增曲线。
图8幽门螺杆菌的克拉霉素耐药性突变检测复合扩增体系,23srrna扩增曲线。
图9幽门螺杆菌的左氧氟沙星耐药性突变检测复合扩增体系,gyra扩增曲线。
图10幽门螺杆菌鉴定结果判定:本检测样品为幽门螺杆菌阳性样品。
图11幽门螺杆菌分型结果判定:本检测样品中幽门螺杆菌类型为东亚型。
图12幽门螺杆菌的克拉霉素耐药性突变检测结果判定:本检测样品中幽门螺杆菌类型为克拉霉素耐药型。
图13幽门螺杆菌的左氧氟沙星耐药性突变检测结果判定:本检测样品中幽门螺杆菌类型为左氧氟沙星耐药型。
具体实施方式
实施例1
以下是我们采用本发明对一例粪便样本dna检测的具体实施情况,粪便样本来源于经临床确诊为幽门螺杆菌阳性的胃癌患者。
1.dna提取
采用商品化的粪便基因组dna提取试剂盒提取粪便样本dna,操作步骤依照说明书,dna提取完毕用hsqubit试剂盒定量,并稀释成25ng/μl。
2.qpcr检测
2.1qpcr反应体系:
按照说明书分别配置4个反应体系。
1)对菌种鉴定基因16srrna和23srrna进行检测的引物及探针的幽门螺杆菌鉴定复合扩增体系;
其中,引物为:
16srrna引物:
16-f:gagaagcaatactgcgaagtggag
16-r:gcgattccagcttcatgca
23srrna引物:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
16s-p:(ned)-ctcagttcggattgtaggctgcaac-(mgb)
23s-p:(vic)-caagacggaaagacc-(mgb);
2)对菌株分型基因caga进行检测的引物及探针的幽门螺杆菌分型鉴定扩增体系;
其中,引物为:
caga-f:agcaggtaaaggagtgggcg
caga-r:tttgcctcatcaaaatcaattgtag
探针为:
caga-p:(fam)-gctagccctgaacccatttacgctac-(mgb);
3)包括对菌株克拉霉素耐药基因23srrna的2个突变位点进行检测的引物及探针的幽门螺杆菌克拉霉素耐药性突变检测复合扩增体系;
其中,引物为:
23-f:ttcagtgaaattgtagtggaggtg
23-r:tcccattagcagtgctaagttgta
探针为:
23s-p2142m:(fam)-caagacgggaagacc-(mgb)
23s-p2143m:(fam)-caagacggagagacc-(mgb);
4)包括对菌株左氧氟沙星耐药基因gyra的3个突变位点进行检测的引物及探针的幽门螺杆菌左氧氟沙星耐药性突变检测复合扩增体系;
其中,引物为:
gyra-f:atcgtgggtgatgtgattggt
gyra-r:catagaaaaatcttgcgccattc
体系中涉及的探针为:
gyra-p261m:(fam)-atggcgacacrgcagt-(mgb)
gyra-p271m:(fam)-cagtttatwatgcgttagtg-(mgb)
gyra-p272m:(fam)-cagtttatggtgcgttagtg-(mgb)
gyra-p271+272m:(fam)-cagtttatagtgcgttagtg-(mgb)。
幽门螺杆菌鉴定复合扩增体系,总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物16-f、16-r、23-f、23-r各1μl,所述探针16s-p、23s-p各0.6μl,模板dna体积4μl,ddh2o体积0.8μl。
幽门螺杆菌分型鉴定扩增体系,总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物caga-f、caga-r各1μl,所述探针caga-p0.6μl,模板dna体积4μl,ddh2o体积3.4μl。
幽门螺杆菌的克拉霉素耐药性突变检测复合扩增体系,总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物23-f、23-r各1μl,所述探针23s-p2142m、23s-p2143m各0.6μl,模板dna体积4μl,ddh2o体积2.8μl。
幽门螺杆菌的左氧氟沙星耐药性突变检测复合扩增体系,总体积为20μl,其中2×qpcrpremix体积为10μl,所述引物gyra-f、gyra-r各1μl,所述探针gyra-p261m、gyra-p271m、gyra-p272m、gyra-p271+272m各0.6μl,模板dna体积4μl,ddh2o体积1.6μl。
2.2qpcr反应程序:
将pcr反应管置于qpcr扩增仪7500上,并设计如下程序:(1)95℃变性10min;(2)95℃变性15s,64℃退火15s,72℃延伸30s,延伸时收集荧光信号,共40个cycle;(3)25℃保温10s,结束反应。
2.3数据导出与结果判定(附图10-13)
反应完成后,根据仪器设置,在软件中进行荧光阈值和基线的调整后导出实验数据。ct值小于35判定为阳性,ct值大于35判定为阴性。反应完成后,根据仪器设置,在软件中进行荧光阈值和基线的调整后导出实验数据。
2.3.1幽门螺杆菌鉴定结果判定:16srna和23srna的qpcr检测结果显示扩增曲线均起峰正常;两者ct值分别为29.68和31.81,都小于35。因此判定本检测样品为幽门螺杆菌阳性样品。
2.3.2幽门螺杆菌分型结果判定:caga的qpcr检测结果显示扩增曲线起峰正常;ct值为28.37,小于35。因此判定本检测样品中幽门螺杆菌类型为东亚型样品。
2.3.3幽门螺杆菌的克拉霉素耐药性突变检测结果判定:23srna突变型的qpcr检测结果显示扩增曲线起峰正常;ct值为26.59,小于35。因此判定本检测样品中幽门螺杆菌类型为克拉霉素耐药型。
2.3.4幽门螺杆菌的左氧氟沙星耐药性突变检测结果判定:23srna突变型的qpcr检测结果显示扩增曲线起峰正常;ct值为28.83,小于35。因此判定本检测样品中幽门螺杆菌类型为左氧氟沙星耐药型。
2.4已有检测结果与本样本临床诊断结果比较
本检测样本来源于经临床确诊为幽门螺杆菌阳性的胃癌患者,经实验室检测,其结果与临床诊断结果一致。
序列表
<110>杭州和壹基因科技有限公司
<120>幽门螺杆菌检测体系及检测试剂盒和应用
<130>21-2019-0101
<141>2019-01-24
<160>21
<170>siposequencelisting1.0
<210>1
<211>552
<212>dna
<213>人工序列(artificialsequence)
<400>1
gctgtcgtcagctcgtgtcgtgagatgttgggttaagtcccgcaacgagcgcaaccccct60
ttcttagttgctaacaggtcatgctgagaactctaaggatactgcctccgtaaggaggag120
gaaggtggggacgacgtcaagtcatcatggcccttacgcctagggctacacacgtgctac180
aatggggtgcacaaagagaagcaatactgcgaagtggagccaatcttcaaaacacctctc240
agttcggattgtaggctgcaactcgcctgcatgaagctggaatcgctagtaatcgcaaat300
cagccatgttgcggtgaatacgttcccgggtcttgtactcaccgcccgtcacaccatggg360
agttgtgtttgccttaagtcaggatgctaaattagctactgcccacggcacacacagcga420
ctggggtgaagtcgtaacaaggtaaccgtagtgacacctgcggttggatcacctcctttc480
tagagaaaagcttttaacattcgcttattaaaagccaaagagtattacctgacaaaagaa540
cttcttgttgct552
<210>2
<211>552
<212>dna
<213>人工序列(artificialsequence)
<400>2
gtgtgacgcctgcccggtgctcgaaggttaagaggatgcgtcagtcgcaagatgaagcgt60
tgaattgaagcccgagtaaacggcggccgtaactataacggtcctaaggtagcgaaattc120
cttgtcggttaaataccgacctgcatgaatggcgtaacgagatgggagctgtctcaacca180
gagattcagtgaaattgtagtggaggtgaaaattcctcctacccgcggcaagacggaaag240
accccgtggacctttactacaacttagcactgctaatgggaatatcatgcgcaggatagg300
tgggaggctttgaagtaagggctttggctcttatggagtcatccttgagataccaccctt360
gatgtttctgttagctaactggcctgtgttatccacaggcaggacaatgcttggtgggta420
gtttgactggggcggtcgctcctaaaaagtaacggaggcttgcaaaggttggctcattgc480
ggttggaaatcgcaagttgagtgtaatggcacaagccagcctgactgtaagacatacaag540
tcaagcagagac552
<210>3
<211>552
<212>dna
<213>人工序列(artificialsequence)
<400>3
ctcaaaaacaacacagagcctatttatgctcaagttaataaaaagaaaacaggacaagca60
actagccctgaagagcccatttacgctcaagttgctaaaaaggtgagtgcaaaaattgac120
caactcaacgaagctacatcagcaataaatagaaaaattgaccggattaacaaaattgca180
tcagcaggtaaaggagtgggcggtttcagtggagcagggcgatcagctagccctgaaccc240
atttacgctacaattgattttgatgaggcaaatcaagcaggcttccctttgagaagaagt300
gctgcagttaatgatctcagtaaagtagggctttcaagggaacaagaattgactcgtaga360
attggcgatctcagtcaggcagtgtcagaagctaaaacaggtcattttggcaacctagaa420
caaaagatagatgaactcaaagattctacaaaaaagaatgctttgaagctatgggttgaa480
agtgcgaaacaagtgcctactagtttgcaagcgaaattggacaattacgctactaacagc540
cacacacgcatt552
<210>4
<211>552
<212>dna
<213>人工序列(artificialsequence)
<400>4
atgcaagatcatttagtcaatgaaacaaaaaatattgtagaagtggggattgattcttct60
attgaagagagctatttggcttattccatgagcgtgatcatagggcgcgctttaccggac120
gctagagacggcttaaagcctgtgcataggcgtattttgtatgcgatgcatgaattaggc180
cttacttccaaagtcgcttataaaaaaagcgctaggatcgtgggtgatgtgattggtaaa240
taccacccccatggcgacaccgcagtttatgatgcgttagtgagaatggcgcaagatttt300
tctatgcgcttggaattagtggatgggcagggcaactttggctctattgatggcgataac360
gccgcagcgatgcgttacactgaagccagaatgaccaaggcgagtgaagagattttaaga420
gatattgataaagacaccattgattttgtgcctaattacgatgacaccttaaaagagcca480
gatattttaccaagccgtctgcctaaccttttagtcaatggggctaatgggatcgccgta540
gggatggcgact552
<210>5
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>5
gagaagcaatactgcgaagtggag24
<210>6
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>6
gcgattccagcttcatgca19
<210>7
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>7
ttcagtgaaattgtagtggaggtg24
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>8
tcccattagcagtgctaagttgta24
<210>9
<211>30
<212>dna
<213>人工序列(artificialsequence)
<400>9
ndctcagttcggattgtaggctgcaacmgb30
<210>10
<211>15
<212>dna
<213>人工序列(artificialsequence)
<400>10
caagacggaaagacc15
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
agcaggtaaaggagtgggcg20
<210>12
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>12
tttgcctcatcaaaatcaattgtag25
<210>13
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>13
gctagccctgaacccatttacgctac26
<210>14
<211>15
<212>dna
<213>人工序列(artificialsequence)
<400>14
caagacgggaagacc15
<210>15
<211>15
<212>dna
<213>人工序列(artificialsequence)
<400>15
caagacggagagacc15
<210>16
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>16
atcgtgggtgatgtgattggt21
<210>17
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>17
catagaaaaatcttgcgccattc23
<210>18
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>18
atggcgacacrgcagt16
<210>19
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>19
cagtttatwatgcgttagtg20
<210>20
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>20
cagtttatggtgcgttagtg20
<210>21
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>21
cagtttatagtgcgttagtg20
- 还没有人留言评论。精彩留言会获得点赞!