一种高产2-苯乙醇的酵母工程菌及其制备方法与应用与流程
本发明属于基因工程领域,尤其是涉及一种高产2-苯乙醇的酵母工程菌及其制备方法与应用。
背景技术:
2-苯乙醇(2-phenylethylalcohol,2-pe)是一种具有玫瑰花香的芳香醇,常温下是一种无色粘稠液体,天然存在于许多花和植物精油中,如玫瑰、风信子、茉莉、百合等。2-苯乙醇所具有的玫瑰芳香颇受人们欢迎,也因此被广泛应用于食品、化妆品、日用品等行业,它不仅是所有玫瑰型香气的基本组分,而且还因具有协合增效作用而成为多种香型配方所必须的组分。作为香料添加剂,可用于玫瑰、焦糖、蜂蜜及其他果香型食品香精及各种烟用香精、皂用香精的配制,其使用量仅次于香兰素,是世界第二大香料成分。
目前,2-苯乙醇主要通过化学方式生产,但化学合成的风味化合物在某些领域的应用受到限制,特别是在食品工业中,天然风味化合物显然更受消费者的青睐。然而,从花卉和植物的精油中提取天然的2-苯乙醇是昂贵且低效的,市场价格高达1,000美元/kg。
从上世纪末开始,随着人们对生活品质要求的提高和对健康的关注,消费者越来越注重食品、日用品等的安全性,“绿色”、“天然”、“健康”的理念不断加强,使得食品、日用品等行业也越来越倾向于使用天然的添加剂。在美国和欧洲,都有相关规定认为,采用物理方法从天然材料中提取和酶催化或微生物发酵法生产的调味剂或芳香剂可以被标记为“天然”。需求量之大以及生产成本之高,使得从玫瑰或其他精油中提取的天然2-苯乙醇远远不能满足市场的需要。而利用微生物发酵法生产2-苯乙醇就成为了一种极具吸引力的选择,具有产品绿色天然、成本低、周期短、反应条件温和、环境污染小等突出优点,是2-苯乙醇制备研究的主流方向,具有良好的发展前景。
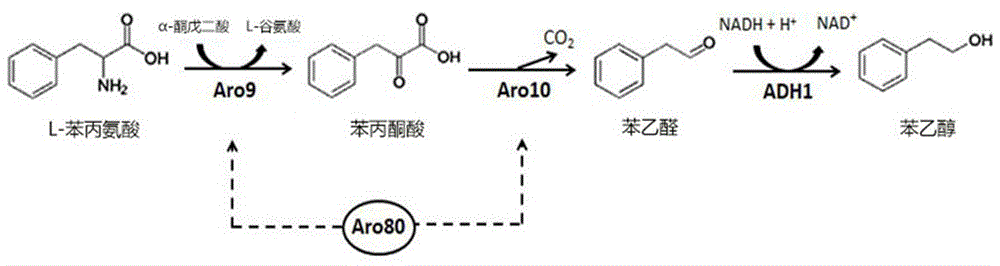
一些微生物本身就具有合成2-苯乙醇的能力,为微生物发酵法生产2-苯乙醇奠定了基础。酿酒酵母具有较高的2-苯乙醇合成能力,同时对多种环境胁迫具有很好的适应性,是微生物发酵法生产2-苯乙醇的理想菌种。酵母菌株通过ehrlich途径生产2-苯乙醇由三个步骤组成:l-苯丙氨酸转氨生成l-苯丙酮酸,l-苯丙酮酸脱羧生成苯乙醛,最后苯乙醛还原成2-苯乙醇。但是目前我国使用酵母发酵法生产2-苯乙醇的技术还不算成熟,产量停留在一个较低水平。已有研究主要集中在菌种的自然筛选、传统的微生物育种或发酵工艺的改进上,存在着2-苯乙醇合成能力有限,难以进行大规模工业化生产的问题。
目前有关于生物生产2-苯乙醇的专利里,提出了3株可用于2-苯乙醇生产的发酵菌株,分别是于2013年8月27日申请的专利里,提出的酿酒酵母whh6菌株,于2010年12月20日保存于中国微生物菌种保藏管理委员会普通微生物中心(申请号为201210309144.6)。和2014年12月8日申请的专利里,也有一株2-苯乙醇产量最高3.39g/l酿酒酵母菌株(申请编号为201410743753.1)。以及在2016年4月22日的专利里,有选取高渗透压的产甘油假丝酵母转化l-苯丙氨酸发酵生产2-苯乙醇(申请编号201610256845.6)该菌株可利用7g/l的l-苯丙氨酸生成5g/l的苯乙醇。但总体而言2-苯乙醇的产量仍受菌株耐受以及底物抑制的限制,产量始终保持在较低水平,无法满足广大的市场需求。
技术实现要素:
为了解决上述问题,本发明提供一种高产2-苯乙醇的酵母工程菌及其制备方法与应用,目的是针对目前市场对天然绿色产品的需要,以及生物法生产2-苯乙醇产物产量低的问题,通过构建转录激活因子(aro80)和谷氨酸脱氢酶(gdha)重组质粒,并将其转入酿酒酵母中,使该工程菌串联表达aro80和gdha。在酿酒酵母中,l-苯丙氨酸通过氨基酸转氨酶(aro90)的作用生成苯丙酮酸,再通过丙酮酸脱羧酶(aro10)的作用生成苯乙醛,苯乙醛经过脱氢酶(adh1)氧化脱氢生成苯乙醇。其中,aro80的过表达会以更高水平激活氨基酸转氨酶(aro9)和丙酮酸脱羧酶(aro10)的表达,从而提高苯乙醇的产量如图1所示。而通过引入gdha的表达,可将l-谷氨酸分解为α-酮戊二酸和nad(p)h,为转化过程提供了氨基受体和还原力,可实现辅因子和还原力的再生和平衡,从而提高苯乙醇的产量,如图2所示。两者的串联表达可使2-苯乙醇的产量得到较大的提升;另外,本发明还通过在酵母的发酵工艺中添加l-谷氨酸、α-酮戊二酸、沸石,以此增加转化体系中的反应物物质浓度以及消除循环过程合成的nh3,以提高2-苯乙醇的合成效率。
本发明的目的是通过以下技术方案实现的:
一种高产2-苯乙醇的酵母工程菌的制备方法,向受体酿酒酵母中导入转录激活因子aro80基因和谷氨酸脱氢酶gdha基因,得到高产2-苯乙醇的酿酒酵母工程菌。
进一步的,所述转录激活因子aro80是如下a)或b)的蛋白质:
a)seqidno.1所示的蛋白质;
b)将seqidno.1所示的氨基酸序列经过一个或几个氨基酸残基的取代/或缺失和/或添加且功能相同的蛋白质。
进一步的,所述谷氨酸脱氢酶gdha是如下a)或b)的蛋白质:
a)seqidno.2所示的蛋白质;
b)将seqidno.2所示的氨基酸序列经过一个或几个氨基酸残基的取代/或缺失和/或添加且功能相同的蛋白质。
进一步的,所述受体酿酒酵母为酿酒酵母ys58。
本发明还保护一种上述方法得到的产2-苯乙醇的酿酒酵母工程菌。
本发明还保护一种一种制备2-苯乙醇的的方法,将上述酿酒酵母工程菌进行发酵培养,得到
进一步的,制备2-苯乙醇的的方法包括如下具体步骤:
(1)种子液制备:将上述酿酒酵母工程菌接种入yepd培养基中,28-32℃活化12h;
(2)发酵液制备:接种步骤(1)制备的种子液于发酵培养基中,接种量10%,28-32℃、220r/min培养36h,得到发酵液;
(3)将步骤(2)制备的发酵液,12000rpm离心10min,取上清液,弃菌体,上清液经0.22μm滤膜过滤后,得到2-苯乙醇;
所述发酵培养基由溶剂和溶质组成,溶剂为水,溶质为l-苯丙氨酸、葡萄糖、硫酸镁和磷酸二氢钾;所述l-苯丙氨酸在发酵培养基中的浓度为5g/l,所述葡萄糖在发酵培养基中的浓度为20g/l,所述硫酸镁在发酵培养基中的浓度为0.5g/l,所述磷酸二氢钾在发酵培养基中的浓度为0.5g/l。
进一步的,所述发酵培养基的溶质还包括0.736g/l-2.944g/l的l-谷氨酸、0g/l-3.497g/lα-酮戊二酸和0g/l-20g/l沸石。
所述发酵体系中所述l-谷氨酸的添加量具体为0.147g/l、0.294g/l、0.736g/l、1.472g/l、2.944g/l、3.675g/l;
所述发酵体系中所述α-酮戊二酸的添加量具体为0.146g/l,0.292g/l,0.731g/l,1.461g/l,2.992g/l;
所述发酵体系中所述沸石的添加量具体为1g/l、2g/l、5g/l、10g/l、20g/l;
进一步的,所述发酵培养温度具体为30℃。
上述所述酿酒酵母工程菌在制备2-苯乙醇中的应用也属于本发明的保护范围。
本发明的有益效果:
1、本发明提供的高产2-苯乙醇的酵母工程菌与宿主菌相比,合成2-苯乙醇的能力显著提高,且通过发酵体系的优化大大提高了底物转化率高,且该工程菌发酵条件简单,发酵周期短,再结合其产量高的特点,有利于其在大规模工业化生产中的应用。
2、本发明通过构建转录激活因子(aro80)和谷氨酸脱氢酶(gdha)重组质粒,并将其转入酿酒酵母中,使该工程菌串联表达aro80和gdha。其中,aro80的过表达会以更高水平激活氨基酸转氨酶(aro9)和丙酮酸脱羧酶(aro10)的表达,如图1所示。而通过引入gdha的表达可实现辅因子和还原力的再生和平衡,如图2所示。两者的串联表达可使2-苯乙醇的产量得到较大的提升。
3、本发明还通过在酵母的发酵工艺中添加l-谷氨酸、α-酮戊二酸、沸石,以此增加转化体系中的反应物物质浓度以及消除循环过程合成的nh3,以提高2-苯乙醇的合成效率。
附图说明
图1是酿酒酵母中ehrlich途径生产2-苯乙醇;
图2是酿酒酵母中加入辅因子谷氨酸脱氢酶,还原力平衡的2-苯乙醇代谢途径;
图3是利用酿酒酵母得到转化产物苯乙醇的hplc分析图;
图4是利用酿酒酵母得到转化产物苯乙醇的标准曲线。
具体实施方式
为了更好的理解本发明,下面通过实施例对本发明进进一步说明,实施例只用于解释本发明,并不会对本发明构成任何限定。
以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段,所用原料均为市售商品。
实施例1、产2-苯乙醇的酵母工程菌的构建
实施例中所用酿酒酵母(saccharomycescerevisiae)ys58菌株,1988年5月1日保藏在中国微生物菌种保藏管理委员会普通微生物中心,,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,保藏编号为cgmccno.2.1526。
实施例中所用大肠杆菌(escherichiacolistr.)k-12菌株,2014年3月28日保藏在中国微生物菌种保藏管理委员会普通微生物中心,,地址:北京市朝阳区北辰西路1号院3号中国科学院微生物研究所,保藏编号为cgmccno.1.12874。
实施例中所用质粒pyx212-kanmx是酿酒酵母中常用的表达载体可自addgene网站上购得。
实施例中所用的材料、试剂、培养基等,如无特殊说明,均可从商业途径得到。
参与编码aro9和aro10基因表达的转录激活因子aro80的获得方法,步骤如下:
(1)根据已知的转录因子aro80序列,设计如下引物:
上游引物p1:ggaattaaccatggcacctgttacaattg(seqidno.5)。
下游引物p2:ccttggtaccctattttttatttcttttaagtgcc(seqidno.6)。
(2)以酿酒酵母saccharomycescerevisiaew303-1b基因组为模板,以p1和p2为引物进行pcr扩增,扩增得到转录激活因子aro80,片段大小约2800bp,经测序分析,结果表明扩增的aro80的基因序列(seqidno.3)所示,该核苷酸序列所编码的转录激活因子aro80的氨基酸序列(seqidno.1)。
pcr反应体系:酿酒酵母saccharomycescerevisiaew303-1b基因组dna100ng,引物p1终浓度0.2μmol/l,引物p2终浓度0.2μmol/l,
pcr反应条件:95℃预变性10分钟;95℃变性30秒,55℃退火30秒,72℃延伸1分钟,共30个循环,72℃延伸10分钟使产物延伸完全。
二、辅因子循环系统关键酶谷氨酸脱氢酶的编码基因gdha的获得
根据已知的谷氨酸脱氢酶的编码基因gdha序列,设计如下引物:
上游引物p3:aggagatataatggatcagacatattctctg(seqidno.7)
下游引物p4:ccttctcgagttaaatcacaccctgcgc(seqidno.8)
以大肠杆菌escherichiacolistr.k-12基因组为模板,以p3和p4为引物进行pcr扩增,得到谷氨酸脱氢酶的编码基因(gdha),片段大小约1300bp,经测序分析,结果表明扩增的gdha的基因序列(seqidno.4),该核苷酸序列所编码的谷氨酸脱氢酶的氨基酸序列(seqidno.2)。
pcr反应体系:大肠杆菌escherichiacolistr.k-12基因组dna100ng,引物p1终浓度0.2μmol/l,引物p2终浓度0.2μmol/l,
pcr反应条件:95℃预变性10分钟;95℃变性30秒,55℃退火30秒,72℃延伸1分钟,共30个循环,72℃延伸10分钟使产物延伸完全。
三、构建重组质粒pyxg80
pyxg80是由pyx212-kanmx载体(购自addgene公司)改造而来,使用ecorⅰ和bamhⅰ双酶切,电泳回收载体大片段。使用gibson连接方法,将酶切回收后的pyx212-kanmx片段和步骤二中获得的谷氨酸脱氢酶gdha按照片段浓度与载体浓度3:1的比例,加入gibson反应体系中。50℃反应1小时,取10μl连接产物转化至感受态细胞dh5α中,使用氨苄青霉素抗性筛选,挑去阳性克隆,将阳性克隆进行液体培养,提取阳性克隆质粒,进行酶切验证,结果表明gdha基因和aro80基因串联插入pyx212-kanmx的多克隆位点中,重组质粒构建正确,将该重组载体命名为pyxg80。
四、构建产2-苯乙醇的酵母工程菌
将重组质粒pyxg80和空载体质粒pyx212-kanmx分别转化酿酒酵母ys58,在含600μg/mlg418(遗传霉素,购自赛默飞公司)的yepd平板(培养基成分可以在中国普通微生物保藏管理中心找到,编号0801)上筛选酵母转化子,因质粒中含有kanmx基因,所以含有质粒pyxg80和pyx212-kanmx的转化菌在含600μg/mlg418的yepd平板上能生长,而宿主菌ys58则不能生长。
提取pyxg80转化子和pyx212-kanmx转化子的质粒,进行酶切验证,测序表明质粒pyxg80和pyx212-kanmx成功转入宿主菌酿酒酵母ys58,将含有重组质粒pyxg80的转化菌株命名为ys58(pyxg80),将含空载质粒pyx212-kanmx的转化菌株命名为ys58(pyx212-kanmx)。
实施例2、酵母菌发酵法生产2-苯乙醇
1.酵母菌发酵培养
种子液制备:将酵母转化子接种入yepd培养基(培养基成分可以在中国普通微生物保藏管理中心找到,编号0801)中(18×180mm试管,5ml装液量),30℃活化12h。
发酵液制备:接种种子液于发酵培养基中(250ml三角瓶,50ml装液量),5ml种子液接种至50ml发酵培养基中,30℃、220r/min培养36h。
上述发酵培养基由溶剂和溶质组成,溶剂为水,溶质为l-苯丙氨酸、葡萄糖、硫酸镁、磷酸二氢钾;所述l-苯丙氨酸在发酵培养基中的浓度为5g/l,所述葡萄糖在发酵培养基中的浓度为20g/l,所述硫酸镁在发酵培养基中的浓度为0.5g/l,所述磷酸二氢钾在发酵培养基中的浓度为0.5g/l。
2.检测2-苯乙醇
将发酵液,12000rpm离心10min,取上清液,弃菌体,上清液经0.22μm滤膜过滤后,使用hplc法测定样品上清液中的2-苯乙醇含量。
测定条件:agilent1200高效液相色谱仪(配四元泵,dad检测器和工作站)。色谱条件:watersx-bridgec-183.5μm4.6*150nmcolumn:流动相:水/乙醇=70/30,流速:0.5ml/min-2,柱温25℃:进样量5μl,检测波长244nm,实验设置三次重复,结果取平均值。
在此条件下,可以同时将底物苯丙氨酸l-phe(3.00min)、中间代谢产物苯丙酮酸ppa(2.68min)、苯乙醛pald(4.83min)以及终产物苯乙醇pea(8.44min)同时检测出来。检测结果如图3所示,苯乙醇峰面积与标准品苯乙醇峰面积相同,证明检测到在重新构建的酿酒酵母ys58中苯乙醇的合成。
实施例3、不同酵母菌株2-苯乙醇产量比较
将工程菌株ys58(pyxg80)、空载体转化菌株ys58(pyx212-kanmx)和ys58分别接入5mlyepd培养基(含300μg/mlg418),30℃活化12小时。再以10%接种量,分别将ys58(pyxg80)和ys58(pyx212-kanmx)接种于发酵培养基(含300μg/mlg418),30℃、220r/min培养36h。
培养结束后,分别取ys58(pyxg80)、ys58(pyx212-kanmx)、ys58的发酵液,12000rpm离心10min,取上清液,弃菌体,上清液经0.22μm滤膜过滤后,使用hplc法测定各个样品上清液中的2-苯乙醇含量。
测定条件:agilent1200高效液相色谱仪(配四元泵,dad检测器和工作站)。色谱条件:watersx-bridgec-183.5μm4.6*150nmcolumn:流动相:水/乙醇=70/30,流速:0.5ml/min-2,柱温25℃:进样量5μl,检测波长244nm,实验设置三次重复,结果取平均值。
各个培养液中2-苯乙醇产量检测结果如表1所示。
实施例4、酵母菌株生产2-苯乙醇发酵工艺的优化
一、谷氨酸对酵母生产2-苯乙醇的影响
使用菌株ys58(pyxg80)按照实施案例2中方法进行发酵生产2-苯乙醇,基础发酵培养基中添加0-20mm的l-谷氨酸,转化体系ph=6.5(使用pbs磷酸缓冲盐溶液进行调配)。在添加不同浓度的l-谷氨酸发酵培养基中,酵母菌株生产2-苯乙醇浓度如表2所示。
表2表明,在发酵培养基中添加5-20mm(即0.736g/l-2.944g/l)l-谷氨酸时,生产的2-苯乙醇浓度较高,当发酵培养基中添加10mm(1.471\2g/l)l-谷氨酸时,所生产的2-苯乙醇浓度最高,达到40.07mm,即3.28g/l。
二、α-酮戊二酸对酵母生产2-苯乙醇的影响
使用菌株ys58(pyxg80)按照实施案例2中方法进行发酵生产2-苯乙醇,基础发酵培养基中添加0-20mm的α-酮戊二酸,转化体系ph=6.5(使用pbs磷酸缓冲盐溶液进行调配)同上。在添加不同浓度的α-酮戊二酸发酵培养基中,酵母菌株生产2-苯乙醇浓度如表3所示。
表3表明,在发酵培养基中添加2.922g/lα-酮戊二酸所生产的2-苯乙醇浓度最高,达到44.83mm,即3.67g/l。
三、沸石对酵母生产2-苯乙醇的影响
使用菌株ys58(pyxg80)按照实施案例2中方法进行发酵生产2-苯乙醇,基础发酵培养基中添加0-20g/l的沸石,转化体系ph=6.5(使用pbs磷酸缓冲盐溶液进行调配)。在添加不同浓度的沸石发酵培养基中,酵母菌株生产2-苯乙醇浓度如表4所示。
表4表明,在发酵培养基中添加5-15g/l时,生产的2-苯乙醇浓度较高,当添加10g/l沸石所生产的2-苯乙醇浓度最高,达到41.05mm,即3.36g/l。
四、l-谷氨酸、α-酮戊二酸和沸石对酵母生产2-苯乙醇的影响
使用菌株ys58(pyxg80)按照实施案例2中方法进行发酵生产2-苯乙醇,基础发酵培养基中添加1.471g/l的l-谷氨酸、2.922g/lα-酮戊二酸和10g/l沸石,转化体系ph=6.5。酵母菌株生产2-苯乙醇浓度如表5所示。
表5表明,向培养基中添加l-谷氨酸、α-酮戊二酸和沸石对2-苯乙醇的产量有较大提升,添加1.471g/l的l-谷氨酸、2.922g/lα-酮戊二酸和10g/l沸石之后,2-苯乙醇的产量从33.41mm提高到了45.2mm。
以上所述仅为本发明的实施例,并非因此限制本发明的专利范围,凡是利用本发明说明书内容所作的等效结构或等效流程变换,或直接或间接运用在其他相关的技术领域,均同理包括在本发明的专利保护范围内。
序列表
<110>福建师范大学
<120>一种高产2-苯乙醇的酵母工程菌及其制备方法与应用
<160>8
<170>siposequencelisting1.0
<210>1
<211>950
<212>prt
<213>人工序列(artificialsequence)
<400>1
metseralalyslysargproserglyasnalaalaphegluleupro
151015
lysargargargthrtyrglnalacysilesercysargserarglys
202530
vallyscysaspleuglyprovalaspasnprohisaspproprocys
354045
alaargcyslysarggluleulyslyscysilepheserserasnlys
505560
glythrserasnaspleuproproasnserileasnalaileserleu
65707580
proserleuglylysserlysglngluileglnasnaspserthrser
859095
proileleuseraspvalproleuserarglysglyileserserglu
100105110
lysserphelyssergluglymetlystrplysleugluleuserser
115120125
metglnasnalaleuglupheleualaglnalaalaglythrvalala
130135140
lysgluglyalalysgluileilelysglulysserthrthrprolys
145150155160
proleulysserserleuaspalathrasnlysseralathraspglu
165170175
glyleulysargleuserlysseraspserthrasnthrleutyrglu
180185190
asnthralaaspmetleuasnhisthrleuasnthrasnarglysthr
195200205
serglnleumetglugluileglylysvalargproproprothrarg
210215220
lysileaspasppheasptyrileglyproaspserleuleuthrlys
225230235240
gluglualailegluleuileglualaphepheleuthrmethispro
245250255
phepheproasnileproleuglnleuhisaspprolysgluleuala
260265270
glutyrproileleuphecysalaileleuthrvalseralaargtyr
275280285
hispropheaspthrleuglyleuaspasnglygluaspglymetarg
290295300
hisilegluvalhisasplysleutrpvaltyrcysglnlysleuile
305310315320
serglnthriletrpalaglualaserthrargserileglythrval
325330335
leualapheileilephethrglutrpasnproargserilehistyr
340345350
lystrpserasptyralaasnaspprogluleuasnasnvalasnala
355360365
argglyserlysasnileserthrarglysaspglugluglyleuthr
370375380
glyvalglyalaileargargseraspargmetsertrpmetleuthr
385390395400
glyseralavalargleualaglnaspmetglypheilegluasnser
405410415
serlysvalpheilevalthrhisilesergluthrthrseralamet
420425430
asnmetasnglnargserleuleualagluserpheservalleuasn
435440445
leuasnleuglylysilegluasnaspglyasngluserasngluasp
450455460
tyrleuglyasnglulysphetyrleuasngluileleuproaspglu
465470475480
gluserlysleuargtrplysargvalphegluasnsergluasnasp
485490495
hisaspasnglulysasnpheleuthrasptrpgluargglupheleu
500505510
asnaspglutyrvalleutyrtyrserasnlyslysaspaspthrasn
515520525
leualaglnasnhisilepropropheproleuargpheserpheala
530535540
glnargalalysilegluileileargileleuserilealatyrglu
545550555560
thriletyrcysglulysasnlysarglysleualathrthraspgln
565570575
arghisasnleuservalleuservalpheserproleuileglugly
580585590
trpleuserasntyrarggluleuleuvalproleuseraspvalpro
595600605
pheserleualaasparglysasnlyslysglnilepheaspasnile
610615620
aspargileasnglygluserileilethrasppheasntyrcysgln
625630635640
leutyrilepheserleualaleuglnvalaspglylysthrserarg
645650655
leuasnmetasngluilevalthrseralaargtyrvalgluleuala
660665670
tyrargseralalysgluileleuserseralalysargvalserarg
675680685
glnglymetleulystyrmetprovalargtrpvalileargileile
690695700
argserilealapheilevallyscystyrleuthrleuthrglyser
705710715720
gluleualathrasnproaspalaargasnileleulysleuserala
725730735
ileservalaspgluthrpheaspileileargaspthralavalthr
740745750
leulysglualathrproaspgluleuhisleucysglnargtyrala
755760765
alaileleumettyrleucysthrglumetlysleuarglyslysser
770775780
tyrleugluargproproleuleuargaspglythrthrproleuglu
785790795800
serasnarggluserserleugluglyglnaspleuthrlyslyspro
805810815
ilepheserlysargileglytyrasnlysthrgluthrthrpheglu
820825830
prosergluargproleuthrglugluileasnserasnserglnasn
835840845
serasnaspthrserserlysglyilevalaspprophevalglugln
850855860
asnasnaspilethrthralaleuleuasnasngluleupheglngly
865870875880
proserleuseraspgluvalthrasptrppheglyalasergluasp
885890895
ileglyleugluphevalgluprotrpthrgluleuilegluglnarg
900905910
tyrmetglncysglyaspglyaspasnasnasnphegluasnleutyr
915920925
asnleuphevalasnserasnasnileasnasnaspileasnasnser
930935940
argproilethrarglys
945950
<210>2
<211>447
<212>prt
<213>人工序列(artificialsequence)
<400>2
metaspglnthrtyrserleugluserpheleuasnhisvalglnlys
151015
argaspproasnglnthrgluphealaglnalavalarggluvalmet
202530
thrthrleutrppropheleugluglnasnprolystyrargglnmet
354045
serleuleugluargleuvalgluprogluargvalileglnphearg
505560
valvaltrpvalaspaspargasnglnileglnvalasnargalatrp
65707580
argvalglnpheserseralaileglyprotyrlysglyglymetarg
859095
phehisproservalasnleuserileleulyspheleuglypheglu
100105110
glnthrphelysasnalaleuthrthrleuprometglyglyglylys
115120125
glyglyserasppheaspprolysglylyssergluglygluvalmet
130135140
argphecysglnalaleumetthrgluleutyrarghisleuglyala
145150155160
aspthraspvalproalaglyaspileglyvalglyglyarggluval
165170175
glyphemetalaglymetmetlyslysleuserasnasnthralacys
180185190
valphethrglylysglyleuserpheglyglyserleuileargpro
195200205
glualathrglytyrglyleuvaltyrphethrglualametleulys
210215220
arghisglymetglyphegluglymetargvalservalserglyser
225230235240
glyasnvalalaglntyralaileglulysalametglupheglyala
245250255
argvalilethralaseraspserserglythrvalvalaspgluser
260265270
glyphethrlysglulysleualaargleuilegluilelysalaser
275280285
argaspglyargvalalaasptyralalysglupheglyleuvaltyr
290295300
leugluglyglnglnprotrpserleuprovalaspilealaleupro
305310315320
cysalathrglnasngluleuaspvalaspalaalahisglnleuile
325330335
alaasnglyvallysalavalalagluglyalaasnmetprothrthr
340345350
ileglualathrgluleupheglnglnalaglyvalleuphealapro
355360365
glylysalaalaasnalaglyglyvalalathrserglyleuglumet
370375380
alaglnasnalaalaargleuglytrplysalaglulysvalaspala
385390395400
argleuhishisilemetleuaspilehishisalacysvalgluhis
405410415
glyglygluglygluglnthrasntyrvalglnglyalaasnileala
420425430
glyphevallysvalalaaspalametleualaglnglyvalile
435440445
<210>3
<211>2853
<212>dna
<213>人工序列(artificialsequence)
<400>3
atgtctgctaagaaaaggccttcgggaaacgcagcatttgaacttccaaaacggagaaga60
acctaccaagcttgcatcagctgcagatcaaggaaggtgaaatgtgatcttggtccggtt120
gataacccacacgacccaccgtgtgcacgttgcaaaagggagctaaaaaaatgtattttt180
agctctaataagggaacttcgaacgacttgcctcctaattcgattaatgcaataagtctg240
ccatctctaggtaagagtaagcaagagatacaaaatgactcaacgagcccaattttatcg300
gatgtacctttgtcaagaaaagggattagcagtgaaaaatcatttaaatcagaagggatg360
aagtggaagcttgaactttcctccatgcagaacgccctagagtttctggcccaagctgct420
ggaactgtagcaaaagaaggcgcaaaagaaatcataaaagagaagtccacaacaccgaaa480
ccactgaaaagttctttggatgcgacaaataaatctgctacggacgaggggttgaaacga540
ctttcaaaaagtgacagtactaacacactttacgaaaatacagcagatatgcttaaccac600
acgctgaatacgaataggaaaacatcacaattaatggaagaaatcgggaaagtgagacct660
ccacctacccggaaaattgacgacttcgattacatcggtccagatagcttgcttactaaa720
gaggaggcgattgaactcatagaggcattttttctcacgatgcacccattctttcccaat780
atcccacttcaattgcatgatccgaaagaattagcagaatatccaattttgttttgtgca840
attttgactgtttctgctcgctatcatcctttcgatactttaggattagacaatggagaa900
gatgggatgagacacattgaagttcacgataaattatgggtgtattgccagaagcttata960
tctcaaacaatttgggcagaggctagtactaggtcaattggcaccgtgcttgctttcata1020
attttcacggaatggaacccaaggagtatccactataaatggtctgactatgcaaatgat1080
cctgagttaaataatgtcaacgctagaggaagtaagaatattagtacgaggaaggacgaa1140
gaaggattgaccggtgtcggcgcaatccgtagaagcgaccgaatgtcatggatgctgaca1200
gggtctgcagtaaggctggcgcaagatatgggattcattgaaaacagctcaaaagtattc1260
attgtcactcatatatctgagactaccagtgctatgaatatgaatcaaagatccttatta1320
gcagaatcattcagcgttttaaacttgaatttaggaaaaattgaaaacgatggaaatgaa1380
agcaatgaggattaccttgggaatgaaaaattttatctgaatgagattttgcctgatgaa1440
gaaagtaaactaaggtggaagagagtttttgaaaactcagaaaatgaccacgacaacgag1500
aaaaattttttgactgattgggagagggagtttttaaacgatgagtatgttctatattat1560
tctaataaaaaagatgatactaatctagcacagaatcacataccaccatttccactaaga1620
ttctcattcgcccaaagagcaaaaatagagatcattcgaatcctatccatagcatatgag1680
acgatttactgtgagaagaataagaggaaattggcaacgacagatcagagacacaacctc1740
tctgtcctaagtgttttttctcccttgatagagggctggctcagtaactacagagagctt1800
cttgtacccctttctgatgtccctttctcactagcagataggaagaacaaaaaacaaata1860
tttgataacatcgatagaataaatggtgaaagtattatcacagatttcaattattgtcaa1920
ctttatatcttttccctcgcactgcaagtggatgggaaaacaagcagattgaatatgaat1980
gaaattgtgacaagcgcgaggtatgtggaattagcatacagatctgcaaaagaaatattg2040
agttctgcaaagcgagtttcaaggcaaggaatgctaaaatacatgcccgtaagatgggta2100
atacgaataattagatccattgcgttcatagtaaaatgctacctaacacttactggtagt2160
gaactggcaacaaatccagatgccagaaacattttaaaattaagtgcaatatcggttgac2220
gaaacattcgatatcatccgtgacactgcggttactttgaaagaagctacaccagatgaa2280
ttacatctatgtcaaagatacgcagccatattaatgtacttatgtacagagatgaaactt2340
cgaaaaaaatcttacttagaacgaccacccttattaagagatggcaccactcctctagag2400
agcaatcgcgaatcctcactggagggacaagacttaactaagaagccaatattctctaaa2460
cgtattggatataataaaaccgagactacttttgaaccctcggaaaggcctttgacggag2520
gaaattaatagcaattctcagaattcaaatgatacatcttctaaaggtatcgttgatcct2580
tttgttgaacaaaacaacgatatcacaacagcgttactgaacaatgaactcttccaaggt2640
ccatctctatctgacgaggttacagattggttcggtgctagtgaagatatcgggcttgaa2700
tttgtagaaccatggacagaacttattgagcaacgatatatgcaatgtggagatggtgat2760
aataataatttcgaaaatttatacaacttgttcgtgaatagtaataacatcaataatgat2820
attaataactcaaggccaataacgcgtaaataa2853
<210>4
<211>1344
<212>dna
<213>人工序列(artificialsequence)
<400>4
atggatcagacatattctctggagtcattcctcaaccatgtccaaaagcgcgacccgaat60
caaaccgagttcgcgcaagccgttcgtgaagtaatgaccacactctggccttttcttgaa120
caaaatccaaaatatcgccagatgtcattactggagcgtctggttgaaccggagcgcgtg180
atccagtttcgcgtggtatgggttgatgatcgcaaccagatacaggtcaaccgtgcatgg240
cgtgtgcagttcagctctgccatcggcccgtacaaaggcggtatgcgcttccatccgtca300
gttaacctttccattctcaaattcctcggctttgaacaaaccttcaaaaatgccctgact360
actctgccgatgggcggtggtaaaggcggcagcgatttcgatccgaaaggaaaaagcgaa420
ggtgaagtgatgcgtttttgccaggcgctgatgactgaactgtatcgccacctgggcgcg480
gataccgacgttccggcaggtgatatcggggttggtggtcgtgaagtcggctttatggcg540
gggatgatgaaaaagctctccaacaataccgcctgcgtcttcaccggtaagggcctttca600
tttggcggcagtcttattcgcccggaagctaccggctacggtctggtttatttcacagaa660
gcaatgctaaaacgccacggtatgggttttgaagggatgcgcgtttccgtttctggctcc720
ggcaacgtcgcccagtacgctatcgaaaaagcgatggaatttggtgctcgtgtgatcact780
gcgtcagactccagcggcactgtagttgatgaaagcggattcacgaaagagaaactggca840
cgtcttatcgaaatcaaagccagccgcgatggtcgagtggcagattacgccaaagaattt900
ggtctggtctatctcgaaggccaacagccgtggtctctaccggttgatatcgccctgcct960
tgcgccacccagaatgaactggatgttgacgccgcgcatcagcttatcgctaatggcgtt1020
aaagccgtcgccgaaggggcaaatatgccgaccaccatcgaagcgactgaactgttccag1080
caggcaggcgtactatttgcaccgggtaaagcggctaatgctggtggcgtcgctacatcg1140
ggcctggaaatggcacaaaacgctgcgcgcctgggctggaaagccgagaaagttgacgca1200
cgtttgcatcacatcatgctggatatccaccatgcctgtgttgagcatggtggtgaaggt1260
gagcaaaccaactacgtgcagggcgcgaacattgccggttttgtgaaggttgccgatgcg1320
atgctggcgcagggtgtgatttaa1344
<210>5
<211>29
<212>prt
<213>人工序列(artificialsequence)
<400>5
glyglyalaalathrthralaalacyscysalathrglyglycysala
151015
cyscysthrglythrthralacysalaalathrthrgly
2025
<210>6
<211>35
<212>prt
<213>人工序列(artificialsequence)
<400>6
cyscysthrthrglyglythralacyscyscysthralathrthrthr
151015
thrthrthralathrthrthrcysthrthrthrthralaalaglythr
202530
glycyscys
35
<210>7
<211>31
<212>prt
<213>人工序列(artificialsequence)
<400>7
alaglyglyalaglyalathralathralaalathrglyglyalathr
151015
cysalaglyalacysalathralathrthrcysthrcysthrgly
202530
<210>8
<211>28
<212>prt
<213>人工序列(artificialsequence)
<400>8
cyscysthrthrcysthrcysglyalaglythrthralaalaalathr
151015
cysalacysalacyscyscysthrglycysglycys
2025
- 还没有人留言评论。精彩留言会获得点赞!