一种可变剪接报告载体及其制备方法与流程
本发明涉及基因克隆技术领域,更具体的说是涉及一种可变剪接报告载体及其制备方法。
背景技术:
可变剪接是基因表达的一种普遍调控机制,它能使单个基因产生多个不同种类的mrna。可变剪接包括外显子跳跃,外显子互斥之间的选择,剪接位点的利用和内含子保留等。通过可变剪接机制,可以产生在非翻译区或编码序列上存在差异的mrna,这些差异可能影响mrna的稳定、定位或者翻译。此外,一些剪接mrna亚型可以改变阅读框,产生具有不同作用或定位的差异蛋白亚型。全基因组的研究预测90-95%的人类基因都会发生某种程度的可变剪接。高分辨质谱分析显示在大约2万个人类蛋白质编码基因中,约有37%的产生多种蛋白亚型,表明可变剪接有利于蛋白质组的复杂化。
现已确定可变剪接有助于细胞分化和细胞谱系的确立,组织形成和维持,以及器官发育。由前体mrna(pre-mrna)(包括5'和3'剪接位点,以及外显子和内含子的增强子或沉默序列)中的顺式作用元件,反式作用因子或剪接体的其他成分突变引起的大量人类疾病进一步突显了可变剪接的生物学重要性。组织和器官发育特别适合研究与剪接网络识别相关的功能,细胞命运决定的调节对组织和器官功能至关重要,并且控制其从胚胎到成人的功能过渡。
然而,目前对于可变剪接的功能性影响及其机制研究仍较为有限,在体外研究可变剪接仍然缺乏直观的检测工具,而目前大多数研究均是对可变剪接的分析方法或与病毒、调控因子靶向调节的报告载体,并没有可用于在细胞水平和活体水平可变剪接研究的报告载体。因此,如何提供一种能够用于在细胞水平和活体水平检测可变剪切事件的报告载体成为本领域技术人员亟待解决的问题。
技术实现要素:
有鉴于此,本发明提供了一种可变剪接报告载体及其制备方法,可用于细胞水平和活体水平可变剪切事件的检测。
为了实现上述目的,本发明采用如下技术方案:
一种可变剪接报告载体,可变剪接报告载体为在报告基因表达载体pegfp-c1的报告基因序列内插入两段内含子序列intron1和intron2,intron1与intron2分别具有5’剪切位点,分支位点和3’剪切位点,以保证u1snrnp可以同5’剪切位点结合从而启动剪接,u2af因子同分支位点和3’剪切位点之间的区域结合,使内含子得以形成套索结构发生剪接。
优选地,报告基因为egfp基因。
优选地,intron1序列如seqidno:1所示:
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaataatgagttgactgtgggaactaaagtgtttttttttctctttag;
其中,下划线部分序列依次为5’剪切位点,分支位点和3’剪切位点。
优选地,intron2序列如seqidno:2所示:
gtaagatatttttatacaaagaaaaaaattaatttaactgtaaaatagtaacagtctctaatgatctggcagaagactcagctaattgtcaatttttatttttcctttatag;
其中,下划线部分序列依次为5’剪切位点,分支位点和3’剪切位点。
优选地,可变剪接报告载体的序列如seqidno:3所示。
一种可变剪接报告载体的制备方法,包括如下步骤:
(1)设计并合成序列intron1,将其克隆到载体pegfp-c1的egfp序列中,构建载体pegfp-c1-intron1;
2)设计并合成序列intron2,将其克隆到载体pegfp-c1-intron1的egfp序列中intron1下游,构建得到载体pegfp-c1-asr。
由上述技术方案可知,本发明根据rna的剪接机制设计合成含有5’剪接位点,3’剪接位点以及分支位点的两段内含子序intron1和intron2,将intron1和intron2分别插入报告基因表达载体的报告序列,用以在细胞水平和活体水平检测可变剪接事件,含有剪接位点的内含子的插入便于转录后mrna的正确拼接。
附图说明
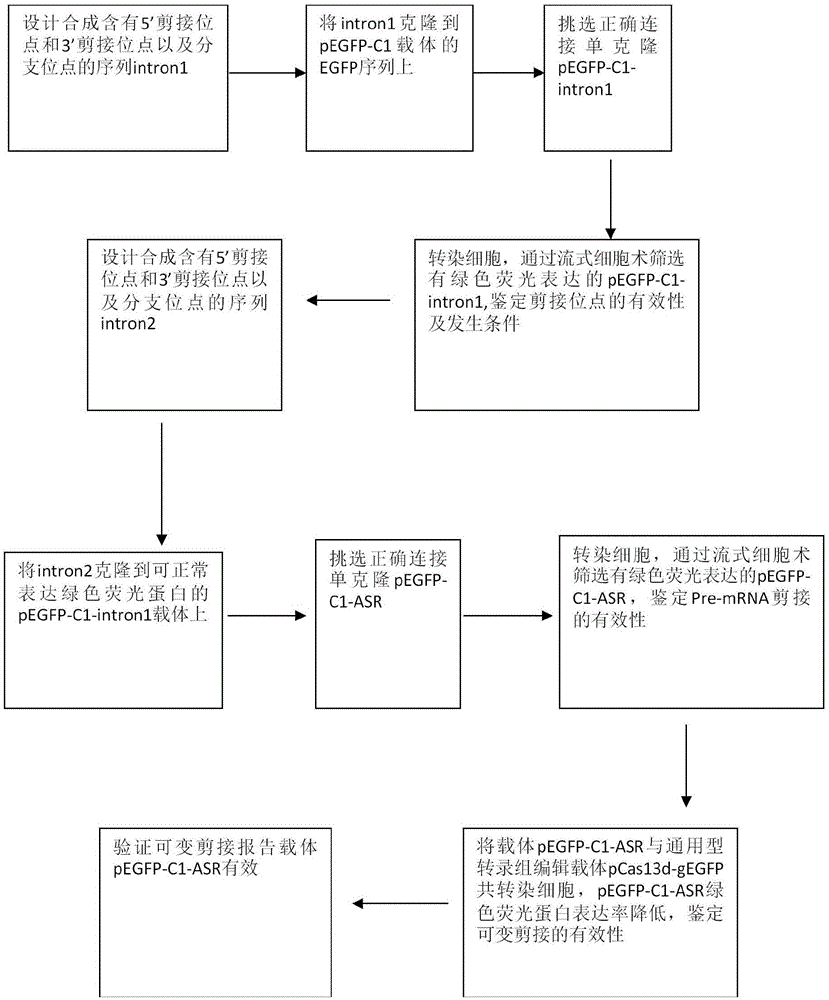
图1所示为可变剪接报告载体的构建流程;
图2所示为载体pegfp-c1-intron1的鉴定电泳图谱;
m:marker;
泳道1:pegfp-c1-intron1双酶切(nhei,xhoi)产物;
泳道2:空载pegfp-c1双酶切(nhei,xhoi)产物;
泳道3:pegfp-c1-intron1质粒;
图3所示为对照载体pegfp-c1-5’ss的鉴定电泳图谱;
m:marker;
泳道1:pegfp-c1-5’ss质粒;
泳道2:pegfp-c1-5’ss双酶切(nhei,xhoi)产物;
泳道3:空载pegfp-c1双酶切(nhei,xhoi)产物;
图4所示为对照载体pegfp-c1-3’ss的鉴定电泳图谱;
m:marker;
泳道1:pegfp-c1-3’ss双酶切(nhei,xhoi)产物;
泳道2:空载pegfp-c1双酶切(nhei,xhoi)产物;
泳道3:pegfp-c1-3’ss质粒;
图5所示为对照载体pegfp-c1-bp的鉴定电泳图谱;
m:marker;
泳道1:pegfp-c1-bp双酶切(nhei,xhoi)产物;
泳道2:空载pegfp-c1双酶切(nhei,xhoi)产物;
泳道3:pegfp-c1-bp质粒;
图6所示为报告载体pegfp-c1-intron1及三个对照载体pegfp-c1-5’ss,pegfp-c1-3’ss,pegfp-c1-bp转染细胞;
a:左图为载体pegfp-c1-intron1明场;右图为载体pegfp-c1-intron1暗场;
b:左图为载体pegfp-c1-5’ss明场;右图为载体pegfp-c1-5’ss暗场;
c:左图为载体pegfp-c1-3’ss明场;右图为载体pegfp-c1-3’ss暗场;
d:左图为载体pegfp-c1-bp明场;右图为载体pegfp-c1-bp暗场;
图7所示为验证intron1剪接功能的pcr电泳图谱;
m:marker;
泳道1:pegfp-c1-intron1质粒;
泳道2:细胞转染pegfp-c1-intron1;
泳道3:细胞转染pegfp-c1-5’ss;
泳道4:细胞转染pegfp-c1-3’ss;
泳道5:细胞转染pegfp-c1-bp;
图8所示为载体pegfp-c1-asr的鉴定电泳图谱;
m:marker;
泳道1:pegfp-c1-asr双酶切(nhei,xhoi)产物;
泳道2:空载pegfp-c1双酶切(nhei,xhoi)产物;
泳道3:pegfp-c1-asr质粒;
图9所示为报告载体pegfp-c1-asr的质粒图谱;
图10所示为报告载体pegfp-c1-asr转染细胞;
左图为明场;右图为暗场;
图11所示为验证pre-mrna剪接功能的pcr电泳图谱;
m:marker;
泳道1:pegfp-c1-asr质粒;
泳道2:细胞转染pegfp-c1-asr;
图12所示为hindiii、noti双酶切鉴定正确连接的单克隆pcas13d-grna;
泳道1:dnamarker;
泳道2、3:正确连接的单克隆;
图13所示为pcas13d-grna的载体图谱;
图14所示为通用型转录组编辑载体pcas13d-gegfp干扰剪切原理图;
图15所示为报告载体pegfp-c1-asr与通用型转录组编辑载体pcas13d-gegfp共转染细胞;
上图为明场;下图为暗场;
图16所示为验证可变剪接功能的pcr电泳图谱;
m:marker;
泳道1:pegfp-c1-asr质粒;
泳道2:细胞共转染pegfp-c1-asr与载体pcas13d-grna。
具体实施方式
下面对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1
如图1所示,本发明构建含有特殊序列intron1和intron2的载体pegfp-c1-asr,用电转染法将载体pegfp-c1-asr转染到细胞,荧光显微镜及流式细胞仪观察标记基因egfp的表达情况,经pcr鉴定,证实基因完成转录后的pre-mrna的能够正确剪切拼接。
将报告载体pegfp-c1-asr与通用型转录组编辑载体pcas13d-gegfp共转染细胞,荧光显微镜观察标记基因egfp的表达情况,经pcr鉴定,证实载体pcas13d-gegfp可以干扰剪接事件,使报告载体发生可变剪接。
可变剪接报告载体构建的具体方法如下:
(1)合成含有5’剪接位点、分支位点以及3’剪接位点的序列intron1,如seqidno:1所示:
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaataatgagttgactgtgggaactaaagtgtttttttttctctttag;
其中,下划线部分序列依次为5’剪切位点,分支位点和3’剪切位点。
该序列合成后使用cloneezpcr克隆试剂盒(南京金斯瑞)将其无缝克隆到载体pegfp-c1(clontech)的egfp序列中,egfp序列被分为egfp-exon1’,egfp-intron1,egfp-exon2’。将连接载体转化到感受态细胞dh5α中,涂板培养,次日挑取阳性克隆,提取质粒用nhei/xhoi双酶切,通过凝胶电泳检测鉴定正确连接单克隆(图2),命名为pegfp-c1-intron1。
由于质粒在大肠杆菌中增殖,用普通的质粒提取方法容易将细菌内毒素带到终产物中,细菌内毒素的存在会影响质粒转染效率和细胞生长,因此,本发明使用去内毒素的质粒提取试剂盒(sigma),以减少内毒素对试验结果的负面影响。
用核酸蛋白测定仪测定质粒的浓度和纯度,经测定,质粒的浓度为974ng/ul,od260/280为1.83,说明质粒较纯,可用于后续的细胞转染。
(2)合成含有5’剪接位点和分支位点的序列5’ss,如seqidno:4所示:
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaataatgagttgactgtgggaactaaagtg
下划线部分序列依次为5’剪接位点和分支位点,该序列合成后克隆到载体pegfp-c1的egfp序列中,鉴定正确连接单克隆(图3),命名为pegfp-c1-5’ss。
合成含有分支位点和3’剪接位点的序列3’ss,如seqidno:5所示:
atacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaataatgagttgactgtgggaactaaagtgtttttttttctctttag
下划线部分序列依次为分支位点以及3’剪接位点,该序列合成后克隆到载体pegfp-c1的egfp序列中,鉴定正确连接单克隆(图4),命名为pegfp-c1-3’ss;
合成含有5’剪接位点和3’剪接位点的序列bp,如seqidno:6所示:
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaagaaagtcatttaccactatctcagtagtaagcttaggaaaatatttaataatgagttgactgtgggaactaaagtgtttttttttctctttag
下划线部分序列依次为5’剪接位点和3’剪接位点,该序列合成后克隆到载体pegfp-c1的egfp序列中,鉴定正确连接单克隆(图5),命名为pegfp-c1-bp;
(3)于含10%fbs的dmem培养液中培养3t3细胞,37℃co2培养箱培养至达80%汇合时,消化,1000r/min离心4min收集细胞,然后用opti-
将cell盐与opti-
向电转染液中分别加入适量报告载体pegfp-c1-intron1及三个对照载体pegfp-c1-5’ss,pegfp-c1-3’ss,pegfp-c1-bp,混匀,添加到洗涤离心后的细胞中,opti-
将上述细胞悬液转移到4mm间隙、黄色盖子、800μl容积的btx电击杯中,静置10min后进行电转染;电转染参数设置如下:电压510v,脉冲时间1ms,电击次数3次。
转染完成后,静置10min,然后将细胞悬液全部转移到90mm培养皿中,加入37℃预温的含10%胎牛血清的dmem完全培养液3ml,置于37℃、5%co2培养箱中培养;待细胞贴壁后,弃去培养液(培养液中含有电转染液影响细胞生长),换成经37℃温育的dmem完全培养液10ml。
培养24h后,转染pegfp-c1-intron1的细胞表达绿色荧光蛋白,转染对照载体的三组细胞不表达绿色荧光蛋白(图6),说明转染pegfp-c1-intron1的细胞中u1snrnp和u2af因子可以与剪切位点及分支位点结合,使目的基因完成转录后能够正确剪切拼接;而转染对照载体的细胞中由于对照载体分别缺乏5’剪接位点、分支位点以及3’剪接位点,导致u1snrnp或u2af因子无法与相应位点正确结合,使内含子序列无法被剪切,绿色荧光蛋白无法正常表达。
转染细胞培养36小时后,经胰蛋白酶消化,离心,pbs悬浮;使用流式细胞仪分析pegfp-c1-intron1转染细胞的egfp荧光强度,并据此对转染细胞进行分选;分选获得的转染细胞2000r/min离心5min后用培养基悬浮,分种于24或96孔培养板,置37℃、5%co2温箱中培养。
使用总rna提取试剂盒(promega公司)分别提取通过流式细胞术筛选得到的成功表达绿色荧光蛋白的细胞rna和转染对照载体的细胞rna,进行rt-pcr,过程参照全式金公司反转录试剂盒进行,每20μl的体系中1μgrna作为模板,按照25℃10min,42℃30min,85℃5s的程序进行反应,反转录所得的cdna于-20℃保存。
利用rna剪接原理在exon1’和exon2’的序列上设计引物:
asr-f:atggtgagcaagggcgagga,seqidno:7;
asr-r:ttatctagatccggtggatc,seqidno:8。
以cdna为模板,设计合成的asr-f、asr-r作为引物,扩增egfp序列,再次对剪接发生条件进行pcr鉴定。转染报告载体pegfp-c1-intron1的细胞中内含子被剪切,转染三个报告载体的细胞中的内含子序列仍然存在,说明只有同时含有5’剪切位点,分支位点和3’剪切位点的内含子才能发生剪接事件(图7)。
(4)合成含有5’剪接位点,分支位点和3’剪接位点的序列intron2,如seqidno:2所示:
gtaagatatttttatacaaagaaaaaaattaatttaactgtaaaatagtaacagtctctaatgatctggcagaagactcagctaattgtcaatttttatttttcctttatag;
其中,下划线部分序列依次为5’剪切位点,分支位点和3’剪切位点,该序列合成后使用cloneezpcr克隆试剂盒将其无缝克隆到载体pegfp-c1-intron1的intron1序列下游,使egfp序列分为egfp-exon1,egfp-intron1,egfp-exon2,egfp-intron2,egfp-exon3。将连接载体转化到感受态细胞dh5α中,涂板培养,次日挑取阳性克隆,提取质粒用nhei/xhoi双酶切,通过凝胶电泳检测,鉴定正确连接单克隆(图8),命名为pegfp-c1-asr,该载体含有可正常发生剪接的两段内含子intron1和intron2,即为可变剪接报告载体(图9),其全序列如seqidno:3所示。
(5)将载体pegfp-c1-asr采用电转染的方法转染细胞,转染24小时后在荧光显微镜下镜检,转染载体的细胞可以观察到绿色荧光蛋白的表达(图10),说明目的基因完成转录后的pre-mrna的能够正确剪切拼接。
转染细胞培养36小时后,通过流式细胞术筛选出成功表达绿色荧光蛋白的细胞,使用总rna提取试剂盒(promega公司)提取细胞rna,进行rt-pcr,过程参照全式金公司反转录试剂盒进行,每20μl的体系中1μgrna作为模板,按照25℃10min,42℃30min,85℃5s的程序进行反应,反转录所得的cdna于-20℃保存。
以其cdna为模板,设计合成的asr-f,asr-r作为引物,扩增egfp序列,再次对pre-mrna的正确剪接进行pcr鉴定,发现成功表达绿色荧光蛋白的细胞中的内含子被剪切,mrna正确拼接(图11)。
(6)对瘤胃球菌cas13d基因的序列进行优化,优化后的cas13d基因如seqidno:9所示。
将优化后的cas13d基因克隆到px459载体上,同源置换出cas9序列,获得载体pcas13d。
设计引物f4(pcii)及引物r4(bglii,clai,ecorv,hindiii,nhei,spei,kpni),以pcas13d为模板,克隆u6promoter原件并且引入酶切位点,得到产物p5。
其中,引物f4及引物r4的序列分别为:
f4:5’-gacatgtgagggcctatttcccatgat-3’;seqidno:10;
r4:5’-cggtacccactagtgctagcaagcttgatatcatcgatagatctggtcttctcgaagacccggg-3’;seqidno:11。
pcii、kpni双酶切pcr产物p5和载体pcas13d,0.8%凝胶回收,t4dna连接酶连接,4℃过夜,转化大肠杆菌dh5α,提质粒,双酶切鉴定正确连接单克隆,结果如图12所示,测序结果正确,命名载体为pcas13d-grna,pcas13d-grna的载体图谱如图13所示。
在pcas13d-grna的多克隆位点插入靶向egfp的guiderna序列,guiderna的序列如下:
caagtaaacccctaccaactggtcggggtttgaaacgtccagctaaagagaaaaaaaaacactttacaagtaaacccctaccaactggtcggggtttgaaacataaaaatatcttaccttgaagttggcctt,seqidno:12,下划线处为dr序列;载体命名为pcas13d-gegfp。
(7)将报告载体pegfp-c1-asr与通用型转录组编辑载体pcas13d-gegfp采用电转染的方法共转染细胞(图14),转染24小时后在荧光显微镜下镜检,发现细胞表达绿色荧光蛋白率降低(图15),细胞培养36h后使用总rna提取试剂盒(promega公司)提取细胞rna,进行rt-pcr,过程参照全式金公司反转录试剂盒进行,每20μl的体系中1μgrna作为模板,按照25℃10min,42℃30min,85℃5s的程序进行反应,反转录所得的cdna于-20℃保存。
以转染细胞的cdna为模板,设计合成的asr-f、asr-r作为引物,扩增egfp序列,pcr鉴定载体pcas13d-gegfp可以干扰剪接事件,使报告载体发生可变剪接,载体插入进去的两段内含子被正确剪切或者受到载体pcas13d-gegfp的干扰,两段内含子及其中间的egfp序列被剪切,导致细胞表达绿色荧光蛋白率降低(图16)。
对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本发明。对这些实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
序列表
<110>西北农林科技大学
<120>一种可变剪接报告载体及其制备方法
<160>12
<170>siposequencelisting1.0
<210>1
<211>224
<212>dna
<213>artificial
<400>1
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaa60
atcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatg120
ctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaat180
aatgagttgactgtgggaactaaagtgtttttttttctctttag224
<210>2
<211>112
<212>dna
<213>artificial
<400>2
gtaagatatttttatacaaagaaaaaaattaatttaactgtaaaatagtaacagtctcta60
atgatctggcagaagactcagctaattgtcaatttttatttttcctttatag112
<210>3
<211>5067
<212>dna
<213>artificial
<400>3
tagttattaatagtaatcaattacggggtcattagttcatagcccatatatggagttccg60
cgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgcccatt120
gacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtca180
atgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgcc240
aagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagta300
catgaccttatgggactttcctacttggcagtacatctacgtattagtcatcgctattac360
catggtgatgcggttttggcagtacatcaatgggcgtggatagcggtttgactcacgggg420
atttccaagtctccaccccattgacgtcaatgggagtttgttttggcaccaaaatcaacg480
ggactttccaaaatgtcgtaacaactccgccccattgacgcaaatgggcggtaggcgtgt540
acggtgggaggtctatataagcagagctggtttagtgaaccgtcagatccgctagcgcta600
ccggtcgccaccatggtgagcaagggcgaggagctgttcaccggggtggtgcccatcctg660
gtcgaggtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtg720
tagaaaatcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttt780
tgaatgctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaata840
tttaataatgagttgactgtgggaactaaagtgtttttttttctctttagctggacggcg900
acgtaaacggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggca960
agctgaccctgaagttcatctgcaccaccggcaagctgcccgtgccctggcccaccctcg1020
tgaccaccctgacctggggcgtgcagtgcttcagccgctaccccgaccacatgaagcagc1080
acgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccatcttcttca1140
aggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtga1200
accgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagc1260
tggagtacaactacatcagccacaacgtctatatcaccgccgacaagcagaagaacggca1320
tcaaggccaacttcaaggtaagatatttttatacaaagaaaaaaattaatttaactgtaa1380
aatagtaacagtctctaatgatctggcagaagactcagctaattgtcaatttttattttt1440
cctttatagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccag1500
cagaacacccccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacc1560
cagtccgccctgagcaaagaccccaacgagaagcgcgatcacatggtcctgctggagttc1620
gtgaccgccgccgggatcactctcggcatggacgagctgtacaagtccggactcagatct1680
cgagctcaagcttcgaattctgcagtcgacggtaccgcgggcccgggatccaccggatct1740
agataactgatcataatcagccataccacatttgtagaggttttacttgctttaaaaaac1800
ctcccacacctccccctgaacctgaaacataaaatgaatgcaattgttgttgttaacttg1860
tttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcacaaataaa1920
gcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttaacgc1980
gtaaattgtaagcgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctc2040
attttttaaccaataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccga2100
gatagggttgagtgttgttccagtttggaacaagagtccactattaaagaacgtggactc2160
caacgtcaaagggcgaaaaaccgtctatcagggcgatggcccactacgtgaaccatcacc2220
ctaatcaagttttttggggtcgaggtgccgtaaagcactaaatcggaaccctaaagggag2280
cccccgatttagagcttgacggggaaagccggcgaacgtggcgagaaaggaagggaagaa2340
agcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcgtaaccac2400
cacacccgccgcgcttaatgcgccgctacagggcgcgtcaggtggcacttttcggggaaa2460
tgtgcgcggaacccctatttgtttatttttctaaatacattcaaatatgtatccgctcat2520
gagacaataaccctgataaatgcttcaataatattgaaaaaggaagagtcctgaggcgga2580
aagaaccagctgtggaatgtgtgtcagttagggtgtggaaagtccccaggctccccagca2640
ggcagaagtatgcaaagcatgcatctcaattagtcagcaaccaggtgtggaaagtcccca2700
ggctccccagcaggcagaagtatgcaaagcatgcatctcaattagtcagcaaccatagtc2760
ccgcccctaactccgcccatcccgcccctaactccgcccagttccgcccattctccgccc2820
catggctgactaattttttttatttatgcagaggccgaggccgcctcggcctctgagcta2880
ttccagaagtagtgaggaggcttttttggaggcctaggcttttgcaaagatcgatcaaga2940
gacaggatgaggatcgtttcgcatgattgaacaagatggattgcacgcaggttctccggc3000
cgcttgggtggagaggctattcggctatgactgggcacaacagacaatcggctgctctga3060
tgccgccgtgttccggctgtcagcgcaggggcgcccggttctttttgtcaagaccgacct3120
gtccggtgccctgaatgaactgcaagacgaggcagcgcggctatcgtggctggccacgac3180
gggcgttccttgcgcagctgtgctcgacgttgtcactgaagcgggaagggactggctgct3240
attgggcgaagtgccggggcaggatctcctgtcatctcaccttgctcctgccgagaaagt3300
atccatcatggctgatgcaatgcggcggctgcatacgcttgatccggctacctgcccatt3360
cgaccaccaagcgaaacatcgcatcgagcgagcacgtactcggatggaagccggtcttgt3420
cgatcaggatgatctggacgaagagcatcaggggctcgcgccagccgaactgttcgccag3480
gctcaaggcgagcatgcccgacggcgaggatctcgtcgtgacccatggcgatgcctgctt3540
gccgaatatcatggtggaaaatggccgcttttctggattcatcgactgtggccggctggg3600
tgtggcggaccgctatcaggacatagcgttggctacccgtgatattgctgaagagcttgg3660
cggcgaatgggctgaccgcttcctcgtgctttacggtatcgccgctcccgattcgcagcg3720
catcgccttctatcgccttcttgacgagttcttctgagcgggactctggggttcgaaatg3780
accgaccaagcgacgcccaacctgccatcacgagatttcgattccaccgccgccttctat3840
gaaaggttgggcttcggaatcgttttccgggacgccggctggatgatcctccagcgcggg3900
gatctcatgctggagttcttcgcccaccctagggggaggctaactgaaacacggaaggag3960
acaataccggaaggaacccgcgctatgacggcaataaaaagacagaataaaacgcacggt4020
gttgggtcgtttgttcataaacgcggggttcggtcccagggctggcactctgtcgatacc4080
ccaccgagaccccattggggccaatacgcccgcgtttcttccttttccccaccccacccc4140
ccaagttcgggtgaaggcccagggctcgcagccaacgtcggggcggcaggccctgccata4200
gcctcaggttactcatatatactttagattgatttaaaacttcatttttaatttaaaagg4260
atctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagttttcg4320
ttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatccttttttt4380
ctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtttg4440
ccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgcagata4500
ccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtagca4560
ccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgataag4620
tcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgggc4680
tgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactgaga4740
tacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggacagg4800
tatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaaac4860
gcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttttg4920
tgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttacgg4980
ttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgattct5040
gtggataaccgtattaccgccatgcat5067
<210>4
<211>207
<212>dna
<213>artificial
<400>4
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaa60
atcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatg120
ctaagaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaat180
aatgagttgactgtgggaactaaagtg207
<210>5
<211>220
<212>dna
<213>artificial
<400>5
atacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaaatcg60
cattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatgctaa120
gaaagtcatttacgttcatccactatctcagtagtaagcttaggaaaatatttaataatg180
agttgactgtgggaactaaagtgtttttttttctctttag220
<210>6
<211>217
<212>dna
<213>artificial
<400>6
gtaaatacagaaaaaatgttgaaataaataagactagtactatctgcctatgtgtagaaa60
atcgcattaccaacattgtaaatgtataaataatgcacaatctcagattttttttgaatg120
ctaagaaagtcatttaccactatctcagtagtaagcttaggaaaatatttaataatgagt180
tgactgtgggaactaaagtgtttttttttctctttag217
<210>7
<211>20
<212>dna
<213>artificial
<400>7
atggtgagcaagggcgagga20
<210>8
<211>20
<212>dna
<213>artificial
<400>8
ttatctagatccggtggatc20
<210>9
<211>2904
<212>dna
<213>artificial
<400>9
atgatcgaaaagaagaagtcatttgcaaagggcatgggagtaaaatcaacacttgtatcc60
ggttcaaaggtatacatgacgacgttcgcagaaggaagcgatgccagacttgaaaagatc120
gttgaaggcgattctatcagatctgtcaacgaaggagaagcgttctcagctgaaatggct180
gataagaatgcaggctacaagatcggtaacgcaaagttcagccacccaaagggctatgct240
gtagttgcaaacaaccccttatacaccggaccggtacagcaggatatgctcggtctgaag300
gaaacgcttgaaaagagatattttggagagtctgccgacggaaatgataatatctgtatt360
caggtcatccataatatcctcgatatcgaaaagatcctcgctgaatatataaccaatgct420
gcttatgcggtaaacaatatttccggtcttgataaggatatcatcggttttggtaagttc480
agtacggtctatacttatgatgagttcaaggatcctgaacatcacagagcagctttcaac540
aataacgataagttaattaatgccatcaaggcacagtatgatgaatttgacaatttcctt600
gataatcctcgtctcggctactttggacaggcttttttcagtaaggaaggcagaaattac660
attatcaattacggcaacgagtgttatgatattcttgctttactcagcggattggctcac720
tgggtagtagctaataatgaggaagaatcaaggatttcccgtacatggctttataatctc780
gacaagaatcttgacaacgaatatatctctactctcaattatctgtatgatagaattaca840
aacgaattaacaaattccttctcaaagaatagtgcagccaacgtaaactatatcgctgaa900
acccttggtattaatcctgctgaatttgcagagcagtatttcagattcagtatcatgaag960
gaacagaagaatctcggtttcaatattactaagctgagagaagtaatgcttgacagaaag1020
gatatgtctgagatccgtaaaaatcataaggtctttgattcaatccgtactaaggtctat1080
actatgatggatttcgttatctacagatattacattgaagaggatgcaaaggttgctgct1140
gccaacaagtctctgccggataacgaaaaaagcctcagtgaaaaggatatctttgttata1200
aatctcagaggaagctttaacgatgatcagaaggatgccctttattatgatgaggccaat1260
cgtatttggagaaagctcgaaaacattatgcacaatatcaaggaattcagaggcaataag1320
acacgtgaatacaagaagaaggatgctccaagactccccagaattcttcctgccggaagg1380
gatgtttccgcgttctcaaagttgatgtacgctcttaccatgttccttgatggtaaggag1440
atcaatgatcttctcaccacgctcatcaataagttcgataacatccagagtttcctcaag1500
gtaatgcctcttatcggagtgaatgcaaagtttgttgaggaatatgccttcttcaaggac1560
agcgcaaagattgctgacgaactcaggctgattaagagctttgccagaatgggagaacct1620
atcgcagatgcaagacgtgctatgtatatcgatgctatcaggattctcggaacaaacctc1680
agctatgatgagcttaaggcccttgccgatactttttcgcttgatgaaaacggcaacaag1740
cttaagaagggcaagcacggcatgagaaacttcatcattaataatgtaatcagtaacaag1800
cgcttccattatctcattcgttacggtgatcctgcacatctccatgagatcgccaagaat1860
gaagctgttgtaaagttcgtcctcggcaggatagctgatatccagaagaagcagggacag1920
aacggaaagaatcagatcgacaggtactatgagacctgtatcggcaaggacaagggcaag1980
tctgtctccgaaaaggttgatgccctcacaaagattatcaccggtatgaactacgatcag2040
ttcgataagaagagaagcgttattgaggatactggaagagaaaacgctgagagagaaaag2100
ttcaagaagatcatcagcctctatcttactgtcatttatcacatccttaagaatattgtt2160
aatatcaatgcgcgttacgttatcggcttccattgcgttgagcgtgatgcacagctctat2220
aaggaaaagggctatgatatcaacctcaagaagctcgaagaaaaggggttttcatcagtc2280
acaaagctgtgtgcaggtattgatgagactgctcctgacaagcgtaaggatgttgaaaag2340
gaaatggctgagcgtgcaaaggaatctatcgatagccttgaatctgcaaatcctaagctt2400
tacgcaaactatatcaagtattctgacgagaagaaggctgaggaatttactagacagatc2460
aaccgtgagaaggcaaagaccgctctgaatgcatatctcagaaatactaagtggaatgtg2520
ataatcagggaagatcttcttagaatcgataataagacatgtacgctctttgcaaataag2580
gccgttgctcttgaagttgcaagatatgttcatgcatatatcaacgatattgccgaagta2640
aacagctatttccagctttatcattacatcatgcagagaatcatcatgaacgaaagatat2700
gaaaagtcttctggaaaggtaagcgaatacttcgatgctgtgaacgatgaaaagaagtac2760
aacgacaggcttctgaagctgttgtgcgttccatttggttactgcatcccgagattcaag2820
aatctctccattgaagctttgttcgacaggaacgaagcagctaagtttgacaaggaaaag2880
aagaaagtatcaggtaattcatag2904
<210>10
<211>27
<212>dna
<213>artificial
<400>10
gacatgtgagggcctatttcccatgat27
<210>11
<211>64
<212>dna
<213>artificial
<400>11
cggtacccactagtgctagcaagcttgatatcatcgatagatctggtcttctcgaagacc60
cggg64
<210>12
<211>132
<212>dna
<213>artificial
<400>12
caagtaaacccctaccaactggtcggggtttgaaacgtccagctaaagagaaaaaaaaac60
actttacaagtaaacccctaccaactggtcggggtttgaaacataaaaatatcttacctt120
gaagttggcctt132
- 还没有人留言评论。精彩留言会获得点赞!