夹杂活性降低的核苷酶剂的制作方法
本发明涉及一种新型核苷酶剂。详细而言涉及对减少啤酒或啤酒类饮料中的嘌呤体有用的核苷酶剂。本申请基于2018年2月9日申请的日本专利申请第2018-022542号主张优先权,以参照的形式引用该专利申请的全部内容。
背景技术:
由于饮食生活的欧美化、酒精摄取量的增加,高尿酸血症和以其为基础的痛风的患病率有上升趋势。作为这些疾病的原因之一,食品或饮料中的嘌呤体被视为问题。例如,肝类、鱼精或鱼卵、一部分鱼贝类等含有大量嘌呤体。另外,酒精饮料、特别是酿造酒(啤酒、葡萄酒等)也含有较多的嘌呤体。由于酒精饮料被日常摄取的情况很多,因此,作为高尿酸血症和痛风的风险因素被认为很重要。例如,对于啤酒、发泡酒等啤酒类饮料,通常在100ml中含有3~8mg左右的来自原料(麦芽等)的嘌呤体,迫切希望提供降低嘌呤体含量的啤酒/啤酒类饮料(低嘌呤体啤酒/啤酒类饮料)。
迄今为止尝试了减少啤酒中的嘌呤体。例如,在专利文献1所记载的方法中,在制造工序中,通过使酶(嘌呤核苷磷酸化酶或核苷酶)与麦芽汁作用,将麦芽汁中的嘌呤核苷分解成核糖和游离嘌呤碱基。游离嘌呤碱基在发酵中被酵母同化。其结果,啤酒产品中的嘌呤体(游离嘌呤碱基、核苷酸、核苷)的量减少。专利文献2中也报告了对减少啤酒中的嘌呤体有用的核苷酶。另外,还提出了通过使用吸附剂而除去嘌呤体的方法(专利文献3)。专利文献4也提及了减少啤酒中的嘌呤体可利用核苷酶,并且公开了各种微生物具有核苷分解活性。专利文献5中公开了来自微生物的核苷酶的一个例子。应予说明,已知在啤酒的制造中酵母发酵的嘌呤体代谢路径的终末物质为黄嘌呤,酵母发酵使黄嘌呤增加(专利文献6)。
现有技术文献
专利文献
专利文献1:日本特许第3824326号公报
专利文献2:日本特许第3824353号公报
专利文献3:日本特开2004-113189号公报
专利文献4:国际公开第96/025483号小册子
专利文献5:日本特开平10-57063号公报
专利文献6:日本特开2012-125205号公报
技术实现要素:
如上所述,虽然报告了用于除去啤酒中的嘌呤体的各种技术,但没有被实用化的例子,对制造低嘌呤体啤酒的技术的需求仍然很高。另外,对于发泡酒等啤酒类饮料,虽然通过减少麦芽使用率或者使用麦芽以外的原料能够减少嘌呤体含量,但使用的原料受限制,因此,不能说是根本性的解决对策。因此,本发明的课题在于提供特别是对减少啤酒或啤酒类饮料中的嘌呤体有用的酶剂及其用途。
在上述课题下,本发明人等为了开发对制造低嘌呤体啤酒/啤酒类饮料有用的酶剂进行了深入研究。嘌呤体存在制造过程中能够被酵母同化的嘌呤体(同化性嘌呤体)和无法同化的嘌呤体(非同化性嘌呤体)。如果利用核苷酶则能够减少啤酒/啤酒类饮料中的嘌呤体,但无法除去非同化性嘌呤体的情况或因夹杂酶活性而生成非同化性嘌呤体的情况下,在最终产品(啤酒)中仍残留嘌呤体。为了解决该问题,想到在啤酒/啤酒类饮料的制造过程中抑制作为非同化性嘌呤体之一的黄嘌呤的生成是有效的,并展开了研究。其结果,成功地获得催化由鸟嘌呤生成黄嘌呤的脱酰胺反应的鸟嘌呤脱氨酶活性低的核苷酶剂。对该新型核苷酶剂的效果进行了评价,结果确认了对减少非同化性嘌呤体(换言之,同化性嘌呤体增多)有用。另外,还成功地确定出制造希望为低嘌呤体产品的啤酒/啤酒类饮料所要求的核苷酶剂的特性(相对于核苷酶活性的鸟嘌呤脱氨酶活性的程度)。
如上进行了深入研究,结果成功地获得了对制造低嘌呤体啤酒/啤酒类饮料极其有用的新型核苷酶剂。基于该成果,提供以下的发明。
[1]一种核苷酶剂,其特征在于,相对于核苷酶活性的鸟嘌呤脱氨酶活性、即活性比(u/u)为0.4以下。
[2]根据[1]所述的核苷酶剂,其中,上述活性比为0.36以下。
[3]根据[1]或[2]所述的核苷酶剂,其中,所含的核苷酶含有序列号1或序列号21的氨基酸序列或者与该序列号1或序列号21的氨基酸序列85%以上相同的氨基酸序列、或者序列号2或序列号22的氨基酸序列或者与该序列号2或序列号22的氨基酸序列88%以上相同的氨基酸序列。
[4]根据[3]所述的核苷酶剂,其中,上述核苷酶的氨基酸序列为与序列号1或序列号21的氨基酸序列或者序列号2或序列号22的氨基酸序列90%以上相同的氨基酸序列。
[5]根据[1]或[2]所述的核苷酶剂,其中,所含的核苷酶具有下述的酶学性质:
(1)作用:催化将嘌呤核苷水解成d-核糖和嘌呤碱基的反应,在腺苷、腺嘌呤、肌苷、次黄嘌呤、鸟苷、鸟嘌呤和黄嘌呤的存在下也显示活性,
(2)分子量:不含n型糖链时的分子量约为49kda(利用sds-page测得),
(3)最适温度:55℃~60℃,
(4)温度稳定性:在55℃以下稳定(ph6.0、30分钟)。
[6]根据[5]所述的核苷酶剂,其中,所含的核苷酶进一步具有下述的酶学性质:
(5)最适ph:3.5,
(6)ph稳定性:在ph3.5~7.5的范围稳定(30℃、30分钟)。
[7]根据[1]或[2]所述的核苷酶剂,其中,所含的核苷酶具有下述的酶学性质:
(1)作用:催化将嘌呤核苷水解成d-核糖和嘌呤碱基的反应,在腺苷、腺嘌呤、肌苷、次黄嘌呤、鸟苷、鸟嘌呤和黄嘌呤的存在下也显示活性,
(2)分子量:不含n型糖链时的分子量约为40kda(利用sds-page测得),
(3)最适温度:50℃~55℃,
(4)温度稳定性:在65℃以下稳定(ph4.5、60分钟)。
[8]根据[7]所述的核苷酶剂,其中,所含的核苷酶进一步具有下述的酶学性质:
(5)最适ph:4.5,
(6)ph稳定性:在ph3.5~7.5的范围稳定(30℃、30分钟)。
[9]根据[3]~[8]中任一项所述的核苷酶剂,其中,上述核苷酶来自多色青霉。
[10]根据[9]所述的核苷酶剂,其中,上述多色青霉为ifo7569株或其变异株。
附图说明
图1是糖化(进料)试验的反应工序。
图2是麦芽汁中的嘌呤体量的比较。通过高效液相色谱分析糖化后的麦芽汁中的各嘌呤体量。
图3是来自多色青霉ifo7569株的核苷酶的作用温度区域。在7种的嘌呤体存在下,在各温度条件下进行酶反应,求出游离嘌呤碱基比率。
图4是来自多色青霉ifo7569株的核苷酶的作用ph区域。在7种的嘌呤体存在下,在各ph条件下进行酶反应,求出游离嘌呤碱基比率。
图5是从多色青霉ifo7569株纯化核苷酶。示出deaehp柱色谱的结果。
图6是各纯化酶(峰1~3)的分子量的测定结果(sds-page)。左边为峰1、2的结果。右边为峰3的结果。将pngasef处理后的样品(“无糖链”的泳道)和无处理的样品(“有糖链”的泳道)进行电泳,进行cbb染色。左端的泳道为分子量标记(肌球蛋白(200kda)、β-半乳糖苷酶(116.3kda)、磷酸化酶b(97.4kda)、bsa(66.3kda)、谷氨酸脱氢酶(55.4kda)、乳酸脱氢酶(36.5kda)、碳酸脱氢酶(31.0kda)、胰蛋白酶抑制剂(21.5kda)、溶菌酶(14.4kda)、抑肽酶(6.0kda)、胰岛素b链(3.5kda)、胰岛素a链(2.5kda))。
图7是各纯化酶(峰1~3)的分子量。也示出了n末端氨基酸分析的结果。
图8是基因克隆中使用的探针的序列。上段:pn1用(序列号18)、下段:pn2用(序列号19)。
图9是基因克隆的结果。示出编码峰3的酶(pn1)的基因组序列(上段,序列号4)和编码峰1、2的酶(pn2)的基因组序列(下段,序列号6)。
图10是基因克隆的结果。示出编码峰3的酶(pn1)的cdna序列(上段,序列号3)和编码峰1、2的酶(pn2)的cdna序列(下段,序列号5)。
图11是基因克隆的结果。示出峰3的酶(pn1)的氨基酸序列(上段,序列号1)和峰1、2的酶(pn2)的氨基酸序列(下段,序列号2)。
图12是基因克隆的结果。将cdna碱基数、内含子数、氨基酸长度、分子量和推定pi在峰3的酶(pn1)与峰1、2的酶(pn2)之间进行比较。
图13是纯化酶(pn1)的最适温度。
图14是纯化酶(pn1)的温度稳定性。
图15是纯化酶(pn1)的最适ph。
图16是纯化酶(pn1)的ph稳定性。
图17是重组生产的酶(pn2)的电泳的结果。
图18是纯化酶(pn2)的最适温度。
图19是纯化酶(pn2)的温度稳定性。
图20是纯化酶(pn2)的最适ph。
图21是纯化酶(pn2)的ph稳定性。
图22是使用重组生产的酶(pn2)的糖化(进料)试验的结果。
图23是糖化(进料)试验的反应工序。
图24是麦芽汁中的嘌呤体量的比较。以ph4.5~5.5进行糖化试验。通过高效液相色谱分析糖化后的麦芽汁中的各嘌呤体量。
图25是相对于核苷酶活性的鸟嘌呤脱氨酶活性与麦芽汁中的同化性嘌呤构成比的关系。
具体实施方式
1.核苷酶剂
本发明涉及对制造低嘌呤体啤酒或啤酒类饮料有用的核苷酶剂。“啤酒类饮料”是指啤酒风味的饮料,本发明中,优选为原料的一部分使用了麦芽的发酵饮料,更优选为通过在发酵工序前包括由麦芽制造麦芽汁的工序的方法制造的发酵饮料。
本发明的核苷酶剂主要用途是制造低嘌呤体啤酒或啤酒类饮料,但其它的用途也可以使用本发明的核苷酶剂。例如,为了减少除啤酒或啤酒类饮料以外的食品或饮料中的嘌呤体,也可以使用本发明的核苷酶剂。如果在食品或饮料的制造过程中使用本发明的核苷酶剂,则能够将来自原料的嘌呤核苷转换成游离嘌呤碱基。如果游离嘌呤碱基在其后的制造过程中被除去,则可获得嘌呤体含量减少的食品·饮料。因此,可以对在制造过程中可除去游离嘌呤碱基的食品或饮料的制造使用本发明的核苷酶剂。对应的食品·饮料的例子为利用了基于能够将游离嘌呤碱基同化的微生物的发酵的食品和饮料,即发酵食品和发酵饮料。具体而言,可举出各种腌渍物、味噌、酱油、酸奶、发酵乳、乳酸菌饮料、绍兴酒、葡萄酒等。在这些食品、饮料的制造中采用本发明时,例如,将本发明的核苷酶剂添加到发酵前或发酵中的原料使其发挥作用,将原料中的嘌呤核苷分解成d-核糖和嘌呤碱基。生成的嘌呤碱基典型的是在发酵中被微生物同化。其结果,得到嘌呤体的总量减少的食品或者饮料。
通过相对于核苷酶活性的鸟嘌呤脱氨酶活性、即活性比(u/u)为0.4以下来表征本发明的核苷酶剂。即,相对于作为主成分的核苷酶的活性的夹杂鸟嘌呤脱氨酶活性低。
使用本发明的核苷酶剂制造低嘌呤体的啤酒时,典型的是在麦芽汁中添加发明的核苷酶剂,将麦芽汁中的嘌呤核苷分解成d-核糖和嘌呤碱基。已知啤酒的发酵中使用的酵母通常无法同化嘌呤核苷,但可同化游离嘌呤碱基中的鸟嘌呤和腺嘌呤。因此,如果通过本发明的核苷酶剂的作用将麦芽汁中的嘌呤核苷转换成游离嘌呤碱基,则在发酵工序中酵母同化游离嘌呤碱基,得到嘌呤体的总量减少的啤酒(制造啤酒类饮料的情况下也以此为准)。
核苷酶剂中夹杂的鸟嘌呤脱氨酶是催化将同化性嘌呤的鸟嘌呤转换成作为非同化性嘌呤的黄嘌呤的反应的酶。如果夹杂鸟嘌呤脱氨酶活性高,则使核苷酶剂作用时非同化性嘌呤(黄嘌呤)的生成量变多,最终产品(啤酒)中的残留嘌呤体量增大。因此,为了获得低嘌呤体的啤酒,优选使用夹杂鸟嘌呤脱氨酶活性低的核苷酶剂,提高麦芽汁中的同化性嘌呤构成比(同化性嘌呤与非同化性嘌呤的比率)。同化性嘌呤构成比取决于利用作为核苷酶剂的主成分的嘌呤核苷酶进行的同化性嘌呤的生成和利用夹杂鸟嘌呤脱氨酶进行的非同化性嘌呤(黄嘌呤)的生成。换言之,相对于核苷酶活性的鸟嘌呤脱氨酶活性(活性比)是同化性嘌呤构成比的决定因素。若考虑最终产品(啤酒)中的残留嘌呤量,则酶反应后的麦芽汁中的同化性嘌呤构成比优选为90%以上。优选使同化性嘌呤构成比为91%以上,更优选为92%以上,更进一步优选为93%以上,进一步优选为94%以上,最优选为95%以上。根据后述的实施例中示出的实验结果,相对于核苷酶活性的鸟嘌呤脱氨酶活性与同化性嘌呤构成比的关系如图25所示,为了使同化性嘌呤体构成比为90%以上,需要使相对于核苷酶活性的鸟嘌呤脱氨酶活性为0.4(u/u)以下。因此本发明的核苷酶剂为了实现其目的,用“相对于核苷酶活性的鸟嘌呤脱氨酶活性、即活性比(u/u)为0.4(u/u)以下”这样的特性进行表征。为了进一步提高同化性嘌呤构成比,相对于核苷酶活性的鸟嘌呤脱氨酶活性优选为0.36(u/u)以下,进一步优选为0.3(u/u)以下,更优选为0.2(u/u)以下,更进一步优选为0.1(u/u)以下。
作为本发明的核苷酶剂的主成分的核苷酶可以通过培养产生核苷酶的微生物(核苷酶产生株)而得到。可采用苍白杆菌(ochrobactrum)属、链球菌(streptococcus)属、片球菌(pediococcus)属、明串珠菌(leuconostoc)属、乳杆菌(lactobacillus)属、埃希菌(escherichia)属、柠檬酸杆菌(citrobacter)属、沙雷氏菌(serratia)属、产碱菌(alcaligenes)属、黄杆菌(flavobacterium)属、芽孢杆菌(bacillus)属、棒杆菌(corynebacterium)属、葡萄球菌(staphylococcus)属、节杆菌(arthrobacter)属、丛毛单胞菌(comamonas)属、假单胞菌(pseudomonas)属、克鲁维酵母(kluyveromyces)属、酵母(saccharomyces)属、德巴利酵母(debaryomyces)属、毕赤酵母(pichia)属、汉逊酵母(hansenula)属、掷孢酵母(sporobolomyces)属、锁掷酵母(sporidiobolus)属、曲霉(aspergillus)属、青霉(penicillium)属等微生物作为核苷酶产生株。核苷酶产生株可以为野生株,也可以为变异株(例如通过紫外线照射得到变异株)。举出核苷酶产生株的具体例,有人苍白杆菌(ochrobactrumanthropi)(fermbp-5377)、嗜柠檬酸链球菌(streptococcuscitrovorum)、戊糖片球菌(pediococcuspentosaceus)(ifo3182)、葡聚糖明串珠菌(leuconostocdextranicum)、植物乳杆菌(lactobacillusplantarum)(阿拉伯糖乳杆菌(lactobacillusarabinosus))(ifo3070)、植物乳杆菌(lactobacillusplantarum)(黄瓜乳杆菌(lactobacilluscucumeris))(ifo3074)、大肠杆菌b无生物素(escherichiacolibbiotinless)、弗氏柠檬酸杆菌(citrobacterfreundii)(ifo13546)、粘质沙雷氏菌(serratiamarcescens)(ifo3736)、粪产碱菌(alcaligenesfaecalis)、脑膜败血黄杆菌(flavobacteriummeningosepticum)(dsm2800)、蜡样芽孢杆菌(bacilluscereus)、谷氨酸棒杆菌(corynebacteriumglutamicum)(atcc13060)、金黃葡萄球菌(staphylococcusaureus)(ifo3060)、产脲节杆菌(arthrobacterureafaciens)(球形节杆菌(arthrobacterglobiformis))(ifo12140)、睾丸酮丛毛单胞菌(comamonastestosteroni)(德阿昆假单孢菌(pseudomonasdacunhae))(ifo12048)、恶臭假单胞菌(pseudomonasputida)、bacillusaneurinolyticus、苏云金芽孢杆菌(bacillusthuringiensis)(ifo3951)、马克斯克鲁维酵母(kluyveromycesmarxianus)(酿酒酵母(saccharomycesmaxianus))(ifo0277)、debaryomycespseudopolymorphus、pichiapseudopolymorpha(ifo1026)、荚膜毕赤酵母(pichiacapsulata)(碎囊汉逊酵母(hansenulacapsulata))(ifo0721)、赭色掷孢酵母(sporobolomycessalmonicolor)(ifo1038)、赭色锁掷酵母(sporidiobulussalmonicolor)(香气掷孢酵母(sporobolomycesodorus))(ifo1035)、黑曲霉(aspergillusniger)(ifo4416)、小刺青霉(penicilliumspinulosum)(iam7047)、泡盛曲霉(aspergillusawamori)(ifo4033)、米曲霉(aspergillusoryzae)(iam2630)、黄曲霉(aspergillusflavus)(ifo5839)、土曲霉(aspergillusterreus)(ifo5445)、酱油曲霉(aspergillussojae)(ifo4386)、寄生曲霉(aspergillusparasiticus)(ifo4082)、青霉属(penicilliumsp.)。应予说明,也可以使用导入了从核苷酶产生株获取的核苷酶基因(或者对该基因进行修饰而得的基因)的宿主微生物作为核苷酶产生株。
从产生株获取的核苷酶的夹杂鸟嘌呤脱氨酶活性高的情况下,例如,可以通过适当地组合各种色谱(离子交换色谱、疏水色谱、亲和色谱等)、热处理、ph处理、盐析等进行纯化,将夹杂鸟嘌呤脱氨酶活性降低至需要的程度。应予说明,也可以通过对产生株实施变异处理(通过紫外线、x射线、γ射线等的照射,利用亚硝酸、羟胺、n-甲基-n’-硝基-n-亚硝基胍等进行的处理等),获得相对于核苷酶活性的夹杂鸟嘌呤脱氨酶活性低的变异株。作为指标的“相对于核苷酶活性的夹杂鸟嘌呤脱氨酶活性”可以通过后述的方法来评价。
优选将来自多色青霉(penicilliummulticolor)的核苷酶作为主成分构成本发明的核苷酶剂。此处的“来自多色青霉的核苷酶”是指被分类为多色青霉的微生物(可以为野生株,也可以为变异株)生产的核苷酶、或者利用多色青霉(可以为野生株,也可以为变异株)的核苷酶基因并通过基因工程学方法得到的核苷酶。因此,利用导入了从多色青霉获取的核苷酶基因(或者对该基因进行修饰而得的基因)的宿主微生物生产的重组体也属于“来自多色青霉的核苷酶”。
特别优选构成本发明的核苷酶剂的核苷酶为从多色青霉(penicilliummulticolor)获取的二种核苷酶(以下与实施例中的表述对应地称为“pn1”和“pn2”中的任一个(也可以为其等效物),在一个方式中,本发明的核苷酶剂中含有这二个核苷酶两者(也可以为其等效物)。此处的等效物是指具有等效的氨基酸序列且发挥同等的功能(具体而言酶活性)的酶。将pn1的氨基酸序列示于序列号1,将pn2的氨基酸序列示于序列号2。另外,将pn1的成熟体的氨基酸序列示于序列号21,将pn2的成熟体的氨基酸序列示于序列号22。构成优选方式的核苷酶剂的核苷酶含有序列号1的氨基酸序列或序列号21的氨基酸序列、或者序列号2的氨基酸序列或序列号22的氨基酸序列、或者与这些氨基酸序列中的任一个等效的氨基酸序列。此处的“等效的氨基酸序列”是指与作为基准的序列(序列号1的氨基酸序列、序列号21的氨基酸序列、序列号2的氨基酸序列或者序列号22的氨基酸序列)一部分不同,但该不同不会对蛋白质的功能(此处指核苷酶活性)造成实质性影响的氨基酸序列。因此,具有由等效的氨基酸序列构成的多肽链的酶显示核苷酶活性。活性的程度只要能够发挥作为核苷酶的功能就没有特别限定。但是,优选与具有由基准序列构成的多肽链的酶同等程度或者比其高。
“氨基酸序列的一部分不同”典型的是通过构成氨基酸序列的1~几个(上限例如为3个、5个、7个、10个)氨基酸的缺失、替换或者1~几个(上限例如为3个、5个、7个、10个)的氨基酸的附加、插入或者它们的组合而使氨基酸序列发生变异(变化)。此处的氨基酸序列的不同只要可保持核苷酶活性就被允许(活性也可以稍微有变动)。只要满足该条件,则氨基酸序列不同的位置没有特别限定,另外,可以在多个位置产生不同。此处的“多个”,对于序列号1或序列号21的氨基酸序列而言是相当于小于全部氨基酸的约15%的数量,优选为相当于小于约10%的数量,进一步优选为相当于小于约5%的数量,更进一步优选为相当于小于约3%的数量,最优选为相当于小于约1%的数量。另一方面,对于序列号2或序列号22的氨基酸序列而言是相当于小于全部氨基酸的约12%的数量,优选为相当于小于约10%的数量,进一步优选为相当于小于约5%的数量,更进一步优选为相当于小于约3%的数量,最优选为相当于小于约1%的数量。即,在序列号1或序列号21的氨基酸序列成为基准的情况下,等效蛋白质具有例如约85%以上、优选约90%以上、进一步优选约95%以上、更进一步优选约98%以上、最优选约99%以上的同源性,在序列号2或序列号22的氨基酸序列成为基准的情况下,具有例如约88%以上、优选约90%以上、进一步优选约95%以上、更进一步优选约98%以上、最优选约99%以上的同源性。应予说明,氨基酸序列的不同可以在多个位置产生。
优选通过在对核苷酶活性非必需的氨基酸残基中产生保守性氨基酸替换而得到等效的氨基酸序列。此处的“保守性氨基酸替换”是指将某个氨基酸残基替换成具有同样性质的侧链的氨基酸残基。氨基酸残基根据其侧链而被分为碱性侧链(例如赖氨酸、精氨酸、组氨酸)、酸性侧链(例如天冬氨酸、谷氨酸)、无电荷极性侧链(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、非极性侧链(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、β分支侧链(例如苏氨酸、缬氨酸、异亮氨酸)、芳香族侧链(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)之类的几个家族。保守性氨基酸替换优选为相同家族内的氨基酸残基间的替换。
然而,两个氨基酸序列或两个核酸序列(以下使用“两个序列”作为包括它们的术语)的同源性(%)例如可以通过以下的步骤来确定。首先,以能够进行最佳的比较的方式将两个序列进行排列(例如,可以在第一序列导入空位(gap)而使其与第二序列的比对最佳化)。第一序列的特定位置的分子(氨基酸残基或核苷酸)与第二序列的对应的位置的分子相同时,可以说该位置的分子相同。两个序列的同源性是该两个序列共同的相同位置的数量的函数(即,同源性(%)=相同位置的数量/位置的总数×100),优选将比对的最佳化所需的空位的数量和尺寸也考虑在内。
两个序列的比较和同源性的确定可以使用数学算法来实现。作为可用于序列比较的数学算法的具体例,有在karlin和altschul(1990)proc.natl.acad.sci.usa87:2264-68中记载的、在karlin和altschul(1993)proc.natl.acad.sci.usa90:5873-77中进行了改变的算法,但并不限定于此。这样的算法已被编入altschul等(1990)j.mol.biol.215:403-10中记载的nblast程序和xblast程序(版本2.0)。为了得到等效的核苷酸序列,例如只要在nblast程序中使score=100、wordlength=12进行blast核苷酸检索即可。为了得到等效的氨基酸序列,例如只要在xblast程序中使score=50、wordlength=3进行blast多肽检索即可。为了得到用于比较的空位比对,可以利用altschul等(1997)aminoacidsresearch25(17):3389-3402中记载的gappedblast。在利用blast和gappedblast的情况下,可以使用对应的程序(例如xblast和nblast)的默认参数。详细而言,请参照http://www.ncbi.nlm.nih.gov。作为可用于序列比较的其它数学算法的例子,有myers和miller(1988)computapplbiosci.4:11-17中记载的算法。这样的算法例如已被编入可在genestream网络服务器(ighmontpellier,法国)或isrec服务器中利用的align程序。在氨基酸序列的比较中利用align程序的情况下,例如可以使用pam120残基质量表,使空位长罚分=12、空位罚分=4。
可以利用gcg软件包的gap程序,并使用blossom62矩阵或pam250矩阵,使空位权重=12、10、8、6或4,空位长权重=2、3或4来确定两个氨基酸序列的同源性。另外,可以利用gcg软件包(可在http://www.gcg.com中利用)的gap程序,使空位权重=50、空位长权重=3来确定两个核酸序列的相同度。
构成本发明的核苷酶剂的核苷酶可以为更大的蛋白质(例如融合蛋白)的一部分。作为在融合蛋白中附加的序列,例如可举出多重组氨酸残基这样的对纯化有用的序列、确保重组生产时的稳定性的附加序列等。
具有上述氨基酸序列的核苷酶可以通过基因工程学方法容易地制备。例如可以通过用编码核苷酶的dna转化适当的宿主细胞(例如大肠杆菌),回收在转化体内表达的蛋白质而制备。所回收的蛋白质可以根据目的适当地纯化。如果这样作为重组蛋白质得到本酶,则可以进行各种修饰。例如,如果将编码核苷酶的dna和其它适当的dna插入相同的载体并使用该载体进行重组蛋白质的生产,则能够得到由连接有任意的肽或蛋白质的重组蛋白质构成的核苷酶。另外,可以实施糖链和/或脂质的附加、或者产生n末端或c末端的处理这样的修饰。通过如上所述的修饰,能够简化重组蛋白质的提取、纯化,或者附加生物学功能等。
也可以用以下的酶学性质来表征pn1和pn2。
<pn1的酶学性质>
(1)作用
pn1为核苷酶,催化将嘌呤核苷水解成d-核糖和嘌呤碱基的反应。嘌呤核苷是嘌呤碱基与糖的还原基以n-糖苷键进行键合而成的糖苷。嘌呤核苷的例子为腺苷、鸟苷、肌苷。另外,嘌呤碱基是具有嘌呤骨架的碱基的统称,具体例为腺嘌呤、鸟嘌呤、次黄嘌呤、黄嘌呤。应予说明,除嘌呤核苷和嘌呤碱基以外,还包含嘌呤核苷酸等,将具有嘌呤骨架的化合物统称为嘌呤体。
pn1在腺苷、腺嘌呤、肌苷、次黄嘌呤、鸟苷、鸟嘌呤和黄嘌呤的存在下也显示活性。即,不会受到由分解产物带来的实质性抑制。该特征在将本酶应用于食品、饮料的制造方面特别重要。利用显示该特征的pn1,在食品、饮料的制造过程中,能够将来自原料的嘌呤核苷有效地分解。
(2)分子量
pn1为天然型时含有糖链(即为糖蛋白),n型糖链除去前的分子量约为53kda(用sds-page测定的分子量)。如果通过凝胶渗透色谱进行测定,则约为126kda,推测形成了二聚体。另一方面,如果在n型糖链除去后用sds-page测定分子量,则显示约为49kda。因此,不含n型糖链时的本酶的分子量约为49kda(用sds-page测定的分子量)。
(3)最适温度
pn1的最适温度为55℃~60℃。如此最适温度高在用于经历较高温下的处理工序的食品、饮料的制造中是有利的。最适温度可以通过使用乙酸缓冲液(ph4.3)并以鸟苷作为底物,对作为反应产物的核糖进行定量来评价。
(4)温度稳定性
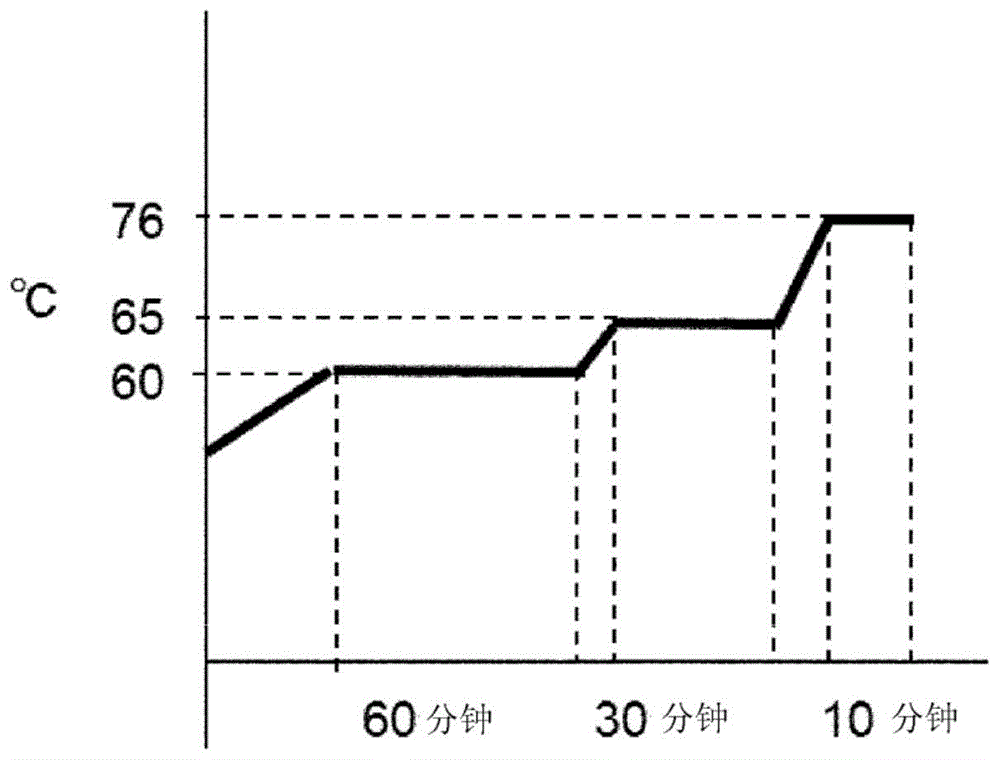
pn1在乙酸缓冲液(ph4.5)中处理60分钟时,在45℃以下的温度条件下维持80%以上的活性。因此,例如如果处理时的温度为5℃~45℃的范围,则处理后的残留活性为80%以上。
另一方面,将pn1在磷酸缓冲液(ph6.0)中处理30分钟时,在55℃以下的温度条件下维持80%以上的活性。因此,例如如果处理时的温度为5℃~55℃的范围,则处理后的残留活性为80%以上。
如此显示优异的温度稳定性的pn1可以在啤酒的进料工序等较高温的条件下也显示高活性。
可以用以下的酶学性质(5)和(6)进一步表征pn1。
(5)最适ph
pn1的最适ph为3.5。最适ph例如可以基于对于ph2.5~3.5的ph区域在柠檬酸缓冲液、对于ph3.5~5.5的ph区域在乙酸缓冲液、对于ph5.5~6.5的ph区域在磷酸钾缓冲液中测得的结果来判断。
(6)ph稳定性
pn1在宽的ph区域显示稳定的活性。例如,如果供于处理的酶溶液的ph为3.5~7.5的范围内,则30℃、30分钟的处理后,显示最大活性的80%以上的活性。另外,在50℃、60分钟的处理的情况下,如果供于处理的酶溶液的ph为3.5~7.5的范围内,则处理后,显示最大活性的80%以上的活性。应予说明,ph稳定性例如可以基于对于ph2.5~3.5的ph区域在柠檬酸缓冲液、对于ph3.5~5.5的ph区域在乙酸缓冲液、对于ph5.5~6.5的ph区域在磷酸钾缓冲液中测得的结果来判断。
<pn2的酶学性质>
(1)作用
pn2为核苷酶,催化将嘌呤核苷分解成d-核糖和嘌呤碱基的反应。
pn2在腺苷、腺嘌呤、肌苷、次黄嘌呤、鸟苷、鸟嘌呤和黄嘌呤的存在下也显示活性。即,不会受到由分解产物带来的实质性抑制。该特征在将本酶用于食品、饮料的制造方面特别重要。利用显示该特征的pn1,在食品、饮料的制造过程中,能够将来自原料的嘌呤核苷有效地分解。
(2)分子量
pn2为天然型时含有糖链(即为糖蛋白),n型糖链除去前的分子量约为51kda(用sds-page测定的分子量)。如果通过凝胶渗透色谱进行测定,则约为230kda。另一方面,如果在n型糖链除去后利用sds-page测定分子量,则显示约为40kda。因此,不含n型糖链时的本酶的分子量约为40kda(用sds-page测定的分子量)。
(3)最适温度
pn2的最适温度为50℃~55℃。如此最适温度高在用于经历较高温下的处理工序的食品、饮料的制造中是有利的。最适温度可以通过使用乙酸缓冲液(ph4.3)并以鸟苷作为底物,对作为反应产物的核糖进行定量来评价。
(4)温度稳定性
pn2在乙酸缓冲液(ph4.5)中处理60分钟时,在65℃以下的温度条件维持80%以上的活性。因此,例如如果处理时的温度为5℃~65℃的范围,则处理后的残留活性为80%以上。
另一方面,将pn2在磷酸缓冲液(ph6.0)中处理30分钟时,在55℃以下的温度条件下维持80%以上的活性。因此,例如如果处理时的温度为5℃~55℃的范围,则处理后的残留活性为80%以上。
如此显示优异的温度稳定性的pn2可以在啤酒的进料工序等较高温的条件下也显示高活性。
可以用以下的酶学性质(5)和(6)进一步表征pn2。
(5)最适ph
pn2的最适ph为4.5。最适ph例如可以基于对于ph2.5~3.5的ph区域在柠檬酸缓冲液、对于ph3.5~5.5的ph区域在乙酸缓冲液、对于ph5.5~6.5的ph区域在磷酸钾缓冲液中测得的结果来判断。
(6)ph稳定性
pn2在宽的ph区域显示稳定的活性。例如,如果供于处理的酶溶液的ph为3.5~7.5的范围内,则30℃、30分钟的处理后,显示最大活性的80%以上的活性。另外,在50℃、60分钟的处理的情况下,如果供于处理的酶溶液的ph为4.5~7.5的范围内,则处理后,显示最大活性的80%以上的活性。应予说明,ph稳定性例如可以基于对于ph2.5~3.5的ph区域在柠檬酸缓冲液、对于ph3.5~5.5的ph区域在乙酸缓冲液、对于ph5.5~6.5的ph区域在磷酸钾缓冲液中测得的结果来判断。
如后述的实施例所示,本发明人等成功地从多色青霉ifo7569株分离·纯化了具备上述性质的核苷酶。应予说明,多色青霉ifo7569株是保存在独立行政法人产品评价技术基础机构(千叶县木更津市上总镰足2-5-8)的菌株(在nbrcculturecatalogue以nbrc7569被登载),可以经过规定的手续而获得。
本发明的核苷酶剂中的核苷酶的含量没有特别限定,例如,每1g核苷酶剂中含有100u~100000u的核苷酶。本发明的核苷酶剂的形态可以为液体状,也可以为固体状(包括粉体状)。本发明的核苷酶剂除包含有效成分(即,本酶)以外,还可以含有赋形剂、缓冲剂、悬浮剂、稳定剂、保存剂、防腐剂、生理食盐水等。作为赋形剂,可以使用乳糖、山梨糖醇、d-甘露醇、麦芽糊精、白糖等。作为缓冲剂,可以使用磷酸盐、柠檬酸盐、乙酸盐等。作为稳定剂,可以使用丙二醇、抗坏血酸等。作为保存剂,可以使用苯酚、苯扎氯铵、苄醇、氯丁醇、对羟基苯甲酸甲酯等。作为防腐剂,可以使用苯扎氯铵、对羟基苯甲酸、氯丁醇等。
2.核苷酶剂的制造方法
本发明的核苷酶剂可以通过以下的方法,即具有下述特征的方法获得:进行培养产生核苷酶的微生物的步骤(步骤(1))和从培养后的培养液和/或菌体回收核苷酶的步骤(步骤(2))。产生核苷酶的微生物例如为多色青霉,优选为多色青霉ifo7569株或其变异株。变异株可以通过紫外线、x射线、γ射线等的照射,利用亚硝酸、羟胺、n-甲基-n’-硝基-n-亚硝基胍等进行的处理等获得。
培养条件、培养法只要可生产核苷酶就没有特别限定。即,可以将生产核苷酶作为条件而适当地设定适合使用的微生物的培养的方法、培养条件。作为培养法,液体培养、固体培养中的任一种均可,优选利用液体培养。以液体培养为例说明其培养条件。
作为培养基,只要是使用的微生物可生长的培养基就没有特别限定。例如可以使用添加有葡萄糖、蔗糖、龙胆二糖、可溶性淀粉、甘油、糊精、糖蜜、有机酸等碳源,进一步添加有硫酸铵、碳酸铵、磷酸铵、乙酸铵、或者蛋白胨、酵母提取物、玉米浆、酪蛋白水解物、麦糠、肉提取物等氮源,进一步添加有钾盐、镁盐、钠盐、磷酸盐、锰盐、铁盐、锌盐等无机盐的培养基。为了促进使用的转化体的生长,可以在培养基中添加维生素、氨基酸等。培养基的ph例如调整为约3~8,优选调整为约4~7左右,培养温度通常为约20~40℃,优选为约25~35℃左右,在需氧条件下培养1~20天、优选培养3~10天左右。作为培养法,例如可利用振荡培养法、基于发酵罐的需氧深层培养法。
在以上的条件下培养后,从培养液或者菌体回收目标酶(核苷酶)(步骤(2))。从培养液回收时,例如通过对培养上清液进行过滤、离心处理等而除去不溶物后,适当地组合利用超滤膜的浓缩、硫酸铵沉淀等盐析、透析、离子交换树脂等各种色谱等而进行分离、纯化,由此能够得到核苷酶。另一方面,从菌体内回收时,例如通过加压处理、超声波处理等将菌体破碎后,与上述同样地进行分离、纯化,由此能够得到核苷酶。应予说明,也可以通过过滤、离心处理等预先从培养液中回收菌体后,进行上述一系列的工序(菌体的破碎、分离、纯化)。
在一个方式中,为了通过简便的操作得到液态的核苷酶剂,利用包括以下的步骤(i)和(ii)的制造方法来制造酶剂。
(i)培养产生核苷酶的微生物的步骤
(ii)在培养后除去菌体的步骤
步骤(i)与上述的步骤(1)同样,因此省略其说明。在接着步骤(i)的步骤(ii)中,利用离心分离、过滤、过滤器处理等除去菌体。如此得到的不含菌体的培养液直接或者经过进一步的处理(即,将除去菌体后的培养液纯化的步骤(步骤(iii))后,作为核苷酶剂使用。
也可以利用导入了核苷酶基因的重组菌制造核苷酶剂。利用重组菌制造核苷酶剂时,通过向适当的宿主微生物导入核苷酶基因而得到重组菌(转化体)后,在产生由导入其中的基因编码的蛋白质的条件下培养该转化体(步骤(i))。关于各种载体宿主体系,转化体的培养条件是公知的,只要是本领域技术人员就能够容易地设定适当的培养条件。在培养步骤之后,将产生的蛋白质(即,核苷酶)回收(步骤(ii))。回收和其后的纯化与上述方式的情况同样地进行即可。
作为核苷酶基因,优选使用含有编码序列号1的氨基酸序列的dna(编码pn1的dna)、编码序列号21的氨基酸序列的dna(编码pn1的成熟体的dna)、编码序列号2的氨基酸序列的dna(编码pn2的dna)、或者编码序列号22的氨基酸序列的dna(编码pn2的成熟体的dna)的基因。该方式的具体例为由序列号3的碱基序列构成的dna(与编码序列号1的氨基酸序列的cdna对应)、由序列号23的碱基序列构成的dna(与编码序列号21的氨基酸序列的cdna对应)、由序列号4的碱基序列构成的dna(与编码序列号1的氨基酸序列的基因组dna对应)、由序列号5的碱基序列构成的dna(与编码序列号2的氨基酸序列的cdna对应)、由序列号24的碱基序列构成的dna(与编码序列号22的氨基酸序列的cdna对应)、由序列号6的碱基序列构成的dna(与编码序列号2的氨基酸序列的基因组dna对应)。
上述的核苷酶基因可以通过参考本说明书或附带的序列表所公开的序列信息,利用标准的基因工程学方法、分子生物学方法、生物化学方法、化学合成、pcr法(例如重叠pcr)或它们的组合而制备成分离的状态。
一般而言,对编码某种蛋白质的dna的一部分实施修饰时,有时修饰后的dna所编码的蛋白质与修饰前的dna所编码的蛋白质具有同等的功能。即有时dna序列的修饰不对所编码的蛋白质的功能造成实质性影响,所编码的蛋白质的功能在修饰前后得以维持。因此,作为核苷酶基因,可以使用含有具有与作为基准的碱基序列(序列号3~6、23、24中的任一种序列)等效的碱基序列且编码具有核苷酶活性的蛋白质的dna(以下也称为“等效dna”)的基因。此处的“等效的碱基序列”是指与作为基准的碱基序列所示的dna一部分不同,但其所编码的蛋白质的功能(此处为核苷酶活性)不会因该不同而受到实质性影响的碱基序列。
等效dna的具体例是在严格条件下对与作为基准的碱基序列互补的碱基序列进行杂交的dna。此处的“严格条件”是指形成所谓的特异性杂交而不形成非特异性杂交的条件。这样的严格条件是本领域技术人员公知的,例如可以参照molecularcloning(thirdedition,coldspringharborlaboratorypress,newyork)、currentprotocolsinmolecularbiology(editedbyfrederickm.ausubeletal.,1987)进行设定。作为严格条件,例如可举出使用杂交液(50%甲酰胺,10×ssc(0.15mnacl,15mm柠檬酸钠,ph7.0)、5×denhardt溶液、1%sds、10%硫酸葡聚糖、10μg/ml的改性鲑鱼精子dna、50mm磷酸缓冲液(ph7.5))在约50℃进行温育,然后,使用0.1×ssc、0.1%sds在约65℃进行清洗的条件。作为进一步优选的严格条件,例如可举出使用作为杂交液的50%甲酰胺、5×ssc(0.15mnacl,15mm柠檬酸钠,ph7.0)、1×denhardt溶液、1%sds、10%硫酸葡聚糖、10μg/ml的改性鲑鱼精子dna、50mm磷酸缓冲液(ph7.5))的条件。
作为等效dna的其它具体例,可举出由对作为基准的碱基序列包含1个或多个(优选为1~几个)碱基的替换、缺失、插入、附加或倒位的碱基序列构成且编码具有核苷酶活性的蛋白质的dna。碱基的替换、缺失等可以发生在多个部位。此处的“多个”根据该dna所编码的蛋白质的立体结构中的氨基酸残基的位置、种类而不同,例如为2~40个碱基,优选为2~20个碱基,更优选为2~10个碱基。
等效dna相对于作为基准的碱基序列(序列号3~6、23、24中的任一个序列),具有例如70%以上、优选80%以上、进一步优选约90%以上、更进一步优选95%以上、最优选99%以上的同源性。
如上的等效dna例如可以通过下述方式得到:利用限制性内切酶处理、介由核酸外切酶或dna连接酶等进行的处理、基于定位突变导入法(molecularcloning,thirdedition,chapter13,coldspringharborlaboratorypress,newyork)或随机突变导入法(molecularcloning,thirdedition,chapter13,coldspringharborlaboratorypress,newyork)的变异的导入等,以包含碱基的替换、缺失、插入、附加和/或倒位的方式对具有基准碱基序列的dna进行修饰。另外,也可以通过紫外线照射等其它方法而得到等效dna。作为等效dna的另一个例子,可举出由于以snp(单核苷酸多态性)为代表的多态性而确认有如上所述的碱基的不同的dna。
为了获得导入有核苷酶基因的重组菌,典型的是构建含有核苷酶基因的表达载体,用该表达载体转化宿主细胞。可以考虑宿主细胞的种类选择适当的载体。作为以大肠杆菌为宿主的载体,可例示m13噬菌体或其修饰体、λ噬菌体或其修饰体、pbr322或其修饰体(pb325、pat153、puc8等)等,作为以酵母为宿主的载体,可例示pyepsec1、pmfa、pyes2等,作为以昆虫细胞为宿主的载体,可举出pac、pvl等,作为以哺乳类细胞为宿主的载体,可例示pcdm8、pmt2pc等。
“表达载体”是指能够将插入该载体的核酸导入到目标细胞(宿主细胞)内且能够在该细胞内表达的载体。表达载体通常含有插入的核酸的表达所需的启动子序列、促进表达的增强子序列等。也可以使用含有选择标记的表达载体。使用该表达载体时,可以利用选择标记来确认有无导入表达载体(及其程度)。
dna向载体的插入、选择标记基因的插入(有需要的情况下)、启动子的插入(有需要的情况下)等可以使用标准的重组dna技术(例如,可以参照molecularcloning,thirdedition,1.84,coldspringharborlaboratorypress,newyork,使用限制性内切酶和dna连接酶的众所周知的方法)进行。
利用上述表达载体的转化体的制备可以利用转染或转化。转染、转化可以通过磷酸钙共沉淀法、电穿孔(potter,h.etal.,proc.natl.acad.sci.u.s.a.81,7161-7165(1984))、脂质转染(felgner,p.l.etal.,proc.natl.acad.sci.u.s.a.84,7413-7417(1984))、显微注射(graessmann,m.&graessmann,a.,proc.natl.acad.sci.u.s.a.73,366-370(1976))、hanahan的方法(hanahan,d.,j.mol.biol.166,557-580(1983))、乙酸锂法(schiestl,r.h.etal.,curr.genet.16,339-346(1989))、原生质体-聚乙二醇法(yelton,m.m.etal.,proc.natl.acad.sci.81,1470-1474(1984))等来实施。
宿主细胞只要表达核苷酶就没有特别限定,例如可以从枯草芽孢杆菌(bacillussubtilis)、地衣芽孢杆菌(bacilluslicheniformis)、环状芽孢杆菌(bacilluscirculans)等芽孢杆菌(bacillus)属细菌,乳球菌(lactococcus)、乳酸杆菌(lactobacillus)、链球菌(streptococcus)、明串珠菌(leuconostoc)、双歧杆菌(bifidobacterium)等乳酸菌,埃希菌(escherichia)、链霉菌(streptomyces)等其它细菌,酵母菌(saccharomyces)、克鲁维酵母(kluyveromyces)、假丝酵母(candida)、圆酵母(torula)、球拟酵母(torulopsis)等酵母,米曲霉(aspergillusoryzae)、黑曲霉(aspergillusniger)等曲霉菌(aspergillus)属,青霉(penicillium)属,木霉(trichoderma)属,镰刀菌(fusarium)属等丝状菌(真菌)等中选择。
实施例
<实施例1>新型核苷酶的获取和鉴定
1.新型核苷酶的获取
为了找出对制造低嘌呤体啤酒有用的酶,以超过一万种的微生物为对象实施了筛选。其结果,鉴定了4株微生物,即多色青霉(penicilliummulticolor)ifo7569株、短芽孢杆菌(bacillusbrevis)ifo15304株、亚麻短杆菌(brevibacilluslinens)ifo12141株、爪哇毛霉(mucorjavanicus)4068株作为有希望的候补。对于这些微生物所产生的核苷酶,假设在啤酒的进料工序中利用,对在进料工序的常规条件(糖化试验)下的作用·效果进行评价。应予说明,其后对多色青霉(penicilliummulticolor)ifo7569株再次鉴定,结果鉴定为penicilliummaximae。
(1)多色青霉ifo7569株的培养方法
将多色青霉ifo7569株接种于下述的培养培养基b100ml,在500ml容量坂口烧瓶中在27℃振荡培养48~72小时。将该前培养液移至下述培养培养基b2l,在27℃通气搅拌培养120~188小时。将该培养液进行硅藻土过滤而除去菌体,将得到的培养上清液利用超滤膜进行浓缩,得到冷冻干燥粉末。
<培养培养基a>
1%lustergenfk(日淀化学)
1%酵母提取物(difco)
0.5%nacl
ph7.0
<培养培养基b>
1%lustergenfk(日淀化学)
1%酵母提取物(difco)
2%玉米粉(matsumotonohsan)
0.5%nacl
ph6.5
(2)短芽孢杆菌ifo15304株、亚麻短杆菌ifo12141株、爪哇毛霉4068株的培养方法
将短芽孢杆菌ifo15304株和亚麻短杆菌ifo12141株接种于上述培养培养基a10ml,在试验管中在30℃振荡培养48小时。另一方面,将爪哇毛霉ifo4068株接种于上述培养培养基b10ml,在相同条件下进行培养。将培养液分别移至相同组成的本培养培养基50ml,在30℃振荡培养120小时。将该培养液通过离心分离除去菌体,由得到的上清液得到冷冻干燥粉末。
(3)核苷酶活性的测定
核苷酶活性通过对由以鸟苷为底物的反应生成的核糖进行定量来定义。反应液1ml中含有0.1m乙酸缓冲液(ph4.3)、8mm鸟苷和适量的酶。反应在添加鸟苷时开始,在55℃反应30分钟。添加1.5ml的0.5%二硝基水杨酸溶液使反应停止后,进行10分钟煮沸处理。测定冷却后的反应溶液的540nm的吸光度,由减去了未添加酶的反应溶液的吸光度的值算出活性值。将30分钟生成1μmol的核糖的酶量定义为酶活性1u。
(4)糖化试验
添加粉碎麦芽80g、水320ml以及320u当量的核苷酶进行糖化试验,制备麦芽汁。将反应工序示于图1。糖化后的麦芽汁中的各嘌呤体量通过高效液相色谱在下述条件下进行定量分析。
<hplc条件>
柱:asahipakgs-220hq
流动相:150mm磷酸钠缓冲液(ph2.5)
温度:35℃
流速:0.5ml/分钟
检测:260nm
将分析结果示于图2。图中也示出由以下的算式算出的游离嘌呤碱基比率。
游离嘌呤碱基比率(%)={嘌呤碱基/(嘌呤核苷+嘌呤碱基)}×100
添加了来自多色青霉ifo7569株(p.multicolor)的核苷酶的麦芽汁中嘌呤核苷减少,嘌呤碱基增加。作为对照,认为来自短芽孢杆菌(bacillusbrevis)ifo15304株、亚麻短杆菌(brevibacilluslinens)ifo12141株、爪哇毛霉(mucorjavanicus)4068株的核苷酶可能受到由分解产物(腺嘌呤、鸟嘌呤、次黄嘌呤、黄嘌呤)带来的抑制,嘌呤核苷的量没有大幅变化。
(5)来自多色青霉ifo7569株的核苷酶(p.multicolor核苷酶)的特性的研究
为了分析多色青霉核苷酶的特性,使用以下组成的溶液(以下为模拟麦芽汁),对作用温度区域和作用ph区域进行了研究。
腺苷0.08mmol/l
腺嘌呤0.43mmol/l
肌苷0.49mmol/l
次黄嘌呤0.08mmol/l
鸟苷0.67mmol/l
鸟嘌呤1.45mmol/l
黄苷0.00mmol/l
黄嘌呤0.08mmol/l
(5-1)作用温度区域
在模拟麦芽汁2ml中添加9u的多色青霉核苷酶,在各温度下以ph5.5进行1小时反应后,用hplc的流动相150mm磷酸钠缓冲液(ph2.5)稀释至10倍并利用高效液相色谱进行定量分析。由以下的算式算出游离嘌呤碱基比率。在50℃~60℃的反应温度下,游离嘌呤碱基比率为90%以上(图3)。
游离嘌呤碱基比率(%)={嘌呤碱基/(嘌呤核苷+嘌呤碱基)}×100
(5-2)作用ph区域
在模拟麦芽汁2ml中添加9u的多色青霉核苷酶,在各ph下以55℃进行1小时反应后,用hplc的流动相150mm磷酸钠缓冲液(ph2.5)稀释至10倍并利用高效液相色谱进行定量分析。ph4.5~ph6.0使用柠檬酸缓冲液,ph6.0~6.5使用mes缓冲液。与作用温度区域的研究的情况同样地算出游离嘌呤碱基比率。为柠檬酸缓冲液时,在ph4.5~ph5.5,游离嘌呤体比率为80%以上。为mes缓冲液时,在ph6.0~ph6.5,游离嘌呤体比率为80%以上(图4)。
(6)来自多色青霉ifo7569株的核苷酶的纯化
通过羟基磷灰石柱、阴离子交换柱、疏水柱、凝胶过滤柱色谱将核苷酶纯化。以下示出一系列的纯化工序。首先,将由多色青霉ifo7569株的培养液制备的冷冻干燥粉末0.1g用5ml的缓冲液(5mm磷酸钾缓冲液(ph6)+0.3mnacl)溶解,供于用相同缓冲液平衡了的羟基磷灰石柱(biorad)。将吸附的蛋白质用5mm~300mm的磷酸梯度洗脱并回收活性馏分。将得到的活性馏分用缓冲液(20mm磷酸钾缓冲液(ph5.5))透析,供于用相同缓冲液平衡了的deaehp柱(gehealthcare)。将吸附的蛋白质用0mm~500mm的nacl梯度洗脱,结果确认到3个峰(图5)。将fr.2确定为峰1,将fr.8、9确定为峰2,将fr.14、15确定为峰3。
将回收的峰3用缓冲液(20mm乙酸缓冲液(ph4.5)+30%饱和硫酸铵)透析,供于用相同缓冲液平衡了的phenylhp柱(gehealthcare)。将吸附的蛋白质用30%饱和~0%的硫酸铵梯度洗脱并回收活性馏分。将得到的活性馏分用缓冲液(20mm磷酸钠缓冲液(ph6))透析后,使用超滤膜浓缩至0.5ml。将浓缩的活性馏分供于用相同缓冲液平衡了的hiload16/60superdex200(gehealthcare)并回收活性馏分。得到的纯化酶通过sds-page确定为单一条带(图6)。分子量通过sds-page推定为约53kda,通过凝胶过滤色谱推定为约126kda(图7)。将得到的纯化酶的糖链用pngasef(newenglandbiolabs)除去。处理方法依照所附说明。根据处理后的sds-page,示出了通过除去n型糖链而分子量从约53kda变为约49kda(图6、7)。对于回收的峰1、2也同样地纯化,通过sds-page和凝胶过滤色谱来确定分子量。分子量通过sds-page推定为约51kda,通过凝胶过滤色谱推定为约230kda(图7)。将得到的纯化酶的糖链用pngasef(newenglandbiolabs)除去。根据处理后的sds-page,示出了通过除去n型糖链而分子量从约51kda变为约40kda(图6、7)。
用蛋白质测序仪(岛津制作所)分析各纯化酶(峰1~3)的n末端氨基酸序列,结果推定为以下所示的序列。
峰1的n末端氨基酸序列:adkhyaimdndwyta(序列号7)
峰2的n末端氨基酸序列:adkhyaimdndwyta(序列号8)
峰3的n末端氨基酸序列:vetkliflt(序列号9)
峰1和峰2的分子量及n末端氨基酸序列一致,推定为相同的酶(图7)。关于峰3的n末端氨基酸序列,通过其后的解析判明了实际为“veillislt”(序列号20)。在以下的研究中,将该酶称为pn2,将峰3的酶称为pn1。
2.基因克隆
由确定的n末端氨基酸序列和核苷酶保守序列设计以下的简并引物,以多色青霉基因组dna作为模板而实施pcr。
<pn1用简并引物>
fw:aciaartaymgnttyytiac(序列号10)
rv:catnccnckngtccaytgncc(序列号11)
<pn2用简并引物>
fw:gcnathatggayaaygaytggtayac(序列号12)
rv:gcngcngtytcrtcccaraangg(序列号13)
将得到的扩增片段亚克隆于pmd20-t(takara)并进行测序,使用图8所示的探针实施southern印迹和菌落杂交。对得到的片段进行测序而鉴定出pn1和pn2的基因组中的碱基序列(图9)。
接下来,由多色青霉的基因组dna制备mrna,使用smarterrace5’/3’(takara)由该mrna制备cdna。然后,使用以下的引物进行pcr,对扩增片段进行测序,确定cdna中的pn1和pn2的碱基序列(图10)。另外,由确定的碱基序列鉴定pn1和pn2的氨基酸序列(图11)。应予说明,在图12中比较了pn1和pn2。
<pn1用pcr引物>
fw:atggcacctaagaaaatcatcattg(序列号14)
rv:ttagtggaagattctatcgatgagg(序列号15)
<pn2用pcr引物>
fw:atgcatttccctgtttcattgccgc(序列号16)
rv:tcaacgctcatttctcaggtcgg(序列号17)
3.酶pn1的各种性质的研究
(1)最适温度
对从deaehp柱回收的峰3的核苷酶(pn1)的最适温度进行分析。将各温度的结果示于图13。该条件下的最适温度为55℃~60℃。
(2)温度稳定性
对从deaehp柱回收的峰3的核苷酶的温度稳定性进行分析。在各温度下以ph4.5处理60分钟时,到45℃为止显示残留活性80%,以ph6.0处理30分钟时,到55℃为止显示残留活性80%(图14)。
(3)最适ph
对从deaehp柱回收的峰3的核苷酶的最适ph进行分析。ph2.5和ph3.5使用柠檬酸缓冲液,ph3.5、ph4.5和ph5.5使用乙酸缓冲液,ph5.5和ph6.5使用磷酸钾缓冲液。最适ph为ph3.5(图15)。
(4)ph稳定性
对将从deaehp柱回收的峰3的核苷酶在各ph下以30℃处理30分钟时和在50℃处理60分钟时的ph稳定性进行分析。缓冲液使用与最适ph的研究时相同的缓冲液,对于ph7.5,使用磷酸钾缓冲液。在30℃处理30分钟时,在ph3.5~7.5显示80%以上的残留活性,在50℃处理60分钟时,在ph3.5~7.5显示80%以上的残留活性(图16)。
4.酶pn2的重组生产
将pn2的cdna片段插入表达用载体的克隆位点,构建pn2表达载体。用该表达载体转化米曲霉(a.oryzae(pyrg-))。将得到的转化体进行4天液体培养(30℃、300rpm)。回收培养上清液,测定核苷酶活性。其结果,得到显示活性的转化体。另外,对培养上清液进行糖链除去处理,并进行电泳,结果确认了与所推定的分子量一致的尺寸的条带(图17)。
5.酶pn2的各种性质的研究
使用重组生产的pn2研究各种性质。实验方法、条件等与pn1的研究的情况同样。
(1)最适温度
最适温度为50℃~55℃(图18)。
(2)温度稳定性
在各温度下以ph4.5处理60分钟时,到65℃为止显示残留活性80%,以ph6.0处理30分钟时,到55℃为止显示残留活性80%(图19)。
(3)最适ph
ph2.5和ph3.5使用柠檬酸缓冲液,ph3.5、ph4.5和ph5.5使用乙酸缓冲液,ph5.5和ph6.5使用磷酸钾缓冲液。最适ph为ph4.5(图20)。
(4)ph稳定性
对以各ph在30℃处理30分钟时和在50℃处理60分钟时的ph稳定性进行分析。缓冲液使用与最适ph的研究时相同的缓冲液。在30℃处理30分钟时,在ph3.5~7.5显示80%以上的残留活性,在50℃处理60分钟时,在ph4.5~7.5显示80%以上的残留活性(图21)。
6.酶pn2的糖化试验
使用重组生产的pn2进行糖化试验。试验方法、条件等与上述1.(4)同样。用高效液相色谱对糖化后的麦芽汁中的各嘌呤体量进行定量分析。将分析结果示于图22。可知添加了pn2(核苷酶)的麦芽汁中嘌呤核苷减少,嘌呤碱基增加。
7.在低ph条件下的糖化试验
为了进一步确认来自多色青霉ifo7569株的核苷酶在啤酒或啤酒类饮料的制造中的有用性,在该酶的最适ph附近(ph4.5~5.5)进行糖化试验,研究是否可得到期望的效果(即,嘌呤体减少)。添加粉碎麦芽80g、水320ml以及2400u当量的核苷酶,将初始(52℃)和60℃工序的ph调整为ph4.5、ph5.0或ph5.5进行糖化试验,制备麦芽汁。将反应工序示于图23。糖化后的麦芽汁中的各嘌呤体量利用高效液相色谱在下述条件下进行定量分析。将分析结果示于图24。可知本核苷酶即使在ph4.5~5.5进行糖化,游离嘌呤碱基比率也为90%以上,嘌呤核苷充分减少,嘌呤碱基增加(图24)。
<hplc条件>
柱:asahipakgs-220hq
流动相:150mm磷酸钠缓冲液(ph2.5)
温度:35℃
流速:0.5ml/分钟
检测:260nm
8.总结
来自多色青霉ifo7569株的核苷酶显示出适合在啤酒的进料工序中采用的最适温度和温度稳定性。另外,判明了ph稳定性和温度稳定性优异,不限于啤酒或啤酒类饮料的制造,还可用于各种用途。如此,成功地获取了对减少饮料·食品中的嘌呤体极其有用的新型核苷酶。
<实施例2>核苷酶剂的制备
将多色青霉ifo7569株的变异株接种于下述的培养培养基b100ml,在500ml容量坂口烧瓶中在27℃振荡培养48~72小时。将该前培养液移至下述培养培养基b2l,在27℃通气搅拌培养190~240小时。将该培养液进行硅藻土过滤而除去菌体,将得到的培养上清液利用超滤膜进行浓缩,得到核苷酶酶溶液。应予说明,使用鸟嘌呤脱氨酶生产能力不同的变异株3种制备核苷酶酶溶液(试样1~3)。
<培养培养基a>
1%lustergenfk(日淀化学)
1%酵母提取物(difco)
0.5%nacl
ph7.0
<培养培养基b>
1%lustergenfk(日淀化学)
1%酵母提取物(difco)
2%玉米粉(matsumotonohsan)
0.5%nacl
ph6.5
<实施例3>黄嘌呤生成试验
将溶解于50mm盐酸的1mm鸟嘌呤溶液0.1ml添加到150mm乙酸钠缓冲液(ph4.3)0.8ml中,制成底物溶液。将以核苷酶活性成为500u/g的方式用纯化水稀释的各核苷酶的酶溶液(试样1~3)0.1ml添加到底物溶液0.9ml中,在52℃反应。在1小时后各取样0.1ml的反应液。通过煮沸10分钟使酶反应停止后,用纯化水稀释2倍,使其通过0.45μm的膜过滤器。将过滤器处理后的溶液供于hplc,对黄嘌呤生成量进行定量。
(hplc分析条件)
柱:asahipakgs220-hqno.m211036
溶剂:150mm磷酸二氢钠ph2.5
流速:0.5ml/min
柱温:35℃
注入量:10μl
检测:260nm
分析时间:60分钟
将1分钟将1pmol的鸟嘌呤转换成黄嘌呤的酶量定义为1u,由以下的算式算出核苷酶酶溶液的鸟嘌呤脱氨酶活性。
鸟嘌呤脱氨酶活性(u/ml)=反应60分钟的黄嘌呤生成量(μmol/ml)×1/60×1000000
另一方面,通过实施例1的“(3)核苷酶活性的测定”中记载的方法,求出各核苷酶酶溶液(试样1~3)的核苷酶活性,算出相对于核苷酶活性的鸟嘌呤脱氨酶活性(u/u)(表1)
[表1]
[表1]
鸟嘌呤脱氨酶活性的比较
<实施例4>糖化试验
使用实施例2的核苷酶的酶溶液(试样1~3)进行糖化试验,分析同化性嘌呤构成比。添加粉碎麦芽80g、水320ml以及核苷酶2400u当量,将初始(52℃)和60℃工序的ph调整为ph4.5而进行糖化试验,制备麦芽汁。将反应工序示于图23。糖化后的麦芽汁中的各嘌呤体量通过高效液相色谱在下述条件下进行定量分析。
<hplc条件>
柱:asahipakgs-220hq
流动相:150mm磷酸钠缓冲液(ph2.5)
温度:35℃
流速:0.5ml/分钟
检测:260nm
通过以下的算式算出各核苷酶的酶溶液的同化性嘌呤构成比,进行比较(表2)。应予说明,同化性嘌呤构成比为同化性嘌呤(腺嘌呤、鸟嘌呤)与非同化性嘌呤(腺苷、鸟苷、肌苷、黄苷、黄嘌呤、次黄嘌呤)的比率。
同化性嘌呤构成比(%)=同化性嘌呤量/(同化性嘌呤量+非同化性嘌呤量)×100
[表2]
[表2]
根据以上的实验结果,相对于核苷酶活性的鸟嘌呤脱氨酶活性与同化性嘌呤构成比的关系如图25所示。因此,可确认由于同化性嘌呤构成比为90%以上,所以相对于核苷酶活性的鸟嘌呤脱氨酶活性为0.4(u/u)以下,可得到满足该条件的核苷酶的酶溶液(试样1)。
产业上的可利用性
本发明的核苷酶剂的夹杂鸟嘌呤脱氨酶活性低,对制造低嘌呤体啤酒/啤酒类饮料有用。除低嘌呤体啤酒/啤酒类饮料以外,各种低嘌呤体食品或饮料的制造中也可利用本发明。
本发明完全不受上述发明的实施方式和实施例的说明限定。在不脱离权利要求书的记载的情况下,在本领域技术人员能够容易地想到的范围内进行的各种变形方式也包含在本发明中。本说明书中明示的论文、公开专利公报和专利公报等的内容通过援引而引用其全部内容。
序列表
<110>天野酶制品株式会社
三得利控股株式会社
<120>夹杂活性降低的核苷酶剂
<130>ae17006p
<150>jpp2018-022542
<151>2018-02-09
<160>24
<170>patentinversion3.5
<210>1
<211>424
<212>prt
<213>多色青霉
<400>1
metalaprolyslysileileileaspthraspproglyileaspasp
151015
ileleualaleuleuleualaleuserserlysprogluaspvalglu
202530
ileleuleuileserleuthrpheglyasnilegluvallysasncys
354045
leuargasnvalvalsermetphehisileleugluarggluilegln
505560
trpargargglyasnglylyssergluglytyrglythrmetargala
65707580
pheargprovalvalalavalglyalagluaspproleugluaspgln
859095
lysmetleualaasptyrphehisglythraspglyleuglyglyile
100105110
hisalaserhisprohisleuthrproserlysalatrpgluhisleu
115120125
phethrproalavalaspproglnglyilegluprovalglnthrgly
130135140
alaglyproglyasphisserpheileproserargleuproalahis
145150155160
lysgluileleuargalaleuargglnasngluproaspthrvalthr
165170175
leuvalalavalglyproleuthrasnleualaleualaalaalaglu
180185190
aspprogluthrpheleuargvallysgluvalvalvalmetglygly
195200205
alaileasnglnproglyasnvalthrprovalglyglupheasnala
210215220
tyralaaspalavalalaalaalaargvalphealaleuthrserpro
225230235240
asnproasnserthrleuproprothrthrserproleuleuglyleu
245250255
tyrproalalysleuserargglnleuthrleuargleupheproleu
260265270
aspilethrleuarghisasnleuserargglyglnpheargglnala
275280285
valgluproleuleualathrglyserproleualaglutrpvalthr
290295300
alaphemetglyhisthrpheargthrleugluargleuhisprogly
305310315320
hisgluglyaspglualaglnleuserleuhisaspprovalcysval
325330335
trptyralaleuthralagluaspserhistrpthrproseralaasn
340345350
serprogluaspileargvalgluthrleuglyglntrpthrarggly
355360365
metcysvalileaspglyargasnarghislysileaspglyaspglu
370375380
gluserserserasphisglyleutrpleuseralaargalaglyasn
385390395400
argileleuargmetaspglyserproalagluhisthrpheglylys
405410415
ileleuileaspargilephehis
420
<210>2
<211>380
<212>prt
<213>多色青霉
<400>2
methispheprovalserleuproleuleucysglyserleuleupro
151015
leuilethrglythrleualavalprolysalaserargalaasplys
202530
histyralailemetaspasnasptrptyrthralaglyphevalpro
354045
tyrleuilealaleuaspglyaspvalgluvalleuglyleualaser
505560
aspthralaasnthrtrpglnproglnvalalaleuhisalavalala
65707580
thrleuglualaglyasnleusercysileprovaltyrproglyser
859095
thrtrpproleuileasnthrproasnargpheglnalatrpglumet
100105110
valhisglylysleuprotrpgluglyalaphealaprogluasnlys
115120125
thrleuglualagluglyasnaspprothrserglyasnproasnarg
130135140
ilevallysalaalaphelysgluglypheprolysglylysproglu
145150155160
asnargthrseralaalaasnphemetvalglumetvalhislystyr
165170175
proglyglnvalseriletyrseralaglyalaleuthrasnvalala
180185190
leualavalargmetaspproglnphealaserleualalysgluleu
195200205
valilemetglyglytyrvalaspleuasnmetleuglnalathrgly
210215220
servalleuleualaaspleuglnseraspileasnleumetileasp
225230235240
proglualaserlysilealaleuthralaglupheproasnilethr
245250255
ilealaglyasnvalalaasnglnvalpheprothrlysglupheval
260265270
aspgluilealaservalproasnprotyrserlysleuphehisasp
275280285
tyrtyraspleuserpheprophetrpaspgluthralaalaalaleu
290295300
metvalaspprothrleualathrasnglnthrservalpheleuasp
305310315320
valaspthralatyrglyserproasntyrglyasnilehisvaltyr
325330335
glnlysalaleualaprovalglyilearggluvalasnphevalphe
340345350
glnvalaspglyaspargleulysglnargilelyshisserleugln
355360365
tyrprolyssercysalaaspleuargasngluarg
370375380
<210>3
<211>1275
<212>dna
<213>多色青霉
<400>3
atggcacctaagaaaatcatcattgacactgacccgggtatcgatgacatcctggcactg60
ctgctggctctgtcatctaagccagaggatgttgagattctacttatctctttaacattt120
ggaaacattgaggtgaagaactgtcttcgaaatgtggtctccatgtttcatatcctcgag180
cgcgagatccagtggcgtcgtggtaacggcaagtccgaaggctatggcactatgcgtgct240
ttccgcccagtagtagccgtgggagcggaagatcccttggaagaccagaagatgctcgct300
gattatttccatggaaccgatggccttggtggcatccatgctagtcacccacatctcact360
ccaagcaaggcctgggagcatctattcaccccggccgtggatccccaggggatcgagcct420
gtgcaaacgggagctggtcccggcgaccattcctttatcccatcaagactacctgcacac480
aaggagattcttcgtgcactgcgccagaatgagcctgacaccgtgactctcgtggcggtt540
ggtccactgaccaacttggccttggcagcagcagaggatcccgaaaccttcctacgtgtc600
aaggaggtcgttgtgatgggtggagcaatcaaccagcctggaaatgtcacccccgttgga660
gaattcaacgcctacgcagacgccgttgcagctgcgcgagtctttgcgctgacatcacct720
aatcccaactcgactctaccaccgaccacgagtccactacttggcctgtaccctgcaaag780
ctcagccgacaattgactctgcgtctcttcccgctggacatcaccctgcgccataacctg840
tcccgcggccaattccgccaagcagttgagcctctcctcgcaacaggctcacccctcgct900
gaatgggtgacagcattcatgggacacacgttccgaaccctggaacgcctgcaccccggc960
catgagggcgatgaagcccagctgagtctccacgaccctgtctgtgtgtggtatgccctt1020
acagcagaggattcgcactggactccctccgccaattccccagaggacattcgtgttgag1080
acattgggccagtggacgcgtggtatgtgcgtaatcgatggccgaaaccgccataagatt1140
gatggcgacgaggaaagctcgagtgatcatggtctgtggttgagtgctcgtgcaggaaac1200
cgcattttgcgaatggatggatcgccagccgaacacacgttcggcaagatcctcatcgat1260
agaatcttccactaa1275
<210>4
<211>1016
<212>dna
<213>多色青霉
<400>4
gtacccattttctaacactatctggacagcacccacatctcactccaagcaaggcctggg60
agcatctattcaccccggccgtggatccccaggggatcgagcctgtgcaaacgggagctg120
gtcccggcgaccattcctttatcccatcaagactacctgcacacaaggagattcttcgtg180
cactgcgccagaatgagcctgacaccgtgactctcgtggcggttggtccactgaccaact240
tggccttggcagcagcagaggatcccgaaaccttcctacgtgtcaaggaggtcgttgtga300
tgggtggagcaatcaaccagcctggaaatgtatgaaccccgtcgaaacacccatttgata360
ataagtcattaaccgcgattgactaggtcacccccgttggagaattcaacgcctacgcag420
acgccgttgcagctgcgcgagtctttgcgctgacatcacctaatcccaactcgactctac480
caccgaccacgagtccactacttggcctgtaccctgcaaagctcagccgacaattgactc540
tgcgtctcttcccgctggacatcaccctgcgccataacctgtcccgcggccaattccgcc600
aagcagttgagcctctcctcgcaacaggctcacccctcgctgaatgggtgacagcattca660
tgggacacacgttccgaaccctggaacgcctgcaccccggccatgagggcgatgaagccc720
agctgagtctccacgaccctgtctgtgtgtggtatgcccttacagcagaggattcgcact780
ggactccctccgccaattccccagaggacattcgtgttgagacattgggccagtggacgc840
gtggtatgtgcgtaatcgatggccgaaaccgccataagattgatggcgacgaggaaagct900
cgagtgatcatggtctgtggttgagtgctcgtgcaggaaaccgcattttgcgaatggatg960
gatcgccagccgaacacacgttcggcaagatcctcatcgatagaatcttccactaa1016
<210>5
<211>1143
<212>dna
<213>多色青霉
<400>5
atgcatttccctgtttcattgccgctgttgtgcggctctttgctgcctctcatcaccggc60
accctggcagtgcccaaggcctcgcgtgccgacaagcactatgccatcatggacaatgat120
tggtacacagcgggtttcgtgccttacctgatcgccctcgatggcgatgtggaggttctg180
ggcctagcctctgacaccgcaaacacctggcagcctcaggtcgctctgcacgctgtcgca240
actctggaagctggcaacttgagctgtatccccgtttacccaggctcgacatggccgctc300
atcaacacccccaaccgcttccaggcgtgggaaatggttcatggcaagctgccatgggag360
ggtgcttttgcgccggagaacaagactctcgaggccgagggtaacgatcctacctctggc420
aaccccaaccgtatcgtcaaggccgctttcaaggaagggttccccaagggcaagcccgag480
aacagaacatctgctgccaacttcatggtcgagatggtgcacaagtaccccggccaggtc540
tcgatctactctgctggagccctgaccaatgttgcgctggctgtgcgcatggatccccag600
tttgcatctctggctaaggagttggttatcatgggtggatacgtcgatttgaatatgctc660
caggccactggaagtgtcttgctggctgatcttcaatctgatatcaacttgatgattgat720
cccgaggcctccaagatcgcattgactgccgaattccccaatatcaccatcgccggtaac780
gtcgccaaccaggtctttcctaccaaggagttcgtcgacgagatcgcctccgttccaaac840
ccctacagcaagctcttccacgactactacgatctgtccttccccttctgggatgagacg900
gctgccgcgctgatggttgaccctactcttgctaccaaccagacctctgtcttcctcgac960
gtggataccgcttatggtagccccaactatggtaacattcacgtttaccagaaggctctt1020
gcccctgttggtatccgggaggtcaactttgtcttccaggttgatggggatagacttaag1080
cagcgcatcaagcactctctgcagtaccccaagtcatgcgccgacctgagaaatgagcgt1140
tga1143
<210>6
<211>847
<212>dna
<213>多色青霉
<400>6
acgatcctacctctggcaaccccaaccgtatcgtcaaggccgctttcaaggaagggttcc60
ccaagggcaagcccgagaacagaacatctgctgccaacttcatggtcgagatggtgcaca120
agtaccccggccaggtctcgatctactctgctggagccctgaccaatgttgcgctggctg180
tgcgcatggatccccagtttgcatctctggctaaggagttggttatcatgggtggatacg240
tcgatttgaatatgctccaggccactggaagtgtcttgctggctgatcttcaatctgatg300
tatgtttcattcccggcttctatcagctgtgttcatctgctaacttctctttagatcaac360
ttgatgattgatcccgaggcctccaagatcgcattgactgccgaattccccaatatcacc420
atcgccggtaacgtcgccaaccaggtctttcctaccaaggagttcgtcgacgagatcgcc480
tccgttccaaacccctacagcaagctcttccacgactactacgatctgtccttccccttc540
tgggatgagacggctgccgcgctgatggttgaccctactcttgctaccaaccagacctct600
ggtgagtttaatctcgcattgacacttgtatgaacaaatctaacagcttatagtcttcct660
cgacgtggataccgcttatggtagccccaactatggtaacattcacgtttaccagaacgc720
tcttgcccctgttggtatccgggaggtcaactttgtcttccaggttgatggggatagact780
taagcagcgcatcaagcactctctgcagtaccccaagtcatgcgccgacctgagaaatga840
gcgttga847
<210>7
<211>15
<212>prt
<213>多色青霉
<400>7
alaasplyshistyralailemetaspasnasptrptyrthrala
151015
<210>8
<211>15
<212>prt
<213>多色青霉
<400>8
alaasplyshistyralailemetaspasnasptrptyrthrala
151015
<210>9
<211>9
<212>prt
<213>多色青霉
<400>9
valgluthrlysleuilepheleuthr
15
<210>10
<211>20
<212>dna
<213>人工序列
<220>
<223>引物
<220>
<221>misc_feature
<222>(3)..(3)
<223>肌苷
<220>
<221>misc_feature
<222>(6)..(6)
<223>gora
<220>
<221>misc_feature
<222>(9)..(9)
<223>torc
<220>
<221>misc_feature
<222>(10)..(10)
<223>aorc
<220>
<221>misc_feature
<222>(12)..(12)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(15)..(15)
<223>torc
<220>
<221>misc_feature
<222>(16)..(16)
<223>torc
<220>
<221>misc_feature
<222>(18)..(18)
<223>肌苷
<400>10
acnaartaymgnttyytnac20
<210>11
<211>21
<212>dna
<213>人工序列
<220>
<223>引物
<220>
<221>misc_feature
<222>(4)..(4)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(7)..(7)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(9)..(9)
<223>gort
<220>
<221>misc_feature
<222>(10)..(10)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(16)..(16)
<223>torc
<220>
<221>misc_feature
<222>(19)..(19)
<223>anyofa,g,c,ort
<400>11
catnccnckngtccaytgncc21
<210>12
<211>26
<212>dna
<213>人工序列
<220>
<223>引物
<220>
<221>misc_feature
<222>(3)..(3)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(6)..(6)
<223>a,c,ort
<220>
<221>misc_feature
<222>(12)..(12)
<223>torc
<220>
<221>misc_feature
<222>(15)..(15)
<223>torc
<220>
<221>misc_feature
<222>(18)..(18)
<223>torc
<220>
<221>misc_feature
<222>(24)..(24)
<223>torc
<400>12
gcnathatggayaaygaytggtayac26
<210>13
<211>23
<212>dna
<213>人工序列
<220>
<223>引物
<220>
<221>misc_feature
<222>(3)..(3)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(6)..(6)
<223>anyofa,g,c,ort
<220>
<221>misc_feature
<222>(9)..(9)
<223>torc
<220>
<221>misc_feature
<222>(12)..(12)
<223>gora
<220>
<221>misc_feature
<222>(18)..(18)
<223>gora
<220>
<221>misc_feature
<222>(21)..(21)
<223>anyofa,g,c,ort
<400>13
gcngcngtytcrtcccaraangg23
<210>14
<211>25
<212>dna
<213>人工序列
<220>
<223>引物
<400>14
atggcacctaagaaaatcatcattg25
<210>15
<211>25
<212>dna
<213>人工序列
<220>
<223>引物
<400>15
ttagtggaagattctatcgatgagg25
<210>16
<211>25
<212>dna
<213>人工序列
<220>
<223>引物
<400>16
atgcatttccctgtttcattgccgc25
<210>17
<211>23
<212>dna
<213>人工序列
<220>
<223>引物
<400>17
tcaacgctcatttctcaggtcgg23
<210>18
<211>574
<212>dna
<213>人工序列
<220>
<223>探针
<400>18
gaggatcccgaaaccttcctacgtgtcaaggaggtcgttgtgatgggtggagcaatcaac60
cagcctggaaatgtatgaaccccgtcgaaacacccatttgataataagtcattaaccgcg120
attgactaggtcacccccgttggagaattcaacgcctacgcagacgccgttgcagctgcg180
cgagtctttgcgctgacatcacctaatcccaactcgactctaccaccgaccacgagtcca240
ctacttggcctgtaccctgcaaagctcagccgacaattgactctgcgtctcttcccgctg300
gacatcaccctgcgccataacctgtcccgcggccaattccgccaagcagttgagcctctc360
ctcgcaacaggctcacccctcgctgaatgggtgacagcattcatgggacacacgttccga420
accctggaacgcctgcaccccggccatgagggcgatgaagcccagctgagtctccacgac480
cctgtctgtgtgtggtatgcccttacagcagaggattcgcactggactccctccgccaat540
tccccagaggacattcgtgttgagacattgggcc574
<210>19
<211>509
<212>dna
<213>人工序列
<220>
<223>探针
<400>19
agacaccgcaaacacctggcagcctcaggtcgctctgcacgctgtcgcaactctggaagc60
tggcaacttgagctgtatccccgtttacccaggctcgacatggccgctcatcaacacccc120
caaccgcttccaggcgtgggaaatggttcatggcaagctgccatgggagggtgcttttgc180
gccggagaacaagactctcgaggccgagggtaacgatcctacctctggcaaccccaaccg240
tatcgtcaaggccgctttcaaggaagggttccccaagggcaagcccgagaacagaacatc300
tgctgccaacttcatggtcgagatggtgcacaagtaccccggccaggtctcgatctactc360
tgctggagccctgaccaatgttgcgctggctgtgcgcatggatccccagtttgcatctct420
ggctaaggagttggttatcatgggtggatacgtcgatttgaatatgctccaggccactgg480
aagtgtcttgctggctgatcttcaatctg509
<210>20
<211>9
<212>prt
<213>多色青霉
<400>20
valgluileleuleuileserleuthr
15
<210>21
<211>394
<212>prt
<213>多色青霉
<400>21
valgluileleuleuileserleuthrpheglyasnilegluvallys
151015
asncysleuargasnvalvalsermetphehisileleugluargglu
202530
ileglntrpargargglyasnglylyssergluglytyrglythrmet
354045
argalapheargprovalvalalavalglyalagluaspproleuglu
505560
aspglnlysmetleualaasptyrphehisglythraspglyleugly
65707580
glyilehisalaserhisprohisleuthrproserlysalatrpglu
859095
hisleuphethrproalavalaspproglnglyilegluprovalgln
100105110
thrglyalaglyproglyasphisserpheileproserargleupro
115120125
alahislysgluileleuargalaleuargglnasngluproaspthr
130135140
valthrleuvalalavalglyproleuthrasnleualaleualaala
145150155160
alagluaspprogluthrpheleuargvallysgluvalvalvalmet
165170175
glyglyalaileasnglnproglyasnvalthrprovalglygluphe
180185190
asnalatyralaaspalavalalaalaalaargvalphealaleuthr
195200205
serproasnproasnserthrleuproprothrthrserproleuleu
210215220
glyleutyrproalalysleuserargglnleuthrleuargleuphe
225230235240
proleuaspilethrleuarghisasnleuserargglyglnphearg
245250255
glnalavalgluproleuleualathrglyserproleualaglutrp
260265270
valthralaphemetglyhisthrpheargthrleugluargleuhis
275280285
proglyhisgluglyaspglualaglnleuserleuhisaspproval
290295300
cysvaltrptyralaleuthralagluaspserhistrpthrproser
305310315320
alaasnserprogluaspileargvalgluthrleuglyglntrpthr
325330335
argglymetcysvalileaspglyargasnarghislysileaspgly
340345350
aspglugluserserserasphisglyleutrpleuseralaargala
355360365
glyasnargileleuargmetaspglyserproalagluhisthrphe
370375380
glylysileleuileaspargilephehis
385390
<210>22
<211>351
<212>prt
<213>多色青霉
<400>22
alaasplyshistyralailemetaspasnasptrptyrthralagly
151015
phevalprotyrleuilealaleuaspglyaspvalgluvalleugly
202530
leualaseraspthralaasnthrtrpglnproglnvalalaleuhis
354045
alavalalathrleuglualaglyasnleusercysileprovaltyr
505560
proglyserthrtrpproleuileasnthrproasnargpheglnala
65707580
trpglumetvalhisglylysleuprotrpgluglyalaphealapro
859095
gluasnlysthrleuglualagluglyasnaspprothrserglyasn
100105110
proasnargilevallysalaalaphelysgluglypheprolysgly
115120125
lysprogluasnargthrseralaalaasnphemetvalglumetval
130135140
hislystyrproglyglnvalseriletyrseralaglyalaleuthr
145150155160
asnvalalaleualavalargmetaspproglnphealaserleuala
165170175
lysgluleuvalilemetglyglytyrvalaspleuasnmetleugln
180185190
alathrglyservalleuleualaaspleuglnseraspileasnleu
195200205
metileaspproglualaserlysilealaleuthralagluphepro
210215220
asnilethrilealaglyasnvalalaasnglnvalpheprothrlys
225230235240
gluphevalaspgluilealaservalproasnprotyrserlysleu
245250255
phehisasptyrtyraspleuserpheprophetrpaspgluthrala
260265270
alaalaleumetvalaspprothrleualathrasnglnthrserval
275280285
pheleuaspvalaspthralatyrglyserproasntyrglyasnile
290295300
hisvaltyrglnlysalaleualaprovalglyilearggluvalasn
305310315320
phevalpheglnvalaspglyaspargleulysglnargilelyshis
325330335
serleuglntyrprolyssercysalaaspleuargasngluarg
340345350
<210>23
<211>1185
<212>dna
<213>多色青霉
<400>23
gttgagattctacttatctctttaacatttggaaacattgaggtgaagaactgtcttcga60
aatgtggtctccatgtttcatatcctcgagcgcgagatccagtggcgtcgtggtaacggc120
aagtccgaaggctatggcactatgcgtgctttccgcccagtagtagccgtgggagcggaa180
gatcccttggaagaccagaagatgctcgctgattatttccatggaaccgatggccttggt240
ggcatccatgctagtcacccacatctcactccaagcaaggcctgggagcatctattcacc300
ccggccgtggatccccaggggatcgagcctgtgcaaacgggagctggtcccggcgaccat360
tcctttatcccatcaagactacctgcacacaaggagattcttcgtgcactgcgccagaat420
gagcctgacaccgtgactctcgtggcggttggtccactgaccaacttggccttggcagca480
gcagaggatcccgaaaccttcctacgtgtcaaggaggtcgttgtgatgggtggagcaatc540
aaccagcctggaaatgtcacccccgttggagaattcaacgcctacgcagacgccgttgca600
gctgcgcgagtctttgcgctgacatcacctaatcccaactcgactctaccaccgaccacg660
agtccactacttggcctgtaccctgcaaagctcagccgacaattgactctgcgtctcttc720
ccgctggacatcaccctgcgccataacctgtcccgcggccaattccgccaagcagttgag780
cctctcctcgcaacaggctcacccctcgctgaatgggtgacagcattcatgggacacacg840
ttccgaaccctggaacgcctgcaccccggccatgagggcgatgaagcccagctgagtctc900
cacgaccctgtctgtgtgtggtatgcccttacagcagaggattcgcactggactccctcc960
gccaattccccagaggacattcgtgttgagacattgggccagtggacgcgtggtatgtgc1020
gtaatcgatggccgaaaccgccataagattgatggcgacgaggaaagctcgagtgatcat1080
ggtctgtggttgagtgctcgtgcaggaaaccgcattttgcgaatggatggatcgccagcc1140
gaacacacgttcggcaagatcctcatcgatagaatcttccactaa1185
<210>24
<211>1056
<212>dna
<213>多色青霉
<400>24
gccgacaagcactatgccatcatggacaatgattggtacacagcgggtttcgtgccttac60
ctgatcgccctcgatggcgatgtggaggttctgggcctagcctctgacaccgcaaacacc120
tggcagcctcaggtcgctctgcacgctgtcgcaactctggaagctggcaacttgagctgt180
atccccgtttacccaggctcgacatggccgctcatcaacacccccaaccgcttccaggcg240
tgggaaatggttcatggcaagctgccatgggagggtgcttttgcgccggagaacaagact300
ctcgaggccgagggtaacgatcctacctctggcaaccccaaccgtatcgtcaaggccgct360
ttcaaggaagggttccccaagggcaagcccgagaacagaacatctgctgccaacttcatg420
gtcgagatggtgcacaagtaccccggccaggtctcgatctactctgctggagccctgacc480
aatgttgcgctggctgtgcgcatggatccccagtttgcatctctggctaaggagttggtt540
atcatgggtggatacgtcgatttgaatatgctccaggccactggaagtgtcttgctggct600
gatcttcaatctgatatcaacttgatgattgatcccgaggcctccaagatcgcattgact660
gccgaattccccaatatcaccatcgccggtaacgtcgccaaccaggtctttcctaccaag720
gagttcgtcgacgagatcgcctccgttccaaacccctacagcaagctcttccacgactac780
tacgatctgtccttccccttctgggatgagacggctgccgcgctgatggttgaccctact840
cttgctaccaaccagacctctgtcttcctcgacgtggataccgcttatggtagccccaac900
tatggtaacattcacgtttaccagaaggctcttgcccctgttggtatccgggaggtcaac960
tttgtcttccaggttgatggggatagacttaagcagcgcatcaagcactctctgcagtac1020
cccaagtcatgcgccgacctgagaaatgagcgttga1056
- 还没有人留言评论。精彩留言会获得点赞!