调节植物中的种子和器官尺寸的方法与流程
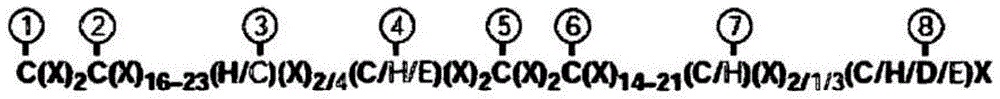
本申请是以下申请的分案申请:申请日:2014年11月05日;申请号:201480061807.4;发明名称“调节植物中的种子和器官尺寸的方法”。
本发明涉及改变植物的种子和器官的尺寸的方法,例如用以提高植物产量。
背景技术:
种子和器官的尺寸是农艺学和生态学上重要的性状,是受到遗传控制的(alonso-blanco,c.pnasusa96,4710-7(1999);song,x.j.natgenet39,623-30(2007);weiss,j.intjdevbiol49,513-25(2005);dinneny,j.r.development131,1101-10(2004);disch,s.currbiol16,272-9(2006);science289,85-8(2000);horiguchi,g.plantj43,68-78(2005);hu,yplantj47,1-9(2006);hu,y.plantcell15,1951-61(2003);krizek,b.a.devgenet25,224-36(1999);mizukami,y.pnasusa97,942-7(2000);nath,u.science299,1404-7(2003);ohno,c.k.development131,1111-22(2004);szecsi,j.emboj25,3912-20(2006);white,d.w.pnasusa103,13238-43(2006);horvath,b.m.emboj25,4909-20(2006);garcia,d.plantcell17,52-60(2005)。在给定的物种中,最终的种子和器官的尺寸是恒定的,而物种间的种子和器官的尺寸变化是非常大的,表明植物具有调节机制,能以协调的和适时地的方式控制种子和器官的生长。尽管种子和器官的尺寸很重要,但是对植物中控制最终器官和种子尺寸的分子和遗传学机制仍知之甚少。
已经在包括番茄、大豆、玉米和水稻的植物中使用数量性状基因座(qtl)定位研究了种子尺寸的遗传学调节。迄今为止,在已公开的文献中,鉴定了两个基因(song,x.j.natgenet39,623-30(2007);fan,c.theor.appl.genet.112,1164-1171(2006)),这两个基因是负责水稻籽粒大小的两个主要的qtl,但是这些基因的分子机制还有待阐明。在拟南芥中,在保藏物ler和cvi的杂交中,已经定位了十一个影响种子重量和/或长度的基因座{alonso-blanco,1999supra},但是还没有鉴定到对应的基因。近期有研究揭露,ap2和arf2参与到种子尺寸的控制中。但遗憾的是,ap2和arf2突变体具有比野生型更低的可育性(schruff,m.c.development137,251-261(2006);ohto,m.a.pnasusa102,3123-3128(2005);jofuku,k.d.pnasusa102,3117-3122(2005))。此外,使用突变体植物的研究已经鉴定了多个正和负调节子,其通过作用于细胞增殖或扩增来影响器官尺寸{krizek,b.a.devgenet25,224-36(1999);mizukami,y.procnatlacadsciusa97,942-7(2000);nath,u.science299,1404-7(2003);ohno,c.k.development131,1111-22(2004);szecsi,j.emboj25,3912-20(2006);white,d.w.pnasusa103,13238-43(2006);horvath,b.m.emboj25,4909-20(2006);garcia,d.plantcell17,52-60(2005).horiguchi,g.plantj43,68-78(2005);hu,yplantj47,1-9(2006)dinneny,j.r.development131,1101-10(2004))。
已了解涉及到泛素化相关活性的几个因子能影响种子尺寸。da1,一种生长限制因子,是一种泛素受体,并且含有两个在体外结合泛素的泛素相互作用基序(uim),并且da1-1突变体通过影响胚珠的母本珠被形成大的种子(li等,2008)。编码e3泛素连接酶bigbrother(bb)(disch等,2006;li等,2008)的da1-1的增强子(eod1)中的突变,协同地增da1-1的种子尺寸表型,表明da1和eod1/bb协同地作用从而控制种子尺寸。
控制种子和器官两者的最终尺寸的进一步的因子的鉴定,不仅会推进植物中尺寸控制机制的理解,而且可能在例如提高农作物产量和提高用于产生生物燃料的植物生物质中具有重要的实际应用。
技术实现要素:
本发明的发明人意外地发现植物da1蛋白中的lim结构域和/或lim类似结构域的打断不会消除da同源二聚化或活性,但是反而给予了显性负性表型。
本发明的一个方面提供了一种提高植物的产量或增强植物中的产量相关性状的方法;包括在所述植物的细胞内表达具有失活的lim结构域或lim类似结构域的da1蛋白。
da1蛋白可包括相对于野生型序列的一个或多个突变,这些突变打断或失活da1蛋白的lim结构域或lim类似结构域。
表达具有打断的或失活的lim结构域或lim类似结构域的da1蛋白增强植物的一个或多个产量相关性状或提高植物的产量。
具有失活的lim结构域或lim类似结构域的da1蛋白可以从植物的一个或多个细胞中的异源核酸编码序列表达,或可以从植物的一个或多个细胞中的内源的核酸编码序列表达。
本发明的另一个方面提供了一种产生具有提高的产量和/或一个或多个增强的产量相关性状的植物的方法,包括:
向植物细胞中引入编码具有失活的lim结构域或lim类似结构域的da1蛋白的异源核酸,或
向编码da1蛋白的植物细胞的核苷酸序列中引入突变,使得da1蛋白的lim结构域或lim类似结构域被失活,以及
从该植物细胞再生该植物。
本发明的另一个方面提供了一种植物细胞,该植物细胞包含编码具有失活的lim结构域或lim类似结构域的da1蛋白的异源核酸。
本发明的另一个方面提供了一种植物,该植物包含一种或多种植物细胞,该一种或多种植物细胞包含编码具有失活的lim结构域或lim类似结构域的da1蛋白的异源核酸。
该植物可显示相对于对照的提高的产量和增强的产量相关性状。
附图说明
图1显示了基于135个人lim序列的分析获得的lim结构域中的八个锌结合残基(1-8)的间隔和一致性。很少观察到的lim结构域的保守序列和拓扑图(topography)的模式(<10%)。
图2显示了lim结构域的锌配位拓扑图。紫色圆圈表示锌结合残基。半保守的脂肪族的/体积大的残基以绿色显示,具有不变的间隔的非保守的残基以红色表示。虚线黄色圆圈表示在间隔内的可能的可变数量的残基(x)。
图3显示了体外免疫沉淀反应,其显示了da1lim8对野生型da1的结合。在用谷胱甘肽琼脂糖珠纯化和对gst和flag进行免疫印迹之前,将大肠杆菌(e.coli)表达的gst标签的诱饵蛋白与大肠杆菌表达的flag标签的捕获蛋白进行温育。flag-da1和flag-da1lim8与gst-da1和gst-da1lim8(泳道5、6、8、9)共纯化,但是不和阴性对照gst-gus(泳道2、3)共纯化;表明da1中的lim结构域的突变不足以消除da1蛋白之间的物理相互作用。
图4显示了在col背景下lim8突变对种子尺寸的作用。
发明的具体实施方式
本发明涉及植物中da1蛋白的表达,在da1蛋白中lim或lim类似结构域被打断或失活(本文中统称为lim打断的da1蛋白)。这在改变影响产量,诸如种子和器官尺寸的植物性状中是有用的。
da1是一种植物泛素受体,已在li等(2008)、wang等(2012)和wo2009/047525中有详细描述。
da1蛋白的特征是存在一个lim结构域、一个lim类似结构域、一个保守的c末端结构域和一个或多个uim结构域。
lim结构域包括两个锌指基序并可以具有氨基酸序列(seqidno:1);
c(x)2c(x)16-23(h/c)(x)2/4(c/h/e)(x)2c(x)2c(x)14-21(c/h)(x)2/1/3(c/h/d/e)x
其中x是任意氨基酸,并且锌配位残基(zncoordinatingresidues)以下划线标示出。
lim结构域中的锌配位残基可以是c、h、d或e,优选为c。
在一些优选的具体实施方式中,lim结构域可以包括cxxc、hxxcxxcxxc和hxxc基序,其中x是任意氨基酸。比例,lim结构域可以包括氨基酸序列(seqidno:2);
c(x)2c(x)16-23(h)(x)2(c)(x)2c(x)2c(x)14-21h(x)2cx
其中x是任意氨基酸,并且锌配位残基以下划线标示出。
在一些具体实施方式中,lim结构域可以包括atda1lim结构域的氨基酸序列;
cagcnmeighgrflnclnslwhpecfrcygcsqpiseyefstsgnypfhkacy
(seqidno:3;锌配位残基以下划线标示出)
包含da1氨基酸序列的lim结构域在内的其它lim结构域如表1(虚线框)所示,例如seqidno:4(si_gi-514815267.pro)的残基141至193、seqidno:5(bd_gi-357157184.pro)的残基123至175、seqidno:6(br_da1b.pro)的残基155至207,seqidno:7(br_da1a.pro)的残基172至224、seqidno:8(at_gi-15221983.pro)的残基172至224、seqidno:9(tc_gi-508722773.pro)的残基117至169、seqidno:10(gm_gi-356564241.pro)的残基117至169、seqidno:11(gm_gi-356552145.pro)的残基121至173、seqidno:12(vv_gi-302142429.pro)的残基119至171、seqidno:13(vv_gi-359492104.pro)的残基122至174、seqidno:14(sl_gi-460385048.pro)的残基125至177、seqidno:15(os_gi-218197709.pro)的残基516至568、seqidno:16(os_gi-115466772.pro)的残基124至176、seqidno:17(bd_gi-357160893.pro)的残基150至202、seqidno:18(bd_gi-357164660.pro)的残基132至184、seqidno:19(sb_gi-242092232.pro)的残基124至176、seqidno:20(zm_gi-212275448.pro)的残基147至199、seqidno:21(at_gi-240256211.pro)的残基190至242、seqidno:22(at_gi-145360806.pro)的残基162至214、seqidno:23(at_gi-22326876.pro)的残基1240至1291、seqidno:24(at_gi-30698242.pro)的残基80至122、seqidno:25(at_gi-30698240.pro)的残基347至402、seqidno:26(at_gi-15240018.pro)的残基286至341或seqidno:27(at_gi-334188680.pro)的残基202至252。
lim结构域序列可以使用标准的序列分析技术(如简单模块结构研究工具(simplemodulararchitectureresearchtool,smart);emblheidelberg,de)来鉴定。
lim类似结构域包括两个锌指基序并可包括cxxc、hxxxxxxxcxxh和cxxc基序,其中,x是任意氨基酸。比例,lim类似结构域可包括氨基酸序列(seqidno:28);
其中x是任意氨基酸,锌配位残基以实线下划线示出,并且推定的锌配位残基以点线下划线示出
优选地,lim类似结构域可以包括氨基酸序列(seqidno:29);
其中x是任意氨基酸,锌配位残基以实线下划线示出,并且推定的锌配位残基以点线下划线示出。
更优选地,lim类似结构域可以包括氨基酸序列(seqidno:30);
其中x是任意氨基酸,锌配位残基以实线下划线示出,并且推定的锌配位残基以点线下划线示出。
在一些具体实施方式中,lim类似结构域可以包括atda1lim类似结构域的氨基酸序列;
包括da1氨基酸序列的lim结构域在内的其它lim类似结构域如表1(实线框)所示,例如seqidno:4(si_gi-514815267.pro)的残基200至266、seqidno:5(bd_gi-357157184.pro)的残基182到248、seqidno:6(br_da1b.pro)的残基214到280、seqidno:7(br_da1a.pro)的残基231到297、seqidno:8(at_gi-15221983.pro)的残基231到297、seqidno:9(tc_gi-508722773.pro)的残基176到242、seqidno:10(gm_gi-356564241.pro)的残基176到242、seqidno:11(gm_gi-356552145.pro)的残基180到246、seqidno:12(vv_gi-302142429.pro)的残基178到244、seqidno:13(vv_gi-359492104.pro)的残基181到247、seqidno:14(sl_gi-460385048.pro)的残基184到250、seqidno:15(os_gi-218197709.pro)的残基575到641、seqidno:16os_gi-115466772.pro)的残基183到149、seqidno:17(bd_gi-357160893.pro)的残基209到275、seqidno:18(bd_gi-357164660.pro)的残基191到257、seqidno:19(sb_gi-242092232.pro)的残基183到249、seqidno:20(zm_gi-212275448.pro)的残基206到272、seqidno:21(at_gi-240256211.pro)的残基249到315、seqidno:22(at_gi-145360806.pro)的残基221到287、seqidno:23(at_gi-22326876.pro)的残基1298到1363、seqidno:24(at_gi-30698242.pro)的残基130到176、seqidno:25(at_gi-30698240.pro)的残基406到465、seqidno:26(at_gi-15240018.pro)的残基345到404或seqidno:27(at_gi-334188680.pro)的残基256到319。
其它da1蛋白中的lim类似结构域序列可以利用上述信息使用标准的序列分析技术(如简单模块结构研究工具(例如simplemodulararchitectureresearchtool,smart);emblheidelberg,de)来鉴定。
除了lim结构域和lim类似结构域外,da1蛋白可以进一步包括羧基末端区域,该羧基末端区域具有与下列序列至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%和至少98%氨基酸一致性的氨基酸序列:seqidno:4的残基198至504、seqidno:5的残基180到487、seqidno:6的残基212到514、seqidno:7的残基229到532、seqidno:8的残基229到532、seqidno:9的残基174到487、seqidno:10的残基174到474、seqidno:11的残基178到478、seqidno:12的残基176到462、seqidno:13的残基179到482、seqidno:14的残基182到486、seqidno:15的残基573到878、seqidno:16的残基181到486、seqidno:17的残基207到512、seqidno:18的残基189到491、seqidno:19的残基181到486、seqidno:20的残基204到508、seqidno:21的残基247到553、seqidno:22的残基219到528、seqidno:23的残基1296到1613、seqidno:24的残基128到450、seqidno:25的残基404到702、seqidno:26的残基343到644或seqidno:27的残基256到587。
da1蛋白的羧基末端区域可以包括金属肽酶活性中心基序hemmh(seqidno:32)。
该羧基末端区域可以进一步包括位于lim结构域和hemmh基序之间的ek(x)8r(x)4seeq(seqidno:33)或ek(x)8r(x)4seq(seqidno:34)基序。
除了lim结构域和保守的羧基末端区域之外,da1蛋白可以包括uim1结构域和uim2结构域。uim1和uim2结构域可以位于da1蛋白的n末端和lim结构域之间。
uim1结构域可以由seqidno:35的序列组成,并且uim2结构域可以由seqidno:36的序列组成。
p---plpbalpb.sbp-.ppp(seqidno:35)
p---plpbalpb.sbp-sppp(seqidno:36)
其中;
p是极性氨基酸残基,例如c、d、e、h、k、n、q、r、s或t;
b是大的氨基酸残基,例如e、f、h、i、k、l、m、q、r、w或y;
s是小的氨基酸残基,例如a、c、d、g、n、p、s、t或v;
l是脂肪族的氨基酸残基,例如i、l或v;
.是缺失或是任意氨基酸,以及
-是任意氨基酸。
uim1和uim2结构域序列的进一步例子可以使用如本文所述的标准的序列分析技术(如简单模块结构研究工具(smart);emblheidelberg,de)来鉴定。
在一些优选的具体实施方式中,da1蛋白可以包括;
seqidno:1的lim结构域,
seqidno:28的lim类似结构域,
c末端区域,该c末端区域具有与seqidno:8的残基229至532至少20%序列一致性或具有与如上列举的seqno4至7或9至27中任意一个相同的区域,并包括ek(x)8r(x)4seeq或ek(x)8r(x)4seq基序以及hemmh基序,
seqidno:35的uim结构域,以及
seqidno:36的uim结构域。
da1蛋白可以包括如表1所示的植物da1蛋白的氨基酸序列(seqidno:4至27)或可以是这些序列中一个的具有da1活性的等位基因或变体。
例如,da1蛋白可以包括atda1、atdar1、atdar2、atdar3、atdar4、atdar5、atdar6、atdar7、brda1a、brda1b、brdar1、brdar2、brdar3-7、brdal1、brdal2、brdal3、osda1、osdar2、osdal3、osdal5、ppdal1、ppdal2、ppdal3、ppdal4、ppdal5、ppdal6、ppdal7、ppdal8、smdal1、smdal2或zmda1(acr35367.1gi:238008664)的氨基酸序列,优选地包括atda1、atdar1brda1a、brda1b、osda1或zmda1的氨基酸序列或这些序列中的一个的等位基因或变体。
在一些优选的具体实施方式中,da1蛋白可以包括atda1(seqidno:8;at1g19270;np_173361.1gi:15221983)的氨基酸序列或可以是这个序列的具有da1活性的等位基因或变体。
在任何感兴趣的植物物种中,包含如上所述的特征和编码da1核酸序列的其它da1蛋白序列可以使用标准的序列分析技术工具来鉴定。
感兴趣的植物物种中的da1蛋白可以具有本文列出的da1蛋白参考氨基酸序列的变体的氨基酸序列。
一种da1蛋白,它是参考植物da1序列,如seqidno:4-27中任意一个的同源物或变体,可以包括具有与参考序列至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或至少98%的序列一致性的氨基酸序列。
出现在一个植物物种中的特定的氨基酸序列变体可以通过1个氨基酸、2、3、4、5-10、10-2020-30、30-50或超过50个氨基酸的插入、增加、替换或缺失与本文列出的参考序列形成差异。
在一些具体实施方式中,seqno:4至27中任意一个的da1多肽,其是atda1序列的变体,可以包括具有seqidno:3的序列的lim结构域和具有seqidno:31的序列的lim类似结构域。
编码da1蛋白的核酸可以包括在选自由nm_101785.3gi:42562170(atda1);nm_001057237.1gi:115454202(osda1);bt085014.1gi:238008663(zmda1)组成的组的数据库条目中选出的核苷酸序列,或可以是这些序列中的一个编码有活性的da1蛋白的等位基因或变体。
在一些优选的具体实施方式中,编码da1蛋白的核酸可以包括atda1(nm_101785.3gi:42562170)、zmda1(bt085014.1gi:238008663)、osda1(nm_001057237.1gi:115454202)的核苷酸序列,或可以是这些序列中的一个编码具有da1活性的蛋白的等位基因或变体。
一种核酸,其编码感兴趣的植物物种中的da1蛋白,可以具有如本文列出的da1蛋白参考核苷酸序列的变体的核苷酸序列。
da1多肽和编码核酸可以使用常规的序列分析技术,在植物物种,尤其是诸如小麦、大麦、玉米、水稻和其它农业植物的作物中鉴定。
例如,变体核苷酸序列可以是本文列出的参考da1序列的同源物,并且可以通过核酸中一个或多个核苷酸的一个或多个增加、插入、缺失或替换,例如核酸中2、3、4、5-10、10-20、20-30、30-50或超过50个核苷酸的增加、插入、缺失或替换,导致编码的蛋白中的一个或多个氨基酸的增加、插入、缺失或替换,从而与参考da1核苷酸序列形成差异。当然,也包括对核酸的不造成编码的氨基酸序列变化的改变。编码da1蛋白的核酸可以包括具有与参考核酸序列至少20%或至少30%序列一致性的序列,优选地,至少40%、至少50%、至少60%、至少65%、至少70%、至少80%、至少90%、至少95%或至少98%的序列一致性。序列一致性如本文描述的。
序列一致性根据算法gap(wisconsinpackage,accelerys,圣地亚哥,美国)进行通常地定义。gap使用needleman和wunsch算法将两个完整的序列进行比对,最大化匹配(match)的数量并最小化空位(gap)的数量。通常,使用默认参数,空位产生罚分=12且空位延伸罚分=4。可以优选使用gap,但是也可以使用其它算法,例如blast(其使用altschul等(1990)j.mol.biol.215:405-410中的方法)、fasta(其使用pearson和lipmanwhich(1988)pnasusa85:2444-2448中的方法)或smith-waterman算法(smith和waterman(1981)j.molbiol.147:195-197)或altschul等(1990)supra中的tblastn程序,通常采用默认参数。特别地,可使用psi-blast算法(nucl.acidsres.(1997)253389-3402)。
可对本文描述的相关序列的全长进行序列比较。
一种da1核苷酸序列,其是本文列出的参考da1核酸序列的变体,可以在严谨条件下与该参考核酸序列或其互补序列选择性地杂交。
严谨条件包括,例如对于大约80-90%一致性的序列的杂交,在ph7.2的0.25mna2hpo4、6.5%sds、10%硫酸葡聚糖中于42℃杂交过夜,并在0.1xssc、0.1%sds中55℃最终洗涤。对于大于大约90%一致性的序列的检测,合适的条件包括在ph7.2的0.25mna2hpo4、6.5%sds、10%硫酸葡聚糖中于65℃杂交过夜,并在0.1xssc、0.1%sds中60℃最终洗涤。
一种可替换的选择,其可能对植物核酸制备尤其适合,是5xsspe溶液(最终0.9mnacl、0.05m磷酸钠、0.005medtaph7.7)、5xdenhardt's溶液、0.5%sds,在50℃或65℃过夜。根据需要,可以在0.2xssc/0.1%sds中,于65℃进行洗涤,或者于50-60℃在1xssc/0.1%sds中进行洗涤。
da1蛋白和编码核酸,可以使用常规的序列分析技术和/或与本文列出的参考序列的比较,在植物物种,尤其是诸如小麦、大麦、玉米、水稻和其它农业植物的作物中鉴定。
da1蛋白的lim结构域、lim类似结构域或lim结构域和lim类似结构域两者,根据本文描述的应用,可能被失活或打断(“lim打断的da1蛋白”)。
lim结构域和lim类似结构域已在上方详细描述,并且可以使用标准序列分析技术在任何da1蛋白中被鉴定。
具有失活的或打断的lim结构域或lim类似结构域的da1蛋白可能显示出异常的肽酶活性,例如提高的或激活的肽酶活性。例如,lim结构域或lim类似结构域的失活或打断可能减少或阻止该结构域与da1蛋白的c末端区域的相互作用,并且抑制da1肽酶活性。
在一些具体实施方式中,与野生型da1蛋白相比,具有失活的或打断的lim结构域或lim类似结构域的da1蛋白在泛素化后可能会显示出在植物细胞中降低的稳定性。
打断的或失活的lim结构域或lim类似结构域可能不能与锌配位(coordinate)或形成锌指基序,以致该结构域的功能被消除,即打断的lim或lim类似结构域不能介导蛋白:蛋白相互作用。例如,打断的lim结构域或lim类似结构域可能不能和da1蛋白的c末端区域进行分子内的相互作用以抑制肽酶活性。
失活的或打断的lim结构域或lim类似结构域可以包括消除lim或lim类似结构域中的一个或多个锌指基序的序列改变或突变。
da1蛋白的氨基酸序列可以通过相对于野生型氨基酸序列的一个或多个氨基酸的插入、替换或缺失改变或突变,以失活lim结构域或lim类似结构域。例如,可以相对于野生型氨基酸序列改变,例如缺失或替换1、2、3、4、5、6、7、8、9或10个或更多个氨基酸。在一些具体实施方式中,可以改变1至30、1至20或1至10个残基。
lim结构域和lim类似结构域中的单个氨基酸的替换已经足够引起lim敲除表型。例如,lim结构域可以通过lim结构域中的锌配位残基或其它残基的突变来失活(mcintosh等(1998)amjhumangenet631651-1658l;clough等(1999)humanmutation14459-465;hamlington等(2001)humanmutation18458-464;taira等,nature1994372,677-9;agulnick等,nature1996384,270-2)。lim类似结构域也可以通过lim类似结构域中的锌配位残基(即保守的cys残基)或其它残基的突变来失活(yang等theplantjournal,(2010),63,283–296)。
合适的失活或打断突变优选地在lim结构域或lim类似结构域中或其附近。
失活的或打断的lim结构域或lim类似结构域可以包括一个或多个锌配位残基、或推定的锌配位残基,例如cxxc或cxxh环境中的半胱氨酸或组氨酸残基的突变、和/或一个或多个非锌配位氨基酸残基的突变。
失活的或打断的lim结构域可以包括如上述seqidno:1或seqidno:2所示的lim结构域中第一个、第二个、第三个、第四个、第五个、第六个、第七个和第八个锌配位残基中的一个或多个的突变。例如,失活的或打断的lim结构域可以包括cxxc基序中的一个或多个半胱氨酸残基或hxxc基序中的半胱氨酸或组氨酸残基处的突变,例如seqidno:3的位点1、4、22、25、28、31、49和52所示的半胱氨酸/组氨酸残基,以及上述seqidno1和2中以下划线标示出的半胱氨酸/组氨酸残基。所述da1蛋白的lim结构域可以包括上方所示的da1lim结构域中的以下划线标示出的残基中的一个或多个的突变,优选地seqidno:4的da1序列的c141、c144、h162、c165、c168、c171、h189和c192,seqidno:5的da1序列的c123、c126、h144、c147、c150、c153、h171和c174,seqidno:6的da1序列的c155、c158、h176、c179、c182、c185、h203和c206,seqidno:7的da1序列的c172、c175、h193、c196、c199、c202、h220和c223,seqidno:8的atda1序列的c172、c175、h193、c196、c199、c202、h220和c223,seqidno:9的da1序列的c117、c120、h138、c141、c144、c147、h165和c168,seqidno:10的da1序列的c177、c180、h198、c201、c204、c207、h225和c228,seqidno:11的da1序列的c121、c124、h142、c145、c148、c151、h169和c172,seqidno:12的da1序列的c119、c122、h140、c143、c146、c149、h167和c170,seqidno:13的da1序列的c122、c125、h143、c146、c149、c152、h170和c173,seqidno:14的da1序列的c125、c128、h146、c149、c152、c155、h173和c176,seqidno:15的da1序列的c516、c519、h537、c540、c543、c546、h564和c567,seqidno:16的da1序列的c124、c127、h145、c148、c151、c154、h172和c175,seqidno:17的da1序列的c150、c153、h171、c174、c177、c180、h198和c201,seqidno:18的da1序列的c132、c135、h153、c156、c159、c162、h180和c183,seqidno:19的da1序列的c124、c127、h145、c148、c151、c154、h172和c175,seqidno:20的da1序列的c147、c150、h168、c172、c175、c178、h196和c199,seqidno:21的da1序列的c190、c193、h211、c204、c207、c210、h228和c231,seqidno:22的da1序列的c162、c165、h183、c186、c189、c192、h210和c213,seqidno:23的da1序列的c1240、c1243、h1261、c1264、c1267、c1270、h1287和c1290,seqidno:25的da1序列的c347、c350、h368、c371、c374、c377、h398和c401,seqidno:26的da1序列的c286、c289、h307、c310、c313、c316、h337和c340,seqidno:27的da1序列的c201、c204、h222、c225、c228、c231、h248和c251,或其它da1蛋白序列中的等价的半胱氨酸残基。
例如,lim打断的da1蛋白可以在这些位点中的一个或多个处具有c到y、c到g或其它替换。
da1蛋白的lim结构域中的锌配位残基可以通过标准序列分析来鉴定。与seqidno:8中c172、c175、h193、c196、c199、c202、h220和c223等价的半胱氨酸和组氨酸残基是一个不同的da1蛋白序列中的相同序列环境中的序列残基,并且可以通过标准序列分析来鉴定,如表1所示。
失活的或打断的lim结构域可以包括如上述seqidno:1或seqidno:2中所示的lim结构域中的一个或多个非锌配位残基的突变。非锌配位残基可以位于lim结构域序列中的锌配位残基中的4个残基之内,或可以位于从锌配位残基起的4个或更多个残基。
失活的或打断的lim类似结构域可以包括如上述seqidno:28-31所示的lim类似结构域中的第一个、第二个、第三个、第四个、第五个、第六个、第七个和第八个锌配位残基或推定的锌配位残基中的一个或多个的突变。例如,失活的或打断的lim类似结构域可以包括cxxc基序中的一个或多个半胱氨酸残基或cxxh基序中的半胱氨酸或组氨酸残基处的突变,例如seqidno:31的位点1、4、29、32、40、43、63或66所示的半胱氨酸/组氨酸残基以及上述seqidno28至31中以下划线标示的半胱氨酸/组氨酸残基。lim类似结构域中的三个推定的锌配位残基h252、c260、h263中的两个负责锌配位(即h252和c260;h252和h263;或c260和h263)。所述da1蛋白的lim类似结构域可以包括上方所示的atda1lim类似结构域中的以下划线标示出的残基中的一个或多个的突变,优选地seqidno:8的atda1序列的c232、c235、h252、c260、h263、c271、c274、c294和/或c297,或其它da1蛋白序列中的等价半胱氨酸残基。例如,lim打断的da1蛋白可以在这些位点中的一个或多个处具有c到y、c到g或其它替换。
与seqidno:8中的c232、c235、h252、c260、h263、c271、c274、c294和c297等价的半胱氨酸残基,是一个不同的da1蛋白序列中的相同序列环境中的序列残基,并且可以通过标准序列分析来鉴定,如表1所示。
失活的或打断的lim类似结构域可以包括如上述seqidno:28至seqidno:31中所示的保守的半胱氨酸或组氨酸残基之外的lim类似结构域中的一个或多个残基的突变。合适的残基可能位于lim类似结构域序列中的保守的半胱氨酸或组氨酸残基的4个残基之内,或可能位于从保守的半胱氨酸或组氨酸残基起的4个或更多个残基。
一些优选的突变包括lim或lim类似结构域中的锌配位残基的转变,诸如半胱氨酸或组氨酸变到诸如甘氨酸的中性氨基酸。
打断锌指基序以及适合于消除da1蛋白中的lim或lim类似功能的其它突变对本领域技术人员而言是显而易见的。不像da1蛋白中的其它结构域中的突变,lim结构域和lim类似结构域突变破坏了植物细胞中在与da1蛋白相互作用的配合体eod1存在下的da1蛋白的稳定。合适的lim结构域和lim类似结构域突变因此可以通过使用标准实验技术,通过测定在eod1存在下突变体da1蛋白的稳定性来鉴定。相对于野生型da1降低的稳定性指示了打断lim或lim类似结构域的突变。
本文描述的lim打断的da1蛋白可以包括保守的r残基,其位于da1氨基酸序列中等价于拟南芥(a.thaliana)da1的seqidno:8的位点358、玉米(z.mays)da1的seqidno:8的位点333、或其它da1氨基酸序列的等价位点的位点处,例如表1的da1序列(保守的r残基以箭头表示)。位于da1氨基酸序列中等价于拟南芥da1的seqidno:8的位点358或seqidno:20的玉米da1的位点333的位点的保守的r残基,是位于da1氨基酸序列内的对应于seqidno:20的r333和seqidno:8的r358的位点处的,即该保守的r残基相对于da1蛋白中的其它基序或结构域是在相同的位点处。该保守的r残基位于lim结构域和c末端区域的hemmh(seqidno:32)肽酶基序之间,并且在da1蛋白的相同序列环境中是完全保守的。该保守的r残基可以被包含在c末端区域内的ek(x)8r(x)4seeq(seqidno:33)或ek(x)8r(x)4seq(seqidno:34)中。
本文的数据显示,lim结构域和lim类似结构域不介导da1同源二聚化,并且lim打断的da1蛋白保持了与野生型da1结合的能力。
植物的一个或多个细胞中的lim-打断的da1蛋白的表达降低了细胞中的da1活性,并增强了产量相关的植物性状,诸如种子和器官尺寸(例如参见li等(2008);wo2009/047525;wang等2012),因此提高了植物产量。表达lim打断的da1蛋白的植物可能具有da1-1或类似da1-1的表型。
在一些具体实施方式中,可以从一个或多个植物细胞中的异源核酸表达lim-打断的da1蛋白。
lim打断的da1蛋白可以通过本领域中任何方便的技术和合适的技术在植物的一个或多个细胞中表达。
编码lim-打断的da1蛋白的核酸可以在来自它最初分离时的同一种植物物种或变种或在不同的植物物种或变种(即异源植物)中重组地表达。
提供的核酸可以是双链或单链、cdna或基因组dna、或rna。该核酸根据设计,可以是全部地或部分地合成的。自然地,技术人员了解当核酸包括rna时,涉及到所示的序列,应当解释为涉及以u替代t的rna等价物。
“异源的”表明讨论中的基因/核苷酸序列或调节讨论中的基因/序列的序列,通过遗传工程或重组方法,即通过人为干涉被引入到所述植物的细胞中或其祖先中。对一个植物细胞是异源的核苷酸序列可以是非自然地出现在那种类型、变体或物种中(即外源的或异质的),或可以是非自然地出现在细胞的亚细胞的或遗传环境中的序列,或可以是细胞中非自然地被调节的序列,即可操作地连接到非自然的调节元件上。
编码lim-打断的da1蛋白的核酸可以可操作地连接到异源调节序列上,诸如启动子,例如如上所述的组成型的、诱导型的、组织特异性的或发育特异性的启动子。
编码lim-打断的da1蛋白的核酸可以包含在核酸构建体或载体中。构建体或载体优选地是适合于植物细胞内的转染进入和/或表达。一种载体是、尤其是双链或单链的线性或环状形式的任何质粒、粘粒、噬菌体或农杆菌双元载体,其可以或不可以自我转移或移动,并且其能通过整合到细胞的基因组或以染色体外的存在(例如具有复制起点的自主复制质粒)转化原核的或真核的宿主,尤其是植物宿主。
特别地包括穿梭载体,使用穿梭载体意味着dna媒介物能自然的或通过设计地在两个不同的有机体中复制,两种不同的有机体可以选自放线菌(actinomyces)和相关的物种、细菌和真核(例如高等植物、哺乳动物、酵母或真菌)细胞。
如上所述的包括核酸的构建体或载体不需要包含启动子或其它调节序列,尤其是当载体被用于将核酸引入细胞中从而重组进入基因组中时。
构建体和载体可以进一步包括可选择的遗传标记,该可选择的遗传标记由给予可选择的表型,诸如抗生素抗性的基因组成,该抗生素诸如卡那霉素(kanamycin)、潮霉素(hygromycin)、膦丝菌素(spectinomycin)、氯磺隆(chlorsulfuron)、甲氨蝶呤(methotrexate)、庆大霉素(gentamycin)、奇霉素(spectinomycin)、咪唑啉酮(imidazolinones)、草甘膦(glyphosate)和d-氨基酸(d-aminoacids)。
本领域技术人员能构建载体及设计方案用于例如在微生物的或植物的细胞中的重组基因表达。可以选择或构建合适的载体,其含有适合的调节序列,包括启动子序列、终止子片段、聚腺苷酸化序列、增强子序列、标记基因和其它合适的序列。进一步地细节参见,例如分子克隆:实验操作指南(molecularcloning:alaboratorymanual):第三版,sambrook等,2001,冷泉港实验室出版社(coldspringharborlaboratorypress)和分子生物学技术(protocolsinmolecularbiology),第二版,ausubel等版本,johnwiley&sons,1992。之前在植物上广泛成功使用的特定方法和载体在bevan,nucl.acidsres.(1984)12,8711-8721)和guerineau及mullineaux(1993)植物转化和表达载体(planttransformationandexpressionvectors)中描述。在:plantmolecularbiologylabfax(croyrrded)oxford,biosscientificpublishers,pp121-148中被描述。
当向细胞中引入选定的基因构建体时,必须要考虑一定的注意事项,本领域技术人员是清楚知道的。待插入的核酸应当组装在一个构建体中,该构建体含有有效的启动转录的调节元件。有可获得的方法将构建体转运入细胞中。一旦构建体位于细胞膜内,将会发生或不会发生与内源的染色体材料的整合。最终,优选地是目的细胞类型,使得细胞能再生成为完整植株。
还适宜使用增强编码lim或lim类似-打断的da1蛋白的核酸的表达的构建体和转化方法。单拷贝的基因在植物细胞的基因组中的整合可以有利于将沉默效应最小化。类似的,控制整合的复杂性也对这一点有益。对这一点特别值得关注的是使用例如根据ep专利号ep1407000b1的最小基因表达构建体转化植物细胞,ep专利号ep1407000b1为了该目的通过引用并入本申请。
可以使用本领域技术人员熟知的技术以向植物细胞中引入核酸构建体和载体,从而产生具有本文描述的特征的转基因植物。
农杆菌转化是本领域技术人员广泛使用的转化植物种类的一种方法。产生稳定的、可育的转基因植物在本领域现在是常规的(参见例如goriyama等(1988)bio/technology6,1072-1074;zhang等(1988)plantcellrep.7,379-384;zhang等(1988)theorapplgenet76,835-840;himamoto等(1989)nature338,274-276;datta等(1990)bio/technology8,736-740;christou等(1991)bio/technology9,957-962;peng等(1991)internationalriceresearchinstitute,manila,philippines563-574;cao等(1992)plantcellrep.11,585-591;li等(1993)plantcellrep.12,250-255;rathore等(1993)plantmolecularbiology21,871-884;fromm等(1990)bio/technology8,833-839;gordon-kamm等(1990)plantcell2,603-618;d'halluin等(1992)plantcell4,1495-1505;walters等(1992)plantmolecularbiology18,189-200;koziel等(1993)biotechnology11,194-200;vasil,i.k.(1994)plantmolecularbiology25,925-937;weeks等(1993)plantphysiology102,1077-1084;somers等(1992)bio/technology10,1589-1594;wo92/14828;nilsson,o.etal(1992)transgenicresearch1,209-220)。
当农杆菌转化效率低或无效时,例如在一些裸子植物物种中,可以优选其它的方法,诸如微弹或颗粒轰击(us5100792、ep-a-444882、ep-a-434616)、电穿孔(ep290395、wo8706614)、显微注射(wo92/09696、wo94/00583、ep331083、ep175966、green等(1987)植物组织和细胞培养(planttissueandcellculture),academicpress)、直接dna摄取(de4005152、wo9012096、us4684611)、脂质体介导的dna摄取(例如freeman等plantcellphysiol.29:1353(1984))或涡旋振荡法(如kindle,pnasu.s.a.87:1228(1990d))。植物细胞转化的物理方法在oard,1991,biotech.adv.9:1-11中有综述。
可替换地,可以采用不同技术的组合以增强转化步骤的效率,例如用农杆菌包被的微粒进行轰击(ep-a-486234)或微弹轰击以引起伤口,随后与农杆菌共培养(ep-a-486233)。
在转化之后,如本领域常规的方法,可以从例如单个细胞、愈伤组织或叶盘来再生植株。几乎任何植物都能从植物的细胞、组织和器官中完整地再生。可获得的技术在vasil等,植物的细胞培养和体细胞遗传学(cellcultureandsomaticcellgeneticsofplants),i,ii和iii卷,实验操作及其应用(laboratoryproceduresandtheirapplications),academicpress,1984,以及weissbach和weissbach,植物分子生物学方法(methodsforplantmolecularbiology),academicpress,1989中有综述。
转化技术的特定选择将由其转化特定植物物种的效率以及采用特定选择方法论的操作本发明的人员的经验和偏好决定。将核酸引入植物细胞中的转化系统的特定选择不是本发明必须的或不是本发明的限制,植物再生技术的选择亦然。
转化之后,表达lim打断的da1蛋白的植物细胞可以被鉴定和/或选择。可以从该植物细胞再生植株。
在另一个具体实施方式中,可以在植物细胞的基因组内的核酸序列中引入突变,该核酸序列编码da1蛋白,从而使得该核酸序列编码lim-打断的da1蛋白。例如,可以在编码da1蛋白的lim结构域或lim类似结构域的序列中引入突变。然后可以从该突变的细胞中再生植株。
编码da1蛋白的核酸可以通过相对于野生型核苷酸序列的一个或多个核苷酸的插入、替换、或缺失进行突变。例如,相对于野生型核苷酸序列,可以改变1、2、3、4、5、6、7、8、9或10个或更多个核苷酸,从而失活编码的lim或lim类似结构域。这些突变失活或敲除lim结构域和/或lim类似结构域,并且优选地在编码lim结构域或lim类似结构域的核酸序列区域中。优选的突变不会引起移码。
目的基因的突变、失活或敲除技术在本领域是公知的(参见例如体外突变技术(invitromutagenesisprotocols);分子生物学方法(methodsinmolecularbiology)(第二版)edjeffbraman;sambrookj等2012.分子克隆:实验室指南(第四版)cshpress;分子生物学现代方法(currentprotocolsinmolecularbiology);edausubel等(2013)wiley)。在一些具体实施方式中,可以通过基因组编辑技术向目标da1编码序列中引入突变,例如rna引导的核酸酶技术,诸如crispr、锌指核酸酶(zfn)和反式激活因子样效应因子核酸酶(talen)(urnov,f.d.等naturereviews.genetics11,636-646(2010);joung,j.k.等naturereviews.molecularcellbiology14,49-55(2013);gasiunas,g.等pnasusa109,e2579-2586(2012);cong,l.等science339,819-823(2013))。
如上所述的表达lim打断的da1蛋白(即具有失活的或打断的lim结构域或lim类似结构域的da1蛋白)的植物可以有性地或无性地繁殖或种植以产生子代或后代。从一个或多个细胞再生的植株的子代或后代可以有性地或无性地繁殖或种植。该植株或其子代或后代可以和其它植物或它自己杂交。
可以相对于对照检测该植株或其子代或后代的种子尺寸、器官尺寸和/或植物产量。
表达本文描述的lim-打断的da1蛋白的植物相对于对照可以表现出增大的种子和/或器官尺寸,并且可以具有更高的植物产量。
显性负性da1等位基因对植物中的产量相关性状的作用在eod1表达或活性缺陷的植物中被增强了(li等(2008),wo2009/047525)。
lim-打断的da1蛋白可以如上所述的在eod1表达和活性缺陷的植物中表达。
eod1蛋白是植物e3泛素连接酶(disch等(2006)、li等(2008)、wo2009/047525)。eod1蛋白包括一个eod结构域。植物eod结构域可以由seqidno:37的氨基酸序列组成;
(e/k)rcvicq(l/m)(k/r/g/t/e)y(k/r)(r/i)(g/k)(d/n/e)(r/q/k/l)q(i/m/v)(k/n/t/a)l(l/p)c(k/s)h(v/a)yh(s/t/g/a)(e/q/d/s/g)c(i/g/t/v)(s/t)(k/r)wl(g/t/s)ink(v/i/a/k)cp(v/i)c(seqidno:37)
在一些优选的具体实施方式中,eod1蛋白可以包括eod结构域,eod结构域具有如下氨基酸序列:seqidno:38的残基150至192、seqidno:39的残基187至229、seqidno:40的残基192至234、seqidno:41的残基189至231、seqidno:42的残基194至236、seqidno:43的残基194至236、seqidno:44的残基194至236、seqidno:45的残基195至237、seqidno:46的残基189至231、seqidno:47的残基195至237、seqidno:48的残基195至237、seqidno:49的残基195至237、seqidno:50的残基218至260、seqidno:51的残基196至238、seqidno:52的残基197至239、或seqidno:53的残基193至235。
进一步适合的eod结构域可以使用本文描述的标准序列分析技术(如简单模块结构研究工具(smart);emblheidelberg,de)来鉴定。
eod1蛋白可以包括如表2中列出的seqidno38至53中的任意一个的氨基酸序列,该eod1蛋白的表达或活性在表达lim打断的da1蛋白的植物细胞中是降低的。在一些优选的具体实施方式中,eod1蛋白可包括seqidno:45(ateod1)或seqidno:50或51(oseod1)的氨基酸序列,或可以是保持e3泛素连接酶活性的这种序列的变体。
一种eod1蛋白,其是seqidno:38至53中任意一个的变体或其它参考eod1序列,可以包括具有与参考eod1序列至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或至少98%的序列一致性的氨基酸序列。
一种eod蛋白,其是seqidno:38至53中任意一个的变体,可以进一步包括一个具有seqidno:37的序列的eod结构域。适合的序列的例子如上方列出的。
编码eod1蛋白的核酸可以包括选自由xm_002299911.1gi:224059639(pteod1);xm_002531864.1gi:255582235(rceod1);xm_002279758.2gi:359487285(vveod1);xm_003542806.1gi:356548934(gmeod1a);xm_003540482.1gi:356544175(gmeod1b);xm_002468372.1gi:242042044(sbeod1);nm_001147247.1gi:226496788(zmeod1);ornp_001030922.1gi:79316205(ateod1;at3g63530)组成的组中的数据库条目中列出的核苷酸序列,或可以是这些序列中的一个的变体。
在一些优选的具体实施方式中,编码植物中的eod1蛋白的核苷酸序列可以编码ateod1或oseod1,或可以是它们的变体。
eod1蛋白和编码核酸,其表达或活性可以如本文描述的被降低,可以通过常规的序列分析技术很容易地在任何感兴趣的植物物种中被鉴定,尤其是在作物植物,诸如小麦、大麦、玉米、水稻和其它农业作物中被鉴定。
降低eod1表达或活性的合适的方法是本领域中公知的。
例如,eod1的活性可以通过引入突变来降低,优选地消除,该突变诸如在对应于seqidno:45的位点44的位点上的缺失、插入或替换,例如,a到t的替换。eod1蛋白序列中与seqidno:45的位点44等价的位点,可以使用标准序列分析和比对工具进行鉴定。
在一些具体实施方式中,可以通过在所述植物的细胞内表达编码或转录抑制子核酸的异源核酸从而降低植物细胞中的eod1蛋白的表达,抑制子核酸例如抑制子rna或rnai分子。抑制子rna抑制表达lim-打断的da1的植物细胞中eod1蛋白的表达。
合适的rnai序列可以对应于本文列出的参考eod1核苷酸序列的片段,或可以是它的变体。
在另一个具体实施方式中,可以在植物细胞的基因组内的核酸序列中引入突变,该核酸序列编码da1蛋白,从而使得该核酸序列编码lim-打断的da1蛋白。然后可以从突变的细胞中再生植株。
编码eod1的核酸可以通过相对于野生型核苷酸序列的一个或多个核苷酸的插入、替换或缺失进行突变。例如,可以相对于野生型核苷酸序列改变1、2、3、4、5、6、7、8、9或10个或更多个核苷酸。
lim-打断的da1蛋白如本文描述的在任何植物物种中表达。根据本文描述的发明的任何方面使用的适合的植物的例子,包括单子叶的和双子叶的高等植物,例如农业的或作物植物,诸如选自由紫草(lithospermumerythrorhizon)、红豆杉属(taxusspp)、烟草、葫芦科植物、胡萝卜、蔬菜芸苔属、瓜类植物、辣椒、葡萄藤、莴苣、草莓、油料种子芸苔属、糖用甜菜、小麦、大麦、玉米、水稻、大豆、豌豆、高粱、向日葵、番茄、土豆、胡椒、菊属、石竹属、亚麻籽、大麻和黑麦组成的组中的植物。
本发明的另一个方面提供了一种表达如上所述的lim打断的da1蛋白的转基因植物。
该植物可以包括编码lim-打断的da1蛋白的外源核酸。
相对于不表达lim-打断的da1蛋白的对照植物,该植物中的一个或多个产量相关性状可以在该植物中改善、提高或增强。产量相关性状可以包括寿命、器官尺寸和种子尺寸。
相对于对照野生型植物(即不表达lim-打断的da1蛋白的相同植物),该植物可以具有提高的产量。例如,相对于对照植物,每单位面积的种子质量(如谷粒)或其它植物产品提高了。
可以通过上述描述的方法产生合适的植物。
除了通过本文描述的方法产生的植物外,本发明还包含这样的植物、种子、自交或杂交子代或后代的任何克隆,以及这些中任意一个的可以用于有性或无性再生或繁殖的任何部分或繁殖体,例如插枝和种子。本发明还包含一种植物,其是这样一种植物、或该植物的任何部分或繁殖体、子代、克隆或后代经有性的或无性的繁殖的子代、克隆或后代。
根据本发明的植物可以是在一个或多个特性上不是纯育的。可将一些植物变种排除在外,特别是根据植物育种者权(plantbreedersrights)可注册的植物变种。
本文使用的“和/或”应理解为确切的公开了两个特定的特征或组成中的每一个与另一个一起或缺失另一个。例如“a和/或b”应理解为确切的公开了(i)a、(ii)b和(iii)a和b中的每一个,就如每一个都单独的在此列出。
除非内容有另外的描述,上述列出的特征的描述和定义不限于本发明中任何特定方面或具体实施方式,并且同样地适用于所描述的所有方面和具体实施方式。
本说明书中提到的所有文件,由于所有的目的,它们的全文通过引用并入本申请。
本说明书中提到的所有数据库条目的内容,由于所有的目的,它们的整体也通过引用并入本申请。这包括在本申请的提交日时的任何序列的版本。
实验
1、方法
1.1共免疫沉淀分析
这些研究的所有诱饵蛋白是gst-标记的,并且它们的下拉使用谷胱甘肽琼脂糖珠(gelifescience17-0756-01)。
在带有合适抗生素的10mllb的烧瓶中接种有合适表达构建体的bl21甘油贮存液,并让其在37℃和220rpm下生长过夜。第二天早晨,该10ml预培养物被用来接种100mllb烧瓶(以1:100的比例),并且这个培养物在37℃、220rpm温育两小时。将该烧瓶从保温箱中移出,添加iptg(melfordmb1008)至终浓度为1mm,然后在28℃(以及220rpm)下继续再温育三小时。
在该生长期之后,该培养物在4500xg离心10分钟,弃上清,并将沉淀在4℃重悬于2.5mltgh缓冲液(50mmhepes(ph7.5),150mmnacl,1%triton-x-100,10%甘油,1mmdtt,1片完全无edta的蛋白酶抑制剂药片(每50ml)(roche11873580001))中。然后将该细菌悬浮液进行超声(在冰上),四次十秒的脉冲以20秒的间隔隔开,然后以12000xg离心20分钟以沉淀细胞碎片。然后将清澈的超声物储存在冰上,同时据生产商说明书准备50%洗涤的谷胱甘肽琼脂糖珠(gelifesciences17-0756-01)悬浮液。随后将20μl的50%谷胱甘肽琼脂糖珠悬浮液与2.5ml来自诱饵蛋白(gst-标记的)表达细胞的蛋白提取物和2.5ml捕获蛋白(ha-/flag-/his-标记的)表达细胞的蛋白提取物结合。将该混合物在旋转轮(rotatingwheel)上于4℃温育30分钟,然后用过量的(500μl)tgh缓冲液(根据生产商说明书)洗涤谷胱甘肽琼脂糖珠五次。洗涤之后,用35μlgst洗脱缓冲液(50mmtris-甘氨酸(ph8.0),10mm还原型谷胱甘肽)在4℃超过30分钟来洗脱蛋白,然后用蛋白质印记分析进行分析。
1.2蛋白质印记法
将20%、12%或4-20%预制sds-聚丙烯酰胺凝胶(runbluenxg02012、nxg01227、nxg42027)浸没在凝胶槽(attojapanae6450)中的runbluesds-tris-tricine运行缓冲液(runbluenxb0500)中。将样品和2xlaemmli样品缓冲液(bio-radltd161-0737)混合,在恒温器(heatblock)上96℃放置10分钟,然后以10μl或20μl等份上样到凝胶的漂洗净的孔中。凝胶与3ul小份的10至250kda的pagerulerplus预染蛋白梯度(fermentas26619)在160v运行60分钟。若合适,凝胶在此阶段染色。
使用bio-radmini
转移之后,将膜于室温在50mlpbs(140mmnacl,2.7mmkcl,10mmna2hpo4,1.8mmkh2po4,ph7.3)洗涤10分钟,然后在50ml封闭液(5%(w/v)奶粉,0.1%(v/v)吐温-20)中于室温摇动一小时或于4℃摇动过夜。将一抗用封闭液稀释到它们的合适浓度(参见表2.9),并在室温下和膜(10ml每张膜,伴随轻柔的摇动)一起温育一小时,然后用50mlpbst(140mmnacl,2.7mmkcl,10mmna2hpo4,1.8mmkh2po4,0.1%(v/v)吐温-20,ph7.3)洗涤五次。如果需要二抗,需要重复染色和洗涤步骤。
洗涤的膜用镊子夹取,并小心地将一个角上放在蓝卷纸上吸干以除去过量的水分。然后将膜放在皮氏培养皿中,并用过氧化物酶底物(supersignalwestfemtomax.sensitivitysubstrate(fisherscientificpn34095))以每张膜800μl的比例进行处理。将膜放在这个底物中五分钟,如前所述吸干并放在x-射线盒中,位于一片x-射线胶片下(fujifilmx-ray18x24cm–(fujifilm497772rxno))。使用konicasrx-101桌面x-射线胶片显影仪(konica106931659)对x-射线胶片进行显影。
在分析之后,如有需要,将膜在50mlpbst中洗涤,并用10ml丽春红s溶液(ponceaussolution,sigma-aldrichp7170)染色30分钟,之后在50mlpbst中单次洗涤并在室温下干燥。
1.3种子尺寸测定
使用种子面积作为种子尺寸的替代性测量(proxymeasurement)。将种子分散在皮氏培养皿中,并使用桌面扫描仪(hewlettpackardscanjet4370)以高分辨率(<3600dpi)对白色背景进行扫描。图像存储为黑色和白色的8位(8-bit)图像,并使用imagej软件对其进行图像分析。imagej是公开的,并且设定阈值(ctrl+shift+t)以使所有种子是完全红的,然后用“矩形选区”工具选择所有的种子,并选择分析选项(分析>分析颗粒)。在对话框中,设定尺寸阈值以排除较小的(非种子)结构以及打的结构,例如种子的聚集体。种子场合和宽度通过对每颗种子拟合椭圆来计算(分析>设定测量>拟合椭圆)。当选择该选项时,分析输出对应于椭圆的长度和宽度的“主要”和“次要”值,代表种子最长和最宽的部分。[j1]
2、结果
lim结构域(prosite:ps00478)是串连的锌指结构域,充当蛋白:蛋白相互作用的平台(图1)。
基于网络的结构域预测软件(pfam、smart、prosite)预测da1(atda1170aa-230aa)中存在单个lim结构域,其被认为参与介导推定的da1-da1同源二聚化(li等,2008)。
然而意外的是,具有突变的lim结构域的da1变体(此后称为‘da1lim8’)诱导了显性负性的器官尺寸表型,等价于在拟南芥中,当向col背景中引入da-1突变(图4)。这表明da1的lim结构域并不参与da1同源二聚化。
4个关键锌配位氨基酸(c172、c175、c199和c202)被改变成甘氨酸以产生da1lim8突变体。这些突变被预测会消除锌指基序,锌指基序是由于通过半胱氨酸(c)残基模式的锌配位。
重组gst-标记的诱饵蛋白与重组flag-标记的捕获蛋白温育,然后在谷胱甘肽琼脂糖珠上沉淀gst-标签的诱饵蛋白.纯化的蛋白之后被洗脱,并进行sds-page和免疫印迹分析。使用β-葡萄糖醛酸酶(gus)形成同源四聚体的能力设计gst-gus对flag-gus的阳性对照。还使用了两组阴性对照;他们是gst-gus对flag-捕获蛋白和gst-诱饵蛋白对flag-gus。
这些体外共免疫沉淀实验表明,da1lim8能够结合野生型da1蛋白(图3),并且在col背景下的lim8种子尺寸表型与da1-1中的等价(图4)。
使用两步结构域预测分析进一步分析da1蛋白的序列。第一,执行起始同源性检测筛选(hhpred)以鉴定具有相似结构域和结构的蛋白。然后进行结构域预测筛选(pfam、smart、prosite),其用这些蛋白作为作为查询序列。这个策略揭示了atda1的230aa-297aa区域共享了其它蛋白(包括鼠lim/同源盒蛋白lhx3)的重要结构同源性。这个新的推断的结构域被称为lim类似结构域。
由于与典型lim模式显著的序列差异,da1的lim类似结构域中假定的第二对锌配位氨基酸不是通过经典的结构域预测软件(pfam、smart、prosite)检测到的。通过考虑atda1序列中位点261aa-264aa处的cxxh配对,很明显第一锌指结构域中和指内区域(inter-fingerregion)中的插入引起了序列与lim一致性模式的明显偏离。这导致了24aa的指长度和7aa的指内区域(而不是分别为16-23aa和2aa)。
因此lim类似结构域代表了da1蛋白内的含第二锌指的lim结构域。
序列表
<110>植物生物科学有限公司
<120>调节植物中的种子和器官尺寸的方法
<130>nrs/lp7065238
<150>gb1319876.7
<151>2013-11-11
<160>54
<170>patentinversion3.3
<210>1
<211>66
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim结构域
<220>
<221>variant
<222>(2)..(3)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(27)
<223>在位点5至27的xaa是任意氨基酸并且它们中多达七个可以缺失;代表16-23个氨基酸的范围。
<220>
<221>variant
<222>(28)..(28)
<223>xaa是his或cys
<220>
<221>variant
<222>(29)..(32)
<223>在位点29至32的xaa是任意氨基酸并且它们中任意两个可以缺失;代表2或4个氨基酸的串。
<220>
<221>variant
<222>(33)..(33)
<223>xaa是his、cys或glu
<220>
<221>variant
<222>(34)..(35)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(37)..(38)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(40)..(60)
<223>在位点40至60的xaa是任意氨基酸并且它们中多达七个可以缺失;代表14-21个氨基酸的范围。
<220>
<221>variant
<222>(61)..(61)
<223>xaa是his或cys
<220>
<221>variant
<222>(62)..(64)
<223>在位点62-64的xaa是任意氨基酸并且它们中一个或两个可以缺失;代表2或1或3个氨基酸。
<220>
<221>variant
<222>(65)..(65)
<223>xaa是cys、his、asp或glu
<220>
<221>variant
<222>(66)..(66)
<223>xaa是任意氨基酸
<400>1
cysxaaxaacysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
151015
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
202530
xaaxaaxaacysxaaxaacysxaaxaaxaaxaaxaaxaaxaaxaaxaa
354045
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
505560
xaaxaa
65
<210>2
<211>63
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim结构域
<220>
<221>variant
<222>(2)..(3)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(27)
<223>在位点5-27的xaa是任意氨基酸并且它们中多达七个可以缺失;代表16-23个氨基酸的范围。
<220>
<221>variant
<222>(29)..(30)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(32)..(33)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(35)..(36)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(38)..(58)
<223>在位点38至58的xaa是任意氨基酸并且它们中多达七个可以缺失;代表14-21个氨基酸的范围。
<220>
<221>variant
<222>(60)..(61)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(63)..(63)
<223>xaa是任意氨基酸
<400>2
cysxaaxaacysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
151015
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaahisxaaxaacysxaa
202530
xaacysxaaxaacysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
354045
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaahisxaaxaacysxaa
505560
<210>3
<211>53
<212>prt
<213>arabidopsisthaliana
<400>3
cysalaglycysasnmetgluileglyhisglyargpheleuasncys
151015
leuasnserleutrphisproglucyspheargcystyrglycysser
202530
glnproileserglutyrglupheserthrserglyasntyrprophe
354045
hislysalacystyr
50
<210>4
<211>504
<212>prt
<213>setariaitalica
<400>4
metglytrpleuserlysilephelysglyservalasnargvalser
151015
argglyhistyrasnglyasnserhisgluglytyrserthrglnhis
202530
thrlyssertyrglyalahisglyasngluaspgluaspmetasphis
354045
alailealaleuserleusergluglnaspglnarglysglylysala
505560
ileaspthrgluhishisleuaspgluaspgluglnleualaargala
65707580
leuglngluasnthrserprothrleuaspgluaspgluglnleuala
859095
argalaleuglnglusermetasnaspgluhisproproargglnhis
100105110
ileproilegluaspvalhissergluseralaproalaserserleu
115120125
proprotyrvalpheprothrasnglyserargvalcysalaglycys
130135140
lysthrproileglyglnglyargpheleusercysmetaspserval
145150155160
trphisproglncyspheargcystyrglycysaspileproileser
165170175
glutyrgluphealavalhisgluasphisalatyrhisargsercys
180185190
tyrlysgluargphehisprolyscysaspvalcysasnserpheile
195200205
prothrasnlysasnglyleuileglutyrargalahisprophetrp
210215220
metglnlystyrcysproserhisgluasnaspglythrproargcys
225230235240
cyssercysgluargmetgluprolyshisserglntyrilethrleu
245250255
aspaspglyargargleucysleuglucysleuhisthralailemet
260265270
aspthrasnglucysglnproleutyrileaspileglngluphetyr
275280285
gluglymetasnmetlysvalgluglnglnvalproleuleuleuval
290295300
gluargglnalaleuasnglualametglualaglulysileglyhis
305310315320
hisleuprogluthrargglyleucysleuserglugluglnileval
325330335
argthrileleuargargproileileglyproglyasnargileile
340345350
aspmetilethrglyprotyrlysleuvalargargcysgluvalthr
355360365
alaileleuileleutyrglyleuproargleuleuthrglyserile
370375380
leualahisglumetmethisalatyrleuargleulysglytyrarg
385390395400
thrleuserprogluvalglugluglyilecysglnvalleualahis
405410415
leutrpleuglusergluilethrserglyserglysermetalathr
420425430
thrseralaalaserserserserserthrserserserserlyslys
435440445
glyalalysthrglupheglulysargleuglygluphephelyshis
450455460
glnilegluthraspproservalalatyrglyaspglypheargala
465470475480
glymetargalavalgluargtyrglyleuargserthrleuasphis
485490495
ilelysleuthrglyserphepro
500
<210>5
<211>487
<212>prt
<213>brachypodiumdistachyon
<400>5
metglytrpleuasnlysilephelysglyservalasnargvalser
151015
argglyasntyraspglyasntrphisaspglyasnsersergluasn
202530
ileargglyalatyraspgluseraspasngluaspileaspargala
354045
ilealaleuserleualaglugluaspproasnlysglylysalaile
505560
ileaspproasptyrserleuglugluaspgluglnleualaargala
65707580
leuhisgluserleuasnthrglyserproprohisglnasnvalpro
859095
valvalaspvalprosergluargvalprothrargglupropropro
100105110
provalpheleuserserglypheargalacysalaglycysasnasn
115120125
proileglyasnglyargpheleusercysmetaspservaltrphis
130135140
proglncyspheargcysphealacysasnlysproileserglutyr
145150155160
gluphealamethisgluasnglnprotyrhislyssercystyrlys
165170175
aspphephehisprolyscysaspvalcyslysasppheileprothr
180185190
asnlysaspglyleuileglutyrargalahisprophetrpmetgln
195200205
lystyrcysproserhisgluaspaspglythrproargcyscysser
210215220
cysgluargmetgluprothraspilelystyrileargleuaspasp
225230235240
glyarglysleucysleuglucysleuthrseralathrmetaspser
245250255
proglucysglnhisleutyrmetaspileglngluphepheglugly
260265270
leuasnmetlysvalgluglnglnvalproleuleuleuvalgluarg
275280285
glnalaleuasnglualaleuglualaglulysserglyhishisleu
290295300
progluthrargglyleucysleuserglugluglnilevalargthr
305310315320
ileleuargargprothrileglyproglyasnargileileaspmet
325330335
ilethrglyprotyrlysleuvalargargcysgluvalthralaile
340345350
leuileleutyrglyleuproargleuglnthrglyserileleuala
355360365
hisglumetmethisalatyrleuargleulysglytyrargserleu
370375380
serproglnvalglugluglyilecysglnvalleuserhismettrp
385390395400
leuglusergluileilealaglyalaserglyasnthralaserthr
405410415
servalproserserserseralaprothrserserlyslysglyala
420425430
lysthrglupheglulysargleuglyalapheilelysasnglnile
435440445
gluthraspserservalglutyrglyaspglypheargalaglyasn
450455460
argalavalgluargtyrglyleuargserthrleuasphismetlys
465470475480
ilethrglyserpheprotyr
485
<210>6
<211>514
<212>prt
<213>brassicarapa
<400>6
metglytrpleuasnlysilephelysglyserasnglnarghispro
151015
leuglyasngluhistyrhishisasnglyglytyrtyrgluasntyr
202530
prohisgluhissergluproseralagluthraspalaasphisthr
354045
glngluproserthrsergluglugluthrtrpasnglylysgluasn
505560
glugluvalaspargvalilealaleuserileleugluglugluasn
65707580
glnargprogluthrasnthrglyalatrplyshisalametmetasp
859095
aspaspgluglnleualaargalaileglnglusermetilealaarg
100105110
asnglythrthrtyrasppheglyasnalatyrglyasnglyhismet
115120125
hisglyglyglyasnvaltyraspasnglyaspiletyrtyrproarg
130135140
proilealaphesermetasppheargilecysalaglycysasnmet
145150155160
gluileglyhisglyargtyrleuasncysleuasnalaleutrphis
165170175
proglncyspheargcystyrglycysserhisproileserglutyr
180185190
glupheserthrserglyasntyrprophehislysalacystyrarg
195200205
gluargphehisprolyscysaspvalcysserleupheileserthr
210215220
asnhisalaglyleuileglutyrargalahisprophetrpvalgln
225230235240
lystyrcysproserhisgluhisaspalathrproargcyscysser
245250255
cysgluargmetgluproargasnthrglytyrphegluleuasnasp
260265270
glyarglysleucysleuglucysleuaspserservalmetaspthr
275280285
pheglncysglnproleutyrleuglnileglngluphetyrglugly
290295300
leuasnmetthrvalgluglngluvalproleuleuleuvalgluarg
305310315320
glnalaleuasnglualaarggluglygluargasnglyhistyrhis
325330335
metprogluthrargglyleucysleuserglugluglnthrvalarg
340345350
thrvalarglysargserlysglyasntrpserglyasnmetilethr
355360365
gluglnphelysleuthrargargcysgluvalthralaileleuile
370375380
leupheglyleuproargleuleuthrglyserileleualahisglu
385390395400
metmethisalatrpmetargleulysglypheargproleusergln
405410415
aspvalglugluglyilecysglnvalmetalahislystrpleuglu
420425430
alagluleualaalaglyserargasnserasnalaalaserserser
435440445
sersersertyrglyglyvallyslysglyproargserglntyrglu
450455460
arglysleuglygluphephelyshisglnilegluseraspalaser
465470475480
provaltyrglyaspglypheargalaglyargleualavalasnlys
485490495
tyrglyleutrpargthrleugluhisileglnmetthrglyargphe
500505510
proval
<210>7
<211>532
<212>prt
<213>brassicarapa
<400>7
metglytrppheasnlysilephelysglyserthrglnargphearg
151015
leuglyasnasphisasphisasnglytyrtyrglnsertyrprohis
202530
aspgluproseralaaspthraspproaspproaspproaspproasp
354045
gluthrhisthrglngluproserthrserglugluaspthrsergly
505560
glngluasngluaspileaspargalailealaleuserleuileglu
65707580
asnserglnglyglnthrasnasnthrcysalaalaasnalaglylys
859095
tyralametvalaspgluaspgluglnleualaargalaileglnglu
100105110
sermetvalvalglyasnthrproargglnlyshisglysersertyr
115120125
aspileglyasnalatyrglyalaglyaspvaltyrglyasnglyhis
130135140
methisglyglyglyasnvaltyralaasnglyaspiletyrtyrpro
145150155160
argprothralapheprometasppheargilecysalaglycysasn
165170175
metgluileglyhisglyargtyrleuasncysleuasnalaleutrp
180185190
hisproglucyspheargcystyrglycysarghisproileserglu
195200205
tyrglupheserthrserglyasntyrprophehislysalacystyr
210215220
arggluargtyrhisprolyscysaspvalcysserleupheilepro
225230235240
thrasnhisalaglyleuileglytyrargalahisprophetrpval
245250255
glnlystyrcysproserhisgluhisaspalathrproargcyscys
260265270
sercysgluargmetgluproargasnthrglytyrvalgluleuasn
275280285
aspglyarglysleucysleuglucysleuaspseralavalmetasp
290295300
thrpheglncysglnproleutyrleuglnileglngluphetyrglu
305310315320
glyleuphemetlysvalgluglnaspvalproleuleuleuvalglu
325330335
argglnalaleuasnglualaarggluglyglulysasnglyhistyr
340345350
hismetprogluthrargglyleucysleuserglugluglnthrval
355360365
serthrvalarglysargserlyshisglythrglyasntrpalagly
370375380
asnmetilethrgluprotyrlysleuthrargglncysgluvalthr
385390395400
alaileleuileleupheglyleuproargleuleuthrglyserile
405410415
leualahisglumetmethisalatrpmetargleulysglyphearg
420425430
thrleuserglnaspvalglugluglyilecysglnvalmetalahis
435440445
lystrpleuglualagluleualaalaglyserargasnserasnval
450455460
alaserserserserserargglyvallyslysglyproargsergln
465470475480
tyrgluarglysleuglygluphephelyshisglnilegluserasp
485490495
alaserprovaltyrglyaspglypheargalaglyargleualaval
500505510
asnlystyrglyleuprolysthrleugluhisileglnmetthrgly
515520525
argpheproval
530
<210>8
<211>532
<212>prt
<213>arabidopsisthaliana
<400>8
metglytrppheasnlysilephelysglyserasnglnargleuarg
151015
valglyasnasnlyshisasnhisasnvaltyrtyraspasntyrpro
202530
thralaserhisaspaspgluproseralaalaaspthraspalaasp
354045
asnaspgluprohishisthrglngluproserthrsergluaspasn
505560
thrserasnaspglngluasngluaspileaspargalailealaleu
65707580
serleuleuglugluasnglngluglnthrserileserglylystyr
859095
sermetprovalaspgluaspgluglnleualaargalaleuglnglu
100105110
sermetvalvalglyasnserproarghislysserglyserthrtyr
115120125
aspasnglyasnalatyrglyalaglyaspleutyrglyasnglyhis
130135140
mettyrglyglyglyasnvaltyralaasnglyaspiletyrtyrpro
145150155160
argproilethrpheglnmetasppheargilecysalaglycysasn
165170175
metgluileglyhisglyargpheleuasncysleuasnserleutrp
180185190
hisproglucyspheargcystyrglycysserglnproileserglu
195200205
tyrglupheserthrserglyasntyrprophehislysalacystyr
210215220
arggluargtyrhisprolyscysaspvalcysserhispheilepro
225230235240
thrasnhisalaglyleuileglutyrargalahisprophetrpval
245250255
glnlystyrcysproserhisgluhisaspalathrproargcyscys
260265270
sercysgluargmetgluproargasnthrargtyrvalgluleuasn
275280285
aspglyarglysleucysleuglucysleuaspseralavalmetasp
290295300
thrmetglncysglnproleutyrleuglnileglnasnphetyrglu
305310315320
glyleuasnmetlysvalgluglngluvalproleuleuleuvalglu
325330335
argglnalaleuasnglualaarggluglyglulysasnglyhistyr
340345350
hismetprogluthrargglyleucysleuserglugluglnthrval
355360365
serthrvalarglysargserlyshisglythrglylystrpalagly
370375380
asnilethrgluprotyrlysleuthrargglncysgluvalthrala
385390395400
ileleuileleupheglyleuproargleuleuthrglyserileleu
405410415
alahisglumetmethisalatrpmetargleulysglypheargthr
420425430
leuserglnaspvalglugluglyilecysglnvalmetalahislys
435440445
trpleuaspalagluleualaalaglyserthrasnserasnalaala
450455460
serserserserserserglnglyleulyslysglyproargsergln
465470475480
tyrgluarglysleuglygluphephelyshisglnilegluserasp
485490495
alaserprovaltyrglyaspglypheargalaglyargleualaval
500505510
hislystyrglyleuarglysthrleugluhisileglnmetthrgly
515520525
argpheproval
530
<210>9
<211>478
<212>prt
<213>theobromacacao
<400>9
metasptrpilelyslysilephelysglycysalahislyspheser
151015
gluglyhishishisglyasntyrvalgluaspprohisproglnphe
202530
asnalaproservalserglyaspalatrpglngluleugluasnglu
354045
aspvalaspargalailealaleuserleuleuglygluserglnlys
505560
glyarglysvalileaspaspglutyrglnleuglugluaspglugln
65707580
leualaargalaleuglngluserleuasnphegluproproprogln
859095
tyrgluasnalaasnmettyrglnprometprovalhisphepromet
100105110
glytyrargilecysalaglycysasnthrgluileglyhisglyarg
115120125
pheleuasncysleuasnalaphetrphisproglucyspheargcys
130135140
hisalacysasnleuproileserasptyrgluphesermetsergly
145150155160
asntyrargphehislyssercystyrlysgluargtyrhisprolys
165170175
cysaspvalcysasnasppheileprothrasnproalaglyleuile
180185190
glutyrargalahisprophetrpileglnlystyrcysproserhis
195200205
gluhisaspserthrproargcyscyssercysgluargmetglupro
210215220
glnaspthrglytyrvalalaleuasnaspglyarglysleucysleu
225230235240
glucysleuaspseralavalmetaspthrlysglncysglnproleu
245250255
tyrleuaspileleugluphetyrgluglyleuasnmetlysvalglu
260265270
glnglnvalproleuleuleuvalgluargglnalaleuasngluala
275280285
arggluglyglulysasnglyhistyrhismetprogluthrarggly
290295300
leucysleuserglugluglnthrvalserthrileleuargglnpro
305310315320
argpheglythrglyasnargalametaspmetilethrgluprocys
325330335
lysleuthrargargcysgluvalthralaileleuileleutyrgly
340345350
leuproargleuleuthrglyserileleualahisglumetmethis
355360365
alatrpmetargleuglnglypheargthrleuserglnaspvalglu
370375380
gluglyilecysglnvalleualahismettrpleuleuthrglnleu
385390395400
glutyralaserserserasnvalalaseralaserserseralaser
405410415
serargleuglnlysglylysargproglnphegluglylysleugly
420425430
gluphephelyshisglnilegluseraspthrserprovaltyrgly
435440445
aspglypheargalaglyhisglnalavaltyrlystyrglyleuarg
450455460
argthrleugluhisileargmetthrglyargpheprotyr
465470475
<210>10
<211>474
<212>prt
<213>glycinemax
<400>10
metglytrpleuserargilephelysglyserasphisasnlysleu
151015
sergluglyhistyrtyrlysgluaspalaglytyrtyrleuproser
202530
thrserglyvalthrasnasnglnasngluasngluaspileasparg
354045
alailealaleuserleuvalglugluserargargalaasnasnasn
505560
valasnglygluargileleuserleuglnthrleuleuglugluasp
65707580
gluglnleualaargalailegluglnserleuasnleugluserpro
859095
proargtyrglyasngluasnmettyrglnproproileglntyrphe
100105110
proleuglyilecysalaglycystyrthrgluileglypheglyarg
115120125
tyrleuasncysleuasnalaphetrphisproglucyspheargcys
130135140
argalacysasnleuproileserasptyrglupheserthrsergly
145150155160
asntyrprotyrhislyssercystyrlysglusertyrhisprolys
165170175
cysaspvalcyslyshispheileprothrasnproalaglyleuile
180185190
glutyrargalahisprophetrpileglnlystyrcysprothrhis
195200205
gluhisaspglythrproargcyscyssercysgluargmetgluser
210215220
glnglualaglytyrilealaleulysaspglyarglysleucysleu
225230235240
glucysleuaspserserilemetaspthrasnglucysglnproleu
245250255
hisalaaspileglnargphetyraspserleuasnmetlysleuasp
260265270
glnglnileproleuleuleuvalgluargglnalaleuasngluala
275280285
arggluglyglulysasnglyhistyrhismetprogluthrarggly
290295300
leucysleuserglugluleuserthrpheserargargproargleu
305310315320
glythralametaspmetargalaglnprotyrargprothrthrarg
325330335
cysaspvalthralaileleuvalleutyrglyleuproargleuleu
340345350
thrglyserileleualahisglumetmethisalatrpleuargleu
355360365
lysglytyrargthrleuserglnaspvalglugluglyilecysgln
370375380
valleualahismettrpleuglusergluleuserseralasergly
385390395400
serasnphevalseralaserserserseralaserhisthrserarg
405410415
lysglylysargproglnphegluarglysleuglygluphephelys
420425430
hisglnilegluseraspileserprovaltyrglyaspglyphearg
435440445
alaglyglnlysalavalarglystyrglyleuglnargthrleuhis
450455460
hisileargmetthrglythrpheprotyr
465470
<210>11
<211>478
<212>prt
<213>glycinemax
<400>11
metglytrpleuserargilephelysglyserasphisasnlysleu
151015
sergluglyhistyrtyrlysgluaspalaglytyrtyrleuproser
202530
thrserglyvalthrasnaspalatrpasnglnserglnasnglnasn
354045
gluasngluaspileaspargalailealaleuserleuvalgluglu
505560
thrglnlysalaasnasnasnvalasnasptyrargserglnleuglu
65707580
gluaspgluglnleualaargalailegluglnserleuasnleuglu
859095
serproproargtyrglyasngluasnmettyrglnproproilegln
100105110
tyrpheprometglyserargilecysalaglycystyrthrgluile
115120125
glytyrglyargtyrleuasncysleuasnalaphetrphisproglu
130135140
cyspheargcysargalacysasnleuproileserasptyrgluphe
145150155160
serthrserglyasntyrprotyrhislyssercystyrlysgluser
165170175
tyrhisprolyscysaspvalcyslyshispheileprothrasnpro
180185190
alaglyleuileglutyrargalahisprophetrpileglnlystyr
195200205
cysprothrhisgluhisaspglythrthrargcyscyssercysglu
210215220
argmetgluserglnglualaglytyrilealaleulysaspglyarg
225230235240
lysleucysleuglucysleuaspseralailemetaspthrasnglu
245250255
cysglnproleuhisalaaspileglnargphetyrgluserleuasn
260265270
metlysleuaspglnglnileproleuleuleuvalgluargglnala
275280285
leuasnglualaarggluglyglulysasnglyhistyrhismetpro
290295300
gluthrargglyleucysleuserglugluleuserthrpheserarg
305310315320
argproargleuglythrthrmetaspmetargalaglnprotyrarg
325330335
prothrthrargcysaspvalthralaileleuileleutyrglyleu
340345350
proargleuleuthrglyserileleualahisglumetmethisala
355360365
trpleuargleulysglytyrargthrleuserglnaspvalgluglu
370375380
glyilecysglnvalleuserhismettrpleuglusergluleuser
385390395400
seralaserglyserasnphevalseralaserserserseralaser
405410415
histhrserarglysglylysargproglnphegluarglysleugly
420425430
gluphephelyshisglnilegluseraspileserprovaltyrgly
435440445
glyglypheargalaglyglnlysalavalserlystyrglyleugln
450455460
argthrleuhishisileargmetthrglythrpheprotyr
465470475
<210>12
<211>462
<212>prt
<213>vitisvinifera
<400>12
metglytrpleuasnlysilephelysglyserserhislysileser
151015
gluglyasntyrhisglyargtyrglnglyaspthrvalglnasnglu
202530
prosercysserglyaspvaltrpalagluthrgluasngluaspile
354045
aspargalailealaleuserleusergluglugluglnlysglylys
505560
lysvalileaspasnglupheglnleuglugluaspgluglnleuala
65707580
argalaileglngluserleuasnilegluserproproglnhisgly
859095
asnglyasnglyasnglyasniletyrglnproilepropheprotyr
100105110
serthrglypheargilecysalaglycysasnthrgluileglyhis
115120125
glyargpheleusercysmetglyalavaltrphisproglucysphe
130135140
argcyshisglycysglytyrproileserasptyrglutyrsermet
145150155160
asnglyasntyrprotyrhislyssercystyrlysgluhistyrhis
165170175
prolyscysaspvalcyslyshispheileprothrasnproalagly
180185190
leuileglutyrargalahisprophetrpvalglnlystyrcyspro
195200205
serhisgluhisaspargthrproargcyscyssercysgluargmet
210215220
gluproargaspthrargtyrvalalaleuasnaspglyarglysleu
225230235240
cysleuglucysleuaspseralailemetaspthrasnglucysgln
245250255
proleutyrleuaspileglngluphetyrgluglyleuasnmetlys
260265270
valglnglnglnvalproleuleuleuvalgluargglnalaleuasn
275280285
glualametgluglyglulysserglyhishishismetprogluthr
290295300
argglyleucysleuserglugluglnthrvalserthrileleuarg
305310315320
argprolysileglythrglyasnargvalmetasnmetilethrglu
325330335
procyslysleuthrargargcysaspvalthralavalleuileleu
340345350
tyrglyleuproargleuleuthrglyserileleualahisglumet
355360365
methisalatrpleuargleuasnglytyrargthrleualaglnasp
370375380
valglugluglyilecysglnvalleualatyrmettrpleuaspala
385390395400
gluleuthrserglyserglyargserglncysgluarglysleugly
405410415
glnphephelyshisglnilegluseraspthrserleuvaltyrgly
420425430
alaglypheargalaglyhisglnalavalleulystyrglyleupro
435440445
alathrleulyshisilehisleuthrglyasnpheprotyr
450455460
<210>13
<211>482
<212>prt
<213>vitisvinifera
<400>13
metglytrpleuasnlysilephelysglyserserhislysileser
151015
gluglyasntyrhisglyargtyrglnglyaspthrvalglnasnglu
202530
prosercysserglyaspvaltrpalagluthrgluasngluaspile
354045
aspargalailealaleuserleusergluglugluglnlysglylys
505560
lysvalileaspgluleuaspasnglupheglnleuglugluaspglu
65707580
glnleualaargalaileglngluserleuasnilegluserpropro
859095
glnhisglyasnglyasnglyasnglyasniletyrglnproilepro
100105110
pheprotyrserthrglypheargilecysalaglycysasnthrglu
115120125
ileglyhisglyargpheleusercysmetglyalavaltrphispro
130135140
glucyspheargcyshisglycysglytyrproileserasptyrglu
145150155160
tyrsermetasnglyasntyrprotyrhislyssercystyrlysglu
165170175
histyrhisprolyscysaspvalcyslyshispheileprothrasn
180185190
proalaglyleuileglutyrargalahisprophetrpvalglnlys
195200205
tyrcysproserhisgluhisaspargthrproargcyscyssercys
210215220
gluargmetgluproargaspthrargtyrvalalaleuasnaspgly
225230235240
arglysleucysleuglucysleuaspseralailemetaspthrasn
245250255
glucysglnproleutyrleuaspileglngluphetyrgluglyleu
260265270
asnmetlysvalglnglnglnvalproleuleuleuvalgluarggln
275280285
alaleuasnglualametgluglyglulysserglyhishishismet
290295300
progluthrargglyleucysleuserglugluglnthrvalserthr
305310315320
ileleuargargprolysileglythrglyasnargvalmetasnmet
325330335
ilethrgluprocyslysleuthrargargcysaspvalthralaval
340345350
leuileleutyrglyleuproargleuleuthrglyserileleuala
355360365
hisglumetmethisalatrpleuargleuasnglytyrargthrleu
370375380
alaglnaspvalglugluglyilecysglnvalleualatyrmettrp
385390395400
leuaspalagluleuthrserglyserglyserasnvalproserthr
405410415
serseralaserthrserserlyslysglyalaglyserglncysglu
420425430
arglysleuglyglnphephelyshisglnilegluseraspthrser
435440445
leuvaltyrglyalaglypheargalaglyhisglnalavalleulys
450455460
tyrglyleuproalathrleulyshisilehisleuthrglyasnphe
465470475480
protyr
<210>14
<211>486
<212>prt
<213>solanumlycopersicum
<400>14
metglytrpleuasnlysilepheargglyserserhislysileser
151015
gluglyglntyrasptrpargcysgluglyhisthrglugluaspasp
202530
proserthralagluaspsertrpsergluileglugluileasparg
354045
alailealaileserleusergluglugluglnlysglylysileval
505560
ileaspsergluserglnleulysgluaspgluglnleualaargala
65707580
leuglngluserleuasnvalgluserproproglnhisvalserarg
859095
asnasphisglyglyglyasnvaltyrglyasnglyasnphetyrhis
100105110
provalpropheprotyrseralaserpheargvalcysalaglycys
115120125
serthrgluileglyhisglyargpheleusercysmetglyalaval
130135140
trphisproglucyspheargcyshisalacysasnglnproileser
145150155160
asptyrgluphesermetserglyasntyrprotyrhislysthrcys
165170175
tyrlysgluhistyrhisprolyscysaspvalcyslyshispheile
180185190
prothrasnalaalaglyleuileglutyrargalahisprophetrp
195200205
serglnlystyrcysprophehisgluhisaspglythrproargcys
210215220
cyssercysgluargmetgluproargaspthrargtyrilealaleu
225230235240
aspaspglyarglysleucysleuglucysleuaspseralailemet
245250255
aspthrserglncysglnproleutyrtyraspileglngluphetyr
260265270
gluglyleuasnmetlysvalgluglnlysvalproleuleuleuval
275280285
gluargglnalaleuasnglualametaspglygluarghisglytyr
290295300
hishismetprogluthrargglyleucysleuserglugluglnthr
305310315320
ileserthrileglnargargproargileglyalaglyasnargval
325330335
metaspmetargthrgluprotyrlysleuthrargargcysgluval
340345350
thralaileleuileleutyrglyleuproargleuleuthrglyser
355360365
ileleualahisglumetmethisalatrpleuargleuargglytyr
370375380
argthrleuserglnaspvalglugluglyilecysglnvalleuala
385390395400
hismettrpleugluthrglnilealaserileserserserasngly
405410415
glyalaserthrserserglymetserserserlysglnglyilearg
420425430
serprophegluarglysleuglyaspphephelyshisglnileglu
435440445
seraspthrserproiletyrglyasnglypheargalaglyasngln
450455460
alavalleulystyrglyleugluargthrleuasphisileargmet
465470475480
thrglythrpheprotyr
485
<210>15
<211>878
<212>prt
<213>oryzasativa
<400>15
metglyaspargproaspmetglyalaglyvalalaleuargpheser
151015
hisasnasptrpthrleuglugluaspserlysalaleuhispheleu
202530
glnproaspleuvalleuphethrglyasptyrglyasngluasnval
354045
glnleuvallysserileseraspleuglnleuprolysalaalaile
505560
leuglyasnhisaspcystrphisthrtyrglnpheserglulyslys
65707580
valaspargvalglnleuglnleugluserleuglygluglnhisval
859095
glytyrlyscysleuasppheprothrilelysleuservalvalgly
100105110
glyargprophesercysglyglyasnargilepheargprolysleu
115120125
leuserlystrptyrglyvalasnaspmetalagluseralalysarg
130135140
iletyraspalaalathrasnalaprolysgluhisalavalileleu
145150155160
leualahisasnglyprothrglyleuglyserargmetgluaspile
165170175
cysglyargasptrpvalalaglyglyglyasphisglyaspproasp
180185190
leugluglnalaileseraspleuglnarggluthrglyvalserile
195200205
proleuvalvalpheglyhismethislysserleualatyrglyarg
210215220
glyleuarglysmetilealapheglyalaasnargthriletyrleu
225230235240
asnglyalavalvalproargvalasnhisalaglnserserarggln
245250255
proalaileserthrserglulysthrglyleugluglyleuthrgly
260265270
leumetvalprothrserargalaphethrilevalaspleupheglu
275280285
glyalavalglulysilesergluvaltrpvalthrvalglyaspala
290295300
argthrgluleugluglngluleuvalleutyrlysglnprohislys
305310315320
servalproserasnilealailetrpserthrmetglytrpleuthr
325330335
lysphepheargglyserthrhislysilesergluglyglntyrhis
340345350
serlysproalaglugluthriletrpasnglyproserasnserala
355360365
valvalthrmetvaltyrproleugluserthrpheglyglnleuasp
370375380
leuleuleuleualathraspleuargglnleuvalileaspaspval
385390395400
aspcyscyslysleuargglnglnalaglnprovalleuhisleumet
405410415
tyrserglnleuglnleuleuglnthrserhisalahisglnhisgly
420425430
aspvalproserglupheaspasngluaspilealaargalaileser
435440445
leuserleuleugluglugluglnarglysalalysalaileglulys
450455460
aspmethisleuglugluaspgluglnleualaargalaileglnglu
465470475480
serleuasnvalgluserproproargalaarggluasnglyasnala
485490495
asnglyglyasnmettyrglnproleuprophemetphesersergly
500505510
pheargthrcysalaglycyshissergluileglyhisglyargphe
515520525
leusercysmetglyalavaltrphisproglucyspheargcyshis
530535540
alacysasnglnproiletyrasptyrgluphesermetserglyasn
545550555560
hisprotyrhislysthrcystyrlysgluargphehisprolyscys
565570575
aspvalcyslysglnpheileprothrasnmetasnglyleuileglu
580585590
tyrargalahisprophetrpleuglnlystyrcysproserhisglu
595600605
valaspglythrproargcyscyssercysgluargmetgluproarg
610615620
gluserargtyrvalleuleuaspaspglyarglysleucysleuglu
625630635640
cysleuaspseralavalmetaspthrserglucysglnproleutyr
645650655
leugluileglngluphetyrgluglyleuasnmetlysvalglugln
660665670
glnvalproleuleuleuvalgluargglnalaleuasnglualamet
675680685
gluglyglulysthrglyhishishisleuprogluthrargglyleu
690695700
cysleuserglugluglnthrvalserthrileleuargargproarg
705710715720
metalaglyasnlysvalmetglumetilethrgluprotyrargleu
725730735
thrargargcysgluvalthralaileleuileleutyrglyleupro
740745750
argleuleuthrglyserileleualahisglumetmethisalatrp
755760765
leuargleulysglytyrargthrleuserproaspvalgluglugly
770775780
ilecysglnvalleualahismettrpileglusergluileileala
785790795800
glyserglyserasnglyalaserthrserserserserseralaser
805810815
thrserserlyslysglyglyargserglnphegluarglysleugly
820825830
aspphephelyshisglnilegluseraspthrsermetalatyrgly
835840845
aspglypheargalaglyasnargalavalleuglntyrglyleulys
850855860
argthrleugluhisileargleuthrglythrpheprophe
865870875
<210>16
<211>486
<212>prt
<213>oryzasativa
<400>16
metglytrpleuthrlysphepheargglyserthrhislysileser
151015
gluglyglntyrhisserlysproalaglugluthriletrpasngly
202530
proserasnseralavalvalthraspvalproserglupheaspasn
354045
gluaspilealaargalaileserleuserleuleuglugluglugln
505560
arglysalalysalaileglulysaspmethisleuglugluaspglu
65707580
glnleualaargalaileglngluserleuasnvalgluserpropro
859095
argalaarggluasnglyasnalaasnglyglyasnmettyrglnpro
100105110
leuprophemetpheserserglypheargthrcysalaglycyshis
115120125
sergluileglyhisglyargpheleusercysmetglyalavaltrp
130135140
hisproglucyspheargcyshisalacysasnglnproiletyrasp
145150155160
tyrgluphesermetserglyasnhisprotyrhislysthrcystyr
165170175
lysgluargphehisprolyscysaspvalcyslysglnpheilepro
180185190
thrasnmetasnglyleuileglutyrargalahisprophetrpleu
195200205
glnlystyrcysproserhisgluvalaspglythrproargcyscys
210215220
sercysgluargmetgluproarggluserargtyrvalleuleuasp
225230235240
aspglyarglysleucysleuglucysleuaspseralavalmetasp
245250255
thrserglucysglnproleutyrleugluileglngluphetyrglu
260265270
glyleuasnmetlysvalgluglnglnvalproleuleuleuvalglu
275280285
argglnalaleuasnglualametgluglyglulysthrglyhishis
290295300
hisleuprogluthrargglyleucysleuserglugluglnthrval
305310315320
serthrileleuargargproargmetalaglyasnlysvalmetglu
325330335
metilethrgluprotyrargleuthrargargcysgluvalthrala
340345350
ileleuileleutyrglyleuproargleuleuthrglyserileleu
355360365
alahisglumetmethisalatrpleuargleulysglytyrargthr
370375380
leuserproaspvalglugluglyilecysglnvalleualahismet
385390395400
trpileglusergluileilealaglyserglyserasnglyalaser
405410415
thrserserserserseralaserthrserserlyslysglyglyarg
420425430
serglnphegluarglysleuglyaspphephelyshisglnileglu
435440445
seraspthrsermetalatyrglyaspglypheargalaglyasnarg
450455460
alavalleuglntyrglyleulysargthrleugluhisileargleu
465470475480
thrglythrpheprophe
485
<210>17
<211>512
<212>prt
<213>brachypodiumdistachyon
<400>17
metglytrpleuthrlysilepheargglyserthrtyrlysileser
151015
gluglyglnargglnserargproalagluglualavaltrpasnglu
202530
proserserserthrvalvalthraspvalleuserglupheaspasn
354045
gluaspileaspargalailealaleuserleuserglugluglnarg
505560
lysserlysglythrglylysaspleuhisleuaspgluaspglugln
65707580
leualaargalailehisgluserleuasnvalgluserproprocys
859095
alaargaspasnglyserproprohisalaargaspasnserserpro
100105110
prohisalaarggluasnserserhisproargalaarggluasngly
115120125
ilealaasnglyglyasnserileglnhisserprophemetpheser
130135140
serglypheargthrcysalaglycyshissergluileglyhisgly
145150155160
argpheleusercysmetglyalavaltrphisproglucysphecys
165170175
cyshisalacysserglnproiletyrasptyrgluphesermetser
180185190
glyasnhisprotyrhislysthrcystyrlysgluargphehispro
195200205
lyscysaspvalcyslysglnpheileprothrasnmetasnglyleu
210215220
ileglutyrargalahisprophetrpleuglnlystyrcysproser
225230235240
hisgluvalaspglythrproargcyscyssercysgluargmetglu
245250255
proarggluserargtyrvalleuleuaspaspglyarglysleucys
260265270
leuglucysleuaspseralavalmetaspthrthrglucysglnpro
275280285
leutyrleugluileglngluphetyrgluglyleuasnmetlysval
290295300
gluglnglnvalproleuleuleuvalgluargglnalaleuasnglu
305310315320
alametgluglyglulysthrglyhishishisleuprogluthrarg
325330335
glyleucysleuserglugluglnthrvalserthrileleuargarg
340345350
proargmetthrglyasnlysilemetglumetilethrgluprotyr
355360365
argleuthrargargcysgluvalthralaileleuileleutyrgly
370375380
leuproargleuleuthrglyserileleualahisglumetmethis
385390395400
alatrpleuargleulysglytyrargthrleuserprogluileglu
405410415
gluglyilecysglnvalleualahismettrpileglusergluile
420425430
metalaglyserserserasnalaalaserthrserserserserser
435440445
serserileserserlyslysglyglyargserglnphegluarglys
450455460
leuglyaspphephelyshisglnilegluseraspthrservalala
465470475480
tyrglyasnglypheargserglyasnglnalavalleuglntyrgly
485490495
leulysargthrleugluhisiletrpleuthrglythrtrpprophe
500505510
<210>18
<211>491
<212>prt
<213>brachypodiumdistachyon
<400>18
metglytrpleuthrlysphepheargglyserthrhisasnileser
151015
gluglyglnaspglnserlysproalaglugluthrvaltrpasnglu
202530
proserserserthralavalasntyralaleuserglupheaspasn
354045
gluaspileaspargalailealaleuserleuserglugluglugln
505560
arglysserlysglythrglylysaspglnhisleuaspgluaspglu
65707580
glnleualaargalaileglngluserleuasnvalgluserpropro
859095
argalaargglulysserserhisproargalaarggluasnglyser
100105110
alaasnglyglyasnsertyrglnleuproleumetphesersergly
115120125
pheargthrcysalaglycyshissergluileglyhisglyargphe
130135140
leusercysmetglyalavaltrphisproglucysphecyscyshis
145150155160
glycysserglnproiletyrasptyrgluphesermetserglyasn
165170175
hisprotyrhislysthrcystyrlysgluargphehisprolyscys
180185190
aspvalcysglnglnpheileprothrasnthrasnglyleuileglu
195200205
tyrargalahisprophetrpleuglnlystyrcysproserhisglu
210215220
valaspglythrproargcyscyssercysgluargmetgluproarg
225230235240
gluserargtyrvalleuleuaspaspglyarglysleucysleuglu
245250255
cysleuaspseralavalmetaspthrthrglucysglnproleutyr
260265270
leugluileglngluphetyrgluglyleuasnmetlysvalglugln
275280285
glnvalproleuleuleuvalgluargglnalaleuasnglualamet
290295300
gluglyglulysthrglyhishishisleuprogluthrargglyleu
305310315320
cysleuserglugluglnthrvalserthrileleuargargproarg
325330335
metalaglyasnlysilemetglumetargthrgluprotyrargleu
340345350
thrargargcysgluvalthralaileleuileleutyrglyleupro
355360365
argleuleuthrglyserileleualahisglumetmethisalatrp
370375380
leuargleulysglytyrargthrleuserproaspilegluglugly
385390395400
ilecysglnvalleualahismettrpileglusergluilethrala
405410415
glyserglyserasnalaalaserthrserserserserthrserser
420425430
lyslysglyglyargserglnphegluarglysleuglyaspphephe
435440445
lyshisglnilegluseraspthrservalalatyrglyaspglyphe
450455460
argalaglyasnglnalavalleuglntyrglyleulysargthrleu
465470475480
gluhisileargleuthrglythrleuprophe
485490
<210>19
<211>486
<212>prt
<213>sorghumbicolor
<400>19
metglytrpleuthrlysphepheargglyserthrhisasnileser
151015
gluglyglntyrhisserargproalagluaspthralatrpasnglu
202530
proserserserprovalvalthraspilepheserglupheasnasn
354045
gluaspileaspargalailealaleuserleuserglugluglugln
505560
arglysalalysthrileasplysaspmethisleuglugluaspglu
65707580
glnleualaargalaileglngluserleuasnvalgluserpropro
859095
proserarggluasnglyseralaasnglyglyasnalatyrhispro
100105110
leuprophemetpheserserglypheargalacysalaglycyshis
115120125
arggluileglyhisglyargpheleusercysmetglyalavaltrp
130135140
hisproglucyspheargcyshisalacysserglnproiletyrasp
145150155160
tyrgluphesermetserglyasnhisprotyrhislysthrcystyr
165170175
lysgluglnphehisprolyscysaspvalcyslysglnpheilepro
180185190
thrasnmetasnglyleuileglutyrargalahisprophetrpleu
195200205
glnlystyrcysproserhisgluvalaspglythrproargcyscys
210215220
sercysgluargmetgluproarggluserargtyrvalleuleuasp
225230235240
aspglyarglysleucysleuglucysleuaspseralavalmetasp
245250255
thrasnglucysglnproleutyrleugluileglngluphetyrglu
260265270
glyleuasnmetlysvalgluglnglnvalproleuleuleuvalglu
275280285
argglnalaleuasnglualametgluglyglulysalaglyhishis
290295300
hisleuprogluthrargglyleucysleuserglugluglnthrval
305310315320
serthrileleuargargproargmetalaglyasnlysilemetgly
325330335
metilethrgluprotyrargleuthrargargcysgluvalthrala
340345350
ileleuileleutyrglyleuproargleuleuthrglyserileleu
355360365
alahisglumetmethisalatrpleuargleulysglytyrargthr
370375380
leuserproaspvalglugluglyilecysglnvalleualahisleu
385390395400
trpileglusergluilemetalaglyserglyserglyalaalaser
405410415
serserserglysersersersermetserserlyslysalaglyarg
420425430
serglnphegluhislysleuglyaspphephelyshisglnileglu
435440445
thraspthrsermetalatyrglygluglypheargalaglyasnarg
450455460
alavalleuglntyrglyleulysargthrleugluhisileargleu
465470475480
thrglythrpheprophe
485
<210>20
<211>508
<212>prt
<213>zeamays
<400>20
metglytrpleuthrlysphepheargglyserthrhisasnileser
151015
glugluglntyrhisserargproalagluaspthralatrpasnglu
202530
proserserserprovalvalthraspileleuserglupheasnasn
354045
gluaspileaspargalailealaleuserleuserglugluglugln
505560
arglysglulysalaileasplysaspmethisleuglugluaspglu
65707580
glnleualaargalaileglngluserleuasnvalgluserpropro
859095
argargasnglyseralaasnglyglythrmettyrhisproproarg
100105110
gluthrglyasnalatyrglnproproarggluasnglyseralaasn
115120125
glyglyasnalatyrhisproleuprophemetpheserserglyphe
130135140
argalacysalaglycyshisarggluileglyhisglyargpheleu
145150155160
sercysmetglyalavaltrphisproglucyspheargcyshisala
165170175
cysserglnproiletyrasptyrgluphesermetserglyasnhis
180185190
protyrhislysthrcystyrlysgluglnphehisprolyscysasp
195200205
valcyslysglnpheileprothrasnmetasnglyleuileglutyr
210215220
argalahisprophetrpvalglnlystyrcysproserhisglumet
225230235240
aspglythrproargcyscyssercysgluargmetgluproargglu
245250255
serlystyrvalleuleuaspaspglyarglysleucysleuglucys
260265270
leuaspseralavalmetaspthrasnaspcysglnproleutyrleu
275280285
gluileglngluphetyrgluglyleuasnmetlysvalgluglngln
290295300
valproleuleuleuvalgluargglnalaleuasnglualametglu
305310315320
glyglulysalaglyhishishisleuprogluthrargglyleucys
325330335
leuserglugluglnthrvalserthrileleuargproargmetala
340345350
glyasnlysilemetglymetilethrgluprotyrargleuthrarg
355360365
argcysgluvalthralaileleuileleutyrglyleuproargleu
370375380
leuthrglyserileleualahisglumetmethisalatrpleuarg
385390395400
leulysglytyrargthrleuserproaspvalglugluglyilecys
405410415
glnvalleualahismettrpileglusergluilemetalaglyser
420425430
glyserseralaalaserserserserglyserserserserthrser
435440445
serlyslysglyglyargserglnphegluhisargleuglyaspphe
450455460
phelyshisglnilegluthraspthrsermetalatyrglyaspgly
465470475480
pheargthrglyasnargalavalleuhistyrglyleulysargthr
485490495
leugluhisileargleuthrglythrpheprophe
500505
<210>21
<211>553
<212>prt
<213>arabidopsisthaliana
<400>21
metglytrpleuthrlysileleulysglyserserhislyspheser
151015
aspglyglncysasnglyargtyrarggluaspargasnleuglugly
202530
proargtyrseralagluglyserasppheasplysglugluileglu
354045
cysalailealaleuserleusergluglngluhisvalileprogln
505560
aspasplysglylyslysileileglutyrlyssergluthrgluglu
65707580
aspaspaspaspaspgluaspgluaspgluglutyrmetargalagln
859095
leuglualaalaglugluglugluargargvalalaglnalaglnile
100105110
gluglugluglulysargargalaglualaglnleuglugluthrglu
115120125
lysleuleualalysalaargleugluglugluglumetargargser
130135140
lysalaglnleuglugluaspgluleuleualalysalaleuglnglu
145150155160
sermetasnvalglyserproproargtyraspproglyasnileleu
165170175
glnprotyrpropheleuileproserserhisargilecysvalgly
180185190
cysglnalagluileglyhisglyargpheleusercysmetglygly
195200205
valtrphisproglucysphecyscysasnalacysasplysproile
210215220
ileasptyrgluphesermetserglyasnargprotyrhislysleu
225230235240
cystyrlysgluglnhishisprolyscysaspvalcyshisasnphe
245250255
ileprothrasnproalaglyleuileglutyrargalahisprophe
260265270
trpmetglnlystyrcysproserhisgluargaspglythrproarg
275280285
cyscyssercysgluargmetgluprolysaspthrlystyrleuile
290295300
leuaspaspglyarglysleucysleuglucysleuaspseralaile
305310315320
metaspthrhisglucysglnproleutyrleugluilearggluphe
325330335
tyrgluglyleuhismetlysvalgluglnglnileprometleuleu
340345350
valgluargseralaleuasnglualametgluglyglulyshisgly
355360365
hishishisleuprogluthrargglyleucysleusergluglugln
370375380
thrvalthrthrvalleuargargproargileglyalaglytyrlys
385390395400
leuileaspmetilethrgluprocysargleuileargargcysglu
405410415
valthralaileleuileleutyrglyleuproargleuleuthrgly
420425430
serileleualahisglumetmethisalatrpleuargleuasngly
435440445
tyrproasnleuargprogluvalglugluglyilecysglnvalleu
450455460
alahismettrpleuglusergluthrtyralaglyserthrleuval
465470475480
aspilealaserserserserseralavalvalseralaserserlys
485490495
lysglygluargserasppheglulyslysleuglygluphephelys
500505510
hisglnilegluseraspserserseralatyrglyaspglyphearg
515520525
glnglyasnglnalavalleulyshisglyleuargargthrleuasp
530535540
hisileargleuthrglythrphepro
545550
<210>22
<211>528
<212>prt
<213>arabidopsisthaliana
<400>22
metaspserserserserserserserserserprosersersertyr
151015
glyvalalaargvalserhisileserasnprocysilepheglyglu
202530
valglyserserserserserthrtyrargasplyslystrplysleu
354045
metlystrpvalserlysleuphelysserglyserasnglyglygly
505560
serglyalahisthrasnhishisproproglnpheglngluaspglu
65707580
asnmetvalpheproleuproproserserleuaspaspargserarg
859095
glyalaargasplysglugluleuaspargserileserleuserleu
100105110
alaaspasnthrlysargprohisglytyrglytrpsermetaspasn
115120125
asnargasppheproargprophehisglyglyleuasnproserser
130135140
pheileproprotyrgluprosertyrglntyrargargargglnarg
145150155160
ilecysglyglycysasnseraspileglyserglyasntyrleugly
165170175
cysmetglythrphephehisproglucyspheargcyshissercys
180185190
glytyralailethrgluhisglupheserleuserglythrlyspro
195200205
tyrhislysleucysphelysgluleuthrhisprolyscysgluval
210215220
cyshishispheileprothrasnaspalaglyleuileglutyrarg
225230235240
cyshisprophetrpasnglnlystyrcysproserhisglutyrasp
245250255
lysthralaargcyscyssercysgluargleuglusertrpaspval
260265270
argtyrtyrthrleugluaspglyargserleucysleuglucysmet
275280285
gluthralailethraspthrglyglucysglnproleutyrhisala
290295300
ileargasptyrtyrgluglymettyrmetlysleuaspglnglnile
305310315320
prometleuleuvalglnargglualaleuasnaspalailevalgly
325330335
glulysasnglytyrhishismetprogluthrargglyleucysleu
340345350
serglugluglnthrvalthrservalleuargargproargleugly
355360365
alahisargleuvalglymetargthrglnproglnargleuthrarg
370375380
lyscysgluvalthralaileleuvalleutyrglyleuproargleu
385390395400
leuthrglyalaileleualahisgluleumethisglytrpleuarg
405410415
leuasnglypheargasnleuasnprogluvalglugluglyilecys
420425430
glnvalleusertyrmettrpleuglusergluvalleuserasppro
435440445
serthrargasnleuproserthrserservalalathrserserser
450455460
serserpheserasnlyslysglyglylysserasnvalglulyslys
465470475480
leuglygluphephelyshisglnilealahisaspalaserproala
485490495
tyrglyglyglypheargalaalaasnalaalaalacyslystyrgly
500505510
leuargargthrleuasphisileargleuthrglythrpheproleu
515520525
<210>23
<211>1613
<212>prt
<213>arabidopsisthaliana
<400>23
metgluproproalaalaargvalthrproserilelysalaaspcys
151015
serhisservalasnileilecysglugluthrvalleuhisserleu
202530
valserhisleuseralaalaleuargarggluglyileservalphe
354045
valaspalacysglyleuglngluthrlysphepheserilelysgln
505560
asnglnproleuthraspglyalaargvalleuvalvalvalileser
65707580
aspgluvalgluphetyraspprotrppheprolyspheleulysval
859095
ileglnglytrpglnasnasnglyhisvalvalvalprovalphetyr
100105110
glyvalaspserleuthrargvaltyrglytrpalaasnsertrpleu
115120125
glualaglulysleuthrserhisglnserlysileleuserasnasn
130135140
valleuthraspsergluleuvalglugluilevalargaspvaltyr
145150155160
glylysleutyrproalagluargvalglyiletyralaargleuleu
165170175
gluileglulysleuleutyrlysglnhisargaspileargserile
180185190
glyiletrpglymetproglyileglylysthrthrleualalysala
195200205
valpheasnhismetserthrasptyraspalasercyspheileglu
210215220
asnpheaspglualaphehislysgluglyleuhisargleuleulys
225230235240
gluargileglylysileleulysaspglupheaspilegluserser
245250255
tyrilemetargprothrleuhisargasplysleutyrasplysarg
260265270
ileleuvalvalleuaspaspvalargaspserleualaalagluser
275280285
pheleulysargleuasptrppheglyserglyserleuileileile
290295300
thrservalasplysglnvalphealaphecysglnileasnglnile
305310315320
tyrthrvalglnglyleuasnvalhisglualaleuglnleupheser
325330335
glnservalpheglyileasngluprogluglnasnasparglysleu
340345350
sermetlysvalileasptyrvalasnglyasnproleualaleuser
355360365
iletyrglyarggluleumetglylyslysserglumetgluthrala
370375380
phephegluleulyshiscysproproleulysileglnaspvalleu
385390395400
lysasnalatyrseralaleuseraspasnglulysasnilevalleu
405410415
aspilealaphephephelysglygluthrvalasntyrvalmetgln
420425430
leuleuglugluserhistyrpheproargleualaileaspvalleu
435440445
valasplyscysvalleuthrilesergluasnthrvalglnmetasn
450455460
asnleuileglnaspthrcysglngluilepheasnglygluileglu
465470475480
thrcysthrargmettrpgluproserargileargtyrleuleuglu
485490495
tyraspgluleugluglyserglygluthrlysalametprolysser
500505510
glyleuvalalagluhisilegluserilepheleuaspthrserasn
515520525
vallyspheaspvallyshisaspalaphelysasnmetpheasnleu
530535540
lyspheleulysiletyrasnsercysserlystyrileserglyleu
545550555560
asnpheprolysglyleuaspserleuprotyrgluleuargleuleu
565570575
histrpgluasntyrproleuglnserleuproglnasppheaspphe
580585590
glyhisleuvallysleusermetprotyrserglnleuhislysleu
595600605
glythrargvallysaspleuvalmetleulysargleuileleuser
610615620
hisserleuglnleuvalglucysaspileleuiletyralaglnasn
625630635640
ilegluleuileaspleuglnglycysthrglyleuglnargphepro
645650655
aspthrserglnleuglnasnleuargvalvalasnleuserglycys
660665670
thrgluilelyscyspheserglyvalproproasnileglugluleu
675680685
hisleuglnglythrargilearggluileproilepheasnalathr
690695700
hisproprolysvallysleuasparglyslysleutrpasnleuleu
705710715720
gluasnpheseraspvalgluhisileaspleuglucysvalthrasn
725730735
leualathrvalthrserasnasnhisvalmetglylysleuvalcys
740745750
leuasnmetlystyrcysserasnleuargglyleuproaspmetval
755760765
serleugluserleulysvalleutyrleuserglycyssergluleu
770775780
glulysilemetglypheproargasnleulyslysleutyrvalgly
785790795800
glythralailearggluleuproglnleuproasnserleugluphe
805810815
leuasnalahisglycyslyshisleulysserileasnleuaspphe
820825830
gluglnleuproarghispheilepheserasncystyrargpheser
835840845
serglnvalilealagluphevalglulysglyleuvalalaserleu
850855860
alaargalalysglnglugluleuilelysalaprogluvalileile
865870875880
cysileprometaspthrargglnargserserpheargleuglnala
885890895
glyargasnalametthraspleuvalprotrpmetglnlysproile
900905910
serglyphesermetservalvalvalserpheglnaspasptyrhis
915920925
asnaspvalglyleuargileargcysvalglythrtrplysthrtrp
930935940
asnasnglnproaspargilevalgluargphepheglncystrpala
945950955960
prothrglualaprolysvalvalalaasphisilephevalleutyr
965970975
aspthrlysmethisproseraspserglugluasnhisilesermet
980985990
trpalahisgluvallysphegluphehisthrvalserglygluasn
99510001005
asnproleuglyalasercyslysvalthrglucysglyvalglu
101010151020
valilethralaalathrglyaspthrservalserglyileile
102510301035
argglusergluthrilethrileileglulysgluaspthrile
104010451050
ileaspglugluaspthrproleuleuserarglysprogluglu
105510601065
thrasnargserargsersersergluleuglnlysleuserser
107010751080
thrserserlysvalargserlysglyasnvalphetrplystrp
108510901095
leuglycyspheproleuglnprolysasnleuargserargser
110011051110
argargthrthralaleugluglualaleugluglualaleulys
111511201125
gluargglulysleugluaspthrarggluleuglnilealaleu
113011351140
ilegluserlyslysilelyslysilelysglnalaaspgluarg
114511501155
aspglnilelyshisalaaspgluarggluglnarglyshisser
116011651170
lysasphisglugluglugluilegluserasnglulysgluglu
117511801185
argarghisserlysasptyrvalileglugluleuvalleulys
119011951200
glylysglylysarglysglnleuaspaspasplysalaaspglu
120512101215
lysgluglnilelyshisserlysasphisvalgluglugluval
122012251230
asnproproleuserlyscyslysaspcyslysseralaileglu
123512401245
aspglyileserileasnalatyrglyservaltrphisprogln
125012551260
cysphecyscysleuargcysarggluproilealametasnglu
126512701275
ileseraspleuargglymettyrhislysprocystyrlysglu
128012851290
leuarghisproasncystyrvalcysglulyslysileproarg
129513001305
thralagluglyleulystyrhisgluhisprophetrpmetglu
131013151320
thrtyrcysproserhisaspglyaspglythrprolyscyscys
132513301335
sercysgluargleugluhiscysglythrglntyrvalmetleu
134013451350
alaasppheargtrpleucysargglucysmetaspseralaile
135513601365
metaspseraspglucysglnproleuhisphegluileargglu
137013751380
phephegluglyleuhismetlysileglugluglupheproval
138513901395
tyrleuvalglulysasnalaleuasnlysalaglulysgluglu
140014051410
lysileasplysglnglyaspglncysleumetvalvalarggly
141514201425
ilecysleuserglugluglnilevalthrservalserglngly
143014351440
valargargmetleuasnlysglnileleuaspthrvalthrglu
144514501455
serglnargvalvalarglyscysgluvalthralaileleuile
146014651470
leutyrglyleuproargleuleuthrglytyrileleualahis
147514801485
glumetmethisalatyrleuargleuasnglytyrargasnleu
149014951500
asnmetvalleuglugluglyleucysglnvalleuglytyrmet
150515101515
trpleuglucysglnthrtyrvalpheaspthralathrileala
152015251530
serserserserserserargthrproleuserthrthrthrser
153515401545
lyslysvalaspproserasppheglulysargleuvalasnphe
155015551560
cyslyshisglnilegluthraspgluserprophepheglyasp
156515701575
glyphearglysvalasnlysmetmetalaserasnasnhisser
158015851590
leulysaspthrleulysgluileileserileserlysthrpro
159516001605
glntyrserlysleu
1610
<210>24
<211>450
<212>prt
<213>arabidopsisthaliana
<400>24
metvalargarglysargglnglugluaspglulysilegluileglu
151015
argvallysglugluserleulysleualalysglnalagluglulys
202530
argargleuglugluserlysgluglnglylysargileglnvalasp
354045
aspaspglnleualalysthrthrserlysasplysglyglnileasn
505560
hisserlysaspvalvalglugluaspvalasnproproproserile
65707580
aspglylyssergluileglyaspglythrservalasnproargcys
859095
leucyscysphehiscyshisargprophevalmethisgluileleu
100105110
lyslysglylysphehisileaspcystyrlysglutyrtyrargasn
115120125
argasncystyrvalcysglnglnlysileprovalasnalaglugly
130135140
ilearglysphesergluhisprophetrplysglulystyrcyspro
145150155160
ilehisaspgluaspglythralalyscyscyssercysgluargleu
165170175
gluproargglythrasntyrvalmetleuglyasppheargtrpleu
180185190
cysileglucysmetglyseralavalmetaspthrasngluvalgln
195200205
proleuhisphegluilearggluphephegluglyleupheleulys
210215220
valasplysgluphealaleuleuleuvalglulysglnalaleuasn
225230235240
lysalagluglugluglulysileasptyrhisargalaalavalthr
245250255
argglyleucysmetserglugluglnilevalproserileilelys
260265270
glyproargmetglyproaspasnglnleuilethraspilevalthr
275280285
gluserglnargvalserglyphegluvalthrglyileleuileile
290295300
tyrglyleuproargleuleuthrglytyrileleualahisglumet
305310315320
methisalatrpleuargleuasnglytyrlysasnleulysleuglu
325330335
leuglugluglyleucysglnalaleuglyleuargtrpleugluser
340345350
glnthrphealaserthraspalaalaalaalaalaalavalalaser
355360365
serserserpheserserserthralaproproalaalailethrser
370375380
lyslysseraspasptrpserilepheglulyslysleuvalgluphe
385390395400
cysmetasnglnilelysgluaspaspserprovaltyrglyleugly
405410415
phelysglnvaltyrglumetmetvalserasnasntyrasnilelys
420425430
aspthrleulysaspilevalseralaserasnalathrproaspser
435440445
thrval
450
<210>25
<211>702
<212>prt
<213>arabidopsisthaliana
<400>25
metproileseraspvalalaserleuvalglyglyalaalaleugly
151015
alaproleusergluilephelysleuvalilegluglualalyslys
202530
vallysaspphelysproleuserglnaspleualaserthrmetglu
354045
argleuvalproilepheasngluileaspmetmetglnglnglyser
505560
asnargglythrsergluleulysvalleuthrgluthrmetgluarg
65707580
alaglyglumetvalhislyscysserargileglntrptyrserile
859095
alalyslysalaleutyrthrarggluilelysalaileasnglnasp
100105110
pheleulysphecysglnilegluleuglnleuileglnhisargasn
115120125
glnleuglntyrmetargsermetglymetalaservalserthrlys
130135140
alaaspleuleuseraspileglyasnglupheserlysleucysleu
145150155160
valalaglnprogluvalvalthrlysphetrpleulysargproleu
165170175
metgluleulyslysmetleuphegluaspglyvalvalthrvalval
180185190
valseralaprotyralaleuglylysthrthrleuvalthrlysleu
195200205
cyshisaspalaaspvallysglulysphelysglnilephepheile
210215220
servalserlyspheproasnvalargleuileglyhislysleuleu
225230235240
gluhisileglycyslysalaasnglutyrgluasnaspleuaspala
245250255
metleutyrileglnglnleuleulysglnleuglyargasnglyser
260265270
ileleuleuvalleuaspaspvaltrpalagluglugluserleuleu
275280285
glnlyspheleuileglnleuproasptyrlysileleuvalthrser
290295300
argphegluphethrserpheglyprothrphehisleulysproleu
305310315320
ileaspaspgluvalglucysargaspgluileglugluasnglulys
325330335
leuprogluvalasnproproleusermetcysglyglycysasnser
340345350
alavallyshisglugluservalasnileleuglyvalleutrphis
355360365
proglycysphecyscysargsercysasplysproilealailehis
370375380
gluleugluasnhisvalserasnserargglylysphehislysser
385390395400
cystyrgluargtyrcystyrvalcyslysglulyslysmetlysthr
405410415
tyrasnilehisprophetrpglugluargtyrcysprovalhisglu
420425430
alaaspglythrprolyscyscyssercysgluargleugluproarg
435440445
glythrlystyrglylysleuseraspglyargtrpleucysleuglu
450455460
cysglylysseralametaspseraspglucysglnproleutyrphe
465470475480
aspmetargaspphephegluserleuasnmetlysileglulysglu
485490495
pheproleuileleuvalarglysgluleuleuasnlyslysgluglu
500505510
lysileaspasnhistyrgluvalleuileargalatyrcysmetser
515520525
gluglnlysilemetthrtyrvalserglugluproargthrglygln
530535540
asnlysglnleuileaspmetaspthrgluproglnglyvalvalhis
545550555560
glucyslysvalthralaileleuileleutyrglyleuproargleu
565570575
leuthrglytyrileleualahisglumetmethisalatrpleuarg
580585590
leuasnglyhismetasnleuasnasnileleuglugluglyilecys
595600605
glnvalleuglyhisleutrpleugluserglnthrtyralathrala
610615620
aspthrthralaaspalaalaseralaserserserserserargthr
625630635640
proproalaalaseralaserlyslysglyglutrpserasppheasp
645650655
lyslysleuvalgluphecyslysasnglnilegluthraspgluser
660665670
provaltyrglyleuglypheargthrvalasnglumetvalthrasn
675680685
serserleuglngluthrleulysgluileleuargargarg
690695700
<210>26
<211>644
<212>prt
<213>arabidopsisthaliana
<400>26
metalaserasptyrtyrserseraspaspgluglypheglyglulys
151015
valglyleuileglyglulysaspargpheglualagluthrilehis
202530
valilegluvalserglnhisglualaaspileglnlysalalysgln
354045
argserleualathrhisglualaglulysleuaspleualathrhis
505560
glualagluglnleuaspleualaileglnglupheserargglnglu
65707580
glugluglugluargargargthrarggluleugluasnaspalagln
859095
ilealaasnvalleuglnhisglugluarggluargleuileasnlys
100105110
lysthralaleugluaspglugluaspgluleuleualaargthrleu
115120125
glugluserleulysgluasnasnargarglysmetphegluglugln
130135140
valasnlysaspgluglnleualaleuilevalglngluserleuasn
145150155160
metgluglutyrproileargleugluglutyrlysserileserarg
165170175
argalaproleuaspvalaspgluglnphealalysalavallysglu
180185190
serleulysasnlysglylysglylysglnphegluaspgluglnval
195200205
lyslysaspgluglnleualaleuilevalglngluserleuasnmet
210215220
valgluserproproargleuglugluasnasnasnileserthrarg
225230235240
alaprovalaspgluaspgluglnleualalysalavalglugluser
245250255
leulysglylysglyglnilelysglnserlysaspgluvalglugly
260265270
aspglymetleuleugluleuasnproproproserleucysglygly
275280285
cysasnphealavalgluhisglyglyservalasnileleuglyval
290295300
leutrphisproglycysphecyscysargalacyshislysproile
305310315320
alailehisaspilegluasnhisvalserasnserargglylysphe
325330335
hislyssercystyrgluargtyrcystyrvalcyslysglulyslys
340345350
metlysthrtyrasnasnhisprophetrpglugluargtyrcyspro
355360365
valhisglualaaspglythrprolyscyscyssercysgluargleu
370375380
gluproarggluserasntyrvalmetleualaaspglyargtrpleu
385390395400
cysleuglucysmetasnseralavalmetaspseraspglucysgln
405410415
proleuhispheaspmetargaspphephegluglyleuasnmetlys
420425430
ileglulysgluphepropheleuleuvalglulysglnalaleuasn
435440445
lysalaglulysgluglulysileasptyrglntyrgluvalvalthr
450455460
argglyilecysleuserglugluglnilevalaspservalsergln
465470475480
argprovalargglyproasnasnlysleuvalglymetalathrglu
485490495
serglnlysvalthrargglucysgluvalthralaileleuileleu
500505510
tyrglyleuproargleuleuthrglytyrileleualahisglumet
515520525
methisalatyrleuargleuasnglyhisargasnleuasnasnile
530535540
leuglugluglyilecysglnvalleuglyhisleutrpleuaspser
545550555560
glnthrtyralathralaaspalathralaaspalaserserserala
565570575
serserserserargthrproproalaalaseralaserlyslysgly
580585590
glutrpserasppheasplyslysleuvalgluphecyslysasngln
595600605
ilegluthraspaspserprovaltyrglyleuglypheargthrval
610615620
asnglumetvalthrasnserserleuglngluthrleulysgluile
625630635640
leuargglnarg
<210>27
<211>587
<212>prt
<213>arabidopsisthaliana
<400>27
mettrpcysleusercysphelysproserthrlyshisaspproser
151015
gluaspargphegluglugluthrasnilevalthrglyileserleu
202530
tyrgluaspvalileleuargglnargargserglualaaspglnile
354045
glutrpalaileglnaspserpheasnproglngluthrserargcys
505560
argglnargglugluaspaspglnilealaargglyleuglntyrval
65707580
glugluthrgluleuasplysservalvalaspglugluaspglngln
859095
leuserlysilevalglugluserleulysglulysglylysserlys
100105110
glnphegluaspaspglnvalgluasnaspgluglnglnalaleumet
115120125
valglngluserleutyrmetvalgluleuseralaglnleugluglu
130135140
asplysasnileserthrileproproleuasngluaspalaglnleu
145150155160
glnlysvaliletrpgluseralalysglylysglyglnilegluhis
165170175
phelysaspprovalglugluaspglyasnleuproargvalaspleu
180185190
asnvalasnhisprohisserilecysaspglycyslysseralaile
195200205
glutyrglyargservalhisalaleuglyvalasntrphisproglu
210215220
cysphecyscysargtyrcysasplysproilealamethisgluphe
225230235240
serasnthrlysglyargcyshisilethrcystyrgluargserhis
245250255
proasncyshisvalcyslyslyslyspheproglyarglystyrlys
260265270
gluhisprophetrplysglulystyrcysprophehisgluvalasp
275280285
glythrprolyscyscyssercysgluargleugluprotrpglythr
290295300
lystyrvalmetleualaaspasnargtrpleucysvallyscysmet
305310315320
glucysalavalmetaspthrtyrglucysglnproleuhispheglu
325330335
ilearggluphepheglyserleuasnmetlysvalglulysgluphe
340345350
proleuleuleuvalglulysglualaleulyslysalaglualagln
355360365
glulysileaspasnglnhisglyvalvalthrargglyilecysleu
370375380
sergluglyglnilevalasnservalphelyslysprothrmetgly
385390395400
proasnglygluleuvalserleuglythrgluproglnlysvalval
405410415
glyglycysgluvalthralaileleuileleutyrglyleuproarg
420425430
leuleuthrglytyrileleualahisglumetmethisalatrpleu
435440445
argleuasnglythrthrserthrglnphevalphealaasnglntyr
450455460
glygluserserglnleulysvalleupheglyleuilethrglytyr
465470475480
argasnleulysleugluleuglugluglyilecysglnvalleugly
485490495
hismettrpleugluserglnthrtyrserserseralaalaalaser
500505510
seralaserserserserargthrproalaalaasnalaserlyslys
515520525
glyalaglnserasptyrglulyslysleuvalgluphecyslysasp
530535540
glnilegluthraspaspserprovaltyrglyvalglyphearglys
545550555560
valasnglnmetvalseraspserserleuhislysileleulysser
565570575
ileglnhistrpthrlysproaspserasnleu
580585
<210>28
<211>73
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim类似结构域
<220>
<221>variant
<222>(2)..(3)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(27)
<223>在位点5至27的xaa是任意氨基酸并且它们中多达七个可以缺失;代表16-23个氨基酸的范围。
<220>
<221>variant
<222>(29)..(35)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(37)..(38)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(40)..(46)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(48)..(49)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(51)..(69)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(71)..(72)
<223>xaa是任意氨基酸
<400>28
cysxaaxaacysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
151015
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaahisxaaxaaxaaxaa
202530
xaaxaaxaacysxaaxaahisxaaxaaxaaxaaxaaxaaxaacysxaa
354045
xaacysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
505560
xaaxaaxaaxaaxaacysxaaxaacys
6570
<210>29
<211>73
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim类似结构域
<220>
<221>variant
<222>(2)..(2)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(27)
<223>在位点5至27的xaa是任意氨基酸并且它们中多达七个可以缺失;代表16-23个氨基酸的范围。
<220>
<221>variant
<222>(32)..(34)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(38)..(38)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(40)..(46)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(53)..(53)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(55)..(59)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(61)..(62)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(64)..(64)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(66)..(66)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(68)..(68)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(71)..(72)
<223>xaa是任意氨基酸
<400>29
cysxaavalcysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
151015
xaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaahisprophetrpxaa
202530
xaaxaatyrcysproxaahisxaaxaaxaaxaaxaaxaaxaacyscys
354045
sercysgluargxaagluxaaxaaxaaxaaxaatyrxaaxaaleuxaa
505560
aspxaaargxaaleucysxaaxaacys
6570
<210>30
<211>66
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim类似结构域
<220>
<221>variant
<222>(2)..(2)
<223>xaa是asp、glu、tyr或his
<220>
<221>variant
<222>(5)..(6)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(7)..(7)
<223>xaa是phe或lys
<220>
<221>variant
<222>(8)..(8)
<223>xaa是ile、lys或phe
<220>
<221>variant
<222>(9)..(9)
<223>可以存在或缺失。若存在,xaa是pro或ser
<220>
<221>variant
<222>(10)..(10)
<223>可以存在或缺失。若存在,xaa是thr、arg或val
<220>
<221>variant
<222>(11)..(11)
<223>可以存在或缺失。若存在,xaa是asn或thr
<220>
<221>variant
<222>(12)..(13)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(14)..(14)
<223>可以存在或缺失。若存在,xaa是gly
<220>
<221>variant
<222>(15)..(15)
<223>xaa是leu、ile、met或gly
<220>
<221>variant
<222>(16)..(16)
<223>xaa是arg、lys或ile
<220>
<221>variant
<222>(17)..(17)
<223>xaa是glu、gly、lys或thr
<220>
<221>variant
<222>(18)..(18)
<223>xaa是tyr或phe
<220>
<221>variant
<222>(19)..(19)
<223>xaa是arg、his、ser、asn或lys
<220>
<221>variant
<222>(20)..(20)
<223>xaa是ala、cys、glu、ile或asn
<220>
<221>variant
<222>(25)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(26)..(26)
<223>xaa是gln或glu
<220>
<221>variant
<222>(27)..(27)
<223>xaa是lys、thr或arg
<220>
<221>variant
<222>(31)..(31)
<223>xaa是phe、val、ile、ser或thr
<220>
<221>variant
<222>(33)..(33)
<223>xaa是glu或asp
<220>
<221>variant
<222>(34)..(34)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(36)..(36)
<223>xaa是gly、lys、arg、ser或ala
<220>
<221>variant
<222>(38)..(38)
<223>xaa是pro、thr或ala
<220>
<221>variant
<222>(39)..(39)
<223>xaa是arg或lys
<220>
<221>variant
<222>(46)..(46)
<223>xaa是met或leu
<220>
<221>variant
<222>(48)..(48)
<223>xaa是pro、ser或his
<220>
<221>variant
<222>(49)..(52)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(54)..(55)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(57)..(57)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(59)..(59)
<223>xaa是gly、phe或asn
<220>
<221>variant
<222>(61)..(61)
<223>xaa是arg、lys、ser或trp
<220>
<221>variant
<222>(64)..(64)
<223>xaa是leu、arg或val
<220>
<221>variant
<222>(65)..(65)
<223>xaa是glu或lys
<400>30
cysxaavalcysxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaaxaa
151015
xaaxaaxaaxaahisprophetrpxaaxaaxaatyrcysproxaahis
202530
xaaxaaaspxaathrxaaxaacyscyssercysgluargxaagluxaa
354045
xaaxaaxaaxaatyrxaaxaaleuxaaaspxaaargxaaleucysxaa
505560
xaacys
65
<210>31
<211>66
<212>prt
<213>arabidopsisthaliana
<400>31
cysaspvalcysserhispheileprothrasnhisalaglyleuile
151015
glutyrargalahisprophetrpvalglnlystyrcysproserhis
202530
gluhisaspalathrproargcyscyssercysgluargmetglupro
354045
argasnthrargtyrvalgluleuasnaspglyarglysleucysleu
505560
glucys
65
<210>32
<211>5
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>syntheticsequence:metallopeptidaseactivesitemotif
<400>32
hisglumetmethis
15
<210>33
<211>19
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>syntheticsequence:carboxylterminalregionmotif
<220>
<221>variant
<222>(3)..(10)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(12)..(15)
<223>xaa是任意氨基酸
<400>33
glulysxaaxaaxaaxaaxaaxaaxaaxaaargxaaxaaxaaxaaser
151015
gluglugln
<210>34
<211>18
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>syntheticsequence:carboxylterminalregionmotif
<220>
<221>variant
<222>(3)..(10)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(12)..(15)
<223>xaa是任意氨基酸
<400>34
glulysxaaxaaxaaxaaxaaxaaxaaxaaargxaaxaaxaaxaaser
151015
glugln
<210>35
<211>21
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:uim1结构域
<220>
<221>variant
<222>(1)..(1)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(2)..(4)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(5)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(7)..(7)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(8)..(8)
<223>xaa是大的核酸残基,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(10)..(10)
<223>xaa是脂肪族氨基酸残基,比如ile、leu或val
<220>
<221>variant
<222>(11)..(11)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(12)..(12)
<223>xaa是大的核酸残基,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(13)..(13)
<223>可以存在或缺失。若存在,xaa是任意氨基酸.
<220>
<221>variant
<222>(15)..(15)
<223>xaa是大的核酸残基,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(16)..(16)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(17)..(17)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(18)..(18)
<223>可以存在或缺失。若存在,xaa是任意氨基酸.
<220>
<221>variant
<222>(19)..(21)
<223>xaa是极性氨基酸残基,比如cys,asp,glu,
his,lys,asn,gln,arg,ser或thr
<400>35
xaaxaaxaaxaaxaaleuxaaxaaalaxaaxaaxaaxaaserxaaxaa
151015
xaaxaaxaaxaaxaa
20
<210>36
<211>21
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:uim2结构域
<220>
<221>variant
<222>(1)..(1)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(2)..(4)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(5)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(7)..(7)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(8)..(8)
<223>xaa是大的氨基酸,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(10)..(10)
<223>xaa是脂肪族氨基酸,比如ile、leu或val
<220>
<221>variant
<222>(11)..(11)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(12)..(12)
<223>xaa是大的氨基酸,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(13)..(13)
<223>可以存在或缺失。若存在,xaa是任意氨基酸.
<220>
<221>variant
<222>(15)..(15)
<223>xaa是大的氨基酸,比如glu、phe、his、ile、lys、met、gln、arg、trp或tyr
<220>
<221>variant
<222>(16)..(16)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<220>
<221>variant
<222>(17)..(17)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(18)..(18)
<223>xaa是小的氨基酸残基,比如ala、cys、asp、gly、asn、pro、ser、thr或val
<220>
<221>variant
<222>(19)..(21)
<223>xaa是极性氨基酸残基,比如cys、asp、glu、his、lys、asn、gln、arg、ser或thr
<400>36
xaaxaaxaaxaaxaaleuxaaxaaalaxaaxaaxaaxaaserxaaxaa
151015
xaaxaaxaaxaaxaa
20
<210>37
<211>43
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:植物eod结构域
<220>
<221>variant
<222>(1)..(1)
<223>xaa是glu或lys
<220>
<221>variant
<222>(8)..(8)
<223>xaa是leu或met
<220>
<221>variant
<222>(9)..(9)
<223>xaa是lys、arg、gly、thr或glu
<220>
<221>variant
<222>(11)..(11)
<223>xaa是lys或arg
<220>
<221>variant
<222>(12)..(12)
<223>xaa是arg或ile
<220>
<221>variant
<222>(13)..(13)
<223>xaa是gly或lys
<220>
<221>variant
<222>(14)..(14)
<223>xaa是asp、asn或glu
<220>
<221>variant
<222>(15)..(15)
<223>xaa是arg、gln、lys或leu
<220>
<221>variant
<222>(17)..(17)
<223>xaa是ile、met或val
<220>
<221>variant
<222>(18)..(18)
<223>xaa是lys、asn、thr或ala
<220>
<221>variant
<222>(20)..(20)
<223>xaa是leu或pro
<220>
<221>variant
<222>(22)..(22)
<223>xaa是lys或ser
<220>
<221>variant
<222>(24)..(24)
<223>xaa是val或ala
<220>
<221>variant
<222>(27)..(27)
<223>xaa是ser、thr、gly或ala
<220>
<221>variant
<222>(28)..(28)
<223>xaa是glu、gln、asp、ser或gly
<220>
<221>variant
<222>(30)..(30)
<223>xaa是ile、gly、thr或val
<220>
<221>variant
<222>(31)..(31)
<223>xaa是ser或thr
<220>
<221>variant
<222>(32)..(32)
<223>xaa是lys或arg
<220>
<221>variant
<222>(35)..(35)
<223>xaa是gly、thr或ser
<220>
<221>variant
<222>(39)..(39)
<223>xaa是val、ile、ala或lys
<220>
<221>variant
<222>(42)..(42)
<223>xaa是val或ile
<400>37
xaaargcysvalilecysglnxaaxaatyrxaaxaaxaaxaaxaagln
151015
xaaxaaleuxaacysxaahisxaatyrhisxaaxaacysxaaxaaxaa
202530
trpleuxaaileasnlysxaacysproxaacys
3540
<210>38
<211>203
<212>prt
<213>populustrichocarpa
<400>38
metgluvalhistyrmetasnthrasppheprotyrthrthrthrglu
151015
serphemetaspphephegluglyleuthrhisalaprovalasntyr
202530
alahisasnglypromethisaspglnaspasnalatyrtrpsermet
354045
asnmetasnalatyrlyspheglypheserglyleuglyserthrser
505560
tyrtyrserprotyrgluvalasnaspasnleuproargmetaspval
65707580
serargmetalatrpglutyrproservalvalilelysalaleutrp
859095
glnaspaspvalaspproaspthrmetthrtyrglugluleuvalasp
100105110
leuglygluthrvalglythrglnserlysglyleuserprogluleu
115120125
ileserleuleuprothrserlyscyslyspheglyserphepheser
130135140
arglysargserglygluargcysvalilecysglnmetlystyrlys
145150155160
argglyasplysglnilelysleuleucyslyshisalatyrhisser
165170175
glucysilethrlystrpleuglyileasnlysvalcysprovalcys
180185190
asnaspgluvalpheglyglugluserargasn
195200
<210>39
<211>240
<212>prt
<213>ricinuscommunis
<400>39
metgluvalhistyrileasnthrglypheprotyrthrvalthrglu
151015
serpheleuaspphephegluglyleuserhisvalprovalhistyr
202530
alahisthrglyglnvalleuaspglnvalglngluasnalatyrtrp
354045
sermetasnmetasnalatyrlystyrglypheserglyproglyser
505560
thrtyrtyraspprotyrgluvalasnaspasnleuproargmetasp
65707580
valserargserthrtrpglutyrproservalvalasnmetgluglu
859095
alathrthrthraspthrglnsergluglyaspalavalvalglyval
100105110
hisalaserprogluglucysileproasnhisthrserglyaspser
115120125
proglnglyvaltrpglnaspaspvalaspproaspasnmetthrtyr
130135140
glugluleuleuaspleuglygluthrvalglythrglnserarggly
145150155160
leuserglngluleuileserleuleuprothrserlyscyslysphe
165170175
argserphepheleuarglyslysalaglygluargcysvalilecys
180185190
glnmetargtyrlysargglyasplysglnmetlysleuprocyslys
195200205
hisvaltyrhisserglucysileserlystrpleuglyileasnlys
210215220
valcysprovalcysasnasngluvalpheglygluaspserarghis
225230235240
<210>40
<211>245
<212>prt
<213>prunuspersica
<400>40
metasnglyasnglyglnmetaspvalhistyrileaspthraspphe
151015
protyrthrprothrgluserphemetaspphepheglyglyvalthr
202530
hisvalprometasntyrglyhisalametpromethisaspglnglu
354045
thralatyrtrpsermetasnmethissertyrlyspheglyproser
505560
glyproglyserasnsertyrtyrglyasntyrtyrgluvalasnasp
65707580
hisleuproargmetaspvalserargargthrtrpgluhisproser
859095
valmetasnserglugluproalaasnileaspserhisprogluglu
100105110
gluaspalavalalaglualaalaprogluglucysileglnasngln
115120125
glnasnthrasnthrserglnvalvaltrpglngluaspileasppro
130135140
aspasnmetthrtyrglugluleuleuaspleuglyglualavalgly
145150155160
thrglnserargglyleuseraspgluleuileserleuleuprothr
165170175
serlystyrlyscysglyserphepheserarglyslysserglyglu
180185190
argcysvalilecysglnmetargtyrlysargglyaspargglnile
195200205
asnleuprocyslyshisvaltyrhisserglucysileserlystrp
210215220
leuglyileasnlysvalcysprovalcysasnleugluvalsergly
225230235240
glugluserarghis
245
<210>41
<211>242
<212>prt
<213>theobromacacao
<400>41
metasnglyasnargglnmetgluvalhistyrileaspthrglyphe
151015
protyrthralathrgluserphemetaspphephegluglyleuthr
202530
hisvalprovalasntyrthrhisthrvalprometglnaspglnglu
354045
asniletyrtrpsermetsermetasnalatyrlyspheglypheser
505560
glyprogluserthrphetyrserprotyrgluvalserasphisleu
65707580
proargmetaspvalserargargthrtrpasptyrproserthrleu
859095
asnserglugluproalathrileaspmetglnproglyglygluala
100105110
valvalglyilehisalaileprogluglucysilethrasnhisgln
115120125
serasnserasnserglnvalvaltrpglnaspasnileaspproasp
130135140
asnmetthrtyrglugluleuleuaspleuglygluthrileglyser
145150155160
glnserargglyleuserglngluleuileaspleuleuprothrser
165170175
lyscyslyspheglyserphepheserthrlysarggluargcysval
180185190
ilecysglnmetargtyrlysargglygluglnglnmetlysleupro
195200205
cyslyshisvaltyrhisserglncysilethrlystrpleuserile
210215220
asnlysilecysprovalcysasnasngluvalpheglyglugluser
225230235240
arghis
<210>42
<211>247
<212>prt
<213>vitisvinifera
<400>42
metasnglyasnargglnmetgluvalhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleugly
202530
hisvalprovalasntyralaglnalaglualamethisasnglnser
354045
ileglngluasnphetyrtrpthrmetasnmetasnsertyrlysphe
505560
glypheserglyproglyserthrtyrtyrglyprotyraspvalasn
65707580
gluhisvalproglyilegluvalserargargprotrpglutyrpro
859095
sersermetilevalglugluprothrthrilegluthrglnprothr
100105110
glyasngluvalmetasnvalhisalaileprogluglucysserpro
115120125
asnhistyrseralathrserserglnalailetrpglnaspasnval
130135140
aspproaspasnmetthrtyrglugluleuleuaspleuglygluala
145150155160
valglythrglnserargglyleuserglngluhisileasnleuleu
165170175
prothrcysargtyrlysserglyargleupheserarglysargser
180185190
alagluargcysvalilecysglnmetglytyrlysargglyasparg
195200205
glnilelysleuprocyslyshisvaltyrhisthraspcysglythr
210215220
lystrpleuthrileasnlysvalcysprovalcysasnilegluval
225230235240
pheglyglugluserarghis
245
<210>43
<211>247
<212>prt
<213>glycinemax
<400>43
metasnaspglyargglnmetglyvalhistyrvalaspalaglyphe
151015
protyralavalasnaspasnphevalaspphepheglnglyphethr
202530
hisvalprovalasntyralaphealaglyserileproaspglnglu
354045
servaltyrtrpsermetasnmetasnprotyrlyspheglyleuser
505560
glyproglyserthrsertyrtyrsersertyrgluvalasnglyhis
65707580
leuproargmetgluileaspargalaglutrpglutyrproserthr
859095
ilethrthrvalglugluproalathrthraspserproproargarg
100105110
aspglyvalthrsermetglnthrileprogluglucysserproasn
115120125
hishisgluserasnserserserglnvaliletrpglnaspasnile
130135140
tyrproaspaspmetthrtyrglugluleuleuaspleuglygluala
145150155160
valglythrglnserargglyleuserglngluleuileaspmetleu
165170175
prothrserlystyrlyspheglyserleuphelysarglysasnser
180185190
glylysargcysvalilecysglnmetthrtyrargargglyaspgln
195200205
glnmetlysleuprocysserhisvaltyrhisglyglucysilethr
210215220
lystrpleuserileasnlyslyscysprovalcysasnthrgluval
225230235240
pheglyglugluserthrhis
245
<210>44
<211>247
<212>prt
<213>glycinemax
<400>44
metasnaspglyargglnmetglyvalasntyrvalaspalaglyphe
151015
protyralavalasngluasnphevalaspphepheglnglyphethr
202530
provalprovalasntyralaphealaglyserileproaspglnglu
354045
servaltyrtrpsermetasnmetasnprotyrlyspheglyleuser
505560
glyproglyserthrsertyrtyrsersertyrgluvalasnglyhis
65707580
leuproargmetgluileaspargalaglutrpglutyrproserthr
859095
ilethrthrvalglugluproalathrthraspserproproargarg
100105110
aspglyvalthrasnmetglnthrileprogluglucysserproasn
115120125
hishisgluserasnserserserglnvaliletrpglnaspasnile
130135140
aspproaspasnmetthrtyrglugluleuleuaspleuglygluala
145150155160
valglythrglnserargglyleuserglngluleuileaspmetleu
165170175
prothrserlystyrlyspheglyasnleuphelysarglysasnser
180185190
glylysargcysvalilecysglnmetthrtyrargargglyaspgln
195200205
glnmetlysleuprocysserhisvaltyrhisglyglucysilethr
210215220
lystrpleuserileasnlyslyscysprovalcysasnthrgluval
225230235240
pheglyglugluserthrhis
245
<210>45
<211>248
<212>prt
<213>arabidopsisthaliana
<400>45
metasnglyaspasnargprovalgluaspalahistyrthrgluthr
151015
glypheprotyralaalathrglysertyrmetaspphetyrglygly
202530
alaalaglnglyproleuasntyrasphisalaalathrmethispro
354045
glnaspasnleutyrtrpthrmetasnthrasnalatyrlysphegly
505560
pheserglyseraspasnalaserphetyrglysertyraspmetasn
65707580
asphisleuserargmetserileglyargthrasntrpasptyrhis
859095
prometvalasnvalalaaspaspprogluasnthrvalalaargser
100105110
valglnileglyaspthraspgluhisserglualagluglucysile
115120125
alaasngluhisaspproaspserproglnvalsertrpglnaspasp
130135140
ileaspproaspthrmetthrtyrglugluleuvalgluleuglyglu
145150155160
alavalglythrgluserargglyleuserglngluleuilegluthr
165170175
leuprothrlyslystyrlyspheglyserilepheserarglysarg
180185190
alaglygluargcysvalilecysglnleulystyrlysileglyglu
195200205
argglnmetasnleuprocyslyshisvaltyrhisserglucysile
210215220
serlystrpleuserileasnlysvalcysprovalcysasnserglu
225230235240
valpheglygluproserilehis
245
<210>46
<211>243
<212>prt
<213>capsellarubella
<400>46
metasnglyaspargprovalgluaspalahistyrthrglualaglu
151015
pheprotyralaalaserglysertyrileaspphetyrglyglyala
202530
proglnglyproleuasntyralahisalaglythrmetaspasnleu
354045
tyrtrpthrmetasnthrasnalatyrlyspheglypheserglyser
505560
aspasnproserphetyrasnsertyraspmetthrasphisleuser
65707580
argmetserileglyargthrasntrpglutyrhisprometvalasn
859095
valaspaspproaspilethrleualaargservalglnileglyasp
100105110
seraspgluhisserglualagluaspcysilealaasngluhisasp
115120125
proaspserproglnvalsertrpglnaspaspileaspproaspthr
130135140
metthrtyrglugluleuvalgluleuglyglualavalglythrglu
145150155160
serargglyleuserglngluleuilegluthrleuprothrarglys
165170175
phelyspheglyserilepheserarglysargalaglygluargcys
180185190
valilecysglnleulystyrlysileglygluargglnmetasnleu
195200205
procyslyshisvaltyrhisserglucysileserlystrpleuser
210215220
ileasnlysvalcysprovalcysasnthrgluvalpheglyasppro
225230235240
serilehis
<210>47
<211>249
<212>prt
<213>sorghumbicolor
<400>47
metasnsercysargglnmetgluleuhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleuthr
202530
tyralahisalaaspphealaleumetaspglypheglnaspglngly
354045
asnprotyrtrpalametmethisthrasnsertyrlystyrglytyr
505560
serglyproglyasntyrtyrthrtyralahisvaltyraspileasp
65707580
asptyrmethisargalaaspglyglyargargvaltrpaspasnthr
859095
thrproalaasnasnvalaspseralaasnvalvalleuglnglyser
100105110
glualaproargthrthralaasnthrthrthrgluglucysilegln
115120125
glnvalhisglnserproglyserprohisvalvaltrpglnaspasn
130135140
ileaspproaspasnmetthrtyrglugluleuleuaspleuglyglu
145150155160
valvalglythrglnserargglyleuserglngluargileserser
165170175
leuprovalthrlystyrlyscysglyphepheserarglyslysthr
180185190
argarggluargcysvalilecysglnmetglutyrargargglyasn
195200205
leuglnmetthrleuprocyslyshisvaltyrhisalasercysval
210215220
thrargtrpleuserileasnlysvalcysprovalcysphealaglu
225230235240
valproglyaspgluprolysarggln
245
<210>48
<211>253
<212>prt
<213>zeamays
<400>48
metasnserserargglnmetgluleuhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleuthr
202530
tyralahisalaaspphealaleuthraspglypheglnaspglngly
354045
asnprotyrtrpalametmethisthrasnsertyrlystyrglytyr
505560
serglyproglyasntyrtyrsertyralahisvaltyraspileasp
65707580
asptyrmetargargalaaspglyglyargargiletrpaspasnthr
859095
thrprovalasnasnvalaspseralaasnvalvalleuglnglygly
100105110
glualaprohisthrthrthrasnthrileasnlysglucysilegln
115120125
glnvalhisglnserproglyserproglnvalvaltrpglnaspasn
130135140
ilegluproaspasnmetthrtyrglugluleuleuaspleuglyglu
145150155160
alavalglythrglnserargglyleuserglngluargileserser
165170175
leuprovalthrlystyrlyscysglyphepheserarglyslysthr
180185190
argarggluargcysvalilecysglnmetglutyrargargglyasn
195200205
leuglnmetthrleuprocyslyshisvaltyrhisalasercysval
210215220
thrargtrpleuglyileasnlysvalcysprovalcysphealaglu
225230235240
valproglygluaspproglualametserglnglnleu
245250
<210>49
<211>249
<212>prt
<213>zeamays
<400>49
metthrserserargglnmetgluleuhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleuthr
202530
tyralahisalaaspphealaleumetaspglypheglnaspglngly
354045
asnprotyrtrpthrmetmethisthrasnsertyrlystyrglytyr
505560
serglyserglyasntyrtyrsertyralahisalatyraspileasp
65707580
asptyrmethisargthraspglyglyargargthrtrpaspasnthr
859095
thrprovalasnasnvalaspseralaasnvalvalleuglnglygly
100105110
glualaproargthrthralaasnthrthrsergluaspcysilegln
115120125
glnvalhisglnserproglyserproglnvalvaltrpglnaspasn
130135140
ileaspproaspasnmetthrtyrglugluleuleuaspleuglyglu
145150155160
alavalglythrglnserargglyleuserglnglucysileserleu
165170175
leuproilethrlystyrlyscysglyphepheserarglyslysthr
180185190
argarggluargcysvalilecysglnmetglutyrargargglyasn
195200205
leuglnilethrleuprocyslyshisvaltyrhisalasercysval
210215220
thrargtrpleuserileasnlysvalcysprovalcysphealaglu
225230235240
valproglygluaspserleuarggln
245
<210>50
<211>272
<212>prt
<213>oryzasativa
<400>50
metthrgluserhisgluargaspthrgluvalthrargtrpglnval
151015
hisaspprosergluglymetasnglyserargglnmetgluleuhis
202530
tyrileasnthrglypheprotyrthrilethrgluserphemetasp
354045
phephegluglyleuthrtyralahisalaaspphealailealaasp
505560
alaphehisaspglnalaasnprotyrtrpalametmethisthrasn
65707580
sertyrlystyrglytyrserglyalaglyasntyrtyrsertyrgly
859095
hisvaltyraspmetasnasptyrmethisargalaaspglyglyarg
100105110
argiletrpaspasnalathrprovalasnasnthrgluserproasn
115120125
valvalleuglnglyglygluthrprohisalaasnthrserserthr
130135140
thrgluglucysileglnglnglnvalhisglnasnserserserpro
145150155160
glnvaliletrpglnaspasnileaspproaspasnmetthrtyrglu
165170175
gluleuleuaspleuglyglualavalglythrglnserargglyleu
180185190
serglngluargileserleuleuprovalthrlystyrlyscysgly
195200205
phepheserarglyslysthrargarggluargcysvalilecysgln
210215220
metglutyrargargglyasnleuglnmetthrleuprocyslyshis
225230235240
valtyrhisalasercysvalthrargtrpleuserileasnlysval
245250255
cysprovalcysphealagluvalproglyaspgluprolysarggln
260265270
<210>51
<211>250
<212>prt
<213>oryzasativa
<400>51
metasnglyserargglnmetgluleuhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleuthr
202530
tyralahisalaaspphealailealaaspalaphehisaspglnala
354045
asnprotyrtrpalametmethisthrasnsertyrlystyrglytyr
505560
serglyalaglyasntyrtyrsertyrglyhisvaltyraspmetasn
65707580
asptyrmethisargalaaspglyglyargargiletrpaspasnala
859095
thrprovalasnasnthrgluserproasnvalvalleuglnglygly
100105110
gluthrprohisalaasnthrserserthrthrgluglucysilegln
115120125
glnglnvalhisglnasnserserserproglnvaliletrpglnasp
130135140
asnileaspproaspasnmetthrtyrglugluleuleuaspleugly
145150155160
glualavalglythrglnserargglyleuserglngluargileser
165170175
leuleuprovalthrlystyrlyscysglyphepheserarglyslys
180185190
thrargarggluargcysvalilecysglnmetglutyrargarggly
195200205
asnleuglnmetthrleuprocyslyshisvaltyrhisalasercys
210215220
valthrargtrpleuserileasnlysvalcysprovalcyspheala
225230235240
gluvalproglyaspgluprolysarggln
245250
<210>52
<211>251
<212>prt
<213>brachypodiumdistachyon
<400>52
metasnglyserargglnmetgluleuhistyrileasnthrglyphe
151015
protyrthrilethrgluserphemetaspphephegluglyleuthr
202530
tyralahisalaaspphealaleualaaspalapheglnaspglnala
354045
asnprotyrtrpthrmetmetglnthrasnsertyrlystyrglytyr
505560
serglyalaserasntyrtyrsertyrglyhisvaltyraspmetasn
65707580
asptyrmethisargalaaspglyglyargargiletrpaspasnpro
859095
thrproalaserasnthraspserproasnvalvalleuglnglyala
100105110
alaglualaprohisproargalaserserthrthrgluglucysile
115120125
glnglnprovalhisglnasnserserserproglnvalvaltrpgln
130135140
aspasnvalaspproaspasnmetthrtyrglugluleuleuaspleu
145150155160
glyglualavalglythrglnserargglyleuserglngluargile
165170175
serserleuprovalthrlystyrlyscysglyphepheserarglys
180185190
lysthrargarggluargcysvalilecysglnmetglutyrargarg
195200205
glyaspleuglnmetalaleuprocyslyshisvaltyrhisalaser
210215220
cysvalthrargtrpleuserileasnlysvalcysprovalcysphe
225230235240
alagluvalproserglugluproserarggln
245250
<210>53
<211>246
<212>prt
<213>solanumlycopersicum
<400>53
metasntrpasnglnglnthrgluiletyrtyrthrasnglyalamet
151015
protyrasnserileglyserphemetaspphepheglyglyvalthr
202530
tyrasphisvalasntyrilephealaaspproprotyralaglnglu
354045
serleutyrproserileserthrasnprotyrlyspheglytyrser
505560
glualaglyserphesertyrtyrasptyraspargglutyrvalval
65707580
asnasphisvalserglyileglugluhisasparghisleugluasn
859095
proserthrthrthrvalasnvalalaalaasnvalhisarggluglu
100105110
ileserglyserasnserleuthrasnservalglucysproarggly
115120125
glnileasnthrargaspsergluvalvaltrpglnaspasnileasp
130135140
proaspasnmetthrtyrglugluleuleugluleuglyglualaval
145150155160
glythrglnserargglyleuserglnasnglnileserleuleupro
165170175
valthrlysphelyscysglyphepheserarglyslysserarglys
180185190
gluargcysvalilecysglnmetglutyrlysarglysaspglngln
195200205
valthrleuprocyslyshisvaltyrhisalaglycysglyserarg
210215220
trpleuserileasnlysalacysproilecystyrthrgluvalval
225230235240
ileasnthrserlysarg
245
<210>54
<211>10
<212>prt
<213>人工序列(artificialsequence)
<220>
<223>合成序列:lim结构域基序
<220>
<221>variant
<222>(2)..(3)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(5)..(6)
<223>xaa是任意氨基酸
<220>
<221>variant
<222>(8)..(9)
<223>xaa是任意氨基酸
<400>54
hisxaaxaacysxaaxaacysxaaxaacys
1510
- 还没有人留言评论。精彩留言会获得点赞!