一种烯醇还原酶突变体及其在制备(R)-香茅醛中的应用的制作方法
(一)技术领域
本发明涉及一种烯醇还原酶突变体oye3-mut及其在生物酶法不对称合成(r)-香茅醛的应用。
(二)
背景技术:
香茅醛存在于植物的代谢产物中,可以通过对植物的精油进行蒸馏或者从溶剂中直接进行提取得到,多以(r/s)-香茅醛混合物的形式出现。单一构型的(r)-香茅醛是植物精油中的重要组成成分,在香料原料、食品香精和减少咖啡因的苦涩方面具有广泛的应用。(r)-香茅醛是合成l-薄荷醇的关键中间体。近年研究还发现,在对机体细胞影响作用较小的状况下,(r)-香茅醛能够对乳腺癌细胞产生特异性抑制。同时,(r)-香茅醛也可以作为雄性大蜡蛾性信息素((5r/11r)二甲基二十二烷)和胃酸分泌抑制剂(1,2,3,4,4a,9,9a,10-八氢吖叮)等重要手性药物合成的中间体。
(r)-香茅醛的合成方法包括化学法和生物酶法。由日本takasago公司发明的化学合成法中,月桂烯首先和二乙胺合成香叶基胺,随后香叶基胺经过异构化和水解之后最终合成(r)-香茅醛。在该生产工艺下,(r)-香茅醛的得率可以达到97%以上。在诸多合成(r)-香茅醛的化学法中,有些方法在产物的e.e.值上不能达到工业生产的要求,另外一些方法则存在催化剂昂贵、催化剂回收利用难度大、合成比较困难以及能耗高等一系列问题。与使用月桂烯为原料进行三步法不对称合成(r)-香茅醛相比,生物酶法从天然柠檬醛一步不对称还原合成(r)-香茅醛是更为简捷、经济的方法。天然柠檬醛是柠檬醛顺式和反式异构体的混合物,顺反异构体比例约为2:3。在生物酶法中,烯醇还原酶是不对称还原柠檬醛合成(r)-香茅醛的关键酶,该酶催化柠檬醛顺式和反式异构体的加氢反应,所得产物的立体选择性通常是互补的,所以产物e.e.值往往很低。针对此问题,可以通过偶联氨基酸催化的顺反异构反应予以改善。通过偶联氨基酸催化的柠檬醛顺反异构反应,将部分顺式柠檬醛转化为反式柠檬醛,显著地提高了产物(r)-香茅醛的e.e.值;在10ml催化体系中,添加100mg/ml的甘氨酸,50mm柠檬醛经4h的催化反应后,(r)-香茅醛的e.e.值达65.4%,与不偶联顺反异构化反应时(r)-香茅醛的e.e.值(16.7%)相比,提高了48.7%(应向贤,汪钊,孟淑敏,等.一种酶法不对称还原柠檬醛提高(r)-香茅醛光学纯度的方法,专利号:zl201610438016.x)。此外,通过野生酶的大规模筛选以及酶的定向进化与改造获得具有高立体选择性的烯醇还原酶,也是行之有效的策略。相比于野生酶的筛选,基于结构信息对酶进行定向进化和理性设计可克服天然酶的先天不足,理性高效地改善酶的催化性能,极大地降低了筛选工作量。课题组前期工作对源自酿酒酵母的烯醇还原酶oye2y进行了单点突变、点饱和突变以及多点组合突变,成功获得了三个双突变子p76m/r330h、p76g/r330h和p76s/r330h,这些突变子催化(e/z)-柠檬醛不对称还原产物e.e.值均>99%,而未经改造的oye2y产物e.e.值仅为38.13%(r)(xiangxianying*,shihuayu,meijuanhuang,ranwei,shuminmeng,fengcheng,meilanyu,meirongying,manzhao,zhaowang.engineeringtheenantioselectivityofyeastoldyellowenzymeoye2yinasymmetricreductionof(e/z)-citralto(r)-citronellal,molecules,2019,24:1057)。
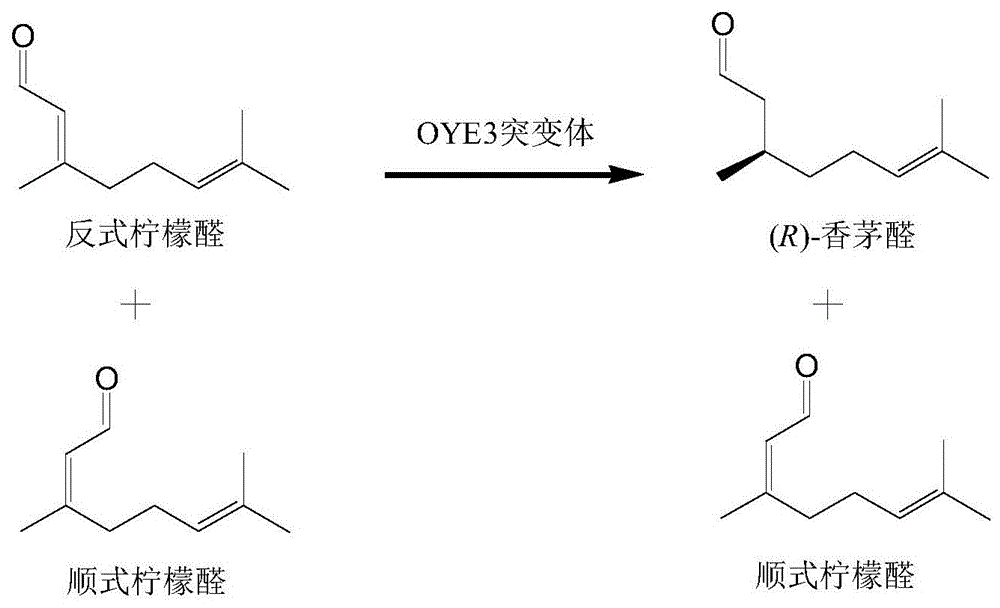
源自酿酒酵母的烯醇还原酶oye3对顺式和反式柠檬醛的催化效率不同,并且其对应氢化产物分别为(s)-和(r)-香茅醛,当以(e/z)-柠檬醛为底物时(r)-香茅醛光学纯度极低。前期研究通过分子改造改变oye2y对底物的结合模式,强化酶对顺式柠檬醛的利用,通过手性反转,使其产物构型从(s)-香茅醛转变为(r)-香茅醛,从而提高其在不对称氢化(e/z)-柠檬醛中的立体选择性。除此之外,也可以反其道而为之,即弱化酶对顺式柠檬醛的利用,使其专一利用反式柠檬醛而不利用顺式柠檬醛,与此同时保留酶对反式柠檬醛的严格立体选择性,从而提高产物(r)-香茅醛的光学纯度;在不对称氢化的同时,还可以实现底物柠檬醛顺反异构体的选择性拆分(图1)。目前,未见对oye3的296位丝氨酸突变为苯丙氨酸同时将第116位色氨酸突变位甘氨酸的分子改造报道,也未见利用oye3-mut催化不对称还原(e)-柠檬醛或(e/z)-柠檬醛合成(r)-香茅醛的报道。
(三)
技术实现要素:
本发明目的是提供一种高立体选择性的烯醇还原酶突变体oye3-mut,及其在催化不对称还原柠檬醛合成高光学纯度(r)-香茅醛中的应用。通过对源自酿酒酵母的烯醇还原酶oye3的分子改造,成功获得了不能利用顺式柠檬醛为底物的突变子oye3-mut,并运用于不对称还原(e/z)-柠檬醛合成(r)-香茅醛,产物e.e.值显著高于未经改造的野生型烯醇还原酶oye3。
本发明采用的技术方案是:
本发明提供一种烯醇还原酶突变体(记为oye3-mut),所述烯醇还原酶突变体oye3-mut是将seqidno.3所示氨基酸序列中第296位丝氨酸突变为苯丙氨酸,同时将第116位色氨酸突变为甘氨酸获得的;所述烯醇还原酶突变体oye3-mut的氨基酸序列为seqidno.5所示。
本发明还提供一种所述烯醇还原酶突变体oye3-mut编码基因,所述编码基因的核苷酸序列为seqidno.4所示。
本发明还涉及一种含所述烯醇还原酶突变体oye3-mut编码基因的载体及所述载体构建的重组基因工程菌。所述含烯醇还原酶突变体oye3-mut编码基因载体由如下方法得到:seqidno.1所示烯醇还原酶oye3基因进行密码子优化,获得优化后的oye3基因,核苷酸序列如seqidno.2所示,对应烯醇还原酶oye3的氨基酸序列如seqidno.3所示。将密码子优化后的oye3基因(seqidno.2)经人工合成后插入pet28b的ncoi和xhoi之间得到重组质粒pet28b-oye3。以重组质粒pet28b-oye3为模板,利用带有突变碱基的引物经过反向pcr扩增全质粒,得到的pcr产物经dpnⅰ酶消化甲基化的模板,酶切产物转化至大肠杆菌e.colibl21(de3)中,即可得到所述含烯醇还原酶突变体oye3-mut基因的重组基因工程菌e.colibl21(de3)/pet28b-oye3-mut,其中含重组烯醇还原酶突变体编码基因的质粒命名为pet28b-oye3-mut。
此外,本发明还提供一种所述烯醇还原酶突变体oye3-mut在催化柠檬醛制备(r)-香茅醛中的应用,所述应用为:将含烯醇还原酶突变体编码基因的工程菌(优选e.colibl21(de3)/pet28b-oye3-mut)经发酵培养获得的湿菌体超声破碎,取破碎液分离纯化,获得纯酶液,以纯酶液为催化剂,加入底物和辅酶nadp+,以d-葡萄糖为辅助底物,以葡萄糖脱氢酶为辅助酶,以ph7.0、50mmpipes缓冲液为反应介质构成反应体系,在30℃、300rpm条件下反应,反应完全后,反应液用乙酸乙酯萃取,取乙酸乙酯层分离纯化,获得(r)-香茅醛;所述底物为(e)-柠檬醛、(z)-柠檬醛或(e/z)-柠檬醛;所述底物以200mm底物异丙醇溶液的形式加入;所述反应体系中,催化剂用量以纯酶计0.96u/ml,所述底物终浓度为20mm,d-葡萄糖终浓度为50mm,nadp+终浓度为0.6mm,葡萄糖脱氢酶用量为0.96u/ml。
进一步,所述湿菌体按如下方法制备:将含烯醇还原酶突变体oye3-mut编码基因的工程菌(优选e.colibl21(de3)/pet28b-oye3-mut)接种至含100μg/ml卡那霉素的lb液体培养基中,37℃培养12h,获得种子液,将种子液以体积浓度2%的接种量接种至新鲜的含100μg/ml卡那霉素的lb液体培养基中,37℃培养至od600为0.5~0.7,再加入终浓度为0.2mm的iptg,25℃诱导12h,获得诱导培养液,再将诱导培养液于4℃和10000rpm下离心10min,弃去上清液,收集湿菌体。
进一步,所述纯酶液按如下方法制备:将湿菌体以1g:20ml的比例溶解到50mm的tris-hcl(ph8.0)中,然后再使用细胞超声破碎仪,以工作2s,间隔6s,功率125w的程序破碎15min。在4℃和8000rpm下离心10min,上清即为粗酶液。将粗酶液通过层析仪转移到已经平衡好的ni2+柱中,上样完成后首先用含5mm咪唑的洗脱液洗脱大量的杂蛋白,接着再用含40mm咪唑的洗脱液洗脱残留的杂蛋白和部分目的蛋白,再用含100mm咪唑的洗脱液进行洗脱,此时大量的目的蛋白被洗脱下来,收集含目的蛋白的流出液,最后用含250mm咪唑的洗脱液洗脱柱子中残留的全部蛋白,将含目的蛋白的流出液用截留分子量为10kda的超滤管在4℃和5000rpm下离心30min进行脱盐浓缩,取截留液即为纯酶液;所述洗脱液组成:相应浓度(即5mm、40mm、100mm、250mm)的咪唑、300mm氯化钠,溶剂为50mm的tris-hcl缓冲液,ph8.0。
本发明所述葡萄糖脱氢酶是将含葡萄糖脱氢酶基因的工程菌经发酵培养的湿菌体超声破碎,取破碎液分离纯化,获得纯酶液;所述葡萄糖脱氢酶基因核苷酸序列为seqidno.6所示,编码蛋白氨基酸序列为seqidno.7所示。所述纯酶液的制备方法同催化剂的制备。
与现有技术相比,本发明的有益效果主要体现在:本发明所述立体选择性专一的烯醇还原酶突变体oye3-mut与烯醇还原酶oye3相比,在生物酶法不对称合成(r)-香茅醛中具有更高的立体选择性。利用突变体oye3-mut为生物催化剂,以nadp+为辅酶,以葡萄糖脱氢酶和d-葡萄糖驱动辅酶循环,成功构建双酶级联催化不对称合成光学纯(r)-香茅醛的反应体系。oye3-mut不能利用(z)-柠檬醛为底物。在双酶催化体系中,20mm(e)-柠檬醛和(e/z)-柠檬醛分别经oye3-mut酶法催化其不对称还原反应11h后,与烯醇还原酶oye3相比,(e)-柠檬醛催化产物e.e.值从63.4%(r)提高到了>99%(r),(e/z)-柠檬醛催化产物e.e.值从23.6%(r)提高到了>99%(r)。
(四)附图说明
图1为生物酶法不对称合成(r)-香茅醛示意图。
图2为烯醇还原酶oye3编码基因经pcr扩增后的琼脂糖凝胶电泳图;泳道m为marker;泳道1为烯醇还原酶oye3编码基因。
图3为质粒pet-28b-oye3经反向pcr扩增后的琼脂糖凝胶电泳图;泳道m为marker;泳道1为质粒反向pcr产物。
图4为分离纯化后烯醇还原酶突变体oye3-mut和烯醇还原酶oye3的sds-page图;泳道m为marker;泳道1为分离纯化后的烯醇还原酶突变体oye3-mut;泳道2为分离纯化后的烯醇还原酶oye3。
(五)具体实施方式
下面结合具体实施例对本发明进行进一步描述,但本发明的保护范围并不仅限于此:基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
实施例1:基因工程菌e.colibl21(de3)/pet28b-oye3的构建
将源自酿酒酵母的烯醇还原酶oye3编码基因(核苷酸序列如seqidno.1所示)进行密码子优化,密码子优化后的oye3基因的核苷酸序列如seqidno.2所示,对应烯醇还原酶oye3的氨基酸序列如seqidno.3所示,由杭州擎科生物技术有限公司合成。密码子优化后的oye3基因的核苷酸序列(seqidno.2)经人工合成后插入pet28b的ncoi和xhoi之间得到重组质粒pet28b-oye3,电泳图见图2所示。
将合成的重组表达质粒pet28b-oye3取出5μl加入到50μle.colibl21(de3)感受态中,轻弹管壁混匀,冰上放置30min。42℃水浴热击45s,立即置于冰上2min。向管中加入1mllb液体培养基,37℃摇床培养1h。将培养液4500rpm离心4min,取出800μl上清。用剩下的培养基将菌体悬浮,取出100μl涂在含有100μg/ml卡那霉素的lb固体培养基上。37℃培养箱过夜培养12-14h,得到基因工程菌e.colibl21(de3)/pet28b-oye3,并提取质粒pet28b-oye3。
lb液体培养基组成:酵母提取物5g/l,胰蛋白胨10g/l,nacl10g/l,溶剂为蒸馏水,ph7.0-7.5。
实施例2:重组表达质粒pet28b-oye3-mut与基因工程菌e.colibl21(de3)/pet28b-oye3-mut的构建
1、重组表达质粒pet28b-oye3-mut
以实施例1制备的质粒pet28b-oye3为模板,设计引物f1和r1,利用反向pcr技术克隆出全质粒,转化于大肠杆菌e.colibl21(de3)。提取质粒测序,并利用软件对测序结果进行分析,该序列含有一个长为1200bp的开放阅读框,第296位氨基酸成功由丝氨酸变为苯丙氨酸。再以该突变质粒为模板,设计f2和r2,利用反向pcr克隆出全质粒,得到第296位丝氨酸突变为苯丙氨酸,同时第116位色氨酸突变为甘氨酸的突变质粒,再将突变质粒转化于大肠杆菌e.colibl21(de3),提取质粒测序,成功得到突变质粒pet28b-oye3-mut,核苷酸序列如seqidno.4所示,其氨基酸序列如seqidno.5所示。
引物如下:
f1:5’-acgatccgttcctggttgaaggtgaaggtgaatatagcg-3’;
r1:5’-aaccaggaacggatcggtaacacgcggttcaaccagatg-3’;
f2:5’-gtccagctgggtagtctgggttgggcgagttttccggat-3’;
r2:5’-cagactacccagctggacccatgcaaagctctgacagtc-3’。
反向pcr扩增体系见表1所示。
表1pcr扩增反应体系
pcr反应进程为:95℃预变性5min;之后,以95℃变性15s,60℃复性15s,72℃保持1min为一个循环,重复这样循环30次;最后,72℃保持5min。pcr产物经0.8%琼脂糖凝胶电泳检测,从图3中可看到约7000bp处有明亮的条带,条带与质粒的理论值相符。
2、基因工程菌e.colibl21(de3)/pet28b-oye3-mut
pcr产物经37℃酶切1h去除甲基化的模板,酶切体系如表2所示。
表2pcr产物中甲基化模板的消化体系
dpnⅰ酶切后的pcr产物(质粒所携带的突变体基因oye3-mut的核苷酸序列为seqidno.4所示,氨基酸序列为seqidno.5所示),直接转化表达宿主菌大肠杆菌e.colibl21(de3),得到大肠杆菌基因工程菌e.colibl21(de3)/pet28b-oye3-mut。转化子经菌落pcr验证后接入含100μg/ml卡那霉素的lb液体培养基中,37℃培养12h,离心收集菌体,提取质粒,送去测序。测序结果经软件分析氨基酸序列,296位由丝氨酸成功突变为苯丙氨酸同时116位点由色氨酸突变为甘氨酸。
实施例3:基因工程菌e.colibl21(de3)/pet28b-gdh的构建
源自微小杆菌重组葡萄糖脱氢酶gdh编码基因(核苷酸序列如seqidno.6所示,氨基酸序列如seqidno.7所示),由杭州擎科生物技术有限公司合成。gdh基因的核苷酸序列(seqidno.6)经人工合成后插入pet28b的ndei和xhoi之间得到重组质粒pet28b-gdh。
将合成的重组表达质粒pet28b-gdh取出5μl加入到50μle.colibl21(de3)感受态中,轻弹管壁混匀,冰上放置30min。42℃水浴热击45s,立即置于冰上2min。向管中加入1mllb液体培养基,37℃摇床培养1h。将培养液4500rpm离心4min,取出800μl上清。用剩下的培养基将菌体悬浮,取出100μl涂在含有100μg/ml卡那霉素的lb固体培养基上。37℃培养箱过夜培养12-14h,得到基因工程菌e.colibl21(de3)/pet28b-gdh。
实施例4:烯醇还原酶oye3及其突变体oye3-mut的诱导表达及分离纯化
将实施例2构建的基因工程菌e.colibl21(de3)/pet28b-oye3-mut接种至含100μg/ml卡那霉素的lb液体培养基中,37℃培养12h,获得种子液,将种子液以体积浓度2%的接种量接种至新鲜的含100μg/ml卡那霉素的lb液体培养基中,37℃培养至od600为0.5~0.7,再加入终浓度为0.2mm的iptg,25℃诱导12h,获得诱导培养液,再将培养液于4℃和10000rpm下离心10min,弃去上清液,收集湿菌体。
将上述湿菌体按1g湿菌体加20mltris-hcl缓冲液(ph8.0)的比例加入适量tris-hcl(ph8.0)缓冲液,在125w下超声破碎15min(工作2s,间歇6s)后,破碎液于4℃和10000rpm下离心10min,重复离心三次后得到上清粗酶液。
按照ni-nta金属螯合亲和层析使用说明,取上清粗酶液上样至预平衡ni2+柱中,再依次用含5mm咪唑、40mm咪唑、100mm咪唑、250mm咪唑的洗脱液(相应浓度的咪唑、300mm的氯化钠,溶剂为50mm的tris-hcl缓冲液,ph8.0)洗脱杂蛋白和目的蛋白,收集含100mm咪唑的洗脱液对应的流出液,并用截留分子量为10kda的超滤管在4℃和5000rpm下离心30min进行脱盐浓缩,取截留液即为突变体oye3-mut纯酶液,存于-20℃备用。
将实施例2构建的基因工程菌e.colibl21(de3)/pet28b-oye3-mut替换为实施例1构建的基因工程菌e.colibl21(de3)/pet28b-oye3,其他操作相同,获得烯醇还原酶oye3纯酶液。
烯醇还原酶oye3及其突变体oye3-mut纯酶液的纯度经sds-page凝胶电泳验证,sds-page电泳结果如图4所示。烯醇还原酶oye3及其突变体oye3-mut经sds-page电泳后均为单一条带,表明分离纯化后的烯醇还原酶oye3及其突变体oye3-mut均为电泳纯。烯醇还原酶oye3及其突变体oye3-mut的亚基理论大小分别为44kda,而在sds-page电泳上的表观大小约为48~49kda。
实施例5:重组葡萄糖脱氢酶gdh的诱导表达及分离纯化
将实施例3构建的基因工程菌e.colibl21(de3)/pet28b-gdh接种至含100μg/ml卡那霉素的lb液体培养基中,37℃培养12h,获得种子液,将种子液以体积浓度2%的接种量接种至新鲜的含100μg/ml卡那霉素的lb液体培养基中,37℃培养至od600为0.5~0.7,再加入终浓度为0.2mm的iptg,25℃诱导12h,获得诱导培养液,再将培养液于4℃和10000rpm下离心10min,弃去上清液收集湿菌体。
将上述湿菌体按1g湿菌体加20mltris-hcl缓冲液(ph8.0)的比例加入适量tris-hcl(ph8.0)缓冲液,在125w下超声破碎15min(工作2s,间歇6s)后,破碎液于4℃和10000rpm下离心10min,重复离心三次后得到上清粗酶液。
按照ni-nta金属螯合亲和层析使用说明,取上清粗酶液上样至预平衡ni2+柱中,再依次用含5mm咪唑、40mm咪唑、100mm咪唑、250mm咪唑的洗脱液(相应浓度的咪唑、300mm的氯化钠,溶剂为50mm的tris-hcl缓冲液,ph8.0)洗脱杂蛋白和目的蛋白。收集含100mm咪唑的洗脱液对应的流出液,并用截留分子量为10kda的超滤管在4℃和5000rpm下离心30min进行脱盐浓缩,取截留液即为重组葡萄糖脱氢酶纯酶液,存于-20℃备用。
实施例6:烯醇还原酶和葡萄糖脱氢酶的比酶活测定
1、烯醇还原酶的比酶活测定
烯醇还原酶的酶活力采用分光光度计的单因素动力学方法测定在340nm处吸光值的变化来计算酶活。比酶活测定体系:2mm(z)-柠檬醛,0.26mmnadph,100μl纯酶液,加50mm磷酸盐缓冲液(ph7.0)补足1ml。(z)-柠檬醛先以异丙醇为溶剂配成200mm的底物溶液,再取需要的量加入比酶活测定体系。酶活力单位(u)定义:在30℃下,每分钟氧化1μmolnadph所需的酶量。每次做三组平行实验,计算平均值和标准误差。烯醇还原酶的体积酶活力及比活力计算公式如公式1和公式2:
①δa为1min之内吸光值的变化;
②v1、v2分别为反应液的总体积和添加的酶液体积,ml;
③6220为nad(p)h在340nm下的摩尔消光系数,
④l为光程距离,为1cm;t为反应时间,1min;
2、葡萄糖脱氢酶的比酶活测定
葡萄糖脱氢酶的酶活力采用分光光度计的单因素动力学方法测定340nm处吸光值的变化来计算酶活。酶活检测体系为:5mm葡萄糖,0.4mmnadp+,100μl纯酶液,加50mm磷酸盐缓冲液(ph7.0)补足1ml。酶活力单位(u)定义:在30℃下,每分钟还原1μmolnadp+所需的酶量。每次做三组平行实验,计算平均值和标准误差。葡萄糖脱氢酶的体积酶活力及比活力计算公式如上公式1和公式2。
实施例7:烯醇还原酶突变体oye3-mut和烯醇还原酶oye3的催化表现比较
以实施例4制备的烯醇还原酶突变体oye3-mut纯酶液作为催化剂,以实施例5方法制备的重组葡萄糖脱氢酶纯酶液为辅助酶,分别加入0.96u/ml催化剂、20mm(e)-柠檬醛、(z)-柠檬醛或(e/z)-柠檬醛为底物,50mmd-葡萄糖为辅底物,0.6mmnadp+和0.96u/ml辅助酶,于50mmpipes缓冲液(ph7.0)中构成1ml反应体系。底物(e)-柠檬醛、(z)-柠檬醛或(e/z)-柠檬醛先以异丙醇为溶剂配成200mm的底物溶液,再取需要的量加入反应体系。
在30℃和300rpm的条件下反应11h,反应结束后,反应液加1ml的乙酸乙酯萃取1.5-2h,取上层有机相除水后,样品用气相色谱检测底物和产物,并计算e.e值和得率。
同样条件下,以实施例4方法制备的烯醇还原酶oye3纯酶液作为对照催化剂。
结果如表3所示。当还原反应进行11h后,oye3-mut催化(e)-柠檬醛或(e/z)-柠檬醛得到的(r)-香茅醛产物e.e值均>99%,而催化(z)-柠檬醛无催化产物生成。酶法催化结果表明,当利用oye3-mut催化(e)-柠檬醛或(e/z)-柠檬醛的不对称还原合成(r)-香茅醛时,其立体选择性显著地优于突变前的烯醇还原酶oye3。
气相色谱检测条件为:色谱柱,bgb174,30.0m×250μm×0.25μm;检测器fid,250℃;载气,n2;载气流量,1.38ml/min;分流比,1:100;柱温程序为初始温度90℃,维持25min,20℃/min升至160℃保持2min,然后20℃/min升至180℃保持3min。进样量,1μl;进口温度,250℃。底物(z)-柠檬醛和(e)-柠檬醛的保留时间分别为29.2min和30.2min。(s)-香茅醛和(r)-香茅醛的保留时间分别为22.5min和23.0min。
表3烯醇还原酶突变体oye3-mut和烯醇还原酶oye3催化效果比较
“/”表示没有催化活力。
序列表
<110>浙江工业大学
<120>一种烯醇还原酶突变体及其在制备(r)-香茅醛中的应用
<160>7
<170>siposequencelisting1.0
<210>1
<211>1203
<212>dna
<213>未知(unknown)
<400>1
atgccatttgtaaaaggttttgagccgatctccctaagagacacaaacctttttgaacca60
attaagattggtaacactcagcttgcacatcgtgcggttatgcccccattgaccagaatg120
agggccactcaccccggaaatattccaaataaggagtgggctgctgtgtattatggtcag180
cgtgctcaaagacctggtaccatgatcatcacggaaggtacgtttatttcccctcaagcc240
ggcggctatgacaacgcccctgggatttggtctgatgagcaggtcgctgagtggaagaat300
atctttttagccatccatgattgtcagtcgttcgcgtgggtacaactttggtctttaggc360
tgggcatccttcccagacgtattggcaagagacgggttacgctatgactgtgcatctgac420
agagtgtatatgaatgctacgttacaagaaaaggccaaagatgcgaataatctcgaacat480
agtttgactaaagacgacattaaacagtatatcaaggattacatccatgcggctaagaat540
tctatcgcggctggcgccgatggtgtagaaattcatagcgccaatgggtacttgttgaat600
cagttcttggatccacattctaataagaggaccgacgaatacggcggaacgatcgaaaac660
agggcccgctttacactggaggttgtcgatgctcttatcgaaactatcggtcctgaacgg720
gtgggtttgaggttgtcgccgtacggcacttttaacagtatgtctgggggtgctgaacca780
ggtattatcgctcaatattcgtatgttttgggtgaattagagaagagggcaaaggctggt840
aagcgtttggcctttgtgcacctcgttgaaccacgtgtcacggacccatcgttggtggag900
ggcgaaggagaatattccgagggtactaacgattttgcctactctatatggaagggtcca960
atcatcagagctggtaattacgctcttcatccagaagtggttagagaacaagtaaaggat1020
cccagaaccttgataggctatggtagattcttcatctctaacccagatttagtctaccgt1080
ttagaagagggcctgccattgaacaagtatgacagaagtaccttctacaccatgtccgcg1140
gaaggttataccgactacccaacatatgaagaggcagtagatttaggttggaacaagaac1200
tga1203
<210>2
<211>1203
<212>dna
<213>未知(unknown)
<400>2
atgccgtttgttaaagggtttgaaccgattagcctgcgtgacaccaacctgttcgaaccg60
attaaaattgggaacacccagctggcccaccgtgccgttatgccgccgctgacccgcatg120
cgtgcaacccacccgggtaatattccgaataaagaatgggcagcagtttattatggtcag180
cgcgcacagcgtccgggtactatgattattacagagggcacatttatttcaccgcaggca240
ggaggctatgataatgcgccgggtatctggtcagatgaacaggtggcagagtggaaaaac300
atctttctggcaattcacgactgtcagagctttgcatgggtccagctgtggagtctgggt360
tgggcgagttttccggatgttctggcgcgtgatgggctgcgttatgattgtgcaagcgat420
cgtgtttatatgaatgcaacgctgcaggagaaagcgaaagatgcaaataatctggagcat480
agcctgaccaaagatgatattaaacagtatatcaaagactacatccacgcagcaaaaaat540
agcattgcagcaggtgcagatggtgttgaaattcatagcgcaaatggttatctgctgaat600
cagtttctggatccgcatagcaataaacgtaccgatgaatatggtggtaccattgaaaat660
cgtgcacgttttaccctggaagttgttgatgcactgattgaaaccattggtccggaacgt720
gttggtctgcgtctgagcccgtatggtacctttaatagcatgagcggtggtgcagaaccg780
ggtattattgcacagtatagctatgttctgggtgaactggaaaaacgtgcaaaagcaggt840
aaacgtctggcatttgttcatctggttgaaccgcgtgttaccgatccgagcctggttgaa900
ggtgaaggtgaatatagcgaaggtaccaatgattttgcatatagcatttggaaaggtccg960
attattcgtgcaggtaattatgcactgcatccggaagttgttcgtgaacaggttaaagat1020
ccgcgtaccctgattggttatggtcgtttttttattagcaatccggatctggtttatcgt1080
ctggaagaaggtctgccgctgaataaatatgatcgtagcaccttttataccatgagcgca1140
gaaggttataccgattatccgacctatgaagaagcagttgatctgggttggaataaaaat1200
taa1203
<210>3
<211>400
<212>prt
<213>未知(unknown)
<400>3
metprophevallysglyphegluproileserleuargaspthrasn
151015
leuphegluproilelysileglyasnthrglnleualahisargala
202530
valmetproproleuthrargmetargalathrhisproglyasnile
354045
proasnlysglutrpalaalavaltyrtyrglyglnargalaglnarg
505560
proglythrmetileilethrgluglythrpheileserproglnala
65707580
glyglytyraspasnalaproglyiletrpseraspgluglnvalala
859095
glutrplysasnilepheleualailehisaspcysglnserpheala
100105110
trpvalglnleutrpserleuglytrpalaserpheproaspvalleu
115120125
alaargaspglyleuargtyraspcysalaseraspargvaltyrmet
130135140
asnalathrleuglnglulysalalysaspalaasnasnleugluhis
145150155160
serleuthrlysaspaspilelysglntyrilelysasptyrilehis
165170175
alaalalysasnserilealaalaglyalaaspglyvalgluilehis
180185190
seralaasnglytyrleuleuasnglnpheleuaspprohisserasn
195200205
lysargthraspglutyrglyglythrilegluasnargalaargphe
210215220
thrleugluvalvalaspalaleuilegluthrileglyprogluarg
225230235240
valglyleuargleuserprotyrglythrpheasnsermetsergly
245250255
glyalagluproglyileilealaglntyrsertyrvalleuglyglu
260265270
leuglulysargalalysalaglylysargleualaphevalhisleu
275280285
valgluproargvalthraspproserleuvalgluglygluglyglu
290295300
tyrsergluglythrasnaspphealatyrseriletrplysglypro
305310315320
ileileargalaglyasntyralaleuhisprogluvalvalargglu
325330335
glnvallysaspproargthrleuileglytyrglyargphepheile
340345350
serasnproaspleuvaltyrargleuglugluglyleuproleuasn
355360365
lystyraspargserthrphetyrthrmetseralagluglytyrthr
370375380
asptyrprothrtyrgluglualavalaspleuglytrpasnlysasn
385390395400
<210>4
<211>1203
<212>dna
<213>未知(unknown)
<400>4
atgccgtttgttaaagggtttgaaccgattagcctgcgtgacaccaacctgttcgaaccg60
attaaaattgggaacacccagctggcccaccgtgccgttatgccgccgctgacccgcatg120
cgtgcaacccacccgggtaatattccgaataaagaatgggcagcagtttattatggtcag180
cgcgcacagcgtccgggtactatgattattacagagggcacatttatttcaccgcaggca240
ggaggctatgataatgcgccgggtatctggtcagatgaacaggtggcagagtggaaaaac300
atctttctggcaattcacgactgtcagagctttgcatgggtccagctggttagtctgggt360
tgggcgagttttccggatgttctggcgcgtgatgggctgcgttatgattgtgcaagcgat420
cgtgtttatatgaatgcaacgctgcaggagaaagcgaaagatgcaaataatctggagcat480
agcctgaccaaagatgatattaaacagtatatcaaagactacatccacgcagcaaaaaat540
agcattgcagcaggtgcagatggtgttgaaattcatagcgcaaatggttatctgctgaat600
cagtttctggatccgcatagcaataaacgtaccgatgaatatggtggtaccattgaaaat660
cgtgcacgttttaccctggaagttgttgatgcactgattgaaaccattggtccggaacgt720
gttggtctgcgtctgagcccgtatggtacctttaatagcatgagcggtggtgcagaaccg780
ggtattattgcacagtatagctatgttctgggtgaactggaaaaacgtgcaaaagcaggt840
aaacgtctggcatttgttcatctggttgaaccgcgtgttaccgatccgttcctggttgaa900
ggtgaaggtgaatatagcgaaggtaccaatgattttgcatatagcatttggaaaggtccg960
attattcgtgcaggtaattatgcactgcatccggaagttgttcgtgaacaggttaaagat1020
ccgcgtaccctgattggttatggtcgtttttttattagcaatccggatctggtttatcgt1080
ctggaagaaggtctgccgctgaataaatatgatcgtagcaccttttataccatgagcgca1140
gaaggttataccgattatccgacctatgaagaagcagttgatctgggttggaataaaaat1200
taa1203
<210>5
<211>400
<212>prt
<213>未知(unknown)
<400>5
metprophevallysglyphegluproileserleuargaspthrasn
151015
leuphegluproilelysileglyasnthrglnleualahisargala
202530
valmetproproleuthrargmetargalathrhisproglyasnile
354045
proasnlysglutrpalaalavaltyrtyrglyglnargalaglnarg
505560
proglythrmetileilethrgluglythrpheileserproglnala
65707580
glyglytyraspasnalaproglyiletrpseraspgluglnvalala
859095
glutrplysasnilepheleualailehisaspcysglnserpheala
100105110
trpvalglnleuglyserleuglytrpalaserpheproaspvalleu
115120125
alaargaspglyleuargtyraspcysalaseraspargvaltyrmet
130135140
asnalathrleuglnglulysalalysaspalaasnasnleugluhis
145150155160
serleuthrlysaspaspilelysglntyrilelysasptyrilehis
165170175
alaalalysasnserilealaalaglyalaaspglyvalgluilehis
180185190
seralaasnglytyrleuleuasnglnpheleuaspprohisserasn
195200205
lysargthraspglutyrglyglythrilegluasnargalaargphe
210215220
thrleugluvalvalaspalaleuilegluthrileglyprogluarg
225230235240
valglyleuargleuserprotyrglythrpheasnsermetsergly
245250255
glyalagluproglyileilealaglntyrsertyrvalleuglyglu
260265270
leuglulysargalalysalaglylysargleualaphevalhisleu
275280285
valgluproargvalthrasppropheleuvalgluglygluglyglu
290295300
tyrsergluglythrasnaspphealatyrseriletrplysglypro
305310315320
ileileargalaglyasntyralaleuhisprogluvalvalargglu
325330335
glnvallysaspproargthrleuileglytyrglyargphepheile
340345350
serasnproaspleuvaltyrargleuglugluglyleuproleuasn
355360365
lystyraspargserthrphetyrthrmetseralagluglytyrthr
370375380
asptyrprothrtyrgluglualavalaspleuglytrpasnlysasn
385390395400
<210>6
<211>789
<212>dna
<213>未知(unknown)
<400>6
atgggttataattctctgaaaggcaaagtcgcgattgttactggtggtagcatgggcatt60
ggcgaagcgatcatccgtcgctatgcagaagaaggcatgcgcgttgttatcaactatcgt120
agccatccggaggaagccaaaaagatcgccgaagatattaaacaggcaggtggtgaagcc180
ctgaccgtccagggtgacgtttctaaagaggaagacatgatcaacctggtgaaacagact240
gttgatcacttcggtcagctggacgtctttgtgaacaacgctggcgttgagatgccttct300
ccgtcccacgaaatgtccctggaagactggcagaaagtgatcgatgttaatctgacgggt360
gcgttcctgggcgctcgtgaagctctgaaatacttcgttgaacataacgtgaaaggcaac420
attatcaatatgtctagcgtccacgaaatcatcccgtggcctactttcgtacattacgct480
gcttctaagggtggcgttaaactgatgacccagactctggctatggaatatgcaccgaaa540
ggtatccgcattaacgctatcggtccaggcgcgatcaacactccaattaatgcagaaaaa600
ttcgaggatccgaaacagcgtgcagacgtggaaagcatgatcccgatgggcaacatcggc660
aagccagaggagatttccgctgtcgcggcatggctggcttctgacgaagcgtcttacgtt720
accggcatcaccctgttcgcagatggtggcatgaccctgtacccgagctttcaggctggc780
cgtggttga789
<210>7
<211>262
<212>prt
<213>未知(unknown)
<400>7
metglytyrasnserleulysglylysvalalailevalthrglygly
151015
sermetglyileglyglualaileileargargtyralagluglugly
202530
metargvalvalileasntyrargserhisprogluglualalyslys
354045
ilealagluaspilelysglnalaglyglyglualaleuthrvalgln
505560
glyaspvalserlysglugluaspmetileasnleuvallysglnthr
65707580
valasphispheglyglnleuaspvalphevalasnasnalaglyval
859095
glumetproserproserhisglumetserleugluasptrpglnlys
100105110
valileaspvalasnleuthrglyalapheleuglyalaarggluala
115120125
leulystyrphevalgluhisasnvallysglyasnileileasnmet
130135140
serservalhisgluileileprotrpprothrphevalhistyrala
145150155160
alaserlysglyglyvallysleumetthrglnthrleualametglu
165170175
tyralaprolysglyileargileasnalaileglyproglyalaile
180185190
asnthrproileasnalaglulysphegluaspprolysglnargala
195200205
aspvalglusermetileprometglyasnileglylysprogluglu
210215220
ileseralavalalaalatrpleualaseraspglualasertyrval
225230235240
thrglyilethrleuphealaaspglyglymetthrleutyrproser
245250255
pheglnalaglyarggly
260
- 还没有人留言评论。精彩留言会获得点赞!