脊柱侧弯动物模型的建立方法与流程
[0001]
本申请涉及生物医学领域,更具体地说,涉及脊柱侧弯动物模型的建立方法。
背景技术:
[0002]
脊柱侧弯分为先天性脊柱侧弯和特发性脊柱侧弯,先天性脊柱侧弯(congenital scoliosis,cs)是由椎体发育异常所致,多见于椎体形成障碍和分节不良,前者所致半椎体畸形是导致先天性脊柱侧弯的主要原因。特发性脊柱侧弯(idiopathic scoliosis,is)是指没有明确的病因导致的脊柱侧弯,是造成人类脊柱弯曲最普遍的原因,影响全世界约3%的儿童,其致病机理尚不明确。因此,治疗方式仅限于通过进行体外辅助支撑或矫正手术治疗脊柱畸形。特发性脊柱侧弯常见于青少年时期并随着年龄增长而加重,在发病进程严重程度上具有性别偏向性,女性患特发性脊柱侧弯时往往发病进程更快,程度也更严重。基因组学关联分析已经鉴定出不同的特发性脊柱侧弯病人群体具有多态性,而表型和遗传的异质性导致难以确定引起病变的具体遗传突变。
[0003]
脊柱侧弯动物模型的匮乏限制了对特发性脊柱侧弯病理的研究,在人类中导致脊椎侧弯的致病基因,在小鼠敲除模型中往往不呈现脊椎侧弯的表型。小鼠等啮齿类动物以及兔、猴等模式动物是四足动物,它们的脊柱结构和运动生物力学跟人类不同。人类早已适应直立行走,而啮齿类动物被迫直立行走将严重压迫脊柱,导致脊柱畸形。因此亟需寻找一种合适的动物建立脊柱侧弯动物模型。
技术实现要素:
[0004]
本申请实施例的目的在于提供一种脊柱侧弯动物模型的建立方法,以解决现有技术中的脊柱侧弯动物模型的匮乏的技术问题。
[0005]
为实现上述目的,本申请采用的技术方案是:提供一种脊柱侧弯动物模型的建立方法,包括以下步骤:
[0006]
基于靶点基因,合成grna和cas9 mrna,靶点基因与ccdc57基因的外显子对应,且序列如seq id no.1所示;
[0007]
将grna和cas9 mrna载入至斑马鱼的胚胎中,获得f0胚胎;
[0008]
f0胚胎培育至性成熟后与野生型斑马鱼交配获得f1胚胎,筛选有效突变胚胎培育至成年后获得相同基因型的有效敲除斑马鱼,将有效敲除斑马鱼进行自交获得f2胚胎,培育至成年筛选获得ccdc57纯合敲除斑马鱼。
[0009]
优选地,grna的合成包括以下步骤:
[0010]
将t7启动子序列、靶点序列和grna scaffold前20个碱基序列融合,得到正向引物,正向引物的序列如seq id no.2所示;将grna scaffold后20个碱基序列设计为反向引物,反向引物的序列如seq id no.3所示,grna scaffold的序列如seq id no.4所示;
[0011]
以插入gdna scaffold的pmd-18t质粒为模板,以seq id no.2和seq id no.3为引物,进行第一次扩增,得到gdna;
[0012]
以gdna为模板合成grna。
[0013]
优选地,第一次扩增的步骤为:94℃下保持2分钟后,进入32个循环,每个循环经历10秒98℃变性,30秒56℃退火,10秒68℃延伸。
[0014]
优选地,cas9 mrna合成包括以下步骤:
[0015]
以xbai内切酶线性化pt3.cas9-utrglobin质粒,再以线性化的质粒为模板,合成cas9 mrna。
[0016]
优选地,将grna和cas9 mrna载入至斑马鱼的胚胎中的步骤包括:
[0017]
将grna和cas9 mrna混合稀释,使得各自最终浓度为50ng/ul和100ng/ul,将混合物注射到1-2细胞期野生型斑马鱼胚胎中。
[0018]
优选地,获得f0胚胎和f0胚胎培育至性成熟之间还包括对f0胚胎进行敲除检测,敲除检测包括:
[0019]
将f0胚胎培养至出膜阶段,挑取胚胎,逐个提取基因组进行第二次扩增,测序检测靶点序列情况。
[0020]
优选地,第二次扩增中以f0胚胎基因组为模板,正向引物的序列如seq id no.5所示,反向引物的序列如seq id no.6所示。
[0021]
优选地,第二次扩增的步骤为:
[0022]
94℃下保持5分钟后,进入32个循环,每个循环经历95℃变性30秒、56℃退火30秒,72℃延伸30s,最后在72℃延伸5分钟,使产物充分延伸并完成加a尾过程。
[0023]
优选地,筛选有效突变胚胎的步骤为:
[0024]
将f1胚胎培养至出膜阶段,挑取胚胎,逐个提取基因组进行第三次扩增,测序确定非三倍数突变的f1胚胎为有效突变胚胎。
[0025]
优选地,培育至成年筛选可获ccdc57纯合敲除斑马鱼的步骤为:
[0026]
将具有相同非三倍数突变的f1斑马鱼进行交配,获得f2,经剪尾筛选可获得纯合非三倍数碱基插入/缺失的斑马鱼,即为纯合有效的ccdc57突变体斑马鱼。
[0027]
本申请实施例提供的脊柱侧弯动物模型的建立方法,以ccdc57基因为目标,在其外显子上设计靶点基因。在此基础上,获得敲除嵌合体的f0斑马鱼、敲除杂合子f1斑马鱼和纯合f2斑马鱼。纯合f2斑马鱼表现为脊椎侧弯性状,且可以稳定繁殖传代。上述脊柱侧弯动物模型的建立方法,以斑马鱼建立脊柱侧弯动物模型,方法简单可靠。并且由于鱼类脊椎受到的流水阻力与人类直立行走脊椎所受重力相似,因此,斑马鱼建立的脊柱侧弯动物模型更适合对脊椎侧弯致病机理的挖掘和治疗方案的探究。
附图说明
[0028]
为了更清楚地说明本申请实施例中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
[0029]
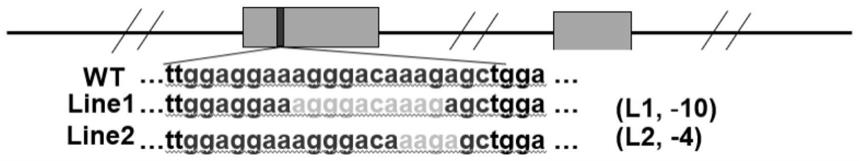
图1为ccdc57敲除靶点设计及突变品系靶点序列图:ccdc57敲除靶点设计在外显子上,具体序列如图红色部分。line1(l1)和line2(l2)为两个敲除品系,其中l1品系靶点序列丢失10个碱基,l2品系靶点序列丢失4个碱基;
[0030]
图2为野生型及两个ccdc57敲除品系斑马鱼侧面观和背面观图:野生型斑马鱼体轴呈直线型,两个敲除品系斑马鱼体轴呈s型或波浪形;
[0031]
图3为micro ct显示野生型及l1品系斑马鱼的骨骼特征图:e-f显示野生型斑马鱼骨骼呈直线型。g-l显示l1敲除品系斑马鱼骨骼呈s型或波浪形,其中g-h弯曲程度较轻,i-j弯曲程度中等,k-l弯曲程度严重;
[0032]
图4为野生型及l1品系回补注射ccdc57 mrna后胚胎外观图:野生型斑马鱼72hpf胚胎体轴直线型,注射正常的ccdc57 mrna后使得部分胚胎体轴向背侧翘起。l1品系斑马鱼72hpf胚胎体轴向腹侧弯曲,注射正常的ccdc57mrna后使得部分胚胎体轴变直或者向背侧翘起,说明注射回补正常的ccdc57mrna能够有效挽救l1品系胚胎早期体轴向腹侧弯曲的表型,该敲除品系的脊柱侧弯表型确实由ccdc57蛋白缺失引起;
[0033]
图5为q-pcr检测野生型、l1品系和回补l1品系尾加压肽及其受体表达水平图:缺失ccdc57蛋白的l1品系尾加压肽1和2及其受体的表达均降低,当回补过量正常的ccdc57 mrna后能显著提高l1品系尾加压肽1和2及其受体的表达,说明ccdc57通过影响尾加压肽信号通路调控体轴发育。
具体实施方式
[0034]
为了使本申请所要解决的技术问题、技术方案及有益效果更加清楚明白,以下结合附图及实施例,对本申请进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本申请,并不用于限定本申请。
[0035]
需要说明的是,当元件被称为“固定于”或“设置于”另一个元件,它可以直接在另一个元件上或者间接在该另一个元件上。当一个元件被称为是“连接于”另一个元件,它可以是直接连接到另一个元件或间接连接至该另一个元件上。
[0036]
需要理解的是,术语“长度”、“宽度”、“上”、“下”、“前”、“后”、“左”、“右”、“竖直”、“水平”、“顶”、“底”、“内”、“外”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本申请和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本申请的限制。
[0037]
此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括一个或者更多个该特征。在本申请的描述中,“多个”的含义是两个或两个以上,除非另有明确具体的限定。
[0038]
除非另有定义,下文中所使用的所有专业术语与本领域技术人员通常理解的含义相同。本文中所使用的专业术语只是为了描述具体实施例的目的,并不是旨在限制本发明的保护范围。
[0039]
除非另有特别说明,本发明所使用的任何试剂、原料均为可从市场上购买的商品或者可以通过公知的方法制得的产品。
[0040]
在寻找一种合适的动物建立脊柱侧弯动物模型时,用于研究的动物应当具有与人类相同或相似的运动生物力学,鱼类脊椎受到的流水阻力与人类直立行走脊椎所受重力相似,在鱼类养殖过程中容易出现可遗传的脊椎侧弯畸形,因此鱼类比四足动物更适合用于建立脊椎侧弯疾病模型。
[0041]
2016年,有文章首次提出纤毛发育缺陷引起的脑脊液流动障碍能够引起斑马鱼脊椎侧弯。2019年,有报道进一步解析了脑脊液流动障碍导致传递给脊椎管中神经细胞的肾上腺素信号减弱,从而影响下游尾加压素信号通路对背部慢肌的收缩刺激,导致脊椎背部收缩力减弱,背腹受力不均衡导致体轴向腹侧弯曲。
[0042]
ccdc57属于卷曲螺旋结构域(coiled-coil domain-containing,ccdc)结构家族,2019年之前未见与其功能相关的报道。ccdc家族中的部分基因与被报道与纤毛发育调控相关,其中ccdc114、ccdc103、ccdc65、ccdc39/40被证实突变可导致原发性纤毛运动障碍。2019年gheiratmand首次提出ccdc57是中心粒随体互作组分之一,可能与中心体复制相关。一方面ccdc57蛋白能够招募cep63,cep152,plk4分子向中心体聚集。另一方面ccdc57蛋白定位在中心体微管上且与微管蛋白相互作用,影响微管集结、稳定和有丝分裂进程。总体而言,ccdc57蛋白是调控中心粒复制的多效因子,能够进一步调控纤毛形成和有丝分裂进程。发明人在研究过程中发现ccdc57在斑马鱼脑室表达,而脑室连通脊神经管内布满纤毛,暗示斑马鱼ccdc57可能涉及脑室内纤毛发育的调控。
[0043]
在此基础上,设计了一种脊柱侧弯动物模型的建立方法,包括以下步骤:
[0044]
基于靶点基因,合成grna和cas9 mrna,靶点基因与ccdc57基因的外显子对应,且序列如seq id no.1所示;
[0045]
将grna和cas9 mrna载入至斑马鱼的胚胎中,获得f0胚胎;
[0046]
f0胚胎培育至性成熟后与野生型斑马鱼交配获得f1胚胎,筛选有效突变胚胎培育至成年后获得相同基因型的有效敲除斑马鱼,将有效敲除斑马鱼进行自交获得f2胚胎,培育至成年筛选获得ccdc57纯合敲除斑马鱼。
[0047]
脊柱侧弯动物模型的建立方法主要包括构建ccdc57基因敲除斑马鱼和ccdc57有效突变品系的筛选两部分。
[0048]
构建ccdc57基因敲除斑马鱼可采用如下方式:从ncbi数据库(https://www.ncbi.nlm.nih.gov/gene/100318317)获取斑马鱼ccdc57基因序列(gene id:100318317);针对ccdc57基因第一个外显子区域设计靶点,根据靶点设计规则,选取以gg/ggg开头、结尾紧接ngg的16-24bp序列作为靶点基因,其序列具体如seq id no.1所示,为:ggaggaaagggacaaagagc。
[0049]
当靶点基因确定之后,合成grna和cas9 mrna,将其载入至斑马鱼的胚胎中,如通入显微注射至胚胎中,获得f0胚胎。f0斑马鱼为ccdc57基因的敲除嵌合体。
[0050]
f0胚胎培育至性成熟后的斑马鱼与野生型斑马鱼交配获得f1胚胎。f1胚胎中包括有效突变的胚胎和非有效突变的胚胎,因此需要从中筛选有效突变的胚胎,并将其培育至成年。有效突变的f1斑马鱼为ccdc57基因的敲除杂合子,即该基因两个拷贝之一发生移码突变。
[0051]
f1胚胎中的有效突变的胚胎可能出现不同的基因型,将相同基因型的有效敲除斑马鱼进行自交获得f2胚胎,f2斑马鱼为ccdc57纯合敲除斑马鱼,即为该基因两个拷贝均发生移码突变的斑马鱼。
[0052]
本申请实施例提供的脊柱侧弯动物模型的建立方法,发明人发现ccdc57在斑马鱼脑室表达,而脑室连通脊神经管内布满纤毛,暗示斑马鱼ccdc57可能涉及脑室内纤毛发育的调控。以ccdc57基因为目标,在其外显子上设计靶点基因。在此基础上,获得敲除嵌合体
的f0斑马鱼、敲除杂合子f1斑马鱼和纯合f2斑马鱼。纯合f2斑马鱼表现为脊椎侧弯性状,且可以稳定繁殖传代。上述脊柱侧弯动物模型的建立方法,以斑马鱼建立脊柱侧弯动物模型,方法简单可靠。并且由于鱼类脊椎受到的流水阻力与人类直立行走脊椎所受重力相似,因此,斑马鱼建立的脊柱侧弯动物模型更适合对特发性脊柱侧弯病理的研究。
[0053]
在其中一个实施例中,grna的合成包括以下步骤:
[0054]
将t7启动子序列、靶点序列和grna scaffold前20个碱基序列融合,得到正向引物,正向引物的序列如seq id no.2所示;将grna scaffold后20个碱基序列设计为反向引物,反向引物的序列如seq id no.3所示,grna scaffold的序列如seq id no.4;
[0055]
以插入gdna固定序列的pmd18-t质粒为模板,以seq id no.2和seq id no.3为引物,进行第一次扩增,得到gdna;
[0056]
以gdna为模板合成grna。
[0057]
具体的grna的合成可采用以下步骤,当靶点基因确定之后,设计和合成用于扩展gdna模板的引物。通过直接合成长片段引物,将转录所需t7启动子序列、靶点序列和grna scaffold前20个碱基序列融合,设计为正向引物;将grna scaffold后20个碱基序列设计为反向引物。
[0058]
正向引物:taatacgactcactatagggaggaaagggacaaagagcgttttagagctagaaatagc(seq id no.2);
[0059]
反向引物:agcaccgactcggtgccact(seq id no.3)。
[0060]
然后以插入grna scaffold的pmd18-t质粒为模板,以seq id no.2和seq id no.3为引物,进行第一次扩增,如采用pcr方式扩增。
[0061]
pcr扩增完成后可获得t7启动子连接靶点和grna scaffold的gdna模板片段,可通过电泳分离目的条带,进一步通过胶回收试剂盒(omega d2500-01)进行纯化回收,获得gdna模板片段。在获得gdna模板片段后,可利用t7高效转录试剂盒合成grna,再用licl沉淀法纯化grna。
[0062]
在其中一个实施例中,第一次扩增的步骤为:94℃下保持2分钟后,进入32个循环,每个循环经历10秒98℃变性,30秒56℃退火,10秒68℃延伸。
[0063]
该次pcr的具体步骤为:以pmd18t-grna质粒为模板,以seq id no.2和seq id no.3为引物,用kod-fx高保真酶(phanta super-fidelity dna polymerase)进行扩增,配制50ul反应体系,包括25ul 2x pcr缓冲液、10ul 2mm dntps、1.5ul正向引物、1.5ul反向引物、2ul 400ng/ul模板质粒、1ul kod-fx高保真酶。采用三步法程序如:94
°
下保持2分钟后,进入32个循环,每个循环经历10秒98
°
变性,30秒56
°
退火,10秒68
°
延伸。
[0064]
在其中一个实施例中,cas9 mrna合成包括以下步骤:
[0065]
以xbai内切酶线性化pt3.cas9-utrglobin质粒,再以线性化的质粒为模板,合成cas9 mrna。
[0066]
其中,cas9线性化模板可先纯化,再以纯化后的线性化的质粒为模板,合成cas9 mrna。cas9得到的线性化产物可用酚氯仿抽提法提纯。
[0067]
线性化cas9模板,可利用t3 mrna合成试剂盒(mmessage mmachinetmt3 transcription kit,am1348)合成cas9 mrna,合成cas9 mrna。
[0068]
在上述条件下合成cas9 mrna后,还可以采用licl沉淀法纯化cas9mrna
[0069]
在其中一个实施例中,将grna和cas9 mrna载入至斑马鱼的胚胎中的步骤包括:
[0070]
将grna和cas9 mrna混合稀释,使得各自最终浓度为50ng/ul和100ng/ul,将混合物注射到1-2细胞期野生型斑马鱼胚胎中。
[0071]
其中,将纯化的grna和cas9 mrna按照一定比例混合稀释,使得各自最终浓度为50ng/ul和100ng/ul,如ccdc57_grna(100ng/ul,3ul)和cas9 mrna(400ng/ul,3ul)按1:1混合,获得终浓度分别为50ng/ul和100ng/ul的ccdc57_grna和cas9 mrna混合物。再利用wpi公司p830显微注射仪将rna混合物注射到1-2细胞期的斑马鱼胚胎中,即可获得敲除f0胚胎。
[0072]
在其中一个实施例中,获得f0胚胎和f0胚胎培育至性成熟之间还包括对f0胚胎进行敲除检测,敲除检测包括:
[0073]
将f0胚胎培养至出膜阶段,挑取胚胎,逐个提取基因组进行第二次扩增,测序检测靶点序列情况。
[0074]
在f0胚胎培育至性成熟之前,有必要对f0胚胎进行敲除检测,检测f0胚胎的有效敲除情况。如将敲除f0胚胎培养至出膜阶段,随机挑取一定数量的胚胎,逐个提取基因组进行第二次扩增如pcr扩增,测序检测靶点序列情况。如可采用naoh法快速提取基因组。
[0075]
在其中一个实施例中,第二次扩增中以f0胚胎基因组为模板,正向引物的序列如seq id no.5所示,反向引物的序列如seq id no.6所示。
[0076]
靶点序列检测可利用ncbi在线blast软件设计检测靶点片段的引物,保持靶点距离上下游引物120bp以上,pcr产物控制在400-1000bp以内。检测引物序列具体如下:
[0077]
正向引物:tgctggaagctgtggacctg(seq id no.5)
[0078]
反向引物:ctgctcctctagggcattctg(seq id no.6)
[0079]
在其中一个实施例中,第二次扩增的步骤为:
[0080]
94℃下保持5分钟后,进入32个循环,每个循环经历95℃变性30秒、56℃退火30秒,72℃延伸30s,最后在72℃延伸5分钟,使产物充分延伸并完成加a尾过程。
[0081]
第二次扩增可采用pcr扩增,pcr体系每20ul包括10ul pcr mix、1ul如seq id no.5所示的正向引物(10um)、1ul如seq id no.6所示的反向引物(10um)、2ul基因组模板(如上述naoh法提取)。采用普通pcr程序进行扩增,具体程序如:94℃下保持5分钟后,进入32个循环,每个循环经历95℃变性30秒、56℃退火30秒,72℃延伸30s,最后在72℃延伸5分钟,使产物充分延伸并完成加a尾过程。整个扩增过程历时1小时30分钟左右,完成扩增后,取2ul样品进行电泳,确定扩出目的片段后则将剩下的样品送测序。
[0082]
在其中一个实施例中,筛选有效突变胚胎的步骤为:
[0083]
将f1胚胎培养至出膜阶段,挑取胚胎,逐个提取基因组进行第三次扩增,测序确定非三倍数突变的f1胚胎为有效突变胚胎。
[0084]
经3个月左右的培育,f0斑马鱼可与野生型斑马鱼进行交配,获得f1胚胎。待孵化出膜后,随机挑取一定数量的胚胎,采用naoh法快速提取基因组,即将单个胚胎对应加入50ul naoh溶液,浓度为50mm,置于95℃消化10-15分钟,振荡混匀,再加热5分钟后取出,放置冷却后加入5ul tris-hcl溶液(浓度为1m,ph=8.0),混匀即可作为pcr模板。经测序后确定f1胚胎中有无非三倍数,将包含有非三倍数突变的f1胚胎群体培育至成年,再经剪尾筛选非三倍数突变的f1个体,即:将成年f1群体逐个剪尾,一个样品对应加入100ul naoh溶
液,加热消化均匀后加入10ul tris-hcl溶液,混匀作为pcr模板。扩增出检测片段后测序确认基因型。
[0085]
在其中一个实施例中,培育至成年筛选可获ccdc57纯合敲除斑马鱼的步骤为:
[0086]
将具有相同非三倍数突变的f1斑马鱼进行交配,获得f2,经剪尾筛选可获得纯合非三倍数碱基插入/缺失的斑马鱼,即为纯合有效的ccdc57突变斑马鱼。
[0087]
同一条f0产出的f1,可能包含多种突变,需要筛选确认,将相同的突变归为一个品系,进行自交获得f2。同时f2是由1/4++、1/4
--
、1/2+-组成,其中1/4
--
才是纯合有效的突变体,因此在f2需要进一步筛选纯合子斑马鱼。
[0088]
实施例1
[0089]
1)构建ccdc57基因敲除斑马鱼
[0090]
1-1)从ncbi数据库(https://www.ncbi.nlm.nih.gov/gene/100318317)获取斑马鱼ccdc57基因序列(gene id:100318317);针对ccdc57基因第一个外显子区域设计靶点,根据靶点设计规则,选取以gg/ggg开头、结尾紧接ngg的16-24bp序列作为靶点,其序列具体如seq id no.1所示,为:ggaggaaagggacaaagagc;
[0091]
1-2)设计和合成用于扩展gdna模板的引物。通过直接合成长片段引物,将转录所需t7启动子序列、靶点序列和grna scaffold前20个碱基序列融合,设计为正向引物;将grna scaffold后20个碱基序列设计为反向引物。具体如seq id no.2和seq id no.3所示,为:
[0092]
正向引物:taatacgactcactatagggaggaaagggacaaagagcgttttagagctagaaatagc(seq id no.2);
[0093]
反向引物:agcaccgactcggtgccact(seq id no.3)。
[0094]
1-3)扩增和纯化gdna模板:以pmd18t-grna质粒为模板,以seq id no.2和seq id no.3为引物,用kod-fx高保真酶(phanta super-fidelity dna polymerase)进行扩增,配制50ul反应体系,包括25ul 2x pcr缓冲液、10ul 2mm dntps、1.5ul正向引物、1.5ul反向引物、2ul 400ng/ul模板质粒、1ul kod-fx高保真酶。采用三步法程序如:94
°
下保持2分钟后,进入32个循环,每个循环经历10秒98
°
变性,30秒56
°
退火,10秒68
°
延伸。
[0095]
pcr扩增完成后可获得t7启动子连接靶点和grna scaffold的gdna模板片段,通过电泳分离目的条带,进一步通过胶回收试剂盒(omega d2500-01)进行纯化回收,具体步骤如下:1)在紫外灯下将含有目的片段的琼脂糖凝胶切下转移至ep管中,并加入400μl binding buffer;2)将ep管置于65℃水浴锅中加热至凝胶块完全融化;3)将dna回收纯化柱与2ml收集管组装好,加入300μl binding buffer,以13000g室温离心1分钟,弃滤液;4)将步骤2)中的溶液加入回收柱中,以13000g室温离心1分钟,弃滤液;5)向回收柱中加入700ul洗脱液,以13000g室温离心1分钟,弃滤液。该步骤可重复1-2次;6)以15000g室温离心2分钟,弃滤液和收集管,将回收柱盖子打开,晾置2分钟。7)将纯化柱装在无rna酶的1.5mlep管上,加入30ul depc水,静置2-3分钟,以13000g室温离心1分钟。重复一次该步骤可提高回收效率;8)跑胶检测产物有无降解,测定浓度后将胶回收产物保存在-20℃冰箱。
[0096]
1-4)grna合成和纯化:以gdna为模板,利用t7高效转录试剂盒合成grna,合成步骤参照t7 rna高效合成试剂盒(transcriptaid t7 high yield transcription kit,k0441)说明:20ul合成体系包括4ul 5
×
transcript aid reaction buffer、8ul 2.5mm ntp mix、
2ul transcript aid enzyme mix、1ug ccdc57_gdna。37℃条件下反应2-4小时后,加入dna酶处理15分钟,加入30ul depc水和30ul 7.5m licl溶液,混匀后置于-20℃沉淀过夜。第二天以13200rmp 4℃离心10分钟,去上清,加入700ul 75%乙醇溶液洗涤后以13200rmp 4℃离心5分钟,去上清,打开盖子直至彻底晾干,加入50ul depc水溶解。经分光光度计测量浓度后,将ccdc57_grna稀释到100ng/ul,3ul每管分装保存于-80℃。
[0097]
1-5)cas9 mrna合成和纯化:
[0098]
cas9线性化模板制备:用fast-xbai内切酶线性化pt3.cas9-utrglobin质粒,50ul酶切体系包括5ul 10xbuffer、1ul xbai内切酶和5ug质粒,于37℃水浴酶切2小时。
[0099]
cas9线性化模板纯化:得到的线性化产物用酚氯仿抽提法提纯:50μl酶切体系补水至100μl,加入等体积的平衡酚/氯仿,混匀后以7500g离心5分钟,取上层水相,加入十分之一体积的naac和2倍体积无水乙醇,置于-20℃沉淀2h后,在4℃条件下离心,以13000g离心15分钟,去上清,加入700ul75%乙醇,以13000g离心5分钟,去上清留沉淀,打开ep管盖子晾干,加入depc水溶解,经分光光度计测浓度,保存于-20℃冰箱。
[0100]
cas9 mrna合成及纯化:
[0101]
以上步骤得到线性化cas9模板,利用t3 mrna合成试剂盒(mmessage mmachinetmt3 transcription kit,am1348)合成cas9 mrna,合成步骤参照试剂盒说明:20ul合成体系包括4ul 5
×
transcript aid reaction buffer、10ul 2
×
ntp mix、1ul transcript aid enzyme mix、1ug cas9线性化质粒。37℃条件下反应2小时,电泳检测mrna是否合成或降解。
[0102]
cas9 mrna纯化:将反应体系加入dna酶处理15分钟去除质粒模板,再加入30ul depc水和30ul 7.5m licl溶液,混匀后置于-20℃沉淀过夜。第二天以13200rmp 4℃离心10分钟,去上清,加入700ul 75%乙醇溶液洗涤后以13200rmp 4℃离心5分钟,去上清,打开盖子直至彻底晾干,加入50ul depc水溶解。经分光光度计测量浓度后,将cas9 mrna稀释到200ng/ul,3ul/管分装保存于-80℃冰箱。
[0103]
1-6)显微注射获得敲除f0胚胎:将ccdc57_grna(100ng/ul,3ul)和cas9mrna(400ng/ul,3ul)按1:1混合,获得终浓度分别为50ng/ul和100ng/ul的ccdc57_grna和cas9 mrna混合物,利用wpi公司p830显微注射仪将rna混合物注射到1-2细胞期的斑马鱼胚胎中,即可获得敲除f0胚胎。
[0104]
1-7)f0胚胎敲除效率估算:
[0105]
提取基因组:将敲除f0胚胎培养至出膜阶段,随机挑取15个胚胎,分成3组,采用naoh法快速提取基因组,即每组胚胎对应加入100ul naoh溶液,浓度为50mm,置于95℃消化10-15分钟,振荡混匀,再加热5分钟后取出,放置冷却后加入10ul tris-hcl溶液(浓度为1m,ph=8.0),混匀即可作为pcr模板。
[0106]
靶点序列检测:利用ncbi在线blast软件设计检测靶点片段的引物,保持靶点距离上下游引物120bp以上,pcr产物控制在400-1000bp以内。检测引物序列具体如下:
[0107]
正向引物:tgctggaagctgtggacctg(seq id no.5)
[0108]
反向引物:ctgctcctctagggcattctg(seq id no.6)
[0109]
pcr体系每20ul包括10ul pcr mix、1ul正向引物(10um)、1ul反向引物(10um)、2ul基因组模板(如上述naoh法提取)。采用普通pcr程序进行扩增,具体程序如:94℃下保持5分
钟后,进入32个循环,每个循环经历95℃变性30秒、56℃退火30秒,72℃延伸30s,最后在72℃延伸5分钟,使产物充分延伸并完成加a尾过程。整个扩增过程历时1小时30分钟左右,完成扩增后,取2ul样品进行电泳,确定扩出目的片段后则将剩下的样品送测序。
[0110]
2)ccdc57有效突变品系的筛选
[0111]
2-1)f1杂合有效突变体获得:经3个月左右的培育,f0斑马鱼可与野生型斑马鱼进行交配,获得f1胚胎。
[0112]
f1胚胎采用1-7)f0胚胎敲除效率估算步骤的方式进行提取基因组和靶点序列检测,通过pcr扩增出检测片段并测序。经测序后确定f1胚胎中有无非三倍数,将包含有非三倍数突变的f1胚胎群体培育至成年,再经剪尾筛选非三倍数突变的f1个体,即:将成年f1群体逐个剪尾,一个样品采用1-7)f0胚胎敲除效率估算步骤的方式进行提取基因组和靶点序列检测,通过pcr出检测片段后测序确认基因型。筛选出l1和l2两个在ccdc57设计的靶点区域呈现不同序列的品系。如图1所示,ccdc57敲除靶点设计及突变品系靶点序列:ccdc57敲除靶点设计在外显子上,具体序列如wt(wild type即野生)的灰色部分标注的序列,如seq id no.20所示。line1(l1)和line2(l2)为两个敲除品系,其中l1品系靶点序列丢失10个碱基(颜色较灰色浅的灰白部分),其序列如seq id no.21所示。l2品系靶点序列丢失4个碱基(颜色较灰色浅的灰白部分),其序列如seq id no.22所示。
[0113]
2-2)f2纯合有效突变体获得:将具有相同非三倍数碱基插入/缺失的f1斑马鱼进行交配,获得f2,经剪尾筛选可获得纯合非三倍数碱基插入/缺失的斑马鱼,即为纯合有效的ccdc57突变斑马鱼。剪尾筛选的具体方法与2-1)中的剪尾筛选方法相同。
[0114]
3)ccdc57敲除斑马鱼脊柱侧弯表型分析
[0115]
3-1)敲除斑马鱼外观特征收集:将野生型和l1品系斑马鱼浸泡于0.016%的三卡因溶液中使其麻醉,再置于体式显微镜下进行拍照。野生型及两个ccdc57敲除品系斑马鱼侧面观和背面观如图2所示:野生型斑马鱼体轴呈直线型,两个敲除品系斑马鱼体轴呈s型或波浪形。
[0116]
3-2)敲除斑马鱼骨骼特征收集:将野生型和l1品系斑马鱼浸泡于0.016%的三卡因溶液中使其麻醉,再置于冰上10分钟使其死亡,浸泡于4%的多聚甲醛溶液中固定2小时,转至100%甲醇溶液中洗脱10分钟,再置于100%甲醇溶液中保存。进行micro ct扫描前将保存于甲醇溶液中的样品取出,用pbs溶液洗脱2次,每次5分钟。之后将样品置于载台上进行扫描。如图3所示,micro ct显示野生型及l1品系斑马鱼的骨骼特征:e-f显示野生型斑马鱼骨骼呈直线型。g-l显示l1敲除品系斑马鱼骨骼呈s型或波浪形,其中g-h弯曲程度较轻,i-j弯曲程度中等,k-l弯曲程度严重。
[0117]
3-3)显微注射回补正常的ccdc57 mrna:设计引物(seq id no.7和no.8)将野生型斑马鱼ccdc57的cds序列(coding sequence、编码序列)扩增出来,连接到psp64载体上,进行体外转录合成野生型的ccdc57 mrna,纯化后用分光光度计测定浓度,分装保存于-80℃冰箱。预注射前将ccdc57 mrna取出,分别稀释至100ng/ul,50ng/ul,25ng/ul,采用3个浓度梯度进行注射,探索最佳浓度,最终确定25ng/ul的浓度具有挽救效果且致死率较低。正式回补注射实验中,取0.1ul 25ng/ul ccdc57 mrna加入显微注射针中,注射到200个受精卵中,其中100个为野生型受精卵,另100个为l1敲除品系的受精卵。待孵化出苗后,将注射和未注射的野生型及l1品系胚胎浸泡于0.016%的三卡因溶液中使其麻醉,置于体式显微镜
下进行拍照。结果如图4所示,显示注射回补正常的ccdc57 mrna能够有效挽救l1品系胚胎早期体轴向腹侧弯曲的表型,说明该敲除品系的脊柱侧弯表型确实由ccdc57蛋白缺失引起。
[0118]
3-4)荧光定量pcr检测肾上腺素及其受体表达变化:收集72hpf的野生型、l1突变品系和经回补l1突变品系胚胎,采用trizol法提取总rna,进一步逆转录合成cdna文库。以cdna文库为模板,以扩增gapdh、ccdc57、urp1、urp2、uts2r和uts2ra的特异性片段的引物(seq id no.9-seq id no.20)和sybr green预混液和去离子水配置pcr体系,利用abi的荧光定量pcr仪进行pcr扩增和荧光信号监测,最终以gapdh为内参,将不同样品的各个目标基因的表达量进行相对比较。结果如图5所示,显示缺失ccdc57蛋白的l1品系尾加压肽1和2及其受体的表达均降低,当回补过量正常的ccdc57 mrna后能显著提高l1品系尾加压肽1和2及其受体的表达,说明ccdc57通过影响尾加压肽信号通路调控体轴发育。
[0119]
需要说明的是,为方便描述,扩展gapdh、ccdc57、urp1、urp2、uts2r和uts2ra的特异性片段的对应正向引物和反向引物,分别用相应的片段和字母f、r表示,f:forward primer正向引物、r:reversed primer反向引物。如gapdhf表示gapdh的正向引物,具体序列如序列表seq id no.9所示,gapdhr表示gapdh的反向引物,具体序列如序列表seq id no.10所示。
[0120]
以上所述仅为本申请的较佳实施例而已,并不用以限制本申请,凡在本申请的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本申请的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1