一种用于迟缓爱德华氏菌检测的基因芯片探针组
本发明涉及食品安全和食源性致病微生物检测技术领域,特别是涉及一种用于迟缓爱德华氏菌检测的基因芯片探针组。
背景技术:
农产品和食品安全一直是全社会广泛关注的问题,而这其中,食源性致病微生物往往是导致食品安全问题的重要因素。用于食源性致病微生物检测的传统方法,一般存在着操作方法步骤多、检测时间长、结果灵敏度低等问题。随着生物技术的飞速发展,一些基于酶联免疫、生物传感器、荧光检测、pcr技术的新的食源性致病微生物检测技术也应运而生,检测方法呈现出多样性。在食品安全领域,开发食源性致病微生物快速、高效、准确的检测方法,是满足社会不断提高的食品安全要求的需要,是满足不同领域的检测研究的需要,这也进一步推动了现代化检测技术的革新。
基因芯片技术作为新型检测手段,结合了基因探针、荧光检测技术,在理论上可以在一次实验中检出所有潜在的致病原,也可以用同一张芯片检测某一致病原的多种遗传学指标,具有检测速度快、准确度高、微量化等特点,受到食品安全研究者的关注,是食品安全检测的有力工具。
迟缓爱德华氏菌(edwardsiellatarda)是革兰氏阴性细菌,在分类学上属于肠杆菌科(enterobacteriaceae)爱德华氏菌属(edwardsiella)。它是一种常见的水产病原菌,也是一类危害性较强的食源性致病微生物。在形态上,该细菌呈现短杆状,无荚膜,以周生鞭毛作为运动器官。爱德华氏菌感染的宿主类群比较广泛,从鱼类、爬行类、鸟类、哺乳动物和人类,危害性较大,其防治研究也有很多。在水产养殖领域,由爱德华氏菌引起的鱼类疾病,统称为鱼类的爱德华氏菌病(edwardsiellasis),该病流行面积大、发病率和死亡率高、能引起多种鱼类感染发病,是一种重要的病害。迟缓爱德华氏菌毒力因子的研究为揭示其致病过程提供了基础资料,主要涉及的毒力因子有铁载体、过氧化氢酶、溶血素等方面。研究发现,迟缓爱德华氏菌ⅲ型分泌系统与其致病性密切相关,其分泌的不同致病性蛋白可以作用于宿主细胞和相关分子。基于现有技术检测手段的发展,以及迟缓爱德华氏菌与致病性的关系,提供一种新的检测迟缓爱德华氏菌的方法,对于解决传统检测方法所存在的弊端意义重大。
技术实现要素:
本发明的目的是提供一种用于迟缓爱德华氏菌检测的基因芯片探针组,以解决上述现有技术存在的问题,通过选取迟缓爱德华氏菌ⅲ型分泌系统相关基因作为靶序列,设计特异性检测探针,为准确检查致病性迟缓爱德华氏菌提供新的技术方法。
为实现上述目的,本发明提供了如下方案:
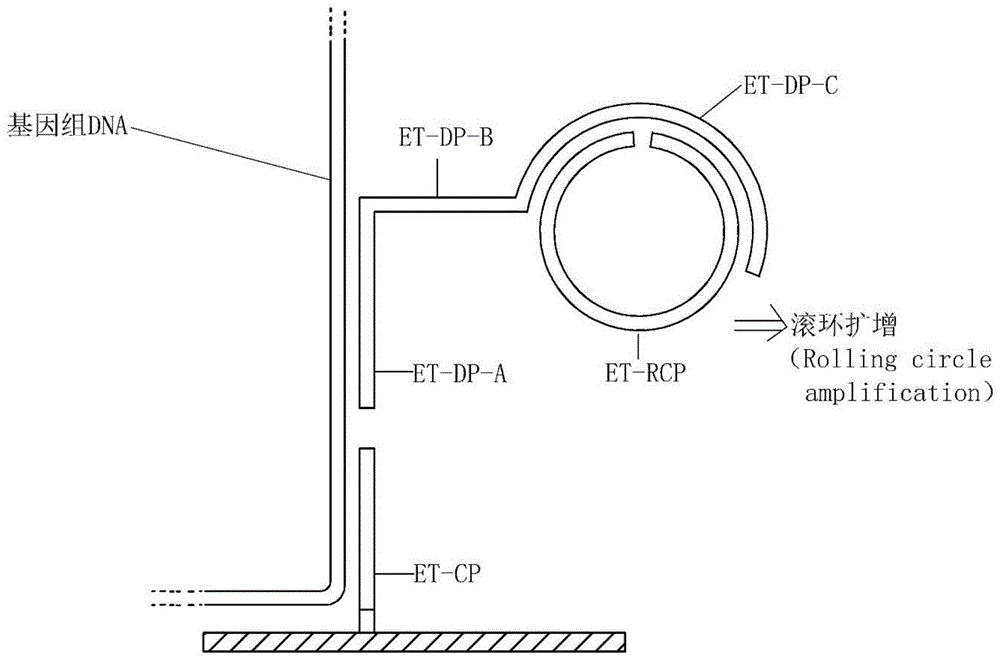
本发明提供一种用于迟缓爱德华氏菌检测的基因芯片探针组,包括捕获探针et-cp、检测探针et-dp、滚环探针et-rcp,其中所述检测探针et-dp是由et-dpa,et-dpb和et-dpc按照5’-3’方向依次连接而成。
优选的是,所述捕获探针et-cp和所述检测探针et-dp序列包括如下任意一组所示的序列:
(1)et-cp1(seqidno:1):5’-nh2-tttttttgcgtcgatctgattagcca-3’;
et-dp1:5’-cctgctgggagtttctggatgtcagaacacacgggatacagga-3’,其中,et-dp1a((seqidno:2):cctgctgggagtttctg与迟缓爱德华氏菌基因组dna互补;et-dp1b(seqidno:3):gatgtc;et-dp1c(seqidno:4):agaacacacgggatacagga,且与所述滚环探针et-rcp的5’端和3’端序列互补,同时是所述滚环探针et-rcp的引物和连接区域;
(2)et-cp2(seqidno:5):5’-nh2-tttttttccttaatcacgtcgag-3’;
et-dp2:5’-ctcccctttatccagc-acttac-agaacacacgggatacagga-3’,其中,et-dp2a(seqidno:6):ctcccctttatccagc与迟缓爱德华氏菌基因组dna互补;et-dp2b(seqidno:7):acttac;et-dp2c(seqidno:8):agaacacacgggatacagga,且与所述滚环探针et-rcp的5’端和3’端序列互补,同时是所述滚环探针et-rcp的引物和连接区域;
(3)et-cp3(seqidno:9):5’-nh2-tttttttcatggtcttctggatctgca-3’;
et-dp3:5’-gctgcgcctgggtgacga-gatgtc-agaacacacgggatacagga-3’;其中,et-dp3a(seqidno:10):gctgcgcctgggtgacga与迟缓爱德华氏菌基因组dna互补;et-dp3b(seqidno:11):gatgtc;et-dp3c(seqidno:12):agaacacacgggatacagga,且与所述滚环探针et-rcp的5’端和3’端序列互补,同时是所述滚环探针et-rcp的引物和连接区域;
(4)et-cp4(seqidno:13):5’-nh2-gcgtcgatctgattagcca-3’;
et-dp4:5’-cagacgttataggactgacttacagaacacacgggatacagga-3’;其中,et-dp4a(seqidno:14):cagacgttataggactg与迟缓爱德华氏菌基因组dna互补;et-dp4b(seqidno:15):acttac;et-dp4c(seqidno:16):agaacacacgggatacagga,且与所述滚环探针et-rcp的5’端和3’端序列互补,同时是所述滚环探针et-rcp的引物和连接区域;
所述滚环探针et-rcp(seqidno:17):5’-cgtgtgttctattctactgtattctgtatgtctccgtctgcctgtcacctgtgtatctttgtcccgtcagtcctgtatcc-3’,其中所述滚环探针et-rcp5’和3’序列与所述et-dp1c、et-dp2c、et-dp3c和et-dp4c序列互补。
进一步地,et-dpb、et-dpc和et-rcp不含有迟缓爱德华氏菌基因组dna序列。
优选的是,所述et-cp1、et-cp2、et-cp3、et-cp4序列与迟缓爱德华氏菌基因组dna互补;所述et-dp1a、et-dp2a、et-dp3a、et-dp4a序列与迟缓爱德华氏菌基因组dna互补;et-cp系列探针中的et-cp1、et-cp2、et-cp3、et-cp4序列,与et-dp系列探针中的et-dp1a、et-dp2a、et-dp3a、et-dp4a序列在迟缓爱德华氏菌基因组dna存在时,相应的在t4dna连接酶作用下连接(即:et-cp1与et-dp1a、et-cp2与et-dp2a、et-cp3与et-dp3a、et-cp4与et-dp4a连接)。上述迟缓爱德华氏菌基因组dna为ncbi公开的dna基因组,也可以是其他任一项细菌的dna基因组。
进一步地,et-cp探针5′端修饰有氨基,5′端添加7个t碱基,作为探针与基片的连接区。
所述基因芯片探针组序列还包括阴性对照探针et-nc和阳性对照探针et-pc,et-nc和et-pc序列不含有与迟缓爱德华氏菌基因组dna序列互补的序列;
进一步地,et-nc和et-pc的5′端修饰氨基,et-pc的3′端修饰biotin;et-nc和et-pc的5′端添加11个t碱基;et-nc和et-pc序列分别如5′-seqidno.18-biotin-3′和5′-seqidno.19-3′所示。
本发明还提供根据所述的基因芯片探针组检测迟缓爱德华氏菌的方法,包括如下步骤:
步骤(1),探针设计:根据待检测样品中检测的迟缓爱德华氏菌的基因序列与ncbi数据库中迟缓爱德华氏菌的基因组dna进行比对,选择高度保守性序列作为探针,所述探针分别具有所述捕获探针et-cp、检测探针et-dp、滚环探针et-rcp的碱基序列;
步骤(2),探针合成:人工合成所述捕获探针et-cp、检测探针et-dp、滚环探针et-rcp的碱基序列,制备目的探针;
步骤(3),芯片点制:把所合成的5’-氨基修饰的所述捕获探针et-cp与芯片点样液混合,进行点阵排布,点制在醛基基片上,制备成微阵列;
步骤(4),芯片杂交:把所述检测探针et-dp、滚环探针et-rcp与迟缓爱德华氏菌基因组dna混合,经过变性处理,加到所述微阵列上,进行退火反应和连接反应;
步骤(5),滚环扩增:将退火的微阵列进行洗脱,加入含有biotin-dutp的滚环扩增反应溶液,进行滚环扩增,加入链亲和素与滚环扩增产物结合;
步骤(6),结果扫描:采用荧光仪扫描芯片,若产生荧光信号即为阳性结果,所述待测样品中有迟缓爱德华氏菌;若无荧光信号即说明结果为阴性,待测样品中没有迟缓爱德华氏菌。
优选的是,步骤(4)中,所述退火反应为95℃变性5min,50℃杂交反应4h。
优选的是,连接反应系包括:t4dna连接酶1μl、10×t4缓冲液2.5μl、无菌ddh2o21.5μl;
连接条件为:35-40℃连接反应20-40min。
优选的是,步骤(5)中滚环扩增反应体系包括:dna聚合酶1μl、10×dna聚合酶缓冲液2.5μl、dntpmixture2μl、biotin-dutp0.2μl、100×bsa0.5μl、无菌ddh2o18.8μl;
滚环扩增条件为:25-35℃密闭扩增1.5-3.5h。
本发明还提供根据所述的基因芯片探针组在制备检测迟缓爱德华氏菌产品中的应用。
本发明公开了以下技术效果:
(1)本发明提供的迟缓爱德华氏菌基因芯片检测探针组用于基因芯片方法检测迟缓爱德华氏菌,具有高特异性、不依赖抗体等优点。
(2)本发明采用滚环扩增(rca)技术和芯片技术相结合,且线性rca对模板的扩增能力高达100000倍,具有非常好的灵敏度,能够实现检测到单分子水平。
(3)本发明中基于rca是一种等温核酸扩增技术,不需要热循环仪完成扩增,为商业化应用和推广提供了广阔的前景。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为利用基因芯片探针组序列检测迟缓爱德华氏菌的检测原理。
具体实施方式
现详细说明本发明的多种示例性实施方式,该详细说明不应认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
应理解本发明中所述的术语仅仅是为描述特别的实施方式,并非用于限制本发明。另外,对于本发明中的数值范围,应理解为还具体公开了该范围的上限和下限之间的每个中间值。在任何陈述值或陈述范围内的中间值以及任何其他陈述值或在所述范围内的中间值之间的每个较小的范围也包括在本发明内。这些较小范围的上限和下限可独立地包括或排除在范围内。
除非另有说明,否则本文使用的所有技术和科学术语具有本发明所述领域的常规技术人员通常理解的相同含义。虽然本发明仅描述了优选的方法和材料,但是在本发明的实施或测试中也可以使用与本文所述相似或等同的任何方法和材料。本说明书中提到的所有文献通过引用并入,用以公开和描述与所述文献相关的方法和/或材料。在与任何并入的文献冲突时,以本说明书的内容为准。
在不背离本发明的范围或精神的情况下,可对本发明说明书的具体实施方式做多种改进和变化,这对本领域技术人员而言是显而易见的。由本发明的说明书得到的其他实施方式对技术人员而言是显而易见的。本申请说明书和实施例仅是示例性的。
关于本文中所使用的“包含”、“包括”、“具有”、“含有”等等,均为开放性的用语,即意指包含但不限于。
本发明公开了一种用于基因芯片检测迟缓爱德华氏菌的探针组,属于食品安全问题中的食源性致病微生物检测技术领域。所采用的检测探针组涉及:捕获探针et-cp、检测探针et-dp、滚环探针et-rcp。此探针组序列根据genbank中迟缓爱德华氏菌的全基因组序列中的毒力相关基因设计而成,以上述探针组开发的基因芯片et-rca检测迟缓爱德华氏菌的方法具有灵敏度高、特异性强和准确性高等特点,可以用于实验室研究和食品安全、水产养殖、海产品质量安全检测以及海洋环境监测等方面。
本发明提供用于基因芯片检测迟缓爱德华氏菌的特异性探针组是根据迟缓爱德华氏菌基因组序列中的毒力相关基因设计而成。
本发明提供用于基因芯片检测迟缓爱德华氏菌的探针组是:
捕获探针et-cp和检测探针et-dp序列有4组,每个et-cp和相应et-dp的a组探针序列与迟缓爱德华氏菌基因组dna序列互补,et-cp和相应et-dp的a组探针序列在迟缓爱德华氏菌基因组dna序列存在时,可被t4连接酶催化发生连接反应。
每个et-cp探针5′端修饰有氨基,5′端添加7个t碱基,作为探针与基片的连接区。
检测探针et-dp从5’到3’依次由a、b和c三段序列组成。其中,et-dpa序列与迟缓爱德华氏菌基因组dna序列互补,检测时et-dpa和基因组dna结合;et-dpb是et-dpa和et-dpc的连接区;et-dpc序列与滚环探针et-rcp的5′端和3′端序列互补,也是滚环探针et-rcp的引物和连接区。
探针组序列中,et-dpb、et-dpc和et-rcp等三组序列不含迟缓爱德华氏菌基因组dna序列。阴性对照探针et-nc和阳性对照探针et-pc序列不含有与迟缓爱德华氏菌基因组dna序列互补的序列,et-nc和et-pc的5′端修饰氨基,et-pc的3′端修饰biotin。
为了进一步说明所设计的探针组检测迟缓爱德华氏菌的方法及其应用,以下将以实施例的方式进行说明。
实施例1利用基因芯片探针组检测迟缓爱德华氏菌的方法验证
本实施例中所用迟缓爱德华氏菌(edwardsiellatarda)、大肠埃希氏菌(escherichiacoli)、沙门氏菌(salmonellasp.)、嗜水气单胞菌(aeromonashydrophila)、腐败希瓦氏菌(shewanellaputrefaciens)、假单胞菌(pseudomonassp.)和枯草芽孢杆菌(bacillussubtilis)均为本实验室保藏。
(1)采用5%的戊二醛磷酸缓冲液(0.1mph7.4)处理硅烷化的玻片,随后依次用丙酮、乙醇、去离子蒸馏水彻底清洗,干燥后得到醛基修饰的玻片,置于4℃保存备用。
(2)生物公司合成序列表中seqidno:1-seqidno:19所示序列及特征合成检测探针。
(3)使用te缓冲液将et-cp、et-pc和et-nc配置为100μm的贮存液,保存于-20℃备用。点样前,使用te缓冲液将100μmet-cp、et-pc和et-nc稀释为20μm,再用50%点样液稀释至2μm,瞬时离心混匀,然后取至少35μl至384微孔板中,将384微孔板置于点样仪中的吸样位置进行点样。
(4)点样后,将玻片置于密闭湿盒中,湿盒中提前放置pbs溶液浸湿的滤纸,37℃水浴过夜(大于12h),去离子水洗涤2min、0.2%sds洗涤5min、去离子水洗涤2min、0.15%~0.3%硼氢化钠溶液处理5min、去离子水洗涤3遍,2min/遍、玻片离心机离心甩干,储存于4℃备用。
(5)先用细菌dna提取试剂盒提取迟缓爱德华氏菌、大肠埃希氏菌、沙门氏菌、嗜水气单胞菌、腐败希瓦氏菌、假单胞菌和枯草芽孢杆菌(上述菌株菌来源于徐州工程学院微生物实验室)的基因组dna,然后又将基因组dna分别与et-dp(终浓度0.05μm)与et-rcp(5μl)加入ep管杂交液中,在95℃变性5min,将25μl杂交反应体系迅速转移到贴于醛基基片的25μl基因结构(geneframe)中,盖好盖玻片,置于杂交炉中50℃杂交反应4h。
(6)杂交反应结束后,使用去离子水洗涤1min,干燥。
(7)按如下表1的标准加入试剂,在37℃进行连接反应30min,将25μl连接反应体系贴于基因结构中。连接反应后,使用去离子水洗涤1min,吹干。
表1连接反应体系
(8)醛基基片的固相et-rca体系,反应体系如下表2所示:
表2固相et-rca反应体系
将配制好的25μlet-rca扩增反应体系转移到上述基因结构中,将玻片置于30℃杂交炉的密闭湿盒中,扩增2h。
(9)待et-rca反应结束,去除基因结构,用无菌ddh2o洗涤玻片2min。
(10)瞬时离心玻片,加入1×封闭液浸没玻片,室温条件下,在摇床慢速摇动封闭30min。
(11)按照链亲和素(streptavidin-irdye800):pbs=1:15000的比例,配制streptavidin-irdye800的pbs溶液,浸没玻片,室温条件下,在摇床慢速摇动结合30min。
(12)无菌ddh2o洗涤玻片10min,瞬时离心玻片,使用odyssey800nm激光通道扫描信号。
结果显示,阳性探针(et-pc)荧光信号正常,在检测的微生物中,迟缓爱德华氏菌完成滚环扩增,显示荧光信号正常。阴性探针(et-nc)和其它待测微生物样品没有检测到荧光信号,说明不能进行滚环扩增。实验结果表明,本发明的检测迟缓爱德华氏菌特异性探针组检测序列在迟缓爱德华氏菌检测中具有较高的特异性,可以从样品中检测到迟缓爱德华氏菌。
以上所述的实施例仅是对本发明的优选方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
序列表
<110>徐州工程学院
<120>一种用于迟缓爱德华氏菌检测的基因芯片探针组
<160>19
<170>siposequencelisting1.0
<210>1
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>1
tttttttgcgtcgatctgattagcca26
<210>2
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>2
cctgctgggagtttctg17
<210>3
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>3
gatgtc6
<210>4
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>4
agaacacacgggatacagga20
<210>5
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>5
tttttttccttaatcacgtcgag23
<210>6
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>6
ctcccctttatccagc16
<210>7
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>7
acttac6
<210>8
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>8
agaacacacgggatacagga20
<210>9
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>9
tttttttcatggtcttctggatctgca27
<210>10
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>10
gctgcgcctgggtgacga18
<210>11
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>11
gatgtc6
<210>12
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>12
agaacacacgggatacagga20
<210>13
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>13
gcgtcgatctgattagcca19
<210>14
<211>17
<212>dna
<213>人工序列(artificialsequence)
<400>14
cagacgttataggactg17
<210>15
<211>6
<212>dna
<213>人工序列(artificialsequence)
<400>15
acttac6
<210>16
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>16
agaacacacgggatacagga20
<210>17
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>17
cgtgtgttctattctactgtattctgtatgtctccgtctgcctgtcacctgtgtatcttt60
gtcccgtcagtcctgtatcc80
<210>18
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>18
tttttttttttgcatatgagcgtctcgacttt32
<210>19
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>19
tttttttttttcgttactagtacatgcttcggttt35
- 还没有人留言评论。精彩留言会获得点赞!