用于可靠的无创胚胎植入前基因检测的方法与流程
本发明涉及一种或多种用于无创胚胎植入前基因检测的准确方法。具体地,本发明涉及用于扩增溶液中的微量dna的系统和方法,以及分析来自体外培养胚胎的培养液中的生物样本以确定胚胎中遗传病的易感性的连锁分析方法。
背景技术:
1、如本领域已知的胚胎植入前基因检测(pgt)(handyside等人,1989和1990,针对第一胎出生)已被广泛用于临床体外授精(ivf)以避免选择患有单基因病(monogenic disease)(kerem等人,1989;handyside等人,1992;liu等人,1994;harton等人,1996;ao等人,1998;sermon等人,1998;xu等人,1999;hussey等人,1999;ray等人,2000;de rycke等人,2001;moutou等人,2001;verlinsky等人,2001;sermon等人,2001;girardet等人,2003;fiorentino等人,2006;kahraman等人,2014)、非整倍体(munné等人,1993;wilton等人,2001;wells等人,2002;treff等人,2010;gutiérrez-mateo等人,2011;yang等人,2012;scott等人,2013;forman等人,2013;wells等人,2014;rubio等人,2017)、结构变异(conn等人,1998;scriven等人,1998;coonen等人,2000;munné等人,2000;escudero等人,2003;melotte等人,2004;le caignec等人,2006;traversa等人,2010;fiorentino等人,2010;fiorentino等人,2010;rius等人,2011;fiorentino等人,2011;treff等人,2016;zhang等人,2017;tan等人,2013;chow等人,2018)或多基因病(polygenic disease)(treff等人,2019;kumar等人,2022)的胚胎。据报道,使用一种基于ngs的检测,可以同时进行染色体拷贝数分析和连锁分析以及针对突变的靶向单体型分析(yan等人,2015)。目前,对于pgt,需要进行滋养外胚层(te)活检(dokras等人,1990;handyside等人,1990;kokkali等人,2005),这种方法是有创的并且受制于采样偏差。
2、使用囊胚腔液(bf)中的游离dna进行扩增和基因检测被报道为微创胚胎植入前基因检测(palini 2013;gianaroli等人,2014;tobler等人,2015;magli等人,2016;zhang等人,2016;shangguan等人,2017;capalbo等人,2018;tsuiko等人,2018;magli等人,2018)。
3、本发明描述了一种使用废弃的胚胎培养液中的游离dna来对非整倍体和单基因病的完全无创的胚胎植入前基因检测方式(galluzzi等人,2015;wu等人,2015;xu等人,2016;shamonki等人,2016;feichtinger等人,2017;lane等人,2017;liu等人,2017;ho等人,2018;vera-rodriguez等人,2018;capalbo等人,2018;fang等人,2019;huang等人,2019;rubio等人,2019;yeung等人,2019;jiao等人,2019;ou等人,2022)。
4、微创胚胎植入前基因检测和无创胚胎植入前基因检测是避免胚胎植入前基因检测技术中的活检损伤的两种主要方法。胚胎培养方法和废培养液(scm)收集方法,以及用于微量游离dna的单细胞全基因组扩增(scwga)方法,可以直接影响结果。来自囊胚腔液(bf)和废弃的胚胎培养液的样本的扩增成功率在不同的临床中心有很大差异。当使用相同的扩增方法时,bf中的扩增成功率远低于废培养液(scm)中的扩增成功率,如galluzzi等人(2015)报道的62.5% scm vs 44.4% bf,capalbo等人(2018)报道的89.7% scm vs27.4% bf。虽然scm的扩增成功率高于bf,但scm的结果受到母源污染以及胚胎发育培养液中外源蛋白的添加的影响。研究表明,母体血中的母体dna片段比胎儿dna片段更长(chan等人,2004),并且扩增技术对片段长度的偏倚使得其对胎儿dna扩增低效。此外,在维持临床检测用途的产量的同时,确保扩增的准确度也很重要。目前,可用的商业单细胞扩增试剂盒基于dop-pcr、mda或malbac技术,并且这些商业试剂盒在扩增效率和保真度方面有差异(huang等人,2015)。这些方法都是指数式扩增,会引起较高的假阴性和假阳性。在2017年,发明了一种新型扩增方法,即线性扩增(chen等人,2017,us10,894,980,称为“lianti”)。snp检测的假阳性率远低于其它方法。然而,这些试剂盒并未针对培养液中所含的dna片段长度特征和高蛋白质含量进行改善,而只是扩大了反应体系,导致培养液的扩增效率不高。迫切需要一种高效且准确的扩增方法以在scm或/和bf系统中获得更多胚胎dna信息。然而,原始lianti方法的产量太低,并且可重复性较低,由于胚胎较珍贵,因此并不适合临床应用。
5、就分析而言,存在许多关于非整倍体的无创胚胎植入前基因检测(nipgt-a)的报道(leaver等人,2020中综述),但仅有几篇关于无创胚胎植入前单基因疾病基因检测(nipgt-m)的报告(galluzzi等人,2015;zhang等人,2016;shangguan等人,2018;wu等人,2015;liu等人,2017;capalbo等人,2018;ou等人,2022)。在用scm进行nipgt-m检测的报道中,与滋养外胚层活检结果相比,成功扩增的scm中的基因型一致性也有很大差异,从21%到88%不等(capalbo等人,2018;liu等人,2017;ou等人,2022)。这些论文中并未提及假阳性和假阴性率这些评估检测方法准确度的重要指标。然而,基于文献报道的基因型一致率,可以判断其假阴性和假阳性率非常高,这些结果对无创pgt-m在临床上应用是不利的(capalbo等人,2018;cimadomo等人,2020)。pgt-m评估的最重要标准不是基因型一致率,而是误诊率。eshre pgt联盟公布的误诊率非常低(<0.1%)(de rycke等人,2017)。应当对新型检测方法的误诊风险进行评估,并与传统pgt-m进行比较。
6、除了检测疾病相关突变位点之外,已报道的论文中使用的分析方法是连锁分析方法。在等位基因脱扣(allele drop-out,ado)率低于5%时,建议应用突变位点上下游的至少各两个有效信息str标记或snp位点(欧洲人类生殖和胚胎学协会(eshre)2020)。ou等人使用了4个含有有效信息的snp位点,liu等人使用了10个有效snp位点。由于scm或bf的ado率远高于5%,因此仅仅使用少数有效位点进行连锁分析会很容易地导致误诊。所使用的有效snp标记与致病基因的距离还要考虑发生重组的风险。
7、此外,母源污染也会导致诊断错误。广泛使用的连锁分析方法基于lander-green算法及其变体,该算法不考虑母源污染并且需要高质量的测序数据(lander等人,1987;kruglyak等人,1996;idury等人,1997;kruglyak等人,1998;abecasis等人,2002)。直接应用这些方法会导致高误诊率(capalbo等人,2018;cimadomo等人,2020)。将与母体细胞的混合或污染问题纳入考虑的连锁分析方法很少(fan等人,2002;nabieva等人,2020)。然而,它们需要比来自培养液的那些测序数据更高质量的测序数据,并且它们的主要应用是基于妊娠期间母亲的血液样本或胎盘或羊水中的细胞(fan等人,2002;nabieva等人,2020)。这些现有方法在应用于来自培养液的测序数据时,将会导致更多的诊断错误。nipgt-m的高假阳性和假阴性率主要与scm中初始dna含量低和母源污染的存在以及ado率高有关,这使得所有这些现有的nipgt-m方法都不适于临床应用。将nipgt-m应用于临床ivf的关键在于该技术的准确度可达到当前pgt-m的水平。对于nipgt-m,通过检测胚胎中携带致病突变的等位基因的存在,准确率应当大于97%。据我们所知,目前还没有对于无创诊断的这样一种连锁分析方法。其最关键的解决方案是对在母源污染存在下且具有高ado率的样本开发出更准确的连锁分析方法。
技术实现思路
1、本发明提供了一种用于扩增溶液中低输入量dna的dna扩增方法,以及一种基于贝叶斯模型(bayesian model)的新型连锁分析方法,以在存在母源污染和单倍型丢失(haplotype loss)的情况下执行pgt-m和胚胎植入前多基因疾病的基因检测(pgt-p)分析。
2、本公开的实施方案提供了用于对有限样本(例如,临床废培养液、囊胚腔液、单细胞或有限数量的细胞)进行dna裂解、全基因组扩增(wga)并进行二代测序(ngs)的改进的湿实验方法,以实现高覆盖率、高准确度,以及高扩增成功率。本文所述的系统和方法旨在在具有高蛋白质含量的复杂系统中有效地扩增不同片段分布的dna。
3、该方法是根据本领域所述的lianti方法改良的,以有效地提高扩增效率,实现更高的产物产量。lianti的测序数据展示出高覆盖率(鉴于测序深度)、高准确度(如由低假阳性率所表明的),以及低嵌合率。单细胞基因组扩增期间的高准确度是单核苷酸变异(snv)检测的关键要求,而低嵌合率是结构变异(sv)检测的关键要求,这两者对于许多基于单细胞基因组学的应用均非常重要。
4、例如,在一些实施方案中,本公开提供了一种在扩增前裂解临床样本的方法,以及一种使用基于转座子的方法均匀且有效地扩增全基因组样本的方法,包括a)去除临床样本中的血清蛋白以及改变裂解温度和时间以使dna尽可能多的暴露。在一些实施方案中,游离dna的样本是在废培养液中。在一些实施方案中游离dna的样本是在囊胚腔液中。本公开的方面包括b)改善后续扩增技术,包括但不限于调节蛋白酶和/或转座体浓度、添加用于逆转录的dna引物、增加第二链dna扩增步骤中的扩增周期等。因此,可以实现准确的扩增,并且可以增加产量以满足测序分析。扩增产物可用于下一代测序的文库制备方法,或用于致病基因座区域的靶向扩增,或用于芯片测序。
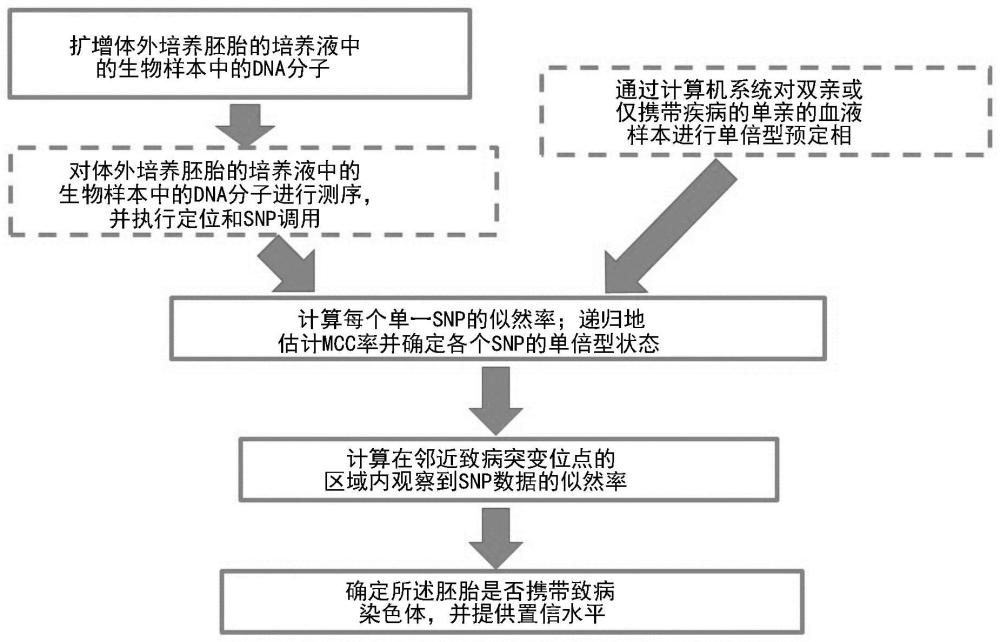
5、根据一个方面,本公开提供了一种解决单倍型大片段丢失(large fraction lossof haplotype,lfloh)率高、覆盖率低,以及母源细胞污染(maternal cellcontamination,mcc)率高的问题的连锁分析方法,这些问题可能阻碍在致病突变位点处遗传双亲染色体的推断。根据本公开,本文所述的连锁分析与现有方法相比增加了snp或标记的数量以解决高ado率,并且进一步提供了一套基于贝叶斯分析的连锁分析方法,包括:a)结合snp的mcc率和单倍型状态,计算每个单一snp的似然率;(b)递归估计每个snp的mcc率和单倍型状态;(c)计算在邻近致病突变位点的区域内观察到snp数据的似然率;(d)确定胚胎是否携带致病染色体并提供置信水平。
6、本公开的实施方案提供了一种对具有低输入量dna的废培养液和/或囊胚腔液和/或样本的准确连锁分析。本公开提供了一种用于扩增样本(如废培养液或单细胞)中的较低量dna的扩增方法。本公开还提供了一种用于辅助pgt-m和pgt-p疾病携带分析以提高准确度并降低误诊风险的贝叶斯模型,该连锁分析结合snp的mcc率和单倍型状态,迭代地计算遗传携带疾病染色体与无疾病染色体的似然比,并输出疾病携带分析的置信水平。分析的工作流程显示于图1中。本文所述的方法使输出疾病携带分析的假阴性率最小化,并确定携带健康染色体的可能性最高的胚胎。贝叶斯模型采用测序数据以及由merlin算法定相的双亲单倍型作为输入。
7、为了提高数据质量,过滤掉质量极低的snp。在计算似然比之前估计了几个参数:测序错误率、mcc率以及单倍型状态。mcc率被定义为来源于母本染色体的dna片段的比率。单倍型状态被定义为各个snp处dna的真实双亲来源,并且分为“仅父本染色体”、“仅母本染色体”或“双亲染色体”。例如,仅父本染色体表明,在该snp处检测到的dna仅来自父亲。mcc率和单倍型状态被重复校准直至收敛(convergence)。
8、然后,递归地计算在距致病突变位点特定物理距离内测序数据的似然率。为了完成递归,确定相邻snp之间的重组概率,并计算单个snp的似然率。重组概率可以从长期存在的decode数据集获得,而单一snp似然率从二项式模型获取,其参数由估计的测序错误率、mcc率以及单倍型状态确定。
9、与先前发表的论文中所考虑的固定snp的数量不同,本公开的实施方案描述了从致病突变位点逐个添加snp并计算每个snp子集的对数似然比,从而产生了对数似然比与末端snp的物理距离的关系曲线。典型地,该曲线从纵坐标接近零的点开始,并且随着添加足够的snp而最终稳定在一个平台。基于该曲线的特性,确定遗传了哪条染色体,并将我们的疾病携带分析的置信水平分为四类:高置信度、中等置信度、可能以及不确定。使用该方法,可以在母源污染的存在下准确地区分等位基因。该方法可以使nipgt-m的检测准确度达到100%。
- 还没有人留言评论。精彩留言会获得点赞!