一种我国小麦根部禾谷多黏菌分型及其鉴定的方法与流程
本发明涉及农作物病毒病害传播介体的分子检测技术,属于农业科学技术领域。
背景技术:
禾谷多黏菌(Polymyxa graminis)是一种禾本科植物根部的专性寄生低等真核生物,自身对植物的生长不会产生明显的危害,但是它作为至少11种病毒的传播介体,传播的病毒可以在小麦、大麦、水稻等作物上引起多种病毒病害。因其活体寄生特性,不能进行室内离体培养,休眠孢子壁厚,抗逆性强,能在土壤中存活十多年,导致对禾谷多黏菌的研究十分困难,进展缓慢。通过世界范围内的大量采样和鉴定,明确了禾谷多黏菌传播的麦类病毒主要隶属于Bymovirus属和Furovirus属。在我国的冬麦区,禾谷多黏菌主要传播小麦上的小麦黄花叶病毒(WYMV)和中国小麦花叶病毒(CWMV),引起小麦植株分蘖减少,矮化发黄等症状,发病严重时可导致减产50-70%,危害巨大。
多黏菌下有两个种,禾谷多黏菌和甜菜多黏菌。这两个物种最初主要是通过形态及宿主区分,禾谷多黏菌主要侵染禾本科作物,而甜菜多黏菌的寄主则是藜科植物。由于禾谷多黏菌基因组信息未知,目前其种下的分类主要依赖于核糖体DNA(rDNA)的ITS1和ITS2区域序列。1994年Ward等利用限制性片段长度多态性(RFLP)技术,首次通过ITS区分了这两种多黏菌,同时通过序列分析发现禾谷多黏菌存在两种核糖体分型。随后,Ward和Adams在印度高粱上发现了禾谷多黏菌的第三种分型。随着越来越多的禾谷多黏菌ITS的揭示,Ward等将非洲地区、亚洲日本地区样品和其余地区已知的禾谷多黏菌ITS序列进行系统进化分析后发现目前共有6种禾谷多黏菌分型(I型–VI型)。2003年Legreve及她同事的研究则表明禾谷多黏菌的分型依据除了ITS序列外,不同地区来源的禾谷多黏菌分型对宿主及温度也有不同要求,在分型中也需予以考虑。来源于印度的禾谷多黏菌在高粱上完成整个生活史的最佳温度是27-30℃,23-26℃休眠孢子的萌发及各个阶段的发育速度减缓,19-22℃侵染不显著,19℃以下无侵染。而温带地区来源的禾谷多黏菌在大麦上的最佳温度是15-18℃,19-22℃侵染率明显降低,22℃以上侵染不显著。因此,Legreve根据其研究结果对禾谷多黏菌进行分型的时候除考虑ITS序列外,还加入了宿主及温度因素,将禾谷多黏菌分为了五种类型(f.sp.temperate,f.sp.tepida,f.sp.tropicalis,f.sp.subtropicalis,f.sp.colombiana)。虽然目前这两个分类系统未统一,但其对应关系非常明显。
虽然早在上世纪80年代已经明确我国冬小麦区的小麦黄花叶病的传播媒介是土壤中的禾谷多黏菌,但对禾谷多黏菌的研究极少,我国禾谷多黏菌存在哪些分型目前也未知,同时不清楚其分型与传播的小麦病毒种类是否有关。因此,明确我国小麦黄花叶病爆发区域禾谷多黏菌的分型有助于为土传小麦黄花叶病的防控提供依据。
技术实现要素:
本研究提供了一种中国禾谷多黏菌分型快速鉴定和区分的方法。根据禾谷多黏菌核糖体DNA序列,利用已知的禾谷多黏菌引物对我国的冬麦区小麦根部DNA进行扩增,筛选适用于我国禾谷多黏菌分型的引物,同时针对我国特有的禾谷多黏菌分型设计特异性的反向引物ITS.CN,并对PCR的反应体系及扩增条件作大量优化,最终获得能稳定扩增和鉴别我国两种禾谷多黏菌分型的方法。
本发明的研究人员首先利用欧洲地区检测禾谷多黏菌核糖体DNA(rDNA)ITS2区域的通用引物PSP2(f)和ITS4(r)对源于我国小麦黄花叶病区的小麦根部基因组DNA进行PCR扩增(PCR产物约250bp),扩增产物测序并根据ITS2区域进行系统进化分析,初步明确我国冬麦区禾谷多黏菌至少存在两种分型,一种和已报道的I型具有一定同源性,为我国特有的分型,命名为I-CN型菌,另一种为II型菌。为进一步得到禾谷多黏菌rDNA的完整ITS序列(包括ITS1和ITS2)用于区分两种菌型,使用欧洲地区已报道的ITS序列的通用正向引物NS5(f),NS7(f),PSP1(f),ITS5(f)和反向引物ITS4(r)组合进行扩增。但主要扩增到小麦的rDNA序列,均未能稳定特异地扩增到禾谷多黏菌ITS序列。因此,本发明的研究人员设计了大量的引物,最终筛选得到一条反向引物ITS.CN(r),利用正向引物NS7扩增包含ITS1的约800bp序列,可以用于检测I-CN型菌的特异性引物。另外,利用II型菌引物Pg.F2(f)和Pg.R2(r)扩增得到II型菌特异的ITS序列,PCR产物长度为430bp。具体的本发明采用如下技术方案:
1.样品的采集
采集田间发病小麦样品,通过RT-PCR检测叶片病毒以及显微镜观察根部禾谷多黏菌的休眠孢子情况,明确样品根部含有禾谷多黏菌。
2.我国禾谷多黏菌分型的确定
首先利用引物组合PSP2(f)和ITS4(r)扩增小麦根部禾谷多黏菌ITS2序列,测序明确具体的扩增序列,利用NCBI上已知的禾谷多黏菌分型构建进化树,明确我国禾谷多黏菌分型为I-CN型和II型。
3.分型的特异性引物设计
根据我国特有的分型I-CN型的序列特点设计引物ITS.CN(r),结合正向引物NS7(f)PCR特异性扩增I-CN型。利用Pg.F2(f)和Pg.R2(r)PCR特异性扩增II型菌。
4.引物的特异性验证
提取含有不同分型禾谷多黏菌的小麦病根总DNA作为模版PCR扩增验证引物特异性,最终明确检测禾谷多黏菌的特异性引物为PSP2(f)和ITS4(r),检测禾谷多黏菌I型菌的特异性引物为Ns7(f)和ITS.CN(r),检测禾谷多黏菌II型菌的特异性引物为Pg.F2(f)和Pg.R2(r)。
5.反应体系及扩增条件的优化
对影响PCR扩增的各个条件:包括退火温度、引物浓度、HiFi DNA聚合酶浓度、dNTP浓度、延伸时间及循环次数等方面进行优化,最终确定适合本研究的最佳PCR体系为:10×PCR Buffer 1μL,dNTP 2mM 1μL,上游引物10μM 0.25μL,下游引物10μM 0.25μL,DNA 1.0μL,HiFi DNA polymerase 5U/μL 0.25μL,ddH2O 6.25μL,终体积为10μL;最佳PCR扩增程序为:94℃预变性3min;94℃变性30s,58℃退火50s,72℃延伸时间根据引物不同分别为:PSP2(f)和ITS4(r)延伸20s,Ns7(f)和ITS.CN(r)延伸60s,Pg.F2(f)和Pg.R2(r)延伸30s,共进行35个循环;最后72℃延伸10min。
5.在研究中的应用
应用该技术对我国冬小麦区的禾谷多黏菌分型情况进行了较全面的调查,样品来源包括山东、河南、江苏、安徽和湖北五个省份土传小麦黄花叶病病区的66个小麦病根样品,明确了各地样品带禾谷多黏菌及其分型的情况。
采用本发明的技术方案,可以获得如下的技术效果:
1.获得了鉴定禾谷多黏菌I-CN型和II型的特异性引物;
2.获得了稳定的检测禾谷多黏菌I-CN型和II型菌的PCR体系。
附图说明
图1用于检测的三对引物在禾谷多黏菌核糖体DNA上的位置示意图(PSP2和ITS4为禾谷多黏菌特异性引物;NS7和ITS.CN为禾谷多黏菌I-CN型特异性引物;Pg.F2和Pg.R2为禾谷多黏菌II型特异性引物;→表示正向引物;←表示反向引物)
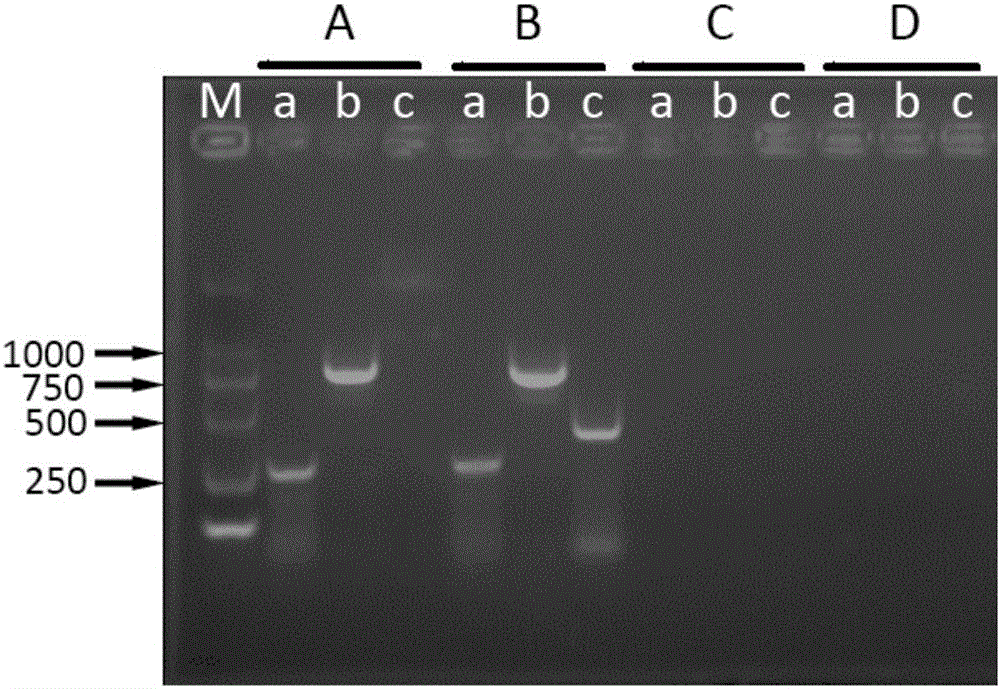
图2禾谷多黏菌分型特异性引物的样品检测效果(M:Marker;a:PSP2/ITS4引物组合特异性扩增禾谷多黏菌;NS7/ITS.CN引物组合特异性扩增I-CN型禾谷多黏菌;Pg.F2/Pg.R2引物组合特异性扩增II型禾谷多黏菌;样品A:I-CN型禾谷多黏菌侵染的小麦根部;样品B:I-CN型和II型禾谷多黏菌共同侵染的小麦根部;样品C:无禾谷多黏菌侵染的小麦根部;样品D:空白对照(H2O))
具体实施方式
下面通过具体实施方式的详细描述来进一步阐明本发明,但并不是本发明的限制,仅仅作示例说明。
实施例1:特异性引物的设计、合成及PCR初步检测
根据已报道的禾谷多黏菌通用和分型特异性引物序列设计如下正向引物:NS5,NS7,PSP1,ITS5,PSP2,Pg.F2;以及反向引物:ITS4,Pg.R2。初步PCR检测病区小麦根部样品后,将电泳出现条带的序列进行割胶回收并连接T载体测序。引物的配对、序列及初步的PCR结果如下表1所示:
表1本研究所用引物序列及其PCR结果
注:引物委托北京擎科新业生物技术有限公司合成。
测序结果表明禾谷多黏菌通用引物PSP2(f)/ITS4(r)能顺利扩增到禾谷多黏菌rDNA ITS2序列,对我国冬麦区大量小麦样品(五省共66份样品,详见实施例3)的ITS2测序结果分析初步明确我国冬麦区禾谷多黏菌至少存在两种分型,一种和已报道的I型具有一定同源性,为我国特有的分型,命名为I-CN型菌,另一种为II型菌。Pg.F2(f)/Pg.R2(r)同样能特异性的扩增到禾谷多黏菌序列(II型)。
同时,NS5(f)/ITS4(r),NS7(f)/ITS4(r),PSP1(f)/ITS4(r),ITS5(f)/ITS4(r),PSP2(f)/ITS4(r)引物组合虽然能顺利扩增到正确大小条带,但测序结果为小麦rDNA,而非禾谷多黏菌rDNA。进一步的序列比对结果表明,反向引物ITS4序列和小麦rDNA上的一段序列完全匹配。因此虽然已报道的ITS4序列在欧洲能广泛应用于禾谷多黏菌检测,但在我国小麦上不适用,这可能是因为目前欧洲用于禾谷多黏菌检测的作物主要为大麦、黑麦等作物,而非小麦。
基于以上结果,根据小麦与禾谷多黏菌I-CN型rDNA序列的同源性,在两者的差异位置设计了一条反向引物ITS.CN,利用正向引物NS7扩增包含禾谷多黏菌ITS1的约800bp序列,用于检测I-CN型菌的特异性引物(见表1)。因此,通过对禾谷多黏菌引物的筛选,获得了用于我国禾谷多黏菌检测的通用引物PSP2(f)/ITS4(r),新发现的我国I-CN型禾谷多黏菌特异性引物NS7(f)/ITS.CN(r),以及II型菌的特异性引物Pg.F2(f)/Pg.R2(r)。
实施例2:建立检测禾谷多黏菌及其分型的PCR体系与条件
剪取干病根每份30mg,采用TIANGEN公司的DNAsecure Plant Kit试剂盒方法提取小麦病根样品的总DNA,溶于50μL ddH2O中。
进行50μL体系的PCR反应,体系为:10×PCR Buffer 5μL,dNTP 2mM 5μL,上游引物10μM 1.25μL,下游引物10μM 1.25μL,DNA5μL,HiFi DNA polymerase 5U/μL 0.5μL,ddH2O 32μL,终体积为50μL;PCR扩增程序为:94℃预变性3min;94℃变性30s,58℃退火50s,72℃延伸时间根据引物不同分别为:PSP2(f)和ITS4(r)延伸20s,Ns7(f)和ITS.CN(r)延伸60s,Pg.F2(f)和Pg.R2(r)延伸30s,共进行35个循环;最后72℃延伸10min。将PCR产物进行电泳分析并在紫外灯下观察,可以观察到引物PSP2(f)和ITS4(r)扩增出的条带大小约为250bp,引物Ns7(f)和ITS.CN(r)扩增出的条带大小约为800bp,引物Pg.F2(f)和Pg.R2(r)扩增出的条带大小约为430bp。
分别将特异性片段进行凝胶回收,进行克隆转化,将获得的阳性克隆交由北京擎科新业生物技术有限公司进行测序分析。测序结果分别为:
引物PSP2(f)和ITS4(r)的扩增序列(SEQ ID NO:10):
TTGCGCTTTCGAGAGCCCTCGGAAGCATGCCTCTCTGAGTATCGGTTTCTATATCTCTCACATACTCACTGGTGGTGTGTGTGGTGGAGAATGGGAGTTGCGCACGTCTCTCTGCATCTCCTGAAGTCAGCGTCGGCCGAAGTCCATCGGGTTCTTGGAACGATAATCCGTCATGGCGCCCAAACCATACACAAAAGATCTCAGATGAGGTAAGACTACCCGCCGAATTTAAGCATATCAATAAGCGGAGGA
引物Ns7(f)和ITS.CN(r)的扩增序列(SEQ ID NO:11):
GAGGCAATAACAGGTCTGTGATGCCCTTAGATGTTCTGGGCCGCACGCGCGCTACAATGCTAGGTTCAACGAGTCGATTTCGTACTTGGCCGAGAGGCCTGGTTAATCTTCTGAAATCCTAGCGTGCTTGGGCTTGCCTCTTGCAACTAGAGGCACCAACGAGGAATTCCTAGTAGACGCAGGTCATCAACCTGCATCGATTACGTCCCTGCCCTTTGTACACACCGCCCGTCGCTTCTACCGATTGGTCGTCCCGGTGAACAATCGGGAGGCCGTGCTCATGGTCAGCAATGGCCGGAGCGCGGTCGAACTTTTGTAAATTTGGACGACTAGAGGAAGAAGAAGTCGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTAGCGTTGATTGGTCTTGGTGCCATGCGAAAAACATGTGGATCGTGGGCTATGTGACCCCTCTGTGGGGGGTATTTTGTCGCGAACTTGGAGATTTGGCTGATGGATATACATGATAGATAAACAAAATACAACTCTTAGCAATGGATATCTTGGTTCCCACAACGATGAAGAACGCAGCGAAATGCGATACGTAATGCGAATTGCAGAATTCAGTGAATCATCGAATCTTTGAACGCAAGTTGCGCTTTCGAGAGCCCTCGGAAGCATGCCTCTCTGAGTATCGGTTTCTATATCTCTCACACACTCACTGGTGGTGTGTGTGGTGGAGAATGGGAGTTGCGCACGTCTCTCTGCATCTCCTGAAGTCAGCGTCGGCCGAAGTCCATCGGGTTCTTGGAACGATAATCCGC
引物Pg.F2(f)和Pg.R2(r)的扩增序列(SEQ ID NO:12):
ATGTGGATCGTCTCTGTTGCTGGATACCGGGATGGAACGCCCTCGTGGTGGTTCTTGTCTTTCACGAATTGGATCAAACGGTGGCTAAAGTCGAATTGGTTTCATAACCTACAAACAATATATACAACTCTTAGCAATGGATATCTTGGTTCCCACAACGATGAAGAACGCAGCGAAATGCGATACGTAATGCGAATTGCAGAATTCAGTGAATCATCGAATCTTTGAACGCAAGTTGCGCTTTCGAGAGCCCTCGGAAGCATGCCTCTCTGAGTATCGGTTTCCCATCTCACACCCGGTGTGGAGAATGAGGGTCGGGCGCCTCGGCGCCGGTCCCTTCAAATCAGGCCGGCCTGGTCAAGTCCATCGGATTGCTGGTACGATAGTCCGTCATGGCGCAAGGAACCACTTGGCAAGATCTCAGATGAGG
实施例3:
本发明中的检测方法在实际研究中的应用
利用该技术检测了包括来自山东、河南、江苏、安徽、湖北五个省份小麦黄叶病病区的62个小麦病根样品,明确了各地样品带禾谷多黏菌及其I型和II型菌的情况。从表中可以看出,在这五个省份中都分布有I型禾谷多黏菌,少部分地点同时分布有II型菌,但是并没有发现单独存在II型菌的情况。
氨基酸序列表
<110> 浙江省农业科学院
<120> 一种我国小麦根部禾谷多黏菌分型及其鉴定的方法
<130> NKY001
<160> 12
<170> PatentIn version 3.5
<210> 1
<211> 22
<212> DNA
<213> Artificial
<220>
<223> NS5(f)
<400> 1
aacttaaagg aattgacgga ag 22
<210> 2
<211> 20
<212> DNA
<213> Artificial
<220>
<223> ITS4(r)
<400> 2
tcctccgctt attgatatgc 20
<210> 3
<211> 23
<212> DNA
<213> Artificial
<220>
<223> NS7(f)
<400> 3
gaggcaataa caggtctgtg atg 23
<210> 4
<211> 20
<212> DNA
<213> Artificial
<220>
<223> PSP1(f)
<400> 4
tagacgcagg tcatcaacct 20
<210> 5
<211> 22
<212> DNA
<213> Artificial
<220>
<223> ITS5(f)
<400> 5
ggaagtaaaa gtcgtaacaa gg 22
<210> 6
<211> 19
<212> DNA
<213> Artificial
<220>
<223> PSP2(f)
<400> 6
ttgcgctttc gagagccct 19
<210> 7
<211> 19
<212> DNA
<213> Artificial
<220>
<223> Pg.F2(f)
<400> 7
ttgcgctttc gagagccct 19
<210> 8
<211> 23
<212> DNA
<213> Artificial
<220>
<223> Pg.R2(r)
<400> 8
cctcatctga gatcttgcca agt 23
<210> 9
<211> 20
<212> DNA
<213> Artificial
<220>
<223> ITS.CN(r)
<400> 9
gcggattatc gttccaagaa 20
<210> 10
<211> 252
<212> DNA
<213> 禾谷多黏菌(Polymyxa graminis)
<400> 10
ttgcgctttc gagagccctc ggaagcatgc ctctctgagt atcggtttct atatctctca 60
catactcact ggtggtgtgt gtggtggaga atgggagttg cgcacgtctc tctgcatctc 120
ctgaagtcag cgtcggccga agtccatcgg gttcttggaa cgataatccg tcatggcgcc 180
caaaccatac acaaaagatc tcagatgagg taagactacc cgccgaattt aagcatatca 240
ataagcggag ga 252
<210> 11
<211> 798
<212> DNA
<213> 禾谷多黏菌(Polymyxa graminis)
<400> 11
gaggcaataa caggtctgtg atgcccttag atgttctggg ccgcacgcgc gctacaatgc 60
taggttcaac gagtcgattt cgtacttggc cgagaggcct ggttaatctt ctgaaatcct 120
agcgtgcttg ggcttgcctc ttgcaactag aggcaccaac gaggaattcc tagtagacgc 180
aggtcatcaa cctgcatcga ttacgtccct gccctttgta cacaccgccc gtcgcttcta 240
ccgattggtc gtcccggtga acaatcggga ggccgtgctc atggtcagca atggccggag 300
cgcggtcgaa cttttgtaaa tttggacgac tagaggaaga agaagtcgta acaaggtttc 360
cgtaggtgaa cctgcggaag gatcattagc gttgattggt cttggtgcca tgcgaaaaac 420
atgtggatcg tgggctatgt gacccctctg tggggggtat tttgtcgcga acttggagat 480
ttggctgatg gatatacatg atagataaac aaaatacaac tcttagcaat ggatatcttg 540
gttcccacaa cgatgaagaa cgcagcgaaa tgcgatacgt aatgcgaatt gcagaattca 600
gtgaatcatc gaatctttga acgcaagttg cgctttcgag agccctcgga agcatgcctc 660
tctgagtatc ggtttctata tctctcacac actcactggt ggtgtgtgtg gtggagaatg 720
ggagttgcgc acgtctctct gcatctcctg aagtcagcgt cggccgaagt ccatcgggtt 780
cttggaacga taatccgc 798
<210> 12
<211> 430
<212> DNA
<213> 禾谷多黏菌(Polymyxa graminis)
<400> 12
atgtggatcg tctctgttgc tggataccgg gatggaacgc cctcgtggtg gttcttgtct 60
ttcacgaatt ggatcaaacg gtggctaaag tcgaattggt ttcataacct acaaacaata 120
tatacaactc ttagcaatgg atatcttggt tcccacaacg atgaagaacg cagcgaaatg 180
cgatacgtaa tgcgaattgc agaattcagt gaatcatcga atctttgaac gcaagttgcg 240
ctttcgagag ccctcggaag catgcctctc tgagtatcgg tttcccatct cacacccggt 300
gtggagaatg agggtcgggc gcctcggcgc cggtcccttc aaatcaggcc ggcctggtca 360
agtccatcgg attgctggta cgatagtccg tcatggcgca aggaaccact tggcaagatc 420
tcagatgagg 430
- 还没有人留言评论。精彩留言会获得点赞!