酶复合物及自组装催化纳米线的制作方法
本发明涉及酶催化剂,特别涉及一种酶复合物及自组装催化纳米线。
背景技术:
酶作为一种生物催化剂,已广泛应用于生产、生活的各个领域。近几十年来,随着酶工程不断的技术性突破,在工业、农业、医药卫生、能源开发及环境工程等方面的应用越来越广泛。在对酶催化过程的研究和应用过程中,人们总是期望酶的活力和稳定性越高越好,并具备良好的催化性能。如何提升酶蛋白的催化能力是学术界的研究热点之一,同时也是工业生产实践中迫切需要解决的问题。传统的各种提升酶蛋白催化能力的方法主要包括化学修饰、分子酶工程等。
酶化学修饰是应用化学方法主要包括1)在酶蛋白特殊位点引入特定分子进行修饰的定点突变方法;2)使用双功能基团试剂如戊二醛、peg等将酶蛋白分子之间、亚基之间或分子内不同肽链部分,进行共价交联的交联技术;3)利用小分子化合物对酶活性部位或活性部位之外的侧链基团进行化学修饰的小分子化合物修饰等方法对酶分子施行种种化学修饰,通过主链的切割、剪接和侧链基团的化学修饰对酶蛋白进行分子改造,以改变其理化性质及生物活性。酶化学修饰主要是通过化学基团的引入或除去,且修饰大都发生在酶活性部位或必需基团上,这样不仅使酶蛋白共价结构发生改变,而且使得酶蛋白活性随着修饰程度的加深而逐渐减弱,即酶化学修饰并不能有效的提高酶自身的催化效率,反而会降低酶活。
分子酶工程是采用基因工程和蛋白质工程的方法和技术,研究酶基因的克隆和表达、酶蛋白的结构与功能的关系,并以其为基础对酶分子进行再设计和定向加工。分子酶工程需要从一个或多个已经存在的亲本酶出发,经过基因的突变和重组,构建一个人工突变酶库,通过一定的筛选或选择方法最终获得具有某些特性的进化酶。分子酶工程方法不仅依赖于亲本酶分子的结构生物学数据的完善,还需要费时费力得构建人工突变酶库,后期的筛选过程更是复杂繁琐。
综合酶化学修饰和分子酶工程技术来看,二者的工作都集中于加工单个酶分子,并没有考虑到酶分子在活细胞内的整体分布情况。越来越多的研究表明酶分子在细菌体内并不是像在溶液里一样均匀分散的,而是成簇存在的,这种以超分子聚集体形态出现酶分子簇具有更高的催化活性。但是通过基因克隆所表达出来的酶分子往往以单分散的形式出现,这可能是所制备的酶分子与天然状态酶分子相比活性降低的原因之一。
技术实现要素:
本发明为克服现有技术中存在的酶的催化特性和稳定性提高有限、制备复杂、成本高且针对特定的酶需要寻找适合于该酶的试剂、材料或方法等缺点,提供了成纤维蛋白在增强酶催化活性和/或酶稳定性中的应用。本发明所述的成纤维蛋白为amyloidogenesisfibrils(淀粉样成纤维蛋白),简称成纤维蛋白。
为了实现本发明的目的,本发明采用如下技术方案:
本发明一方面提供成纤维蛋白在增强酶催化活性和/或酶稳定性中的应用。
示例性地,所述成纤维蛋白与所述酶形成酶复合物,所述酶直接或间接连接于所述成纤维蛋白。
示例性地,所述酶通过连接肽和/或接头蛋白连接于所述成纤维蛋白。
在本发明一具体实施方式中,所述酶通过连接肽连接于所述成纤维蛋白。
在本发明一具体实施方式中,所述成纤维蛋白形成纤维状纳米结构。
在本发明一具体实施方式中,所述成纤维蛋白与所述酶形成的酶复合物自组装形成纤维状纳米结构。
在本发明一具体实施方式中,所述纤维状纳米结构由成纤维蛋白自组装形成。
示例性地,所述成纤维蛋白可为任何能够形成纤维状,并能够与酶相连接的蛋白。
示例性地,所述成纤维蛋白为酵母朊蛋白或淀粉样蛋白。例如可为酵母朊蛋白sup35、淀粉样蛋白ure2、丝素蛋白等。
示例性地,所述成纤维蛋白为酵母朊蛋白sup35,优选地,成纤维蛋白为酵母朊蛋白sup35的第1-61位氨基酸形成的自组装结构域。
在本发明一具体实施方式中,所述酶选自蛋白酶、核酸酶或人工酶。
在本发明一具体实施方式中,所述酶选自氧化还原酶、转移酶、水解酶、裂合酶、异构酶或合成酶。
示例性地,氧化还原酶如葡萄糖氧化酶、过氧化氢酶、氢化酶、加氧酶等。
示例性地,转移酶如转甲基酶、转氨酶、己糖激酶、磷酸化酶等。具体地,如西塔列汀转氨酶。
示例性地,水解酶如酯酶、糖基酶、肽酶、核酸酶等。具体地,如甲基对硫磷水解酶。
示例性地,裂合酶如醛缩酶、水化酶、脱氨酶、碳酸酐酶、丙酮酸脱羧酶、碱性磷酸酶等。
示例性地,异构酶如消旋酶、差向异构酶、变位酶等。
示例性地,合成酶如氨酰trna合成酶、谷氨酰胺合成酶、海藻糖合成酶等。
在本发明一具体实施方式中,所述酶包括以碳水化合物、蛋白质、脂肪、核酸、生物碱为底物的酶。
示例性地,以碳水化合物为底物的酶如淀粉酶,葡萄糖异构酶,乳糖酶,凝乳酶,纤维素酶,聚糖酶,乳糖酶等;以蛋白质为底物的酶如菠萝蛋白酶、胃蛋白酶、胰蛋白酶等;以脂肪为底物的酶如脂肪酶、酯酶等;以核酸、生物碱为底物的酶如碱性磷酸酶等。
本发明另一方面提供上述酶复合物的制备方法,其包括通过分子克隆,将成纤维蛋白与酶分子融合或者将成纤维蛋白、连接肽(或街头蛋白)、酶分子融合形成融合蛋白基因,并将该融合蛋白基因进行表达。
在本发明一具体实施方式中,上述酶复合物的制备方法由以下步骤组成:
通过分子克隆,将成纤维蛋白、连接肽、酶分子融合形成融合蛋白基因,并将该融合蛋白基因进行表达、纯化。
在本发明一具体实施方式中,所述成纤维蛋白为酵母朊蛋白自组装结构域sup35,所述酶为甲基对硫磷水解酶mph(或西塔列汀转氨酶ata-117)。酶复合物具体的制备方法包括:
通过分子克隆将酵母朊蛋白自组装结构域sup35和甲基对硫磷水解酶mph的基因mph(或西塔列汀转氨酶ata-117的基因ata-117)通过连接肽(例如:ggggs)融合连接形成融合蛋白基因sup35-mph(或sup35-ata-117);
将融合蛋白基因sup35-mph(或sup35-ata-117)克隆入表达载体中,将表达载体转入表达宿主中培养,诱导表达融合蛋白sup35-mph(或sup35-ata-117)并纯化。
在具体实施方式中,融合蛋白基因的表达、纯化可采用本领域常规用于蛋白表达纯化的方法,例如将融合蛋白基因克隆入表达载体,将表达载体转入表达宿主中培养,活化至对数生长期后加入诱导表白蛋白,经破碎、纯化后得到融合蛋白。其中,本发明对表达载体、表达宿主的种类和类别不作限定,可选用本领域常规用于遗传修饰的载体和宿主。具体的,表达载体可为pet-28、pet-32、pet-15或pet-11等;表达宿主可选自大肠杆菌、枯草芽孢杆菌、巨大芽孢杆菌、棒状杆菌、酿酒酵母、毕赤酵母或哺乳动物细胞。
在具体实施方式中,接头蛋白可代替连接肽,同样可以减少融合蛋白可能存在的位阻的影响。
本发明又一方面提供一种自组装催化纳米线,其包括本发明中的酶复合物或上述制备方法制得的酶复合物,其中,所述酶复合物中50%以上的成纤维蛋白直接或间接连接有酶。
示例性地,55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的成纤维蛋白直接或间接连接有酶,具体比例可依据实际生产需要及成本而确定。
示例性地,成纤维蛋白通过连接肽和/或接头蛋白连接于酶。
示例性地,所述连接肽为ggggs。
在本发明一具体实施方式中,成纤维蛋白自组装形成纤维纳米结构。
本发明还一方面提供自组装催化纳米线的制备方法,包括如下步骤:
将上述一部分酶复合物或者将该部分酶复合物与没有酶复合的成纤维蛋白相混合形成的混合物作为单体孵育,形成纳米线,通过超声将纳米线破碎为纳米线片段,制备纳米线种子;
将制备好的纳米线种子与另一部分酶复合物或者该部分酶复合物与没有酶复合的成纤维蛋白相混合形成的混合物混合孵育,进行种子诱导自组装形成自组装催化纳米线。
在本发明的一个具体的实施方式中,所述酶复合物为sup35-mph(或sup35-ata-117),自组装催化纳米线的制备包括如下步骤:
将一部分sup35-mph(或sup35-ata-117)作为单体置于4℃孵育一周后,形成纳米线,通过超声将纳米线破碎为纳米线片段,制备sup35-mph种子(或sup35-ata-117种子);
将制备好的sup35-mph种子(或sup35-ata-117种子)与另一部分sup35-mph(或sup35-ata-117)按照一定摩尔比例混合,于4℃混合孵育,进行种子诱导自组装形成自组装催化纳米线,该纳米线中100%的sup35直接或间接连接有mph(或ata-117)。
在本发明的一个具体的实施方式中,所述酶复合物为sup35-mph(或sup35-ata-117),自组装催化纳米线的制备包括如下步骤:
将一部分sup35-mph(或sup35-ata-117)作为单体置于4℃孵育一周后,形成纳米线,通过超声将纳米线破碎为纳米线片段,制备sup35-mph种子(或sup35-ata-117种子)。
将另一部分sup35-mph(或sup35-ata-117)与未复合sup35-mph(或sup35-ata-117)的sup35按照一定摩尔比例混合形成混合单体。
将制备好的sup35-mph种子(或sup35-ata-117种子)与上述混合单体按照一定摩尔比例混合,于4℃混合孵育,进行种子诱导自组装形成自组装催化纳米线。可通过调整混合单体中sup35-mph(或sup35-ata-117)与sup35的比例控制所制备的纳米线中sup35直接或通过连接肽连接mph(或ata-117)的比例。
在本发明的另一个具体的实施方式中,所述酶复合物为sup35-mph(或sup35-ata-117),自组装催化纳米线的制备包括如下步骤:
将一部分sup35-mph(或sup35-ata-117)与未复合sup35-mph(或sup35-ata-117)的sup35按照一定摩尔比例混合形成的混合物作为单体置于4℃孵育一周后,形成纳米线,通过超声将纳米线破碎为纳米线片段,制备sup35-mph种子(或sup35-ata-117种子)。
将另一部分sup35-mph(或sup35-ata-117)与未复合sup35-mph(或sup35-ata-117)的sup35(这里不存在任何单独的sup35蛋白分子,仅存在sup35-mph和sup35-ata-117蛋白分子或分子形成的纳米线)按照一定摩尔比例混合形成混合单体。
将制备好的sup35-mph种子(或sup35-ata-117种子)与上述混合单体按照一定摩尔比例混合,于4℃混合孵育,进行种子诱导自组装形成自组装催化纳米线。可通过调整种子中sup35-mph(或sup35-ata-117)与sup35的比例和/或混合单体中sup35-mph(或sup35-ata-117)与sup35的比例控制所制备的纳米线中sup35直接或通过连接肽连接mph(或ata-117)的比例。
本发明采用种子诱导自组装方法,种子可以快速的诱发融合有自组装结构域的融合蛋白组装在其末端,通过控制种子与单体(或混合单体)融合蛋白的组装比,可实现对纳米线长度的控制。
本发明还一方面提供上述酶复合物或自组装催化纳米线作为催化剂的应用。
本发明还一方面提供成纤维蛋白在制备用于增强酶催化作用和/酶稳定性的自组装催化纳米线中的应用。
本发明还一方面提供纤维状纳米结构在增强酶催化活性和/或没稳定性中的应用。
本发明还一方面提供一种增加酶活性和/或稳定性的方法,其包括:
(1)将酶直接或间接连接于成纤维蛋白得到酶复合物;
(2)将(1)中得到的酶复合物自组装成纳米线,进而使得所述酶展示在自组装纳米线上。
示例性的,本发明至少具有以下优势之一:
本发明通过基因操纵,将成纤维蛋白例如酵母朊蛋白sup35的自组装决定域与酶的基因相连,经过基因表达将酶分子结合在酵母朊蛋白sup35的c末端,利用酵母朊蛋白sup35自组装结构域的聚合形成纳米结构,而将大量的酶分子高密度、阵列化地展示在纳米结构的表面,形成具有催化能力的线状纳米结构。该纳米结构模拟了酶分子的天然区域化状态,从而在不对酶分子本身进行改变的情况下,提高酶的催化活性和/或稳定性。例如,本发明实施例中,相较于溶液中自由分散的游离mph,与成成纤维蛋白相结合的mph,其米氏常数km不足游离酶的1/5,催化常数kcat提高了1倍,最大速率vmax提高了26.5倍,比活力提高了4.8倍,显著地提高了甲基对硫磷水解酶mph与底物的亲和能力和催化能力;对于西塔列汀转氨酶ata-117,在提高西塔列汀转氨酶ata-117对底物的转化率的同时,缩短了底物转化率的时间,在相同的反应时间内其底物转化率最高可提高30%左右,达到最高底物转化率的时间自组装催化纳米线sup35-ata-117相较于游离酶ata-117缩短了4倍多。
附图说明
图1.本发明实施例中融合蛋白sup35-mph(sup35-ata-117)表达和纳米线酶蛋白自组装结构示意图。
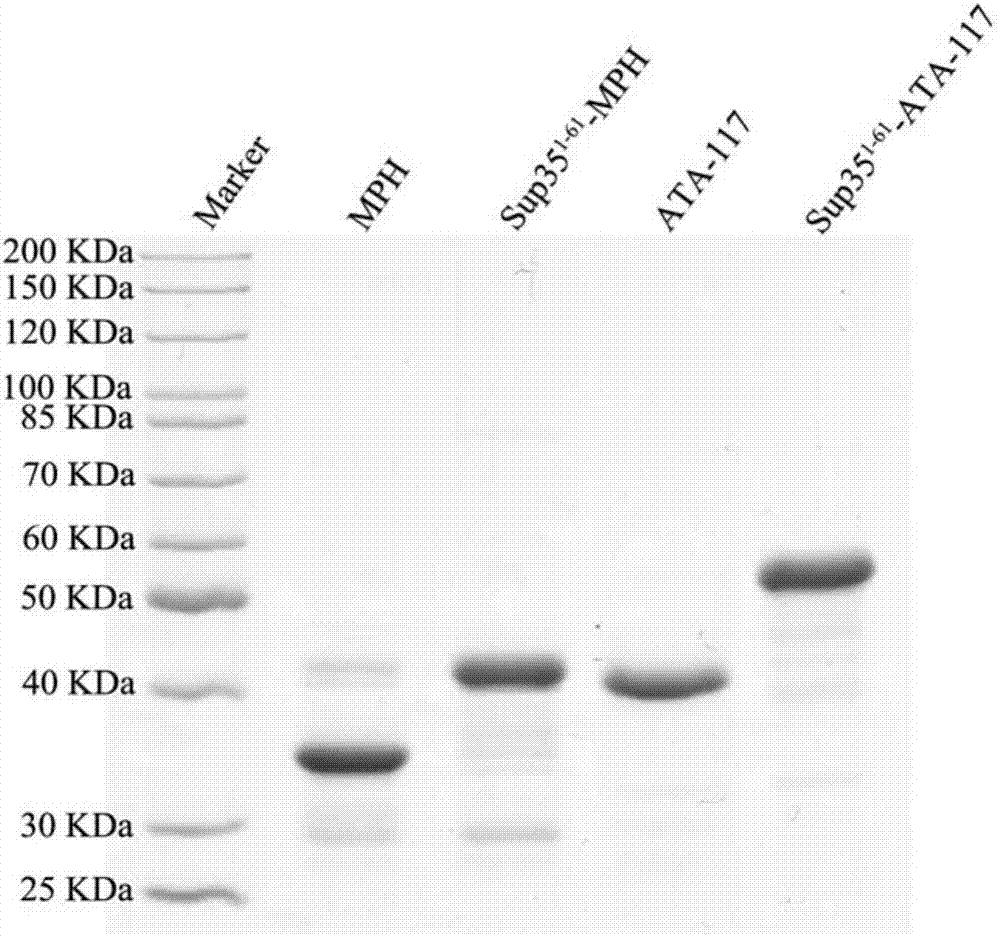
图2.本发明实施例中sup35-mph酶复合物及sup35-ata-117酶复合物sds-page凝胶电泳检测图。
图3a.本发明实施例中自组装形成的sup35-mph纳米线的电镜图。
图3b.本发明实施例中自组装形成的sup35-ata-117纳米线的电镜图。
图4a.本发明实施例中游离酶mph和纳米线状态的sup35-mph反应过程中,产物od405值随时间的变化状况图。
图4b.本发明实施例中游离酶mph和纳米线状态的酶蛋白sup35-mph动力学比较结果图。
图5.本发明实施例中游离酶ata-117和纳米线状态的酶蛋白sup35-ata-117催化能力比较结果图。
具体实施方式
说明:本发明具体实施方式、实施例中缩写“mph”、“ata-117”、“sup35-mph”、“sup35-ata-117”不区分大小写,可代表融合基因、融合蛋白或自组装纳米线,具体所代表的含义依据上下文理解。
“sup35-mph”即可代表酵母朊蛋白sup35与mph直接连接,也可代表酵母朊蛋白sup35与mph通过连接肽或街头蛋白连接,具体所代表的含义依据上下文理解。
“sup35-ata-117”即可代表酵母朊蛋白sup35与ata-117直接连接,也可代表酵母朊蛋白sup35与ata-117通过连接肽或接头蛋白连接,具体所代表的含义依据上下文理解。
本发明具体实施例中的连接肽,可根据需要替换成为接头蛋白。例如,亮氨酸拉链等。
下面结合具体实施例,以sup35-mph/sup35-ata-117为例,对本发明的酶复合物、自组装催化纳米线及其制备方法作进一步的阐述。
实施例1sup35-mph酶复合物的制备
1、sup35-mph酶复合物克隆
通过分子克隆将酵母朊蛋白自组装结构域(sup35的第1-61个氨基酸,简称sup35,具体序列参见seqidno.1所示)与甲基对硫磷水解酶(mph的第36-331个氨基酸,具体序列参见seqidno.2所示)通过柔性连接肽(具体序列参见seqidno.3所示)融合连接形成融合蛋白sup35-mph,然后在其n末端融合his标签。以单独的甲基对硫磷水解酶(mph)为对照。具体步骤如下:
1)以质粒pet28a为模板,利用引物mph-primer-1进行pcr扩增,得到含有引物mph-primer-1序列的质粒pet28a;
2)以mph基因片段为模板,利用引物mph-primer-3进行pcr扩增,得到n端带his-tag的mph基因片段;
3)以本实验室存在的c端带histag的质粒pet28a-sup351-61–linker–mph-histag(具体可参见文献:门冬,基于朊蛋白自组装的多功能纳米线及超灵敏生物传感,博士毕业论文)为模板,利用mph-primer-2进行pcr扩增,得到n端带his-tag的sup351-61-mph片段;
4)将步骤1)与2)中得到的基因片段进行同源重组,即可得到表达载体pet28a-his-mph;
5)和步骤1)与3)中得到的基因片段做同源重组反应,即可得到表达载体pet28a-histag-sup351-61-linker-mph。
2、sup35-mph酶复合物表达、纯化
分别将表达载体pet28a-his-mph和pet28a-histag-sup351-61-linker-mph转化入大肠杆菌表达菌株bl21,卡那霉素抗性平板挑取阳性克隆。将挑取的阳性克隆e.colibl21二次活化到卡那霉素抗性lb培养基,37℃、200rpm振荡培养至对数生长期(od值约为0.5)。向培养物中加入工作终浓度为1mm的iptg和工作中浓度为50μm(kan的终浓度是50μg/ml)的卡那霉素,25℃,120rpm振荡培养诱导蛋白表达8小时。8000rpm离心收集菌体5分钟,超声破碎菌体,10000×g离心30分钟去除细胞碎片,取上清ni亲和层析纯化目标蛋白,即获得纯化的mph和sup35-mph(如图1所示)。mph为游离的甲基对硫磷水解酶蛋白,作为对照。
本领域技术人员知晓,本发明上述具体实施方式中蛋白的表达与纯化中具体参数(例如浓度、时间、温度等)数值并不用于限制本发明,本领域技术人员可以依据实际需求作调整。
3、自组装催化纳米线sup35-mph的可控制备
(1)sup35-mph种子的制备:将一部分酶复合物sup35-mph作为单体蛋白置于4℃孵育一周后,通过超声所产生的剪切力,将长的纳米线打断成为纳米线片段制备种子,该种子可以快速的诱发融合有自组装结构域的融合蛋白组装在其末端。
(2)sup35-mph纳米线的制备:将制备好的sup35-mph种子,与另外一部分sup35-mph酶复合物按照一定的摩尔比混合,于4℃孵育8个小时,使其发生种子诱导的快速组装(如图1所示)。sup35-mph种子与sup35-mph酶复合物的摩尔比可为任意比,本实施例中选择1:4的比例。以游离的甲基对硫磷水解酶蛋白(mph)为对照。
实施例2sup35-mph酶复合物及纳米线的检测
对实施例1制得的mph和sup35-mph进行sds-page电泳,对得到的凝胶进行考马斯亮蓝染色,其结果如图2所示。由图2可知,在30kda与40kda之间,40kda附近有清晰明显的条带,其分别对应于游离的甲基对硫磷水解酶蛋白(mph)和sup35-mph酶复合物。电泳显示无明显的杂蛋白条带,说明实施例1制备的甲基对硫磷水解酶蛋白(mph)和sup35-mph酶复合物的纯度和表达量都很高。
对实施例1制备的自组装催化纳米线sup35-mph进行电镜检测,其结果如图3a所示。由图3a可知,自组装形成的纳米线浓度和长度分布均匀。
实施例3sup35-ata-117酶复合物的制备
1、sup35-ata-117酶复合物克隆
通过分子克隆将酵母朊蛋白自组装结构域(sup35的第1-61个氨基酸,简称sup35,具体序列参见seqidno.1所示)与西塔列汀转氨酶(ata-117,具体序列参见seqidno.4所示)通过柔性连接肽(具体的序列参见seqidno.3所示)融合连接形成融合蛋白sup35-ata-117,然后在其n末端融合his标签。以单独的西塔列汀转氨酶(ata-117)为对照。具体步骤如下:
1)以质粒pet28a为模板,利用引物ata-primer-1进行pcr扩增,得到含有引物ata-primer-1序列的pet28a载体;
2)以质粒puc57-ata-117为模板,利用引物ata-primer-2进行pcr扩增,得到n端带his-tag的histag-ata-117基因片段;
3)以质粒pet28a-histag-sup351-61-linker-mph为模板,利用引物ata-primer-3进行pcr扩增,得到n端带his-tag的histag-sup351-61-linker基因片段;
4)以puc57-ata-117为模板,利用引物ata-primer-4进行pcr反应,得到linker-ata-117基因片段;
5)以步骤3)所得的histag-sup351-61-linker片段和步骤4)所得linker-ata-117片段为模板,利用引物ata-primer-5进行pcr扩增,扩增得到sup351-61-linker-ata-117基因片段;
6)将步骤1)与2)中得到的基因片段进行同源重组,即可得到表达载体pet28a-histag-ata-117;
7)将步骤1)与5)中得到的基因片段进行同源重组,即可得到表达载体pet28a-histag-sup351-61-ata-117。
2、sup35-ata-117酶复合物表达、纯化
分别将表达载体pet28a-histag-ata-117、pet28a-histag-sup351-61-ata-117转化入大肠杆菌表达菌株bl21,卡那霉素抗性平板挑取阳性克隆。将挑取的阳性克隆e.colibl21二次活化到卡那霉素抗性lb培养基,37℃、200rpm振荡培养至对数生长期(od值约为0.5)。向培养物中加入工作终浓度为1mm的iptg和工作中浓度为50μm的卡那霉素,25℃、120rpm振荡培养诱导蛋白表达8小时。8000rpm离心收集菌体5分钟,超声破碎菌体,10000×g离心30分钟去除细胞碎片,取上清ni亲和层析纯化目标蛋白,即获得纯化的ata-117和sup35-ata-117(如图1所示)。ata-117为游离的西塔列汀转氨酶蛋白,作为对照。
本领域技术人员知晓,本发明上述具体实施方式中蛋白的表达与纯化中具体参数(例如浓度、时间、温度等)数值并不用于限制本发明,本领域技术人员可以依据实际需求作调整。
3、自组装催化纳米线sup35-ata-117的可控制备
(1)sup35-ata-117种子的制备:将一部分酶复合物sup35-ata-117作为单体蛋白置于4℃孵育一周后,通过超声所产生的剪切力,将长的纳米线打断成为纳米线片段制备种子,该种子可以快速的诱发融合有自组装结构域的融合蛋白组装在其末端。
(2)sup35-ata-117纳米线的制备:将制备好的sup35-ata-117种子,与另外一部分sup35-ata-117酶复合物按照一定的摩尔比混合,于4℃孵育8个小时,使其发生种子诱导的快速组装(如图1所示)。sup35-ata-117种子与sup35-ata-117酶复合物的摩尔比可为任意比,本实施例中选择1:6的比例。以游离的西塔列汀转氨酶蛋白(ata-117)为对照。
实施例4sup35-ata-117酶复合物及纳米线的检测
对实施例3制得的ata-117和sup35-ata-117进行sds-page电泳,对得到的凝胶进行考马斯亮蓝染色,其结果如图2所示。由图2可知,在40kda附近,50kda与60kda之间有清晰明显的条带,其分别对应于游离的西塔列汀转氨酶蛋白(ata-117)和sup35-ata-117酶复合物。电泳显示无明显的杂蛋白条带,说明实施例3制备的西塔列汀转氨酶蛋白(ata-117)和sup35-ata-117酶复合物的纯度和表达量均很高。
对实施例3制备的自组装催化纳米线sup35-ata-117进行电镜检测,其结果如图3b所示。由图3b可知,自组装形成的纳米线浓度和长度的分布都很均匀。
分别以实施例1和实施例3所制备的自组装催化纳米线sup35-mph和sup35-ata-117进行酶动力学和酶稳定性分析研究。
实施例5自组装催化纳米线sup35-mph酶动力学及稳定性研究
实施例1制备的自组装催化纳米线sup35-mph(记为smph)可催化底物如甲基对硫磷发生反应,其产物为黄色,且在波长为405nm具有光吸收,利用酶标仪检测其产物的产生及产物量的变化情况,以分析自组装催化纳米线sup35-mph的酶学动力学和稳定性变化情况,实验结果如图4a和图4b。以游离的mph为对照(记为mph)。
从图4a中可以看出,游离酶mph的反应速率远远低于纳米线状态酶sup351-61-mph即smph,smph在很短时间内即达到反应的平衡点。
图4b左上图为米氏常数测试结果图,从图4b左上图中可以看出,纳米线状态酶smph的米氏常数km较游离酶mph降低了5.4倍,即纳米线状态酶smph更易与底物结合,其与底物的亲和力明显增强。
图4b右上图为催化常数测试结果图,从图4b右上图中可以看出,纳米线状态酶smph的催化常数kcat较游离酶mph提高了1倍,即底物浓度饱和状态下,纳米线状态酶smph催化相同反应的速率远远高于游离酶mph。
图4b左下图为最大催化速率测试结果图,从图4b左下图中可以看出,在相同条件下,纳米线状态酶smph的最大速率vmax较游离酶mph提高了26.5倍。
图4b右下图为酶活性测试结果图,从图4b右下图中可以看出,纳米线状态酶smph的比活力(enzymespecificactivity)较游离酶mph提高了4.8倍,即每毫克纳米线状态酶smph蛋白中所含酶活力单位数远远高于每毫克游离酶mph所含的酶活力单位数。
综上所述,自组装催化纳米线sup35-mph相较于游离的mph其催化特性和稳定性均明显提高。
实施例6自组装催化纳米线sup35-mph酶动力学及稳定性研究
实施例3制备的自组装催化纳米线sup35-ata-117可催化西塔列汀中间体4(西塔列汀前体酮)转化成西塔列汀。利用hplc分离检测西塔列汀的产生及产物量的变化情况,以分析自组装催化纳米线sup35-ata-117的催化能力,实验结果如图5。以游离ata-117酶为对照。
从图5中可以看出,自组装催化纳米线sup35-ata-117的底物转化率明显高于游离酶ata-117的底物转化率。在相同的反应时间内其底物转化率最高可提高30%左右,达到最高底物转化率的时间自组装催化纳米线sup35-ata-117相较于游离酶ata-117缩短了4倍多。
综上所述,成纤维蛋白(例如sup35)能够提高酶分子的催化伙子能够和稳定性。成纤维蛋白利用其自身的特性组装成蛋白纳米结构,将酶分子高密度、阵列化地展示在蛋白纳米结构的表面,形成具有催化能力的纳米结构。该纳米结构模拟了细胞内酶分子的天然区域化状态,从而在不对酶分子本身进行改变的情况下,提高酶的催化活性和稳定性。
本发明提供的方法或应用可作为酶分子的定向进化技术(directedevolution),简称酶定向进化。酶分子在自然进化过程中,保证了酶对环境任何改变的适应能力,但是自然进化既没有特定的方向,也没有特定的目标,它是在整个生物的繁殖和生存过程中自发进行。酶的自然进化主要不是表现为某个酶分子的活力和稳定性的不断提高,而是在于生物整体的适应能力,调控能力的增强,因此,通常只要求酶在生物体内对特定的生物学功能有专一性。
定向进化技术可使发生在自然界中漫长的进化过程能在实验室中得以模拟,使人类可以按照自己的意愿和需要改造酶分子,甚至设计出自然界中原来并不存在的全新酶分子(全新蛋白质)。与自然进化相比,酶分子的定向进化过程完全是在人为控制下进行的,使酶分子朝向人们期望的特定目标进化。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换等,均应包含在本发明的保护范围之内。
序列表
<110>中国科学院武汉病毒研究所
<120>酶复合物及自组装催化纳米线
<130>2017
<160>20
<170>siposequencelisting1.0
<210>1
<211>61
<212>prt
<213>酵母朊蛋白(prion)
<400>1
metseraspserasnglnglyasnasnglnglnasntyrglnglntyr
151015
serglnasnglyasnglnglnglnglyasnasnargtyrglnglytyr
202530
glnalatyrasnalaglnalaglnproalaglyglytyrtyrglnasn
354045
tyrglnglytyrserglytyrglnglnglyglytyrgln
505560
<210>2
<211>296
<212>prt
<213>甲基对硫磷水解酶(pseudomonassp.)
<400>2
alaalaproglnvalargthrseralaproglytyrtyrargmetleu
151015
leuglyaspphegluilethralaleuseraspglythrvalalaleu
202530
provalasplysargleuasnglnproalaprolysthrglnserala
354045
leualalysserpheglnlysalaproleugluthrservalthrgly
505560
tyrleuvalasnthrglyserlysleuvalleuvalaspthrglyala
65707580
alaglyleupheglyprothrleuglyargleualaalaasnleulys
859095
alaalaglytyrglnprogluglnvalaspgluiletyrilethrhis
100105110
methisproasphisvalglyglyleumetvalglygluglnleuala
115120125
pheproasnalavalvalargalaaspglnlysglualaaspphetrp
130135140
leuserglnthrasnleuasplysalaproaspaspgluserlysgly
145150155160
phephelysglyalametalaserleuasnprotyrvallysalagly
165170175
lysphelyspropheserglyasnthraspleuvalproglyilelys
180185190
alaleualaserhisglyhisthrproglyhisthrthrtyrvalval
195200205
gluserglnglyglnlysleualaleuleuglyaspleuileleuval
210215220
alaalavalglnpheaspaspproservalthrthrglnleuaspser
225230235240
aspserlysservalalavalgluarglyslysalaphealaaspala
245250255
alalysglyglytyrleuilealaalaserhisleuserpheprogly
260265270
ileglyhisileargalagluglylysglytyrargphevalproval
275280285
asntyrservalvalasnprolys
290295
<210>3
<211>5
<212>prt
<213>人工序列(artificialsequence)
<400>3
glyglyglyglyser
15
<210>4
<211>330
<212>prt
<213>人工序列(artificialsequence)
<400>4
metalapheseralaaspthrprogluilevaltyrthrhisaspthr
151015
glyleuasptyrilethrtyrserasptyrgluleuaspproalaasn
202530
proleualaglyglyalaalatrpilegluglyalaphevalpropro
354045
serglualaargileserilepheaspglnglyphetyrasnserasp
505560
alathrtyrthrthrphehisvaltrpasnglyasnalapheargleu
65707580
glyasphisilegluargleupheserasnalagluserileargleu
859095
ileproproleuthrglnaspgluvallysgluilealaleugluleu
100105110
valalalysthrgluleuargglualametvalthrvalthrilethr
115120125
argglytyrserserthrprophegluargaspilethrlyshisarg
130135140
proglnvaltyrmetseralacysprotyrglntrpilevalprophe
145150155160
aspargileargaspglyvalhisleumetvalalaglnservalarg
165170175
argthrproargserserileaspproglnvallysasnpheglntrp
180185190
glyaspleuileargalaileglngluthrhisaspargglypheglu
195200205
leuproleuleuleuaspcysaspasnleuleualagluglyprogly
210215220
pheasnvalvalvalilelysaspglyvalvalargserproglyarg
225230235240
alaalaleuproglyilethrarglysthrvalleugluilealaglu
245250255
serleuglyhisglualaileleualaaspilethrproalagluleu
260265270
tyraspalaaspgluvalleuglycysserthrglyglyglyvaltrp
275280285
prophevalservalaspglyasnserileseraspglyvalprogly
290295300
provalthrglnserileileargargtyrtrpgluleuasnvalglu
305310315320
proserserleuleuthrprovalglntyr
325330
<210>6
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>6
tgagatccggctgctaac18
<210>6
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>6
gtgatgatgatgatgatggctgctgcccatgg32
<210>7
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>7
catcatcatcatcacatgtcggattcaaaccaaggc36
<210>8
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>8
agcagccggatctcactattacttggggttgacgaccgagt41
<210>9
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>9
catcatcatcatcacgccgcaccgcaggtgcgcac35
<210>10
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>10
agcagccggatctcactattacttggggttgacgaccgagt41
<210>11
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>11
tgagatccggctgctaac18
<210>12
<211>32
<212>dna
<213>人工序列(artificialsequence)
<400>12
gtgatgatgatgatgatggctgctgcccatgg32
<210>13
<211>35
<212>dna
<213>人工序列(artificialsequence)
<400>13
catcatcatcatcacatggcattcagcgccgatac35
<210>14
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>14
agcagccggatctcactattagtactgcacaggcgtaagc40
<210>15
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>15
catcatcatcatcacatgtcggattcaaaccaaggc36
<210>16
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>16
tccggtggaggtggatcgatggcattcagcgccgatacc39
<210>17
<211>26
<212>dna
<213>人工序列(artificialsequence)
<400>17
cgatccacctccaccggatccttgat26
<210>18
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>18
agcagccggatctcactattagtactgcacaggcgtaagc40
<210>19
<211>36
<212>dna
<213>人工序列(artificialsequence)
<400>19
catcatcatcatcacatgtcggattcaaaccaaggc36
<210>20
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>20
agcagccggatctcactattagtactgcacaggcgtaagc40
- 还没有人留言评论。精彩留言会获得点赞!
- 一种基于纳米材料自组装的土霉素sers检测方法
- 暴露高能晶面纳米片自组装的三维网络结构α?Fe<sub>2</sub>O<sub>3</sub>的制备方法
- 一种三氧化钨纳米带自组装成微纳米星的制备方法
- 一种葡萄糖和温度敏感性的自组装纳米粒子及其制备方法和应用
- 允许通过嵌段共聚物的自组装产生纳米结构体的方法
- 一种自组装纳米止血剂、其制备方法及应用
- 一种纳米片自组装棱台状(NH<sub>4</sub>)<sub>2</sub>V<sub>3</sub>O<sub>8</sub>的制备方法
- 一种干燥介导金纳米颗粒自组装制备金基底的方法及其应用
- 一种以杆菌肽为模板自组装合成球状金纳米粒子的方法
- 一种果胶-多臂聚乙二醇自组装制备纳米药物的方法
- 一种具有葡萄糖敏感性和温度敏感性的自组装纳米粒子及其制备方法
- 一种基于纳米材料自组装的土霉素sers检测方法
- 暴露高能晶面纳米片自组装的三维网络结构α?Fe<sub>2</sub>O<sub>3</sub>的制备方法
- 一种三氧化钨纳米带自组装成微纳米星的制备方法
- 一种葡萄糖和温度敏感性的自组装纳米粒子及其制备方法和应用
- 允许通过嵌段共聚物的自组装产生纳米结构体的方法
- 一种自组装纳米止血剂、其制备方法及应用
- 一种纳米片自组装棱台状(NH<sub>4</sub>)<sub>2</sub>V<sub>3</sub>O<sub>8</sub>的制备方法
- 一种干燥介导金纳米颗粒自组装制备金基底的方法及其应用
- 一种以杆菌肽为模板自组装合成球状金纳米粒子的方法