一种含重组人表皮生长因子受体抗体基因的CHO细胞株、筛选方法及其生产工艺与流程
本发明涉及“一种含重组人表皮生长因子受体抗体基因的CHO细胞株、筛选方法及其生产工艺”属于生物技术领域。
背景技术:
近年来,恶性肿瘤一直作为一种顽固性疾病危害着人类健康。目前,癌症的治疗主要还是以放疗和化疗为主,但放化疗给人体带来的损伤往往是不可修复的,如放射性肝、肺纤维化等。表皮生长因子受体(epidermal growth factor receptor,EGFR)是一种膜表面受体,其具有酪氨酸激酶活性,能够进行细胞信号传递,已有研究证实,EGFR过度表达与某些肿瘤,如非小细胞肺癌(NSCLC)、结直肠癌、乳腺癌等密切相关。在癌症患者中,通过人为干预使EGFR降低后,可以明显改善癌症患者的病情。
编码表皮生长因子受体的基因位于7号染色体的短臂1区2带,隶属于表皮生长因子(erbB)家族,该家族共有四个成员:ErbB1(EGFR)、ErbB2、ErbB3、ErbB4,EGFR在许多如头颈部癌、皮肤鳞癌、食管癌、结直肠癌等实体瘤组织和器官中过度表达。EGF结合EGFR后,促使EGFR单体进行二聚化作用而形成二聚体,受体的二聚化导致胞内区的酪氨酸蛋白激酶活性激活,随后,信号传导到细胞核,通过对核内基因的转录调控,发挥相应的生物学作用。相关研究发现,EGF途径主要包括RAS/RAF/MEK/ERK和PI3K/AKT通路,这两个通路作为一个复杂的网络而运行。EGFR的过度表达和活化导致胞内若干信号活化,不仅使细胞增殖达到高潮,而且对细胞凋亡、细胞迁移,血管生成等其他癌症的进展过程也至关重要。表皮生长因子受体(EGFR)的异常信号传导在恶性表型的发展和维持起着重要的作用,因此,该受体是一种合理的抗肿瘤靶点。
目前,临床上针对于EGFR异常表达所引起的肿瘤所使用的治疗药物主要为小分子酪氨酸激酶拮抗剂和单克隆抗体。小分子化合物酪氨酸激酶拮抗剂主要为吉非替尼和埃罗替尼。目前,已批准上市的用于治疗癌症抗体药物有20种,其中,以EGFR为靶点的治疗性单克隆抗体主要为Cetuximab、Panitumumab和Nimotuzumab三种,这些EGFR单克隆抗体可以封闭EGFR胞外区对应的配体的结合位点,抑制其特有的酪氨酸激酶活性,减慢因该信号通路所引起的癌症的恶化速度。FDA批准上市的抗hEGFR抗体中,其中,Cetuximab为人鼠嵌合单克隆抗体,其虽然可以特异性的与EGFR结合,较好的抑制癌症的发展,但在人体应用的过程中往往表现出较强的免疫原性,易产生人抗鼠反应;Panitumumab是100%的人源性单克隆抗体,其采用转基因小鼠技术制备,由balb/c小鼠细胞分泌表达及修饰,鼠源修饰可造成糖基化减弱,超敏反应,半衰期短等缺点;Nimotuzumab是一种通过人源化改造的而获得的IgG1型单克隆抗体,改造过程中将鼠源IgG2a的互补决定域(CDR)移植到人的抗体结构中,大大降低了鼠源抗体成分,然而在表达载体的构建过程中,抗体的轻、重链基因被分别连至不同的载体而使之分别表达,这种方法往往会导致轻、重链的表达比例处于非均衡状态,同时降低抗体的组装效率,使具有完整结构的抗体的产率下降。本发明中,抗体轻、重链连接到同一表达载体peedual中,实现抗体轻、重链的共表达。
全长抗体分子是由两条完整的重链和两条完整的轻链通过二硫键连接而成的四聚体蛋白,在生物表达过程中经过细胞器严格的加工修饰才能使其具有高亲和力以及与抗原特异性结合能力,因此,对其生产从质和量上的要求就更加的严格,表达系统的选择也至关重要。原核生物不含蛋白加工修饰的众多细胞器,因此,大肠杆菌表达系统只可以用于生产体积小、结构简单、不需要糖基化的抗体片断如Fab、Fab'、scFv等;低等真核生物表达系统,如酵母及丝状真菌,可用于全长抗体的生产,但其糖基化类型与人类相比存在差异,如酵母糖基化类型为多麦芽糖类型,该类型糖蛋白抗体半衰期较短、生理活性有限甚至对人体有毒性。在蛋白修饰的系统中最接近人体的是哺乳动物表达系统,该系统可以对蛋白进行适当的折叠、装配和翻译后修饰,在临床应用中占主导地位,目前,中国仓鼠卵巢细胞(Chinese hamster ovary cells,CHO cells)是最理想的真核表达宿主,通过该系统表达的蛋白在构型和构象上更接近于天然蛋白,是重组糖基化蛋白生产的首选体系。
目前,获FDA批准的抗体药物中许多是由CHO细胞生产的,CHO-K1细胞是可以悬浮生长的谷氨酰胺合成酶(GS)缺陷型细胞,因此,转染后的CHO细胞可运用共扩增基因进行筛选,即在表达载体上游加入GS基因作为筛选标记,同时利用GS抑制物甲硫胺酸亚砜(L-Methionine sulfoxide,MSX)进行加压筛选,随培养基中MSX浓度的逐渐增加,GS基因带动与之串联在一起的外源基因共同转录,使目的基因的转录效率增加,从而实现外源基因的高效表达。由于GS筛选标记不能够直观的监测及分选阳性细胞,因此,本发明在轻链的下游引入增强型绿色荧光蛋白(EGFP)基因,EGFP是GFP的突变体,具有更高的荧光强度,在488nm波长光激发下可产生绿色荧光,从而通过流式细胞仪进行检测和分选。
由于常规CHO细胞需要在有牛血清或类似成分存在的情况下贴壁生长,细胞培养基中的血清或蛋白质成分对重组蛋白在细胞中的表达以及后期的蛋白纯化过程有很大的影响。如果进行产业化,还需要首先进行无血清驯化,使细胞适应无血清培养基并在其中生长良好并持续分泌产生重组蛋白质以降低成本并方便下游纯化工艺的开发。
无血清悬浮驯化技术被广泛应用于基因工程制药。一方面是为了获得能够满足生产要求的无血清细胞株;另一方面,是为了获得无血清悬浮生长的宿主细胞,为后续细胞株的开发奠定基础,常用的宿主细胞,如CHO细胞、骨髓瘤细胞。本发明中工程细胞驯化方法采用两个阶段的驯化过程,第一步适应无血清培养条件培养,第二步使细胞适应悬浮。
技术实现要素:
本发明提供了一种可以稳定,高效表达anti-hEGFR的悬浮CHO-K1单克隆细胞株,可以用于hEGFR抗体的产业化生产,得到的anti-hEGFR可以封闭EGFR胞外区对应的配体的结合位点,抑制其特有的酪氨酸激酶活性,减慢因该信号通路所引起的癌症的恶化速度。
本发明提供的稳定,高效表达anti-hEGFR的悬浮CHO-K1单克隆细胞株,由不含内毒素的peedual-IRES-EGFP-hEGFR质粒转染进入CHO-K1细胞株,并通过甲硫胺酸亚砜加压和流式细胞仪多次筛选、流式细胞仪分选单克隆后悬浮驯化得到。
本发明还提供了稳定,高效表达anti-hEGFR的悬浮CHO-K1单克隆细胞株悬浮培养的生产工艺,即37℃、5%CO2、150rpm培养8天,分别于第3天、第5天和第7天补加10%培养体积的含1g/L Pluronic F-68OptiCHO无血清培养基,培养结束后收集上清,得到anti-hEGFR。
附图说明
图1为转染前后的CHO-K1细胞
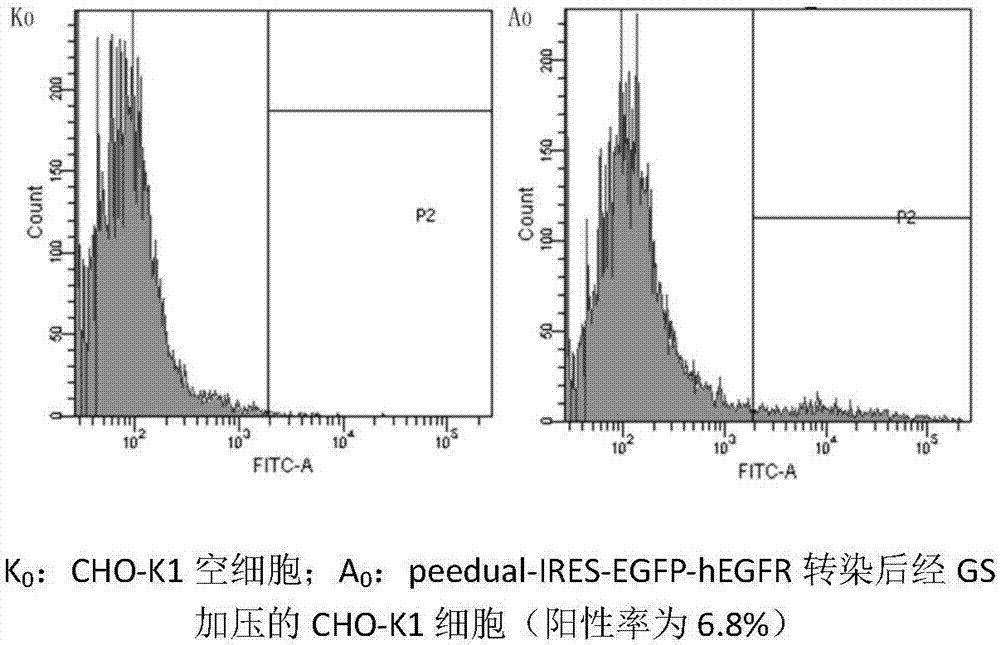
图2为转染后的经MSX加压CHO-K1细胞
图3为转染后CHO-K1阳性细胞的筛选
图4为单克隆细胞阳性率检测
图5为单克隆细胞的Dot Blot检测
图6为表达anti-EGFR重组蛋白的悬浮细胞和贴壁细胞形态
图7为悬浮培养表达anti-hEGFR CHO-K1单克隆细胞株SDS-PAGE分析
图8为anti-hEGFR纯化结果
图9为anti-hEGFR与抗原结合的ELISA分析
图10为anti-hEGFR对A431细胞周期的影响
图11为anti-hEGFR对A431细胞侵袭的抑制作用
图12为anti-hEGFR对A431细胞中MMP2、TIMP2、MMP9、TIMP1基因mRNA表达影响
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。CHO-K1细胞:本实验室保存。胎牛血清,IMDM培养基,DMEM培养基,无血清MEM培养基,OptiCHO无血清培养基:GIBCO公司。L-Glutamine,甲硫胺酸亚砜:Sigma公司。
实施例1、获得anti-hEGFR CHO-K1细胞株
CHO-K1细胞是谷氨酰胺合成酶(GS)缺陷的中国仓鼠卵巢细胞系,从细胞库中复苏一支CHO-K1细胞株,用含有10%胎牛血清含谷氨酰胺的DMEM培养基传代两次,当细胞处在对数生长期时,以1×10^6cells/ml细胞密度接种于细胞培养瓶,于37℃,5%CO2培养箱中培养直至汇合度为70~80%,用胰蛋白酶消化,加血清终止消化,1000rpm,5min,离心后用PBS洗细胞一次,将细胞用无血清MEM培养基(GIBCO公司MEM转染用无血清培养基)稀释计数,使其浓度达1×10^7cells/mL。取纯化后且不含内毒素的peedual-IRES-EGFP-hEGFR质粒DNA 20μg加入400μL细胞悬液中,250V、1 000Ω、1 000μF参数下进行电转。将转染后的细胞转入含有10%胎牛血清的完全培养基中复苏培养。电转24-30h后更换含50μM GS抑制物甲硫胺酸亚砜(L-Methionine sulfoxide,MSX),无谷氨酰胺的10%胎牛血清IMDM培养基进行加压培养。转染前后细胞形态如图1所示。转染后经MSX加压培养后anti-hEGFR CHO-K1细胞株流式细胞仪检验阳性率如图2所示。
实施例2、稳定、高效表达anti-hEGFR CHO-K1细胞株的筛选
本发明在轻链的下游引入增强型绿色荧光蛋白(EGFP)基因,在488nm波长光激发下可产生绿色荧光,可通过流式细胞仪进行检测和分选。转染后用含50μM MSX,无谷氨酰胺的10%胎牛血清IMDM培养基进行加压培养使未转入和瞬时转染的CHO-K1细胞淘汰,待稳定转染的克隆长满细胞培养瓶后进行流式细胞仪无菌分选。
分选的步骤为:将细胞用胰蛋白酶消化,加血清终止消化,1000rpm,5min,离心后用PBS洗细胞两次,500μL PBS重悬细胞,同样处理未转染的空白细胞作对照。流式细胞仪识别出的阳性率较高的细胞分选到含50μM MSX,无谷氨酰胺的10%胎牛血清IMDM培养基的六孔板中,37℃,5%CO2培养箱中培养并观察,细胞长至约5~10×10^6cells后,进行下一轮筛选。
四轮筛选完成后,得到阳性率90%以上的混合克隆群,扩大培养,冻存细胞,建立种子细胞库。四轮筛选流式细胞仪检验anti-hEGFR CHO-K1细胞株阳性率结果如图3所示。
实施例3、稳定,高效表达anti-hEGFR CHO-K1单克隆细胞株的获得
用96孔板单克隆分选方法从四筛得到的混合克隆群中筛选出稳定、高效表达的单克隆细胞株。
单克隆分选方法如下:先将96孔板每孔加入200μL、37℃提前预热的无菌含50μM MSX,无谷氨酰胺的10%胎牛血清IMDM培养基,边孔加入PBS。细胞处理步骤如实施例2分选步骤,筛选完成后将96孔板置于37℃、5%CO2恒温培养箱中培养,1~2周后利用荧光显微镜观察细胞生长情况,并更换培养基,待单克隆细胞增殖到1 000cells以上时,传至24孔培养板,根据细胞增殖数量的增加依次扩大培养,利用流式细胞仪在488nm激发光条件下,检测各单克隆细胞株的荧光强度。流式细胞仪筛选得到anti-hEGFR单克隆细胞株检验阳性率如图4所示。选择流式检测值最高的5个细胞株进行Dot blot方法检测。Dot blot检测结果如图5所示。选取荧光强度既强、Dot blot值又高的一个细胞株作为anti-hEGFR表达细胞株,扩增培养,冻存细胞。建立种子细胞库。
实施例4、稳定,高效表达anti-hEGFR CHO-K1单克隆细胞株悬浮驯化
从细胞库中复苏一支重组anti-hEGFR表达细胞株,用50μM MSX、无谷氨酰胺的10%胎牛血清IMDM培养基传代两次,当细胞处在对数生长期时,用胰蛋白酶消化,加血清终止消化,1000rpm,5min,离心,去上清,加入OptiCHO无血清培养基(GIBCO公司CHO无血清培养基),把细胞制成悬液,以5~6×10^5cells/ml接种到细胞培养瓶中,当细胞密度达到2~3×10^6cells/ml时按1:5细胞传代,培养两周,使细胞适应在无血清培养基中生长。而后1000rpm,5min,离心,去上清,加入1g/L Pluronic F-68OptiCHO无血清培养基(GIBCO公司CHO无血清培养基),制成5~6×10^5cells/ml细胞悬液,接种到无菌摇瓶中,摇床转速为150rpm,当细胞密度达到2~3×10^6cells/ml,按1:3传代,传代5~6次时,当细胞在无血清中生长速度与含血清中生长速度接近,冻存细胞。建立种子细胞库。驯化前后细胞形态对比见图6。
实施例5、稳定,高效表达anti-hEGFR CHO-K1单克隆细胞株悬浮培养
从细胞库中复苏一支重组anti-hEGFR CHO-K1细胞株到无菌摇瓶中,用含1g/LPluronic F-68OptiCHO无血清培养基传代两次,当细胞处在对数生长期时,以3~5×10^5cells/ml细胞密度接种,37℃、5%CO2、150rpm培养8天,分别于第3天、第5天和第7天补加10%培养体积的含1g/L Pluronic F-68OptiCHO无血清培养基,培养结束后收集上清。即得到anti-hEGFR。培养期间每天取微量上清,经非还原性SDS-PAGE(图7)分析可见,随着培养时间的增加,anti-hEGFR表达量不断增加,可达到0.4mg/ml。
实施例6、anti-hEGFR纯化
收集细胞培养上清约400mL后,使用AKTA Purifier 100蛋白层析系统(购自GE公司)进行抗体纯化。上样前以PH 7.0的磷酸盐缓冲液平衡proteinA柱子(购自GE公司),上样后以PH 3.0的柠檬酸-柠檬酸盐缓冲液洗脱抗体蛋白,然后将洗脱的蛋白于PBS中过夜透析。纯化后的蛋白用SDS-PAGE分析(图8)。
实施例7、Anti-hEGFR的生物学活性检测
(1)ELISA检测anti-hEGFR的亲和力
将hEGFR抗原蛋白用包被液稀释至20μg/mL后包被酶标板,4℃过夜。分别加入二倍梯度稀释的anti-hEGFR(初始浓度3μg/mL),不加抗体作为阴性对照,加Cetuximab(浓度3μg/mL)作为阳性对照,每个样品设置3个复孔。加入HRP标记的羊抗人抗体作为第二抗体,用酶标仪检测450nm波长下的A值。结果如图9所示,随着hEGFR全长抗体浓度的降低,吸光值也随之降低,在0.047μg/mL浓度时OD450值达到最低,但仍然是阴性对照的4.5倍。结果表明hEGFR全长抗体与hEGFR的作用呈现抗体浓度依赖性,并且抗体效价较高。
(2)PI染色检测细胞周期
经常规胰酶消化处理Control、anti-hEGFR和Cetuximab组A431细胞,用70%乙醇固定后并经PI染色后,进行流式细胞仪检测,细胞周期拟和软件ModFit分析结果如图10所示:未经蛋白处理的A431细胞中G1/G0期的细胞所占比重为62.43%,而anti-hEGFR组与Cetuximab组G1/G0期的细胞所占比重分别为77.96%和79.15%。anti-hEGFR组、Cetuximab组G1/G0期的细胞所占比重均显著低于空白对照组,差异显著(p<0.01);而anti-hEGFR组与Cetuximab组相比,差异不显著。
(3)全长抗体对A431细胞侵袭的影响
1)Boyden法检测Anti-hEGFR对A431细胞侵袭的抑制作用
Boyden体外侵袭实验中,anti-hEGFR、Cetuximab分别作用A431细胞48h后检测细胞穿透Matrigel人工基底膜的能力,结果(图11)显示,anti-hEGFR能够明显地减少A431细胞的穿膜细胞数(穿膜细胞数由空白对照组的34.0±2.26个减少至27.3±3.05个,p<0.01),而与Cetuximab组(穿膜细胞数为26.3±2.54个)相比无差异。结果表明,癌细胞可以移动并具有体外侵袭的能力,anti-hEGFR可以通过与癌细胞表面EGFR结合来降低A431细胞侵袭转移能力。
2)Anti-hEGFR调节A431细胞中MMP2、MMP9及TIMP2、TIMP1的表达
MMPs和TIMPs在多种组织和器官中均出现表达,主要参与细胞外基质的代谢作用,MMPs对细胞外机制有破坏性作用,而TIMPs则可以抑制MMPs的活性,如MMP2和MMP9在肿瘤细胞浸润、转移的过程中起关键性作用。MMP2/TIMP2、MMP9/TIMP1Real-time PCR结果(图12)可见,anti-hEGFR组中MMP2与MMP9表达的量高于Cetuximab组,但与空白对照相比,anti-hEGFR组中,MMP2与MMP9表达量分别是空白对照组的0.718756和0.0403273倍,差异达显著水平(p<0.05);MMP2与MMP9对应的抑制剂TIMP2、TIMP1表达情况为:anti-hEGFR组TIMP2、TIMP1表达量分别为空白对照组的4.67576倍和757.155倍,且分别为Cetuximab组的1.93281倍和2.28798倍,差异均达极显著水平(p<0.01)。可以说明,anti-hEGFR能够通过调节细胞外基质破坏性酶类因子来维持细胞外基质的稳定,从而抑制肿瘤细胞的侵袭。
序列表
<110> 东北农业大学
<120> 一种含重组人表皮生长因子受体抗体基因的CHO细胞株、筛选方法及其生产工艺
<160> 7
<170> SIPOSequenceListing 1.0
<210> 1
<211> 798
<212> DNA
<213> Artifical sequence
<220>
<221> CDS
<222> (1)..(798)
<400> 1
atg gtg agc aag ggc gag gag ctg ttc acc ggg gtg gtg ccc atc ctg 48
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
gtc gag ctg gac ggc gac gta aac ggc cac aag ttc agc gtg tcc ggc 96
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
gag ggc gag ggc gat gcc acc tac ggc aag ctg acc ctg aag ttc atc 144
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
tgc acc acc ggc aag ctg ccc gtg ccc tgg ccc acc ctc gtg acc acc 192
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
ctg acc tac ggc gtg cag tgc ttc agc cgc tac ccc gac cac atg aag 240
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys
65 70 75 80
cag cac gac ttc ttc aag tcc gcc atg ccc gaa ggc tac gtc cag gag 288
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
85 90 95
cgc acc atc ttc ttc aag gac gac ggc aac tac aag acc cgc gcc gag 336
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
gtg aag ttc gag ggc gac acc ctg gtg aac cgc atc gag ctg aag ggc 384
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
atc gac ttc aag gag gac ggc aac atc ctg ggg cac aag ctg gag tac 432
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
aac tac aac agc cac aac gtc tat atc atg gcc gac aag cag aag aac 480
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
ggc atc aag gtg aac ttc aag atc cgc cac aac atc gag gac ggc agc 528
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
gtg cag ctc gcc gac cac tac cag cag aac acc ccc atc ggc gac ggc 576
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
ccc gtg ctg ctg ccc gac aac cac tac ctg agc acc cag tcc gcc ctg 624
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
195 200 205
agc aaa gac ccc aac gag aag cgc gat cac atg gtc ctg ctg gag ttc 672
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
gtg acc gcc gcc ggg atc act ctc ggc atg gac gag ctg tac aag tcc 720
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys Ser
225 230 235 240
gga ctc aga tct cga gct caa gct tcg aat tct gca gtc gac ggt acc 768
Gly Leu Arg Ser Arg Ala Gln Ala Ser Asn Ser Ala Val Asp Gly Thr
245 250 255
gcg ggc ccg gga tcc acc gga tct aga taa 798
Ala Gly Pro Gly Ser Thr Gly Ser Arg
260 265
<210> 2
<211> 275
<212> PRT
<213> Artifical sequence
<400> 2
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Glu
65 70 75 80
Thr Lys Gln His Asp Phe Phe Lys Ser Ala Met Glu Thr Pro Glu Gly
85 90 95
Tyr Val Gln Glu Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys
100 105 110
Thr Arg Ala Glu Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile
115 120 125
Glu Leu Lys Gly Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His
130 135 140
Lys Leu Glu Tyr Asn Tyr Asn Ser His Asn Val Tyr Ile Met Glu Thr
145 150 155 160
Ala Asp Lys Gln Lys Asn Gly Ile Lys Val Asn Phe Lys Ile Arg His
165 170 175
Asn Ile Glu Asp Gly Ser Val Gln Leu Ala Asp His Tyr Gln Gln Asn
180 185 190
Thr Pro Ile Gly Asp Gly Pro Val Leu Leu Pro Asp Asn His Tyr Leu
195 200 205
Ser Thr Gln Ser Ala Leu Ser Lys Asp Pro Asn Glu Lys Arg Asp His
210 215 220
Met Glu Thr Val Leu Leu Glu Phe Val Thr Ala Ala Gly Ile Thr Leu
225 230 235 240
Gly Met Glu Thr Asp Glu Leu Tyr Lys Ser Gly Leu Arg Ser Arg Ala
245 250 255
Gln Ala Ser Asn Ser Ala Val Asp Gly Thr Ala Gly Pro Gly Ser Thr
260 265 270
Gly Ser Arg
275
<210> 3
<211> 1385
<212> DNA
<213> Artifical sequence
<400> 3
gcccctctcc ctcccccccc cctaacgtta ctggccgaag ccgcttggaa taaggccggt 60
gtgcgtttgt ctatatgtta ttttccacca tattgccgtc ttttggcaat gtgagggccc 120
ggaaacctgg ccctgtcttc ttgacgagca ttcctagggg tctttcccct ctcgccaaag 180
gaatgcaagg tctgttgaat gtcgtgaagg aagcagttcc tctggaagct tcttgaagac 240
aaacaacgtc tgtagcgacc ctttgcaggc agcggaaccc cccacctggc gacaggtgcc 300
tctgcggcca aaagccacgt gtataagata cacctgcaaa ggcggcacaa ccccagtgcc 360
acgttgtgag ttggatagtt gtggaaagag tcaaatggct ctcctcaagc gtattcaaca 420
aggggctgaa ggatgcccag aaggtacccc attgtatggg atctgatctg gggcctcggt 480
gcacatgctt tacatgtgtt tagtcgaggt taaaaaaacg tctaggcccc ccgaaccacg 540
gggacgtggt tttcctttga aaaacacgat gatatatggc cacaaccatg gtgagcaagg 600
gcgaggagct gttcaccggg gtggtgccca tcctggtcga gctggacggc gacgtaaacg 660
gccacaagtt cagcgtgtcc ggcgagggcg agggcgatgc cacctacggc aagctgaccc 720
tgaagttcat ctgcaccacc ggcaagctgc ccgtgccctg gcccaccctc gtgaccaccc 780
tgacctacgg cgtgcagtgc ttcagccgct accccgacca catgaagcag cacgacttct 840
tcaagtccgc catgcccgaa ggctacgtcc aggagcgcac catcttcttc aaggacgacg 900
gcaactacaa gacccgcgcc gaggtgaagt tcgagggcga caccctggtg aaccgcatcg 960
agctgaaggg catcgacttc aaggaggacg gcaacatcct ggggcacaag ctggagtaca 1020
actacaacag ccacaacgtc tatatcatgg ccgacaagca gaagaacggc atcaaggtga 1080
acttcaagat ccgccacaac atcgaggacg gcagcgtgca gctcgccgac cactaccagc 1140
agaacacccc catcggcgac ggccccgtgc tgctgcccga caaccactac ctgagcaccc 1200
agtccgccct gagcaaagac cccaacgaga agcgcgatca catggtcctg ctggagttcg 1260
tgaccgccgc cgggatcact ctcggcatgg acgagctgta caagtccgga ctcagatctc 1320
gagctcaagc ttcgaattct gcagtcgacg gtaccgcggg cccgggatcc accggatcta 1380
gataa 1385
<210> 4
<211> 645
<212> DNA
<213> Artifical sequence
<220>
<221> CDS
<222> (1)..(645)
<400> 4
gac atc ttg ctg acc cag tct cca gtc atc ctg tcc gtg agt cca gga 48
Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly
1 5 10 15
gaa aga gtc agt ttc tcc tgc agg gcc agt cag agt att ggc aca aac 96
Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn
20 25 30
ata cac tgg tat cag caa aga aca aat ggt tct cca agg ctt ctc ata 144
Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile
35 40 45
aag tat gct tct gag tct atc tct ggg atc cct tcc agg ttt agt ggc 192
Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
agt gga tca ggg aca gat ttt act ctt agc atc aac agt gtg gag tct 240
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser
65 70 75 80
gaa gat att gca gat tat tac tgt caa caa aat aat aac tgg cca acc 288
Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr
85 90 95
acg ttc ggt gct ggc acc aag ctg gag ctg aag cga act gtg gct gca 336
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
cca tct gtc ttc atc ttc cct cca tct gat gag cag ttg aaa tct gga 384
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
act gcc tct gtt gtg tgc ctg ctg aat aac ttc tat ccc aga gag gcc 432
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
aaa gtg cag tgg aag gtg gat aac gcc ctc caa tcc ggt aac tcc cag 480
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
gag agt gtc aca gag cag gac agc aag gac agc acc tac agc ctc agc 528
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
agc acc ctg acg ctg agc aaa gca gac tac gag aaa cac aaa gtc tac 576
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
gcc tgc gaa gtc acc cat cag ggc ctg agc tct ccc gtc aca aag agc 624
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
ttc aac agg gga gag tgt tga 645
Phe Asn Arg Gly Glu Cys
210
<210> 5
<211> 214
<212> PRT
<213> Artifical sequence
<400> 5
Asp Ile Leu Leu Thr Gln Ser Pro Val Ile Leu Ser Val Ser Pro Gly
1 5 10 15
Glu Arg Val Ser Phe Ser Cys Arg Ala Ser Gln Ser Ile Gly Thr Asn
20 25 30
Ile His Trp Tyr Gln Gln Arg Thr Asn Gly Ser Pro Arg Leu Leu Ile
35 40 45
Lys Tyr Ala Ser Glu Ser Ile Ser Gly Ile Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Ser Ile Asn Ser Val Glu Ser
65 70 75 80
Glu Asp Ile Ala Asp Tyr Tyr Cys Gln Gln Asn Asn Asn Trp Pro Thr
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 6
<211> 1350
<212> DNA
<213> Artifical sequence
<220>
<221> CDS
<222> (1)..(1350)
<400> 6
cag gtg cag ctg aag cag tca gga cct ggc ctc gtg cag ccc tca cag 48
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
agc ctg tcc atc acc tgc aca gtc tct ggt ttc tca tta act aac tat 96
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
ggt gta cac tgg gtt cgc cag tct cca gga aag ggt ctg gag tgg ctg 144
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
gga gtg ata tgg agt ggt gga aac aca gac tat aat aca cct ttc aca 192
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
tcc aga ctg agc atc aac aag gac aat tcc aag agc caa gtt ttc ttt 240
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
aaa atg aac agt ctg caa tct aat gac aca gcc ata tat tac tgt gcc 288
Lys Met Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
aga gcc ctc acc tac tat gat tac gag ttt gct tac tgg ggc caa ggg 336
Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly Gln Gly
100 105 110
act ctg gtc act gtc tct gca gcc tcc acc aag ggc cca tcc gtc ttc 384
Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser Val Phe
115 120 125
ccc ctg gca ccc tcc tcc aag agc acc tct ggg ggc aca gcc gcc ctg 432
Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu
130 135 140
ggc tgc ctg gtc aag gac tac ttc ccc gaa cct gtg acg gtg tcc tgg 480
Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp
145 150 155 160
aac tca ggc gcc ctg acc agc ggc gtg cac acc ttc ccg gct gtc ctc 528
Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu
165 170 175
cag tcc tca gga ctc tac tcc ctc agc agc gtg gtg acc gtg ccc tcc 576
Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser
180 185 190
agc agc ttg ggc acc cag acc tac atc tgc aac gtg aat cac aag ccc 624
Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro
195 200 205
agc aac acc aag gtg gac aag cgc gtt gag ccc aaa tct tgt gac aaa 672
Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys Asp Lys
210 215 220
act cac aca tgc cca cct tgc cca gca cct gaa ctc ctg ggg gga cct 720
Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro
225 230 235 240
tca gtc ttc ctc ttc ccc cca aaa ccc aag gac acc ctc atg atc tcc 768
Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser
245 250 255
cgg acc cct gag gtc aca tgc gtg gtg gtg gac gtg agc cac gaa gac 816
Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp
260 265 270
cct gag gtc aag ttc aac tgg tac gtg gac ggc gtg gag gtg cat aat 864
Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn
275 280 285
gcc aag aca aag cct cgg gag gag cag tac aac agc acg tac cgg gtg 912
Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val
290 295 300
gtc agc gtc ctc acc gtc ctg cac cag gac tgg ctg aat ggc aag gag 960
Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu
305 310 315 320
tac aag tgc aag gtc tcc aac aaa gcc ctc cca gcc ccc atc gag aaa 1008
Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys
325 330 335
acc atc tcc aaa gcc aaa ggg cag ccc cga gaa cca cag gtg tac acc 1056
Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr
340 345 350
ctg ccc cca tcc cgg gag gag atg acc aag aac cag gtc agc ctg acc 1104
Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr
355 360 365
tgc ctg gtc aaa ggc ttc tat ccc agc gac atc gcc gtg gag tgg gag 1152
Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu
370 375 380
agc aat ggg cag ccc gag aac aac tac aag acc acg cct ccc gtg ctg 1200
Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu
385 390 395 400
gac tcc gac ggc tcc ttc ttc ctc tat agc aag ctc acc gtg gac aag 1248
Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys
405 410 415
agc agg tgg cag cag ggg aac gtc ttc tca tgc tcc gtg atg cat gag 1296
Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu
420 425 430
gct ctg cac aac cac tac acg cag aag agc ctc tcc ctg tct ccc ggt 1344
Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly
435 440 445
aaa tga 1350
Lys
<210> 7
<211> 457
<212> PRT
<213> Artifical sequence
<400> 7
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Asn Tyr
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Asn Thr Asp Tyr Asn Thr Pro Phe Thr
50 55 60
Ser Arg Leu Ser Ile Asn Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Glu Thr Asn Ser Leu Gln Ser Asn Asp Thr Ala Ile Tyr Tyr
85 90 95
Cys Ala Arg Ala Leu Thr Tyr Tyr Asp Tyr Glu Phe Ala Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Arg Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Glu Thr Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Glu Thr Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met Glu Thr His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
- 还没有人留言评论。精彩留言会获得点赞!
- 一种阿法替尼有关物质的分离检测方法与流程
- 用于检测人血清中HER2含量的ELISA试剂盒、使用方法及用途与流程
- 用于预防或治疗皮疹的药物组合物的制造方法与工艺
- 用于汽车内饰表皮的30ST/R双面布的制造方法与工艺
- 检测EGFR耐药性突变的方法和组合物与制造工艺
- 一种肝素结合表皮生长因子的单域抗体及其应用的制造方法与工艺
- 3‑[(3‑{[4‑(4‑吗啉基甲基)‑1H‑吡咯‑2‑基]亚甲基}‑2‑氧代‑2,3‑二氢‑1H‑吲哚‑5‑基)甲基]‑1,3‑噻唑烷‑2,4‑二酮和EGFR TYR激酶抑制剂的组合的制造方法与工艺
- 一种二甲苯磺酸拉帕替尼片剂及其制备方法与制造工艺
- 苯并咪唑衍生物作为ERBB酪氨酸激酶抑制剂用于治疗癌症的制造方法与工艺
- 包含STP和生长因子的活性组合物及其化妆品或护肤品的制造方法与工艺