与节球藻毒素-R特异性结合的适配体及其应用的制作方法
本发明涉及生物医药工程技术领域,具体涉及一种与节球藻毒素-r特异性结合的适配体及其应用。
背景技术
节球藻毒素-r(nodularin-r,nod-r)是一种强烈的蓝藻肝毒素,主要由丝状浮游蓝藻节球藻(nodulariaspumigena)产生。研究表明,nod-r不仅具有促癌作用,而且还是一种致癌剂,其作用机理为通过抑制丝氨酸/苏氨酸蛋白磷酸酶1(pp1)和2a(pp2a)的活性,进而发挥毒性作用。自1988年起,研究者们共发现了十种nod,其中,nod-r是含量最多的,也是唯一商业化的毒素。nod-r易溶于水,而且耐热性强,加热煮沸都不能将毒素破坏。人们主要因饮用被污染的水和误食被污染的水产品而中毒,中毒症状包括腹泻、呕吐和口眼分泌物增多等,严重时可导致死亡。国内外卫生组织都将饮用水中nod-r的最高含量规定为1μg/l。随着水体富营养化的加剧,蓝藻水华事件愈加频繁,nod-r的污染也越来越严重。因此,亟需建立一种准确、灵敏的nod-r检测方法,以便更好的监控水体和水产品中nod-r的水平,进而保障人们和水生生物的安全。
目前,常用的nod-r的检测方法主要分为三大类,即生物分析法、物理化学分析法和免疫化学法。其中,小鼠生物法(mba)是应用最广的生物分析方法。然而,该方法不仅灵敏度低、重现性差,而且还存在伦理道德问题。化学分析法,如高效液相色谱法(hplc)、毛细管电泳法(ce)和液质联用(lc-ms)等也逐渐建立。其中,多种化学仪器联用的方法因能够大大提高检测的灵敏度而获得了越来越多的研究者们的青睐。然而,该方法需要进行复杂的样品前处理过程,所用仪器设备昂贵,而且还需要专业的技术人员。免疫化学法因灵敏度低、特异性高而被广泛应用,但是,该方法不仅步骤繁琐,而且还存在交叉反应。因此,亟需建立一种快速、灵敏的新型检测方法。
近年来,生物传感器检测法因能克服传统检测方法的众多缺点而备受研究者们的关注。生物传感器的核心部分是分子识别元件,抗体是一种很好的分子识别元件,但是,抗体不仅价格昂贵,而且容易变性。因此,需要寻找一种新的分子识别元件。
1990年,tuerk和gold首次通过指数富集的配基系统进化技术(systematicevolutionofligandsbyexponentialenrichment,selex),成功筛选到了亲和力高、特异性强的t4dna聚合酶适配体。适配体,即具有功能的单链寡核苷酸,包括ssdna和rna,能够形成发夹、假结和g-四链体等空间结构,并通过分子间的相互作用,如疏水作用、范德华力和氢键等特异性识别并结合靶分子。作为一种新型分子识别元件,适配体与抗体相似,但优于抗体。例如,适配体能够识别多种靶标,如蛋白质、氨基酸以及金属离子等,甚至细胞和病毒;适配体能够与多种基团连接,修饰方便;适配体化学性质稳定,不容易变性;适配体可以直接化学合成,成本低廉。
目前,已经有很多研究者们将适配体与多种生物传感器平台,如电化学、比色分析和表面等离子共振等相结合,制备快速、灵敏的新型蓝藻毒素检测方法。然而,还没有关于nod-r适配体的相关报道。
技术实现要素:
本发明是为解决上述技术问题进行的,目的在于提供一种与节球藻毒素-r特异性结合的适配体及其应用。
本发明的目的在于提供多条能够与nod-r以很高的亲和力特异性结合的单链dna适配体,并对该多条适配体与nod-r之间的亲和力进行测试,得到亲和力常数(kd)值最小的一条适配体n13。本发明的第二目的在于以截短的方式对该适配体n13进行优化,提供结合能力相当,但是长度更短的nod-r适配体(n13-t-02)。本发明的第三目的在于提供适配体的应用,如适配体在制备节球藻毒素-r分离富集试剂中的应用,在制备节球藻毒素-r检测试剂、试剂盒或传感器中的应用,在制备治疗节球藻毒素-r中毒药物中的应用,以及在自来水样品中nod-r的快速检测中的应用,并为预防或治疗nod-r中毒药物的制备和水体或水产品中nod-r的去除奠定基础。
本发明的主要技术方案是:通过磁珠-selex技术筛选并得到一条能够与nod-r以很高的亲和力特异性结合的单链dna适配体(n13)。根据在线工具themfoldwebserver对其二级结构的预测结果,对适配体n13进行截短,并得到优化后的适配体n13-t-02。通过与生物传感器平台相结合,可以制备nod-r适配体传感器,并将其用于nod-r的快速检测。此外,nod-r适配体还可以为预防或治疗nod-r中毒药物的制备以及水体或水产品中nod-r的去除奠定基础。
本发明的第一方面提供了与节球藻毒素-r特异性结合的适配体,该适配体的序列通式为:5’-aaggagcagcgtggaggata-n40-ttagggtgtgtcgtcgtggt-3’;其中,n为a、t、g、c四种脱氧核糖核苷酸碱基中的任意一个,40代表随机碱基的数量。
通过磁珠-selex技术筛选,得到如下代表性序列:
n7:如seqidno.1所示;
n13:如seqidno.2所示;
n32:如seqidno.3所示;
n36:如seqidno.4所示;
n49:如seqidno.5所示;
n62:如seqidno.6所示;
n71:如seqidno.7所示。
本发明的第二方面是以截短的方式对适配体n13进行优化,提供结合能力相当,但是长度更短的四条nod-r适配体,分别被命名为适配体n13-t-o1、适配体n13-t-o2、适配体n13-t-o3和适配体n13-t-o4。在这4条适配体中,除了适配体n13-t-o4,其余适配体都能和nod-r结合,而且亲和力均有所提高,其中,适配体n13-t-o2与nod-r之间的亲和力最高,达到了29.6nm。
适配体n13-t-o1~适配体n13-t-o3的序列如seqidno.9~seqidno.11所示,优选n13-t-o2序列。
优选的,上述适配体n13-t-o2,可以在其3’端或5’端进行生物素、fitc和巯基等化学修饰。
本发明的第三方面提供了适配体的应用,如在制备节球藻毒素-r分离富集试剂中的应用;在制备节球藻毒素-r检测试剂、试剂盒或传感器中的应用;在制备治疗节球藻毒素-r中毒药物中的应用。
优选的,治疗节球藻毒素-r中毒药物是以与节球藻毒素-r特异性结合的适配体作为唯一活性成份或者是包含与节球藻毒素-r特异性结合适配体的药物组合物。
本发明的第四方面,提供了一种与节球藻毒素-r特异性结合的适配体的药物组合物,以与节球藻毒素-r特异性结合的适配体作为活性成份,还包括医学上可接受的药物载体。
优选的,药物组合物为去除水体或水产品中节球藻毒素-r制剂。
本发明的药物组合物和药学上可以接受的辅料一起组成药物制剂组合物,从而更稳定地发挥疗效,这些制剂可以保证本发明公开的适配体核心序列的构像完整性,同时还能保护蛋白质的多官能团,防止其降解(包括但不限于凝聚、脱氨或氧化)。
本发明的第五方面是提供了适配体n13-t-02在自来水样品中nod-r的快速检测中的应用,防止人们主要因饮用被污染的水和误食被污染的水产品而中毒。
本发明的有益保障及效果:
本发明提供了一种与节球藻毒素-r特异性结合的适配体及其应用,通过实验验证,本发明的适配体能够快速特异性地与节球藻毒素-r结合,其中适配体n13-t-o2与nod-r之间的亲和力最高,达到了29.6nm。因此,本发明筛选得到的适配体可以制备成适配体传感器或检测试剂,并将其应用于饮用水体样品中nod-r的检测。另外,这些适配体还可以为预防或治疗nod-r中毒药物的制备以及水体或水产品中nod-r的去除奠定基础。
此外,本发明根据nod-r分子的特点,设计了磁珠-selex,通过正向筛选和反向筛选,获得了能够与节球藻毒素-r以很高的亲和力特异性结合的单链dna适配体,具有操作简便、重复性高等特点,大大简化了构建技术路线,生产成本低、纯化周期短。适配体作为一种新型分子识别探针,具有成本低廉、性质稳定和方便修饰等优点,适宜于在生物医药工业化生产中大规模应用。
附图说明
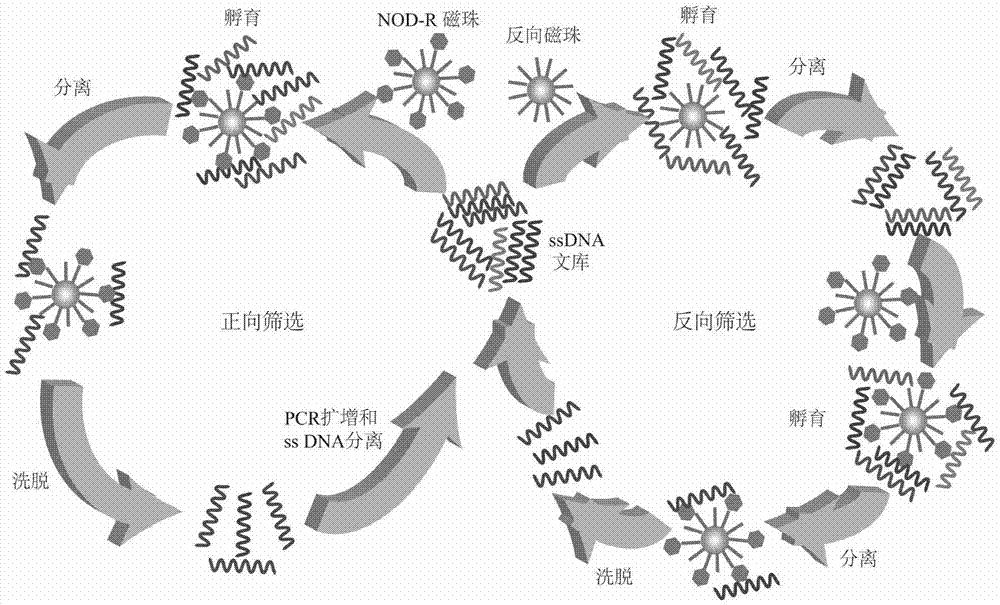
图1是nod-r适配体筛选的流程图;
图2是筛选过程中每一轮的单链dna回收率结果图;
图3是截短后适配体n13的二级结构预测图,其中a为n13、b为n13-t、c为n13-t-o1、d为n13-t-o2、e为n13-t-o3、f为n13-t-o4;
图4是适配体n13-t-02的亲和力和特异性鉴定图。
具体实施方式
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。
下列实施例中未注明具体条件的实验方法,通常按照常规条件如sambrook等人,分子克隆:实验室指南(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数按重量计算。除非另行定义,文中所使用的所有专业与科学用语与本领域熟练人员所熟悉的意义相同。此外,任何与所记载内容相似或均等的方法及材料皆可应用于本发明中。文中所述的较佳实施方法与材料仅作示范之用。
实施例1.随机ssdna文库及其引物的设计
1.ssdna文库的设计
nod-r适配体文库由80个碱基组成,两端均为含有20个碱基的固定区域,中间则为含有40个碱基的随机区域:5’-aaggagcagcgtggaggata-n40-ttagggtgtgtcgtcgtggt-3’(n为a、t、g、c四种脱氧核糖核苷酸碱基中的任意一种,40代表随机碱基的数量)。
2.引物的设计
上游引物:5’-aaggagcagcgtggaggata-3’(seqidno.13)
下游引物a:5’-accacgacgacacaccctaa-3’(seqidno.14)
下游引物b:5’-poly(da20)-spacer18-accacgacgacacaccctaa-3’(seqidno.15)。
其中,下游引物a主要用于筛选中ssdna的扩增,下游引物b主要用于克隆测序。
实施例2.nod-r适配体的筛选
根据nod-r分子的特点,采用经典的适配体筛选方法——磁珠法进行筛选。
如图1所示,筛选流程主要包括四个步骤,即孵育、分离、洗脱和扩增,表1给出了具体筛选方案,具体过程如下:
(1)孵育:取3nmolssdna文库(第二轮到第五轮文库量为200pmol,第六轮到第十二轮文库量为120pmol)进行变复性处理,即先在95℃水浴中热浴10min;然后,冰浴猝冷5min;最后,在室温下放置5min;同时,用筛选缓冲液洗涤nod-r磁珠数次,并取20μl加入到经过上述处理的ssdna文库中;加入一定量的筛选缓冲液,使总体积为600μl,并在室温下旋转孵育2h。
(2)分离:2h后,利用磁力架实现溶液中的ssdna和结合在磁珠上的ssdna的分离。为了去除非特异性吸附的ssdna,在磁性分离后,需要用筛选缓冲液洗涤磁珠3-5次。
(3)洗脱:洗涤结束后,向磁珠中加入80μl无酶水,并在95℃水浴中变性20min,回收特异性结合的ssdna(共三次)。
(4)扩增:以特异性结合的ssdna作为pcr模板对其进行扩增。pcr反应体系(50μl)为:模板2.5μl;hotstartpremix(2×)25μl;上游引物(10μm)2.5μl;下游引物(10μm)2.5μl;无酶水17.5μl。pcr反应条件为:95℃,5min;95℃,30s;62℃,45s;72℃,30s;72℃,5min,共25个循环。气溶胶的存在是筛选失败的重要原因之一,因此,在每次pcr扩增时都需要设置空白对照。
为了提高筛选的效率,从第六轮开始进行反向筛选。与正向筛选的不同之处在于:在用nod-r磁珠与ssdna文库进行孵育以前,先将ssdna文库与空白磁珠在室温下旋转孵育,孵育结束以后,用磁力架分离溶液中的ssdna和结合在空白磁珠上的ssdna,回收得到的溶液中的ssdna即为正向筛选的ssdna文库。
表1nod-r适配体筛选方案
经上述pcr扩增得到的dna是双链的,适配体则是单链的dna。为了获得ssdna次级文库,特意设计了含有20个鸟嘌呤的下游引物b,使pcr后的两条链相差20个碱基,从而通过尿素变性聚丙烯酰胺凝胶电泳对目标链进行切胶回收。
尿素聚丙烯酰胺凝胶电泳的操作步骤如下:(1)将等体积的尿素上样缓冲液(2×)和pcr产物混合,充分混匀后在95℃下变性10min。(2)将上述样品缓慢加入到凝胶上样孔中,接通电泳仪,并使样品在恒定电压(230v)下电泳至溴酚蓝迁移到凝胶底端。(3)电泳结束后,首先,用剥胶板将凝胶剥离,然后,将凝胶放入盛有核酸染料(gel-red,10000×)的玻璃皿中,并在四维旋转混匀仪上染色20min。(4)染色完成后,将凝胶置于凝胶成像仪下显像并观察。如果空白对照中能观察到目的条带,则该轮筛选失败;如果没有,则可以对实验组中的目的条带进行切胶回收。
将回收的凝胶离心破碎后,加入2倍体积的煮胶缓冲液,并在50℃水浴锅中孵育1h。孵育结束后,对样品进行离心(12000rpm,5min),收集上清。采用凝胶提取试剂盒对回收得到的ssdna进行纯化。纯化完成后,用核酸定量荧光计
如图2所示,当筛选到第12轮的时候,ssdna的回收率不再上升,预示筛选达到了终点。于是,利用上游引物和下游引物a对第12轮洗脱下来的ssdna进行pcr扩增,并用核酸回收试剂盒对经聚丙烯酰胺凝胶电泳回收的dsdna进行纯化以后,委托上海生工生物工程股份有限公司进行克隆测序,总共测得了80条序列。在用clustalx2.1软件对所有序列进行比对分析以后,从中挑选了10条具有代表性的序列进行亲和力测定(生物膜干涉技术),具体如表2所示。
表2mb-selex筛选出的代表性序列的信息及亲和力值
实施例3.适配体n13的优化
在选取的10条适配体中,适配体n13与nod-r之间亲和力常数(kd)值最小,为0.138μm。为了得到适配体n13的核心结构,对该适配体进行了截短优化。
图3显示了在线工具themfoldwebserver对截短后适配体n13的二级结构预测图。
当适配体n13两端的固定区域被去除以后(n13-t),适配体依然能够与nod-r结合,而且kd值与截短前相当。因此,可以推断:适配体n13两端的引物结合区域不参与其与nod-r之间的结合。
根据在线工具themfoldwebserver对适配体n13-t二级结构的预测结果,对其进行了进一步的截短,并获得了适配体n13-t-o1、适配体n13-t-o2、适配体n13-t-o3和适配体n13-t-o4。在这4条适配体中,除了适配体n13-t-o4,其余适配体都能和nod-r结合,而且亲和力均有所提高。其中,适配体n13-t-o2与nod-r之间的亲和力最高,达到了29.6nm。通过比较适配体n13-t-o1、适配体n13-t-o2、适配体n13-t-o3和适配体n13-t-o4的二级结构,可以推断:适配体n13-t中较大的茎环结构是其核心结构。考虑到时间和成本问题,我们没有进行进一步优化,而是以亲和力最高的适配体n13-t-o2作为最佳适配体,并将该适配体作为今后研究的对象。
本实施例中,n13及其截短后的序列信息和亲和力信息值如表3所示:
表3n13及其截短后的序列信息和亲和力值
实施例4.生物膜干涉技术分析分子间的相互作用
生物膜干涉技术是一种不需要标记的技术,可以提供实时、高通量的生物分子相互作用信息。其基本原理是仪器发射白光到传感器表面并收集光学膜层两个表面的反射光。因为不同频率的反射光受到光学膜层厚度和质量密度的影响有差异,一些频率的反射光形成了相长干涉(蓝色),另一些则形成了相消干涉(红色)。这些干涉光被光谱仪检测到以后就形成了一幅干涉光谱,并以干涉光谱的相位位移强度(δλ)显示。因此,当结合到传感器表面的分子有数量上的变化的时候,光谱仪就会检测到干涉光谱的位移。而这种位移能够直接反映传感器表面生物膜厚度和质量密度的变化,从而对待测分子间的相互作用过程进行精确的定量测定。作为一种新型的非标记技术,生物膜干涉技术已经在生物分子间的相互作用研究中占据着重要地位。
将适配体的5’端用生物素修饰,通过生物素和链霉亲合素相互作用将适配体固定在超级链霉亲和素(ssa)传感器表面。固定前,首先要对适配体进行变复性处理,帮助适配体重新折叠成稳定的空间结构。在用生物膜干涉仪octetred96进行检测前,依次将缓冲液、生物素标记的适配体溶液、缓冲液以及nod-r溶液加入96孔板的不同列中,加样体积为200μl。octetred96系统的程序设定为:(1)传感器平衡(2min);(2)适配体耦合(3min);(3)传感器再平衡(2min);(4)nod-r结合(5min);(5)nod-r解离(5min)。所有的步骤都是在室温下完成的,震荡的速度为1000rpm。检测完成后,利用octet数据分析软件cfrpart11version6.x将实验组传感器的响应值中扣除对照组传感器的响应值,得到校正后的实际响应值。另外,采用1∶1的结合模式对响应数据进行拟合,从而得到适配体与nod-r的结合-解离曲线以及各种动力学参数,包括结合速率常数kon、解离速率常数kdis和亲和常数kd值。
利用上述方法对适配体n13-t-o2和nod-r、gtx、stx、btx、ptx、mc-lr以及随机序列(randomsequence)之间的相互作用进行测试,结果如图4所示。根据图4可知,n13-t-o2仅能够和nod-r特异性结合,结合速率常数为5.44e+041/ms,解离速率常数为1.61e-031/s,是nod-r典型的快结合慢解离的配体,具有巨大的实际应用的价值。
以上已对本发明的实例进行了具体说明,但本发明并不限于所述实施例,熟悉本领域的技术人员在不违背本发明创造精神的前提下还可做出种种的等同的变型或替换,这些等同的变型或替换均包含在本申请权利要求所限定的范围内。
序列表
<110>中国人民解放军第二军医大学
<120>与节球藻毒素-r特异性结合的适配体及其应用
<130>权利要求书说明书
<160>15
<170>siposequencelisting1.0
<210>1
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>1
aaggagcagcgtggaggatatgggcgggacaaaaagggtttggaggataggtcagttcac60
ttagggtgtgtcgtcgtggt80
<210>2
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>2
aaggagcagcgtggaggataccgtgtggtatgattctaggctcgaagtcgtgcatctgca60
ttagggtgtgtcgtcgtggt80
<210>3
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>3
aaggagcagcgtggaggatagctgcgacgagtctatcgcttctgcaatggtccgtcttga60
ttagggtgtgtcgtcgtggt80
<210>4
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>4
aaggagcagcgtggaggataggtggtgattgtctctataggtctttgatttgttgttggg60
ttagggtgtgtcgtcgtggt80
<210>5
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>5
aaggagcagcgtggaggatacagctcagaagcttgatcctcgtcgccttattgattgtag60
ttagggtgtgtcgtcgtggt80
<210>6
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>6
aaggagcagcgtggaggatagctggcacaggtcccgggtaatttagtgtgggtcataggg60
ttagggtgtgtcgtcgtggt80
<210>7
<211>80
<212>dna
<213>人工序列(artificialsequence)
<400>7
aaggagcagcgtggaggatatatccgccggccagatatccccgataaatttacgccacgt60
ttagggtgtgtcgtcgtggt80
<210>8
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>8
ccgtgtggtatgattctaggctcgaagtcgtgcatctgca40
<210>9
<211>34
<212>dna
<213>人工序列(artificialsequence)
<400>9
ggtatgattctaggctcgaagtcgtgcatctgca34
<210>10
<211>27
<212>dna
<213>人工序列(artificialsequence)
<400>10
tgattctaggctcgaagtcgtgcatct27
<210>11
<211>16
<212>dna
<213>人工序列(artificialsequence)
<400>11
ggctcgaagtcgtgca16
<210>12
<211>12
<212>dna
<213>人工序列(artificialsequence)
<400>12
taccgtgtggta12
<210>13
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>13
aaggagcagcgtggaggata20
<210>14
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>14
accacgacgacacaccctaa20
<210>15
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>15
aaaaaaaaaaaaaaaaaaaaaccacgacgacacaccctaa40
- 还没有人留言评论。精彩留言会获得点赞!