一种丙二酸盐克罗诺杆菌CRISPR分型方法与流程
本发明属于分子流行病学领域,具体涉及一种丙二酸盐克罗诺杆菌crispr分型方法。
背景技术:
婴幼儿的健康一直是国家和社会关注的重点,对于人工喂养的新生儿,奶粉及其替代食品的安全至关重要。克罗诺杆菌又名阪崎肠杆菌(cronobacter(enterobactersakazakii))为奶粉中检出的一种强致病性细菌,可导致婴幼儿脑膜炎,坏死性小肠结肠炎和菌血症,死亡率高达40%到80%,已经引起了全球的高度重视。目前认为婴幼儿配方奶粉污染克罗诺杆菌是新生儿感染该菌的主要渠道,国际粮农组织和世界卫生组织把克罗诺杆菌属列为婴儿配方奶粉中的a类致病菌。然而近期亦出现母乳喂养的婴幼儿感染克罗诺杆菌的案例报道,时至今日,该菌的宿主和传播模型尚不清晰。同时克罗诺杆菌也可以感染老年人及免疫力低下的成年人,症状相对较轻,近期有克罗诺杆菌引发食物中毒的报道。克罗诺杆菌属包含7个种,丙二酸克罗诺杆菌(cronobactersakazakii)是其中一个重要的致病种。我们前期开展的全国食源性致病菌污染调结果显示熟食,蔬菜和食用菌等食品中均发现丙二酸盐克罗诺杆菌污染的情况,建立有效的分型方法对于早期分子溯源十分必要。
近年来,随着技术的进步,除了脉冲场凝胶电泳(pfge)技术分辨率高,重复性好,被誉为病原微生物分子分型的“金标准”之外,其他以电泳为基础的分型手段已经少用于病原菌的分子溯源了。大量基于基因测序的分型手段应用于病原菌的分子溯源如应用广泛的多序列分型方法(multi-locussequencetyping,mlst),核心基因组多序列分型方法(coregenomemulti-locussequencetyping,cgmlst),全基因组多序列分型方法(wholegenomemulti-locussequencetyping,wgmlst)等,基于全基因组分型的效果是最佳的,但是基因组测序成本高,操作人员需要有较好的生物信息学分析基础,推广应用有一定的难度。
crispr-cas系统是近期发现的一种新的原核生物防御外来基因组包括噬菌体入侵的有效免疫机制。crispr-cas系统由crispr分子和cas基因组成。由于crispr-cas系统中的crispr分子保存了细菌遭受噬菌体等基因水平转移形式的外源dna分子入侵的信息,且间隔序列的排列顺序包含时间信息,对于追溯细菌遗传进化规律具有其他分型方法无法企及的优势,因此已经被用于一些病原菌的基因分型和分子溯源研究中,如结核分枝杆菌菌株的分型,沙门氏菌,鼠疫耶尔森菌和空肠弯曲杆菌等。
技术实现要素:
本发明针对现有技术的不足,提供一种丙二酸盐克罗诺杆菌的crispr分型方法。
本发明的目的是提供一种非疾病的诊断和治疗目的的丙二酸盐克罗诺杆菌crispr分型方法,包括以下步骤:
a.提取待检测菌株培养物的dna;
b.分别以步骤a的dna作为模板,进行crispr1、crispr2位点和crispr3位点的pcr扩增,其中crispr1位点的扩增引物为e-1f和e-1r,crispr2位点的扩增引物为e-2f和e-2r、crispr3位点的扩增引物为e-3f和e-3r;对crispr3位点进行pcr扩增,若e-3f和e-3r扩增出条带,则加扩crispr6位点,若无扩增出条带,则不需要加扩crispr6位点,其中crispr6位点的扩增引物为e-6f-mal和e-3r6r;
所述的引物e-1f,其序列如sedidno.1所示,
所述的引物e-1r,其序列如sedidno.2所示,
所述的引物e-2f,其序列如sedidno.3所示,
所述的引物e-2r,其序列如sedidno.4所示;
所述的引物e-3f,其序列如sedidno.5所示,
所述的引物e-3r,其序列如sedidno.6所示;
所述的引物e-6f-mal,其序列如sedidno.7所示,
所述的引物e-3r6r,其序列如sedidno.8所示;
c.分别对步骤b所述的crispr1、crispr2、crispr3和crispr6位点的pcr扩增产物进行dna测序;
d.提取crispr1、crispr2、crispr3和crispr6位点序列中的间隔序列,根据间隔序列的组合确定丙二酸盐克罗诺杆菌的crispr型别。
优选,采用50μl的pcr扩增体系,其中含有
优选,所述的步骤b中,若利用引物e-3f和e-3r经pcr扩增反应后检测无条带,则利用引物e-3f和e-3r6r进行pcr扩增crispr3位点。
优选,所述的步骤c的dna测序,其测序引物为步骤b的pcr扩增引物。
本发明人在丙二酸盐克罗诺杆菌crispr分子中均发现靶向噬菌体和质粒等外源物质的间隔序列,说明这些序列中含有菌株的重要侵染信息,可以应用于该菌感染来源的追踪和传播模型的构建,并可联合数据库实现全国甚至全球标准化分子溯源,可以推广至食品、检验检疫等领域。本发明方法省时,经济,分辨率高,易于操作,对实验室设备及软件要求低,且含有重要的时间顺序信息,利于菌株分子溯源。
对于奶粉中丙二酸盐克罗诺杆菌的污染溯源及婴幼儿感染该菌的监测是疾病预防控制机构的重要任务之一,但目前的疾病控制机构存在高端分型设备不足及技术人员水平参差不齐等诸多问题。本发明的分型方法由于原理简单,操作容易,成本低廉,反应快速,高效,准确,无环境污染,且对实验室的设备要求不高,因此能在更广泛的应用于食品、检验检疫等领域,同时可以进行疾病的监测和预警。
本发明的crispr分型方法具有以下优势:
1.操作简单方便,对设备要求低;
2.分型成本低,不到mlst分型成本的三分之一;
3.与mlst分型方法相比具有更好的分辨率;
4.由于crispr间隔序列具有特殊的时间顺序,使得该分型方法具有细菌遗传分化及地域等多重信息的优势,更适合用于疾病爆发或食品安全事件的分子流行病学溯源。
本发明的分型方法可在未能配备复杂昂贵分型设备的实验室及基层疾控单位实施,具有较好的推广及应用价值。
附图说明
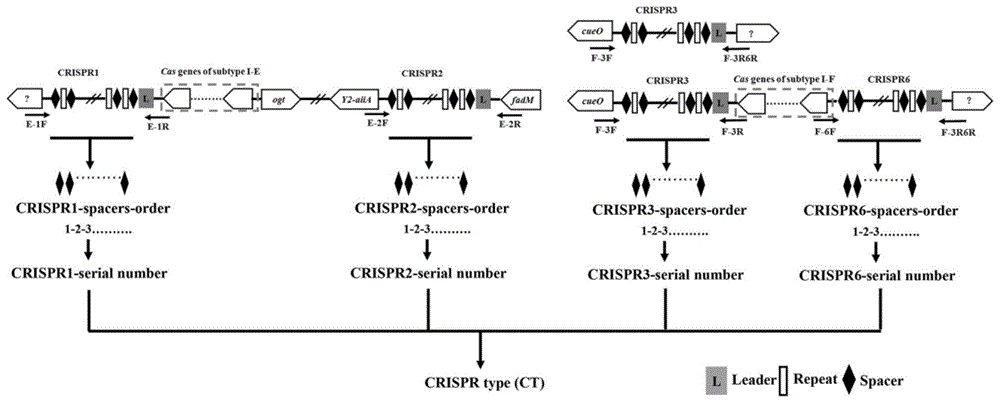
图1是丙二酸盐克罗诺杆菌的crispr分型示意图。
图2是丙二酸盐克罗诺杆菌crispr的pcr扩增电泳图谱。
具体实施方式
以下实施例是对本发明的进一步说明,而不是对本发明的限制。
实施例164株丙二酸盐克罗诺杆菌的crispr分型方法
(1)菌株
本次实验用到的64株菌株分离自我国多个地区的奶粉,食用菌和蔬菜样,已完成传统生化鉴定和分子检测,确定为丙二酸盐克罗诺杆菌。
(2)细菌dna提取
采用magen公司的细菌基因组dna提取试剂盒分别提取上述待测64株菌株的基因组dna,提取的dna于-20℃保存。
(3)引物合成
表1crispr分子扩增的引物表
pcr扩增中所用的引物浓度均为10μmol/l。
(4)pcr扩增
4.1crispr1扩增pcr反应体系:
crispr1扩增pcr反应条件为:98℃预变性1min,然后98℃10s,58℃5s,72℃4min,共30次循环后,72℃最后延伸5min。
4.2crispr2扩增pcr反应体系:
crispr2扩增pcr反应条件为:98℃预变性1min,然后98℃10s,58℃5s,72℃4min,共30次循环后,72℃最后延伸5min。
4.3crispr3扩增pcr反应体系:
crispr3扩增pcr反应条件为:98℃预变性1min,然后98℃10s,57℃5s,72℃4min,共30次循环后,72℃最后延伸5min。
4.4若e-3f和e-3r扩增出条带,则加扩crispr6;若无条带,则不需要扩增crispr6,换成上游引物e-3f和e-3r6r扩增crispr3,体系和条件同4.3。
crispr6扩增pcr反应体系:
crispr6扩增pcr反应条件为:98℃预变性1min,然后98℃10s,57℃5s,72℃4min,共30次循环后,72℃最后延伸5min。
反应结束后,取5μlpcr反应产物在1%琼脂糖凝胶上进行电泳,在凝胶成像系统上观察结果。图2为扩增出的丙二酸盐克罗诺杆菌crispr位点电泳图,marker为dl5000,由图2可见,不同菌株中的crispr位点序列长短不一。分型结果各不相同,详见表2。
(5)pcr扩增产物测序
利用扩增的引物进行crispr分子测序,测序任务由华大基因完成。
(6)crispr分型
丙二酸盐克罗诺杆菌的crispr分型示意图如图1所示。
利用crisprfinder提取crispr位点序列中的间隔序列(spacer),通过重复序列(repeat)判断crispr分子的正反方向,富含at碱基的序列为前导序列,从离该前导序列最远处的spacer开始计算顺序号。分别将不同crispr分子中提取的spacer去冗余后存为单独的序列库,每个spacer赋予一个序号。每株菌的crispr分子的spacer通过blast找到序列库中的对应序号,未能找到对应序号的新spacer分子,则加入到序列库,赋予新的序号。根据crispr分子的spacer排列组合给予一个特定编号,其中缺失的crispr分子记为编号零,最后综合crispr分子的组合编号确定丙二酸盐克罗诺杆菌的crispr型别。
表264株丙二酸盐克罗诺杆菌的crispr分型结果
实施例2丙二酸盐克罗诺杆菌的crispr分型与mlst分型方法的分辨率比较
(1)mlst分型
主要选取了丙二酸盐克罗诺杆菌七个管家基因进行扩增测序,将所得序列信息上传至专门的网站(https://pubmlst.org/cronobacter/)获取相应的mlst型别,具体实验操作方法见mlst网站。
(2)两种分型方法的分辨率的比较
表3丙二酸盐克罗诺杆菌的mlst分型和crispr分型结果比较
分辨率评价用simpson多样性指数(d)量化表示,其计算公式为:d=1-∑[nj(nj-1)]/[n(n-1)],其中nj表示第j带型的菌株数量,n表示实验菌株总数。d值越大说明分辨率越强。
两种分型方法的分辨率分别为:
多位点序列分型mlst:d=0.8953
crispr分型方法:d=0.9727
由此可见crispr分型方法的分辨率高于mlst分型方法。
同时如表3所示,13株丙二酸盐克罗诺杆菌mlst分型为st7,crispr分型可以将其细分为ct12~ct15,ct17~ct19七种型别,11株丙二酸盐克罗诺杆菌mlst分型为st60,crispr分型进一步分为ct23,ct24,ct25和ct26四种型别,9株丙二酸盐克罗诺杆菌mlst分型为st211,crispr分型进一步分为ct2,ct3和ct4三种型别。说明crispr分型方法较之mlst分型具有更强的分子溯源能力,对于疾病爆发或污染溯源时的分子流行病学追踪具有重要的意义。
综上所述,本发明的crispr分型方法具有以下优势:
1.操作简单方便,对设备要求低;
2.平均分型成本低;
3.与mlst分型方法相比具有更好的分辨率;
4.由于crispr间隔序列具有特殊的时间顺序,使得该分型方法具有细菌遗传分化及地域等多重信息的优势,更适合用于疾病爆发或食品安全事件的分子流行病学溯源。
基于上述的优势,该分型方法可在未能配备复杂昂贵分型设备的实验室及基层疾控单位实施,具有较好的推广及应用价值。
以上仅是本发明的优选实施方式,应当指出的是,上述优选实施方式不应视为对本发明的限制,本发明的保护范围应当以权利要求所限定的范围为准。对于本技术领域的普通技术人员来说,在不脱离本发明的精神和范围内,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>广东省微生物研究所(广东省微生物分析检测中心)
广东环凯微生物科技有限公司
<120>一种丙二酸盐克罗诺杆菌crispr分型方法
<160>8
<170>siposequencelisting1.0
<210>1
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>1
cctgacctggtaaacagagtagcg24
<210>2
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>2
cgatttccagacgtwcggcgttaa24
<210>3
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>3
cagttragatggtgtacycgcata24
<210>4
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>4
aragggcagccgrtctttaacaag24
<210>5
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>5
gttgagcttaaaccctccccttgc24
<210>6
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>6
gtcagcggyaccttcagcagtt22
<210>7
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>7
gctattagcacctgactgatgtacg25
<210>8
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>8
caggcattccggtaatattcgctc24
- 还没有人留言评论。精彩留言会获得点赞!