利用脂质生物标志物预测乳腺癌的方法与流程
本发明涉及生物技术领域,尤其涉及一种利用脂质生物标志物预测乳腺癌的方法,能够准确预测乳腺癌。
背景技术:
与其他大多数国家一样,乳腺癌也成为了中国女性最常见的癌症;每年中国乳腺癌新发数量和死亡数量分别占全世界的12.2%和9.6%。中国对全球的“贡献率”逐步增加,主要归因于中国社会经济地位的提高和特殊的生育模式。
90年代以来,中国的乳腺癌发病率增长速度是全球的两倍多,城市地区尤为显著。目前,乳腺癌是中国女性发病率最高的癌症,癌症死亡原因位居第六。截至2008年,中国总计169452例新发乳腺癌,44908例死于乳腺癌,分别占到全世界的12.2%和9.6%。如果这一趋势保持不变,到2021年,中国乳腺癌患者将高达250万,发病率将从不到60例/10万女性(年龄在55岁到69岁之间)增加到超过100例/10万女性。
目前对乳腺癌的筛查,主要通过x线检查。然而,中国57%的患者都在一个年龄段发作。这一结果也暗示乳腺x线检查,不是非常有效的筛查手段,并且具有无法实施的障碍,包括:缺乏令人信服的成本效果分析数据;人群分布广泛;器材设备缺乏;医疗保险未覆盖此项目。同时,中国还没有全国范围内的筛查项目;所以,开发快速而且高效的乳腺癌生物标志物,具有重要的意义和市场前景。
研究表明,脂质代谢与组织病变有非常直接的联系,也包括乳腺癌。迄今为止,利用脂质和代谢组的分析手段,发现了39个代谢物与乳腺癌发生有关,包括:黄嘌呤(xanthine),葡萄糖6-磷酸(glucose-6-phosphate),甘露糖-6-磷酸(mannose-6-phosphate),鸟嘌呤(guanine),腺嘌呤(adenine)等等。另外,研究也发现,大部分的磷脂含量在乳腺癌细胞中都显著升高。特别地,磷脂酰丝氨酸phosphatidylserine(ps)18:0/20:4,磷脂酰肌醇phosphatidylinositol(pi)18:0/20:4,磷脂酰胆碱phosphatidylcholine(pc)18:0/20:4,在乳腺癌细胞中显著增加;而磷脂酰乙醇胺phosphatidylethanolamine(pe)18:1/18:1和磷脂酰肌醇pi18:0/18:1显著降低.
但是,目前为止,还没有出现能够利用这些标志物准确预测乳腺癌的预测模型,尤其是针对中国人群的乳腺癌预测模型。
技术实现要素:
本发明的一个目的是解决至少上述问题,并提供至少后面将说明的优点。
本发明还有一个目的是提供一种利用脂质生物标志物预测乳腺癌的方法,能够快捷、准确、高效地预测乳腺癌。
本发明还有一个目的是提供一种针对中国人群的利用脂质生物标志物预测乳腺癌的方法。
本发明还有一个目的是提供一组针对中国人群的与乳腺癌密切相关的化合物。
为了实现根据本发明的这些目的和其它优点,提供了以下技术方案:
一种利用脂质生物标志物预测乳腺癌的方法,其中,主要包括以下步骤:
步骤1、筛选出正常人组群和乳腺癌组群之间vip值大于5的排名前9位的差异性化合物r1-r9,分别为表1所示:
表1
步骤2、利用逻辑回归模型2进行计算,得到tc值,所述逻辑回归模型2的计算公式为:
tc=3.564+6.135*r6+5.375*r7-1.814*r8-2.943*r9+1.151*r10;
步骤3、根据所得tc值进行判断,tc=0为否;tc=1为是。
优选的是,步骤1中利用opls-da模型对差异性化合物进行筛选,筛选条件vip>1。
优选的是,步骤1中筛选的具体方法为:
步骤1.1将样品进行超高效液相色谱和质谱分析,得到脂质组学数据,将正常人组群和乳腺癌组群分别计为ck及rc;
步骤1.2对脂质组学数据进行标准化操作,利用opls-da模型对ck及rc进行s-plot分布得到s形曲线,并进行强制分组,计算影响ck及rc分组的变量重要性,即得vip值;
步骤1.3按照vip值大于1的标准得到10个化合物,并将该10个化合物作为与乳腺癌相关度最高的差异性化合物;
步骤1.4将所得10个化合物按照vip值大小从高到低排列,取前9位,即得步骤1中所述差异性化合物r1-r9。
优选的是,步骤2中tc值或者用逻辑回归模型1进行计算,所述逻辑回归模型1的计算公式为:
tc=-8.673-32.803*r1+19.399*r2+8.738*r3-20.88*r8+2.217*r9+1.607*r10。
优选的是,步骤2中tc值或者用逻辑回归模型3进行计算,所述逻辑回归模型3的计算公式为:
tc=0.7564-2.6459*r8+2.524*r9+1.5554*r10。
本发明至少包括以下有益效果:
本发明通过筛选,首次发现与乳腺癌相关的一组化合物,即与乳腺癌相关的脂质生物标志物,并通过构建逻辑回归模型,得到根据这些脂质标志物预测乳腺癌的方法,快捷方便,准确度高。通过aic值初步判断,并进行roc曲线绘制,auc值达到0.939。
本发明的其它优点、目标和特征将部分通过下面的说明体现,部分还将通过对本发明的研究和实践而为本领域的技术人员所理解。
附图说明
图1为本发明中所述的opls-da模型的s-plot分布图;
图2为本发明中利用opls-da模型对ck和rc进行强制分组的结果;
图3为本发明中用来考察筛选出的化合物的火山图;
图4为本发明中roc曲线图。
具体实施方式
下面结合附图对本发明做进一步的详细说明,以令本领域技术人员参照说明书文字能够据以实施。
应当理解,本文所使用的诸如“具有”、“包含”以及“包括”术语并不配出一个或多个其它元件或其组合的存在或添加。
一种利用脂质生物标志物预测乳腺癌的方法,其中,主要包括以下步骤:
步骤1、筛选出正常人组群和乳腺癌组群之间vip值大于5的排名前9位的差异性化合物r1-r9,分别为表1所示。其中,所述vip值表示差异性的大小,所述r1-r9按照vip值从高到低顺次排列。
表1
步骤2、利用逻辑回归模型2进行计算,得到tc值,所述逻辑回归模型2的计算公式为:
tc=3.564+6.135*r6+5.375*r7-1.814*r8-2.943*r9+1.151*r10;
经过roc曲线绘制,模型2的auc值为0.939,准确度高达93.9%。
步骤3、根据所得tc值进行判断,tc=0为否;tc=1为是。
绘制roc曲线,模型1所得auc值为0.939,即准确度为93.9%,准确度高。其中,公式中r1-r10均为测量样品中各种脂质的含量。
一个优选方案中,步骤1中利用opls-da模型对差异性化合物进行筛选,筛选条件vip>1。
一个优选方案中,步骤1中筛选的具体方法为:
步骤1.1将样品进行超高效液相色谱和质谱分析,得到脂质组学数据,将正常人组群和乳腺癌组群分别计为ck及rc;
步骤1.2对脂质组学数据进行标准化操作,利用opls-da模型对ck及rc进行s-plot分布得到s形曲线,并进行强制分组,计算影响ck及rc分组的变量重要性,即得vip值;
步骤1.3按照vip值大于1的标准得到10个化合物,并将该10个化合物作为与乳腺癌相关度最高的差异性化合物;
步骤1.4将所得10个化合物按照vip值大小从高到低排列,取前9位,即得步骤1中所述差异性化合物r1-r9。
一个优选方案中,步骤2中tc值或者用逻辑回归模型1进行计算,绘制roc曲线,模型1所得auc值为0.939。所述逻辑回归模型1的计算公式为:
tc=-8.673-32.803*r1+19.399*r2+8.738*r3-20.88*r8+2.217*r9+1.607*r10。
经过roc曲线绘制,模型1的auc值为0.907,准确度90.7%。
一个优选方案中,步骤2中tc值或者用逻辑回归模型3进行计算,绘制roc曲线,模型3所得auc值为0.899。所述逻辑回归模型3的计算公式为:
tc=0.7564-2.6459*r8+2.524*r9+1.5554*r10。
经过roc曲线绘制,模型3的auc值为0.899,准确度89.9%。
本发明通过筛选,首次发现与乳腺癌相关的一组化合物,即与乳腺癌相关的脂质生物标志物,并通过构建逻辑回归模型,得到根据这些脂质标志物预测乳腺癌的方法,快捷方便,准确度高。通过aic值初步判断,并进行roc曲线绘制,auc值达到0.939。
实施例1
材料和方法
1.实验对象(均选自中国人):14位正常人,28位乳腺癌患者,抽取静脉血5ml。准确量取100μl的血液,加入0.9ml的提取液(100%异丙醇),转入2ml离心管(进口离心管,塑料不易溶于有机溶剂中;axygen品牌)中,漩涡振荡10s以上,超声10min,然后在-20度冰箱中冷冻1小时,取出后在室温下漩涡振荡,用冷冻离心机10000rpm离心10min,然后去上清液1ml并过0.22μm的有机相滤膜到玻璃进样品种,保存在冰箱中待测。
2.主要仪器
2.1.1冷冻离心机:型号d3024r,scilogex公司,美国
2.1.2漩涡振荡器:型号mx-s,scilogex公司,美国
2.1.3高分辨质谱仪:esi-qtof/ms;型号:xevog2-sq-tof;厂家:waters
2.1.4超高效液相色谱:uplc;型号:acquityuplci-class系统;厂家:waters
2.1.4数据采集软件:masslynx4.1;厂家:waters
2.1.5分析鉴定软件:progenesisqi;厂家:water
2.1.6作图软件:ezinfo;hemi;simca-p
3.主要试剂
甲醇、乙腈、甲酸、甲酸铵、亮氨酸脑啡肽、甲酸钠。厂家均为fisher。
4.实验设置
以提取液(100%异丙醇)作为空白对照(blank)样品;从每个样品的进样品种取出100μl混合到新的进样品种作为质控(qc)样品;正式样品按照每组样品间隔进样,例如先是blank1,然后是qc1,然后是w1,然后是w2这个顺序,然后再进行下一轮进样。
液相方法
色谱柱:acquityuplccshc18column,
柱温:55度
流速:0.4ml/min
流动相:
a:acn/h2o(60%/40%),含有10mm甲酸铵和0.1%甲酸
b:ipa/acn(90%/10%),含有10mm甲酸铵和0.1%甲酸
(注:acn为乙腈,ipa为异丙醇)
进样体积:0.2μl
洗脱程序:
质谱方法
数据采集方式:mse;分子量扫描范围:50-1500m/z;分辨率模式(轮廓图)。
正负离子模式各采集一次。
离子源:电喷雾电离源(esi)
毛细管电压:3kv
锥孔电压为:25v
碰撞能:15-60v
源温度:120度
脱溶剂温度:500度
锥孔气体速度:50l/h
脱溶剂气体速度:500l/h
扫描时间:0.2s
使用亮氨酸脑啡肽(m/z556.2771,正离子;554.2615,负离子)进行实时校正。使用甲酸钠进行校正。
脂质组学数据分析
progenesisqi软件(waters,massachusetts,usa)用于结果分析,抽提非靶向脂质分子的特征峰,进行比对和筛选。同时,用qc(quantifyingcontrol)和blank(空白)来筛选背景数据。最终的数据,导入ezinfo3.0,并进行principalcomponentanalysis(pca)分析,orthogonalsignalcorrectionpartialleastsquarediscriminationanalysis(opls-da)建模,variableimportanceinprojection(vip)的计算,同时得到火山图(coefficientsvs.vipspots)。其中,逻辑回归模型以及roc曲线通过r语言进行建设和绘制。
结果描述
正常人群与乳腺癌人群脂质差异物质的鉴定。
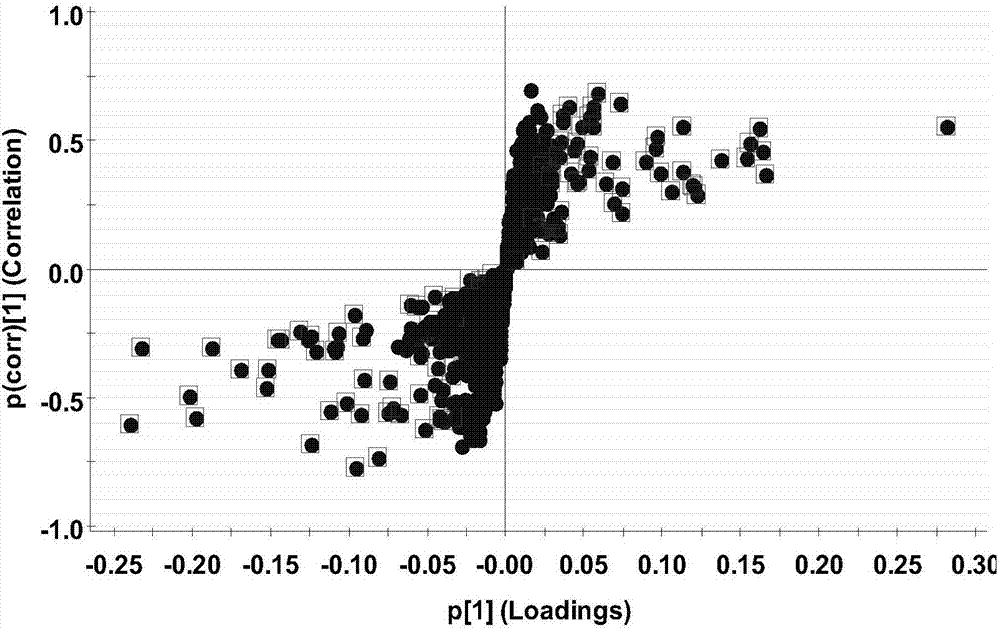
我们首先建立opls-da模型,对正常人(ck)和乳腺癌(rc)组群进行分类,并研究造成他们出现差异的原因。我们看到在opls-da模型中,利用相关性(correlation)和协方差(covarience)的p值作出的s-plot形成了非常好的s形曲线,如图1所示。
利用opls-da将两组数据进行强制分组,如图2所示。
计算影响ck和rc分组的变量重要性,即vip(variableimportanceinprojection)值。一共筛选到132个化合物,他们的vip值大于1;我们挑选vip>5,贡献率大的10个化合物,如表2所示。
表2.变量重要性投影
我们通过vip>1筛选出来的化合物在s-plot上用红色方框标出,发现他们均匀的分布在两侧。同时,我们利用火山图,如图3所示,来考察筛选出的化合物的分布,如图所示,发现筛选出来的化合物都分布在火山图的外围。这些结果都说明,借由opls-da模型,我们成功筛选出造成ck和rc差异的化合物。
建立逻辑回归模型以及roc曲线。
我们对脂质组学的数据进行标准化操作(rproject:scale)。然后将vip>1的排名前9位变量的数据提取出来,建立逻辑回归模型和roc曲线。
公式中,tc:是否患有肿瘤,0为否,1为是
r1:pc(16:0/18:2)
r2:pe(p-20:0/22:6)
r3:pa(21:0/18:1)
r4:pa(p-16:0/21:0)
r5:pa(19:0/18:0)
r6:pc(16:0/18:1)
r7:sm(d18:1/24:1)
r8:sm(d18:1/16:0)
r9:ps(o-20:0/18:1)
r10:pe(22:0/18:4)
模型2:
tc=3.564+6.135*r6+5.375*r7-1.814*r8-2.943*r9+1.151*r10
aic:35.898
signif.codes:‘***’0.001;‘**’0.01;‘*’0.05;‘.’0.1;
模型1:
tc=-8.673-32.803*r1+19.399*r2+8.738*r3-20.88*r8+2.217*r9+1.607*r10
aic:39.564
signif.codes:‘***’0.001;‘**’0.01;‘*’0.05;‘.’0.1;
模型3:
tc=0.7564-2.6459*r8+2.524*r9+1.5554*r10
aic:37.067
signif.codes:‘***’0.001;‘**’0.01;‘*’0.05;‘.’0.1;
针对以上三个逻辑回归模型,进行roc曲线绘制,如图4所示,模型1为m1,auc值为0.907;模型2为m2,auc值为0.939;模型3为m3,auc值为0.899。我们发现模型2最靠近左上角定点,同时auc值最高,最终,确定模型2为基于脂质指标来预测糖尿病血脂异常较好的预测模型。也可以根据所测样品数据情况,选择模型1或者模型3进行计算预测,或者优先选择模型2进行计算,同时使用1和3模型进行辅助验证,协同分析预测。
本发明利用opls-da模型,首次发现与乳腺癌相关的10个biomarker。利用逻辑回归模型以及roc曲线,首次提出了基于脂质生物标志物的乳腺癌预测模型。
尽管本发明的实施方案已公开如上,但其并不仅仅限于说明书和实施方式中所列运用,它完全可以被适用于各种适合本发明的领域,对于熟悉本领域的人员而言,可容易地实现另外的修改,因此在不背离权利要求及等同范围所限定的一般概念下,本发明并不限于特定的细节和这里示出与描述的图例。
- 还没有人留言评论。精彩留言会获得点赞!