一种基因组变异数据的计算方法与流程
本发明属于高通量测序的生物信息领域,尤其涉及到一种基因组变异数据的计算方法。
背景技术:
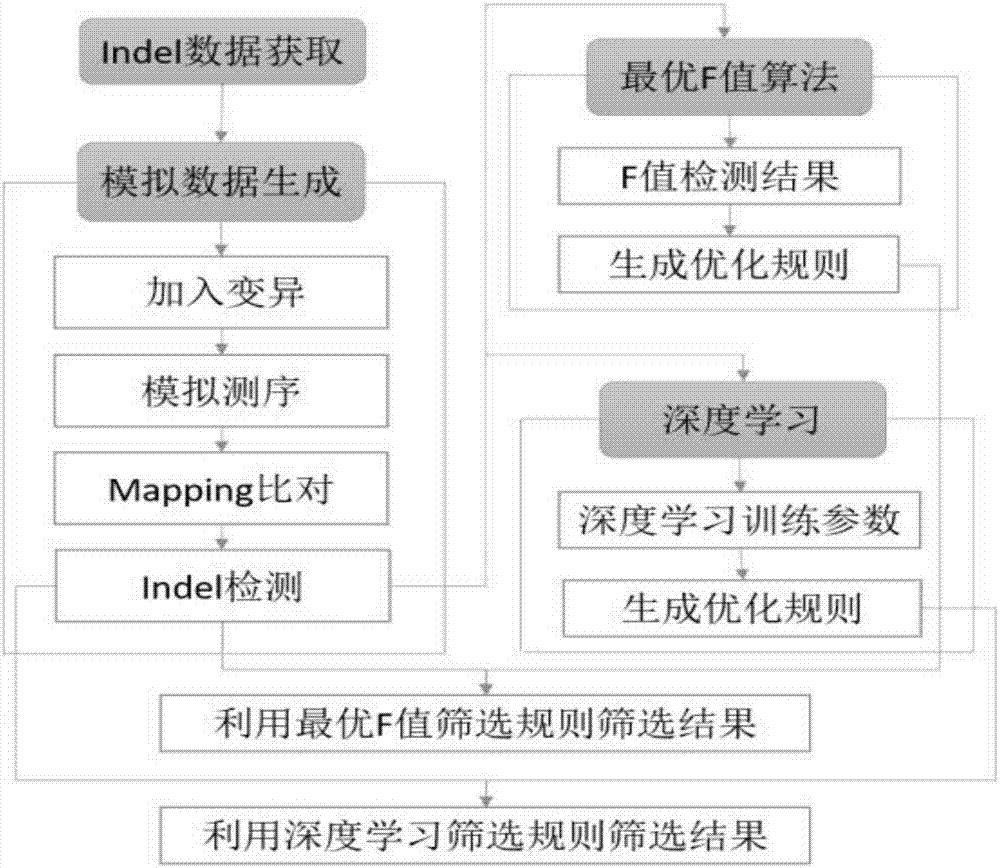
:在重测序技术中变异检测是基因组功能分析的基础,因此检测结果是否准确直接影响分析结果的准确性。近些年在变异检测领域为了弥补单个软件检测结果的不足,发表了一些基于多个软件的整合算法。利用合并软件结果提高回收率,利用提取软件的一致检测结果提高正确率。技术实现要素:本发明所要解决的技术问题是提供一种基因组变异数据的计算方法。本发明分析了indel的大小和基因组序列特征对变异检测结果正确率和回收率的影响,提出了基于最优f值策略的优化算法。本发明提出的indel检测算法是整合多个软件检测结果的优化筛选算法,选择samtools、gatk(unifidgenotyper)、varscan、pindel和soapindel生成原始indel数据。这五个软件分别采用了四种不同的算法检测indel变异。(1)samtools和gatk(unifiedgenotyper)是基于测序数据与参考基因组的比对结果,利用贝叶斯统计模型计算每个位点基因型的后验概率检测indel。(2)pindel是基于比对结果中未匹配的读段(unmappedreads)数据,利用模式生长算法检测插入/删除变异。(3)varscan是基于samtools的pileup数据,利用稳定的启发式算法检测indel变异,并且能够处理极端读段深度、混池测序数据和测序数据受污染等问题。(4)soapindel是利用debruijngraph算法将所有未匹配的读段重组,通过和参考基因组比对检测插入/删除变异。基于最优f值策略的优化算法如下:1)建立最优化规则选择samtools、gatk(unifidgenotyper)、varscan、pindel和soapindel软件对模拟数据进行indel检测,生成原始indel数据,计算每两个软件的联合的f值,通过最优的f值建立一个最优选择的规则。2)根据优化规则选择indel利用软件samtools、gatk(unifidgenotyper)、varscan、pindel和soapindel对待测数据进行indel检测,根据ds,rt,ss,st进行分组。根据最优化规则选择indel。本发明提出了indel检测算法,可以提高结果的正确率、回收率和f值。附图说明图1,f值趋势图。图2,基因组变异数据获取流程示意图。具体实施方式本发明分析了indel的大小和基因组序列特征对变异检测结果正确率和回收率的影响,提出了基于最优f值策略的优化算法。一.软件的选择本发明提出的indel检测算法是整合多个软件检测结果的优化筛选算法,选择samtools、gatk(unifidgenotyper)、varscan、pindel和soapindel生成原始indel数据。这五个软件分别采用了四种不同的算法检测indel变异。(1)samtools和gatk(unifiedgenotyper)是基于测序数据与参考基因组的比对结果,利用贝叶斯统计模型计算每个位点基因型的后验概率检测indel。(2)pindel是基于比对结果中未匹配的读段(unmappedreads)数据,利用模式生长算法检测插入/删除变异。(3)varscan是基于samtools的pileup数据,利用稳定的启发式算法检测indel变异,并且能够处理极端读段深度、混池测序数据和测序数据受污染等问题。(4)soapindel是利用debruijngraph算法将所有未匹配的读段重组,通过和参考基因组比对检测插入/删除变异。二.模拟数据为了详细的研究每个软件indel检测结果的正确率、回收率以及基因组的序列特征对检测到结果的影响,需要已知所有变异的具体信息,包括变异的位置、大小以及所处基因组区域的特征。为此本发明利用计算机模拟技术将已知变异加入到参考基因组中生成新基因组序列,再利用模拟测序技术生成测序数据。模拟数据如表1所示。表1变异分布变异类型大小(bp)数量snp11/1000比例indel1-502792000deletion/insertion51-50020000duplication100-5001000inversion100-5001000translocation100-5001000三.比对与检测利用bwa[lianddurbin,2009]将测序数据和大豆参考基因组(william82)比对生成sam文件,用samtoolsview将sam文件转换为bam文件,用samtoolssort把bam文件按坐标排序并用samtoolsrmdup去重复并用samtoolsindex建立索引。然后用五个软件检测变异,将varscan的参数“最小测序深度”设置为2,其余软件使用软件默认参数。最后抽取结果中1-50bp的indel。四.一致结果的判定标准为了分析软件之间相互补足和相互验证的关系,需要明确两个软件一致性结果的判定标准。对于该问题有文献提出了两个标准,一个是相互重叠率超过50%,另一个是有一个以上的碱基重叠[lametal.,2012]。这两个标准只考虑检测结果坐标重叠的情况。但是由于软件算法的不同会导致结果坐标的差异甚至大小的不同。本发明为了保证检测结果的准确性,规定只有大小相同才能判定为同一个indel。另外本发明经过模拟实验发现,对于同一个indel变异不同软件检测结果的坐标存在差异,导致该偏差的原因主要是序列相似性,例如序列atatat中删除at,软件报告的结果可能是三个中的任何一个。我们利用下面公式计算软件结果之间的坐标偏差d:d=|p1-p2|其中p1是indel1的起始坐标,p2是indel2起始坐标。多次模拟实验的统计结果表明,在大豆基因组中非重复序列区域坐标偏差范围为[1,31],在重复序列区域坐标偏差值最大等于重复序列长度。五.四个重要indel属性的分析indel有四个重要属性——变异类型(st)、变异大小(ss)、所处重复区域类型(rt)和检测软件(ds)。将检测结果按这四个属性分组,为了方便描述,本发明定义g(f,s)表示集合s按属性f分组的结果,例如g(st,s)表示集合s按变异类型分组,g(ss,g(st,s))表示集合s先按变异类型分组,然后再按变异大小分组以此类推。模拟实验数据表明在分组g(ds,g(rt,g(ss,g(st,检测结果))))中,对处于相同类型重复序列和相同大小的indel,不同的软件的正确率和回收率存在较大差异。五个软件对处于非重复序列的1bp删除变异的检测正确率和回收率的分布,其中gatk拥有最大的正确率(99.83%)而同时拥有最小的回收率(41.92%),varscan拥有最大的回收率(88.42%)。这说明软件是影响检测精度的重要因素。同一个软件对处于不同类型重复序列和不同大小的indel检测的正确率和回收率也存在较大差异。以上分析说明变异类型、变异大小、所处重复区域类型和检测软件四个属性是影响indel检测正确率和回收率的重要因素。六.基于最优f值策略的优化筛选方法根据上文的分析可知,从宏观看提取多个软件的一致结果会提高正确率。然而模拟数据表明检测结果中,g(ss(st,两个软件一致检测结果))中有些分组的正确率和回收率都较高,有些分组正确率高而回收率低,有些分组的正确率和回收率很低甚至为零。因此直接合并每两个软件的一致结果并不能得到最优的正确率。f值是用来评估正确率和回收率平衡性的指标,f值计算公式如下式所示。f=2×p×r/(p+r)其中p为正确率,r为回收率。本发明通过模拟实验发现g(rt,g(ss,g(st,两个软件的一致结果)))中两个软件一致结果(ir)的f值的具有稳定的变化规律,同时对于不同的分组的最优f值出现在不同的ir上(图1)。该图中g1是各ir对tir类型区域中1bp删除变异检测结果的f值,g2是各ir对ssr类型区域中9bp删除变异检测结果的f值。g1的最优f值出现在samtools和varscan的一致结果中,g2的最优f值出现在samtools和soapindel的一致结果中。基于以上的分析,我们给出一个直观简单的基于分组最优f值的优化策略:1.建立最优化规则选择indel检测软件,模拟染色体变异和序列。利用工具进行indel检测,计算每两个软件的联合的f值。通过最优的f值建立一个最优选择的规则。2.根据优化规则选择indel利用软件进行indel检测,根据ds,rt,ss,st进行分组。根据最优化规则选择indel。从全体变异看,该方法的正确率(99.32%)高于samtools(97.46%)、pindel(94.69%)、soapindel(97.24%)和varscan(98.59%),回收率(65.20%)高于gatkunifiedgenotyper(25.50%)和pindel(41.36%)。七.基于深度学习的筛选方法最优f值的方法是基于软件的一致结果,因而会舍弃仅由单个软件检测到的indel,而从模拟数据可知仅由单个软件检测的indel接近占整体数量的20%,全部舍弃严重影响回收率。为了能够更加全面的利用所有软件的结果从而获得更高的回收率兼顾平衡性,本发明设计了基于深度学习(deeplearning)的方法筛选所有软件的检测结果,我们以所有原始数据为训练集,以检测indel的所用软件,indel的类型,indel所处重复序列类型,支持indel检测结果的读段数量(覆盖度)为训练特征,准确率和召回率为训练目标。利用训练集我们可以训练得到一个使回收率和召回率尽可能高的模型。我们采用tensorlayer来进行深度学习程序的开发,tensorlayer是建立在googletensorflow上的深度学习(deeplearning)与增强学习(reinforcementlearning)软件库。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 油菜BnaSPT1基因在促进双子叶植物角果生长中的应用的制造方法与工艺
- 油菜BnaSPT3基因在促进双子叶植物角果生长中的应用的制造方法与工艺
- 一种OsGA3ox1基因在水稻雄性不育株系创制中的应用的制造方法与工艺
- 水稻紫叶基因PL1的分子标记及其鉴定方法和应用与制造工艺
- 一种番茄PSY 1基因的CRISPR‑Cas9体系构建及其应用的制造方法与工艺
- OsARD1基因在提高水稻耐水淹方面的应用的制造方法与工艺
- 一种烟草干旱响应基因NtXERICO及其克隆方法与应用与制造工艺
- 一种小麦耐盐基因及其应用的制造方法与工艺
- 水稻FAH基因在水稻育性控制中的应用的制造方法与工艺
- 水稻大粒基因GS12紧密连锁的分子标记及应用的制造方法与工艺