具有乳酸耐受性提高的酵母及其用途的制作方法
1.涉及具有乳酸耐受性提高的酵母及其用途的生物技术,特别是基因工程。
背景技术:
2.由可再生资源生产的有机酸正成为几个行业的有吸引力的分子。在能源工业中,生物乙醇和生物丁醇已经得到长期开发和商业化应用。乳酸和琥珀酸也是以生物方式生产的,并作为原料用在多种应用中,例如食品工业中的食品和饲料添加剂、个人护理生产中的温和溶剂应用以及生物基树脂生产中的生物聚合物合成。所有上述分子都是通过被称为发酵的众所周知的过程产生的。发酵过程最大的挑战是生产成本。
3.已经进行了几种降低生产成本的尝试。为了降低发酵原料(fermentation feedback)的成本,已经提议农业废物,例如来自制糖工业的甘蔗渣(bagasse)。来自甘蔗渣的纤维素可以物理分解和化学分解成可发酵的糖,以供细菌利用。但是,预处理过程中的多种杂质会对细菌产生抑制作用。例如,半纤维素分解释放的乙酸盐/酯(acetate)是众所周知的大多数微生物的抑制剂。此外,在甘蔗渣的储存过程中,一些细菌的污染所产生的非期望的乳酸也会影响发酵过程。
4.另一方面,在发酵过程中防止细胞损伤的问题的成本也很高昂。例如,它发生在弱酸(例如乳酸和琥珀酸)生产的情况下。在这些酸的发酵过程中,重要的是要中和已产生的乳酸并对产生的乳酸盐(lactate)脱盐,以防止降低ph值对细胞活动造成损害。在生物技术工艺中使用的强大细菌中,酵母因其健壮性(robustness)和对低ph值的耐受性而被广泛使用。尽管如此,ph值低于2.8,工程化的酵母菌株的乳酸产量下降。因此,赋予酵母更高的乳酸抗性将提高非中和条件下的乳酸生产力。
5.已经在酿酒酵母(saccharomyces cerevisiae)的酵母中进行了几项研究,其中清楚地表明酵母细胞通过由haa1基因编码的转录因子的调节而对酸作出响应。
6.杉山(sugiyama)等人(应用与环境微生物学(applied and environmental microbiology),2014,80,3488
–
3495)尝试过度表达编码转录因子的haa1基因,以模拟其蛋白质haa1p重新定位到核酸酶中,从而诱导弱酸适应机制,以期提高酿酒酵母中的乳酸抗性。本研究还发现,与亲本菌株相比,haa1缺失菌株(disruptant)引起了严重的乳酸敏感性。
7.田中(tanaka)等人(应用与环境微生物学(applied and environmental microbiology),2012,78,8161
–
8163)公开了关于haa1在酿酒酵母中的过度表达可以提高乙酸耐受性的类似结果。
8.us9085781b2以及思文(swinnen)等人(微生物细胞工厂(microbial cell factories),2017,16:7,1-15)试图诱导点突变来微调酿酒酵母中的haa1表达;因此,与野生型等位基因相比,它导致了更高的乙酸抗性。
9.wo2016083397a1公开了haa1及其旁系同源物(paralog)cup2的组合以实现酵母(特别是酿酒酵母)中耐酸性的提高。这些复杂性主要导致了将这种方法用于商业化生产规
模中的障碍。
10.此外,在此研究领域,已经对酵母菌株(特别是酿酒酵母)进行了一些研究,以提高乳酸耐受性但不涉及haa1基因。
11.us9994877b2公开了一种基因工程化的酵母细胞增加了sul1、str3、aad10、mxr1、grx8、mrk1、gre1、htx7、err1或它们的组合的活性。与其亲本细胞相比,获得的酵母菌株具有增加的耐酸性,并且有助于生产乳酸。
12.us9758564b2公开了一种抗酸酵母细胞,其降低了编码fps1的多核苷酸的表达以增加乳酸抗性。
13.us10053714b2公开了一种耐酸基因工程化的酵母细胞,该酵母细胞包括以下基因修饰:增加催化磷脂酰肌醇(pi)和神经酰胺转化为肌醇磷酸神经酰胺(ipc)和二酰基甘油(dg)的酶的活性,或增加催化将双键引入脂肪酰基-coa的脂肪酰基位点的酶的活性,和/或降低催化从dg形成三酰基甘油(tg)的酶的活性或它们的组合,以及可选地降低外部线粒体nadh脱氢酶nde1和/或nde2的活性,nde1和/或nde2可以增加对具有1-20个碳原子的有机酸(尤其是乳酸)的耐受性。
14.铃木(suzuki等人)(生物科学与生物工程期刊(journal of bioscience and bioengineering),2013,115,467-474)报道了通过结合导致乳酸抗性的基因破坏来在酿酒酵母中构建抗酸酵母。本研究发现,94个基因缺失菌株体显示出对乳酸的抗性,并发现通过结合几个基因的破坏而进一步增强了乳酸抗性,导致高乳酸生产力而无需中和。
15.基于上述原因,本发明公开了一种简单的方法来提高其他非常规酵母的乳酸耐受性,特别是提高克鲁维酵母菌属的种(kluyveromyces sp.)的乳酸耐受性,其中,缺失haa1足以导致其乳酸耐受性、乳酸产量或它们的组合的提高。
16.本发明的改良酵母可用于广泛的方面,例如但不限于,通过使用含有任何酸性杂质的低成本原料的发酵过程,并进行发酵以产生具有高水平游离乳酸的发酵液。这些优点是成本效益(cost-effectiveness)生产。
技术实现要素:
17.本发明涉及一种基因工程化的克鲁维酵母菌属的种(kluyveromyces sp.)的酵母细胞,该酵母细胞至少包含使编码氨基酸seq id no.2(特别是haa1)的核苷酸序列失活或缺失的基因修饰。
18.与亲本相比,根据本发明的基因工程化的酵母细胞具有乳酸耐受性、乳酸产量或它们的组合的提高。
附图说明
19.图1 phaa1del2的结构,一种包含用于缺失来自菌株myr2297和myr2787的haa1基因的盒(cassette)的质粒。
20.图2用于缺失来自菌株myr2297和myr2787的haa1基因的过程示意图。
21.图3 myr2297亲本细胞(myr2297 haa1)和携带有δhaa1破坏(disruption)的myr2297(myr2297 δhaa1)在cm减尿嘧啶的固体培养基(cm minus uracil solid medium)(补充0.5%(ph 2.7)、1.0%(ph 2.5)、1.5%(ph 2.4)、2.0%(ph 2.3)的乳酸或未补充乳
酸(ph 3.5))上、在30℃下孵育2天的生长性能。
22.图4 myr2787亲本细胞(myr2787 haa1)、携带有δhaa1破坏的myr2787(myr2787 δhaa1)和携带有haa1过度表达的myr2787(myr2787 2x haa1)在cm减尿嘧啶的固体培养基(补充0.5%(ph 2.7)、1.0%(ph 2.5)、1.5%(ph 2.4)、2.0%(ph 2.3)乳酸或未补充乳酸(ph 3.5))上、在30℃下孵育2天的生长性能。
23.图5 myr2787亲本细胞(myr2787 haa1)、携带有δhaa1破坏的myr2787(myr2787 δhaa1)和携带有haa1过度表达的myr2787(myr2787 2x haa1)在微型生物反应器(biolector)中发酵2天的生长性能。
24.图6 myr2787亲本细胞(myr2787 haa1)、携带有δhaa1破坏的myr2787(myr2787 δhaa1)和携带有haa1过度表达的myr2787(myr2787 2x haa1)在微型生物反应器(biolector)中发酵2天产生的d-乳酸滴度。
具体实施方式
25.定义
26.除非另有说明,否则本文使用的技术术语或科学术语具有本领域普通技术人员所理解的定义。为了便于理解本发明,下面提供对命名法的描述。
27.本文提及的设备、装备、方法或化学品是指本领域技术人员通常操作或使用的设备、装备、方法或化学品,除非另有明确说明它们是本发明中专门使用的设备、装备、方法或化学品。
28.权利要求书或说明书中使用的“包括(comprising)”一词的单数或复数名词是指“一个”,也包括“一个或多个”、“至少一个”和“一个或不止一个”。
29.本技术中公开的所有组合物和/或方法和权利要求旨在涵盖来自任何动作、性能、修改或调整的实施方式,而无需进行任何与本发明明显不同的实验,并且这些实施方式根据本领域普通技术人员的意见,虽然没有在权利要求中具体说明,都能获得目的和用途并且与本实施方式的结果相同。因此,本实施方式的可替代或类似的客体,包括本领域技术人员清楚地看到的任何微小的修改或调整,应被解释为仍位于所附权利要求中出现的发明的精神、范围和概念内。
30.贯穿本技术,术语“约”用于表示本文呈现的任何值可能潜在地变化或偏离。这种变化或偏差可能是由计算中使用的装备、方法的错误或由执行设备或方法的个体操作员造成的。这些包括由物理性质变化引起的变化或偏差。
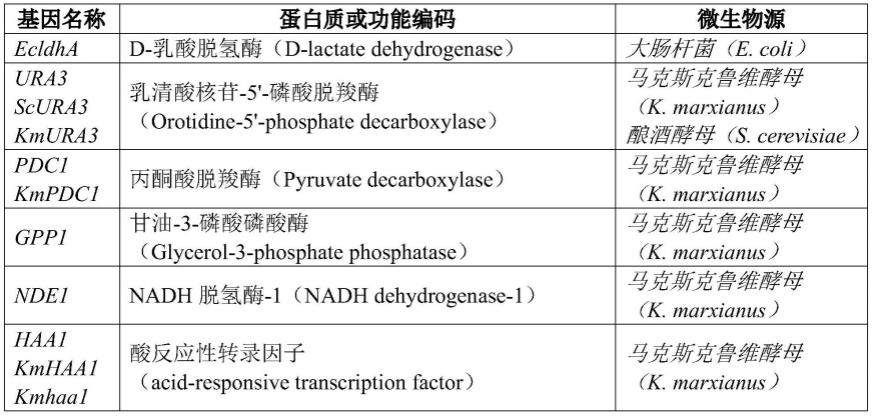
31.关于命名法,细菌基因或编码区通常以斜体小写字母命名,例如来自大肠杆菌(e.coli)的“ldha”,而由基因编码的酶或蛋白质可以用相同的字母命名,但首字母大写,并且不加斜体,例如“ldha”。酵母基因或编码区通常以斜体大写字母命名,例如“haa1”,而由基因编码的酶或蛋白质可以用相同的字母命名,但首字母大写,并且不加斜体,例如“haa1”。对于包含特定基因的突变或具有突变表型的酵母菌株,基因或菌株由小写斜体字母定名,例如,haha1或者δhaa1——用于缺乏功能性haa1基因的菌株。为了指定特定基因的来源生物体,基因名称最前面可以用两个字母表示属和种。例如,kmura3基因来源于马克斯克鲁维酵母(kluyveromyces marxianus),scura3基因来源于酿酒酵母(saccharomyces cerevisiae)。
[0032]“酵母”是指在某些条件下能够以单细胞状态生长的任何真菌生物体。一些酵母菌株在某些条件下(例如在饥饿状态下)也可以以菌丝状态(hyphal state)或假菌丝(pseudohyphal)(即短菌丝(short hyphae))状态生长。
[0033]“乳酸”或“d-lac”或“d-乳酸”是指乳酸,包括其盐,例如乳酸盐。
[0034]“基因工程”或“基因工程化的”是指将一种或多种基因修饰引入细胞或由此产生的细胞的活动。
[0035]“基因修饰”是指对细胞遗传物质的特点或结构的人工改变。基因修饰包括将编码多肽的多核苷酸引入细胞中,用于相对于亲本细胞的遗传物质进行替换、添加、插入或缺失一个或多个核苷酸或对亲本细胞的遗传物质进行化学修饰。
[0036]“亲本细胞”或“亲本”或“亲本的”是指原始细胞或没有特定基因修饰的细胞,并且可以用作起始材料以产生具有增加或减少的活性或产量的基因工程化的细胞。
[0037]“野生型”是指没有特定基因修饰的多肽或多核苷酸。
[0038]“转化株(transformant)”是指通过将所需的dna序列(线性或环状、以及自主复制或非自主复制)安装到宿主或亲本菌株中而产生的细胞或菌株。
[0039]“突变”是指天然或亲本dna序列的任何变化,例如倒位、重复、一个或多个碱基对的插入、一个或多个碱基对的缺失、点突变导致碱基变化产生过早的终止密码子,或一个错义突变改变了在该位置编码的氨基酸。
[0040]“突变体”是指与天然的、野生型、亲本或前体菌株相比包含一个或多个突变的菌株。
[0041]“盒(cassette)”是指一种脱氧核糖核酸(dna)序列,当安装在宿主生物体中时,该dna序列能够编码、产生或过量产生或替代地消除一种或多种所需蛋白质或酶或降低一种或多种所需蛋白质或酶的活性。用于产生蛋白质或酶的盒通常包含至少一个启动子、至少一个蛋白质编码序列和任选地至少一个转录终止子。盒可以构建到质粒中,它可以是环状的,或者它可以是通过聚合酶链式反应(pcr)、引物延伸pcr或者体内或体外dna片段末端之间的同源重组产生的线性dna,每个片段都是所需最终盒的子集,其中每个子集片段在一端或两端具有重叠同源性,旨在于体内或体外通过同源重组连接自然力(od)相邻片段。盒可以设计成包括可选标记基因或dna序列,其在整合后被约30个或更多的碱基对的正向重复序列(direct repeat sequence)(在整合的可选择的基因的两端都存在的取向中的相同序列)包围,使得在包含可选标记的初始盒已整合到染色体或质粒中之后,能通过在正向重复之间的同源重组缺失该可选标记(也称为“环出(looping out)”)。有用的可选标记基因包括但不限于抗生素g418抗性(kan或kanr)、潮霉素抗性(hyg或hygr)、博莱霉素(zeocin)抗性(zeo或者zeor)、天然植物素(naturicin)抗性(nat或natr)以及生物合成基因,例如ura3、trp1、trp5、leu2和his3。对于用作可选标记的生物合成基因,宿主菌株必须在相应基因中包含突变,优选地为非回复性无效突变(non-reverting null mutation)。例如,如果ura3被用作可选标记基因,那么待转化的菌株必须表型地为ura3。
[0042]“质粒”是指线状或环状dna分子,其基本上小于染色体,从微生物多个染色体的染色体分离,并且与染色体或多个染色体分开复制。质粒可以以每个细胞约一个拷贝(copy)或每个细胞多于一个拷贝的形式存在。在微生物细胞内维持质粒通常需要在选择质粒存在的培养基中生长,例如使用染色体营养缺陷体(auxotrophy)或抗生素抗性基因的互补。
[0043]“染色体”或“染色体的”是指比质粒大很多且通常不需要任何抗生素或营养选择的线性或环状dna分子。
[0044]“破坏(disruption)”是指导致由基因或编码区编码的酶或蛋白质在宿主微生物中不产生或产生的水平低于在相同或相似生长条件下在宿主微生物的野生型版本中发现的水平。
[0045]“过度表达”是指使由基因或编码区编码的酶或蛋白质在宿主微生物中产生的水平高于在相同或相似生长条件下在宿主微生物的野生型版本中发现的水平。
[0046]“惯性压力法(吉布森法)(gibson method)”是指一种在体外将两个或多个在它们的末端具有短的(约15-40个碱基对)重叠同源性的线性dna片段连接在一起的方法。该方法可用于从合成的线性dna片段、pcr片段或由限制酶产生的片段构建质粒。可以购买用于执行惯性压力法(gibson method)的试剂盒,例如nebuilder hifi dna组装克隆试剂盒(新英格兰生物实验室(new england biolabs,neb),美国马萨诸塞州伊普斯威奇市),并按照制造商的指示使用。
[0047]“滴度”是指一种化合物在发酵液中的浓度,通常以克每升(g/l)或每体积重量%(%)表示。
[0048]
为了便于理解本发明,表1中列出了多种基因。
[0049]
表1基因名称和描述
[0050][0051]
除非另有说明,本发明中的重组dna和基因工程均采用本领域熟知的方法和材料进行。根据制造商的方案,使用“惯性压力法(gibson method)”组装质粒和线性dna盒,或通过如上所述的体内同源重组。
[0052]
为了缺失dna序列或整合表达盒,我们通常使用的方法是在质粒上组装盒,该质粒可以在大肠杆菌(e.coli)中复制,或通过共同转化盒的两个或多个子部分(subsections)在体内将盒组装在目标酵母菌株中,相邻的子部分设计为在待连接的末端的重叠碱基对,以及在组装盒末端的与染色体靶序列同源的碱基对。本文所述的所有用于整合在克鲁维酵母菌属的种(kluyveromyces sp.)的染色体中的盒被设计为用来表达酵母ura3基因(通常是scura3基因或天然kmura3基因),并且受体宿主生物体具有非回复性ura3-表型,通常是由于天然kmura3基因座(locus)处的缺失。为了能够在随后的工程步骤中再次使用ura3+选
择,在每个盒中,ura3基因被正向重复的dna序列(direct repeat dna sequence)包围,该正向重复的dna序列允许从已被整合的盒中缺失ura3基因,通过所述正向重复的dna序列之间的同源重组,在第二步中通过在含有5'-氟乳清酸(5'-fluoroorotic acid,5'-foa)的基本培养基上针对ura3基因进行选择。
[0053]
因此,用于整合破坏的盒的一般设计按以下顺序具有以下特征:1)与目标染色体序列同源的碱基对的“上(up)”序列,该目标染色体序列正好位于所需整合目标位点的上游2)与目标染色体序列同源的碱基对的“下(down)”序列,该目标染色体序列正好位于所需缺失端点的下游3)可选标记基因,例如ura3基因(和任选地为反向可选择的)4)与所需待缺失的染色体靶序列的至少一部分同源的碱基对的“中间(middle)”序列。一经转化和选择,组装的盒通过“上”序列和“中间”序列之间的同源双重组整合到染色体靶位点。包含正确整合的转化子通过诊断pcr进行鉴定,该诊断pcr显示具有正确预期大小的上游和下游连接片段。在第二步中,通过反向选择(counterselection)以及在盒内部的“下”序列以及逻辑上存在于整合的盒下游的染色体中的与“下”同源的序列之间的同源重组,可选标记基因被“环出”。
[0054]
另一方面,用于整合的表达盒的一般设计按以下顺序具有以下特征:1)与目标染色体序列同源的更多个碱基对的“上”序列,该目标染色体序列正好位于所需整合目标位点的上游2)被设计为待整合的序列,例如,启动子-orf-终止子组合3)与目标染色体序列同源的碱基对的“下”序列,该目标染色体序列正好位于所需缺失端点的下游4)可选标记基因,例如ura3基因(和任选地为可反向选择的)5)与所需待缺失的染色体靶序列的至少一部分同源的碱基对的“中间”序列。如果要缺失目标染色体序列而不伴随插入,则省略上述通用设计的第二个片段2)。包含正确整合的转化子通过诊断pcr进行鉴定,该诊断pcr显示具有正确预期大小的上游和下游连接片段。在第二步中,通过反向选择以及在盒内部的“下”序列以及逻辑上存在于整合的盒下游的染色体中的与“下”同源的序列之间的同源重组,可选标记基因被“环出”。
[0055]
在下文中显示的本发明的实施方式并非出于限制本发明的任何范围的任何目的。
[0056]
本发明涉及一种基因工程化的克鲁维酵母菌属的种的酵母细胞,其中,所述酵母细胞至少包含使编码氨基酸seq id no.2的核苷酸序列失活或缺失的基因修饰。
[0057]
在一个实施方式中,根据本发明的基因工程化的克鲁维酵母菌属的种(kluyveromyces sp.)的酵母细胞包含编码氨基酸seq id no.2的核苷酸序列,其是一种酸反应性转录因子haa1。
[0058]
在一个实施例中,与亲本相比,根据本发明的基因工程化的克鲁维酵母菌属的种(kluyveromyces sp.)的酵母细胞包含增加乳酸耐受性的基因修饰。
[0059]
在一个实施方案中,与亲本相比,根据本发明的基因工程化的克鲁维酵母菌属的种(kluyveromyces sp.)的酵母细胞包含增加乳酸产量的基因修饰。
[0060]
在一个实施方案中,根据本发明的基因工程化的克鲁维酵母菌属的种(kluyveromyces sp.)的酵母细胞可以选自乳酸克鲁维酵母(kluyveromyces lactis)、马克斯克鲁维酵母(kluyveromyces marxianus)或耐热克鲁维酵母(kluyveromyces thermotolerans)。
[0061]
在另一个实施方案中,根据本发明的基因工程化的克鲁维酵母菌属的种
(kluyveromyces sp.)的酵母细胞可用在用于乳酸生产的所有发酵过程中。
[0062]
为了评估kmhaa1基因对乳酸耐受性和乳酸产量的影响,研究了用于haa1基因的破坏和过度表达的基因修饰,并将结果与亲本菌株进行了比较。结果示于图3-6中。
[0063]
提供以下实施例是为了进一步解释本发明,但并非旨在限制本发明的范围。
[0064]
然后,为了确认kmhaa1基因对乳酸耐受性的破坏效果,本发明用了马克斯克鲁维酵母(k.marxianus)的2个菌株——能够产生d-lac的myr2787以及不能够产生d-lac的myr2297。
[0065]
用于马克斯克鲁维酵母(k.marxianus)菌株myr2297和myr2787的dna转化的方法
[0066]
以下基于化学的dna转化方法改编自阿卜杜勒-巴纳特(abdel-banat)等人发表的方案(abdel-banat,2010),以针对菌株myr2297和myr2787进行提高,其中许多在本文描述的实施例中得到命名和使用。
[0067]
将待转化菌株的新鲜单菌落接种到5ml ypd培养基中,该ypd培养基每升包含10g酵母提取物、20g蛋白胨和3g葡萄糖。该起始培养物在30℃的振荡培养箱中过夜培养至饱和。
[0068]
之后,将5ml培养的酵母接种到45ml含2%葡萄糖的ypd培养基中,在30℃的振荡培养箱中以225rpm的转速经过约4至8小时,直到od 600nm为约4到5。
[0069]
在培养物的生长过程中,通过在热循环仪中加热10分钟,然后在冰水浴中快速冷却试管来制备10mg/ml单链鲑鱼精子(ssdna)的溶液。通过将10μl ssdna溶液添加到每个试管中,然后添加5至10μl用于转化至菌株的实验dna,从而为每个单独的转化制备微量离心管(eppendorf tube)。理想情况下,实验dna的浓度应为每个质粒约500至1,000ng dna。
[0070]
制备无菌转化混合物(tm)以化学方法制备细胞用于转化,该混合物包含最终浓度的peg 3350(40%聚乙二醇)、2m乙酸锂(liac)和1m二硫苏糖醇(dtt)。在实践中,这种tm是通过在转化当天结合三种原液制备的。每1ml的tm组成为800μl的50%peg3350、100μl的2m liac和100μl的1m dtt。
[0071]
一旦待转化的培养物达到约4至5的od 600nm,细胞在室温下以7500转每分钟(rpm)离心5分钟。倒掉上清液,将细胞团块重悬于1ml tm中,并在相同条件下再次离心。用微量移液器除去上清液并重新悬浮于1ml tm中。对于后续步骤,将15μl混合dna和85μl的等分细胞悬液加入到每个转化管中,并彻底混合约1至2分钟。将试管置于42℃水浴或加热器(heating block)中1小时,对每次转化进行热冲击。
[0072]
热冲击后,然后用1ml cm减尿嘧啶培养基(cm minus uracil medium)在微量离心机(microfuge)中全速团块化(pelleted)并冲洗细胞。将每次转化的细胞离心沉降(span down)并吸去上清液。用1ml的新鲜cm减尿嘧啶培养基重新悬浮每个团块,并将100μl细胞悬液分散在含有cm减尿嘧啶培养基的板上。将板在30℃下孵育直到出现菌落,通常需要2至4天。
[0073]
携带有δhaa1破坏的菌株myr2297(myr2297 δhaa1)的构建
[0074]
表2生长培养基的组成。所有列出的都是每升的数量。对于培养皿(petri plate),添加20g/l琼脂。
[0075]
配料cm减尿嘧啶sdm2ypd葡萄糖 可变的(20-200g)20g
蔗糖 可变的(20-200g) 天惠华(teknova)cm-ura混合物一个1l包装
ꢀꢀ
磷酸二氢钾
ꢀꢀꢀ
磷酸二氢铵 13.8g 磷酸氢二铵 3.96g 硫酸镁7h2o 0.493g 酵母提取物
ꢀꢀ
10g蛋白胨
ꢀꢀ
20g盐酸硫胺素 200mcg 烟酸 3mg 生物素 10mcg 泛酸钙 400mcg 1000x微量元素* 1ml ph(用氢氧化铵或磷酸) 6.2 mes(2-(n-吗啉代)乙磺酸)
ꢀꢀꢀ
甜菜碱(betaine)
ꢀꢀꢀ
氯化钠 0.234g 氯化钾 0.521g [0076]
*1000x微量元素,每升:1.6g六水氯化铁(fecl3·
6h2o)、0.1g二水氯化铜(cucl2·
2h2o)、0.2g氯化锌(zncl2)、0.05g硼酸(h3bo3)、0.55g四水氯化锰(mncl2·
4h2o)、10ml 85%磷酸
[0077]
起始菌株是马克斯克鲁维酵母(k.marxianus),马克斯克鲁维酵母具有尿嘧啶营养缺陷型酵母菌株和野生型haa1基因,即myr2297 haa1。通过整合构建在质粒phaa1del2上的破坏盒,缺失haa1基因。该破坏盒包括1)“haa上”序列2)“haa下”序列3)作为可选标记的scura3序列和4)“haa中间”序列。图1给出了显示phaa1del2的结构图,其序列如seq id no.1所示。图2中给出了kmhaa1缺失的程序。破坏盒被设计成整合在目标haa1基因座(loci),其通过同源重组编码氨基酸seq id no.2。通过从scura3基因中选择,在cm减尿嘧啶培养基上选择多个缺失菌株。然后,ura3基因通过含有5'-foa的培养基上的下游侧翼的正向重复之间的同源重组环出。然后,必要时将所得的单个菌落重新划线(restreaked)一次或多次,以从背景细胞中释放正确的菌株并消除杂合二倍体。使用合适的引物通过pcr识别正确的插入和正确的环出,该引物将盒末端与正好在整合盒的上游或正好在整合盒的下游的靶基因座处的染色体序列之间的边界括起来。pcr诊断无法区分单倍体中正确整合的盒与正确整合的纯合二倍体,因此,在构建的任何步骤都没有进行此类区分。在确认环出后,天然kmura3基因通过线性dna片段的转化重新安装,该线性dna片段是通过从马克斯克鲁维酵母野生型菌株染色体dna作为模板pcr获得的,通过在cm减尿嘧啶板上选择,产生尿嘧啶原养型微生物(prototroph)。由此产生的菌株,其现在包含haa1基因破坏,被命名为myr2297 δhaa1。
[0078]
携带有δhaa1破坏的菌株myr2787(myr2787 δhaa1)的构建
[0079]
起始菌株是马克斯克鲁维酵母(k.marxianus),马克斯克鲁维酵母具有尿嘧啶营
养缺陷型酵母菌株和能够产生d-lac的野生型haa1基因,即myr2787 haa1。myr2787 haa1的构建描述如下。被设计成表达ecldha基因的三种不同的盒被构建在质粒上。在所有三种情况下,ldha基因都是从kmpdc1启动子上表达的。通过同源重组,这三个ecldha盒被设计并插入靶基因座处,即kmpdc1、kmgpp1和kmnde1基因座处。通过从scura3基因中选择,在cm减尿嘧啶培养基上选择缺失菌株(disruptant)。然后ura3基因通过含有5'-foa的培养基上的下游侧翼的正向重复之间的同源重组环出,以将ura3基因重复用于后续的转化。在每个转化步骤和每个环出步骤,必要时将单个菌落重新划线一次或多次,以从背景细胞中释放正确的菌株并消除杂合二倍体。使用合适的引物通过pcr识别正确的插入和正确的环出,该引物将盒末端与正好在整合盒的上游或正好在整合盒的下游的靶基因座处的染色体序列之间的边界括起来。pcr诊断无法区分单倍体中正确整合的盒与正确整合的纯合二倍体,因此,在构建的任何步骤都没有进行此类区分。以菌株myr2297作为起始,安装三个ldha盒,按上文列出的顺序一次安装一个。在每次初始整合盒后,ura3基因通过5'-foa反向选择环出。这样安装第三个盒后,天然kmura3基因通过线性dna片段的转化重新安装,该线性dna片段是通过从马克斯克鲁维酵母野生型菌株染色体dna作为模板pcr获得的,通过在cm减尿嘧啶板上选择,产生尿嘧啶原养型微生物。由此产生的菌株,其现在包含整合的ldha基因的三个拷贝,被被命名为myr2787 haa1。
[0080]
haa1的破坏盒被设计成通过同源重组整合在靶基因座,其程序与上文描述的构建myr2297 δhaa1中的程序相同。由此产生的d-lac生产菌株,其现在包含haa1基因破坏,被命名为myr2787 δhaa1。
[0081]
携带有haa1过度表达的酵母菌株(myr2787 2x haa1)的构建
[0082]
haa1过度盒被设计成通过交换到ldha盒来整合至菌株myr2287。质粒按以下顺序具有以下特征:1)强的组成型启动子2)haa1编码区3)终止子4)作为可选标记的scura3和5)与ldha基因座的中间序列有同源性。尽管盒被设计成能够整合到三个ldha表达基因座中的任何一个中,gpp1基因座中的haa1过度表达盒的交换整合被选定以用于进一步检查。由此产生的d-lac生产菌株,其现在在gpp1基因座包含haa1表达盒(而非ldha表达盒),被命名为myr2787 2x haa1。
[0083]
补充乳酸的培养基上的酵母生长比较
[0084]
为了确定和比较如上所述获得的所有酵母菌株在低ph条件下的生长性能,在不含l-乳酸和含有0.5%、1.0%、1.5%和2.0%重量/体积百分比(w/v)的l-乳酸的cm减尿嘧啶培养基中培养在myr2297 haa1、myr2297 δhaa1、myr2787 haa1、myr2787 δhaa1和myr2787 2x haa1中的菌株。每种培养基的起始ph值分别为3.5、2.7、2.5、2.4和2.3。所有固体培养基均含有2%的琼脂。通过点样观察(spotting)连续10倍稀释液对酵母菌株之间的生长差异进行定量比较,从107细胞开始,在同一琼脂板上彼此并排,其在30℃下孵育2天。
[0085]
biolector(2升)中酵母生长和d-lac产量的比较
[0086]
为了确定和比较酵母生长性能及其d-lac产量,在biolector微型发酵器中的花板(flower plate)中培养myr2787 haa1、myr2787 δhaa1和myr2787 2x haa1的d-lac生产菌株。myr2787 haa1、myr2787 δhaa1和myr2787 2x haa1的酵母菌株的单菌落被分别接种到试管中的5ml cm减尿嘧啶中,并在37℃下通过使用旋转孵育以200rpm的速度孵育12-18小时。之后,将0.1ml接种物接种到biolector培养板中的0.9ml sdm2培养基(如表2所示)中,
其中起始od 600nm为0.1-0.2并且ph为6.0-6.2。分别将各花板以1200rpm的速度振荡并在37℃孵育48小时。湿度控制在50%-80%,以消除实验过程中的水分蒸发。在发酵过程中,未对ph进行控制。因此,由于d-lac的产生,每种培养物的ph值随着发酵而下降。培养开始后,每30分钟自动测量一次od 600nm的生物量。48小时后,实验停止。通过离心将每个菌株的细胞团(cell mass)沉降(spin down)。收集上清液并分析乳酸含量的量。
[0087]
如图3和图4中所示,与亲本菌株相比,所有具有δhaha1破坏(myr2297δhaa1和myr2787 δhaa1)的酵母细胞均提高了其在低ph条件下的生长。此外,如图4中所示,与亲本菌株相比,具有haa1基因过度表达的酵母细胞(myr2787 2x haa1)在低ph条件下显示出敏感的响应性。这些结果说明,与亲本菌株相比,具有δhaha1破坏的酵母细胞增加了乳酸耐受性。
[0088]
如图5中所示,亲本菌株(myr2787 haa1)和具有haa1基因的过度表达的酵母细胞(myr2787 2x haa1)的生长在培养12小时后停止,而具有δhaa1破坏的酵母细胞(myr2787 δhaa1)的生长在继续。这表明具有δhaa1破坏(myr2787 δhaa1)的酵母细胞在低ph条件下具有提高的生长性能。该结果有力地证实了,与亲本菌株相比,具有δhaa1破坏的酵母细胞增加了乳酸耐受性。
[0089]
如图6中所示,由myr2787亲本细胞(myr2787 haa1)、携带有δhaa1破坏的myr2787(myr2787 δhaa1)和携带有haa1过度表达的myr2787(myr2787 2x haa1)在48小时时产生的d-lac滴度分别为3.8g/l、7.5g/l和0.8g/l。该结果清楚地表明,与亲本菌株相比,具有δhaa1破坏的酵母细胞(myr2787 δhaa1)在没有ph控制的情况下具有乳酸产量的提高。另一方面,具有haa1基因过度表达的酵母细胞(myr2787 2x haa1)产生的乳酸少于亲本菌株和具有δhaa1破坏的酵母细胞(myr2787 δhaa1)。
[0090]
从上述所有实施例的结果可以看出,本发明中公开的具有δhaa1破坏的基因工程化的酵母细胞具有乳酸耐受性、乳酸产量或它们的组合的提高,如发明简述中所提到的。本发明是首次发现。
[0091]
本发明的最佳方式
[0092]
本发明的最佳方式如详细说明书中所公开。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1