人胶质母细胞瘤的预后靶点基因的筛选方法及应用与流程
本发明涉及一种人胶质母细胞瘤的预后靶点基因的筛选方法及应用,属于生物信息学领域。
背景技术:
脑肿瘤是最具破坏性的癌症之一,胶质母细胞瘤(gbm)是这种疾病最具侵袭性和致死性的形式,中位生存期为14个月。肿瘤异质性(包括瘤间和瘤内)非常高,大多数肿瘤对治疗产生抗药性,很快就会复发。癌症基因组图谱(tcga)它是目前为止可以获得的公开数据库里面数据相对全面的一个,在各个领域得到了广泛的应用,为肿瘤基础医学和转化医学研究者提供了海量的基因组数据和与其关联的临床数据。中国胶质瘤基因组图谱(cgga)是一个由2000多个中国胶质瘤样本组成的数据库,它为胶质瘤提供了大量的基因组和临床数据,为更好地了解胶质瘤的生物学和病理学提供了可能。
技术实现要素:
为了寻找gbm诊断和预后特异性好的靶点的基因,本发明以人胶质母细胞瘤干细胞单细胞rna测序数据与癌症基因组图谱(tcga)和中国胶质瘤基因组图谱(cgga)的rna-seq数据、mirna微阵列数据、临床生存数据,利用微阵列分析技术,构建一个共表达网络,筛选出靶点基因,得到与gbm患者生存期显著关联的gbm干细胞相关基因。
本发明提供了一种人胶质母细胞瘤的预后靶点基因的筛选方法,具体步骤包括:
1)以人胶质母细胞瘤干细胞单细胞rna测序数据信息与ii级到ⅳ级的弥散性胶质瘤标本的rna-seq数据、mirna微阵列数据、临床生存数据作为原始数据,将原始数据进行预处理,得到mirna微阵列数据;
2)分析步骤1)中得到的mirna微阵列数据,用r的limma软件包来识别在肿瘤和正常组织之间显著差异表达的基因或mirnas,构建一个共表达网络,确定最佳mirna截止点,筛选靶点基因。
进一步地限定,步骤1)所述的ii级到ⅳ级的弥散性胶质瘤标本的数据来自于cgga和tcga数据库。
进一步地限定,步骤1)所述的预处理采用的是rma方法,包括将探针id转换成基因符号,对于多个探针对应于同一基因符号的情况,选取探针的平均值作为最终的基因表达值。
进一步地限定,步骤2)所述的共表达网络是皮尔森相关性网络,所述最佳的mirna截止点为r=0.3。
进一步地限定,步骤2)中选择显著差异表达的基因或mirnas的临界值是选择p<0.05和log2倍变化率φfc≥0.58。
本发明提供一种人胶质母细胞瘤标记物,其特征在于,所述的标记物为本发明提供的筛选方法筛选出的靶基因,所述的靶基因是与gbm患者生存期显著关联的gbm干细胞相关基因包括abcc3、ap1s3、c1s、cd44、cfi、cp、emp3、fzd7、gclm、lgals1、lgals3、nrp1、osmr、s100a10、steap3、timp1和tnfrsf11b。
本发明还提供了人胶质母细胞瘤标记物在制备gbm的预后的检测试剂盒中的应用。
本发明还提供了人胶质母细胞瘤标记物在制备gbm的治疗药物中的应用。
本发明还提供了人胶质母细胞瘤标记物在制备检测gbm预后效果的试剂盒的应用。
本发明还提供了人胶质母细胞瘤标记物在制备预测人神经胶质瘤母细胞患者生存期的试剂盒中的应用。
有益效果:由于肿瘤异质性的存在,现有的gbm标志物基因对于预后的特异性不高,本发明的生物信息分析的数据包括人胶质母细胞瘤干细胞单细胞测序数据,以至于本发明筛选出的靶点基因都是与gbm患者生存期显著关联的gbm干细胞相关基因,肿瘤异质性是由于干细胞造成的,所述的基因对于gbm预后预测的特异性高。
附图说明
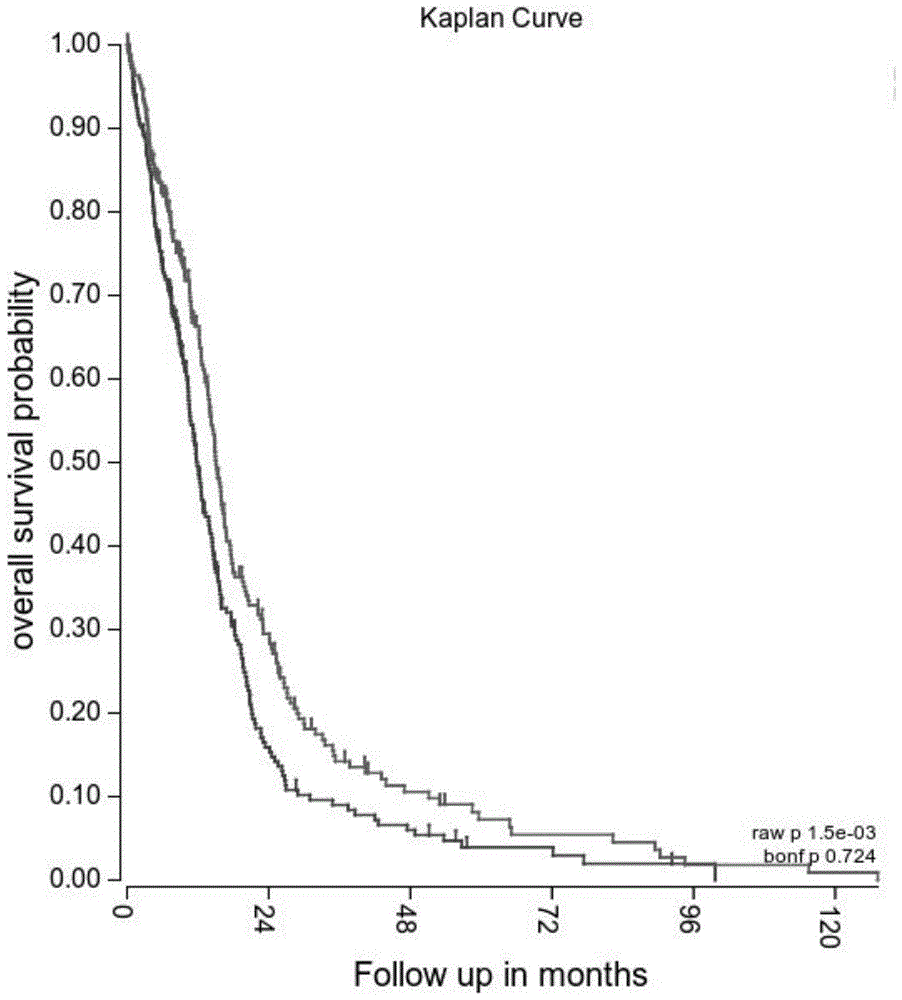
图1.nrp1基因的kaplan-meier分析图;
图2.细胞干性与生存期相关性分析图。
实施例1.
1.以胶质母细胞瘤干细胞单细胞rna测序数据和从cgga、tcga数据库下载的世卫组织ii级到ⅳ级的弥散性胶质瘤标本的基因级rna测序结果、微阵列数据和临床生存数据作为原始数据。将原始数据使用raffy软件包(版本:1.46.1)以标准化的rma方法进行预处理,探针id被转换成基因符号,没有相应基因符号的探针被移除,对于多个探针对应于同一基因符号的情况,选取探针的平均值作为最终的基因表达值。
2.对步骤1中经过数据处理得到的mirna微阵列数据进行分析,利用affy公司的mirna芯片平台,用r(版本:3.30.2)的limma软件包来识别在肿瘤和正常组织之间显著差异表达的基因或mirnas,选择p<0.05和log2倍变化率
3.生存分析采用(2.42-6版)r软件包,以p值<0.05为阈值。筛选与预后相关的mirnas,绘制生存曲线,单因素生存分析,使用rbsurvr软件包来考察单变量生存分析的稳健性,在cgga和tcga数据库以1:1比例选取498例中随机选择3/4样本作为训练数据,其余1/4样本作为验证数据。经过验证gbm中高表达这17种基因的患者,其生存期较空白组又明显的降低,本实施例以nrp1靶点基因为例,对nrp1基因进行了kaplan-meier分析,结果如图1所示,靠下曲线为nrp1高表达的gbm患者,靠上曲线为nrp1低表达的gbm患者,可见在gbm中高表达这nrp1基因的患者,其生存期较空白组又明显的降低,细胞干性与生存期相关性分析如图2所示,横坐标为干细胞(stemness)分化后的细胞类型个数,纵坐标为survival生存期的天数,可见tcgaidh1-wttmzpatient的生存率同神经胶质母细胞瘤的细胞干性有关,细胞分化的越多,患者的生存越差,蓝色曲线代表nrp1富集组患者,其生存期明显缩短。
- 还没有人留言评论。精彩留言会获得点赞!