一种基因型数据压缩方法、系统、计算机设备及存储介质与流程
本发明涉及生物信息学
技术领域:
,尤其涉及一种基因型数据压缩方法、系统、计算机设备及存储介质。
背景技术:
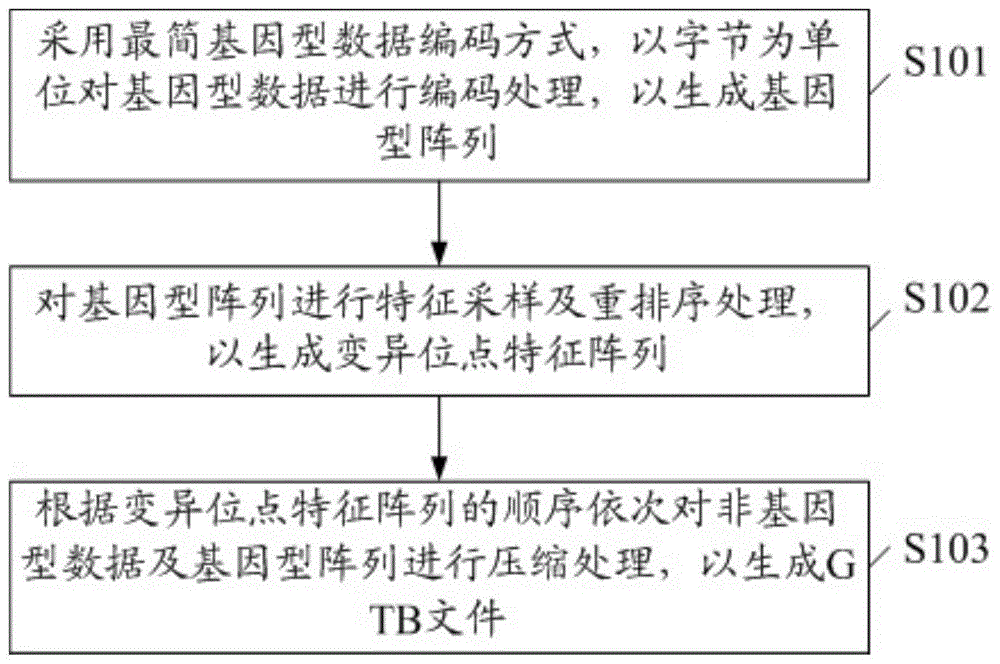
:基因组文件是基因组变异研究的重要基本文件。大型测序项目产生的基因组文件十分庞大,以千人基因组计划为例,项目的最终阶段采集了2504人的基因序列,涵盖了8800万变异位点,这些数据文件经过通用的gzip(gnu-zip)压缩算法压缩后仍然超过25gb大小。因此,结合基因组数据的特点,需设计专用压缩算法来缩减空间大小。目前,基因组文件压缩研究的重心在占据庞大空间的基因型数据上,压缩的基本手段包括:对基因型数据重新表示(编码)、使用排列算法增强数据间的一致性(重排序)、使用字典压缩算法进行压缩(压缩)。自2016年问世的pbwt(参见durbinr.efficienthaplotypematchingandstorageusingthepositionalburrows–wheelertransform(pbwt)[j].bioinformatics,2014,30(9):1266-1272.)、bgt(参见lih.bgt:efficientandflexiblegenotypequeryacrossmanysamples[j].bioinformatics,2016,32(4):590-592)、gtc(参见daneka,deorowiczs.gtc:howtomaintainhugegenotypecollectionsinacompressedform[j].bioinformatics,2018,34(11):1834-1840)压缩算法是兼顾高效压缩与随机访问的优秀技术。编码上,pbwt、bgt、gtc都将基因型数据转为2个单倍型,并将每个单倍型都编码为2个位。重排序上,pbwt、bgt都使用了pbwt变换,而gtc使用了基于数据特征的最近邻启发式算法。压缩上,pbwt、bgt、gtc主要采用字典类压缩技术(如,lzma压缩、huffman压缩)。但是,上述压缩算法具有以下缺点:(1)基因组数据压解不一致:pbwt变换只能对01序列进行,这些算法需要将基因组数据转为定长01编码序列,因而需要将复等位基因位点转为双等位基因位点,再对双等位基因位点编码为4个位(每个单倍型都是2个位),破坏了基因组数据的原始信息;(2)压解速度慢:所使用的排列算法复杂度高,在大样本基因组数据中推进效率低;lzma算法在大型数据上压缩速度较慢;检索结构不合理、不可复用检索缓存结构;(3)压缩比上:bgt编码导致数据过度预压缩,降低了后续字典压缩技术的功效;(4)文件管理上:这些工具将基因组文件的不同组件分别存放到不同的子文件中,随着数据文件越来越多,它们产生的压缩存档也越来越庞杂,无法应对大规模人群基因组中的集群文件管理需求;同时,非解耦式结构使得局部压缩存档的修改、保留、合并都需要先将压缩存档还原回基因组文件,再重新压缩。技术实现要素:本发明所要解决的技术问题在于,提供一种基因型数据压缩方法及计算机设备,可实现更为高效、统一的大规模基因型数据区块压缩。为了解决上述技术问题,本发明提供了一种基因型数据压缩方法,包括:采用最简基因型数据编码方式,以字节为单位对基因型数据进行编码处理,以生成基因型阵列;对所述基因型阵列进行特征采样及重排序处理,以生成变异位点特征阵列;根据所述变异位点特征阵列的顺序依次对非基因型数据及所述基因型阵列进行压缩处理,以生成gtb文件。作为上述方案的改进,所述采用最简基因型数据编码方式,以字节为单位对基因型数据进行编码处理,以生成基因型阵列的步骤包括:判断变异位点的等位基因个数是否处于预设范围内;当所述变异位点中等位基因个数小于预设范围时,采用单字节模式将所述变异位点的每个基因型a|b编码为:当所述变异位点中等位基因个数处于预设范围内时,采用双字节模式将所述变异位点的每个基因型a|b编码为:作为上述方案的改进,所述对基因型阵列进行特征采样及重排序处理,以生成变异位点特征阵列的步骤包括:定义所述基因型阵列中变异位点的特征向量λv,其中,在单字节模式下,λv=[1,f(qv0),-f(qv1),index],“1”表示编码位长为1字节,f(qv0)为0等位基因的频率映射值,-f(qv1)为1等位基因的频率映射值,index为该位点在该压缩块中的原始位置,在双字节模式下,λv=[2,0,0,index],“2”表示编码位长为2字节;根据所述特征向量的字典序对压缩块中的变异位点进行排序,生成新的位点排列顺序。作为上述方案的改进,所述等位基因的频率映射值的计算步骤包括:基因型a|b中具有的等位基因个数表示为:si[a×15+b]=i(a=i)+i(b=i),其中,i为等位基因;每个变异位点的等位基因频率表示为:其中,v为变异位点;根据公式将等位基因频率进行变换,并映射到子区间上,其中,f(q)为等位基因的频率映射值。作为上述方案的改进,所述根据变异位点特征阵列的顺序依次对非基因型数据及基因型阵列进行压缩处理,以生成gtb文件的步骤包括:将非基因型数据中的位置数据转换为整数,并进行拼接处理;将非基因型数据中的碱基序列进行拼接处理;将拼接后的位置数据、拼接后的碱基序列及变异位点特征阵列通过zstd压缩方法进行压缩处理,以生成gtb文件。作为上述方案的改进,所述gtb文件包括文件基本信息、样本名称序列、第一数据分隔符、压缩块摘要信息、第二数据分隔符及压缩块数据段;所述压缩块摘要信息包括染色体编号、块最小位点、块最大位点、块变异位点数量及块数据段长度。作为上述方案的改进,所述基因型数据压缩方法还包括:解压所述gtb文件时,扫描所述gtb文件的压缩块摘要信息以构建gtbroot检索树,并利用所述gtbroot检索树对所述gtb文件进行解压;所述gtbroot检索树以所述染色体编号作为一级节点,并以同一染色体编号下的每个压缩块摘要信息作为子叶。相应地,本发明还提供了一种基因型数据压缩系统,包括:编码模块,用于采用最简基因型数据编码方式,以字节为单位对基因型数据进行编码处理,以生成基因型阵列;采样及重排序模块,用于对所述基因型阵列进行特征采样及重排序处理,以生成变异位点特征阵列;压缩模块,用于根据所述变异位点特征阵列的顺序依次对非基因型数据及所述基因型阵列进行压缩处理,以生成gtb文件。相应地,本发明还提供了一种计算机设备,包括存储器和处理器,所述存储器存储有计算机程序,所述处理器执行上述基因型数据压缩方法的步骤。相应地,本发明还提供了一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述基因型数据压缩方法的步骤。实施本发明,具有如下有益效果:本发明提出以“字节”为粒度(单位)进行编码,构建了新的最简基因型数据编码方式,能够充分保留基因型数据间的规律;同时,本发明还引入重排序技术及zstd压缩方法,使得压缩比、压解速度提升至更高的水平;另外,本发明还构建了新型的区块状储存结构——gtb文件,可使一个vcf文件可以仅产生一个压缩存档,而对于同一个测序群体的多个vcf文件,也可以将这些vcf文件压缩为一个整体,统一性、灵活性强;进一步,本发明还构建了新型的gtbroot检索树,重复进行多次检索时,只需要构建一次gtbroot检索树,减少了频繁查询数据的预处理时间开销,具有极高的检索效率。附图说明图1是本发明基因型数据压缩方法的实施例流程图;图2是本发明基因型数据压缩方法的实施例示意图;图3是本发明中gtbroot检索树的结构示意图;图4是本发明基因型数据压缩系统的结构示意图。具体实施方式为使本发明的目的、技术方案和优点更加清楚,下面将结合附图对本发明作进一步地详细描述。参见图1,图1显示了本发明基因型数据压缩方法(genotypeblockedcompressor,gbc)的实施例流程图,其包括:s101,采用最简基因型数据编码方式,以字节为单位对基因型数据进行编码处理,以生成基因型阵列。现有基因型数据编码方法都是将基因型数据以“位”为粒度进行编码,但这样的编码无法直接处理多等位基因型数据,也会掩盖基因型数据的内部规律。与现有技术不同的是,本发明提出以“字节”为粒度(单位)进行编码,构建了新的最简基因型数据编码(themostparsimoniousbyteencodingofgenotype,mpbeg)方式。具体地,所述采用最简基因型数据编码方式对基因型数据进行编码处理,以生成基因型阵列的步骤包括:(1)判断变异位点的等位基因个数是否处于预设范围内。需要说明的是,识别字节模式的唯一标准是变异位点的ref碱基字段和alt碱基字段所具有的等位基因个数,因此,本发明根据等位基因个数对变异位点进行分类编码。(2)当所述变异位点中等位基因个数小于预设范围时,采用单字节模式将所述变异位点的每个基因型a|b编码为:优选地,所述预设范围为16~255,也就是说,当所述变异位点中等位基因个数nv<16时,采用单字节模式将所述变异位点的每个基因型进行编码。需要说明的是,数值范围在0~225之间,可以在1字节内进行表示,并定义此模式为单字节模式。实际上,单字节模式已经可以表示足够多等位基因的变异位点。因此,默认情况下,只采用单字节模式进行编码,以降低压缩时的内存空间需求和压缩后的硬盘存储空间需求。(3)当所述变异位点中等位基因个数处于预设范围内时,采用双字节模式将所述变异位点的每个基因型a|b编码为:优选地,所述预设范围为16~255,也就是说,当所述变异位点中等位基因个数nv满足条件:16≤nv≤255时,采用双字节模式将所述变异位点的每个基因型编码。需要说明的是,数值范围在0~65025之间,可以在2字节内进行表示,并定义此模式为双字节模式。另外,为了加速编码/解码过程,本发明将编码/解码过程中所采用的编码表储存于线性数组中,其中,所述编码/解码表以编码值作为索引,可直接提取数据的字节表示。例如,解码过程的0对应基因型0|0,在解码表中取得的结果是字节数组{9,48,124,48}。因此,本发明所采用的最简基因型数据编码方式,是保存单个基因型数据的最简字节形式,能够充分保留基因型数据间的规律,使得压缩比、压解速度提升至更高的水平。s102,对所述基因型阵列进行特征采样及重排序处理,以生成变异位点特征阵列。具体地,对基因型阵列进行特征采样的方法为:定义所述基因型阵列中变异位点的特征向量λv。下面分别针对单字节模式及双字节模式进行分别说明。(1)单字节模式对于单字节模式,基因型a|b中具有的等位基因个数表示为:si[a×15+b]=i(a=i)+i(b=i)。每个变异位点的等位基因频率表示为:可将等位基因频率按如下公式进行变换,并映射到8个子区间上:其中,i为等位基因,v为变异位点,f(q)为等位基因的频率映射值。相应地,在单字节模式下,变异位点的特征向量λv=[1,f(qv0),-f(qv1),index。其中,第一项参数“1”表示编码位长为1字节,第二项参数fqv0为0等位基因的频率映射值,第三项参数-f(qv1)为1等位基因的频率映射值,第四项参数index为该位点在该压缩块中的原始位置。(2)在双字节模式下,变异位点的特征向量λv=[2,0,0,index]。其中,第一项参数“2”表示编码位长为2字节,第四项参数index为该位点在该压缩块中的原始位置。另外,对特征采样后的基因型阵列进行重排序的方法为:根据所述特征向量的字典序对压缩块中的变异位点进行排序,生成新的位点排列顺序。s103,根据变异位点特征阵列的顺序依次对非基因型数据及基因型阵列进行压缩处理,以生成gtb文件。具体地,所述根据变异位点特征阵列的顺序依次对非基因型数据及基因型阵列进行压缩处理,以生成gtb文件的步骤包括:(1)将非基因型数据中的位置数据转换为整数,并进行拼接处理。如图2所示,非基因型数据中,将位置数据(pos字段)按照int类型保存(即每个位置数据占用4字节),并拼接到一起。(2)将非基因型数据中的碱基序列进行拼接处理。如图2所示,非基因型数据中,将ref碱基序列和alt碱基序列使用“/”作为分隔符直接拼接在一起。(3)将拼接后的位置数据、拼接后的碱基序列及变异位点特征阵列通过zstd压缩方法进行压缩处理,以生成gtb文件。需要说明的是,zstd压缩方法与lzma压缩方法都是经典的字典类压缩算法,其中,zstd压缩方法具有更高的吞吐速度,而lzma压缩方法具有更高的压缩比。由于本发明所采用的最简基因型数据编码和重排序技术,使得压缩比提升至更高的水平,因此,本发明使用具有更高吞吐速度的zstd压缩方法进行压缩。如图2所示,所述gtb文件包括文件基本信息、样本名称序列、第一数据分隔符、压缩块摘要信息、第二数据分隔符及压缩块数据段;其中,所述压缩块摘要信息包括染色体编号、块最小位点、块最大位点、块变异位点数量及块数据段长度;所述压缩块数据段包括经过处理的位置数据阵列、经过处理的碱基序列(ref碱基序列、alt碱基序列)、数据分隔符及经过处理的基因型阵列。现有的基因组文件压缩算法都将不同的字段拆分到多个子文件中进行储存。与现有技术不同的是,本发明设计了一种新型的区块状储存结构——gtb文件。gtb文件可使一个vcf文件可以仅产生一个压缩存档,而对于同一个测序群体的多个vcf文件,gtb文件也可以将这些vcf文件压缩为一个整体,统一性、灵活性强。因此,通过gtb文件可使大型基因组文件的管理变得更为简便,能够灵活应用于多种数据分析、存储场景。同时,gtb文件还便于传输、使用,支持增量压缩、局部提取数据、局部修改数据,也支持并行地进行压解,可充分利用计算机多核特性,提高算法的推进速度。因此,本发明设计了新型的编码方式、新型的基因型阵列排列算法、新型的存储架构,并结合更快速的字典类压缩技术,实现一种更为高效、统一的基因型数据压缩方法,在压解速度、压缩比上相比过去的工具具有重大提升。进一步,所述基因型数据压缩方法还包括:解压所述gtb文件时,扫描所述gtb文件的压缩块摘要信息以构建gtbroot检索树,并利用所述gtbroot检索树对所述gtb文件进行解压,速度快,在数据解压时具有极高的检索效率。如图3所示,所述gtbroot检索树以所述染色体编号作为一级节点,并以同一染色体编号下的每个压缩块摘要信息作为子叶,形成树形结构。需要说明的是,每个压缩块的变异位点都具有相同的染色体编号,而每个压缩块的染色体编号、块内最小pos、块内最大pos、变异位点总数、数据段大小构成了块的摘要信息。需要说明的是,经典数据结构中已经证实树形结构是一种高效的检索方式。gtbroot检索树的构建只需要采集gtb文件非常短的一段数据,在交互模式下重复检索同一个文件时,这样的gtbroot检索树只需要构建一次,便可重复使用,减少了频繁查询数据的预处理时间开销。同时,依托于树状的gtbroot检索树,gtb文件可以实现增量压缩、局部数据修改、局部数据提取,而不必再解压全部数据后重新进行压缩。下面使用千人基因组计划常染色体数据集对现有技术(bgt、pbwt、gtc)及本发明基因型数据压缩方法(gbc)进行测试对比,测试设备都是macbookpro15'2018(512gb固态硬盘,32gb内存),提取数据采用4线程并行解压(其他软件均不支持并行解压):一、基本性能对比(全部数据,2504样本,81271745位点,816.13gb大小)平台存档大小[gb]压缩比压缩时间[s]解压时间[s]存档数量bgt10.781.88145804287440pbwt8.6102.0389556259330gtc4.1214.07124177432440gbc2.9306.81170313195二、提取指定数量的变异位点用时对比(单位:秒,amr-chr1,347样本,6468094位点)三、提取指定数量的样本全部位点用时对比(单位:秒,afr-chr1,661样本6468094位点)样本数量bgtpbwtgtcgbc14.4459.8985.3200.713105.54512.5508.8450.9055010.53221.91719.3471.61210015.68533.51635.6482.54030037.47778.17066.8989.56450058.206126.113101.87314.003四、过滤性能用时对比(单位:秒,afr-chr2,661样本,7081600位点)等位基因频数范围bgtgtcgbc0-1322(100%)90.416129.90918.370132-1190(80%)23.97427.4183.879264-1058(60%)21.18724.1213.125396-926(40%)19.23523.8402.869528-794(20%)17.76421.6012.734594-728(10%)17.13120.6542.597注:多线程环境下,gbc解压产生的变异位点顺序是无序的;单线程环境下,gbc产生的变异位点顺序是有序的,此时也仍能以1倍以上速度快于现有的工具。由上可知,本发明基因型数据压缩方法通过创新的编码方式、基因型阵列排列方式、索引结构、存储架构,提供极快的压解速度和精简统一的文件储存方式,并可成为本领域先进数据分析工具的基础组件之一。如图,本发明还提供了一种基因型数据压缩系统100,其包括:编码模块1,用于采用最简基因型数据编码方式,以字节为单位对基因型数据进行编码处理,以生成基因型阵列。具体地,所述编码模块1执行上述步骤s101,在此不再重复描述。采样及重排序模块2,用于对所述基因型阵列进行特征采样及重排序处理,以生成变异位点特征阵列。具体地,所述采样及重排序模块2执行上述步骤s102,在此不再重复描述。压缩模块3,用于根据所述变异位点特征阵列的顺序依次对非基因型数据及所述基因型阵列进行压缩处理,以生成gtb文件。具体地,所述压缩模块3执行上述步骤s103,在此不再重复描述。相应地,本发明还提供了一种计算机设备,包括存储器和处理器,所述存储器存储有计算机程序,所述处理器执行上述基因型数据压缩方法的步骤。同时,本发明还提供了一种计算机可读存储介质,其上存储有计算机程序,所述计算机程序被处理器执行时实现上述基因型数据压缩方法的步骤。以上所述是本发明的优选实施方式,应当指出,对于本
技术领域:
的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也视为本发明的保护范围。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1