用于在猪中检测产仔数的基因标记的方法和试剂盒与流程
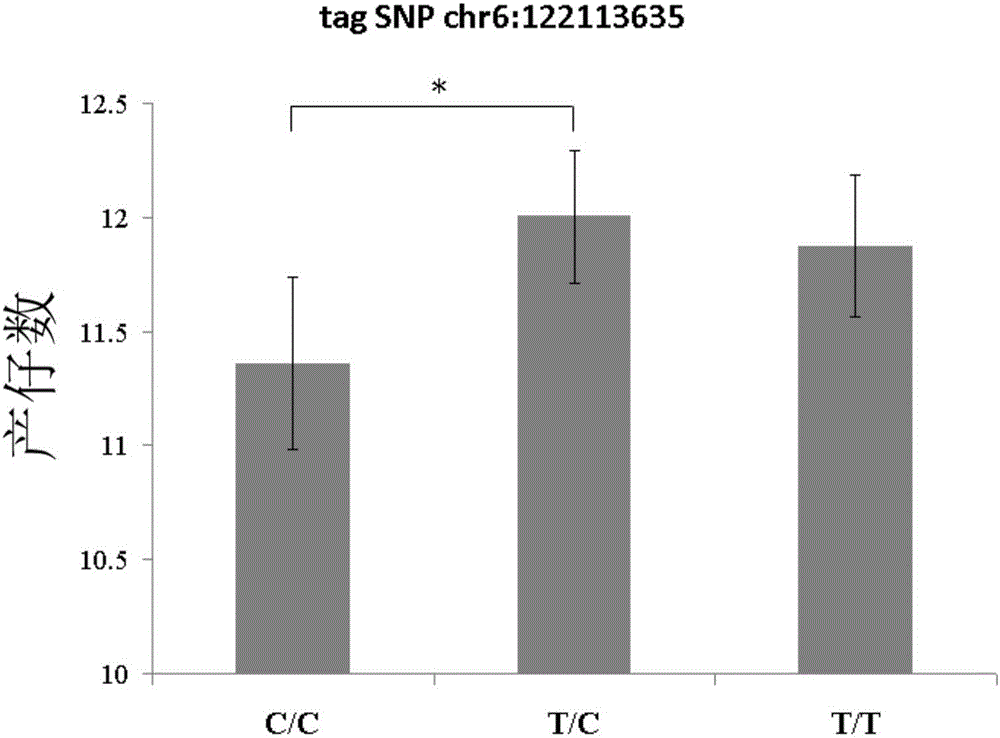
猪是全球性的经济动物并且是重要的肉类来源。在2009年,全世界饲养了约9.66亿头猪,猪肉生产的估计价值达1630亿美元,并且近半数的猪(4.76亿)都在中国(http://www.fao.org)。随着全球市场需求的提升,其群体数已经逐年增加。通过饲养与总群体成比例的母猪和公猪群来生产仔猪是至关重要的。在猪繁殖过程中,公猪的数量比母猪的数量少得多。通常,母猪群大小占总群大小的比例稍大于1/10,其相当于全世界数量约为1亿的相当大的母猪群。一般地,猪的繁殖成本主要来自于饲料。对于猪的繁殖,在怀孕和哺乳期需要特别的照料,这提升了饲养母猪的成本。减少母猪群数量但仍能提供足够的仔猪,这将有利于养猪业。将母猪群数量减少10%将能每年节约掉饲养1000万头母猪的成本,这可以通过母猪每窝生产1个额外的仔猪来实现。体现为母猪产仔数的繁殖效率的提高是养猪业在经济上的考量。中国太湖猪由几个来自地域紧密相连的太湖流域地区的地方品种(包括二花脸猪、梅山猪和枫泾猪等)组成。太湖猪因其繁殖力而闻名;例如,太湖猪每窝可以比大多数其他中国地方猪种和西方商业品种多生产4-5个额外的仔猪[1]。太湖猪较大的产仔数背后的基因序列/变体是改善猪的产仔数性状方面的宝贵资源。与产仔数相关的基因标记已经在几个基因,例如OPN(US6410227B1)、PRLR(US7081335B2)、FSHβ(US006291174B1)、ESR(US005550024A)和RBP4[2]中得到了很好的描述。数量性状位点(QTL)定位已经鉴定了很多造成太湖猪高产仔数的区域,其跨越6号、7号、8号和15号染色体[3,4]。然而,QTL通常跨越较大的基因组区域,并且还没有很好地解决遗传变异体的问题。鉴定太湖猪和其他猪之间生殖能力差异背后的遗传变异体,能用来更高效地繁殖母猪。因此,非常需要在二花脸猪中找到人工选择的基因组区域,以及鉴定能在猪的分子育种中使用的高产仔数遗传变异体。发明概述本发明一方面涉及鉴定猪能生产增加的产仔数的方法,所述方法包括在猪基因组中检测基因组区域内的至少一个单核苷酸多态性(SNP)位点处E等位基因SNP标记的存在或缺失,所述基因组区域选自SweepA、SweepB、SweepC、SweepD和SweepE,其中所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述基因组区域是SweepA。在这些实施方案中,所述方法进一步包括在一个或者两个拷贝的染色体的所述SNP位点处检测所述E等位基因SNP标记的存在或缺失。在某些实施方案中,在一个或两个拷贝的染色体的所述SNP位点处所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述SweepA基因组区域跨越自Chr6:122097788至Chr6:122217096的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:A1-A13。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点包括SNPA2,或者与SNPA2连锁不平衡。在某些实施方案中,所述基因组区域是SweepB。在这些实施方案中,所述方法进一步包括在两个拷贝的染色体的所述SNP位点处检测所述E等位基因SNP标记的存在或缺失。在某些实施方案中,在两个拷贝的染色体的所述SNP位点处所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述SweepB基因组区域跨越自Chr6:89403626至Chr6:90311616的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:B1-B91。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点包括SNPB13,或者与SNPB13连锁不平衡。在某些实施方案中,所述基因组区域是SweepC。在在这些实施方案中,所述方法进一步包括在一个或者两个拷贝的染色体的所述SNP位点处检测所述E等位基因SNP标记的存在或缺失。在某些实施方案中,在一个或两个拷贝的染色体的所述SNP位点处所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述SweepC基因组区域位于Chr7:63714553(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepC基因组区域内的所述SNP位点是表1中所列的SNPC1。在某些实施方案中,所述基因组区域是SweepD。在这些实施方案中,所述方法进一步包括在两个拷贝的染色体的所述SNP位点处检测所述E等位基因SNP标记的存在或缺失。在某些实施方案中,在一个拷贝的染色体的所述SNP位点处所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述SweepD基因组区域跨越自Chr15:51799437至Chr15:51800356的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:D1-D3。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点包括SNPD1,或者与SNPD1连锁不平衡。在某些实施方案中,所述基因组区域是SweepE。在这些实施方案中,所述方法进一步包括在一个或者两个拷贝的染色体的所述SNP位点处检测所述E等位基因SNP标记的存在或缺失。在某些实施方案中,在一个或两个拷贝的染色体的所述SNP位点处所述E等位基因SNP标记的存在指示了所述猪的增加的产仔数。在某些实施方案中,所述SweepE基因组区域跨越自Chr3:72655441至Chr3:72795872的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:E1-E23。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点包括SNPE10,或者与SNPE10连锁不平衡。在某些实施方案中,所述E等位基因SNP标记是位于二花脸猪基因组相应SNP位点处的高频核苷酸。在某些实施方案中,所述E等位基因SNP标记如表1中所示。在某些实施方案中,所述方法进一步包括在所述猪被鉴定为生产增加的产仔数的条件下选择所述猪用于繁殖。在某些实施方案中,所述方法进一步包括在所述猪被鉴定为生产增加的产仔数的条件下从所述猪获得生殖细胞用于繁殖。可以使用本发明所公开的方法测试任意猪。在某些实施方案中,所述猪是母猪、太湖猪的后代、或者二花脸猪的后代。可以使用任意合适的方法来实施本文所述的检测。在某些实施方案中,所述检测包括测序来自所述猪的核酸样本中的至少含有所述SNP位点的核酸片段。在某些实施方案中,所述检测包括检测扩增产物,所述扩增产物是来自所述猪的核酸样本中至少含有所述SNP位点的片段的扩增产物。在某些实施方案中,所述检测包括检测探针和来自所述猪的核酸样本中至少含有所述SNP位点的片段的杂交。在某些实施方案中,所述检测包括检测来自所述猪的核酸样本中至少含有所述SNP位点的片段的引物延伸产物。在某些实施方案中,所述检测包括检测来自所述猪的核酸样本中至少含有所述SNP位点的片段的限制酶切产物。在某些实施方案中,所述检测包括检测来自所述猪的核酸样本中至少含有所述SNP位点的片段的凝胶电泳结果。在某些实施方案中,所述检测包括检测蛋白与来自所述猪的核酸样本中至少含有所述SNP位点的片段的结合亲和力。另一方面,本发明提供了检测猪SweepA区域内SNP位点处的所述SNP标记的方法,所述方法包括:在猪基因组中的SweepA基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失。在某些实施方案中,所述SweepA基因组区域跨越自Chr6:122097788至Chr6:122217096的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:A1-A13。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点包括SNPA2,或者与SNPA2连锁不平衡。另一方面,本发明提供了检测猪SweepB区域内SNP位点处的所述SNP标记的方法,所述方法包括:在猪基因组中的SweepB基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失。在某些实施方案中,所述SweepB基因组区域跨越自Chr6:89403626至Chr6:90311616的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:B1-B91。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点包括SNPB13,或者与SNPB13连锁不平衡。另一方面,本发明提供了检测猪SweepC区域内SNP位点处的所述SNP标记的方法,所述方法包括:在猪基因组中的SweepC基因组区域内的所述单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失。在某些实施方案中,所述SweepC基因组区域位于Chr7:63714553(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepC基因组区域内的所述SNP位点是表1中所列的SNPC1。另一方面,本发明提供了检测猪SweepD区域内SNP位点处的所述SNP标记的方法,所述方法包括:在猪基因组中的SweepD基因组区域内的至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失。在某些实施方案中,所述SweepD基因组区域跨越自Chr15:51799437至Chr15:51800356的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:D1-D3。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点包括SNPD1,或者与SNPD1连锁不平衡。另一方面,本发明提供了检测猪SweepE区域内SNP位点处的所述SNP标记的方法,所述方法包括:在猪基因组中的SweepE基因组区域内的至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失。在某些实施方案中,所述SweepE基因组区域跨越自Chr3:72655441至Chr3:72795872的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:E1-E23。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点包括SNPE10,或者与SNPE10连锁不平衡。另一方面,本发明提供了分离的寡核苷酸引物,其用于选择性扩增多核苷酸片段,所述多核苷酸片段在表1中所列的SNP位点中的至少一个处含有E等位基因SNP标记,或者选择性扩增多核苷酸片段,所述多核苷酸片段在表1中所列的SNP位点中的至少一个处缺乏E等位基因SNP标记(例如含有O等位基因SNP标记)。另一方面,本发明提供了分离的寡核苷酸探针,其用于选择性杂交多核苷酸片段,所述多核苷酸片段在表1中所列的一个或多个SNP位点处含有E等位基因SNP标记或者选择性杂交多核苷酸片段,所述多核苷酸片段在表1中所列的一个或多个SNP位点处缺乏E等位基因SNP标记(例如含有O等位基因SNP标记)。另一方面,本发明提供了用于本发明所提供的方法的试剂盒。所述试剂盒包括本文所提供的分离的寡核苷酸引物,或者本文所提供的分离的寡核苷酸探针。附图简述图1.对于白色杜洛克×二花脸F2代母猪群的个体,使用标签SNPchr6:122,113,635检测了3种基因型的产仔数。*表示p<0.05的统计学显著性差异;并且**表示p<0.01的统计学显著性差异。图2.对于苏泰母猪群的个体,使用标签SNPchr6:122,113,635检测了3种基因型的产仔数。*表示p<0.05的统计学显著性差异。图3.对于白色杜洛克×二花脸F2代母猪群的个体,使用标签SNPchr6:89,899,151检测了3种基因型的产仔数。*表示p<0.05的统计学显著性差异。图4.对于白色杜洛克×二花脸F2代母猪群的个体,使用标签SNPchr7:63,714,553检测了3种基因型的产仔数。**表示p<0.01的统计学显著性差异。图5.对于苏泰母猪群的个体,使用标签SNPchr7:63,714,553检测了3种基因型的产仔数。*表示p<0.05的统计学显著性差异;并且**表示p<0.01的统计学显著性差异。图6.对于白色杜洛克×二花脸F2代母猪群的个体,使用标签SNPchr15:51,799,437检测了3种基因型的产仔数。**表示p<0.01的统计学显著性差异。图7.对于苏泰母猪群的个体,使用标签SNPchr15:51,799,437检测了3种基因型的产仔数。*表示p<0.05的统计学显著性差异。图8.对于大白母猪群的个体,使用标签SNPchr3:72,759,645检测了3种基因型的产仔数。**表示p<0.01的统计学显著性差异;并且*表示p<0.05的统计学显著性差异。图9.对于大白猪×长白猪F1代杂交母猪群的个体,使用标签SNPchr3:72,759,645检测了3种基因型的产仔数。**表示p<0.01的统计学显著性差异。发明详述在详细描述本申请的实施方案之前,应当理解,除非另外指出,否则本申请并不限于特定的材料、试剂、反应材料、制造工艺等,因为这些是可以变化的。也应当理解,本文所使用的术语仅用于描述具体实施方案的目的,并且不旨在限制。只要逻辑上可行,在本申请中还可能以不同的顺序实施步骤。本说明书中所引用的所有出版物和专利都通过引用并入本文,如同特别地和单独地指明每个单独的出版物或者专利通过引用并入并且通过引用并入本文来公开和描述与所述出版物所引用的内容相联系的方法和/或材料。猪中增加的产仔数的遗传标记一方面,本发明提供了与猪的增加的产仔数相关的遗传标记。本文使用的术语“遗传标记”是指在基因组或者染色体的已知位置上的核苷酸序列。本文提供的遗传标记与涉及猪产仔数的表型(例如,增加的产仔数表型)相关。“产仔数”是指同一个母亲一次生产动物的后代数。增加的产仔数是大于猪群的平均产仔数的产仔数。例如,增加的产仔数比猪群体的平均产仔数多至少2%、至少3%、至少5%、至少7%、至少8%、至少10%、至少15%、至少20%、至少25%、或者更多。又例如,增加的产仔数比猪群生产的平均产仔数每窝多至少0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9、1.0、1.1、1.2、1.3、1.4、1.5、1.6、1.7、1.8、1.9、2.0、2.1、2.2、2.3、2.4、2.5或者更多。本文使用的术语“基因组”包括来自具有两个拷贝的染色体的猪体细胞的基因组DNA序列,并且还包括来自具有单拷贝的染色体的猪生殖细胞的基因组DNA序列。在某些实施方案中,本文提供的遗传标记是单核苷酸多态性(SNP)标记。SNP是在群体中发生的位于基因组或染色体的特定位置(即SNP位点)处的单核苷酸变化,换句话说,不同的个体可以在特定的SNP位点具有不同的核苷酸,例如,一些可能在SNP位点处具有A,而另一些可能在所述位点具有C。在SNP位点处的核苷酸是SNP标记。对于给定的群体,SNP标记可以在A、T、G和C中变化,每个具有相同的频率或者不同的频率,例如,某些SNP标记(如A或T)可以以高于平均概率的高频率被发现,而某些其他SNP标记(如G或C)可以以低于平均概率的低频率被发现。在不同的群体中,在SNP位点处每个SNP标记的频率可以是保守的或者可以是不保守的。例如,对于给定的SNP位点,在A群体中以高频率发现SNP标记,而在B群体中,有可能以或者不以相似的高频率发现这样的标记,或者甚至在B群体中以低频率发现这样的标记。不束缚于任何理论,可以预期在某些情况下这种SNP位点处的高频SNP标记的差异可以与不同群体中的表型差异相关联。例如,假设:1)A群体具有比B群体更高的产仔数(即不同表型);和2)发现A群体在给定的SNP位点处具有高频SNP标记“C”,但是B群体在给定的SNP位点处具有高频率“T”(即不同的高频SNP标记);如果A群体的表型(高产仔数)可以与SNP位点处“C”的存在(即A群体中的高频SNP标记)相关,那么在SNP位点处的SNP标记可被用作遗传标记以确定候选者是否具有高产仔数表型。在某些实施方案中,本文提供的遗传标记涉及发现于二花脸猪基因组中的高频SNP标记。二花脸猪是主要分布于中国东部的本土猪品种,详见于由中国农业出版社出版的《中国动物遗传资源:猪》(AnimalgeneticresourcesinChina:Pigs),第41-44页,2011。二花脸猪是著名的多产品种,其比其他猪种生产更多的产仔数。本发明人意外地发现,二花脸猪的高产仔数表型与二花脸猪基因组中一些SNP位点处的某些高频SNP标记相关。在这些SNP位点处,二花脸猪基因组中的高频SNP标记不同于来自其他的已知比二花脸猪生产更低产仔数的猪种(例如藏族猪种、巴马香猪种、莱芜猪种以及滇南小耳猪和野生猪)的基因组中的高频SNP标记。这使得本文公开的SNP标记能用作遗传标记来鉴定具有高产仔数表型的猪。在某些实施方案中,本文提供的遗传标记是猪基因组6号染色体上SweepA基因组区域内的SNP标记。本文使用的SweepA基因组区域是指跨越自Chr6:122097788至Chr6:122217096的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,本文提供的SweepA区域内的遗传标记是SNPA1-A13,其位置总结于表1中。为了更好地示意位置,表1还示出了每个SNP位点紧邻的20bp序列,使得本领域人员还能够通过序列同源性检索在给定的猪基因组上找到这些SNP位点。在某些实施方案中,本文提供的遗传标记是猪基因组6号染色体上SweepB基因组区域内的SNP标记。本文使用的SweepB基因组区域是指跨越自Chr6:89403626至Chr6:90311616的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,本文提供的SweepB区域内的遗传标记是SNPB1-B91,其位置总结于表1中。为了更好地示意所述位置,表1还示出了每个SNP位点紧邻的20bp序列,使得本领域人员还能够通过序列同源性检索在给定的猪基因组上找到这些SNP位点。在某些实施方案中,本文提供的遗传标记是猪基因组7号染色体上SweepC基因组区域内的SNP标记。本文使用的SweepC基因组区域是指位于Chr7:63714553(NCBIbuildSscrofa10.2版本)处的核苷酸位点。在某些实施方案中,本文提供的SweepC区域内的遗传标记是SNPC1,其位置总结于表1中。为了更好地示意所述位置,表1还示出了每个SNP位点紧邻的20bp序列,使得本领域人员还能够通过序列同源性检索在给定的猪基因组上找到这些SNP位点。在某些实施方案中,本文提供的遗传标记是猪基因组15号染色体上SweepD基因组区域内的SNP标记。本文使用的SweepD基因组区域是指跨越自Chr15:51799437至Chr15:51800356的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,本文提供的SweepD区域内的遗传标记是SNPD1-D3,其位置总结于表1中。为了更好地示意所述位置,表1还示出了每个SNP位点紧邻的20bp序列,使得本领域人员还能够通过序列同源性检索在给定的猪基因组上找到这些SNP位点。在某些实施方案中,本文提供的遗传标记是猪基因组3号染色体上SweepE基因组区域内的SNP标记。本文使用的SweepE基因组区域是指跨越自Chr3:72655441至Chr3:72795872的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,本文提供的SweepE区域内的遗传标记是SNPE1-E23,其位置总结于表1中。为了更好地示意所述位置,表1还示出了每个SNP位点紧邻的20bp序列,使得本领域人员还能够通过序列同源性检索在给定的猪基因组上找到这些SNP位点。对于SNP位点A1-A13、B1-B91、C1、D1-D3、E1-E23,在二花脸猪基因组中发现的高频SNP标记(即E等位基因,代表二花脸等位基因,示于表1中)不同于发现于非二花脸猪基因组中的高频SNP标记(即O等位基因,代表其他猪等位基因,示于表1中)。还已经表明,带有E等位基因SNP标记的猪与高产仔数表型相关联,而带有O等位基因SNP标记的猪与相对较低的产仔数相关联。通过检测E等位基因SNP标记的存在或缺失,可以鉴定具有高产仔数表型的猪。SNP标记可以连锁不平衡。本文使用的术语“连锁不平衡”是指与一个以上的SNP标记非随机关联。例如,对于给定的群体,如果SNP位点X上的SNP标记“A”以50%的频率存在,并且SNP位点Y上的SNP标记“T”以50%的频率存在,那么预测在一个个体中存在SNP位点X上的SNP标记“A”并且SNP位点Y上的SNP标记“T”的发生频率为25%。然而,如果发现所述两个SNP标记以显著高于25%的比例共同存在,那么所述两个SNP标记都趋向于以比基于其独立的发生频率而预测的比例更高的比例共同传递。因此,处于连锁不平衡中的SNP标记倾向于以组合的方式(例如,类似其通过连锁而关联)被继承或者遗传传递给后代。例如,如果SNP标记中的一个被传递给后代,那么有可能与该标记连锁不平衡的其他SNP标记也由此后代遗传。换句话说,对于处于连锁不平衡中的SNP标记,这些标记中任何一个的存在将指示其他标记存在的可能性。因此,如果SNP位点处存在SNP标记指示所需的表型(例如,增加的产仔数),那么在处于连锁不平衡中的SNP位点处存在任意其他SNP遗传标记将有可能指示相同的表型。在某些实施方案中,表1中归组于相同的Sweep内的SNP标记都处于连锁不平衡中。在某些实施方案中,遗传标记是SNPA2。在某些实施方案中,遗传标记是表1中任何与SNPA2连锁不平衡的标记,或者是与SNPA2强连锁不平衡的标记。在某些实施方案中,遗传标记是SNPB13。在某些实施方案中,遗传标记是表1中任何与SNPB13连锁不平衡的标记,或者是与SNPB13强连锁不平衡的标记。在某些实施方案中,遗传标记是SNPC1。在某些实施方案中,遗传标记是SNPD1。在某些实施方案中,遗传标记是表1中任何与SNPD1连锁不平衡的标记,或者是与SNPD1强连锁不平衡的标记。在某些实施方案中,遗传标记是SNPE10。在某些实施方案中,遗传标记是表1中任何与SNPE10连锁不平衡的标记,或者是与SNPE10强连锁不平衡的标记。可以通过本领域已知的合适的参数,例如,D值和r2(详述参见HartlDL,ClarkAGPrinciplesofPopulationGenetics,第四版.SinauerAssociates,Inc)评估连锁不平衡性。D值和r2都代表两个位点之间的统计相关性,并且评估所述两个位点之间的关联强度。在某些实施方案中,r2值不低于0.8指示两个位点之间有很强的关联(并且因此具有强连锁不平衡性)。在某些实施方案中,与SNPA2强连锁不平衡的SNP标记包括SNPA1、SNPA3、SNPA4、SNPA5、SNPA6、SNPA7、SNPA8、SNPA9、SNPA10、SNPA11、SNPA12和SNPA13。在某些实施方案中,与SNPB13强连锁不平衡的SNP标记包括SNPB1、SNPB2、SNPB3、SNPB4、SNPB5、SNPB6、SNPB7、SNPB8、SNPB9、SNPB10、SNPB11、SNPB12、SNPB14、SNPB15、SNPB16、SNPB17、SNPB18、SNPB19、SNPB20、SNPB21、SNPB22、SNPB23、SNPB24、SNPB25、SNPB26、SNPB27、SNPB28、SNPB29、SNPB30、SNPB31、SNPB32、SNPB33、SNPB34、SNPB35、SNPB36、SNPB37、SNPB38、SNPB39、SNPB40、SNPB41、SNPB42、SNPB43、SNPB44、SNPB45、SNPB46、SNPB47、SNPB48、SNPB49、SNPB50、SNPB51、SNPB52、SNPB53、SNPB54、SNPB55、SNPB56、SNPB57、SNPB58、SNPB59、SNPB60、SNPB61、SNPB62、SNPB63、SNPB64、SNPB65、SNPB66、SNPB67、SNPB68、SNPB69、SNPB70、SNPB71、SNPB72、SNPB73、SNPB74、SNPB75、SNPB76、SNPB77、SNPB78、SNPB79、SNPB80、SNPB81、SNPB82、SNPB83、SNPB84、SNPB85、SNPB86、SNPB87、SNPB88、SNPB89、SNPB90和SNPB91。在某些实施方案中,与SNPD1强连锁不平衡的SNP标记包括SNPD2和SNPD3。在某些实施方案中,与SNPE10强连锁不平衡的SNP标记包括SNPE1、SNPE2、SNPE3、SNPE4、SNPE5、SNPE6、SNPE7、SNPE8、SNPE9、SNPE11、SNPE12、SNPE13、SNPE14、SNPE15、SNPE16、SNPE17、SNPE18、SNPE19、SNPE20、SNPE21、SNPE22和SNPE23。猪是二倍体生物,并且其体细胞中具有两个拷贝的染色体。对于染色体上给定的SNP位点,可能在染色体两个拷贝的SNP位点处存在E等位基因SNP标记(即,在SNP位点处具有E等位基因SNP标记的双拷贝),这指示E/E的纯合基因型;或者,还可能仅在染色体的两个拷贝中的一个上存在E等位基因SNP标记(即,在SNP位点处具有E等位基因SNP标记的单拷贝),并且在染色体的另一个拷贝上存在O等位基因SNP标记,这指示E/O的杂合基因型;还可能在染色体的两个拷贝上都缺失E等位基因SNP标记(即,在SNP位点处不具有E等位基因SNP标记的拷贝),并且在两个拷贝上都存在O等位基因SNP标记,这指示O/O的纯合基因型。在某些实施方案中,在表1中SweepA所示的SNP位点中的一个位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。在某些实施方案中,在表1中SweepB所示的SNP位点中的一个位点处存在E/E基因型指示了增加的产仔数的表型。在某些实施方案中,在表1中SweepC所示的SNP位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。在某些实施方案中,在表1中SweepD所示的SNP位点中的一个位点处存在E/O基因型指示了增加的产仔数的表型。在某些实施方案中,在表1中SweepE所示的SNP位点中的一个位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。对于仅含有一个拷贝的染色体的生殖细胞,在SNP位点处存在E等位基因SNP标记指示了增加的产仔数的表型。用于鉴定具有增加的产仔数的猪的方法本发明的另一方面涉及鉴定猪能生产增加的产仔数的方法。在某些实施方案中,所述方法包括检测基因组区域内的SNP位点中的一个位点处E等位基因SNP标记的存在或缺失,所述基因组区域选自SweepA、SweepB、SweepC、SweepD和SweepE,并且所述E等位基因SNP标记的存在指示了猪的增加的产仔数。在某些实施方案中,SweepA基因组区域跨越自Chr6:122097788至Chr6:122217096的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepA基因组区域内的至少一个SNP位点选自表1中所列的SNP:A1-A13。在某些实施方案中,SweepA基因组区域内的至少一个SNP位点包括SNPA2,或者与SNPA2连锁不平衡。在某些实施方案中,在SweepA的SNP位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。在某些实施方案中,SweepB基因组区域跨越自Chr6:89403626至Chr6:90311616的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepB基因组区域内的至少一个SNP位点选自表1中所列的SNP:B1-B91。在某些实施方案中,SweepB基因组区域内的至少一个SNP位点包括SNPB13,或者与SNPB13连锁不平衡。在某些实施方案中,在SweepB的SNP位点处存在E/E基因型指示了增加的产仔数的表型。在某些实施方案中,SweepC基因组区域位于Chr7:63714553(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepC基因组区域内的SNP位点是表1中所列的SNPC1。在某些实施方案中,在SweepC的SNP位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。在某些实施方案中,SweepD基因组区域跨越自Chr15:51799437至Chr15:51800356的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepD基因组区域内的至少一个SNP位点选自表1中所列的SNP:D1-D3。在某些实施方案中,SweepD基因组区域内的至少一个SNP位点包括SNPD1,或者与SNPD1连锁不平衡。在某些实施方案中,在SweepD的SNP位点处存在E/O基因型指示了增加的产仔数的表型。在某些实施方案中,SweepE基因组区域跨越自Chr3:72655441至Chr3:72795872的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepE基因组区域内的至少一个SNP位点选自表1中所列的SNP:E1-E23。在某些实施方案中,SweepE基因组区域内的至少一个SNP位点包括SNPE10,或者与SNPE10连锁不平衡。在某些实施方案中,在SweepE的SNP位点处存在E/E基因型和/或E/O基因型指示了增加的产仔数的表型。在某些实施方案中,E等位基因SNP标记是在二花脸猪基因组相应的SNP位点处的高频SNP标记。在某些实施方案中,E等位基因SNP标记是指在相应的SNP位点处的表1中“E”列下所示的核苷酸。在某些实施方案中,O等位基因SNP标记是指在相应的SNP位点处的表1中“O”列下所示的核苷酸。可以使用本文提供的方法测试任意猪,包括但不限于,公猪、母猪和仔猪。所述猪可以是任何合适的品种,例如,但不限于,阿克塞黑斑猪、美国长白猪、美国约克郡猪、昂格尔恩白肩猪、阿巴拉契亚英国猪、阿拉帕瓦岛猪、奥克兰群岛猪、澳大利亚约克郡猪、巴比甘榜猪、巴旭银猪、班图猪、班图猪、巴斯克猪、巴兹纳猪、北京黑猪、白俄罗斯黑斑猪、比利时长白猪、孟加拉棕Shannaj(BengaliBrownShannaj)猪、本特海姆黑斑猪、巴克夏猪、比萨罗(Bisaro)猪、黑斯拉夫尼亚(BlackSlavonian)猪、黑色加那利猪、Breitovo猪、英国长白猪、英国罗布泊猪、英国白肩猪、保加利亚白猪、广东(Cantonese)猪、查托穆尔西亚猪、切斯特白猪、乔克托猪、克里奥尔猪、克里奥尔猪、坎伯兰猪、捷克改良白猪、丹麦长白猪、丹麦Protest猪、Dermantsi花衣猪、杜洛克猪、荷兰长白猪、东巴尔干猪、埃塞克斯猪、爱沙尼亚培根猪、枫泾猪、芬兰长白猪、林山猪、法国长白猪、加斯科涅(Gascon)猪、德国长白猪、格洛斯特郡花猪、格莱斯(Grice)猪、几内亚猪、汉普夏猪、汉特猪、海福特猪、Hezuo猪、伊比利亚猪、意大利长白猪、日本长白猪、济州黑猪、金华猪、卡克黑提安猪、可乐猪、克麦罗沃猪、韩国本土猪、Krskopolje猪、酷你酷你猪、南江(Lamcombe)猪、大黑猪、大黑白猪、大白猪、拉脱维亚白猪、Leicoma猪、立陶宛本土猪、立陶宛白猪、林肯郡卷毛猪、利夫内猪、Malhadode猪、曼加利察猪、梅山猪、中白猪、民猪、中白猪、民猪、MinokawaButa猪、蒙开猪、MoraRomagnola猪、莫拉猪、Mukota猪、缪尔福特(Mulefoot)猪、穆罗姆(Murom)猪、米尔霍罗德(Myrhorod)猪、内江猪、宁乡猪、北高加索猪、北西伯利亚猪、挪威长白猪、挪威约克郡猪、Ossabaw岛猪、牛津桑迪黑猪、菲律宾本土猪、Piétrain猪、波中猪、红垂(RedWattle)猪、Semirechye猪、西伯利亚黑斑猪、小黑猪、小白猪、斑点猪、SurabayaBabi猪、施韦比施哈尔猪、瑞典长白猪、太湖猪、太姆华斯猪、ThuocNhieu猪、藏猪、东京-X猪、齐维利斯克(Tsivilsk)猪、Turopolje猪、乌斑草原猪、乌白草原猪、乌尔茹姆(Urzhum)猪、越南大肚猪、威尔士猪、威赛克斯白肩猪、西法白猪、Windsnyer猪、五指山猪、雅南猪、约克郡蓝白猪。在某些实施方案中,所述猪是二花脸猪、太湖猪、苏太猪、梅山猪、枫泾猪、嘉兴黑猪。在某些实施方案中,所述猪是太湖猪的后代。例如,所述猪可以是太湖猪与以上列出的任意其他猪种杂交的后代,或者是纯种太湖猪。在某些实施方案中,所述猪是二花脸猪的后代,例如,二花脸猪与以上列出的任意其他猪种杂交的后代,或者是纯种二花脸猪。可以从猪中得到含有携带待检测的SNP位点的核酸分子的样本。所述样本可以是组织样本、体细胞、生殖细胞(例如,精子细胞和卵细胞)、细胞提取物、细胞核提取物、基因组DNA样本、或者所述基因组DNA样本的片段。任选地,可以例如通过细胞裂解、蛋白酶消化、离心和DNA纯化等处理所述样本以释放核酸。这样的技术在本领域中是公知的,并且可以由本领域技术人员视情况采用。在某些实施方案中,所述样本含有猪的基因组DNA,或者至少含有SNP位点的所述基因组DNA的片段。所述样本可以用于检测本文公开的SNP标记的存在或缺失。可以使用任意合适的方法来进行本文所述的检测。可以使用本领域已知的方法检测所述样本,包括例如,测序法、基于PCR的方法、基于杂交的方法、基于限制性酶切的方法、基于凝胶电泳的方法和结合亲和力法。在某些实施方案中,所述检测包括在来自猪的核酸样本中测序至少含有SNP位点的片段。例如,可以从猪中分离核酸并且至少测序含有SNP位点的部分。可以使用本领域中已知的任意合适的测序法。在某些实施方案中,所述检测包括检测扩增产物,所述扩增产物是来自猪的核酸样本中至少含有SNP位点的片段的扩增产物。可以使用本领域已知的方法,例如,通过在存在与包含SNP位点的部分紧邻结合的引物对的条件下进行PCR反应,扩增核酸样本。简要地说,可以将来自猪的核酸样本用作模板,然后在存在合适的聚合酶、缓冲液和温度循环的条件下对其进行扩增,其中每个循环包括模板变性步骤、引物退火步骤和引物延伸步骤等。引物紧邻结合的部分可以呈指数扩增。可以使用,例如,凝胶电泳来检测扩增产物的存在或缺失以及大小(或者分子量)。在某些实施方案中,通过PCR扩增产物的存在(或者缺失)来揭示E等位基因SNP标记的存在或者缺失。任选地,可以进一步测序扩增产物以揭示SNP位点处的核苷酸的特性,以便可以通过SNP位点处的特定序列来鉴定E等位基因SNP标记的存在或者缺失。在某些实施方案中,所述检测包括检测探针和来自猪的核酸样本中至少含有SNP位点的片段的杂交。本文使用的“杂交”是指在引物与模板DNA之间通过碱基配对形成氢键的过程,例如,A和T对之间,或者G和C对之间。探针可以是寡核苷酸分子,其包含天然核苷酸或者非天然核苷酸。可以任选地用可检测部分例如放射性配体、荧光分子等标记探针。可以使样本接触探针并且发生杂交。可以用本领域已知的方法检测杂交,例如,通过用双链DNA染料检测双链杂交产物的存在或者缺失,或者通过检测与固定在芯片上的靶序列结合的已标记的探针,或者通过检测杂交产物的熔解温度(Tm),即所述杂交产物有一半解离成单链时的温度,或者通过检测作为杂交结果而产生的荧光信号。在某些实施方案中,设计探针来识别SNP位点处不同的SNP标记,例如,特异性地杂交E等位基因SNP标记而非其他标记,反之亦然,或者作为替代,以与其他标记的Tm可检测地不同的Tm值杂交E等位基因SNP标记。通过检测探针与样本中的核酸的杂交,本领域人员可以知道在目标SNP位点处E等位基因SNP标记的存在或缺失。在某些实施方案中,所述检测包括检测来自猪的核酸样本中至少含有SNP位点的片段的引物延伸产物。可以用DNA聚合酶和双脱氧核苷酸(ddNTP)执行引物的延伸,其可被添加到引物的3’端,但是由于其不具有3’-羟基从而阻止了进一步的延伸。为了检测SNP标记,可以将引物设计成杂交SNP位点紧邻的上游,使得在引物上延伸的下个核苷酸与模板上的SNP标记互补。对于这种新掺入的核苷酸,可以分析其质量(例如,通过MALDI-TOF质谱分析),或者通过其独特的荧光标记进行分析。或者,如果存在选定的SNP标记,可以将引物的3’端设计成仅杂交所述选定的SNP标记(例如,E等位基因SNP标记),那么引物的3’端将与模板互补并允许引物延伸以掺入标记的核苷酸,但是在不存在所选择的SNP标记的情况下,引物的3’端将错配并且因此将允许引物延伸。在某些实施方案中,所述检测包括检测来自猪的核酸样本中至少含有SNP位点的片段的限制酶切产物。在某些SNP位点处,SNP标记连同其相邻核苷酸一起形成限制性位点,所述限制性位点可被限制性酶识别和剪切。因此,通过用这种限制性酶处理核酸样本,在核酸样本上的限制性位点(包括含有SNP位点的限制性位点),将被切割以得到具有预期的尺寸模式的片段。然而,如果SNP位点具有不同的不形成这种限制性位点的SNP标记,那么限制性酶对核酸样本的消化将导致至少一个片段具有大于预期的尺寸。在某些实施方案中,所述检测包括检测来自猪的核酸样本中至少含有SNP位点的片段的凝胶电泳结果。在某些实施方案中,所述检测基于温度梯度凝胶电泳。简单地说,将核酸样本和对照核酸的混合物进行变性和退火。所述核酸样本和对照核酸除了所述样本在SNP位点处含有未知SNP标记而所述对照在SNP位点处含有已知的SNP标记(例如E等位基因SNP标)外,其余都是相同的。如果在样本中的SNP标记与对照中的SNP标记相同,那么会有一个退火产物,其在电泳中显示一个条带。否则,将有四个不同的退火产物(对照产物、样本产物、对照的正链和样本的负链形成的杂化物,以及对照的负链和样本的正链形成的杂化物),其在电泳中显示可被检测的不同条带。以这种方式,可以相应地检测到目标SNP标记(例如等位基因SNP标记)的存在或者缺失。在某些实施方案中,所述检测包括检测蛋白与来自猪的核酸样本中至少含有SNP位点的片段的结合亲和力。已经发现某些DNA结合蛋白(来自水生栖热菌的MutS蛋白)差异性结合具有不同序列的DNA分子,并且由此可被用来区分SNP标记。在某些实施方案中,在一个测定法中可以检测一个以上的SNP位点。例如,可以使用微阵列以允许在同一时间检测多个SNP标记,其中针对多个SNP位点的多个探针或者引物被固定在预定的位置。通过检测每个预定的位置上的信号,在一个测定法中可以确定在多个SNP位点处的SNP标记。在某些实施方案中,在所述方法中测试来自猪对象的生殖细胞。例如,从雄性猪收集精液样本,并且从所述样本中得到基因组DNA用于进一步的分析和检测。如果检测到E等位基因SNP标记存在于表1中所列的SNP位点中的至少一个上,那么可以鉴定源自该精液样本的猪生产增加的产仔数。可以选择该被鉴定的雄性猪用于进一步的繁殖,并且还可以收集来源于该被鉴定的雄性猪的精液样本用于进一步的繁殖,例如,通过人工受精或者体外受精进行繁殖。同样地,也可以从雌性猪收集卵子样本,并且获得并测试基因组DNA。如果检测到E等位基因SNP标记存在于表1中所列的SNP位点中的至少一个上,那么可以鉴定源自该卵子样本的猪生产增加的产仔数。可以选择该被鉴定的雌性猪用于进一步的繁殖,并且还可以收集来源于该雌性猪的卵子样本用于进一步的繁殖,例如通过人工受精或者体外受精进行繁殖。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处检测E等位基因SNP标记的存在或缺失。来源于体细胞的样本可以用于这种检测。可以同时检测SNP位点在两个拷贝的染色体上的存在或缺失。例如,当两个拷贝的染色体都含有E等位基因SNP标记(即E/E基因型),当仅有一个拷贝的染色体含有E等位基因SNP标记(如E/O基因型),或者当两个拷贝都不含有E等位基因SNP标记(如O/O基因型)时,能区别出检测信号是不同的。在某些实施方案中,E等位基因SNP标记的存在导致产生可检测的信号,对于O/O基因型检测不到所述信号,但是对于E/O基因型和E/E基因型能够检测到所述信号,并且E/E基因型具有较高量级的信号。在某些实施方案中,E等位基因SNP标记的存在导致产生X信号(例如红色),O等位基因SNP标记的存在导致产生Y信号(例如绿色),并且E/O基因型会导致产生X和Y的混合信号(例如红色和绿色,其在测定法中会叠加显示成黄色)。在某些实施方案中,对于SweepA的SNP标记,在SNP位点处存在E等位基因SNP标记的单拷贝或者双拷贝指示猪的增加的产仔数。在SweepA的SNP位点处,E/O和E/E基因型都与增加的产仔数的表型相关。在某些实施方案中,对于SweepB的SNP标记,在SNP位点处存在E等位基因SNP标记的双拷贝指示猪的增加的产仔数。E/E基因型与增加的产仔数的表型相关。在某些实施方案中,对于SweepC的SNP标记,在SNP位点处存在E等位基因SNP标记的单拷贝或者双拷贝指示猪的增加的产仔数。E/O和E/E基因型都与增加的产仔数的表型相关。在某些实施方案中,对于SweepD的SNP标记,在SNP位点处存在E等位基因SNP标记的单拷贝指示猪的增加的产仔数。E/O基因型与增加的产仔数的表型相关。在某些实施方案中,对于SweepE的SNP标记,在SNP位点处存在E等位基因SNP标记的单拷贝或者双拷贝指示猪的增加的产仔数。E/O和E/E基因型都与增加的产仔数的表型相关。在某些实施方案中,所述方法进一步包括在猪被鉴定为生产增加的产仔数时选择该猪用于繁殖。所选择的猪可以是公猪或者母猪。在某些实施方案中,相较于在相应SNP位点不具有E等位基因拷贝数的猪,在相应的SNP位点处具有双拷贝数或者单拷贝数E等位基因标记的猪每窝至少多生产0.2、0.3、0.4.0.5、0.6、0.7、0.8、0.9、1.0、1.1、1.2、1.3、1.4、1.5、1.6、1.7、1.8、1.9、2.0、2.1、2.2、2.3、2.4、2.5或者更多的猪。在某些实施方案中,相较于在相应SNP位点不具有E等位基因拷贝数的猪,在相应的SNP位点处具有双拷贝数或者单拷贝数E等位基因标记的猪每窝至少多生产2%、3%、4%、5%、6%、7%、8%、9%、10%、11%、12%、13%、14%、15%、16%、17%、18%、19%、20%、21%、22%、23%、24%或者25%的猪。检测SweepA区域内SNP位点处的SNP标记的方法另一方面,本发明提供了检测猪SweepA区域内SNP位点处的SNP标记的方法。所述方法包括:在猪基因组的SweepA基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或者缺失。在某些实施方案中,所述SweepA基因组区域跨越自Chr6:122097788至Chr6:122217096的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:A1-A13。在某些实施方案中,SweepA基因组区域内的所述至少一个SNP位点包括SNPA2,或者与SNPA2连锁不平衡。E等位基因SNP标记是在二花脸猪基因组相应的SNP位点处的高频SNP标记,并且E等位基因SNP标记如表1的“E”列所示。在SNP位点处存在E等位基因SNP标记指示猪的增加的产仔数。在某些实施方案中,在SNP位点处缺失E等位基因SNP标记(即E等位基因SNP标记没有拷贝数)可能指示缺失增加的产仔数的表型。在某些实施方案中,认为被鉴定为在一个拷贝或者两个拷贝的染色体的至少一个SNP位点处具有E等位基因SNP标记(即在SNP位点处具有单拷贝或者双拷贝的E等位基因SNP标记)的猪能生产增加的产仔数,以及任选地,被选择用于繁殖。O等位基因SNP标记是在非二花脸猪基因组相应的SNP位点处的高频SNP标记,并且O等位基因SNP标记如表1的“O”列所示。在某些实施方案中,在SNP位点处,例如在两个拷贝的染色体上存在O等位基因SNP标记(即在SNP位点处的双拷贝O等位基因SNP标记),指示缺失增加的产仔数的表型。在某些实施方案中,认为被鉴定为在两个拷贝的染色体的SNP位点处具有O等位基因SNP标记(即在SNP位点处的双拷贝O等位基因SNP标记)的猪不能生产增加的产仔数,以及任选地,不用于繁殖。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失,其中所述SNP位点选自表1中所示的SNPA1-A13。在两个拷贝的染色体上存在E等位基因SNP标记(即E/E基因型),或者在染色体的其中一个拷贝上存在E等位基因SNP标记(即E/O基因型),指示增加的产仔数的表型。在两个拷贝的染色体上都存在O等位基因SNP标记(即O/O基因型)指示缺失增加的产仔数的表型。检测SweepB区域内SNP位点处的SNP标记的方法另一方面,本发明提供了检测猪SweepB区域内SNP位点处的SNP标记的方法。所述方法包括:在猪基因组的SweepB基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或者缺失。在某些实施方案中,所述SweepB基因组区域跨越自Chr6:89403626至Chr6:90311616的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:B1-B91。在某些实施方案中,SweepB基因组区域内的所述至少一个SNP位点包括SNPB13,或者与SNPB13连锁不平衡。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失,其中所述SNP位点选自表1中所示的SNPB1-B91。在两个拷贝的染色体上都存在E等位基因SNP标记(即E/E基因型)指示增加的产仔数的表型。在两个拷贝的染色体上都存在O等位基因SNP标记(即O/O基因型),或者在单拷贝的染色体上存在O等位基因SNP标记(即E/O基因型),指示缺失增加的产仔数的表型。检测SweepC区域内SNP位点处的SNP标记的方法另一方面,本发明提供了检测猪SweepC区域内SNP位点处的SNP标记的方法。所述方法包括:在猪基因组的SweepC基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或者缺失。在某些实施方案中,所述SweepC基因组区域位于Chr7:63714553(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepC基因组区域内的SNP位点选自表1中所列的SNP:C1。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失,其中所述SNP位点为如表1中所示的SNPC1。在两个拷贝的染色体上都存在E等位基因SNP标记(即E/E基因型),或者在染色体的其中一个拷贝上存在E等位基因SNP标记(即E/O基因型),指示增加的产仔数的表型。在两个拷贝的染色体上都存在O等位基因SNP标记(即O/O基因型)指示缺失增加的产仔数的表型。检测SweepD区域内SNP位点处的SNP标记的方法另一方面,本发明提供了检测猪SweepD区域内SNP位点处的SNP标记的方法。所述方法包括:在猪基因组的SweepD基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或者缺失。在某些实施方案中,所述SweepD基因组区域跨越自Chr15:51799437至Chr15:51800356的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:D1-D3。在某些实施方案中,SweepD基因组区域内的所述至少一个SNP位点包括SNPD1,或者与SNPD1连锁不平衡。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失,其中所述SNP位点选自表1中所示的SNPD1-D3。在染色体的单拷贝上存在E等位基因SNP标记(即E/O基因型)指示增加的产仔数的表型。在两个拷贝的染色体上都存在O等位基因SNP标记(即O/O基因型),或者在所述染色体上都不存在O等位基因SNP标记(即E/E基因型),指示缺失增加的产仔数的表型。检测SweepE区域内SNP位点处的SNP标记的方法另一方面,本发明提供了检测猪SweepE区域内SNP位点处的SNP标记的方法。所述方法包括:在猪基因组的SweepE基因组区域内至少一个单核苷酸等位基因(SNP)位点处,确定E等位基因SNP标记或者O等位基因SNP标记的存在或者缺失。在某些实施方案中,所述SweepE基因组区域跨越自Chr3:72655441至Chr3:72795872的区域(NCBIbuildSscrofa10.2版本)。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点选自表1中所列的SNP:E1-E23。在某些实施方案中,SweepE基因组区域内的所述至少一个SNP位点包括SNPE10,或者与SNPE10连锁不平衡。在某些实施方案中,所述方法进一步包括在两个拷贝的染色体的SNP位点处确定E等位基因SNP标记或者O等位基因SNP标记的存在或缺失,其中所述SNP位点选自表1中所示的SNPE1-E23。在两个拷贝的染色体上都存在E等位基因SNP标记(即E/E基因型),或者在染色体的其中一个拷贝上存在E等位基因SNP标记(即E/O基因型),指示增加的产仔数的表型。上述任意方法都能用于所述检测。在两个拷贝的染色体上都存在O等位基因SNP标记(即O/O基因型)指示缺失增加的产仔数的表型。可以直接地(例如通过测序)或者间接地(例如,通过从已知的对照序列进行推断)确定SNP位点处的核苷酸。可以同时检测多个SNP标记。引物另一方面,本发明提供了分离的寡核苷酸引物,其能用于区别携带E等位基因SNP标记和不携带E等位基因SNP标记的核酸样本。在某些实施方案中,可以将引物设计成无论E等位基因SNP标记存在与否,都能扩增靶序列。以此,可以进一步测序扩增产物来确定E等位基因SNP标记在SNP位点处的存在或缺失。可以将这种引物设计成在SNP位点的紧邻位置,并且离所述SNP位点至少有几个碱基的距离。为了提供想要的PCR产物,扩增子可以具有合适的大小,例如至少100bp、至少200bp、至少500bp等。在某些实施方案中,引物选择性地扩增在表1中所列的SNP位点中的至少一个处含有E等位基因SNP标记的多核苷酸片段。在这种情况下,E等位基因SNP标记的存在导致存在能被检测或者进一步测序的扩增产物,而样本中缺失E等位基因SNP标记会导致缺失扩增产物。可以将这种引物设计成在具有正好在SNP位点处杂交的3’端,并且所述3’端与E等位基因SNP标记互补。因此,在SNP位点处存在E等位基因的SNP标记会导致引物的延伸和扩增子的产生,而缺失E等位基因SNP标记将导致在引物的3’端发生错配,其会阻止扩增子的形成。在某些实施方案中,引物选择性地扩增在表1中所列的SNP位点中的至少一个处缺失E等位基因SNP标记的多核苷酸片段。在某些实施方案中,引物选择性地扩增在表1中所列的SNP位点中的至少一个处含有O等位基因SNP标记的多核苷酸片段。在这种情况下,E等位基因SNP标记的存在导致扩增产物的缺失而缺失E等位基因SNP标记(或者,存在O等位基因SNP标记)导致扩增产物的存在。同样地,可以将引物设计成具有正好在SNP位点处杂交并且与O等位基因SNP标记互补的3’端。因此,O等位基因的存在会导致扩增而E等位基因的存在则不会导致扩增。在某些实施方案中,将引物设计成具有与SNP位点的紧邻上游杂交的3’端。这种引物能用于引物延伸方法,其中所述引物的3’端仅掺入了一个核苷酸,并且所述核苷酸与SNP位点处的核苷酸互补(即SNP标记)。探针另一方面,本发明提供了分离的寡核苷酸探针,其基于与样本的杂交能用于区分携带E等位基因SNP标记的核酸样本和不携带E等位基因SNP标记的核酸样本。在某些实施方案中,将寡核苷酸探针设计成选择性地杂交在表1中所列的一个或多个SNP位点处含有E等位基因SNP标记的多核苷酸片段。例如,所述探针以与其结合O等位基因SNP标记的Tm值不同的Tm值结合E等位基因SNP位点。如本领域中公知的,在短寡核苷酸探针中的一个或多个错配会降低由此形成的DNA双螺旋体的Tm值。因此,如果寡核苷酸探针在SNP位点处杂交靶序列,那么Tm值会根据存在于SNP位点处的特定的SNP标记而变化,从而指示SNP标记的鉴定。可以使用本领域已知的方法来确定不同的Tm值。在某些实施方案中,可以将探针设计成在彼此分离的端部具有缀合的荧光团和猝灭剂,使得在完整的探针中,荧光团被淬灭剂淬灭。这种探针可以用于TaqMan测定法,其中在存在所述探针的条件下使用TaqDNA聚合酶通过PCR扩增样本。如果样本含有E等位基因SNP标记,那么探针将与样本杂交并且由此在TaqDNA聚合酶延伸DNA链时被其降解。这将导致探针中的荧光团和猝灭剂分离,从而发出指示E等位基因SNP标记存在的荧光。同样地,也可以设计探针来以同样的方式检测O等位基因SNP标记的存在。试剂盒另一方面,本发明提供了用于本文所提供的方法的试剂盒。试剂盒包括本文所提供的分离的寡核苷酸引物,或者本文所提供的分离的寡核苷酸探针。试剂盒可以进一步包括缓冲液、酶或者检测标签。试剂盒可以进一步包括使用说明。实施例实施例1全基因组重测序在得到了本地猪基因组(杜洛克猪)后,我们对东亚野猪和五个中国本土猪种(包括二花脸猪、莱芜猪、巴马香猪、滇南小耳猪和藏猪)进行了种群全基因组测序。所选择的猪种代表两组具有不同的产仔数能力的种群。将由中国太湖当地猪的一个品种组成的二花脸猪选作具有较大的产仔数的品种,并且与其他四个中国地方猪种和野猪进行比较。对于二花脸猪、藏猪、巴马香猪、莱芜猪和滇南小耳猪,分别以种群大小11、11、9、10和10对猪群进行测序,对于野猪,以种群大小10对猪群进行测序。这样做的目的是鉴定二花脸猪基因组中重要的单核苷酸多态性(SNP),其能潜在地用于标记辅助性选择(MAS)来改善猪的产仔数性状。二花脸猪基因组中假定的选择性清除东亚本地猪与当地的野猪拥有共同的起源[5,6]。因此,二花脸猪与莱芜猪、巴马香猪、滇南小耳猪和藏猪拥有共同的祖先(东亚野猪)。二花脸猪生产较高产仔数的能力是历史上人类驱动的在驯养繁殖过程中为了追求更多经济效益而进行的筛选的结果。检测人类驱动的筛选在二花脸基因组上的印迹将有助于鉴定潜在造成增加的产仔数的遗传变异。将基因组读取结果与对照猪基因组(NCBIbuildSscrofa10.2版本)进行比对。使用SAMtools软件包调出基因组变异体。具有强烈需要的人类会选择二花脸猪中导致增加的产仔数的突变。在这种筛选力的推动下,在很少的几代内,二花脸猪中的因果突变(causativemutation)会达到非常高的频率。在种群基因组中,这种人类驱动的筛选的印迹是跨越这种有利突变的选择性清除(selectivesweep)。选择性清除(selectivesweep)是指在强的正向自然选择或者人工筛选条件下,含有有益突变的DNA序列变异的减少或者消除。选择性清除(selectivesweep)是由含有有利突变的基因组区域中缺乏重组造成的。鉴于在侧翼SNP和有利的突变位点之间缺乏重组,作为对有利突变选择的副产物,对于位于有利突变中心的侧翼SNP,来自每个侧翼SNP位点的某个等位基因也将增加其在群体中的等位基因频率。其结果是,对于选择性清除(selectivesweep)下的基因组区域,预期在目的群体中具有降低的基因多样性,以及在所述群体与其他群体之间具有升高的分化水平。从基因组的分化中,通过实施马尔可夫链蒙特卡尔理论(BayesianMarkovChainMonteCarlo),在二花脸猪和其他猪之间比较衍生等位基因频率的后验分布,我们开发了统计方法来在二花脸猪基因组中检测近期选择性清除(selectivesweep)。在二花脸猪基因组的18个常染色体上集中筛选选择性清除(selectivesweep)。鉴定了五个假定的选择性清除(selectivesweep)。第一个假定的选择性清除(selectivesweep)(SweepA)位于猪6号染色体上,跨越122097788-122217096。在所述清除中一共鉴定出了13个SNP位点。第二个假定的选择性清除(selectivesweep)(SweepB)位于猪6号染色体上,跨越89403626-90311616。在所述清除中一共鉴定出了91个SNP位点。第三个假定的选择性清除(selectivesweep)(SweepC)位于猪7号染色体上,位于Chr7:63714553处。在所述清除中鉴定出了一个SNP位点。第四个假定的选择性清除(selectivesweep)(SweepD)位于猪15号染色体上,跨越51799437-51800356。在所述清除中一共鉴定出了3个SNP位点。第五个假定的选择性清除(selectivesweep)(SweepE)位于猪3号染色体上,跨越72655441-72795872。在所述清除中一共鉴定出了23个SNP位点。清除中SNP的共传递由于重组的稀缺性,在选择性清除中一直同时选择侧翼“搭便车”(“hitchhiked”)SNP和因果(“causative”)SNP位点处的有利突变。因此,侧翼“搭便车”SNP总是和有利突变一起共同从亲代传递给后代。因此,除了有利突变本身,侧翼“搭便车”SNP还能用于在标记辅助性种选择中改善产仔数性状。实际上,没有必要区分侧翼“搭便车”SNP和因果SNP。侧翼“搭便车”SNP和因果SNP之间的强关联性将在定量产仔数调控方面的效应时呈现出类似的效能。作为对选择性清除中有条理的所有SNP的代表,无论其是因果的还是“搭便车的”,都可以随机选择一个标签SNP来验证在产仔数调控方面的效应。如果标签SNP可用作改善产仔数性状方面的有效的分子标记,标签SNP和其他SNP之间的关联是其他SNP在促进标记辅助性选择方面具有效能的重要指标。紧密关联的SNP可以用于标记辅助性选择,并且具有与标签SNP同样高的可信度。可以用连锁不平衡(LD)度测量标签SNP和其他SNP之间的关联,所述连锁不平衡度反应存在来自两个SNP等位基因的一些组合,其以比从基于它们的频率而预测的从等位基因随机形成的概率更常出现在群体中。强LD指示来自两个SNP等位基因的组合总是一起传递给后代。在总群体中,考虑SNPX具有两个等位基因A和a,频率为pA和pa,其中pA+pa=1。同样地,具有等位基因B和b的不同的SNPY具有频率pB和pb,其中pB+pb=1。如果SNPX与SNPY随机关联,那么携带等位基因的任何特定组合的配子的频率等于那些等位基因频率的产物:AB:pA×pBab:pa×pbAb:pA×pbaB:pa×pB在随机关联中的SNP处于连锁平衡中,并且衍生自所述随机关联的SNP处于连锁不平衡中。例如,在pAB>pA×pB条件下,指示SNPX上的A等位基因与SNPY上的B等位基因以高于预期的频率一起进行传递,其中pAB是携带SNPX上的A等位基因和SNPY上的B等位基因的组合的染色体的频率。广泛使用测试来验证两个SNP是否连锁不平衡。将连锁不平衡参数D[7]给定为:D=pAB*pab–pAb*paB如果SNPX和SNPY强连锁不平衡,预期得到高D值,因为我们将观察到非重组配子(AB和ab基因型)的富集,以及重组配子(Aa和aB基因型)的缺乏。作为连锁不平衡的量度,D值具有取决于等位基因频率的局限性。在不同等位基因频率下的相同的D值表示不同程度的连锁不平衡性。相关但不同的参数是r2[7]:r2=D2/(pA*pB*pa*pb)其中r2不依赖于两个目标SNP上的等位基因频率。r2的平方根是SNPX和Y之间的等位基因状态的相关系数。可以对r2参数执行统计显著性检验(卡方检验)。χ2的值在数值上等于r2*N,其中N是检查的染色体的总数[7]。此χ2具有一个自由度,并且可以计算相关的概率。为了验证在产仔数调控上的效应,我们从每个选择性SweepA到E中随机选择标签SNP。SweepA的标签SNP是6号染色体上的chr6:122,113,635。SweepB的标签SNP是6号染色体上的chr6:89,899,151。SweepC的标签SNP是7号染色体上的chr7:63,714,553。SweepD的标签SNP是15号染色体上的chr15:51,799,437。SweepE的标签SNP是3号染色体上的chr3:72,759,645。与标签SNP紧密关联的SNP示于表1中,并且标签SNP以粗体示出。标签SNP和侧翼SNP之间的关联示于表2中。在白色杜洛克×二花脸F2代母猪中验证SweepA在产仔数调控方面的效应从我们以前开发的白色杜洛克×二花脸F2代母猪群体中选择了192头F2代母猪[3]。如Ren等人[8]中所述,通过将2头白色杜洛克公猪和17头二花脸母猪进行杂交,产生了F2代来源群体。在所述F2代来源群体中,在6号、7号、8号和15号染色体上鉴定了针对产仔数的QTL[3]。使用SNAPSHOT基因分型平台在所述F2代母猪上确定了标签SNPchr6:122,113,635上的基因型。在186头F2代母猪中成功分型了基因型。C/C(即O/O基因型),T/C(即E/O基因型)和T/T(即E/E基因型)三种基因型的个体数分别是58、85和43。携带C/C基因型的F2代母猪的平均产仔数是10.0676,标准差为0.9348;携带T/C基因型的F2代母猪的平均产仔数是11.0982,标准差为0.9206;携带T/T基因型的F2代母猪的平均产仔数是11.8184,标准差为0.9587。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现从SweepA中选择的标签SNP与产仔数统计显著性相关(图1)。平均来说,携带T/T基因型的F2代母猪比携带C/C基因型的F2代母猪每窝多生产1.75头仔猪(p=0.0018);并且携带T/C基因型的F2代母猪比携带C/C基因型的F2代母猪每窝多生产1.03头仔猪(p=0.0225);观察到这样的趋势:即携带C/C基因型的F2代母猪生产最少产仔数。在苏泰母猪中验证SweepA在产仔数调控方面的效应我们进一步在苏泰(杜洛克×梅山)母猪群中检测了SweepA的效应。选择了192个个体的苏泰母猪群。在其中的185头母猪中成功分型了标签SNPchr6:122,113,635。C/C(即O/O基因型)、T/C(即E/O基因型)和T/T(即E/E基因型)三种基因型的个体数分别是24、103和58。如图2中所示,携带C/C基因型的苏泰母猪的平均产仔数是11.359,标准差为0.3789;携带T/C基因型的苏泰母猪的平均产仔数是12.0029,标准差为0.2912;携带T/T基因型的苏泰母猪的平均产仔数是11.874,标准差为0.3095。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。在苏泰母猪群中证实了标签SNP和产仔数性状之间的关联。平均来说,携带T/C基因型的苏泰母猪比携带C/C基因型的苏泰母猪每窝多生产0.64头仔猪(p=0.0251);尽管在携带T/T基因型或者C/C基因型的苏泰母猪中差异并不具有统计显著性(p=0.0873),携带T/T基因型的个体仍然比携带C/C基因型的个体每窝多生产0.51头仔猪。观察到这样的趋势:即携带C/C基因型的苏泰母猪生产最小产仔数,这与在白色杜洛克×二花脸F2代母猪中观察到的趋势类似。在白色杜洛克×二花脸F2代母猪中验证SweepB在产仔数调控方面的效应从我们以前开发的白色杜洛克×二花脸F2代母猪群体中选择了192头F2代母猪[3]。如Ren等人[8]中所述,通过将2头白色杜洛克公猪和17头二花脸母猪进行杂交,产生了F2代来源群体。在所述F2代来源群体中,在6号、7号、8号和15号染色体上鉴定了针对产仔数的QTL[3]。使用SNAPSHOT基因分型平台在192头F2代母猪上确定了标签SNPchr6:89,899,151上的基因型。在其中161个个体上成功分型了基因型。如表1中所示,在所述标签SNPchr6:89,899,151上,E等位基因SNP标记是C,O等位基因SNP标记是T。C/C(即E/E基因型),T/C(即E/O基因型)和T/T(即O/O基因型)三种基因型的个体数分别是43、69和49。如图3中所示,携带C/C基因型的F2代母猪的平均产仔数是11.7932,标准差为0.9316;携带T/C基因型的F2代母猪的平均产仔数是11.1217,标准差为0.9001;携带T/T基因型的F2代母猪的平均产仔数是10.3489,标准差为0.9244。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现标签SNP与产仔数统计显著性相关。平均来说,携带C/C基因型的F2代母猪可以比携带T/T基因型的F2代母猪每窝多生产1.44头仔猪(p=0.0127);虽然当将携带T/C基因型的F2代母猪和携带T/T基因型的F2代母猪进行比较时,统计性测试呈现出非显著性结果(p=0.1226),携带T/C基因型的个体比携带T/T基因型的个体每窝多生产0.77头仔猪。观察到这样的趋势:即携带T/T基因型的F2代母猪具有最小产仔数。在白色杜洛克×二花脸F2代母猪中验证SweepC在产仔数调控方面的效应从我们以前开发的白色杜洛克×二花脸F2代母猪群体中选择了192头F2代母猪[3]。如Ren等人[8]中所述,通过将2头白色杜洛克公猪和17头二花脸母猪进行杂交,产生了F2代来源群体。在所述F2代来源群体中,在6号、7号、8号和15号染色体上鉴定了针对产仔数的QTL[3]。使用SNAPSHOT基因分型平台在192头F2代母猪上确定了标签SNPchr7:63,714,553上的基因型。在其中185个个体上成功分型了基因型。如表1中所示,在所述标签SNPchr7:63,714,553上,E等位基因SNP标记是A,O等位基因SNP标记是G。G/G(即O/O基因型),A/G(即E/O基因型)和A/A(即O/O基因型)三种基因型的个体数分别是6、58和121。如图4中所示,携带G/G基因型的F2代母猪的平均产仔数是7.6198,标准差为1.3942;携带A/G基因型的F2代母猪的平均产仔数是10.7951,标准差为0.9178;携带A/A基因型的F2代母猪的平均产仔数是10.9955,标准差为0.8884。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现标签SNP与产仔数性状统计显著性相关。平均来说,携带A/G基因型的F2代母猪比携带G/G基因型的F2代母猪每窝能多生产3.17头仔猪(p=0.0086);并且当将携带G/G基因型的F2代母猪和携带A/A基因型的F2代母猪进行比较时,统计性测试呈现出显著性结果(p=0.0045),携带A/A基因型的个体比携带G/G基因型的个体每窝多生产3.37头仔猪。观察到这样的趋势:即携带G/G基因型的F2代母猪具有最小产仔数。在苏泰母猪中验证SweepC在产仔数调控方面的效应我们进一步在苏泰(杜洛克×梅山)母猪群中检测了SweepC的效应。选择了192个个体的苏泰母猪群。在其中的181头母猪中成功分型了标签SNPchr7:63,714,553。G/G(即O/O基因型)、G/A(即E/O基因型)和A/A(即E/E基因型)三种基因型的个体数分别是53、97和31。如图5中所示,携带A/A基因型的苏泰母猪的平均产仔数是12.0069,标准差为0.3598;携带A/G基因型的苏泰母猪的平均产仔数是12.0076,标准差为0.2905;携带G/G基因型的苏泰母猪的平均产仔数是11.4269,标准差为0.3199。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijkl其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。在苏泰母猪群中证实了标签SNPchr7:63,714,55和产仔数性状之间的关联。平均来说,携带A/G基因型的苏泰母猪比携带G/G基因型的苏泰母猪每窝能多生产0.58头仔猪(p=0.0034);并且当将携带A/A基因型的F2代母猪和携带G/G基因型的F2代母猪进行比较时,统计性测试呈现出显著性结果(p=0.0384),携带A/A基因型的个体比携带G/G基因型的个体每窝多生产0.57头仔猪。观察到这样的趋势:即携带G/G基因型的苏泰母猪生产最小产仔数,这与在白色杜洛克×二花脸F2代母猪中观察到的趋势类似。在白色杜洛克×二花脸F2代母猪中验证SweepD在产仔数调控方面的效应从我们以前开发的白色杜洛克×二花脸F2代母猪群体中选择了192头F2代母猪[3]。如Ren等人[8]中所述,通过将2头白色杜洛克公猪和17头二花脸母猪进行杂交,产生了所述F2代来源群体。在所述F2代来源群体中,在6号、7号、8号和15号染色体上鉴定了针对产仔数的QTL[3]。在其中183个个体上成功分型了标签SNPchr15:51,799,437上的基因型。如表1中所示,在所述标签SNPchr15:51,799,437上,E等位基因SNP标记是T,O等位基因SNP标记是C。C/C(即O/O基因型),T/C(即E/O基因型)和T/T(即E/E基因型)三种基因型的个体数分别是53、87和43。如图6中所示,携带C/C基因型的F2代母猪的平均产仔数是9.8743,标准差为0.9046;携带T/C基因型的F2代母猪的平均产仔数是11.3673,标准差为0.8657;携带T/T基因型的F2代母猪的平均产仔数是10.7179,标准差为0.9227。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现标签SNP与产仔数性状统计显著性相关。平均来说,携带T/C基因型的F2代母猪比携带C/C基因型的F2代母猪每窝能多生产1.49头仔猪(p=0.0018);尽管当将携带C/C基因型的F2代母猪和携带T/T基因型的F2代母猪进行比较时,统计性测试呈现出非显著性结果(p=0.1211),携带T/T基因型的个体比携带C/C基因型的个体每窝多生产0.84头仔猪。观察到这样的趋势:即携带C/C基因型的F2代母猪具有最小产仔数。在苏泰母猪中验证SweepD在产仔数调控方面的效应我们进一步在苏泰(杜洛克×梅山)母猪群中检测了SweepD的效应。选择了192个个体的苏泰母猪群。在其中的185头母猪中成功分型了标签SNPchr15:51,799,437。C/C、T/C和T/T三种基因型的个体数分别是84、87和14。如图7中所示,携带C/C基因型的苏泰母猪的平均产仔数是11.742,标准差为0.2919;携带T/C基因型的苏泰母猪的平均产仔数是12.0997,标准差为0.3037;携带T/T基因型的苏泰母猪的平均产仔数是12.2924,标准差为0.4272。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。在苏泰母猪群中证实了标签SNPchr15:51,799,437和产仔数性状之间的关联。平均来说,携带T/C基因型的苏泰母猪比携带C/C基因型的苏泰母猪每窝能多生产0.36头仔猪(p=0.0436);尽管在携带T/T基因型或者C/C基因型的苏泰母猪中差异并没有统计显著性(p=0.1153),携带T/T基因型的个体还是比携带C/C基因型的个体每窝多生产0.55头仔猪。观察到这样的趋势:即携带C/C基因型的苏泰母猪生产最小产仔数,这与在白色杜洛克×二花脸F2代母猪中观察到的趋势类似。在大白母猪中验证SweepE在产仔数调控方面的效应选择了434头大白母猪来验证SweepE在产仔数调控方面的效应。在所述母猪群中成功分型了在标签SNPchr3:72,759,645上的基因型。C/C(即O/O基因型)、T/C(即E/O基因型)和T/T(即E/E基因型)三种基因型的个体数分别是84、218和132。如图8中所示,携带C/C基因型的大白母猪的平均产仔数是11.4712,标准差为0.3469;携带T/C基因型的大白母猪的平均产仔数是11.9252,标准差为0.3182;携带T/T基因型的大白母猪的平均产仔数是12.1636,标准差为0.325。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现标签SNP和产仔数性状之间具有统计显著性关联。平均来说,携带T/T基因型的大白母猪比携带C/C基因型的大白母猪每窝能多生产0.69头仔猪(p=0.0015);携带T/C基因型的大白母猪比携带C/C基因型的大白母猪每窝多生产0.45头仔猪(p=0.0221)。观察到这样的趋势:即携带T/T基因型的大白母猪具有最高产仔数。在大白猪×长白猪(LL)F1代杂交母猪中验证SweepE在产仔数调控方面的效应选择了404头LLF1代母猪来验证SweepE在产仔数调控方面的效应。在LLF1代母猪群中成功分型了标签SNPchr3:72,759,645上的基因型。C/C、T/C和T/T三种基因型的个体数分别是84、218和132。如图9中所示,携带C/C基因型的LLF1代母猪的平均产仔数是10.9214,标准差为0.4841;携带T/C基因型的LLF1代母猪的平均产仔数是11.6953,标准差为0.3551;携带T/T基因型的LLF1代母猪的平均产仔数是11.9334,标准差为0.327。使用下面的混合模型,采用混合的SAS程序来分析标签SNP位点和产仔数之间的关联:Yijklm=u+Ai+Gj+Bk+Pl+eijklm其中:Yijkl是猪产仔数的总数或者活猪数;Ai是第i个个体的加性效应(多基因效应);Gj是第j种SNP基因型的固定效应;Bk是系列批次的固定效应(k=1,2,3,4);Pl是第l个校验(parity)的随机效应(l=1,2,3),将三个校验(parity)视为重复性测量。发现标签SNP和产仔数性状之间具有统计显著性关联。平均来说,携带T/T基因型的LLF1代母猪比携带C/C基因型的LLF1代母猪每窝能多生产1.012头仔猪(p=0.0015);携带T/C基因型的LLF1代母猪比携带C/C基因型的LLF1代母猪每窝能多生产0.7739头仔猪(p=0.0558);观察到这样的趋势:即携带T/T基因型的LLF1代母猪具有最高产仔数。参考文献1.ZhangZ(1986)PigBreedsinChina.ShanghaiScientificandTechnicalPublishers,Shanghai,China.2.RothschildMF,MesserL,DayA,WalesR,ShortT,etal.(2000)Investigationoftheretinol-bindingprotein4(RBP4)geneasacandidategeneforincreasedlittersizeinpigs.MammGenome11:75-77.3.LiK,RenJ,XingY,ZhangZ,MaJ,etal.(2009)QuantitativetraitlociforlittersizeandprenatallossinaWhiteDurocxChineseErhualianresourcepopulation.AnimGenet40:963-966.4.WilkiePJ,PaszekAA,BeattieCW,AlexanderLJ,WheelerMB,etal.(1999)Agenomicscanofporcinereproductivetraitsrevealspossiblequantitativetraitloci(QTLs)fornumberofcorporalutea.MammGenome10:573-578.5.LarsonG,DobneyK,AlbarellaU,FangM,Matisoo-SmithE,etal.(2005)Worldwidephylogeographyofwildboarrevealsmultiplecentersofpigdomestication.Science307:1618-1621.6.WuGS,YaoYG,QuKX,DingZL,LiH,etal.(2007)PopulationphylogenomicanalysisofmitochondrialDNAinwildboarsanddomesticpigsrevealedmultipledomesticationeventsinEastAsia.GenomeBiol8:R245.7.HartlDL,ClarkAGPrinciplesofPopulationGenetics,FourthEdition.SinauerAssociates,Inc8.RenDR,RenJ,XingYY,GuoYM,WuYB,etal.(2009)AgenomescanforquantitativetraitlociaffectingmalereproductivetraitsinaWhiteDurocxChineseErhualianresourcepopulation.JAnimSci87:17-23.表1:染色体6、7、15和3上的SNP标记表2:染色体6上的SNP位点与SNPA2的关联性染色体位置r2P值(χ2,df=1)61220977880.9470.00E+00612211363561221231400.8060.00E+0061221252280.8060.00E+0061221266200.8580.00E+0061221267990.8490.00E+0061221486360.8440.00E+0061221788480.8490.00E+0061221788620.8490.00E+0061221788700.9470.00E+0061221975410.8950.00E+0061221975880.9470.00E+0061222170960.9470.00E+00表3:染色体6上的SNP位点与SNPB13的关联性表4:染色体15上的SNP位点与SNPD1的关联性染色体位置r2P值(χ2,df=1)155179943715518000220.915015518003560.8760表5:染色体3上的SNP位点与SNPE10的关联性染色体位置r2P值(χ2,df=1)37265544112.73E-0637265556112.73E-0637265557412.73E-0637265751612.73E-0637270427312.73E-0637270471312.73E-0637273725612.73E-0637274054112.73E-0637274653912.73E-0637275964537276146812.73E-0637276546412.73E-0637276579712.73E-0637276586112.73E-0637276959412.73E-0637277478912.73E-0637278428912.73E-0637278632812.73E-0637279035012.73E-0637279067812.73E-0637279077812.73E-0637279564212.73E-0637279587212.73E-06当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 用于基因产品的检索方法与流程
- 一种用于检测TNF‑a基因SNP位点rs1799724基因型的扩增引物、试剂盒、方法及其应用与流程
- 用于基因样本储存的多功能系统的制作方法与工艺
- 一种适用于鼠尾藻新型采苗附着基的缓释肥营养制剂的制作方法与工艺
- 一种适用于水域软基的上覆泥密封真空预压装置的制作方法
- 一种适用于发电机组的液压注胶枪的制作方法与工艺
- 一种适用于软基条件下的丁坝的制作方法与工艺
- 用于检测CYP2C19基因SNP位点rs4986893基因型的试剂盒的制作方法与工艺
- 用于检测JAG2基因SNP位点rs2238286基因型的试剂盒的制作方法与工艺
- 用于检测GATA4基因SNP位点rs867858基因型的试剂盒的制作方法与工艺