一种高危人乳头瘤病毒分型检测方法与试剂盒与流程
本发明为一种高危人乳头瘤病毒分型检测方法与试剂盒,属于生物技术领域。
背景技术:
人乳头瘤病毒(humanpapillomavirus,hpv)属于乳头瘤病毒科,是一种小分子的、无被膜包被的、环状双链dna病毒,基因组长约8000碱基对(bp),分为3个功能区,即早期转录区(e区)、晚期转录区(l区)和非转录区(长控制区,lcr)。hpv通过直接或间接接触污染物品或性传播感染人类。该病毒不但具有宿主特异性,而且具有组织特异性,只能感染人的皮肤和粘膜上皮细胞,能引起人体皮肤黏膜的鳞状上皮增殖,目前已分离出130多种引起人类皮肤的多种乳头瘤或疣及生殖道上皮增生性损伤。不同的型别引起不同的临床表现,根据侵犯的组织部位不同可分为:(1)皮肤低危型:包括hpv1、2、3、4、7、10、12、15等型与寻常疣、扁平疣、跖疣等相关;(2)皮肤高危型:包括hpv5、8、14、17、20、36、38型与疣状表皮发育不良有关,其它还与可能hpv感染有关的恶性肿瘤有关,包括:外阴癌、阴茎癌、肛门癌、前列腺癌、膀胱癌;(3)黏膜低危型如hpv6、11、13、32、34、40、42、43、44、53、54等与感染生殖器、肛门、口咽部、食道黏膜相关;(4)黏膜高危型hpv16、18、30、31、33、35、39与宫颈癌、直肠癌、口腔癌、扁桃体癌等相关。
对于感染生殖道和肛门的hpv,根据各基因型别致病力大小或致癌危险性大小可分为低危型和高危型两大类。在有性生活史的女性中生殖道hpv感染具有普遍性,据统计70%~80%的女性在其一生中会有至少一次的hpv感染,但大多数感染为自限性,超过90%的感染的女性会出现一种有效的免疫应答,在没有任何长期的健康干预时在6到24个月之间可以清除感染。而持续性的高危型hpv感染则是导致宫颈上皮内瘤变及宫颈癌的主要原因。全球范围的研究结果显示,在99.7%的宫颈癌患者体内检测到高危型hpvdna的存在,其中hpv16型、18型、45型和31型感染占80%。低危型hpv一般与尖锐湿疣或低度鳞状上皮内病变相关,极少引起浸润癌。依据who国际癌症研究机构(iarc)及其他国际组织的研究成果,国家食品药品监督管理局相关hpv检测产品注册指导原则建议将hpv16、18、31、33、35、39、45、51、52、56、58、59、68这13种基因型列为高危型别,26、53、66、73、82等5种基因型列为中等风险型别。
高危hpv感染是宫颈癌的主要致病因素,99.7%宫颈癌患者存在高危型hpv感染。哈拉尔德•楚尔•豪森于1976年发现hpv是导致女性第二常见癌症——宫颈癌的罪魁祸首。1995年,国际癌症研究机构讨论确认hpv感染是宫颈癌的主要原因,2005年,iarc/who推荐hpv检测可以用于宫颈癌筛查。研究表明,hpv感染阳性者有15%~28%在2年内进展为宫颈鳞状上皮内病变,尤其是hpv16和18型感染危险性更高。由子宫颈鳞状上皮内病变发展到浸润癌大约需要10~20年的时间,现在所需年限正逐渐缩短。hpv感染率高低主要取决于年龄和性行为习惯,年轻性活跃的妇女感染率最高,可达40%。大部分妇女都是短暂性的hpv感染,通常在6-12个月消失,只有少数妇女会持续感染乃至最终发展成宫颈癌。多年来,国际上通用的宫颈上皮内瘤变及宫颈癌的诊断主要遵循“三阶梯式”诊断程序,即宫颈细胞学、阴道镜及组织病理学检查。宫颈细胞学检查是普遍应用的宫颈上皮内瘤变及宫颈癌的筛查方法,可发现早期病变,但宫颈细胞学检查存在其固有的局限性,并且对原位癌的检出率较低。高危型hpv感染的检测对于预防和早期发现宫颈癌有非常重要的意义。
基于taqman探针技术的荧光定量pcr是融汇了pcr高敏感性、dna杂交技术高特异性和光谱技术高精确定量三大技术的一种新的检测方法。taqman探针是一种寡核苷酸探针,荧光基团连接在探针的5’-末端,淬灭基团在探针3’-末端。当探针完整及其与靶序列配对时,荧光基团发射的荧光因与3’-端的淬灭基团接近而被淬灭,无荧光信号产生。在进行pcr反应延伸过程时,taqdna聚合酶的5'→3'外切核酸酶活性将探针降解,使得荧光基团与淬灭基团分离,即产生荧光信号。每经历一个pcr反应循环,就有一分子的扩增产物生成,并伴随着一分子的荧光信号的产生。随着扩增循环数的增加,释放出来的荧光基团信号不断积累,为一个指数同步增长的过程,荧光信号的强度就代表了扩增产物的拷贝数。该技术具有特异性强、自动化程度高等特点、有效解决了pcr污染等问题。单重探针实时定量pcr,因每条探针都是针对特异的靶位点设计,具有较高的特异性,每条探针只能检测一种靶序列,如需检测多种靶序列,需要加入多条探针,由于探针荧光标记吸收波长因素的限制,基因序列检测种类受到制约限制。
本发明提供一种高危人乳头瘤病毒分型检测方法与试剂盒,该方法采用多条同源上游引物、多条同源下游引物以及多条探针的方法,即在上下游引物设计位点,设计多条同源引物覆盖扩增更多的同源序列,多条探针标记可标记相同的荧光发光基团,在荧光扩增后结合熔解曲线进行多型别hpv分型。根据taqman引物探针设计原则,选择在人乳头瘤病毒基因组的l1段序列作为靶位点设计通用扩增引物和人乳头瘤病毒特异性的taqman探针,根据taqman探针序列设计与其不完全互补的杂交探针——pco(partiallycomplementaryoligonucleotide,部分互补序列),杂交探针目的用于扩增后产物熔解曲线分析,杂交探针的3’端可根据目的进行荧光淬灭基团标记(即blackholequencher,缩写bhq,包括bhq1、bhq2、bhq3等)或者磷酸化基团(phosphate,pho)标记,杂交探针标记后3’端不能再延伸。同时提供一种人乳头瘤病毒核酸分型检测试剂盒,通过荧光pcr扩增结合单波长熔解曲线分析鉴别hpv16、18、31、52、59型。本发明还引入内源性的内参照系统,用于监测样本取样质量以避免检测结果的假阴性。和现有人乳头瘤病毒核酸分型检测公开技术比较,本发明的检测结果稳定可靠,试剂盒使用方便,检测灵敏度高,单个波长进行荧光pcr反应即可鉴别更多的hpv基因亚型。
技术实现要素:
本发明的目的之一是提供一种高危人乳头瘤病毒分型检测方法,另一目的是提供一种hpv16、18、31、52、59型分型检测试剂盒。
本发明在markusschmitt等人(jclinmicrobiol,2008,46(3):1050–1059)设计的通用引物bsgp5+/6+(bs)基础上,综合考虑试剂盒所覆盖检测的高危hpv型别以及荧光pcr扩增退火温度较高的特点,设计适合本试剂盒的通用引物序列seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5、seqidno.6、seqidno.7、seqidno.8、seqidno.9。
本发明中hpv16、18、31、52、59型检测基于l1基因,优选的靶序列分别为:seqidno.10、seqidno.11、seqidno.12、seqidno.13、seqidno.14,并根据靶序列设计hpv16、18、31、52、59型特异性探针序列分别为:seqidno.15、seqidno.16、seqidno.17、seqidno.18、seqidno.19,taqman探针5’端均标记相同的荧光基团标记(fam、vic/joe/hex、rox或者cy5),3’端均标记相同的bhq2淬灭基团。
在本发明中,根据taqman探针的互补序列进行pco杂交探针设计,pco杂交探针设计原则为:(1)序列与相应的taqman探针序列不完全互补(一般有3-6个碱基的错配);(2)不同的taqman探针可以与同一条pco探针杂交进行熔解曲线分析;(3)pco探针3’端必须进行标记封闭,防止pco探针3’端延伸,3’端标记的淬灭基团可以为bhq1、bhq2、bhq3、pho等;(4)pco探针设计完成后需对其进行熔解曲线模拟分析tm值。根据hpv特异性探针序列设计出三条pco探针,pco探针序列分别为:seqidno.20、seqidno.21、seqidno.22,其中序列seqidno.203’端以pho标记修饰封闭,seqidno.21、seqidno.223’端以bhq2淬灭基团修饰封闭。
本发明还提供一种人内源性内参照系统,内参引物为seqidno.23、seqidno.24,内参探针为seqidno.25,内参探针5’端均标记与hpv目的检测探针不同的荧光基团标记(fam、vic/joe/hex、rox或者cy5),3’端标记bhq2淬灭基团。
本发明提供一种hpv16、18、31、52、59型分型检测试剂盒,采用荧光pcr结合熔解曲线技术对高危人乳头瘤病毒16、18、31、52、59型进行核酸定性检测。在使用上海复星长征医学科学有限公司生产的磁珠法纯化试剂(核酸提取及纯化试剂(产品型号:拭子dna))对宫颈脱落细胞样本进行全自动核酸提取后,直接配制反应体系进行扩增,并通过熔解曲线进行结果分型分析,反应体系配制方便,pcr产物无需开盖分析,通过单波长的熔解曲线分析即可区分5种高危型别,并且体系中引入了dutp-ung酶系统防止pcr产物污染。
本发明提供了一种通过荧光pcr扩增并结合单波长熔解曲线分析检测多种高危hpv亚型的方法,同时提供了一种高危人乳头瘤病毒16、18、31、52、59型分型检测试剂盒。
1.实时荧光pcr引物的设计
通过对文献检索确定:在markusschmitt等人设计的通用引物bsgp5+/6+(bs)基础上进行引物优化设计。荧光pcr扩增检测技术中引物设计的规则为:(1)避免引物自身或引物之间形成4个或4个以上连续配对,避免引物自身形成环状发夹结构;(2)典型的引物18-24个核苷酸长,引物需足够长,保证序列独特性,并降低非特异性;(3)tm值在55-65℃,gc含量在40%-60%;(4)引物之间的tm值避免相差2℃;(5)引物的3’端避免使用碱基a,引物3’端避免出现3个或者3个以上连续相同的碱基;(6)taqman探针技术要求片段长度在50-250bp之间;(7)引物末端(最后5个核苷酸)不能有超过2个的g和c。
按照taqman荧光pcr设计的原则,基于bsgp5+/6+(bs)通用引物组的基础设计出本发明hpv扩增通用引物:seqidno.1、seqidno.2、seqidno.3、seqidno.4、seqidno.5、seqidno.6、seqidno.7、seqidno.8、seqidno.9,具体序列如下:
上游引物为:
5’-caactatttgttactgttgttgatactac-3’如seqidno.1所示;
5’-caattatttgttactgtggtagataccac-3’如seqidno.2所示;
5’-cagttgtttgtcacagttgtggataccac-3’如seqidno.3所示;
5’-caattgtttttaacagttgtagatcctac-3’,如seqidno.4所示;
下游引物为:
5’-gaaatataaactgcaaatcaaattcctc-3’,如seqidno.5所示;
5’-gaaaaataaattgtaaatcaaattcctc-3’,如seqidno.6所示;
5’-gaaatataaattgtaaatcaaattcctc-3’,如seqidno.7所示;
5’-gaaaaataaactgcaaatcatattcctc-3’,如seqidno.8所示;
5’-gaaaaataaactgtaaatcatattcctc-3’,如seqidno.9所示。
2.hpv分型探针的设计
taqman探针设计原则为:(1)探针长度应在15bp-45bp(最好20-30bp),保证结合特异性;(2)探针的dna折叠和二级结构尽量避开;(3)tm值在65-70℃,通常比引物tm值高5-10℃(至少5℃),以确保在退火过程中探针先于引物与目的片段结合,gc含量在40%-60%;(4)探针的5’端尽量避免使用g鸟嘌呤;(5)整条探针中碱基c的含量要明显高于g含量。hpv16、18、31、52、59型特异性探针分别以基因组l1片段序列设计,l1片段分别为:seqidno.10、seqidno.11、seqidno.12、seqidno.13、seqidno.14,根据l1片段设计的taqman探针序列分别为:seqidno.15、seqidno.16、seqidno.17、seqidno.18、seqidno.19。
hpv16型探针序列为:
5’-tgctgccatatctacttcagaaactacatataa-3’,5’端标记fam(6-羧基荧光素),3’端标记bhq2,如seqidno.15所示。
hpv18型探针序列为:
5’-tgcttctacacagtctcctgtacctggg-3’,5’端标记fam,3’端标记bhq2,如seqidno.16所示。
hpv31型探针序列为:
5’-tgctgcaattgcaaacagtgatactacatttaa-3’,5’端标记fam,3’端标记bhq2,如seqidno.17所示。
hpv52型探针序列为:
5’-tgctgaggttaaaaaggaaagcacatataa-3’,5’端标记fam,3’端标记bhq2,如seqidno.18所示。
hpv59型探针序列为:
5’-tgcttctactacttcttctattcctaatgtac-3’,5’端标记fam,3’端标记bhq2,如seqidno.19所示。
3.hpvpco探针的设计及验证
本发明的提供一种pco探针序列设计方法,pco探针是一种与靶序列不完全匹配的探针,设计目的是为了在熔解曲线分析中区分不同的靶核酸序列,且一条pco探针可以区分多条靶核酸序列。根据seqidno.15、seqidno.16、seqidno.17、seqidno.18、seqidno.19探针的互补序列设计pco探针,以taqman探针seqidno.18为例设计其pco探针:
seqidno.18序列:5’-tgctgccatatctacttcagaaactacatataa-3’
其互补序列为:5’-ttatatgtagtttctgaagtagatatggcagca-3’
在其互补序列基础上设计5个碱基错配,经过设计错配的互补序列为:5’-ttatatgtagtttcagatgtaaatatgcgagca-3’,3’端标记bhq2淬灭基团,如seqidno.21所示。
seqidno.18序列(5’--->3’方向)与seqidno.21序列(3’--->5’方向)互补情况如下:
seqidno.21探针序列设计完成之后进行模拟探针杂交熔解曲线分析,模拟分析结果见附图1,再通过实验室或者第三方引物探针合成服务公司合成引物探针,进行实验验证,验证结果见附图2。以同样的方法设计pco探针seqidno.20和seqidno.22,序列如下:
5’-ttatatgtagtatcactgttagctatcgcagca-3’,3’端标记pho,如seqidno.20所示;
5’-gtatatatttggtacaggagactctgtagagg-3’,3’端标记bhq2,如seqidno.22所示;
seqidno.20探针分别与seqidno.15探针和seqidno.17探针杂交互补,seqidno.22探针分别与seqidno.16探针和seqidno.19探针互补。
hpv16、18、31、52、59型的taqman探针分别与各自的pco探针熔解曲线分析,结果分别见图3、4、5、2、6,之后混合到一管中进行荧光pcr扩增结合单波长熔解曲线分析验证,结果见图7~图11。
4.内参照系统设计
本发明中按taqman探针荧光pcr原则设计了内源性内参照系统,用于监测样本的取样、提取和扩增,避免检测结果假阴性,内参照系统包括内参引物和探针,根据taqman荧光pcr设计原则,设计的引物和探针序列如下:
5’-ctaagcagaactcttccccat-3’,如seqidno.23所示;
5’-ctcagccagtcctccacat-3’,如seqidno.24所示;
5’-tctgcctatggcttgaccaggcag-3’,5’端标记cy5荧光基团,3’端标记bhq2,如seqidno.25所示。
5.试剂盒制备
含有上述引物和探针的人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法)(32人份),其主要组分如下:
(1)pcr缓冲液,288μl
配制方法:在14.2ml超纯水中依次加入10*pcrbuffer(含100mmph8.3的tris-hcl、500mmkcl)30.0ml,5mmdntps(包括datp、dgtp、dctp、dutp)各12ml,0.05mol/lmgcl233ml,10mg/mlbsa0.8ml,振荡混匀,终体积为90.0ml,以288μl/管分装后即为试剂盒中pcr缓冲液。
(2)引物探针,256μl
配制方法:在76.31ml灭菌超纯水中分别加入156μl200pmol/μl引物seqidno.1,156μl200pmol/μl引物seqidno.2,156μl200pmol/μl引物seqidno.3,156μl200pmol/μl引物seqidno.4,156μl200pmol/μl引物seqidno.5,156μl200pmol/μl引物seqidno.6,156μl200pmol/μl引物seqidno.7,156μl200pmol/μl引物seqidno.8,156μl200pmol/μl引物seqidno.9,112.5μl200pmol/μl引物seqidno.15,139.5μl200pmol/μl引物seqidno.16,84μl200pmol/μl引物seqidno.17,97.5μl200pmol/μl引物seqidno.18,84μl200pmol/μl引物seqidno.19,555μl200pmol/μl引物seqidno.20,284.8μl200pmol/μl引物seqidno.21,474μl200pmol/μl引物seqidno.22,60μl200pmol/μl引物seqidno.23,90μl200pmol/μl引物seqidno.24,300μl200pmol/μl引物seqidno.25,震荡混匀后以256μl/管分装,即为试剂盒中引物探针。
(3)dna聚合酶,96μl
配制方法:在24.8ml酶稀释液(从上海宏谊生物技术有限公司外购)中加入taq-dna聚合酶(5u/μl)1.5ml和ung酶(1u/μl)3.7ml,颠倒多次混匀,终体积为30.0ml,以96μl/管分装即为试剂盒dna聚合酶。
(4)阴性对照,400μl
用含有0.9%nacl,1mmol/ledta,10mmol/ltris-hcl和0.1%tritonx-100的缓冲液稀释正常人类基因组dna至10ng/ml,以400μl/管分装即为阴性对照。
(5)阳性对照和临界阳性对照,各400μl
用含有0.9%nacl,1mmol/ledta,10mmol/ltris-hcl、0.1%tritonx-100和10ng/ml正常人类基因组dna的缓冲液分别稀释hpv阳性质粒至104copies/µl、102copies/µl,旋涡震荡混匀10分钟,以400μl/管分装后即为阳性对照和临界阳性对照。
将上述组分(1)~(5)分别进行抽检,检验合格后进行包装。
上述组装的试剂盒在-20℃保存,冻融次数应小于3次。
由(1)~(3)及模板配制的30μl反应体系,每个反应中加入pcr缓冲液9μl、引物探针8μl以及dna聚合酶3μl,或者按比例配制公共体系后以20μl/反应进行分装,分装后每管中加入10μl提取的核酸样本。
3.试剂盒使用方法
本发明试剂盒在样本检测过程中使用方法之一如下:
(1)样本提取
取临床采集的宫颈脱落细胞样本,吸取含有宫颈脱落细胞的细胞保存液200μl置于上海复星长征医学科学有限公司生产的磁珠法纯化试剂(核酸提取及纯化试剂(产品型号:拭子dna))96孔核酸提取板的a1-h1、a7-h7孔中,按照提取试剂盒说明书的要求设置自动提取程序进行核酸提取,提取板的a5-h5、a11-h11孔为提取的核酸用于荧光pcr扩增检测。试剂盒阴性、临界阳性对照和阳性对照处理方法和样品液相同。
(2)扩增检测
按照发明内容中的方法,启封试剂盒,将各组分冻融振荡混匀后配制反应预混液,其配制方法如下:
a按反应样本数(反应样本数=待检样品数+对照品3个+1)n配制反应液:取pcr反应液n×9μl、引物探针n×8μl、dna聚合酶n×3μl于一离心管中混匀;低速离心数秒,按20μl/管分装到反应管中,再分别加入10μl提取待检测的核酸。
b取样本提取物、对照品提取物各10μl分别加入反应管中,低速离心数秒,取出置全自动荧光pcr仪上。
c荧光pcr反应程序如下:
[1]50℃2min
[2]95℃3min
[3]95℃6s
[4]66℃45s
[5]goto[3],9cycles
[6]95℃3s
[7]60℃33s
[8]63℃15s
[9]goto[6],42cycles
在第[7]步采集fam和cy5通道荧光信号
[10]95℃15s
[11]25℃1min
[12]25℃——75℃熔解曲线分析
[13]75℃15s
[14]end。
注:rochelightcycler480荧光pcr仪上反应程序具体参数设置要求如下:
1)检测模式选择“multicolorhydrolysisprobe”,且同时选择fam及cy5通道;
2)扩增及荧光采集步骤,选择在60℃采集荧光;
3)熔解曲线步骤,熔解温度范围25~75℃,其中参数“raperate(℃/s)”选择0.03,“acquisitions(per℃)”选择5,并且连续采集fam及cy5通道的荧光。
d仪器pcr程序运行完成后按仪器及软件要求进行结果保存和数据分析。对照图7-11进行熔解曲线分型分析。
cy5通道检测结果应符合下表要求:32<ctcy5
4.有益效果
本发明根据在markusschmitt等人设计的通用引物bsgp5+/6+(bs)基础上进行引物优化设计,设计适合本试剂盒的引物组合,在人乳头瘤病毒特异的taqman探针的互补序列基础上设计pco探针,通过模拟验证和实验验证,最后组研制出人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法),其主要特点如下:
(1)采用人乳头瘤病毒保守l1片段序列,根据文献报道优化设计引物。
(2)根据taqman探针的互补序列设计pco探针,根据pco探针和taqman探针进行熔解曲线分析进行分型检测hpv16、18、31、52、59型核酸,以相同荧光标记的taqman探针单管单波长定性检测不同hpv型别,试剂盒检测灵敏度高,结果稳定可靠。
(2)引入内源性内参照系统,可以监测提取和扩增整个过程,可以尽可能的避免假阳性和假阴性结果。
(3)扩增体系中的dutp-ung酶系统更进一步避免了因扩增产物污染导致的结果假阳性。
本发明方法原理是基于taqman水解探针荧光pcr原理,结合探针杂交熔解曲线分析技术,以人乳头瘤病毒l1段基因序列为靶序列,采用相同荧光标记的hpv16、18、31、52、59型特异的探针,经荧光pcr扩增结合熔解曲线分析,可快速、准确的判断hpv16、18、31、52、59型核酸有无并进行分型鉴别。
附图说明
为了使本发明的目的、技术方案和有益效果更加清楚,本发明提供如下附图进行说明。
图1seqidno.18探针与seqidno.21探针在终浓度分别为0.1mm、0.2mm,na+浓度0.1m,mg2+浓度为4.5mm条件下模拟熔解曲线结果。模拟结果为:tm(conc)61.8°c,δg12.9kcal/mol,δh123.5kcal/mol,δs374.6cal/mol/k,tm(cp)61.8°c,tm(ext1)60.1°c,tm(ext2)58.4°c。
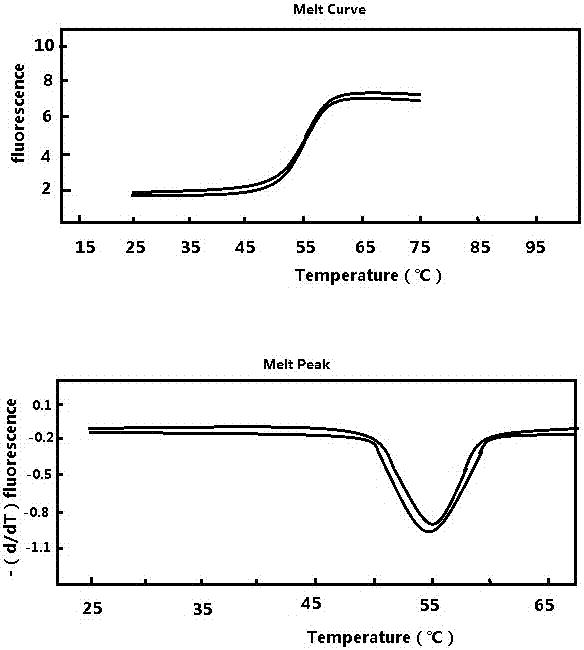
图2seqidno.18探针与seqidno.21探针进行复孔熔解曲线试验分析。
图3seqidno.15探针与seqidno.20探针进行复孔熔解曲线试验分析。
图4seqidno.16探针与seqidno.22探针进行复孔熔解曲线试验分析。
图5seqidno.17探针与seqidno.20探针进行复孔熔解曲线试验分析。
图6seqidno.19探针与seqidno.22探针进行复孔熔解曲线试验分析。
图7a为引物探针混合后复孔进行熔解曲线分析。
图7b为试剂盒hpv16型熔解曲线分型结果,圆圈位置标示曲线为hpv16型特征峰。
图8hpv18型熔解曲线分型结果,圆圈位置标示曲线为hpv18型特征峰。
图9hpv31型熔解曲线分型结果,圆圈位置标示曲线为hpv31型特征峰。
图10hpv52型熔解曲线分型结果,圆圈位置标示曲线为hpv52型特征峰。
图11hpv59型熔解曲线分型结果,圆圈位置标示曲线为hpv59型特征峰。
具体实施方式
以下为该发明结合具体的实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。此外应理解,在阅读了本发明讲授的内容之后,本领域技术人员可以通过技术常识对本发明作各种技术性改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
实施例1人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法)
1.引物探针合成
委托英潍捷基(上海)贸易有限公司合成引物探针,合成的引物探针序列具体如下:
上游引物为:
5’-caactatttgttactgttgttgatactac-3’如seqidno.1所示;
5’-caattatttgttactgtggtagataccac-3’如seqidno.2所示;
5’-cagttgtttgtcacagttgtggataccac-3’如seqidno.3所示;
5’-caattgtttttaacagttgtagatcctac-3’,如seqidno.4所示;
下游引物为:
5’-gaaatataaactgcaaatcaaattcctc-3’,如seqidno.5所示;
5’-gaaaaataaattgtaaatcaaattcctc-3’,如seqidno.6所示;
5’-gaaatataaattgtaaatcaaattcctc-3’,如seqidno.7所示;
5’-gaaaaataaactgcaaatcatattcctc-3’,如seqidno.8所示;
5’-gaaaaataaactgtaaatcatattcctc-3’,如seqidno.9所示。
taqman探针序列为:
5’-tgctgccatatctacttcagaaactacatataa-3’,5’端标记fam,3’端标记bhq2,如seqidno.15所示。
5’-tgcttctacacagtctcctgtacctggg-3’,5’端标记fam,3’端标记bhq2,如seqidno.16所示。
5’-tgctgcaattgcaaacagtgatactacatttaa-3’,5’端标记fam,3’端标记bhq2,如seqidno.17所示。
5’-tgctgaggttaaaaaggaaagcacatataa-3’,5’端标记fam,3’端标记bhq2,如seqidno.18所示。
5’-tgcttctactacttcttctattcctaatgtac-3’,5’端标记fam,3’端标记bhq2,如seqidno.19所示。
pco探针序列为:
5’-ttatatgtagtatcactgttagctatcgcagca-3’,3’端标记pho,如seqidno.20所示。
5’-ttatatgtagtttcagatgtaaatatgcgagca-3’,如3’端标记bhq2淬灭基团,如seqidno.21所示。
5’-gtatatatttggtacaggagactctgtagagg-3’,3’端标记bhq2,如seqidno.22所示。
合成后用无菌双蒸水溶解至浓度为200μm或者500μm,-20℃保存。
2.pcr缓冲液,288μl
配制方法:在14.2ml超纯水中依次加入10*pcrbuffer(含100mmph8.3的tris-hcl、500mmkcl)30.0ml,5mmdntps(包括datp、dgtp、dctp、dutp)各12ml,0.05mol/lmgcl233ml,10mg/mlbsa0.8ml,振荡混匀,终体积为90.0ml,以288μl/管分装后即为试剂盒中pcr缓冲液。
3.引物探针,256μl
配制方法:在76.31ml灭菌超纯水中分别加入156μl200pmol/μl引物seqidno.1,156μl200pmol/μl引物seqidno.2,156μl200pmol/μl引物seqidno.3,156μl200pmol/μl引物seqidno.4,156μl200pmol/μl引物seqidno.5,156μl200pmol/μl引物seqidno.6,156μl200pmol/μl引物seqidno.7,156μl200pmol/μl引物seqidno.8,156μl200pmol/μl引物seqidno.9,112.5μl200pmol/μl引物seqidno.15,139.5μl200pmol/μl引物seqidno.16,84μl200pmol/μl引物seqidno.17,97.5μl200pmol/μl引物seqidno.18,84μl200pmol/μl引物seqidno.19,555μl200pmol/μl引物seqidno.20,284.8μl200pmol/μl引物seqidno.21,474μl200pmol/μl引物seqidno.22,60μl200pmol/μl引物seqidno.23,90μl200pmol/μl引物seqidno.24,300μl200pmol/μl引物seqidno.25,震荡混匀后以256μl/管分装,即为试剂盒中引物探针。
4.dna聚合酶,96μl
配制方法:在24.8ml酶稀释液(从上海宏谊生物技术有限公司外购)中加入taq-dna聚合酶(5u/μl)1.5ml和ung酶(1u/μl)3.7ml,颠倒多次混匀,终体积为30.0ml,以96μl/管分装即为试剂盒dna聚合酶。
5.阴性对照,400μl
用含有0.9%nacl,1mmol/ledta,10mmol/ltris-hcl和0.1%tritonx-100的缓冲液稀释正常人类基因组dna至10ng/ml,以400μl/管分装即为阴性对照。
6.阳性对照和临界阳性对照各400μl
用含有0.9%nacl,1mmol/ledta,10mmol/ltris-hcl、0.1%tritonx-100和10ng/ml正常人类基因组dna的缓冲液分别稀释hpv阳性质粒至104copies/µl、102copies/µl,旋涡震荡混匀10分钟,以400μl/管分装后即为阳性对照和临界阳性对照。
将上述组分2-6分别进行抽样检验,检验合格后进行包装。
上述组装的试剂盒在-20℃保存,冻融次数应小于3次。
试剂盒制备工艺简述如下:
(1)合成的引物探针定量配制、浓度检测、抽样质检。
(2)将pcr缓冲液、taq酶、核酸提取液、内参照无菌分装,抽样质检。
(3)将阴性、阳性对照品进行浓度测定、分装,抽样质检。
(4)组装成品试剂盒,保存于-20℃。
实施例2人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法)的应用
采用本发明所得试剂盒对20例临床样本进行测试,同时用艾康生物技术有限公司生产的人乳头瘤病毒基因分型(25型)检测试剂盒(pcr-反向点杂交法)进行平行对比试验,评价试剂盒效果。
1.人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法)检测临床样本
(1)试剂配制
按样本数n(样本数=待检样本数+对照品3个+1)配制反应液:
取pcr缓冲液n×9μl、酶混合液n×3μl、引物探针n×8μl于一离心管中混匀;低速离心数秒,按20μl/管分装到反应管中。分装后反应管可在2~8℃放置3小时。
(2)核酸提取
将取样管中的宫颈刷充分振荡洗涤,取400μl洗涤液,采用上海复星长征医学科学有限公司生产的磁珠法纯化试剂(核酸提取及纯化试剂(产品型号:拭子dna))进行核酸提取。
核酸提取完成后,核酸提取液用于pcr检测,或将核酸提取液转移到离心管中于-20℃保存。
(3)样本及对照品加样
取阴性对照、阳性对照、临界阳性对照以及待检样本核酸提取液各10μl分别加入反应管中,低速离心数秒,取出置实时荧光定量pcr仪上。
(4)pcr扩增及熔解曲线分析
将pcr扩增管按顺序放入pcr仪,设置如下图的反应程序
[1]50℃2min
[2]95℃3min
[3]95℃6s
[4]66℃45s
[5]goto[3],9cycles
[6]95℃3s
[7]60℃33s
[8]63℃15s
[9]goto[6],42cycles
在第[7]步采集fam和cy5通道荧光信号
[10]95℃15s
[11]25℃1min
[12]25℃——75℃熔解曲线分析
[13]75℃15s
[14]end。
注:rochelightcycler480荧光pcr仪上反应程序具体参数设置要求如下:
1)检测模式选择“multicolorhydrolysisprobe”,且同时选择fam及cy5通道;
2)扩增及荧光采集步骤,选择在60℃采集荧光;
3)熔解曲线步骤,熔解温度范围25~75℃,其中参数“raperate(℃/s)”选择0.03,“acquisitions(per℃)”选择5,并且连续采集fam及cy5通道的荧光。
2.人乳头瘤病毒基因分型(25型)检测试剂盒(pcr-反向点杂交法)检测临床样本
临床操作方法见艾康生物技术有限公司生产的人乳头瘤病毒基因分型(25型)检测试剂盒(pcr-反向点杂交法)说明书。
3.结果分析
对比人乳头瘤病毒16、18、31、52、59型核酸分型检测试剂盒(荧光pcr法)和人乳头瘤病毒基因分型(25型)检测试剂盒(pcr-反向点杂交法)检测20例临床样本结果,检测结果显示两种试剂盒对hpv16、18、31、52、59型的分型结果相同,采用本发明试剂盒具有操作步骤少、检测时间短的优点。
sequencelisting
<110>上海星耀医学科技发展有限公司
<120>一种高危人乳头瘤病毒分型检测方法与试剂盒
<160>25
<170>patentinversion3.3
<210>1
<211>29
<212>dna
<213>artifical
<220>
<221>seqidno.1
<222>(1)..(29)
<400>1
caactatttgttactgttgttgatactac29
<210>2
<211>29
<212>dna
<213>artifical
<220>
<221>seqidno.2
<222>(1)..(29)
<400>2
caattatttgttactgtggtagataccac29
<210>3
<211>29
<212>dna
<213>artifical
<220>
<221>seqidno.3
<222>(1)..(29)
<400>3
cagttgtttgtcacagttgtggataccac29
<210>4
<211>29
<212>dna
<213>artifical
<220>
<221>seqidno.4
<222>(1)..(29)
<400>4
caattgtttttaacagttgtagatcctac29
<210>5
<211>28
<212>dna
<213>artifical
<220>
<221>seqidno.5
<222>(1)..(28)
<400>5
gaaatataaactgcaaatcaaattcctc28
<210>6
<211>28
<212>dna
<213>artifical
<220>
<221>seqidno.6
<222>(1)..(28)
<400>6
gaaaaataaattgtaaatcaaattcctc28
<210>7
<211>28
<212>dna
<213>artifical
<220>
<221>seqidno.7
<222>(1)..(28)
<400>7
gaaatataaattgtaaatcaaattcctc28
<210>8
<211>28
<212>dna
<213>artifical
<220>
<221>seqidno.8
<222>(1)..(28)
<400>8
gaaaaataaactgcaaatcatattcctc28
<210>9
<211>28
<212>dna
<213>artifical
<220>
<221>seqidno.9
<222>(1)..(28)
<400>9
gaaaaataaactgtaaatcatattcctc28
<210>10
<211>277
<212>dna
<213>humanpapillomavirustype16
<220>
<221>seqidno.10
<222>(1)..(277)
<400>10
ctgatgcccaaatattcaataaaccttattggttacaacgagcacagggccacaataatg60
gcatttgttggggtaaccaactatttgttactgttgttgatactacacgcagtacaaata120
tgtcattatgtgctgccatatctacttcagaaactacatataaaaatactaactttaagg180
agtacctacgacatggggaggaatatgatttacagtttatttttcaactgtgcaaaataa240
ccttaactgcagacgttatgacatacatacattctat277
<210>11
<211>281
<212>dna
<213>humanpapillomavirustype18
<220>
<221>seqidno.11
<222>(1)..(281)
<400>11
gctctattgttacctctgactcccagttgtttaataaaccatattggttacataaggcac60
agggtcataacaatggtgtttgctggcataatcaattatttgttactgtggtagatacca120
ctcccagtaccaatttaacaatatgtgcttctacacagtctcctgtacctgggcaatatg180
atgctaccaaatttaagcagtatagcagacatgttgaggaatatgatttgcagtttattt240
ttcagttgtgtactattactttaactgcagatgttatgtcc281
<210>12
<211>259
<212>dna
<213>humanpapillomavirustype31
<220>
<221>seqidno.12
<222>(1)..(259)
<400>12
caaatttttaataaaccatattggatgcaacgtgctcagggacacaataatggtatttgt60
tggggcaatcagttatttgttactgtggtagataccacacgtagtaccaatatgtctgtt120
tgtgctgcaattgcaaacagtgatactacatttaaaagtagtaattttaaagagtattta180
agacatggtgaggaatttgatttacaatttatatttcagttatgcaaaataacattatct240
gcagacataatgacatata259
<210>13
<211>262
<212>dna
<213>humanpapillomavirustype52
<220>
<221>seqidno.13
<222>(1)..(262)
<400>13
aaaccgtactggttacaacgtgcgcagggccacaataatggcatatgttggggcaatcag60
ttgtttgtcacagttgtggataccactcgtagcactaacatgactttatgtgctgaggtt120
aaaaaggaaagcacatataaaaatgaaaattttaaggaataccttcgtcatggcgaggaa180
tttgatttacaatttatttttcaattgtgcaaaattacattaacagctgatgttatgaca240
tacattcataagatggatgcca262
<210>14
<211>308
<212>dna
<213>humanpapillomavirustype59
<220>
<221>seqidno.14
<222>(1)..(308)
<400>14
atttaataaaccatattggctgcacaaggctcagggtttaaacaatggtatatgttggca60
caatcaattgtttttaacagttgtagatcctactcgcagcaccaatctttctgtgtgtgc120
ttctactacttcttctattcctaatgtacacacacctaccagttttaaagaatatgccag180
acatgtggaggaatttgatttgcagtttatatttcaactgtgtaaaataacattaactac240
agaggtaatgtcatacattcataatatgaataccactattttggaggattggaattttgg300
tgttacac308
<210>15
<211>35
<212>dna
<213>artifical
<220>
<221>seqidno.15
<222>(2)..(34)
<223>b=fam,d=bhq2
<400>15
btgctgccatatctacttcagaaactacatataad35
<210>16
<211>30
<212>dna
<213>artifical
<220>
<221>seqidno.16
<222>(2)..(29)
<223>b=fam,d=bhq2
<400>16
btgcttctacacagtctcctgtacctgggd30
<210>17
<211>35
<212>dna
<213>artifical
<220>
<221>seqidno.17
<222>(2)..(34)
<223>b=fam,d=bhq2
<400>17
btgctgcaattgcaaacagtgatactacatttaad35
<210>18
<211>32
<212>dna
<213>artifical
<220>
<221>seqidno.18
<222>(2)..(31)
<223>b=fam,d=bhq2
<400>18
btgctgaggttaaaaaggaaagcacatataad32
<210>19
<211>34
<212>dna
<213>artifical
<220>
<221>seqidno.19
<222>(2)..(33)
<223>b=fam,d=bhq2
<400>19
btgcttctactacttcttctattcctaatgtacd34
<210>20
<211>34
<212>dna
<213>artifical
<220>
<221>seqidno.20
<222>(1)..(33)
<223>d=pho
<400>20
ttatatgtagtatcactgttagctatcgcagcad34
<210>21
<211>34
<212>dna
<213>artifical
<220>
<221>seqidno.21
<222>(1)..(33)
<223>d=bhq2
<400>21
ttatatgtagtttcagatgtaaatatgcgagcad34
<210>22
<211>33
<212>dna
<213>artifical
<220>
<221>seqidno.22
<222>(1)..(32)
<223>d=bhq2
<400>22
gtatatatttggtacaggagactctgtagaggd33
<210>23
<211>21
<212>dna
<213>artifical
<220>
<221>seqidno.23
<222>(1)..(21)
<400>23
ctaagcagaactcttccccat21
<210>24
<211>19
<212>dna
<213>artifical
<220>
<221>seqidno.24
<222>(1)..(19)
<400>24
ctcagccagtcctccacat19
<210>25
<211>26
<212>dna
<213>artifical
<220>
<221>seqidno.25
<222>(2)..(25)
<223>b=cy5,d=bhq2
<400>25
btctgcctatggcttgaccaggcagd26
- 还没有人留言评论。精彩留言会获得点赞!