一种玉米抗逆相关基因ZmDi19‑5及其应用的制作方法
本发明涉及的是基因工程技术领域,尤其涉及的是一种玉米抗逆相关基因ZmDi19-5及其应用。
背景技术:
玉米不仅作为三大粮食物之一,也可作为重要的能源材料。从玉米中生产出的乳酸,经过化学反应可生成高分子材料(聚乳酸),能替代化工塑料,是能够被生物降解的环保材料,在社会和经济发展中具有重要的战略意义。目前,我国玉米生产总量已超过小麦产量,位于我国第二大粮食作物,其重要性已不言而喻。我国大部分玉米种植地区受到干旱和盐渍的胁迫,每年因此而减产,直接影响了旱区和盐渍地的农业生产。因此,增强玉米抗旱和耐盐的能力已成为我国农业研究重要问题之一。
目前选育抗旱和耐盐的新品种方法有很多,相比于传统育种,转基因技术是实现育种目标见效快的方法。该技术关键是挖掘出与干旱和盐胁迫相关的基因,并分析生物学功能和抗逆机理。在模式植物拟南芥中,通过基因缺失突变体进行分析其基因功能已成为主流,相比较玉米而言,获得单基因缺失突变体的方法比较困难,使得对基因功能研究受到一定的限制,因此通过异源转化获得转基因材料方法,在抗逆基因的功能研究中发挥了重要的作用。
植物的抗逆应答机制离不开转录因子的调控。有报道显示,植物的抗逆性能的体现并不是某一个转录因子单独调控的结果,是由多个转录因子协同表达共同影响决定的复杂生物学性状。转录因子的功能调控和植物抗逆机制的应答,为作物分子抗逆育种的开展提供了有力支持,为作物综合抗逆性能的提高和改良提供了理论依据。
研究表明C2H2锌指蛋白家族的转录因子主要参与调控植物的生长发育以及植物的抗逆性相关活动。Di19蛋白不仅是C2H2锌指蛋白家族的一员,同时也是干旱诱导蛋白的成员。目前已经在拟南芥、棉花、大豆、小麦、水稻等植物中克隆出Di19基因,但在玉米中有关Di19基因的转录因子报道还没有。
技术实现要素:
本发明的目的在于克服现有技术的不足,提供了一种玉米抗逆相关基因ZmDi19-5及其应用,以提供一种新的可用于玉米抗逆遗传改良的基因。
本发明是通过以下技术方案实现的:
本发明提供了一种玉米抗逆相关基因ZmDi19-5,其核苷酸序列如SEQ ID NO.1所示,全长720bp。
本发明还提供了上述玉米抗逆相关基因ZmDi19-5在降低或提高植物抗逆性中的应用。
本发明还提供了上述玉米抗逆相关基因ZmDi19-5的编码蛋白,其氨基酸序列如SEQ ID NO.2所示,具有239个氨基酸。
本发明还提供了一种植物过量表达载体,所述植物过量表达载体为pCAMBIA1301载体,所述pCAMBIA1301载体的多克隆位点区域依次连接有35S启动子、ZmDi19-5基因和终止子。
本发明还提供了一种遗传工程化的宿主细胞,所述宿主细胞含有所述植物过量表达载体,或其基因组中整合有外源的所述ZmDi19-5基因的正向或反向序列。
本发明还提供了一种提高植物抗逆性的方法,所述方法是将所述ZmDi19-5基因导入植物的细胞、组织或器官中,获得抗逆性强的转基因植株新品种。
进一步地,所述植物包括玉米、水稻、小麦、拟南芥。
本发明相比现有技术具有以下优点:本发明提供了一种玉米抗逆相关基因ZmDi19-5及其应用,该基因为一种新的植物抗逆相关基因,丰富了抗逆相关基因的资源,对玉米抗逆新品种的选育具有重要意义。
附图说明
图1为ZmDi19-5基因凝胶电泳检测结果图;
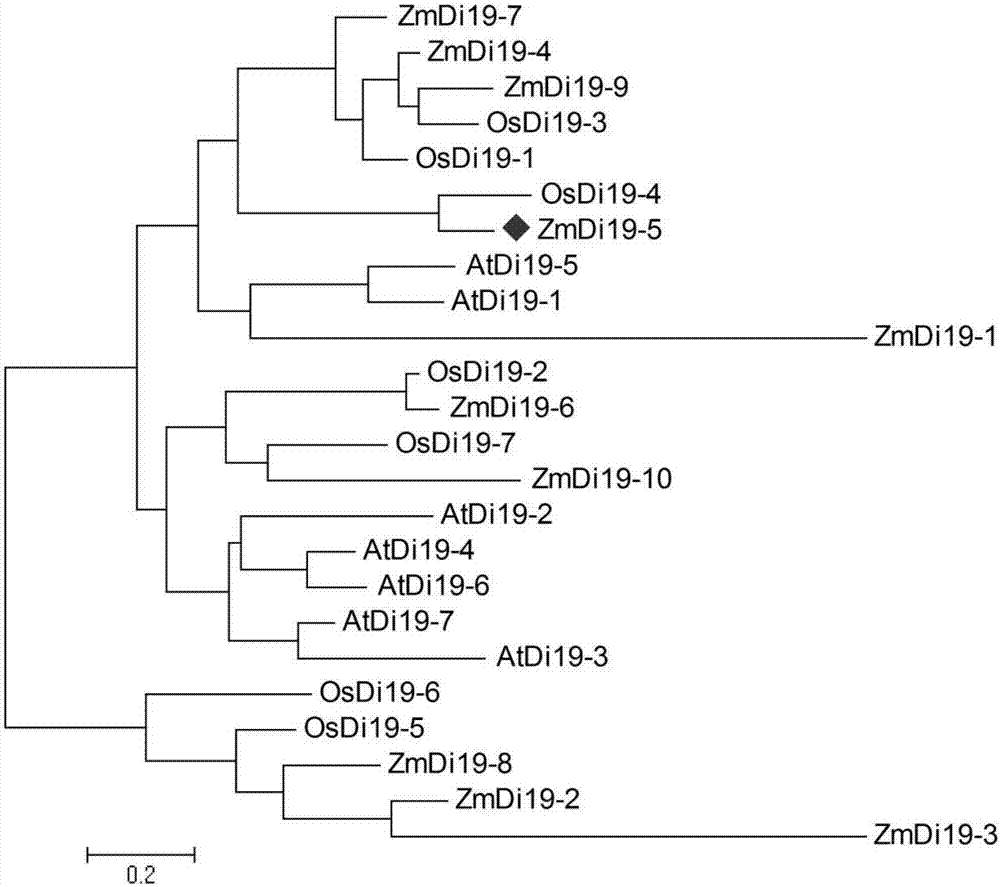
图2为ZmDi19-5蛋白与其他作物的Di19蛋白家族成员的系统进化树分析结果图;
图3为ZmDi19-5基因过表达载体构建图谱;
图4为盐胁迫下转基因拟南芥与野生型拟南芥根生长情况柱状分析图;
图5为盐胁迫下转基因拟南芥与野生型拟南芥株高情况柱状分析图;
图6为盐胁迫下转基因拟南芥与野生型拟南芥丙二醛含量测定结果柱状图;
图7为盐胁迫下转基因拟南芥与野生型拟南芥相对电导率的测定结果柱状图;
图8为干旱处理下转基因拟南芥与野生型拟南芥过氧化氢酶测定结果柱状图;
图9为干旱处理下转基因拟南芥与野生型拟南芥丙二醛含量测定结果柱状图;
图10为干旱处理下转基因拟南芥与野生型拟南芥叶片相对含水量测定结果柱状图。
具体实施方式
实施例1
1、材料
本实施例所用方法如无特别说明均为本领域的技术人员所知晓的常规方法,所用的试剂等材料,如无特别说明,均为市售购买产品。
2、方法
2.1ZmDi19-5基因的克隆
选择玉米B73自交系为实验材料,提取玉米叶片组织RNA,并反转录成cDNA备用。
从玉米数据库检索获取ZmDi19-5的生物学信息,以ZmDi19-5基因序列为模板设计特异性扩增引物,同时结合克隆载体上的多克隆酶切位点,选择Kpn I和BamHI酶切位点引入ZmDi19-5序列两端,特异性扩增引物序列如下所示:
SEQ ID NO.3:ZmDi19-5-F:5’-GGGGTACCATGGATTCCGACCTCTGGAT-3’
SEQ ID NO.4:ZmDi19-5-R:5’-CGGGATCCCTAGTCTTCAAAAATGGTGGTG-3’
以cDNA为模板,ZmDi19-5-F和ZmDi19-5-R为引物,利用TAKARA公司生产的高保真酶PrimerSTAR Max Premix(2×)进行PCR扩增反应,PCR扩增反应体系如下:
PCR扩增程序为:98℃预变性10min,98℃变性10s,62℃退火5s,72℃延伸1min,共30个循环后,加入0.5μL的Taq酶,以给3’端加上PolyA尾巴,最后,72℃下继续延伸10min完成反应,获得的PCR产物用质量比为1%的琼脂糖凝胶电泳检测拍照。
结果如图1所示,图中可以看出,目标基因条带清晰,且大小与预测结果一致,说明PCR结果良好。
进一步地,将目标基因用Axygen公司的胶回收试剂盒进行切胶回收,与T1Simple载体连接后转化到大肠杆菌Trans5α感受态细胞中,用Kpn I和BamHI进行双酶切验证,筛选阳性克隆子,获得T1+ZmDi19-5载体,并送去上海生工公司测序。
2.2ZmDi19-5基因的生物信息学分析
测序结果经Mega4.0软件与数据库中的序列比对后,与SEQ ID NO.1所示的核苷酸序列一致,说明成功克隆获得ZmDi19-5基因。通过在线程序PFAM对ZmDi19-5基因的编码蛋白进行结构域特征分析,结果表明其蛋白结构域含有zf-Di19(位于第49个到102个氨基酸位置)和Di19_C(位于第123个到236个氨基酸位置),说明其为一种Di19蛋白。利用DNAMAN软件,将其氨基酸序列与已知的水稻OsDi19-4的氨基酸序列比对,结果表明,ZmDi19-5基因不仅含有两个Cys2/His2型锌指蛋白结构域,还与OsDi19-4的氨基酸序列的同源性达到74%左右,而OsDi19-4基因被报道是转录因子。由此预测ZmDi19-5属于干旱诱导蛋白家族的转录因子一类。
利用Mega4.0软件,将已报道功能的拟南芥和水稻的Di19蛋白,与玉米Di19蛋白进行系统进化树分析,结果如图2所示,图中,ZmDi19-5与OsDi19-4的亲缘关系最近,而OsDi19-4基因已经被证明参与调控植物的抗旱机制,这一发现为后续的ZmDi19-5基因功能验证奠定基础。
2.3ZmDi19-5基因过表达载体的构建
以pCAMBIA1301为原始载体,在其多克隆位点的EcoRI和SalI酶切位点之间连接一个35S启动子,并在SphI和HindIII酶切位点之间加上一段终止子,获得改造的载体pCAMBIA1301a。
用Kpn I、BamHI双酶切T1+ZmDi19-5载体得到小的目的片段,同时用Kpn I、BamHI双酶切p1301a得到大片段。
连接反应体系为:
连接反应在16℃下进行3h后,转化至大肠杆菌感受态细胞中,挑斑、摇菌、提质粒,并通过双酶切验证,获得如图3所示的ZmDi19-5基因过表达载体p1301a-ZmDi19-5。
2.4ZmDi19-5基因的拟南芥遗传转化
2.4.1拟南芥种子的清洗:
先取12ml次氯酸钠溶液于88ml水中,配置成配置12%的次氯酸钠溶液。取待清洗种子于无菌离心管中,每管加入1ml的12%的次氯酸钠溶液,浸泡清洗15min,期间间隔摇晃;再用无菌水漂洗5次,期间摇晃混匀,以洗净种子表面次氯酸钠溶液和杂质;将清洗杀菌之后的哥伦比亚野生型拟南芥种子在4℃冰箱处理2天,备用。
2.4.2拟南芥的培养
取出清洗之后种子,借助移液枪均匀铺散在MS固体培养基的方形培养皿上,拟南芥培养室培养;将培养基上生长10d左右的幼苗移入混合后的营养土中生长,营养黑土和蛭石体积比为1:3,继续温室培养;需要注意的是,刚刚移栽的幼苗需保持水分,采用透光塑料膜覆盖法,2-3d后揭开。
2.4.3农杆菌介导转化拟南芥
将构建的过表达载体p1301a-ZmDi19-5导入农杆菌EHA105感受态细胞中,获得侵染菌。待移栽的拟南芥大部分准备第一次开花时,即为拟南芥侵染最佳时期。为了促进拟南芥有花枝的增生,可修剪提前开花的花苞;同时将已经有的角果剪去,提高转基因种子的阳性率。转化方法如下:将侵染菌进行扩大培养至OD600值为0.8。同时配置转化Buffer溶液(处理液):1L的Buffer溶液需要2.3g的MS,50g蔗糖,0.5g的MES,用NaOH调节pH值到5.8。处理液使用前还需加入表面活性剂。取搅拌后的混合Buffer溶液悬浮离心得到的菌块,即得到侵染溶液。
浸染拟南芥花序:拟南芥抽薹约10厘米高时,用农杆菌浸染液浸湿拟南芥花序,重复2-3次,用保鲜膜包裹住植株地上部分,在黑暗下培养2-3天。一个礼拜后,再次重复浸染。约两个月,收获成熟拟南芥的种子(T0代),干燥后置于4℃下保存。
2.4.5转基因拟南芥的筛选
将上述收获的拟南芥种子(T0代)经消毒(用12%的花王漂白水漂洗9分钟,然后用灭菌的蒸馏水冲洗5次,每次清洗时间约1分钟),4℃春化两天(注意避光),种子均匀洒在含潮霉素的MS培养基平板上,同时以野生型拟南芥种子作为对照。如果野生型拟南芥种子在不含抗性的MS平板上能够正常萌发生长,在含抗性的平板上均不能正常萌发生长,而转基因拟南芥中却有种子在含抗性的平板上能够正常萌发生长,则认为筛选阳性拟南芥植株是有效的。
以ZmDi19-5基因为目的基因,对筛选的阳性植株进行PCR分子检测,初步鉴定转基因拟南芥是否转化成功。
2.4.6转基因拟南芥后代的获得
按照步骤2.4.5的方法,将初步鉴定为转基因拟南芥植株的种子(T1代)用含潮霉素的MS培养基平板进行筛选,获得T1代植株,待T1代植株果荚成熟时,收获转基因T2代种子,以此类推,最终获得了稳定遗传的转基因拟南芥种子(至少T3代)。
2.5ZmDi19-5转基因拟南芥的耐盐性分析
2.5.1ZmDi19-5转基因拟南芥幼苗期的耐盐性分析
将稳定遗传的转基因拟南芥L1、L2、L5株系种子和野生型WT株系种子消毒后,均匀铺在含有MS培养基的平板上,4天左右(根生长约2-3厘米)将其移至含0、130mM NaCl的MS平板上,将这些平板竖直放于22℃人工温室中,观察根生长情况。
结果如图4所示,图中可以看出,盐胁迫条件下,转基因拟南芥植株的根系要比野生型的根系长,说明ZmDi19-5基因的过量表达增强了转基因植物对高盐的非生物胁迫。
2.5.2ZmDi19-5转基因拟南芥成苗期的耐盐性分析
将稳定遗传的转基因拟南芥L1、L2、L5株系种子和野生型WT株系种子消毒后,均匀铺在含有MS培养基的平板上,放在4℃冰箱里春化2天(便于同步发芽)。然后,置于条件为22℃,16/8h光/暗培养的拟南芥专用培养温室里培养。萌发10天左右,选取大小一致的转基因株系和野生型幼苗移入营养土和蛭石体积比为1:3混合的营养土中生长。使用220mM的NaCl溶液处理3周龄的转基因拟南芥和野生型拟南芥,连续处理4周,作为试验组,对照组用水代替220mM的NaCl溶液(正常生长)。
2.5.2.1表型分析
观察处理前后拟南芥的表型,结果发现,NaCl溶液处理之前,ZmDi19-5转基因植株和野生型拟南芥的生长状态一致,但是用220mM NaCl处理3周后,野生型拟南芥的大部分叶片则发白枯萎,表现出对高盐胁迫的敏感性,而ZmDi19-5转基因拟南芥植株的大部分叶片仍然保持绿色,表明ZmDi19-5基因的表达能增强植物对高盐的胁迫。
进一步地,统计正常生长条件下和盐胁迫条件下野生型和转基因拟南芥的株高,结果如图5所示,图中可以看出,正常生长的野生型与转基因拟南芥的株高和大小差不多,但经过盐胁迫处理后,野生型显著低于转基因植株,进一步表明过量表达的ZmDi19-5基因增强了拟南芥植株对高盐的胁迫。
2.5.2.2丙二醛(MDA)含量的测定:
(1)称取实验组和对照组同等重量的组织,加入磷酸钠缓冲液并借助于石英砂,研磨成组织匀浆;
(2)取匀浆于离心管中,离心15min,观察匀浆是否分层;
(3)取分层的匀浆上清液于新的试管中,先加入3ml的0.5%硫代巴比妥酸混匀,再加入5%三氯乙酸溶液,摇晃混匀,水浴锅(100℃)10min,并用清水冲洗试管以快速降温,然后离心,以去离子水为对照,测量离心上清在532nm、600nm波长下吸光值A532、A600。
(4)数据的处理按照下面运算公式:
式中,A为吸光度,V1为反应液总量,V为提取液总量,V2为反应液中的提取液量,W为植物样品的重量,1.55×10-1为丙二醛的微摩尔吸光系数。
结果如图6所示,图中可以看出,处理后转基因株系和野生型植株的丙二醛含量都有得到增加,但在上升幅度上,野生型植株明显高于转基因植株,说明转基因植株中积累的丙二醛含量较少,细胞受伤程度较轻。
2.5.2.3相对电导率(REL)的测定
(1)取处理前后的实验组和野生型WT各株系同位置叶片,用ddH2O冲洗两次,打孔叶片10-15个圆片;
(2)各组叶片分别放入试管中,同时选用纱布和透气封口胶带将试管罩住并固定,抽取试管中空气,30min;
(3)试管放于摇床内1h,校准完成的电导仪测量初电导值K1;
(4)再将包含样品试管于水浴锅(100℃)煮沸16min,。沸水浴后用自来冷冲洗试管至室温,室温下静置10min后,摇匀后测其终电导值K2;
(5)以ddH2O作为对照,电导仪测量值为空白电导值K0;
(6)相对电导率的计算可按照下面公式计算:
结果如图7所示,图中可以看出,处理前各组植株相对电导率的是无明显差异的,处理后野生型与转基因拟南芥的相对电导率均显著升高,但转基因拟南芥的升高幅度显著低于野生型,说明转基因拟南芥比野生型拟南芥的叶细胞间的电解质含量少,膜受损情况较轻。
2.6ZmDi19-5转基因拟南芥的干旱性分析
将稳定遗传的转基因拟南芥L1、L2、L5株系种子和野生型WT株系种子消毒后,均匀铺在含有MS培养基的平板上,放在4℃冰箱里春化2天(便于同步发芽)。然后,置于条件为22℃,16/8h光/暗培养的拟南芥专用培养温室里培养。萌发10天左右,选取大小一致的转基因株系和野生型幼苗移入营养土和蛭石体积比为1:3混合的营养土中生长。待幼苗有活力时,进行缺水干旱处理15天,再复水5天。
2.6.1表型分析
观察表型,结果发现,干旱处理15天后,野生型拟南芥植株叶片发黄并且严重卷曲,而转基因拟南芥叶片大部分还是绿色,且转基因拟南芥叶片比野生型叶片展开程度大;复水5天后,野生型全部死亡,而转基因植株全部恢复正常生长。表明过量表达的ZmDi19-5基因增强了转基因拟南芥植株对干旱胁迫的适应性和耐受性。
2.6.2过氧化氢酶(CAT)测定
CAT测定采用南京建成生物研究所购买的过氧化氢酶(CAT)测定试剂盒(可见光法)和总蛋白(TP)测定试剂盒(考马斯亮蓝法)进行测定,具体方法参照试剂盒说明书进行,具体为:
分别取干旱胁迫处理前后和野生型WT各实验组株系同一叶位的莲座叶,用ddH2O冲洗两次,除去叶面上的土壤和杂质。
(1)组织样本的检测
a.液氮预冷研钵和研磨棒,加入样品研磨至粉末状,称取1g粉末,并加入9倍体积的生理盐水,由此得到的组织匀浆,并于离心机2500rpm离心10分钟,取上清。
b.取5ml离心管按下面体积加入相应的试剂,其中,测定管添加组织原浆,对照管以水代替:
混匀,水浴锅中37℃温浴1min,再按下表加入相应体积的试剂三、试剂四:
c.摇匀各试剂的混样,波长405nm,测定各管吸光度值,双蒸水调零。
(2)蛋白定量测试
a.取(1)中a.步上清,取上清液用生理盐水按1:9比例稀释成1%浓度组织匀浆,待测;
b.将待测组织匀浆加入5ml离心管中,并按下表体积加入相应体积的试剂。其中,考马斯亮蓝显色液需要现配现用,配置方法:将考马斯亮蓝贮备液和双蒸水按1:4的比例混匀。注意:0.563/L蛋白标准品(ml)需放-20℃保存,可保存6个月。
混匀各试剂成分,室温放置10min,测量前双蒸水调零,测量各试管样品在调波长595nm的吸光值。
c.实验组样本蛋白浓度计算公式如:
本实施例中,标准品浓度为0.563g/L。
(3)组织中CAT活力的计算:
为了准确分析各组数据,我们选用SPSS10.0软件完成本部分数据的处理和统计分析。差异显著性t检验是基于P<0.01(**)和P<0.05(*)两个水平上进行。
在逆境条件下,植株会产生过多的活性氧如过氧化氢(H2O2),影响植株的生长发育,但是植株体内的过氧化氢酶能消除体内过多的过氧化氢,保护细胞免受过氧化氢的毒害。本发明对转基因株系和野生型植株叶片中的氧化氢酶活力进行测定,结果如图8所示,图中可以看出,干旱处理后,转基因植株和野生型植株的过氧化氢酶活力均有所下降,但野生型中的过氧化氢酶活力极显著低于转基因植株中的活力,说明野生型植株的细胞活力明显没有转基因植株的活力大。
2.6.3丙二醛含量检测
检测方法同步骤2.5.2.2,结果如图9所示,图中可以看出,转基因植株和野生型植株的丙二醛含量均有所上升,但转基因植株的丙二醛含量极显著低于野生型植株,说明野生型植株的细胞受伤程度较大;
2.6.4叶片相对含水量(RWC)测定
(1)分别取干旱处理前、后的转基因植株和对照植株相同部位叶片。
(2)用ddH2O冲洗两次,除去叶面上的土壤和杂质,再用滤纸吸尽表面水分,称鲜重W0;
(3)取称重后叶片浸泡在去离子水中5-6h,直至吸水饱和重量不变时,称鲜重W1;
(4)将(3)中称重的叶片用锡箔纸包好,放入180℃的烘箱,烘干水分,称干重W2;
(5)相对含水量的计算公式:
结果图如10所示,转基因植株和野生型植株的相对含水量均有所下降,但转基因植株的相对含水量极显著高于野生型植株,说明野生型植株的细胞已大部分死亡。
2.7结论
野生型植株受到盐胁迫和干旱胁迫程度均大于转基因植株,说明ZmDi19-5基因能明显提高转基因植物的抗逆性。
以上为本发明一种详细的实施方式和具体的操作过程,是以本发明技术方案为前提下进行实施,但本发明的保护范围不限于上述的实施例。
SEQUENCE LISTING
<110> 安徽农业大学
<120> 一种玉米抗逆相关基因ZmDi19-5及其应用
<130> /
<160> 4
<170> PatentIn version 3.3
<210> 1
<211> 720
<212> DNA
<213> Zea mays
<400> 1
atggattccg acctctggat ctcgcgtctc acggccgcca agcggcagtt tgcgctgcag 60
cgcgcgcagc gccagcacgc cgccccggca tcccatcacg atcgatttgg gtatgatgat 120
atcgaacctg aggatgattt gcactcagac ttcccatgcc cttactgtta tgaagatcat 180
gatgttgctt ctctctgcgc ccatttggag gacgagcacc ctttcgagtc taaaattgtg 240
tcttgcccag tttgctcagc taggatttcg aaggacttgg tggatcatat aacccttcag 300
catggctacc tatttaaatt gcagaaacat caaagagtac gcagagttac tggtaatggc 360
aaccacaatc tctcctatgc agggagagat cttcaactgc aagagaccta cttaaaggtg 420
cttcttggaa atagcagtcg aagcagcagt accaatgcat caagtacagt caccgattca 480
ctgctaccct cactagtttt aaatttatca tcaccagaag tagaagacgc atcaaagttt 540
tcagctcctg ctgtggtgga aaataattgg tttaaacgat cactgccttc aaaaacttgg 600
aaattaagaa ctgttgattc cacccttagt catgaagaga gggagcgtag gaggaggaga 660
gctgctgtga gatcagcttt tgtccagcat ctccttgtca ccaccatttt tgaagactag 720
<210> 2
<211> 239
<212> PRT
<213> Zea mays
<400> 2
Met Asp Ser Asp Leu Trp Ile Ser Arg Leu Thr Ala Ala Lys Arg Gln
1 5 10 15
Phe Ala Leu Gln Arg Ala Gln Arg Gln His Ala Ala Pro Ala Ser His
20 25 30
His Asp Arg Phe Gly Tyr Asp Asp Ile Glu Pro Glu Asp Asp Leu His
35 40 45
Ser Asp Phe Pro Cys Pro Tyr Cys Tyr Glu Asp His Asp Val Ala Ser
50 55 60
Leu Cys Ala His Leu Glu Asp Glu His Pro Phe Glu Ser Lys Ile Val
65 70 75 80
Ser Cys Pro Val Cys Ser Ala Arg Ile Ser Lys Asp Leu Val Asp His
85 90 95
Ile Thr Leu Gln His Gly Tyr Leu Phe Lys Leu Gln Lys His Gln Arg
100 105 110
Val Arg Arg Val Thr Gly Asn Gly Asn His Asn Leu Ser Tyr Ala Gly
115 120 125
Arg Asp Leu Gln Leu Gln Glu Thr Tyr Leu Lys Val Leu Leu Gly Asn
130 135 140
Ser Ser Arg Ser Ser Ser Thr Asn Ala Ser Ser Thr Val Thr Asp Ser
145 150 155 160
Leu Leu Pro Ser Leu Val Leu Asn Leu Ser Ser Pro Glu Val Glu Asp
165 170 175
Ala Ser Lys Phe Ser Ala Pro Ala Val Val Glu Asn Asn Trp Phe Lys
180 185 190
Arg Ser Leu Pro Ser Lys Thr Trp Lys Leu Arg Thr Val Asp Ser Thr
195 200 205
Leu Ser His Glu Glu Arg Glu Arg Arg Arg Arg Arg Ala Ala Val Arg
210 215 220
Ser Ala Phe Val Gln His Leu Leu Val Thr Thr Ile Phe Glu Asp
225 230 235
<210> 3
<211> 28
<212> DNA
<213> 人工序列
<400> 3
ggggtaccat ggattccgac ctctggat 28
<210> 4
<211> 25
<212> DNA
<213> 人工序列
<400> 4
cgggatccct agtcttcaaa aatggtggtg 30
- 还没有人留言评论。精彩留言会获得点赞!