早发性结直肠癌辅助诊断的遗传生物标志物及其应用的制作方法
技术领域:
本发明属于基因工程及肿瘤学领域,涉及与早发性结直肠癌辅助诊断相关的遗传生物标志物(snps)及其应用。
背景技术:
:
结直肠癌是全球范围内常见的消化系统恶性肿瘤,每年新诊断病例接近600,000例。据世界卫生组织(who)最新资料显示,全球结直肠癌发病率排位占男性第四位,女性第三位。近年来,我国结直肠癌的发病率和死亡率均呈不断上升趋势。流行病学研究表明,早发性结直肠癌(诊断年龄≤50岁)约占结直肠癌总发病的2%~8%,且预后转归较差。因此,早发性结直肠癌受到研究者更多关注。
目前,针对结直肠癌发生的危险因素已有多项研究报道。研究认为,结直肠癌是由不良饮食(如高脂饮食)、不良生活方式(如久坐)以及遗传特征等因素共同作用的结果。其中,遗传因素对结直肠癌的发生起着不可忽视的关键作用。研究表明,若近亲中有一人为结直肠癌者,其本人患此癌的危险度约为正常人的2倍。因此,我们着重关注个体罹患结直肠癌的遗传风险评估,早期识别高危人群,进而实现结直肠癌的早期预防、早期诊断和早期治疗。
近十年来,随着全基因组关联性研究(genome-wideassociationstudy,gwas)的不断深入,越来越多的结直肠癌相关的单核苷酸多态性位点(singlenucleotidepolymorphism,snp)被人们发现,从而为学者在遗传水平制定结直肠癌风险预测模型奠定基础。遗传风险评分(geneticriskscore,grs)是流行病学疾病风险研究中评价遗传风险的最常用方法之一。纳入遗传风险位点构建grs模型,是评估目标人群结直肠癌遗传风险的有效手段。
然而,目前尚未见grs模型应用于早发性结直肠癌的遗传风险预测的研究报道。若能筛选出与早发性结直肠癌发生密切相关的风险位点,构建grs风险预测模型,将有利于对目标人群进行早发性结直肠癌的遗传风险评定。本专利将制备相应的辅助诊断试剂盒,对我国早发性结直肠癌的早期诊断发挥极大的推动作用。
技术实现要素:
:
解决的技术问题:本发明的目的之一是针对上述研究背景,提出一种早发性结直肠癌辅助诊断的遗传生物标志物及其应用。
本发明的第二个目的是构建grs风险预测模型,对目标人群进行早发性结直肠癌的遗传风险评定。
本发明的第三个目的是提供上述snp标志物的特异性引物及其在制备早发性结直肠癌辅助诊断试剂盒中的应用。
本发明的第四个目的是提供早发性结直肠癌辅助诊断试剂盒。
发明人通过开展早发性结直肠癌病例对照研究,鉴定早发性结直肠癌患者及其健康对照外周血dna中的风险snps位点,同时采用医学统计学相关方法,在调整年龄、性别等相关混杂因素后,最终筛选出一组与早发性结直肠癌密切相关的风险snps位点;在此基础上,构建grs模型,评价风险snps位点对早发性结直肠癌发病风险预测能力;最终制备早发性结直肠癌辅助诊断试剂盒,为早发性结直肠癌的早期诊断和预测提供技术支持。
技术方案:早发性结直肠癌辅助诊断的遗传生物标志物,所述标志物为rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530的组合。
上述的早发性结直肠癌辅助诊断的遗传生物标志物的特异性扩增引物,引物分别为:rs10904012的引物序列为seqidno.1和seqidno.2;rs12635144的引物序列为seqidno.5和seqidno.6;rs2964283的引物序列为seqidno.9和seqidno.10;rs3747926的引物序列为seqidno.13和seqidno.14;rs59306779的引物序列为seqidno.17和seqidno.18;rs6135530的引物序列为seqidno.21和seqidno.22。
上述的早发性结直肠癌辅助诊断的遗传生物标志物的特异性探针引物,探针引物分别为:rs10904012的引物序列为seqidno.3和seqidno.4;rs12635144的引物序列为seqidno.7和seqidno.8;rs2964283的引物序列为seqidno.11和seqidno.12;rs3747926的引物序列为seqidno.15和seqidno.16;rs59306779的引物序列为seqidno.19和seqidno.20;rs6135530的引物序列为seqidno.23和seqidno.24。
上述早发性结直肠癌辅助诊断的遗传生物标志物在制备早发性结直肠癌辅助诊断试剂盒的应用。
上述早发性结直肠癌辅助诊断的遗传生物标志物在构建grs模型中的应用。
上述早发性结直肠癌辅助诊断的遗传生物标志物的特异性扩增引物在制备早发性结直肠癌辅助诊断试剂盒的应用。
上述早发性结直肠癌辅助诊断的遗传生物标志物的特异性探针引物在制备早发性结直肠癌辅助诊断试剂盒的应用。
早发性结直肠癌辅助诊断试剂盒,含有rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530组合的特异性扩增引物和特异性探针引物。
上述试剂盒还包括pcr常用的试剂以及标准品和对照品。
总体来说,本发明的技术方案包括:
(1)确定纳入标准和排除标准,系统收集符合标准的血液样本;
(2)基因型检测:选择早发性结直肠癌病例及其健康对照,利用全基因组高密度snp芯片和taqman探针基因分型法,鉴定出与早发性结直肠癌发病风险相关的snps;
(3)基于鉴定出具有风险效应的snps构建grs模型,利用受试者工作特征曲线(roc)判断模型的预测精度,以检测风险snps位点对早发性结直肠癌风险预测能力;
(4)早发性结直肠癌辅助诊断试剂盒的制备:基于上述筛选出的snp遗传标志物构建辅助诊断试剂盒。
实验方法:
1、明确研究对象
(1)经病理确诊的早发性结直肠癌病例;
(2)发病年龄≤50岁。
本研究共纳入687个病例和3650个对照进行风险位点的鉴定。
2、酚-氯仿法提取外周血基因组dna,按常规方法操作。通常能得到20-50ng/μldna,纯度(紫外260od与280od比值)在1.6-2.0。
3、全基因组snps位点检测
(1)取受试者全基因组dna样本,包括397个病例和1795个对照;
(2)利用illuminahumanomnizhonghuabeadchips芯片进行全基因组snps位点扫描;
(3)通过拟合logistic回归分析,比较各基因型在早发性结直肠癌病例与对照中的分布差异。
4、单个snp的taqmanmgb探针法基因分型
(1)取受试者全基因组dna样本,包括281个病例和1855个对照;
(2)设计单个snp的特异性扩增引物和探针引物;
(3)pcr扩增反应;
(4)通过拟合logistic回归分析,比较早发性结直肠癌与健康对照中不同基因型的分布差异。
5、构建grs模型
(1)基于上述全基因组扫描和单个snp检测策略,通过拟合logistic回归模型鉴定出与早发性结直肠癌发病风险密切相关snps位点,包括rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530。
(2)以现有回归系数作为权重,构建加权遗传风险得分模型(wgrs模型)。
(3)利用wgrs模型对总人群样本进行风险评估,利用roc曲线对模型的区分效能进行评价,以检测鉴定出的风险snps对早发性结直肠癌发病风险的预测能力。
6、早发性结直肠癌诊断试剂盒的制备
诊断试剂盒由上述筛选出的snps(rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530)构成。诊断试剂包括所述的snp遗传标志物的特异性扩增及探针引物;还包括pcr常用的试剂,如taq酶,dntp混合液,去离子水等;以及标准品和对照品。
7、统计学分析
研究对象的人口学特征(如性别、年龄)在病例和对照中的差异由卡方检验及studentt检验进行分析。采用logistic回归的相加模型分析snps和早发性结直肠癌发病风险的关联强度,同时调整混杂变量(如性别、年龄)。
通过计算wgrs得分来拟合grs风险预测模型。基本步骤包括:
(1)每个snp的三种基因型进行定量评分,如野生纯合型为“0”,杂合型为“1”,纯合突变型为“2”,且所有位点的效应评分均调整为正向关联;
(2)每个snp的权重系数即为logistic回归模型中获取的回归系数,构建方程式为:wgrs=rs12635144×0.378+rs2964283×0.654+rs59306779×0.653+rs10904012×0.397+rs3747926×0.403+rs6135530×0.477;
(3)利用roc曲线和计算auc值来评定grs模型的区分效能。
所有统计学分析采用plink1.07和r3.3.2软件完成。统计学显著性p值为0.05,且均为双侧检验。
结果说明:
(1)在已有的687个病例和3650个对照中,年龄存在显著性差异,性别频率分布无差异性。
(2)经全基因组snps扫描和taqman探针基因分型,最终鉴定出6个snps与早发性结直肠癌存在显著关联性,包括rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530。其中与疾病发生呈现正向关联的有:rs10904012,rs12635144和rs6135530;负性关联的有rs2964283,rs3747926和rs59306779。见表1。
表1.6个风险snps位点与结直肠癌易感性的关联
a哈迪-温伯格平衡检验
b两阶段合并结果,调整协变量(年龄和性别)
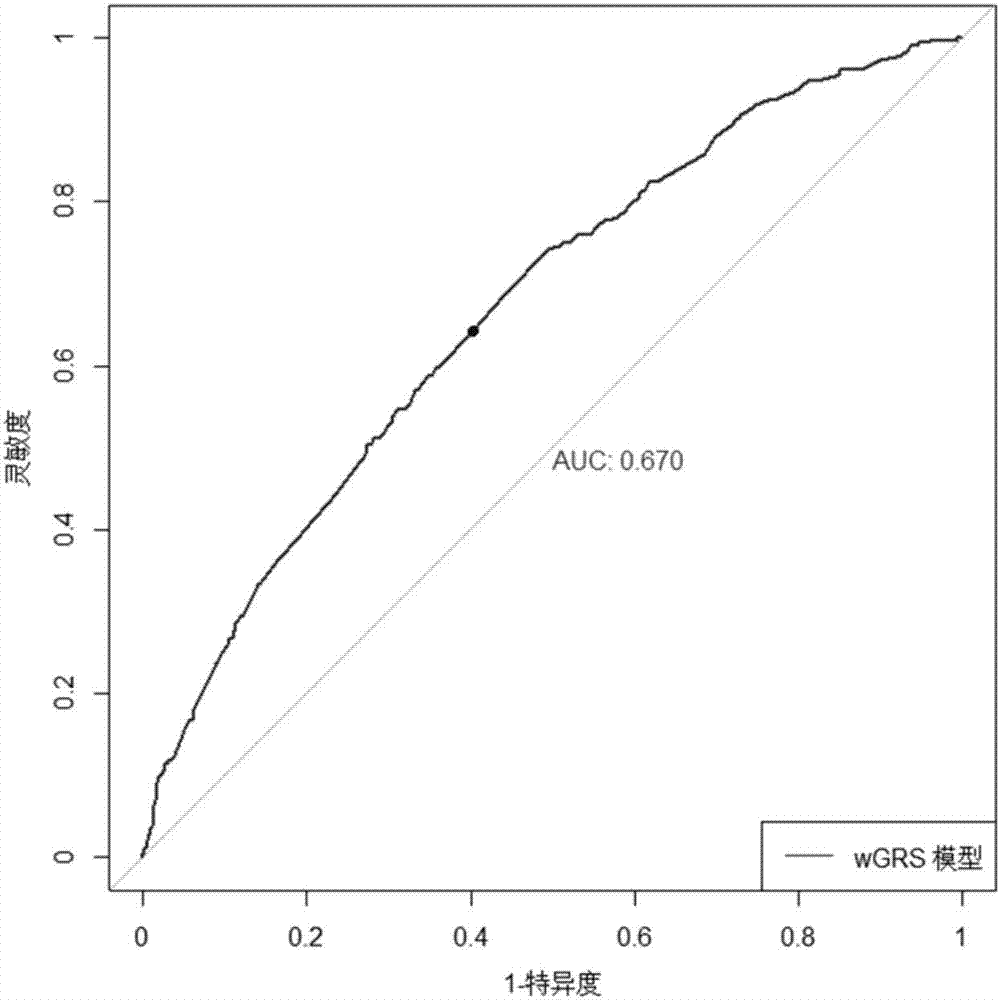
(3)进一步通过拟合wgrs对在现有总人群样本中进行风险预测,通过roc曲线和计算auc值检测模型效能,发现该6个snps组合能够很好地区分病例和对照,auc=0.670,p=3.81e-26(见图1和2)。
基于上述结果,本发明人制备了早发性结直肠癌辅助诊断试剂盒,包含鉴定的6个snps的特异性引物及其他试剂。
总的来说,该6个snps的特异性引物制备的早发性结直肠癌辅助诊断试剂盒有助于临床医生快速掌握患者发生疾病的遗传风险,为及时采取治疗措施提供有力的支持。
有益效果:(1)snp遗传标志物区别于传统的生物标志物,稳定性好,易于检测。(2)早发性结直肠癌辅助诊断试剂盒有助于帮助临床医生了解患者的疾病状态,为采取有效的防治措施提供支持。
附图说明
图1:描述wgrs评分在病例和对照中的分布图。
图2:显示早发性结直肠癌病例组和对照组的roc曲线图。
具体实施方式
以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改和替换,均属于本发明的范围。
若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段。
实施例1
样品的收集和资料的整理
发明人于2010年至2016年从南京医科大学附属医院收集结直肠癌的血液样本,并进行整理,从中包含687例早发性结直肠癌(发病年龄≤50岁)样本,以及同时在医院健康体检的对照3650例,并采集相关的流行病学和临床资料。
外周血dna中snps的全基因组扫描
在符合要求的397个病例及1795个对照中,通过illuminahumanomnizhonghuabeadchips芯片检测获得相应结果。具体步骤如下:
(1)裂解液的配置:配置40份量,即219.72g蔗糖、2.02g氯化镁、20ml曲拉通x-100混合,并采用trishcl溶液定容至2000ml。
(2)于2ml冻存管中的外周血加入裂解液,颠倒混匀。
(3)红细胞的去除:用裂解液将5ml离心管补至4ml刻度,混匀,4000rpm离心10分钟,弃去上清液。并向沉淀中加入4ml裂解液,再次混匀、清洗一次,4000rpm离心10分钟,弃去上清液。
(4)配置抽提液:每300ml中含有122.5ml0.2m氯化钠,14.4ml0.5m乙二胺四乙酸,15ml10%十二烷基硫酸钠及148.1ml双蒸水。
(5)dna提取:向得到的沉淀中加入1ml抽提液和8μl蛋白酶k,震荡器上充分震荡混匀,37℃水浴过夜。
(6)蛋白质去除:加入1ml饱和酚,并充分混匀,4000rpm离心10分钟,取上清液转入新的5ml离心管中。在上清液中加入等体积氯仿与异戊醇混合液(氯仿:异戊醇=24:1,v/v),充分混勾后,4000rpm离心10分钟,取上清液,分别放入两个1.5ml的离心管。
(7)dna沉淀:在上清液中加入60μl3m的醋酸钠,并加入与上清液等体积的冰无水乙醇,上下轻摇,可见白色絮状沉淀物,再以12000rpm离心10min。
(8)dna洗绦:在沉淀中加入冰无水乙醇1ml,12000rpm离心10min,弃去上清液。最后置于清洁干燥环境中蒸干。
(9)测量浓度:通常能得到20-50ng/μldna,纯度(紫外260od与280od比值)在1.6-2.0。
(10)全基因组扫描:在illuminahumanomnizhonghuabeadchips芯片上进行全基因组扫描。
(11)数据分析与处理:通过拟合logistic回归模型,在早发性结直肠癌病例组和健康对照组中筛选出有显著差异的snps。
单个snp的taqman探针法基因分型
在符合要求的281个病例及1855个对照中进行相应检测。
(1)dna提取方法同上。
(2)taqman探针法基因分型:
①针对已筛选出的6个snps位点设计探针和引物,均符合探针及引物要求,且能分型成功。
②引物稀释为10倍的工作浓度,探针稀释为20倍的工作浓度。
③制备real-timepcr反应体系:每份用量5μl,其中包括无菌双蒸水1.25μl、1×qpcrmix(thunderbirdprobeqpcrmix,购自toyobd公司)2.5μl、上下游引物(1pmol/μl)各0.25μl、fam探针(0.25pmol/μl)0.125μl、hex探针(0.25pmol/μl)0.125μl、1.25×rox溶液(0.25pmol/μl)0.125μl和dna模板(10ng/μl)0.5μl。
④将配好的real-timepcr反应体系加入384孔板中(axygen),并进行封膜处理。
⑤snps分型:将封好膜的384孔板置于abiprism7900ht型荧光定量pcr仪进行分型,实验程序如下:
1)95℃,2min,使酶活化;
2)95℃,15s,使dna变性;
3)60℃,60s,使引物和探针退火及延伸,共40个循环。
⑥利用sds2.4软件进行基因分型。其中,蓝色和红色分别表示两种不同的纯合子,绿色代表杂合子,灰色代表阴性对照(ntc),×代表分型失败。
(3)数据分析与处理:通过拟合logistic回归模型,结合全基因组阶段和二阶段的结果,鉴定候选snps的关联性强度,结果见表1。
利用wgrs构建遗传风险预测模型
基于上述鉴定的6个风险snps位点,以拟合的logistic回归系数作为权重,构建wgrs模型,利用roc曲线对其评估,进而评价风险位点对早发性结直肠癌发生风险的预测能力。结果表明,鉴定出的6个snps(rs10904012,rs12635144,rs2964283,rs3747926,rs59306779和rs6135530)能够较好地区分早发性结直肠癌病例和对照(见图1),其grs模型的roc曲线下面积为66.98%,最佳临界点的灵敏度为64.30%,特异度为59.76%(见图2),因此,能够较好地预测疾病发生的遗传风险。
制备早发性结直肠癌辅助诊断试剂盒
试剂盒含有一批snps特异性扩增引物和特异性探针引物(见表2),还包括pcr常用的试剂,如taq酶,dntp混合液,去离子水等。以及标准品和对照品。该试剂盒相对于其他诊断仪器,具有精简,稳定性高,能够预测疾病状态和遗传风险的优势。因此建议将此试剂盒投入临床工作,帮助高危人群进行早期预防。
表2.snp位点分型引物信息
f:forwardprimer,上游引物;r:reverseprimer,下游引物;fam和hex:不同等位基因的荧光信号。
sequencelisting
<110>南京医科大学
<120>早发性结直肠癌辅助诊断的遗传生物标志物及其应用
<130>
<160>24
<170>patentinversion3.3
<210>1
<211>25
<212>dna
<213>人工序列
<400>1
cctcagagccatgtagaaaaagcta25
<210>2
<211>16
<212>dna
<213>人工序列
<400>2
ccggccttgcgtgttg16
<210>3
<211>16
<212>dna
<213>人工序列
<400>3
ctggtctgtcagagga16
<210>4
<211>16
<212>dna
<213>人工序列
<400>4
tggtctatcagaggaa16
<210>5
<211>24
<212>dna
<213>人工序列
<400>5
actcaagtcctatcatagctacct24
<210>6
<211>25
<212>dna
<213>人工序列
<400>6
ggaaagtgtgagatgagataatgca25
<210>7
<211>18
<212>dna
<213>人工序列
<400>7
acagtgtggtgtctctac18
<210>8
<211>15
<212>dna
<213>人工序列
<400>8
agggacagtgcggtg15
<210>9
<211>27
<212>dna
<213>人工序列
<400>9
gccctgtttaggacacaattcttatag27
<210>10
<211>27
<212>dna
<213>人工序列
<400>10
tgagttggaggtgctttaatatgtatg27
<210>11
<211>19
<212>dna
<213>人工序列
<400>11
cagaatgtctagaaatgta19
<210>12
<211>19
<212>dna
<213>人工序列
<400>12
cagaatgtctagaaatata19
<210>13
<211>28
<212>dna
<213>人工序列
<400>13
tccaattttactaaaggatacagcactt28
<210>14
<211>23
<212>dna
<213>人工序列
<400>14
gaaatggactttcatcctggaaa23
<210>15
<211>22
<212>dna
<213>人工序列
<400>15
cattttcaatgttttattttta22
<210>16
<211>20
<212>dna
<213>人工序列
<400>16
attttcaatgttttcttttt20
<210>17
<211>24
<212>dna
<213>人工序列
<400>17
tgactttcctcatgctccattatg24
<210>18
<211>20
<212>dna
<213>人工序列
<400>18
aaccaaggcagaggcagtgt20
<210>19
<211>16
<212>dna
<213>人工序列
<400>19
cccagtctctctggaa16
<210>20
<211>16
<212>dna
<213>人工序列
<400>20
ccccagtctctctaga16
<210>21
<211>26
<212>dna
<213>人工序列
<400>21
ccaactgaaacagtcacctatcttaa26
<210>22
<211>24
<212>dna
<213>人工序列
<400>22
tttgaccataatccctgtttctca24
<210>23
<211>15
<212>dna
<213>人工序列
<400>23
accttagtcttcccc15
<210>24
<211>15
<212>dna
<213>人工序列
<400>24
accttagtctccccc15
- 还没有人留言评论。精彩留言会获得点赞!