用于生产3‑羟基丙酸的组合物和方法与流程
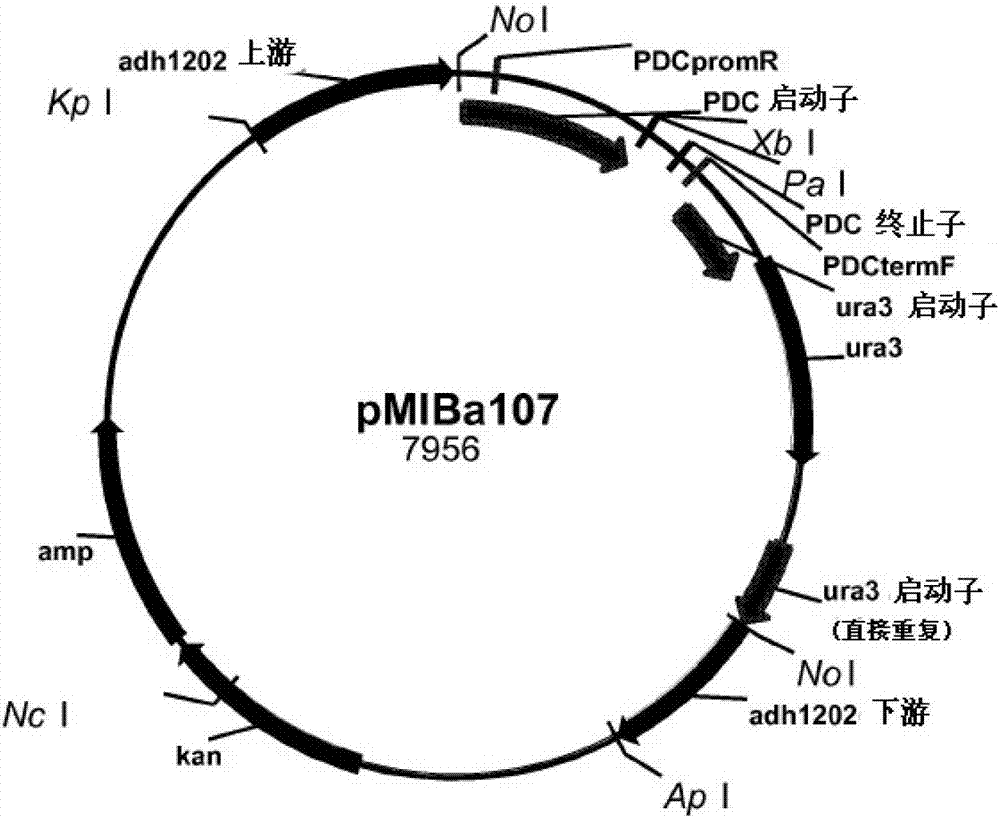
本发明申请是基于申请日为2011年11月21日、申请号为201180065738.0(国际申请号为pct/us2011/061717)、名称为“用于生产3-羟基丙酸的组合物和方法”的发明专利申请的分案申请。序列表的引用本申请含有计算机可读取形式的序列表,其通过提述并入本文。
背景技术:
:3-羟基丙酸(3-hp)是一种3碳羧酸,其由u.s.departmentofenergy鉴定为12种高潜力的建造模块(buildingblock)化学品之一,可以通过发酵制备。3-hp,一种乳酸(2-羟基丙酸)的异构体,的其它名称是包括亚甲基乳酸(ethylenelacticacid)和3-羟基丙酸根(3-hydroxypropionate)。3-hp是有吸引力的可再生的平台化学品,具有由葡萄糖生产的100%理论产率、使其参与多种化学反应的多个功能基团以及低毒性。3-hp可以用作底物,以形成几种商品化学品,例如1,3-丙二醇、丙二酸、丙烯酰胺和丙烯酸。丙烯酸是大体积的化学品(>70亿lbs/年),其用于制备丙烯酸酯和具有超强吸收能力的聚合物,且目前来源于丙烯的催化性氧化。3-hp的发酵生产将提供可持续的石油化学品替代物,作为这些商业上重要的化学品的原料,因而减少了能量消耗、美国对外来原油的依赖性和温室效应气体的产生。细菌可以用于将糖发酵成有机酸。然而,对于大规模的有机酸生产,细菌显示出某些缺点。随着有机酸产生,发酵培养基变得酸性越来越强。事实上,较低的ph条件是优选的,因为所得产物部分或完全是酸性形式的。然而,产生有机酸的大部分细菌在强酸环境中表现不佳,因此或者死亡或者开始产生缓慢以致其随着培养基变得酸性更强而变得经济上不可行。为了防止这种情况,需要对培养基进行缓冲以维持较高的ph。然而,这使得有机酸产物的回收更加困难和昂贵。近些年来,围绕利用酵母将糖发酵成有机酸的兴趣日益增加。酵母在一些工业发酵中用作生物催化剂,并显示出超越细菌的几项优势。尽管很多细菌不能合成其生长和有效代谢糖类所需要的某些氨基酸或蛋白,但大多数酵母菌种能够由无机氮化合物合成其必需的氨基酸或蛋白。酵母还不易受到在细菌中导致生产力或整体发酵运行过程受损的细菌噬菌体感染。虽然酵母是有机酸生产的引人注目的候选者,但其显现出几项困难。首先,酵母中的途径工厂改造通常比细菌中更加困难。酵母中的酶被分隔在胞质、线粒体或过氧化物酶体中,而在细菌中其汇集于胞质中。这意味着可能需要去除靶向信号,以确保生物合成途径的所有酶共存于单个细胞内的相同区室中。还需要对途径中间物在区室之间的转运进行调控,以使到达目标产物的碳流最大化。其次,并非所有的酵母菌种符合大规模经济发酵的必要标准。事实上,仅小部分酵母具有足够高的体积和特定的糖利用与在低ph条件下快速生长的能力的结合。u.s.departmentofenergy已经估测,大约2.5g/l/小时的生长速率对于几种有机酸的经济发酵来说是必需的,包括3-hp(http://www1.eere.energy.gov/biomass/pdfs/35523.pdf)。虽然很多酵母菌种天然地将己糖发酵为乙醇,几乎没有任何酵母天然地产生显著产量的有机酸。这导致尝试对多种酵母菌种进行遗传修饰以生产有机酸。先前已经通过破坏或缺失天然的丙酮酸脱羧酶(pdc)基因并插入乳酸脱氢酶(ldh)基因以消除乙醇的产生,从而开发了产生乳酸的经遗传修饰的酵母株(参见例如w099/14335、wo00/71738、wo02/42471、wo03/049525、wo03/102152和wo03/102201)。这种改变使糖代谢由产生乙醇转为产生乳酸。酵母的发酵产物和途径不同于细菌,因而需要不同的工程改造方法以使产率最大化。增加有机酸产物的产率或纯度可能需要消除或减少的其他天然产物是甘油、醋酸盐和二醇。经遗传改变的酵母株中甘油的减少描述于例如wo07/106524中。与乳酸不同,3-hp不是自然界任何已知途径的主要终产物,其在一些细菌和真菌中仅以痕量发现。因此,需要进行更多的遗传工程改造,以生成产生3-hp的酵母。先前对酿酒酵母(saccharomycescerevisiae)菌株进行了工程改造,以经由乳酸盐中间物产生非常低水平的3-hp(参见wo02/042418)。然而,野生型酿酒酵母的耐受水平不足以使其成为生产3-hp的最佳宿主。因此,需要在工业化规模上以更具成本效率的方式生产3-hp的改进的酵母菌株。技术实现要素:在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep、丙酮酸和/或甘油产生3-hp的活性3-hp发酵途径。在某些实施方案中,本文中提供的细胞含有一个或多个3-hp途径基因,所述基因编码具有ppc、pyc、aat、adc、baat、gabt、3-hpdh、hibadh、4-羟基丁酸脱氢酶、acc、aam、丙氨酸脱氢酶、醛脱氢酶、bcka、kgd、4-氨基丁酸转氨酶、β-丙氨酰-coa氨解酶、co-a酰化丙二酸半醛脱氢酶、coa合成酶、coa转移酶、甘油脱水酶、ipda、ldh、乳酰-coa脱水酶、苹果酸脱羧酶、苹果酸脱氢酶、丙二酰-coa还原酶、oaa甲酸裂合酶(formatelyase)、oaa脱氢酶、丙酮酸/丙氨酸氨基转移酶、pdh、2-酮酸脱羧酶、3-hp-coa脱水酶、3-hp-coa水解酶或3-羟基异丁酰-coa水解酶活性的酶。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep或丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有ppc、pyc、aat、adc、baat、gabt、3-hpdh、hibadh和4-羟基丁酸脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,且在这些实施方案中,所述基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。例如,细胞可以含有来源于东方伊萨酵母(i.orientalis)的酵母pyc基因或来源于类球红细菌(r.sphaeroides)、菜豆根瘤菌(r.etli)、荧光假单胞菌(p.fluorescens)、谷氨酸棒杆菌(c.glutamicum)或苜蓿中华根瘤菌(s.meliloti)的细菌pyc基因;来源于大肠杆菌(e.coli)、嗜热自养甲烷杆菌(m.thermoautotrophicum)或产气荚膜梭菌(c.perfringens)的细菌ppc基因;来源于东方伊萨酵母或酿酒酵母的酵母aat基因或来源于大肠杆菌的细菌aat基因;来源于阿维链霉菌(s.avermitilis)、丙酮丁醇梭菌(c.acetobutylicum)、幽门螺旋杆菌(h.pylori)、地衣芽孢杆菌(b.licheniformis)或谷氨酸棒杆菌的细菌adc基因;来源于东方伊萨酵母或克鲁维酵母(s.kluyveri)的酵母baat基因或来源于阿维链霉菌的细菌baat基因;来源于酿酒酵母的酵母gabt基因或来源于阿维链霉菌的细菌gabt基因;来源于东方伊萨酵母或酿酒酵母的酵母3-hpdh基因或来源于大肠杆菌或m.sedula的细菌3-hpdh基因;来源于粪产碱菌(a.faecalis)、恶臭假单胞菌(p.putida)或铜绿假单胞菌(p.aeruginosa)的细菌hibadh基因;和/或来源于克鲁维酵母的酵母4-羟基丁酸脱氢酶基因或来源于r.eutropha的细菌4-羟基丁酸脱氢酶基因。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep或丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有ppc、苹果酸脱氢酶和苹果酸脱羧酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep或丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有ppc、2-酮酸脱羧酶、kgd、bcka、吲哚丙酮酸脱羧酶、3-hpdh、hibadh或4-羟基丁酸脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep或丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有ppc、oaa甲酸裂合酶、丙二酰-coa还原酶、co-a酰化丙二酸半醛脱氢酶、3-hpdh、hibadh和4-羟基丁酸脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有pdh、酰基-coa羧化酶、丙二酰-coa还原酶、coa酰化丙二酸半醛脱氢酶、3-hpdh、hibadh和4-羟基丁酸脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有丙氨酸脱氢酶、丙酮酸/丙氨酸氨基转移酶、丙氨酸2,3氨基变位酶、coa转移酶、coa合成酶、β-丙氨酰-coa氨解酶、3-hp-coa脱水酶、3-hp-coa水解酶、3-羟基异丁酰-coa水解酶、baat、3-hpdh、hibadh和4-羟基丁酸脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有ldh、coa转移酶、乳酰-coa脱水酶、3-hp-coa脱水酶、3-hp-coa水解酶和3-羟基异丁酰-coa水解酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。在某些实施方案中,本文提供了经遗传修饰的酵母细胞,其包含由pep或丙酮酸产生3-hp的活性3-hp发酵途径,其中所述细胞含有编码具有甘油脱水酶和醛脱氢酶活性的酶的一个或多个基因。在某些实施方案中,一个或多个3-hp途径基因是外源的,在这些实施方案中,基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动物来源。本文提供的经遗传修饰的酵母细胞可以是任何酵母菌种。在某些实施方案中,细胞是crabtree阴性的,在这些实施方案的某些中,其属于伊萨酵母属(issatchenkia)、假丝酵母属(candida)、克鲁维酵母属(kluyveromyces)、毕赤酵母属(pichia)、裂殖酵母属(schizosaccharomyces)、有孢圆酵母属(torulaspora)、接合酵母属(zygosaccharomyces)或酵母属(saccharomyces)。在这些实施方案的某些中,细胞可以属于东方伊萨酵母p.fermentans进化枝或酵母属进化枝,在这些实施方案中,其可以是东方伊萨酵母、郎比可假丝酵母(c.lambica)或博伊丁酵母(s.bulderi)。在某些实施方案中,酵母细胞可以是3-hp抗性酵母细胞。3-hp抗性可以是细胞的天然特性或其是细胞在引入与活性3-hp发酵途径相关的遗传修饰之前、过程中或之后或其组合经过了突变和/或选择所致。在某些实施方案中,酵母细胞可以表现出对除了3-hp、其他发酵产物或副产物和/或多种培养基组分以外的有机酸具有高于由相同菌种的野生型酵母细胞所表现出的耐受性程度。在某些实施方案中,酵母细胞已经经过了突变和/或选择,以致经突变和/或选择的细胞具有高于相同菌种野生型细胞的3-hp抗性程度。在这些实施方案的一些中,细胞在用一个或多个外源3-hp途径基因进行遗传修饰之前已经经过了突变和/或选择。在一些实施方案中,细胞经过了乳酸或3-hp存在下的选择。在一些实施方案中,选择是恒化选择。除了与活性3-hp发酵途径相关的修饰之外,本文提供的细胞可能含有一个或多个天然基因的缺失或破坏。例如,细胞可以含有一个或多个pdc、adh、gal6、cyb2a、cyb2b、gpd、gpp、ald或pck基因的缺失或破坏。在某些实施方案中,这些缺失或破坏可以与引入与活性3-hp发酵途径相关的一个或多个基因偶联。在某些实施方案中,本文提供了利用本文提供的经遗传修饰的酵母细胞,通过在至少一种碳源存在下培养细胞并从培养基中分离3-hp从而产生3-hp的方法。在这些实施方案的某些中,碳源可以选自葡萄糖、木糖、阿拉伯糖、蔗糖、果糖、纤维素、葡萄糖寡聚物和甘油的一种或多种。具体地,本发明涉及如下各项:1.包含活性3-hp发酵途径的经遗传修饰的酵母细胞,其中细胞包含选自:外源ppc基因;外源pyc基因;外源aat基因;外源adc基因;外源baat或gabt基因;和外源3-hpdh基因的一种或多种外源3-hp途径基因。2.项1的经遗传修饰的酵母细胞,其包含外源pyc基因。3.项2的经遗传修饰的酵母细胞,其中pyc基因编码与选自seqidno:2、3、4、5、6、7和8的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。4.项2或3的经遗传修饰的酵母细胞,其中pyc基因包含与seqidno:1的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。5.项1-4任一项的经遗传修饰的酵母细胞,其包含外源aat基因。6.项5任一项的经遗传修饰的酵母细胞,其中aat基因编码与选自seqidno:14、15和16的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。7.项5或6的经遗传修饰的酵母细胞,其中aat基因包含与seqidno:13的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。8.项1-7任一项的经遗传修饰的酵母细胞,其包含外源adc基因。9.项9的经遗传修饰的酵母细胞,其中adc基因编码与选自seqidno:17、18、133、135、137和139的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。10.项8或9的经遗传修饰的酵母细胞,其中adc基因包含与选自seqidno:130、131、132、134、136和138的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。11.项1-10任一项的经遗传修饰的酵母细胞,其包含外源baat基因或外源gabt基因。12.项11的经遗传修饰的酵母细胞,其中外源baat基因或gabt基因编码与选自seqidno:20、21、22、23、和24的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。13.项11或12的经遗传修饰的酵母细胞,其中baat基因或gabt基因包含与选自seqidno:19、140、141、和142的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。14.项11-13任一项的经遗传修饰的酵母细胞,其中所述baat基因或gabt为也是gabt基因的baat基因。15.项1-14任一项的经遗传修饰的酵母细胞,其包含外源3-hpdh基因。16.项15的经遗传修饰的酵母细胞,其中3-hpdh基因编码与选自seqidno:26、27、28、29、30、31、32、33、34和129的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。17.项15或16的经遗传修饰的酵母细胞,其中3-hpdh基因包含与选自seqidno:25、143、144和343的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。18.项15-17任一项的经遗传修饰的酵母细胞,其中3-hpdh基因也是hibadh基因。19.项15-18任一项的经遗传修饰的酵母细胞,其中3-hpdh基因也是4-羟基丁酸脱氢酶基因。20.项1-19任一项的经遗传修饰的酵母细胞,其包含外源ppc基因。21.项20的经遗传修饰的酵母细胞,其中ppc基因编码与选自seqidno:10、11和12的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。22.项20或21的经遗传修饰的酵母细胞,其中ppc基因包含与seqidno:9的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。23.项1-22任一项的经遗传修饰的酵母细胞,其中所述酵母细胞是crabtree阴性的。24.项1-22任一项的经遗传修饰的酵母细胞,其中酵母细胞属于选自伊萨酵母属(issatchenkia)、假丝酵母属(candida)、克鲁维酵母属(kluyveromyces)、毕赤酵母属(pichia)、裂殖酵母属(schizosaccharomyces)、有孢圆酵母属(torulaspora)、接合酵母属(zygosaccharomyces)和酵母属(saccharomyces)的属。25.项24的经遗传修饰的酵母细胞,其中酵母细胞属于选自东方伊萨酵母/发酵毕赤酵母(i.orientalis/p.fermentans)进化枝和酵母属进化枝的进化枝。26.项24的经遗传修饰的酵母细胞,其中酵母细胞选自东方伊萨酵母(i.orientalis)、郎比可假丝酵母(c.lambica)和博伊丁酵母(s.bulderi)。27.项1-26任一项的经遗传修饰的酵母细胞,其中所述细胞进一步包含选自pdc、adh、gal6、cyb2a、cyb2b、gpd、gpp、ald和pck基因的天然基因的一个或多个缺失或破坏。28.项27的经遗传修饰的酵母细胞,其中一个或多个缺失或破坏得自一个或多个外源3-hp途径基因的插入。29.项1-28任一项的经遗传修饰的酵母细胞,其中一个或多个外源3-hp途径基因可操作连接到一个或多个外源调节元件上。30.项1-29任一项的经遗传修饰的酵母细胞,其中细胞能够在含75g/l或更多3-hp的培养基中在小于4的ph下生长。31.项1-30任一项的经遗传修饰的酵母细胞,其中细胞是3-hp抗性酵母细胞。32.项1-31任一项的经遗传修饰的酵母细胞,其中细胞已经经过了突变和/或选择,以致经突变的和/或选择的细胞相比同种的野生型细胞具有更高程度的对3-hp的抗性。33.项32的经遗传修饰的酵母细胞,其中细胞在用一个或多个外源3-hp途径基因进行遗传修饰之前已经经过了突变和/或选择。34.项32或33的经遗传修饰的酵母细胞,其中细胞已经经过了乳酸或3-hp存在下的选择。35.项34的经遗传修饰的酵母细胞,其中选择是恒化选择。36.生产3-hp的方法,包括:(i)在含至少一种碳源的培养基的存在下培养项1-35任一项的经遗传修饰的酵母细胞;和(ii)从培养物中分离3-hp。37.项36的方法,其中所述碳源选自葡萄糖、木糖、阿拉伯糖、蔗糖、果糖、纤维素、葡萄糖寡聚物和甘油。38.项36或37的方法,其中培养基的ph小于5,如在约1.5至约4.5、约2.0至约4.0或约2.0至约3.5的范围中。附图简述图1:所选3-hp发酵途径的概要图2:质粒pmiba107图3:靶向整合技术的示意性展示。图4:质粒pgmer125(a)图5:质粒pgmrr125(b)图6:质粒pgmer121图7:质粒pmhct074图8:质粒pmhct083图9:质粒pmhct087图10:质粒pmhct075图11:质粒pmhct077图12:质粒pmhct095图13:质粒pmhct096图14:质粒pmeji310-2图15:质粒pmeji312-2图16:质粒pgmer126图17:质粒pgmer130图18:质粒pgmer137图19:质粒pacn5图20:质粒pacn23图21:质粒phjj27图22:质粒pacn43图23:质粒phjj75图24:质粒phjj76图25:质粒pjlj49图26:质粒pjlj62图27:质粒pmi458图28:质粒pcm208图29:质粒pjy39图30:质粒pmcts64图31:质粒pmcts65图32:质粒pjlj8。发明详述下面的本发明的描述仅仅意在阐述本发明的多个实施方案。因此,所讨论的特定修饰不能视为对本发明范围的限制。对于本领域技术人员来说显而易见的是,可以在不偏离本发明范围的情况下进行多种等价替代、变化和修饰,并且应当理解这种等同的实施方案包含于本文中。本文中引用的所有参考文献作为整体通过提述并入。缩写3-hp,3-羟基丙酸;3-hpa,3-羟基丙醛;3-hpdh,3-羟基丙酸脱氢酶;aam,丙氨酸2,3氨基变位酶;aat,天冬氨酸氨基转移酶;acc,乙酰-coa羧化酶;adc,天冬氨酸1-脱羧酶;akg,α-酮戊二酸;ald,醛脱氢酶;baat,β-丙氨酸氨基转移酶;bcka,支链α-酮酸脱羧酶;bp,碱基对;cyb2,l-(+)-乳酸-细胞色素c氧化还原酶;cyc,异-2-细胞色素c;ems,甲基磺酸乙酯(ethanemethylsulfonase);eno,烯醇酶;gabt,4-氨基丁酸氨基转移酶;gapdh,甘油醛-3-磷酸脱氢酶;gpd,甘油3-磷酸脱氢酶;gpp,甘油-3-磷酸磷酸酶;hibadh,3-羟基异丁酸脱氢酶;ipda,吲哚丙酮酸脱羧酶;kgd,α-酮戊二酸脱羧酶;ldh,乳酸脱氢酶;mae,苹果酸酶;oaa,草酰乙酸;pck,磷酸烯醇丙酮酸羧激酶;pdc,丙酮酸脱羧酶;pdh,丙酮酸脱氢酶;pep,磷酸烯醇丙酮酸;pgk,磷酸甘油酸激酶;ppc,磷酸烯醇丙酮酸羧化酶;pyc,丙酮酸羧化酶;rki,核糖5-磷酸酮醇异构酶;tal,转醛醇酶;tef1,翻译延长因子-1;tef2,翻译延长因子-2;tkl,转酮醇酶;xdh,木糖醇脱氢酶;xr,木糖还原酶;yp,酵母提取物/蛋白胨。描述本文中提供了用于生产3-hp的经遗传修饰的酵母细胞、制备这些酵母细胞的方法以及利用这些细胞生产3-hp的方法。本文中使用的“3-hp”包括3-羟基丙酸的盐和酸形式。一些3-hp发酵途径是本领域已知的(参见例如us专利号6,852,517;us专利号7,309,597;us公开号2001/0021978;us公开号2008/0199926;wo02/42418和wo10/031083,所有通过提述并入本文)。3-hp发酵途径经由一系列中间物运行,所述中间物可以包括磷酸烯醇丙酮酸(pep)、丙酮酸、草酰乙酸(oaa)、天冬氨酸、β-丙氨酸、丙二酸半醛、苹果酸、丙二酰-coa、乙酰-coa、丙氨酸、乳酸、乳酰-coa、丙烯酰-coa、甘油、3-羟基丙醛(3-hpa)、β-丙氨酰-coa、3-hp-coa和甘油酸。几条已知的3-hp发酵途径的概述示于图1中。如本文中所公开,对一组来自多个菌种的酵母细胞进行3-hp抗性的测试。基于在含有不同浓度3-hp的培养基中的生长速率和葡萄糖消耗速率,对呈现3-hp抗性的细胞进行进一步评估。基于这些实验,鉴定出了一组用于3-hp生产的理想的宿主细胞。接着对这些宿主细胞进行遗传修饰,以含有活性3-hp发酵途径,获得在低ph条件下产生3-hp的经遗传修饰的酵母细胞。本文在某些实施方案中提供了经遗传修饰的酵母细胞,其具有至少一条由pep、丙酮酸和/或甘油产生3-hp的活性3-hp发酵途径。本文中使用的具有“活性3-hp发酵途径”的酵母细胞产生催化3-hp发酵途径中的每个反应所必需的活性酶,因此当在至少一种可发酵糖存在时在发酵条件下进行培养时,能够产生可测量的产量的3-hp。具有活性3-hp发酵途径的酵母细胞包含一个或多个3-hp途径基因。本文中使用的“3-hp途径基因”是指编码参与3-hp发酵途径的酶的核苷酸序列编码区。在某些实施方案中,本文中提供的酵母细胞具有经由pep或丙酮酸、oaa、天冬氨酸、β-丙氨酸和丙二酸半醛中间物进行的活性3-hp发酵途径(参见例如us公开号2010/0021978,图1)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括丙酮酸羧化酶(pyc)、pep羧化酶(ppc)、天冬氨酸氨基转移酶(aat)、天冬氨酸1-脱羧酶(adc)、β-丙氨酸氨基转移酶(baat)、氨基丁酸氨基转移酶(gabt)、3-hp脱氢酶(3-hpdh)、3-羟基异丁酸脱氢酶(hibadh)和4-羟基丁酸脱氢酶基因中的一个或多个。3-hp发酵途径基因还可以包括pep羧激酶(pck)基因,其已经修饰以产生优选催化pep转化为oaa的多肽(天然pck基因一般产生优选催化oaa至pep的逆反应的多肽)。在某些实施方案中,本文中提供的酵母细胞具有经由pep或丙酮酸、oaa和苹果酸中间物进行的活性3-hp发酵途径(参见例如us公开号2010/0021978,图4)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括ppc、pyc、苹果酸脱氢酶和苹果酸脱羧酶基因中的一个或多个。3-hp发酵途径基因还可以包括pck基因,其已经修饰以产生优选催化pep转化为oaa的多肽。在某些实施方案中,本文中提供的酵母细胞具有经由pep或丙酮酸、oaa和丙二酸半醛中间物进行的活性3-hp发酵途径(参见例如us公开号2010/0021978,图1)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括ppc、pyc、2-酮酸脱羧酶、α-酮戊二酸(akg)脱羧酶(kgd)、支链α-酮酸脱羧酶(bcka)、吲哚丙酮酸脱羧酶(ipda)、3-hpdh、hibadh和4-羟基丁酸脱氢酶基因中的一个或多个。3-hp发酵途径基因还可以包括pck基因,其已经修饰以产生优选催化pep转化为oaa的多肽。3-hp发酵途径基因还可以包括pdc基因和/或甲酸苄酯脱羧酶基因,其已经被修饰以编码能够催化oaa转化为丙二酸半醛的多肽。在某些实施方案中,本文中提供的酵母细胞具有经由pep或丙酮酸、oaa、丙二酰-coa和丙二酸半醛中间物进行的活性3-hp发酵途径(参见例如us公开号2010/0021978,图2)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括ppc、pyc、oaa甲酸裂合酶、丙二酰-coa还原酶、coa酰化丙二酸半醛脱氢酶、3-hpdh、hibadh和4-羟基丁酸脱氢酶基因中的一个或多个。3-hp发酵途径基因还可以包括pck基因,其已经修饰以产生优选催化pep转化为oaa的多肽。此外,3-hp发酵途径基因还可以包括通过修饰2-酮酸脱氢酶基因而获得的oaa脱氢酶基因,以产生催化oaa转化为丙二酰-coa的多肽。在某些实施方案中,本文中提供的酵母细胞具有经由丙酮酸、乙酰-coa、丙二酰-coa和丙二酸半醛中间物进行的活性3-hp发酵途径,其中丙二酸半醛中间物是任选的(参见例如wo02/042418,图44)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括丙酮酸脱氢酶(pdh)、乙酰-coa羧化酶(acc)、丙二酰-coa还原酶、coa酰化丙二酸半醛脱氢酶、3-hpdh、hibadh和4-羟基丁酸脱氢酶基因中的一个或多个。在某些实施方案中,本文中提供的酵母细胞具有经由丙酮酸、丙氨酸、β-丙氨酸、β-丙氨酰-coa、丙烯酰-coa、3-hp-coa和丙二酸半醛中间物进行的活性3-hp发酵途径,其中β-丙氨酰-coa、丙烯酰-coa、3-hp-coa和丙二酸半醛中间物是任选的(β-丙氨酸能够经由丙二酸半醛中间物或经由β-丙氨酰-coa、丙烯酰-coa和3-hp-coa中间物转化为3-hp)(参见例如us专利7,309,597,图1)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括丙氨酸脱氢酶、丙酮酸/丙氨酸氨基转移酶、丙氨酸2,3氨基变位酶、coa转移酶、coa合成酶、β-丙氨酰-coa氨解酶、3-hp-coa脱水酶、3-hp-coa水解酶、3-羟基异丁酰-coa水解酶、baat、3-hpdh、hibadh和4-羟基丁酸脱氢酶基因中的一个或多个。在某些实施方案中,本文中提供的酵母细胞具有经由丙酮酸、乳酸、乳酰-coa、丙烯酰-coa和3-hp-coa中间物进行的活性3-hp发酵途径(参见例如wo02/042418,图1)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括ldh、coa转移酶、coa合成酶、乳酰-coa脱水酶、3-hp-coa脱水酶、3-hp-coa水解酶和3-羟基异丁酰-coa水解酶基因中的一个或多个。在某些实施方案中,本文中提供的酵母细胞具有经由甘油和3-hpa中间物进行的活性3-hp发酵途径(参见例如us专利6,852,517)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括甘油脱水酶和醛脱氢酶基因中的一个或多个。在某些实施方案中,本文中提供的酵母细胞具有经由pep或丙酮酸、oaa、天冬氨酸、β-丙氨酸、β-丙氨酰-coa、丙烯酰-coa、3-hp-coa和丙氨酸中间物进行的活性3-hp发酵途径,其中oaa、天冬氨酸和丙氨酸中间物是任选的(pep或丙酮酸可以经由oaa和天冬氨酸或经由丙氨酸转化为β-丙氨酸)(参见wo02/042418,图54;us专利7,309,597,图1)。在这些实施方案中,酵母细胞包含一组3-hp发酵途径基因,其包括ppc、pyc、aat、adc、coa转移酶、coa合成酶、β-丙氨酰-coa氨解酶、3-hp-coa脱水酶、3-hp-coa水解酶、3-羟基异丁酰-coa水解酶、丙氨酸脱氢酶、丙酮酸/丙氨酸氨基转移酶和aam基因中的一个或多个。3-hp发酵途径基因还可以包括pck基因,其已经修饰以产生优选催化pep转化为oaa的多肽。本文中提供的酵母细胞中的3-hp发酵途径基因可以是内源的或外源的。本文中关于基因元件例如基因、启动子和终止子序列所使用的“内源的”是指基因元件存在于天然形式的特定酵母细胞的基因组中的特定位置。本文中关于基因元件使用的“外源的”是指基因元件不存在于天然形式的特定酵母细胞的基因组中的特定位置。本文中关于酵母细胞使用的“天然的”是指特定酵母菌种的野生型酵母细胞。本文中关于代谢途径使用的“天然的”是指天然酵母细胞中存在且具有活性的代谢途径。外源基因元件可以具有天然的或非天然的序列。具有天然序列的外源基因元件包含与天然细胞基因组中存在的基因元件相同(除了不影响功能的个体-个体突变以外)的序列(即外源基因元件与内源基因元件相同)。然而,外源元件在宿主细胞基因组中存在的位置与内源元件不同。例如,与内源pyc基因相同的外源pyc基因可以插入到酵母细胞中,产生具有非天然(增加的)数量的pyc基因拷贝。具有非天然序列的外源基因元件包含天然细胞基因组中未发现的序列。例如,来自特定菌种的外源pyc基因可以插入到另一菌种的酵母细胞中。外源基因优选以功能性方式整合到宿主细胞基因组中,这意味着其能够在宿主细胞中产生活性蛋白。然而,在某些实施方案中,外源基因可以作为载体的一部分引入细胞中,所述载体稳定保持于宿主胞质中。在某些实施方案中,本文中提供的酵母细胞包含一个或多个外源3-hp发酵途径基因。在某些实施方案中,本文中公开的经遗传修饰的酵母细胞包含单个外源基因。在其他实施方案中,酵母细胞包含多个外源基因。在这些实施方案中,酵母细胞可包含单个外源基因的多个拷贝和/或两个或多个不同外源基因的拷贝。包含多个外源基因的酵母细胞可包含任意数量的外源基因。例如,这些酵母细胞可包含1至20个外源基因,在某些优选的实施方案中,其可包含1至7个外源基因。外源基因的多个拷贝可整合到单个基因座中,以致其彼此相邻。或者,其可整合到宿主细胞基因组内分离的基因座中。在某些实施方案中,本文中提供的酵母细胞包含一个或多个内源3-hp发酵途径基因。在这些实施方案的某些中,可以对细胞进行工程改造,以过量表达这些内源基因中的一个或多个,这意味着在至少相同的条件下,经修饰的细胞使内源基因以高于天然细胞的水平表达。在这些实施方案的某些中,过量表达的内源基因可以与一个或多个外源调节元件可操作连接。例如,可以将一个或多个天然或非天然外源强效启动子引入细胞中,以致其与一个或多个内源3-hp途径基因可操作连接。本文中提供的酵母细胞中的3-hp发酵途径基因可以与一个或多个调节元件例如启动子或终止子可操作连接。如本文中所使用,术语“启动子”是指位于基因翻译起始密码子上游(即5’)的非翻译序列(一般约1至1000碱基对(bp)内,优选约1至500bp内),其调控基因转录的起始。本文中使用的术语“终止子”是指位于基因翻译终止密码子下游(即3’)的非翻译序列(一般约1至1000bp内,优选约1至500bp内,尤其是约1至100bp内),其调控基因转录的结束。如果启动子或终止子在基因组中相对于基因的位置使得启动子或终止子(视情况而定)行使其转录调控功能,则其与基因“可操作连接”。适宜的启动子和终止子描述于例如wo99/14335、wo00/71738、wo02/42471、wo03/102201、wo03/102152和wo03/049525中(所有作为整体通过提述并入)。本文中提供的细胞中与3-hp发酵途径基因连接的调节元件可以是内源的或外源的。例如,外源3-hp发酵途径基因可以插入到酵母细胞中,使其处于内源启动子和/或终止子的转录调控下。或者,外源3-hp发酵途径基因可以与一个或多个外源调节元件连接。例如,可以将外源基因作为基因表达构建体的一部分引入细胞中,所述构建体包含一个或多个外源调节元件。在某些实施方案中,外源调节元件或至少外源调节元件的功能性部分,可包含天然序列。在其他实施方案中,外源调节元件可包含非天然序列。在这些实施方案中,外源调节元件可包含与天然调节元件具有相对较高程度的序列同一性的序列。例如,外源基因可以与天然启动子或终止子具有至少50%、至少60%、至少70%、至少80%或至少90%序列同一性的外源启动子或终止子连接。核苷酸或氨基酸序列的序列同一性百分比可以通过本领域已知的方法计算,例如利用具有缺省参数的blast(nationalcenterforbiologicalinformation(ncbi)basiclocalalighmentsearchtool)第2.2.1版软件。例如,利用具有缺省参数的blast第2.2.1版运算方法得到至少90%相同性分值的序列被认为具有至少90%的序列同一性。blast软件可以从ncbi,bethesda,maryland获得。在某些方面中,本文中提供的细胞中与3-hp发酵途径基因连接的调节元件(例如启动子)对于途径基因来说可以是外来的。对于途径基因来说是外来的调节元件是不以天然形式与基因连接的调节元件。本领域技术人员可以领会的是,对于途径基因来说是外来的调节元件可以是内源的或外源的,这取决于途径基因及其与酵母细胞的关系。在一些情况下,内源3-hp发酵途径基因与对于途径基因来说是外来的调节元件(例如启动子)可操作连接。在其他情况下,外源3-hp发酵途径基因与对于途径基因来说是外来的外源调节元件(例如启动子)可操作连接。在其中多个外源基因插入宿主细胞的那些实施方案中,每个外源基因可以处于不同调节元件的控制下,或者两个或多个外源基因可以处于相同调节元件的控制下。例如,当第一外源基因与第一调节元件连接时,第二外源基因也可以与第一调节元件连接,或者其可以与第二调节元件连接。第一和第二调节元件可以是相同的或享有高度的序列同一性,或者其完全无关。本文中提供的酵母细胞中可以与一个或多个3-hp发酵途径基因连接的启动子的示例包括但不限于pdc1、磷酸甘油酸激酶(pgk)、木糖还原酶(xr)、木糖醇脱氢酶(xdh)、l-(+)-乳酸-细胞色素c氧化还原酶(cyb2)、翻译延长因子-1(tef1)、翻译延长因子-2(tef2)、烯醇酶(eno1)、甘油醛-3-磷酸脱氢酶(gapdh)和乳清苷5’-磷酸脱羧酶(ura3)基因的启动子。在这些示例中,3-hp发酵途径基因可以与pdc1、pgk、xr、xdh、cyb2、tef1、tef2、eno1、gapdh或ura3基因的内源或外源启动子连接。当启动子是外源的时,其与pdc1、pgk、xr、xdh、cyb2、tef1、tef2、eno1、gapdh或ura3基因的天然启动子可以是相同的或享有高度的序列同一性(即至少约80%、至少约85%、至少约90%、至少约95%或至少约99%)。本文中提供的酵母细胞中可以与一个或多个3-hp发酵途径基因连接的终止子的示例包括但不限于pdc1、xr、xdh、转醛醇酶(tal)、转酮醇酶(tkl)、核糖5-磷酸酮醇异构酶(rki)、cyb2或异-2-细胞色素c(cyc)基因或者半乳糖家族基因(特别是gal10终止子)的终止子。在这些示例中,3-hp发酵途径基因可以与pdc1、xr、xdh、tal、tkl、rki、cyb2或cyc基因或者半乳糖家族基因的内源或外源终止子连接。当终止子是外源的时,其与pdc1、xr、xdh、tal、tkl、rki、cyb2或cyc基因或者半乳糖家族基因的天然终止子可以是相同的或享有高度的序列同一性(即至少约80%、至少约85%、至少约90%、至少约95%或至少约99%)。在某些实施方案中,3-hp发酵途径基因与包含宿主细胞天然的天然gal10基因的功能性部分或者与天然gal10终止子享有至少80%、至少85%、至少90%或至少95%序列同一性的序列的终止子连接。外源基因可以通过本领域任何已知的方法插入到酵母宿主细胞中。在优选的实施方案中,基因整合到宿主细胞基因组中。外源基因可以以靶向或随机方式整合到基因组中。在其中基因以靶向方式整合的那些实施方案中,其可以整合到特定基因的基因座,以致外源基因的整合与天然基因的缺失或破坏偶联。例如,外源3-hp途径基因的引入可以与一个或多个编码参与其他发酵产物途径的酶的基因的缺失或破坏偶联。或者,外源基因可以整合到不对应于基因的基因组部分中。靶向整合和/或缺失可以利用整合构建体。本文中使用的术语“构建体”是指用于转化宿主细胞的dna序列。构建体可以是例如环状质粒或载体、环状质粒或载体的一部分(例如限制性酶消化产物)、线性质粒或载体或者利用质粒或基因组dna作为模板制备的pcr产物。用外源构建体转化酵母细胞的方法描述于例如wo99/14335、wo00/71738、wo02/42471、wo03/102201、wo03/102152和wo03/049525中。整合构建体可以利用来自插入位点靶标的两条克隆的靶dna序列进行组装。两条靶dna序列在天然宿主基因组中可以是相邻的或非相邻的。在本文中,“非相邻的”是指dna序列在天然基因组中不是互相紧邻的,而是由将要被缺失的区域分隔开。本文中使用的“相邻的”序列在天然基因组中直接互相紧邻。当靶向整合将与靶基因的缺失或破坏偶联时,整合构建体还可以被称为缺失构建体。在缺失构建体中,一条靶序列可包含位于靶基因启动子5’端的区域、所有或一部分启动子区域、所有或一部分靶基因编码序列或者其一些组合。另一条靶序列可包含位于靶基因终止子3’端的区域、所有或一部分终止子区域和/或所有或一部分靶基因编码序列。当靶向整合不与天然基因的缺失或破坏偶联时,靶序列的选择使得干扰序列的插入将不会破坏天然基因的表达。制备整合或缺失构建体,以致两条靶序列彼此之间定向于相同方向,如其天然存在于宿主细胞基因组中时。当利用整合或缺失构建体将外源基因引入到宿主细胞中时,将基因表达盒克隆到构建体的两条靶基因序列之间,以使外源基因能够表达。基因表达盒含有外源基因,并且可以进一步包含与外源基因可操作连接的一个或多个调节序列,例如启动子或终止子。缺失构建体还可以构建为不含基因表达盒。设计这种构建体,以在不插入外源基因的情况下使基因序列缺失或破坏。整合或缺失构建体可包含克隆到质粒的两条靶基因序列之间的一个或多个选择标志物盒。选择标志物盒含有使得能够进行转化子选择的至少一个选择标志物基因。“选择标志物基因”是编码经转化的细胞在选择培养基中存活和/或生长所需蛋白的基因,因此能够用于对细胞施加选择压力。成功的转化子将含有选择标志物基因,其给予成功转化的细胞提供选择基础的至少一种特性。通常的选择标志物基因编码下列蛋白:(a)赋予对抗生素或其他毒素的抗性(例如对博来霉素或zeomycin(例如印度斯坦链异壁菌(streptoalloteichushindustanus)ble基因)、氨基糖苷类例如g418或卡那霉素(例如来自转座子tn903的卡那霉素抗性基因)或潮霉素(例如来自大肠杆菌的氨基糖苷类抗生素抗性基因)的抗性),(b)补充细胞的营养缺陷型缺陷(例如亮氨酸(例如马克斯克鲁维酵母(k.marxianus)leu2基因)、尿嘧啶(例如马克斯克鲁维酵母、酿酒酵母或东方伊萨酵母ura3基因)或色氨酸(例如马克斯克鲁维酵母、酿酒酵母或东方伊萨酵母trp基因)的缺陷),(c)能够使细胞合成从简易培养基中不可获得的关键营养成分,或(d)赋予细胞在特定碳源上生长的能力(例如来自酿酒酵母的mel5基因,其编码α-半乳糖苷酶(蜜二糖酶)并赋予在蜜二糖作为唯一碳源上生长的能力)。优选的选择标志物包括ura3基因、zeocin抗性基因、g418抗性基因、mel5基因和潮霉素抗性基因。另一种优选的选择标志物是l-乳酸:亚铁细胞色素c氧化还原酶(cyb2)基因盒,前提是宿主细胞天然地缺少这种基因或其天然的cyb2基因首先被缺失或破坏。选择标志物与在宿主细胞中可操作的一个或多个启动子和/或终止子序列可操作连接。在某些实施方案中,这些启动子和/或终止子序列是包含于选择标志物盒中的外源启动子和/或终止子序列。适宜的启动子和终止子如本文中所描述。整合或缺失构建体用于转化宿主细胞。可以利用例如电穿孔和/或化学转化(例如基于氯化钙、醋酸锂等)方法完成转化。可以基于选择标志物的存在或缺少进行选择或筛选,以鉴定成功的转化子。在成功的转化子中,在靶位点的基因座的同源重组事件导致靶位点序列的破坏或缺失。当构建体靶向天然基因以供缺失或破坏时,可以在该重组事件过程中缺失所有或一部分天然靶基因、其启动子和/或其终止子。将整合构建体中的表达盒、选择标志物盒和靶序列之间的任何其他基因物质插入到宿主基因组靶序列的对应基因座中。可以进行pcr分析或southern分析,以确认发生了期望的插入/缺失。在一些实施方案中,可以利用来自两个或多个构建体、pcr产物或其组合的dna进行细胞转化,而非利用单个构建体或pcr产物。在这些实施方案中,一个整合片段的3’末端与另一个整合片段的5’末端重叠。在一个实例中,一个构建体将含有来自靶序列基因座的第一序列和标志物基因盒的非功能性部分,而另一个将含有来自靶序列基因座的第二序列和标志物基因盒的第二非功能性部分。选择标志物基因盒的部分,以致其能够组合,以形成完整的盒。同时用这些碎片转化细胞,使得形成完整的功能性标志物或结构性基因盒。可以基于由选择标志物给予的特性选择成功的转化子。在另一个实施例中,选择标志物存在于一个片段上,而靶序列在另外的片段上,以使整合片段非常有可能在目标位点整合。在其他实施方案中,可以利用来自三条线性dnas的转化整合外源基因物质。在这些实施方案中,一个片段在5’末端与第二片段并且在3’末端与第三片段重叠。可以设计整合或缺失构建体,以致选择标志物基因和其调节元件的一些或所有能够由于随后的同源重组事件而自发缺失。完成此的便利方法是设计构建体使得选择标志物基因和/或调节元件的侧翼为重复序列。重复序列是相同的dna序列,其对于宿主细胞是天然或非天然的,并且在构建体上以对于彼此相同或相反的方向取向。重复序列的长度有利地为50至1500bp,并且无需编码任何物质。包含重复序列允许发生同源重组,这导致选择标志物基因和一条重复序列的缺失。由于同源重组发生的频率相对较低,使转化子在非选择培养基上生长数轮,以使一些细胞中发生自发性同源重组是必需的。已经自发缺失选择标志物基因的细胞可以基于其由选择标志物基因所给予的选择特性的丧失而进行选择或筛选。在某些情况下,重组酶的表达可以增强重复位点之间的重组。本文中提供的经修饰的酵母细胞中的外源3-hp发酵途径基因可来源于来自任何适宜来源的来源基因。例如,外源基因可来源于酵母、真菌、细菌、植物、昆虫或哺乳动来源。如本文中所使用,“来源于”天然来源基因的外源基因编码以下多肽:1)其与由天然基因所编码的多肽相同,2)其与由天然基因所编码的多肽享有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%的序列同一性和/或3)在3-hp发酵途径中与由天然基因所编码的多肽具有相同的功能。例如,来源于东方伊萨酵母pyc基因的pyc基因可以编码包含seqidno:2的氨基酸序列的多肽,与seqidno:2的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的多肽和/或能够催化丙酮酸转化oaa的多肽。来源于天然基因的基因可包含与天然基因的编码区具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在某些实施方案中,来源于天然基因的基因可包含与来源基因的编码区相同的核苷酸序列。例如,来源于东方伊萨酵母pyc基因的pyc基因可包含seqidno:1的核苷酸序列或与seqidno:1的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在本文中提供的经修饰的酵母细胞的某些实施方案中,外源3-hp发酵途径基因源自的天然来源基因产生参与3-hp发酵途径的多肽。然而在其他实施方案中,天然来源基因可以编码不参与3-hp发酵途径或催化3-hp发酵途径中的逆反应的多肽。在这些实施方案中,外源3-hp途径基因相对于天然来源基因将具有已经经过了一个或多个靶向或随机的突变,这引起修饰的活性和/或底物偏好。例如,可以使天然来源基因突变,以产生编码在3-hp发酵途径中期望的反应方向中活性增加和/或非期望的方向中活性降低的多肽的基因。例如,当天然来源基因编码能够同时催化3-hp发酵途径中的正向和逆向反应的多肽时,可以对基因进行修饰,以致所得外源基因在正向反应中活性增加并且在逆向反应中活性降低。类似地,可以使天然来源基因突变,以产生编码与天然多肽具有不同底物偏好的多肽的基因。例如,可以使3-hp途径基因突变,以产生能够作用于天然多肽不偏好或根本不起作用的底物的多肽。在这些实施方案中,由外源3-hp途径基因所编码的多肽可以催化由天然来源基因所编码的多肽完全不能催化的反应。还可以使天然来源基因突变,以致所得3-hp途径基因在一种或多种下游3-hp途径中间物或副产物的存在下在dna、rna或蛋白水平上呈现为反馈抑制减少。在本文中提供的经修饰的酵母细胞的某些实施方案中,外源3-hp途径基因可来源于宿主酵母菌种。例如,当宿主细胞是东方伊萨酵母时,外源基因可来源于东方伊萨酵母基因。在这些实施方案中,外源基因可包含与天然基因的编码区相同的核苷酸序列,以致如果处于与野生型细胞中驱动基因表达的启动子不同的启动子的控制下,外源基因掺入到宿主细胞中使得天然基因序列的拷贝数增加和/或基因的调节或表达水平发生改变。在其他实施方案中,外源3-hp途径基因可包含与天然3-hp途径基因的编码区不同的核苷酸序列,然而即使如此仍然编码与由天然3-hp途径基因所编码的多肽相同的多肽。仍在其他实施方案中,外源3-hp途径基因可包含编码与由一个或多个天然3-hp途径基因所编码的多肽具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的多肽的核苷酸序列。在这些实施方案的某些中,外源基因包含与一个或多个天然基因具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。仍在其他实施方案中,外源3-hp基因可以编码与由天然3-hp途径基因所编码的多肽具有少于50%的序列同一性然而即使如此仍然与3-hp发酵途径中的天然多肽具有相同功能(即能够催化相同的反应)的多肽。如果需要,天然来源基因可以经过突变,以提供以通用真核细胞起始密码子(atg)起始的编码序列,或出于其他目的。在其他实施方案中,外源3-hp途径基因可来源于与宿主酵母细胞菌种不同的菌种。在这些实施方案的某些中,外源3-hp途径基因可来源于与宿主细胞不同的酵母菌种。例如,当宿主细胞是东方伊萨酵母时,外源基因可来源于酿酒酵母。在其他实施方案中,外源3-hp途径基因可来源于真菌、细菌、植物、昆虫或哺乳动物来源。例如,当宿主细胞是东方伊萨酵母时,外源基因可来源于细菌来源,例如大肠杆菌。在外源3-hp途径基因来源于非酵母来源的那些实施方案中,外源基因序列可以经密码子优化,以在酵母宿主细胞中表达。在外源3-hp途径基因来源于宿主细胞菌种以外的其他菌种的那些实施方案中,外源基因可以编码与由来源生物的天然3-hp途径基因所编码的多肽相同的多肽。在这些实施方案的某些中,外源3-hp途径基因可以与来自来源生物的天然3-hp途径基因是相同的。在其他实施方案中,外源基因可以与来自来源生物的天然3-hp途径基因享有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%的序列同一性。在其他实施方案中,外源3-hp途径基因可以编码与由来自来源生物的天然3-hp途径基因所编码的多肽享有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的多肽。在这些实施方案的某些中,外源基因可包含与来自来源生物的一个或多个天然3-hp途径基因具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。仍在其他实施方案中,外源3-hp基因可以编码与由来自来源生物的天然3-hp途径基因所编码的多肽具有少于50%的序列同一性然而即使如此仍然与3-hp发酵途径中来自来源生物的天然多肽具有相同功能的多肽。在某些实施方案中,本文中提供的酵母细胞表达一个或多个3-hp途径基因,其编码选自下组的酶:acc(催化乙酰-coa转化为丙二酰-coa),丙氨酸2,3氨基变位酶(aam,催化丙氨酸转化为β-丙氨酸),丙氨酸脱氢酶(催化丙酮酸转化为丙氨酸),醛脱氢酶(催化3-hpa转化为3-hp),kgd(催化oaa转化为丙二酸半醛),att(催化oaa转化为天冬氨酸),adc(催化天冬氨酸转化为β-丙氨酸),bcka(催化oaa转化为丙二酸半醛),baat(催化β-丙氨酸转化为丙二酸半醛),4-氨基丁酸氨基转移酶(gabt,催化β-丙氨酸转化为丙二酸半醛),β-丙氨酰-coa氨解酶(催化β-丙氨酰-coa转化为丙烯酰-coa),co-a酰化丙二酸半醛脱氢酶(催化丙二酰-coa转化为丙二酸半醛),coa合成酶(催化β-丙氨酸转化为β-丙氨酰-coa或乳酸转化为乳酰-coa),coa转移酶(催化β-丙氨酸转化为β-丙氨酰-coa和/或乳酸转化为乳酰-coa),甘油脱水酶(催化甘油转化为3-hpa),ipda(催化oaa转化为丙二酸半醛),ldh(催化丙酮酸转化为乳酸),乳酰-coa脱水酶(催化乳酰-coa转化为丙烯酰-coa),苹果酸脱羧酶(催化苹果酸转化为3-hp),苹果酸脱氢酶(催化oaa转化为苹果酸),丙二酰-coa还原酶(催化丙二酰-coa转化为丙二酸半醛或3-hp),oaa甲酸裂合酶(还被称为丙酮酸-甲酸裂合酶和酮酸甲酸裂合酶,催化oaa转化为丙二酰-coa),oaa脱氢酶(催化oaa转化为丙二酰-coa),ppc(催化pep转化为oaa),丙酮酸/甘氨酸氨基转移酶(催化丙酮酸转化为甘氨酸),pyc(催化丙酮酸转化为oaa),pdh(催化丙酮酸转化为乙酰-coa),2-酮酸脱羧酶(催化oaa转化为丙二酸半醛),3-hp-coa脱水酶(也被称为丙烯酰-coa水合酶,催化丙烯酰-coa转化为3-hp-coa),3-hpdh(催化丙二酸半醛转化为3-hp),3-hp-coa水解酶(催化3-hp-coa转化为3-hp),hibadh(催化丙二酸半醛转化为3-hp),3-羟基异丁酰-coa水解酶(催化3-hp-coa转化为3-hp)和4-羟基丁酸脱氢酶(催化丙二酸半醛转化为3-hp)。对于这些酶活性的每个,圆括号中的目标反应可以是天然或非天然活性的结果。本文中使用的“丙酮酸羧化酶基因”或“pyc基因”是指编码具有丙酮酸羧化酶活性的多肽的任何基因,意味着能够催化丙酮酸、co2、atp转化为oaa、adp和磷酸。在某些实施方案中,pyc基因可来源于酵母来源。例如,pyc基因可来源于编码seqidno:2所示氨基酸序列的东方伊萨酵母pyc基因。在其他实施方案中,基因可以编码与seqidno:2的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于东方伊萨酵母的pyc基因可包含seqidno:1所示的核苷酸序列或者与seqidno:1所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,pyc基因可来源于细菌来源。例如,pyc基因可来源于仅利用pyc而非ppc(参见下面)进行糖回补(anaplerosis)的少数几个细菌菌种之一,例如类球红细菌,或者来源于同时具有pyc和ppc的细菌菌种,例如菜豆根瘤菌。由类球红细菌和菜豆根瘤菌的pyc基因所编码的氨基酸序列分别示于seqidno:3和4。pyc基因可来源于编码seqidno:3或4的氨基酸序列的基因,或来源于编码与seqidno:3或4的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列的基因。或者,pyc基因可来源于编码不依赖于乙酰-coa活化的酶的pyc基因,例如编码seqidno:5(羧基转移酶亚基)或seqidno:6(生物素羧化酶亚基)所示氨基酸序列的荧光假单胞菌pyc基因、编码seqidno:7所示氨基酸序列的谷氨酸棒杆菌pyc基因,或编码与seqidno:5、6或7的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列的基因。pyc基因还可来源于编码不被天冬氨酸抑制的酶的pyc基因,例如编码seqidno:8所示氨基酸序列的苜蓿中华根瘤菌pyc基因(sauerfemsmicrobiolrev29:765(2005)),或者来源于编码与seqidno:8的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列的基因。本文中使用的“pep羧化酶基因”或“ppc基因”是指编码具有pep羧化酶活性的多肽的任何基因,意味着能够催化pep和co2转化为oaa和磷酸。在某些实施方案中,ppc基因可来源于细菌ppc基因。例如,ppc基因可来源于大肠杆菌ppc基因,其编码seqidno:10所示的氨基酸序列或者与seqidno:10的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于大肠杆菌的ppc基因可包含seqidno:9所示的核苷酸序列或者与seqidno:9所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,ppc基因可来源于在很多古菌(archea)和有限数量的细菌中发现的“a”型ppc,其不被乙酰coa活化并且较少被天冬氨酸抑制。例如,ppc基因可来源于编码seqidno:11所示氨基酸序列的嗜热自养甲烷杆菌ppca基因、编码seqidno:12所示氨基酸序列的产气荚膜梭菌ppca基因或编码与seqidno:11或12的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列的基因。在这些实施方案的某些中,基因相对于天然基因已经经过了一个或多个突变,以产生具有改进的特性的酶。例如,基因可以已经进行突变,以编码对天冬氨酸反馈的抗性相对于天然多肽增加的ppc多肽。在其他实施方案中,ppc基因可来源于植物来源。本文中使用的“天冬氨酸氨基转移酶基因”或“aat基因”是指编码具有天冬氨酸氨基转移酶活性的多肽的任何基因,意味着能够催化ooa转化为天冬氨酸。具有天冬氨酸氨基转移酶活性的酶被分类为ec2.6.1.1。在某些实施方案中,aat基因可来源于酵母来源,例如东方伊萨酵母或酿酒酵母。例如,aat基因可来源于编码seqidno:14所示氨基酸序列的东方伊萨酵母aat基因或编码seqidno:15所示氨基酸序列的酿酒酵母aat2基因。在其他实施方案中,基因可以编码与seqidno:14或15的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于东方伊萨酵母的aat基因可包含seqidno:13所示的核苷酸序列或者与seqidno:13所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,aat基因可来源于细菌来源。例如,aat基因可来源于大肠杆菌aspc基因,其编码包含seqidno:16所示氨基酸序列的多肽。在其他实施方案中,基因可以编码与seqidno:16的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。本文中使用的“天冬氨酸脱羧酶”或“adc基因”是指编码具有天冬氨酸脱羧酶活性的多肽的任何基因,意味着能够催化天冬氨酸转化为β-丙氨酸。具有天冬氨酸脱羧酶活性的酶被分类为ec4.1.1.11。在某些实施方案中,adc基因可来源于细菌来源。由于活性天冬氨酸脱羧酶需要对非活性酶原进行蛋白水解加工,在这些实施方案中,应当选择能够支持由细菌adc基因所编码的活性酶形成的酵母宿主细胞。在一些实施方案中,adc基因可来源于编码seqidno:17所示氨基酸序列的阿维链霉菌pand基因。在一些实施方案中,adc基因可以编码与seqidno:17的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于阿维链霉菌的adc基因可包含seqidno:130、145、146或147任一项所示的核苷酸序列或者与seqidno:130、145、146或147任一项所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,adc基因可来源于编码seqidno:18所示氨基酸序列的丙酮丁醇梭菌pand基因。在一些实施方案中,adc基因可以编码与seqidno:18的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于丙酮丁醇梭菌的adc基因可包含seqidno:131所示的核苷酸序列或者与seqidno:131所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,adc基因可来源于编码seqidno:133所示氨基酸序列的幽门螺旋杆菌adc基因。在一些实施方案中,adc基因可以编码与seqidno:133的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于幽门螺旋杆菌的adc基因可包含seqidno:133所示的核苷酸序列或者与seqidno:133所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,adc基因可来源于编码seqidno:135所示氨基酸序列的芽孢杆菌种ts25(bacillussp.ts25)adc基因。在一些实施方案中,adc基因可以编码与seqidno:135的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于芽孢杆菌种ts25的adc基因可包含seqidno:134所示的核苷酸序列或者与seqidno:134所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,adc基因可来源于编码seqidno:137所示氨基酸序列的谷氨酸棒杆菌adc基因。在一些实施方案中,adc基因可以编码与seqidno:137的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于谷氨酸棒杆菌的adc基因可包含seqidno:136所示的核苷酸序列或者与seqidno:136所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,adc基因可来源于编码seqidno:139所示氨基酸序列的地衣芽孢杆菌adc基因。在一些实施方案中,adc基因可以编码与seqidno:139的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于地衣芽孢杆菌的adc基因可包含seqidno:138、148、149、150或151任一项所示的核苷酸序列或者与seqidno:138、148、149、150或151任一项所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。本文中使用的“β-丙氨酸氨基转移酶基因”或“baat基因”是指编码具有β-丙氨酸氨基转移酶活性的多肽的任何基因,意味着能够催化β-丙氨酸转化为丙二酸半醛。具有β-丙氨酸氨基转移酶活性的酶被分类为ec2.6.1.19。在某些实施方案中,baat基因可来源于酵母来源,例如,baat基因可来源于pyd4基因的东方伊萨酵母同源物,其编码seqidno:20所示的氨基酸序列。在一些实施方案中,baat基因可以编码与seqidno:20的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于东方伊萨酵母的baat基因可包含seqidno:19所示的核苷酸序列或者与seqidno:19所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,baat基因可来源于编码seqidno:21所示氨基酸序列的克鲁维酵母pyd4基因。在一些实施方案中,baat基因可以编码与seqidno:21的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于克鲁维酵母的baat基因可包含seqidno:142所示的核苷酸序列或者与seqidno:142所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,baat基因可来源于细菌来源。例如,baat基因可来源于编码seqidno:22所示氨基酸序列的阿维链霉菌baat基因。在一些实施方案中,baat基因可以编码与seqidno:22的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于阿维链霉菌的baat基因可包含seqidno:140所示的核苷酸序列或者与seqidno:140所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。baat基因还可以是“4-氨基丁酸氨基转移酶”或“gabt基因”,意味着其对4-氨基丁酸以及β-丙氨酸具有天然活性。或者,baat基因可来源于对来自细菌或酵母来源的天然gabt基因的随机或定向工程改造,以编码具有baat活性的多肽。例如,baat基因可来源于编码seqidno:23所示氨基酸序列的阿维链霉菌gabt。在一些实施方案中,来源于阿维链霉菌的baat基因可以编码与seqidno:23的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在其他实施方案中,baat基因可来源于编码seqidno:24所示氨基酸序列的酿酒酵母gabt基因uga1。在一些实施方案中,来源于酿酒酵母的baat基因可以编码与seqidno:24的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于酿酒酵母的baat基因可包含seqidno:141所示的核苷酸序列或者与seqidno:141所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。本文中使用的“3-hp脱氢酶基因”或“3-hpdh基因”是指编码具有3-hp脱氢酶活性的多肽的任何基因,意味着能够催化丙二酸半醛转化为3-hp。具有3-hp脱氢酶活性的酶如果利用nad(h)转助因子则被分类为ec1.1.1.59,如果其利用nadp(h)转助因子则被分类为ec1.1.1.298。被分类为ec1.1.1.298的酶也可被称为丙二酸半醛还原酶。在某些实施方案中,3-hpdh基因可来源于酵母来源。例如,3-hpdh基因可来源于ymr226c基因的东方伊萨酵母同源物,其编码seqidno:26所示的氨基酸序列。在一些实施方案中,3-hpdh基因可以编码与seqidno:26的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于东方伊萨酵母的3-hpdh基因可包含seqidno:25所示的核苷酸序列或者与seqidno:25所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,3-hpdh基因可来源于编码seqidno:129所示氨基酸序列的酿酒酵母ymr226c基因。在一些实施方案中,3-hpdh基因可以编码与seqidno:129的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于酿酒酵母的3-hpdh基因可包含seqidno:144所示的核苷酸序列或者与seqidno:144所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,3-hpdh基因可来源于细菌来源。例如,3-hpdh基因可来源于编码seqidno:27所示氨基酸序列的大肠杆菌ydfg基因。在一些实施方案中,基因可以编码与seqidno:27的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于大肠杆菌的3-hpdh基因可包含seqidno:143所示的核苷酸序列或者与seqidno:143所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,3-hpdh基因可来源于编码seqidno:29所示氨基酸序列的m.sedula丙二酸半醛还原酶基因。在一些实施方案中,3-hpdh基因可以编码与seqidno:29所示的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。在某些实施方案中,来源于m.sedula的3-hpdh基因可包含seqidno:343所示的核苷酸序列或者与seqidno:343所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。本文中使用的“3-羟基异丁酸脱氢酶基因”或“hibadh基因”是指编码具有3-羟基异丁酸脱氢酶活性的多肽的任何基因,意味着能够催化3-羟基异丁酸转化为丙二酸半醛。具有3-羟基异丁酸脱氢酶活性的酶被分类为ec1.1.1.31。一些3-羟基异丁酸脱氢酶还具有3-hpdh活性。在某些实施方案中,hibadh基因可来源于细菌来源。例如,hibadh基因可来源于编码seqidno:28所示氨基酸序列的粪产碱菌m3a基因,分别编码seqidno:30或seqidno:31所示氨基酸序列的恶臭假单胞菌kt2440或e23440mmsb基因,或者编码seqidno:32所示氨基酸序列的铜绿假单胞菌pao1mmsb基因。在某些实施方案中,hibadh基因可以编码与seqidno:28、30、31或32所示的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。本文中使用的“4-羟基丁酸脱氢酶基因”是指编码具有4-羟基丁酸脱氢酶活性的多肽的任何基因,意味着能够催化4-羟基丁酸转化为琥珀酸半醛。具有4-羟基丁酸脱氢酶活性的酶被分类为ec1.1.1.61。一些4-羟基丁酸脱氢酶还具有3-hpdh活性。在某些实施方案中,4-羟基丁酸脱氢酶基因可来源于细菌来源。例如,4-羟基丁酸脱氢酶基因可来源于编码seqidno:33所示氨基酸序列的r.eutrophah164hbd基因或者编码seqidno:34所示氨基酸序列的克鲁维梭菌(c.kluyveri)dsm555hbd基因。在其他实施方案中,基因可以编码与seqidno:33或34所示的氨基酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的氨基酸序列。本文中使用的“pep羧激酶基因”或“pck基因”是指编码具有pep羧激酶活性的多肽的任何基因,意味着能够催化pep、co2和adp或gdp转化为oaa和atp或gtp,或者反之亦然。具有pep羧激酶活性的酶被分类为ec4.1.1.32(利用gtp/gdp)和ec4.1.1.49(利用atp/adp)。在某些实施方案中,pck基因可来源于酵母来源。在其他实施方案中,pck基因可来源于细菌来源,并且在这些实施方案的某些中,基因可来源于其中pck反应有利于oaa的产生而非脱羧化占主导的更常见反应形式的细菌中。例如,pck基因可来源于编码seqidno:35所示氨基酸序列的产琥珀酸曼氏杆菌(m.succiniciproducens)pck基因、编码seqidno:36所示氨基酸序列的产琥珀酸厌氧螺菌(a.succiniciproducens)pck基因、编码seqidno:37所示氨基酸序列的产琥珀酸放线杆菌(a.succinogenes)pck基因或者编码seqidno:38所示氨基酸序列的r.eutrophapck基因。在其他实施方案中,pck基因相对于其所来源的天然基因已经经过了一个或多个突变,以致所得基因编码优选催化pep转化为oaa的多肽。例如,pck基因可来源于编码seqidno:39所示氨基酸序列的大肠杆菌k12菌株pck基因,其中已经使基因突变,以优选催化pep转化为oaa。在其他实施方案中,pep向oaa的转化由例如在丙酸菌(例如谢曼氏丙酸杆菌(p.shermanii)、伍氏醋酸杆菌(a.woodii))中发现的pep羧基转磷酸酶催化,所述丙酸菌利用无机磷酸盐和二磷酸盐而非atp/adp或gtp/gdp。本文中使用的“苹果酸脱氢酶基因”是指编码具有苹果酸脱氢酶活性的多肽的任何基因,意味着能够催化oaa转化为苹果酸。在某些实施方案中,苹果酸脱氢酶基因可来源于细菌或酵母来源。本文中使用的“苹果酸脱羧酶基因”是指编码具有苹果酸脱羧酶活性的多肽的任何基因,意味着能够催化苹果酸转化为3-hp。已知苹果酸脱羧酶活性不能天然发生。因此,苹果酸脱羧酶基因可以通过在编码具有乙酰乳酸脱羧酶活性的多肽的天然来源基因中掺入一个或多个突变而得到。具有乙酰乳酸脱羧酶活性的多肽催化2-羟基-2-甲基-3-氧代丁酸转化为2-乙偶姻(2-acetoin),其被分类为ec4.1.1.5。在某些实施方案中,苹果酸脱羧酶基因可来源于细菌来源。例如,苹果酸脱羧酶基因可来源于编码seqidno:40所示氨基酸序列的乳酸乳球菌(l.lactis)aldb基因、编码seqidno:41所示氨基酸序列的嗜热链球菌(s.thermophilus)aldb基因、编码seqidno:42所示氨基酸序列的短芽孢杆菌(b.brevis)aldb基因或者编码seqidno:43所示氨基酸序列的产气肠杆菌(e.aerogenes)buda基因。本文中使用的“α-酮戊二酸(akg)脱羧酶基因”或“kgd基因”是指编码具有α-酮戊二酸脱羧酶活性的多肽的任何基因,意味着能够催化α-酮戊二酸(2-氧化戊二酸)转化为琥珀酸半醛。具有α-酮戊二酸脱羧酶活性的酶被分类为ec4.1.1.71。kgd基因可以用于得到编码能够催化oaa转化为丙二酸半醛的多肽的基因。这种活性可以在天然kgd基因中找到,或者其可以通过在天然kgd基因中掺入一个或多个突变而得到。在某些实施方案中,kgd基因可来源于细菌来源。例如,kgd基因可来源于编码seqidno:44所示氨基酸序列的结核分枝杆菌(m.tuberculosis)kgd基因、编码seqidno:45所示氨基酸序列的b.japonicumkgd基因或者编码seqidno:46所示氨基酸序列的m.loti(即rhizobiumloti)kgd基因。本文中使用的“支链α-酮酸脱羧酶基因”或“bcka基因”是指编码具有支链α-酮酸脱羧酶活性的多肽的任何基因,其能够用于使长度为3至6个碳原子的一类α-酮酸脱羧。具有bcka活性的酶被分类为ec4.1.1.72。bcka基因可以用于得到编码能够催化oaa转化为丙二酸半醛的多肽的基因。这种活性可以在天然bcka基因中找到,或者其可以通过在天然bcka基因中掺入一个或多个突变而得到。在某些实施方案中,bcka基因可来源于细菌来源。例如,bcka基因可来源于编码seqidno:47所示氨基酸序列的乳酸乳球菌kdca基因。本文中使用的“吲哚丙酮酸脱羧酶基因”或“ipda基因”是指编码具有吲哚丙酮酸脱羧酶活性的多肽的任何基因,意味着能够催化吲哚丙酮酸转化为吲哚乙醛。具有ipda活性的酶被分类为ec4.1.1.74。ipda基因可以用于得到编码能够催化oaa转化为丙二酸半醛的多肽的基因。这种活性可以在天然ipda基因中找到,或者其可以通过在天然ipda基因中掺入一个或多个突变而得到。在某些实施方案中,吲哚丙酮酸脱羧酶基因可来源于酵母、细菌或植物来源。本文中使用的“丙酮酸脱羧酶基因”或“pdc基因”是指编码具有丙酮酸脱羧酶活性的多肽的任何基因,意味着能够催化丙酮酸转化为乙醛。具有pdc活性的酶被分类为ec4.1.1.1。在优选的实施方案中,掺入到如本文中所提供的经修饰的酵母细胞中的pdc基因相对于其所来源的天然基因已经经过了一个或多个突变,以致所得基因编码能够催化oaa转化为丙二酸半醛的多肽。在某些实施方案中,pdc基因可来源于酵母来源。例如,pdc基因可来源于编码seqidno:49所示氨基酸序列的东方伊萨酵母pdc基因、编码seqidno:50所示氨基酸序列的酿酒酵母pdc1基因或者编码seqidno:51所示氨基酸序列的乳酸克鲁维酵母(k.lactis)pdc。在某些实施方案中,来源于东方伊萨酵母pdc基因的pdc基因可包含seqidno:48所示的核苷酸序列或者与seqidno:48所示的核苷酸序列具有至少50%、至少60%、至少70%、至少80%、至少85%、至少90%、至少95%、至少97%或至少99%序列同一性的核苷酸序列。在其他实施方案中,pdc基因可来源于细菌来源。例如,pdc基因可来源于编码seqidno:52所示氨基酸序列的运动发酵单胞菌(z.mobilis)pdc基因或者编码seqidno:53所示氨基酸序列的巴斯德醋酸杆菌(a.pasteurianus)pdc基因。本文中使用的甲酸苄酯脱羧酶”基因是指编码具有甲酸苄酯脱羧酶活性的多肽的任何基因,意味着能够催化甲酸苄酯转化为苯甲醛。具有甲酸苄酯脱羧酶活性的酶被分类为ec4.1.1.7。在优选的实施方案中,掺入到如本文中所提供的经修饰的酵母细胞中的甲酸苄酯脱羧酶基因相对于其所来源的天然基因已经经过了一个或多个突变,以致所得基因编码能够催化oaa转化为丙二酸半醛的多肽。在某些实施方案中,甲酸苄酯脱羧酶基因可来源于细菌来源。例如,甲酸苄酯脱羧酶基因可来源于编码seqidno:54所示氨基酸序列的恶臭假单胞菌mdlc基因、编码seqidno:55所示氨基酸序列的铜绿假单胞菌mdlc基因、编码seqidno:56所示氨基酸序列的施氏假单胞菌(p.stutzeri)dpgb基因或者编码seqidno:57所示氨基酸序列的荧光假单胞菌ilvb-1基因。本文中使用的“oaa甲酸裂合酶”是指编码具有oaa甲酸裂合酶活性的多肽的任何基因,意味着能够催化酰化酮酸转化为其相应的coa衍生物。由oaa甲酸裂合酶基因所编码的多肽可以对丙酮酸或对另一种酮酸具有活性。在某些实施方案中,oaa甲酸裂合酶基因编码将oaa转化为丙二酰-coa的多肽。本文中使用的“丙二酰-coa还原酶基因”是指编码具有丙二酰-coa还原酶活性的多肽的任何基因,意味着能够催化丙二酰-coa转化为丙二酸半醛(也被称为co-a酰化丙二酸半醛脱氢酶活性)。在某些实施方案中,丙二酰-coa还原酶基因可来源于双功能性丙二酰-coa还原酶基因,其还能够催化丙二酸半醛转化为3-hp。在这些实施方案的某些中,丙二酰-coa还原酶基因可来源于细菌来源。例如,丙二酰-coa还原酶基因可来源于编码seqidno:58所示氨基酸序列的橙色绿屈挠菌(c.aurantiacus)丙二酰-coa还原酶基因、编码seqidno:59所示氨基酸序列的r.castenholzii丙二酰-coa还原酶基因或者编码seqidno:60所示氨基酸序列的赤杆菌属nap1(erythrobactersp.nap1)丙二酰-coa还原酶基因。在其他实施方案中,丙二酰-coa还原酶基因可来源于编码仅催化丙二酰-coa转化为丙二酸半醛的多肽的丙二酰-coa还原酶基因。例如,丙二酰-coa还原酶基因可来源于编码seqidno:61所示氨基酸序列的m.sedulamsed_0709基因或者编码seqidno:62所示氨基酸序列的硫化叶菌(s.tokodaii)丙二酰-coa还原酶。本文中使用的“丙酮酸脱氢酶基因”或“pdh基因”是指编码具有丙酮酸脱氢酶活性的多肽的任何基因,意味着能够催化丙酮酸转化为乙酰-coa。在某些实施方案中,pdh基因可来源于酵母来源。例如,pdh基因可来源于分别编码seqidno:63-66所示氨基酸序列的酿酒酵母lat1、pda1、pdb1或lpd基因。在其他实施方案中,pdh基因可来源于细菌来源。例如,pdh基因可来源于分别编码seqidno:67-69所示氨基酸序列的大肠杆菌k12亚株mg1655acee、acef或ipd基因或者分别编码seqidno:70-73所示氨基酸序列的枯草芽孢杆菌(b.subtilis)pdha、pdhb、pdhc或pdhd基因。本文中使用的“乙酰-coa羧化酶基因”或“acc基因”是指编码具有乙酰-coa羧化酶活性的多肽的任何基因,意味着能够催化乙酰-coa转化为丙二酰-coa。具有乙酰-coa羧化酶活性的酶被分类为ec6.4.1.2。在某些实施方案中,乙酰-coa羧化酶基因可来源于酵母来源。例如,乙酰-coa羧化酶基因可来源于编码seqidno:74所示氨基酸序列的酿酒酵母acc1基因。在其他实施方案中,乙酰-coa羧化酶基因可来源于细菌来源。例如,乙酰-coa羧化酶基因可来源于分别编码seqidno:75-78所示氨基酸序列的大肠杆菌acca、accb、accc或accd基因或者分别编码seqidno:79-82所示氨基酸序列的橙色绿屈挠菌acca、accb、accc或accd基因。本文中使用的“丙氨酸脱氢酶基因”是指编码具有丙氨酸脱氢酶活性的多肽的任何基因,意味着能够催化丙酮酸nad依赖性还原胺化为丙氨酸。具有丙氨酸脱氢酶活性的酶被分类为ec1.4.1.1。在某些实施方案中,丙氨酸脱氢酶基因可来源于细菌来源。例如,丙氨酸脱氢酶基因可来源于编码seqidno:83所示氨基酸序列的枯草芽孢杆菌丙氨酸脱氢酶基因。本文中使用的“丙酮酸/丙氨酸氨基转移酶基因”是指编码具有丙酮酸/丙氨酸氨基转移酶活性的多肽的任何基因,意味着能够催化丙酮酸和l-谷氨酸转化为丙氨酸和2-氧代戊二酸。在某些实施方案中,丙酮酸/丙氨酸氨基转移酶基因来源于酵母来源。例如,丙酮酸/丙氨酸氨基转移酶基因可来源于编码seqidno:84所示氨基酸序列的粟酒裂殖酵母(s.pombe)丙酮酸/丙氨酸氨基转移酶基因或者编码seqidno:85所示氨基酸序列的酿酒酵母alt2基因。本文中使用的“丙氨酸2,3氨基变位酶基因”或“aam基因”是指编码具有丙氨酸2,3氨基变位酶活性的多肽的任何基因,意味着能够催化丙氨酸转化为β-丙氨酸。已知丙氨酸2,3氨基变位酶活性不能天然发生。因此,丙氨酸2,3氨基变位酶基因可以通过在编码具有类似活性例如赖氨酸2,3氨基变位酶活性(参见例如us专利7,309,597)的多肽的天然来源基因中掺入一个或多个突变而得到。在某些实施方案中,天然来源基因可以是编码seqidno:86所示氨基酸序列的枯草芽孢杆菌赖氨酸2,3氨基变位酶基因、编码seqidno:87所示氨基酸序列的牙龈卟啉单胞菌(p.gingivalis)赖氨酸2,3氨基变位酶基因或者编码seqidno:88所示氨基酸序列的具核梭杆菌(f.nucleatum)(atcc-10953)赖氨酸2,3氨基变位酶基因。本文中使用的“coa转移酶基因”是指编码具有coa转移酶活性的多肽的任何基因,所述coa转移酶活性在一个实施例中包括催化β-丙氨酸转化为β-丙氨酰-coa和/或乳酸转化为乳酰-coa的能力。在某些实施方案中,coa转移酶基因可来源于酵母来源。在其他实施方案中,coa转移酶基因可来源于细菌来源。例如,coa转移酶基因可来源于编码seqidno:89所示氨基酸序列的埃氏巨球形菌(m.elsdenii)coa转移酶基因。本文中使用的“coa合成酶基因”是指编码具有coa合成酶活性的多肽的任何基因。在一个实例中,这包括催化β-丙氨酸转化为β-丙氨酰-coa的能力。在另一个实例中,这包括催化乳酸转化为乳酰-coa的能力。在某些实施方案中,coa合成酶基因可来源于酵母来源。例如,coa合成酶基因可来源于酿酒酵母coa合成酶基因。在其他实施方案中,coa合成酶基因可来源于细菌来源。例如,coa合成酶基因可来源于大肠杆菌coa合成酶、类球红细菌或肠道沙门氏菌(s.enterica)coa合成酶基因。本文中使用的“β-丙氨酰-coa氨解酶基因”是指编码具有β-丙氨酰-coa氨解酶活性的多肽的任何基因,意味着能够催化β-丙氨酰-coa转化为丙烯酰-coa。在某些实施方案中,β-丙氨酰-coa氨解酶基因可来源于细菌来源,例如编码seqidno:90所示氨基酸序列的丙酸梭菌(c.propionicum)β-丙氨酰-coa氨解酶基因。本文中使用的“3-hp-coa脱水酶基因”或“丙烯酰-coa水合酶基因”是指编码具有3-hp-coa脱水酶活性的多肽的任何基因,意味着能够催化丙烯酰-coa转化为3-hp-coa。具有3-hp-coa脱水酶活性的酶被分类为ec4.2.1.116。在某些实施方案中,3-hp-coa脱水酶基因可来源于酵母或真菌来源,例如编码seqidno:91所示氨基酸序列的大豆疫霉根腐菌(p.sojae)3-hp-coa脱水酶基因。在其他实施方案中,3-hp-coa脱水酶基因可来源于细菌来源。例如,3-hp-coa脱水酶基因可来源于编码seqidno:92所示氨基酸序列的橙色绿屈挠菌3-hp-coa脱水酶基因、编码seqidno:93所示氨基酸序列的深红红螺菌(r.rubrum)3-hp-coa脱水酶基因或者编码seqidno:94所示氨基酸序列的r.capsulates3-hp-coa脱水酶基因。仍在其他实施方案中,3-hp-coa脱水酶基因可来源于哺乳动物来源。例如,3-hp-coa脱水酶基因可来源于编码seqidno:95所示氨基酸序列的智人(h.sapiens)3-hp-coa脱水酶基因。本文中使用的“3-hp-coa水解酶基因”是指编码具有3-hp-coa水解酶活性的多肽的任何基因,意味着能够催化3-hp-coa转化为3-hp。在某些实施方案中,3-hp-coa基因可来源于酵母或真菌来源。在其他实施方案中,3-hp-coa基因可来源于细菌或哺乳动物来源。本文中使用的“3-羟基异丁酰-coa水解酶基因”是指编码具有3-羟基异丁酰-coa水解酶活性的多肽的任何基因,所述3-羟基异丁酰-coa水解酶活性在一个实例中包括催化3-hp-coa转化为3-hp的能力。在某些实施方案中,3-羟基异丁酰-coa水解酶基因可来源于细菌来源,例如编码seqidno:96所示氨基酸序列的荧光假单胞菌3-羟基异丁酰-coa水解酶基因或者编码seqidno:97所示氨基酸序列的蜡状芽孢杆菌(b.cereus)3-羟基异丁酰-coa水解酶基因。在其他实施方案中,3-羟基异丁酰-coa水解酶基因可来源于哺乳动物来源,例如编码seqidno:98所示氨基酸序列的智人3-羟基异丁酰-coa水解酶基因。本文中使用的“乳酸脱氢酶基因”或“ldh基因”是指编码具有乳酸脱氢酶活性的多肽的任何基因,意味着能够催化丙酮酸转化为乳酸。在某些实施方案中,ldh基因可来源于真菌、细菌或哺乳动物来源。本文中使用的“乳酰-coa脱水酶基因”是指编码具有乳酰-coa脱水酶活性的多肽的任何基因,意味着能够催化乳酰-coa转化为丙烯酰-coa。在某些实施方案中,乳酰-coa脱水酶基因可来源于细菌来源。例如,乳酰-coa脱水酶基因可来源于编码seqidno:99-101所示氨基酸序列的埃氏巨球形菌乳酰-coa脱水酶e1、ella或ellb亚基基因。本文中使用的“醛脱氢酶基因”是指编码具有醛脱氢酶活性的多肽的任何基因,所述醛脱氢酶活性在一个实例中包括催化3-hpa转化为3-hp及其相反转化的能力。在某些实施方案中,醛脱氢酶基因可来源于酵母来源,例如编码seqidno:102所示氨基酸序列的酿酒酵母醛脱氢酶基因或者编码seqidno:122、124或126所示氨基酸序列的东方伊萨酵母醛脱氢酶基因。在其他实施方案中,醛脱氢酶可来源于细菌来源,例如编码seqidno:103所示氨基酸序列的大肠杆菌aldh基因或者编码seqidno:104所示氨基酸序列的肺炎克雷伯氏菌(k.pneumoniae)醛脱氢酶基因。本文中使用的“甘油脱水酶基因”是指编码具有甘油脱水酶活性的多肽的任何基因,意味着能够催化甘油转化为3-hpa。在某些实施方案中,甘油脱水酶基因可来源于细菌来源,例如肺炎克雷伯氏菌或弗氏柠檬酸杆菌(c.freundii)甘油脱水酶基因。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞进一步包含一个或多个天然基因的缺失或破坏。关于天然基因的“缺失或破坏”是指或者基因的整个编码区被去除(缺失),或者基因的编码区、启动子和/或其终止子区域被修饰(例如通过缺失、插入或突变),以致基因不再产生活性酶,产生显著减少的量(至少减少75%,优选至少减少90%)的活性酶,或产生活性显著降低(至少降低75%,优选至少降低90%)的酶。在某些实施方案中,一个或多个天然基因的缺失或破坏导致一个或多个天然代谢途径的缺失或破坏。关于代谢途径的“缺失或破坏”是指该途径或者无法运行,或者表现出相对于天然途径降低至少75%、至少85%或至少95%的活性。在某些实施方案中,天然代谢途径的缺失或破坏通过掺入导致一个或多个天然基因的表达减少的一个或多个遗传修饰而达到,所述表达减少使得3-hp的产生减少。在某些实施方案中,天然基因的缺失或破坏可以通过强制进化(forcedevolution)、诱变或基因工程改造方法,随后进行适宜的选择或筛选以鉴定期望突变体而达到。在某些实施方案中,天然宿主细胞基因的缺失或破坏可以与在宿主细胞中掺入一个或多个外源基因偶联,即可以利用也是缺失构建体的基因表达整合构建体来掺入外源基因。在其他实施方案中,缺失或破坏可以利用不含外源基因的缺失构建体或者通过本领域已知的其他方法而达到。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞包含一个或多个天然基因的缺失或破坏,所述天然基因编码参与乙醇发酵的酶,包括例如丙酮酸脱羧酶(pdc,将丙酮酸转化为乙醛)和/或乙醇脱氢酶(adh,将乙醛转化为乙醇)基因。这些修饰降低了酵母细胞生产乙醇的能力,从而使3-hp的生产最大化。然而,在某些实施方案中,可以对本文中提供的经遗传修饰的酵母细胞进行工程改造,以同时产生3-hp和乙醇。在那些实施方案中,编码参与乙醇发酵的酶的天然基因优选不被缺失或破坏,并且在某些实施方案中,酵母细胞可包含使乙醇生产增加的一个或多个外源基因。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞包含一个或多个天然基因的缺失或破坏,所述天然基因编码参与产生另外的发酵产物例如甘油或其他副产物例如乙酸盐或二醇的酶。例如,本文中提供的细胞可包含一个或多个以下基因的缺失或破坏:甘油3-磷酸脱氢酶(gpd,催化二羟基丙酮至甘油3-磷酸的反应)、甘油3-磷酸酶(gpp,催化甘油3-磷酸转化为甘油)、甘油激酶(催化甘油3-磷酸酶转化为甘油)、二羟基丙酮激酶(催化二羟基丙酮磷酸转化为二羟基丙酮)、甘油脱氢酶(催化二羟基丙酮转化为甘油)、醛脱氢酶(ald,例如,将乙醛转化为乙酸或将3-hp转化为3-hpa)或丁二醇脱氢酶(催化丁二醇转化为乙偶姻和相反转化)基因。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞包含一个或多个天然基因的缺失或破坏,所述天然基因编码催化3-hp发酵途径中的逆反应的酶,包括例如pep羧激酶(pck)、具有oaa脱羧酶活性的酶或cyb2a或cyb2b(催化乳酸转化为丙酮酸)。pck催化pep转化为oaa和相反转化,但是表现为优选oaa至opp的反应。为了减少oaa至pep的转化,一个或多个拷贝的天然pck基因可以被缺失或破坏。在某些实施方案中,其中一个或多个天然pck基因已被缺失或破坏的酵母细胞可以表达一个或多个外源pck基因,所述外源pck基因已经进行突变,以编码有利于pep转化为oaa的多肽。oaa脱羧酶催化oaa转化为丙酮酸。已经鉴定出了具有oaa脱羧酶活性的酶,例如大肠杆菌中由eda基因所编码的酶以及酵母和真菌中的苹果酸酶(mae)。为了降低oaa脱羧酶活性,编码具有oaa脱羧酶活性的酶的一个或多个拷贝的天然基因可以被缺失或破坏。在某些实施方案中,其中一个或多个天然oaa脱羧化基因已被缺失或破坏的酵母细胞可以表达一个或多个外源oaa脱羧化基因,所述外源oaa脱羧化基因已经进行突变,以编码催化丙酮酸转化为oaa的多肽。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞包含一个或多个天然基因的缺失或破坏,所述天然基因编码参与利用3-hp发酵途径产物或中间物的不期望反应的酶。这些基因的示例包括编码将3hp转化为3hp醛的酶的那些,所述3hp醛已知对细菌细胞是有毒性的。在某些实施方案中,本文中提供的经遗传修饰的酵母细胞包含一个或多个天然基因的缺失或破坏,所述天然基因编码对3-hp发酵途径具有中性作用的酶,包括例如gal6(将半乳糖转化为葡萄糖的gal系统的负性调节物)。中性基因的缺失或破坏使得能够在不影响天然发酵途径的情况下插入一个或多个外源基因。在某些实施方案中,本文中提供的酵母细胞是3-hp抗性酵母细胞。本文中使用的“3-hp抗性酵母细胞”是指在含有75g/l或更多3-hp的培养基中在小于4.0的ph下呈现至少2.5g/l/hr的平均糖酵解速率的酵母细胞。这些速率和条件代表生产3-hp的经济的过程。在这些实施方案的某些中,酵母细胞可以以其天然形式呈现3-hp抗性。在其他实施方案中,细胞可以在引入与活性3-hp发酵途径相关的遗传修饰之前、过程中或之后已经经过了突变和/或选择(例如恒化选择或重复的系列亚培养),以致经突变和/或选择的细胞具有高于相同菌种野生型细胞的3-hp抗性程度。例如,在一些实施方案中,细胞在用一个或多个外源3-hp途径基因进行遗传修饰之前已经经过了3-hp或乳酸存在下的突变和/或选择。在某些实施方案中,可以对以其天然形式表现出3-hp抗性的细胞进行突变和/或选择。可以在不同水平的3-hp存在下对已经经过了突变和/或选择的细胞进行糖消耗和其他特性的测试,以确定其作为3-hp生产的工业宿主的潜力。除了3-hp抗性,本文中提供的酵母细胞可以已经经过了对一种或多种额外的有机酸(例如乳酸)或者对其他发酵产物、副产物或培养基组分的抗性的突变和/或选择。可以利用本领域公知的方法进行选择,例如对3-hp或其他化合物的抗性的选择。例如,如上面所提及,选择可以是恒化选择。恒化选择利用使得能够持续培养微生物(例如酵母)的恒化器,其中特定的生长速率和细胞数量可以被独立地控制。持续培养是基本上恒定容积的流动系统,其中持续加入培养基,任何溢出可以持续移除。一旦这种系统处于平衡,细胞数量和营养成分状态保持恒定,并且系统处于稳定状态。恒化器使得能够通过稀释速率和限制性营养成分例如碳源或氮源的浓度变化来调控培养物的群体密度和特定生长速率。通过随着培养物生长而改变条件(例如,降低接种菌株生长所必需的第二碳源的浓度,以及其它),群体中能够在改变的条件下生长较快的微生物将被选择,并且其生长将超过在新条件下表现不佳的微生物。通常这种选择需要在恒化培养物的生长过程中不断增加或减少至少一种培养物组分。恒化器的操作及其在微生物定向进化中的用途是本领域公知的(参见例如novickprocnatlacadsciusa36:708-719(1950),harderjapplbacteriol43:1-24(1977))。其他选择方法包括但不限于例如u.s.专利号7,629,162中所描述的在选择性条件下重复的系列亚培养。这种方法可以用于替代或附加于上面所描述的利用葡萄糖限制性恒化方法。在如实施例中所公开的3-hp培养基中呈现生长和葡萄糖消耗的最佳组合的酵母菌株是用于与3-hp发酵途径相关的多种遗传修饰的优选宿主细胞。如通过在75g/l或更高3-hp的存在下在ph小于4下的生长所显示,具有相对较高程度的3-hp抗性的有潜力的酵母属包括例如假丝酵母属(candida)、克鲁维酵母属(kluyveromyces)、伊萨酵母属(issatchenkia)、酵母属(saccharomyces)、毕赤酵母属(pichia)、裂殖酵母属(schizosaccharomyces)、有孢圆酵母属(torulaspora)和接合酵母属(zygosaccharomyces)。呈现3-hp抗性的菌种包括东方伊萨酵母(也被称为克鲁斯假丝酵母(c.krusei))、郎比可假丝酵母(c.lambica)(也被称为发酵毕赤酵母(pichiafermentans))和博伊丁酵母(s.bulderi)(也被称为kazachstaniabulderi)。东方伊萨酵母和郎比可假丝酵母来自东方伊萨酵母/发酵毕赤酵母进化枝,而博伊丁酵母来自酵母属进化枝。呈现3-hp抗性的特定菌株包括东方伊萨酵母株24210、pta-6658、60585和cd1822,博伊丁酵母株mya-402和mya-404,以及郎比可假丝酵母株atcc38617。其他野生型酵母或真菌可以相同方式测试并进行鉴定,以在如本文中所描述的高水平3-hp存在下具有可接受的生长和葡萄糖利用水平。例如,gross和robbins(hydrobiologia433(103):91-109)已经汇编了在低ph(<4)的环境中鉴定的81种真菌菌种的一份名单,其能够作为潜力生产宿主进行相关测试。在某些实施方案中,本文中提供的经修饰的酵母细胞通过将一个或多个遗传修饰掺入crabtree阴性宿主酵母细胞中而制备。在这些实施方案的某些中,宿主酵母细胞属于伊萨酵母属、假丝酵母属或酵母属,在这些实施方案的某些中,宿主酵母细胞属于东方伊萨酵母/发酵毕赤酵母或酵母进化枝。在某些实施方案中,宿主细胞是东方伊萨酵母或郎比可假丝酵母或博伊丁酵母。东方伊萨酵母/发酵毕赤酵母进化枝是含有至少东方伊萨酵母、盔形毕赤酵母(p.galeiformis)、毕赤酵母菌种yb-4149(nrrl命名)、嗜酒假丝酵母(c.ethanolica)、(p.deserticola)、膜璞毕赤酵母(p.membranifaciens)和发酵毕赤酿母的最末端的进化枝。通过对酵母菌种26s核糖体dna可变d1/d2结构域的分析,鉴定东方伊萨酵母/发酵毕赤酵母进化枝的成员,其中利用由kurtzman和robnett在通过提述并入通过提述并入本文的“identificationandphylogenyofascomycetousyeastsfromanalysisofnuclearlargesubunit(26s)ribosomaldnapartialsequences,"antonievanleeuwenhoek73:331-371,1998中所描述的方法(具体参见第349页)。对成百上千子囊菌26s核糖体dna可变d1/d2结构域的分析已经揭示,东方伊萨酵母/发酵毕赤酵母进化枝含有非常密切相关的菌种。东方伊萨酵母/发酵毕赤酵母进化枝的成员表现为与进化枝其他成员在26s核糖体dna可变d1/d2结构域中的相似性高于进化枝外酵母菌种。因此,东方伊萨酵母/发酵毕赤酵母进化枝的其他成员可以通过利用kurtzman和robnett的方法比较其相应核糖体dna的d1/d2结构域并与进化枝其他成员和进化枝外密切相关的菌种作对比而进行鉴定。在某些实施方案中,本文中提供的经修饰的酵母细胞属于伊萨酵母属,在这些实施方案的某些中,酵母细胞是东方伊萨酵母。当最初鉴定时,东方伊萨酵母菌种被命名为库德毕赤酵母(pichiakudriavzevii)。无性型(无性生殖形式)的东方伊萨酵母被称为克鲁斯假丝酵母。东方伊萨酵母菌种的大量其它同义词已经列于别处(kurtzmanandfell,theyeasts,ataxonomicstudy.section35.issatchenkiakudryavtsev,pp222-223(1998))。生产3-hp的理想的酵母细胞理想地能够在低ph水平下生长。在低ph下进行发酵的能力降低了下游回收的成本,使得生产更经济。因此,在某些实施方案中,酵母宿主细胞能够在低ph水平下生长(例如在小于7、6、5、4或3的水平下)。除了3-hp抗性和/或低ph生长能力之外,适宜的宿主细胞可以具有一个或多个有利的特性。例如,可以进一步基于糖酵解速率、特定生长速率、热耐受性、对生物质水解产物抑制物的耐受性、过程的总体鲁棒性(robustness)等等对呈现3-hp抗性的潜在宿主细胞进行选择。可以在与3-hp发酵途径相关的任何遗传修饰前对这些标准进行评估,或者在一个或多个这种修饰已经发生后对其进行评估。由于大多数酵母是乙醇的天然生产者,因此将催化由丙酮酸产生乙醇中第一个步骤的酶(pdc)去除或者显著减少,对于产生足够的替代性产物来说是必需的。在crabtree阳性酵母例如酵母属中,pdc基因的缺失或破坏使得宿主获得对二碳化合物例如乙醇或乙酸盐的营养缺陷型,并且不能在含有葡萄糖的培养基中生长。可以利用乙酸盐非依赖性和葡萄糖耐受性的进行性选择获得能够克服这些局限的突变体(参见例如vanmarisapplenvironmicrobiol70:159(2004))。因此,在某些实施方案中,优选的酵母宿主细胞是crabtree阴性的酵母细胞,其中pdc缺失菌株能够在葡萄糖上生长,并且保持c2原养型。基因表达水平和/或给定细胞中将要利用的外源基因数目将根据所选择的酵母菌种而变化。对于经全基因组测序的酵母,可以利用全基因组化学计量模型来确定应当表达哪种酶以开发期望的3-hp发酵途径。全基因组化学计量模型量描述于例如hjersted等,"genome-scaleanalysisofsaccharomycescerevisiaemetabolismandethanolproductioninfed-batchculture,"biotechnol.bioeng.2007;和famili等,"saccharomycescerevisiaephenotypescanbepredictedbyusingconstraint-basedanalysisofagenome-scalereconstructedmetabolicnetwork,"proc.natl.acad.sci.2003,100(23):13134-9。对于不具有已知的基因组序列酵母,通常可以利用例如下面实施例2a中描述的技术获得目标基因的序列(作为过量表达的候选物或者作为插入位点)。可以进行常规的实验设计,以测试多种基因的表达和多种酶的活性,包括在3-hp途径中发挥功能的基因和酶。可进行实验,其中每种酶在酵母中独立表达,和形成酶区块,所述酶区块直至和优选包括所有途径酶,以确立何者为改进的3-hp产生所需(或所期望)。一种说明性实验设计测试每种单独的酶以及每个独特酶对的表达,并且能够进一步测试所有所需酶或者每个独特酶组合的表达。将要领会的是,可以采取多种方法。在某些实施方案中,提供了由如本文中所提供的经遗传修饰的酵母细胞生产3-hp的方法。在某些实施方案中,这些方法包括在至少一种碳源存在下培养如本文中所提供的经遗传修饰的酵母细胞,使细胞能够生产3-hp一段时间,接着从培养物中分离由细胞生产的3-hp。碳源可以是能够被所提供的酵母发酵的任何碳源。碳源可以是十二碳糖例如蔗糖,己糖例如葡萄糖或果糖,葡聚糖(glycan)或其他葡萄糖多聚物,葡萄糖寡聚物例如麦芽糖、麦芽三糖和异麦芽三糖、潘糖,以及果糖寡聚物。如果细胞经修饰以赋予发酵戊糖的能力,则发酵培养基可包含戊糖,例如木糖、木聚糖或木糖的其他寡聚物和/或阿拉伯糖。这些戊糖是含半纤维素的生物质的适宜水解产物。在寡聚糖的情况下,为了将这些消化为相应的单糖以由细胞发酵,向发酵液中加入酶可能是必需的。在某些实施方案中,培养物中可以存在多于一种类型的经遗传修饰的酵母细胞。同样地,在某些实施方案中,培养物中可以存在与经遗传修饰的酵母细胞相同或不同菌种的一种或多种天然酵母细胞。在某些实施方案中,对本文中提供的细胞进行培养以生产3-hp可以分为多个阶段。例如,细胞培养过程可以分为培养期、生产期和回收期。本领域普通技术人员将认识到,这些时期中使用的条件可以基于例如所使用的酵母菌种、酵母所利用的特定3-hp发酵途径、期望产率或其他因素的因素而变化。培养基将通常含有特定细胞所需的营养成分,包括氮源(例如氨基酸、蛋白、无机氮源例如氨或铵盐,等)以及多种维生素、矿物质等。在一些实施方案中,本发明的细胞可以在化学限定性培养基中进行培养。在一个实施例中,培养基含有大约5g/l硫酸铵、大约3g/l磷酸二氢钾、大约0.5g/l硫酸镁、微量元素、维生素以及大约150g/l葡萄糖。可以使ph在培养过程中自由变化,或者如果需要,进行缓冲以防止ph降至预设水平以下或升至预设水平以上。在某些实施方案中,发酵培养基接种足够的作为评估对象的酵母细胞,以产生大约1.0的od600。除非另有详细说明,本文中使用的od600是指利用ud600型分光光度仪(beckmancoulter)以1cm路径长度在600nm波长下所测得的光密度值。培养温度可以在大约30-40℃范围内,培养时间可以长达大约120小时。在一个实施例中,生产期过程中,发酵培养基中的细胞浓度通常为大约0.1至20,优选0.1至5,甚至更优选1至3g干燥细胞/升发酵培养基的范围内。根据途径所需,发酵可以在需氧、微氧或厌氧条件下进行。如果需要,氧摄取速率(our)可以作为过程控制环节在发酵期间变化(参见例如wo03/102200)。在一些实施方案中,本文中提供的经修饰的酵母细胞在微氧条件下进行培养,其特征在于氧摄取速率为2至45mmol/l/hr,例如2至25、2至20、2至15、2至10、10至45、15至40、20至35或25至35mmol/l/hr。在某些实施方案中,当在微氧条件下培养时,本文中提供的经修饰的酵母细胞可以表现尤其出色,所述微氧条件的特征在于氧摄取速率为2至25mmol/l/hr。在生产期过程中,可以对培养基进行缓冲,以致ph保持在大约3.0至大约7.0或大约4.0至大约6.0的范围内。适宜的缓冲剂是随着酸的形成能够将其中和的碱性物质,包括例如氢氧化钙、碳酸钙、氢氧化钠、氢氧化钾、碳酸钾、碳酸钠、碳酸铵、氨水、氢氧化铵等等。总的来讲,已经用于常规发酵过程中的那些缓冲剂也适用于此处。在利用经缓冲的发酵的那些实施方案中,可以随着酸性发酵产物的形成将其中和为相应的盐。在这些实施方案中,酸的回收包括游离酸的再生。这可以通过去除细胞并用强酸例如硫酸使发酵液酸化来进行。这导致形成盐类副产物。例如,当钙盐用作中和剂并且硫酸用作酸化剂时,产生盐类副产物石膏。将该副产物从肉汤中分离,并利用例如液-液萃取、蒸馏、吸收以及其他技术回收酸(参见例如t.b.vickroy,vol.3,chapter38ofcomprehensivebiotechnology,(m.moo-young编),pergamon,oxford,1985;r.datta等,femsmicrobiolrev,1995,16:221-231;u.s.专利nos.4,275,234、4,771,001、5,132,456、5,420,304、5,510,526、5,641,406和5,831,122以及wo93/00440)。在其他实施方案中,可以允许发酵培养基的ph由3-hppka处或以上,通常为4.5或更高的起始ph,降至酸性发酵产物的pka处或以下,例如低于4.5或4.0,例如在大约1.5至大约4.5的范围内、大约2.0至大约4.0的范围内或大约2.0至大约3.5的范围内。在其他实施方案中,可以进行发酵,以通过在发酵过程之前或初始将发酵液的ph调节至产物酸的pka处或以下,从而产生产物酸。其后在培养期间,ph可以保持在产物酸的pka处或以下。在某些实施方案中,ph可以保持在低于4.5或4.0,例如在大约1.5至大约4.5的范围内、大约2.0至大约4.0的范围内或大约2.0至大约3.5的范围内。在本文中提供的方法的某些实施方案中,经遗传修饰的酵母细胞产生相对低水平的乙醇。在某些实施方案中,可以产生产率为10%或更低的乙醇,优选产率为2%或更低。在这些实施方案的某些中,产生的乙醇不可检测出。然而在其他实施方案中,3-hp和乙醇可以同时产生。在这些实施方案中,可以产生产率为高于10%、高于25%或高于50%的乙醇。在本文中提供的方法的某些实施方案中,3-hp在碳源上的终产率是至少10%、至少20%、至少30%、至少40%、至少50%或高于50%的理论产率。3-hp的浓度或效价将是产率以及碳源起始浓度的函数。在某些实施方案中,在发酵过程中的某些时间点,优选发酵结束时,效价可以达到至少1-3、至少5、至少10、至少20、至少30、至少40、至少50或高于50g/l。在某些实施方案中,可以通过尤其是在生产期过程中改变发酵培养基的温度,从而使3-hp的终产率增加。一旦产生,可以使用本领域已知的任何方法从发酵培养基中分离3-hp。例如,可以利用普通的分离技术从培养液中去除生物质,并且可以利用普通的离析步骤(例如萃取、蒸馏和离子交换步骤)从不含微生物的培养液中获得3-hp。此外,3-hp可以在其产生时进行分离,或者其可以在产物生产期已经结束后从培养液中分离。可以将利用本文中公开的方法产生的3-hp化学性地转化为其他有机化合物。例如,可以使3-hp氢化,以形成1,3丙二醇,这其是一种有价值的聚酯单体。丙二醇还可以利用具有氧化还原酶活性的多肽在体外或体内由3-hp产生。可以利用任何方法使有机酸例如3-hp氢化,例如用于使琥珀酸和/或乳酸氢化的那些。例如,可以利用金属催化剂使3-hp氢化。在另一个实例中,可以利用任何已知进行脱氢反应的方法使3-hp脱氢,从而形成丙烯酸。例如,可以在催化剂(例如金属或无机酸催化剂)存在下对3-hp进行加热,以形成丙烯酸。提供下面的示例来更好地说明所请求保护的本发明,且不应被理解为对本发明范围的限制。特定材料所被提及的程度,仅仅用于说明的目的,而并不是打算对本发明进行限制。在不行使创造性能力和不超出本发明范围的情况下,本领域技术人员可以开发出等同的方法和反应物。应当理解,可以保持在本发明的范围之内,对本文所描述的步骤作出许多变化。发明人的意为这些变化都包括在本发明的范围之内。实施例培养基和溶液te由10mmtris碱和1mmedta,ph8.0构成。2xyt+amp平板由16g/l胰蛋白胨,10g/l的酵母提取物,5g/l的nacl,100mg/l的氨苄青霉素和15g/l的bacto琼脂构成。ura选择平板由6.7g含硫酸铵酵母基础氮源,5g酪蛋白氨基酸,100ml0.5m,ph5的琥珀酸,20gnoble琼脂以及855ml去离子水构成。高压蒸汽灭菌后,加入40ml的无菌50%葡萄糖和2ml10mg/ml的氯霉素(chloraphenicol),然后倾倒平板。ura选择培养基由6.7g含硫酸铵酵母基础氮源,5g酪蛋白氨基酸,100ml0.5m,ph5的琥珀酸以及855ml去离子水构成。高压蒸汽灭菌后,加入40ml的无菌50%葡萄糖和2ml10mg/ml的氯霉素。yp+10%葡萄糖培养基由500mlyp培养液和100ml无菌50%葡萄糖构成。yp培养液由10g/l的酵母提取物,20g/l的蛋白胨构成。ypd平板由10g酵母提取物,20g蛋白胨,20gbacto琼脂,以及去离子水加至960ml构成。高压灭菌后,加入40ml的无菌50%葡萄糖,并倾倒平板。tae由4.84g/l的tris碱,1.14ml/l冰醋酸和2ml/l的0.5medta,ph8.0构成。tbe由10.8g/l的tris碱,5.5g/l的硼酸,和4ml/l的0.5medta,ph8.0构成。lioac/te溶液由为8份的无菌水,1份的1mlioac以及1份的10×te构成。10×te(200ml)由2.42gtris碱,4ml0.5m的edta,ph8.0构成,用5m的hcl调整ph值至7.5,用高压蒸汽灭菌器对溶液进行灭菌。peg/lioac/te溶液由8份50%的peg3350,1份1m的lioac,以及1份10×te构成。50%peg3350的制备方法为:将100gpeg3350加入到150ml水中,并加热和搅拌直至溶解。然后用水将体积补至200ml,高压蒸汽灭菌。scdfoa平板由275ml的2x-scd2xfoa液体培养基和275ml的2xscd2xfoa平板培养基构成,熔化并冷却至65℃.2x-scd2xfoa液体培养基由6.66g不含氨基酸的酵母基础氮源,1.54gura-do补充物(clontech,moutainview,ca,usa),20g葡萄糖,50mg尿嘧啶,以及2g5-foa(5-氟乳清酸,一水合物;torontoresearchchemicals,northyork,on,canada)和水加至1l构成。将得到的溶液过滤除菌。2xscd2xfoa平板培养基由11gbacto琼脂和275ml水构成。将得到的溶液高压蒸汽灭菌。dm2培养基由硫酸铵(5.0g/l),七水硫酸镁(4.50.5g/l),磷酸二氢钾(3.0g/l),微量元素溶液(1ml/l)和维生素溶液(1ml/l)构成。在所有培养基组分溶解之后,用合适的碱(例如,koh)将培养基的ph调节到所需的初始ph值。微量元素溶液由edta(15.0g/l),七水硫酸锌(4.5g/l),脱水氯化镁(1.0g/l),六水氯化钴(ii)(0.3g/l),五水硫酸铜(ii)(0.3g/l),脱水二钠钼(disodiummolybdenumdehydrate)(0.4g/l),脱水氯化钙(4.5g/l),七水硫酸亚铁(3g/l),硼酸(1.0g/l),以及碘化钾(0.1g/l)构成。维生素溶液由生物素(d-,0.05g/l),泛酸钙(d+;1g/l),烟酸(5g/l),myo-肌醇(25g/l),盐酸吡哆醇(1g/l),对氨基苯甲酸(0.2g/l),以及盐酸硫胺(1g/l)构成。dm1x-α-gal平板由dm1盐,20g/l的葡萄糖,微量元素溶液,维生素溶液,2ml/l的x-α-gal(16mg/ml),20g/l的琼脂构成。dm1盐溶液由2.28g/l的尿素,3g/l的磷酸二氢钾,以及0.5g/l的七水硫酸镁构成。butterfield磷酸缓冲液由1.25ml/l的储备溶液(26.22g/l磷酸二氢钾和7.78g/l的碳酸钠),以及5ml/l的氯化镁溶液(81.1g/lmgcl2-6h2o)构成。将得到的溶液高压蒸汽灭菌,调节ph至7.2。cnb1摇瓶培养基由尿素(2.3g/l),七水硫酸镁(0.5g/l),磷酸二氢钾(3.0g/l),微量元素溶液(1ml/l)和维生素溶液(1ml/l),葡萄糖(120.0g/l),2-(n-吗啉代)乙磺酸(mes)(97.6g/l)构成。在所有培养基组分溶解后,用合适的碱(例如,koh)将培养基的ph调节为初始ph5.8。表0:引物序列各种序列启动子:在实施例中描述的pdc,tdh3,eno1和pgk1启动子分别来源于东方伊萨酵母的seqidno:244,245,246和247序列。终止子:实施例中描述的tal,tkl,rki和pdc终止子分别来源于东方伊萨酵母的seqidno:248,249,250和251序列。实施例中描述的ura3启动子,orf和终止子来源于东方伊萨酵母seqidno:252序列。实施例1a:基于3-hp抗性选择酵母宿主细胞检测一组野生型酵母株在3-hp存在的情况下的生长能力。通过评估7个野生酵母株(表1,组a)在含有不同水平3-hp的培养基上的生长能力来确定在初筛过程中所使用的3-hp浓度范围。这些实验中使用的3-hp是由丙烯酸水溶液(30%)和co2催化剂在200psi和175℃下通过化学合成得到的,如wo04/076398中所描述的。用异丙醚在室温下进行逆向萃取以除去残留的丙烯酸(见wo05/021470)。细胞在ypd平板上划线,培养过夜。在ph3.0的ypd培养基中制备od600约为4的细胞浆。该浆状物用于接种到含有不同浓度3-hp(ph3.9)的微滴定孔中,至od600为0.05。用透气膜覆盖平板,在30℃/300rpm的摇床中孵育过夜。在genios模型平板读数器(tecan)中对每个孔测量600nm波长处的光密度,并肉眼观察平板的生长情况。一个或多个菌株所生长的最高3-hp浓度(125g/l)被选为初筛步骤的上限值。初筛对于初筛步骤,利用与范围确定相同的步骤,对生长在含有0g/l,75g/l,100g/l或125g/l3-hp(ph3.9)的微滴定板上的89个野生型酵母菌株进行筛选。对于每个菌株都使用新鲜的ypd平板,并且在ph3.0的ypd培养基中制备od600约为4的细胞浆。该浆状物用于接种到含有不同浓度3-hp(ph3.9)的微滴定孔中至od600为0.05。用透气膜覆盖平板,在30℃/300rpm的摇床中孵育过夜。在genios模型平板读数器中对每个孔测量600nm波长处的光密度,并肉眼观察平板的生长情况。实施类似的步骤,以评估在乳酸浓度为0g/l,30g/l,45g/l和60g/l时的生长情况。表1总结了观察到生长的最高3-hp或乳酸浓度。鉴定出15个能在100g/l的3-hp上生长或者在75g/l的3-hp上生长良好的菌株(表1,b组)。为了进一步缩小菌株的范围,实行了第二微滴定板检测。该检测与第一次的检测类似,但是所使用的3-hp浓度为100g/l,112.5g/l,和125g/l(ph3.9)。从这个检测中鉴定出了11个在75g/l的3-hp上生长良好或在75和112.5g/l的3-hp上都展现出一些生长的菌株(表1,c组)。将这11个菌株进行二次筛选。四株在75g/l的3-hp上生长情况不好以及在112.5g/l的3-hp上没有生长的菌株并没有进行二次筛选。据预测,没有进行二次筛选的菌株在商业发酵中也展示出较差的经济表现。不过可能一个或多个这样的菌株均能满足商业上可行的发酵过程的最低要求。表1:在3-hp或乳酸上的生长上面所测试的东方伊萨酵母菌种cd1822是通过在葡萄糖限制恒化器中使东方伊萨酵母atccpta-6658进化91天产生的。用含有15g/l右旋葡萄糖的dm培养基供给该系统,通过将乳酸加入到供给培养基中,在ph=3,稀释率为0.06h-1下进行操作。用大概2mmoll-1h-1的低氧传输速率来维持该条件,而溶解氧的浓度保持恒定在0%的空气饱和度。初始时间点的单菌落分离物在两个摇瓶分析中被表征出。在第一个分析中,菌种的特征在于它们在25g/l的总乳酸存在下,在无需对ph校正的dm1确定培养基(dm1definedmedium)中,能够将葡萄糖发酵成乙醇。在第二分析中,在存在25,32和45g/l的总乳酸的情况下,在无需对ph校正的dm1确定培养基中测定分离物的生长速率。cd1822菌株是基于所测量的发酵速率和生长速率所选择出来的单一分离物。其他进化东方伊萨酵母的方法包括,但不限于在选择条件下重复系列传代培养,如us专利号7629162中所描述。此类方法可以用来代替,或补充上述的葡萄糖限制恒化器法。本领域技术人员可以理解的是,可以使用类似的进化步骤,添加3-hp而不是乳酸来开发改进的3-hp抗性,来产生菌株。此外,在选择前也可以对菌株进行诱变,如本文所述(例如,见实施例1b)。二次筛选对于二次筛选的第一部分,在ph3.9,含有0g/l,35g/l,或75g/l3-hp的ypd培养基中测量生长速率。用收获自种子瓶的生物质接种摇瓶,所述种子瓶过夜生长至od600为6至10。将250ml生长速率受困的瓶子(50ml工作体积)接种至od6000.1,在250rpm和30℃下生长。在分析的整个时间进程中获取样品,并通过od600对生物质生长进行分析。绘制所得到的od600数据,确立生长速率。表2:3-hp中的生长速率(μ)菌株0g/l3-hp35g/l3-hp75g/l3-hp东方伊萨酵母atcc605850.560.510.29东方伊萨酵母cd18220.620.520.28郎比可假丝酵母atcc386170.650.530.30粗壮假丝酵母0.510.320.14东方伊萨酵母pta-66580.610.530.32东方伊萨酵母242100.580.510.26博伊丁酵母mya4020.530.450.28膜酺毕赤酵母0.410.390.32博伊丁酵母mya4040.550.440.27栗酒裂殖酵母0.410.350.21zygosaccharomyceslentus0.610.410.20对于二次筛选的第二部分,测量ph3.9,含有100g/l葡萄糖和0g/l,35g/l或75g/l3-hp的ypd培养基中相同10个菌种的葡萄糖消耗量。用收获自种子瓶的生物质接种摇瓶,所述种子瓶过夜生长至od600为6至10。将250ml生长速率受困的瓶子(50ml工作体积)接种至od6000.1,在250rpm和30℃下生长。在分析的整个时间进程中获取样品,并利用yellowspringsinstruments(ysi)的2700biochemistryanalyzer分析葡萄糖消耗量。绘制所得到的od600数据,确立葡萄糖消耗速率。表3:3-hp中的葡萄糖消耗速率(g/l/hr)在75g/l的3-hp(ph3.9)条件下,有四个菌种(膜酺毕赤酵母,栗酒裂殖酵母,粗壮假丝酵母和z.lentus)并未达到2.5g/l/hr的葡萄糖利用率,而该条件则是经济的发酵过程所必需的。为了鉴定3-hp的主要菌株,将菌株的表现分为三类等级。这些类别中的两个是基于生长速率的不同方面:1)在最高酸浓度上的生长速率和2)所绘制的生长速率相对于酸浓度的斜率。第三个类别是在最高酸浓度时的糖酵解速率。该分级是通过将每个评级的最高和最低值作为标准化的边界,在标准化的范围内完成的。因此,对于每个类别,每个菌株都获得一个0和1之间的评级,其中1是可能的最高得分。一个菌株的总评级值是三个类别标准化值的总和。所制定权重值中生长速率和糖酵解速率权重相同。在这种情况下,在最高酸浓度下的糖酵解速率权重为50%,而两个生长速率评级每个的权重为25%。每个类别的标准化数值和总和以及权重值都归纳在表4中。表4:3-hp中的菌株级别在所检测的菌株中,来自于东方伊萨酵母,郎比可假丝酵母,和博伊丁酵母种的菌株呈现在低ph下作为3-hp生产宿主的最大潜力。利用相同的方法,用ph值为2.85,含有0,25,和50g/l乳酸(~80%游离酸)的培养基,来对初筛中的原始的91个野生型酵母菌株进行筛选,评级和评分。对来自于13个菌株的标准化值和权重和总得分值进行二次筛选。表5:乳酸中的菌株级别对于乳酸,只有s.javensis在ph2.85,含有50g/l乳酸的培养基中没能达到2.5g/l/hr的葡萄糖利用率。而东方伊萨酵母,郎比可假丝酵母和博伊丁酵母对3-hp和乳酸都展示出酸耐受,很多菌株只是对一种酸产生了耐受。这些也可以在初筛的结果中看到(表1)。例如,c.milleri,皱褶假丝酵母,c.vanderwaltii,k.ohmeri,贝酵母,s.javensis,y.lipolytica,z.bisporus和z.kombuchaensis都表现在45-60g/l乳酸上生长,而不在甚至是所检测的最低浓度的3-hp上生长(35g/l)。因此,菌株对于一种有机酸的耐受性并不能明确地用于预测其对于其他酸类的耐受性。这得到了进一步的凸显,即通过将上述呈现3-hp抗性的菌株与wo03/049525中所鉴定的8个优选能作为有机酸生产宿主的菌株列表进行对比。其中那些菌株中的两株(c.diddensiae和c.entomophila)无法获得来进行测试,其他6株都包括在上述的初筛之中。在这6株中,只有c.krusei(作为东方伊萨酵母进行测试)能够在35g/l的3-hp存在的情况下生长。实施例1b:诱变和选择具有3-hp抗性的突变菌株对在实施例1a中选择出来的酵母细胞进行诱变,并将其暴露于选择压力下,以便鉴定具有高3-hp抗性的突变株。例如,新鲜yp(酵母提取物/蛋白胨)+20g/l的葡萄糖平板或液体培养(od600为1-4)中的酵母细胞被悬浮于无菌水中,至od600为10左右。200μl该细胞悬液的等分试样被滴加入各试管中,暴露于3μl的甲磺酸乙酯(ems)中,约1小时,这杀灭约65%的细胞。可以使用更高的ems浓度来提高杀灭率。在暴露之后,用5%的硫代硫酸钠中和细胞,在pbs缓冲液中洗涤,在营养充足的培养基中恢复约4小时,并在选择培养基中培养。同样进行模拟样本(无ems),以确保条件是选择性的。另外,也可以使用紫外线照射来诱变细胞。0为了选择3-hp抗性的突变菌株,将ems-处理过的细胞悬液(约2×108个诱变细胞)的等分试样接种到马铃薯葡萄糖琼脂(pda)或另外的培养基上,该培养基所含有的3-hp的水平使亲本菌株所不能生长的或生长得很慢。将这些平板孵育几天,直至菌落出现。纯化单个菌落,在非选择培养基上划线,以消除选择的任何适应性影响,然后在选择培养基上重新测试,以确认抗性的提高。然后通过对产物和底物定期取样进行hplc分析,在摇瓶中测试抗性菌株。另外,对3-hp抗性的选择可以通过恒化器或系列摇瓶进化来完成。可以再进行几轮诱变和选择。诱变可用于提高天然并不能满足3-hp的生产需求的宿主的抗性,以便它具有用于商业生产3-hp所需的属性。实施例2a:将dna转化进宿主基因组的步骤将dna转化进酵母宿主的基因组中,以产生在下面实施例中所述的经修饰的酵母菌株,是基于下述的特定步骤来实施的。将4mlyp+10%葡萄糖的培养基加入到14mlfalon管中,使用无菌环将所需的菌株接种到该培养基中。培养物在37℃下以250rpm振荡培养过夜(~16hr)。1ml的过夜培养物被加入到250ml含有50ml液体yp+10%葡萄糖培养基的挡板烧瓶中。烧瓶在37℃下以250rpm振荡培养。以约每小时的间隔时间取出少量等量的培养物,并测量od600。培养物生长至od600为0.6-1.0。室温下以2279×g离心收集细胞,将沉淀物重悬于25ml无菌水中,然后在室温下以2279×g离心。将沉淀重悬于1ml无菌水中,将重悬细胞转移至1.5ml管中,然后在16100×g下沉淀。将细胞重悬于1mllioac/te溶液中,然后在16100×g下沉淀。细胞沉淀物重悬于500μllioac/te溶液中。将下列组分加入到1.5ml管中:100μl上述细胞,10μl新鲜煮沸再冷冻的鲑鱼精子dna(agilenttechnologies,santaclara,ca,usa),以及10μl所需的线性转化dna。制备用水代替了dna的对照反应。对于每个转化反应,加入600μlpeg/lioac/te溶液,在30℃下,250rpm的摇床中每个反应在其一侧孵育30分钟。将40μldmso加入到每个反应中,然后在42℃的水浴中孵育5分钟。在5400×g下沉淀细胞1分钟。细胞重悬于水中,分为两份,将转化反应的每一半都涂布到ura选择培养平板上。平板被放置在37℃下。经过18至24hr的生长后,一般都可见菌落,这取决于菌株背景。用无菌环将小量的酵母从培养皿转移到含有300μl酵母裂解溶液(biotechenologies,madison,wi,usa)的1.5ml管中,并使用masterpuretmyeastdnapurificationkit(biotechenologies)根据制造商说明提取基因组dna。用masterpuretmyeastdnapurificationkit(biotechenologies)制备的基因组dna被用于pcr反应,以确定在分离的转化体中是否发生了正确的整合事件。pcr反应体系(25μl)包含0.5μl待筛选菌株的基因组dna,1×crimsontaqtm反应缓冲液(newenglandbiolabs,ipswich,ma,usa),有义和反义引物各25pmol,datp,dctp,dgtp和dttp各200μm,以及0.625单位的crimsontaqtmdna聚合酶(newenglandbiolabs)。在(eppendorfscientific,westbury,newyork,usa)中进行pcr,其程序为一个循环的95℃30秒,接着是30个循环,每个循环95℃30秒,50℃30秒,68℃最大预期产物的每kbp1分钟,最后68℃延伸10分钟。热循环之后,在tae或tbe中用1%的琼脂糖凝胶电泳分离pcr反应产物,观察和解释如之前描述的特异性引物对所产生的条带的大小。实施例2b:插入位点的选择适合于将外源基因掺入到宿主酵母细胞中的插入位点可以是基因的基因座,当从酵母宿主细胞中缺失所述基因的基因座时,将会对3-hp的生产带来有益的或中性的影响。对于所选择的酵母菌株的合适插入位点的非限制性示例描述在本文所进行的实施例中。本领域技术人员可以很容易地应用本发明的教导来使用这些和其他的插入位点,例如,一个或多个pdc的基因座(例如,编码如seqidno:49所示的氨基酸序列和/或包括如seqidno:48所示的核苷酸序列的编码区域的东方伊萨酵母pdc基因),adh基因座(例如,编码如seqidno:106,108或110所示的氨基酸序列和/或包含如seqidno:105,107或109所示的核苷酸序列的编码区域的东方伊萨酵母adh基因),gal6基因座(例如,编码如seqidno:112所示的氨基酸序列和/或包含如seqidno:111所示的核苷酸序列的编码区域的东方伊萨酵母gal6基因),cyb2a基因座(例如,编码如seqidno:114所示的氨基酸序列和/或包含如seqidno:113所示的核苷酸序列的编码区域的东方伊萨酵母cyb2a基因),cyb2b基因座(例如,编码如seqidno:116所示的氨基酸序列和/或包含如seqidno:115所示的核苷酸序列的编码区域的东方伊萨酵母cyb2b基因),gpd基因座(例如,编码如seqidno:118所示的氨基酸序列和/或包含如seqidno:117所示的核苷酸序列的编码区域的东方伊萨酵母gpd基因),ald基因座(例如,编码如seqidno:120所示的氨基酸序列和/或包含如seqidno:119所示的核苷酸序列的编码区域的东方伊萨酵母ald同源基因5680,编码如seqidno:122所示的氨基酸序列和/或包含如seqidno:121所示的核苷酸序列的编码区域的东方伊萨酵母ald同源基因42026,编码如seqidno:124所示的氨基酸序列和/或包含如seqidno:123所示的核苷酸序列的编码区域的东方伊萨酵母ald同源基因42426,编码如seqidno:126所示的氨基酸序列和/或包含如seqidno:125所示的核苷酸序列的编码区域的东方伊萨酵母ald同源基因42727),或pck基因座(编码如seqidno:128所示的氨基酸序列和/或包含如seqidno:127所示的核苷酸序列的编码区域的东方伊萨酵母pck基因)或其同源物。如果这些基因的序列并未公开,则可以利用标准步骤获得其序列,例如基因组测序,基因组或cdna文库的探针杂交,或者基于已知同源序列设计的简并引物扩增基因片段,然后再进行基因组步行以获得全序列。插入位点的其他的合适位置包括不含有开放阅读框的基因间隔区。实施例2c:插入载体,选择标志物盒,基因表达盒和整合构建体的技术产生插入位点载体以将一个或多个外源性基因整合进宿主酵母细胞中。宿主酵母细胞可以是经历过如实施例1中所描述的选择过程的细胞,或其可为未经诱变和/或选择的细胞。为了产生插入位点载体,用宿主基因组dna作为模板扩增所需插入位点的上游区域(5’)和下游区域(3’)。上游区域优选大于70bp并小于1.5kbp。将所得的靶序列同时或顺序连接到克隆载体中,以获得含有每条片段的一个拷贝的载体,使得这些片段是连续的或者几乎是连续的。可在片段之间掺入一个独特地限制性位点,使得能够插入基因表达盒和/或选择标志物盒。独特地限制性酶切位点可以掺入至或者靠近上游片段的5’端,以及下游片段的3’端,以允许将来可以将这些位点间的dna从克隆载体中移除。用于掺入至插入位点载体的选择标志物盒由常规克隆技术所产生。这些选择标志物盒包含可选择性标记的基因,并且也可含有上游启动子和/或下游终止子序列。合适的选择标志物基因的示例包括ura3,trp1,his,mel5,cyb2a,leu2,和g418基因。侧翼序列可以在启动子/标记基因/终止子序列的任一侧被掺入表达盒,以允许将来通过重组敲除该标记。这些侧翼序列可以包括直接的或倒置的重复序列(可以是功能性的或非功能性的序列)或一个或多个loxp位点。基因表达盒利用常规克隆技术所产生。这些基因表达盒含有待过表达的基因,也可含有上游启动子和/或下游终止子序列。在某些实施方式中,可以将这些启动子/基因/终止子组合的两个或多个拷贝掺入到单个基因表达盒中。为了促进其在宿主酵母菌株中的表达,异源基因可以经过密码子优化。本文中所述的选择标志物盒可以克隆进基因表达盒中,使得它与待过表达的基因以及任何相关的启动子和/或终止子是连续的或几乎连续的。或者,为了将天然启动子替换为外源启动子,表达盒可以将待整合的启动子上游的选择盒整合进靶向序列之间。可以利用常规克隆技术将基因表达盒插入到本文所述插入位点载体的两个靶位点序列之间,以产生基因表达整合构建体。一个或多个选择标志物盒也可以插入到靶序列之间,无论是作为基因表达盒的一个部分还是独立插入。在某些变化方式中,基因表达盒的片段可以被克隆到不同的插入位点载体中,以使得这些整合片段之间有共同的重叠片段。例如,一个载体可能含有上游插入片段,启动子,基因和终止子,第二个载体可能含有终止子,选择标志物盒和下游插入片段。在另一个实例中,为了允许同时插入两个基因,一个载体可能含有上游插入片段,启动子,基因,终止子以及所有或部分的选择标志物盒,第二个载体可能含有所有或部分的选择标志物盒,第二启动子,基因,终止子,以及下游插入片段。为了产生基因敲除构建体,使用来源于待缺失或破坏的基因上游和下游侧翼区域的靶dna序列来制作插入位点载体。所选择的靶序列可以包括靶基因的上游和下游侧翼区域和/或所有或部分的靶基因编码序列或其调节元件(例如,启动子或终止子)。一个或多个选择标志物盒可以被掺入到插入位点载体中的两个靶序列之间。当敲除与外源基因的表达偶联时,还将一个或多个基因表达盒被掺入插入位点载体中。可以通过限制性酶消化来自克隆载体的片段或来自多个载体的重叠片段来使待整合到宿主酵母基因组中的dna片段线性化。或者,可以使用pcr,或者pcr与限制性酶切的组合来产生线性的整合片段。插入位点侧翼区域可以是由于其本身存在于载体模板中或掺入到扩增引物中而掺入到整合片段中。在后一种情况中,优选侧翼区域最少是70个核苷酸掺入到引物中。合适的插入载体,选择标志物盒,基因表达盒,以及所选酵母菌株的整合构建体的非限制性示例描述于下面的具体实施例中。本领域技术人员能够很容易地应用从这些实施例中得到的教导以及前面的描述,从而产生其他修饰的能够生产3-hp的酵母菌株。实施例2d:在adh1202基因座表达外源基因的插入载体的构建构造质粒pmiba107以允许在东方伊萨酵母adh1202基因座掺入单个基因,该基因位于pdc启动子和终止子的控制之下,使用ura3作为选择性标记。pcr扩增有ura选择性标记pdc启动子和终止子,并克隆进如下所述的pcr4tm4blunt(invitrogen,lajolla,ca,usa)中。含有pdc启动子,终止子和ura3选择性标记的pcr片段由soepcr构建。pdc启动子的扩增引物在pcr产物的3’端含有pdc终止子的同源物,pdc终止子和ura3选择标志物的扩增引物在产物的5’端与pdc启动子同源。然后将这两个片段通过soepcr置于一起。用引物0611184和0611195从pacn5(图19)中扩增pdc启动子。引物0611184将noti限制性位点引入到pcr产物的5’端。引物0611195则将xbai限制性位点引入至pdc启动子后,并在pcr产物的3’端引入pdc终止子的同源物。根据制造商说明使用pfxdna聚合酶(invitrogen)来实施扩增反应。每个pcr反应含有0.5μl稀释的pacn5(图19),引物0611184和0611195各25pm,1×pfx扩增缓冲液(invitrogen),2mm的mgso4,0.2mmdntp混合物,1.25单位的pfxdna聚合酶(invitrogen),终体积为50μl。在(eppendorfscientific)中孵育扩增反应物,程序为95℃2分钟的1个循环,随后是每个为95℃30秒,55℃30秒和72℃2分钟的30个循环,及72℃3分钟的最后延伸。以89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)中1%琼脂糖凝胶电泳来纯化pcr产物。一条约700bp的片段被从胶上切下,并根据制造商的说明使用gelextracttion试剂盒(qiagen,valencia,ca,usa)从琼脂糖中提取。用引物0611189和0611185从phjj76(图24)中扩增pdc终止子和ura3选择标志物。引物0611189在pcr产物的5’端引入了pdc启动子的同源物以及直接在pdc终止子前面引入paci位点。引物0611185将一个noti限制性位点引入到pcr产物的3’端。根据制造商说明使用pfxdna聚合酶(invitrogen)来实施扩增反应。每个pcr反应含有0.5μl稀释的phjj76,引物0611189和0611185各25pm,1×pfx扩增缓冲液(invitrogen),2mm的mgso4,0.2mmdntp混合物,1.25单位的pfxdna聚合酶(invitrogen),终体积为50μl。在(eppendorfscientific)中孵育扩增反应物,程序为95℃2分钟的1个循环,随后是每个为95℃30秒,55℃30秒和72℃2分钟的30个循环,及72℃3分钟的最后延伸。以使用89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)的1%琼脂糖凝胶电泳来纯化pcr产物。一条约2000bp的片段被从胶上切下,并根据制造商的说明使用gelextracttion试剂盒(qiagen,valencia,ca,usa)从琼脂糖中提取。使用soe-pcr将2000bp的pdc终止子和ura3选择标志物pcr产物以及700bp的pdc启动子pcr产物融合。根据制造商说明使用pfxdna聚合酶(invitrogen)来实施扩增反应。每个pcr反应含有8ng2000bp的pdc终止子和ura3选择标志物pcr产物,24ng700bp的pdc启动子pcr产物,引物0611184和0611185各50pm,1×pfx扩增缓冲液(invitrogen),2mm的mgso4,0.2mmdntp混合物,2.5单位的pfxdna聚合酶(invitrogen),终体积为100μl。在(eppendorfscientific)中孵育扩增反应反应物,程序为95℃2分钟的1个循环,随后是每个为95℃30秒,55℃30秒和68℃3分钟的30个循环,和68℃3分钟的1个循环。以使用89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)的1%琼脂糖凝胶电泳来纯化2700bppcr产物。约2700bp的片段被从胶上切下,并根据制造商的说明使用gelextracttion试剂盒(qiagen,valencia,ca,usa)从琼脂糖中提取。根据使用制造商说明,用测序用zeropcr克隆试剂盒(invitrogen)将2700bp的pcr产物克隆进pcrtm4blunt(invitrogen)载体中。在6μl的反应总体积中,将1μl或4μl2700bp的pcr产物,1μl盐溶液(invitrogen)和1μlpcrtm4blunt(invitrogen)在室温下一起孵育15分钟。根据实验制造商说明,将每个克隆反应中取出2μl转化至onetop10chemicallycompetent大肠杆菌细胞(invitrogen)中。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。通过noti消化来筛选所得到的一些转化体,以筛选出所需pcr产物的合适插入。用dna测序来证实产生所需大小条带的克隆的正确性并将其命名为pmiba100。质粒phjj76(图24)含有同源性,以允许将基因整合到adh1202基因座上。用noti消化质粒phjj76,以移除存在于adh1202同源序列内部的ura3选择标志物。用89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)中1%琼脂糖凝胶电泳来纯化消化后的phjj76。使用gelextracttion试剂盒(qiagen,valencia,ca,usa)从琼脂糖中提取出一条5.2kbp长的片段,然后用t4dna连接酶连接到一起。根据制造商说明,将连接产物转化至onetop10chemicallycompetent大肠杆菌细胞(invitrogen)中。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。通过apai和saci消化来筛选所得到的一些转化体。产生所需消化产物的克隆被命名为phjj76-noura。将来自于pmiba100(见上)的pdc启动子和终止子和ura3选择标志物克隆进phjj76-noura中,以产生质粒,在该质粒中,为了在adh1202基因座进行整合,基因可以被置于pdc启动子和终止子的调控之下。用noti消化phjj76-noura,接着用cip处理。用pcrpurification试剂盒(qiagen)纯化该线性的5.2kbp片段。用noti消化pmiba100,并用89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)来实施1%琼脂糖凝胶电泳。约2742bp的片段被从胶上切下,使用gelextracttion试剂盒(qiagen,valencia,ca,usa)提取,然后用t4dna连接酶将其连接至phjj76-noura的5.2kbp片段上。根据制造商说明,将连接产物转化至onetop10chemicallycompetent大肠杆菌细胞(invitrogen)中。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。通过kpni和xbai消化来筛选所得到的一些转化体。产生所需消化产物的克隆命名为pmiba107(图2)。实施例2e:在pdc基因座表达多个外源基因的插入载体片段的构造可以使用下列的插入载体片段来产生预定的dna构建体,所述构建体是将内源性的东方伊萨酵母pdc基因替换为表达多个基因的表达盒,例如,本文所描述的从pdc,eno1和tdh3启动子表达的三个基因。左端构建体(pmhct068及其衍生体)和右端构建体(pmhct069及其衍生体)发生同源重组使ura3蛋白表达,导致该菌株由尿嘧啶营养缺陷型转化为尿嘧啶原养型,而允许对所需要的整合体进行选择。每个左端构建体的5’末端与pdc基因座的上游dna同源,而每个右端构建体的3’末端则与pdc基因座的下游dna同源。这些同源区域用于将表达盒靶向pdc基因座。在图3中示意性地示出了该靶向方法,并且可以利用本文所述的多个基因的任意组合来进行改进来靶向任意合适的位点,例如,上述的任意位点,例如adh基因座(见下述示例)或ald基因座。左端片段的构造对空的左端构建体载体,pmhct068,进行如下所述多个步骤的克隆。用引物0611166和0611167从东方伊萨酵母的基因组dna中扩增含有pdc启动子区域和所需的其它限制性位点以及侧翼dna的pcr片段。pcr反应体系(50μl)含有100ng东方伊萨酵母的基因组dna(例如,可使用来自于biotechnologies的masterpuretmyeastdnapurificationkit制备),1xthermopolreaction缓冲液(newenglandbiolabs),引物0611166和0611167每种100pmol,datp、dctp、dgtp和dttp每种200μm,2μl100mmmgso4,以及2个单位的(exo-)dna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为94℃2分钟的1个循环,随后是每个为94℃30秒,54℃30秒和72℃1分钟的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约780碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel,bethlehem,pa,usa)进行纯化。用引物0611168和0611169从pacn5(图19)中扩增含有tal终止子区域和所需的其它限制性位点以及侧翼dna的pcr片段。pcr反应体系(50μl)含有1μlpacn5小量制备质粒dna,1xiprooftmhf缓冲液(bio-radlaboratories,hercules,ca,usa),引物0611168和0611169每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso,以及1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的324个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约460碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。然后用pcr来构建融合了上述产物的单一扩增产物。pcr反应体系(50μl)含有125ng包含pcr产物的pdc启动子,76ng包含pcr产物的tal终止子,1xthermopolreaction缓冲液(newenglandbiolabs),引物0611166和0611169每种100pmol,datp、dctp、dgtp和dttp每种200μm,2μl100mmmgso4,以及2个单位的(exo-)dna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为94℃2分钟的1个循环,随后是每个为94℃30秒,54℃30秒和72℃1分30秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1250碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel,bethlehem,pa,usa)进行纯化。用引物0611170和0611171从pacn43(图22)中扩增含有eno1启动子区域和所需的其它限制性位点以及侧翼dna的pcr片段。pcr反应体系(50μl)含有1μlpacn43小量制备质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611170和0611171每种100pmol,datp、dctp、dgtp和dttp每种200μm,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1050碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。用引物0611172和0611173从pacn43(图22)中扩增pcr片段以及其它限制性位点和侧翼dna,该片段含有rki终止子区域,其后是ura3启动子区域以及ura3orf的5’末端。pcr反应体系(50μl)含有1μlpacn43小量制备质粒dna,1xiprooftmhf缓冲液(bio-radlaboratories,hercules,ca,usa),引物0611172和0611173每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso,以及1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1400碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。用pcr来构建融合了两个上述产物的单一扩增产物。pcr反应体系(50μl)含有93ng包含pcr产物的eno1启动子(见上),125ngrki终止子,ura3启动子和包含pcr产物的部分orf(见上),1xphusionhf缓冲液(newenglandbiolabs),引物0611170和0611173每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,56℃20秒和72℃2分45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约2460碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)进行纯化。为了为pcr产物创建接收载体,使用hindiii和ecori消化质粒pmhct017(具有一个无关插入的标准克隆载体puc19),用10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,和在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约2.6kbp的条带被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。所得到的hindiii至ecori纯化片段与puc18中发现的相同(yanisch-perron,c.,vieira,j.andmessing,j.(1985)gene,33,103-119)。用in-fusiontmadvantagepcrcloningkit(clontech)将上述纯化的1250bp和2460bppcr产物插入到消化后的pmhct017片段中,总反应体积为10μl,由125ngpmhct017hindiii至ecori载体片段,92ngpdc启动子和tal终止子pcr产物,165ngeno1启动子和ura3启动子以及含有pcr产物的部分orf,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。将该反应孵育在37℃下15分钟,50℃下15分钟,然后置于冰上。将该反应体系用40μlte缓冲液稀释,取出2.5μl根据制造商说明用于转化solopackglodsupercompetentcells(agilenttechnologies)。将转化体体涂布于2xyt+amp平板上,37℃孵育过夜。通过apali消化来筛选所得到的一些转化体,其中有所需pcr产物的正确插入。用dna测序命名来证实产生所需大小条带的克隆的正确性,并命名为pmhct068。质粒pmhct068含有pdc启动子区域,之后是nhei和asci限制性位点,所述限制性位点可用于本文所述的感兴趣的异位基因的加入,tal终止子,eno1启动子区域,在其后面的xbai和paci限制性位点(该限制性位点用于克隆本文所述的感兴趣的第二异位基因),rki终止子,东方伊萨酵母ura3启动子和东方伊萨酵母ura3orf的5’端。发现质粒pmhct068在进入pdc启动子中大约200bp处有一个a至t的核苷酸改变,在进入pdc启动子的约2/3处有g至t的改变,以及位于nhei限制性位点的5’侧翼有一个提前起始密码子(atg)。因此,pmhct068的正确版本如下构建。用引物0611166和0611828从pacn5(图19)中pcr扩增pdc启动子区域,该扩增并没有引入上述不期望的起始密码子。pcr反应体系(50μl)含有1μl1至50稀释度的pacn5小量制备质粒dna,1xthermopolreaction缓冲液(newenglandbiolabs),引物0611166和0611828每种100pmol,datp、dctp、dgtp和dttp每种200μm,2μl100mmmgso4,以及2个单位的(exo-)dna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为94℃2分钟的1个循环,随后是每个为94℃30秒,54℃30秒和72℃1分钟的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约780碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。如上所述的方法将含有pcr产物的pdc启动子融合到含有pcr产物的tal终止子上。由于tal终止子pcr片段是由0611168引物所得到的,因此所得到的pcr融合产物应当是混合物,即缺失提前起始密码子的产物和含有不期望的起始密码子的产物。纯化所得到的~1250bp的pcr产物,并如前所述,通过in-fusiontmadvantagepcrcloingkit(clontech)将其与rki终止子,ura3启动子和含有融合pcr产物和puc18的部分orf组合。dna测序显示产生预期apali消化图谱的克隆的正确性,包括pdc启动子中不存在突变和nhei限制性位点5’端的提前atg的缺失(这些是期望的),并命名为pmhct082。右端片段的构造对空的右端构建体载体,pmhct069,进行如下所述多个步骤的克隆。用引物0611174和0611175从pacn43(图22)中扩增含有东方伊萨酵母ura3orf3’末端,ura3终止子(ura3终止密码子下游275bp),ura3启动子(当整合进酵母宿主后,用作重复区域以环出标志物)以及期望的其它限制性位点和侧翼dna的pcr片段。pcr反应体系(50μl)含有1μlpacn43小量制备质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611174和0611175每种100pmol,datp、dctp、dgtp和dttp每种200μm,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1210bp的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。使用引物0611176和0611177从pacn23(图20)中扩增含有tdh3启动子区域和期望的其它限制性位点和侧翼dna的pcr片段。pcr反应体系(50μl)含有1μlpacn23小量制备质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611176和0611177每种100pmol,datp、dctp、dgtp和dttp每种200μm,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1028碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。用引物0611178和0611179从东方伊萨酵母基因组dna中扩增pcr片段,所述片段含有东方伊萨酵母pdc基因区域终止密码子的3’端区域(pdc终止子区域)和期望的另外限制性位点以及侧翼dna。pcr反应体系(50μl)含有100ng东方伊萨酵母的基因组dna,1xiprooftmhf缓冲液(bio-radlaboratories),引物0611178和0611179每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso,以及1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约938碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)纯化。用pcr来构建融合了两个上述最后产物的单一扩增产物。pcr反应体系(50μl)含有125ng包含pcr产物的tdh3启动子,114ng含有pcr产物的pdc终止子,1xphusionhf缓冲液(newenglandbiolabs),引物0611176和0611179每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,56℃20秒和72℃2分30秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1966碱基对的pcr产物被从胶上切下并根据制造商的说明使用extractii试剂盒(macherey-nagel)进行纯化。随后用in-fusiontmadvantagepcrcloingkit(clontech)将上述纯化的1210bp和1966bppcr融合产物插入到见上所述的hindiii和ecori消化后的pmhct017片段中,总反应体积为10μl,由125ngpmhct017hindiii至ecori载体片段,54ng含有ura3orf3’末端及其后ura3终止子的pcr产物,200ngtdh3启动子和pdc终止子融合pcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。将该反应孵育在37℃下15分钟,50℃下15分钟,然后置于冰上。将该反应体系用40μlte缓冲液稀释,取出2.5μl根据制造商说明用于转化solopackglodsupercompetentcells(agilenttechnologies)。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过apali消化来筛选所到一些转化体有所期望的pcr产物合适地插入。用dna测序来证实产生所需大小条带的克隆的正确性,并命名为pmhct069。质粒pmhct069含有东方伊萨酵母ura3标记的3’末端,相应的ura3终止子,ura3启动子(用于将来将ura3标记环出),tdh3启动子,用于亚克隆所需基因使其异位表达的xbai和paci限制性位点,以及pdc位点的3’侧翼区域。实施例2f:在adh9091基因座表达多个外源基因的插入载体片段的构造设计下面的插入载体片段的方法与实施例2e中所描述的类似,是为了将内源性的东方伊萨酵母adh9091基因替换为表达多个本文所述的感兴趣的基因的表达盒。左端片段的构造对空的左端构建体载体,pgrer125,进行如下所述多个步骤的克隆。含有在东方伊萨酵母adh9091基因座发生同源重组所需的5’端侧翼的构建体,以及pdc启动子/tal终止子空表达盒被pcr克隆进载体质粒pcr2.1-topo(invitrogen)。用引物0611250和0611251从质粒pacn5(图19)中pcr扩增pdc启动子片段。pcr反应体系(50μl)含有15ngpacn5质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611250和0611251每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约900bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约753bp,在该片段的5’末端有noti限制性位点,其3’末端有paci和xbai限制性位点。产生第二pcr片段以使用引物0611252和0611253从质粒pacn5(图19)中扩增tal终止子区域,所述第二片段含有上述pcr产物的5’同源物,包括xbai和paci限制性位点。pcr反应体系(50μl)含有15ng质粒pacn5dna(见上),1xphusionhf缓冲液(newenglandbiolabs),引物0611252和0611253每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,有72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约900bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约435bp,在该片段的5’末端有paci和xbai限制性位点,3’末端有pmei限制性位点。用引物0611250和0611253通过pcr将753bp的片段和435bp的片段融合,结果得到一条1149bp的片段,其中,pdc启动子在tal终止子的上游。pcr反应体系(50μl)含有125ng753bp片段,75ng435bp片段,1xphusionhf缓冲液(newenglandbiolabs),引物0611250和0611253每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1149bp对的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。产生pcr片段以使用引物0611254和0611255扩增东方伊萨酵母adh9091基因座的5’侧翼,所述第二片段含有上述1149bppcr产物的3’同源物,包括noti限制性位点。pcr反应体系(50μl)含有15ng质粒phjj27(图21)作为模板dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611254和0611255每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,有72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约900bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约891bp,在该片段的5’末端有hpai限制性位点,3’末端有noti限制性位点。用引物0611254和0611253,利用pcr将891bp片段融合到1149bp的pdc启动子/tal终止子片段上,产生约2005bp的片段。pcr反应体系(50μl)含有125ng的1149bp片段,95ng的891bp片段,1xphusionhf缓冲液(newenglandbiolabs),引物0611254和0611253每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃2分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约2005bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的2005bp片段,包含用于在adh9091基因座整合的5’侧翼,pdc启动子和tal终止子,被克隆进pcr2.1-topo载体中,使用topotacloningkit(invitrogen)根据操作说明转化进one-shottop10大肠杆菌细胞中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过avai消化来筛选到一些转化体有所期望的pcr产物的正确插入。产生所需大小条带的克隆被确认并命名为pgmer112。质粒pgmer112包含用于在adh9091基因座进行同源重组的5’侧翼,其后是空的表达盒pdc启动子/tal终止子。用引物0611283和0611263通过pcr从质粒phjj27(图21)中扩增由ura3启动子片段驱动的5’截短的ura3标记基因。pcr反应体系(50μl)含有15ng质粒phjj27,1xphusionhf缓冲液(newenglandbiolabs),引物0611263和0611283每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso,以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约900bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约885bp,在该片段的5’末端有hpai和pmei限制性位点,3’末端有nhei限制性位点。将所得到的885bp片段克隆进pcr2.1-topo载体中,使用topotacloningkit(invitrogen)根据制造商说明转化进one-shottop10大肠杆菌细胞中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过bgli消化来筛选到一些有所期望的pcr产物的正确插入的转化体。产生所需大小条带的克隆被确认并命名为pgmer108。质粒pgmer108包括ura3启动子,其后是ura3基因的5’截短片段,该片段的侧翼5’末端是hpai和pmei限制性位点,3’末端则是noti限制性位点。将来自于质粒pgmer112(见上)的1998bp的hpai和pmei限制性片段连接至由hpai和pmei线性化的来自于pgmer108(见上)的4806bp载体上,所述限制性片段包含5’adh9091侧翼,其后是构建体pdc启动子/tal终止子。以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离双限制性酶切反应产物,其中约1998bp的插入片段和4806bp的载体片段被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。使用1:3的载体:插入片段比例来进行连接反应,特别的,建立的反应体系有2μl4806bp线性化载体,6μl1998bp插入片段,9μl2xquickligation反应缓冲液以及1μlquickt4dna连接酶(newenglandbiolabs),根据制造商说明来实施。根据制造商说明,将5μl连接产物转化到大肠杆菌ultracompetentcells(agilenttechnogogies)中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过hindiii消化来筛选到一些有所期望的pcr产物的正确插入的转化体。产生所需大小条带的克隆被确认并命名为pgmer117。质粒pgmer117包括5’adh9091侧翼,其后是空的表达盒pdc启动子/tal终止子以及由ura3驱动的截短的5’ura3基因。此外,质粒pgmer117有两个不同的xbai限制性位点:第一个限制性位点位于pdc启动子和tal终止子之间(靠近限制性位点paci),该位点可用于插入感兴趣的基因,第二个xbai限制性位点则来自于原始pcr2.1-topo的骨架。为了消除这第二个xbai限制性位点,用apai消化质粒pgmer117,然后根据制造商说明用酶dna聚合酶i,大(klenow)片段(newenglandbiolabs)处理线性化的质粒。将得到的线性化质粒(含有平末端)用限制性酶ecorv消化,其从含有xbai限制性位点载体中将一条43bp的片段切割出来。以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离限制性酶切反应产物,并将约6761bp的载体片段被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。建立自连接反应体系,其具有3μl线性化载体,6μl灭菌双蒸水,10μl2xquickligation反应缓冲液以及1μlquickt4dna连接酶(newenglandbiolabs),根据制造商说明来实施。根据制造商说明,将5μl连接产物转化到大肠杆菌ultracompetentcells(agilenttechnogogies)中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过xbai消化来筛选到一些有所期望的pcr产物的正确插入的转化体。产生所需大小条带的克隆被确认并命名为pgmer122。用引物0611295和0611296从质粒pacn43(图22)中pcr扩增eno1启动子片段。pcr反应体系(50μl)含有15ngpacn43质粒,1xphusionhf缓冲液(newenglandbiolabs),引物0611295和0611296每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs)以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1009bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)纯化。所得到的pcr片段的总长度约为1009bp,在片段的5’末端具有pmei限制性位点,在3’末端有apai和nrui限制性位点。产生第二pcr片段以使用引物0611297和0611298从质粒pacn43(图22)中扩增rki终止子,所述第二片段含有上述pcr产物的5’同源物,包括nrui和apai限制性位点。pcr反应体系(50μl)含有15ng质粒pacn43dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611297和0611298每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约438bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约438bp,在该片段的5’末端有nrui和apai限制性位点,3’末端有pmei限制性位点。用引物0611295和0611298通过pcr将1009bp的启动子片段和438bp的终止子片段融合在一起,产生一条约1447bp的片段,该片段中eno1启动子位于rki终止子上游。pcr反应体系(50μl)含有125ng1009bp片段,65ng438bp片段,1xphusionhf缓冲液(newenglandbiolabs),引物0611295和0611298每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃2分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1447bp对的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的1447bp的片段,包含eno1启动子/rki终止子构建体,克隆进pcr2.1-topo载体中,使用topotacloningkit(invitrogen)根据制造商说明转化进one-shottop10大肠杆菌细胞中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过bamhi消化来筛选到一些有所期望的pcr产物的正确插入的转化体。产生所需大小条带的克隆被确认并命名为pgmer114,其包含空的表达盒eno1启动子/rki终止子。用限制性酶pmei在37℃下对质粒pgmer122和pgmer114消化3小时。在终止各个消化反应前1小时,向每个消化管中加入1μl牛肠碱性磷酸酯酶(newenglandbiolabs),以使得末端去磷酸化防止自连接。所得到的来自于质粒pgmer122的6761bp载体片段,以及包含来自于质粒pgmer144的构建体eno1启动子/tal终止子(1439bp)的插入片段,在1xtbe缓冲液中通过0.8%琼脂糖凝胶电泳来分离,并从凝胶中切割下来,根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。随后制备后续的连接反应,其包括3μl来自于质粒pgmer122的载体片段,4μl来自于质粒pgmer114的插入片段,2μl无菌dd水,10μl2xquickligation反应缓冲液以及1μlquickt4dna连接酶(newenglandbiolabs),根据制造商说明来实施。根据制造商说明,将5μl上述连接反应的等分试样转化到大肠杆菌ultracompetentcells(agilenttechnogogies)中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过xbai和bstbi消化来筛选到一些有所期望的pcr产物的正确插入的转化体。产生所需大小条带的克隆通过测序被确认并命名为pgmer125。eno1启动子/rki终止子构建体以相反的取向被插入,导致了两个版本的命名为(a)和(b)的质粒pgmer125(图4和5)。质粒pgmer125a和pgmer125b含有pdc启动子区域,tal终止子,eno1启动子区域,rki终止子,东方伊萨酵母ura3启动子,相应的ura3标记的5’末端以及东方伊萨酵母adh9091基因座的5’侧翼区域。右端片段的构建对空的右端构建体载体,pgrer121,进行如下所述多个步骤的克隆。用引物0611256和0611257从质粒pacn23(图20)中pcr扩增tdh3启动子片段。pcr反应体系(50μl)含有15ngpacn23质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611256和0611257每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约994bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约994bp,在该片段的5’末端有sfoi限制性位点,其3’末端有paci和nrui限制性位点。产生第二pcr片段以使用引物0611258和0611259从质粒pacn23(图20)中扩增tkl终止子,所述第二片段含有上述pcr产物的5’同源物,包括nrui和paci限制性位点。pcr反应体系(50μl)含有15ng质粒pacn23dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611258和0611259每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约469bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约469bp,在该片段的5’末端有nrui和paci限制性位点,3’末端有noti限制性位点。用引物0611256和0611259通过pcr将上述994bp和469bp的片段融合在一起,产生一条约1433bp的片段,该片段中tdh3启动子位于tkl终止子上游。pcr反应体系(50μl)含有125ng994bp片段,60ng469bp片段,1xphusionhf缓冲液(newenglandbiolabs),引物0611256和0611259每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1433bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。产生第二pcr片段以使用引物0611260和0611261扩增adh9091基因座的3’侧翼,所述第二片段含有上述1433bp的pcr产物3’末端的5’同源物,包括noti限制性位点。pcr反应体系(50μl)含有15ng质粒phjj27dna作为模板dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611260和0611261每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1019bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。所得到的pcr片段的总长度为约1019bp,在该片段的5’末端有noti限制性位点,3’末端有apai限制性位点。用引物0611256和0611261通过pcr将1019bp的片段融合至1433bp的tdh3启动子/tkl终止子片段下游,产生约2405bp的片段。pcr反应体系(50μl)含有125ng1433bp片段,90ng1019bp片段,1xphusionhf缓冲液(newenglandbiolabs),引物0611256和0611261每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃2分钟的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约2405bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。将所得到的2405bp的片段,其包含用于在位于tdh3启动子/tal终止子构建体下游adh9091基因座发生整合的3’侧翼,克隆进pcr2.1-topo载体中,并使用topotacloningkit(invitrogen)根据制造商说明转化进one-shottop10大肠杆菌细胞中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过avai消化来筛选到一些有所期望的pcr产物的正确插入的所得的转化体。产生所需大小条带的克隆被确认并命名为pgmer113。质粒pgmer113包含用于在东方伊萨酵母adh9091位点发生同源重组的3’侧翼,及其前面的空的表达盒tdh3启动子/tkl终止子。用引物0611264和0611284从质粒phjj27(图21)中扩增ura3orf的截短的3’片段,ura3终止子和ura3启动子(如上所述,当整合进酵母宿主后,用作重复区域以环出标记)。pcr反应体系(50μl)含有15ngphjj27质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611264和0611284每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs),以及1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1324bp的pcr产物被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)纯化。所得到的pcr片段的总长度为约1324bp,在其5’端有nhei限制性位点,3’端则有apai和sfoi限制性位点。将上述凝胶纯化的1324bp片段克隆进pcr2.1-topo载体中,使用topotacloningkit(invitrogen)根据制造商说明转化进one-shottop10大肠杆菌细胞中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过hindiii消化来筛选到一些有所期望的pcr产物的正确插入的所得的转化体。产生所需大小条带的克隆被确认并命名为pgmer109。质粒pgmer109包含ura3orf的3’片段和ura3终止子,其后是ura3启动子。质粒pgmer109中ura3基因3’片段的上游部分与克隆进质粒pgmer108的截短的5’ura3片段的末端具有460bp的同源性。该同源区域允许该基因的这两个部分发生重组,以在用含有这两个片段的构建体对宿主进行共转化时,能产生功能性的选择标志物。用kpni消化质粒pgmer109,然后根据制造商说明用酶dna聚合酶i,大(klenow)片段(newenglandbiolabs)进行处理。将得到的线性化质粒(含有平末端)用限制性酶bamhi消化。以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离产物,其中5247bp的载体片段被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。用bamhi和ecorv来消化质粒pgmer113,得到2466bp的片段,该片段具有构建体tdh3启动子/tkl终止子,其后为ura3orf的截短3’片段和ura3终止子,其后为ura3启动子。以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离双限制性酶切反应产物,其中约2466bp的载体片段被从胶上切下并根据制造商的说明使用gelextracttion试剂盒(qiagen)从琼脂糖中纯化。2644bpbamhi和ecorv消化片段随后被连接进来自质粒pgmer109的5247bp载体片段。连接反应设置为包含3μl5247bp的线性载体,3μl2466bp的插入片段,3μl无菌dd水,10μl2xquickligation反应缓冲液以及1μlquickt4dna连接酶(newenglandbiolabs),根据制造商说明来实施。根据制造商说明,将5μl连接反应产物转化到大肠杆菌ultracompetentcells(agilenttechnogogies)中。将转化体涂布于2xyt+amp平板上,37℃孵育过夜。通过xbai和paci消化来筛选到一些有所期望的pcr产物的正确插入的所得的的转化体。产生所需大小条带的克隆通过测序被确认并命名为pgmer121(图6)。质粒pgmer121含有东方伊萨酵母ura3标记的3’末端,其后为相应的ura3启动子,tdh3启动子,tkl终止子和adh9091位点的3’侧翼区域。实施例2g:东方伊萨酵母cnb1的构造如下所述,从东方伊萨酵母cd1822中构建东方伊萨酵母cnb1(对于从东方伊萨酵母atccpta-6658中生产东方伊萨酵母cd1822,见实施例1a)。菌株cd1822中含有的ura3基因的两个拷贝都被缺失以允许该基因能够成为遗传工程的选择标志物。ura3是酵母遗传学中的一种多用途标志物,这是由于该基因无论是存在(在尿嘧啶缺失的培养基上生长)还是缺失(在5-氟乳清酸存在的情况下生长)都可以用于选择。通过利用选择盒进行替换来破坏其中一个ura3基因,所述选择盒含有mel5选择标志物,其侧翼为重复dna序列。对检测为mel5选择盒阳性的菌株就mel5标记基因的缺失进行筛选。然后通过在5-氟乳清酸存在的情况下生长来选择第二个ura3基因缺失。用2.8μg经saci/pspomi消化的载体pmi458(图27)的dna转化cd1822。质粒pmi458含有酿酒酵母(s.cerevisiae)mel5基因(seqidno:255),该基因位于东方伊萨酵母pgk启动子的调控之下(p-lopgk,seqidno:247),其侧翼分别是与东方伊萨酵母ura3基因上游序列(p-loura3,seqidno:253)和下游序列(t-loura3,seqidno:254)同源的dna片段。p-loura3和t-loura3的相对取向与在东方伊萨酵母基因组中相同的。在30℃下,5天后获得了大约500个mel+菌落。通过接种至10mlbfp(butterfield磷酸缓冲液)管中,取出25μl涂布在dm1-x-α-gal平板上而单菌落分离了10个菌落。然后挑取从各个初步分离物中得到的单个蓝色菌落,置于ypd上以进行进一步分析。使用pcr来从转化体中筛选所期望的基因事件。为了获得在pcr筛选中作为模板使用的基因组dna,从1.5ml过夜培养物中将细胞旋转沉降至带螺旋帽的微量离心管中,重悬于0.2ml2%的tritonx-100,1%sds,100mmnacl,10mmtris-hcl,ph为8.0,1mm的乙二胺四乙酸二钠ph8.0的溶液中。添加用10mmtrisph8.0/1mmedta(sigma)平衡0.2ml苯酚:氯仿:异戊醇(25:24:1)混合物和0.3g玻璃珠。用mini-beadbeater-8(biospec)将管子全速振荡2分钟。加入0.2mlte,简短地旋涡振荡管子。通过在16100×g下离心5分钟来分离水相。将上清液移到新的管中,加入1ml100%的乙醇。将该管在20℃下放置30分钟,在16100×g下离心5分钟,将液体倒出。风干dna,重悬于500μlte中。利用引物oca405和oca406来对所期望的5’交叉进行pcr筛选,这将产生1.5kbp的产物。对于所期望的3’交叉的筛选则使用引物wg26和cm647,这产生1.6kbp的产物。用来构建pmi458的ura3区域外的引物(上游或下游远端)是oca405和cm647,其在野生型中产生3.2kbp的产物,而在pmi458被破坏的等位基因中产生5.0kbp的产物,当选择标志物被环出时产生2.2kbp的产物。这些pcr反应都使用55℃的退火温度。同样可以使用四引物方法通过pcr来筛选ura3开放阅读框的缺失。引物pjlj28和pjlj29扩增肌动蛋白基因的800bp片段,引物pjlj30和pjlj31扩增ura3基因的600bp片段。在一个反应中使用所有这些引物提供阳性内部对照(肌动蛋白片段)。按照生产商的操作步骤使用来自于roche的taqdna聚合酶,退火温度为61℃。确认mel5选择盒整合到ura3位点上的是菌株1822urahetmel-1和1822urahetmel-2。然后通过允许在ktseq1a(seqidno:256)和ktseq1b(seqidno:257)之间发生重组,将mel-5标志物从1822urahetmel-1和1822urahetmel-2的基因组中移除。mel+菌株在ypd培养基中生长过夜,直至od600约为0.5至2.0。然后在ypd培养基中稀释培养物,使其od600值回到约0.00001。将200μl培养物稀释液转移到96孔微量滴定板的每个孔中。用具有黏性的覆盖物覆盖平板,在30℃培养箱中孵育,以最大搅拌6-7小时(约6次细胞分裂,取决于菌株的生长速率)。从每个孔中取出100μl(约为~1000cfu每平板)涂布在dm1+x-α-gal培养基上。将平板孵育在30℃下过夜,或者在室温下2天,观察不同的颜色。将白色菌落(推定为mel-)在类似的培养基上划线,如上所述通过pcr进行筛选。发现了两个独立的环出,一个来自于1822urahetmel-1,保藏为1822urahetmel-1,另一个来自于1822urahetmel-2,保藏为1822urahetmel-2。奇怪的是,所获得的大部分白色菌落均没有给出预期的2.2kbp条带。为了得到ura-衍生体,在yp5d培养基上(yp+100g/l右旋糖)培养1822urahetmel-1和1822urahetmel-1过夜,过夜培养物的等分试样(0.5,5和50μl)涂布于scd-2xfoa平板上,对foa-抗性的菌落划线以得到单菌落,通过在scd-ura平板上涂布来证实ura-表型。从1822urahetmel-1中挑取2个菌落,从1822urahetmel-2中挑取6个菌落以进行进一步分析。这些菌落在ypd中生长过夜,使用上述的酚/氯仿法提取基因组dna。通过pcr筛选ura3开放阅读框的存在,8个菌株中均不含有ura3基因。1822urahetmel-1的两个ura后代命名为yjlj3(cnb1)和yjlj4。基于基因组测序,确定yjlj3(cnb1)和yjlj4含有ura3基因及附近透酶基因的缺失;优选只有ura3基因被缺失。为了构建cd1822的ura3营养缺陷型而不破坏该透酶基因,用1μgsaci/apai消化的pcm208(图28)转化cd1822。质粒pcm208含有与东方伊萨酵母ura3基因的上游(5’ura侧翼(附近),seqidno:258)和下游(3’ura侧翼(附近),seqidno:259)侧翼区域同源的dna序列。在30℃下培养5天后,获得大约200个mel+菌落。通过在scdx-α-gal平板上划线分离出8个蓝色菌落。使用pcr来就期望的基因事件筛选转化体。使用引物ojy11和ojy12来进行5’交叉筛选,这将在所期望的转化体中产生0.9kbp的产物。使用引物ojy13和ojy14来进行3’交叉筛选,这将在所期望的转化体中产生1.0kbp的产物。8个克隆中有3个显示了所期望的pcr产物。这些菌落的mel标记可被环出,并如上文所述,缺失第二ura3基因。或者,衍生自yjlj3或yjlj4的菌株中的ura3和透酶基因的缺失能够在一步转化中被修复,如实施例2h中所述。实施例2h:含有ura3选择标志物的mbin500对照菌株的构建如前所述,由于其ura3基因的纯合缺失,本文所使用的命名为cnb1的东方伊萨酵母菌株,是尿苷营养缺陷型。使用ura修复载体,pjlj62(图26),能够对ura3基因座进行杂合修复,该载体含有ura修复盒,其由ura3基因及691bp的5’侧翼dna,1500bp的3’侧翼dna构成。ura修复盒的侧翼是5’的noti限制性位点,3’的apai限制性位点。使用noti和apai进行限制性消化,以从载体骨架中移除2796bp的ura修复盒。使用qiaquickpcrpurificationkit(qiagen),根据制造商的说明对消化物进行纯化。将dna洗脱至玻璃蒸馏水中,用1μg来转化东方伊萨酵母cnb1。在ura选择平板上选择转化体,不需要补充尿苷的单菌落被命名为mbin500。实施例2i:移除ura3选择标志物为了分离其中ura3选择标志物通过存在于整合盒中的两个ura3启动子区域的重组而被移除的菌株,将目的ura+菌株接种至3mlyp+10%葡萄糖的培养基中,在37℃下,以250rpm振荡培养至少4小时至过夜。取出50-100μl培养物涂布在scdfoa平板上,在37℃下生长48-60h直到菌落出现。由于foa可被ura3蛋白转化成毒性化合物,导致ura+细胞的死亡,因此可在foa上生长来选择ura3标记的移除。通过在ypd平板上,37℃下生长来对几个foa-抗性菌落纯化两次。如本文所述,然后通过pcr对这些纯化的分离物进行ura3适当环出的筛选。实施例2j:对摇瓶中生长的修饰的酵母菌株进行十二烷基磺酸钠聚丙烯酰胺凝胶电泳(sds-page)分析和酶分析的步骤将4mlura选择培养基加入到14mlfalcon管中,用无菌环将所期望的菌株接种到该培养基中。在37℃下以250rpm振荡培养该培养物过夜(~16hrs)。对于含有至少一个东方伊萨酵母基因座野生型拷贝的菌株,将500μl过夜培养物加入到125ml含有25mlyp+10%葡萄糖培养基的挡板瓶中。对于pdcδ/pdcδ菌株,将1ml过夜培养物加入到125ml含有25ml液体yp+100g/l葡萄糖培养基的挡板瓶中。在37℃下以250rpm振荡培养该瓶培养物。大约每隔一小时从培养物中取出小量等分试液,测量od600。使培养物生长,直至od600值为4至6之间。为了制备用于sds-page分析的细胞的小样本,从培养物中取出对应2.5od单位的体积的培养物,并置于1.5ml管中。在16100×g下沉淀细胞,移除上清液,细胞沉淀储藏于-20℃直至使用。在室温下以2279×g离心来收获培养瓶中剩余的细胞,将沉淀重悬于12.5ml0.85mnacl中,然后在室温下以2279×g离心。将沉淀重悬于1ml0.85mnacl中,将重悬细胞转移到2.0ml管中,然后在16100×g下沉淀。然后移除上清液,如果能在一周之内就可以使用以进行酶测定,则将沉淀储藏于-20℃下,或者在-80℃下进行更长期储藏。对于对应2.5od单位的细胞沉淀的sds-page分析,将细胞重悬于100dh2o中,然后加入100μl0.2mnaoh。将样品在室温下孵育5分钟,然后16100×g离心沉淀细胞,重悬于100μlsds样品缓冲液中(bio-radlaboratories)。将样品在95℃下加热5分钟,简单地离心以沉淀细胞。根据制造商的说明,在criterion8-16%pre-cast凝胶(bio-radlaboratories)上对1至5μl的上清液进行分析。用instantbluetmcoomassie-basedstainingsolution(expedeonproteinsolutions,sandiego,ca,usa)显色条带。实施例2k:用于产品分析的修饰酵母菌株的摇瓶培养步骤在ura选择平板上对菌株进行划线以得到单菌落,在30℃下培养1-2天。将来自于ura选择平板上的1-2个菌落接种到含有50mlcnb1摇瓶培养基的250ml挡板瓶中,以制备种子培养物。种子培养物在30℃下,以200rpm振荡培养约18小时。然后取出培养物的小量等分试液来测定od600值,直至od600达到4至6。使用reagentstrip(bayer,elkhart,in,usa)来测量残余存在的葡萄糖。使用种子瓶培养来接种到含有50mlcnb1摇瓶培养基的125ml挡板瓶中,至od600=0.2。培养物在30℃下,140rpm下振荡孵育20小时。移除培养液的样品以进行如下所述的分析。使用样品的等分试样来测量培养物的光密度(od),并使用reagentstrip来测量残余的葡萄糖。然后将剩余的样品进行离心,使用上清液进行产品分析。实施例2l:用于产品分析的修饰酵母菌株发酵的步骤使用种子繁殖阶段来培养本文所描述的菌株,然后在2l的生物反应器(applikon,fostercity,ca,usa)中再进行单阶段发酵。对于种子阶段的制备,将25ml1xdm2培养基(用koh调整至所需的ph值)加入到125ml挡板瓶中,然后用无菌环接种目的菌株。在所需的温度下,250rpm振荡培养培养物过夜约16hr。然后每隔约一小时取出培养物的小量等分试液,测定od600值直至od600达到4-6。使用reagentstrip(bayer,elkhart,in,usa)来测量存在的残余的葡萄糖。然后将12ml培养物加入到4ml无菌冷冻的75%甘油中,彻底混匀,在冰上孵育10分钟。随后将培养物和甘油的混合物混匀,等分为10份,每份1.0ml放置于1.8ml冷冻管中(thermoscientific,rochester,ny,usa),储藏在-80℃下。使用25ml种子瓶培养接种到含有1.5ldm2培养基的2l生物反应器中。在约30℃-40℃的温度下,控制ph值在2.0-7.0的范围内,在搅拌条件下以及导致氧气吸收速率(our)在2-45mmol/l/hr范围内的通气条件下,在该生物反应器中进行发酵。在本文所示的实施例中,生物反应器中培养物的温度,ph和our分别是30℃,4.0和25-30。定期移除发酵液样品以进行分析。简单地说,使用样品的等分试液测量培养物的光密度(od),葡萄糖浓度和ph。然后将剩下的样品离心。沉淀物储藏于-80℃用于酶分析,上清液用于进行3-hp和其他胞外化合物的分析。除另有指明之外,本文中所报告的所有3-hp生产值均是发酵的48-小时时间点的生产值。在发酵过程中的二氧化碳生成量和氧气消耗是通过确定从生物反应器中排出的气体中的二氧化碳含量和氧气含量来进行测定的。实施例2m:对修饰酵母菌株所产生的3-hp和β-丙氨酸的分析步骤通过在1%甲酸中稀释10倍来酸化培养物样品,并且通过0.46μm的96孔滤板来进行过滤。根据样品中的分析物浓度在水中进行进一步稀释。在样品缓冲液中进一步稀释10倍,该缓冲液为1mmnh4ac,0.1%nh3和5mg/l的一律用13c标记的3-hp(作为3-hp的内标),或者1%甲酸和3mg/l一律用13c标记的β-丙氨酸(作为β-丙氨酸的内标)。总稀释倍数为约100至1000,取决于β-丙氨酸或3-hp的浓度。将2μl样品注入agilent1200hplc(agilent)中,由masshunter程序与agilent6410triplequadms/ms检测器控制,使用的仪器设置和栏目在表6中列出。定量离子碎片峰面积与其稳定同位素对应物(来自于内标)之间的比值可用于定量,以消除离子的抑制效应和仪器漂移。在日常检测中,标准偏差为低于5%。表6:用于β-丙氨酸和3-hp分析的lc/ms/ms设置实施例3:表达3-hp发酵途径基因的修饰酵母菌株在酵母宿主细胞中表达编码涉及3-hp不同发酵途径中的酶的一个或多个基因,其为单独表达或组合表达。3-hp途径酶可以从外源基因,内源基因或其一些组合来进行表达。利用基因表达构建体将要表达的外源基因引入酵母细胞中,例如,如实施例2中所描述的表达构建体。外源基因可在一个单一的位点或多个位点整合进宿主酵母的基因组中,并且外源基因的整合伴随着插入位点的目的基因的缺失或破坏,如下所述。实施例3a:表达天冬氨酸/丙二酸半醛途径基因的修饰酵母菌株通过利用pep和/或丙酮酸,oaa,天冬氨酸,β-丙氨酸,丙二酸半醛中间体的途径来产生3-hp的酵母细胞可以通过表达一个或多个参与该途径的酶来进行工程改造。表达的基因包括一个或多个ppc,pyc,aat,adc,baat,gabt,3-hpdh(包括丙二酸半醛还原酶),hibadh,或4-羟基丁酸酯脱氢酶基因。所表达的基因可以衍生自宿主细胞的天然基因。例如,当宿主酵母细胞是东方伊萨酵母时,所表达的基因可以衍生自东方伊萨酵母pyc(例如,编码如seqidno:2所示的氨基酸序列的和/或包括如seqidno:1所示核苷酸序列编码区的东方伊萨酵母pyc基因),aat(例如,编码如seqidno:14所示的氨基酸序列的和/或包括如seqidno:13所示核苷酸序列编码区的东方伊萨酵母aat基因),baat(例如,编码如seqidno:20所示的氨基酸序列的和/或包括如seqidno:19所示核苷酸序列编码区的东方伊萨酵母pyd4同源基因),或3-hpdh(例如,编码如seqidno:26所示的氨基酸序列的和/或包括如seqidno:25所示核苷酸序列编码区的与东方伊萨酵母ymr226c同源的基因)。当酵母宿主细胞是另外的3-hp耐受酵母菌株时,可以使用本领域已知技术来获得基因序列,途径基因的天然同源物能够被外源表达或者与外源调节元件连接来进行表达。天然途径基因可以包括一个或多个ppc,pyc,aat,baat和/或3-hpdh基因。或者,一个或多个表达的3-hp基因可来自于源基因,该源基因对于宿主细胞而言是非天然的。例如,当酵母宿主细胞是东方伊萨酵母时,可以对该细胞进行工程改造以表达一个或多个非天然的pyc基因,如编码如seqidno:3所示氨基酸序列的类球红细菌(r.sphaeroides)pyc基因,编码如seqidno:4所示氨基酸序列的菜豆根瘤菌(r.etli)pyc基因,编码如seqidno:5或6所示氨基酸序列的荧光假单胞菌(p.fluorescens)pyc基因,编码如seqidno:7所示氨基酸序列的谷氨酸棒杆菌(c.glutamicum)pyc基因,或编码如seqidno:8所示氨基酸序列的苜蓿中华根瘤菌(s.melioti)pyc基因;一个或多个非天然ppc基因,例如编码如seqidno:10所示氨基酸序列的大肠杆菌(e.coli)ppc基因,编码如seqidno:11所示氨基酸序列的嗜热碱甲烷杆菌(m.thermoautotrophicum)ppc基因,编码如seqidno:12所示氨基酸序列的产气荚膜梭菌(c.perfringens)ppc基因;一个或多个非天然aat基因,如编码如seqidno:16所示氨基酸序列的大肠杆菌aspc基因,或编码如seqidno:15所示氨基酸序列的酿酒酵母(s.cerevisiae)aat2基因;一个或多个非天然adc基因,如编码如seqidno:17所示氨基酸序列的(和/或包含如seqidno:130,145,146或147所示核苷酸序列的编码区的)阿维链霉菌(s.avermitilis)pand基因,编码如seqidno:18所示氨基酸序列的(和/或包含如seqidno:131所示核苷酸序列的编码区的)丙酮丁醇梭菌(c.acetobutylicum)pand基因,编码如seqidno:133所示氨基酸序列的(和/或包含如seqidno:132所示核苷酸序列的编码区的)幽门螺旋杆菌(h.pylori)adc基因,编码如seqidno:135所示氨基酸序列的(和/或包含如seqidno:134所示核苷酸序列的编码区的)芽孢杆菌属(bacillussp)ts25adc基因,编码如seqidno:137所示氨基酸序列的(和/或包含如seqidno:136所示核苷酸序列的编码区的)谷氨酸棒杆菌(c.glutamicum)adc基因,编码如seqidno:139所示氨基酸序列的(和/或包含如seqidno:138,148,149,150或151所示核苷酸序列的编码区的)地衣芽孢杆菌(b.licheniformis)adc基因;一个或多个非天然baat或gabt基因,如编码如seqidno:21所示氨基酸序列的(和/或包含如seqidno:142所示核苷酸序列的编码区的)克鲁维酵母pyd4基因,编码如seqidno:22所示氨基酸序列的(和/或包含如seqidno:140所示核苷酸序列的编码区的)阿维链霉菌baat基因,编码如seqidno:23所示氨基酸序列的阿维链霉菌gabt基因,或编码如seqidno:24所示氨基酸序列的(和/或包含如seqidno:141所示核苷酸序列的编码区的)酿酒酵母uga1基因;一个或多个非天然3-hpdh基因,如编码如seqidno:27所示氨基酸序列的(和/或包含如seqidno:143所示核苷酸序列的编码区的)大肠杆菌ydfg基因,或编码如seqidno:129所示氨基酸序列的(和/或包含如seqidno:144所示核苷酸序列的编码区的)酿酒酵母ymr226c基因;一个或多个非天然的丙二酸半醛还原酶基因,如编码如seqidno:29所示氨基酸序列的(和/或包含如seqidno:343所示核苷酸序列的编码区的)嗜热金属球菌丙二酸半醛还原酶基因;一个或多个非天然hibadh基因,如编码如seqidno:28所示氨基酸序列的粪产碱菌(a.faecalis)m3a基因,编码分别如seqidno:30或seqidno:31所示氨基酸序列的恶臭假单胞菌(p.putida)kt2440或e23440mmsb基因,或者编码如seqidno:32所示氨基酸序列的铜绿假单胞菌(p.aeruginosa)pao1mmsb基因;和/或一个或多个非天然4-羟基丁酸脱氢酶基因,如编码如seqidno:33所示氨基酸序列的r.eutropha(r.eutropha)h164hbd基因,或编码如seqidno:34所示氨基酸序列c.kluyverdsm555hbd基因。实施例3a-0:表达天冬氨酸/丙二酸半醛途径基因的修饰酵母菌株的酶活测定制备用于酶测定的粗制无细胞提取物(cfe)通过离心来收集本发明所示的来自于摇瓶或生物反应器培养物中的细胞,弃去上清液,如上所述,将细胞沉淀储藏于-80℃下。为了制备cfe,将细胞沉淀物解冻,用磷酸盐缓冲液(pbs)洗涤,再一次离心收集。弃去上清液,在2.0ml微量离心管中,将细胞沉淀重悬于大约等体积的裂解缓冲液中,该裂解缓冲液含有1%的蛋白酶抑制剂cocktail,来自于sigma的p8215。加入约300μl0.5mm的氧化锆珠(biospec),在裂解器(mpbiomedicals)中以6/20秒的设定进行3轮细胞裂解。在每轮之间,将样品管置于冰上冷却5分钟。裂解之后,在微量离心管中,4℃下,以最大速度对样品离心15分钟。将上清液转移到新的1.5ml管中,并保持在冰上。根据制造商提供的说明,使用bio-rad蛋白质测定试剂(bradford测定)对裂解液中的总蛋白浓度进行确定,用牛血清蛋白作为标准品。丙酮酸羧化酶活性(pyc)活性:本文所示细胞的cfe中的丙酮酸羧化酶的活性测定如下。制备储备反应混合溶液,当在测定反应混合物中与cfe组合时,该混合溶液提供的组分的最终浓度如下:tris(ph8.0),100mm;nahco3,10mm;mgcl2,5mm;nadh,0.2mm;atp,1mm;乙酰coa,1mm;丙酮酸,1mm;生物素(如果测定pyc酶时需要),5μm;牛心苹果酸脱氢酶,0.02单位/ml。将270μl该混合物加入到96孔微量滴定板的孔中,加入30μl经过适当稀释的cfe以开始反应。使用spectramax340pc酶标仪监测340nm处的nadh消耗量。丙酮酸羧化酶的活性表示为nmolnadh消耗/秒/mg蛋白质。磷酸烯醇丙酮酸羧化酶(ppc)活性:cfe中的磷酸烯醇丙酮酸羧化酶(ppc)的活性测定如下。制备储备反应混合溶液,当在测定反应混合物中与cfe组合时,该混合溶液提供的组分的最终浓度如下:tris(ph8.0),100mm;nahco3,10mm;mgcl2,5mm;nadh,0.1mm;乙酰coa,0.5mm;磷酸烯醇丙酮酸,3.3mm;牛心(或猪心)苹果酸脱氢酶,0.02单位/ml。将270μl该混合物加入到96孔微量滴定板的孔中,加入30μl经过适当稀释的cfe以开始反应。使用spectramax340pc酶标仪监测340nm处的nadh消耗量。天冬氨酸转氨酶(aat)活性:本文所示细胞的cfe中的天冬氨酸转氨酶的活性测定如下。制备储备反应混合溶液,当在测定反应混合物中与cfe组合时,该混合溶液提供的组分的最终浓度如下:100mmtris(ph8.0),100mm;nahco3,10mm;mgcl2,5mm;nadh,0.1mm;天冬氨酸,1mm;α-酮戊二酸,1mm;苹果酸脱氢酶,0.02单位/ml。在一些测定中,储备反应混合物还含有吡哆醛5’磷酸(0.1mm)。将270μl该混合物加入到96孔微量滴定板的孔中,加入30μl经过适当稀释的cfe以开始反应。使用spectramax340pc酶标仪监测340nm处的nadh消耗量。天冬氨酸转氨酶的活性表示为nmolesnadh消耗/秒/mg蛋白质。天冬氨酸脱羧酶(adc)活性:本文所示细胞的cfe中的天冬氨酸脱羧酶的活性测定如下。在恒温37℃下,将165μl100mmnh4ac缓冲液(ph6.8)和25μl80mm的天冬氨酸加入到96孔微量滴定板的每个孔中。加入10μlcfe以起始反应。在不同的时间间隔(5,10,15,20,25,30,40,60分钟),从反应混合物中取出20μl样品,加入到180μl淬灭缓冲液中(2%的甲酸以及2mg/l的13c标记β-丙氨酸作为内标)。过滤之后,用lc/ms/ms分析样品中的β-丙氨酸。从β-丙氨酸相对于时间的曲线图中获取斜率。通过斜率除以反应中总细胞蛋白浓度来计算活性。adc的活性表示为μmolβ-丙氨酸形成/秒/g蛋白质。在一些具体实验中则使用修改的adc测定法。在这些情况下,本文所示细胞的cfe中的天冬氨酸转氨酶的活性测定如下。将110μl100mmnh4ac缓冲液(ph7.6)和80μl25mm的天冬氨酸(用naoh中和之后)加入到恒温40℃的96孔微量滴定板的每个孔中。加入10μlcfe以起始反应。在不同的时间间隔(2,4,6,8,10分钟),从反应混合物中取出20μl样品,加入到180μl淬灭缓冲液中(2%的甲酸以及2mg/l的13c标记β-丙氨酸作为内标或者在2%的甲酸中淬灭,然后按比例1:10转移至含有2mg/l的13c标记β-丙氨酸作为内标的20%甲醇/80%水中)。过滤之后,用lc/ms/ms分析样品中的β-丙氨酸。从β-丙氨酸相对于时间的曲线图中获取斜率。通过斜率除以反应中总细胞蛋白浓度来计算活性。adc的活性表示为μmolβ-丙氨酸形成/秒/g蛋白质。β-丙氨酸转氨酶(baat)活性cfe中的β-丙氨酸转氨酶(baat)的活性测定如下。在室温下,将190μl含有100mmnh4hco3(ph7.6),8mmα-酮戊二酸,0.5mm乙酰coa,0.1mm吡哆醛-5'-磷酸和200mmβ-丙氨酸的反应混合物加入到96孔微量滴定板中。加入10μlcfe以起始反应。在2,4,6,8,10,12,15和20分钟时,从反应混合物中取出20μl样品,并加入到75μl淬灭缓冲液中(2.5%的甲酸)。中和样品,通过加入5μl10m的naoh和50μl100mm的naco3(ph10)来控制ph值。在注射时将过滤后的样品与opa(临苯二甲醛)试剂,10mg/ml(agilenttechnologies5061-3335)混合,以衍生化样品。在hplc分离之后,通过荧光检测(激发波长340nm;发射波长460nm)对用opa衍生化的谷氨酸进行定量。将15μl样品注射到5μm包装的反相geminic18分析柱中(phenomonex150×4.6mm)。在62.5%20mm的磷酸缓冲液(ph7.8)(a)和37.5%甲醇(b)中平衡该柱。线性梯度如下所示:坡增(ramp)至40%b,0-0.3分钟;40%b,0.3-1分钟;坡增至85%b,1-1.75分钟;85%b,1.75-2.25分钟;坡增至37.5%,2.25-3分钟;37.5%b,3-4分钟。流速为2ml/分钟。反应缓冲液中谷氨酸的标准曲线用于确定样品中的浓度。从[谷氨酸]相对于时间的曲线图中获取斜率。通过斜率除以反应中总细胞蛋白浓度来计算活性。3-hp脱氢酶(3-hpdh)活性本文所示细胞的cfe中的3-hp脱氢酶的活性测定如下。将190μl稀释于(通常是100倍稀释)100mmnh4hco3(ph7.6)中的cfe和nadph加入到恒温37℃的96孔微量滴定板的每个孔中。通过加入10μl60mm的丙二酸半醛(从在10mmh2so4中的200mmmsa储备溶液在10mmh2so4中制备)来起始反应。在1,2,4,6,8,10和12分钟时,从反应混合物中取出每份20μl样品,在80μl沸水中淬火。冷却之后,用75μl含有2mmnh4ac(ph6.8)和3mg/l13c标记的3-hp的缓冲液与75μl淬火混合物进行混合。过滤之后,用lc/ms/ms定量样品中的3-hp。从3-hp相对于时间的曲线图中获取斜率。通过斜率除以反应中总细胞蛋白浓度来计算活性。3-hp脱氢酶的活性表示为nmolnadph形成/秒/mg蛋白质。在一些具体实施方式中使用修改的3-hpdh测定法。在这些情况下,本文所示细胞的cfe中的3-hpdh的活性测定如下。通过测量在340nm处,nadph随着时间推移而消失来测量丙二酸半醛的还原。根据yamada和jzcoby所发现的方法自己合成丙二酸半醛(yamada,e.w.,jacoby,w.b.,1960,directconversionofmalonicsemialdehydetoacetyl-coenzymea,journalofbiologicalchemistry,volume235,number3,pp.589-594)。该测定在96孔板中实施,最终体积为200μl。将30μlcce加入到170μl的测定缓冲液(2mm的丙二酸半醛,100nmtrisph8.0和0.5mm的nadph)中来起始反应。在室温下(~25℃),在微板读数仪(spectramax340pc,moleculardevicesllc,sunnyvale,ca)上追踪340nm处的吸光度10分钟。1个单位的3-hpdh活性被定义为在丙二酸半醛存在的情况下,1分钟内氧化1μmol的nadph所需的酶量。实施例3a-1:在adh1202基因座表达天冬氨酸脱羧酶(adc)的插入载体为了在东方伊萨酵母中表达,对一些天冬氨酸脱羧酶基因进行了密码子优化,并通过(burlingame,ca,usa)合成,得到了表7中所列出的质粒。通过xbai和paci限制性消化,可使合成基因到达载体pma-t中,也能使其从载体中引出(elicit)。然后将限制性片段克隆进pmiba107的相同位置中,将基因置于pdc启动子和终止子的控制之下,并允许其在东方伊萨酵母的adh1202基因座发生整合。表7:转化体构建体质粒1045172,105387,105388,105389,105390,105391用xbai和paci进行消化,使用tbe缓冲液进行1.3%琼脂糖凝胶电泳。每个消化反应中,对应于adc(pand)基因的400-500bp片段被从凝胶中切割下来,并使用gelextractkit(qiagen)从琼脂糖中提取。用xbai和paci消化质粒pmiba107,用小牛肠磷酸酶(newenglandbiolabs)处理,在tbe缓冲液中进行琼脂糖凝胶电泳后,纯化载体条带。将xbai和paci消化后的pand片段用t4dna连接酶和quickligationkit(newenglandbiolabs)根据制造商的说明,连接到这个纯化的pmiba107载体上。根据制造商的说明,将连接产物转化进xl10-goldultra细胞中(agilenttechnologies)。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。每个反应挑取24个转化体至2xyt+amp平板上。通过apai,ncoi和saci消化筛选出所得到的四个转换体每个的小量制备dna。通过dna测序来验证产生所需条带大小的克隆的正确性,并如表7所示被命名。所得到的质粒允许以表达盒正向取向的方式使每个adc基因整合到adh1202基因座上。大约10μg的每种整合构建体用apai和kpni消化,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。每个质粒大约4450bp的片段从凝胶中切割下来,根据制造商的说明使用gelextractkit(qiagen)提取。发现纯化产物的浓度在39-138ng/ul之间。用0.39-1.4μg来自于整合构建体(用apai和kpni消化)的片段转化进东方伊萨酵母cnb1中,如上所述。将转化体涂布在ura选择培养基上,37℃下孵育,在ura选择培养基上重新划线,37℃下孵育。从ura3+菌落中制备基因组dna,通过pcr检测以验证整合。使用引物0611718和0611632扩增2.5kbp片段来验证整合。每个pcr反应含有2.5μl基因组dna,引物0611718和0611632各12.5pm,1xcrimsontaqtm反应缓冲液(newenglandbiolabs),0.2mmdntp混合物,0.625单位crimsontaqtmdna聚合酶(newenglandbiolabs),最终体积为25μl。扩增反应孵育在(eppendorfscientific)中,程序为1个循环95℃30秒;30个循环的95℃30秒,50℃30秒和68℃3分钟;以及1个循环的68℃10分钟。对于每个构建体,两个证实为ura3的转化体被命名如表7所示。这些菌株在adh1202对于所示的adc基因是杂合的,所述基因由来自于东方伊萨酵母的pdc启动子和终止子驱动表达。检测表7中所列出的菌株中(以及mbin500菌株,如上,作为阴性对照)pand的表达和酶活性。每个菌株的过夜培养物在ypdon中,37℃下过夜培养,在37℃下,在125ml的挡板瓶中,以1:50稀释到25ml新鲜ypd中,生长至od600~2-8。然后使用细胞沉淀物来制备cfe,然后如上所述使用该cfe进行adc活性测定。代表性的结果示于表8中。表8:转化体酶活性数据接下来,创建ywty5-17和ywty7-25的纯合子版本。首先,如上所述分离ura-衍生的ywty5-17和ywty7-25。从foa-抗性菌落中制备基因组dna,如上所述用pcr进行检查以验证ura3选择标志物的缺失。使用引物0611718和0611632,对于ura标志存在的整合,扩增出2.4kbp的片段,对于ura标志的缺失,则扩增出1100bp的片段。使用引物0611718和0611632,产生1100bppcr片段的ura-菌株ywty5-17和ywty7-25被分别命名为miba331和miba332。pwty10-0033-5和pwty10-0033-7各10μg用apai,kpni和ncoi进行消化,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。每个质粒大约4450bp的片段从凝胶中切割下来,使用gelextractkit(qiagen)提取。将来自pwty10-0033-5和pwty10-0033-7纯化片段分别转化进miba331和miba332中,如上所述。将转化体涂布在ura选择培养基上,37℃下孵育过夜,然后在ura选择培养基上重新划线,37℃下孵育过夜。制备基因组dna,实施crimsontaqtmpcr来验证整合,如上所述。使用引物0611718和0611632,对于ura标志存在的整合,扩增出2.4kbp的片段,对于ura标志的缺失,则扩增出1100bp的片段。使用引物0611718和0611632,产生1100bp和2.4kbppcr片段的miba331和miba332转化体被保存并分别命名为miba338和miba337。miba337在adh1202基因座是来自于阿维链霉菌的adc基因纯合子,miba338则在adh1202基因座是来自于地衣芽孢杆菌的adc基因纯合子。两个菌株都具有各自的adc位于来自于东方伊萨酵母的pdc启动子和终止子的调控之下。将来自于pand纯合菌株miba337和miba338与来自于pand杂合菌株ywty5-17和ywty7-25的adc表达和酶活进行比较。培养物在ypd中以37℃下培养过夜,然后在37℃下,在125ml的挡板瓶中,以1:50稀释到25ml新鲜ypd中,生长至od600~2-8。使用细胞沉淀物来制备cfe,然后如上所述使用该cfe进行adc活性测定。两个独立实验的代表性的结果示于表9a中。表9a:转化体酶活数据第三个独立实验,即从菌株miba337和miba338制备的cfe中的adc活性比较的结果示于表9b中。表9b:转化体酶活数据上述样品的sds-page分析表明miba338中的pand表达量在这些菌株中是最高的。使用本发明所描述的方法,在生物反应器中评估菌株mbin500,miba337和miba338的3-hp产生。对照菌株mbin500没有产生可检测的3-hp(两个独立发酵的平均值)。菌株miba337产生了1.33g/l3-hp(实施了一个发酵),菌株miba338产生了3.15g/l3-hp(三个独立发酵的平均值)。在菌株miba337和miba338的各个独立发酵中,进一步比较它们的3-hp生产性能和adc活性(表10)。考虑到这些发酵中细胞量的不同,3-hp生产性能在表10中被记录为3-hp浓度/每单位的细胞量(表示为[g/l3-hp]/[g/l细胞干重])。该结果显示,使用地衣芽孢杆菌pand基因(菌株miba338)要比使用阿维链霉菌的pand基因(菌株miba337)具有改进的adc活性和3-hp生产性能。表10:菌株miba337和miba338的3-hp生产性能和adc活性实施例3a-2:在adh1202基因座表达β-丙氨酸转氨酶(baat)或3-羟基丙酸脱氢酶(3-hpdh)的插入载体来自于阿维链霉菌的baat,来自于酿酒酵母的uga1,来自于克鲁维酵母的pyd4,来自于酿酒酵母的ymr226c以及来自于大肠杆菌的ydfg,这些基因的东方伊萨酵母密码子优化型式由合成,所得到的质粒在下面列出。这些合成基因通过使用xbai和paci消化能够进入到载体pma-t中,也可以从载体中引出。然后将经消化的片段克隆进pmiba107的相同位置中,将基因置于pdc启动子和终止子的调控之下,并允许其在adh1202基因座发生整合。表11:转化体构建体质粒1054168,1054169,1054170,1054171,1054172和1054173用xbai和paci进行消化,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。将来自于1045168的761bp片段(ydfg),来自于1045169的1430bp片段(uga1),来自于1045170的1370bp片段(baat),来自于1045171的1442bp片段(pyd4)或来自于1045173的814bp片段(ymr226c)从凝胶中切割下来,使用gelextractkit(qiagen)从琼脂糖中提取。质粒pmiba107用xbai和paci消化,接着用cip进行处理,产生7.9kbp的线性片段。消化物用pcrpurificationkit(qiagen)进行纯化。如本文所述,然后利用t4dna连接酶将消化后的ydfg,uga1,baat,pyd4或ymr226c片段连接到pmiba107上(用xbai和paci消化,并用cip处理)。根据制造商的说明将连接产物转化至onetop10chemicallycompetent大肠杆菌细胞(invitrogen)中。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。通过xbai和paci消化筛选到所得转化体中。产生所需片段大小的克隆通过dna测序来确认正确,并分别对于ydfg,baat,uga1,ymr226c或pyd4命名为pmiba120,pmiba121,pmiba122,pmiba123和pmiba124。所得到的质粒允许以表达盒正向取向的方式使所需基因整合到adh1202基因组上。表11中的整合构建体用于将为在东方伊萨酵母中表达而密码子优化的目的基因整合到东方伊萨酵母的adh1202基因座上,在pdc启动子和终止子的调控之下。如本文所述,表达盒还包括ura3选择标志物以允许对ura-宿主中的转化体进行选择。表达盒和adh1202同源区域的侧翼是apai和kpni限制性位点,以允许片段从质粒中释放出来。表11中的各个整合构建体各15μg用apai,kpni和ncoi消化,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。用ncoi消化破坏了载体的骨架,使得更容易从琼脂糖凝胶中提取目的片段。将分别来自于pmiba120,pmiba121,pmiba122,pmiba123和pmiba124的4884bp,5493bp,5553bp,4937bp和5565bp的片段,从凝胶中切割下来,使用gelextractkit(qiagen)从琼脂糖中提取。发现纯化后产物的浓度在80-120ng/μl之间。如本文所述,将0.8-1.2μg来自于pmiba120-4的限制性片段转化进东方伊萨酵母cnb1(ura-)中。然后将转化体涂布在ura选择培养基上,室温下生长60小时。将转化体在ura选择培养基上重新划线,在37℃下孵育过夜。通过菌落pcr对几个转化体各进行检查以验证整合。使用引物对来验证正确的整合,所述引物对检查整合的5’和3’端,并列在下面。引物0611717以反向退火于pdc启动子中,而引物0611225则以正向退火于ura3选择标志物上。引物0611631和0611632分别以正向和反向在整合位点的外面进行退火;引物0611717和0611631在正确的整合体中扩增出976bp的片段;引物0611225和0611632在正确的整合体中扩增出1.4kbp的片段;引物0611631和0611632扩增出2.7kbp的片段则意味着野生型染色体,而如果整合,则扩增出~5kbp的片段。为了构建基因组dna,将每个转化体的一个菌落在50μl的含0,05u/μl溶细胞酶(sigma,st.louis,mo,usa)的te中,在37℃下孵育30分钟,接着在95℃下孵育10分钟。如本文所述实施pcr以验证整合。使用0611717和0611631产生976bppcr片段,使用0611225和0611632产生1.4kbp片段,和使用0611631和0611632产生2.7kbp片段的每个杂合整合体的一个转化体被保存,并命名为miba308,miba309,miba310,miba311和miba312,如表11中所示。转化体miba308,miba309,miba310,miba311和miba312的培养物在ypd中,37℃下生长过夜。在125ml挡板瓶中,在37℃以1:50倍将培养物稀释到25ml新鲜ypd中,生长至od600为~4-10。使用本文所述的方法,通过sds-page来分析细胞样品的蛋白表达。从培养物的细胞沉淀中制备cfe,使用本文所述的方法测量cfe中3hp脱氢酶的活性。通过sds-page是否存在~53kda的条带来检测miba310和miba312中是否分别有uga1和pyd4的表达,所述条带在没有整合任何基因的菌株中是不存在的。而在这些条件下,sds-page并不能检测到miba309中baat的表达。表12a显示了菌株的cfe中3-hp脱氢酶(3-hpdh)的活性。表12a:转化体酶活数据在采用改进的测定条件的独立实验中,比较从菌株mbin500(对照),miba310,miba309和miba312中制备的cfe中baat的活性。该实验的结果示于表12b中。表12b:转化体酶活数据菌株过表达基因基因来源基因seqidnobaat活性mbin500n/an/an/a0.67miba310gabt(uga1)酿酒酵母1419.05miba309baat阿维链霉菌1400.42miba312baat(pyd4)克鲁维酵母142105.85质粒pmiba120-4(如上)含有noti限制性位点,位于表达盒的侧翼,如下所示:pdc启动子,目的基因(baat或3-hpdh),pdc终止子,以及ura3选择标志物。用于在adh1202基因座整合的同源区在noti限制性位点之外。这些质粒全都具有正向取向的表达盒。构建具有反向取向表达盒的新质粒,以允许方便地筛选纯合整合菌株。用noti消化质粒pmiba120-4,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。3.5kbp片段(pmiba120),4.2kbp片段(pmiba121),4.2kbp片段(pmiba122),3.5kbp片段(pmiba123)和4.2kbp片段(pmiba124)从凝胶中切割下来,使用gelextractkit(qiagen)从琼脂糖中提取。使用t4dna连接酶如上所述,将这些片段分别连接到5.2kbp线性化的用noti/cip处理过的phjj76-无ura上。根据制造商的说明将连接产物转化至onetop10chemicallycompetent大肠杆菌细胞(invitrogen)中。将转化体涂布在2xyt+amp平板上,37℃孵育过夜。通过xbai和kpni消化来筛选数个所得转化体。产生所需片段大小的克隆分别对于uga1,ymr226c,pyd4,ydfg和baat命名为pmiba131,pmiba132,pmiba133,pmiba134和pmiba135。所得到的质粒允许以表达盒反向取向的方式使所需基因整合到adh1202基因座上。如本文所述,分离miba308-12的ura-衍生体。数个miba308-12的foa抗性菌落是通过37℃下生长在ypd平板上以纯化两次的菌落。从foa-抗性菌落中制备基因组dna,通过pcr进行检查仪验证ura3选择标志物的缺失,如本文所述。引物0611631和0611632在整合位点的外部,分别以正向和反向进行退火。引物0611718退火于ura选择标志物上游的pdc终止子上;引物0611632和0611631在野生型染色体上扩增出2.7kbp的片段;引物0611718和0611632对于ura标记存在的整合则扩增出2.4kbp的片段,如果不存在ura标记则扩增出1100bp片段。使用引物0611718和0611632产生1100bppcr片段,使用0611631和0611632产生2.7kbp片段的miba308-12的其中一个ura-菌株被保存,命名为miba314(miba310的ura-菌株),miba315(miba312的ura-菌株),miba316(miba311的ura-菌株),miba326(miba308的ura-菌株)和miba328(miba309的ura-菌株)。pmiba131,pmiba132,pmiba133和pmiba135各10-15μg,用apai,kpni和ncoi消化,使用89mmtirs碱-89mm硼酸-2mm乙二胺四乙酸钠(tbe)缓冲液,在1%琼脂糖凝胶上进行电泳。用ncoi消化后,将目的片段从琼脂糖凝胶中提取变得更方便。将分别来自于pmiba131-3,pmiba135的5553bp,4937bp,5565bp,5493bp片段从凝胶中切割下来,使用gelextractkit(qiagen)从琼脂糖中提取。发现纯化后产物的浓度在67-80ng/μl之间。将0.67-0.8μg来自于pmiba131-3和pmiba135的限制性片段转化进miba314,miba316,miba315或miba328中,如本文所述。将转化体涂布在ura选择培养基上,在37℃下孵育过夜。然后将转化体在ura选择培养基上重新划线,在37℃下孵育过夜。从ura3+菌落中制备基因组dna,如上所述使用pcr进行检查以验证第二表达盒的整合,使得菌株对于目的基因是纯合的。如上所述,引物0611718和0611632对于第一个整合扩增出1100bp片段,而引物0611632和0611717则对于反向取向的第二整合扩增出814bp的片段。使用引物0611718和0611632扩增出1100bp,以及使用引物0611717和0611632扩增出814bp的每个品系的ura3+转化体被命名为miba317,miba318,miba319和miba329(见表13)。表13:转化体基因型确定ymr226c,uga1,pyd4和baat纯合或杂合菌株中的表达和酶活。miba309-12,miba317-9和miba329的过夜培养物在ypdon中,37℃下生长,然后在37℃在125ml挡板瓶中,以1:50倍将培养物稀释到25ml新鲜ypd中,生长至od600为~4-10。使用本文所述的方法,通过sds-page来分析细胞样品的蛋白表达。也从培养物的细胞沉淀中制备cfe,使用本文所述的方法测量cfe中3hp脱氢酶的活性。基于sds-page的结果,miba310,miba318,miba312和miba319含有分子量为~53kda的蛋白(由uga1或pyd4基因编码的蛋白质的期望大小)。在对菌株mbin500进行sds-page分析时并没有观察到对应于该蛋白质的条带。另外,纯合菌株miba318和miba319中uga1和pyd4的表达分别比相应的杂合菌株miba310或miba312中的表达要高(通过sds-page分析来判断)。在这些条件下在菌株miba309或miba329中没有检测(通过sds-page)到baat的表达。表14a显示了菌株mbin500,miba311和miba317的cfe中,3-hp脱氢酶(3-hpdh)的活性。表14a:转化体酶活性数据在使用改进的测定条件的独立实验中,比较从菌株mbin500(对照),miba319和miba329中制备的cfe中baat的活性。该实验的结果示于表14b中。表14b:转化体酶活数据菌株过表达基因基因seqidno来源baat活性mbin500(对照)n/an/an/a0.67miba319baat(pyd4)142克鲁维酵母228.01miba329baat140阿维链霉菌0.38如本文所述,分离菌株miba317,miba318和miba319的ura-衍生体。数个miba317,miba318和miba319的foa抗性菌落是通过37℃下生长在ypd平板上以纯化菌落。从foa-抗性菌落中制备基因组dna,通过pcr进行检查以验证ura3选择标志物的缺失,如本文中所述。引物0611718和0611632扩增出1100bp片段表明如上述的第一整合,引物0611632和0611717扩增出814bp片段表明在反向取向存在第二整合。引物0611718和0611631扩增出2.6kbp片段表明第二整合具有ura标志,而1200bp片段表明没有ura标志。使用0611718和0611632产生1100bppcr片段,使用0611632和0611717产生814bp片段,或者使用0611718和0611631产生1200bpmiba307和miba308ura-菌株被保存,分别命名为miba320和miba321。当ura标志从miba319中移除时,可能发生基因转换事件,从而产生miba322,如pcr所示(用引物0611632和0611631不产生2.7kbp片段,或者用引物0611632和0611717不产生814bp片段,但是用0611718和0611632则扩增出1100bp片段),以至于两个表达盒都是正向取向的。实施例3a-3:在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh)的插入载体的左端片段的构建含有阿维链霉菌adc(seqidno:130)和阿维链霉菌baat(seqidno:140)的左端片段为了允许在eno1启动子和pdc终止子区域之间插入供表达的基因,用xbai和paci消化pmhct068载体,用10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约6.1kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。然后,用引物0611196和0611186扩增克鲁维酵母baat(pyd4)基因(seqidno:142),所述引物包括后续亚克隆步骤所需的限制性位点。pcr反应体系(50μl)包含1μl1比50稀释的小量制备含有克鲁维酵母pyd4基因的质粒,1xpfx扩增缓冲液(invitrogen),引物0611196和0611186每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso和2.5个单位的pfxdna聚合酶(invitrogen)。在(eppendorfscientific)中进行pcr,程序为95℃2分钟的1个循环,随后是每个为95℃30秒,40.8℃30秒和72℃1分30秒的34个循环,及72℃5分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1428bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。上面所产生的pyd4pcr产物用xbai和paci消化,在tae缓冲液中通过0.9%琼脂糖凝胶电泳纯化,约1.4kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。在连接反应中,将该纯化的dna克隆进上述被xbai和paci限制性消化过的pmhct068载体中,所述连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),100ngxbai/pacipmhct068载体,70.5ngxbai/pacipyd4插入物,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后在冰上冷却。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过xbai和paci消化筛选到数个有所需pcr产物的合适插入的所得转化体,其中确认的一个分离物被命名为“左+pyd4#1”。用引物0611376和0611377(注意当通过in-fusion插入到pmhct068时,引物0611376会导致在nhei限制性位点的5’末端的“t”碱基被移除,这将除去存在于初始pmhct068克隆中的不期望的atg起始密码子)扩增编码seqidno:17的阿维链霉菌adc,并且为了在大肠杆菌中表达而经过密码子优化的多聚核苷酸。pcr反应体系(50μl)包含1μl1比50稀释的小量制备的含有为大肠杆菌表达经密码子优化的阿维链霉菌adc基因的质粒,1xthermopol反应缓冲液(newenglandbiolabs),引物0611376和0611377每种100pmol,datp、dctp、dgtp和dttp每种200μm,2μl100mmmgso4,和2个单位的(exo-)dna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为94℃2分钟的1个循环,随后是每个为94℃30秒,54℃30秒和72℃1分钟的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约420bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。然后将“左+pyd4#1”质粒用nhei和asci消化,再用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约7.5kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。上述纯化的含有对于大肠杆菌经密码子优化的阿维链霉菌adc基因的pcr片段,用nhei和asci消化,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约420bp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。随后在连接反应中,将得到的片段连接到线性的“左+pyd4#1”载体中,所述连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),100ngnhei/asci“左+pyd4#1”载体,31ngnhei和ascii消化过的pand插入片段,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后置于冰上。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。如本文所述,通过asci和pvuii消化筛选到数个有对于大肠杆菌经密码子优化的阿维链霉菌adc基因片段的合适插入的所得的转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct070。质粒pmhct070用作基础载体,用于添加为在酵母宿主中表达而经过密码子优化的adc和baat同源物。pmhct070用xbai和paci消化,用10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约6.5kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。将如上所述的经xbai和paci消化的片段,所述片段含有编码阿维链霉菌baat(seqidno:140),并为了在东方伊萨酵母中表达而经过密码子优化的多核苷酸,连接到pmhct070切割载体上,如下所示:连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),42ng经xbai和paci消化的pmhct070载体,4μl密码子优化的阿维链霉菌baatxbai和paci消化的插入片段,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后置于冰上。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。如本文所述,通过xbai,paci和ecorv消化筛选到数个有所需baatorf的正确插入的所得的转化体。产生所需片段大小的克隆被通过dna测序确认正确并命名为pmhct072。将在pmhct072中的为在大肠杆菌中表达而进行密码子优化的阿维链霉菌adc基因,用为在东方伊萨酵母中表达而进行的密码子优化的型式(seqidno:130)进行替换。东方伊萨酵母密码子优化adc基因(seqidno:130),连同所需另外的限制性位点和侧翼dna用引物0611378和0611379来扩增。pcr反应体系(50μl)包含1μl1比50稀释的小量制备的含有密码子优化的阿维链霉菌pand基因的质粒1xthermopol反应缓冲液(newenglandbiolabs),引物0611378和0611379每种100pmol,datp、dctp、dgtp和dttp每种200μm,2μl100mmmgso4,和2个单位的(exo-)dna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为94℃2分钟的1个循环,随后是每个为94℃30秒,54℃30秒和72℃45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约420bp的pcr产物被从胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。5μl小量制备的pmhct072用xbai和paci消化,再用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过1%的琼脂糖凝胶电泳纯化,约7.5kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。在随后的in-fusiontmadvantagepcrcloningkit(clontech)反应中,将含有来自于上面的密码子优化的阿维链霉菌adc基因的分离pcr产物加入到载体中:10μl反应体积由6μlpmhct072消化和纯化载体,1μl纯化的密码子优化的pandpcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过nhei,asci和clai消化筛选到数个合适地插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct074(图7)。pmhct074是左端pdc靶向构建体,含有驱动密码子优化的阿维链霉菌adc(pand,seqidno:130)表达的pdc启动子,tal终止子,驱动密码子优化的阿维链霉菌baat(seqidno:140)表达的eno1启动子,rki终止子,东方伊萨酵母ura3启动子和东方伊萨酵母ura3orf的5’片段。含有阿维链霉菌adc(seqidno:130)和克鲁维酵母baat(seqidno:142)的左端片段为了构建表达克鲁维酵母baat(pyd4)的左端dna构建体,将含有为在东方伊萨酵母中表达而经过密码子优化的克鲁维酵母baat序列(seqidno:142,如上)的xbai和paci消化的片段连接到上面的pmhct070消化载体上,如下所示:连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),42ng经xbai和paci消化的pmhct070载体,4μl密码子优化的克鲁维酵母baatxbai和paci消化的插入物,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后置于冰上。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被置于2xyt+amp平板上并在37℃下孵育过夜。如本文所述,通过xbai,paci和ecorv消化筛选到数个有所需pyd4orf的合适插入所得的转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct073。质粒pmhct073含有所需的,为在东方伊萨酵母中表达而经过密码子优化的克鲁维酵母baat(pyd4)序列,但是并不含有所需的为在东方伊萨酵母中表达而经过密码子优化的阿维链霉菌adc(pand)序列。为了移入orf,5μl小量制备的pmhct073用xbai和paci消化,再用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过1%的琼脂糖凝胶电泳纯化,约7.5kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。在随后的in-fusiontmadvantagepcrcloningkit(clontech)反应中,将含有来自于上面的密码子优化的阿维链霉菌pand基因(如上)的分离pcr产物加入到载体中:10μl反应体积由6μlpmhct073消化和纯化载体,1μl纯化的密码子优化的pandpcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过nhei,asci和clai消化筛选到数个合适地插入了所需pcr产物所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct076。质粒pmhct076是左端pdc靶向构建体,含有驱动为在东方伊萨酵母中表达而经密码子优化的阿维链霉菌adc(pand,seqidno:130)表达的pdc启动子,tal终止子,驱动为在东方伊萨酵母中表达而经密码子优化的克鲁维酵母baat(seqidno:142)表达的eno1启动子,rki终止子,东方伊萨酵母ura3启动子和东方伊萨酵母ura3orf的5’片段。测序确定,质粒pmhct076在到pdc启动子中约200bp处有a至t的核苷酸改变,以及在pdc启动子的~2/3处有g至t的核苷酸改变,而这些都是存在于pmhct068亲本载体中的(如上)。为了解决潜在基因表达改变的担忧,如下所述克隆一个类似pmhct076,但是含有正确pdc启动子的构建体。5μl小量制备的pmhct082用nhei和paci消化,再用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约4.7kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。4μl小量制备的pmhct076用nhei和paci消化,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约3.3kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。在连接反应中将纯化的4.7kbp载体和3.3kbp插入片段连接起来,所述反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),3μlpmhct082载体,6μlpmhct076插入物,1μlxquickt4dna连接酶(newenglandbiolabs)。反应物在室温下孵育5分钟,随后被置于冰上。根据制造商的说明使用5μl该反应来转化onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。通过stui和noti消化来筛选到数个合适地插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct083(图8)。质粒pmhct083与pmhct076相同,除了前者含有正确的pdc启动子序列,而后者如上所述具有一个a至t的核苷酸改变和一个g至t的核苷酸改变。测试表明,与pmhct083和pmhct077相比,来自于pmhct076和pmhct077整合的表达阿维链霉菌pand的菌株中,pand酶活性并没有差异。含有阿维链霉菌adc(seqidno:130)和酿酒酵母gabt(seqidno:141)的左端片段4μl小量制备的pmhct083用xbai和paci消化,用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约6.5kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。将经xbai和paci消化的片段,所述片段含有编码酿酒酵母gabt并为了在东方伊萨酵母中表达而经过密码子优化(uga1,seqidno:141),连接到pmhct083切割载体上,如下所示:连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),1μl经xbai和paci消化的纯化pmhct083载体,3μl密码子优化的酿酒酵母uga1xbai和paci消化的插入物,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后在冰上冷却。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。如本文所述,通过xbai和bglii消化筛选到数个有所需baatorf的合适插入的所得的转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct087(图9)。pmhct087是左端pdc靶向构建体,含有驱动为在东方伊萨酵母中表达而经密码子优化的阿维链霉菌adc(pand,seqidno:130)表达的pdc启动子,tal终止子,驱动为在东方伊萨酵母中表达而经密码子优化的酿酒酵母gabt(uga1,seqidno:141)表达的eno1启动子,rki终止子,东方伊萨酵母ura3启动子和东方伊萨酵母ura3orf的5’片段。实施例3-4:在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh)的插入载体的右端片段的构建含有大肠杆菌3-hpdh(seqidno:143)的右端片段2μl小量制备的pmhct069(如上)用xbai和paci消化,再用10单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,在tae缓冲液中通过0.9%的琼脂糖凝胶电泳纯化,约2.2kbp的条带从凝胶上切下,根据制造商的说明使用extractiikit(macherey-nagel)纯化。将经xbai和paci消化的片段,所述片段含有为了在东方伊萨酵母中表达而经过密码子优化(ydfg,seqidno:143)的大肠杆菌3-hpdh基因,连接到pmhct069切割载体上,如下所示:连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),2μl经xbai和paci消化的纯化pmhct069载体,4μlxbai和paci消化的密码子优化的大肠杆菌ydfg插入物,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后在冰上冷却。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。如本文所述,通过xbai,paci和ecorv消化筛选到数个有所需ydfgorf的合适插入的所得的转化体。如本发明中所述产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct075(图10)。质粒pmhct075含有东方伊萨酵母ura3orf的3’片段,来自于东方伊萨酵母的ura3终止子,其后是ura3启动子(用于将来将ura3标志环出),为了在东方伊萨酵母中表达而经密码子优化的大肠杆菌3-hpdh基因(ydfg,seqidno:143),其由东方伊萨酵母tdh3启动子所驱动,以及东方伊萨酵母pdc终止子区域。含有酿酒酵母3-hpdh(seqidno:144)的右端片段将用xbai和paci消化的,含有为了在东方伊萨酵母中表达而经过密码子优化的酿酒酵母ymr226c基因(如上)的片段连接到pmhct069切割载体中,如下所示:连接反应(20μl)含有1xquick连接缓冲液(newenglandbiolabs),2μl经xbai和paci消化的纯化pmhct069载体,4μlxbai和paci消化的密码子优化的酿酒酵母3-hpdh(ymr226c,seqidno:144)插入物,以及1μlquickt4dna连接酶(newenglandbiolabs)。连接反应在室温下孵育5分钟,然后置于冰上。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。如本文所述,通过xbai,paci和ecorv消化筛选到数个有所需ymr226corf的合适插入的所得的转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct077(图11)。质粒pmhct077含有东方伊萨酵母ura3orf的3’片段,来自于东方伊萨酵母的ura3终止子,其后是ura3启动子(用于将来将ura3标志环出),为了在东方伊萨酵母中表达而经密码子优化的酿酒酵母3-hpdh基因(ymr226c,seqidno:144),其由东方伊萨酵母tdh3启动子所驱动,以及东方伊萨酵母pdc终止子区域。实施例3a-5:在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh)的杂合和纯合酵母菌株上面的实施例3a-3和3a-4描述了为使三种异位基因同时在东方伊萨酵母pdc基因座中靶向表达的各种左端或右端构建体的构建。在转化前,每种构建体(一个所需的左端构建体和一个所需的右端构建体)各约10μg用noti消化,以将所需的转化dna从puc18骨架载体中释放出来;对于大多数消化,在noti消化中也包括限制性酶pvui。限制性酶pvui将puc18载体片段消化为两半,使得用凝胶电泳能够更容易地分离较大的所需dna片段。含有较大的表达盒的条带可通过凝胶电泳从puc18骨架dna中分离出来,并从凝胶中切割,根据制造商的说明使用extractiikit(macherey-nagel)纯化。30μl洗脱缓冲液被用于洗脱步骤。等摩尔比的左端和右端构建体,共10μl用于转化东方伊萨酵母菌株cnb1或合适的衍生体。在ura选择平板上选择转化体,置于37℃下生长。第二天,挑取出约12个转化体,并在ura选择平板上重新划线以得到单个菌落,在37℃下生长。翌日,从每个起始转化体生成的每条线中挑取单独的菌落,在ura选择平板上重新划线以得到单个菌落。37℃下再生长一个晚上后,从每条线中挑取最终的单菌落,在ura选择平板上重新划线,在37℃下生长过夜。经过这种两轮单菌落纯化和生长之后,制备用于pcr的基因组dna,所述pcr是为了验证如上所述的所需的靶向整合的发生。为了使用左-和右端构建体靶向到pdc,使用引物0611814,0611554和0611555来进行所需整合事件的验证。引物0611554结合到东方伊萨酵母基因组dna中正好是pdc终止子区域的3’端,该区域存在于右端pdc靶向构建体中;引物0611555结合到pdcorf中,进行扩增直至停止;引物0611814结合到存在于右端构建体中的tdh3启动子区域的3’末端附近,并朝3’方向进行扩增。从含有引物0611814和0611554的pcr中产生约1.9kbp的条带表明在pdc基因座发生了所需整合事件。从含引物0611555和0611554的pcr中产生约1.4kbp的条带表明存在野生型pdc基因座。由于该整合事件是二倍体东方伊萨酵母cnb1中的第一个靶向事件,所需整合体对引物0611814和0611554将显示出1.9kbp的条带,对引物0611555和0611554则显示出1.4kbp的条带。对每个质粒具有所需条带谱型的两个独立转化体被命名,如表15中所示。表15:转化体基因型接下来,如上所述分离ymhct004或ymhct005的ura-衍生体。使用引物0611815和0611817,用pcr对每个亲本菌株的几个foa抗性菌落的基因组dna进行所需环出事件的筛选。引物0611815在左端构建体的rki终止子上进行退火,朝着ura3启动子扩增。引物0611817在tdh3启动子中退火,反方向朝着ura3盒扩增。存在828bp的条带意味着只存在所期望的ura3残留位点(在亲本菌株的两个ura3启动子之间发生同源重组后,所留下的单个ura3启动子),而约2.2kbp的条带则意味着存在完整的ura3启动子-ura3orf-ura3终止子-ura3启动子盒,这表明所期望的重组事件并没有发生。如上所述,实施crimsontaqtmdna聚合酶(newenglandbiolabs)的pcr反应。来自于亲本菌株ymhct004的一个foa抗性菌落,被命名为ymhct012,来自于亲本菌株ymhct005的一个foa抗性菌落,命名为ymhct007,都具有所期望的828bp条带。接下来转化菌株ymhct012和ymhct007,以构建pdc基因被缺失并且分别被pand,pyd4,和ymr226c或者pand,uga1和ymr226c的表达盒替换的纯合菌株。用来自于pmhct083和pmhct077的线性dna转化菌株ymhct012,用来自于pmhct087和pmhct077的线性dna转化菌株ymhct007。经过两轮的单菌落纯化后,用引物0611815和0611817,用pcr筛选每个亲本菌株的几个转化体的基因组dna,如上所述。来自于各个亲本菌株的两个独立分离的整合体都具有828bp条带(扩增来自于原始靶向pdc基因座的ura3残留区域得到的)和2.2kbp条带(在另一条染色体上发生第二次整合的ura3启动子-ura3orf-ura3终止子-ura3启动子盒整合所得到的),命名所述整合体,如表16所示。表16:转化体基因型如上所述分离ymhct008的ura-衍生体。使用引物0611815和0611817,用pcr对ymhct008亲本菌株的几个foa抗性菌落的基因组dna进行所需环出事件的筛选。存在828bp的条带意味着只存在所期望的ura3残留位点(在亲本菌株的两个ura3启动子之间发生同源重组后,所留下的单个ura3启动子)。只显示828bp条带的分离物用引物0611555和0611554进行进一步的筛选,如本文所述。从含有引物0611555和0611554的pcr中产生约1.4kbp的条带意味着存在野生型pdc基因座。缺少该条带的分离物,这表明两条染色体上的pdc基因座都丢失,被命名为ymhct010。在摇瓶中培养菌株,制备cfe,测定天冬氨酸脱羧酶(adc)活性和3-hp脱氢酶(3-hpdh)活性,如本文所述。这几个测定系列的实验结果(记为试验1-4)如表17中所示。还对来自于表17中试验1的菌株进行sds-page分析,如本文所述。在sds-page分析中,试验1中的菌株74/75#1和菌株ymhct002在27kd处产生了条带,而该条带在试验1的对照菌株中(mbin500)并不存在。该蛋白质条带的大小与其作为ydfg基因编码的蛋白质的身份是吻合的。表17:转化体酶活数据另一个测定系列的实验结果示于表17中(试验2)。表17中试验2的菌株也用sds-page进行分析,如本文所述。在sds-page分析中,除mbin500之外,试验2中的所有菌株在29kd处都具有条带。该蛋白质条带的大小与其作为ymr226c基因编码的蛋白质的身份是吻合的。试验2中的ymhct005和87/77#2在53kd处具有条带,而该试验中的其他三个样品中却不存在该条带。该蛋白质条带的大小与其作为uga1基因编码的蛋白质的身份是吻合的。另一个测定系列的实验结果示于表17中(试验3)。表17中试验3的菌株也用sds-page进行分析,如本文所述。在sds-page分析中,除mbin500之外,试验3中的所有菌株在53kd和29kd处都具有条带。该蛋白质条带的大小分别与其作为uga1和ymr226c基因编码的蛋白质的身份是吻合的。在sds-page分析中,试验3中的mbin500和ymhct005在64kd处具有条带,而该试验中的ymhct008和ymhct009中却不存在该条带。该蛋白质条带的大小与其作为东方伊萨酵母酵母cnb1的天然丙酮酸脱羧酶(pdc)基因编码的蛋白质的身份是吻合的。另一个测定系列的实验结果示于表17中(试验4)。表17中试验4的菌株也用sds-page进行分析,如本文所述。在sds-page分析中,除mbin500之外,试验4中的所有菌株在29kd处都具有条带。该蛋白质条带的大小与其作为ymr226c基因编码的蛋白质的身份是吻合的。试验4中的ymhct005,ymhct008和ymhct009在53kd处具有条带,该蛋白质条带的大小与uga1基因编码的蛋白质是吻合的。试验4中的ymhct013和ymhct014菌株在53kd处也有一条微弱的条带,该条带的大小与pyd4基因编码的蛋白质是吻合的。试验4中的mbin500,ymhct004和ymhct005在64kd处具有条带,而该条带在菌株ymhct008,ymhct009,ymhct013和ymhct014中并不存在。该蛋白质条带的大小与其作为东方伊萨酵母酵母cnb1的天然丙酮酸脱羧酶(pdc)基因编码的蛋白质的身份是吻合的。使用本文所述的方法,在生物反应器中测试菌株mbin500和ymhct008,评估3-hp生产。对照菌株mbin500并没有产生可检测的3-hp(两个独立发酵的平均值)。菌株ymhct008产生2.45g/l的3-hp(12个独立发酵的平均值)。实施例3a-6:在adh1202基因座表达β-丙氨酸氨基转移酶(baat)或3-羟基丙酸脱氢酶(3-hpdh),在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh)的酵母菌株用noti和pvui消化20μgpmhct077,pmhct083和pmhct087(如上),然后以89mmtris碱-89mm硼酸-2mmedta二钠缓冲液(tbe)中进行1%琼脂糖凝胶电泳。noti消化片段3815bp,5332bp或5320bp分别来自于pmhct077,pmhct083和pmhct087,从凝胶中切割下来,并使用gelextractkit(qiagen)从琼脂糖中提取。将560ngnoti消化后的pmhct077和560ngnoti消化后的pmhct083或pmhct087转化进菌株miba320,miba321和miba322。miba320用pmhct077/83以及pmhct077/87组合转化。miba321用pmhct077/87转化,miba322用pmhct077/83转化,如本文所述。将转化体涂布于ura选择培养基上,室温孵育约60小时。转化体在ura选择培养基上重新划线,在37℃下孵育过夜。从ura3+菌落中制备基因组dna,如本文所述使用pcr检查以验证表达盒的整合。引物对611814和611554扩增出1.9kbp片段表明整合。引物对611555和611554扩增出1.4kbp片段表明是野生型基因座。用611554和611814扩增出1.9kbppcr片段,用611555和611554扩增出1.4kbp片段的每个品系中的一个ura3+转化体被保存,它们被命名为miba323,miba324,miba325和miba327(见表18的基因型)。启动子和终止子来源于东方伊萨酵母基因。表18:转化体基因型菌株在摇瓶培养,如本发明中所述制备cfe并测试3-hp脱氢酶(3-hpdh)活性。结果示于表19中。表19:转化体酶活性数据如本发明所描述分离miba323、miba324、miba325和miba327的ura-衍生体。如本发明所描述的,从foa抗性菌落制备基因组dna并以pcr检查来确定失去了ura3选择标志物。引物0611815在左端构建体rki终止子退火并向ura3启动子扩增,而引物0611817在tdh3启动子退火并反向朝ura3盒扩增。828bp条带的存在表示仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.2kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。以引物0611815和0611817获得了828bppcr片段的miba323、miba324、miba325和miba327的ura-菌株被保存并被分别命名为miba335、miba333、miba334和miba336。按上面miba320-2转化部分中所描述,以来自pmhct077和pmhct087的片段转化菌株miba333和miba334,并以来自pmhct077和pmhct083的片段转化菌株miba335和miba336。如本发明中所描述通过在ura选择培养基上生长来选择转化体。如本发明所描述的,从ura3+菌落制备基因组dna并以pcr检查来确定表达盒的整合。引物0611815在左端构建体rki终止子退火并向ura3启动子扩增。引物0611817在tdh3启动子退火并反向朝ura3盒扩增。对第一整合所期望的828bp条带的存在表示仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而对第二整合约2.2kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在。当ura标志存在时,引物对0611815和0611816扩增625bp片段。当pdc基因座存在时,引物0611555和0611554扩增1.4kbp片段。纯合的整合株不应该能以这些引物扩增片段。保存以引物0611815和0611817扩增出828bp和2.2kbppcr片段的,以引物0611815和0611816扩增出625bp的,以引物0611555和0611554未扩增出片段的每个品系的一个ura3+转化体;这些被命名为miba340、miba341、miba345和miba348(见表20a)。启动子和终止子来自东方伊萨酵母基因。表20a:转化体基因型比较从菌株mbin500(对照)、miba345、miba348、miba340和miba341制备的cfe中的天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-hp脱氢酶(3-hpdh)活性。该实验的结果显示于表20b中。表20b:转化体酶活性数据实施例3a-7:具有用于在adh1202基因座表达天冬氨酸1-脱羧酶(adc)的多个核苷酸序列的插入载体左端片段设计构建体以在东方伊萨酵母adh1202基因座掺入编码adc(seqidno:17)的四个核苷酸拷贝。使用与本发明所描述的对pdc基因座所用方法类似的方法,设计左端和右端构建体以允许同源重组。整合载体的一般设计和所需的重组事件在图3中显示。该方法也被用于表达如下面实施例中所描述的可选的adc(seqidno:139)。为了防止重组在编码相同adc序列的多个核苷酸拷贝之间发生,设计四种不同的为在东方伊萨酵母中表达而密码子优化的核苷酸序列(seqidno:130、145、146和147)以编码相同的seqidno:17的adc序列。此外,由于构建体的初始设置被设计为靶向东方伊萨酵母的ald5680基因座,adh1202靶向序列在克隆的后面步骤中被掺入这些载体。ald5680构建体可以被用于靶向东方伊萨酵母cnb1菌株中的第二基因座,该菌株中在adh1202异位的四个pand拷贝与在ald5680的四个另外的pand拷贝已纯合。如下构建左端ald5680靶向载体。以引物0612271和0612272扩增含紧接着ald5680orf的5’端的序列,连同有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的phjj75小量制备质粒dna(图23),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612271和0612272每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的32个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约930碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。为允许纯化更大量的dna,使用纯化的930bppcr产物作为模板dna来重复上述pcr反应。设置5个50μl的反应并以上述条件扩增,除了以1μl纯化的930bppcr产物替代phjj75质粒(如上)作为模板dna。热循环后,如上纯化扩增的930bp产物。含pdcpromo-optpand-eno1-uga1的片段(其含有seqidno:130的为东方伊萨酵母密码子优化的阿维链霉菌adc编码序列)通过noti和ecori消化,从pmhct087(如上)上切下。10μgpmhct087的中量制备品被以noti和ecori消化并随后通过tae缓冲液中0.9%琼脂糖凝胶电泳纯化。约4.4kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用引物0612273和0612274扩增含ura3分裂标志的5’半边,并有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的pmhct082小量制备质粒dna(如上),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612273和0612274每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的32个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约960碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。为构建上述dna片段的接收载体,质粒puc19(yanisch-perron,c,vieira,j.和messing,j.(1985)gene,33,103-119)被以hindiii和ecori消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化,约2.6kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。来自上面的纯化的930bp、4.4kbp和960bp的dna片段随后被使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中插入消化的puc19片段,10μl总反应体积由150ng以hindiii和ecori消化的puc19载体,56ng含ald5680侧翼dna的930bpdna,250ng来自以noti和ecori消化的pmhct087的pdcpromo-optpand-eno1-uga1片段,55ng含ura3分裂标志5’半边的960bppcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。随后以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。通过sall和hpal消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct089。接下来,以编码seqidno:17adc的,为在东方伊萨酵母中表达而密码子优化的阿维链霉菌pand基因来替代pmhct089中的uga1orf。通过在pma-t载体中合成阿维链霉菌pand型式r1(seqidno:145)。以xbal和pacl消化质粒pma-t,得到的片段被以tae缓冲液中0.9%琼脂糖凝胶电泳分离,约434bp的条带被从胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒pmhct089被以xbal和pacl消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,而得到的片段被以tae缓冲液中0.9%琼脂糖凝胶电泳分离,约4.5kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。在含1xquick连接缓冲液(newenglandbiolabs),2μlxbal/paclpmhct089载体,2μlxbal/pacl阿维链霉菌pand型式r1插入物,和1μlquickt4dna连接酶(newenglandbiolabs)的连接体系中(20μl)将pmhct089载体和阿维链霉菌pand型式r1结合。连接反应物被在室温下孵育5分钟,并随后被置于冰上。5μl该反应物被用于根据制造商的说明转化onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。通过xbal和pacl消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct092。左端构建体的最后克隆步骤是以adh12025’同源区域替代存在于pmhct092中的ald56805’同源区域。以引物0612470和0612471扩增含ald1202orf5’端序列,连同有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的pgmer140小量制备质粒dna(本发明所描述pmiba107的衍生体,其中pcr扩增的区域相同),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612470和0612471每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃30秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约790bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。为构建上述dna片段的接收载体,质粒pmhct092被以hpal和noti消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化,约7.0kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中将pcr产物和线性载体联结,10μl总反应体积由120ng以hpal和noti消化的pmhct092载体,30ng含adh12025’同源区的pcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育过夜。通过bamhi和pstl消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct095(图12)。质粒pmhct095是左端东方伊萨酵母adh1202靶向构建体,其含有驱动为在东方伊萨酵母中表达而密码子优化的阿维链霉菌adc基因(seqidno:130)表达的pdc启动子,tal终止子,驱动为在东方伊萨酵母中表达而密码子优化的第二阿维链霉菌adc基因(seqidno:145)表达的eno1启动子,东方伊萨酵母rki终止子,东方伊萨酵母ura3启动子和东方伊萨酵母ura3orf的5’片段。实施例3a-8:具有用于在adh1202基因座表达天冬氨酸1-脱羧酶(adc)的多个核苷酸序列的插入载体右端片段以引物0612275和0612276扩增含东方伊萨酵母ura3orf3’片段,连同有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的pmhct069小量制备质粒dna(如上),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612275和0612276每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的32个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约1155bp的pcr产物被从胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。含tdh3启动子,用于异位基因插入的xbal和pacl限制性位点,和pdc终止子的片段通过以noti和pmel消化被从pmhct069上切下。10μgpmhct069的中量制备品被以noti和pmel消化,并随后通过tae缓冲液中0.9%琼脂糖凝胶电泳纯化。约1.85kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。以引物0612277和0612278扩增含ald5680orf3’序列,连同有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的phjj75小量制备质粒dna(图23),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612277和0612278每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃45秒的32个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约844bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。来自上面的纯化的1155bp、1.85kbp和844bp的dna片段随后被使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中插入以ecori和hindiii消化的puc19,10μl总反应体积由150ng以hindiii和ecori消化的puc19载体,66ng含ura3分裂标志3’部分的1155bpdna;106ng来自以pmel和noti消化的并含tdh3启动子,用于异位基因插入的xbal和pacl限制性位点,和pdc终止子的来自pmhct069的1.85kbp片段;48ng含3’ald5680侧翼dna的844bppcr产物;1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。通过sall和hpal消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为ald5680右#20。如下在tdh3启动子和ald5680右#20的3’ald5680侧翼dna之间加入tkl终止子,pgk1启动子,xbal和pacl限制性位点以及pdc终止子区域的较短型式。以引物0612356和0612357扩增tkl终止子连同所需的另外限制性位点和克隆用的侧翼dna。使用退火温度的温度梯度和在某些反应中的dmso来扩增所需的pcr产物。准备四个相同的pcr反应体系,每个pcr反应体系(50μl)包含1μl1比50稀释的pacn23小量制备质粒dna(图20),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612356和0612357每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。如上设置第二组四个管,除了每个反应包括加入1.5μl的dmso。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,x℃20秒,其中x=47.6℃、51.8℃、57.1℃或62.1℃,和72℃45秒的32个循环,及72℃10分钟的最后延伸。在每个退火温度进行有和没有dmso的pcr。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离每种10μl的pcr反应物。对该凝胶的观察显示有dmso在两个最高的退火温度进行的,以及没有dmso在两个最低退火温度进行的pcr反应给出了所需844bp产物的最高产量。将这四个pcr反应物结合,以tae缓冲液中1%琼脂糖凝胶电泳来分离,其中约844碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。以两步骤过程来完成所需pgk1启动子区域的pcr扩增。首先,以下面引物0612150和0612151扩增之后克隆含pgk1启动子的pcr产物。pcr反应体系(50μl)包含3μlpjlj49小量制备质粒dna(图25),1xpfx扩增缓冲液(invitrogen),2mmmgso4,引物0612150和0612151每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1.25个单位的pfxdna聚合酶(invitrogenbio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为95℃2分钟的1个循环,随后是每个为95℃1分钟,55℃1分钟和72℃3分钟的25个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约630bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。根据制造商的说明,使用测序用zeropcrcloningkit(invitrogen)将约630bp的pcr产物克隆进pcr4tmblunt载体(invitrogen)。在6μl总反应体积中,0.5或4μl630bppcr产物,1μl盐溶液(invitrogen)和1μlpcr4tmblunt(invitrogen)一起在室温孵育15分钟。根据制造商的说明每种克隆反应物2μl被转化入onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被置于lb+kan平板上并在37℃孵育过夜。通过ecori消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pgk1_in_topo。用作pcr模板之前,来自pgk1_in_topo的pgk1启动子被如下分离并纯化。25μl的pgk1_in_topo中量制备品以xbal和pacl消化并通过tae缓冲液中0.9%琼脂糖凝胶电泳来纯化,而约640bp的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用温度梯度以引物0612358和0612359扩增pgk1启动子连同所需的另外限制性位点和克隆用的侧翼dna。设置八个相同的pcr反应,每个pcr反应体系(50μl)包含20ng通过pgk1_in_topo的xbal和pacl消化得到的纯化pgk1启动子dna,1xherculase反应缓冲液(agilenttechnologies),引物0612358和0612359每种100pmol,datp、dctp、dgtp和dttp每种200μm,和2.5个单位的herculasehotstartdna聚合酶(agilenttechnologies)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,x℃20秒,其中x=53.7℃、55.4℃、57.6℃、60.0℃、62.4℃、64.8℃、66.9℃、68.6℃,和72℃45秒的32个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离每种10μl的pcr反应物。该胶的观察显示以最高退火温度进行的pcr反应给出了所需约700bp产物的最高产量。将这四个pcr反应物结合,以tae缓冲液中1%琼脂糖凝胶电泳来分离,其中约700碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒ald5680右#20包含来自东方伊萨酵母pdcorf的约870bp的下游区域作为pdc终止子区域。然而,该区域可能比作为终止子适合功能所需的大,而如果保持在其目前的大小,该区域可能起对不想要的pdc基因座重组的催化剂的作用。因此,以引物0612360和0612361扩增以较小型式替换ald5680右#20中pdc终止子的pcr产物。使用退火温度的温度梯度和在某些反应中的dmso来扩增所需的pcr产物。建立四个相同的pcr反应体系,每个pcr反应体系(50μl)包含1μl1比50稀释的pjlj49小量制备质粒dna(图25),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612360和0612361每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。如上设置第二组四个管,除了每个反应包括加入1.5μl的dmso。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,x℃20秒,其中x=47.6℃、51.8℃、57.1℃或62.1℃,和72℃45秒的32个循环,及72℃10分钟的最后延伸。对于所示的每个退火温度,都实施了有和没有dmso的pcr反应。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离每种10μl的pcr反应物。对该凝胶的观察显示有dmso进行的四个pcr反应,无论退火温度,给出了所需338bp产物的最高产量。将这四个pcr反应物结合,以tae缓冲液中1%琼脂糖凝胶电泳来分离,其中约338碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用pcr构建将约700bp含pgk1的pcr产物的融合到338bp的pdc终止子产物上的单独扩增产物。pcr反应体系(50μl)包含107ng含pgk1的pcr产物,56ng含pdc终止子的pcr产物,1xphusionhf缓冲液(newenglandbiolabs),引物0612358和0612361每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的phusiontmhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,56℃20秒和72℃2分钟45秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约1020碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒ald5680右#20被以xbal和notl消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化。约5.6kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。来自上面的纯化的487bp和1020bp的pcr产物随后被使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中插入消化的ald5680右#20片段,10μl总反应体积由150ng以xbal和notl消化的ald5680右#20载体,13ngtkl终止子pcr产物,28ngpgk1启动子-pdc终止子pcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过accl消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct091。质粒pmhct091是空的右端东方伊萨酵母ald5680靶向构建体,其包含东方伊萨酵母ura3orf的3’片段,随后有用于加入感兴趣基因的nhel和ascl限制性位点的东方伊萨酵母tdh3启动子,东方伊萨酵母tkl终止子,随后有用于加入第二感兴趣基因的xbal和pacl限制性位点的东方伊萨酵母pgk1启动子,东方伊萨酵母pdc终止子,和将同源重组靶向3’ald5680基因座的侧翼dna。通过在载体1075328_sapand_r5中合成阿维链霉菌pand型式r5(seqidno:146)。xbal和pacl限制性位点被改变为通过pcr克隆进pmhct0915’克隆位点所需的nhel和ascl位点。pcr反应体系(50μl)包含1μl1比50稀释的1075328_sapand_r5小量制备质粒dna1xiprooftmhf缓冲液(bio-radlaboratories),引物0612378和0612379每种100pmol,datp、dctp、dgtp和dttp每种200μm,1.5μldmso和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃30秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中0.9%琼脂糖凝胶电泳来分离pcr反应产物,其中约471个碱基对的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒pmhct091(如上)被以nhel和ascl消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化。约6.9kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。来自上面的含pandr5的纯化pcr产物随后被使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中插入消化的pmhct091片段,10μl总反应体积由150ng以nhel和ascl消化的pmhct091载体,19ngpandr5pcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过smal消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct093。质粒pmhct093被以xbal和pacl消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化。约7.4kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。通过在载体pma-t中合成为在东方伊萨酵母中表达而被密码子优化的阿维链霉菌pand型式r2(seqidno:147)。以xbal和pacl消化质粒并以tae缓冲液中0.9%琼脂糖凝胶电泳分离得到的片段。约434bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。上面纯化的~434bp片段在连接反应体系(20μl)中被克隆进xbal和pacl消化的pmhct093载体,连接反应体系(20μl)含1xquick连接缓冲液(newenglandbiolabs),2μl以xbal和pacl消化的pmhct093载体,2μl上述的~434bp片段,和1μlquickt4dna连接酶(newenglandbiolabs)。连接反应物在室温下孵育5分钟,随后管被置于冰上。5μl该反应物被用于根据制造商的说明来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在室温下孵育3天。通过xbal和pacl消化筛选到数个合适插入了所需pcr产物的所得转化体。选择分离的pmhct094来进行之后的工作。右端构建体的最后克隆步骤是以adh12023’同源区域替代存在于pmhct094中的ald56803’同源区域。以引物612472和612473扩增含紧接着ald1202orf的3’端的序列,连同有所需的另外限制性位点和克隆用的侧翼dna的pcr产物。pcr反应体系(50μl)包含1μl1比50稀释的pgmer140小量制备质粒dna(如上),1xiprooftmhf缓冲液(bio-radlaboratories),引物0612472和0612473每种100pmol,datp、dctp、dgtp和dttp每种200μm,和1个单位的iprooftmhighfidelitydna聚合酶(bio-radlaboratories)。在(eppendorfscientific)中进行pcr,程序为98℃30秒的1个循环,随后是每个为98℃10秒,59℃20秒和72℃30秒的34个循环,及72℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物,其中约620碱基对的的pcr产物被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。为构建上述dna片段的接收载体,质粒pmhct094被以sacii和noti消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)在37℃下处理30分钟,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化,约7.0kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用in-fusiontmadvantagepcrcloningkit(clontech)在10μl总反应体积中将pcr产物和线性载体联结,10μl总反应体积由191ng以sacll和noti消化的pmhct094载体,36ng含adh12023’同源区的pcr产物,1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)构成。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。以40μlte缓冲液稀释反应物并根据制造商的说明使用2.5μl来转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过nsil和pvul消化筛选到数个合适插入了所需pcr产物的所得转化体。产生所需片段大小的克隆通过dna测序来确认正确并命名为pmhct096(图13)。质粒pmhct096是右端东方伊萨酵母adh1202靶向构建体,其含有东方伊萨酵母ura3orf的3’片段,驱动为在东方伊萨酵母中表达而密码子优化的第三阿维链霉菌adc基因(seqidno:146)表达的东方伊萨酵母tdh3启动子,东方伊萨酵母tkl终止子,驱动为在东方伊萨酵母中表达而密码子优化的第四阿维链霉菌adc基因(seqidno:147)表达的东方伊萨酵母rgk1pgk1启动子,东方伊萨酵母pdc终止子和将同源重组靶向到3’adh1202基因座的侧翼dna。实施例3a-9在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh);和在adh1202基因座表达来自四种核苷酸序列的天冬氨酸1-脱羧酶(adc)的酵母菌株上面的实施例3a-7和3a-8描述了左端和右端构建体的构建,左端和右端构建体是用于在adh1202基因座靶向表达为在东方伊萨酵母中表达而密码子优化的阿维链霉菌adc基因的四种核苷酸变体。在转化进东方伊萨酵母cnb1之前,以hpal和sacll消化10μgpmhct095以从puc19骨架载体中释放所需转化dna。类似的,以ecori和sacll消化10μgpmhct096以从puc19骨架载体中释放所需转化dna。含~5kbp表达盒的条带被通过凝胶电泳从puc19骨架dna中分离,从凝胶上切下,并根据制造商的说明使用extractiikit(macherey-nagel)纯化。30μl洗脱缓冲液被用于洗脱步骤。总计10μl的等摩尔比的pmhct095和pmhct096线性转化dna被用于转化菌株ymhct010(如上)。在ura选择平板上选择转化体并将其置于37℃下生长。翌日挑出约12个转化体并将其再划线到ura选择平板上以获取单菌落并在37℃下生长过夜,随后从每个初始转化体生成的每个划线中挑取单菌落并再划线到ura选择平板上。在37℃下生长再一夜之后,从每个划线中挑取最终单菌落并再划线到ura选择平板上,并37℃下生长过夜。在这第二轮单菌落纯化和生长之后,制备基因组dna来用于如本发明中所述验证所需靶向整合发生的pcr。使用引物0611718和0611632(如上)来验证pmhct095和pmhct096片段正确靶向到adh1202基因座。引物0611718结合到存在于pmhct096中的pdc终止子区域,而引物0611632结合到靶向区域adh1202基因座dna3’端并以反义方向扩增。以这些引物从pcr中生成约727bp条带表明了在adh1202基因座所需整合事件的发生。pcr反应体系(25μl)包含0.5μl要被筛选的菌株的基因组dna,1xcrimsontaqtm反应缓冲液(newenglandbiolabs),25pmol有义引物,25pmol反义引物,datp、dctp、dgtp和dttp每种200μm,和0.625个单位的crimsontaqtmdna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为95℃30秒的1个循环,随后是每个为95℃30秒,50℃30秒和68℃2.5分钟的30个循环,及68℃10分钟的最后延伸。热循环后,以tae缓冲液中1%琼脂糖凝胶电泳来分离pcr反应产物并如上所述观察和解释条带的大小。给出所需727bp条带的两个独立分离的转化体被命名为ymhct019或95/962(见表21中的基因型)。表21:转化体基因型随后如本发明所描述分离ymhct019的ura-衍生体。使用引物0611815和0612795通过pcr筛选到来自ymhct019数个foa抗性菌落的基因组dna有所需环出事件。引物0611815在左端构建体rki终止子处退火并向ura3启动子扩增,而引物0612795在3’adh1202同源区域中(pmhct096的或者内源adh1202基因座)反向朝着5’区域退火。如上所述对分离的ymhct019进行pcr反应,除了延伸阶段的长度被变为3.5分钟。以这些引物生成3.7kbp条带表明所需的环出事件已经发生而仅ura3启动子残留剩余在修饰的adh1202基因座,而约5.1kbp的条带会表明完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表示所需重组事件没有发生。给出所需3.7kp条带的菌株被保存并命名为ymhct021。为了分离多个在adh1202的pand表达盒的纯合的ymhct021衍生体,如上所述以线性化的pmhct095和pmhct096转化ymhct021。在两轮单菌落纯化和生长之后,制备基因组dna来用于验证所需靶向整合发生的pcr。使用引物0612891和0612893来验证pmhct095和pmhct096片段正确靶向到ymhct021剩余野生型的adh1202基因座。引物0612891在sapandr1加上pmhct095r1终止后的pacl位点半边的3’区域退火。引物0612893在sapandr5的最5’区域退火,包括nhel位点和pmhct096的前导序列,并在反向互补的方向扩增。按照从ymhct021剩余野生型的adh1202基因座上经pmhct095和pmhct096的第二重组事件所预期,这些引物生成3.2kbp条带表明完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,而约1.7kbp条带表明在其他adh1202基因座处ura3残留位点的存在(来自初始整合事件和随后的ura3标记环出)。按上面对分离ymhct019所描述的进行pcr反应,除了延伸阶段的长度被变为3.5分钟。给出两个这些条带的菌株被命名为ymhct022(见表22中的基因型)。表22:转化体基因型菌株在摇瓶中培养,如本发明中所述制备cfe并测试天冬氨酸脱羧酶(adc)活性。实验结果在表23a中显示。表23a:转化体酶活性数据比较了从菌株mbin500(对照),ymhct019、95/96-2、ymhct008和ymhct022制备的cfe中的天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-hp脱氢酶(3-hpdh)活性。该实验的结果在表23b中显示。表23b:转化体的酶活性数据也如本发明所述通过sds-page分析了菌株ymhct019和95/962。两个菌株都显示在53kd、29kd、~14kd和~3kd的蛋白条带。53kd和29kd蛋白条带的大小分别与uga1和ymr226c基因所编码蛋白的大小相符。14和3kd蛋白条带组合的大小与pand基因所编码的经翻译后切割的蛋白相符。在对照菌株mbin500的sds-page分析中没有观察到53kd、29kd,14kd和3kd蛋白。使用本发明描述的方法,在生物反应器中评价菌株mbin500和ymhct019的3-hp产生。对照菌株mbin500没有产生可检测到的3-hp(两个独立的发酵的平均)。菌株ymhct019产生5.23g/l的3-hp(三个独立的发酵的平均)。实施例3a-10:在adh1202基因座从四种核苷酸序列表达天冬氨酸1-脱羧酶(adc)的酵母菌株设计另外的构建体以在adh1202基因座掺入编码来自地衣芽孢杆菌的替代adc(seqidno:139)的四个核苷酸拷贝。以与上述方式类似的方式,设计左端和右端构建体以允许在东方伊萨酵母adh1202基因座的同源重组。左端片段的构建质粒pmhct095(如上)被如本发明中所述以xbal和pacl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约7.3kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。地衣芽孢杆菌天冬氨酸脱羧酶(adc)pand基因为在东方伊萨酵母中表达而被密码子优化(型式1,seqidno:149)并合成构建入质粒1110206质粒1110206被如本发明中所述以xbal和pacl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约380bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将~380bp的纯化片段连接进7.3kbp的pmhct095线性化载体,10μl总反应体积由60.5ng消化的pmhct095,6.3ng来自1110206的380bp片段,有10mmatp的1μl10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育1.5小时,而3μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用xbal和pacl限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmeji309。质粒pmeji309被以nhel和ascl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约7.3kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。地衣芽孢杆菌天冬氨酸脱羧酶pand基因为在东方伊萨酵母中表达而被再次密码子优化(型式2,seqidno:148)并合成构建入质粒1110205在含3μl1110205,引物0612695和0612724每种25pm,1xpfx扩增缓冲液(invitrogen),2mmmgso4,1.25个单位pfxdna聚合酶(invitrogen)的终体积50μl的混合物中进行pcr。扩增反应物在(eppendorfscientific)中孵育,程序为95℃2分钟的1个循环;每个为95℃1分钟,55℃1分钟和72℃1分钟的25个循环;以及72℃3分钟的1个循环。扩增反应的pcr产物被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约400bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。纯化的pcr产物被使用in-fusiontmadvantagepcrcloningkit(clontech)在反应中插入上面消化的pmeji309载体,反应体系包括93ngpmeji309载体片段,52ng上述pcr产物,2μl1xin-fusiontmin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。2.5μl反应物样品根据制造商的说明转化入onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过使用xbal和pacl的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆通过dna测序来确认正确并命名为pmeji310-2(图14)。右端片段的构建质粒pmhct096(如上)被以xbal和pacl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约4.8kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。地衣芽孢杆菌天冬氨酸脱羧酶pand基因为在东方伊萨酵母中表达而被再次密码子优化(型式3,seqidno:151)并合成构建入质粒1110208质粒1110208被以xbal和pacl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约380bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将上面380bp的片段连接进7.3kbp的pmhct096线性化载体,10μl总反应体积由72.2ng消化的pmhct096,6.9ng来自1110208的380bp片段,有10mmatp的1μl10x连接缓冲液,和1μlt4连接酶构成。反应物在室温下孵育1个半小时,而3μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用nhel和ascl限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmeji311。质粒pmeji311被以nhel和ascl消化并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约7.3kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。地衣芽孢杆菌天冬氨酸脱羧酶pand基因为在东方伊萨酵母中表达而被再次密码子优化(型式4,seqidno:150)并合成构建入质粒1110207在含3μl1110207,引物0612698和0612725每种25pm,1xpfx扩增缓冲液(invitrogen),2mmmgso4,1.25个单位pfxdna聚合酶(invitrogen)的终体积50μl的混合物中进行pcr。扩增反应物在(eppendorfscientific)中孵育,程序为95℃2分钟的1个循环;每个为95℃1分钟,55℃1分钟和72℃1分钟的25个循环;以及72℃3分钟的1个循环。扩增反应的pcr产物被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约400bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。纯化的pcr产物被使用in-fusiontmadvantagepcrcloningkit(clontech)在反应中插入上面消化的pmeji311载体,反应体系包括65.1ngpmeji311nhel到ascl消化的载体片段,85ng上述pcr产物,2μl1xin-fusion反应缓冲液(clontech)和1μlin-fusiontm酶(clontech)。反应物在37℃孵育15分钟,在50℃15分钟,随后被置于冰上。2.5μl反应物样品被根据制造商的说明转化入onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在室温孵育2天。通过使用nhel和ascl的限制性消化筛选到数个有合适插的入所得转化体。产生正确消化条带大小的克隆通过dna测序来确认正确并命名为pmeji312-2(图15)。左端和右端片段的整合如本发明所述质粒pmeji310-2被以hpal和sacll消化而质粒pmeji312-2被以ecori和sacll消化。这些以tbe缓冲液中1%琼脂糖凝胶电泳来纯化,而两个约5kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。以消化的pmeji310-2和pmeji312-2dna转化东方伊萨酵母cnb1,并如本发明中所述通过crimsontaq(newenglandbiolabs)pcr来验证正确的基因座靶向和转化。引物0612794和0611245产生约3.17kbp的条带;引物612479和0611632产生约1.48kbp的条带;而引物611248和612795产生约2.3kbp的条带。给出表达盒正确整合预期条带的菌株被命名为meji409-2。随后如上所述获得meji409-2的ura-衍生体。使用本发明描述的方法,在发酵生物反应器中评价菌株mbin500和meji409-2的3-hp产生。对照菌株mbin500没有产生可检测到的3-hp(两个独立的发酵的平均)。菌株meji409-2(1个发酵)产生4.62g/l。为考虑到相比其他(如,未来)发酵,这些发酵中的细胞质量量上的差异,meji409-2每单位细胞质量的3-hp浓度(表示为[g/l3-hp]/[g/l干细胞重])被计算为0.20。实施例3a-11:在adh9091基因座表达天冬氨酸1-脱羧酶(adc)和天冬氨酸氨基转移酶(aat)的酵母菌株使用引物0611268和0611269从东方伊萨酵母基因组dna中pcr扩增编码seqidno:14的东方伊萨酵母氨基转移酶(aat)的核苷酸序列(seqidno:13)。pcr反应体系(50μl)包含50ng东方伊萨酵母菌株的基因组dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611268和0611269每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs)和1个单位的phusionhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约1278bp的pcr产物被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。所得pcr片段的总长度约1278bp,在片段5’末端有nrul限制性位点而在其3’末端有pacl限制性位点。上面得到的包含aat基因cds(seqidno:13)的1278bp片段随后被克隆进pcr2.1-topo载体并根据制造商的说明转化进one-shottop10大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过bamhi消化筛选到数个有所需片段的合适插入的所得转化体。产生所需条带大小的克隆通过测序来确认并命名为pgmer111。质粒pgmer121和pgmer111被以限制性酶pacl和nrul双消化。所得的来自质粒pgmer121的7695bp载体片段,和所得的来自质粒pgmer111的含aat编码序列的1272bp插入片段,被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,从凝胶上切下,并根据制造商的说明使用gelextractionkit(qiagen)纯化。随后以3μl载体片段,4μl插入片段,2μl无菌双蒸水,10μl2xquick连接酶缓冲液和1μlquickt4连接酶(quickligationkit,newenglandbiolabs)来建立连接反应体系并根据制造商说明进行反应。5μl等份的上述连接反应物被根据制造商说明转化入ultracompetent大肠杆菌细胞(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过smai/ppumi双消化筛选到数个有所需插入物的合适插入的所得转化体。产生所需条带大小的克隆通过测序来确认并命名为pgmer126(图16)。质粒pgmer126包括东方伊萨酵母aat表达盒,其中基因转录由东方伊萨酵母tdh3启动子和tkl终止子调控,其上游侧翼有截短的ura3编码序列3’区域和ura3启动子;下游侧翼有东方伊萨酵母adh9091基因座3’同源区域。使用引物0611661和0611662从获自的pma-t载体pcr扩增为在东方伊萨酵母中表达而密码子优化的阿维链霉菌pand基因(seqidno:130)。pcr反应体系(50μl)包含50ng菌株质粒dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611661和0611662每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs)和1个单位的phusionhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约453bp的pcr产物被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。所得pcr片段的总长度约453bp,在片段5’末端有nrul限制性位点而在3’末端有apal限制性位点。包含阿维链霉菌pand基因密码子优化型式的453bp片段被克隆进pcr2.1-topo载体并根据制造商的说明转化进one-shottop10大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过ecori消化筛选到数个有所需片段的合适插入的所得转化体。产生所需条带大小的克隆通过测序来确认并命名为pgmer127。质粒pgmer127和pgmer125(a)被以限制性酶nrul和apal消化。在终止消化反应之前将1μl小牛肠碱性磷酸酯酶(newenglandbiolabs)加入到pgmer125(a)消化体系中以将末端去磷酸化并防止自连接。所得的来自质粒pgmer125(a)(如上)的8188bp载体片段,和来自质粒pgmer127(如上)的含阿维链霉菌pand基因密码子优化型式(seqidno:130)的440bp插入片段,被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,从凝胶上切下,并根据制造商的说明使用gelextractionkit(qiagen)纯化。随后以4μl载体片段,4μl插入片段,9μl2xquick连接酶缓冲液和1μlquickt4连接酶(newenglandbiolabs)来建立连接反应体系并根据制造商说明进行反应。5μl等份的上述连接反应物被根据制造商说明转化入ultracompetent大肠杆菌细胞(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过bamhi消化筛选到数个有所需插入物的合适插入的所得转化体。产生所需条带大小的克隆被确认并命名为pgmer130(图17)。质粒pgmer130包含由下面片段制成的构建体:东方伊萨酵母adh9091基因座的5’侧翼,有东方伊萨酵母pdc启动子/tal终止子的空表达盒,东方伊萨酵母eno1启动子和rki终止子调控下的pand表达盒(含seqidno:130),和东方伊萨酵母ura3启动子调控下的截短的ura3标记基因5’片段。为确定酵母菌株东方伊萨酵母cnb1是否能表达阿维链霉菌pand基因的东方伊萨酵母密码子优化型式(seqidno:130)和东方伊萨酵母aat基因(seqidno:13),构建了表达质粒pgmer130和pgmer126。质粒pgmer130包括(从5’到3’)在东方伊萨酵母adh9091基因座构建体基因组整合用的5’侧翼区域,eno1启动子和rki终止子调控下的pand表达盒,由ura3启动子驱动的截短的ura3选择标志物5’部分。质粒pgmer126包括(从5’到3’)ura3选择标志物3’部分,tdh3启动子和tkl终止子调控下的aat基因表达盒,和在东方伊萨酵母adh9091基因座构建体基因组整合用的3’侧翼区域。所有启动子和终止子源自东方伊萨酵母。pgmer126被以限制性酶ecori消化,其切割出感兴趣的4758bp片段,而pgmer130被以限制性酶hindiii消化,构建转化所需的5034bp片段。4758bp和5034bp片段被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,从凝胶上切下,并根据制造商的说明使用gelextractionkit(qiagen)纯化。培养东方伊萨酵母cnb1并如本发明中所述以约500ng的4758bp和5034bp线性片段共转化。获得8个转化菌株并随后在摇瓶中培养。将得到的培养液用于跑sds-page,tris-hcl(bio-radlaboratories)凝胶以检测为在东方伊萨酵母中表达而密码子优化的阿维链霉菌pand基因(seqidno:130)和东方伊萨酵母aat基因(seqidno:13)的表达。阳性菌株被命名为ygmer008,而如上描述其培养液也被用于确定adc和aat活性水平。实施例3a-12:在adh9091基因座表达丙酮酸羧化酶(pyc),天冬氨酸1-脱羧酶(adc)和天冬氨酸氨基转移酶(aat)的酵母菌株使用引物0611266和0611267从东方伊萨酵母基因组dnapcr扩增编码seqidno:2的东方伊萨酵母丙酮酸脱羧酶(pyc)的核苷酸序列。pcr反应体系(50μl)包含50ng东方伊萨酵母菌株的基因组dna,1xphusionhf缓冲液(newenglandbiolabs),引物0611266和0611267每种50pmol,datp、dctp、dgtp和dttp每种200μm,1.5μl100%dmso(newenglandbiolabs)和1个单位的phusionhighfidelitydna聚合酶(newenglandbiolabs)。在(eppendorfscientific)中进行pcr,程序为98℃2分钟的1个循环,随后是每个为98℃30秒,55℃30秒和72℃1分钟30秒的35个循环,及72℃7分钟的最后延伸。热循环后,以tbe缓冲液中0.8%琼脂糖凝胶电泳来分离pcr反应产物,其中约3557bp的pcr产物被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。所得pcr片段在片段5’末端有xbal限制性位点而在其3’末端有pacl限制性位点。得到的包含东方伊萨酵母pyc基因cds(seqidno:1)的3557bp片段被克隆进pcr2.1-topo载体并根据制造商的说明转化进one-shottop10大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过ecori消化筛选到数个有所需片段的合适插入的所得转化体。产生所需条带大小的6个克隆被确认并命名为pgmer132.7、pgmer132.14、pgmer132.16、pgmerl32.25、pgmer132.27和pgmerl32.30。序列分析显示质粒pgmer132.14有合适的pyccds但丢失了cds5’端的xbal限制性位点。由于需要该限制性位点以将pyccds插入表达质粒pgmer125,质粒pgmer132.14的315bphindiii片段(包含pyccds上的5’端,有改变的xbal位点)被以来自质粒pgmer132.7的315bphindiii片段所替代,其具有包括正确xbal位点在内的未变化的pyccds5’端。所得来自pgmer132.14的7173bphindiii载体片段,和来自pgmer132.7的315bpbphindiii插入物被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,从凝胶上切下,并根据制造商的说明使用gelextractionkit(qiagen)纯化。随后以4μl载体片段,5μl插入片段,10μl2xquick连接酶缓冲液和1μlquickt4连接酶(newenglandbiolabs)来建立连接反应体系并根据制造商说明进行反应。5μl等份的上述连接反应物被根据制造商说明转化入one-shottop10大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过bamhi和xbal双消化筛选到数个有所需插入物的合适插入和取消的所得转化体。产生所需条带大小的克隆被确认并命名为pgmer133。为了将东方伊萨酵母pyccds插入到质粒pgmer125(b)中pdc启动子下游。质粒pgmer125(b)和pgmer133(如上)被以pacl和xbal消化。所得的来自质粒pgmer125(b)的8188bp载体片段,和来自质粒pgmer133的包含东方伊萨酵母pyccds(seqidno:1)的3553bp插入片段被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,从凝胶上切下,并根据制造商的说明使用gelextractionkit(qiagen)纯化。随后以3μl载体片段,6μl插入片段,10μl2xquick连接酶缓冲液和1μlquickt4连接酶(newenglandbiolabs)来建立连接反应体系并根据制造商说明进行反应。5μl等份的上述连接反应物被根据制造商说明转化入one-shottop10大肠杆菌细胞(invitrogen)大肠杆菌细胞(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过bamhi消化筛选到数个有所需片段的合适插入的所得转化体。产生所需条带大小的克隆被确认并命名为pgmer136。质粒pgmer136包括东方伊萨酵母adh9091基因座的5’侧翼,东方伊萨酵母pdc启动子和tal终止子调控下的东方伊萨酵母pyc基因表达盒(seqidno:1),有东方伊萨酵母eno1启动子/rki终止子的空表达盒,和ura3启动子调控下的截短的东方伊萨酵母ura3标志基因5’片段。约5μg的质粒pgmer136(如上)和4μg的质粒pgmer127被以限制性酶apal和nrul消化。所得来自质粒pgmer136的11729bp载体片段,和所得来自质粒pgmer127的包含为在东方伊萨酵母中表达而密码子优化的阿维链霉菌pand基因(seqidno:130)的插入片段(436bp)被根据制造商的说明使用extractii(macherey-nagel)以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来纯化。随后以5μl载体片段,4μl插入物,9μl2xquick连接酶缓冲液和1μlquickt4连接酶(newenglandbiolabs)来建立连接反应体系。反应物被在室温下孵育1小时。5μl等份的上述连接反应物被根据制造商说明转化入onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃孵育过夜。通过bamhi消化筛选到数个有所需插入物的所得转化体。产生所需条带大小的克隆被选出并命名为pgmer137(图18)。质粒pgmer137包含在东方伊萨酵母pdc启动子和tal终止子基因转录调控下的东方伊萨酵母pyc基因(seqidno:1),在东方伊萨酵母eno1启动子和rki终止子转录调控下的为在东方伊萨酵母中表达而密码子优化的阿维链霉菌pand基因(seqidno:130),随后有ura3标志5’端和东方伊萨酵母adh9091基因座5’侧翼区域的ura3启动子。质粒pgmer126(如上)被以限制性酶ecori消化,其切下感兴趣的4758bp片段;而质粒pgmer137(如上)被以限制性酶hpal和nhel消化,构建8400bp片段。4758bp和8400bp的片段被以1xtbe缓冲液中0.8%琼脂糖凝胶电泳来分离,条带被从凝胶上切下并根据制造商的说明使用extractii(macherey-nagel)来纯化。培养东方伊萨酵母cnb1并如本发明所述以约500ng4758bp和8400bp线性片段共转化,得到转化体ygmer009。实施例3a-13:在pdc基因座表达天冬氨酸1-脱羧酶(adc),β-丙氨酸氨基转移酶(baat)和3-羟基丙酸脱氢酶(3-hpdh);并在adh9091基因座表达丙酮酸羧化酶(pyc),天冬氨酸1-脱羧酶(adc)和天冬氨酸氨基转移酶(aat)的酵母菌株为了在已经过表达天冬氨酸氨基转移酶(aat),β-丙氨酸氨基转移酶(baat)和3-hp脱氢酶(3-hpdh)的菌株中增加丙酮酸羧化酶(pyc)和天冬氨酸氨基转移酶(aat)活性,如上所述以pgmer137(如上)和pgmer126(如上)线性片段转化ymhct010(如上)。在两轮单菌落纯化和生长之后,如上所述制备基因组dna来用于验证所需靶向整合发生的pcr。使用引物0611814和0612055来确认pgmer137和pgmer126片段正确靶向到adh9091基因座。引物0611814在pgmer126tdh3启动子3’端退火并在3’方向扩增,引物0612055退火于存在于pgmer126中的在adh90913’侧翼同源区的3’,所以以该引物对进行的pcr产物扩增仅在整合dna通过同源重组靶向正确位点时发生。来自含引物0611814和0612055的pcr的约3066bp条带的存在表示pgmer126和pgmer137片段在adh9091基因座发生所需整合。在以pgmer137和pgmer126线性片段转化的ymhct010数个独立转化体两轮单菌落纯化和生长之后,如上所述制备基因组dna来用于验证所需靶向整合发生的pcr。给出包含引物0611814和0612055的约3066bppcr条带的三个独立分离的菌株被命名为ymhct020,gmerin010#2,gmerin010#3。这些菌株包含在两个pdc基因座和一个adh9091基因座编码对应adc(seqidno:17)的多核苷酸(seqidno:130);在两个pdc基因座编码对应gabt(seqidno:24)的多核苷酸(seqidno:141);在两个pdc基因座编码对应3-hpdh(seqidno:129)的多核苷酸(seqidno:144);在一个adh9091基因座编码对应pyc(seqidno:2)的多核苷酸(seqidno:1);在一个adh9091基因座编码对应aat(seqidno:14)的多核苷酸(seqidno:13)(见表24)。表24:转化体基因型菌株ymhct020,gmerin010#2,和gmerin010#3在摇瓶中培养,如本发明所述制备cfe并测试pyc、aat和3-hpdh活性。实验结果在表25中显示。表25:转化体酶活性数据也如本发明中描述的以sds-page分析了表25中的菌株。mbin500和gmer009-2显示在四个其他样品中没有的~64kd蛋白条带。这些蛋白的质量与其作为东方伊萨酵母cnb1中天然丙酮酸脱羧酶的身份相符。菌株ymhct008、ymhct020、gmerin010#2和gmerin010#3显示53kd和29kd的条带。这些蛋白的质量分别与uga1和ymr226c基因所编码的蛋白的质量相符。菌株gmer009-2、ymhct020、gmerin010#2,和gmerin010#3都显示46.3kd的条带。这些蛋白的质量与aat基因所编码蛋白的质量相符。实施例3a-14:在两个1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc)的酵母菌株。使用上面描述的foa逆选择环出方案分离包含编码seqidno:139地衣芽孢杆菌adc(如上)的四个核苷酸拷贝的meji409-2ura3-衍生体。使用引物0611815和0611817通过pcr筛选亲本菌株meji409-2数个foa抗性菌落的基因组dna有所需环出事件。引物0611815在左端构建体rki终止子处退火并向ura3启动子扩增。引物0611817在tdh3启动子处退火并反向朝ura3盒扩增。828bp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.2kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。如上所述进行以crimsontaqtmdna聚合酶(newenglandbiolabs)的pcr反应。来自亲本菌株meji409-2的一个foa抗性菌落,命名为meji411,给出了所需的828bp条带。左端和右端片段的整合如本发明中所述质粒pmeji310-2被以hpal和sacll消化而质粒pmeji312-2被以ecori和sacll消化。这些被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化,两个约5kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)来纯化。以消化的pmeji310-2和pmeji312-2dna来转化meji411,并如本发明所述以crimsontaq(newenglandbiolabs)pcr来验证正确的位点靶向和转化。引物0611225和0611632产生了约5kbp的条带;引物0611815和0611632产生了约6kbp有ura标志的条带,和4.5kbp没有该标志的条带。当野生型adh1202基因座仍然完整时引物0611631和0612579得到了约936bp的条带(选择不显示该条带的菌株)。给出表达盒正确整合预期条带的菌株被命名为meji412。实施例3a-15:在adh1202基因座表达编码天冬氨酸1-脱羧酶(adc)的四个核苷酸拷贝的酵母菌株,其中有在pdc启动子的调控下编码天冬氨酸1-脱羧酶(adc)的两个核苷酸拷贝和在tdh3启动子调控下的两个拷贝。本实施例描述了构建体,其设计为要在adh1202基因座掺入编码seqidno:139地衣芽孢杆菌adc的四个核苷酸拷贝,所述拷贝有在东方伊萨酵母pdc启动子调控下的两个拷贝和在东方伊萨酵母tdh31启动子调控下的两个拷贝。以与上面描述方式类似的方式,设计左端和右端构建体以允许在东方伊萨酵母cnb1adh1202基因座的同源重组。左端片段的构建质粒pmeji310-2(如上,见图14)被以xbal和stul消化随后如本发明中所述以cip处理并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约6.7kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。通过以notl的消化,随后以klenow的补平反应和以nhel的后续消化来将pdc启动子从pmeji310-2上切下。约708bp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将708bp的纯化片段连接进6.7kbp的pmeji310-2线性化载体,10μl总反应体积由1μl来自pmeji310-2的6.7kbp片段,1或5μl来自pmeji310-2的708bp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育过夜,而4μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用apali的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba137。右端片段的构建质粒pmeji312-2(如上)被以xbal和stul消化随后如本发明中所述以cip处理并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约6.8kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。以pmel和nhel消化从pmeji312-2上切下tdh3启动子。约966bp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将966bp的纯化片段连接进6.8kbp的pmeji312-2线性化载体,10μl总反应体积由1μl来自pmeji312-2的6.8kbp片段,1或5μl来自pmeji312-2的966bp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育约6小时,而4μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用sall的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba136。左端和右端片段的整合如本发明中所述质粒pmiba137被以hpal和sacll消化而质粒pmiba136被以ecori和sacll消化。这些被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化,两个约5kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。以消化的pmiba137和pmiba136dna来转化东方伊萨酵母cnb1,并如本发明所述以crimsontaq(newenglandbiolabs)pcr来验证正确的基因座靶向和转化。引物0611717和0611631产生约2.5kbp和955bp的条带;引物0611718和0611632产生约733bp的条带;引物0612794和0611245产生约2.7kbp的条带;引物0611225和0612795产生约4.2kbp的条带。给出表达盒正确整合预期条带的菌株被命名为miba351。从miba351去除ura标志如上所述分离miba351的ura-衍生体。使用引物0611815和0611817通过pcr筛选到miba351数个foa抗性菌落的基因组dna有所需环出事件。引物0611815在左端构建体rki终止子处退火并向ura3启动子扩增,而引物0611817在tdh3启动子处退火并反向朝ura3盒扩增。828bp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.2kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。如上所述进行用crimsontaqtmdna聚合酶(newenglandbiolabs)的pcr反应。以上述引物得到828bp片段的foa抗性菌落被以产生2.7kbp产物的引物0612794和0611245以及测试4kbp产物的引物0611815和0612795进一步测试,以确认编码seqidno:139的地衣芽孢杆菌adc的核苷酸序列seqidno:138的四个拷贝保持完整。一个来自亲本菌株miba351的foa抗性菌落,命名为miba353,以所有三个引物组给出了所需的pcr产物。反向表达盒右端片段的构建质粒pmiba136包含两个朝正向取向的表达盒。为简化同源菌株的筛选,构建了新的质粒,其中pmiba136的pandbl表达盒被置于反向取向。质粒pmiba136(如上)被以notl和pmel消化,如本发明中所述随后碱性用klenow的补平反应,并以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约3.4kbp和4.4kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。来自pmiba136的的4.4kbp片段被以cip处理并根据制造商的说明使用pcrpurificationkit(qiagen)来纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pmiba136的3.4kbp的纯化片段连接进4.4kbp的pmiba136载体,10μl总反应体积由1μl来自pmiba136的4.4kbp片段,1或5μl来自pmiba136的3.4kbp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育过夜,而4μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用pacll和ecori的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba138。左端和右端片段的整合如本发明中所述质粒pmiba137被以hpal和sacll消化而质粒pmiba138被以ecori和sacll消化。这些被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化,两个约5kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。以消化的pmiba137和pmiba138dna来转化miba353,并使用plantdirectpcrkit(finnzymes)来通过pcr来验证正确的基因座靶向和转化。引物0611718和0611632产生约733bp的条带(以确认第一整合仍然存在);引物0612367和0611632产生约960bp的条带(以确认整合的第二拷贝);当野生型adh1202基因座仍然存在时引物0611631和06112579产生约936bp的条带(缺少该条带确认野生型adh1202基因座的丧失)。给出表达盒正确整合预期条带的两个菌株被保存并被命名为miba355和miba356。meji409-2,meji412、miba351和miba355中天冬氨酸1-脱羧酶活性菌株meji409-2,meji412、miba351和miba355被在摇瓶中培养,如本发明中所述制备cfe并测试天冬氨酸1-脱羧酶(adc)活性。结果显示于表26中。每个菌株的活性为两个独立摇瓶培养物的平均值。也使用本发明描述的方法,在生物反应器中测试菌株meji409-2、meji412、miba351和miba355的3-hp产生。这些生物反应器实验的结果也显示于表26中。考虑到这些发酵中的细胞质量的差异,表26中显示的3-hp产生性能被表示为每单位细胞质量的3-hp浓度(表示为g/l3-hp/g/l干细胞重)。结果显示随着细胞中adc活性水平的增加,3-hp产生性能增加。表26:转化体adc活性和3-hp产生数据实施例3a-16:在pdc基因座表达丙酮酸羧化酶(pyc)的质粒构建如下所述构建用于在pdc基因座整合的,含seqidno:1核苷酸序列(编码seqidno:2的东方伊萨酵母pyc)的质粒pann28。根据制造商的说明使用基因组dna作为模板来pcr扩增东方伊萨酵母pdc的上游和下游侧翼区域(pfu聚合酶stratagene)。引物oann7和oann8允许掺入上游区域侧翼独特的限制位点而引物oann9和oann10允许掺入下游区域侧翼独特的限制位点。如本发明中所述,pcr产物被以tbe缓冲液中1%琼脂糖凝胶电泳纯化。每个pcr产物约800bp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。纯化的pcr产物被克隆进topo载体(invitrogen)并根据制造商的说明转化进电感受态大肠杆菌dh10b细胞(invitrogen)。使用与用于构建pcr产物的引物相同的引物通过菌落pcr筛选到中的数个有合适插入的所得转化体。产生正确pdc下游侧翼的克隆被命名为pann04。产生正确pdc上游侧翼的克隆被命名为pann07。质粒pann04被以apal和sacl消化(用作载体/骨架);质粒pann04被以notl和sacl消化;质粒pann07被以notl和apal消化。如本发明中所述,每个片段被以tbe缓冲液中1%琼脂糖凝胶电泳纯化。对载体约3.5kbp的条带,和对每个插入物约1kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将纯化产物连接,10μl总反应体积由49ng载体,120ng下游插入物,41ng上游插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而2μl等份的反应物被根据制造商的说明转化进电感受态大肠杆菌oneshottop10细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物oann7和oann8(产生约1.7kbp的片段)通过菌落pcr筛选到数个有合适插入的所得转化体。产生正确插入的克隆被命名为pann12。来自pgmer137(如上)的东方伊萨酵母pyc编码序列(seqidno:1)被以位点定向诱变修饰,以消除不改变所编码酶氨基酸序列的三个ecori限制性位点。质粒pgmer137被用作引物oann13、oann14和oann15的模板,根据制造商的说明来使用multichangekit(stratagene),使用这些引物消除上述限制性位点。通过使用ecori的限制性消化筛选到数个所得转化体。阳性克隆通过测序进一步确认。得到正确pyc编码序列的克隆被命名为pann14。质粒pjy39(图29)被以xhol和pacl消化;质粒pacn5(如上,见图19)被以xhol和xbal消化;质粒pann14被以xball和pacl消化。如本发明中所述,每个片段被以tbe缓冲液中1%琼脂糖凝胶电泳纯化。对载体约8kbp的条带,对第一插入物约700bp的条带和对编码pyc的第二插入物约3.6kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将纯化产物连接,10μl总反应体积由51ng载体,49ng第一插入物,210ng第二插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而2μl等份的反应物被根据制造商的说明转化进电感受态大肠杆菌oneshottop10细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物ojlj57和ojlj43(产生约1kbp的片段),引物ojlj45和oann16(产生约730bp的片段),和引物oann20和ojy45(产生约1.2kbp的片段)通过菌落pcr筛选到数个有合适插入的所得转化体。得到正确插入的克隆被命名为pann15。质粒pann12和pann15被以notl消化。质粒pann15被另外地以ncol消化以将骨架进一步片段化并改进所需片段的分离。消化的pann12被根据制造商的说明使用qiagen试剂盒纯化。如本发明中所述notl片段以tbe缓冲液中的琼脂糖凝胶电泳纯化。约5kbp的片段(来自pann12)和约6.3kbp的片段(来自pann15)被根据制造商的说明使用gelextractionkit(qiagen)来凝胶纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pann15的纯化产物连接进pann12线性化载体,10μl总反应体积由50ng载体,115ng插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育1.5小时,而2μl等份的反应物被用于根据制造商的说明转化电感受态大肠杆菌oneshottop10细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物oann20和ojy45(产生约1.2kbp的片段)通过菌落pcr筛选到数个有合适插入的所得转化体。产生正确插入的克隆被通过以sacl/ecori和以sacl/ecorv的限制性酶消化来进一步筛选以区分插入取向。产生上游pdc侧翼附近ura3标志的克隆被命名为pann27。产生下游pdc侧翼附近ura3标志的克隆被命名为pann28。实施例3a-17:在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc)和在pdc基因座表达丙酮酸羧化酶(pyc)的酵母菌株本实施例描述了酵母菌株的构建,该酵母菌株在adh1202基因座表达编码seqidno:139地衣芽孢杆菌adc的四个核苷酸拷贝并在pdc基因座表达编码seqidno:2东方伊萨酵母pyc的核苷酸。从meji412去除ura标志如上所述分离meji412的ura-衍生体。使用引物0611815和0611817通过菌落pcr筛选到meji412数个foa抗性菌落有所需环出事件。引物0611815在左端构建体rki终止子处退火并向ura3启动子扩增,而引物0611817在tdh3启动子处退火并反向朝ura3盒扩增。869bp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.6kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。如上所述进行用plantdirectpcrkit(finnzymes)的pcr反应。以上述引物产生869bp片段的foa抗性菌落被以产生3.8kbp产物的引物0612794和0611817以及产生3.7kbp产物的引物0611815和0612795进一步测试,以确认编码seqidno:139地衣芽孢杆菌adc的核苷酸序列seqidno:138四个拷贝保持完整。一个来自亲本菌株meji412的foa抗性菌落,命名为meji413,以所有三个引物组给出了所需的pcr产物。片段的整合质粒pann28(如上)被以ascl和sacl消化并通过tbe缓冲液中的琼脂糖凝胶电泳纯化。约7.1kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。以来自菌株pann28的消化并纯化片段来转化菌株meji413,并根据制造商说明通过菌落pcr(failsafe,mixe,epicenter)来验证正确的基因座靶向和转化。引物oann12和ojlj44产生约1kbp的条带;引物oann11和oann16产生约1.3kbp的条带。给出表达盒正确整合预期条带的菌株被命名为yann35。随后如上所述分离yann35的ura-衍生体。使用引物oann12和ojy44通过菌落pcr筛选到数个foa抗性菌落有所需环出事件。引物oann12在下游侧翼区域外退火。引物ojy44在tal终止子处退火。1.5kbp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.8kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。如上所述进行用failsafedna聚合酶(epicenter)的pcr反应。对该事件阳性的分离株被进一步使用引物oann16和oann11通过菌落pcr来确认。一个foa抗性菌落被命名为yann37。以来自菌株pann28的消化并纯化片段来转化菌株yann37,并根据制造商说明通过菌落pcr(failsafe,mixe,epicenter)来验证正确的基因座靶向和转化。使用对pdc基因特异的引物ohjj116和ohjj117进行初步筛选。约500bp的条带表示基因的存在并因此是所需整合的阴性结果。对pdc缺失阳性的分离株被以另外的pcr反应进一步确认。引物oann11和oann16产生约1.3kbp的条带;引物oann12和ojy44ojlj44产生约1kbp的条带;引物oann12和ojy44产生约1.5kbp的条带和约2.9kbp的条带(分别对应第一个第二整合事件)。此外,如上所述通过菌落pcr来确认之前在adh1202基因座处的整合事件。引物0611631和0611245产生约3.8kbp的条带。引物0611245和onovo3产生约3kbp的条带。引物0611815和0612795产生约3.6kbp的条带。给出表达盒正确整合预期条带的菌株被命名为yann41。实施例3a-18:在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc)和在pdc基因座表达丙酮酸羧化酶(pyc)的酵母菌株本实施例描述了酵母菌株的构建,该酵母菌株在adh1202基因座表达编码seqidno:139地衣芽孢杆菌adc的四个核苷酸拷贝(有两个在pdc启动子调控下的拷贝和两个在tdh3启动子调控下的拷贝)并在pdc基因座表达编码seqidno:2东方伊萨酵母pyc的核苷酸。从miba355去除ura标志如上所述分离miba355的ura-衍生体。使用引物0611815和0611718通过pcr筛选到miba355数个foa抗性菌落的基因组dna有所需环出事件。约500bp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约1.9kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。根据制造商的说明使用plantdirectpcrkit(finnzymes)进行pcr。以上述引物产生约500bp片段的foa抗性菌落被以产生3.5kbp产物的引物0611631和0611245以及产生4.5kbp产物的引物0611815和0611632进一步测试,以确认编码seqidno:139地衣芽孢杆菌adc的核苷酸序列seqidno:138四个拷贝保持完整。其也被使用引物0611815和0611817以pcr测试,以确认adh1202处第一修饰的存在。这些pcr引物产生828bp的片段。一个来自亲本菌株miba355的foa抗性菌落,命名为miba357,以所有四个引物组给出了所需的pcr产物。质粒pann28(如上)被以ascl和sacl消化并以tbe缓冲液中的1%琼脂糖凝胶电泳纯化。约7.1kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。miba357被以消化的pann28dna转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。引物0611622和0611552产生约850bp条带;引物0611245和0612794产生约2.8kbp的条带;引物0611815和0612795产生约3.9kbp的条带。给出表达盒正确整合预期条带的菌株被命名为mcts241。随后如前所述分离mcts241的ura-衍生体。使用引物0614233和0611554通过pcr筛选到mcts241数个foa抗性菌落有所需的环出事件,并在减除了ura的平板上缺乏生长。约4.6kbp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约5.9kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。如上所述进行使用phireplantdirectpcrkit(finnzymes)的pcr反应。一个来自亲本菌株mcts241的,具有所需环出事件的foa抗性菌落被命名为mcts247。为构建纯合整合体,mcts247被以消化的pann28dna转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。作为第一次筛选,使用引物0611552和0611553通过pcr来筛选转化体,其仅当pdc基因座完整并因此pdc基因座处pyc的纯合重组没有发生时应产生约850bp的条带。对该pcr的条带阴性的那些随后被使用引物0611555和0611554通过另外pcr来筛选。使用这些引物,当pdc完整并因此pdc基因座处无pyc的纯合重组时产物应仅扩增1.4kbp条带。使用产生约850bp条带的引物0611622和0611552;产生约2.8kbp条带的引物0611245和0612794;产生约3.9kbp条带的引物0611815和0612795通过pcr进行转化体的进一步筛选。给出表达盒正确整合预期条带的菌株被命名为mcts253。实施例3a-19:菌株meji412、yann35、yann41、miba355、mcts241和mcts253的pyc活性、adc活性和3-hp产生性能。菌株meji412、yann35、yann41、miba355、mcts241和mcts253被在摇瓶中生长,如本发明中所述制备cfe并测试丙酮酸羧化酶(pyc)活性和天冬氨酸1-脱羧酶(adc)活性。结果显示于表27中。也使用本发明描述的方法,在生物反应器中测试菌株meji412、yann35、yann41、miba355、mcts241和mcts253的3-hp产生。这些生物反应器实验的结果也显示于表27中。考虑到这些发酵中的细胞质量的差异,显示的3-hp产生性能被表示为每单位细胞质量的3-hp浓度(表示为g/l3-hp/g/l干细胞重)。结果显示随着细胞中pyc活性水平的增加,3-hp产生性能增加。表27:转化体pyc和adc活性和3-hp产生性能实施例3a-20:在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc)并缺失了β-丙氨酸氨基转移酶(baat)的酵母菌株本实施例描述了酵母菌株的构建和性能,所述酵母菌株在adh1202基因座表达编码seqidno:139地衣芽孢杆菌adc的四个核苷酸拷贝并缺失了编码seqidno:20的baat(pyd4)的天然东方伊萨酵母基因。东方伊萨酵母baat(pyd4)缺失质粒的构建质粒pmiba123(如上)被以notl、kpnl、apal消化并如本发明中所述通过tbe缓冲液中1%的琼脂糖凝胶电泳来纯化。约3.6kbp和3.8kbp的两个条带被被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。这两片段包含质粒骨架和有seqidno:144的酿酒酵母3-hpdh基因(ymr226c)的ura选择盒以引物0613178和0613180使用东方伊萨酵母mbin500基因组dna通过pcr扩增生成上游东方伊萨酵母pyd4同源区的pcr产物。使用引物0613179和0613181通过pcr扩增从mbin500基因组dna制备下游东方伊萨酵母pyd4同源区片段。每种引物50pmol被用于含0.5μl作为模板的mbin500基因组dna,datp、dctp、dgtp和dttp每种0.2mm,1xexpandhighfidelitybuffer(roche),3.5uexpandhighfidelityenzymemix(roche)的终体积50μl的pcr反应中。在5333(eppendorf)中进行扩增反应,程序为95℃3分钟的1个循环;和每个为95℃30秒,55℃30秒和72℃1分钟的30个循环。循环后,反应物被在72℃孵育5分钟并随后在10℃冷却直至进一步处理。使用引物0613178和0613180的约800bp的pcr条带,以及使用引物0613179和0613181的约900bp的pcr条带被以tbe缓冲液中1%的琼脂糖凝胶电泳纯化。条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。pyd4上游pcr产物,pyd4下游pcr产物,和pmiba123notl/kpnl/apal消化的质粒被根据制造商说明在用in-fusionhdtm(clontechlabboratories,inc.)的反应中组装。来自in-fusion反应的2μl被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化和测序来分析。有正确序列的质粒通过测序验证并命名为pmcts61。质粒pmcts61仍然含pdc启动子,来自酿酒酵母的ymr226c基因,和pdc终止子。为去除这些不需要的片段,pmcts61被以ecori和xhol消化随后加入klenow片段以构建平末端。7.1kbp的片段被以tbe缓冲液中1%的琼脂糖凝胶电泳纯化。条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs,ipswich,ma,usa)将消化的平末端质粒连接在一起。反应混合物在总体积20μl中含1xt4连接酶缓冲液,1μlt4dna连接酶,5μlpmcts61消化和平末端化的纯化dna。反应物在室温下孵育2小时。10μl连接反应样品被用于根据制造商的说明转化onetop10化学感受态大肠杆菌细胞(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆。通过菌落pcr分析克隆。通过将1个菌落溶解于50μl无菌水中,在95℃加热10分钟,随后在冰上冷却直至使用来制备每个菌落的模板dna。引物0612911和0612909被用于筛选转化体。如果质粒正确,以这些引物的pcr反应会扩增出1kbp的条带。每种引物10pmol被用于含2μl菌落dna模板,datp、dctp、dgtp和dttp每种0.1mm,1xcrimsontaq反应缓冲液(newenglandbiolabs),1ucrimsontaqdna聚合酶(newenglandbiolabs)的终体积20μl的pcr反应中。在5333(eppendorf)中进行扩增反应,程序为95℃3分钟的1个循环;和每个为95℃30秒,55℃30秒和72℃3分钟的30个循环。循环后,反应物被在72℃孵育5分钟并随后在10℃冷却直至进一步处理。以tae缓冲液中的溴化乙啶来在1%tae-琼脂糖凝胶中观察来自5μlpcr反应物的1kbp片段。有正确大小pcr产物的一个转化体被选出并命名为pmcts64(图30)。使用9600(qiagen)制备pmcts64的质粒dna。使用pmcts64构建体从meji413缺失天然东方伊萨酵母baat(pyd4)质粒pmcts64(如上,见图30)被以apal、ncol、kpnl消化并通过tbe缓冲液中1%的琼脂糖凝胶电泳来纯化。约3.3kbp的条带被被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。菌株meji413(如上)被以消化的pmcts64dna转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。引物0612908和0613242产生约1.7kbp条带;引物0613241和0612909产生约1.5kbp的条带以确认缺失盒的整合;引物0611815和0611632产生约4.2kbp的条带;引物0611817和0611631产生约4.8kbp的条带以确认adh1202基因座的adc表达盒仍然完整。给出缺失盒和adc盒正确整合预期条带的菌株被命名为mcts225。随后如前所述分离mcts225的ura-衍生体。使用引物0612911和0612910通过pcr筛选到mcts225数个foa抗性菌落有所需的环出事件。约1.1kbp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.5kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。引物0611815和0611632产生约4.2kbp的条带;引物0611817和0611631产生约4.8kbp的条带以确认adh1202基因座的adc表达盒仍然完整。如上所述进行使用phireplantdirectpcrkit(finnzymes)的pcr反应。一个来自亲本菌株mcts225的,具有所需环出事件的foa抗性菌落被命名为mcts228。为构建编码seqidno:20的东方伊萨酵母baat(pyd4)的天然基因的纯合缺失体,mcts228被以消化的pmcts64转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。两组引物被用于通过pcr筛选pyd4位点缺失。仅当pyd4位点完整时,引物0613550和0612910产生约700bp的条带,表明pyd4纯合缺失没有发生。此外转化体被以在pyd4没有被缺失时产生约600bp条带的引物0612911和0613551来筛选。对东方伊萨酵母pyd4基因座阴性的转化体被以产生约3.5kbp和2.1kbp条带的引物0613242和0613243,产生约1.7kbp条带的引物0612908和0613243;产生约950bp条带的引物0612909和0612911来进一步筛选。以产生约4.8kbp条带的引物0611817和0611631以及产生约4.2kbp条带的引物611815和612712来确认adh1202处adc盒仍然完整。给出表达盒正确整合预期条带的菌株被命名为mcts236。随后如前所述分离mcts236的ura-衍生体。使用产生约2.1kbp条带的引物0613242和0613243通过pcr筛选到mcts236数个foa抗性菌落有所需的环出事件。使用产生约3kbp条带的引物0611245和0612794以及产生约3.6kbp条带的引物0611815和0612795来确认adh1202基因座的adc盒仍然完整。给出表达盒正确整合预期条带的菌株被命名为mcts245。使用本发明描述的方法,在生物反应器中测试菌株miba372和mcts245的3-hp产生。考虑到这些发酵中的细胞质量的差异,3-hp产生性能被表示为每单位细胞质量的3-hp浓度(表示为g/l3-hp/g/l干细胞重)。菌株miba372和mcts245的g/l3-hp/g/l干细胞重分别为1.66和0.16。这些结果表明由于缺失该基因导致3-hp产生性能降低至10分之1,因此是东方伊萨酵母中的天然pyd4基因负责将β-丙氨酸转化成丙二酸半醛。实施例3a-21在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc)并缺失了3-hp脱氢酶(3-hpdh)的酵母菌株本实施例描述了酵母菌株的构建和性能,所述酵母菌株在adh1202基因座表达编码seqidno:139地衣芽孢杆菌adc的四个核苷酸拷贝并缺失了编码seqidno:26的3-hpdh的天然东方伊萨酵母基因。东方伊萨酵母3-hpdh缺失质粒的构建质粒pmiba123(如上)被以notl、kpnl、apal消化并如本发明中所述通过tbe缓冲液中1%的琼脂糖凝胶电泳来纯化。约3.6kbp和3.8kbp的两个条带被被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。这两片段包含质粒骨架和有seqidno:144酿酒酵母3-hpdh基因的ura选择盒。使用引物0613183和0613184从如前所述制备的东方伊萨酵母mbin500基因组dna扩增上游东方伊萨酵母3-hpdh同源区的pcr产物。使用引物0613185和0613186从mbin500基因组dna扩增下游东方伊萨酵母3-hpdh同源区片段。每种引物50pmol被用于含0.5μl作为模板的mbin500基因组dna,datp、dgtp、dctp、和dttp每种0.2mm,2%dmso,1xphusionhf缓冲液(finnzymes),2uhotstarthighfidelitydna聚合酶(finnzymes)的终体积50μl的pcr反应中。在5333(eppendorfscientificinc.,westbury,ny,usa)中进行扩增反应,程序为95℃3分钟的1个循环;和每个为95℃30秒,55℃30秒和72℃1分钟的30个循环。循环后,反应物被在72℃孵育5分钟并随后在4℃冷却直至进一步处理。引物0613183和0613184的约640bp的pcr条带,以及引物0613185和0613186的约670bp的pcr条带被以tbe缓冲液中1%的琼脂糖凝胶电泳纯化。条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。东方伊萨酵母3-hpdh上游pcr产物,东方伊萨酵母3-hpdh下游pcr产物,和pmiba123notl/kpnl/apal消化的质粒被根据制造商说明在用in-fusionhdtm(clontechlabboratories,inc.)的反应中组装。来自in-fusion反应的2μl被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化和测序来分析。有正确序列的质粒通过测序验证并命名为pmcts60。质粒pmcts60仍然含pdc启动子,来自酿酒酵母的ymr226c基因,和pdc终止子。为去除这些不需要的片段,pmcts60被以notl和xbal消化,含3-hpdh同源区和质粒骨架片段的约5kbp条带被以tbe缓冲液中1%的琼脂糖凝胶电泳纯化。条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。以两个pcr反应,一个用引物0613416和0613417另一个用引物0613418和0613419来扩增ura3选择盒。每种引物50pmol被用于含0.5μl作为模板的pmcts60质粒dna,datp、dgtp、dctp和dttp每种0.2mm,1xexpandhighfidelitybuffer(roche),3.5uexpandhighfidelityenzymemix(roche)的终体积50μl的pcr反应中。在5333(eppendorfscientificinc.)中进行扩增反应,程序为95℃3分钟的1个循环;和每个为95℃30秒,55℃30秒和72℃1分钟的30个循环。循环后,反应物被在72℃孵育5分钟并随后在4℃冷却直至进一步处理。引物0613416和0613417的约700bp的pcr条带,以及引物0613418和0613419的1kbp的pcr条带被以tbe缓冲液中1%的琼脂糖凝胶电泳纯化。条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。含东方伊萨酵母3-hpdh同源区的质粒骨架和ura3盒pcr产物被根据制造商说明在用in-fusionhdtm(clontechlabboratories,inc.)的反应中组装。来自in-fusion反应的2μl被根据制造商的说明转化进solopackgoldsupercompetentcells(stratagene)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化来分析,有正确限制性消化方式的质粒被命名为pmcts65(图31)。使用pmcts65构建体从meji413缺失天然东方伊萨酵母3-hpdh质粒pmcts65(如上,见图31)被以apal、sph、kpnl消化并通过tbe缓冲液中1%的琼脂糖凝胶电泳来纯化。约2.9kbp的条带被被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。菌株meji413(如上)被以消化的pmcts65dna转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。引物0613034和0613035产生约2.7kbp条带以确认缺失盒的整合;引物0611815和0611632产生约4.2kbp的条带;引物0611817和0611631产生约4.8kbp的条带以确认adh1202基因座的adc表达盒仍然完整。给出缺失盒和adc盒正确整合预期条带的菌株被命名为mcts229。随后如前所述分离mcts229的ura-衍生体。使用引物0613034和0613241通过pcr筛选到mcts229数个foa抗性菌落有所需的环出事件。约1.4kbp条带的存在表示如所需的仅ura3残留位点的存在(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约2.8kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需的重组事件没有发生。约1.9kbp条带的存在表示存在于这些转化体中的野生型基因盒,因为这些是缺失的杂合。引物0611815和0611632产生约4.2kbp的条带;引物0611631和0612366产生约4.5kbp的条带以确认adh1202基因座的adc表达盒仍然完整。如上所述进行使用phireplantdirectpcrkit(finnzymes)的pcr反应。一个来自亲本菌株mcts225的,具有所需环出事件的foa抗性菌落被命名为mcts238。为构建编码seqidno:26的东方伊萨酵母3-hpdh的天然基因的纯合缺失体,mcts238被以消化的pmcts65转化,并使用phireplantdirectpcrkit(finnzymes)通过pcr验证正确的基因座靶向和转化。两组引物被用于通过pcr筛选ymr226c基因座缺失。当3-hpdh位点完整时引物0613034和0613747产生约500bp的条带以表明3-hpdh纯合缺失没有发生。此外转化体被以引物0613746和0613241来筛选,其在3-hpdh没有被缺失时产生约660bp的条带。对东方伊萨酵母3-hpdh基因座野生型阴性的转化体被以产生约2.8kbp和1.4kbp条带的引物0613034和0613241,产生约1.5kbp条带的引物0612908和0613241;产生约1kbp条带的引物0613034和0612909来进一步筛选。以产生约3kbp条带的引物0611245和0612794以及产生约3.6kbp条带的引物0611815和0612795来确认adh1202基因座处adc盒仍然完整。有表达盒正确整合预期条带的菌株被命名为mcts244。随后如前所述分离mcts244的ura-衍生体。使用产生约1.4kbp条带的引物0613034和0613241通过pcr筛选到mcts244数个foa抗性菌落有所需的环出事件。使用产生约3kbp条带的引物0611245和0612794以及产生约3.6kbp条带的引物0611815和0612795来确认adh1202基因座的adc表达盒仍然完整。一个来自亲本菌株mcts244的,具有所需环出事件的foa抗性菌落被命名为mcts259。使用本发明描述的方法,在生物反应器中测试菌株miba372和mcts244的3-hp产生。考虑到这些发酵中的细胞质量的差异,3-hp产生性能被表示为每单位细胞质量的3-hp浓度(表示为g/l3-hp/g/l干细胞重)。菌株miba372和mcts259的g/l3-hp/g/l干细胞重分别为1.66和<0.1。这些结果表明由于缺失该基因消除了3-hp产生,因此是东方伊萨酵母中的天然3-hpdh基因负责将丙二酸半醛转化为3-hp。实施例3a-22:在pdc基因座表达丙酮酸羧化酶(pyc),天冬氨酸氨基转移酶(aat),β-丙氨酸氨基转移酶(baat),和3-羟基丙酸脱氢酶(3-hpdh);并在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱氢酶(adc)的酵母菌株设计另外的构建体以在菌株中东方伊萨酵母pdc基因座掺入编码seqidno:2的东方伊萨酵母pyc的核苷酸序列seqidno:1,编码seqidno:14的东方伊萨酵母att的核苷酸序列seqidno:13,编码seqidno:21的克鲁维酵母baat的核苷酸序列seqidno:142,和编码seqidno:129的酿酒酵母3-hpdh的核苷酸序列seqidno:144,菌株也在adh1202基因座包含编码seqidno:139的地衣芽孢杆菌adc的四个核苷酸拷贝,以与上面描述的方式类似的方式,设计左端和右端构建体以允许在东方伊萨酵母cnb1pdc基因座的同源重组。如下所述,这些构建体被制备并转化进miba357。左端片段的构建使用质粒pgmer126(图16)作为模板按制造商的说明通过pcr扩增(pfu聚合酶,stratagene)编码seqidno:14的东方伊萨酵母att的核苷酸序列seqidno:13。引物oann1和oann2允许掺入基因编码序列侧翼独特的限制性位点。pcr产物被以tbe缓冲液中1%琼脂糖凝胶电泳纯化。约1.3kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。纯化的pcr产物被以apal和nrul消化并如本发明中所述被凝胶纯化。质粒pgmer135(与上面pgmer136相同,除了eno1启动子/rki终止子插入物是相反取向)被以apal和nrul消化并如本发明中所述以tbe缓冲液中琼脂糖凝胶电泳纯化。约11.7kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将纯化的1.3kbppcr产物连接进11.7kbp的pgmer135线性化载体,10μl总反应体积由49.8ng载体,354ng1.3kbp插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而2μl等份的反应物被根据制造商的说明转化进电感受态大肠杆菌dh10b细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物ohj2和oann1(产生约2.3kbp条带)以及引物oann5和oann6(产生约877bp条带)通过菌落pcr筛选到数个有合适插入的所得转化体。pcr扩增aat片段的序列也被确认。产生正确插入和序列的克隆以apal和nrul消化并如本发明中所述被凝胶纯化。约1.3kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。包含编码seqidno:2的东方伊萨酵母pyc的所需核苷酸序列seqidno:1的质粒pgmer137(如上,见图18)被以apal和nrul消化并如本发明中所述以tbe缓冲液中琼脂糖凝胶电泳纯化。约11.7kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将纯化的1.3kbppcr产物连接进11.7kbp的pgmer137线性化载体,10μl总反应体积由49.4ng载体,54ng1.3kbp插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而2μl等份的反应物被用于根据制造商的说明转化进电感受态大肠杆菌dh10b细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物ohj2和oann1(产生约2.3kbp条带)以及引物oann5和oann6(产生约877bp条带)通过菌落pcr筛选到数个有合适插入所得转化体。产生正确插入和序列的克隆被命名为pann02。具有相反朝向aat编码序列的左端片段的构建质粒pann02被以pmel消化并如本发明中所述以tbe缓冲液中琼脂糖凝胶电泳纯化。约10.3kbp的条带和约2.7kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。来自pann2的10.3kbp载体片段被以cip(newenglandbiolabs)去磷酸化并根据制造商的说明使用纯化试剂盒(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pann02的2.7kbp片段连接进来自pann02的去磷酸化10.3kbp线性化载体,10μl总反应体积由36ng载体,28ng插入物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而2μl等份的反应物被根据制造商的说明转化进电感受态大肠杆菌top10细胞(invitrogen)。转化体被涂布于lb+卡那霉素平板上并在37℃下孵育过夜。使用引物ojy44和ohj1(产生约1.3kbp条带)通过菌落pcr筛选到数个有合适插入的所得转化体。产生正确插入的克隆被命名为pann5。右端片段的构建如下构建含两个地衣芽孢杆菌adc编码区和东方伊萨酵母pdc基因座3’靶向侧翼dna的右端构建体。质粒pmhct071(与上述pmhct077相同的质粒,除了酿酒酵母3-hpdhorf没有为东方伊萨酵母密码子优化)被以pmel和pacl消化,以10个单位的小牛肠磷酸酶(newenglandbiolabs)处理,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化,约4.7kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒pmeji312-2(如上,见图15)被以pmel和pacl消化以提取两个地衣芽孢杆菌adc表达盒,并以tae缓冲液中0.9%琼脂糖凝胶电泳纯化,约2.8kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。在含1xquick连接缓冲液(newenglandbiolabs),1μl4.7kbppmhct071载体片段,3μl来自pmeji312-2的2.8kbp插入物,和1μlquickt4dna连接酶(newenglandbiolabs)的连接体系中(20μl),将来自pmeji312-2的含两个地衣芽孢杆菌adc编码区的片段连接进线性化的pmhct071载体片段。连接反应物在室温孵育5分钟,随后将管置于冰上。5μl反应物根据制造商的说明被用于转化solopackgoldsupercompetentcells(agilenttechnologies)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过apali消化筛选到数个有所需片段的正确连接的所得转化体。产生所需片段大小的克隆被保存并命名为pmhct110。质粒pmhct110被以xbal和pacl消化,随后如本发明所述以cip处理并以tbe缓冲液中1%琼脂糖凝胶电泳纯化。约7.1kbp的片段被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。seqidno:144的密码子优化酿酒酵母3-hpdh编码序列被通过以xbal和pacl消化从pmiba123(如上)上切下并如本发明所述以tbe缓冲液中1%琼脂糖凝胶电泳纯化。约814bp的片段被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将814bp的纯化片段连接进来自pmhct110的7.1kbp片段,10μl总反应体积由1μl消化的pmhct110,1或7μl来自pmiba123的814bp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育30分钟,而4μl等份的反应物被用于根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在室温下孵育过周末。通过使用xbal和pacl以及ascl和pacl的2个酶组合的限制性消化筛选到数个有合适插入的所得转化体。从每个消化中产生正确消化条带大小的克隆被命名为pmiba142。质粒pmiba142被以ascl消化,随后进行用klenow的补平反应和以nhel的随后消化以及cip处理。如本发明所述消化物以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约7.5kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。编码seqidno:21的克鲁维酵母baat的核苷酸序列seqidno:142通过以pacl的消化被从pmiba124(如上)上切下,随后进行用klenow的补平反应和以xbal的随后消化。如本发明所述消化物以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约1.4kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pmiba124的1.4kbp纯化片段连接进来自pmiba142的7.5kbp片段,10μl总反应体积由1μl消化的pmiba142,7μl来自pmiba124的1.4kbp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育2.5小时,而整个连接物被根据制造商的说明转化进sure细胞(agilent)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用stul和pmel的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba144。左端和右端片段整合进miba357如本发明中所述质粒pann5被以notl和nhel消化而质粒pmiba144被以notl消化。这些被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。来自pann5约8.2kbp片段和来自pmiba144的约6kbp片段被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。以消化的pann5和pmiba144dna来转化miba357,并根据制造商说明使用plantdirectpcrkit(finnzymes)来通过pcr来验证正确的基因座靶向和转化。引物0611552和0613695产生约4.1kbp的条带(以确认pdc基因座处左半边整合);引物0612358和0611554产生约2.5kbp的条带(以确认pdc基因座处右半边整合);引物0611245和0611631产生约3.5kbp的条带(以确认左半边4xadc整合仍然在adh1202基因座);而引物0611815和0611632产生约4.6kbp的条带(以确认右半边4xadc整合仍然在adh1202基因座)。给出pdc基因座表达盒正确整合预期条带并在adh1202基因座保留表达盒的一个分离株被保存并命名为miba360。从miba360去除ura标志如上所描述分离miba360的ura-衍生体。使用引物0611815和0613689通过pcr筛选到来自miba360数个foa抗性菌落的基因组dna有所需环出事件。约1.9kbp条带的存在表示如所需地去除了ura标志而有ura3残留位点(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约3.3kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需重组事件没有发生。按照制造商的说明使用plantdirectpcrkit(finnzymes)进行pcr。以上述引物产生约1.9kbp片段的两个foa抗性菌落被保存并命名为miba363和miba364。反向表达盒右端片段的构建质粒pmiba144包含所需朝正向取向前进的adc和3-hpdh表达盒。为简化同源菌株的筛选,构建了新的质粒,其中pmiba144的adc表达盒被置于反向取向。质粒pmiba144(如上)被以stul和pmel消化,如本发明中所述以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。约6.1kbp和2.8kbp的条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)来纯化。来自pmiba144的的6.1kbp片段被以cip处理并根据制造商的说明使用pcrpurificationkit(qiagen)来纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pmiba144的2.8kbp的纯化片段连接进6.1kbp的pmiba144线性化载体,10μl总反应体积由1μl来自pmiba144的6.1kbp片段,7μl来自pmiba144的2.8kbp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育4小时,而4μl等份的反应物被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用sphl和xbal的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba146。将左端和右端片段整合进miba363如本发明中所述质粒pann5被以notl和nhel消化而质粒pmiba146被以notl消化。这些被以tbe缓冲液中1%琼脂糖凝胶电泳来纯化。来自pann5约8.2kbp片段和来自pmiba146的约6.2kbp片段被从凝胶上切下并根据制造商的说明使用pcrpurificationkit(qiagen)纯化。以消化的pann5和pmiba144dna来转化miba363,并根据制造商说明使用plantdirectpcrkit(finnzymes)或kaparobustdna聚合酶来通过pcr来验证正确的基因座靶向和转化。为确认在pdc基因座的整合,使用了下列引物对。引物0613689和0611815产生约1.9kbp的条带;引物0612366和0611554产生约2.5kbp的条带;引物0613688和0611815产生约3.2kbp的条带;引物0611622和0611552产生约945bp的条带。为检查在adh1202基因座的整合,使用了下列引物对。引物0611245和0612794产生约2.8kbp的条带;引物0611815和0612795产生约3.9kbp的条带。给出在pdc基因座表达盒正确整合预期条带并在adh1202基因座保留表达盒的2个分离株被保存并命名为miba372和miba373。从miba372去除ura标志物如上所述分离miba372的ura-衍生体。使用引物0611815和0613688通过pcr筛选到来自miba372数个foa抗性菌落的基因组dna有所需环出事件。约2.1kbp条带的存在表示如需要地去除了ura标志而有ura3残留位点(在亲本菌株中两个ura3启动子间同源重组后留下的单个ura3启动子),而约3.3kbp条带表示完整ura3启动子-ura3orf-ura3终止子-ura3启动子盒的存在,表明所需重组事件没有发生。也使用确认第一染色体修饰的引物0611815和0613689(扩增1.9kbp片段)和确认pdc基因座丢失的0611552和0611553(如果pdc基因座存在扩增850bp片段)来通过pcr筛选miba372的foa抗性菌落。按照制造商的说明使用plantdirectpcrkit(finnzymes)进行pcr。以0611815和0613689产生约1.9kbp片段,但以0613688和0611815或者0611552和0611553不扩增片段的foa抗性菌落被保存并命名为miba375。miba375的基因型显示于表28中。表28:转化体的基因型实施例3a-23:缺失了甘油3-磷酸脱氢酶(gpd)基因的酵母菌株。菌株miba375中gpd基因的缺失设计另外的构建体以缺失来自宿主东方伊萨酵母基因组的甘油3-磷酸脱氢酶基因(seqidno:117,其编码seqidno:118的gpd)的两个拷贝,这些构建体包含与gpd基因上游序列同源的约1003bp核苷酸序列,和与gpd基因下游序列同源的约852bp序列,有克隆在之间的tpdc-ura3标志盒(pdc终止子-ura3启动子-ura3基因-ura3终止子-ura3启动子)。根据制造商的说明书使用pfudna聚合酶从东方伊萨酵母cnb1基因组dna扩增gpd的上游和下游区域。上游区域包含apal位点;使用重叠引物通过pcr消除该位点,所述引物设计有对apal识别序列中一个核苷酸的错配。引物对oacn48/oacn51以及oacn49/oacn50被用于扩增对上游区域的这两个重叠片段。这些pcr产物被在1%琼脂糖凝胶中分离并从中切下,并根据制造商的说明使用qiaquickgelextractionkit(qiagen)纯化,并被用作以引物oacn48/oacn49(具有正向apal/反向notl位点)的第二轮pcr的模板。根据制造商的说明书使用pfudna聚合酶以引物oacn52/oacn53(具有正向notl/反向sacl位点)从东方伊萨酵母基因组dna扩增下游区域。两个pcr产物被凝胶纯化并分别克隆进载体(invitrogen)。分离株通过质粒dna的限制性消化被确认具有所需插入物,并通过测序验证。载体pacn58包含克隆的上游片段而pacn59包含下游片段。质粒pacn58被以apal/notl消化以释放上游侧翼,质粒pacn59被以sacl/notl消化以释放下游侧翼,而pacn59被以apal/sacl消化以提供载体骨架。这三个所需的片段被在1%琼脂糖凝胶中分离,切下和纯化,并在使用t4连接酶(newenglandbiolabs)的3-片段连接反应中连接。连接反应物被转化进大肠杆菌top10电感受态细胞,并通过质粒dna的限制性消化来确认转化体。在该过程中,检测到下游区域中另外的sacl位点,其产生853bp的下游区域(相对于1kbp)。有所需插入物的两个分离株被命名为pacn62和pacn63。使用notl消化从载体pjlj8(图32)分离tpdc1-ura3盒并凝胶纯化。该片段随后被连接进已经被以notl消化并去磷酸化的pacn62(如上),连接物被转化进大肠杆菌dh10b电感受态细胞(invitrogen)。使用引物oacn48/ojlj44和oacn48/ojlj46通过pcr确认有ura3插入物的菌落。引物ojlj44在ura3启动子下游末端退火并从tpdc1-ura3盒向外扩增。引物ojlj46在pdc终止子5‘末端退火并从tpdc1-ura3盒向外扩增。载体phjj56包含面向下游gpd区域的ura3而phjj57包含面向上游gpd区域的ura3。通过以kpnl和apal消化来将phjj56和phjj57线性化,而含有缺失盒的片段被通过凝胶提取纯化。线性化的phjj56被转化进ura-菌株miba375。单菌落被再划线以纯化,并使用引物ojlj44,ojlj46,oacn54和oacn55通过pcr测试所需的gpd缺失。在40μly-裂解缓冲液和2μlzymolyase(zymoresearch)中37℃裂解细胞30分钟,而1μl裂解反应物被用于25μlpcr反应。根据制造商的说明书pcr反应使用failsafedna聚合酶和buffere,有55℃的退火温度和下列循环过程:94℃2分钟的1个循环;每个94℃30秒,55℃30秒,和72℃1.5分钟的29个循环;以及72℃3分钟的1个循环。有一个gpd拷贝敲除的菌株产生约0.9和1.2kbp的条带并被命名为yhjn1和yhjn2。菌株yhjn1和yhjn2在ypd培养基中培养过夜并被置于scd-2xfoa培养基上以选择ura3标志的缺失。单菌落被在ypd上纯化并贴到scd-ura和ypd培养基上以确认ura-表型。使用与用来确认第一整合的pcr反应相同的反应来确认ura-菌落已经保留了敲除。yhjn1的ura-衍生体被命名为yhjn3而yhjn2的ura-衍生体被命名为yhjn4。线性化的phjj57被转化进yhjn3和yhjn4,而单菌落被在scd-ura培养基上纯化。在一个反应中使用引物ojlj44,oacn54和oacn55,在第二反应中使用引物ojlj46,oacn54和oacn55通过pcr确认两个gpd拷贝敲除的存在。引物oacn54在gpd上游侧翼序列上游约37bp区域上退火,而oacn55退火至下游侧翼下游约24bp区域。如果两个gpd拷贝被缺失则前面的反应产生约900和1050bp的条带,而后面的反应产生约1025和1200bp的条带。在scd-ura平板上,有两个gpd拷贝敲除的菌落比有单个拷贝缺失的那些生长更慢。有两个gpd基因拷贝缺失的菌株被命名为yhjn7(源自yhjn3)和yhjn8(源自yhjn4)。使用本发明中描述的方法,在生物反应器中测试菌株miba372,yhjn7和yhjn8的甘油生产。菌株miba372在48小时中产生29.5g/l甘油。发酵期间菌株yhjn7或yhjn8没有产生可检测的甘油。最终发酵液中甘油的缺失可以为从发酵液中回收和纯化3-hp提供优势。实施例3a-24:在adh1202基因座从四个核苷酸序列表达天冬氨酸1-脱羧酶(adc),在adh9091基因座表达3-hp脱氢酶(3-hpdh)并缺失了天然3-hp脱氢酶(3-hpdh)的酵母菌株在adh9091基因座整合东方伊萨酵母3-hpdh的质粒构建使用引物0611954和0611957(对3-hpdh)或0611997和0611998(pyd4)从如前所述制备的mbin500东方伊萨酵母基因组dna扩增编码seqidno:26的东方伊萨酵母3-hpdh的seqidno:25以及编码seqidno:20的东方伊萨酵母baat(pyd4)的seqidno:19核苷酸序列。扩增期间引物0611954向3-hpdh5’末端加入kozak序列(taaa)和nhel位点,而引物0611957向3-hpdh3’端加入pacl位点。扩增期间引物0611997向pyd45’末端加入kozak序列(taaa)和pacl位点,而引物0611998向pyd4的3’端加入pacl位点。每种引物50pmol被用于含50ng作为模板的mbin500基因组dna,datp、dgtp、dctp和dttp每种0.2mm,1xexpandhighfidelitybuffer(roche),2.6单位expandhighfidelitydna聚合酶(roche)的终体积50μl的pcr反应中。在(eppendorfscientific)中进行扩增反应,程序为95℃3分钟的1个循环;随后每个为95℃1分钟,55℃1分钟和72℃1分钟(3-hpdhpcr)或2分钟(pyd4pcr)的30个循环,及72℃5分钟的最终延伸。热循环后,pcr反应产物被以tae缓冲液中1.0%的琼脂糖凝胶电泳分离,其中约831bp的3-hpdh或1.4kbp的pyd4pcr产物被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。5μl纯化的3-hpdh或pyd4被根据制造商的说明使用topo-tacloningkit克隆进pcr2.1(invitrogen)。转化物被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过ecori消化筛选到数个所得转化体。产生所需插入大小的克隆被通过dna测序来确认正确。一个含3-hpdh的克隆被命名为pmbin190,而另一个含pyd4的被命名为pmbin193。质粒pmbin193被以xbal和pacl消化并在tae缓冲液中1.0%的琼脂糖凝胶上跑电泳,其中约1.4kbppyd4条带被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。消化的pyd4片段被使用t4dna连接酶连接进xbal和pacl限制性消化的线性pmiba107质粒(如上)。连接产物被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过snabi或ecori消化筛选到数个的所得转化体。产生产生所需条带大小的一个克隆被命名为pmbin203。质粒pmbin203含在下列表达盒侧翼的notl位点:pyd4cds上游或下游的pdc启动子和终止子,ura3启动子,ura3orf,和随后有ura3启动子的ura3终止子。质粒pmbin203被以notl消化并在tae缓冲液中1.0%的琼脂糖凝胶上分离,其中约4.1kbp的片段(含pdc启动子,pyd4cds,pdc终止子,和ura3标志选择标志物)被从凝胶上切下并根据制造商的说明使用gelextractionkit(qiagen)纯化。质粒phjj27(含与adh9091基因座同源的5’和3’区域;见图21)被以notl消化,如上所述以cip处理并在tae缓冲液中1%琼脂糖凝胶上分离,其中约5.7kbp的线性质粒被纯化。如上所述来自pmbin203的片段随后被使用t4dna连接酶连接进notl限制性消化的phjj27。连接产物被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过pstl消化筛选到数个所得转化体。产生产生所需条带大小的一个克隆被命名为pmbin204。质粒pmbin204允许pyd4靶向到adh9091基因座。如本发明中所述,通过nhel和pacl消化编码seqidno:26的东方伊萨酵母3-hpdh的seqidno:25核苷酸序列被从质粒pmbin190(如上)去除,并通过tbe缓冲液中琼脂糖凝胶电泳纯化。约827bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。质粒pmbin204(如上)被以xbal和pacl消化并如本发明中所述通过tbe缓冲液中琼脂糖凝胶电泳纯化。约8.4kbp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在20μl总反应体积中将上面纯化的约827bp东方伊萨酵母3-hpdh基因产物连接进8.4kbp的pmbin204线性化载体,20μl总反应体积由1μl8.4kbp载体,10μl827bp插入物,有10mmatp的2μl10x连接缓冲液(newenglandbiolabs)和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃孵育18小时,10μl等份的反应物被根据制造商的说明转化进oneshottop10细胞(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化来分析,有正确限制性消化方式的质粒被命名为pmcts90。在adh9091基因座整合酿酒酵母3-hpdh的质粒构建编码seqidno:129的酿酒酵母3-hpdh的826bp野生型核苷酸序列被从jgi69基因组dnapcr扩增,并在基因5’端加上xbal位点和在基因3’端加上pacl位点。扩增反应按照制造商的说明使用pfxdna聚合酶(invitrogen)进行。masterpcr反应含1.125μl酿酒酵母基因组dna,引物611191和611199每种112.5pm,1xpfx扩增缓冲液(invitrogen),2mmmgso4,0.2mmdntp混合物,5个单位的pfxdna聚合酶(invitrogen),终体积200μl。混合物被等分到8个管中并进行梯度pcr。扩增反应物在(eppendorfscientificinc.)中孵育,程序为95℃2分钟的1个循环;每个为95℃30秒,40-55℃梯度30秒,和72℃2分钟的30个循环;和72℃3分钟的1个循环。如本发明所述826bp野生型ymr226cpcr基因产物被通过tbe缓冲液中1%的琼脂糖凝胶电泳纯化。约826bp片段被从凝胶上切下并使用gelextractionkit(qiagen)从凝胶中提取。pcr产物被以xbal和pacl在37℃消化过夜并随后使用pcrpurificationkit(qiagen)纯化。质粒pmiba100(如上)被以xbal和pacl消化随后以cip处理,得到约6.8kbp的线性片段。消化物按照制造商的说明使用pcrpurificationkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将826bpymr226c纯化并消化的pcr片段连接进6.8kbppmiba100线性化载体,10μl总反应体积由1μl来自pmiba100的6.7kbp片段,1或7μl826bpymr226cpcr产物,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育约4小时,而整个反应物被根据制造商的说明转化进sure化学感受态细胞(agilent)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用xbal和pacl的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小和dna序列的克隆被命名为pmiba101。质粒phjj76-noura(如上)被以notl消化随后以cip处理。线性5.2kbp片段使用pcrpurificationkit(qiagen)纯化。ymr226c表达盒通过以notl消化被从pmiba101上切下。约3546bp的条带被从凝胶上切下并按照制造商的说明使用pcrpurificationkit(qiagen)纯化。使用t4连接酶(newenglandbiolabs)在10μl总反应体积中将来自pmiba101的3546bp纯化片段连接进5.2kbpphjj76-noura线性化载体,10μl总反应体积由1μl来自phjj76-noura的5.2kbp片段,1或5μl来自pmiba101的3546bp片段,1μl有10mmatp的10x连接缓冲液(newenglandbiolabs),和1μlt4连接酶(newenglandbiolabs)构成。反应物在16℃下孵育过夜,而约4μl等份的反应物被根据制造商的说明转化进onetop10化学感受态大肠杆菌细胞(invitrogen)。转化体被涂布于2xyt+amp平板上并在37℃下孵育过夜。通过使用xbal和knpl的限制性消化筛选到数个有合适插入的所得转化体。产生正确消化条带大小的克隆被命名为pmiba109。编码seqidno:129的酿酒酵母3-hpdh的野生型核苷酸序列被通过xbal和pacl消化被从质粒pmiba109中去除,并如本发明所述在tbe缓冲液中通过琼脂糖凝胶电泳纯化。约818bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在20μl总反应体积中将纯化的约818bp酿酒酵母3-hpdh基因产物连接进上面8.4kbp的pmbin204线性化载体,20μl总反应体积由1μl8.4kbp载体,10μl818bp插入物,2μl有10mmatp的10x连接缓冲液(newenglandbiolabs)和1μlt4连接酶(newenglandbiolabs)构成成。反应物在16℃孵育18小时,10μl等份的反应物被根据制造商的说明转化进oneshottop10细胞(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化来分析,有正确限制性消化方式的质粒被命名为pmcts91。在adh9091基因座整合m.sedula3-hpdh的质粒构建编码seqidno:29的m.sedula3-hpdh的seqidno:343东方伊萨酵母密码子优化核苷酸序列由合成,得到质粒11aae2ap。合成的基因被以xbal和pacl从质粒中消化出来并如本发明所述通过tbe缓冲液中的琼脂糖凝胶电泳纯化。约959bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在20μl总反应体积中将纯化的约956bpm.sedula3-hpdh基因产物连接进上面8.4kbp的pmbin204线性化载体,20μl总反应体积由1μl8.4kbp载体,16μl956bp插入物,2μl有10mmatp的10x连接缓冲液(newenglandbiolabs)和1μlt4连接酶(newenglandbiolabs)构成。反应物在室温下孵育1小时,10μl等份的反应物被根据制造商的说明转化进solopackgoldsupercompetentcells(invitrogen)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化来分析,有正确限制性消化方式的质粒被命名为pmcts76。在adh9091基因座整合大肠杆菌3-hpdh的质粒构建编码seqidno:27的大肠杆菌3-hpdh的seqidno:143东方伊萨酵母密码子优化核苷酸序列由合成,得到质粒1045168。合成的基因被以xbal和pacl从质粒中消化出来并如本发明所述通过tbe中的琼脂糖凝胶电泳纯化。约761bp的条带被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。使用t4连接酶(newenglandbiolabs)在20μl总反应体积中将纯化的约761bp大肠杆菌3-hpdh基因产物连接进上面8.4kbp的pmbin204线性化载体,20μl总反应体积由1μl8.4kbp载体,16μl761bp插入物,2μl有10mmatp的10x连接缓冲液(newenglandbiolabs)和1μlt4连接酶(newenglandbiolabs)组成。反应物在室温下孵育1小时,10μl等份的反应物被根据制造商的说明转化进solopackgoldsupercompetentcells(invitrogenagilent)。在恢复期后,来自转化反应的两份100μl等份被涂布于添加有每ml100μg氨苄青霉素的150mm2xyt平板上。平板在37℃孵育过夜。从选择平板上选出推定的重组克隆并使用9600(qiagen)从每一个制备质粒dna。克隆通过限制性消化来分析,有正确限制性消化方式的质粒被命名为pmcts77。在mcts259中在adh9091整合3-hpdh片段如本发明中所述质粒pmcts76、pmcts77、pmcts91被以kpnl和apal消化,而质粒pmcts90被以sacl和apal消化。得到的消化产物被通过tbe缓冲液中1%琼脂糖凝胶电泳纯化,而来自pmcts76的5.5kbp片段,来自pmcts77的5.3kbp片段,来自pmcts90的5.4kbp片段和来自pmcts91的5.4kbp片段被从凝胶上切下并根据制造商的说明使用extractiikit(macherey-nagel)纯化。在adh1202基因座表达编码seqidno:139的地衣芽孢杆菌adc的四个核苷酸拷贝并缺失了编码seqidno:26的3-hpdh(如上)的天然东方伊萨酵母基因的菌株mcts259,被以消化的pmcts76、pmcts77、pmcts90或pmcts91dna转化。按照制造商的说明使用plantdirectpcrkit(finnzymes)来通过pcr来验证正确的基因座靶向和转化。为确认在adh9091基因座的整合,使用下列引物对。引物0614627和0612909对来自pmcts76的整合片段产生约3.47kbp条带,对来自pmcts77的整合片段产生约3.27kbp条带,对来自pmcts90的整合片段产生约3.34kbp条带,对来自pmcts91的整合片段产生约3.33kbp条带;引物0612908和0614626产生约1.97kbp条带。为检查在adh1202的整合,使用了下列引物对。引物0611245和0612794产生约3.0kbp条带而引物0611815和0612795产生约3.6kbp的条带。为检查天然东方伊萨酵母3-hpdh基因的缺失,使用了下列引物。引物0613034和0613241产生约1.4kbp条带。如表29中所示,给出在adh9091基因座表达盒正确整合预期条带,在adh1202基因座保留表达盒并保留东方伊萨酵母3-hpdh位点缺失的分离株被保存并命名为mcts261(pmcts76片段),mcts263(pmcts77片段),mcts267(pmcts90片段)和mcts269(pmcts91片段)。表29:转化体构建使用上述摇瓶方法测试转化菌株的3-hp产生。杂合转化体mcts267、mcts269和mcts263分别产生0.149(+/-0.024),0.168(+/-0.052),0.162(+/-0.018)g/l3-hp每g/l干细胞重。天然菌株meji412产生0.263(+/-0.026)g/l3-hp每g/l干细胞重,而3-hpdh缺失菌株不产生可检测的3-hp。本试验中杂合转化体mcts261不产生可检测的3-hp。这些结果显示使用外源或内源3-hpdh基因序列,甚至杂合的3-hpdh转化体也可以恢复一些3-hpdh缺失菌株的3-hpdh活性。实施例3b:表达苹果酸途径基因的修饰的酵母菌株通过利用pep、oaa和苹果酸中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个ppc,苹果酸脱氢酶和苹果酸脱羧酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3c:表达丙二酸半醛途径基因的修饰的酵母菌株通过利用pep、oaa和丙二酸半醛中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可包括一个或多个ppc,2-酮酸脱羧酶,kgd,bcka,吲哚丙酮酸脱羧酶,3-hpdh(包括丙二酸半醛还原酶),hibadh和4-羟基丁酸脱氢酶基因。表达的基因可源自对宿主细胞是天然的基因,或者其可源自对宿主细胞非天然的来源基因。实施例3d:表达丙二酰coa途径基因的修饰的酵母菌株通过利用pep、oaa,丙二酰coa,和可选的,丙二酸半醛中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个ppc,oaa甲酸裂解酶,丙二酰coa还原酶,coa酰化丙二酸半醛脱氢酶,3-hpdh(包括丙二酸半醛还原酶),hibadh和4-羟基丁酸脱氢酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3e:表达丙二酰coa途径基因的修饰的酵母菌株通过利用丙酮酸,乙酰coa,丙二酰coa,和可选的,丙二酸半醛中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个pdh,乙酰coa羧化酶,丙二酰coa还原酶,coa酰化丙二酸半醛脱氢酶,3-hpdh(包括丙二酸半醛还原酶),hibadh和4-羟基丁酸脱氢酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3f:表达丙氨酸途径基因的修饰的酵母菌株通过利用丙酮酸,丙氨酸,β-丙氨酸,和可选的,丙二酸半醛,β-丙氨酰coa,丙烯酰coa以及3-hp-coa中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个丙氨酸脱氢酶,丙酮酸/丙氨酸氨基转移酶,丙氨酸2,3氨基变位酶,coa转移酶,coa合成酶,β-丙氨酰coa氨解酶,3-hp-coa脱水酶,3-hp-coa水解酶,3-羟异丁酰coa水解酶,baat,3-hpdh(包括丙二酸半醛还原酶),hibadh和4-羟基丁酸脱氢酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3g:表达乳酸途径基因的修饰的酵母菌株通过利用丙酮酸,乳酸,乳酰coa,丙烯酰coa和3-hp-coa中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个ldh,coa转移酶,coa合成酶,乳酰coa脱水酶,3-hp-coa脱水酶,3-hp-coa水解酶和3-羟异丁酰coa水解酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3h:表达甘油途径基因的修饰的酵母菌株通过利用甘油和3-hpa中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个甘油脱水酶和醛脱氢酶基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。实施例3i:表达β-丙氨酰coa途径基因的修饰的酵母菌株通过利用pep或丙酮酸,β-丙氨酸,β-丙氨酰coa,丙烯酰coa,3-hp-coa和可选的oaa,天冬氨酸和丙氨酸中间体的途径来产生3-hp的酵母细胞被工程改造以表达一个或多个涉及该途径中的酶。表达的基因可以包括一个或多个ppc,pyc,aat,adc,coa转移酶,coa合成酶,β-丙氨酰coa氨解酶,3-hp-coa脱水酶,3-hp-coa水解酶,3-羟异丁酰coa水解酶,丙氨酸脱氢酶,丙酮酸/丙氨酸氨基转移酶和aam基因。表达的基因可以源自对宿主细胞是天然的基因,或者其可以源自对宿主细胞非天然的来源基因。在某些方面中,本发明使用的酵母细胞或方法可由下面编号的段落描述:[b1]包含活性3-hp发酵途径的经遗传修饰的酵母细胞,其中细胞包含选自:外源ppc基因;外源pyc基因;外源aat基因;外源adc基因;外源baat或gabt基因;和外源3-hpdh基因的一种或多种外源3-hp途径基因。[b2]段落b1的经遗传修饰的酵母细胞,其包含外源aat基因。[b3]段落b1或b2的经遗传修饰的酵母细胞,其包含外源pyc基因。[b4]段落b1-b3任一项的经遗传修饰的酵母细胞,其包含外源adc基因。[b5]段落b1-b4任一项的经遗传修饰的酵母细胞,其包含外源baat或gabt基因。[b6]段落b1-b5任一项的经遗传修饰的酵母细胞,其包含外源3-hpdh基因。[b7]段落b1的经遗传修饰的酵母细胞,其包含:外源pyc基因;外源aat基因;外源adc基因;外源baat或gabt基因;和外源3-hpdh基因。[b8]段落b1-b7任一项的经遗传修饰的酵母细胞,其包含外源ppc基因。[b9]段落b1-b7任一项的经遗传修饰的酵母细胞,其中外源pyc基因编码与选自seqidno:2、3、4、5、6、7和8的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b10]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与选自seqidno:2的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b11]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:3的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b12]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:4的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b13]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:5的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b14]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:6的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b15]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:7的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b16]段落b9的经遗传修饰的酵母细胞,其中外源pyc基因编码与seqidno:8的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b17]段落b1-b16任一项的经遗传修饰的酵母细胞,其中外源pyc基因包含与seqidno:1的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b18]段落b1-b17任一项的经遗传修饰的酵母细胞,其中aat基因编码与选自seqidno:14、15和16的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b19]段落b18的经遗传修饰的酵母细胞,其中aat基因编码与seqidno:14的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b20]段落b18的经遗传修饰的酵母细胞,其中aat基因编码与seqidno:15的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b21]段落b18的经遗传修饰的酵母细胞,其中aat基因编码与seqidno:16的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b22]段落b1-b21任一项的经遗传修饰的酵母细胞,其中aat基因包含与seqidno:13的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b23]段落b1-b22任一项的经遗传修饰的酵母细胞,其中adc基因编码与选自seqidno:17、18、133、135、137和139的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b24]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:17的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b25]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:18的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b26]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:133的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b27]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:135的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b28]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:137的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b29]段落b23的经遗传修饰的酵母细胞,其中adc基因编码与seqidno:139的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b30]段落b1-b29任一项的经遗传修饰的酵母细胞,其中adc基因包含与选自seqidno:130、131、132、134、136和138的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b31]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:130的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b32]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:131的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b33]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:132的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b34]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:134的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b35]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:136的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b36]段落b30的经遗传修饰的酵母细胞,其中adc基因包含与seqidno:138的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b37]段落b1-b36任一项的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与选自seqidno:20、21、22、23和24的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b38]段落b37的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与seqidno:20的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b39]段落b37的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与seqidno:21的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b40]段落b37的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与seqidno:22的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b41]段落b37的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与seqidno:23的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b42]段落b37的经遗传修饰的酵母细胞,其中外源baat或gabt基因编码与seqidno:24的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b43]段落b1-b42任一项的经遗传修饰的酵母细胞,其中baat或gabt基因包含与选自seqidno:19、140、141和142的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b44]段落b43的经遗传修饰的酵母细胞,其中baat基因或gabt基因包含与seqidno:19的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b45]段落b43的经遗传修饰的酵母细胞,其中baat基因或gabt基因包含与seqidno:140的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b46]段落b43的经遗传修饰的酵母细胞,其中baat基因或gabt基因包含与seqidno:141的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b47]段落b43的经遗传修饰的酵母细胞,其中baat基因或gabt基因包含与seqidno:142的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b48]段落b1-b47任一项的经遗传修饰的酵母细胞,其中所述外源baat基因或外源gabt为也是gabt基因的baat基因。[b49]段落b1-b48任一项的经遗传修饰的酵母细胞,其中3-hpdh基因编码与选自由seqidno:26、27、28、29、30、31、32、33、34和129组成的组的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b50]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:26的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b51]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:27的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b52]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:28的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b53]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:29的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b54]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:30的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b55]段落的b49经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:31的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b56]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:32的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b57]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:33的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b58]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:34的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b59]段落b49的经遗传修饰的酵母细胞,其中3-hpdh基因编码与seqidno:129的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b60]段落b1-b59任一项的经遗传修饰的酵母细胞,其中3-hpdh基因包含与选自seqidno:25、143、144和343的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b61]段落b60的经遗传修饰的酵母细胞,其中3-hpdh基因包含与seqidno:25的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b62]段落b60的经遗传修饰的酵母细胞,其中3-hpdh基因包含与seqidno:143的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b63]段落b60的经遗传修饰的酵母细胞,其中3-hpdh基因包含与seqidno:144的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b64]段落b60的经遗传修饰的酵母细胞,其中3-hpdh基因包含与seqidno:343的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b65]段落b1-b64任一项的经遗传修饰的酵母细胞,其中3-hpdh基因也是hibadh基因。[b66]段落b1-b65任一项的经遗传修饰的酵母细胞,其中3-hpdh基因也是4-羟基丁酸脱氢酶基因。[b67]段落b1-b66任一项的经遗传修饰的酵母细胞,其中ppc基因编码与选自seqidno:10、11和12的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b68]段落b67的经遗传修饰的酵母细胞,其中ppc基因编码与seqidno:10的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b69]段落b67的经遗传修饰的酵母细胞,其中ppc基因编码与seqidno:11的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b70]段落b67的经遗传修饰的酵母细胞,其中ppc基因编码与seqidno:12的氨基酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的多肽。[b71]段落b1-b70任一项的经遗传修饰的酵母细胞,其中ppc基因包含与seqidno:9的核苷酸序列具有至少60%,如至少65%、70%、75%、80%、85%、90%、95%、97%、98%、99%或100%序列同一性的核苷酸序列。[b72]段落b1-b71任一项的经遗传修饰的酵母细胞,其中所述酵母细胞是crabtree阴性的。[b73]段落b1-b72任一项的经遗传修饰的酵母细胞,其中酵母细胞属于选自伊萨酵母、假丝酵母、克鲁维酵母属、毕赤酵母、裂殖酵母、有孢圆酵母属、接合酵母属和酵母属的属。[b74]段落b73的经遗传修饰的酵母细胞,其中酵母细胞属于选自东方伊萨酵母/发酵毕赤酵母进化枝和酵母属进化枝的进化枝。[b75]段落b73的经遗传修饰的酵母细胞,其中酵母细胞选自东方伊萨酵母、郎比可假丝酵母和博伊丁酵母。[b76]段落b1-b75任一项的经遗传修饰的酵母细胞,其中所述细胞进一步包含选自pdc、adh、gal6、cyb2a、cyb2b、gpd、gpp、ald和pck基因的天然基因的一个或多个缺失或破坏。[b77]段落b76的经遗传修饰的酵母细胞,其中一个或多个缺失或破坏得自一个或多个外源3-hp途径基因的插入。[b78]段落b1-b77任一项的经遗传修饰的酵母细胞,其中一个或多个外源3-hp途径基因可操作连接到一个或多个外源调节元件上。[b79]段落b78的经遗传修饰的酵母细胞,其中一个或多个调节元件对于一个或多个3-hp途径基因是外来的。[b80]段落b1-b79任一项的经遗传修饰的酵母细胞,其中外源pyc基因可操作连接到对于pyc基因是外来的外源启动子上。[b81]段落b1-b80任一项的经遗传修饰的酵母细胞,其中外源att基因可操作连接到对于att基因是外来的外源启动子上。[b82]段落b1-b81任一项的经遗传修饰的酵母细胞,其中外源adc基因可操作连接到对于adc基因是外来的外源启动子上。[b83]段落b1-b82任一项的经遗传修饰的酵母细胞,其中外源baat或gabt基因可操作连接到对于baat或gabt基因是外来的外源启动子上。[b84]段落b1-b83任一项的经遗传修饰的酵母细胞,其中外源3-hpdh基因可操作连接到对于3-hpdh基因是外来的外源启动子上。[b85]段落b1-b84任一项的经遗传修饰的酵母细胞,其中外源ppc基因可操作连接到对于ppc基因是外来的外源启动子上。[b86]段落b1-b85任一项的经遗传修饰的酵母细胞,其中细胞能够在含75g/l或更多3-hp的培养基中在小于4的ph下生长。[b87]段落b1-b86任一项的经遗传修饰的酵母细胞,其中细胞是3-hp抗性酵母细胞。[b88]段落b1-b87任一项的经遗传修饰的酵母细胞,其中细胞已经经过了突变和/或选择,以致经突变和/或选择的细胞相比同种的野生型细胞具有更高程度的3-hp抗性。[b89]段落b88的经遗传修饰的酵母细胞,其中细胞在用一个或多个外源3-hp途径基因进行遗传修饰之前已经经过了突变和/或选择。[b90]段落b88或b89的经遗传修饰的酵母细胞,其中细胞已经经过了乳酸或3-hp存在下的选择。[b91]段落b91的经遗传修饰的酵母细胞,其中选择是恒化选择。[b92]生产3-hp的方法,包括:(i)在含至少一种碳源的培养基的存在下培养段落b1-b91任一项的经遗传修饰的酵母细胞;和(ii)从培养物中分离3-hp。[b93]段落b92的方法,其中所述碳源选自葡萄糖、木糖、阿拉伯糖、蔗糖、果糖、纤维素、葡萄糖寡聚物和甘油。[b94]段落b92或b93的方法,其中培养基的ph小于5,如在约1.5至约4.5、约2.0至约4.0或约2.0至约3.5的范围中。序列表<110>卡吉尔公司(cargill,incorporated)jessen,hollyrush,brianhuryta,jeanettemastel,bethberry,alanyaver,debbie<120>用于生产3-羟基丙酸的组合物和方法<130>n00096-cn-pcd<150>us61/416,199<151>2010-11-22<150>us61/535,181<151>2011-09-15<160>351<170>patentinversion3.5<210>1<211>3543<212>dna<213>东方伊萨酵母<400>1atgtcaactgtggaagatcactcctccctacataaattgagaaaggaatctgagattctt60tccaatgcaaacaaaatcttagtggctaatagaggtgaaattccaattagaattttcagg120tcagcccatgaattgtcaatgcatactgtggcgatctattcccatgaagatcggttgtcc180atgcataggttgaaggccgacgaggcttatgcaatcggtaagactggtcaatattcgcca240gttcaagcttatctacaaattgacgaaattatcaaaatagcaaaggaacatgatgtttcc300atgatccatccaggttatggtttcttatctgaaaactccgaattcgcaaagaaggttgaa360gaatccggtatgatttgggttgggcctcctgctgaagttattgattctgttggtgacaag420gtttctgcaagaaatttggcaattaaatgtgacgttcctgttgttcctggtaccgatggt480ccaattgaagacattgaacaggctaaacagtttgtggaacaatatggttatcctgtcatt540ataaaggctgcatttggtggtggtggtagaggtatgagagttgttagagaaggtgatgat600atagttgatgctttccaaagagcgtcatctgaagcaaagtctgcctttggtaatggtact660tgttttattgaaagatttttggataagccaaaacatattgaggttcaattattggctgat720aattatggtaacacaatccatctctttgaaagagattgttctgttcaaagaagacatcaa780aaggttgttgaaattgcacctgccaaaactttacctgttgaagttagaaatgctatatta840aaggatgctgtaacgttagctaaaaccgctaactatagaaatgctggtactgcagaattt900ttagttgattcccaaaacagacattattttattgaaattaatccaagaattcaagttgaa960catacaattactgaagaaatcacgggtgttgatattgttgccgctcaaattcaaattgct1020gcaggtgcatcattggaacaattgggtctattacaaaacaaaattacaactagaggtttt1080gcaattcaatgtagaattacaaccgaggatcctgctaagaattttgccccagatacaggt1140aaaattgaggtttatagatctgcaggtggtaacggtgtcagattagatggtggtaatggg1200tttgccggtgctgttatatctcctcattatgactcgatgttggttaaatgttcaacatct1260ggttctaactatgaaattgccagaagaaagatgattagagctttagttgaatttagaatc1320agaggtgtcaagaccaatattcctttcttattggcattgctaactcatccagttttcatt1380tcgggtgattgttggacaacttttattgatgataccccttcgttattcgaaatggtttct1440tcaaagaatagagcccaaaaattattggcatatattggtgacttgtgtgtcaatggttct1500tcaattaaaggtcaaattggtttccctaaattgaacaaggaagcagaaatcccagatttg1560ttggatccaaatgatgaggttattgatgtttctaaaccttctaccaatggtctaagaccg1620tatctattaaagtatggaccagatgcgttttccaaaaaagttcgtgaattcgatggttgt1680atgattatggataccacctggagagatgcacatcaatcattattggctacaagagttaga1740actattgatttactgagaattgctccaacgactagtcatgccttacaaaatgcatttgca1800ttagaatgttggggtggcgcaacatttgatgttgcgatgaggttcctctatgaagatcct1860tgggagagattaagacaacttagaaaggcagttccaaatattcctttccaaatgttattg1920agaggtgctaatggtgttgcttattcgtcattacctgataatgcaattgatcattttgtt1980aagcaagcaaaggataatggtgttgatattttcagagtctttgatgctttgaacgatttg2040gaacaattgaaggttggtgttgatgctgtcaagaaagccggaggtgttgttgaagctaca2100gtttgttactcaggtgatatgttaattccaggtaaaaagtataacttggattattattta2160gagactgttggaaagattgtggaaatgggtacccatattttaggtattaaggatatggct2220ggcacgttaaagccaaaggctgctaagttgttgattggctcgatcagatcaaaataccct2280gacttggttatccatgtccatacccatgactctgctggtaccggtatttcaacttatgtt2340gcatgcgcattggcaggtgccgacattgtcgattgtgcaatcaattcgatgtctggttta2400acctctcaaccttcaatgagtgcttttattgctgctttagatggtgatatcgaaactggt2460gttccagaacattttgcaagacaattagatgcatactgggcagaaatgagattgttatac2520tcatgtttcgaagccgacttgaagggaccagacccagaagtttataaacatgaaattcca2580ggtggacagttgactaacctaatcttccaagcccaacaagttggtttgggtgaacaatgg2640gaagaaactaagaagaagtatgaagatgctaacatgttgttgggtgatattgtcaaggtt2700accccaacctccaaggttgttggtgatttagcccaatttatggtttctaataaattagaa2760aaagaagatgttgaaaaacttgctaatgaattagatttcccagattcagttcttgatttc2820tttgaaggattaatgggtacaccatatggtggattcccagagcctttgagaacaaatgtc2880atttccggcaagagaagaaaattaaagggtagaccaggtttagaattagaacctttcaac2940ctcgaggaaatcagagaaaatttggtttccagatttggtccaggtattactgaatgtgat3000gttgcatcttataacatgtatccaaaggtttacgagcaatatcgtaaggtggttgaaaaa3060tatggtgatttatctgttttaccaacaaaagcatttttggctcctccaactattggtgaa3120gaagttcatgtggaaattgagcaaggtaagactttgattattaagttattagccatttct3180gacttgtctaaatctcatggtacaagagaagtatactttgaattgaatggtgaaatgaga3240aaggttacaattgaagataaaacagctgcaattgagactgttacaagagcaaaggctgac3300ggacacaatccaaatgaagttggtgcgccaatggctggtgtcgttgttgaagttagagtg3360aagcatggaacagaagttaagaagggtgatccattagccgttttgagtgcaatgaaaatg3420gaaatggttatttctgctcctgttagtggtagggtcggtgaagtttttgtcaacgaaggc3480gattccgttgatatgggtgatttgcttgtgaaaattgccaaagatgaagcgccagcagct3540taa3543<210>2<211>1180<212>prt<213>东方伊萨酵母<400>2metserthrvalgluasphisserserleuhislysleuarglysglu151015sergluileleuserasnalaasnlysileleuvalalaasnarggly202530gluileproileargilepheargseralahisgluleusermethis354045thrvalalailetyrserhisgluaspargleusermethisargleu505560lysalaaspglualatyralaileglylysthrglyglntyrserpro65707580valglnalatyrleuglnileaspgluileilelysilealalysglu859095hisaspvalsermetilehisproglytyrglypheleusergluasn100105110sergluphealalyslysvalglugluserglymetiletrpvalgly115120125proproalagluvalileaspservalglyasplysvalseralaarg130135140asnleualailelyscysaspvalprovalvalproglythraspgly145150155160proilegluaspilegluglnalalysglnphevalgluglntyrgly165170175tyrprovalileilelysalaalapheglyglyglyglyargglymet180185190argvalvalarggluglyaspaspilevalaspalapheglnargala195200205serserglualalysseralapheglyasnglythrcyspheileglu210215220argpheleuasplysprolyshisilegluvalglnleuleualaasp225230235240asntyrglyasnthrilehisleuphegluargaspcysservalgln245250255argarghisglnlysvalvalgluilealaproalalysthrleupro260265270valgluvalargasnalaileleulysaspalavalthrleualalys275280285thralaasntyrargasnalaglythralaglupheleuvalaspser290295300glnasnarghistyrpheilegluileasnproargileglnvalglu305310315320histhrilethrglugluilethrglyvalaspilevalalaalagln325330335ileglnilealaalaglyalaserleugluglnleuglyleuleugln340345350asnlysilethrthrargglyphealaileglncysargilethrthr355360365gluaspproalalysasnphealaproaspthrglylysilegluval370375380tyrargseralaglyglyasnglyvalargleuaspglyglyasngly385390395400phealaglyalavalileserprohistyraspsermetleuvallys405410415cysserthrserglyserasntyrgluilealaargarglysmetile420425430argalaleuvalglupheargileargglyvallysthrasnilepro435440445pheleuleualaleuleuthrhisprovalpheileserglyaspcys450455460trpthrthrpheileaspaspthrproserleupheglumetvalser465470475480serlysasnargalaglnlysleuleualatyrileglyaspleucys485490495valasnglyserserilelysglyglnileglypheprolysleuasn500505510lysglualagluileproaspleuleuaspproasnaspgluvalile515520525aspvalserlysproserthrasnglyleuargprotyrleuleulys530535540tyrglyproaspalapheserlyslysvalargglupheaspglycys545550555560metilemetaspthrthrtrpargaspalahisglnserleuleuala565570575thrargvalargthrileaspleuleuargilealaprothrthrser580585590hisalaleuglnasnalaphealaleuglucystrpglyglyalathr595600605pheaspvalalametargpheleutyrgluaspprotrpgluargleu610615620argglnleuarglysalavalproasnilepropheglnmetleuleu625630635640argglyalaasnglyvalalatyrserserleuproaspasnalaile645650655asphisphevallysglnalalysaspasnglyvalaspilephearg660665670valpheaspalaleuasnaspleugluglnleulysvalglyvalasp675680685alavallyslysalaglyglyvalvalglualathrvalcystyrser690695700glyaspmetleuileproglylyslystyrasnleuasptyrtyrleu705710715720gluthrvalglylysilevalglumetglythrhisileleuglyile725730735lysaspmetalaglythrleulysprolysalaalalysleuleuile740745750glyserileargserlystyrproaspleuvalilehisvalhisthr755760765hisaspseralaglythrglyileserthrtyrvalalacysalaleu770775780alaglyalaaspilevalaspcysalaileasnsermetserglyleu785790795800thrserglnprosermetseralapheilealaalaleuaspglyasp805810815ilegluthrglyvalprogluhisphealaargglnleuaspalatyr820825830trpalaglumetargleuleutyrsercyspheglualaaspleulys835840845glyproaspprogluvaltyrlyshisgluileproglyglyglnleu850855860thrasnleuilepheglnalaglnglnvalglyleuglygluglntrp865870875880glugluthrlyslyslystyrgluaspalaasnmetleuleuglyasp885890895ilevallysvalthrprothrserlysvalvalglyaspleualagln900905910phemetvalserasnlysleuglulysgluaspvalglulysleuala915920925asngluleuasppheproaspservalleuaspphephegluglyleu930935940metglythrprotyrglyglypheprogluproleuargthrasnval945950955960ileserglylysargarglysleulysglyargproglyleugluleu965970975glupropheasnleuglugluilearggluasnleuvalserargphe980985990glyproglyilethrglucysaspvalalasertyrasnmettyrpro99510001005lysvaltyrgluglntyrarglysvalvalglulystyrglyasp101010151020leuservalleuprothrlysalapheleualaproprothrile102510301035glyglugluvalhisvalgluilegluglnglylysthrleuile104010451050ilelysleuleualaileseraspleuserlysserhisglythr105510601065arggluvaltyrphegluleuasnglyglumetarglysvalthr107010751080ilegluasplysthralaalailegluthrvalthrargalalys108510901095alaaspglyhisasnproasngluvalglyalaprometalagly110011051110valvalvalgluvalargvallyshisglythrgluvallyslys111511201125glyaspproleualavalleuseralametlysmetglumetval113011351140ileseralaprovalserglyargvalglygluvalphevalasn114511501155gluglyaspservalaspmetglyaspleuleuvallysileala116011651170lysaspglualaproalaala11751180<210>3<211>1154<212>prt<213>类球红细菌(rhodobactersphaeroides)<400>3metalagluphearglysileleuilealaasnargglygluileala151015ileargvalmetargalaalaasnglumetglylyslysthrvalala202530valtyralaglugluasplysleuserleuhisargphelysalaasp354045glualatyrargileglygluglyleuserprovalglyalatyrleu505560serileprogluileileargvalalaglnmetserglyalaaspala65707580ilehisproglytyrglyleuleusergluasnproaspphevalglu859095alacysaspalaalaglyilealapheileglyprolysalagluthr100105110metargalaleuglyasplysalaseralaargargvalalametala115120125alaglyvalprovalileproalathrgluvalleuglyaspaspmet130135140glugluilelysargglnalaalagluileglytyrproleumetleu145150155160lysalasertrpglyglyglyglyargglymetargproilethrser165170175glualagluleualaasplysvalarggluglyargargglualaglu180185190alaalapheglyasnglygluglytyrleuglulysmetileglnarg195200205alaarghisvalgluvalglnileleuglyasplystyrglyalaile210215220tyrhisleutyrgluargaspcysthrvalglnargargasnglnlys225230235240valvalgluargalaproalaprotyrleuthrglugluglnargthr245250255gluilecysgluleuglyargargilecysalahisvalasntyrglu260265270cysalaglythrvalglupheleumetaspmetaspserglulysphe275280285tyrpheilegluvalasnproargvalglnvalgluhisthrvalthr290295300glugluvalthrglyileaspilevalglnserglnileargileala305310315320gluglyalathrleualaglualathrglycysproserglnaspasp325330335ilelysleuserglyhisalaleuglncysargvalthrthrgluasp340345350proglnasnasnpheileproasptyrglyargleuthralatyrarg355360365seralathrglymetglyileargleuaspglyglythralatyrala370375380glyglyvalilethrargtyrtyraspserleuleuvallysvalthr385390395400alatrpalaprothrproglulysalailealaargmetaspargala405410415leuargglupheargileargglyvalalathrasnilealapheval420425430gluasnleuleulyshisproserpheleuasptyrsertyrthrthr435440445lyspheileaspthrthrproaspleupheasnphelysproargarg450455460aspargalathrlysileleuthrtyrilealaaspilethrvalasn465470475480glyhisprogluthralaglyargvalargproseralagluleulys485490495aspprolysalaprogluprolysglyalaproglnproglythrarg500505510thrleuleugluglulysglyproglnalavalalaasptrpmetala515520525alaglnthrargvalleumetthraspthrthrmetargaspglyhis530535540glnserleuleualathrargmetargserileaspmetilelysval545550555560thrproalatyralaalaasnleuglyglyleupheservalglucys565570575trpglyglyalathrpheaspvalalatyrargpheleuglnglucys580585590protrpglnargleuargaspileargalaargleuproasnvalmet595600605thrglnmetleuleuargalaserasnglyvalglytyrthrasntyr610615620proaspasnvalvalglngluphevalargglnalaalagluthrgly625630635640valaspvalpheargvalpheaspserleuasntrpvalgluasnmet645650655argvalalametaspalavalileglualaasnlysvalcysglugly660665670thrilecystyrthrglyaspleuleuaspproaspargserlystyr675680685aspleuasntyrtyrvalglymetglyargalaleuargaspalagly690695700alahisvalleuglyleulysaspmetalaglyleuleulysproala705710715720alaalaargvalleuvallysalaleulysglugluvalglyleupro725730735ilehisphehisthrhisaspthrserglyilealaglyalathrval740745750leualaalacysaspalaglyvalaspalavalaspalaalametasp755760765alapheserglyglythrserglnprocysleuglyserilevalglu770775780alaleulyshisthraspargaspthrglyleuaspilealaalaile785790795800arggluileserasptyrtrpglyhisvalargglnglntyrserala805810815phegluserglyleuproserproalasergluvaltyrleuhisglu820825830metproglyglyglnphethrasnleulysalaglnalaargsermet835840845glyleuglugluargtrpsergluvalalaglnalatyralaaspala850855860asnargmetpheglyaspilevallysvalthrproserserlysval865870875880valglyaspmetalaleumetmetvalalaglnglyleuthrargglu885890895gluvalgluaspprogluvalgluvalserpheproaspservalval900905910aspmetleulysglyasnleuglyglnprohisglyglytrpproglu915920925proileleulyslysvalleulysglyglualaproserthrgluarg930935940proglyalahisleuproprovalaspilealaalaalaargglulys945950955960leuleusergluilelysglnglyaspaspaspproleuaspthrala965970975valaspalagluaspleuasnglytyrleumettyrprolysvalphe980985990thrasptyrargalaarghisargiletyrglyprovalargthrleu99510001005prothrargthrphephetyrglymetgluproglyglugluile101010151020seralagluileaspproglylysthrleugluileargleuser102510301035alavalglygluthrseraspaspglyaspalalysvalphephe104010451050gluleuasnglyglnproargvalileargvalalaasnargala105510601065vallysalalysthralathrargprolysalaglnaspglyasn107010751080proalahisvalglyalaprometproglyservalalaserval108510901095alavalseralaglyglnlysvallysproglyaspleuleuval110011051110thrileglualametlysmetgluthrglyleuhisalaasparg111511201125alaalathrvallysalavalhisvalglyproglyalaglnile113011351140glualalysaspleuleuvalgluleugluasp11451150<210>4<211>1154<212>prt<213>菜豆根瘤菌<400>4metproileserlysileleuvalalaasnargsergluilealaile151015argvalpheargalaalaasngluleuglyilelysthrvalalaile202530trpalaglugluasplysleualaleuhisargphelysalaaspglu354045sertyrglnvalglyargglyprohisleualaargaspleuglypro505560ileglusertyrleuserileaspgluvalileargvalalalysleu65707580serglyalaaspalailehisproglytyrglyleuleusergluser859095progluphevalaspalacysasnlysalaglyileilepheilegly100105110prolysalaaspthrmetargglnleuglyasnlysvalalaalaarg115120125asnleualaileservalglyvalprovalvalproalathrglupro130135140leuproaspaspmetalagluvalalalysmetalaalaalailegly145150155160tyrprovalmetleulysalasertrpglyglyglyglyargglymet165170175argvalileargserglualaaspleualalysgluvalthrgluala180185190lysargglualametalaalapheglylysaspgluvaltyrleuglu195200205lysleuvalgluargalaarghisvalgluserglnileleuglyasp210215220thrhisglyasnvalvalhisleuphegluargaspcysservalgln225230235240argargasnglnlysvalvalgluargalaproalaprotyrleuser245250255glualaglnargglngluleualaalatyrserleulysilealagly260265270alathrasntyrileglyalaglythrvalglutyrleumetaspala275280285aspthrglylysphetyrpheilegluvalasnproargileglnval290295300gluhisthrvalthrgluvalvalthrglyileaspilevallysala305310315320glnilehisileleuaspglyalaalaileglythrproglnsergly325330335valproasnglngluaspileargleuasnglyhisalaleuglncys340345350argvalthrthrgluaspprogluhisasnpheileproasptyrgly355360365argilethralatyrargseralaserglypheglyileargleuasp370375380glyglythrsertyrserglyalaileilethrargtyrtyrasppro385390395400leuleuvallysvalthralatrpalaproasnproleuglualaile405410415serargmetaspargalaleuargglupheargileargglyvalala420425430thrasnleuthrpheleuglualaileileglyhisprolysphearg435440445aspasnsertyrthrthrargpheileaspthrthrprogluleuphe450455460glnglnvallysargglnaspargalathrlysleuleuthrtyrleu465470475480alaaspvalthrvalasnglyhisproglualalysaspargprolys485490495proleugluasnalaalaargprovalvalprotyralaasnglyasn500505510glyvallysaspglythrlysglnleuleuaspthrleuglyprolys515520525lyspheglyglutrpmetargasnglulysargvalleuleuthrasp530535540thrthrmetargaspglyhisglnserleuleualathrargmetarg545550555560thrtyraspilealaargilealaglythrtyrserhisalaleupro565570575asnleuleuserleuglucystrpglyglyalathrpheaspvalser580585590metargpheleuthrgluaspprotrpgluargleualaleuilearg595600605gluglyalaproasnleuleuleuglnmetleuleuargglyalaasn610615620glyvalglytyrthrasntyrproaspasnvalvallystyrpheval625630635640argglnalaalalysglyglyileaspleupheargvalpheaspcys645650655leuasntrpvalgluasnmetargvalsermetaspalailealaglu660665670gluasnlysleucysglualaalailecystyrthrglyaspileleu675680685asnseralaargprolystyraspleulystyrtyrthrasnleuala690695700valgluleuglulysalaglyalahisileilealavallysaspmet705710715720alaglyleuleulysproalaalaalalysvalleuphelysalaleu725730735argglualathrglyleuproilehisphehisthrhisaspthrser740745750glyilealaalaalathrvalleualaalavalglualaglyvalasp755760765alavalaspalaalametaspalaleuserglyasnthrserglnpro770775780cysleuglyserilevalglualaleuserglysergluargasppro785790795800glyleuaspproalatrpileargargileserphetyrtrpgluala805810815valargasnglntyralaalaphegluseraspleulysglyproala820825830sergluvaltyrleuhisglumetproglyglyglnphethrasnleu835840845lysgluglnalaargserleuglyleugluthrargtrphisglnval850855860alaglnalatyralaaspalaasnglnmetpheglyaspilevallys865870875880valthrproserserlysvalvalglyaspmetalaleumetmetval885890895serglnaspleuthrvalalaaspvalvalserproasparggluval900905910serpheprogluservalvalsermetleulysglyaspleuglygln915920925proproserglytrpproglualaleuglnlyslysalaleulysgly930935940glulysprotyrthrvalargproglyserleuleulysglualaasp945950955960leuaspalagluarglysvalileglulyslysleugluarggluval965970975seraspphegluphealasertyrleumettyrprolysvalphethr980985990aspphealaleualaseraspthrtyrglyprovalservalleupro99510001005thrproalatyrphetyrglyleualaaspglyglugluleuphe101010151020alaaspileglulysglylysthrleuvalilevalasnglnala102510301035valseralathraspserglnglymetvalthrvalphepheglu104010451050leuasnglyglnproargargilelysvalproaspargalahis105510601065glyalathrglyalaalavalargarglysalagluproglyasn107010751080alaalahisvalglyalaprometproglyvalileserargval108510901095phevalserserglyglnalavalasnalaglyaspvalleuval110011051110serileglualametlysmetgluthralailehisalaglulys111511201125aspglythrilealagluvalleuvallysalaglyaspglnile113011351140aspalalysaspleuleualavaltyrglygly11451150<210>5<211>602<212>prt<213>荧光假单胞菌<400>5metthrlyslysilephevalthraspthrileleuargaspalahis151015glnserleuleualathrargmetargthrgluaspmetleuproile202530cysasplysleuasplysvalglytyrtrpserleuglucystrpgly354045glyalathrpheaspalacysvalargpheleulysgluaspprotrp505560gluargleuargglnleuargalaalaleuproasnthrargleugln65707580metleuleuargglyglnasnleuleuglytyrarghistyrserasp859095aspvalvallysalaphevalalalysalaalavalasnglyileasp100105110valpheargilepheaspalametasnaspvalargasnleuargval115120125alaileglualavallysalaalaglylyshisalaglnglythrile130135140alatyrthrthrserprovalhisthrileaspalaphevalalagln145150155160alalysglnmetglualametglycysaspservalalailelysasp165170175metalaglyleuleuthrprotyralathrglygluleuvalargala180185190leulysalagluglnserleuprovalpheilehisserhisaspthr195200205alaglyleualaalametcysglnleulysalailegluasnglyala210215220asphisileaspthralaileserserphealaserglythrserhis225230235240proglythrglusermetvalalaalaleulysglythrglupheasp245250255thrglyleuasnleugluleuleuglngluileglyleutyrphetyr260265270alavalarglyslystyrhisglnpheglusergluphethralaval275280285aspthrargvalglnvalasnglnvalproglyglymetileserasn290295300leualaasnglnleulysgluglnglyalaleuasnargmetglyglu305310315320valleualagluileproargvalarggluaspleuglyphepropro325330335leuvalthrprothrserglnilevalglythrglnalaphepheasn340345350valleualaglygluargtyrlysthrilethrasngluvallysleu355360365tyrleuglnglyglytyrglylysalaproglythrvalasnglulys370375380leuargargglnalaileglyserglugluvalileaspvalargpro385390395400alaaspleuleulysproglumetthrlysleuargalaaspilegly405410415alaleualalysserglugluaspvalleuthrphealametphepro420425430aspileglyarglyspheleuglugluargalaalaglythrleuthr435440445progluvalleuleuproileproglualaglylysvalalaserala450455460glyglygluglyvalprothrgluphevalileaspvalhisglyglu465470475480thrtyrargvalaspilethrglyvalglyvallysalagluglylys485490495arghisphetyrleuserileaspglymetproglugluvalvalphe500505510gluproleuasngluphevalglyglyglyserserlysarglysgln515520525alaseralaproglyhisvalserthrthrmetproglyasnileval530535540aspvalleuvallysgluglyaspthrvallysalaglyglnalaval545550555560leuilethrglualametlysmetgluthrgluvalglnalaalaile565570575alaglylysvalthralailehisvalalalysglyaspargvalasn580585590proglygluileleuilegluileglugly595600<210>6<211>481<212>prt<213>荧光假单胞菌<400>6metvalproproalaglnglyasnleuglnvalilethrlysileleu151015ilealaasnargglygluilealavalargilevalargalacysala202530glumetglyileargservalalailetyrseraspalaasparghis354045alaleuhisvallysargalaaspglualahisserileglyalaglu505560proleualaglytyrleuasnproarglysleuvalasnleualaval65707580gluthrglycysaspalaleuhisproglytyrglypheleuserglu859095asnalagluleualaaspilecysalagluargglyilelyspheile100105110glyproseralagluvalileargargmetglyasplysthrgluala115120125argargsermetilelysalaglyvalprovalthrproglythrglu130135140glyasnvalserglyilegluglualaleusergluglyaspargile145150155160glytyrprovalmetleulysalathrserglyglyglyglyarggly165170175ileargargcysasnserargglugluleugluglnasnpheproarg180185190valileserglualathrlysalapheglyseralagluvalpheleu195200205glulyscysilevalasnprolyshisileglualaglnileleugly210215220aspserpheglyasnvalvalhisleuphegluargaspcysserile225230235240glnargargasnglnlysleuilegluilealaproserproglnleu245250255thrprogluglnargalatyrileglyaspleuservalargalaala260265270lysalavalglytyrgluasnalaglythrvalglupheleuleuala275280285gluglygluvaltyrphemetglumetasnthrargvalglnvalglu290295300histhrilethrglugluilethrglyileaspilevalargglugln305310315320ileargilealaserglyleuproleuservallysglngluaspile325330335glnhisargglyphealaleuglnpheargileasnalagluasppro340345350lysasnasnpheleuproserpheglylysilethrargtyrtyrala355360365proglyglyproglyvalargthraspthralailetyrthrglytyr370375380thrileproprophetyraspsermetcysleulysleuvalvaltrp385390395400alaleuthrtrpgluglualametaspargglyleuargalaleuasp405410415aspmetargleuglnglyvallysthrthralaalatyrtyrglnglu420425430ileleuargasnproglupheargserglyglnpheasnthrserphe435440445valgluserhisprogluleuthrasntyrserilelysarglyspro450455460glugluleualaleualailealaalaalailealaalahisalagly465470475480leu<210>7<211>1140<212>prt<213>谷氨酸棒杆菌<400>7metserthrhisthrserserthrleuproalaphelyslysileleu151015valalaasnargglygluilealavalargalapheargalaalaleu202530gluthrglyalaalathrvalalailetyrproarggluasparggly354045serphehisargserphealaserglualavalargileglythrglu505560glyserprovallysalatyrleuaspileaspgluileileglyala65707580alalyslysvallysalaaspalailetyrproglytyrglypheleu859095sergluasnalaglnleualaargglucysalagluasnglyilethr100105110pheileglyprothrprogluvalleuaspleuthrglyasplysser115120125argalavalthralaalalyslysalaglyleuprovalleualaglu130135140serthrproserlysasnileaspgluilevallysseralaglugly145150155160glnthrtyrproilephevallysalavalalaglyglyglyglyarg165170175glymetargphevalalaserproaspgluleuarglysleualathr180185190glualaserargglualaglualaalapheglyaspglyalavaltyr195200205valgluargalavalileasnproglnhisilegluvalglnileleu210215220glyasphisthrglygluvalvalhisleutyrgluargaspcysser225230235240leuglnargarghisglnlysvalvalgluilealaproalaglnhis245250255leuaspprogluleuargaspargilecysalaaspalavallysphe260265270cysargserileglytyrglnglyalaglythrvalglupheleuval275280285aspglulysglyasnhisvalpheileglumetasnproargilegln290295300valgluhisthrvalthrglugluvalthrgluvalaspleuvallys305310315320alaglnmetargleualaalaglyalathrleulysgluleuglyleu325330335thrglnasplysilelysthrhisglyalaalaleuglncysargile340345350thrthrgluaspproasnasnglypheargproaspthrglythrile355360365thralatyrargserproglyglyalaglyvalargleuaspglyala370375380alaglnleuglyglygluilethralahispheaspsermetleuval385390395400lysmetthrcysargglyseraspphegluthralavalalaargala405410415glnargalaleualagluphethrvalserglyvalalathrasnile420425430glypheleuargalaleuleuargglugluaspphethrserlysarg435440445ilealathrglypheilealaasphisprohisleuleuglnalapro450455460proalaaspaspgluglnglyargileleuasptyrleualaaspval465470475480thrvalasnlysprohisglyvalargprolysaspvalalaalapro485490495ileasplysleuproasnilelysaspleuproleuproargglyser500505510argaspargleulysglnleuglyproalaalaphealaargaspleu515520525arggluglnaspalaleualavalthraspthrthrpheargaspala530535540hisglnserleuleualathrargvalargserphealaleulyspro545550555560alaalaglualavalalalysleuthrprogluleuleuservalglu565570575alatrpglyglyalathrtyraspvalalametargpheleupheglu580585590aspprotrpaspargleuaspgluleuargglualametproasnval595600605asnileglnmetleuleuargglyargasnthrvalglytyrthrpro610615620tyrproaspservalcysargalaphevallysglualaalaserser625630635640glyvalaspilepheargilepheaspalaleuasnaspvalsergln645650655metargproalaileaspalavalleugluthrasnthralavalala660665670gluvalalametalatyrserglyaspleuseraspproasnglulys675680685leutyrthrleuasptyrtyrleulysmetalaglugluilevallys690695700serglyalahisileleualailelysaspmetalaglyleuleuarg705710715720proalaalavalthrlysleuvalthralaleuargargglupheasp725730735leuprovalhisvalhisthrhisaspthralaglyglyglnleuala740745750thrtyrphealaalaalaglnalaglyalaaspalavalaspglyala755760765seralaproleuserglythrthrserglnproserleuseralaile770775780valalaalaphealahisthrargargaspthrglyleuserleuglu785790795800alavalseraspleugluprotyrtrpglualavalargglyleutyr805810815leuprophegluserglythrproglyprothrglyargvaltyrarg820825830hisgluileproglyglyglnleuserasnleuargalaglnalathr835840845alaleuglyleualaaspargphegluleuilegluaspasntyrala850855860alavalasnglumetleuglyargprothrlysvalthrproserser865870875880lysvalvalglyaspleualaleuhisleuvalglyalaglyvalasp885890895proalaaspphealaalaaspproglnlystyraspileproaspser900905910valilealapheleuargglygluleuglyasnproproglyglytrp915920925progluproleuargthrargalaleugluglyargsergluglylys930935940alaproleuthrgluvalprogluglugluglnalahisleuaspala945950955960aspaspserlysgluargargasnserleuasnargleuleuphepro965970975lysprothrgluglupheleugluhisargargargpheglyasnthr980985990seralaleuaspasparggluphephetyrglyleuvalgluglyarg99510001005gluthrleuileargleuproaspvalargthrproleuleuval101010151020argleuaspalailesergluproaspasplysglymetargasn102510301035valvalalaasnvalasnglyglnileargprometargvalarg104010451050aspargservalgluservalthralathralaglulysalaasp105510601065serserasnlysglyhisvalalaalaprophealaglyvalval107010751080thrvalthrvalalagluglyaspgluvallysalaglyaspala108510901095valalaileileglualametlysmetglualathrilethrala110011051110servalaspglylysileaspargvalvalvalproalaalathr111511201125lysvalgluglyglyaspleuilevalvalvalser113011351140<210>8<211>1152<212>prt<213>苜蓿中华根瘤菌<400>8metserileserlysileleuvalalaasnargsergluilealaile151015argvalpheargalaalaasngluleuglyleulysthrvalalaile202530trpalaglugluasplysleualaleuhisargphelysalaaspglu354045sertyrglnvalglyargglyprohisleuproargaspleuglypro505560ilemetsertyrleuserileaspgluvalileargvalalalysleu65707580serglyalaaspalailehisproglytyrglyleuleusergluser859095progluphealaglualacysalaalaasnglyilethrpheilegly100105110prolysprogluthrmetargglnleuglyasnlysvalalaalaarg115120125asnleualaileserileglyvalprovalvalproalathrglupro130135140leuproaspaspproglugluilelysargleualaglugluilegly145150155160tyrprovalmetleulysalasertrpglyglyglyglyargglymet165170175argalaileargaspprolysaspleuilearggluvalthrgluala180185190lysargglualalysalaalapheglylysaspgluvaltyrleuglu195200205lysleuvalgluargalaarghisvalgluserglnileleuglyasp210215220thrhisglyasnvalvalhisleuphegluargaspcysserilegln225230235240argargasnglnlysvalvalgluargalaproalaprotyrleuasn245250255aspalaglnargglngluleualaasptyrserleulysilealaarg260265270alathrasntyrileglyalaglythrvalglutyrleumetaspser275280285aspthrglylysphetyrpheilegluvalasnproargileglnval290295300gluhisthrvalthrgluvalvalthrglyileaspilevallysala305310315320glnilehisileleuaspglyphealaileglyalaproglusergly325330335valproargglngluaspileargleuasnglyhisalaleuglncys340345350argilethrthrgluaspprogluglnasnpheileproasptyrgly355360365argilethralatyrargglyalathrglypheglyileargleuasp370375380glyglythralatyrserglyalavalilethrargtyrtyrasppro385390395400leuleuglulysvalthralatrpalaproasnproglyglualaile405410415glnargmetileargalaleuargglupheargileargglyvalala420425430thrasnleuthrpheleuglualaileileserhisprolysphehis435440445aspasnsertyrthrthrargpheileaspthrthrprogluleuphe450455460glnglnvallysargglnaspargalathrlysleuleuthrtyrleu465470475480alaaspvalthrvalasnglyhisprogluvallysglyargprolys485490495proseraspaspilealaalaprovalvalprophethrglyglyasp500505510vallysproglythrlysglnargleuaspglnleuglyprolyslys515520525phealaglutrpvallysalaglnprogluvalleuilethraspthr530535540thrmetargaspglyhisglnserleuleualathrargmetargthr545550555560tyraspilealaargilealaglythrtyralaargalaleuproasn565570575leupheserleuglucystrpglyglyalathrpheaspvalsermet580585590argpheleuthrgluaspprotrpgluargleualametvalargglu595600605glyalaproasnleuleuleuglnmetleuleuargglyalaasngly610615620valglytyrlysasntyrproaspasnvalvallystyrphevalarg625630635640glnalaalalysglyglyileaspvalpheargvalpheaspcysleu645650655asntrpvalgluasnmetargvalalametaspalavalalagluglu660665670aspargilecysglualaalailecystyrthrglyaspileleuasn675680685seralaargprolystyraspleulystyrtyrthralaleualaala690695700gluleuglulysalaglyalahismetilealavallysaspmetala705710715720glyleuleulysproalaalaalaargvalleuphelysalaleulys725730735glualathrglyleuproilehisphehisthrhisaspthrsergly740745750ilealaalaalathrvalleualaalavalgluserglyvalaspval755760765valaspalaalametaspalaleuserglyasnthrserglnprocys770775780leuglyserilevalglualaleuserglysergluargaspprogly785790795800leuaspproglutrpileargargileserphetyrtrpglualaval805810815arghisglntyralaalaphegluseraspleulysglyproalaser820825830gluvaltyrleuhisglumetproglyglyglnphethrasnleulys835840845gluglnalaargserleuglyleugluthrargtrphisgluvalala850855860glnalatyralaaspalaasnargmetpheglyaspilevallysval865870875880thrproserserlysvalvalglyaspmetalaleumetmetvalser885890895glnaspleuthrvalalaaspvalgluasnproglylysaspileala900905910pheprogluservalvalsermetleulysglyaspleuglyglnpro915920925proglyglytrpproglualaleuglnlyslysalaleulysglyglu930935940gluprotyraspalaargproglyserleuleugluaspalaaspleu945950955960aspalagluarglysglyilegluglulysleuglyarggluvalthr965970975aspphegluphealasertyrleumettyrprolysvalphethrasp980985990tyralavalalacysgluthrtyrglyprovalservalleuprothr99510001005proalatyrphetyrglymetalaproglyglugluleupheala101010151020aspileglulysglylysthrleuvalileleuasnglnalagln102510301035glygluileaspglulysglymetvallysmetphepheglumet104010451050asnglyglnproargserilelysvalproaspargasnarggly105510601065alaseralaalavalargarglysalaglualaglyasnalaala107010751080hisleuglyalaprometproglyvalileserthrvalalaval108510901095alaserglyglnservallysalaglyaspvalleuleuserile110011051110glualametlysmetgluthralaleuhisalaglulysaspgly111511201125valilesergluvalleuvalargalaglyaspglnileaspala113011351140lysaspleuleuvalvalpheglygly11451150<210>9<211>2652<212>dna<213>大肠杆菌<400>9atgaacgaacaatattccgcattgcgtagtaatgtcagtatgctcggcaaagtgctggga60gaaaccatcaaggatgcgttgggagaacacattcttgaacgcgtagaaactatccgtaag120ttgtcgaaatcttcacgcgctggcaatgatgctaaccgccaggagttgctcaccacctta180caaaatttgtcgaacgacgagctgctgcccgttgcgcgtgcgtttagtcagttcctgaac240ctggccaacaccgccgagcaataccacagcatttcgccgaaaggcgaagctgccagcaac300ccggaagtgatcgcccgcaccctgcgtaaactgaaaaaccagccggaactgagcgaagac360accatcaaaaaagcagtggaatcgctgtcgctggaactggtcctcacggctcacccaacc420gaaattacccgtcgtacactgatccacaaaatggtggaagtgaacgcctgtttaaaacag480ctcgataacaaagatatcgctgactacgaacacaaccagctgatgcgtcgcctgcgccag540ttgatcgcccagtcatggcataccgatgaaatccgtaagctgcgtccaagcccggtagat600gaagccaaatggggctttgccgtagtggaaaacagcctgtggcaaggcgtaccaaattac660ctgcgcgaactgaacgaacaactggaagagaacctcggctacaaactgcccgtcgaattt720gttccggtccgttttacttcgtggatgggcggcgaccgcgacggcaacccgaacgtcact780gccgatatcacccgccacgtcctgctactcagccgctggaaagccaccgatttgttcctg840aaagatattcaggtgctggtttctgaactgtcgatggttgaagcgacccctgaactgctg900gcgctggttggcgaagaaggtgccgcagaaccgtatcgctatctgatgaaaaacctgcgt960tctcgcctgatggcgacacaggcatggctggaagcgcgcctgaaaggcgaagaactgcca1020aaaccagaaggcctgctgacacaaaacgaagaactgtgggaaccgctctacgcttgctac1080cagtcacttcaggcgtgtggcatgggtattatcgccaacggcgatctgctcgacaccctg1140cgccgcgtgaaatgtttcggcgtaccgctggtccgtattgatatccgtcaggagagcacg1200cgtcataccgaagcgctgggcgagctgacccgctacctcggtatcggcgactacgaaagc1260tggtcagaggccgacaaacaggcgttcctgatccgcgaactgaactccaaacgtccgctt1320ctgccgcgcaactggcaaccaagcgccgaaacgcgcgaagtgctcgatacctgccaggtg1380attgccgaagcaccgcaaggctccattgccgcctacgtgatctcgatggcgaaaacgccg1440tccgacgtactggctgtccacctgctgctgaaagaagcgggtatcgggtttgcgatgccg1500gttgctccgctgtttgaaaccctcgatgatctgaacaacgccaacgatgtcatgacccag1560ctgctcaatattgactggtatcgtggcctgattcagggcaaacagatggtgatgattggc1620tattccgactcagcaaaagatgcgggagtgatggcagcttcctgggcgcaatatcaggca1680caggatgcattaatcaaaacctgcgaaaaagcgggtattgagctgacgttgttccacggt1740cgcggcggttccattggtcgcggcggcgcacctgctcatgcggcgctgctgtcacaaccg1800ccaggaagcctgaaaggcggcctgcgcgtaaccgaacagggcgagatgatccgctttaaa1860tatggtctgccagaaatcaccgtcagcagcctgtcgctttataccggggcgattctggaa1920gccaacctgctgccaccgccggagccgaaagagagctggcgtcgcattatggatgaactg1980tcagtcatctcctgcgatgtctaccgcggctacgtacgtgaaaacaaagattttgtgcct2040tacttccgctccgctacgccggaacaagaactgggcaaactgccgttgggttcacgtccg2100gcgaaacgtcgcccaaccggcggcgtcgagtcactacgcgccattccgtggatcttcgcc2160tggacgcaaaaccgtctgatgctccccgcctggctgggtgcaggtacggcgctgcaaaaa2220gtggtcgaagacggcaaacagagcgagctggaggctatgtgccgcgattggccattcttc2280tcgacgcgtctcggcatgctggagatggtcttcgccaaagcagacctgtggctggcggaa2340tactatgaccaacgcctggtagacaaagcactgtggccgttaggtaaagagttacgcaac2400ctgcaagaagaagacatcaaagtggtgctggcgattgccaacgattcccatctgatggcc2460gatctgccgtggattgcagagtctattcagctacggaatatttacaccgacccgctgaac2520gtattgcaggccgagttgctgcaccgctcccgccaggcagaaaaagaaggccaggaaccg2580gatcctcgcgtcgaacaagcgttaatggtcactattgccgggattgcggcaggtatgcgt2640aataccggctaa2652<210>10<211>883<212>prt<213>大肠杆菌<400>10metasngluglntyrseralaleuargserasnvalsermetleugly151015lysvalleuglygluthrilelysaspalaleuglygluhisileleu202530gluargvalgluthrilearglysleuserlysserserargalagly354045asnaspalaasnargglngluleuleuthrthrleuglnasnleuser505560asnaspgluleuleuprovalalaargalapheserglnpheleuasn65707580leualaasnthralagluglntyrhisserileserprolysglyglu859095alaalaserasnprogluvalilealaargthrleuarglysleulys100105110asnglnprogluleusergluaspthrilelyslysalavalgluser115120125leuserleugluleuvalleuthralahisprothrgluilethrarg130135140argthrleuilehislysmetvalgluvalasnalacysleulysgln145150155160leuaspasnlysaspilealaasptyrgluhisasnglnleumetarg165170175argleuargglnleuilealaglnsertrphisthraspgluilearg180185190lysleuargproserprovalaspglualalystrpglyphealaval195200205valgluasnserleutrpglnglyvalproasntyrleuarggluleu210215220asngluglnleuglugluasnleuglytyrlysleuprovalgluphe225230235240valprovalargphethrsertrpmetglyglyaspargaspglyasn245250255proasnvalthralaaspilethrarghisvalleuleuleuserarg260265270trplysalathraspleupheleulysaspileglnvalleuvalser275280285gluleusermetvalglualathrprogluleuleualaleuvalgly290295300glugluglyalaalagluprotyrargtyrleumetlysasnleuarg305310315320serargleumetalathrglnalatrpleuglualaargleulysgly325330335glugluleuprolysprogluglyleuleuthrglnasnglugluleu340345350trpgluproleutyralacystyrglnserleuglnalacysglymet355360365glyileilealaasnglyaspleuleuaspthrleuargargvallys370375380cyspheglyvalproleuvalargileaspileargglngluserthr385390395400arghisthrglualaleuglygluleuthrargtyrleuglyilegly405410415asptyrglusertrpserglualaasplysglnalapheleuilearg420425430gluleuasnserlysargproleuleuproargasntrpglnproser435440445alagluthrarggluvalleuaspthrcysglnvalilealagluala450455460proglnglyserilealaalatyrvalilesermetalalysthrpro465470475480seraspvalleualavalhisleuleuleulysglualaglyilegly485490495phealametprovalalaproleuphegluthrleuaspaspleuasn500505510asnalaasnaspvalmetthrglnleuleuasnileasptrptyrarg515520525glyleuileglnglylysglnmetvalmetileglytyrseraspser530535540alalysaspalaglyvalmetalaalasertrpalaglntyrglnala545550555560glnaspalaleuilelysthrcysglulysalaglyilegluleuthr565570575leuphehisglyargglyglyserileglyargglyglyalaproala580585590hisalaalaleuleuserglnproproglyserleulysglyglyleu595600605argvalthrgluglnglyglumetileargphelystyrglyleupro610615620gluilethrvalserserleuserleutyrthrglyalaileleuglu625630635640alaasnleuleuproproprogluprolysglusertrpargargile645650655metaspgluleuservalilesercysaspvaltyrargglytyrval660665670arggluasnlysaspphevalprotyrpheargseralathrproglu675680685glngluleuglylysleuproleuglyserargproalalysargarg690695700prothrglyglyvalgluserleuargalaileprotrpilepheala705710715720trpthrglnasnargleumetleuproalatrpleuglyalaglythr725730735alaleuglnlysvalvalgluaspglylysglnsergluleugluala740745750metcysargasptrpprophepheserthrargleuglymetleuglu755760765metvalphealalysalaaspleutrpleualaglutyrtyraspgln770775780argleuvalasplysalaleutrpproleuglylysgluleuargasn785790795800leuglnglugluaspilelysvalvalleualailealaasnaspser805810815hisleumetalaaspleuprotrpilealagluserileglnleuarg820825830asniletyrthraspproleuasnvalleuglnalagluleuleuhis835840845argserargglnalaglulysgluglyglngluproaspproargval850855860gluglnalaleumetvalthrilealaglyilealaalaglymetarg865870875880asnthrgly<210>11<211>483<212>prt<213>嗜热自养甲烷杆菌<400>11metlysvalproargcysmetserthrglnhisproaspasnvalasn151015proprophephealaglugluprogluleuglyglygluaspgluile202530argglualatyrtyrvalpheserhisleuglycysaspgluglnmet354045trpaspcysgluglylysgluvalaspasntyrvalvallyslysleu505560leuthrlystyrglnalaphepheargasphisvalleuglygluasp65707580leuargleuthrleuargvalproasnprothrvalgluargalaglu859095alalysileleuleugluthrleugluserileproargsertyrasp100105110thralaserleuphetyrglymetaspalaalaprovalphegluval115120125ileleuprometthrsersersersercysleuasnargilehisser130135140tyrtyrleuaspphevallysglylysgluargleuglnleualaasp145150155160glyvalthrvallysglutrpileglyglupheargproaspgluile165170175asnvalileproleuphegluasphisgluglymetleuasnalaala180185190lysilethrglyglutyrleuaspglylysaspileglngluglnarg195200205valpheleualaargseraspproalametasntyrglymetileser210215220alathrleuleuasnargilealaleuserasppheargaspleuglu225230235240glugluserglyvallysleutyrproileileglymetglyserala245250255propheargglyasnleuargproaspasnvalgluaspvalthrtrp260265270glutyrargglyalatyrthrphethrvalglnserserphelystyr275280285asphisgluproseraspvalileargglyilelyslysleuargser290295300vallysproglyargalaalagluilegluarggluservalleuglu305310315320ileileseralatyrcysargglutyrargargglnvalmetaspleu325330335valaspileileasnargvalalaargtyrvalproglyargarglys340345350arglysleuhisileglyleupheglytyrserargsermetglyasn355360365valserleuproargalailethrphethralaalaleutyrserleu370375380glyvalproprogluleuleuglypheasnalaleuserserglyasp385390395400leuglupheileglugluvaltyrproglyleuglyargaspleuhis405410415aspalaalaargtyralaasnprogluserpropheleuserproglu420425430vallysserserphegluglutyrleugluproglutyraspglugly435440445hismetlysthrthrglugluileileargalaleuargileasnarg450455460thralaasnleuglngluleuileleuglualaalaserglnarglys465470475480pheleugly<210>12<211>537<212>prt<213>产气荚膜梭菌<400>12metlysileprocyssermetmetthrglnhisproaspasnvalglu151015thrtyrileserileglnglngluproalaglualailelysglyleu202530thrproglnasplysglyglyleuglyileglugluvalmetileasp354045phegluglylysleuthrprotyrhisglnthrserglnilealaleu505560glyleuileserasnglyileileproglylysaspvalargvalthr65707580proargileproasnalaasnlysgluservalpheargglnleumet859095serilemetserileilegluthrasnvalglnserlysgluleuthr100105110glythrproalailesergluvalvalvalprometilegluthrgly115120125lysgluileserglupheglnaspargvalasnservalvalaspmet130135140glyasnlysasntyrlysthrlysleuaspleuasnservalargile145150155160ileproleuvalgluaspvalproalaleualaasnileaspargile165170175leuaspgluhistyrgluileglulysserlysglyhisileleulys180185190aspleuargilemetilealaargseraspthralametsertyrgly195200205leuileserglyvalleuservalleumetalavalaspglyalatyr210215220lystrpglyglulyshisglyvalthrileserproileleuglycys225230235240glyserleupropheargglyhispheserglugluasnileaspglu245250255ileleualathrtyrserglyilelysthrphethrpheglnserala260265270leuargtyrasphisglygluglualathrlyshisalavalargglu275280285leulysglulysilealaglnserlysproargasnphesergluglu290295300asplysaspleumetlysglupheileglyilecysserlyshistyr305310315320leuglnthrpheleulysvalileaspthrvalserphevalserasp325330335pheileprolysasnargaspargleuthrlysalalysthrglyleu340345350glutyrasnarggluvalalaasnleuaspasnvalalaaspleuval355360365lysaspgluvalleulysglngluileleuserileaspasnserlys370375380glutyralavalproargalaileserphethrglyalamettyrthr385390395400leuglymetproprogluleumetglymetglyargalaleuasnglu405410415ilelysthrlystyrglyglngluglyileasplysleuleugluile420425430tyrproileleuarglysaspleualaphealaalaargphealaasn435440445glyglyvalserlyslysileileaspgluglualaargglnglutyr450455460lysgluaspmetlystyrvalasngluileleuasnleuglyleuasp465470475480tyrasppheleuasngluasngluphetyrhisthrleuleulysthr485490495thrlysproileilemethisleumetglyleuglugluasnvalmet500505510argasnserthrglugluleulysileleuasnglutrpilevalarg515520525metglylysvalargglyserilegly530535<210>13<211>1260<212>dna<213>东方伊萨酵母<400>13atgtccagaggcttctttactgagaacattacgcaattgccaccagaccctttgtttggt60cttaaggccaggttcagcaatgactcacgtgaaaacaaggtcgatttaggtattggggca120tatagggacgacaacggtaagccatggatcttaccatctgtcaggttggccgaaaacttg180attcagaactccccagactacaaccatgagtacctaccaatcggtggacttgctgatttc240acttctgctgcggcaagagttgtatttggaggcgattctaaagccatttcgcaaaaccgt300cttgtctccatccagagtttgtcaggtacaggtgccttacatgttgctggtctatttatc360aagcgccaatacaagtctcttgatggcacttccgaagaccctctaatatatctatcggaa420cctacatgggccaaccacgttcaaatctttgaagttattggtctcaagcctgtattctat480ccatattggcatgccgcaagcaagaccttggatctgaagggctacttaaaggcaataaac540gatgctccagaagggtcggtttttgtattgcatgcaacggctcataaccctactggtttg600gatccaacacaagaacaatggatggagattttggccgctataagtgccaaaaagcatctg660ccattatttgattgtgcatatcagggtttcacctccgggtctctagatagagatgcttgg720gctgttcgagaagctgtcaacaatgacaagtacgaattcccgggaattattgtctgtcaa780tcgtttgcgaaaaatgttggcatgtatggtgaacggattggtgcagttcatattgttcta840cctgaatcagacgcttccctaaacagcgccatcttctcccaattgcaaaagacaatcaga900tcggagatttccaatccaccaggatacggtgcaaagattgtgtctaaagttttgaacact960ccggaactttacaaacagtgggagcaagatttgatcaccatgtcttcgagaatcactgca1020atgagaaaggagctagtaaatgagctcgagcgtcttggaacccctggcacttggagacac1080atcaccgagcaacagggtatgttttcctttactggtttgaacccggagcaggttgccaag1140ctagagaaggagcatggtgtttatcttgttcgtagtggacgtgcaagtattgcaggcctc1200aacatgggaaacgtcaagtatgttgccaaggccattgactctgtcgtgagagacctttag1260<210>14<211>419<212>prt<213>东方伊萨酵母<400>14metserargglyphephethrgluasnilethrglnleuproproasp151015proleupheglyleulysalaargpheserasnaspserarggluasn202530lysvalaspleuglyileglyalatyrargaspaspasnglylyspro354045trpileleuproservalargleualagluasnleuileglnasnser505560proasptyrasnhisglutyrleuproileglyglyleualaaspphe65707580thrseralaalaalaargvalvalpheglyglyaspserlysalaile859095serglnasnargleuvalserileglnserleuserglythrglyala100105110leuhisvalalaglyleupheilelysargglntyrlysserleuasp115120125glythrsergluaspproleuiletyrleusergluprothrtrpala130135140asnhisvalglnilephegluvalileglyleulysprovalphetyr145150155160protyrtrphisalaalaserlysthrleuaspleulysglytyrleu165170175lysalaileasnaspalaprogluglyservalphevalleuhisala180185190thralahisasnprothrglyleuaspprothrglngluglntrpmet195200205gluileleualaalaileseralalyslyshisleuproleupheasp210215220cysalatyrglnglyphethrserglyserleuaspargaspalatrp225230235240alavalargglualavalasnasnasplystyrglupheproglyile245250255ilevalcysglnserphealalysasnvalglymettyrglygluarg260265270ileglyalavalhisilevalleuprogluseraspalaserleuasn275280285seralailepheserglnleuglnlysthrileargsergluileser290295300asnproproglytyrglyalalysilevalserlysvalleuasnthr305310315320progluleutyrlysglntrpgluglnaspleuilethrmetserser325330335argilethralametarglysgluleuvalasngluleugluargleu340345350glythrproglythrtrparghisilethrgluglnglnglymetphe355360365serphethrglyleuasnprogluglnvalalalysleuglulysglu370375380hisglyvaltyrleuvalargserglyargalaserilealaglyleu385390395400asnmetglyasnvallystyrvalalalysalaileaspservalval405410415argaspleu<210>15<211>418<212>prt<213>酿酒酵母<400>15metseralathrleupheasnasnilegluleuleuproproaspala151015leupheglyilelysglnargtyrglyglnaspglnargalathrlys202530valaspleuglyileglyalatyrargaspaspasnglylysprotrp354045valleuproservallysalaalaglulysleuilehisasnaspser505560sertyrasnhisglutyrleuglyilethrglyleuproserleuthr65707580serasnalaalalysileilepheglythrglnseraspalaphegln859095gluaspargvalileservalglnserleuserglythrglyalaleu100105110hisileseralalysphepheserlysphepheproasplysleuval115120125tyrleuserlysprothrtrpalaasnhismetalailephegluasn130135140glnglyleulysthralathrtyrprotyrtrpalaasngluthrlys145150155160serleuaspleuasnglypheleuasnalaileglnlysalaproglu165170175glyserilephevalleuhissercysalahisasnprothrglyleu180185190aspprothrsergluglntrpvalglnilevalaspalailealaser195200205lysasnhisilealaleupheaspthralatyrglnglyphealathr210215220glyaspleuasplysaspalatyralavalargleuglyvalglulys225230235240leuserthrvalserprovalphevalcysglnserphealalysasn245250255alaglymettyrglygluargvalglycysphehisleualaleuthr260265270lysglnalaglnasnlysthrilelysproalavalthrserglnleu275280285alalysileileargsergluvalserasnproproalatyrglyala290295300lysilevalalalysleuleugluthrprogluleuthrgluglntrp305310315320hislysaspmetvalthrmetserserargilethrlysmetarghis325330335alaleuargasphisleuvallysleuglythrproglyasntrpasp340345350hisilevalasnglncysglymetpheserphethrglyleuthrpro355360365glnmetvallysargleuglugluthrhisalavaltyrleuvalala370375380serglyargalaserilealaglyleuasnglnglyasnvalglutyr385390395400valalalysalaileaspgluvalvalargphetyrthrilegluala405410415lysleu<210>16<211>396<212>prt<213>大肠杆菌<400>16metphegluasnilethralaalaproalaaspproileleuglyleu151015alaaspleupheargalaaspgluargproglylysileasnleugly202530ileglyvaltyrlysaspgluthrglylysthrprovalleuthrser354045vallyslysalagluglntyrleuleugluasngluthrthrlysasn505560tyrleuglyileaspglyileproglupheglyargcysthrglnglu65707580leuleupheglylysglyseralaleuileasnasplysargalaarg859095thralaglnthrproglyglythrglyalaleuargvalalaalaasp100105110pheleualalysasnthrservallysargvaltrpvalserasnpro115120125sertrpproasnhislysservalpheasnseralaglyleugluval130135140argglutyralatyrtyraspalagluasnhisthrleuasppheasp145150155160alaleuileasnserleuasnglualaglnalaglyaspvalvalleu165170175phehisglycyscyshisasnprothrglyileaspprothrleuglu180185190glntrpglnthrleualaglnleuservalglulysglytrpleupro195200205leupheaspphealatyrglnglyphealaargglyleuglugluasp210215220alagluglyleuargalaphealaalamethislysgluleuileval225230235240alasersertyrserlysasnpheglyleutyrasngluargvalgly245250255alacysthrleuvalalaalaaspsergluthrvalaspargalaphe260265270serglnmetlysalaalaileargalaasntyrserasnproproala275280285hisglyalaservalvalalathrileleuserasnaspalaleuarg290295300alailetrpgluglngluleuthraspmetargglnargileglnarg305310315320metargglnleuphevalasnthrleuglnglulysglyalaasnarg325330335asppheserpheileilelysglnasnglymetpheserphesergly340345350leuthrlysgluglnvalleuargleuarggluglupheglyvaltyr355360365alavalalaserglyargvalasnvalalaglymetthrproaspasn370375380metalaproleucysglualailevalalavalleu385390395<210>17<211>139<212>prt<213>阿维链霉菌<400>17metleuargthrmetphelysserlysilehisargalathrvalthr151015glnalaaspleuhistyrvalglyservalthrileaspalaaspleu202530leuaspalaalaaspleuleuproglygluleuvalhisilevalasp354045ilethrasnglyalaargleugluthrtyrvalilegluglygluarg505560glyserglyvalvalglyileasnglyalaalaalahisleuvalhis65707580proglyaspleuvalileileilesertyralaglnvalseraspala859095glualaargalaleuargproargvalvalhisvalaspargaspasn100105110argvalvalalaleuglyalaaspproalagluprovalproglyser115120125aspglnalaargserproglnalavalthrala130135<210>18<211>127<212>prt<213>丙酮丁醇梭菌<400>18methisleuasnmetleulysserlysilehisargalathrvalval151015glnalaaspleuasntyrvalglyserilethrileaspargasnleu202530metasplysalaasnileleuglutyrglulysvalgluilealaasn354045ileasnasnglyalaargphegluthrtyrvalilealaglygluala505560glyserglyileilecysleuasnglyalaalaalaargcysalagln65707580alaglyasplysvalileilemetcystyrcysserleuthrproglu859095glualasergluhisargprolysvalvalphevalasnaspaspasn100105110serileserasnvalthrglutyrglulyshisglythrilegly115120125<210>19<211>1404<212>dna<213>东方伊萨酵母<400>19atgtctattagtgaaaaatattttcctcaagaacctcaatctcctgaattgaaaactgca60attccaggtcctcaatcaaaggcaaagctcgaggaattatctgctgtctatgatacaaag120gctgcatattttgttaccgactactacaaatctcttggtaactatattgtggatgcagat180ggcaacaagctactagattcttattgccaaatctcttctatcgcattgggttacaataat240ccagcattattaaaagtagcacattctgatgaaatgacagttgctttatgtaacagacct300gctttggcatgttttccatccactgattactatgaaatactaaagaagggattgttgtcc360gttgctccaaagggattagataaggtttgtactgcacacacgggatctgatgccaatgaa420atggcatttaaggctgcatttttgtttcaagcaagtaagaagagaggtgacaaaccattt480accagcgaagagctggaatctgtcatggagaacaagttgccaggcacctctgacatggtt540atcctgtcatttgaaaaagggttccatggtagattgtttggatctttatctaccactaga600tctaaagctattcacaaactggatattcctgcgtttgaatggccaaaggctccattccct660cagttaaagtatcctctggatcaattccaagctgaaaacaaagcagaagaagaaagatgt720ttgaaggctttagaggaaattattgtcaactctcctgccaaaattgcagctgcaatcatt780gaaccggtccaatctgaaggtggtgataatcatgcttcaccagaattcttccaaggtatt840agagaaatcaccaaaaagcacggtgtcattcttattgttgatgaagttcaaacaggaggt900ggtgcttctggtaagatgtggttacatgaacactatggcattgtcccagacatcatgact960ttttctaaaaaaatgcaaaatgcaggtttctttttcagtgaagcaggtcttgctggggac1020caaccattcagacaattcaatacctggtgcggtgatccatcaaaagctctaattgcaaga1080accataattgaagaaattaaagataagaacctattgactagtgttaccgaaacaggtgac1140tacctatattcaaagctcgaagcaatttcagcaaagtatgacaaaatgatcaacttgaga1200ggtaagggaagaggtttctttattgcatttgatgccccaacaccggagttaagaaacaaa1260tttattgctgaatgtaagaaattaggtttaaacattggtggatgcggtgaacaaggtgtt1320agattgagacctgcattagtttttgaaaagaagcatgctgatatcttagcctccattatt1380gatcaagctttttccaaaatttaa1404<210>20<211>467<212>prt<213>东方伊萨酵母<400>20metserileserglulystyrpheproglngluproglnserproglu151015leulysthralaileproglyproglnserlysalalysleugluglu202530leuseralavaltyraspthrlysalaalatyrphevalthrasptyr354045tyrlysserleuglyasntyrilevalaspalaaspglyasnlysleu505560leuaspsertyrcysglnileserserilealaleuglytyrasnasn65707580proalaleuleulysvalalahisseraspglumetthrvalalaleu859095cysasnargproalaleualacyspheproserthrasptyrtyrglu100105110ileleulyslysglyleuleuservalalaprolysglyleuasplys115120125valcysthralahisthrglyseraspalaasnglumetalaphelys130135140alaalapheleupheglnalaserlyslysargglyasplysprophe145150155160thrserglugluleugluservalmetgluasnlysleuproglythr165170175seraspmetvalileleuserpheglulysglyphehisglyargleu180185190pheglyserleuserthrthrargserlysalailehislysleuasp195200205ileproalapheglutrpprolysalapropheproglnleulystyr210215220proleuaspglnpheglnalagluasnlysalagluglugluargcys225230235240leulysalaleuglugluileilevalasnserproalalysileala245250255alaalaileilegluprovalglnsergluglyglyaspasnhisala260265270serprogluphepheglnglyilearggluilethrlyslyshisgly275280285valileleuilevalaspgluvalglnthrglyglyglyalasergly290295300lysmettrpleuhisgluhistyrglyilevalproaspilemetthr305310315320pheserlyslysmetglnasnalaglyphephepheserglualagly325330335leualaglyaspglnpropheargglnpheasnthrtrpcysglyasp340345350proserlysalaleuilealaargthrileileglugluilelysasp355360365lysasnleuleuthrservalthrgluthrglyasptyrleutyrser370375380lysleuglualaileseralalystyrasplysmetileasnleuarg385390395400glylysglyargglyphepheilealapheaspalaprothrproglu405410415leuargasnlyspheilealaglucyslyslysleuglyleuasnile420425430glyglycysglygluglnglyvalargleuargproalaleuvalphe435440445glulyslyshisalaaspileleualaserileileaspglnalaphe450455460serlysile465<210>21<211>475<212>prt<213>克鲁维酵母<400>21metprosertyrservalalagluleutyrtyrproaspgluprothr151015gluprolysileserthrsersertyrproglyprolysalalysgln202530gluleuglulysleuserasnvalpheaspthrargalaalatyrleu354045leualaasptyrtyrlysserargglyasntyrilevalaspglnasp505560glyasnvalleuleuaspvaltyralaglnileserserilealaleu65707580glytyrasnasnprogluileleulysvalalalysseraspalamet859095servalalaleualaasnargproalaleualacyspheproserasn100105110asptyrglyglnleuleugluaspglyleuleulysalaalaprogln115120125glyglnasplysiletrpthralaleuserglyseraspalaasnglu130135140thralaphelysalacysphemettyrglnalaalalyslysargasn145150155160glyargserpheserthrglugluleugluservalmetaspasngln165170175leuproglythrserglumetvalilecysserpheglulysglyphe180185190hisglyargleupheglyserleuserthrthrargserlysproile195200205hislysleuaspileproalapheasntrpprolysalaprophepro210215220aspleulystyrproleuglugluasnlysglualaasnlysalaglu225230235240glusersercysileglulyspheserglnilevalglnglutrpgln245250255glylysilealaalavalileilegluproileglnsergluglygly260265270aspasnhisalaserseraspphepheglnlysleuarggluilethr275280285ilegluasnglyileleumetilevalaspgluvalglnthrglyval290295300glyalathrglylysmettrpalahisgluhistrpasnleuserasn305310315320proproaspleuvalthrpheserlyslyspheglnalaalaglyphe325330335tyrtyrhisaspprolysleuglnproaspglnpropheargglnphe340345350asnthrtrpcysglyaspproserlysalaleuilealalysvalile355360365tyrglugluilevallyshisaspleuvalthrargthralagluval370375380glyasntyrleupheasnargleuglulysleuphegluglylysasn385390395400tyrileglnasnleuargglylysglyglnglythrtyrilealaphe405410415asppheglythrsersergluargaspserpheleuserargleuarg420425430cysasnglyalaasnvalalaglycysglyaspseralavalargleu435440445argproserleuthrphegluglulyshisalaaspvalleuvalser450455460ilepheasplysthrleuargglnleutyrgly465470475<210>22<211>451<212>prt<213>阿维链霉菌<400>22metthrproglnproasnproglnvalglyalaalavallysalaala151015aspargalahisvalphehissertrpseralaglngluleuileasp202530proleualavalalaglyalagluglysertyrphetrpasptyrasp354045glyargargtyrleuaspphethrserglyleuvalphethrasnile505560glytyrglnhisprolysvalvalalaalaileglngluglnalaala65707580serleuthrthrphealaproalaphealavalglualaargserglu859095alaalaargleuilealagluargthrproglyaspleuasplysile100105110phephethrasnglyglyalaaspalailegluhisalavalargmet115120125alaargilehisthrglyargprolysvalleuseralatyrargser130135140tyrhisglyglythrglnglnalavalasnilethrglyaspproarg145150155160argtrpalaseraspseralaseralaglyvalvalhisphetrpala165170175protyrleutyrargserargphetyralagluthrgluglnglnglu180185190cysgluargalaleugluhisleugluthrthrilealapheglugly195200205proglythrilealaalailevalleugluthrvalproglythrala210215220glyilemetvalproproproglytyrleualaglyvalarggluleu225230235240cysasplystyrglyilevalphevalleuaspgluvalmetalagly245250255pheglyargthrglyglutrpphealaalaaspleupheaspvalthr260265270proaspleumetthrphealalysglyvalasnserglytyrvalpro275280285leuglyglyvalalaileserglylysilealagluthrpheglylys290295300argalatyrproglyglyleuthrtyrserglyhisproleualacys305310315320alaalaalavalalathrileasnvalmetalaglugluglyvalval325330335gluasnalaalaasnleuglyalaargvalilegluproglyleuarg340345350gluleualagluarghisproservalglygluvalargglyvalgly355360365metphetrpalaleugluleuvallysasparggluthrargglupro370375380leuvalprotyrasnalaalaglyglualaasnalaprometalaala385390395400pheglyalaalaalalysalaasnglyleutrppropheileasnmet405410415asnargthrhisvalvalproprocysasnvalthrglualagluala420425430lysgluglyleualaalaleuaspalaalaleuservalalaaspglu435440445tyrthrval450<210>23<211>419<212>prt<213>阿维链霉菌<400>23metseralaleuserprohisleuargglnalathrprovalvalala151015valargglygluglyvalhisleutyrglygluaspglyargargtyr202530leuaspphethralaglyileglyvalthrserthrglyhiscyshis354045proargvalvalalaalaalaglngluglnalaglythrleuvalhis505560glyglntyrthrthrvalleuhisproproleuargargleuvalasp65707580argleuglygluvalleuproalaglyleuaspserleuphephethr859095asnserglyserglualavalglualaalaleuargleualaarggln100105110alathrglyargproasnvalleuvalcyshisglyglyphehisgly115120125argthrvalalaalaalaalametthrthrserglythrargphearg130135140serglypheserproleumetserglyvalvalvalthrprophepro145150155160thralapheargtyrglytrpaspglugluthralathrargpheala165170175leuglngluleuasptyrthrleuargthrileserserproaspasp180185190thralaalaileilevalgluprovalleuglygluglyglytyrval195200205proalathrargalapheleugluglyleuarggluargalaasparg210215220hisglyphevalleuileleuaspgluvalglnthrglyvalglyarg225230235240thrglyargphetrpglyhisasphispheglyvalthrproaspile245250255leuilethralalysglyleualaserglypheproleuserglyile260265270alaalaseralagluleumetglylysalatrpproglyserglngly275280285glythrtyrglyalaasnalavalalacysalaalaalacysalathr290295300leuaspvalvalargaspglulysleuvalaspasnalaglualamet305310315320glyalaargleuargalaglyleualaalavalalaalathrthrpro325330335alaileglyaspvalargglyleuglyleumetleualasergluphe340345350valthrgluaspglyglyproaspprogluthralaalaargvalgln355360365argalaalavalaspgluglyleuleuleuleuleucysglyalatrp370375380asnglnvalvalargmetileproalaleuvalileaspglualaglu385390395400valaspgluglyleuargalatrpseralaalavalgluvalglyval405410415proalaarg<210>24<211>471<212>prt<213>酿酒酵母<400>24metserilecysgluglntyrtyrproglugluprothrlysprothr151015vallysthrgluserileproglyprogluserglnlysglnleulys202530gluleuglygluvalpheaspthrargproalatyrpheleualaasp354045tyrglulysserleuglyasntyrilethraspvalaspglyasnthr505560tyrleuaspleutyralaglnileserserilealaleuglytyrasn65707580asnproalaleuilelysalaalaglnserproglumetileargala859095leuvalaspargproalaleuglyasnpheproserlysaspleuasp100105110lysileleulysglnileleulysseralaprolysglyglnasphis115120125valtrpserglyleuserglyalaaspalaasngluleualaphelys130135140alaalapheiletyrtyrargalalysglnargglytyraspalaasp145150155160pheserglulysgluasnleuservalmetaspasnaspalaprogly165170175alaprohisleualavalleuserphelysargalaphehisglyarg180185190leuphealaserglyserthrthrcysserlysproilehislysleu195200205asppheproalaphehistrpprohisalaglutyrprosertyrgln210215220tyrproleuaspgluasnseraspalaasnarglysgluaspasphis225230235240cysleualailevalglugluleuilelysthrtrpserileproval245250255alaalaleuileilegluproileglnsergluglyglyaspasnhis260265270alaserlystyrpheleuglnlysleuargaspilethrleulystyr275280285asnvalvaltyrileileaspgluvalglnthrglyvalglyalathr290295300glylysleutrpcyshisglutyralaaspileglnproprovalasp305310315320leuvalthrpheserlyslyspheglnseralaglytyrphephehis325330335aspprolyspheileproasnlysprotyrargglnpheasnthrtrp340345350cysglygluproalaargmetileilealaglyalaileglyglnglu355360365ileserasplyslysleuthrgluglncysserargvalglyasptyr370375380leuphelyslysleugluglyleuglnlyslystyrprogluasnphe385390395400glnasnleuargglylysglyargglythrpheilealatrpaspleu405410415prothrglyglulysargaspleuleuleulyslysleulysleuasn420425430glycysasnvalglyglycysalavalhisalavalargleuargpro435440445serleuthrphegluglulyshisalaaspilepheileglualaleu450455460alalysservalasngluleu465470<210>25<211>813<212>dna<213>东方伊萨酵母<400>25atgtttggtaatatttcccaaagacttgcaggcaagaacatcctaattacaggtgcgtcc60actggtatcggataccatacagcaaagtattttgcagaagctgcaaatggagacttgaag120ttggttttggctgcaagaagaaaggagaagctggaggcactaaaggcagacttgcttgcc180aagtatccatccatcaaagtccatattgagagtttggatgtctccaaaacggaaaccatt240gcacctttcttaaaaggtttacctgaggaattttcaattgtcgacgtgttggtcaacaat300gcaggtaaggcgcttggtttggatccaattggctctgtcgatccaaaggacgtggatgaa360atgttccagaccaatgttttgggtatgattcaattgacccagttggttgtacagcaaatg420aaggagagaaactccggggacattgtccaactaggttcagtggctggtagaaacccatac480ccaggtggtggtatctactgtgcctccaaggccgcattgagatcttttacacatgtattg540agagaggaattgattaataccaagattagagtgattgaaatcgagcctggaaatgttgca600actgaggaattttctttgaccagattcaaaggtgataagtccaaggccgaaaaggtctat660gagggaaccgagccattgtatggtaccgatattgcagaattgattctatttgcagtttct720agacctcaaaacactgttattgcagaaacacttgtttttgctagtaaccaagcttctgct780taccatattttcagaggatcattagataaatag813<210>26<211>270<212>prt<213>东方伊萨酵母<400>26metpheglyasnileserglnargleualaglylysasnileleuile151015thrglyalaserthrglyileglytyrhisthralalystyrpheala202530glualaalaasnglyaspleulysleuvalleualaalaargarglys354045glulysleuglualaleulysalaaspleuleualalystyrproser505560ilelysvalhisilegluserleuaspvalserlysthrgluthrile65707580alapropheleulysglyleuprogluglupheserilevalaspval859095leuvalasnasnalaglylysalaleuglyleuaspproileglyser100105110valaspprolysaspvalaspglumetpheglnthrasnvalleugly115120125metileglnleuthrglnleuvalvalglnglnmetlysgluargasn130135140serglyaspilevalglnleuglyservalalaglyargasnprotyr145150155160proglyglyglyiletyrcysalaserlysalaalaleuargserphe165170175thrhisvalleuargglugluleuileasnthrlysileargvalile180185190gluilegluproglyasnvalalathrgluglupheserleuthrarg195200205phelysglyasplysserlysalaglulysvaltyrgluglythrglu210215220proleutyrglythraspilealagluleuileleuphealavalser225230235240argproglnasnthrvalilealagluthrleuvalphealaserasn245250255glnalaseralatyrhisilepheargglyserleuasplys260265270<210>27<211>248<212>prt<213>大肠杆菌<400>27metilevalleuvalthrglyalathralaglypheglyglucysile151015thrargargpheileglnglnglyhislysvalilealathrglyarg202530argglngluargleuglngluleulysaspgluleuglyaspasnleu354045tyrilealaglnleuaspvalargasnargalaalailegluglumet505560leualaserleuproalaglutrpcysasnileaspileleuvalasn65707580asnalaglyleualaleuglymetgluproalahislysalaserval859095gluasptrpgluthrmetileaspthrasnasnlysglyleuvaltyr100105110metthrargalavalleuproglymetvalgluargasnhisglyhis115120125ileileasnileglyserthralaglysertrpprotyralaglygly130135140asnvaltyrglyalathrlysalaphevalargglnpheserleuasn145150155160leuargthraspleuhisglythralavalargvalthraspileglu165170175proglyleuvalglyglythrglupheserasnvalargphelysgly180185190aspaspglylysalaglulysthrtyrglnasnthrvalalaleuthr195200205progluaspvalserglualavaltrptrpvalserthrleuproala210215220hisvalasnileasnthrleuglumetmetprovalthrglnsertyr225230235240alaglyleuasnvalhisarggln245<210>28<211>298<212>prt<213>粪产碱菌<400>28metserasnthrilealapheileglyleuglyhismetglylyspro151015metalaleuasnleuleulysalaglyhisserleuasnvalpheasp202530leuasnalaglnalametglngluleuglnalaalaglyalaglnval354045glygluseralavalglnilealaglnaspalaglnmetvalphethr505560metleuproalaglyarghisvalargglnvaltyrgluglygluasn65707580glyleuleuglnthrvalalaproglythrvalleuvalaspcysser859095thrileaspalaglnthrserglnaspleualaalalysalaserlys100105110leuglyleuphemetleuaspalaprovalserglyglythrglygly115120125alailealaglythrleuthrphemetvalglyglygluaspglnala130135140leuglulysalaargprotyrleuaspalametglylysasnilephe145150155160hisalaglylysalaglyalaglyglnvalalalysilecysasnasn165170175metleuleuglyileleumetalaglythralaglualaleualaleu180185190glyvalalahisglyleuaspproalavalleuserthrilemetala195200205argserserglyargasntrpalathrgluleutyrasnprotrppro210215220glyvalmetproaspvalproalaserargasptyrglnglyglyphe225230235240alathrglyleumetleulysaspleuglyleualaalaaspalaala245250255valserglnasnseralathrproleuglygluleualaargasnleu260265270phealaleuhisalaalaglnglyglnasnalaglyleuasppheser275280285serileleuasnleutyrargglnlyshis290295<210>29<211>314<212>prt<213>metallosphaerasedula<400>29metthrglulysvalservalvalglyalaglyvalileglyvalgly151015trpalathrleuphealaserlysglytyrservalserleutyrthr202530glulyslysgluthrleuasplysglyileglulysleuargasntyr354045valglnvalmetlysasnasnserglnilethrgluaspvalasnthr505560valileserargvalserprothrthrasnleuaspglualavalarg65707580glyalaasnphevalileglualavalilegluasptyraspalalys859095lyslysilepheglytyrleuaspservalleuasplysgluvalile100105110leualaserserthrserglyleuleuilethrgluvalglnlysala115120125metserlyshisprogluargalavalilealahisprotrpasnpro130135140prohisleuleuproleuvalgluilevalproglyglulysthrser145150155160metgluvalvalgluargthrlysserleumetglulysleuasparg165170175ilevalvalvalleulyslysgluileproglypheileglyasnarg180185190leualaphealaleupheargglualavaltyrleuvalaspglugly195200205valalathrvalgluaspileasplysvalmetthralaalailegly210215220leuargtrpalaphemetglypropheleuthrtyrhisleuglygly225230235240glygluglyglyleuglutyrphepheasnargglypheglytyrgly245250255alaasnglutrpmethisthrleualalystyrasplyspheprotyr260265270thrglyvalthrlysalaileglnglnmetlysglutyrserpheile275280285lysglylysthrpheglngluileserlystrpargaspglulysleu290295300leulysvaltyrlysleuvaltrpglulys305310<210>30<211>295<212>prt<213>恶臭假单胞菌<400>30metargilealapheileglyleuglyasnmetglyalaprometala151015argasnleuilelysalaglyhisglnleuasnleupheaspleuasn202530lysalavalleualagluleualagluleuglyglyglnileserpro354045serprolysaspalaalaalaasnsergluleuvalilethrmetleu505560proalaalaalahisvalargservaltyrleuasngluaspglyval65707580leualaglyileargproglythrprothrvalaspcysserthrile859095aspproglnthralaargaspvalserlysalaalaalaalalysgly100105110valaspmetglyaspalaprovalserglyglythrglyglyalaala115120125alaglythrleuthrphemetvalglyalaserthrgluleupheala130135140serleulysprovalleugluglnmetglyargasnilevalhiscys145150155160glygluvalglythrglyglnilealalysilecysasnasnleuleu165170175leuglyilesermetileglyvalserglualametalaleuglyasn180185190alaleuglyileaspthrlysvalleualaglyileileasnserser195200205thrglyargcystrpserseraspthrtyrasnprotrpproglyile210215220ilegluthralaproalaserargglytyrthrglyglypheglyala225230235240gluleumetleulysaspleuglyleualathrglualaalaarggln245250255alahisglnprovalileleuglyalavalalaglnglnleutyrgln260265270alametserleuargglygluglyglylysasppheseralaileval275280285gluglytyrarglyslysasp290295<210>31<211>295<212>prt<213>恶臭假单胞菌<400>31metargilealapheileglyleuglyasnmetglyalaprometala151015argasnleuilelysalaglyhisglnleuasnleupheaspleuasn202530lysthrvalleualagluleualagluleuglyglyglnileserpro354045serprolysaspalaalaalasersergluleuvalilethrmetleu505560proalaalaalahisvalargservaltyrleuasnaspaspglyval65707580leualaglyileargproglythrprothrvalaspcysserthrile859095aspproglnthralaargaspvalserlysalaalaalaalalysgly100105110valaspmetglyaspalaprovalserglyglythrglyglyalaala115120125alaglythrleuthrphemetvalglyalaseralagluleupheala130135140serleulysprovalleugluglnmetglyargasnilevalhiscys145150155160glygluvalglythrglyglnilealalysilecysasnasnleuleu165170175leuglyilesermetileglyvalserglualametalaleuglyasn180185190alaleuglyileaspthrlysvalleualaglyileileasnserser195200205thrglyargcystrpserseraspthrtyrasnprotrpproglyile210215220ilegluthralaproalaserargglytyrthrglyglypheglyala225230235240gluleumetleulysaspleuglyleualathrglualaalaarggln245250255alahisglnprovalileleuglyalavalalaglnglnleutyrgln260265270alametserleuargglygluglyglylysasppheseralaileval275280285gluglytyrarglyslysasp290295<210>32<211>298<212>prt<213>铜绿假单胞菌<400>32metthraspilealapheleuglyleuglyasnmetglyglypromet151015alaalaasnleuleulysalaglyhisargvalasnvalpheaspleu202530glnprolysalavalleuglyleuvalgluglnglyalaglnglyala354045aspseralaleuglncyscysgluglyalagluvalvalilesermet505560leuproalaglyglnhisvalgluserleutyrleuglyaspaspgly65707580leuleualaargvalalaglylysproleuleuileaspcysserthr859095ilealaprogluthralaarglysvalalaglualaalaalaalalys100105110glyleuthrleuleuaspalaprovalserglyglyvalglyglyala115120125argalaglythrleuserpheilevalglyglyproalagluglyphe130135140alaargalaargprovalleugluasnmetglyargasnilephehis145150155160alaglyasphisglyalaglyglnvalalalysilecysasnasnmet165170175leuleuglyileleumetalaglythralaglualaleualaleugly180185190vallysasnglyleuaspproalavalleusergluvalmetlysgln195200205serserglyglyasntrpalaleuasnleutyrasnprotrpprogly210215220valmetproglnalaproalaserasnglytyralaglyglyphegln225230235240valargleumetasnlysaspleuglyleualaleualaasnalagln245250255alavalglnalaserthrproleuglyalaleualaargasnleuphe260265270serleuhisalaglnalaaspalagluhisgluglyleuasppheser275280285serileglnlysleutyrargglylysasp290295<210>33<211>382<212>prt<213>ralstoniseutropha<400>33metalapheiletyrtyrleuthrhisilehisleuasppheglyala151015valserleuleulysserglucysgluargileglyileargargpro202530leuleuvalthrasplysglyvalvalalaalaglyvalalaglnarg354045alaileaspalametglnglyleuglnvalalavalpheaspgluthr505560proserasnprothrglualametvalarglysalaalaalaglntyr65707580argglualaglycysaspglyleuvalalavalglyglyglyserser859095ileaspleualalysglyilealaileleualathrhisgluglyglu100105110leuthrthrtyralathrilegluglyglyseralaargilethrasp115120125lysalaalaproleuilealavalprothrthrserglythrglyser130135140gluvalalaargglyalaileileileleuaspaspglyarglysleu145150155160glyphehissertrphisleuleuprolysseralavalcysasppro165170175gluleuthrleuglyleuproalaglyleuthralaalathrglymet180185190aspalailealahiscysilegluthrpheleualaproalapheasn195200205proproalaaspglyilealaleuaspglyleugluargglytrpgly210215220hisilegluargalathrargaspglyglnaspargaspalaargleu225230235240asnmetmetseralasermetglnglyalametalapheglnlysgly245250255leuglycysvalhisserleuserhisproleuglyglyleulysile260265270aspglyargthrglyleuhishisglythrleuasnalavalvalmet275280285proalavalleuargpheasnalaaspalaprothrvalvalargasp290295300aspargtyralaargleuargargalamethisleuproaspglyala305310315320aspilealaglnalavalhisaspmetthrvalargleuglyleupro325330335thrglyleuargglnmetglyvalthrgluaspmetpheasplysval340345350ilealaglyalaleuvalasphiscyshislysthrasnprolysglu355360365alaseralaalaasptyrargargmetleugluglnsermet370375380<210>34<211>371<212>prt<213>克鲁维梭菌<400>34metlysleuleulysleualaproaspvaltyrlyspheaspthrala151015glugluphemetlystyrphelysvalglylysglyasppheileleu202530thrasnglupheleutyrlyspropheleuglulyspheasnaspgly354045alaaspalavalpheglnglulystyrglyleuglygluproserasp505560glumetileasnasnileilelysaspileglyasplysglntyrasn65707580argileilealavalglyglyglyservalileaspilealalysile859095leuserleulystyrthraspaspserleuaspleuphegluglylys100105110valproleuvallysasnlysgluleuileilevalprothrthrcys115120125glythrglysergluvalthrasnvalservalalagluleulysarg130135140arghisthrlyslysglyilealaseraspgluleutyralathrtyr145150155160alavalleuvalproglupheilelysglyleuprotyrlysphephe165170175valthrserservalaspalaleuilehisalathrglualatyrval180185190serproasnalaasnprotyrthraspmetpheservallysalamet195200205gluleuileleuasnglytyrmetglnmetvalglulysglyasnasp210215220tyrargvalgluileilegluaspphevalileglyserasntyrala225230235240glyilealapheglyasnalaglyvalglyalavalhisalaleuser245250255tyrproileglyglyasntyrhisvalprohisglyglualaasntyr260265270leuphephethrgluilephelysthrtyrtyrglulysasnproasn275280285glylysilelysaspvalasnlysleuleualaglyileleulyscys290295300aspgluserglualatyraspserleuserglnleuleuasplysleu305310315320leuserarglysproleuargglutyrglymetlysgluglugluile325330335gluthrphealaaspservalilegluglyglnglnargleuleuval340345350asnasntyrglupropheserarggluaspilevalasnthrtyrlys355360365lysleutyr370<210>35<211>538<212>prt<213>产琥珀酸曼氏杆菌(mannheimiasucciniciproducens)<400>35metthraspleuasnglnleuthrglngluleuglyalaleuglyile151015hisaspvalglngluvalvaltyrasnprosertyrgluleuleuphe202530alaglugluthrlysproglyleugluglytyrglulysglythrval354045thrasnglnglyalavalalavalasnthrglyilephethrglyarg505560serprolysasplystyrilevalleuaspasplysthrlysaspthr65707580valtrptrpthrserglulysvallysasnaspasnlysprometser859095glnaspthrtrpasnserleulysglyleuvalalaaspglnleuser100105110glylysargleuphevalvalaspalaphecysglyalaasnlysasp115120125thrargleualavalargvalvalthrgluvalalatrpglnalahis130135140phevalthrasnmetpheileargproseralaglugluleulysgly145150155160phelysproaspphevalvalmetasnglyalalyscysthrasnpro165170175asntrplysgluglnglyleuasnsergluasnphevalalapheasn180185190ilethrgluglyvalglnleuileglyglythrtrptyrglyglyglu195200205metlyslysglymetphesermetmetasntyrpheleuproleuarg210215220glyilealasermethiscysseralaasnvalglylysaspglyasp225230235240thralailephepheglyleuserglythrglylysthrthrleuser245250255thraspprolysargglnleuileglyaspaspgluhisglytrpasp260265270aspgluglyvalpheasnphegluglyglycystyralalysthrile275280285asnleuseralagluasngluproaspiletyrglyalailelysarg290295300aspalaleuleugluasnvalvalvalleuaspasnglyaspvalasp305310315320tyralaaspglyserlysthrgluasnthrargvalsertyrproile325330335tyrhisileglnasnilevallysprovalserlysalaglyproala340345350thrlysvalilepheleuseralaaspalapheglyvalleupropro355360365valserlysleuthrprogluglnthrlystyrtyrpheleusergly370375380phethralalysleualaglythrgluargglyilethrgluprothr385390395400prothrpheseralacyspheglyalaalapheleuserleuhispro405410415thrglntyralagluvalleuvallysargmetglngluserglyala420425430glualatyrleuvalasnthrglytrpasnglythrglylysargile435440445serilelysaspthrargglyileileaspalaileleuaspglyser450455460ileasplysalaglumetglyserleuproilepheasppheserile465470475480prolysalaleuproglyvalasnproalaileleuaspproargasp485490495thrtyralaasplysalaglntrpgluglulysalaglnaspleuala500505510glyargphevallysasnpheglulystyrthrglythralaglugly515520525glnalaleuvalalaalaglyprolysala530535<210>36<211>532<212>prt<213>产琥珀酸厌氧螺菌(anaerobiospirillumsucciniciproducens)<400>36metserleusergluserleualalystyrglyilethrglyalathr151015asnilevalhisasnproserhisglugluleuphealaalagluthr202530glnalaserleugluglypheglulysglythrvalthrglumetgly354045alavalasnvalmetthrglyvaltyrthrglyargserprolysasp505560lyspheilevallysasnglualaserlysgluiletrptrpthrser65707580aspgluphelysasnaspasnlysprovalthrgluglualatrpala859095glnleulysalaleualaglylysgluleuserasnlysproleutyr100105110valvalaspleuphecysglyalaasngluasnthrargleulysile115120125argphevalmetgluvalalatrpglnalahisphevalthrasnmet130135140pheileargprothrgluglugluleulysglyphegluproaspphe145150155160valvalleuasnalaserlysalalysvalgluasnphelysgluleu165170175glyleuasnsergluthralavalvalpheasnleualaglulysmet180185190glnileileleuasnthrtrptyrglyglyglumetlyslysglymet195200205phesermetmetasnphetyrleuproleuglnglyilealaalamet210215220hiscysseralaasnthraspleugluglylysasnthralailephe225230235240pheglyleuserglythrglylysthrthrleuserthraspprolys245250255argleuleuileglyaspaspgluhisglytrpaspaspaspglyval260265270pheasnphegluglyglycystyralalysvalileasnleuserlys275280285gluasngluproaspiletrpglyalailelysargasnalaleuleu290295300gluasnvalthrvalaspalaasnglylysvalaspphealaasplys305310315320servalthrgluasnthrargvalsertyrproilephehisilelys325330335asnilevallysprovalserlysalaproalaalalysargvalile340345350pheleuseralaaspalapheglyvalleuproprovalserileleu355360365serlysgluglnthrlystyrtyrpheleuserglyphethralalys370375380leualaglythrgluargglyilethrgluprothrprothrpheser385390395400sercyspheglyalaalapheleuthrleuproprothrlystyrala405410415gluvalleuvallysargmetglualaserglyalalysalatyrleu420425430valasnthrglytrpasnglythrglylysargileserilelysasp435440445thrargglyileileaspalaileleuaspglyserileaspthrala450455460asnthralathrileprotyrpheasnphethrvalprothrgluleu465470475480lysglyvalaspthrlysileleuaspproargasnthrtyralaasp485490495alaserglutrpgluvallysalalysaspleualagluargphegln500505510lysasnphelyslysphegluserleuglyglyaspleuvallysala515520525glyproglnleu530<210>37<211>538<212>prt<213>产琥珀酸放线杆菌(actinobacillussuccinogenes)<400>37metthraspleuasnlysleuvallysgluleuasnaspleuglyleu151015thraspvallysgluilevaltyrasnprosertyrgluglnleuphe202530gluglugluthrlysproglyleugluglypheasplysglythrleu354045thrthrleuglyalavalalavalaspthrglyilephethrglyarg505560serprolysasplystyrilevalcysaspgluthrthrlysaspthr65707580valtrptrpasnserglualaalalysasnaspasnlysprometthr859095glngluthrtrplysserleuarggluleuvalalalysglnleuser100105110glylysargleuphevalvalgluglytyrcysglyalaserglulys115120125hisargileglyvalargmetvalthrgluvalalatrpglnalahis130135140phevallysasnmetpheileargprothraspglugluleulysasn145150155160phelysalaaspphethrvalleuasnglyalalyscysthrasnpro165170175asntrplysgluglnglyleuasnsergluasnphevalalapheasn180185190ilethrgluglyileglnleuileglyglythrtrptyrglyglyglu195200205metlyslysglymetphesermetmetasntyrpheleuproleulys210215220glyvalalasermethiscysseralaasnvalglylysaspglyasp225230235240valalailephepheglyleuserglythrglylysthrthrleuser245250255thraspprolysargglnleuileglyaspaspgluhisglytrpasp260265270gluserglyvalpheasnphegluglyglycystyralalysthrile275280285asnleuserglngluasngluproaspiletyrglyalaileargarg290295300aspalaleuleugluasnvalvalvalargalaaspglyservalasp305310315320pheaspaspglyserlysthrgluasnthrargvalsertyrproile325330335tyrhisileaspasnilevalargprovalserlysalaglyhisala340345350thrlysvalilepheleuthralaaspalapheglyvalleupropro355360365valserlysleuthrprogluglnthrglutyrtyrpheleusergly370375380phethralalysleualaglythrgluargglyvalthrgluprothr385390395400prothrpheseralacyspheglyalaalapheleuserleuhispro405410415ileglntyralaaspvalleuvalgluargmetlysalaserglyala420425430glualatyrleuvalasnthrglytrpasnglythrglylysargile435440445serilelysaspthrargglyileileaspalaileleuaspglyser450455460ileglulysalaglumetglygluleuproilepheasnleualaile465470475480prolysalaleuproglyvalaspproalaileleuaspproargasp485490495thrtyralaasplysalaglntrpglnvallysalagluaspleuala500505510asnargphevallysasnphevallystyrthralaasnprogluala515520525alalysleuvalglyalaglyprolysala530535<210>38<211>618<212>prt<213>ralstoniseutropha<400>38metasnhisprosermetglnglythrthralaleuasnvalproala151015trpvalargasnglnlysleuvalalatrpvalalagluilealaala202530leuthrlysprogluargilehistrpcysaspglyserglngluglu354045tyraspargleucysgluglnmetvalalaalaglythrleulysarg505560leuasnproalalysarglysasnsertyrleualaleuserasppro65707580seraspvalalaargvalgluaspargthrpheilecysserglnlys859095lysgluaspalaglyprothrasnasntrpvalalaproalaglumet100105110argthrthrleuasnglyleupheaspglycysmetargglyargthr115120125leutyrvalvalprophesermetglyproleuglyserproileala130135140hisileglyvalgluleuseraspserprotyrvalalavalasnmet145150155160argilemetthrargmetglylysalavaltyraspvalleuglythr165170175aspglyaspphevalprocysvalhisthrvalglylysproleuala180185190alaglyglulysaspvalprotrpprocysasnprothrlystyrile195200205valhispheprogluserarggluiletrpserpheglyserglytyr210215220glyglyasnalaleuleuglylyslyscysphealaleuargileala225230235240serthrmetglyargaspgluglytrpleualagluhismetleuile245250255leuglyvalthrserprogluglylyslysphehisvalalaalaala260265270pheproseralacysglylysthrasnphealametleuilepropro275280285lysglyphegluglytrplysvalthrthrileglyaspaspileala290295300trpilelysproglylysaspglyargleutyralaileasnproglu305310315320alaglytyrpheglyvalalaproglythrserglulysthrasnphe325330335asnalametalathrleulysgluasnvalilephethrasnvalala340345350leuthraspaspglyaspvaltrptrpgluglymetthrlysgluala355360365proalahisleuthrasptrpglnglylysasptrpthrprogluile370375380alalysalathrglyalalysalaalahisproasnalaargphethr385390395400alaproalaserglncysproserileaspgluasntrpaspasnpro405410415alaglyvalproileaspalapheilepheglyglyargargserthr420425430thrvalproleuvalthrglualaargasntrpthrgluglyvaltyr435440445metalaalathrmetglysergluthrthralaalaalaalaglygln450455460glnglyvalvalargargaspprophealametleuprophecysgly465470475480tyrasnmetserasptyrpheglyhistrpleualaleuglyglnlys485490495leuglualaalaglyalalysleuprolysiletyrcysvalasntrp500505510phearglysaspalaaspglyasnphevaltrpproglypheglyglu515520525asnmetargvalleusertrpmetileaspargvalgluglylysgly530535540gluglyalagluhisvalpheglythrserproargtyrgluaspleu545550555560asntrpserglyvalglupheservalalaglnphethrglnvalthr565570575serileaspalaaspalatrplysglngluleualaleuhisaspglu580585590leuphethrglnleulyshisasnleuproglnalaleualagluala595600605argalaalaleuglylysargleuglugly610615<210>39<211>540<212>prt<213>大肠杆菌<400>39metargvalasnasnglyleuthrproglngluleuglualatyrgly151015ileseraspvalhisaspilevaltyrasnprosertyraspleuleu202530tyrglnglugluleuaspproserleuthrglytyrgluargglyval354045leuthrasnleuglyalavalalavalaspthrglyilephethrgly505560argserprolysasplystyrilevalargaspaspthrthrargasp65707580thrphetrptrpalaasplysglylysglylysasnaspasnlyspro859095leuserprogluthrtrpglnhisleulysglyleuvalthrarggln100105110leuserglylysargleuphevalvalaspalaphecysglyalaasn115120125proaspthrargleuservalargpheilethrgluvalalatrpgln130135140alahisphevallysasnmetpheileargproseraspglugluleu145150155160alaglyphelysproasppheilevalmetasnglyalalyscysthr165170175asnproglntrplysgluglnglyleuasnsergluasnphevalala180185190pheasnleuthrgluargmetglnleuileglyglythrtrptyrgly195200205glyglumetlyslysglymetphesermetmetasntyrleuleupro210215220leulysglyilealasermethiscysseralaasnvalglyglulys225230235240glyaspvalalavalphepheglyleuserglythrglylysthrthr245250255leuserthraspprolysargargleuileglyaspaspgluhisgly260265270trpaspaspaspglyvalpheasnphegluglyglycystyralalys275280285thrilelysleuserlysglualagluprogluiletyrasnalaile290295300argargaspalaleuleugluasnvalthrvalarggluaspglythr305310315320ileasppheaspaspglyserlysthrgluasnthrargvalsertyr325330335proiletyrhisileaspasnilevallysprovalserlysalagly340345350hisalathrlysvalilepheleuthralaaspalapheglyvalleu355360365proprovalserargleuthralaaspglnthrglntyrhispheleu370375380serglyphethralalysleualaglythrgluargglyilethrglu385390395400prothrprothrpheseralacyspheglyalaalapheleuserleu405410415hisprothrglntyralagluvalleuvallysargmetglnalaala420425430glyalaglnalatyrleuvalasnthrglytrpasnglythrglylys435440445argileserilelysaspthrargalaileileaspalaileleuasn450455460glyserleuaspasnalagluthrphethrleuprometpheasnleu465470475480alaileprothrgluleuproglyvalaspthrlysileleuasppro485490495argasnthrtyralaserprogluglntrpglnglulysalagluthr500505510leualalysleupheileaspasnpheasplystyrthraspthrpro515520525alaglyalaalaleuvalalaalaglyprolysleu530535540<210>40<211>236<212>prt<213>乳酸乳球菌<400>40metsergluilethrglnleupheglntyrasnthrleuglyalaleu151015metalaglyleutyrgluglythrmetthrileglygluleuleulys202530hisglyaspleuglyileglythrleuaspserileaspglygluleu354045ilevalleuaspglylysalatyrglnalalysglyasplysthrile505560valgluleuthraspaspilelysvalprotyralaalavalvalpro65707580hisglnalagluvalvalphelysglnlysphethrvalserasplys859095gluleugluaspargileglusertyrpheaspglyglnasnleuphe100105110argserilelysilethrglylyspheprolysmethisvalargmet115120125ileproargalalysserglythrlysphevalgluvalserglnasn130135140glnproglutyrthrglugluasnilelysglythrilevalglyile145150155160trpthrproglumetphehisglyvalservalalaglytyrhisleu165170175hispheilesergluaspphethrpheglyglyhisvalleuaspphe180185190ileileaspasnglythrvalgluileglyalaileaspglnleuasn195200205glnserpheprovalglnasparglyspheleuphealaaspleuasp210215220ileglualaleulyslysaspileaspvalalaglu225230235<210>41<211>239<212>prt<213>嗜热链球菌<400>41metserglualailelysleupheglntyrasnthrleuglyalaleu151015metalaglyleutyrglyglythrleuthrvalglygluleuleuglu202530hisglyaspleuglyleuglythrleuaspserileaspglygluleu354045ilevalleuaspglylysalatyrglnalalysglysergluglylys505560valgluvalvalgluvalserproaspglulysvalprotyralaala65707580valvalprohisglnalagluvalilepheargglnargtyrglumet859095thrasplysgluleugluaspargileglusertyrtyraspglyval100105110asnleupheargserilelysilelysglyhisphelyshismethis115120125valargmetileprolysserasnalaaspilelysphealaaspval130135140alathrargglnproglutyrgluvalaspaspileserglythrile145150155160valglyiletrpthrproglumetphehisglyvalservalalagly165170175tyrhisleuhispheileseraspaspleuthrpheglyglyhisval180185190metaspphevalilegluasnglyileilegluvalglyprovalasp195200205glnleuaspglnargpheprovalglnaspargglntyrleupheala210215220lyspheasnvalaspglumetarglysaspilethrlysalaglu225230235<210>42<211>285<212>prt<213>短芽孢杆菌<400>42metlyslysasnileilethrserilethrserleualaleuvalala151015glyleuserleuthralaphealaalathrthralathrvalproala202530proproalalysglngluserlysproalavalalaalaasnproala354045prolysasnvalleupheglntyrserthrileasnalaleumetleu505560glyglnphegluglyaspleuthrleulysaspleulysleuarggly65707580aspmetglyleuglythrileasnaspleuaspglyglumetilegln859095metglythrlysphetyrglnileaspserthrglylysleuserglu100105110leuprogluservallysthrprophealavalthrthrhispheglu115120125prolysglulysthrthrleuthrasnvalglnasptyrasnglnleu130135140thrlysmetleugluglulysphegluasnlysasnvalphetyrala145150155160vallysleuthrglythrphelysmetvallysalaargthrvalpro165170175lysglnthrargprotyrproglnleuthrgluvalthrlyslysgln180185190sergluphegluphelysasnvallysglythrleuileglyphetyr195200205thrproasntyralaalaalaleuasnvalproglyphehisleuhis210215220pheilethrgluasplysthrserglyglyhisvalleuasnleugln225230235240pheaspasnalaasnleugluileserproilehisglupheaspval245250255glnleuprohisthraspaspphealahisseraspleuthrglnval260265270thrthrserglnvalhisglnalaglusergluarglys275280285<210>43<211>259<212>prt<213>产气肠杆菌<400>43metmetmethisserseralacysaspcysglualaserleucysglu151015thrleuargglypheseralaglnhisproaspservaliletyrgln202530thrserleumetseralaleuleuserglyvaltyrvalglygluthr354045thrilealaaspleuleualahisglyasppheglyleuglythrphe505560asngluleuaspglyglumetilealapheserserglnvaltyrgln65707580leuargalaaspglyseralaargalaalalysprogluglnlysthr859095prophealavalmetthrtrppheglnproglntyrarglysthrphe100105110asnglyprovalserargglnglnilehisaspvalileaspglngln115120125ileproseraspasnleuphecysvalargileaspglyasnphearg130135140hisalahisthrargthrvalproargglnthrproprotyrargala145150155160metthraspvalleuaspaspglnprovalpheargpheasnglnarg165170175gluglyvalleuvalglypheargthrproglnhismetglnglyile180185190asnvalalaglytyrhisgluhispheilethraspaspargglngly195200205glyglyhisleuleuasptyrglnleugluserglyvalleuthrphe210215220glygluilehislysleumetileaspleuproalaaspseralaphe225230235240leuglnalaasnleuhisproserasnleuaspalaalaileargala245250255valgluasn<210>44<211>1231<212>prt<213>结核分枝杆菌<400>44metalaasnileserserpropheglyglnasnglutrpleuvalglu151015glumettyrarglyspheargaspaspproserservalaspproser202530trphisglupheleuvalasptyrserprogluprothrserglnpro354045alaalagluprothrargvalthrserproleuvalalagluargala505560alaalaalaalaproglnalaproprolysproalaaspthralaala65707580alaglyasnglyvalvalalaalaleualaalalysthralavalpro859095proproalagluglyaspgluvalalavalleuargglyalaalaala100105110alavalvallysasnmetseralaserleugluvalprothralathr115120125servalargalavalproalalysleuleuileaspasnargileval130135140ileasnasnglnleulysargthrargglyglylysileserphethr145150155160hisleuleuglytyralaleuvalglnalavallyslyspheproasn165170175metasnarghistyrthrgluvalaspglylysprothralavalthr180185190proalahisthrasnleuglyleualaileaspleuglnglylysasp195200205glylysargserleuvalvalalaglyilelysargcysgluthrmet210215220argphealaglnphevalthralatyrgluaspilevalargargala225230235240argaspglylysleuthrthrgluaspphealaglyvalthrileser245250255leuthrasnproglythrileglythrvalhisservalproargleu260265270metproglyglnglyalaileileglyvalglyalametglutyrpro275280285alaglupheglnglyalaserglugluargilealagluleuglyile290295300glylysleuilethrleuthrserthrtyrasphisargileilegln305310315320glyalagluserglyasppheleuargthrilehisgluleuleuleu325330335seraspglyphetrpaspgluvalphearggluleuserileprotyr340345350leuprovalargtrpserthraspasnproaspserilevalasplys355360365asnalaargvalmetasnleuilealaalatyrargasnargglyhis370375380leumetalaaspthraspproleuargleuasplysalaargphearg385390395400serhisproaspleugluvalleuthrhisglyleuthrleutrpasp405410415leuaspargvalphelysvalaspglyphealaglyalaglntyrlys420425430lysleuargaspvalleuglyleuleuargaspalatyrcysarghis435440445ileglyvalglutyralahisileleuaspprogluglnlysglutrp450455460leugluglnargvalgluthrlyshisvallysprothrvalalagln465470475480glnlystyrileleuserlysleuasnalaalaglualaphegluthr485490495pheleuglnthrlystyrvalglyglnlysargpheserleuglugly500505510alagluservalileprometmetaspalaalaileaspglncysala515520525gluhisglyleuaspgluvalvalileglymetprohisargglyarg530535540leuasnvalleualaasnilevalglylysprotyrserglnilephe545550555560thrgluphegluglyasnleuasnproserglnalahisglysergly565570575aspvallystyrhisleuglyalathrglyleutyrleuglnmetphe580585590glyaspasnaspileglnvalserleuthralaasnproserhisleu595600605glualavalaspprovalleugluglyleuvalargalalysglnasp610615620leuleuasphisglyserileaspseraspglyglnargalapheser625630635640valvalproleumetleuhisglyaspalaalaphealaglyglngly645650655valvalalagluthrleuasnleualaasnleuproglytyrargval660665670glyglythrilehisileilevalasnasnglnileglyphethrthr675680685alaproglutyrserargserserglutyrcysthraspvalalalys690695700metileglyalaproilephehisvalasnglyaspaspprogluala705710715720cysvaltrpvalalaargleualavalasppheargglnargphelys725730735lysaspvalvalileaspmetleucystyrargargargglyhisasn740745750gluglyaspaspprosermetthrasnprotyrvaltyraspvalval755760765aspthrlysargglyalaarglyssertyrthrglualaleuilegly770775780argglyaspilesermetlysglualagluaspalaleuargasptyr785790795800glnglyglnleugluargvalpheasngluvalarggluleuglulys805810815hisglyvalglnprosergluservalgluseraspglnmetilepro820825830alaglyleualathralavalasplysserleuleualaargilegly835840845aspalapheleualaleuproasnglyphethralahisproargval850855860glnprovalleuglulysargargglumetalatyrgluglylysile865870875880asptrpalapheglygluleuleualaleuglyserleuvalalaglu885890895glylysleuvalargleuserglyglnaspserargargglythrphe900905910serglnarghisservalleuileasparghisthrglyglugluphe915920925thrproleuglnleuleualathrasnseraspglyserprothrgly930935940glylyspheleuvaltyraspserproleuserglutyralaalaval945950955960glypheglutyrglytyrthrvalglyasnproaspalavalvalleu965970975trpglualaglnpheglyaspphevalasnglyalaglnserileile980985990aspglupheileserserglyglualalystrpglyglnleuserasn99510001005valvalleuleuleuprohisglyhisgluglyglnglyproasp101010151020histhrseralaargilegluargpheleuglnleutrpalaglu102510301035glysermetthrilealametproserthrproserasntyrphe104010451050hisleuleuargarghisalaleuaspglyileglnargproleu105510601065ilevalphethrprolyssermetleuarghislysalaalaval107010751080sergluilelysaspphethrgluilelyspheargservalleu108510901095glugluprothrtyrgluaspglyileglyaspargasnlysval110011051110serargileleuleuthrserglylysleutyrtyrgluleuala111511201125alaarglysalalysaspasnargasnaspleualailevalarg113011351140leugluglnleualaproleuproargargargleuarggluthr114511501155leuaspargtyrgluasnvallysgluphephetrpvalglnglu116011651170gluproalaasnglnglyalatrpproargpheglyleugluleu117511801185progluleuleuproasplysleualaglyilelysargileser119011951200argargalametseralaproserserglyserserlysvalhis120512101215alavalgluglnglngluileleuaspglualaphegly122012251230<210>45<211>985<212>prt<213>bradyrhizobiumjaponicum<400>45metserargglnaspalaasnalaalaphealaleuserserpheleu151015glnglythrasnalathrtyrileaspgluiletyralaargtyrglu202530lysaspproserservalaspalaglutrpglngluphephelysser354045leulysaspglnproaspaspvalargargasnalagluglyproser505560trpgluargalaasntrpproleuthrproglnaspaspleuthrser65707580alaleuaspglyasntrpalagluvalglulysalavalglyglylys859095ilealaalalysalaglnalalysglyalaaspileserseralaasp100105110leuleuglnalathrargaspservalargalaleumetleuilearg115120125sertyrargmetargglyhisphehisalalysleuaspproleugly130135140ileglualaproargasnargglugluleuaspproargthrtyrgly145150155160pheserglualaasppheasparglysilepheleuasphisvalleu165170175glyleuglutyrglythrleuarggluilethralailecysgluarg180185190thrtyrcysglnthrleuglyvalgluphemethisileserasnala195200205alaglnlysalatrpileglngluargilegluglyproasplysglu210215220ileserphethrarggluglyargargalaileleuthrlysleuval225230235240glualagluglypheglulysphecysaspthrlysphethrglythr245250255lysargpheglyleuaspglyalagluserleuileproalaleuglu260265270glnileilelysargglyglyasnleuglyvallysgluilevalleu275280285glymetprohisargglyargleuasnvalleuthrglnvalmetgly290295300lysalahisargalaleuphehisgluphelysglyglyseralaasn305310315320proaspalavalgluglyserglyaspvallystyrhisleuglyala325330335serseraspargglupheaspglyasnargilehisleuserleuthr340345350alaasnproserhisleugluilevalaspprovalvalleuglylys355360365valargalalysglnaspglnhisglyaspproproaspmetargile370375380servalmetproleuleumethisglyaspalaalaphealaglygln385390395400glyvalvalalaglucyspheglyleuseraspleulysglytyrarg405410415thrglyglyservalhispheilevalasnasnglnileglyphethr420425430thrtyrproargtyrserargserserprotyrproseraspvalala435440445lysmetileaspalaproilephehisvalasnglyaspaspproglu450455460alavalvalphealaalalysvalalathrglupheargglnlysphe465470475480hislysprovalvalileaspmetphecystyrargarghisglyhis485490495asngluglyaspgluproalaphethrglnprovalmettyrlyslys500505510ilealaalahisproserthrleugluleutyralaargargleuile515520525sergluglyvalmetthrgluglygluvalasplysalalysalaasp530535540trpargalaargleuaspalaglupheglualaglythrsertyrlys545550555560proasnlysalaasptrpleuaspglylystrpalaglyphelysile565570575alaaspglnglugluaspalaargargglyvalthrglyvalaspile580585590thralaleulysaspileglyarglysilethrlysvalproaspgly595600605pheargvalhisargthrileglnargpheleugluasnargserlys610615620alaileaspserglyalaglyileasptrpalathrglyglualaleu625630635640alaphecysserleuleuasngluasnhishisvalargleusergly645650655glnaspsergluargglythrpheserglnarghisservalleuile660665670aspglngluaspgluserargtyrthrpropheasnhisleuglyhis675680685gluglnglyhistyrgluvalileasnserleuleuserglugluala690695700valleuglypheglutyrglytyrserleualagluproasnthrleu705710715720thrleutrpglualaglnpheglyaspphealaasnglyalaglnval725730735valpheaspglnpheileserserglygluarglystrpleuargmet740745750serglyleuvalcysleuleuprohisglytyrgluglyglnglypro755760765gluhisserseralaargleugluargtyrleuglnmetcysalaglu770775780aspasnmetglnvalvaltyrprothrthrproalaasntyrphehis785790795800valleuargargglnleuhisarggluilearglysproleuileleu805810815metthrprolysserleuleuarghislysargalavalserargleu820825830glugluleualalysglythrthrphehisargileleutyraspasp835840845alaglnmetleuprothraspalailelysleuvalproaspglulys850855860ileargargilevalleucysserglylysvaltyrtyraspleutyr865870875880glugluargglulysargglyileaspaspiletyrleumetargval885890895gluglnleutyrprovalproleulysalaleuvalalagluleuser900905910argphelyslysalagluvalvaltrpcysglnglugluproargasn915920925metglyalatrphispheilegluprotyrleuglutrpvalleuasn930935940glnvalasnglyvalserargargproargtyrvalglyargalaala945950955960seralaalathralathrglyleumetserlyshisglnalaglnleu965970975lysalapheleuaspglualaleuser980985<210>46<211>995<212>prt<213>rhizobiumloti<400>46metalaargglnaspglnthrasnaspglnpheserleuthrserphe151015leutyrglyglyasnalaasptyrileaspalaleutyralaalatyr202530gluaspaspproalaservalasnproglutrpglngluphepheala354045glyleulysaspaspalaglyaspvalargargasnalalysglyala505560sertrpalalysprosertrpproleuglnalaasnglygluleuval65707580seralaleuaspglyasntrpglyilevalglulyshisleuglulys859095lysvallysasplysalavalthrasnglyvalvalleuseraspala100105110aspvalhisglnalathrargaspservalargalailemetmetile115120125argalatyrargmetargglyhisleuhisalaasnleuaspproleu130135140glyilealalysproleugluasptyrasngluleuserprogluasn145150155160tyrglyphethralaalaasptyraspargproilepheleuaspasn165170175valleuglyleuglupheglythrileargglnmetleugluileleu180185190thrargthrtyrcysserthrleuglyvalgluphemethisileser195200205aspprogluglulysalatrpileglnalaargilegluglyalaasp210215220lysgluileserphethrasnthrglylyslysalaileleuglnlys225230235240leuvalglualagluglyphegluglnpheileaspvallystyrlys245250255glythrlysargpheglyleuaspglyglyglualaleuileproala260265270leugluglnilevallysargglyglyglnleuglymetlysgluile275280285valleuglymetalahisargglyargleuasnvalleuserglnval290295300metalalysprohisargalailephehisgluphelysglyglyser305310315320alaalaproaspgluvalgluglyserglyaspvallystyrhisleu325330335glyalaserseraspargglupheaspglyasnlysvalhisleuser340345350leuthralaasnproserhisleugluilevalaspprovalvalmet355360365glylysalaargalalysglnasptyrleupheglyargglyargglu370375380gluilevalproleuglugluargalalysvalleuproleuleuleu385390395400hisglyaspalaalaphealaglyglnglyvalilealagluileleu405410415glyleuserglyleuargglyhisargvalalaglythrleuhisphe420425430ileileasnasnglnileglyphethrthrasnproargpheserarg435440445serserprotyrproseraspvalalalysmetileglualaproile450455460phehisvalasnglyaspaspproglualavalvalhisalathrlys465470475480valalaileglupheargmetlysphehislysprovalvalvalasp485490495metphecystyrargargpheglyhisasngluglyaspgluproala500505510phethrglnproilemettyrargasnileargthrhislysthrthr515520525valglniletyralaaspargleuilealagluglyhisilethrgln530535540alagluleuaspglnmetlysalaasptrpargalahisleugluser545550555560glutrpgluvalglyglnhistyrlysproasnlysalaasptrpleu565570575aspglyalatrpserglyleuargthralaaspasnglnaspglugln580585590argargglylysthralavalprovallysthrleulysgluilegly595600605lyslysleuthrgluvalprolysglypheglualahislysthrile610615620ileargpheleugluasnargargglualailegluserglyglugly625630635640ileasptrpserthralaglualaleualapheglyalaileleuleu645650655aspglyasnproileargleuserglyglnaspsergluargglythr660665670pheserglnarghisservalleutyraspglnargaspgluthrarg675680685tyrileproleuasnasnleuseralaalaglnalaglytyrgluval690695700ileasnsermetleusergluglualavalleuglypheglutyrgly705710715720tyrserleualagluprolysalaleuthrleutrpglualaglnphe725730735glyaspphealaasnglyalaglnvalvalpheaspglnpheileser740745750serglygluarglystrpleuargmetserglyleuvalcysleuleu755760765prohisglytyrgluglyglnglyprogluhisserseralaargleu770775780gluargpheleuglnleucysalagluaspasnmetglnvalalaasn785790795800cysthrthrproalaasntyrphehisileleuargargglnleulys805810815argaspphearglysproleuileleumetthrprolysserleuleu820825830arghislysargalavalserthrleuprogluileserglygluser835840845serphehisargleuleutrpaspaspalaglnleuleuproasngln850855860proilelysleuthrlysaspserlysileargargvalvalleucys865870875880serglylysvaltyrtyraspleutyrglugluargglulysarggly885890895ileasnaspiletyrleuleuargvalgluglnleutyrprophepro900905910alalysalaleuilethrgluleuserargpheargasnalaglumet915920925valtrpcysglnglugluprolysasnmetglyalatrpserpheile930935940aspprotyrleuglutrpvalleualahisileaspalalyshisgln945950955960argvalargtyrthrglyargproalaalaalaserproalathrgly965970975leumetserlyshisleualaglnleualaalaleuleugluaspala980985990leuglyglu995<210>47<211>547<212>prt<213>乳酸乳球菌<400>47mettyrthrvalglyasptyrleuleuaspargleuhisgluleugly151015ileglugluilepheglyvalproglyasptyrasnleuglnpheleu202530aspglnileileserarggluaspmetlystrpileglyasnalaasn354045gluleuasnalasertyrmetalaaspglytyralaargthrlyslys505560alaalaalapheleuthrthrpheglyvalglygluleuseralaile65707580asnglyleualaglysertyralagluasnleuprovalvalgluile859095valglyserprothrserlysvalglnasnaspglylysphevalhis100105110histhrleualaaspglyaspphelyshisphemetlysmethisglu115120125provalthralaalaargthrleuleuthralagluasnalathrtyr130135140gluileaspargvalleuserglnleuleulysgluarglysproval145150155160tyrileasnleuprovalaspvalalaalaalalysalaglulyspro165170175alaleuserleuglulysgluserserthrthrasnthrthrglugln180185190valileleuserlysileglugluserleulysasnalaglnlyspro195200205valvalilealaglyhisgluvalileserpheglyleuglulysthr210215220valthrglnphevalsergluthrlysleuproilethrthrleuasn225230235240pheglylysseralavalaspgluserleuproserpheleuglyile245250255tyrasnglylysleusergluileserleulysasnphevalgluser260265270alaasppheileleumetleuglyvallysleuthraspserserthr275280285glyalaphethrhishisleuaspgluasnlysmetileserleuasn290295300ileaspgluglyileilepheasnlysvalvalgluasppheaspphe305310315320argalavalvalserserleusergluleulysglyileglutyrglu325330335glyglntyrileasplysglntyrgluglupheileproserserala340345350proleuserglnaspargleutrpglnalavalgluserleuthrgln355360365serasngluthrilevalalagluglnglythrserphepheglyala370375380serthrilepheleulysserasnserargpheileglyglnproleu385390395400trpglyserileglytyrthrpheproalaalaleuglyserglnile405410415alaasplysgluserarghisleuleupheileglyaspglyserleu420425430glnleuthrvalglngluleuglyleuserileargglulysleuasn435440445proilecyspheileileasnasnaspglytyrthrvalgluargglu450455460ilehisglyprothrglnsertyrasnaspilepromettrpasntyr465470475480serlysleuprogluthrpheglyalathrgluaspargvalvalser485490495lysilevalargthrgluasngluphevalservalmetlysgluala500505510glnalaaspvalasnargmettyrtrpilegluleuvalleuglulys515520525gluaspalaprolysleuleulyslysmetglylysleuphealaglu530535540glnasnlys545<210>48<211>1728<212>dna<213>东方伊萨酵母<400>48atgactgacaaaatctccctaggtacttatctgtttgaaaagttaaaggaagcaggctct60tattccatctttggtgttcctggtgatttcaatttggcattgttggaccacgtcaaggaa120gttgaaggcattagatgggtcggtaacgctaacgagttgaatgccggctacgaagctgat180ggttatgcaagaatcaatggatttgcatccctaatcaccacctttggtgtcggtgaattg240tctgccgtcaatgccattgcaggttcttatgctgaacacgtcccattgatccatattgtt300ggtatgccttccttgtctgctatgaagaacaacttgttgttacaccataccttgggtgac360acaagattcgacaacttcaccgaaatgtcaaagaaaatcagtgcaaaggttgaaattgtt420tacgatttggaatcagctccaaaattaattaataacttgattgaaaccgcttatcacaca480aagagaccagtctacttgggacttccttccaactttgctgatgaattggttccagcggca540ttagttaaggaaaacaagttacatttagaagaacctctaaacaaccccgttgctgaagaa600gaattcattcataacgttgttgaaatggtcaagaaggcagaaaaaccaatcattctcgtt660gacgcttgtgctgcaagacataacatttctaaggaagtgagagagttggctaaattgact720aaattccctgtcttcaccaccccaatgggtaaatctactgttgatgaagatgatgaagaa780ttctttggcttatacttgggttctctatctgctccagatgttaaggacattgttggccca840accgattgtatcttatccttaggtggtttaccttctgatttcaacaccggttccttctca900tatggttacaccactaagaatgtcgttgaattccattccaactactgtaaattcaaatct960gcaacttatgaaaacttgatgatgaagggcgcagtccaaagattgatcagcgaattgaag1020aatattaagtattccaatgtctcaactttatctccaccaaaatctaaatttgcttacgaa1080tctgcaaaggttgctccagaaggtatcatcactcaagattacctgtggaagagattatct1140tacttcttaaagccaagagatatcattgtcactgaaactggtacttcctcctttggtgtc1200ttggctacccacttaccaagagattcaaagtctatctcccaagtcttatggggttccatt1260ggtttctccttaccagctgcagttggtgctgcatttgctgctgaagatgcacacaaacaa1320actggcgaacaagaaagaagaactgttttgtttattggtgatggttctttacaattgact1380gtccaatcaatctcagatgctgcaagatggaacatcaagccatacatcttcatcttaaac1440aacagaggttacactatcgaaaagttgatccacggtcgtcatgaggactacaaccaaatt1500caaccatgggatcaccaattgttattgaagctctttgctgacaagacccaatatgaaaac1560catgttgttaaatccgctaaggacttggacgctttgatgaaggatgaagcattcaacaag1620gaagataagattagagtcattgaattattcttggatgaattcgatgctccagaaatcttg1680gttgctcaagctaaattatctgatgaaatcaactctaaagccgcttaa1728<210>49<211>575<212>prt<213>东方伊萨酵母<400>49metthrasplysileserleuglythrtyrleupheglulysleulys151015glualaglysertyrserilepheglyvalproglyasppheasnleu202530alaleuleuasphisvallysgluvalgluglyileargtrpvalgly354045asnalaasngluleuasnalaglytyrglualaaspglytyralaarg505560ileasnglyphealaserleuilethrthrpheglyvalglygluleu65707580seralavalasnalailealaglysertyralagluhisvalproleu859095ilehisilevalglymetproserleuseralametlysasnasnleu100105110leuleuhishisthrleuglyaspthrargpheaspasnphethrglu115120125metserlyslysileseralalysvalgluilevaltyraspleuglu130135140seralaprolysleuileasnasnleuilegluthralatyrhisthr145150155160lysargprovaltyrleuglyleuproserasnphealaaspgluleu165170175valproalaalaleuvallysgluasnlysleuhisleugluglupro180185190leuasnasnprovalalaglugluglupheilehisasnvalvalglu195200205metvallyslysalaglulysproileileleuvalaspalacysala210215220alaarghisasnileserlysgluvalarggluleualalysleuthr225230235240lyspheprovalphethrthrprometglylysserthrvalaspglu245250255aspaspglugluphepheglyleutyrleuglyserleuseralapro260265270aspvallysaspilevalglyprothraspcysileleuserleugly275280285glyleuproserasppheasnthrglyserphesertyrglytyrthr290295300thrlysasnvalvalgluphehisserasntyrcyslysphelysser305310315320alathrtyrgluasnleumetmetlysglyalavalglnargleuile325330335sergluleulysasnilelystyrserasnvalserthrleuserpro340345350prolysserlysphealatyrgluseralalysvalalaproglugly355360365ileilethrglnasptyrleutrplysargleusertyrpheleulys370375380proargaspileilevalthrgluthrglythrserserpheglyval385390395400leualathrhisleuproargaspserlysserileserglnvalleu405410415trpglyserileglypheserleuproalaalavalglyalaalaphe420425430alaalagluaspalahislysglnthrglygluglngluargargthr435440445valleupheileglyaspglyserleuglnleuthrvalglnserile450455460seraspalaalaargtrpasnilelysprotyrilepheileleuasn465470475480asnargglytyrthrileglulysleuilehisglyarghisgluasp485490495tyrasnglnileglnprotrpasphisglnleuleuleulysleuphe500505510alaasplysthrglntyrgluasnhisvalvallysseralalysasp515520525leuaspalaleumetlysaspglualapheasnlysgluasplysile530535540argvalilegluleupheleuaspglupheaspalaprogluileleu545550555560valalaglnalalysleuseraspgluileasnserlysalaala565570575<210>50<211>563<212>prt<213>酿酒酵母<400>50metsergluilethrleuglylystyrleuphegluargleulysgln151015valasnvalasnthrvalpheglyleuproglyasppheasnleuser202530leuleuasplysiletyrgluvalgluglymetargtrpalaglyasn354045alaasngluleuasnalaalatyralaalaaspglytyralaargile505560lysglymetsercysileilethrthrpheglyvalglygluleuser65707580alaleuasnglyilealaglysertyralagluhisvalglyvalleu859095hisvalvalglyvalproserileseralaglnalalysglnleuleu100105110leuhishisthrleuglyasnglyaspphethrvalphehisargmet115120125seralaasnilesergluthrthralametilethraspilealathr130135140alaproalagluileaspargcysileargthrthrtyrvalthrgln145150155160argprovaltyrleuglyleuproalaasnleuvalaspleuasnval165170175proalalysleuleuglnthrproileaspmetserleulysproasn180185190aspalagluserglulysgluvalileaspthrileleualaleuval195200205lysaspalalysasnprovalileleualaaspalacyscysserarg210215220hisaspvallysalagluthrlyslysleuileaspleuthrglnphe225230235240proalaphevalthrprometglylysglyserileaspgluglnhis245250255proargtyrglyglyvaltyrvalglythrleuserlysprogluval260265270lysglualavalgluseralaaspleuileleuservalglyalaleu275280285leuserasppheasnthrglyserphesertyrsertyrlysthrlys290295300asnilevalgluphehisserasphismetlysileargasnalathr305310315320pheproglyvalglnmetlysphevalleuglnlysleuleuthrthr325330335ilealaaspalaalalysglytyrlysprovalalavalproalaarg340345350thrproalaasnalaalavalproalaserthrproleulysglnglu355360365trpmettrpasnglnleuglyasnpheleuglngluglyaspvalval370375380ilealagluthrglythrseralapheglyileasnglnthrthrphe385390395400proasnasnthrtyrglyileserglnvalleutrpglyserilegly405410415phethrthrglyalathrleuglyalaalaphealaalaglugluile420425430aspprolyslysargvalileleupheileglyaspglyserleugln435440445leuthrvalglngluileserthrmetileargtrpglyleulyspro450455460tyrleuphevalleuasnasnaspglytyrthrileglulysleuile465470475480hisglyprolysalaglntyrasngluileglnglytrpasphisleu485490495serleuleuprothrpheglyalalysasptyrgluthrhisargval500505510alathrthrglyglutrpasplysleuthrglnasplysserpheasn515520525aspasnserlysileargmetilegluilemetleuprovalpheasp530535540alaproglnasnleuvalgluglnalalysleuthralaalathrasn545550555560alalysgln<210>51<211>563<212>prt<213>乳酸克鲁维酵母(kluyveromyceslactis)<400>51metsergluilethrleuglyargtyrleuphegluargleulysgln151015valgluvalglnthrilepheglyleuproglyasppheasnleuser202530leuleuaspasniletyrgluvalproglymetargtrpalaglyasn354045alaasngluleuasnalaalatyralaalaaspglytyralaargleu505560lysglymetsercysileilethrthrpheglyvalglygluleuser65707580alaleuasnglyilealaglysertyralagluhisvalglyvalleu859095hisvalvalglyvalproservalserserglnalalysglnleuleu100105110leuhishisthrleuglyasnglyaspphethrvalphehisargmet115120125serserasnilesergluthrthralametilethraspileasnthr130135140alaproalagluileaspargcysileargthrthrtyrvalsergln145150155160argprovaltyrleuglyleuproalaasnleuvalaspleuthrval165170175proalaserleuleuaspthrproileaspleuserleulysproasn180185190aspproglualagluglugluvalilegluasnvalleuglnleuile195200205lysglualalysasnprovalileleualaaspalacyscysserarg210215220hisaspalalysalagluthrlyslysleuileaspleuthrglnphe225230235240proalaphevalthrprometglylysglyserileaspglulyshis245250255proargpheglyglyvaltyrvalglythrleuserserproalaval260265270lysglualavalgluseralaaspleuvalleuservalglyalaleu275280285leuserasppheasnthrglyserphesertyrsertyrlysthrlys290295300asnilevalgluphehisserasptyrthrlysileargseralathr305310315320pheproglyvalglnmetlysphealaleuglnlysleuleuthrlys325330335valalaaspalaalalysglytyrlysprovalprovalproserglu340345350progluhisasnglualavalalaaspserthrproleulysglnglu355360365trpvaltrpthrglnvalglyglupheleuarggluglyaspvalval370375380ilethrgluthrglythrseralapheglyileasnglnthrhisphe385390395400proasnasnthrtyrglyileserglnvalleutrpglyserilegly405410415phethrthrglyalathrleuglyalaalaphealaalaglugluile420425430aspprolyslysargvalileleupheileglyaspglyserleugln435440445leuthrvalglngluileserthrmetileargtrpglyleulyspro450455460tyrleuphevalleuasnasnaspglytyrthrilegluargleuile465470475480hisglygluthralaglntyrasncysileglnasntrpglnhisleu485490495gluleuleuprothrpheglyalalysasptyrglualavalargval500505510serthrthrglyglutrpasnlysleuthrthraspglulysphegln515520525aspasnthrargileargleuilegluvalmetleuprothrmetasp530535540alaproserasnleuvallysglnalaglnleuthralaalathrasn545550555560alalysasn<210>52<211>568<212>prt<213>运动发酵单胞菌(zymomonasmobilis)<400>52metsertyrthrvalglythrtyrleualagluargleuvalglnile151015glyleulyshishisphealavalalaglyasptyrasnleuvalleu202530leuaspasnleuleuleuasnlysasnmetgluglnvaltyrcyscys354045asngluleuasncysglypheseralagluglytyralaargalalys505560glyalaalaalaalavalvalthrtyrservalglyalaleuserala65707580pheaspalaileglyglyalatyralagluasnleuprovalileleu859095ileserglyalaproasnasnasnasphisalaalaglyhisvalleu100105110hishisalaleuglylysthrasptyrhistyrglnleuglumetala115120125lysasnilethralaalaalaglualailetyrthrproglugluala130135140proalalysileasphisvalilelysthralaleuargglulyslys145150155160provaltyrleugluilealacysasnilealasermetprocysala165170175alaproglyproalaseralaleupheasnaspglualaseraspglu180185190alaserleuasnalaalavalglugluthrleulyspheilealaasn195200205argasplysvalalavalleuvalglyserlysleuargalaalagly210215220alagluglualaalavallysphealaaspalaleuglyglyalaval225230235240alathrmetalaalaalalysserphepheproglugluasnprohis245250255tyrileglythrsertrpglygluvalsertyrproglyvalglulys260265270thrmetlysglualaaspalavalilealaleualaprovalpheasn275280285asptyrserthrthrglytrpthraspileproaspprolyslysleu290295300valleualagluproargservalvalvalasnglyileargphepro305310315320servalhisleulysasptyrleuthrargleualaglnlysvalser325330335lyslysthrglyalaleuaspphephelysserleuasnalaglyglu340345350leulyslysalaalaproalaaspproseralaproleuvalasnala355360365gluilealaargglnvalglualaleuleuthrproasnthrthrval370375380ilealagluthrglyaspsertrppheasnalaglnargmetlysleu385390395400proasnglyalaargvalglutyrglumetglntrpglyhisilegly405410415trpservalproalaalapheglytyralavalglyalaprogluarg420425430argasnileleumetvalglyaspglyserpheglnleuthralagln435440445gluvalalaglnmetvalargleulysleuprovalileilepheleu450455460ileasnasntyrglytyrthrilegluvalmetilehisaspglypro465470475480tyrasnasnilelysasntrpasptyralaglyleumetgluvalphe485490495asnglyasnglyglytyraspserglyalaglylysglyleulysala500505510lysthrglyglygluleualaglualailelysvalalaleualaasn515520525thraspglyprothrleuileglucyspheileglyarggluaspcys530535540thrglugluleuvallystrpglylysargvalalaalaalaasnser545550555560arglysprovalasnlysleuleu565<210>53<211>557<212>prt<213>巴斯德醋酸杆菌(acetobacterpasteurianus)<400>53metthrtyrthrvalglymettyrleualagluargleuvalglnile151015glyleulyshishisphealavalglyglyasptyrasnleuvalleu202530leuaspglnleuleuleuasnlysaspmetlysglniletyrcyscys354045asngluleuasncysglypheseralagluglytyralaargserasn505560glyalaalaalaalavalvalthrpheservalglyalaileserala65707580metasnalaleuglyglyalatyralagluasnleuprovalileleu859095ileserglyalaproasnserasnaspglnglythrglyhisileleu100105110hishisthrileglylysthrasptyrsertyrglnleuglumetala115120125argglnvalthrcysalaalagluserilethraspalahisserala130135140proalalysileasphisvalileargthralaleuarggluarglys145150155160proalatyrleuaspilealacysasnilealasergluprocysval165170175argproglyprovalserserleuleusergluprogluileasphis180185190thrserleulysalaalavalaspalathrvalalaleuleulysasn195200205argproalaprovalmetleuleuglyserlysleuargalaalaasn210215220alaleualaalathrgluthrleualaasplysleuglncysalaval225230235240thrilemetalaalaalalysglyphepheprogluasphisalagly245250255pheargglyleutyrtrpglygluvalserasnproglyvalglnglu260265270leuvalgluthrseraspalaleuleucysilealaprovalpheasn275280285asptyrserthrvalglytrpserglymetprolysglyproasnval290295300ileleualagluproaspargvalthrvalaspglyargalatyrasp305310315320glyphethrleuargalapheleuglnalaleualaglulysalapro325330335alaargproalaseralaglnlysserservalprothrcysserleu340345350thralathrseraspglualaglyleuthrasnaspgluilevalarg355360365hisileasnalaleuleuthrserasnthrthrleuvalalagluthr370375380glyaspsertrppheasnalametargmetthrleualaglyalaarg385390395400valgluleuglumetglntrpglyhisileglytrpservalproser405410415alapheglyasnalametglyserglnaspargglnhisvalvalmet420425430valglyaspglyserpheglnleuthralaglngluvalalaglnmet435440445valargtyrgluleuprovalileilepheleuileasnasnarggly450455460tyrvalilegluilealailehisaspglyprotyrasntyrilelys465470475480asntrpasptyralaglyleumetgluvalpheasnalaglyglugly485490495hisglyleuglyleulysalathrthrprolysgluleuthrgluala500505510ilealaargalalysalaasnthrargglyprothrleuileglucys515520525glnileaspargthraspcysthraspmetleuvalglntrpglyarg530535540lysvalalaserthrasnalaarglysthrthrleuala545550555<210>54<211>528<212>prt<213>恶臭假单胞菌<400>54metalaservalhisglythrthrtyrgluleuleuargargglngly151015ileaspthrvalpheglyasnproglyserasngluleupropheleu202530lysasppheprogluasppheargtyrileleualaleuglngluala354045cysvalvalglyilealaaspglytyralaglnalaserarglyspro505560alapheileasnleuhisseralaalaglythrglyasnalametgly65707580alaleuserasnalatrpasnserhisserproleuilevalthrala859095glyglnglnthrargalametileglyvalglualaleuleuthrasn100105110valaspalaalaasnleuproargproleuvallystrpsertyrglu115120125proalaseralaalagluvalprohisalametserargalailehis130135140metalasermetalaproglnglyprovaltyrleuservalprotyr145150155160aspasptrpasplysaspalaaspproglnserhishisleupheasp165170175arghisvalserserservalargleuasnaspglnaspleuaspile180185190leuvallysalaleuasnseralaserasnproalailevalleugly195200205proaspvalaspalaalaasnalaasnalaaspcysvalmetleuala210215220gluargleulysalaprovaltrpvalalaproseralaproargcys225230235240propheprothrarghisprocyspheargglyleumetproalagly245250255ilealaalaileserglnleuleugluglyhisaspvalvalleuval260265270ileglyalaprovalpheargtyrhisglntyraspproglyglntyr275280285leulysproglythrargleuileservalthrcysaspproleuglu290295300alaalaargalaprometglyaspalailevalalaaspileglyala305310315320metalaseralaleualaasnleuvalglugluserserargglnleu325330335prothralaalaprogluproalalysvalaspglnaspalaglyarg340345350leuhisprogluthrvalpheaspthrleuasnaspmetalaproglu355360365asnalailetyrleuasngluserthrserthrthralaglnmettrp370375380glnargleuasnmetargasnproglysertyrtyrphecysalaala385390395400glyglyleuglyphealaleuproalaalaileglyvalglnleuala405410415gluprogluargglnvalilealavalileglyaspglyseralaasn420425430tyrserileseralaleutrpthralaalaglntyrasnileprothr435440445ilephevalilemetasnasnglythrtyrglyalaleuargtrpphe450455460alaglyvalleuglualagluasnvalproglyleuaspvalprogly465470475480ileasppheargalaleualalysglytyrglyvalglnalaleulys485490495alaaspasnleugluglnleulysglyserleuglnglualaleuser500505510alalysglyprovalleuilegluvalserthrvalserprovallys515520525<210>55<211>528<212>prt<213>铜绿假单胞菌<400>55metlysthrvalhisseralasertyrgluileleuargarghisgly151015leuthrthrvalpheglyasnproglyserasngluleupropheleu202530lysasppheprogluasppheargtyrileleuglyleuhisglugly354045alavalvalglymetalaaspglyphealaleualaserglyargpro505560alaphevalasnleuhisalaalaalaglythrglyasnglymetgly65707580alaleuthrasnalatrptyrserhisserproleuvalilethrala859095glyglnglnvalargsermetileglyvalglualametleualaasn100105110valaspalaglyglnleuprolysproleuvallystrpserhisglu115120125proalacysalaglnaspvalproargalaleuserglnalailegln130135140thralaserleuproproargalaprovaltyrleuserileprotyr145150155160aspasptrpalaglnproalaproalaglyvalgluhisleualaala165170175argglnvalserglyalaalaleuproalaproalaleuleualaglu180185190leuglygluargleuserargserargasnprovalleuvalleugly195200205proaspvalaspglyalaasnalaasnglyleualavalgluleuala210215220glulysleuargmetproalatrpvalalaproseralaserargcys225230235240propheprothrarghisalacyspheargglyvalleuproalaala245250255ilealaglyileserargleuleuaspglyhisaspleuileleuval260265270valglyalaprovalpheargtyrhisglnphealaproglyasptyr275280285leuproalaglyalagluleuvalglnvalthrcysaspproglyglu290295300alaalaargalaprometglyaspalaleuvalglyaspilealaleu305310315320thrleuglualaleuleugluglnvalargproseralaargproleu325330335proglualaleuproargproproalaleualaglugluglyglypro340345350leuargprogluthrvalpheaspvalileaspalaleualaproarg355360365aspalailephevallysgluserthrserthrvalthralaphetrp370375380glnargvalglumetarggluproglysertyrphepheproalaala385390395400glyglyleuglypheglyleuproalaalavalglyalaglnleuala405410415glnproargargglnvalileglyileileglyaspglyseralaasn420425430tyrglyilethralaleutrpseralaalaglntyrargvalproala435440445valpheileileleulysasnglythrtyrglyalaleuargtrpphe450455460alaglyvalleugluvalproaspalaproglyleuaspvalprogly465470475480leuaspphecysalailealaargglytyrglyvalglualaleuhis485490495alaalathrargglugluleugluglyalaleulyshisalaleuala500505510alaaspargprovalleuilegluvalprothrglnthrileglupro515520525<210>56<211>526<212>prt<213>施氏假单胞菌(pseudomonasstutzeri)<400>56metalaservalhisserilethrtyrgluleuleuargargglngly151015ileaspthrvalpheglyasnproglyserasngluleupropheleu202530lysasppheprogluasppheargtyrileleualaleuglngluala354045cysvalvalglyilealaaspglytyralaglnalaserarglyspro505560alapheileasnleuhisseralaalaglythrglyasnalametgly65707580alametserasnalatrpasncyshisserproleuilevalthrala859095glyglnglnasnargalametileglyvalglualaleuleuthrasn100105110valaspalaalaserleuproargproleuvallystrpsertyrglu115120125proalaseralaalagluvalprohisalametserargalailehis130135140metalasermetalaproargglyprovaltyrleuservalprotyr145150155160aspasptrpasplysglualaaspproglnserhishisleutyrasp165170175argservalasnseralavalargleuasnaspglnaspleugluval180185190leuvalglualaleuasnseralaserasnproalailevalleugly195200205proaspvalaspseralaasnalaasnalaaspcysvalthrleuala210215220gluargleulysalaprovaltrpvalalaproseralaproargcys225230235240propheprothrarghisprocyspheargglyleumetproalagly245250255ilealaalaileserglnleuleugluglyhisaspvalvalleuval260265270ileglyalaprovalpheargtyrhisglntyraspproglyglntyr275280285leulysproglythrargleuileserilethrcysaspproleuglu290295300alaalaargalaprometglyaspalailevalalaaspileglythr305310315320metthralaalaleualaserargileglyglusergluargglnleu325330335proalavalleuproserprogluargvalasnglnaspalaglyarg340345350leuargprogluthrvalpheaspthrleuasnglumetalaproglu355360365aspalailetyrleuasngluserthrserthrthralaglnmettrp370375380glnargleuasnmetargasnproglysertyrtyrphecysalaala385390395400glyglyleuglyphealaleuproalaalaileglyvalglnleuala405410415gluproaspargglnvalilealavalileglyaspglyseralaasn420425430tyrserileseralaleutrpthralaalahistyrasnileproala435440445ilepheleuilemetasnasnglythrtyrglyalaleuargtrpphe450455460alaglyvalleuglualagluasnvalproglyleuaspvalprogly465470475480ileaspphecysalailealalysglytyrglyileproalaleulys485490495alaaspasnleugluglnleulysglyserilehisglualaleuser500505510alalysglyprovalleuilegluvalserthrvalserleu515520525<210>57<211>528<212>prt<213>荧光假单胞菌<400>57metlysthrvalhisseralasertyraspileleuargglnglngly151015leuthrthrvalpheglyasnproglyserasngluleupropheleu202530lysglypheprogluasppheargtyrileleuglyleuhisglugly354045alavalvalglymetalaaspglyphealaleualaserglyglnpro505560alaphevalasnleuhisalaalaalaglythrglyasnglymetgly65707580alaleuthrasnalatrptyrserhisserproleuvalilethrala859095glyglnglnvalargsermetileglyvalglualametleualaasn100105110valaspalaproglnleuprolysproleuvallystrpseralaglu115120125proalacysalagluaspvalproargalaleuserglnalailehis130135140metalaasnglnalaprolysglyprovaltyrleuserileprotyr145150155160aspasptrpalaargproalaproalaglyvalgluhisleualaarg165170175argglnvalalathralaglyleuproseralaalaglnleuargser180185190leuvalglnargleualaalaalaargasnprovalleuvalleugly195200205proaspvalaspglyserargserasnhisleualavalglnleuala210215220glulysleuargmetproalatrpvalalaproseralaserargcys225230235240propheprothrarghisproserpheargglyvalleuproalaala245250255ilealaglyileserargcysleualaasphisaspleuileleuval260265270valglyalaprovalpheargtyrhisglnphealaproglyasptyr275280285leuproalaglythrgluleuleuhisilethrcysaspproglyglu290295300alaalaargalaprometglyaspalaleuvalglyaspilevalglu305310315320thrleuglnalaleuvaltrpalaleuproaspcysaspargprogln325330335proglnalaleuproproalaalaprovalglugluleuglyglyleu340345350leuargprogluthrvalpheaspvalileaspgluleualaprolys355360365aspalailetyrvallysgluserthrserthrvalglyalaphetrp370375380glnargvalglumetarggluproglysertyrtyrpheproalaala385390395400glyglyleuglypheglyleuproalaalavalglyvalglnleuala405410415argprogluargargvalileglyvalileglyaspglyseralaasn420425430tyrglyilethralaleutrpthralaalaglntyrglnileproval435440445valpheileileleulysasnglythrtyrglyalaleuargtrpphe450455460alaglyvalleuglnvalseraspalaproglyleuaspvalprogly465470475480leuaspphecysalaileglyargglytyrglyvalhisservalgln485490495alaasnthrargglualaphealaglnalaleuserglualaleuala500505510glyaspargprovalleuilegluvalprothrleuthrileglupro515520525<210>58<211>1220<212>prt<213>橙色绿屈挠菌(chloroflexusaurantiacus)<400>58metalathrglyglusermetserglythrglyargleualaglylys151015ilealaleuilethrglyglyalaglyasnileglysergluleuthr202530argargpheleualagluglyalathrvalileileserglyargasn354045argalalysleuthralaleualaglumetglnalaglualaglyval505560proalalysargileaspleugluvalmetaspglyseraspproval65707580alavalargalaglyileglualailevalalaarghisglyglnile859095aspileleuvalasnasnalaglyseralaglyalaglnargargleu100105110alagluileproleuthrglualagluleuglyproglyalagluglu115120125thrleuhisalaserilealaasnleuleuglymetglytrphisleu130135140metargilealaalaprohismetprovalglyseralavalileasn145150155160valserthrilepheserargalaglutyrtyrglyargileprotyr165170175valthrprolysalaalaleuasnalaleuserglnleualaalaarg180185190gluleuglyalaargglyileargvalasnthrilepheproglypro195200205ilegluseraspargileargthrvalpheglnargmetaspglnleu210215220lysglyargprogluglyaspthralahishispheleuasnthrmet225230235240argleucysargalaasnaspglnglyalaleugluargargphepro245250255servalglyaspvalalaaspalaalavalpheleualaseralaglu260265270seralaalaleuserglygluthrilegluvalthrhisglymetglu275280285leuproalacyssergluthrserleuleualaargthraspleuarg290295300thrileaspalaserglyargthrthrleuilecysalaglyaspgln305310315320ileglugluvalmetalaleuthrglymetleuargthrcysglyser325330335gluvalileileglypheargseralaalaalaleualaglnpheglu340345350glnalavalasngluserargargleualaglyalaaspphethrpro355360365proilealaleuproleuaspproargaspproalathrileaspala370375380valpheasptrpalaglygluasnthrglyglyilehistrpileleu385390395400proalathrserhisgluproalaprocysvalilegluvalaspasp405410415gluargvalleuasnpheleualaaspgluilethrglythrileval420425430ilealaserargleualaargtyrtrpglnserglnargleuthrpro435440445glyalaargalaargglyproargvalilepheleuserasnglyala450455460aspglnasnglyasnvaltyrglyargileglnseralaalailegly465470475480glnleuileargvaltrparghisglualagluleuasptyrglnarg485490495alaseralaalaglyasphisvalleuproprovaltrpalaasngln500505510ilevalargphealaasnargserleugluglyleugluphealacys515520525alatrpthralaglnleuleuhisserglnarghisileasngluile530535540thrleuasnileproalaasnileseralathrthrglyalaargser545550555560alaservalglytrpalagluserleuileglyleuhisleuglylys565570575valalaleuilethrglyglyseralaglyileglyglyglnilegly580585590argleuleualaleuserglyalaargvalmetleualaalaargasp595600605arghislysleugluglnmetglnalametileglnsergluleuala610615620gluvalglytyrthraspvalgluaspargvalhisilealaprogly625630635640cysaspvalserserglualaglnleualaaspleuvalgluargthr645650655leuseralapheglythrvalasptyrleuileasnasnalaglyile660665670alaglyvalgluglumetvalileaspmetprovalgluglytrparg675680685histhrleuphealaasnleuileserasntyrserleumetarglys690695700leualaproleumetlyslysglnglyserglytyrileleuasnval705710715720sersertyrpheglyglyglulysaspalaalaileprotyrproasn725730735argalaasptyralavalserlysalaglyglnargalametalaglu740745750valphealaargpheleuglyprogluileglnileasnalaileala755760765proglyprovalgluglyaspargleuargglythrglygluargpro770775780glyleuphealaargargalaargleuileleugluasnlysargleu785790795800asngluleuhisalaalaleuilealaalaalaargthraspgluarg805810815sermethisgluleuvalgluleuleuleuproasnaspvalalaala820825830leugluglnasnproalaalaprothralaleuarggluleualaarg835840845argpheargsergluglyaspproalaalaserserserseralaleu850855860leuasnargserilealaalalysleuleualaargleuhisasngly865870875880glytyrvalleuproalaaspilephealaasnleuproasnpropro885890895aspprophephethrargalaglnileaspargglualaarglysval900905910argaspglyilemetglymetleutyrleuglnargmetprothrglu915920925pheaspvalalametalathrvaltyrtyrleualaaspargasnval930935940serglygluthrphehisproserglyglyleuargtyrgluargthr945950955960prothrglyglygluleupheglyleuproserprogluargleuala965970975gluleuvalglyserthrvaltyrleuileglygluhisleuthrglu980985990hisleuasnleuleualamettyrleugluargtyrglyalaarggln99510001005valtrpilevalgluthrgluthrglyalagluthrmetargarg101010151020leuleuhisasphisvalglualaglyargleumetthrileval102510301035alaglyaspglnileglualaalaileaspglnalailethrarg104010451050tyrglyargproglyprovalvalcysthrpropheargproleu105510601065prothrvalproleuvalglyarglysaspserasptrpserthr107010751080valleuserglualagluphealagluleucysgluhisglnleu108510901095thrhishispheargvalalaarglysilealaleuseraspgly110011051110alaserleualaleuvalthrprogluthrthralathrserthr111511201125thrgluglnphealaleualaasnpheilelysthrthrleuhis113011351140alaphethralathrileglyvalglusergluargthralagln114511501155argileleuileasnglnvalaspleuthrargargalaargala116011651170glugluproargaspprohisgluargglnglngluleugluarg117511801185pheileglualavalleuleuvalthralaproleuproproglu119011951200alaaspthrargtyralaglyargilehisargglyargalaile120512101215thrval1220<210>59<211>1229<212>prt<213>roseiflexuscastenholzii<400>59metserthrvalargargleugluglylysvalalaleuilethrgly151015glyalaglyasnileglygluvalilethrargargpheleualaglu202530glyalathrvalvalilethrglyargasnalaglulysleualaval354045tyrargargargleuileaspglugluargvalalaprogluargval505560valalaleuargmetaspglyseraspilealaglnvalargalagly65707580valalaglnilevalhisglyglythraspvalproileproleuhis859095argileaspileleuvalasnasnalaglyseralaglyproargarg100105110argleuvalaspileproleugluprosergluvalglnproproasp115120125sergluthrleualaglnalavalglyasnleuvalglyilethrtrp130135140asnleuthrargalaalaalaprohismetproserglyserserval145150155160ileasnileserthrilepheserargthrasptyrtyrglyargile165170175alatyrvalalaprolysalaalaleuasnalaleuseraspglyleu180185190alaarggluleuglyvalargglyileargvalasnthriletyrpro195200205glyproileglusergluargiletyrthrmetpheglnalametasp210215220alaleulysglyglnprogluglyaspthralaserglypheleuarg225230235240metmetargleuserargileaspglnasnglygluvalvallysarg245250255pheproserprovalaspvalalaasnthralavalpheleualaser260265270aspgluseralaalaphethrglyhisalaphegluvalthrhisgly275280285metgluvalprothrgluserargthrthrphevalserargprogly290295300leuargservalaspalathrglylysvalileleuilecysalagly305310315320aspglnvalaspaspalavalalaleualaaspthrleuargsercys325330335argalathrvalvalileglypheargaspproargalaleuglulys340345350alaservalleuleuarggluproarghisalaleualaalaaspmet355360365tyrglyargprothrmetthralaglualaargleuvalargleuasp370375380proleuaspproargalaalaalaglnthrleugluglnilehisala385390395400gluleuglyalailehishisalavalvalleuproglyglnserarg405410415hisalaproseralaserleuilegluvalaspaspglnvalvalglu420425430argpheleuhisglngluleuvalglythrilealaleualaargglu435440445leualaargphetrpgluglutyrproserglysersermethisarg450455460valleuphevalserasnproaspaspglnglnglyasnglntyrser465470475480hisileleuargalaalavalgluglnleuvalargvaltrparghis485490495gluserglutyraspservalasnproalahisglnglngluglygln500505510serseralaalavaltrpalaasnglnleuileargtyrvalasnasn515520525glumetalaasnleuaspphethrcysalatrpvalalalysleuleu530535540glyseraspargargilealagluileasnleutyrleuprogluglu545550555560ilevalglythrileglyvalhisasnproglypheglytrpalaglu565570575serleupheglyleuhismetglylysvalalaleuilethrglygly580585590seralaglyileglyglyglnileglyargleuleualaleusergly595600605alahisvalmetleualaalaargasnalaaspglnleugluglnmet610615620argalaserilevalarggluvalargaspalasertyrproaspala625630635640gluserargvalalailepheproglyseraspvalseraspileasp645650655glyleugluargleuvalasnhisthrvalargvalpheglylysval660665670asptyrleuileasnasnalaglyilealaglyalagluglumetval675680685ileaspmetprovalaspalatrparghisthrleuargalaasnleu690695700ileserasntyralaleuleuargargleualaproglnmetlysala705710715720alaglyglyalatyrvalleuasnvalsersertyrpheglyglyglu725730735lystyrvalalaileprotyrproasnargserasptyralavalser740745750lysalaglyglnargalametvalgluserleualaargpheleugly755760765progluileglnileasnalailealaproglyprovalgluglyglu770775780argleulysglyalaglyserargproglyleuphemetargargala785790795800argleuileleugluasnlysargleuasngluvalphealaalaleu805810815leualaalaarghisgluglyalathrilealaaspleuleuproasp820825830leuphealaasnaspileglnserilealaasnseralaalametpro835840845alaproleuargargleualathrmetleuarggluthrseraspala850855860glyglyseralaglnsertyrleumetasnalathrilealaarglys865870875880leuleuasnargleugluasnglyglytyrilethrleuhisasparg885890895argalaleuthrvalgluproprogluprophephethrglualagln900905910ilegluargglualailelysvalargaspglyileleuglymetleu915920925hisleuglnargmetprothrglupheaspvalalaleualathrval930935940phetyrleualaaspargasnvalthrglygluthrphehisproser945950955960glyglyleuargphegluargthrvalthrgluglygluleuphegly965970975lysproglyglnglnargleugluargleulysglyservalvaltyr980985990leuileglygluhisleuargglnhisleuvalleuleualaargthr99510001005pheleuaspgluilehisvalalaargvalvalleuleuthrglu101010151020thrthrglnalaalathraspleualaalagluleuserasptyr102510301035glualaalaglyargphevalvalileprothrcysglyaspile104010451050gluglyglyileaspargalametalaglutyrglyargprogly105510601065provalileserthrpropheargproleuproaspargalaleu107010751080seralaargasnglyasptrpserservalleuthrthralaglu108510901095pheglugluleuvalgluglnglnilethrhishispheargval110011051110alaarglysalaglyleuilegluglyalaasnvalthrleuval111511201125thrproprothrseralaargserthrserglugluphealaleu113011351140alaasnphevallysthrthrleuhisalaleuthralathrala114511501155glyalaglusergluargthrvalprohisvalprovalasngln116011651170valaspleuthrargargalaargserglugluproargthrpro117511801185sergluglugluglugluleuglnargphevalasnalavalleu119011951200leuthrseralaproleuprothrproleugluserargtyrarg120512101215alaargiletyrargglyasnalailethrval12201225<210>60<211>1217<212>prt<213>erythrobactersp.<400>60metserlysgluglyasnalaalalysglyargleugluglylysval151015alaleuilethrglyalaalaglyasnleuglyasngluileserarg202530alaphealaarggluglyalaphevalvalmetthrglyargthrglu354045gluargileseralaalaarggluglnleuilealaaspthrglyval505560alaprogluargileaspthralavalleuaspglyglyasnproasp65707580serileargalaalametalalysleuarglysglutyrglyargile859095aspileleuileasnasnalaglyseralaglyprolysglnproleu100105110hisasnvalproleuserproglnglumetglualacysglyaspthr115120125gluthrvalargaspalametleuasnileleuglyvalthrtrpasn130135140metalaargilevalalaprometmetprovalglyglyalametval145150155160asnileserthrilepheserhisthrargtyrtyrglyargthrala165170175tyrvalvalprolysalaalaleuasnalaleuserasnglnleuala180185190sergluleuglyproargglyileargvalasnthrvalpheprogly195200205proilegluseraspargileargthrvalphealaalametaspglu210215220valglnserglnprolysaspthrthralaasntyrphethrglyarg225230235240metalaleuthrargservalasnglylysvalaspglylysproleu245250255proasnprolysaspilealaglythrcysleupheleualaserglu260265270glualaalaglyilealaglyglugluvalaspvalthrhisglyleu275280285seralaasnargthrseralaserthrtyrmetthrargprosermet290295300argserleuaspglyalaglyleuasnilepheilevalserglyglu305310315320asntrpaspaspalaleuvalalaalahisthrleuileglysergly325330335alalysvalargleuglyleualaargasnalaaspvalalaglnala340345350asnalaargleulysalaglnglyileglyglugluleuthrvalthr355360365argpheasnargalagluproaspalametgluaspalaleualaala370375380pheserglyaspvalaspglyalailethrglyalaileileleupro385390395400vallysproserglyhisphethrglyserleuleualaalaaspasp405410415aspthrvalthrlysphemetaspthrgluleuvalglyalaileala420425430valserargserleualaargtyrtrphisglyarggluaspleugln435440445serproproargcysvalphemetthrasnproglyaspproleugly450455460asnserphealaservalleuseralaglyilethrglnleuilearg465470475480iletrpargaspglugluargvalglnalaglyasnglyserthrglu485490495hisalavaltrpserasnglnilevalarghisthrasnthrgluasp500505510gluasnthrargphealaserglyhisalathrargvalleuphearg515520525gluglnhisilealagluileaspleulysleuproalaasnileser530535540glugluthrglyserarglysalametvalglyphealagluasnile545550555560thrglyleuhisleuglylysvalalapheilethrglyglyserala565570575glyileglyglyglnvalalaargleuleualaleualaglyalalys580585590valmetmetvalalaargargglusergluleuvalalaalaargasp595600605argilevalglygluleuglnaspileglyphealaglyvalgluarg610615620argvallystyrmetalaaspileaspvalseraspphealaserleu625630635640asplysalavalaspalathrleugluglupheglyargileasptyr645650655leuileasnasnalaglyvalalaglyalagluaspmetvalileasp660665670metgluproglualatrpargphethrleuaspalaasnleuileser675680685asntyrhisleumetglnargvalvalproleumetlysgluglngly690695700serglytyrvalleuasnvalsersertyrpheglyglyglulysphe705710715720leualavalalatyrproasnargalaasptyrglyleuserlysala725730735glyglnargalametvalglualapheserpropheleuglyproglu740745750valglncysasnalailealaproglyprovalaspglyaspargleu755760765serglythrglyglylysproglyleupheglnargargalalysleu770775780ileleugluasnlysargleuasnalavaltyrseralavalilehis785790795800alailearggluglyglyaspalaalalysileleuthrargleuser805810815argasnserthrserthrleuserhisaspalaglualaprogluglu820825830leuarglysleualaleuaspphealaserglnglyaspglyleucys835840845thrtrpaspglntyrleuleuthraspalametalaglnargleuleu850855860valargleuglnleuglyglypheleuleuglyserasnglutrpala865870875880serleusersersergluglnthrtrpleulysleuserproproasp885890895asplyspropheleuproalaalaglnvalasplysvalalaasngly900905910valglylysglyvalileserglnleuhisleuglyalametprothr915920925glualagluvalalaglnalathrvalphepheleualaaspargala930935940valserglygluthrphemetproserglyglyleuargvalgluarg945950955960serasnthrgluargglumetpheglyserprolysglngluargile965970975asplysmetlysglylysthrvaltrpileileglygluhisleuser980985990asptyrvalalaalathrileglugluleuvalserglycysglyval99510001005alalysvalvalleuilealalysasplysserglyglulysala101010151020valargaspglnleuproasnaspleuserlysaspalaleuglu102510301035valleuilealaglyaspglyleugluglualametaspgluala104010451050leuglyhistrpglylysprothrthrvalleusermetprogly105510601065gluproleuproasphisleuphegluglyglyasnproleuser107010751080thrlysaspphealahismetvalglualaasnilethrarghis108510901095tyrargvalthrarglysalaserleutyraspglycysglnval110011051110valleuvalserproaspvalprotyrglyseraspglyprogly111511201125valalaleualaasnphevallysthrserleuhisalaphethr113011351140alathrvalalavalgluasngluargleuvalhisaspvalpro114511501155valasnglnileasnleuthrargargvalsersergluglupro116011651170argaspalaaspgluhisalaglugluleuargargphethrarg117511801185alavalleuleuvalglyalaproleuproaspalaglnaspser119011951200argtyrargserlysiletyrargglythrsermetthrval120512101215<210>61<211>357<212>prt<213>metallosphaerasedula<400>61metargargthrleulysalaalaileleuglyalathrglyleuval151015glyileglutyrvalargmetleualaasphisprotyrilelyspro202530thrtyrleualaglylysglyservalglylysprotyrglygluile354045valargtrpglnthrvalglyasnvalprolysgluvalalaasngln505560gluvallysprothraspprolysleumetaspaspvalaspileile65707580pheserproleuproglnglyalaalaglyprovalglugluglnphe859095alalysleuglypheasnvalileserasnserproasphisargphe100105110aspmetaspvalprometileileprogluvalasnprohisthrval115120125thrleuileaspgluglnarglysargargasptrplysglypheile130135140valthrthrproleucysthralaglnglyalaalaileproleuthr145150155160proiletyrglnasnphelysmetserglyvalmetilethrthrmet165170175glnserleuserglyalaglytyrproglyilealaserleuaspile180185190valaspasnalaleuproleuglyaspglytyraspalalysthrval195200205lysgluilethrargileleusergluvallysargasnvalglnglu210215220proglyvalasngluilethrleuaspalathrthrhisargileala225230235240thrilehisglyhistyrgluvalalatyrvalthrphelysgluasp245250255thraspvalarglysvalmetglusermetgluserphelysglyglu260265270proglnaspleulysleuprothralaproglulysproileileval275280285thrthrglnaspalaargproglnvalphepheaspargtrpalagly290295300asnproproglymetservalvalvalglyargleulysglnvalasn305310315320proargthrileargphevalserleuilehisasnthrvalarggly325330335alaalaglyglyglyvalleuthralagluleuleuvalglulysgly340345350tyrileasplysarg355<210>62<211>356<212>prt<213>sulfolobustokodaii<400>62metargargthrleulysalaalaileleuglyalathrglyleuval151015glyileglutyrvalargmetleuserasnhisprotyrilelyspro202530alatyrleualaglylysglyservalglylysprotyrglygluval354045valargtrpglnthrvalglyglnvalprolysgluilealaaspmet505560gluilelysprothraspprolysleumetaspaspvalaspileile65707580pheserproleuproglnglyalaalaglyprovalglugluglnphe859095alalysgluglypheprovalileserasnserproasphisargphe100105110aspproaspvalproleuleuvalprogluleuasnprohisthrile115120125serleuileaspgluglnarglysargargglutrplysglypheile130135140valthrthrproleucysthralaglnglyalaalaileproleugly145150155160alailephelysasptyrlysmetaspglyalapheilethrthrile165170175glnserleuserglyalaglytyrproglyileproserleuaspval180185190valaspasnileleuproleuglyaspglytyraspalalysthrile195200205lysgluilepheargileleusergluvallysargasnvalaspglu210215220prolysleugluaspvalserleualaalathrthrhisargileala225230235240thrilehisglyhistyrgluvalleutyrvalserphelysgluglu245250255thralaalaglulysvallysgluthrleugluasnpheargglyglu260265270proglnaspleulysleuprothralaproserlysproileileval275280285metasngluaspthrargproglnvaltyrpheaspargtrpalagly290295300aspileproglymetservalvalvalglyargleulysglnvalasn305310315320lysargmetileargleuvalserleuilehisasnthrvalarggly325330335alaalaglyglyglyileleualaalagluleuleuvalglulysgly340345350tyrileglulys355<210>63<211>482<212>prt<213>酿酒酵母<400>63metseralaphevalargvalvalproargileserargserserval151015leuthrargserleuargleuglnleuargcystyralasertyrpro202530gluhisthrileileglymetproalaleuserprothrmetthrgln354045glyasnleualaalatrpthrlyslysgluglyaspglnleuserpro505560glygluvalilealagluilegluthrasplysalaglnmetaspphe65707580glupheglngluaspglytyrleualalysileleuvalproglugly859095thrlysaspileprovalasnlysproilealavaltyrvalgluasp100105110lysalaaspvalproalaphelysaspphelysleugluaspsergly115120125seraspserlysthrserthrlysalaglnproalagluproglnala130135140glulyslysglnglualaproalaglugluthrlysthrseralapro145150155160glualalyslysseraspvalalaalaproglnglyargilepheala165170175serproleualalysthrilealaleuglulysglyileserleulys180185190aspvalhisglythrglyproargglyargilethrlysalaaspile195200205glusertyrleuglulysserserlysglnserserglnthrsergly210215220alaalaalaalathrproalaalaalathrserserthrthralagly225230235240seralaproserproserserthralasertyrgluaspvalproile245250255serthrmetargserileileglygluargleuleuglnserthrgln260265270glyileprosertyrilevalserserlysileserileserlysleu275280285leulysleuargglnserleuasnalathralaasnasplystyrlys290295300leuserileasnaspleuleuvallysalailethrvalalaalalys305310315320argvalproaspalaasnalatyrtrpleuproasngluasnvalile325330335arglysphelysasnvalaspvalservalalavalalathrprothr340345350glyleuleuthrproilevallysasncysglualalysglyleuser355360365glnileserasngluilelysgluleuvallysargalaargileasn370375380lysleualaprogluglupheglnglyglythrilecysileserasn385390395400metglymetasnasnalavalasnmetphethrserileileasnpro405410415proglnserthrileleualailealathrvalgluargvalalaval420425430gluaspalaalaalagluasnglypheserpheaspasnglnvalthr435440445ilethrglythrpheasphisargthrileaspglyalalysglyala450455460gluphemetlysgluleulysthrvalilegluasnproleuglumet465470475480leuleu<210>64<211>420<212>prt<213>酿酒酵母<400>64metleualaalaserphelysargglnproserglnleuvalarggly151015leuglyalavalleuargthrprothrargileglyhisvalargthr202530metalathrleulysthrthrasplyslysalaprogluaspileglu354045glyseraspthrvalglnilegluleuprogluserserphegluser505560tyrmetleugluproproaspleusertyrgluthrserlysalathr65707580leuleuglnmettyrlysaspmetvalileileargargmetglumet859095alacysaspalaleutyrlysalalyslysileargglyphecyshis100105110leuservalglyglnglualailealavalglyilegluasnalaile115120125thrlysleuaspserileilethrsertyrargcyshisglyphethr130135140phemetargglyalaservallysalavalleualagluleumetgly145150155160argargalaglyvalsertyrglylysglyglysermethisleutyr165170175alaproglyphetyrglyglyasnglyilevalglyalaglnvalpro180185190leuglyalaglyleualaphealahisglntyrlysasngluaspala195200205cysserphethrleutyrglyaspglyalaserasnglnglyglnval210215220phegluserpheasnmetalalysleutrpasnleuprovalvalphe225230235240cyscysgluasnasnlystyrglymetglythralaalaserargser245250255seralametthrglutyrphelysargglyglntyrileproglyleu260265270lysvalasnglymetaspileleualavaltyrglnalaserlysphe275280285alalysasptrpcysleuserglylysglyproleuvalleuglutyr290295300gluthrtyrargtyrglyglyhissermetseraspproglythrthr305310315320tyrargthrargaspgluileglnhismetargserlysasnasppro325330335ilealaglyleulysmethisleuileaspleuglyilealathrglu340345350alagluvallysalatyrasplysseralaarglystyrvalaspglu355360365glnvalgluleualaaspalaalaproproproglualalysleuser370375380ileleuphegluaspvaltyrvallysglythrgluthrprothrleu385390395400argglyargileprogluaspthrtrpaspphelyslysglnglyphe405410415alaserargasp420<210>65<211>366<212>prt<213>酿酒酵母<400>65metpheserargleuprothrserleualaargasnvalalaargarg151015alaprothrserphevalargproseralaalaalaalaalaleuarg202530pheserserthrlysthrmetthrvalargglualaleuasnserala354045metalaglugluleuaspargaspaspaspvalpheleuileglyglu505560gluvalalaglntyrasnglyalatyrlysvalserlysglyleuleu65707580aspargpheglygluargargvalvalaspthrproilethrglutyr859095glyphethrglyleualavalglyalaalaleulysglyleulyspro100105110ilevalgluphemetserpheasnphesermetglnalaileasphis115120125valvalasnseralaalalysthrhistyrmetserglyglythrgln130135140lyscysglnmetvalpheargglyproasnglyalaalavalglyval145150155160glyalaglnhisserglnasppheserprotrptyrglyserilepro165170175glyleulysvalleuvalprotyrseralagluaspalaargglyleu180185190leulysalaalaileargaspproasnprovalvalpheleugluasn195200205gluleuleutyrglygluserphegluilesergluglualaleuser210215220progluphethrleuprotyrlysalalysilegluarggluglythr225230235240aspileserilevalthrtyrthrargasnvalglnpheserleuglu245250255alaalagluileleuglnlyslystyrglyvalseralagluvalile260265270asnleuargserileargproleuaspthrglualaileilelysthr275280285vallyslysthrasnhisleuilethrvalgluserthrpheproser290295300pheglyvalglyalagluilevalalaglnvalmetglusergluala305310315320pheasptyrleuaspalaproileglnargvalthrglyalaaspval325330335prothrprotyralalysgluleugluaspphealapheproaspthr340345350prothrilevallysalavallysgluvalleuserileglu355360365<210>66<211>499<212>prt<213>酿酒酵母<400>66metleuargileargserleuleuasnasnlysargalapheserser151015thrvalargthrleuthrileasnlysserhisaspvalvalileile202530glyglyglyproalaglytyrvalalaalailelysalaalaglnleu354045glypheasnthralacysvalglulysargglylysleuglyglythr505560cysleuasnvalglycysileproserlysalaleuleuasnasnser65707580hisleuphehisglnmethisthrglualaglnlysargglyileasp859095valasnglyaspilelysileasnvalalaasnpheglnlysalalys100105110aspaspalavallysglnleuthrglyglyilegluleuleuphelys115120125lysasnlysvalthrtyrtyrlysglyasnglyserphegluaspglu130135140thrlysileargvalthrprovalaspglyleugluglythrvallys145150155160gluasphisileleuaspvallysasnileilevalalathrglyser165170175gluvalthrpropheproglyilegluileaspgluglulysileval180185190serserthrglyalaleuserleulysgluileprolysargleuthr195200205ileileglyglyglyileileglyleuglumetglyservaltyrser210215220argleuglyserlysvalthrvalvalglupheglnproglnilegly225230235240alasermetaspglygluvalalalysalathrglnlyspheleulys245250255lysglnglyleuaspphelysleuserthrlysvalileseralalys260265270argasnaspasplysasnvalvalgluilevalvalgluaspthrlys275280285thrasnlysglngluasnleuglualagluvalleuleuvalalaval290295300glyargargprotyrilealaglyleuglyalaglulysileglyleu305310315320gluvalasplysargglyargleuvalileaspaspglnpheasnser325330335lyspheprohisilelysvalvalglyaspvalthrpheglypromet340345350leualahislysalagluglugluglyilealaalavalglumetleu355360365lysthrglyhisglyhisvalasntyrasnasnileproservalmet370375380tyrserhisprogluvalalatrpvalglylysthrglugluglnleu385390395400lysglualaglyileasptyrlysileglylyspheprophealaala405410415asnserargalalysthrasnglnaspthrgluglyphevallysile420425430leuileaspserlysthrgluargileleuglyalahisileilegly435440445proasnalaglyglumetilealaglualaglyleualaleuglutyr450455460glyalaseralagluaspvalalaargvalcyshisalahisprothr465470475480leuserglualaphelysglualaasnmetalaalatyrasplysala485490495ilehiscys<210>67<211>887<212>prt<213>大肠杆菌<400>67metsergluargpheproasnaspvalaspproilegluthrargasp151015trpleuglnalailegluservalileargglugluglyvalgluarg202530alaglntyrleuileaspglnleuleualaglualaarglysglygly354045valasnvalalaalaglythrglyileserasntyrileasnthrile505560provalglugluglnproglutyrproglyasnleugluleugluarg65707580argileargseralaileargtrpasnalailemetthrvalleuarg859095alaserlyslysaspleugluleuglyglyhismetalaserphegln100105110serseralathriletyraspvalcyspheasnhisphepheargala115120125argasngluglnaspglyglyaspleuvaltyrpheglnglyhisile130135140serproglyvaltyralaargalapheleugluglyargleuthrgln145150155160gluglnleuaspasnpheargglngluvalhisglyasnglyleuser165170175sertyrprohisprolysleumetprogluphetrpglnpheprothr180185190valsermetglyleuglyproileglyalailetyrglnalalysphe195200205leulystyrleugluhisargglyleulysaspthrserlysglnthr210215220valtyralapheleuglyaspglyglumetaspgluprogluserlys225230235240glyalailethrilealathrargglulysleuaspasnleuvalphe245250255valileasncysasnleuglnargleuaspglyprovalthrglyasn260265270glylysileileasngluleugluglyilephegluglyalaglytrp275280285asnvalilelysvalmettrpglyserargtrpaspgluleuleuarg290295300lysaspthrserglylysleuileglnleumetasngluthrvalasp305310315320glyasptyrglnthrphelysserlysaspglyalatyrvalargglu325330335hisphepheglylystyrprogluthralaalaleuvalalaasptrp340345350thraspgluglniletrpalaleuasnargglyglyhisaspprolys355360365lysiletyralaalaphelyslysalaglngluthrlysglylysala370375380thrvalileleualahisthrilelysglytyrglymetglyaspala385390395400alagluglylysasnilealahisglnvallyslysmetasnmetasp405410415glyvalarghisileargaspargpheasnvalprovalseraspala420425430aspileglulysleuprotyrilethrpheprogluglysergluglu435440445histhrtyrleuhisalaglnargglnlysleuhisglytyrleupro450455460serargglnproasnphethrglulysleugluleuproserleugln465470475480asppheglyalaleuleuglugluglnserlysgluileserthrthr485490495ilealaphevalargalaleuasnvalmetleulysasnlysserile500505510lysaspargleuvalproileilealaaspglualaargthrphegly515520525metgluglyleupheargglnileglyiletyrserproasnglygln530535540glntyrthrproglnasparggluglnvalalatyrtyrlysgluasp545550555560glulysglyglnileleuglngluglyileasngluleuglyalagly565570575cyssertrpleualaalaalathrsertyrserthrasnasnleupro580585590metileprophetyriletyrtyrsermetpheglypheglnargile595600605glyaspleucystrpalaalaglyaspglnglnalaargglypheleu610615620ileglyglythrserglyargthrthrleuasnglygluglyleugln625630635640hisgluaspglyhisserhisileglnserleuthrileproasncys645650655ilesertyraspproalatyralatyrgluvalalavalilemethis660665670aspglyleugluargmettyrglyglulysglngluasnvaltyrtyr675680685tyrilethrthrleuasngluasntyrhismetproalametproglu690695700glyalaglugluglyilearglysglyiletyrlysleugluthrile705710715720gluglyserlysglylysvalglnleuleuglyserglyserileleu725730735arghisvalargglualaalagluileleualalysasptyrglyval740745750glyseraspvaltyrservalthrserphethrgluleualaargasp755760765glyglnaspcysgluargtrpasnmetleuhisproleugluthrpro770775780argvalprotyrilealaglnvalmetasnaspalaproalavalala785790795800serthrasptyrmetlysleuphealagluglnvalargthrtyrval805810815proalaaspasptyrargvalleuglythraspglypheglyargser820825830aspserarggluasnleuarghishisphegluvalaspalasertyr835840845valvalvalalaalaleuglygluleualalysargglygluileasp850855860lyslysvalvalalaaspalailealalyspheasnileaspalaasp865870875880lysvalasnproargleuala885<210>68<211>630<212>prt<213>大肠杆菌<400>68metalailegluilelysvalproaspileglyalaaspgluvalglu151015ilethrgluileleuvallysvalglyasplysvalglualaglugln202530serleuilethrvalgluglyasplysalasermetgluvalproser354045proglnalaglyilevallysgluilelysvalservalglyasplys505560thrglnthrglyalaleuilemetilepheaspseralaaspglyala65707580alaaspalaalaproalaglnalagluglulyslysglualaalapro859095alaalaalaproalaalaalaalaalalysaspvalasnvalproasp100105110ileglyseraspgluvalgluvalthrgluileleuvallysvalgly115120125asplysvalglualagluglnserleuilethrvalgluglyasplys130135140alasermetgluvalproalaprophealaglythrvallysgluile145150155160lysvalasnvalglyasplysvalserthrglyserleuilemetval165170175phegluvalalaglyglualaglyalaalaalaproalaalalysgln180185190glualaalaproalaalaalaproalaproalaalaglyvallysglu195200205valasnvalproaspileglyglyaspgluvalgluvalthrgluval210215220metvallysvalglyasplysvalalaalagluglnserleuilethr225230235240valgluglyasplysalasermetgluvalproalaprophealagly245250255valvallysgluleulysvalasnvalglyasplysvallysthrgly260265270serleuilemetilephegluvalgluglyalaalaproalaalaala275280285proalalysglnglualaalaalaproalaproalaalalysalaglu290295300alaproalaalaalaproalaalalysalagluglylyssergluphe305310315320alagluasnaspalatyrvalhisalathrproleuileargargleu325330335alaargglupheglyvalasnleualalysvallysglythrglyarg340345350lysglyargileleuarggluaspvalglnalatyrvallysgluala355360365ilelysargalaglualaalaproalaalathrglyglyglyilepro370375380glymetleuprotrpprolysvalasppheserlyspheglygluile385390395400glugluvalgluleuglyargileglnlysileserglyalaasnleu405410415serargasntrpvalmetileprohisvalthrhispheasplysthr420425430aspilethrgluleuglualaphearglysglnglnasnglugluala435440445alalysarglysleuaspvallysilethrprovalvalpheilemet450455460lysalavalalaalaalaleugluglnmetproargpheasnserser465470475480leusergluaspglyglnargleuthrleulyslystyrileasnile485490495glyvalalavalaspthrproasnglyleuvalvalprovalphelys500505510aspvalasnlyslysglyileilegluleuserarggluleumetthr515520525ileserlyslysalaargaspglylysleuthralaglyglumetgln530535540glyglycysphethrileserserileglyglyleuglythrthrhis545550555560phealaproilevalasnalaprogluvalalaileleuglyvalser565570575lysseralametgluprovaltrpasnglylysgluphevalproarg580585590leumetleuproileserleuserpheasphisargvalileaspgly595600605alaaspglyalaargpheilethrileileasnasnthrleuserasp610615620ileargargleuvalmet625630<210>69<211>474<212>prt<213>大肠杆菌<400>69metserthrgluilelysthrglnvalvalvalleuglyalaglypro151015alaglytyrseralaalapheargcysalaaspleuglyleugluthr202530valilevalgluargtyrasnthrleuglyglyvalcysleuasnval354045glycysileproserlysalaleuleuhisvalalalysvalileglu505560glualalysalaleualagluhisglyilevalpheglygluprolys65707580thraspileasplysileargthrtrplysglulysvalileasngln859095leuthrglyglyleualaglymetalalysglyarglysvallysval100105110valasnglyleuglylysphethrglyalaasnthrleugluvalglu115120125glygluasnglylysthrvalileasnpheaspasnalaileileala130135140alaglyserargproileglnleupropheileprohisgluasppro145150155160argiletrpaspserthraspalaleugluleulysgluvalproglu165170175argleuleuvalmetglyglyglyileileglyleuglumetglythr180185190valtyrhisalaleuglyserglnileaspvalvalglumetpheasp195200205glnvalileproalaalaasplysaspilevallysvalphethrlys210215220argileserlyslyspheasnleumetleugluthrlysvalthrala225230235240valglualalysgluaspglyiletyrvalthrmetgluglylyslys245250255alaproalagluproglnargtyraspalavalleuvalalailegly260265270argvalproasnglylysasnleuaspalaglylysalaglyvalglu275280285valaspaspargglypheileargvalasplysglnleuargthrasn290295300valprohisilephealaileglyaspilevalglyglnprometleu305310315320alahislysglyvalhisgluglyhisvalalaalagluvalileala325330335glylyslyshistyrpheaspprolysvalileproserilealatyr340345350thrgluprogluvalalatrpvalglyleuthrglulysglualalys355360365glulysglyilesertyrgluthralathrpheprotrpalaalaser370375380glyargalailealaseraspcysalaaspglymetthrlysleuile385390395400pheasplysgluserhisargvalileglyglyalailevalglythr405410415asnglyglygluleuleuglygluileglyleualaileglumetgly420425430cysaspalagluaspilealaleuthrilehisalahisprothrleu435440445hisgluservalglyleualaalagluvalphegluglyserilethr450455460aspleuproasnprolysalalyslyslys465470<210>70<211>371<212>prt<213>枯草芽孢杆菌<400>70metalaalalysthrlyslysalailevalaspserlyslysglnphe151015aspalailelyslysglnphegluthrpheglnileleuasnglulys202530glygluvalvalasnglualaalametproaspleuthraspaspgln354045leulysgluleumetargargmetvalphethrargvalleuaspgln505560argserileserleuasnargglnglyargleuglyphetyralapro65707580thralaglyglnglualaserglnilealathrhisphealaleuglu859095lysgluaspphevalleuproglytyrargaspvalproglnleuile100105110trphisglyleuproleutyrglnalapheleupheserargglyhis115120125pheargglyasnglnmetproaspaspvalasnalaleuserprogln130135140ileileileglyalaglntyrileglnthralaglyvalalaleugly145150155160leulyslysargglylyslysalavalalailethrtyrthrglyasp165170175glyglyalaserglnglyaspphetyrgluglyileasnphealagly180185190alatyrlysalaproalailephevalvalglnasnasnargtyrala195200205ileserthrprovalglulysglnseralaalagluthrilealagln210215220lysalavalalaalaglyilevalglyvalglnvalaspglymetasp225230235240proleualavaltyralaalathralaglualaarggluargalaile245250255asnglygluglyprothrleuilegluthrleuthrpheargtyrgly260265270prohisthrmetalaglyaspaspprothrlystyrargthrlysglu275280285ilegluasnglutrpgluglnlysaspproleuvalargpheargala290295300pheleugluasnlysglyleutrpsergluglugluglualalysval305310315320ilegluaspalalysglugluilelysglnalailelyslysalaasp325330335alagluprolysglnlysvalthraspleumetlysilemettyrglu340345350lysmetprohisasnleuglugluglnphegluiletyrthrglnlys355360365gluserlys370<210>71<211>325<212>prt<213>枯草芽孢杆菌<400>71metalaglnmetthrmetileglnalailethraspalaleuargthr151015gluleulysasnaspgluasnvalleuvalpheglygluaspvalgly202530valasnglyglyvalpheargalathrgluglyleuglnlysgluphe354045glygluaspargvalpheaspthrproleualagluserglyilegly505560glyleualaleuglyleuglyleuasnglypheargprovalmetglu65707580ileglnphepheglyphevaltyrgluvalmetaspservalsergly859095glnmetalaargmetargtyrargserglyglyargtrpthrserpro100105110valthrileargserpropheglyglyglyvalhisthrprogluleu115120125hisalaaspserleugluglyleuvalalaglnglnproglyilelys130135140valvalileproserthrprotyraspalalysglyleuleuileser145150155160alaileargaspasnaspprovalvalpheleugluhismetlysleu165170175tyrargserpheargglngluvalproglugluglutyrthrileglu180185190leuglylysalaaspvallysarggluglythraspleuserileile195200205thrtyrglyalametvalhisgluserleulysalaalaaspgluleu210215220glulysaspglyileseralagluvalvalaspleuargthrvalser225230235240proleuaspileaspthrileilealaservalglulysthrglyarg245250255alailevalvalglnglualaglnlysglnalaglyilealaalaasn260265270valvalalagluileasnaspargalaileleuserleuglualapro275280285valleuargvalalaalaproaspthrvalphepropheserglnala290295300gluservaltrpleuproasnhislysaspvalleugluthralaarg305310315320lysvalleugluphe325<210>72<211>442<212>prt<213>枯草芽孢杆菌<400>72metalaphegluphelysleuproaspileglygluglyilehisglu151015glygluilevallystrpphevallysproasnaspgluvalaspglu202530aspaspvalleualagluvalglnasnasplysalavalvalgluile354045proserprovallysglylysvalleugluleulysvalgluglugly505560thrvalalathrvalglyglnthrileilethrpheaspalaprogly65707580tyrgluaspleuglnphelysglyseraspgluseraspaspalalys859095thrglualaglnvalglnserthralaglualaglyglnaspvalala100105110lysglugluglnalaglngluproalalysalathrglyalaglygln115120125glnaspglnalagluvalaspproasnlysargvalilealametpro130135140servalarglystyralaargglulysglyvalaspilearglysval145150155160thrglyserglyasnasnglyargvalvallysgluaspileaspser165170175phevalasnglyglyalaglnglualaalaproglngluthralaala180185190proglngluthralaalalysproalaalaalaproalaproglugly195200205glupheprogluthrargglulysmetserglyilearglysalaile210215220alalysalametvalasnserlyshisthralaprohisvalthrleu225230235240metaspgluvalaspvalthrasnleuvalalahisarglysglnphe245250255lysglnvalalaalaaspglnglyilelysleuthrtyrleuprotyr260265270valvallysalaleuthrseralaleulyslyspheprovalleuasn275280285thrserileaspasplysthraspgluvalileglnlyshistyrphe290295300asnileglyilealaalaaspthrglulysglyleuleuvalproval305310315320vallysasnalaasparglysservalphegluileseraspgluile325330335asnglyleualathrlysalaarggluglylysleualaproalaglu340345350metlysglyalasercysthrilethrasnileglyseralaglygly355360365glntrpphethrprovalileasnhisprogluvalalaileleugly370375380ileglyargilealaglulysalailevalargaspglygluileval385390395400alaalaprovalleualaleuserleuserpheasphisargmetile405410415aspglyalathralaglnasnalaleuasnhisilelysargleuleu420425430asnaspproglnleuileleumetgluala435440<210>73<211>470<212>prt<213>枯草芽孢杆菌<400>73metvalvalglyasppheproilegluthraspthrleuvalilegly151015alaglyproglyglytyrvalalaalaileargalaalaglnleugly202530glnlysvalthrvalvalglulysalathrleuglyglyvalcysleu354045asnvalglycysileproserlysalaleuileasnalaglyhisarg505560tyrgluasnalalyshisseraspaspmetglyilethralagluasn65707580valthrvalaspphethrlysvalglnglutrplysalaservalval859095asnlysleuthrglyglyvalalaglyleuleulysglyasnlysval100105110aspvalvallysglyglualatyrphevalaspserasnservalarg115120125valmetaspgluasnseralaglnthrtyrthrphelysasnalaile130135140ilealathrglyserargproilegluleuproasnphelystyrser145150155160gluargvalleuasnserthrglyalaleualaleulysgluilepro165170175lyslysleuvalvalileglyglyglytyrileglythrgluleugly180185190thralatyralaasnpheglythrgluleuvalileleugluglygly195200205aspgluileleuproglypheglulysglnmetserserleuvalthr210215220argargleulyslyslysglyasnvalgluilehisthrasnalamet225230235240alalysglyvalglugluargproaspglyvalthrvalthrpheglu245250255vallysglygluglulysthrvalaspalaasptyrvalleuilethr260265270valglyargargproasnthraspgluleuglyleugluglnvalgly275280285ileglumetthraspargglyilevallysthrasplysglncysarg290295300thrasnvalproasniletyralaileglyaspileilegluglypro305310315320proleualahislysalasertyrgluglylysilealaalagluala325330335ilealaglygluproalagluileasptyrleuglyileproalaval340345350valphesergluprogluleualaservalglytyrthrglualagln355360365alalysglugluglyleuaspilevalalaalalysphepropheala370375380alaasnglyargalaleuserleuasngluthraspglyphemetlys385390395400leuilethrarglysgluaspglyleuvalileglyalaglnileala405410415glyalaseralaseraspmetilesergluleuserleualaileglu420425430glyglymetthralagluaspilealametthrilehisalahispro435440445thrleuglygluilethrmetglualaalagluvalalaileglyser450455460proilehisilevallys465470<210>74<211>2123<212>prt<213>酿酒酵母<400>74metargserilearglystrpalatyrgluthrpheasnaspglulys151015ileileglnphevalvalmetalathrproaspaspleuhisalaasn202530serglutyrileargmetalaaspglntyrvalglnvalproglygly354045thrasnasnasnasntyralaasnileaspleuileleuaspvalala505560gluglnthraspvalaspalavaltrpalaglytrpglyhisalaser65707580gluasnprocysleuprogluleuleualaserserglnarglysile859095leupheileglyproproglyargalametargserleuglyasplys100105110ileserserthrilevalalaglnseralalysileprocysilepro115120125trpserglyserhisileaspthrilehisileaspasnlysthrasn130135140phevalservalproaspaspvaltyrvalargglycyscysserser145150155160progluaspalaleuglulysalalysleuileglypheprovalmet165170175ilelysalasergluglyglyglyglylysglyileargargvalasp180185190asngluaspasppheilealaleutyrargglnalavalasngluthr195200205proglyserprometphevalmetlysvalvalthraspalaarghis210215220leugluvalglnleuleualaaspglntyrglythrasnilethrleu225230235240pheglyargaspcysserileglnargarghisglnlysileileglu245250255glualaprovalthrilethrlysprogluthrpheglnargmetglu260265270argalaalaileargleuglygluleuvalglytyrvalseralagly275280285thrvalglutyrleutyrserprolysaspasplysphetyrpheleu290295300gluleuasnproargleuglnvalgluhisprothrthrglumetile305310315320serglyvalasnleuproalathrglnleuglnilealametglyile325330335promethismetileseraspilearglysleutyrglyleuasppro340345350thrglythrsertyrileaspphelysasnleulysargproserpro355360365lysglyhiscysilesercysargilethrsergluaspproasnglu370375380glyphelysproserthrglylysilehisgluleuasnpheargser385390395400serserasnvaltrpglytyrpheservalglyasnasnglyalaile405410415hisserpheseraspserglnpheglyhisilephealavalglyasn420425430aspargglnaspalalysglnasnmetvalleualaleulysaspphe435440445serileargglygluphelysthrproileglutyrleuilegluleu450455460leugluthrargaspphegluserasnasnileserthrglytrpleu465470475480aspaspleuileleulysasnleuserseraspserlysleuasppro485490495thrleualaileilecysglyalaalametlysalatyrvalphethr500505510glulysvalargasnlystyrleugluleuleuargargglyglnval515520525proprolysasppheleulysthrlyspheprovalasppheilephe530535540aspasnasnargtyrleupheasnvalalaglnsersergluglugln545550555560pheileleuserileasnlysserglncysgluvalasnvalglnlys565570575leuserseraspcysleuleuileservalaspglylyscyshisthr580585590valtyrtrplysaspaspileargglythrargleuserileaspser595600605asnthrilepheleuglualagluleuasnprothrglnvalileser610615620prothrproglylysleuvallystyrleuvalargserglyasphis625630635640valphealaglyglnglntyralagluilegluilemetlysmetgln645650655metproleuvalalalysseraspglyvalilegluleuleuarggln660665670proglyserileileglualaglyaspvalilealalysleuthrleu675680685aspserproserlysalaasngluserserleutyrargglygluleu690695700provalleuglyproproleuilegluglyserargproasnhislys705710715720leuargvalleuileasnargleugluasnileleuasnglytyrhis725730735gluasnserglyilegluthrthrleulysgluleuilelysileleu740745750argaspglyargleuprotyrserglutrpaspserglnileserthr755760765valargasnargleuproargglnleuasngluglyleuglyasnleu770775780vallyslysservalserpheproalalysgluleuhislysleumet785790795800lysargtyrleuglugluasnthrasnasphisvalvaltyrvalala805810815leuglnproleuleulysilesergluargtyrsergluglyleuala820825830asnhisglucysgluilepheleulysleuilelyslystyrtyrala835840845valglulysilephegluasnhisaspilehisglugluargasnleu850855860leuasnleuargarglysaspleuthrasnleulyslysileleucys865870875880ileserleuserhisalaasnvalvalalalysasnlysleuvalthr885890895alaileleuhisglutyrgluproleucysglnaspserserlysmet900905910serleulyspheargalavalilehisaspleualaserleugluser915920925lystrpalalysgluvalalavallysalaargservalleuleuarg930935940glyilepheproproilelyslysarglysgluhisilelysthrleu945950955960leuglnleuhisilelysaspthrglyalagluasnilehisserarg965970975asniletyrsercysmetargasppheglyasnleuilehisserasn980985990leuileglnleuglnaspleuphephephepheglyhisglnaspthr99510001005alaleuserserilealasergluiletyralaargtyralatyr101010151020glyasntyrglnleulysserilelysilehislysglyalapro102510301035aspleuleumetsertrpglnpheserserleuargasntyrleu104010451050valasnseraspglygluseraspgluphethrlysleuserlys105510601065proproserthrserglylysserseralaasnserpheglyleu107010751080leuvalasnmetargalaleugluserleuglulysthrleuasp108510901095gluvaltyrgluglnilehisileproglugluargleuserser110011051110glygluasnserleuilevalasnileleuserproileargtyr111511201125argsergluasnaspleuilelysthrleulysilelysleuhis113011351140gluasngluargglyleuserlysleulysvalasnargilethr114511501155phealapheilealaalaasnalaproalavallysphetyrser116011651170pheaspglythrthrtyraspgluileserglnileargasnmet117511801185aspprosertyrglualaproleugluleuglylysmetserasn119011951200tyrlysileargserleuprothrtyraspserserileargile120512101215phegluglyileserlysphethrproleuasplysargphephe122012251230valarglysileileasnserphemettyrasnaspglnlysthr123512401245thrglugluasnleulysalagluileasnalaglnvalvaltyr125012551260metleugluhisleuglyalavalaspileserasnseraspleu126512701275asnhisilepheleuserpheasnthrvalleuasnileproval128012851290hisargleuglugluilevalserthrileleulysthrhisglu129513001305thrargleupheglngluargilethraspvalgluilecysile131013151320servalglucysleugluthrlyslysproalaproleuargleu132513301335leuileserasnlysserglytyrvalvallysilegluthrtyr134013451350tyrglulysileglylysasnglyasnleuileleugluprocys135513601365sergluglnserhistyrserglnlysserleuserleuprotyr137013751380servallysasptrpleuglnprolysargtyrlysalaglnphe138513901395metglythrthrtyrvaltyrasppheproglyleuphehisgln140014051410alaalaileglnglntrplysargtyrpheprolyshislysleu141514201425asnaspserphephesertrpvalgluleuilegluglnasngly143014351440asnleuilelysvalasnarggluproglyleuasnasnilegly144514501455metvalalaphegluilemetvalglnthrproglutyrproglu146014651470glyargasnmetilevalileserasnaspilethrtyrasnile147514801485glyserpheglyproarggluaspleuphepheaspargvalthr149014951500asntyralaarggluargglyileproargiletyrleualaala150515101515asnserglyalalysleuglyilealaglugluleuileproleu152015251530pheargvalalatrpasnaspproseraspprothrlysglyphe153515401545glntyrleutyrleualaprolysaspmetglnleuleulysasp155015551560serglylysglyasnservalvalvalgluhislysmetvaltyr156515701575glyglugluargtyrileilelysalailevalglyphegluglu158015851590glyleuglyvalglucysleuglnglyserglyleuilealagly159516001605alathrserlysalatyrargaspilephethrilethralaval161016151620thrcysargservalglyileglysertyrleuvalargleugly162516301635glnargthrileglnvalgluasplysproileileleuthrgly164016451650alaseralaileasnlysvalleuglythraspiletyrthrser165516601665asnleuglnileglyglythrglnilemettyrlysasnglyile167016751680alahisleuthralaserasnaspmetlysalaileglulysile168516901695metthrtrpleusertyrvalproalalysargaspmetserpro170017051710proleuleugluthrmetaspargtrpaspargaspvalaspphe171517201725lysproalalysglnvalprotyrglualaargtrpleuileglu173017351740glylystrpaspserasnasnasnpheglnserglyleupheasp174517501755lysaspserphephegluthrleuserglytrpalalysglyval176017651770ilevalglyargalaargleuglyglyileprovalglyvalile177517801785alavalgluthrlysthrileglugluileileproalaasppro179017951800alaasnleuaspserserglupheservallysglualaglygln180518101815valtrptyrproasnseralaphelysthralaglnthrileasn182018251830asppheasntyrglygluglnleuproleuileileleualaasn183518401845trpargglypheserglyglyglnargaspmettyrasngluval185018551860leulystyrglyserpheilevalaspalaleuvalasptyrlys186518701875glnproileleuiletyrilepropropheglygluleuarggly188018851890glysertrpvalvalileaspprothrileasnprogluglnmet189519001905glumettyralaaspvalgluserargglyglyvalleuglupro191019151920aspglyvalvalserilelystyrarglysglulysmetileglu192519301935thrmetileargleuaspserthrtyrglyhisleuargargthr194019451950leuthrglulyslysleuserleuglulysglnasnaspleuthr195519601965lysargleulysilearggluargglnleuileproiletyrasn197019751980glnileserileglnphealaaspleuhisaspargserthrarg198519901995metleuvallysglyvalileargasngluleuglutrplyslys200020052010serargargpheleutyrtrpargleuargargargleuasnglu201520202025glyglnvalilelysargleuglnlyslysthrcysaspasnlys203020352040thrlysmetlystyraspaspleuleulysilevalglnsertrp204520502055tyrasnaspleuaspvalasnaspaspargalavalvalgluphe206020652070ilegluargasnserlyslysileasplysasnileglugluphe207520802085gluileserleuleuileaspgluleulyslyslysphegluasp209020952100argargglyasnilevalleuglugluleuthrargleuvalasp210521102115serlysarglysarg2120<210>75<211>319<212>prt<213>大肠杆菌<400>75metserleuasnpheleuaspphegluglnproilealagluleuglu151015alalysileaspserleuthralavalserargglnaspglulysleu202530aspileasnileaspglugluvalhisargleuargglulysserval354045gluleuthrarglysilephealaaspleuglyalatrpglnileala505560glnleualaarghisproglnargprotyrthrleuasptyrvalarg65707580leualapheaspglupheaspgluleualaglyaspargalatyrala859095aspasplysalailevalglyglyilealaargleuaspglyargpro100105110valmetileileglyhisglnlysglyarggluthrlysglulysile115120125argargasnpheglymetproalaprogluglytyrarglysalaleu130135140argleumetglnmetalagluargphelysmetproileilethrphe145150155160ileaspthrproglyalatyrproglyvalglyalaglugluarggly165170175glnserglualailealaargasnleuargglumetserargleugly180185190valprovalvalcysthrvalileglygluglyglyserglyglyala195200205leualaileglyvalglyasplysvalasnmetleuglntyrserthr210215220tyrservalileserprogluglycysalaserileleutrplysser225230235240alaasplysalaproleualaalaglualametglyileilealapro245250255argleulysgluleulysleuileaspserileileprogluproleu260265270glyglyalahisargasnproglualametalaalaserleulysala275280285glnleuleualaaspleualaaspleuaspvalleuserthrgluasp290295300leulysasnargargtyrglnargleumetsertyrglytyrala305310315<210>76<211>156<212>prt<213>大肠杆菌<400>76metaspilearglysilelyslysleuilegluleuvalglugluser151015glyilesergluleugluilesergluglyglugluservalargile202530serargalaalaproalaalaserpheprovalmetglnglnalatyr354045alaalaprometmetglnglnproalaglnserasnalaalaalapro505560alathrvalprosermetglualaproalaalaalagluilesergly65707580hisilevalargserprometvalglythrphetyrargthrproser859095proaspalalysalapheilegluvalglyglnlysvalasnvalgly100105110aspthrleucysilevalglualametlysmetmetasnglnileglu115120125alaasplysserglythrvallysalaileleuvalgluserglygln130135140provalglupheaspgluproleuvalvalileglu145150155<210>77<211>449<212>prt<213>大肠杆菌<400>77metleuasplysilevalilealaasnargglygluilealaleuarg151015ileleuargalacyslysgluleuglyilelysthrvalalavalhis202530serseralaaspargaspleulyshisvalleuleualaaspgluthr354045valcysileglyproalaproservallyssertyrleuasnilepro505560alaileileseralaalagluilethrglyalavalalailehispro65707580glytyrglypheleusergluasnalaasnphealagluglnvalglu859095argserglypheilepheileglyprolysalagluthrileargleu100105110metglyasplysvalseralailealaalametlyslysalaglyval115120125procysvalproglyseraspglyproleuglyaspaspmetasplys130135140asnargalailealalysargileglytyrprovalileilelysala145150155160serglyglyglyglyglyargglymetargvalvalargglyaspala165170175gluleualaglnserilesermetthrargalaglualalysalaala180185190pheserasnaspmetvaltyrmetglulystyrleugluasnproarg195200205hisvalgluileglnvalleualaaspglyglnglyasnalailetyr210215220leualagluargaspcyssermetglnargarghisglnlysvalval225230235240gluglualaproalaproglyilethrprogluleuargargtyrile245250255glygluargcysalalysalacysvalaspileglytyrargglyala260265270glythrpheglupheleuphegluasnglygluphetyrpheileglu275280285metasnthrargileglnvalgluhisprovalthrglumetilethr290295300glyvalaspleuilelysgluglnleuargilealaalaglyglnpro305310315320leuserilelysglnglugluvalhisvalargglyhisalavalglu325330335cysargileasnalagluaspproasnthrpheleuproserprogly340345350lysilethrargphehisalaproglyglypheglyvalargtrpglu355360365serhisiletyralaglytyrthrvalproprotyrtyraspsermet370375380ileglylysleuilecystyrglygluasnargaspvalalaileala385390395400argmetlysasnalaleuglngluleuileileaspglyilelysthr405410415asnvalaspleuglnileargilemetasnaspgluasnpheglnhis420425430glyglythrasnilehistyrleuglulyslysleuglyleuglnglu435440445lys<210>78<211>304<212>prt<213>大肠杆菌<400>78metsertrpilegluargilelysserasnilethrprothrarglys151015alaserileprogluglyvaltrpthrlyscysaspsercysglygln202530valleutyrargalagluleugluargasnleugluvalcysprolys354045cysasphishismetargmetthralaargasnargleuhisserleu505560leuaspgluglyserleuvalgluleuglysergluleugluprolys65707580aspvalleulyspheargaspserlyslystyrlysaspargleuala859095seralaglnlysgluthrglyglulysaspalaleuvalvalmetlys100105110glythrleutyrglymetprovalvalalaalaalapheglupheala115120125phemetglyglysermetglyservalvalglyalaargphevalarg130135140alavalgluglnalaleugluaspasncysproleuilecyspheser145150155160alaserglyglyalaargmetglnglualaleumetserleumetgln165170175metalalysthrseralaalaleualalysmetglngluargglyleu180185190protyrileservalleuthraspprothrmetglyglyvalserala195200205serphealametleuglyaspleuasnilealagluprolysalaleu210215220ileglyphealaglyproargvalilegluglnthrvalargglulys225230235240leuproproglypheglnargserglupheleuileglulysglyala245250255ileaspmetilevalargargproglumetargleulysleualaser260265270ileleualalysleumetasnleuproalaproasnproglualapro275280285arggluglyvalvalvalproprovalproaspglngluprogluala290295300<210>79<211>282<212>prt<213>橙色绿屈挠菌<400>79metglugluthralaileproglnserleuthrprotrpaspargval151015glnleualaarghisproglnargprohisthrleuasptyrileala202530alaleucysgluaspphevalgluleuhisglyaspargargphegly354045aspaspproalametvalglyglymetalathrphealaglyglnthr505560valmetvalileglyhisglnlysglyasnaspthrarggluasnmet65707580argargasnpheglymetprohisprogluglytyrarglysalagln859095argleumetarghisalaglulyspheglyleuprovalilecysphe100105110valaspthrproalaalaaspprothrlysserserglugluarggly115120125glnalaasnalailealagluserilemetleumetthrthrleuarg130135140valproserilealavalvalileglygluglyglyserglyglyala145150155160leualaileservalalaaspargileleumetglngluasnalaile165170175tyrservalalaproproglualaalaalaserileleutrpargasp180185190alaalalysalaproglualaalaargalaleulysleuthralaala195200205aspleutyraspleuargileileaspgluvalileproglupropro210215220glyglyalahisalaaspargleuthralailethrthrvalglyglu225230235240argleuargvalhisleualaaspleuglnglnargaspileaspthr245250255leuleuarggluargtyrarglystyrargsermetglyglntyrgln260265270gluglnglnmetaspphepheglyargmet275280<210>80<211>180<212>prt<213>橙色绿屈挠菌<400>80metmetleutrpglyalametlysaspgluthrthrgluleuproala151015aspglnproasppropheglyleualaalavalargvalleuleugln202530metleugluglnseraspvaltyrgluilethrilegluasnglyasn354045alalysleuhisvallysargglyglnproglyglyvaliletyrser505560alaproleuprothralaprovalproserproserleuproalathr65707580provalthrprophevalglnproproproalaprogluglypropro859095valglumetproalaglyhisthrilethralaprometvalglythr100105110phetyralaalaproserproargaspargprophevalglnglugly115120125aspgluvalargvalglyaspthrvalglyilevalglualametlys130135140metmetasngluilegluseraspvalalaglyargvalalaargile145150155160leuvallysasnglyglnprovalglutyrglyglnproleumetval165170175ilegluproleu180<210>81<211>455<212>prt<213>橙色绿屈挠菌<400>81metilearglysvalleuvalalaasnargglygluilealavalarg151015ileileargalacysglngluleuglyileargthrvalvalalatyr202530serthralaaspargaspserleualavalargleualaaspgluala354045valcysileglyproproproalaalalyssertyrleuasnalapro505560alaleuileseralaalaleuvalserglycysaspalailehispro65707580glytyrglypheleusergluasnprotyrphealaglumetcysala859095aspcyslysleuthrpheileglyproproprogluproileargleu100105110metglyasplysalaileglyarggluthrmetarglysalaglyval115120125prothrvalproglyseraspglygluvalargserleuglugluala130135140ileaspvalalaargglnileglytyrprovalleuleulysproser145150155160glyglyglyglyglyargglymetargvalalatyraspglualaasp165170175leuglnargalapheprothralaargalaglualaglualaalaphe180185190glyasnglyalaleuleuleuglulystyrleuthrargvalarghis195200205valgluileglnvalleualaaspglntyrglyhisalailehisleu210215220glygluargaspcysseralaglnargarghisglnlysilevalglu225230235240glualaproserproalavalthrprogluleuarggluargmetgly245250255alaaspalavalargglyilelysserileglytyrvalasnalagly260265270thrleuglupheleuleuaspglnaspglyasntyrtyrpheileglu275280285metasnthrargileglnvalgluhisprovalthrgluglnvalthr290295300glyileaspleuvalargtrpglnleuleuilealaserglygluarg305310315320leuthrleuargglngluaspilelysilethrarghisalaileglu325330335cysargileasnalagluaspprogluargasppheleuproalaser340345350glygluvalgluphetyrleuproproglyglyproglyvalargval355360365aspserhisleutyrserglytyrthrproproglythrtyraspser370375380leuleualalysileilethrpheglyaspthrargaspglualaleu385390395400asnargmetargargalaleuasnglucysvalilethrglyilelys405410415thrthrilepropheglnleualaleuileaspaspprogluphearg420425430alaglyargilehisthrglytyrvalalagluleuleuargglntrp435440445lysgluthrleuasnproval450455<210>82<211>305<212>prt<213>橙色绿屈挠菌<400>82metlysgluphepheargleuserarglysglyphethrglyargglu151015aspglnaspseralaglnileproaspaspleutrpvallyscysser202530sercysarggluleuiletyrlyslysglnleuasnaspasnleulys354045valcysprolyscysglyhishismetargleuseralahisglutrp505560leuglyleuleuaspvalglyserpheargglumetaspalaasnleu65707580leuprothraspproleuglyphevalthraspgluglusertyrala859095alalysleualalysthrglnglnargthrglymetalaaspalaval100105110ilealaglyileglyalaileserasnmetglnilecysvalalaval115120125alaasppheserphemetglyalasermetglyservaltyrglyglu130135140lysmetalaargseralagluargalaalagluleuglyvalproleu145150155160leuthrileasnthrserglyglyalaargglnglngluglyvalile165170175glyleumetglnmetalalysvalthrmetalaleuthrargleuala180185190aspalaglyglnprohisilealaleuleuvalaspprocystyrgly195200205glyvalthralasertyrproservalalaaspileileilealaglu210215220proglyalaasnileglyphealaglylysargleuilegluglnile225230235240metargglnlysleuproalaglypheglnthralagluphemetleu245250255gluhisglymetileaspmetvalvalproargserglumetargasp260265270thrleualaargileleuargleutyrargglnargserthrserpro275280285alalysalagluleualaglyargargalathrleuproglnproile290295300met305<210>83<211>378<212>prt<213>枯草芽孢杆菌<400>83metileileglyvalprolysgluilelysasnasngluasnargval151015alaleuthrproglyglyvalserglnleuileserasnglyhisarg202530valleuvalgluthrglyalaglyleuglyserglyphegluasnglu354045alatyrgluseralaglyalagluileilealaaspprolysglnval505560trpaspalaglumetvalmetlysvallysgluproleuprogluglu65707580tyrvaltyrphearglysglyleuvalleuphethrtyrleuhisleu859095alaalagluprogluleualaglnalaleulysasplysglyvalthr100105110alailealatyrgluthrvalsergluglyargthrleuproleuleu115120125thrprometsergluvalalaglyargmetalaalaglnileglyala130135140glnpheleuglulysprolysglyglylysglyileleuleualagly145150155160valproglyvalserargglylysvalthrileileglyglyglyval165170175valglythrasnalaalalysmetalavalglyleuglyalaaspval180185190thrileileaspleuasnalaaspargleuargglnleuaspaspile195200205pheglyhisglnilelysthrleuileserasnprovalasnileala210215220aspalavalalaglualaaspleuleuilecysalavalleuilepro225230235240glyalalysalaprothrleuvalthrgluglumetvallysglnmet245250255lysproglyservalilevalaspvalalaileaspglnglyglyile260265270valgluthrvalasphisilethrthrhisaspglnprothrtyrglu275280285lyshisglyvalvalhistyralavalalaasnmetproglyalaval290295300proargthrserthrilealaleuthrasnvalthrvalprotyrala305310315320leuglnilealaasnlysglyalavallysalaleualaaspasnthr325330335alaleuargalaglyleuasnthralaasnglyhisvalthrtyrglu340345350alavalalaargaspleuglytyrglutyrvalproalaglulysala355360365leuglnaspgluserservalalaglyala370375<210>84<211>505<212>prt<213>粟酒裂殖酵母<400>84metphethrasptyrproasnaspileasncysgluserproargmet151015seraspleuaspglyphecysglnasnalapheseraspleuasnser202530leuasnglnglnvalphelysalaasntyralavalargglyalaleu354045alaileleualaaspgluileglnaspaspleuleugluasnproser505560sertyrprophesergluilevaltyralaasnileglyasnprogln65707580glnmetglyglnserproilethrphevalargglnvalleuserleu859095cysglntyrprothrleuleuasphisalagluglulystrpphegln100105110asnleupheprothraspvalvalglnargserlysmetleuleulys115120125gluserglyserleuglyalatyrseralaserglnglyileproleu130135140valargarghisvalalaasppheileargalaargaspglypheasp145150155160cysgluproseraspiletyrleuthrserglyalaserhisalaala165170175argleuilemetthrleuileilealaargprothraspglyvalmet180185190valproalaproglntyrproleutyrglyalaglnileaspleumet195200205serglysermetvalsertyrserleuserglugluasnasntrpasp210215220ileasppheaspglnphelyslysserpheaspglualaserlyslys225230235240glyileasnvalargleucysvalvalileasnproglyasnprothr245250255glyalacysilesergluasnsermetglulysvalleuargpheala260265270lysalalysglyilevalleuleualaaspgluvaltyrglnasnasn275280285iletyrglnasnlysphehisserpheargarglysleuglygluleu290295300argglulysgluproaspasnhistrpaspglnvalserleuileser305310315320valasnservalserlysglyglnpheglyglucysglyglnarggly325330335glytyrleuaspvalvalasnileprogluproalalysaspglnile340345350leulysleualathrileaspilecysproprovalalaglyglnleu355360365leuvalaspmetleuvalasnproprolysproglyaspprosertyr370375380aspleupheilelysgluvalaspgluilehisglualaleuargleu385390395400glncysargglnleutyrgluglythrlysargmetlysargvalser405410415cysleugluprohisglyalamettyrleuhisproservalserleu420425430proglulysleuilethrthralalysalaglnlysileglnproasp435440445gluphetyralailegluleuleulysargserglyilecysvalval450455460proglyserglypheglyglnprogluglyasptyrhisileargile465470475480thrpheleualalysglythrglutyrilegluargphevallysala485490495hisasngluilemetaspleutyrglu500505<210>85<211>507<212>prt<213>酿酒酵母<400>85metthrmetthrhisglnglnaspleulysglyvalphethralalys151015aspleuaspphelysproalaglylysilethrlyslysaspleuasn202530thrglyvalthrlysalaglutyralavalargglyalaileprothr354045argalaaspgluleulysglugluleulyslysasnprogluvalleu505560propheaspaspileileasnalaasnileglyasnproglnglnleu65707580aspglnlysproleuthrphethrargglnvalleualaileleuglu859095tyrprogluileleuargvalglyhisasngluleualaserleuasn100105110leupheserargaspalaleugluargalagluargleuleuasnasp115120125ileglyglyserileglyalatyrserhisserglnglyvalprogly130135140ileargglnthrvalalaasppheilethrargargaspglyglyglu145150155160proalathrprogluaspiletyrleuthrthrglyalaserserala165170175alathrserleuleuserleuleucyslysaspserglnthrglyleu180185190leuileproileproglntyrproleutyrthralaseralaserleu195200205pheasnalaglnvalleuprotyrtyrleuaspglugluserasntrp210215220serthrasnseraspgluileglulysvalvalglnaspalaleulys225230235240lysglnileargproservalleuilevalileasnproglyasnpro245250255thrglyalavalleuserglugluthrilealaargilecysleuile260265270alaalalystyrglyilethrileileseraspgluvaltyrglnglu275280285asnilepheasnaspvallysphehissermetlyslysvalleuarg290295300lysleuglnhisleutyrproglylyspheaspasnvalglnleuala305310315320serleuhisserileserlysglyphemetaspglucysglyglnarg325330335glyglytyrmetgluileileglypheserglngluileargaspala340345350leuphelysleumetserileserilecysservalvalthrglygln355360365alavalvalaspleumetvallysproproglnproglyaspgluser370375380tyrgluglnasphisaspgluargleulysilephehisglumetarg385390395400thrargalaasnleuleutyrgluthrphelysgluleugluglyile405410415glucysglnlysproglnglyalamettyrleupheproargleuval420425430leuprolyslysalaleucysglusergluargleuglyileglupro435440445aspgluphetyrcysthrserleuleugluserthrglyilecysthr450455460valproglyserglypheglyglnargproglythrtyrhisvalarg465470475480thrthrpheleualaproglythrlystrpileglnasptrplysglu485490495phehisglnaspphepheserlystyrargasn500505<210>86<211>471<212>prt<213>枯草芽孢杆菌<400>86metlysasnlystrptyrlysprolysarghistrplysgluileglu151015leutrplysaspvalprogluglulystrpasnasptrpleutrpgln202530leuthrhisthrvalargthrleuaspaspleulyslysvalileasn354045leuthrgluaspgluglugluglyvalargileserthrlysthrile505560proleuasnilethrprotyrtyralaserleumetaspproaspasn65707580proargcysprovalargmetglnservalproleusergluglumet859095hislysthrlystyraspleugluaspproleuhisgluaspgluasp100105110serprovalproglyleuthrhisargtyrproaspargvalleuphe115120125leuvalthrasnglncyssermettyrcysargtyrcysthrargarg130135140argpheserglyglnileglymetglyvalprolyslysglnleuasp145150155160alaalailealatyrilearggluthrprogluileargaspcysleu165170175ileserglyglyaspglyleuleuileasnaspglnileleuglutyr180185190ileleulysgluleuargserileprohisleugluvalileargile195200205glythrargalaprovalvalpheproglnargilethrasphisleu210215220cysgluileleulyslystyrhisprovaltrpleuasnthrhisphe225230235240asnthrserileglumetthrglugluservalglualacysglulys245250255leuvalasnalaglyvalprovalglyasnglnalavalvalleuala260265270glyileasnaspservalproilemetlyslysleumethisaspleu275280285vallysileargvalargprotyrtyriletyrglncysaspleuser290295300gluglyileglyhispheargalaprovalserlysglyleugluile305310315320ilegluglyleuargglyhisthrserglytyralavalprothrphe325330335valvalaspalaproglyglyglyglylysilealaleuglnproasn340345350tyrvalleuserglnserproasplysvalileleuargasnpheglu355360365glyvalilethrsertyrprogluprogluasntyrileproasngln370375380alaaspalatyrphegluservalpheprogluthralaasplyslys385390395400gluproileglyleuseralailephealaasplysgluvalserphe405410415thrprogluasnvalaspargilelysargargglualatyrileala420425430asnprogluhisgluthrleulysaspargargglulysargaspgln435440445leulysglulyslyspheleualaglnglnlyslysglnlysgluthr450455460glucysglyglyaspserser465470<210>87<211>416<212>prt<213>porphyromonasgingivalis<400>87metalagluserargarglystyrtyrpheproaspvalthraspglu151015glntrpasnasptrphistrpglnvalleuasnargilegluthrleu202530aspglnleulyslystyrvalthrleuthralagluglugluglugly354045vallysgluserleulysvalleuargmetalailethrprotyrtyr505560leuserleuileaspprogluasnproasncysproilearglysgln65707580alaileprothrhisglngluleuvalargalaprogluaspglnval859095aspproleusergluaspgluaspserprovalproglyleuthrhis100105110argtyrproaspargvalleupheleuilethrasplyscyssermet115120125tyrcysarghiscysthrargargargphealaglyglnlysaspala130135140serserprosergluargileaspargcysileasptyrilealaasn145150155160thrprothrvalargaspvalleuleuserglyglyaspalaleuleu165170175valseraspgluargleuglutyrileleulysargleuarggluile180185190prohisvalgluilevalargileglyserargthrprovalvalleu195200205proglnargilethrproglnleuvalaspmetleulyslystyrhis210215220provaltrpleuasnthrhispheasnhisproasngluvalthrglu225230235240glualavalglualacysgluargmetalaasnalaglyileproleu245250255glyasnglnthrvalleuleuargglyileasnaspcysthrhisval260265270metlysargleuvalhisleuleuvallysmetargvalargprotyr275280285tyriletyrvalcysaspleuserleuglyileglyhispheargthr290295300provalserlysglyilegluileilegluasnleuargglyhisthr305310315320serglytyralavalprothrphevalvalaspalaproglyglygly325330335glylysileprovalmetproasntyrvalvalserglnserproarg340345350hisvalvalleuargasntyrgluglyvalilethrthrtyrthrglu355360365progluasntyrhisgluglucysaspcysgluaspcysargalagly370375380lyshislysgluglyvalalaalaleuserglyglyglnglnleuala385390395400ilegluproseraspleualaarglyslysarglyspheasplysasn405410415<210>88<211>425<212>prt<213>fusobacteriumnucleatum<400>88metasnthrvalasnthrarglyslysphepheproasnvalthrasp151015gluglutrpasnasptrpthrtrpglnvallysasnargileglulys202530ileaspaspleulyslystyrvalgluleuseralagluglugluglu354045glyvalvalargthrleugluthrleuargmetalailethrprotyr505560tyrpheserleuileaspmetasnseraspargcysproilearglys65707580glnalaileprothrileglngluilehisglnseraspalaaspleu859095leuaspproleuhisgluaspgluaspserprovalproglyleuthr100105110hisargtyrproaspargvalleuleuleuilethraspmetcysser115120125mettyrcysarghiscysthrargargargphealaglyserserasp130135140aspalametprometaspargileasplysalaileglutyrileala145150155160lysthrproglnvalargaspvalleuleuserglyglyaspalaleu165170175leuvalserasplyslysleugluserileileglnlysleuargala180185190ileprohisvalgluileileargileglyserargthrprovalval195200205leuproglnargilethrprogluleucysasnmetleulyslystyr210215220hisproiletrpleuasnthrhispheasnhisproglngluvalthr225230235240proglualalyslysalacysglumetleualaaspalaglyvalpro245250255leuglyasnglnthrvalleuleuargglyileasnaspservalpro260265270valmetlysargleuvalhisaspleuvalmetmetargvalargpro275280285tyrtyriletyrglncysaspleusermetglyleugluhisphearg290295300thrprovalserlysglyilegluileilegluglyleuargglyhis305310315320thrserglytyralavalprothrphevalvalaspalaproglygly325330335glyglylysthrprovalmetproglntyrvalileserglnserpro340345350hisargvalvalleuargasnphegluglyvalilethrthrtyrthr355360365gluprogluasntyrthrhisgluprocystyraspgluglulysphe370375380glulysmettyrgluileserglyvaltyrmetleuaspgluglyleu385390395400lysmetserleugluproserhisleualaarghisgluargasnlys405410415lysargalaglualagluglylyslys420425<210>89<211>517<212>prt<213>megasphaeraelsdenii<400>89metarglysvalgluileilethralagluglnalaalaglnleuval151015lysaspasnaspthrilethrserileglyphevalserseralahis202530proglualaleuthrlysalaleuglulysargpheleuaspthrasn354045thrproglnasnleuthrtyriletyralaglyserglnglylysarg505560aspglyargalaalagluhisleualahisthrglyleuleulysarg65707580alaileileglyhistrpglnthrvalproalaileglylysleuala859095valgluasnlysileglualatyrasnpheserglnglythrleuval100105110histrppheargalaleualaglyhislysleuglyvalphethrasp115120125ileglyleugluthrpheleuaspproargglnleuglyglylysleu130135140asnaspvalthrlysgluaspleuvallysleuilegluvalaspgly145150155160hisgluglnleuphetyrprothrpheprovalasnvalalapheleu165170175argglythrtyralaaspgluserglyasnilethrmetaspgluglu180185190ileglyprophegluserthrservalalaglnalavalhisasncys195200205glyglylysvalvalvalglnvallysaspvalvalalahisglyser210215220leuaspproargmetvallysileproglyiletyrvalasptyrval225230235240valvalalaalaprogluasphisglnglnthrtyraspcysglutyr245250255aspproserleuserglygluhisargalaprogluglyalathrasp260265270alaalaleuprometseralalyslysileileglyargargglyala275280285leugluleuthrgluasnalavalvalasnleuglyvalglyalapro290295300glutyrvalalaservalalaglyglugluglyilealaaspthrile305310315320thrleuthrvalgluglyglyalaileglyglyvalproglnglygly325330335alaargpheglyserserargasnalaaspalaileileasphisthr340345350tyrglnpheaspphetyraspglyglyglyleuaspilealatyrleu355360365glyleualaglncysaspglyserglyasnileasnvalserlysphe370375380glythrasnvalalaglycysglyglypheproasnileserglngln385390395400thrproasnvaltyrphecysglythrphethralaglyglyleulys405410415ilealavalgluaspglylysvallysileleuglngluglylysala420425430lyslyspheilelysalavalaspglnilethrpheasnglysertyr435440445alaalaargasnglylyshisvalleutyrilethrgluargcysval450455460phegluleuthrlysgluglyleulysleuilegluvalalaprogly465470475480ileaspileglulysaspileleualahismetaspphelysproile485490495ileaspasnprolysleumetaspalaargleupheglnaspglypro500505510metglyleulyslys515<210>90<211>145<212>prt<213>丙酸梭菌(clostridiumpropionicum)<400>90metvalglylyslysvalvalhishisleumetmetseralalysasp151015alahistyrthrglyasnleuvalasnglyalaargilevalasngln202530trpglyaspvalglythrgluleumetvaltyrvalaspglyaspile354045serleupheleuglytyrlysaspilegluphethralaprovaltyr505560valglyaspphemetglutyrhisglytrpileglulysvalglyasn65707580glnsertyrthrcyslyspheglualatrplysvalalathrmetval859095aspilethrasnproglnaspthrargalathralacysglupropro100105110valleucysglyargalathrglyserleupheilealalyslysasp115120125glnargglyproglngluserserphelysgluarglyshisprogly130135140glu145<210>91<211>260<212>prt<213>phytophthorasojae<400>91metalaalaglutyrgluserileleuthrgluvalargglylysval151015alaileilethrleuasnargprolysalaleuasnalaleucysser202530proleuileglugluleuasnglyalaalahisalapheaspalaasp354045proserileglyalailevalilethrglyserglyserlysalaphe505560alaalaglyalaaspilelysglumetalathrlysthrphevalasp65707580alatyrlysserasnmetphealaasntrpglyaspilethrlysval859095serlysprovalilealaalavalasnglytyralaleuglyglygly100105110cysgluleualametleucysaspleuileilealaglyaspserala115120125lyspheglyglnprogluilethrleuglythrileproglycysgly130135140glythrglnargleuileargalavalglylysserlysalametglu145150155160metileleuthrglyasnmetileaspalaglnglnalagluargasp165170175glyleuvalalaargvalvalproalaaspglnleuleuaspgluala180185190leulysthralaasnlysilealaserpheserglnprovalvallys195200205metalalysglualavalasnalaalatyrgluglnserleuglnglu210215220glyleulystyrgluserargleuphetrpserserphealathrlys225230235240aspglnlysgluglymetalaalaphevalglulysarglysalaasp245250255phelysaspglu260<210>92<211>258<212>prt<213>橙色绿屈挠菌<400>92metserglugluserleuvalleuserthrilegluglyproileala151015ileleuthrleuasnargproglnalaleuasnalaleuserproala202530leuileaspaspleuilearghisleuglualacysaspalaaspasp354045thrileargvalileileilethrglyalaglyargalaphealaala505560glyalaaspilelysalametalaasnalathrproileaspmetleu65707580thrserglymetilealaargtrpalaargilealaalavalarglys859095provalilealaalavalasnglytyralaleuglyglyglycysglu100105110leualametmetcysaspileileilealasergluasnalaglnphe115120125glyglnprogluileasnleuglyileileproglyalaglyglythr130135140glnargleuthrargalaleuglyprotyrargalametgluleuile145150155160leuthrglyalathrileseralaglnglualaleualahisglyleu165170175valcysargvalcysproprogluserleuleuaspglualaargarg180185190ilealaglnthrilealathrlysserproleualavalglnleuala195200205lysglualavalargmetalaalagluthrthrvalarggluglyleu210215220alailegluleuargasnphetyrleuleuphealaseralaaspgln225230235240lysgluglymetglnalapheileglulysargalaproasnpheser245250255glyarg<210>93<211>288<212>prt<213>rhodospirillumrubrum<400>93metalaalaalaproglyproproalaalaargproargalaalaarg151015glnserargmetileleuproproleuarggluglnalaglnmetala202530tyrgluasnileleuvalgluthrasnglylysvalglyilevalthr354045leuasnargprolysalaleuasnalaleuseralaglyleuvalarg505560aspleuglyalaalaleuaspalapheglualaaspvalasnvalhis65707580valilevalleuthrglyserasplysalaphealaalaglyalaasp859095ilelysglumetalaglulyssertyrmetaspalatyrleugluasp100105110pheilethrlysglytrpgluargvalthrthrcysarglysproile115120125ilealaalavalalaglyphealaleuglyglyglycysglumetala130135140metmetcysasppheileilealaalaglnasnalalyspheglygln145150155160progluileasnleuglythrleuproglyalaglyglythrglnarg165170175leuthrargphevalglylysserlysalametaspmetcysleuthr180185190glyargmetmetaspalaaspglualatrplyscysglyleuvalser195200205argilevalprovalaspaspleulysaspgluvalleulysileala210215220glualailealaasplysserleuproilethrmetmetvallysglu225230235240alavalasnalaalatyrgluthrthrleualaglnglyvalargphe245250255gluargargleupheglnalaserphealathraspaspglnlysglu260265270glymetasnalapheileglulysargglnproserphethrasparg275280285<210>94<211>258<212>prt<213>rhodobactercapsulatus<400>94metsertyrglnthrleuilevalgluilealaaspglyvalalaleu151015ileargleuasnargproglualaleuasnalaleuasnserglnleu202530leuglygluleualaalaalaleuserthrleuaspalaaspproala354045valargcysphevalleuthrglyserasplysalaphealaalagly505560alaaspilelysglumetalaasplysserphevalaspmetleulys65707580leuaspphepheglythrgluglyaspalaileleuargthrarglys859095provalilealaalavalalaglytyralaleuglyglyglycysglu100105110leualametmetcysasppheileleucysalagluasnalalysphe115120125glyglnprogluileasnleuglyvalvalalaglyileglyglythr130135140glnargleuthrargphevalglylysserlyssermetglumethis145150155160leuthrglyargphemetaspalaalaglualagluargserglyleu165170175valserargvalleuproleualaaspleuleuproglualaleuala180185190thralaarglysilealaglulysseralailealathrmetvalala195200205lysaspcysvalasnargalatyrgluthrthrleuarggluglyval210215220leutyrgluargargvalphehisalaleuphealathrgluaspgln225230235240lysgluglymetalaalaphealaglulysargproalalyspheala245250255asplys<210>95<211>290<212>prt<213>智人<400>95metalaalaleuargvalleuleusercysalaargglyproleuarg151015proprovalargcysproalatrpargprophealaserglyalaasn202530pheglutyrileilealaglulysargglylysasnasnthrvalgly354045leuileglnleuasnargprolysalaleuasnalaleucysaspgly505560leuileaspgluleuasnglnalaleulysilepheglugluasppro65707580alavalglyalailevalleuthrglyglyasplysalaphealaala859095glyalaaspilelysglumetglnasnleuserpheglnaspcystyr100105110serserlyspheleulyshistrpasphisleuthrglnvallyslys115120125provalilealaalavalasnglytyralapheglyglyglycysglu130135140leualametmetcysaspileiletyralaglyglulysalaglnphe145150155160alaglnprogluileleuileglythrileproglyalaglyglythr165170175glnargleuthrargalavalglylysserleualametglumetval180185190leuthrglyaspargileseralaglnaspalalysglnalaglyleu195200205valserlysilecysprovalgluthrleuvalgluglualailegln210215220cysalaglulysilealaserasnserlysilevalvalalametala225230235240lysgluservalasnalaalapheglumetthrleuthrgluglyser245250255lysleuglulyslysleuphetyrserthrphealathraspasparg260265270lysgluglymetthralaphevalglulysarglysalaasnphelys275280285aspgln290<210>96<211>399<212>prt<213>荧光假单胞菌<400>96metargvalvalleucyslysphealaleuleuargserhisaspgly151015serseralaasnileleuilelysasnasnlysserarggluleuser202530metthralaglnvalserthrglualaserhisalaalaileleugln354045aspgluvalleualagluvalargasnhisileglyhisleuthrleu505560asnargproalaglyleuasnalaleuthrleuglnmetvalargser65707580leuthrserglnleuglnalatrpseraspaspproglnvaltyrala859095valvalleuargglyalaglyglulysalaphecysalaglyglyasp100105110ileargserleutyraspserphelysasnglyaspthrleuhisgln115120125aspphephevalgluglutyralaleuaspleualailehishistyr130135140arglysprovalleualaleumetaspglyphevalleuglyglygly145150155160metglyleuvalglnglyalaaspleuargvalvalthrgluargser165170175argleualametprogluvalalaileglytyrpheproaspvalgly180185190glysertyrpheleuproargileproglygluleuglyiletyrleu195200205glyvalthrglyvalglnileargalaalaaspalaleutyrcysgly210215220leualaasptrptyrleuaspserhislysleualaaspleuaspgln225230235240lysleuaspasnleuargtrphisaspserproleulysaspleugln245250255glyalaleualaargleualavalglnglnleuproaspalaproleu260265270alaalaleuargproalaileasphisphephealaleuproaspval275280285proserilevalgluglnleuglnglnvalthrvalalaaspserhis290295300glutrpalaleuasnthrvalserleumetglnthrargserproleu305310315320alametalavalthrleuglumetleuargargglyargargleuser325330335leugluglncysphealaleugluleuhisleuaspargglntrpphe340345350gluargglyaspleuilegluglyvalargalaleuileileasplys355360365asplysserproargtrpasnproprothrleuhisglyleualaleu370375380asnhisvalgluserphephehishispheglulysvalvallys385390395<210>97<211>351<212>prt<213>蜡状芽孢杆菌<400>97metthrgluglnvalleupheservalsergluasnglyvalalathr151015ilethrleuasnargprolysalaleuasnserleusertyraspmet202530leuglnproileglyglnlysleulysglutrpgluhisaspgluarg354045ilealaleuilevalleulysglyalaglythrlysglyphecysala505560glyglyaspilelysthrleutyrglualaargserasngluvalala65707580leuglnhisalagluargphepheglugluglutyrgluileaspthr859095tyriletyrglntyrthrlysproileilealacysleuaspglyile100105110valmetglyglyglyvalglyleuthrasnglyalalystyrargile115120125valthrgluargthrlystrpalametproglumetasnileglyphe130135140pheproaspvalglyalaalatyrpheleuasnlysalaproglytyr145150155160thrglyargphevalalaleuthralaserileleulysalaserasp165170175valleupheileasnalaalaasptyrphemetthrseraspserleu180185190proglupheleuthrgluleugluservalasntrphislysgluasp195200205aspvalhisthrasnleulysgluvalileargthrphealathrala210215220proasnleuglusergluleualaproserleugluvalileasnser225230235240hisphealapheaspthrileglugluileilehisserleuglulys245250255aspgluserserphealaleulysthrlysgluileleuleuserlys260265270serproileserleulysvalthrleulysglnpheileaspglygln275280285asplysservalgluglucysphealathraspleuileleualalys290295300asnphemetarghisgluaspphephegluglyvalargservalval305310315320valasplysaspglnasnproasntyrlystyrlysglnleuserasp325330335valserglugluaspvalasnargphepheasnleuleuasnala340345350<210>98<211>381<212>prt<213>智人<400>98mettrpargleumetserargpheasnalaphelysargthrasnthr151015ileleuhishisleuargmetserlyshisthraspalaalagluglu202530valleuleuglulyslysglycysalaglyvalilethrleuasnarg354045prolyspheleuasnalaleuthrleuasnmetileargglniletyr505560proglnleulyslystrpgluglnaspprogluthrphevalileile65707580ilelysglyalaglyglylysalaphecysalaglyglyaspilearg859095valileserglualaglulysalalysglnlysilealaprovalphe100105110phearggluglutyrmetleuasnasnalavalglysercysglnlys115120125protyrvalalaleuilehisglyilethrmetglyglyglyvalgly130135140leuservalhisglyglnpheargvalalathrglulyscysleuphe145150155160alametprogluthralaileglyleupheproaspvalglyglygly165170175tyrphephealathrthrproarglysthrtrpleuleuprocysile180185190asnglypheargleulysglyargaspvaltyrargalaglyileala195200205thrhisphevalaspserglulysleualametleuglugluaspleu210215220leualaleulysserproserlysgluasnilealaservalleuglu225230235240asntyrhisthrgluserlysileaspargasplysserpheileleu245250255glugluhismetasplysileasnsercyspheseralaasnthrval260265270glugluileilegluasnleuglnglnaspglyserserphealaleu275280285gluglnleulysvalileasnlysmetserprothrserleulysile290295300thrleuargglnleumetgluglyserserlysthrleuglngluval305310315320leuthrmetglutyrargleuserglnalacysmetargglyhisasp325330335phehisgluglyvalargalavalleuileasplysaspglnserpro340345350lystrplysproalaaspleulysgluvalthrglugluaspleuasn355360365asnhisphelysserleuglyserseraspleulysphe370375380<210>99<211>261<212>prt<213>megasphaeraelsdenii<400>99vallysthrvaltyrthrleuglyileaspvalglyserserserser151015lysalavalileleugluaspglylyslysilevalalahisalaval202530valgluileglythrglyserthrglyprogluargvalleuaspglu354045valphelysaspthrasnleulysilegluaspmetalaasnileile505560alathrglytyrglyargpheasnvalaspcysalalysglygluval65707580sergluilethrcyshisalalysglyalaleupheglucysprogly859095thrthrthrileleuaspileglyglyglnaspvallysserilelys100105110leuasnglyglnglyleuvalmetglnphealametasnasplyscys115120125alaalaglythrglyargpheleuaspvalmetserlysvalleuglu130135140ileprometserglumetglyasptrptyrphelysserlyshispro145150155160alaalavalserserthrcysthrvalphealaglusergluvalile165170175serleuleuserlysasnvalprolysgluaspilevalalaglyval180185190hisglnserilealaalalysalacysalaleuvalargargvalgly195200205valglygluaspleuthrmetthrglyglyglyserargaspprogly210215220valvalaspalavalserlysgluleuglyileprovalargvalala225230235240leuhisproglnalavalglyalaleuglyalaalaleuilealatyr245250255asplysilelyslys260<210>100<211>428<212>prt<213>megasphaeraelsdenii<400>100metsergluglulysthrvalaspileglusermetserserlysglu151015alaleuglytyrpheleuprolysvalaspgluaspalaarglysala202530lyslysgluglyargleuvalcystrpseralaservalalapropro354045gluphecysthralametaspilealailevaltyrprogluthrhis505560alaalaglyileglyalaarghisglyalaproalametleugluval65707580alagluasnlysglytyrasnglnaspilecyssertyrcysargval859095asnmetglytyrmetgluleuleulysglnglnalaleuthrglyglu100105110thrprogluvalleulysasnserproalaserproileproleupro115120125aspvalvalleuthrcysasnasnilecysasnthrleuleulystrp130135140tyrgluasnleualalysgluleuasnvalproleuileasnileasp145150155160valpropheasnhisglupheprovalthrlyshisalalysglntyr165170175ilevalglygluphelyshisalailelysglnleugluaspleucys180185190glyargpropheasptyrasplysphephegluvalglnlysglnthr195200205glnargserilealaalatrpasnlysilealathrtyrpheglntyr210215220lysproserproleuasnglypheaspleupheasntyrmetglyleu225230235240alavalalaalaargserleuasntyrsergluilethrpheasnlys245250255pheleulysgluleuaspglulysvalalaasnlyslystrpalaphe260265270glygluasnglulysserargvalthrtrpgluglyilealavaltrp275280285ilealaleuglyhisthrphelysgluleulysglyglnglyalaleu290295300metthrglyseralatyrproglymettrpaspvalsertyrglupro305310315320glyaspleuglusermetalaglualatyrserargthrtyrileasn325330335cyscysleugluglnargglyalavalleuglulysvalvalargasp340345350glylyscysaspglyleuilemethisglnasnargsercyslysasn355360365metserleuleuasnasngluglyglyglnargileglnlysasnleu370375380glyvalprotyrvalilepheaspglyaspglnthraspalaargasn385390395400pheserglualaglnpheaspthrargvalglualaleualaglumet405410415metalaasplyslysalaasngluglyglyasnhis420425<210>101<211>372<212>prt<213>megasphaeraelsdenii<400>101metserglnileaspgluleuileserlysleuglngluvalserasn151015hisproglnlysthrvalleuasntyrlyslysglnglylysglyleu202530valglymetmetprotyrtyralaproglugluilevaltyralaala354045glytyrleuprovalglymetpheglyserglnasnproglnileser505560alaalaargthrtyrleuproprophealacysserleumetglnala65707580aspmetgluleuglnleuasnglythrtyraspcysleuaspalaval859095ilepheservalprocysaspthrleuargcysmetserglnlystrp100105110hisglylysalaprovalilevalphethrglnproglnasnarglys115120125ileargproalavalasppheleulysalaglutyrgluhisvalarg130135140thrgluleuglyargileleuasnvallysileseraspleualaile145150155160glnglualailelysvaltyrasngluasnargglnvalmetargglu165170175phecysaspvalalaalaglntyrproglnilephethrproilelys180185190arghisaspvalilelysalaargtrpphemetasplysalagluhis195200205thralaleuvalarggluleuileaspalavallyslysgluproval210215220glnprotrpasnglylyslysvalileleuserglyilemetalaglu225230235240proaspglupheleuaspilepheserglupheasnilealavalval245250255alaaspaspleualaglngluserargglnpheargthraspvalpro260265270serglyileaspproleugluglnleualaglnglntrpglnaspphe275280285aspglycysproleualaleuasngluasplysproargglyglnmet290295300leuileaspmetthrlyslystyrasnalaaspalavalvalilecys305310315320metmetargphecysaspprogluglupheasptyrproiletyrlys325330335proglupheglualaalaglyvalargtyrthrvalleuaspleuasp340345350ilegluserproserleugluglnleuargthrargileglnalaphe355360365sergluileleu370<210>102<211>519<212>prt<213>酿酒酵母<400>102metpheserargserthrleucysleulysthrseralaserserile151015glyargleuglnleuargtyrpheserhisleuprometthrvalpro202530ilelysleuproasnglyleuglutyrgluglnprothrglyleuphe354045ileasnasnlysphevalproserlysglnasnlysthrphegluval505560ileasnproserthrgluglugluilecyshisiletyrgluglyarg65707580gluaspaspvalgluglualavalglnalaalaaspargalapheser859095asnglysertrpasnglyileaspproileaspargglylysalaleu100105110tyrargleualagluleuilegluglnasplysaspvalilealaser115120125ilegluthrleuaspasnglylysalaileserserserargglyasp130135140valaspleuvalileasntyrleulysserseralaglyphealaasp145150155160lysileaspglyargmetileaspthrglyargthrhisphesertyr165170175thrlysargglnproleuglyvalcysglyglnileileprotrpasn180185190pheproleuleumettrpalatrplysilealaproalaleuvalthr195200205glyasnthrvalvalleulysthralagluserthrproleuserala210215220leutyrvalserlystyrileproglnalaglyileproproglyval225230235240ileasnilevalserglypheglylysilevalglyglualailethr245250255asnhisprolysilelyslysvalalaphethrglyserthralathr260265270glyarghisiletyrglnseralaalaalaglyleulyslysvalthr275280285leugluleuglyglylysserproasnilevalphealaaspalaglu290295300leulyslysalavalglnasnileileleuglyiletyrtyrasnser305310315320glygluvalcyscysalaglyserargvaltyrvalglugluserile325330335tyrasplyspheileglugluphelysalaalasergluserilelys340345350valglyasppropheaspgluserthrpheglnglyalaglnthrser355360365glnmetglnleuasnlysileleulystyrvalaspileglylysasn370375380gluglyalathrleuilethrglyglygluargleuglyserlysgly385390395400tyrpheilelysprothrvalpheglyaspvallysgluaspmetarg405410415ilevallysglugluilepheglyprovalvalthrvalthrlysphe420425430lysseralaaspgluvalileasnmetalaasnaspserglutyrgly435440445leualaalaglyilehisthrserasnileasnthralaleulysval450455460alaaspargvalasnalaglythrvaltrpileasnthrtyrasnasp465470475480phehishisalavalpropheglyglypheasnalaserglyleugly485490495argglumetservalaspalaleuglnasntyrleuglnvallysala500505510valargalalysleuaspglu515<210>103<211>495<212>prt<213>大肠杆菌<400>103metasnphehishisleualatyrtrpglnasplysalaleuserleu151015alailegluasnargleupheileasnglyglutyrthralaalaala202530gluasngluthrphegluthrvalaspprovalthrglnalaproleu354045alalysilealaargglylysservalaspileaspargalametser505560alaalaargglyvalphegluargglyasptrpserleuserserpro65707580alalysarglysalavalleuasnlysleualaaspleumetgluala859095hisalaglugluleualaleuleugluthrleuaspthrglylyspro100105110ilearghisserleuargaspaspileproglyalaalaargalaile115120125argtrptyralaglualaileasplysvaltyrglygluvalalathr130135140thrserserhisgluleualametilevalarggluprovalglyval145150155160ilealaalailevalprotrpasnpheproleuleuleuthrcystrp165170175lysleuglyproalaleualaalaglyasnservalileleulyspro180185190serglulysserproleuseralaileargleualaglyleualalys195200205glualaglyleuproaspglyvalleuasnvalvalthrglyphegly210215220hisglualaglyglnalaleuserarghisasnaspileaspalaile225230235240alaphethrglyserthrargthrglylysglnleuleulysaspala245250255glyaspserasnmetlysargvaltrpleuglualaglyglylysser260265270alaasnilevalphealaaspcysproaspleuglnglnalaalaser275280285alathralaalaglyilephetyrasnglnglyglnvalcysileala290295300glythrargleuleuleuglugluserilealaaspglupheleuala305310315320leuleulysglnglnalaglnasntrpglnproglyhisproleuasp325330335proalathrthrmetglythrleuileaspcysalahisalaaspser340345350valhisserpheilearggluglygluserlysglyglnleuleuleu355360365aspglyargasnalaglyleualaalaalaileglyprothrilephe370375380valaspvalaspproasnalaserleuserargglugluilephegly385390395400provalleuvalvalthrargphethrserglugluglnalaleugln405410415leualaasnaspserglntyrglyleuglyalaalavaltrpthrarg420425430aspleuserargalahisargmetserargargleulysalaglyser435440445valphevalasnasntyrasnaspglyaspmetthrvalprophegly450455460glytyrlysglnserglyasnglyargasplysserleuhisalaleu465470475480glulysphethrgluleulysthriletrpileserleugluala485490495<210>104<211>495<212>prt<213>肺炎克雷伯氏菌(klebsiellapneumoniae)<400>104metasnpheglnhisleualatyrtrpglnglulysalalysasnleu151015alailegluthrargleupheileasnglyglutyrcysalaalaala202530aspasnthrthrphegluthrileaspproalaalaglnglnthrleu354045alaglnvalalaargglylyslysalaaspvalgluargalavallys505560alaalaargglnalapheaspasnglyasptrpserglnalaserpro65707580alaglnarglysalaileleuthrargphealaaspleumetgluala859095hisargglugluleualaleuleugluthrleuaspthrglylyspro100105110ilearghisserleuargaspaspileproglyalaalaargalaile115120125argtrptyralaglualaleuasplysvaltyrglygluvalalapro130135140thrglyserasngluleualametilevalarggluproileglyval145150155160ilealaalavalvalprotrpasnpheproleuleuleualacystrp165170175lysleuglyproalaleualaalaglyasnservalileleulyspro180185190serglulysserproleuthralaleuargleualaglyleualalys195200205glualaglyleuproaspglyvalleuasnvalvalserglyphegly210215220hisglualaglyglnalaleualaleuhisproaspvalgluvalile225230235240thrphethrglyserthrargthrglylysglnleuleulysaspala245250255glyaspserasnmetlysargvaltrpleuglualaglyglylysser260265270alaasnilevalphealaaspcysproaspleuglnglnalavalarg275280285alathralaglyglyilephetyrasnglnglyglnvalcysileala290295300glythrargleuleuleuglugluserilealaaspglupheleuala305310315320argleulysalaglualaglnhistrpglnproglyasnproleuasp325330335proaspthrthrmetglymetleuileaspasnthrhisalaaspasn340345350valhisserpheileargglyglygluserglnserthrleupheleu355360365aspglyarglysasnprotrpproalaalavalglyprothrilephe370375380valaspvalaspproalaserthrleuserargglugluilephegly385390395400provalleuvalvalthrargphelysserglugluglualaleulys405410415leualaasnaspserasptyrglyleuglyalaalavaltrpthrarg420425430aspleuserargalahisargmetserargargleulysalaglyser435440445valphevalasnasntyrasnaspglyaspmetthrvalprophegly450455460glytyrlysglnserglyasnglyargasplysserleuhisalaleu465470475480glulysphethrgluleulysthriletrpilealaleugluser485490495<210>105<211>1053<212>dna<213>东方伊萨酵母<400>105atgtcttacgaaatcccacaaacacaaaaggcctgtgtcttttacgaaaacggcggccca60atcacatacaaggacattccagttccaaagccaaaacctactgagattttagtcaaggtt120ctgtactctggtgtctgccacaccgacttgcacgcatggaagggtgactggcctctagct180accaagttgccattggttggtggtcacgaaggtgccggtgttgttgttgccaagggtgaa240aacgtcacctcttttgagattggtgattacgcaggtatcaagtggttgaatggttcatgt300atgggttgtgaattctgtgaacaaggtgctgaaccaaactgtcctaaggccgacttgagt360ggttacacccacgacggttccttccaacagtatgctactgctgacgctattcaagctgca420cacatctccaaggaaaccgacttggctggtgttgctccaatcttgtgtgcaggtgtcact480gtctacaaggctttaaagactgcagaccttagagcaggtgaatgggtttgtatttccggt540gcagctggtggtttaggttctcttgctattcaatatgcaaaggctatgggtctgagagtt600gttggtattgacggtggtgacgaaaagaaggaattgtgtaaatcccttggtgctgaagca660tttattgatttcacaaagaccaaggatatcgtcaaggctgtccaagaggcaaccaatggt720ggtccacatggtgtcatcaatgtctctgtctctgaagctgcaatttctcaatcttgtgaa780tacgttagacctctaggtaaggttgttcttgttggtttaccagcaggcgcacaagtcaaa840actggtgtctttgaagccgttgtcaagtctattgaaattaagggttcttatgtcggtaac900agaaaggataccgccgaagcacttgacttctacactagaggcttggtcaagtctccattc960aagattgtcggtttatccgaattgccaaaagtctttgaactcatggaacagggtaagatt1020ttaggtagaatggtcttagacacctccaaataa1053<210>106<211>350<212>prt<213>东方伊萨酵母<400>106metsertyrgluileproglnthrglnlysalacysvalphetyrglu151015asnglyglyproilethrtyrlysaspileprovalprolysprolys202530prothrgluileleuvallysvalleutyrserglyvalcyshisthr354045aspleuhisalatrplysglyasptrpproleualathrlysleupro505560leuvalglyglyhisgluglyalaglyvalvalvalalalysglyglu65707580asnvalthrserphegluileglyasptyralaglyilelystrpleu859095asnglysercysmetglycysgluphecysgluglnglyalaglupro100105110asncysprolysalaaspleuserglytyrthrhisaspglyserphe115120125glnglntyralathralaaspalaileglnalaalahisileserlys130135140gluthraspleualaglyvalalaproileleucysalaglyvalthr145150155160valtyrlysalaleulysthralaaspleuargalaglyglutrpval165170175cysileserglyalaalaglyglyleuglyserleualaileglntyr180185190alalysalametglyleuargvalvalglyileaspglyglyaspglu195200205lyslysgluleucyslysserleuglyalaglualapheileaspphe210215220thrlysthrlysaspilevallysalavalglnglualathrasngly225230235240glyprohisglyvalileasnvalservalserglualaalaileser245250255glnsercysglutyrvalargproleuglylysvalvalleuvalgly260265270leuproalaglyalaglnvallysthrglyvalpheglualavalval275280285lysserilegluilelysglysertyrvalglyasnarglysaspthr290295300alaglualaleuaspphetyrthrargglyleuvallysserprophe305310315320lysilevalglyleusergluleuprolysvalphegluleumetglu325330335glnglylysileleuglyargmetvalleuaspthrserlys340345350<210>107<211>1131<212>dna<213>东方伊萨酵母<400>107atgtttgcatcaaccttcagaagtcaagctgtaagagctgcaagatttactagattccaa60tccacttttgccattcctgagaagcaaatgggtgttatctttgaaactcatggtggtcct120ttacaatacaaggaaattccagttccaaaaccaaaaccaactgaaattttaatcaatgtt180aaatactctggtgtctgccataccgatttacacgcatggaaaggtgactggccattacca240gcaaagttacccctagttggtggtcacgaaggtgcgggcattgttgttgcgaaaggttct300gcagttaccaactttgagattggcgattatgctggtattaagtggttaaacggttcatgt360atgtcatgtgaattctgtgaacaaggtgatgaatctaactgtgaacatgccgatttgagt420ggttatactcatgatggttctttccaacaatatgccactgctgacgctattcaagctgca480aagatcccaaagggtaccgacttatctgaagttgcgccaattttatgtgctggtgttact540gtctataaagctttgaaaactgctgatttaagagcaggtcaatgggttgcgatttctggt600gccgctggtggtctaggttctcttgctgtccaatatgcaaaggcaatgggtctaagagtt660ttaggtatcgatggtggtgaaggtaaaaaggaactttttgaacaatgtggtggtgatgtg720tttatcgatttcaccagatacccaagagatgcacctgaaaagatggttgctgatattaag780gctgcaactaacggtttgggtccacacggtgttatcaatgtctctgtctccccagctgct840atctctcaatcatgtgactatgttagagcaactggtaaggttgtccttgtcggtatgcca900tctggtgctgtctgtaagtctgatgtcttcactcatgttgttaaatccttacaaattaaa960ggttcttatgttggtaacagagcagataccagagaagctttggaattctttaatgaaggt1020aaggtcagatctccaatcaaggttgtcccattatctactttacctgaaatttacgaattg1080atggagcaaggtaagattttaggtagatacgttgttgatacttctaaataa1131<210>108<211>376<212>prt<213>东方伊萨酵母<400>108metphealaserthrpheargserglnalavalargalaalaargphe151015thrargpheglnserthrphealaileproglulysglnmetglyval202530ilephegluthrhisglyglyproleuglntyrlysgluileproval354045prolysprolysprothrgluileleuileasnvallystyrsergly505560valcyshisthraspleuhisalatrplysglyasptrpproleupro65707580alalysleuproleuvalglyglyhisgluglyalaglyilevalval859095alalysglyseralavalthrasnphegluileglyasptyralagly100105110ilelystrpleuasnglysercysmetsercysgluphecysglugln115120125glyaspgluserasncysgluhisalaaspleuserglytyrthrhis130135140aspglyserpheglnglntyralathralaaspalaileglnalaala145150155160lysileprolysglythraspleusergluvalalaproileleucys165170175alaglyvalthrvaltyrlysalaleulysthralaaspleuargala180185190glyglntrpvalalaileserglyalaalaglyglyleuglyserleu195200205alavalglntyralalysalametglyleuargvalleuglyileasp210215220glyglygluglylyslysgluleuphegluglncysglyglyaspval225230235240pheileaspphethrargtyrproargaspalaproglulysmetval245250255alaaspilelysalaalathrasnglyleuglyprohisglyvalile260265270asnvalservalserproalaalaileserglnsercysasptyrval275280285argalathrglylysvalvalleuvalglymetproserglyalaval290295300cyslysseraspvalphethrhisvalvallysserleuglnilelys305310315320glysertyrvalglyasnargalaaspthrargglualaleugluphe325330335pheasngluglylysvalargserproilelysvalvalproleuser340345350thrleuprogluiletyrgluleumetgluglnglylysileleugly355360365argtyrvalvalaspthrserlys370375<210>109<211>1134<212>dna<213>东方伊萨酵母<400>109atgttatccaagaccatcactgctgcattgaggggcaatacaactcgtactgcattcaga60atcaatgccattagaagtttagcgatcccagctattccagagacacaaaagggtgttatc120ttttatgagaacggaggtgaactattttacaaggacattccagttccaaagccaaagcca180aatgagattttggtgaatgtcaagtattctggtgtttgtcataccgatttacacgcatgg240aaaggtgactggcctttggcgaccaagttgccattggttggtggacatgaaggtgccgga300gttgttgttgctaagggggacaatgtcaccaactttgaaattggcgattatgccggtatc360aagtggttgaatggttcatgtatggggtgtgaattttgccaacaaggtgcagagccaaac420tgtccacaggccgacttgagtggttacacccatgacgggtcctttcaacaatatgccact480gccgatgctgttcaggcagccaagattcctcagggcactgatttggctcaagttgcgcca540attttatgtgcaggtattactgtctataaggctttaaagactgcagaattaagaccaggt600caatgggttgccatttctggtgctgctggaggtttaggttctcttgctgttcaatatgcc660aaggccatgggtttgagagttttgggtattgatggtggtgaggagaagggcaagtttgca720aagtctcttggagctgaagttttcattgatttcaccaaatccaaggacattgtcaaggat780atccaagaggccaccaatggtggtccacatggtgtcattaatgtttctgtttctccagct840gctatttctcaaagtacccagtatgtcagaaccttgggtaaggttgtccttgttggatta900ccagcgcatgctgtatgcgagtcttcggttttcgaccatgttgtcaagtcgattcaaatt960agaggctcttatgttggtaacagggaagatactagtgaggctattgattttttcaccagg1020ggtttagtgaagtcaccaattaagattgttggtttgagtgagttgccaaagatctatgaa1080ttgatggagcaaggtaagattttaggcagatatgttgttgacacttcgaaatga1134<210>110<211>377<212>prt<213>东方伊萨酵母<400>110metleuserlysthrilethralaalaleuargglyasnthrthrarg151015thralapheargileasnalaileargserleualaileproalaile202530progluthrglnlysglyvalilephetyrgluasnglyglygluleu354045phetyrlysaspileprovalprolysprolysproasngluileleu505560valasnvallystyrserglyvalcyshisthraspleuhisalatrp65707580lysglyasptrpproleualathrlysleuproleuvalglyglyhis859095gluglyalaglyvalvalvalalalysglyaspasnvalthrasnphe100105110gluileglyasptyralaglyilelystrpleuasnglysercysmet115120125glycysgluphecysglnglnglyalagluproasncysproglnala130135140aspleuserglytyrthrhisaspglyserpheglnglntyralathr145150155160alaaspalavalglnalaalalysileproglnglythraspleuala165170175glnvalalaproileleucysalaglyilethrvaltyrlysalaleu180185190lysthralagluleuargproglyglntrpvalalaileserglyala195200205alaglyglyleuglyserleualavalglntyralalysalametgly210215220leuargvalleuglyileaspglyglygluglulysglylyspheala225230235240lysserleuglyalagluvalpheileaspphethrlysserlysasp245250255ilevallysaspileglnglualathrasnglyglyprohisglyval260265270ileasnvalservalserproalaalaileserglnserthrglntyr275280285valargthrleuglylysvalvalleuvalglyleuproalahisala290295300valcysgluserservalpheasphisvalvallysserileglnile305310315320argglysertyrvalglyasnarggluaspthrserglualaileasp325330335phephethrargglyleuvallysserproilelysilevalglyleu340345350sergluleuprolysiletyrgluleumetgluglnglylysileleu355360365glyargtyrvalvalaspthrserlys370375<210>111<211>1347<212>dna<213>东方伊萨酵母<400>111atgtctccttcacaaattaacgttgacaacttatctaattggactgaagaattcaaatct60gacgccaagactcaaatcgggggttctgtattgcaacattccaacattgatgaggtcttg120attaacagagatgcagaaatcgccaacaagcatatcttcaaccacaagattgaaattgaa180ggtctacctgtcatggatcagaaggcttctggtagatgttggttgtttgcatcgactaac240ttgatgcgtgttactgcaatgaagaaatacaatttgaaggaaatcaagctttccccatcg300tatttgtttttctatgacaaattggaaagagcaaactatttccttgaacaaatcatcgac360actcataaggaaccaatcgattcaagattggttcaatatttcctgaccaatccagttgaa420gatggtggtcaattcaccatgatggcacaaattgctaccaaatacggtgttgttcctgat480caagtctacccagattctttcaacacaaccacttcgaggattatgaacagattagtcaac540cacagattacgttcttatgcaatgactttacgtaacgctctagatgaaggtaaagatgta600atgtccttgaagaatgagatgcaaaaagaaatttatcgtttgctaacaatgttccttggt660aacccaccaaagccaaacgaagagtttgtctgggaattcaccgataaagatggtaaatat720gaatctattaaaactacaccattaaaatatgcaactgaagttttggatttccatgctcca780gaatatgtttccttgttaaatgacccaagaaataagtataacaagatggttcaagttgaa840agattaggtaatgttgctggtggcgaaccagttgcatacttaaacttagaaattgaaaag900ttatctcaagctgttgttaacagaatcaaaaataacaaaccagttttctttggtaccgat960acacctaaatttatggataaaagtagaggtattatggatatcaatttatgggactatgag1020ttattaggttatgatgtccgtaccatgtcaaagaaggaaagagttgtttttggtgattct1080ttaatgacccacgctatgttgattactgcagtgcacgttgatgaaaatggcaaacctgtc1140agatacagagtcgaaaacagttggggtaccaagagtggtcaagaaggttattacacaatg1200acccaagaatattttgaagagtacgtttatcaagtagtcattgaaaagagtgaatttgct1260gccctaaacctcgatgtttccattctggaggataaagaaccagtcgtcttgccaccttat1320gaccctatgggtgcacttgctttataa1347<210>112<211>448<212>prt<213>东方伊萨酵母<400>112metserproserglnileasnvalaspasnleuserasntrpthrglu151015gluphelysseraspalalysthrglnileglyglyservalleugln202530hisserasnileaspgluvalleuileasnargaspalagluileala354045asnlyshisilepheasnhislysilegluilegluglyleuproval505560metaspglnlysalaserglyargcystrpleuphealaserthrasn65707580leumetargvalthralametlyslystyrasnleulysgluilelys859095leuserprosertyrleuphephetyrasplysleugluargalaasn100105110tyrpheleugluglnileileaspthrhislysgluproileaspser115120125argleuvalglntyrpheleuthrasnprovalgluaspglyglygln130135140phethrmetmetalaglnilealathrlystyrglyvalvalproasp145150155160glnvaltyrproaspserpheasnthrthrthrserargilemetasn165170175argleuvalasnhisargleuargsertyralametthrleuargasn180185190alaleuaspgluglylysaspvalmetserleulysasnglumetgln195200205lysgluiletyrargleuleuthrmetpheleuglyasnproprolys210215220proasnglugluphevaltrpgluphethrasplysaspglylystyr225230235240gluserilelysthrthrproleulystyralathrgluvalleuasp245250255phehisalaproglutyrvalserleuleuasnaspproargasnlys260265270tyrasnlysmetvalglnvalgluargleuglyasnvalalaglygly275280285gluprovalalatyrleuasnleugluileglulysleuserglnala290295300valvalasnargilelysasnasnlysprovalphepheglythrasp305310315320thrprolysphemetasplysserargglyilemetaspileasnleu325330335trpasptyrgluleuleuglytyraspvalargthrmetserlyslys340345350gluargvalvalpheglyaspserleumetthrhisalametleuile355360365thralavalhisvalaspgluasnglylysprovalargtyrargval370375380gluasnsertrpglythrlysserglyglngluglytyrtyrthrmet385390395400thrglnglutyrphegluglutyrvaltyrglnvalvalileglulys405410415sergluphealaalaleuasnleuaspvalserileleugluasplys420425430gluprovalvalleuproprotyraspprometglyalaleualaleu435440445<210>113<211>1737<212>dna<213>东方伊萨酵母<400>113atgttactcagatcactaaactcttctgctcgttgtgtcaaacaaacaaccagaacaaag60gttaggtatctcagccacgtcagtggtgcaagcatggcgaaacctacattgaagaacaac120tcgagagaatccaacaaatccagaaactatctaattgctgctgtgacagcattggctgta180tcaacctcaattggagttgccgtacatgtgaaggaccccttgtataacgatgctaccggc240agtgattctccgagaagtatatctgttgacgagtttgtcaagcataattcacaaaacgac300tgttggattgcaatcaatggcaaggtttatgatttcactgattttattccaaaccatcca360ggtggggtacctccattagttaatcatgctggttatgatggtactaaactttatgagaaa420ttgcatccaaaaggtacaattgagaaattcttgccaaaggataagtttctgggtgtgtta480gatggtgaagcgccaaaattggaagcagactatttggtggacgatgatgaacaagagaga540ctggattatttgaacaacttacctcctttgtcatctattcagaatgtttatgatttcgaa600tacttggccaagaagattttacctaaagatgcctgggcatattattcttgtggtgccgat660gatgaaatcacaatgagagaaaaccattatgcttatcaaagagtttatttcagaccaaga720atttgtgttgatgtcaaggaagttgatacttcttatgaaatgttaggcactaaaacctct780gttcctttttatgtatctgccaccgctttggctaaattaggccatcctgatggtgaatgc840tcaattgctagaggcgctggtaaggaaggtgtcgttcaaatgatttcgaccctttcctca900atgtcattagatgaaattgccgctgctagaattccaggtgcaacccaatggttccaatta960tacattaatgaggatagaaatgtcgctaaaggtctggtcaaacatgcagaagacttgggt1020atgaaggctatctttataactgttgatgctccttctctaggtaacagagaaaaggataaa1080agattaaagtttgttaatgacaccgatgtcgatttgggtgattccgcagatcgaaacagt1140ggtgcttcaaaggcactatcttcgttcattgatgcttctgtctcttggaatgacgtcaaa1200gcggtcaagtcgtggactaaattgcctgtcttagttaaaggtgttcaaacagttgaagac1260gttattgaagcttacgatgctggttgtcaaggtgttgttttgtcaaaccacggtggtagg1320caactagatactgctcctcctccaatcgaattattagctgaaactgttccaactttgaag1380agattgggtaaattaagaccagattttgaaattttaattgacggtggtgtcaaaagaggt1440accgatattttgaaagcagtcgcaatcggtggccaagatgtcagagtttcagttggtatg1500ggtagacctttcttatatgccaactcttgctatggtgaagcaggtgttagaaaattaatt1560caaaatctaaaggatgaattagaaatggatatgagattgttgggtgtcactaaaatggac1620cagctatcttcgaaacatgtcgatactaaacgtttgattggtagagatgcgatcaactat1680ttgtatgataatgtatacagcccaatcgaaaccgttaaattcaacaatgaagattga1737<210>114<211>578<212>prt<213>东方伊萨酵母<400>114metleuleuargserleuasnserseralaargcysvallysglnthr151015thrargthrlysvalargtyrleuserhisvalserglyalasermet202530alalysprothrleulysasnasnserarggluserasnlysserarg354045asntyrleuilealaalavalthralaleualavalserthrserile505560glyvalalavalhisvallysaspproleutyrasnaspalathrgly65707580seraspserproargserileservalaspgluphevallyshisasn859095serglnasnaspcystrpilealaileasnglylysvaltyraspphe100105110thrasppheileproasnhisproglyglyvalproproleuvalasn115120125hisalaglytyraspglythrlysleutyrglulysleuhisprolys130135140glythrileglulyspheleuprolysasplyspheleuglyvalleu145150155160aspglyglualaprolysleuglualaasptyrleuvalaspaspasp165170175gluglngluargleuasptyrleuasnasnleuproproleuserser180185190ileglnasnvaltyrasppheglutyrleualalyslysileleupro195200205lysaspalatrpalatyrtyrsercysglyalaaspaspgluilethr210215220metarggluasnhistyralatyrglnargvaltyrpheargproarg225230235240ilecysvalaspvallysgluvalaspthrsertyrglumetleugly245250255thrlysthrservalprophetyrvalseralathralaleualalys260265270leuglyhisproaspglyglucysserilealaargglyalaglylys275280285gluglyvalvalglnmetileserthrleusersermetserleuasp290295300gluilealaalaalaargileproglyalathrglntrppheglnleu305310315320tyrileasngluaspargasnvalalalysglyleuvallyshisala325330335gluaspleuglymetlysalailepheilethrvalaspalaproser340345350leuglyasnargglulysasplysargleulysphevalasnaspthr355360365aspvalaspleuglyaspseralaaspargasnserglyalaserlys370375380alaleuserserpheileaspalaservalsertrpasnaspvallys385390395400alavallyssertrpthrlysleuprovalleuvallysglyvalgln405410415thrvalgluaspvalileglualatyraspalaglycysglnglyval420425430valleuserasnhisglyglyargglnleuaspthralapropropro435440445ilegluleuleualagluthrvalprothrleulysargleuglylys450455460leuargproaspphegluileleuileaspglyglyvallysarggly465470475480thraspileleulysalavalalaileglyglyglnaspvalargval485490495servalglymetglyargpropheleutyralaasnsercystyrgly500505510glualaglyvalarglysleuileglnasnleulysaspgluleuglu515520525metaspmetargleuleuglyvalthrlysmetaspglnleuserser530535540lyshisvalaspthrlysargleuileglyargaspalaileasntyr545550555560leutyraspasnvaltyrserproilegluthrvallyspheasnasn565570575gluasp<210>115<211>1698<212>dna<213>东方伊萨酵母<220><221>misc_feature<222>(941)..(941)<223>n=a,c,t,或g<220><221>misc_feature<222>(943)..(943)<223>n=a,c,t,或g<400>115atgttaagatcccagttcaaaaacattttgaaaaatgttaacaagaaccattctctaagg60agaacttttacttccagcacctcaaaggctggaaaaaatgcttcatacaatgccaagatt120atatctgcaaccgtggcctcgattgttgcagcagctggctcttatatgttggtccagcct180tcactagctaatgatgaggcacagtctgctaatccaactaggaagatctctgttgacgaa240tttgttaaacacaaccatgccgatgattgttggatcactgttaacggtaacgtctatgac300ttgactgatttcatttcaatgcatccaggtggtactaccccattgattcaaaatgcaggt360cacgacgcaactgaaatttacaacaagattcatccaaagggtacaatcgagaacttctta420ccaaaggaaaagcaattgggtgttttggatggtgaagctcctaaaatcgaagttgtgctt480gacgaaaaggagaaacacagattggagttgttgaatcatctccctgctctttccagaatt540caaaacatttatgatttcgaacatattgcttctagagttttgagcgaccaagcatggaac600tactattcatgtggtgccgaagatgaaatcaccttgagggaaaatcattatgcttaccaa660agaatctactttaagccaaaatgttgtgtcaatgttgcagaagttgatacctctcatgaa720attttaggtacaaaagcttctgttcctttctacgtttccgcagccgcttctgcaaagttg780gggcacgaggatggtgaatgttccattgctagaggtgcaggtaaggaaggcgttattcaa840atgatttcttccttctcttccaactctttggaggaaattgcagaatccagaattcctggt900gcaacacaatggtttcaattatacgttaatgaagacaaggntnttgtgaagaagacttta960aaaagggccgaaaacttgggtatgaaggccatctttgtcactgtggacgctgctagtaga1020ggtaatagagaaaaagacattacaatgagaattaccgaagatacagatgagttaatagac1080gattcttctgttagagctggttctacctctggtgcattgccagctttcattgacaagagg1140ctgacttgggatgaagttaaggatatcatttcatggaccaagttaccagttttgctgaag1200ggtgttcaaagaactgatgatattgagaaggcaattgatattggttgtaagggtgttgtc1260ttgtccaatcatggtggtagacaattagatacttctcctcctccaatagaagttatggct1320gaatctgttccaatcctaaagcaaaagggtaaactggatccaaatttcagtattttcgtt1380gatggtggtgttagaagaggtacagatattttgaaagctttggctattggtggcagagac1440tgtaaagttgctgttggtctgggtagacctttcctttatgcaaatactggttatggtgaa1500aagggtgtcagaaaggccgtgcaaattctaagagaagaattaaaggctgacatgagaatg1560ttgggcgttacctctttgaacgagctagacgactcttacattgacaccagaagattacta1620ggtagagatgctgttaaccacatatacaacaacaactactacccaatgtctaagattcaa1680ttcaaaaacgaaaaataa1698<210>116<211>565<212>prt<213>东方伊萨酵母<220><221>misc_feature<222>(314)..(314)<223>xaa=asp,gly,ala,或val<220><221>misc_feature<222>(315)..(315)<223>xaa=ile,val,leu,或phe<400>116metleuargserglnphelysasnileleulysasnvalasnlysasn151015hisserleuargargthrphethrserserthrserlysalaglylys202530asnalasertyrasnalalysileileseralathrvalalaserile354045valalaalaalaglysertyrmetleuvalglnproserleualaasn505560aspglualaglnseralaasnprothrarglysileservalaspglu65707580phevallyshisasnhisalaaspaspcystrpilethrvalasngly859095asnvaltyraspleuthrasppheilesermethisproglyglythr100105110thrproleuileglnasnalaglyhisaspalathrgluiletyrasn115120125lysilehisprolysglythrilegluasnpheleuprolysglulys130135140glnleuglyvalleuaspglyglualaprolysilegluvalvalleu145150155160aspglulysglulyshisargleugluleuleuasnhisleuproala165170175leuserargileglnasniletyraspphegluhisilealaserarg180185190valleuseraspglnalatrpasntyrtyrsercysglyalagluasp195200205gluilethrleuarggluasnhistyralatyrglnargiletyrphe210215220lysprolyscyscysvalasnvalalagluvalaspthrserhisglu225230235240ileleuglythrlysalaservalprophetyrvalseralaalaala245250255seralalysleuglyhisgluaspglyglucysserilealaarggly260265270alaglylysgluglyvalileglnmetileserserpheserserasn275280285serleuglugluilealagluserargileproglyalathrglntrp290295300pheglnleutyrvalasngluasplysxaaxaavallyslysthrleu305310315320lysargalagluasnleuglymetlysalailephevalthrvalasp325330335alaalaserargglyasnargglulysaspilethrmetargilethr340345350gluaspthraspgluleuileaspaspserservalargalaglyser355360365thrserglyalaleuproalapheileasplysargleuthrtrpasp370375380gluvallysaspileilesertrpthrlysleuprovalleuleulys385390395400glyvalglnargthraspaspileglulysalaileaspileglycys405410415lysglyvalvalleuserasnhisglyglyargglnleuaspthrser420425430proproproilegluvalmetalagluservalproileleulysgln435440445lysglylysleuaspproasnpheserilephevalaspglyglyval450455460argargglythraspileleulysalaleualaileglyglyargasp465470475480cyslysvalalavalglyleuglyargpropheleutyralaasnthr485490495glytyrglyglulysglyvalarglysalavalglnileleuargglu500505510gluleulysalaaspmetargmetleuglyvalthrserleuasnglu515520525leuaspaspsertyrileaspthrargargleuleuglyargaspala530535540valasnhisiletyrasnasnasntyrtyrprometserlysilegln545550555560phelysasnglulys565<210>117<211>1167<212>dna<213>东方伊萨酵母<400>117atggtgtcccctgctgaaagattatctactattgcgtccacaatcaagccaaacagaaaa60gattctacatcattacaaccagaagactatccggaacatccgttcaaggtgacggttgtt120ggttccggtaactgggggtgtacaattgccaaggttatagcggaaaacaccgttgagaga180cctcgtcaatttcaaagagatgttaatatgtgggtctatgaagaattgattgaaggcgaa240aagttgactgaaatcataaataccaaacacgaaaacgtcaagtacttgccaggtatcaag300ttgccagttaacgttgttgcagttccagacattgttgaggcttgtgcaggctcagacttg360attgtctttaatattcctcaccaatttttaccaagaattttatcccaattaaagggtaag420gtgaatccaaaggctagagcaatttcttgtttgaaaggtttggatgtcaatcctaatgga480tgtaagttgctctccactgttattactgaagagttgggtatttattgtggtgccttatca540ggtgctaatttagctcctgaagttgcacaatgtaaatggtcggaaacaactgttgcatat600acaattccggacgatttcagaggtaaaggcaaggatattgaccatcaaattctaaagagt660ttgttccatagaccttatttccatgttcgtgttattagtgatgttgcaggtatttccatt720gccggtgcactcaagaatgtcgttgctatggctgctggatttgtcgaaggtttaggttgg780ggtgataatgcaaaggctgcagtcatgagaataggtttggtggaaaccattcaatttgcc840aagacttttttcgatggctgtcatgctgcaacctttactcatgaatctgcaggtgttgcc900gacctaatcactacctgtgccggcggccgtaacgttagagttggtagatatatggcacaa960cattctgtctctgcaacggaggctgaagaaaagttgttgaatggccaatcctgtcaaggt1020atccacacaactagggaagtttacgagttcctctccaacatgggcaggacagatgagttc1080ccactatttaccaccacctaccgtatcatctacgaaaacttcccaattgagaagctgcca1140gaatgccttgaacctgtggaagattaa1167<210>118<211>388<212>prt<213>东方伊萨酵母<400>118metvalserproalagluargleuserthrilealaserthrilelys151015proasnarglysaspserthrserleuglnprogluasptyrproglu202530hisprophelysvalthrvalvalglyserglyasntrpglycysthr354045ilealalysvalilealagluasnthrvalgluargproargglnphe505560glnargaspvalasnmettrpvaltyrglugluleuilegluglyglu65707580lysleuthrgluileileasnthrlyshisgluasnvallystyrleu859095proglyilelysleuprovalasnvalvalalavalproaspileval100105110glualacysalaglyseraspleuilevalpheasnileprohisgln115120125pheleuproargileleuserglnleulysglylysvalasnprolys130135140alaargalailesercysleulysglyleuaspvalasnproasngly145150155160cyslysleuleuserthrvalilethrglugluleuglyiletyrcys165170175glyalaleuserglyalaasnleualaprogluvalalaglncyslys180185190trpsergluthrthrvalalatyrthrileproaspaspphearggly195200205lysglylysaspileasphisglnileleulysserleuphehisarg210215220protyrphehisvalargvalileseraspvalalaglyileserile225230235240alaglyalaleulysasnvalvalalametalaalaglyphevalglu245250255glyleuglytrpglyaspasnalalysalaalavalmetargilegly260265270leuvalgluthrileglnphealalysthrphepheaspglycyshis275280285alaalathrphethrhisgluseralaglyvalalaaspleuilethr290295300thrcysalaglyglyargasnvalargvalglyargtyrmetalagln305310315320hisservalseralathrglualagluglulysleuleuasnglygln325330335sercysglnglyilehisthrthrarggluvaltyrglupheleuser340345350asnmetglyargthraspglupheproleuphethrthrthrtyrarg355360365ileiletyrgluasnpheproileglulysleuproglucysleuglu370375380provalgluasp385<210>119<211>1566<212>dna<213>东方伊萨酵母<400>119atgttgtccctctctaaacagtcaagaaactttttcaaattgaactatttttcagtcacc60caaatagcaaaaatgtctgcaacttccgtcactttcccaattatcaacgaaacttaccaa120cagccaaccgggcttttcatcaacaatgaatttgttagtgcaaagtcaggtaagactttt180gatgttaacaccccaattgatgagtctctcatttgtaaagtccaacaggccgatgctgaa240gatgttgaaattgccgttcaagcagcatctaaagcttacaagacttggagatttacaccg300ccaaatgaaagaggcagatacttgaacaaattggccgatttgatggacgaaaagagagac360ttacttgccaaaattgaatcccttgataatggtaaggccttacattgtgcaaaattcgat420gtcaatcttgtcattgaatatttcagatactgtgcaggttactgtgataaaatcgatggt480agaacaattacaaccgatgtagaacattttacctacactagaaaggaacctttaggtgtc540tgtggtgcaattacaccttggaacttcccattgctgatgtttgcttggaaaatcggcccg600gctttagcaaccggtaataccattatcttgaagcctgccagtgcaacacctctatcaaac660ctctttacttgtaccttgatcaaggaggcgggcattccagccggtgttgttaatgttgtt720ccaggttccggtagaggctgtggtaactccattttacaacatcctaaaattaagaaggtt780gcgtttaccggatctacagaagttggtaaaactgttatgaaggaatgtgctaattccatc840aaaaaggttactctcgaattgggtggtaagtctccaaacattgttttcaaagactgtaac900gttgaacaaaccattcaaaatttgattactggtattttcttcaatggtggtgaagtctgt960tgtgctggttctagaatttacattgaagcaaccgatgagaaatggtatactgaattcttg1020accaaattcaaggagactgttgaaaaattaaagattggtaacccatttgaagagggtgtt1080ttccaaggtgcacaaaccactccagatcaattccaaactgtcttggactacatcaccgct1140gctaacgaatccagcttgaaactattaactggtggtaaaagaattggcaataagggatac1200tttgttgagccaactatcttctacgatgttcctcaaaattccaagttaactcaagaagaa1260atctttggtccagttgctgttgttttacctttcaagtccactgaagaattgattgaaaag1320gcaaatgattccgattttggcttaggttccggtattcacactgaagatttcaacaaggca1380atttgggtttccgaaaggcttgaagcaggttctgtttggatcaacacttacaatgatttc1440cacccagctgctccattcggtggttacaaggaatccggtattggcagagaaatgggtatt1500gaagctttcgacaactatactcaaaccaagttagttagagctagagttaacaagccagct1560ttttag1566<210>120<211>521<212>prt<213>东方伊萨酵母<400>120metleuserleuserlysglnserargasnphephelysleuasntyr151015pheservalthrglnilealalysmetseralathrservalthrphe202530proileileasngluthrtyrglnglnprothrglyleupheileasn354045asngluphevalseralalysserglylysthrpheaspvalasnthr505560proileaspgluserleuilecyslysvalglnglnalaaspalaglu65707580aspvalgluilealavalglnalaalaserlysalatyrlysthrtrp859095argphethrproproasngluargglyargtyrleuasnlysleuala100105110aspleumetaspglulysargaspleuleualalysilegluserleu115120125aspasnglylysalaleuhiscysalalyspheaspvalasnleuval130135140ileglutyrpheargtyrcysalaglytyrcysasplysileaspgly145150155160argthrilethrthraspvalgluhisphethrtyrthrarglysglu165170175proleuglyvalcysglyalailethrprotrpasnpheproleuleu180185190metphealatrplysileglyproalaleualathrglyasnthrile195200205ileleulysproalaseralathrproleuserasnleuphethrcys210215220thrleuilelysglualaglyileproalaglyvalvalasnvalval225230235240proglyserglyargglycysglyasnserileleuglnhisprolys245250255ilelyslysvalalaphethrglyserthrgluvalglylysthrval260265270metlysglucysalaasnserilelyslysvalthrleugluleugly275280285glylysserproasnilevalphelysaspcysasnvalgluglnthr290295300ileglnasnleuilethrglyilephepheasnglyglygluvalcys305310315320cysalaglyserargiletyrileglualathraspglulystrptyr325330335thrglupheleuthrlysphelysgluthrvalglulysleulysile340345350glyasnpropheglugluglyvalpheglnglyalaglnthrthrpro355360365aspglnpheglnthrvalleuasptyrilethralaalaasngluser370375380serleulysleuleuthrglyglylysargileglyasnlysglytyr385390395400phevalgluprothrilephetyraspvalproglnasnserlysleu405410415thrglnglugluilepheglyprovalalavalvalleuprophelys420425430serthrglugluleuileglulysalaasnaspserasppheglyleu435440445glyserglyilehisthrgluasppheasnlysalailetrpvalser450455460gluargleuglualaglyservaltrpileasnthrtyrasnaspphe465470475480hisproalaalapropheglyglytyrlysgluserglyileglyarg485490495glumetglyileglualapheaspasntyrthrglnthrlysleuval500505510argalaargvalasnlysproalaphe515520<210>121<211>1506<212>dna<213>东方伊萨酵母<400>121atgtcagcactgttcagaaccattgagactccaaacggtaaaaccctggaacaaccactg60ggtctcttcatcgacaatgagtgggtgaaaacaaaccgtacttttgagaccattaatccg120tccacaggtgaggcgatctgtcatgtttaccgtgctggggtccaggaggtgaacgacgct180gtcgaagctgcaaatagagcatttagaaacgaatcttggtcaggtctaactggttctcaa240cgtggcgatttactgtatcgcatgtaccaagttatcaaaagagacgccgagagcattgca300tcgattgagtccatggataatggtaaaccgtatgctgcagaatgcctagatggagattta360ggtgaagctgctgacgttttcaaatattatgccggttgggccgacaagatcaccggtgaa420ctcattggctcgagtgtattgggtaagaataagatgtgttatgtcgagcctacaccactg480ggtgccgttggcggtatagtcccttggaatttcccgtttaccatgatggcatggaaaatt540gccccggcactggcgacgggttgtacagtggttatgaagtcaagtgaagtcacaccgttg600acggcattatggtatggcaagattgcacttgaagtgggtctacctaaaggtgtacttaac660atcctctccggttttggatcggatgttggatcggccatggcttcacatccaaagttggct720aagatagcgttcactggctcaactgcaactggtaaaaaaatcatggaagcagcaggtggt780tccaacttgaaaaaggttacactagagtgtggtggtaaatctccttacattgtttttgat840gatgctgacttagaattggcagtagaatgggcatattggggtatttggtataacaaaggt900gaggtttgtacttcaacttcgagatttttgattcaggaagacatttacgataagtttgtt960gagagttttgttgagttgaccaagacgagagcaatcactgctgatccgtttgatgataga1020tgcactatcgggcctttggtttctagctcacagtacgaaaaagtcaaaaagtacgttgaa1080ataggtaaaaatgaaggagcaaagctactaactggcaaattcatcgacgggccaggctat1140ttctgtgagccatttatcttcagtgaatgcactgacgatatgacaatcatgaaagaggaa1200atctttggccctgttgtggggattactaaattctcaacggttaaagaggcgatcgagaga1260gccaatgctacgacttacggtttaggagctgcgttgttttcctctaacataacaaaggca1320cattctgtggctgccaagttggaggctggaatggtgtggatcaattctaatggtgattct1380gatatccacattccatttggtggttccaaaatgagtggtataggtagggagttggggcca1440tacgcactagacttgtttactgagaaaaaggcagttcatgtcaacttatcgcttccggtc1500aagtga1506<210>122<211>501<212>prt<213>东方伊萨酵母<400>122metseralaleupheargthrilegluthrproasnglylysthrleu151015gluglnproleuglyleupheileaspasnglutrpvallysthrasn202530argthrphegluthrileasnproserthrglyglualailecyshis354045valtyrargalaglyvalglngluvalasnaspalavalglualaala505560asnargalapheargasnglusertrpserglyleuthrglysergln65707580argglyaspleuleutyrargmettyrglnvalilelysargaspala859095gluserilealaserileglusermetaspasnglylysprotyrala100105110alaglucysleuaspglyaspleuglyglualaalaaspvalphelys115120125tyrtyralaglytrpalaasplysilethrglygluleuileglyser130135140servalleuglylysasnlysmetcystyrvalgluprothrproleu145150155160glyalavalglyglyilevalprotrpasnpheprophethrmetmet165170175alatrplysilealaproalaleualathrglycysthrvalvalmet180185190lyssersergluvalthrproleuthralaleutrptyrglylysile195200205alaleugluvalglyleuprolysglyvalleuasnileleusergly210215220pheglyseraspvalglyseralametalaserhisprolysleuala225230235240lysilealaphethrglyserthralathrglylyslysilemetglu245250255alaalaglyglyserasnleulyslysvalthrleuglucysglygly260265270lysserprotyrilevalpheaspaspalaaspleugluleualaval275280285glutrpalatyrtrpglyiletrptyrasnlysglygluvalcysthr290295300serthrserargpheleuileglngluaspiletyrasplyspheval305310315320gluserphevalgluleuthrlysthrargalailethralaasppro325330335pheaspaspargcysthrileglyproleuvalserserserglntyr340345350glulysvallyslystyrvalgluileglylysasngluglyalalys355360365leuleuthrglylyspheileaspglyproglytyrphecysglupro370375380pheilepheserglucysthraspaspmetthrilemetlysgluglu385390395400ilepheglyprovalvalglyilethrlyspheserthrvallysglu405410415alailegluargalaasnalathrthrtyrglyleuglyalaalaleu420425430pheserserasnilethrlysalahisservalalaalalysleuglu435440445alaglymetvaltrpileasnserasnglyaspseraspilehisile450455460propheglyglyserlysmetserglyileglyarggluleuglypro465470475480tyralaleuaspleuphethrglulyslysalavalhisvalasnleu485490495serleuprovallys500<210>123<211>1506<212>dna<213>东方伊萨酵母<400>123atggctcttccacttgcaactacaatctccttatcaagcggcaaaacattagaacagcca60attggtttatttattgataatgaatttgtcaatccaatttctgtttctaatgcaagaaca120ctaacaaccttcaacccaagcacaggtgagccaataaccgatgttcattgtgcctcagct180gcagatgttgatgttgcggtaaatgctgcaaacaaggcaatggaaacatggaaagacatt240gatcctactgttcgtgtcgaacttttactaaaattggccagcttagttgacgagcattcc300caagcaattgctgaaattgaagcactagactcgggtaaaccattgtactcgaatgcactg360gcggatgttcaatcggttgctgagtacttaaggtactgtgccggttgggcggataaatta420cacggtacgcaaattccaataaactctaaggtaatggctattacaaaacgtgtaccctta480gttgtcggctgcatcattccatggaactacccaatttcaatggcctcctggaagttctgt540ccagcattggctgccggatgtactattgtaatgaagtcaagtgagataaccccgttatcg600ttactttattttgcgaatttggtcaaattagcaggtttccctaagggtgtttttaatgtc660gtctctggatttggtgatgatgttggctcagcgctttcaaatcacccaaagttgggtaag720attgcatttacaggctcgaccttgaccgggcaaaaggtgatggcggatgctgccagatca780aatttgaaaagcgtatctttggaatgtggtggtaaatctccacttattgtcttcgaagat840gcagaattggatgaatgcgttaaatgggcaagttttggtgtcatgtataacaccggacaa900aattgtactgccaattctcgtattattgtgcatgataaggtttatgatcaatttatcgaa960aagttcctgtctcaactcaaggaagattggaaaatgggagatgtcatgaatgaaaagact1020acattgggaccacttgtcagccaacaacaatatgagcgtgttcagtcgtatattgatata1080ggtgtcaaagaaggggctacactgattcaaccgcttaaggagagcactccatcaaatgga1140ttctacatctctcctactgtttttactaacgttaaggaagatatgagaattgttaaggag1200gaaatatttggtcctgtcgtaactatctccaaattctcaactgaggaagaggcaatttca1260aaggcgaatgatacaatttatggcttagctgcaatgttatttactactaattttgaacgt1320gccaacagagttgctgataagctggaagctggcagtgtgtacattaatagctctaacaac1380gagagtaccaaagttccatttggaggaatgaagatgagtggtattggaagagagttgggg1440caagaagcatttaatttgtacactgttacaaagagtatttattatagttatggtgctaag1500ctttaa1506<210>124<211>501<212>prt<213>东方伊萨酵母<400>124metalaleuproleualathrthrileserleuserserglylysthr151015leugluglnproileglyleupheileaspasngluphevalasnpro202530ileservalserasnalaargthrleuthrthrpheasnproserthr354045glygluproilethraspvalhiscysalaseralaalaaspvalasp505560valalavalasnalaalaasnlysalametgluthrtrplysaspile65707580aspprothrvalargvalgluleuleuleulysleualaserleuval859095aspgluhisserglnalailealagluileglualaleuaspsergly100105110lysproleutyrserasnalaleualaaspvalglnservalalaglu115120125tyrleuargtyrcysalaglytrpalaasplysleuhisglythrgln130135140ileproileasnserlysvalmetalailethrlysargvalproleu145150155160valvalglycysileileprotrpasntyrproilesermetalaser165170175trplysphecysproalaleualaalaglycysthrilevalmetlys180185190sersergluilethrproleuserleuleutyrphealaasnleuval195200205lysleualaglypheprolysglyvalpheasnvalvalserglyphe210215220glyaspaspvalglyseralaleuserasnhisprolysleuglylys225230235240ilealaphethrglyserthrleuthrglyglnlysvalmetalaasp245250255alaalaargserasnleulysservalserleuglucysglyglylys260265270serproleuilevalphegluaspalagluleuaspglucysvallys275280285trpalaserpheglyvalmettyrasnthrglyglnasncysthrala290295300asnserargileilevalhisasplysvaltyraspglnpheileglu305310315320lyspheleuserglnleulysgluasptrplysmetglyaspvalmet325330335asnglulysthrthrleuglyproleuvalserglnglnglntyrglu340345350argvalglnsertyrileaspileglyvallysgluglyalathrleu355360365ileglnproleulysgluserthrproserasnglyphetyrileser370375380prothrvalphethrasnvallysgluaspmetargilevallysglu385390395400gluilepheglyprovalvalthrileserlyspheserthrgluglu405410415glualaileserlysalaasnaspthriletyrglyleualaalamet420425430leuphethrthrasnphegluargalaasnargvalalaasplysleu435440445glualaglyservaltyrileasnserserasnasngluserthrlys450455460valpropheglyglymetlysmetserglyileglyarggluleugly465470475480glnglualapheasnleutyrthrvalthrlysseriletyrtyrser485490495tyrglyalalysleu500<210>125<211>1544<212>dna<213>东方伊萨酵母<220><221>misc_feature<222>(1319)..(1319)<223>n=a,c,g,或t<400>125atgatgggcgcaactacagcaaaaattagtattccaaatggtaacaaatacgagcaacct60acaggtttgttcatcaatggtgagtttgttgcttcaagtgatggtaaaactgcagaagtt120gagaatccaggcaatggaaacattgtatgttctgtccacttagcttctattgaggatatt180aataccgccgtagaagctgctgaagatgcatttttcaaaaggtgggccaccatcagtggt240aaagccaagggagaatacttgagtaagattgccgatctaatcgttaaatattctgatcaa300ttggcagatctagaggctattgaatcaggtaagccaaaggacaccaatgcaatctttgat360gttttacattcggctgatgttttcagatactatgctggcaaggctgtcactgcacaaagc420ggcaagactatcgagtccgaactctccaaatttacatacacagtttacgagccctatggt480gtttgtgccgctatcatcgcatggaacttcccaatgagcacatttgcgtggaaagttgcg540gcatgtttagctgctggtaatacaatggttgtcaaaacttccgagctgactccgttatct600gcattgttcatgtgtaagattttccaagaagcagatctacctgctggagttataaacgtc660acatgtggtttaggttctgttgcaggtgttcgattgagtgaacatgaaaaggttcagaaa720atttcgtttactggctccactggcgttggtaagttgatccaagaatccgcagcaaagtct780aacttaaagtattgtacgcttgaatgtggtggtaagtctccgttagtgatttacgaggat840gcagatcttgagcaagcagtgaagtgggctgcctttggtatttttttcaacaaaggtgaa900atttgcacagcctcttccagaatatatgttcaagaatcagtctatgacaaatttttgact960atgtacaaggatcatgtggaagaagcctatgttcaaggagaacagtttgccactggtgtt1020aacgttgggcctactgtctgcaaagcccaacaagagaaaatactggcctacattgaaagt1080gccaagcaagaaggtggtagaattatcactggtggtaaaataccatcttacacgaacaaa1140aatggttactatctcgaaccaacaattattgcagattgtaaccaggatatgaaggtagtc1200agggaagagattttcggaccagtcgttactgtatccaaattcactagtgatgaagaagcc1260atcaaattaagcaatgattccgaatatggcttggcagcatatttattcactystsrasna1320ssrgttyrgytaaaatyrththraaggacctcgttagatctcagaattatatcagaaaag1380tgcaaagcggacaggtctttgtcaacttcacctttgcggctgatttcaggttgccatttg1440gcggatataagatgagtggtaacggaagagagcttggtgatgaaggactgagtgctttcc1500agcaagtcaaagcagtacacattaatctcactgggaagttgtaa1544<210>126<211>503<212>prt<213>东方伊萨酵母<400>126metmetglyalathrthralalysileserileproasnglyasnlys151015tyrgluglnprothrglyleupheileasnglygluphevalalaser202530seraspglylysthralagluvalgluasnproglyasnglyasnile354045valcysservalhisleualaserilegluaspileasnthralaval505560glualaalagluaspalaphephelysargtrpalathrilesergly65707580lysalalysglyglutyrleuserlysilealaaspleuilevallys859095tyrseraspglnleualaaspleuglualailegluserglylyspro100105110lysaspthrasnalailepheaspvalleuhisseralaaspvalphe115120125argtyrtyralaglylysalavalthralaglnserglylysthrile130135140glusergluleuserlysphethrtyrthrvaltyrgluprotyrgly145150155160valcysalaalaileilealatrpasnpheprometserthrpheala165170175trplysvalalaalacysleualaalaglyasnthrmetvalvallys180185190thrsergluleuthrproleuseralaleuphemetcyslysilephe195200205glnglualaaspleuproalaglyvalileasnvalthrcysglyleu210215220glyservalalaglyvalargleusergluhisglulysvalglnlys225230235240ileserphethrglyserthrglyvalglylysleuileglngluser245250255alaalalysserasnleulystyrcysthrleuglucysglyglylys260265270serproleuvaliletyrgluaspalaaspleugluglnalavallys275280285trpalaalapheglyilephepheasnlysglygluilecysthrala290295300serserargiletyrvalglngluservaltyrasplyspheleuthr305310315320mettyrlysasphisvalgluglualatyrvalglnglygluglnphe325330335alathrglyvalasnvalglyprothrvalcyslysalaglnglnglu340345350lysileleualatyrilegluseralalysglngluglyglyargile355360365ilethrglyglylysileprosertyrthrasnlysasnglytyrtyr370375380leugluprothrileilealaaspcysasnglnaspmetlysvalval385390395400argglugluilepheglyprovalvalthrvalserlysphethrser405410415aspgluglualailelysleuserasnaspserglutyrglyleuala420425430alatyrleuphethrlysaspleuvalargserglnasntyrilearg435440445lysvalglnserglyglnvalphevalasnphethrphealaalaasp450455460pheargleupropheglyglytyrlysmetserglyasnglyargglu465470475480leuglyaspgluglyleuseralapheglnglnvallysalavalhis485490495ileasnleuthrglylysleu500<210>127<211>1716<212>dna<213>东方伊萨酵母<220><221>misc_feature<222>(655)..(655)<223>nisa,c,g,或t<400>127atggctccaactgctgttgatatccataacgagtacaaacagaatgtttccaacgaacag60gaaattcctttcaacaaaactgaaagaaagtcatcgattgcatctaaattaggactgaat120ccagacgctaagattcactacaattctgctgttcctatattatacgaagatggtttaaag180gaaaaaggtacaaccatttcctcttctggtgcattgattgcattctctggttccaaaaca240ggtagatctccaaaggacaaaagaattgtcgatgaagagacttcaacagacaacatctgg300tggggtccagtcaataagaaggttgatgaaaacacttggaatatctcgaaatctagagcg360attgattatttgagaacaagagagaaggtttacattatcgatgcttttgctggttgggat420ccaagatacagaattaaggttagaattgtctgtgctagagcttaccatgctttgttcatg480aagaatatgttaattagaccaacaacggaagaattaaagaactttggtgagcctgatttc540accatttggaatgcaggtcaattccctgctaatgtttacactaagggtatgacttcttca600acttctgttgaaataaatttcaagtctthrysgymtthrsrsrthrsrvagtasnhyssr660atggaaatggttatcctaggtactgaatacgcaggtgaaatgaagaaaggtatctttacc720gttatgttctacttgatgccaatcagacacaaggttttaactttacactcttctgcaaat780caaggtaaaaaggatggtgatgtcacattattctttggtttatctggtacaggtaaaaca840accttgtctgcagatcctcatagagaattgattggtgatgatgaacattgctggtctgat900catggtgttttcaacattgaaggtggatgttatgctaagtgtttggacttatctgctgaa960agagaacctgagattttcaatgcaattaggtttggatctgtcttggagaatgttgtctat1020gatccagttgatagaactgttgactattccgctgctaatgtcactgaaaatactagatgt1080gcttatcctatcgactttattccttctgctaagatcccatgtctggcagattctcatcca1140aagaatattgttcttttaacttgtgatgcaagaggtgttttgccacctgtctccaagcta1200actaatgcacaagtcatgtatcactttatctctggttacacctccaagatggcaggtacc1260gaagttggtgtcactgaaccagaagcaaccttctctgcatgttttggtcaacctttctta1320gttttacatccaatgaaatacgcacaacaactctctgataaaatggctgaacattcttcc1380accgcttggttattgaataccggttggactggtcaatcttatgttaaaggtggtaagaga1440tgtccattgaagtatactagagcaattttagatgctattcactctggtgagcttgcaaaa1500caggaattcgaaacataccctactttcggtttacaagttccaaaaacttgtccaggtgtc1560ccagaaagtgttctgaacccatctaaacactgggctactggtgaagctgatttcaaggct1620gaagtcactaacttggctaaattatttgctgagaactttgaaaagtattctgcagaatgt1680actgcagaagttgttgctgctggtcctgctttataa1716<210>128<211>560<212>prt<213>东方伊萨酵母<400>128metalaprothralavalaspilehisasnglutyrlysglnasnval151015serasngluglngluilepropheasnlysthrgluarglysserser202530ilealaserlysleuglyleuasnproaspalalysilehistyrasn354045seralavalproileleutyrgluaspglyleulysglulysglythr505560thrileserserserglyalaleuilealapheserglyserlysthr65707580glyargserprolysasplysargilevalaspglugluthrserthr859095aspasniletrptrpglyprovalasnlyslysvalaspgluasnthr100105110trpasnileserlysserargalaileasptyrleuargthrargglu115120125lysvaltyrileileaspalaphealaglytrpaspproargtyrarg130135140ilelysvalargilevalcysalaargalatyrhisalaleuphemet145150155160lysasnmetleuileargprothrthrglugluleulysasnphegly165170175gluproaspphethriletrpasnalaglyglnpheproalaasnval180185190tyrthrlysglymetthrserserthrservalgluileasnphelys195200205sermetglumetvalileleuglythrglutyralaglyglumetlys210215220lysglyilephethrvalmetphetyrleumetproilearghislys225230235240valleuthrleuhisserseralaasnglnglylyslysaspglyasp245250255valthrleuphepheglyleuserglythrglylysthrthrleuser260265270alaaspprohisarggluleuileglyaspaspgluhiscystrpser275280285asphisglyvalpheasnilegluglyglycystyralalyscysleu290295300aspleuseralagluarggluprogluilepheasnalaileargphe305310315320glyservalleugluasnvalvaltyraspprovalaspargthrval325330335asptyrseralaalaasnvalthrgluasnthrargcysalatyrpro340345350ileasppheileproseralalysileprocysleualaaspserhis355360365prolysasnilevalleuleuthrcysaspalaargglyvalleupro370375380provalserlysleuthrasnalaglnvalmettyrhispheileser385390395400glytyrthrserlysmetalaglythrgluvalglyvalthrglupro405410415glualathrpheseralacyspheglyglnpropheleuvalleuhis420425430prometlystyralaglnglnleuserasplysmetalagluhisser435440445serthralatrpleuleuasnthrglytrpthrglyglnsertyrval450455460lysglyglylysargcysproleulystyrthrargalaileleuasp465470475480alailehisserglygluleualalysglngluphegluthrtyrpro485490495thrpheglyleuglnvalprolysthrcysproglyvalprogluser500505510valleuasnproserlyshistrpalathrglyglualaaspphelys515520525alagluvalthrasnleualalysleuphealagluasnpheglulys530535540tyrseralaglucysthralagluvalvalalaalaglyproalaleu545550555560<210>129<211>267<212>prt<213>酿酒酵母<400>129metserglnglyarglysalaalagluargleualalyslysthrval151015leuilethrglyalaseralaglyileglylysalathralaleuglu202530tyrleuglualaserasnglyaspmetlysleuileleualaalaarg354045argleuglulysleuglugluleulyslysthrileaspglngluphe505560proasnalalysvalhisvalalaglnleuaspilethrglnalaglu65707580lysilelyspropheilegluasnleuproglngluphelysaspile859095aspileleuvalasnasnalaglylysalaleuglyseraspargval100105110glyglnilealathrgluaspileglnaspvalpheaspthrasnval115120125thralaleuileasnilethrglnalavalleuproilepheglnala130135140lysasnserglyaspilevalasnleuglyserilealaglyargasp145150155160alatyrprothrglyseriletyrcysalaserlysphealavalgly165170175alaphethraspserleuarglysgluleuileasnthrlysilearg180185190valileleuilealaproglyleuvalgluthrglupheserleuval195200205argtyrargglyasnglugluglnalalysasnvaltyrlysaspthr210215220thrproleumetalaaspaspvalalaaspleuilevaltyralathr225230235240serarglysglnasnthrvalilealaaspthrleuilepheprothr245250255asnglnalaserprohishisilephearggly260265<210>130<211>420<212>dna<213>阿维链霉菌<400>130atgttaagaaccatgttcaaatctaagattcacagagcaactgttactcaagcagatctc60cattatgttggttccgttactattgatgcagacttgttagacgcagcagacttgttgcca120ggtgaattggttcacatcgttgacattacgaacggtgctagattggaaacttacgtcatt180gaaggtgaacgtggttccggtgttgttggtatcaatggtgctgccgctcatttagttcat240cctggtgatcttgttatcattatctcctatgcacaagtttcagatgcagaagcacgtgca300ttgcgtccaagagttgttcacgttgacagagacaatagagttgttgcgcttggtgcggat360ccagccgaaccagtcccaggttccgaccaagctagatccccacaagctgttactgcataa420<210>131<211>384<212>dna<213>丙酮丁醇梭菌<400>131atgcacttgaacatgttgaagtccaagatccacagagctaccgtcgttcaagcagacttg60aactacgtcggttccatcaccatcgacagaaacttgatggacaaggcaaacatcttggaa120tacgaaaaggtcgagatcgcaaacatcaacaacggtgcaagattcgaaacctacgtcatc180gctggtgaggctggttccggtatcatctgtttgaacggtgctgctgcaagatgtgcacaa240gcgggtgacaaggttatcatcatgtgttactgttccttgaccccagaagaagcttccgag300cacagaccaaaggtcgttttcgtcaacgacgacaactccatctccaacgtcaccgaatac360gagaagcacggcaccatcggttaa384<210>132<211>354<212>dna<213>helicobacterpylori<400>132atgaccttcgagatgttgtactccaagatccacagagcaaccatcaccgacgcaaacttg60aactacatcggctccatcaccatcgacgaggacttggctaagttggctaagttgagagag120ggtatgaaggtcgaaatcgtcgacgtcaacaacggcgagagattctccacctacgtcatc180ttgggtaagaagagaggtgaaatctgcgtcaacggtgcagcagccagaaaggtcgctatc240ggtgacgtcgtcatcatcttggcttacgcatccatgaacgaggacgagatcaacgctcac300aagccatccatcgtcttggtcgacgaaaagaacgaaatcttggaaaagggttaa354<210>133<211>117<212>prt<213>helicobacterpylori<400>133metthrpheglumetleutyrserlysilehisargalathrilethr151015aspalaasnleuasntyrileglyserilethrileaspgluaspleu202530alalysleualalysleuarggluglymetlysvalgluilevalasp354045valasnasnglygluargpheserthrtyrvalileleuglylyslys505560argglygluilecysvalasnglyalaalaalaarglysvalalaile65707580glyaspvalvalileileleualatyralasermetasngluaspglu859095ileasnalahislysproserilevalleuvalaspglulysasnglu100105110ileleuglulysgly115<210>134<211>387<212>dna<213>bacillussp.ts25<400>134atgtacagaaccatgatgaagtccaagttgcacagagcgaccgtcaccgaagcaaacttg60aactacgtcggttccatcaccatcgaccaagacttgatggaagctgcagacatcttggaa120aacgagaaggtccaaatcgtcaacaacaacaacggtgctagattcgaaacctacgtcatc180gctggtccaagaggctccggcaccatctgtttgaacggtgcagcagcaagattggtccaa240ccaggtgacaccgttatcatcatctcctacgcaatgttggaagaagccgaggctagaaag300caccaacctgtcgtcgttttgttgaacccagacaacaccatccaagaattgatcagagaa360acccacggtgctaccgctaccgtctaa387<210>135<211>128<212>prt<213>bacillussp.ts25<400>135mettyrargthrmetmetlysserlysleuhisargalathrvalthr151015glualaasnleuasntyrvalglyserilethrileaspglnaspleu202530metglualaalaaspileleugluasnglulysvalglnilevalasn354045asnasnasnglyalaargphegluthrtyrvalilealaglyproarg505560glyserglythrilecysleuasnglyalaalaalaargleuvalgln65707580proglyaspthrvalileileilesertyralametleuglugluala859095glualaarglyshisglnprovalvalvalleuleuasnproaspasn100105110thrileglngluleuilearggluthrhisglyalathralathrval115120125<210>136<211>411<212>dna<213>谷氨酸棒杆菌<400>136atgttgagaaccatcttgggttccaaaatccacagagctaccgttacccaagcagacttg60gactacgttggttcagtcaccatcgatgcagacttggttcatgccgcaggtttgatcgaa120ggtgaaaaggttgccatcgtcgacatcaccaatggcgctagattggaaacctacgttatc180gttggtgatgctggcaccggtaacatctgtatcaacggtgcagcagcacacttgatcaac240ccaggtgatttggttatcatcatgtcctacttgcaagctaccgatgcagaggccaaggct300tacgaaccaaagatcgttcacgtcgacgcagacaacagaatcgttgctttgggtaacgac360ttggcagaagctttgcctggttccggtttgttgacctcaagatctatctaa411<210>137<211>136<212>prt<213>谷氨酸棒杆菌<400>137metleuargthrileleuglyserlysilehisargalathrvalthr151015glnalaaspleuasptyrvalglyservalthrileaspalaaspleu202530valhisalaalaglyleuilegluglyglulysvalalailevalasp354045ilethrasnglyalaargleugluthrtyrvalilevalglyaspala505560glythrglyasnilecysileasnglyalaalaalahisleuileasn65707580proglyaspleuvalileilemetsertyrleuglnalathraspala859095glualalysalatyrgluprolysilevalhisvalaspalaaspasn100105110argilevalalaleuglyasnaspleualaglualaleuproglyser115120125glyleuleuthrserargserile130135<210>138<211>384<212>dna<213>地衣芽孢杆菌<400>138atgtacagaaccttgatgtccgctaagttgcacagagctagagtcaccgaagcaaacttg60aactacgttggttccgtcaccatcgacgaggacttgttggacgcagtcggtatgatggca120aacgaaaaggtccaaatcgtcaacaacaacaacggtgctagattggaaacctacatcatc180cctggtgagagaggttccggtgtcgtctgtttgaacggtgctgcagctagattggtccaa240gttggtgacgtcgttatcatcgtttcctacgccatgatgtccgaagaagaagcaaagacc300cacaagccaaaggtcgctgttttgaacgaaagaaacgaaatcgaagagatgttgggtcaa360gaaccagctagaaccatcttgtaa384<210>139<211>127<212>prt<213>地衣芽孢杆菌<400>139mettyrargthrleumetseralalysleuhisargalaargvalthr151015glualaasnleuasntyrvalglyservalthrileaspgluaspleu202530leuaspalavalglymetmetalaasnglulysvalglnilevalasn354045asnasnasnglyalaargleugluthrtyrileileproglygluarg505560glyserglyvalvalcysleuasnglyalaalaalaargleuvalgln65707580valglyaspvalvalileilevalsertyralametmetsergluglu859095glualalysthrhislysprolysvalalavalleuasngluargasn100105110gluilegluglumetleuglyglngluproalaargthrileleu115120125<210>140<211>1356<212>dna<213>阿维链霉菌<400>140atgacaccacagccaaacccacaagtcggtgcagcagtcaaagctgcagatagagcacac60gtcttccactcttggtctgcacaagaattgatcgatccattggctgttgctggtgcagag120ggttcctacttctgggattacgatggtagacgttaccttgacttcacctccggcttagtc180ttcaccaacatcggttaccaacacccaaaggttgtcgcagctatccaagaacaagccgca240tctttgactacatttgccccagctttcgcagttgaagcaagatccgaagctgcaagattg300atcgctgagcgtactccaggtgatttagacaaaatcttcttcaccaacggtggcgcagat360gctatcgagcacgctgttcgtatggcaagaatccacgctggtagaccaaaggtcttatcc420gcatacagatcataccacggtggtacacaacaggcagtcaacatcactggtgatccaagg480agatgggcatccgattccgcttctgctggcgttgtccacttctgggctccatacttatac540agatccagattctacgccgaaactgagcaacaagaatgtgagcgtgctcttgagcacttg600gaaactactatcgccttcgaaggtccaggtactattgccgctatcgttttggaaactgtc660ccaggtactgctggtatcatggttcctccaccaggttacttagcaggtgttagagaattg720tgtgacaaatacggtatcgtcttcgtcttggatgaagtcatggctggtttcggcagaact780ggcgaatggttcgctgcagacttattcgatgttaccccagacttgatgaccttcgctaag840ggtgtcaactcaggttacgttccattgggtggtgttgctatctccggcaaaatcgcagag900actttcggtaagagagcttacccaggtggtttgacgtactccggtcaccctcttgcttgc960gcagccgctgttgctactatcaacgttatggcagaagaaggtgtcgttgaaaacgctgca1020aacttgggtgctagagttatcgaaccaggtttgagagaacttgcagagagacacccatca1080gttggtgaagttagaggtgttggtatgttctgggctttggaattggttaaggatagagaa1140accagagaacctttggtcccatacaatgccgctggtgaagcgaacgcaccaatggctgct1200ttcggtgcagctgcaaaggcaaacggtttgtggccattcatcaacatgaacagaacccac1260gttgttcctccttgtaacgttaccgaagccgaagctaaagaaggcttggcagcattggat1320gcagctttatctgttgcagatgagtacactgtctaa1356<210>141<211>1416<212>dna<213>酿酒酵母<400>141atgtccatctgtgagcaatactacccagaagaaccaaccaagccaaccgtcaagaccgaa60tccatccctggtccagaatcccaaaagcaattgaaggaattgggtgaagttttcgacacc120agaccagcttacttcttggcagactacgaaaagtccttgggtaactacatcaccgacgtc180gacggtaacacctacttggacttgtacgctcaaatctcctccatcgccttgggttacaac240aacccagcattgatcaaagccgctcaatccccagaaatgatcagagcattggttgacaga300ccagccttgggtaacttcccttccaaggacttggacaagatcttgaagcaaatcttgaag360tccgctccaaagggtcaggaccacgtctggtccggtttgtccggtgcagacgcaaacgaa420ttggctttcaaggctgcattcatctactacagagctaagcaaagaggttacgacgcagac480ttctccgaaaaggaaaacttgtccgttatggacaacgacgcaccaggtgctccacacttg540gcagttttgtccttcaagagagctttccacggtagattgttcgcatccggttccactacc600tgttccaagccaatccacaagttggacttccctgctttccactggccacacgccgaatac660ccatcctaccagtaccctttggacgaaaactccgacgcaaacagaaaggaggacgaccac720tgcttggcaatcgttgaagaattgatcaagacctggtccatccctgttgctgcgttgatc780atcgaaccaatccaatccgagggtggtgacaaccacgcctccaagtacttcttgcaaaag840ttgagagacatcaccttgaagtacaacgtcgtctacatcatcgacgaagtccaaactggc900gttggcgcaaccggtaagttgtggtgtcacgaatacgcagacatccagcctccagtcgac960ttggtcaccttctccaagaagttccaatccgctggctacttcttccacgaccctaagttc1020atcccaaacaagccatacagacaattcaacacctggtgtggtgaaccagcaagaatgatc1080atcgcaggtgcaatcggtcaagagatctccgacaagaagttgaccgaacaatgctccaga1140gtcggtgactacttgttcaagaagttggaaggtttgcaaaagaagtacccagagaacttc1200caaaacttgagaggtaagggtagaggcaccttcatcgcttgggacttgccaactggcgag1260aagagagacttgttgttgaagaagttgaagttgaacggctgtaacgttggtggctgtgct1320gttcacgcagtcagattgagaccttccttgaccttcgaagagaagcacgcagacatcttc1380atcgaagctttggctaagtccgtcaacgaattgtaa1416<210>142<211>1428<212>dna<213>克鲁维酵母<400>142atgccatcctactccgttgcagaattgtactacccagacgaacctaccgaacctaagatc60tccacctcctcctacccaggtccaaaggcaaagcaagaattggaaaagttgtccaacgtc120ttcgacaccagagcagcttacttgttggcagactactacaagtcccgtggtaactacatc180gttgaccaggacggtaacgtcttgttggacgtttacgctcaaatctcctccatcgccttg240ggttacaacaacccagaaatcttgaaggttgcaaagtccgacgcaatgtccgttgcattg300gccaaccgtccagcattggcttgtttcccatccaacgactacggtcaattgttggaagac360ggtttgttgaaggcagcaccacaaggtcaagacaagatctggaccgctttgtccggttcc420gacgcaaacgaaaccgccttcaaagcctgcttcatgtaccaagctgcgaagaagagaaac480ggtagatccttctccaccgaagaattggaatccgttatggacaaccaattgccaggcacc540tccgaaatggttatctgttccttcgaaaagggtttccacggtcgtttgttcggttccttg600tccactaccagatccaagcctatccacaagttggacatcccagctttcaactggcctaaa660gccccattcccagacttgaagtacccattggaagaaaacaaggaagccaacaaggctgaa720gaatcctcctgtatcgaaaagttctcccaaatcgttcaagagtggcaaggtaagatcgct780gcagttatcatcgaaccaatccagtccgagggtggcgacaaccacgcttcctccgacttc840ttccaaaagttgagagaaatcaccatcgaaaacggtatcttgatgatcgtcgacgaagtt900caaaccggtgtcggtgctaccggcaagatgtgggcacacgaacactggaacttgtccaac960cctccagacttggttaccttctccaagaagttccaagcagcaggtttctactaccacgac1020ccaaagttgcaaccagaccagccattcagacagttcaacacctggtgtggtgacccatcc1080aaggctttgatcgccaaggttatctacgaagaaatcgttaagcacgacttggtcaccaga1140actgccgaagtcggtaactacttgttcaacagattggaaaagttgttcgaaggtaagaac1200tacatccagaacttgagaggtaagggtcaaggcacctacatcgctttcgacttcggcacc1260tcctccgagagagactccttcttgtccagattgagatgtaacggtgcaaacgtcgctggt1320tgcggtgactccgctgtcagattgagaccatccttgaccttcgaagagaagcacgcagac1380gtcttggtttccatcttcgacaagaccttgagacaattgtacggctaa1428<210>143<211>747<212>dna<213>大肠杆菌<400>143atgatcgttttggtcaccggtgcaaccgcaggtttcggcgaatgtatcaccagaagattc60atccagcagggtcacaaggttatcgctaccggtagaagacaagagagattgcaagaattg120aaggacgagttgggtgacaacttgtacatcgctcaattggacgttagaaacagagcagct180atcgaagaaatgttggcatccttgccagctgaatggtgcaacatcgacatcttggtcaac240aacgctggtttggcattgggtatggaaccagctcacaaggctagtgttgaggactgggag300accatgatcgacaccaacaacaagggtttggtctacatgaccagagcagttttgcctggt360atggttgaaagaaaccacggtcacatcatcaacatcggttccaccgctggttcctggcca420tacgctggcggtaacgtctacggtgctaccaaggctttcgttagacagttctccttgaac480ttgagaaccgacttgcacggcaccgctgttagagttaccgacatcgaaccaggtttggtt540ggtggcaccgaattctccaacgtcagattcaagggcgacgacggtaaggctgaaaagacc600taccaaaacaccgtcgctttgaccccagaagacgtttcagaggctgtttggtgggtcagt660accttgccagcacacgtcaacatcaacaccttggaaatgatgccagtcacccaatcctac720gcaggtttgaacgttcacagacaataa747<210>144<211>804<212>dna<213>酿酒酵母<400>144atgtcccaaggtagaaaggcagcagaaagattggcaaagaagaccgtcttgatcaccggt60gcgtccgctggtatcggtaaggctaccgcgttggagtacttggaagcatccaacggtgac120atgaagttgatcttggcagcaagaagattggagaagttggaagaattgaagaagaccatc180gaccaagaattcccaaacgctaaggtccacgttgcacaattggacatcacccaagcagag240aagatcaagccattcatcgaaaacttgccacaagaattcaaggacatcgacatcttggtc300aacaacgctggtaaggcgttgggttccgacagagttggtcaaatcgcaaccgaagacatc360caagacgtcttcgacaccaacgtcaccgctttgatcaacatcacccaagctgttttgcca420atcttccaagcgaagaactccggtgacatcgtcaacttgggttccatcgctggtagagac480gcatacccaaccggctccatctactgcgcctccaagttcgctgtcggtgctttcaccgac540tccttgagaaaggaattgatcaacaccaagatcagagtcatcttgattgcccctggtttg600gtcgaaaccgaattctccttggttagatacagaggtaacgaagaacaagcaaagaacgtt660tacaaggacactaccccattgatggccgacgacgttgcagacttgatcgtttacgctacc720tccagaaagcaaaacaccgttatcgcagacaccttgatcttcccaaccaaccaagcatcc780ccacaccacatcttcagaggttaa804<210>145<211>420<212>dna<213>阿维链霉菌<400>145atgcttagaaccatgttcaaatccaagatccacagagcaaccgtcactcaagcagatttg60cattacgttggttctgttactattgacgcagacttacttgatgcagccgatttacttcct120ggtgagcttgttcatattgttgatatcaccaatggtgcgcgtcttgaaacctatgtcatt180gagggtgaacgtggttccggtgtcgtcggtatcaatggcgcagctgcacacctcgtccat240ccaggtgacctggtcatcatcatttcttatgcacaggtctccgatgctgaagcccgtgcc300ttaagaccaagagtcgttcacgtcgacagagataacagagttgtcgctttaggtgcagac360ccagcagagccagttccaggttccgatcaagctagatcaccacaggctgttaccgcctaa420<210>146<211>420<212>dna<213>阿维链霉菌<400>146atgttaagaactatgtttaagtccaagattcacagagctaccgtcacccaagcagatttg60cattacgtcggttccgttaccattgatgcagatttactcgatgcggcagacttgttacct120ggtgaactagttcacattgttgacattaccaatggtgctagattggaaacttacgtcatt180gaaggtgaaagaggtagtggtgtcgttggtatcaatggtgcagctgctcacttagttcac240ccaggtgacttagtcatcatcatttcatatgcacaagtttccgatgcggaagctagagca300ttaagaccaagagttgttcatgttgatagagacaacagagtcgttgcacttggtgcagat360cctgctgaaccagttccaggttcagatcaagctaggtctccacaagcagttactgcataa420<210>147<211>420<212>dna<213>阿维链霉菌<400>147atgttaagaactatgttcaagagtaagattcatagggctaccgttacccaggcagatcta60cattatgttggttctgttaccattgacgcagatttgttggatgcagcagacttgttacca120ggtgaactcgttcacattgtcgatatcaccaacggtgccagactggaaacttatgtcatt180gaaggtgagagaggcagtggtgttgtcggcattaacggtgccgcagcacacttggttcat240ccaggtgacttggtcatcatcatttcttacgcacaagtctccgatgccgaagctagagca300ttgagacctagagtcgttcacgttgacagagacaatagagttgttgctcttggtgcagat360ccagctgagccagttccaggttccgatcaagcgagatctcctcaagctgttactgcataa420<210>148<211>384<212>dna<213>地衣芽孢杆菌<400>148atgtatagaaccttgatgagtgcaaagcttcacagagcaagagtcactgaagcaaacttg60aactacgttggttctgttactattgacgaagacttacttgatgcagtcggtatgatggca120aacgagaaagttcaaattgtcaacaataacaatggtgcgcgtcttgaaacctatatcatt180cctggtgaacgtggttccggtgtcgtctgcttgaatggcgcagctgcaaggctcgtccaa240gttggtgacgtcgtcatcattgtttcttatgcaatgatgtccgaagaagaagccaaaaca300cataagccaaaggtcgctgtcctcaatgaaagaaacgaaattgaggaaatgttaggtcag360gaaccagcgagaaccatcttataa384<210>149<211>384<212>dna<213>地衣芽孢杆菌<400>149atgtatagaaccttgatgagtgcaaagcttcacagagcaagagtcactgaagcaaacttg60aactacgttggttctgttactattgacgaagacttacttgatgcagtcggtatgatggca120aacgagaaagttcaaattgtcaacaataacaatggtgcgcgtcttgaaacctatatcatt180cctggtgaacgtggttccggtgtcgtctgcttgaatggcgcagctgcaaggctcgtccaa240gttggtgacgtcgtcatcattgtttcttatgcaatgatgtccgaagaagaagccaaaaca300cataagccaaaggtcgctgtcctcaatgaaagaaacgaaattgaggaaatgttaggtcag360gaaccagcgagaaccatcttataa384<210>150<211>384<212>dna<213>地衣芽孢杆菌<400>150atgtacagaacgttaatgtctgccaagttacacagagcgagagttactgaagcaaatctc60aactatgttggttcagttaccattgatgaggatctattggatgctgttggtatgatggca120aatgaaaaggttcaaatcgtcaataacaacaatggtgctagattagaaacttacatcatt180cctggtgaaagaggttcaggtgttgtttgcttaaacggtgctgccgcaagattagttcaa240gtcggtgatgttgttatcatcgtctcttatgctatgatgagtgaggaagaagctaagact300cataagcctaaggttgccgttctcaatgagagaaacgaaatcgaagaaatgcttggccaa360gaacctgccagaaccatcctgtaa384<210>151<211>384<212>dna<213>地衣芽孢杆菌<400>151atgtatagaacattgatgtctgcaaagttgcatagggctcgtgttactgaggcaaatcta60aactacgtcggttccgtcacaatcgatgaagatctactcgatgccgttggtatgatggcc120aatgaaaaagttcaaattgtcaacaacaacaacggtgcaagattggagacctatatcatt180cctggtgaaagaggttcaggtgttgtttgtctgaacggtgctgctgcgaggttggtccaa240gtcggtgatgttgtcattatcgtttcttatgcaatgatgtcagaagaagaagctaagacc300cataagccaaaggttgctgttttgaacgaaagaaatgagattgaggaaatgttaggtcaa360gaaccagcaagaacaatcttataa384<210>152<211>54<212>dna<213>东方伊萨酵母<400>152taaaacgacggccagtgaattccgcggcggccgcgagtccatcggttcctgtca54<210>153<211>59<212>dna<213>东方伊萨酵母<400>153cataagaaaatcaaagacagaaggcgcgcctttgctagcatttttgtgttttgctgtgt59<210>154<211>59<212>dna<213>人工序列<220><223>人工引物<400>154acacagcaaaacacaaaaatgctagcaaaggcgcgccttctgtctttgattttcttatg59<210>155<211>45<212>dna<213>人工序列<220><223>人工引物<400>155gggggaaagaactaccaataggcctcctttaattcggagaaaatc45<210>156<211>45<212>dna<213>人工序列<220><223>人工引物<400>156gattttctccgaattaaaggaggcctattggtagttctttccccc45<210>157<211>51<212>dna<213>人工序列<220><223>人工引物<400>157aaaataaactagtaaaataaattaattaattatctagagagggggttatat51<210>158<211>51<212>dna<213>人工序列<220><223>人工引物<400>158atataaccccctctctagataattaattaatttattttactagtttatttt51<210>159<211>53<212>dna<213>人工序列<220><223>人工引物<400>159gaccatgattacgccaagctccgcggcggccgcccagtcaaaaccttcttctc53<210>160<211>52<212>dna<213>人工序列<220><223>人工引物<400>160taaaacgacggccagtgaattccgcggcggccgcctttgaaggagcttgcca52<210>161<211>49<212>dna<213>人工序列<220><223>人工引物<400>161ctattccttcctcaaattgctgtttaaacgcgttgaagatctattctcc49<210>162<211>49<212>dna<213>人工序列<220><223>人工引物<400>162ggagaatagatcttcaacgcgtttaaacagcaatttgaggaaggaatag49<210>163<211>55<212>dna<213>人工序列<220><223>人工引物<400>163aatgttcattttacattcagatgttaattaaggtctagatgtgtttgtttgtgtg55<210>164<211>55<212>dna<213>人工序列<220><223>人工引物<400>164cacacaaacaaacacatctagaccttaattaacatctgaatgtaaaatgaacatt55<210>165<211>53<212>dna<213>人工序列<220><223>人工引物<400>165gaccatgattacgccaagctccgcggcggccgcaatgccaagagttatggggc53<210>166<211>23<212>dna<213>人工序列<220><223>人工引物<400>166ggctaccctatatatggtgagcg23<210>167<211>21<212>dna<213>人工序列<220><223>人工引物<400>167gggtccaagttatccaagcag21<210>168<211>29<212>dna<213>人工序列<220><223>人工引物<400>168ccttaattaaccgtaaagttgtctcaatg29<210>169<211>55<212>dna<213>人工序列<220><223>人工引物<400>169cagcaaaacacaaaaattctagaaaatttaattaacatctgaatgtaaaatgaac55<210>170<211>55<212>dna<213>人工序列<220><223>人工引物<400>170gttcattttacattcagatgttaattaaattttctagaatttttgtgttttgctg55<210>171<211>32<212>dna<213>人工序列<220><223>人工引物<400>171gctctagataaaatgccctcttattctgtcgc32<210>172<211>20<212>dna<213>人工序列<220><223>人工引物<400>172gactggatcattatgactcc20<210>173<211>44<212>dna<213>人工序列<220><223>人工引物<400>173acgccttgccaaatgcggccgcgagtccatcggttcctgtcaga44<210>174<211>49<212>dna<213>人工序列<220><223>人工引物<400>174tcaaagacagaattaattaagctctagaatttttgtgttttgctgtgtt49<210>175<211>49<212>dna<213>人工序列<220><223>人工引物<400>175aacacaaaaattctagagcttaattaattctgtctttgattttcttatg49<210>176<211>32<212>dna<213>人工序列<220><223>人工引物<400>176gtttaaacctttaattcggagaaaatctgatc32<210>177<211>30<212>dna<213>人工序列<220><223>人工引物<400>177gttaacggtaccgagctctaagtagtggtg30<210>178<211>41<212>dna<213>人工序列<220><223>人工引物<400>178aaccgatggactcgcggccgcatttggcaaggcgtatctat41<210>179<211>30<212>dna<213>人工序列<220><223>人工引物<400>179ggcgccagcaatttgaggaaggaataggag30<210>180<211>52<212>dna<213>人工序列<220><223>人工引物<400>180aaactattagattaattaagctcgcgatgtgtttgtttgtgtgttttgtgtg52<210>181<211>52<212>dna<213>人工序列<220><223>人工引物<400>181caaacaaacacatcgcgagcttaattaatctaatagtttaatcacagcttat52<210>182<211>42<212>dna<213>人工序列<220><223>人工引物<400>182tacattatggtagcggccgcgtgtgacattttgatccactcg42<210>183<211>47<212>dna<213>人工序列<220><223>人工引物<400>183tggatcaaaatgtcacacgcggccgctaccataatgtatgcgttgag47<210>184<211>27<212>dna<213>人工序列<220><223>人工引物<400>184gggccctaaaagtgttggtgtattaga27<210>185<211>33<212>dna<213>人工序列<220><223>人工引物<400>185gctagctcaacaaactctttatcagatttagca33<210>186<211>30<212>dna<213>人工序列<220><223>人工引物<400>186gctagcgaggaaaagaagtctaacctttgt30<210>187<211>35<212>dna<213>人工序列<220><223>人工引物<400>187tcgcgataaaatgtcaactgtggaagatcactcct35<210>188<211>27<212>dna<213>人工序列<220><223>人工引物<400>188ttaattaagctgctggcgcttcatctt27<210>189<211>32<212>dna<213>人工序列<220><223>人工引物<400>189tcgcgataaaatgtccagaggcttctttactg32<210>190<211>30<212>dna<213>人工序列<220><223>人工引物<400>190ttaattaactaaaggtctctcacgacagag30<210>191<211>40<212>dna<213>人工序列<220><223>人工引物<400>191gttaacccggtttaaacatagcctcatgaaatcagccatt40<210>192<211>44<212>dna<213>人工序列<220><223>人工引物<400>192gggcccatatggcgcccggggcgttgaagatctattctccagca44<210>193<211>30<212>dna<213>人工序列<220><223>人工引物<400>193gtttaaacgattggtagttctttccccctc30<210>194<211>46<212>dna<213>人工序列<220><223>人工引物<400>194aataaattaagggccctttatcgcgagagggggttatatgtgtaaa46<210>195<211>50<212>dna<213>人工序列<220><223>人工引物<400>195tataaccccctctcgcgataaagggcccttaatttattttactagtttat50<210>196<211>27<212>dna<213>人工序列<220><223>人工引物<400>196gtttaaacttttgtagcacctcctggt27<210>197<211>48<212>dna<213>人工序列<220><223>人工引物<400>197cagcaaaacacaaaaagctagctaaaatgttacgtaccatgttcaaaa48<210>198<211>46<212>dna<213>人工序列<220><223>人工引物<400>198gaaaatcaaagacagaaggcgcgccttatgctgtaacagcctgcgg46<210>199<211>49<212>dna<213>人工序列<220><223>人工引物<400>199cagcaaaacacaaaaagctagctaaaatgttaagaaccatgttcaaatc49<210>200<211>47<212>dna<213>人工序列<220><223>人工引物<400>200gaaaatcaaagacagaaggcgcgccttatgcagtaacagcttgtggg47<210>201<211>20<212>dna<213>人工序列<220><223>人工引物<400>201gcatggtggtgcaagcgacg20<210>202<211>19<212>dna<213>人工序列<220><223>人工引物<400>202ggtgctgcatttgctgctg19<210>203<211>25<212>dna<213>人工序列<220><223>人工引物<400>203tgtatacaggatcgaagaatagaag25<210>204<211>22<212>dna<213>人工序列<220><223>人工引物<400>204gaacgtctacaacgaggtgaac22<210>205<211>21<212>dna<213>人工序列<220><223>人工引物<400>205ggcgcgcctcgcgataaaatg21<210>206<211>32<212>dna<213>人工序列<220><223>人工引物<400>206agggcccttaattaattatgcagtaacagctt32<210>207<211>20<212>dna<213>人工序列<220><223>人工引物<400>207cgctacgatacgctacgata20<210>208<211>20<212>dna<213>人工序列<220><223>人工引物<400>208ctcccttccctgatagaagg20<210>209<211>21<212>dna<213>人工序列<220><223>人工引物<400>209ggggagcaatttgccaccagg21<210>210<211>25<212>dna<213>人工序列<220><223>人工引物<400>210ctccttcatttaactataccagacg25<210>211<211>26<212>dna<213>人工序列<220><223>人工引物<400>211gacagatgtaaggaccaatagtgtcc26<210>212<211>23<212>dna<213>人工序列<220><223>人工引物<400>212ccatatcgaaatctagcccgtcc23<210>213<211>58<212>dna<213>人工序列<220><223>人工引物<400>213cataagaaaatcaaagacagaaggcgcgcctttgctagctttttgtgttttgctgtgt58<210>214<211>19<212>dna<213>人工序列<220><223>人工引物<400>214attggacacaacattatat19<210>215<211>46<212>dna<213>人工序列<220><223>人工引物<400>215gtaaaacgacggccagtgaattgttaacataggctccaacatctcg46<210>216<211>38<212>dna<213>人工序列<220><223>人工引物<400>216ggaaccgatggactcgcggccgcgtgggatattggaag38<210>217<211>22<212>dna<213>人工序列<220><223>人工引物<400>217ggaggtgctacaaaaggaattc22<210>218<211>48<212>dna<213>人工序列<220><223>人工引物<400>218gaccatgattacgccaagctccgcggagtcaaaaccttcttctctacc48<210>219<211>46<212>dna<213>人工序列<220><223>人工引物<400>219gtaaaacgacggccagtgaattctttgaaggagcttgccaagaaac46<210>220<211>23<212>dna<213>人工序列<220><223>人工引物<400>220cctattccttcctcaaattgctg23<210>221<211>38<212>dna<213>人工序列<220><223>人工引物<400>221ataactcttggcattgcggccgccaagttagttagagc38<210>222<211>51<212>dna<213>人工序列<220><223>人工引物<400>222atgaccatgattacgccaagctccgcggcaaagacggtgtattagtgcttg51<210>223<211>64<212>dna<213>人工序列<220><223>人工引物<400>223cacaaaacacacaaacaaacacagctagcaaaggcgcgccatctaatagtttaatcacag60ctta64<210>224<211>47<212>dna<213>人工序列<220><223>人工引物<400>224gccgttgcagcaaatgtcgaggcctgtgtgacattttgatccactcg47<210>225<211>47<212>dna<213>人工序列<220><223>人工引物<400>225cgagtggatcaaaatgtcacacaggcctcgacatttgctgcaacggc47<210>226<211>51<212>dna<213>人工序列<220><223>人工引物<400>226cattttacattcagatgttaattaattatctagatgttgttgttgttgtcg51<210>227<211>51<212>dna<213>人工序列<220><223>人工引物<400>227cgacaacaacaacaacatctagataattaattaacatctgaatgtaaaatg51<210>228<211>51<212>dna<213>人工序列<220><223>人工引物<400>228gctctaactaacttggcggccgcttttattataaaattatatattattctt51<210>229<211>47<212>dna<213>人工序列<220><223>人工引物<400>229aacacacaaacaaacacagctagctaaaatgttaagaactatgttta47<210>230<211>46<212>dna<213>人工序列<220><223>人工引物<400>230gattaaactattagatggcgcgccttatgcagtaactgcttgtgga46<210>231<211>42<212>dna<213>人工序列<220><223>人工引物<400>231cgacggccagtgaattcgttaacccgtttcgatgggattccc42<210>232<211>46<212>dna<213>人工序列<220><223>人工引物<400>232caggaaccgatggactcgcggccgctcccttctctaaatggactgc46<210>233<211>48<212>dna<213>人工序列<220><223>人工引物<400>233tatataattttataataaaagcggccgcaccaggggtttagtgaagtc48<210>234<211>47<212>dna<213>人工序列<220><223>人工引物<400>234catgattacgccaagctccgcggccataactgacatttatggtaagg47<210>235<211>21<212>dna<213>人工序列<220><223>人工引物<400>235ccaacaatcttaattggtgac21<210>236<211>20<212>dna<213>人工序列<220><223>人工引物<400>236ggctgttaccgcctaattaa20<210>237<211>23<212>dna<213>人工序列<220><223>人工引物<400>237gttcttaacattttagctagctg23<210>238<211>30<212>dna<213>人工序列<220><223>人工引物<400>238acgcgtcgactcgacatttgctgcaacggc30<210>239<211>29<212>dna<213>人工序列<220><223>人工引物<400>239ctagtctagatgttgttgttgttgtcgtt29<210>240<211>49<212>dna<213>人工序列<220><223>人工引物<400>240caaaacacagcaaaacacaaaaagctagcatgtatagaaccttgatgag49<210>241<211>40<212>dna<213>人工序列<220><223>人工引物<400>241caaagacagaaggcgcgccttataagatggttctcgctgg40<210>242<211>50<212>dna<213>人工序列<220><223>人工引物<400>242caaaacacacaaacaaacacagctagcatgtacagaacgttaatgtctgc50<210>243<211>47<212>dna<213>人工序列<220><223>人工引物<400>243gtgattaaactattagatggcgcgccttacaggatggttctggcagg47<210>244<211>703<212>dna<213>东方伊萨酵母<400>244gagtccatcggttcctgtcagatgggatactcttgacgtggaaaattcaaacagaaaaaa60aaccccaataatgaaaaataacactacgttatatccgtggtatcctctatcgtatcgtat120cgtagcgtatcgtagcgtaccgtatcacagtatagtctaatattccgtatcttattgtat180cctatcctattcgatcctattgtatttcagtgcaccattttaatttctattgctataatg240tccttattagttgccactgtgaggtgaccaatggacgagggcgagccgttcagaagccgc300gaagggtgttcttcccatgaatttcttaaggagggcggctcagctccgagagtgaggcga360gacgtctcggtcagcgtatcccccttcctcggcttttacaaatgatgcgctcttaatagt420gtgtcgttatccttttggcattgacgggggagggaaattgattgagcgcatccatatttt480tgcggactgctgaggacaatggtggtttttccgggtggcgtgggctacaaatgatacgat540ggtttttttcttttcggagaaggcgtataaaaaggacacggagaacccatttattctaaa600aacagttgagcttctttaattattttttgatataatattctattattatatattttcttc660ccaataaaacaaaataaaacaaaacacagcaaaacacaaaaat703<210>245<211>961<212>dna<213>东方伊萨酵母<400>245agcaatttgaggaaggaataggagaaggagaagcaatttctaggaaagagcaaggtgtgc60aacagcatgctctgaatgatattttcagcaatagttcagttgaagaacctgttggcgtat120ctacatcacttcctacaaacaacaccacgaattgcgtccgtggtgacgcaactacgaatg180gcattgtcaatgccaatgccagtgcacatacacgtgcaagtcccaccggttccctgcccg240gctatggtagagacaagaaggacgataccggcatcgacatcaacagtttcaacagcaatg300cgtttggcgtcgacgcgtcgatggggctgccgtatttggatttggacgggctagatttcg360atatggatatggatatggatatggatatggagatgaatttgaatttagatttgggtcttg420atttggggttggaattaaaaggggataacaatgagggttttcctgttgatttaaacaatg480gacgtgggaggtgattgatttaacctgatccaaaaggggtatgtctattttttagagtgt540gtctttgtgtcaaattatggtagaatgtgtaaagtagtataaactttcctctcaaatgac600gaggtttaaaacaccccccgggtgagccgagccgagaatggggcaattgttcaatgtgaa660atagaagtatcgagtgagaaacttgggtgttggccagccaagggggaaggaaaatggcgc720gaatgctcaggtgagattgttttggaattgggtgaagcgaggaaatgagcgacccggagg780ttgtgactttagtggcggaggaggacggaggaaaagccaagagggaagtgtatataaggg840gagcaatttgccaccaggatagaattggatgagttataattctactgtatttattgtata900atttatttctccttttatatcaaacacattacaaaacacacaaaacacacaaacaaacac960a961<210>246<211>992<212>dna<213>东方伊萨酵母<400>246tattggtagttctttccccctctcaagctggcgtgaaatgcaaccttacggcgtctacgt60tactacaaggtccagaaagtgtaggtattgctactatttttattttttattggttctgga120gaaatgcagacagtcaatgaacacaactgtctcaatatgcatctatgcacatgcacacac180acacacatcacaggtacccctacaaagagaggtctcttgataatgtttcattaccacgtg240gcatccccccccccccccccaataaacaagtggccgagttcccctgttgcagaggaggac300aaaaaaaccgctggtgttggtaccattatgcagcaactagcacaacaaacaaccgaccca360gacatacaaatcaacaacacttcgccaaagacaccctttccagggaggatccactcccaa420cgtctctccataatgtctctgttggcccatgtctctgtcgttgacaccgtaaccacacca480accaacccgtccattgtactgggatggtcgtccatagacacctctccaacggggaacacc540tcattcgtaaaccgccaaggttaccgttcctcctgactcgccccgttgttgatgctgcgc600acctgtggttgcccaacatggttgtatatcgtgtaaccacaccaacacatgtgcagcaca660tgtgtttaaaagagtgtcatggaggtggatcatgatggaagtggactttaccacttggga720actgtctccactcccgggaagaaaagacccggcgtatcacgcggttgcctcaatggggca780atttggaaggagaaatatagggaaaatcacgtcgctctcggacggggaagagttccagac840tatgaggggggggggtggtatataaagacaggagatgtccacccccagagagaggaagaa900gttggaactttagaagagagagataactttccccagtgtccatcaatacacaaccaaaca960caaactctatatttacacatataaccccctct992<210>247<211>613<212>dna<213>东方伊萨酵母<400>247ttgctgcaacggcaacatcaatgtccacgtttacacacctacatttatatctatatttat60atttatatttatttatttatgctacttagcttctatagttagttaatgcactcacgatat120tcaaaattgacacccttcaactactccctactattgtctactactgtctactactcctct180ttactatagctgctcccaataggctccaccaataggctctgtcaatacattttgcgccgc240cacctttcaggttgtgtcactcctgaaggaccatattgggtaatcgtgcaatttctggaa300gagagtccgcgagaagtgaggcccccactgtaaatcctcgagggggcatggagtatgggg360catggaggatggaggatggggggggggggggggaaaataggtagcgaaaggacccgctat420caccccacccggagaactcgttgccgggaagtcatatttcgacactccggggagtctata480aaaggcgggttttgtcttttgccagttgatgttgctgagaggacttgtttgccgtttctt540ccgatttaacagtatagaatcaaccactgttaattatacacgttatactaacacaacaaa600aacaaaaacaacg613<210>248<211>399<212>dna<213>东方伊萨酵母<400>248tctgtctttgattttcttatgttattcaaaacatctgccccaaaatctaacgattatata60tattcctacgtataactgtatagctaattattgatttatttgtacataaaaaccacataa120atgtaaaagcaagaaaaaaaataactaaggagaaggatcaatatctcatttataatgctc180gccaaagcagcgtacgtgaattttaatcaagacatcaacaaatcttgcaacttggttata240tcgcttcttcacccactcacccgcttttctacattgttgaacacaaatatatacaggggt300atgtctcaaggtcaagtgcagtttcaacagagactacctcaaggtacctcttcagaaatg360cagaacttcactcttgatcagattttctccgaattaaag399<210>249<211>422<212>dna<213>东方伊萨酵母<400>249atctaatagtttaatcacagcttatagtctattatagttttcttttttaaacattgttgt60attttgtccccccccctctaattgatgatgattatcctataagaatccaataaaacgatg120gaaactaatactttctcctttgtcatgtggtctttagtatttcttgaacattggctctga180tttctcgactttatagtcctattaaaatctctgttagttctcgatcgttgtatctcgttt240cttgtctctttggtggatgattttgcgtgcgaacatgtttttttccctttctctcaccat300catcgtgtagttcttgtcaccatccccccaccccttccttctctcattgattctataaga360gcttatccacagaggtgcagtaacgaggtagtttaaccttcgagtggatcaaaatgtcac420ac422<210>250<211>421<212>dna<213>东方伊萨酵母<400>250tttattttactagtttatttttgctcctgagaataggattacaaacacttaaagtcttta60attacaactatatataatattctgttggttttcttgaattggttcgctgcgattcatgcc120tcccattcaccaaaggtggagtgggaaataacggttttactgcggtaattagcagaggca180agaacaggatacactttttgatgataaatctgtattatagtcgagcctatttaggaaatc240aaattttcttgtgtttacttttcaaataaataatgttcgaaaatttttactttactcctt300catttaactataccagacgttatatcatcaacaccttctgaccatatacagctcaagatg360tttaagagtctgttaaattttttcaatccatttcatggagtaccaggaggtgctacaaaa420g421<210>251<211>281<212>dna<213>东方伊萨酵母<400>251catctgaatgtaaaatgaacattaaaatgaattactaaactttacgtctactttacaatc60tataaactttgtttaatcatataacgaaatacactaatacacaatcctgtacgtatgtaa120tacttttatccatcaaggattgagaaaaaaaagtaatgattccctgggccattaaaactt180agacccccaagcttggataggtcactctctattttcgtttctcccttccctgatagaagg240gtgatatgtaattaagaataatatataattttataataaaa281<210>252<211>1394<212>dna<213>东方伊萨酵母<400>252catagcctcatgaaatcagccatttgcttttgttcaacgatcttttgaaattgttgttgt60tcttggtagttaagttgatccatcttggcttatgttgtgtgtatgttgtagttattctta120gtatattcctgtcctgagtttagtgaaacataatatcgccttgaaatgaaaatgctgaaa180ttcgtcgacatacaatttttcaaactttttttttttcttggtgcacggacatgtttttaa240aggaagtactctataccagttattcttcacaaatttaattgctggagaatagatcttcaa300cgctttaataaagtagtttgtttgtcaaggatggcgtcatacaaagaaagatcagaatca360cacacttcccctgttgctaggagacttttctccatcatggaggaaaagaagtctaacctt420tgtgcatcattggatattactgaaactgaaaagcttctctctattttggacactattggt480ccttacatctgtctagttaaaacacacatcgatattgtttctgattttacgtatgaagga540actgtgttgcctttgaaggagcttgccaagaaacataattttatgatttttgaagataga600aaatttgctgatattggtaacactgttaaaaatcaatataaatctggtgtcttccgtatt660gccgaatgggctgacatcactaatgcacatggtgtaacgggtgcaggtattgtttctggc720ttgaaggaggcagcccaagaaacaaccagtgaacctagaggtttgctaatgcttgctgag780ttatcatcaaagggttctttagcatatggtgaatatacagaaaaaacagtagaaattgct840aaatctgataaagagtttgtcattggttttattgcgcaacacgatatgggcggtagagaa900gaaggttttgactggatcattatgactccaggggttggtttagatgacaaaggtgatgca960cttggtcaacaatatagaactgttgatgaagttgtaaagactggaacggatatcataatt1020gttggtagaggtttgtacggtcaaggaagagatcctatagagcaagctaaaagataccaa1080caagctggttggaatgcttatttaaacagatttaaatgattcttacacaaagatttgata1140catgtacactagtttaaataagcatgaaaagaattacacaagcaaaaaaaaaaaaataaa1200tgaggtactttacgttcacctacaaccaaaaaaactagatagagtaaaatcttaagattt1260agaaaaagttgtttaacaaaggctttagtatgtgaatttttaatgtagcaaagcgataac1320taataaacataaacaaaagtatggttttctttatcagtcaaatcattatcgattgattgt1380tccgcgtatctgca1394<210>253<211>523<212>dna<213>东方伊萨酵母<400>253gtataaactgtatgtcattataaacagggaaggttgacattgtctagcggcaatcattgt60ctcatttggttcattaactttggttctgttcttggaaacgggtaccaactctctcagagt120gcttcaaaaatttttcagcacatttggttagacatgaactttctctgctggttaaggatt180cagaggtgaagtcttgaacacaatcgttgaaacatctgtccacaagagatgtgtatagcc240tcatgaaatcagccatttgcttttgttcaacgatcttttgaaattgttgttgttcttggt300agttaagttgatccatcttggcttatgttgtgtgtatgttgtagttattcttagtatatt360cctgtcctgagtttagtgaaacataatatcgccttgaaatgaaaatgctgaaattcgtcg420acatacaatttttcaaactttttttttttcttggtgcacggacatgtttttaaaggaagt480actctataccagttattcttcaccctgcagggtacgtagcatg523<210>254<211>626<212>dna<213>东方伊萨酵母<220><221>misc_feature<222>(28)..(28)<223>nisa,c,g,或t<400>254gggcaacaaagcctcccagatttgatanattttcaatttgtgctttgaatcatgacttcc60acctgtttggtccgcaagaacacgtaaatgcgcaatttgtttctcccttctgcttaaaaa120ccatgcacctttaatattatctggaaagataaagaacagaattgttgcgtagaaacaagt180agcagagccgtaaatgagaaaaatatacttccaagctggtaatttcccctttattagtcc240aatacagtgtccgaagaccccaccaagaataccagcaagggtgttgaaatataatgtaga300tcttagtggttgttctgatttcttccaccacattccgctaataatcataaaagacggtaa360tattccggcttcaaatacgccaagaaaaaacctcacggtaaccaaaccaccaaagctatg420acatgcagccatgcacataagtaagccgccccaaatgaacaaacaaatagacacaaattt480gccaattctaactcgtggcaacaaaaaaaaggatatgaactcacctaataaataaccgaa540ataaaaagtagaagcaactgtggaaaattgagaaccatgtaaatttgtgtcttctttcaa600tgtataaacagccgcaatacctaggg626<210>255<211>1416<212>dna<213>酿酒酵母<400>255atgtttgctttctactttctcaccgcatgcaccactttgaagggtgttttcggagtttct60ccgagttacaatggtcttggtctcaccccacagatgggttgggacagctggaatacgttt120gcctgcgatgtcagtgaacagctacttctagacactgctgatagaatttctgacttgggg180ctaaaggatatgggttacaagtatgtcatcctagatgactgttggtctagcggcagggat240tccgacggtttcctcgttgcagacaagcacaaatttcccaacggtatgggccatgttgca300gaccacctgcataataacagctttcttttcggtatgtattcgtctgctggtgagtacacc360tgtgctgggtaccctgggtctctggggcgtgaggaagaagatgctcaattctttgcaaat420aaccgcgttgactacttgaagtatgataattgttacaataaaggtcaatttggtacacca480gacgtttcttaccaccgttacaaggccatgtcagatgctttgaataaaactggtaggcct540attttctattctctatgtaactggggtcaggatttgacattttactggggctctggtatc600gccaattcttggagaatgagcggagatattactgctgagttcacccgtccagatagcaga660tgtccctgtgacggtgacgaatatgattgcaagtacgccggtttccattgttctattatg720aatattcttaacaaggcagctccaatggggcaaaatgcaggtgttggtggttggaacgat780ctggacaatctagaggtcggagtcggtaatttgactgacgatgaggaaaaggcccatttc840tctatgtgggcaatggtaaagtccccacttatcattggtgccgacgtgaatcacttaaag900gcatcttcgtactcgatctacagtcaagcctctgtcatcgcaattaatcaagatccaaag960ggtattccagccacaagagtctggagatattatgtttcagacaccgatgaatatggacaa1020ggtgaaattcaaatgtggagtggtccgcttgacaatggtgaccaagtggttgctttattg1080aatggaggaagcgtagcaagaccaatgaacacgaccttggaagagattttctttgacagc1140aatttgggttcaaaggaactgacatcgacttgggatatttacgacttatgggccaacaga1200gttgacaactctacggcgtctgctatccttgaacagaataaggcagccaccggtattctc1260tacaatgctacagagcagtcttataaagacggtttgtctaagaatgatacaagactgttt1320ggccagaaaattggtagtctttctccaaatgctatacttaacacaactgttccagctcat1380ggtatcgccttctataggttgagaccctcggcttaa1416<210>256<211>707<212>dna<213>kluyveromycesthermotolerans<400>256cactcgcaagctgtgccatcgcccaacggttaattataagaaatcaacatcagccaacaa60ctattttcgtccccctcttttcagtggtaacgagcaattacattagtaagagactatttt120cttcagtgatttgtaattttttttcagtgatttgtaattctttctcgaaatatgcgggct180taacttatccggacattcactacatgcaaggaaaaacgagaaccgcggagatttcctcag240taagtaacaatgatgatctttttacgcttcatcatcactttccaaagttctaagctataa300gttcaagcctagatacgctgaaaaactcctgaccaacaatgtaaagaaaacaattacaat360tgtaaggttgaaaacatctaaaaatgaaatattttattgtacatgcacaccctgatagtc420attctcttacttcatccctgaaagacgtggctgtacaagagttggaatcgcaaggtcatg480aggttaaagttagtgatctttatgctcaaaagtggaaggcctcaatagaccgtgacgact540tcgagcagcttttcgcaagaagagaggttaaaaataccccaagcttcttatgaagcgtat600gccagaggagcattaacaaaagacgtaaatcaggaacaggaaaaacttatttgggcggac660tttgtcattttgtcgtttcctatatggtggtcttctatgccggctag707<210>257<211>655<212>dna<213>kluyveromycesthermotolerans<400>257ggttaattataagaaatcaacatcagccaacaactattttcgtccccctcttttcagtgg60taacgagcaattacattagtaagagactattttcttcagtgatttgtaattttttttcag120tgatttgtaattctttctcgaaatatgcgggcttaacttatccggacattcactacatgc180aaggaaaaacgagaaccgcggagatttcctcagtaagtaacaatgatgatctttttacgc240ttcatcatcactttccaaagttctaagctataagttcaagcctagatacgctgaaaaact300cctgaccaacaatgtaaagaaaacaattacaattgtaaggttgaaaacatctaaaaatga360aatattttattgtacatgcacaccctgatagtcattctcttacttcatccctgaaagacg420tggctgtacaagagttggaatcgcaaggtcatgaggttaaagttagtgatctttatgctc480aaaagtggaaggcctcaatagaccgtgacgacttcgagcagcttttcgcaagaagagagg540ttaaaaataccccaagcttcttatgaagcgtatgccagaggagcattaacaaaagacgta600aatcaggaacaggaaaaacttatttgggcggactttgtcattttgtcgtttccta655<210>258<211>523<212>dna<213>东方伊萨酵母<400>258gtataaactgtatgtcattataaacagggaaggttgacattgtctagcggcaatcattgt60ctcatttggttcattaactttggttctgttcttggaaacgggtaccaactctctcagagt120gcttcaaaaatttttcagcacatttggttagacatgaactttctctgctggttaaggatt180cagaggtgaagtcttgaacacaatcgttgaaacatctgtccacaagagatgtgtatagcc240tcatgaaatcagccatttgcttttgttcaacgatcttttgaaattgttgttgttcttggt300agttaagttgatccatcttggcttatgttgtgtgtatgttgtagttattcttagtatatt360cctgtcctgagtttagtgaaacataatatcgccttgaaatgaaaatgctgaaattcgtcg420acatacaatttttcaaactttttttttttcttggtgcacggacatgtttttaaaggaagt480actctataccagttattcttcaccctgcagggtacgtagcatg523<210>259<211>707<212>dna<213>东方伊萨酵母<400>259tgataacagcaatctgcatgtggattcataatattaggtgtttattgtgggtattaacgt60gtctacctgctttagctggggctattatggtaaataagattgatccacacgaaaatcgac120atgctgcattagctggaatttatttgatgggattctacaatgtgccatggacattaatgt180tggcgttggtatcttcaaacacctcaggatctaccaagaagacgttcatgtatgtttctg240ttgcagtttggtatgcagttggtaacattatcggtccatattttttcaaaggttctcaag300caccaaagtatccgatggggatacatgccatggaagcttccttttcaattatggcgttca360ccggtatcctttactatcttatgctgagaatacagaataaacaaaagcaatcgttaacta420tgaatgatataaaacacttagaatacgcggaacaatcaatcgataatgatttgactgata480aagaaaaccatacttttgtttatgtttattagttatcgctttgctacattaaaaattcac540atactaaagcctttgttaaacaactttttctaaatcataagattttactctatctagttt600ttttggttgtaggtgaacgtaaagtacctcatttaatttttttttgcttgtgtaattctt660ttcatgcttatttaaactagtgtacatgtatcaaatctttgtgtaag707<210>260<211>22<212>dna<213>人工序列<220><223>人工引物<400>260gcaactgatgttcacgaatgcg22<210>261<211>20<212>dna<213>人工序列<220><223>人工引物<400>261ttgccgttgcagcaaatctc20<210>262<211>20<212>dna<213>人工序列<220><223>人工引物<400>262aatgatccatggtccgagag20<210>263<211>21<212>dna<213>人工序列<220><223>人工引物<400>263acggcagtataccctatcagg21<210>264<211>28<212>dna<213>人工序列<220><223>人工引物<400>264gcaccaagagcagttttcccatctattg28<210>265<211>36<212>dna<213>人工序列<220><223>人工引物<400>265ccatatagttcttttctaacatcaacatcacacttc36<210>266<211>37<212>dna<213>人工序列<220><223>人工引物<400>266ggagaatagatcttcaacgctttaataaagtagtttg37<210>267<211>25<212>dna<213>人工序列<220><223>人工引物<400>267cgtgttgcgcaataaaaccaatgac25<210>268<211>20<212>dna<213>人工序列<220><223>人工引物<400>268cctcgaagagcttgaatttg20<210>269<211>20<212>dna<213>人工序列<220><223>人工引物<400>269gtagtgaatgtccggataag20<210>270<211>20<212>dna<213>人工序列<220><223>人工引物<400>270gcaaggtcatgaggttaaag20<210>271<211>20<212>dna<213>人工序列<220><223>人工引物<400>271aacacttatggcgtctcctc20<210>272<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>272cgaaccaattcaagaaaaccaac23<210>273<211>18<212>dna<213>人工序列<220><223>人工dna引物<400>273tctgtcccttggcgacgc18<210>274<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>274cttttcaaacagataagtacc21<210>275<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>275atgggctgacctgaaaattc20<210>276<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>276gctgaaaatatcattcagagcat23<210>277<211>18<212>dna<213>人工序列<220><223>人工dna引物<400>277actgttgatgtcgatgcc18<210>278<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>278tctgaatgcagtacgagttg20<210>279<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>279gaagggggtccaagttatcc20<210>280<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>280gatatgggcggtagagaaga20<210>281<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>281gctccttcaaaggcaacaca20<210>282<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>282tgaactatcacatgaacgta20<210>283<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>283tcaaggtagggtcacttaac20<210>284<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>284tgattccttcaatcacaggt20<210>285<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>285aaccgacctatcgaatgcct20<210>286<211>51<212>dna<213>人工序列<220><223>人工dna引物<400>286accatgattacgccaagcttggtaccttggggtttacgcttacagcgtact51<210>287<211>61<212>dna<213>人工序列<220><223>人工dna引物<400>287tcaacgccccgggggatctggatccgcggccgcaagaaattcctttcttttcccctttat60a61<210>288<211>58<212>dna<213>人工序列<220><223>人工dna引物<400>288aaccgatggactcctcgagggatccgcggccgcgcataacaaaattgtgcctaaccca58<210>289<211>53<212>dna<213>人工序列<220><223>人工dna引物<400>289acgactcactatagggcgaattgggccccgggaaaaggagagagaaaaggaga53<210>290<211>55<212>dna<213>人工序列<220><223>人工dna引物<400>290tatgaccatgattacgccaagcttggtacctcctacagaagtagcaccagcacca55<210>291<211>60<212>dna<213>人工序列<220><223>人工dna引物<400>291ggaaccgatggactcctcgagggatccgcggccgcagactaccgtgtaaataagagtacc60<210>292<211>61<212>dna<213>人工序列<220><223>人工dna引物<400>292tcttcaacgccccgggggatctggatccgcggccgcatttgatataaacgcttctataat60a61<210>293<211>58<212>dna<213>人工序列<220><223>人工dna引物<400>293gtaatacgactcactatagggcgaattgggcccaacatctgctgctgtaatatattca58<210>294<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>294gcatgtctgttaactctcaaaccat25<210>295<211>23<212>dna<213>人工序列<220><223>人工dna引物<400>295tccataatccaatagatcatccc23<210>296<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>296aacacaatggaaccaacctagt22<210>297<211>45<212>dna<213>人工序列<220><223>人工dna引物<400>297atttacacggtagtctgcggccgccatagcctcatgaaatcagcc45<210>298<211>40<212>dna<213>人工序列<220><223>人工dna引物<400>298ctctaggttcactggttgtttcttgggctgcctccttcaa40<210>299<211>40<212>dna<213>人工序列<220><223>人工dna引物<400>299ttgaaggaggcagcccaagaaacaaccagtgaacctagag40<210>300<211>56<212>dna<213>人工序列<220><223>人工dna引物<400>300tattatagaagcgtttatatcaaatgcggccgcggatccagatcccccggggcgtt56<210>301<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>301aatgatcaacttgagaggta20<210>302<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>302caggtctgttacataaagca20<210>303<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>303gtttacgctcaaatctcctcc21<210>304<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>304ggtacatgaagcaggctttgaagg24<210>305<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>305gattgtgtccgtcagcctttgctc24<210>306<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>306taccatattttcagaggatca21<210>307<211>21<212>dna<213>人工序列<220><223>人工dna引物<400>307aggatgttcttgcctgcaagt21<210>308<211>26<212>dna<213>人工序列<220><223>人工dna引物<400>308gatgatatagttgatgctttccaaag26<210>309<211>33<212>dna<213>人工序列<220><223>人工dna引物<400>309gggccccttcatttacgaaataaagtgccgcgg33<210>310<211>40<212>dna<213>人工序列<220><223>人工dna引物<400>310gcggccgcaaataaatttaaaataaacgatatcaaaattc40<210>311<211>30<212>dna<213>人工序列<220><223>人工dna引物<400>311cgacgccaaagaagggctcagccgaaaaag30<210>312<211>32<212>dna<213>人工序列<220><223>人工dna引物<400>312ccatttctttttcggctgagcccttctttggc32<210>313<211>32<212>dna<213>人工序列<220><223>人工dna引物<400>313gcggccgcaataacctcagggagaactttggc32<210>314<211>30<212>dna<213>人工序列<220><223>人工dna引物<400>314gagctcccaaacaaggtctcagtaggatcg30<210>315<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>315cgccataaggagaggctgtagatttgtc28<210>316<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>316ccaggacatcttgcttgcaatgtcg25<210>317<211>34<212>dna<213>人工序列<220><223>人工dna引物<400>317gttccatcgggcccctaaaggtctctcacgacag34<210>318<211>33<212>dna<213>人工序列<220><223>人工dna引物<400>318caaacacatcgcgataaaatgtccagaggcttc33<210>319<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>319gcaagaccttggatctgaaggg22<210>320<211>25<212>dna<213>人工序列<220><223>人工dna引物<400>320cgaaccaattcaagaaaaccaacag25<210>321<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>321gggcccgtcccttggcgacgccctgatc28<210>322<211>33<212>dna<213>人工序列<220><223>人工dna引物<400>322gcggccgctatttttgtgttttgctgtgttttg33<210>323<211>37<212>dna<213>人工序列<220><223>人工dna引物<400>323gcggccgccatctgaatgtaaaatgaacattaaaatg37<210>324<211>29<212>dna<213>人工序列<220><223>人工dna引物<400>324gagctcccccagttgttgttgcaattaac29<210>325<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>325gaagagacgtacaagatccgcc22<210>326<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>326taggaatggtgcatcatccaac22<210>327<211>40<212>dna<213>人工序列<220><223>人工dna引物<400>327ttcttatctgaaaactccgagttcgcaaagaaggttgaag40<210>328<211>45<212>dna<213>人工序列<220><223>人工dna引物<400>328ttattgaaattaatccaaggattcaagttgaacatacaattactg45<210>329<211>42<212>dna<213>人工序列<220><223>人工dna引物<400>329tttccaaaaaagttcgtgagttcgatggttgtatgattatgg42<210>330<211>22<212>dna<213>人工序列<220><223>人工dna引物<400>330cctctaccaccaccaccaaatg22<210>331<211>24<212>dna<213>人工序列<220><223>人工dna引物<400>331gggaagaaactaagaagaagtatg24<210>332<211>44<212>dna<213>人工序列<220><223>人工dna引物<400>332caatccttctagagagggggttatatgtgtaaatatagagtttg44<210>333<211>36<212>dna<213>人工序列<220><223>人工dna引物<400>333ttctaccctcgagattggttctttccccctctcaag36<210>334<211>44<212>dna<213>人工序列<220><223>人工dna引物<400>334gtggtcaatctagaaaatatgactgacaaaatctccctaggtac44<210>335<211>30<212>dna<213>人工序列<220><223>人工dna引物<400>335caattttggagctgattccaaatcgtaaac30<210>336<211>31<212>dna<213>人工序列<220><223>人工dna引物<400>336caagagtatcccatctgacaggaaccgatgg31<210>337<211>27<212>dna<213>人工序列<220><223>人工dna引物<400>337gctggagaatagatcttcaacgccccg27<210>338<211>29<212>dna<213>人工序列<220><223>人工dna引物<400>338cggagaaggcgtataaaaaggacacggag29<210>339<211>47<212>dna<213>人工序列<220><223>人工dna引物<400>339ggataaaagtattacatacgtacaggattgtgtattagtgtatttcg47<210>340<211>40<212>dna<213>人工序列<220><223>人工dna引物<400>340cctccagtgtttttctctctgtctctttgttttttttttc40<210>341<211>46<212>dna<213>人工序列<220><223>人工dna引物<400>341ggttaattaatttatttgtacataaaaaccacataaatgtaaaagc46<210>342<211>35<212>dna<213>人工序列<220><223>人工dna引物<400>342gaattcctttaattcggagaaaatctgatcaagag35<210>343<211>945<212>dna<213>metallosphaerasedula<400>343atgaccgaaaaggtctccgtcgtcggtgcaggcgtcatcggtgtcggttgggctaccttg60ttcgcttccaagggttactccgtctccttgtacaccgaaaagaaggaaaccttggacaag120ggtatcgaaaagttgcgtaactacgtccaagtcatgaagaacaactcccaaatcaccgaa180gacgtcaacaccgtcatctccagagtttcccctactaccaacttggacgaagccgttaga240ggtgcaaacttcgtcatcgaggctgtcatcgaagactacgacgctaagaagaagatcttc300ggttacttggactccgtcttggacaaggaagttatcttggcatcctccacctccggtttg360ttgatcaccgaagtccaaaaggctatgtccaagcacccagaaagagctgtcatcgcccac420ccatggaacccaccacacttgttgcctttggtcgaaatcgttccaggtgagaagacctcc480atggaagtcgttgagagaaccaagtccttgatggaaaagttggacagaatcgtcgtcgtt540ttgaagaaggaaatcccaggtttcatcggtaacagattggcattcgctttgttcagagaa600gctgtctacttggttgacgagggtgtcgcaaccgtcgaggacatcgacaaggttatgact660gccgctatcggcttgagatgggccttcatgggtccattcttgacctaccacttgggtggt720ggtgagggtggtttggaatacttcttcaacagaggtttcggttacggtgccaacgaatgg780atgcacaccttggctaagtacgacaagttcccatacaccggtgtcaccaaggccatccag840caaatgaaggaatactccttcatcaagggtaagaccttccaagagatctccaagtggaga900gacgaaaagttgttgaaggtctacaagttggtctgggaaaagtaa945<210>344<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>344agggtaccttagtacgaagg20<210>345<211>20<212>dna<213>人工序列<220><223>人工dna引物<400>345ctattcttacgatgaaggcg20<210>346<211>31<212>dna<213>人工序列<220><223>人工dna引物<400>346ccttaattaattatccacggaagatatgatg31<210>347<211>32<212>dna<213>人工序列<220><223>人工dna引物<400>347gctctagataaaatgtcccaaggtagaaaagc32<210>348<211>30<212>dna<213>人工序列<220><223>人工dna引物<400>348gctagctaaaatgtttggtaatatttccca30<210>349<211>28<212>dna<213>人工序列<220><223>人工dna引物<400>349ttaattaactatttatctaatgatcctc28<210>350<211>41<212>dna<213>人工序列<220><223>人工dna引物<400>350tctagataaaatgtctattagtgaaaaatattttcctcaag41<210>351<211>43<212>dna<213>人工序列<220><223>人工dna引物<400>351ttaattaacttttaaattttggaaaaagcttgatcaataatgg43当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1