通过细胞色素P450氧化酶突变的细胞核编码雄性不育的制作方法
本发明涉及通过分子生物学方法、标记技术和遗传工程简化费力的育种程序的领域。具体地,提供了植物,其显示通过核基因组中基因区域自发突变的纯合核编码雄性不育,区别在于所述突变通过隐性性状表达获得,与cms(细胞质雄性不育)相反,从而不需要在育种程序中获得不育和可育两种基因型。在这方面,本发明提供植物,特别是甜菜或马铃薯,其中导致上述性状表达的突变通过细胞色素p450氧化酶(cypgst)基因的标记技术和鉴定此突变的相应应方法检测。此外,提供cypgst蛋白、含导致上述性状表达的突变基因的dna分子、提供野生型基因的重组dna分子、用于在植物的花和或果实中特异性表达此基因或异源基因的启动子、和/或编码cypgst基因抑制剂的核苷酸序列,以及相应载体和宿主细胞。
此外,本发明涉及经遗传工程修饰的植物,其通过抑制cypgst基因表达而具有隐性的、核编码的雄性不育表型,涉及各抑制剂以及抑制所述基因的方法和育性恢复方法。本发明还涉及所述植物在杂交育种、抗性育种和/或种子生产中的用途。此外,本发明包括本发明所述植物的种子或后代、器官、植物部分、组织或细胞以及其用途。
发明背景
在甜菜(甜菜属(betavulgaris),普通亚种(subspeciesvulgaris))以及所有其它栽培植物中进行杂交以受控地产生遗传变异。携带花粉的花药从种子亲本仍闭合的花中手工移出。授粉随后也如下手动进行:将花粉从花粉供体施加到种子亲本的柱头。或者,授粉也能在移出种子亲本上的花药后,将花粉供体放置成空间上邻近种子亲本以开花和传送花粉获得。任何情况中,这涉及非常费力的操作,很容易出错,因为如果仅移出花中的部分花药,例如可发生自花授粉。
生成(商业)杂交种子目前常如下发生:在显示cms(细胞质雄性不育)的基因型中杂交育种种子亲本组分,然后回交以增加种子亲本组分的基因部分。所得雄性不育种子亲本组分可随后大规模种植,授粉通过(顶交法)邻近生长的花粉供体发生。由于cms显性基因遗传自雌性,使用cms系需要可育保持系(因而不是cms中包埋的o型)必须同时保持可用,其确保cms系授粉。这需要大量计划和生产努力,以及复杂的组织工作。例如,商业甜菜目前作为三交种杂交,以生成具有足够高品质的种子。在育种程序中生成杂交体同样昂贵和费力,且目前通过安装隔离壁来实施。
在显示天然产生核编码雄性不育(ms:雄性不育)的多种植物类型中有细胞系或基因型的表达。这通常由核基因组中的基因自发突变导致,此突变通过隐性性状表达获得。使用带核编码雄性不育表型的基因型适合简化育种工艺和/或使用这些工艺来产生杂交种子。雄性不育表型因而具有以下优势:花药不必手工移出以用于杂交育种,可育和不育基因型不必同时获得,因为杂合基因型在各生殖周期中通过自花受精而依次分成可育和不育个体。
因此,本发明的目的是提供在作物植物,尤其是甜菜和马铃薯中使用核编码雄性不育的手段和方法。此目的根据本发明通过权利要求书和说明书阐述的实施方式实现。
发明简述
本发明涉及简化费力的育种程序、标记技术和遗传工程的领域。本发明产生通过含细胞色素p450氧化酶(cypgst)dna区段中的突变而显示核编码的、隐性的、雄性不育表型的植物。用标记技术和分子生物学方法鉴定cypgst基因和突变。由于所述突变在隐性形状表达中保持完整,杂合基因型随后在各生殖周期中通过自花受精分成可育和不育基因型,不需要同时获得可育保持系。另外,所述发现能用于产生具有核编码的、隐性的、雄性不育表型的转基因植物,和用于恢复育性。
因此,本发明涉及段落[1]-[36]下所列和描述于实施例的实施方式。
[1]一种显示隐性的、核编码的雄性不育表型的植物,尤其是作物植物,其特征在于,所述表型涉及内源细胞色素p450氧化酶(cypgst)基因所含突变,或涉及相较对应(雄性可育)野生型植物,由野生型cypgst基因编码的功能性cypgst蛋白的缺失或较低含量或较低活性,特征在于,未突变cypgst基因是a)来自甜菜的基因bvcypgst或其同源物、类似物或直系同源物,优选所述基因包括seqidno.1或2中的核苷酸序列,或编码seqidno.3中的氨基酸序列,b)来自马铃薯(solanumruberosum)的基因stcypgst或其同源物、类似物或直系同源物,优选所述基因包括seqidno.12或13中的核苷酸序列,或编码seqidno.14中的氨基酸序列,或c)来自玉米(zeamays)的基因zmcypgst或其同源物、类似物或直系同源物,优选所述基因包括seqidno.9或10中的核苷酸序列,或编码seqidno.11中的氨基酸序列。
[2]如段落[1]所述的植物,其对所述突变而言是杂合的且是雄性不育的,或对所述突变而言是纯合且是雄性可育的,其中在不育植物中功能性花粉的形成受抑制,优选完全抑制。
[3]如段落[1]或[2]所述的植物,其特征在于,所述cypgst基因至少在闭合的花和果实中表达。
[4]如段落[1]-[3]中任一项所述的植物,其特征在于,所述突变防止功能性蛋白转录和/或翻译,优选其中所述突变涉及在cypgst基因的编码核苷酸序列中,在cypgst基因的剪接信号或调节序列,优选启动子序列中的缺失、添加、插入或取代。在一个优选实施方式中,所述核酸分子具有突变,其可以相较seqidno.1中的野生型基因,在seqidno.8的核苷酸序列中发现。具体地,突变可以是seqidno.1的核苷酸位置1560与2095之间或者seqidno.12或9对应位置之间的缺失。缺失能包括至少20、30或50个连续碱基对,优选至少100、150、200或250个连续碱基对,且特别优选至少300、400或500个连续碱基对。一个特别优选的实施方式中,所述核酸分子包括根据seqidno.8的核苷酸序列。在另一个优选实施方式中,所述核酸分子具有表1所述seqidno.1核苷酸序列中的点突变,优选在seqidno.1核苷酸位置1560与2095之间。
[5]如段落[4]所述的植物,其中在甜菜优选甜菜普通亚种中,所述缺失能通过标记基因座sle5983d14(用seqidno.4和5的引物扩增产物)和sle5983d17(用seqidno.6和7的引物扩增产物)之一或两者的不存在,以及通过泛在标记物的存在检测到。
[6]如段落[1]-[5]中任一项所述的植物,其中在甜菜,优选甜菜普通亚种中,所述基因位于位于染色体1上标记基因座sxn2151s01与sle3305s02之间的区段,其中seqidno.24所示sxn2151s01标记序列和seqidno.26所示sle3305s02标记序列显示存在gst基因座,seqidno.25所示sxn2151s01标记序列和seqidno.27所示sle3305s02标记序列显示参照序列。
[7]如段落[6]所述的植物,其特征在于,所述序列为约50-5,000kbp,100-1,000kbp,更优选100-500kbp,特别优选200-250kbp。
[8]如段落[1]-[7]中任一项所述的植物,其特征在于,所述未突变基因是来自甜菜,优选甜菜普通亚种的功能性基因bvcypgst或另一栽培植物或作物植物的功能性同源、类似或直系同源基因。
[9]如段落[8]所述的植物,其特征在于,所述同源、类似或直系同源基因是玉米基因,其优选包括seqidno.9或10中的核苷酸序列或编码seqidno.11中的氨基酸序列,马铃薯(solanumtuberosum)基因,其优选包括seqidno.12或13中所示核苷酸序列或编码seqidno.14中的氨基酸序列,小麦(triticumaestivum)基因,其优选编码seqidno.15中的氨基酸序列,向日葵(helianthusannuus)基因,其优选编码seqidno.16中的氨基酸序列,大麦(hordeumvulgare)基因,其优选编码seqidno.17中的氨基酸序列,甘蓝型油菜(brassicanapus)基因,其优选编码seqidno.18中的氨基酸序列,甘蓝(brassicaoleracea)基因,其优选编码seqidno.19中的氨基酸序列,芜菁(brassicarapa)基因,其优选编码seqidno.20中的氨基酸序列,大豆(glycinemax)基因,其优选编码seqidno.21中的氨基酸序列,棉属(gossypium)基因,其优选编码seqidno.22中的氨基酸序列,或双色高粱(sorghumbicolor)基因,其优选编码seqidno.23中的氨基酸序列。
[10]如段落[1]-[9]中任一项所述的植物,其特征在于,所述未突变基因(野生型基因)具有选自下组的核苷酸序列:
(a)具有seqidno.1或seqidno.2中核苷酸序列或其功能性片段的核苷酸序列(参见例如图4a和4b);
(b)编码seqidno.3中氨基酸序列的核苷酸序列;
(c)能于严格条件下与互补于(a)或(b)所述核苷酸序列的核苷酸序列杂交的核苷酸序列;
(d)编码氨基酸序列的核苷酸序列,所述氨基酸序列与根据seqidno.3的氨基酸序列有差异,所述差异为氨基酸序列中的氨基酸缺失、取代、添加和/或插入的形式,且优选与完整氨基酸序列的至少60%相同;
(e)核苷酸序列,其编码与(a)-(d)核苷酸序列中任一项所述核苷酸序列编码的蛋白具有相同酶促活性的蛋白;和
(f)核苷酸序列,包括来自seqidno.1核酸序列的核苷酸位置1-1518,优选核苷酸位置518-1518,特别优选核苷酸位置1318-1518的启动子或在此区域杂交的序列的至少200或400,优选至少600或800,特别优选至少1,000个连续核苷酸,其中所述核苷酸序列能控制所述基因或与所述核苷酸序列可操作连接的异源核酸分子在闭合的花或果实中的特异性表达。
根据(c)、(d)或(e)的核苷酸序列是这样的核苷酸序列,例如其显示seqidno.12或seqidno.13中的核苷酸序列或其功能性片段,或编码seqidno.14中的氨基酸序列。此外,根据(c)、(d)或(e)的核苷酸序列是这样的核苷酸序列,例如其显示seqidno.9或seqidno.10中的核苷酸序列或其功能性片段,或编码seqidno.11中的氨基酸序列。此外,根据(c)、(d)或(e)的核苷酸序列是这样的核苷酸序列,例如其编码选自seqidno.15-23的氨基酸序列。
[11]如段落[1]-[10]中任一项所述的植物,其特征在于,所述植物是近交植物或杂交植物。
[12]如段落[1]-[11]中任一项所述的植物,其特征在于,所述植物来自玉蜀黍属(zea)、茄属(solanum)、小麦属(triticum)、黑小麦属(triticale)、向日葵属(helianthus)、黑麦属(secale)、大麦属(hordeum)、芸苔属(brassica)、短柄草属(brachypodium)、大豆属(glycine)、棉属(gossypium)、高粱属(sorghum)、甘蔗属(saccharum)、腹腔丝虫属(setaria)、山羊草属(aegilops)、稻属(oryza)、胡萝卜属(daucus)、桉属(eucalyptus)、erythranthe、螺旋狸藻属(genlisea)、芭蕉属(musa)、燕麦属(avena)、烟草属(nicotiana)、咖啡属(coffea)、葡萄属(vitis)、黄瓜属(cucumis)、桑属(morus)、须弥芥属(crucihimalaya)、碎米荠属(cardamine)、独行菜属(lepidium)、荠属(capsella)、无苞芥属(olimarabidopsis)、南芥属(arabis)、萝卜属(raphanus)、芝麻菜属(eruca)、柑橘属(citrus)、麻风树属(jatropha)、科杨属(populus)、或恭属(beta),优选玉米、马铃薯、小麦(triticumaestivum)、硬粒小麦(triticumdurum)、斯佩耳特小麦(triticumspelta)、向日葵(helianthusannuus)、黑麦(secalecereal)、大麦(hordeumvulgare)、球茎大麦(hordeumbulbosum)、甘蓝型油菜(brassicanapus)、甘蓝(brassicaoleracea)、芜菁(brassicarapa)、芥菜(brassicajuncacea)、黑芥(brassicanigra)、大豆(glycinemax)、棉属(gossypiumsp.)、双色高粱(sorghumbicolor)、黑小麦(triticale)、甘蔗(saccharumofficinarum)、小米(setariaitalica)、水稻(oryzasativa)、小粒野生稻(otyzaminuta)、澳洲野生稻(otyzaaustraliensis)、高秆野生稻(oryzaalta)、二穗短柄草(brachypodiumdistachyon)、滨海大麦(hordeummarinum)、山羊草(aegilopstauschii)、胡萝卜(daucusglochidiatus)、小胡萝卜(daucuspusillus)、daucusmuricatus、野胡萝卜(daucuscarota)、巨桉(eucalyptusgrandis)、erythrantheguttate、黄花螺旋狸藻(genliseaaurea)、芭蕉属(musasp.)、燕麦属(avenasp.)、美花烟草(nicotianasylvestris)、普通烟草(nicotianatabacum)、绒毛状烟草(nicotianatomentosiformis)、番茄(solanumlycopersicum)、中果咖啡(coffeacanephora)、酿酒葡萄(vitisvinifera)、黄瓜(cucumissativus)、川桑(morusnotabilis)、须弥芥(crucihimalayahimalaica)、卵叶须弥芥(crucihimalayawallichii)、弯曲碎米荠(cardamineflexuosa)、北美独行菜(lepidiumvirginicum)、荠菜(capsellabursa-pastoris)、小拟南芥(olimarabidopsispumila)、硬毛南芥(arabishirsute)、萝卜(raphanussativus)、芝麻菜属(erucavesicariasativa)、橙(citrusxsinensis)、麻风树(jatrophacurcas)、毛果杨(populustrichocarpa)或甜菜类型的植物。
[13]一种含段落[10]所定义核苷酸序列的核酸分子或重组dna分子。
[14]如段落[13]所述的重组dna分子,其(i)包括具有段落[10](f)所定义的核苷酸序列的启动子,其与异源核酸分子可操作连接,或(ii)包括段落[10](a)-(e)所定义的编码核苷酸序列,其与异源核启动子可操作连接,所述异源启动子优选能控制核苷酸序列在闭合的花或果实中特异性表达。
[15]一种含核苷酸序列的重组dna分子,所述核苷酸序列编码shrna(小发夹rna)、sirna(小干扰rna)、反义rna、有义rna或双链rna,其在植物细胞中表达后或在引入植物细胞后,导致抑制功能性(未突变)cypgst基因的表达。在一个优选实施方式中,所述核苷酸序列具有seqidno.1、2、9、10、12或13有义或反义方向的,或来自seqidno.1核苷酸位置1762-2679的至少一个外显子1和seqidno.1核苷酸位置3507-4142的外显子2的至少15、16、17、18、19或20个,优选至少21、22、23、24或25个,特别优选至少30、35、40、45或50个,极其特别优选至少100、200、300、500或1,000个连续核苷酸。seqidno.12的外显子1从核苷酸位置1762-2032延伸,seqidno.12的外显子2从核苷酸位置2449延伸至2161,seqidno.12的外显子3从核苷酸位置4032延伸至4694。seqidno.9的外显子1从核苷酸位置2001延伸至2927,seqidno.9的外显子2从核苷酸位置3018延伸至3683。在另一个优选实施方式中,所述核苷酸序列具有至少15、16、17、18、19或20个,优选至少21、22、23、24或25个,特别优选至少30、35、40、45或50个,极其特别优选至少100、200、300、500或1,000个连续核苷酸,其能特异性杂交在段落[10]所定义核苷酸序列上。
[16]一种含段落[10]所定义核苷酸序列的核酸分子,具有缺失、添加、插入或取代形式的突变,其中所述突变导致没有功能性cypgst蛋白合成。该突变优选在cypgst基因编码核苷酸序列中,位于cypgst基因的剪接信号或调节序列中,优选在cypgst基因启动子中。在一个优选实施方式中,所述核酸具有也能在根据seqidno.8的核苷酸序列中发现的突变。具体地,突变可以是seqidno.1核苷酸位置1560与2095之间或者seqidno.12或9对应位置之间的缺失。缺失能具有至少20、30或50个连续碱基对,特别优选至少100、150、200或250个连续碱基对,且尤其优选至少300、400或500个连续碱基对的长度。在一个特别优选的实施方式中,所述核酸分子包括根据seqidno.8的核苷酸序列。在另一个优选实施方式中,所述核酸分子在seqidno.1核苷酸序列中,优选在seqidno.1核苷酸位置1560与2095之间具有表1所述的点突变。
[17]一种核酸分子,具有在seqidno.1、2、9、10、12或13的有义和/或反义方向,或者在seqidno.1、9或12的至少一个外显子的有义和/或反义方向的至少15、16、17、18、19或20个,优选至少21、22、23、24或25个,特别优选至少30、35、40、45或50个,极其特别优选至少100、200、300、500或1,000个连续核苷酸。seqidno.1的外显子1从核苷酸位置1762延伸至2679,seqidno.1的外显子2从核苷酸位置3507延伸至4142。seqidno.12的外显子1从核苷酸位置1762延伸至2032,seqidno.12的外显子2从核苷酸位置2449延伸至3161,seqidno.12的外显子3从核苷酸位置4032延伸至4694。seqidno.9的外显子1从核苷酸位置2001延伸至2927,seqidno.9的外显子2从核苷酸位置3018延伸至3683。在一个特定实施方式中,所述核酸分子延伸跨越seqidno.1、seqidno.9或seqidno.12的至少一个内含子,即该核酸分子依次包括i)来自seqidno.1外显子1的3’末端的至少一个核苷酸(优选seqidno.1外显子1的5’-3’方向的最后一个核苷酸;对应于seqidno.2中583位的核苷酸)和来自seqidno.1外显子2的5’末端的至少一个核苷酸(优选seqidno.1外显子2的5’-3’方向的第一个核苷酸;对应于seqidno.2中584位的核苷酸),ii)来自seqidno.12外显子1的3’末端的至少一个核苷酸(优选seqidno.12外显子1的5’-3’方向的最后一个核苷酸;对应于seqidno.13中271位的核苷酸)和来自seqidno.12外显子2的5’末端的至少一个核苷酸(优选seqidno.12外显子2的5’-3’方向的第一个核苷酸;对应于seqidno.13中272位的核苷酸),iii)来自seqidno.12外显子2的3’末端的至少一个核苷酸(优选seqidno.12外显子2的5’-3’方向的最后一个核苷酸;对应于seqidno.13中984位的核苷酸)和seqidno.12外显子3的5’-3’方向的至少一个核苷酸;对应于seqidno.13中985位的核苷酸),或iv)来自seqidno.9外显子1的3’末端的至少一个核苷酸(优选seqidno.9外显子1的5’-3’方向的最后一个核苷酸;对应于seqidno.10中927位的核苷酸)和来自seqidno.9外显子2的5’末端的至少一个核苷酸(优选seqidno.9外显子2的5’-3’方向的第一个核苷酸;对应于seqidno.10中928位的核苷酸)。在另一个优选实施方式中,所述核苷酸序列具有至少15、16、17、18、19或20个,优选至少21、22、23、24或25个,特别优选至少30、35、40、45或50个,极其特别优选至少100、200、300、500或1,000个连续核苷酸,其能特异性杂交在段落[10]或[16]所定义核苷酸序列上。
[18]一种寡核苷酸,优选具有不超过50个核苷酸的长度,其包含段落[17]所述核酸分子或能特异性杂交在根据seqidno.8的核苷酸序列上的核酸分子,和/或优选具有以下核苷酸序列之一:
(i)seqidno.4、6或其互补物,或
(ii)seqidno.5、7或其互补物。
[19]一种载体,优选植物载体,其包含段落[13]-[16]中任一项所述dna分子或核酸分子,或段落[17]所述核酸分子。
[20]如段落[19]所述的载体,其中所述转基因形式的dna分子或核酸分子能表达功能性cypgst,且优选与另一转基因遗传连锁,所述另一转基因防止所述dna分子或核酸分子经花粉转移,优选其中所述载体或所述转基因也具有表达盒,所述表达盒标记种子,优选用荧光标记。
[21]一种宿主细胞,优选植物细胞,其包含段落[13]-[16]中任一项所述重组dna分子或核酸分子或者段落[19]或[20]所述载体。
[22]一种由段落[10]所定义核苷酸序列编码的cypgst蛋白,或者其功能性和/或免疫活性片段。所述cypgst蛋白优选是a)选自seqidno.3、seqidno.11和seqidno.14-23的氨基酸序列,或b)氨基酸序列,其与根据seqidno.3的氨基酸序列至少80%、82%、84%、86%或88%,优选至少90%、91%、92%、93%、94%或95%,特别优选至少96%、97%、98%、99%或99.5%相同,优选在其完整长度上。
[23]一种特异性结合段落[22]所述cypgst蛋白或片段的抗体。
[24]一种试剂盒,包括段落[13]-[16]中任一项所述dna分子或核酸分子、段落[17]所述核酸分子、段落[18]所述寡核苷酸、段落[19]或[20]所述载体、段落[22]所述cypgst蛋白或其片段和/或段落[23]所述抗体,以及潜在的用于基于核酸检测方法或免疫检测方法的试剂。
[25]一种用于产生植物尤其是作物植物的方法,所述植物显示隐性的、核编码的、纯合雄性不育表型,其特征在于,cypgst基因表达受抑制。
[26]如段落[25]所述的方法,其特征在于,所述方法包括引入段落[15]所述重组dna分子、段落[16]或[17]所述核酸分子、或者段落[19]或[20]所述载体的步骤,例如通过农杆菌转化、t-dna标签、同源重组、诱变如tilling和定向诱变,诸如通过使用锌指核酸酶、tale(转录激活因子样效应物)核酸酶、crispr/cas系统,导致所述基因表达的抑制,如通过rnai或共抑制或归因于所引入的突变。
[27]一种使段落[1]-[12]中任一项所述植物或者能通过段落[25]或[26]所述方法获得的植物的育性恢复的方法,包括向所述植物引入功能性cypgst基因。
[28]如段落[27]所述的方法,其中所述cypgst基因通过段落[13]或[14]所述重组dna,或段落[19]或[20]所述载体引入,或通过与携带cypgst野生型基因或功能性cypgst基因,优选处于纯合状态的植物杂交。可选地,可以在后代中针对cypgst野生型基因或功能性cypgst基因的存在进行选择。
[29]一种植物,包含段落[21]所述植物细胞和/或能经段落[25]-[28]中任一项所述方法获得。
[30]一种如段落[1]-[12]或[19]中任一项所述植物的器官、植物部分、组织或细胞。
[31]如段落[1]-[12]或[29]中任一项所述植物的种子或后代,其中所述种子或后代具有段落[1]-[12]中任一项所定义突变和/或段落[13]-[16]中任一项所述重组dna分子或核酸分子或者段落[19]或[20]所述载体。
[32]一种鉴定段落[1]-[12]或[29]中任一项所述植物的方法,其通过检测cypgst基因中的突变或与所述突变连锁的标记。
[33]如段落[13]-[16]中任一项所述dna分子或核酸分子、段落[17]所述核酸分子、段落[18]所述寡核苷酸、段落[19]或[20]所述载体、段落[22]所述cypgst蛋白或其片段、段落[23]所述抗体和/或段落[24]所述用于产生段落[1]-[12]或[29]中任一项所述植物的试剂盒,在产生隐性的、核编码的雄性不育植物,在产生具有恢复育性的植物,在产生杂交植物、在抗性程序或用于种子生产中的用途。
[34]如段落[14]所述dna分子或段落[14]所定义启动子在植物花和/或果实内特异性表达异源核酸分子中的用途。
[35]如段落[1]-[12]或[29]中任一项所述植物,段落[30]所述器官、植物部分、组织或细胞、段落[31]所述种子或后代,或能根据段落[32]所述方法鉴定或通过段落[33]或[34]所述用途获得的植物,或者组织、细胞、后代或种子,在生成食物、活性物质、医药产品或其前体、诊断产品、化妆品、精细化学品、糖、糖浆、生物乙醇或生物气体中的用途。
[36]食物、饲料或活性物质,其包含于段落[1]-[12]或[29]中任一项所述植物,段落[30]所述器官、植物部分、组织或细胞,段落[31]所述种子或后代,或根据段落[32]所述方法鉴定或可以通过段落[33]或[34]所述用途获得的植物,或者其组织、细胞、后代或种子中。
[37]如段落[1]-[12]或[29]中任一项所述植物在育种或产生后代植物中的用途,其特征在于,所述核编码雄性不育表型用于轮回选择。
首先,用于本申请的一些术语应更详细解释:
表述“染色体区段”和该术语的变化形式如“染色体的区段”等同使用,除非另有说明,且指特定染色体的特定染色体dna区段,含有至少一个基因。
编码“cypgst蛋白”的“cypgst基因”或“来自cypgst的野生型基因”在重要的花粉形成中发挥作用,因为cypgst突变通过遏制功能性花粉形成来产生雄性不育植物。这在甜菜(甜菜普通亚种)中经实验证实。与来自拟南芥(arabidopsisthaliana)cyp703基因的相似性可以通过同源性比较建立,其满足根据现有技术的在孢子花粉素合成中的基本功能(morant等,theplantcell,19(2007),1473-1487),并优选催化中链饱和脂肪酸转变成对应简单羟化脂肪酸,优选羟化月桂酸的c-7位。在拟南芥中去除cyp703基因导致部分雄性不育。花粉量实际减少,但仍能生成功能性花粉。出于此原因,来自甜菜普通亚种的基因看来承担另一功能,因为去除其导致植物雄性不育。没有形成理论,以下似乎可信:cypgst基因应被分配到cyp基因不同的类别,或在栽培植物尤其是作物植物如甜菜中,具有不同于低等模型植物拟南芥的功能或意义;参见实施例1的讨论。本领域技术人员能从数据库得到其它cypgst蛋白,使用合适的搜索配置文件和计算机程序以筛选同源序列或序列比较。对所鉴定基因是否实现与甜菜中cypgst基因相同功能的可能评估,可如下评价:通过所鉴定基因的异源表达在雄性不育甜菜植物中恢复cypgst功能,即通过转基因恢复育性。
没有受约束于特定理论,也有可能是来自甜菜普通亚种的cypgst基因和来自拟南芥的cyp703基因属于同一cyp家族,并因而在孢子花粉素合成中实现相同或至少类似功能,但在针对产量、抗虫性、非生物胁迫因素耐受性等以及植物物质含量方面的多年靶向选择和杂交育种中已经被优化的栽培植物中,补偿孢子花粉素缺乏的能力丧失,因而缺失孢子花粉素导致花粉形成受抑制,导致植物雄性不育。
根据本发明,术语“gst基因座”指植物尤其是作物植物中的基因组dna,其中突变与隐性遗传的、核编码的雄性不育相关,其中所述突变包括细胞色素p450氧化酶(cypgst)基因并导致突变包含于以此方式受影响的植物中的gst基因座,且尤其是带有纯合植物的突变,功能性cypgst蛋白的含量或活性相较含野生型基因座(野生型植物)的对应(雄性可育)植物更低,或完全缺失。通常,功能性cypgst蛋白的转录和/或翻译被cypgst基因种的突变阻止。
术语“紧密侧翼”应理解为指2个基因座(如2个标记(标记基因座))在基因图上彼此分开小于15cm、小于12cm、小于10cm、小于8cm、小于7cm、小于6cm、小于5cm、小于4cm、小于3cm、1小于2cm、小于1cm、小于0.5cm、小于0.2cm、1小于0.1cm、小于0.05cm。
术语“使杂交”或“杂交”指这样过程,其中单链核酸分子以最大可能程度附于互补核酸链,即碱基对与之相互作用。杂交的标准方法通过举例方式描述于sambrook等,《分子克隆:实验室手册(molecularcloning;alaboratorymanual)》第3版,纽约冷泉港的冷泉港实验室出版社(coldspringharborlaboratorypress),2001。因而应优选理解,核酸分子碱基的至少60%,更优选至少65%、70%、75%、80%或85%,特别优选90%、91%、92%、93%、94%、95%、96%、97%、98%或99%以最大可能程度参与和互补核酸链的碱基配对。这种附着的可能性取决于杂交条件严格性。术语“严格性”涉及杂交条件。当碱基配对受阻时,存在高严格性,当促进碱基配对时,存在低严格性。杂交条件的严格性取决于盐浓度或离子强度,例如,以及温度。一般,通过提高温度和/或降低盐含量能增加严格性。“严格的杂交条件”指杂交主要仅在同源核酸分子与同源基因之间发生的那些条件。术语“杂交条件”不仅涉及在核酸实际附着期间的当时情况,而且涉及后续生长步骤期间普遍的条件。例如,严格的杂交条件是在所述条件下,主要仅至少70%,优选至少75%、至少80%、至少85%、至少90%或至少95%相同的那些核酸分子杂交。例如,严格的杂交条件是:在65℃于4xssc中杂交,随后在65℃于0.1xssc中多次洗涤,总共约1小时。本文所用术语“严格的杂交条件”也能指:在65℃于0.25m磷酸钠、ph7.2、7%sds、1mmedta和1%bsa中杂交16小时,随后在68℃用2xssc和0.1%sds洗两次。杂交优选在严格条件下发生。
提及双链dna形式的核酸时,“互补”核苷酸序列指在涉及对应第一链碱基的核苷酸碱基配对调节方面,第一dna链与第二dna链互补。
术语“(分子)标记”是用作参照或定向点的核苷酸序列。用于检测重组的标记应适合监测植物群体内的差异或多态性。对于标记,这些差异在dna水平出现,且包括多核苷酸序列差异,如ssr(简单重复序列)、rflp(限制性片段长度多态性)、flp(片段长度多态性)或snp(单核苷酸多态性)。标记能获自基因组或表达的核酸,如剪接rna、cdna或est,也可涉及用作探针或引物对的核酸,且因此适合使用基于pcr的方法扩增序列片段。与群体部分之间遗传多态性相关的标记能通过现有技术中已确定的方法来检测(遗传分析导论(anintroductiontogeneticanalysis),第7版,griffiths,miller,suzuki等,2000)。这些包括,例如:dna测序,基于pcr的序列特异性扩增,检测rflp,通过等位基因特异杂交(ash)方式检测多核苷酸多态性,检测ssr、snp或rflp。此外,也已知检测est(表达序列标签)和rapd(随机扩增多态性dna)的方法。根据上下文,术语“标记”也能指物种基因组中的特定染色体位置,在该处可定位特异性标记(如snp)。本发明中标记也用于检测缺失。
术语“作物植物”涵盖未经栽培的植物以及栽培植物。植物指以任何形式由人直接或间接使用的作物植物,例如作为食物、刺激物或药物以及木材供应或作为牲畜饲料。
与野生植物相反,“栽培植物”是由人种植、栽培和保护的植物,其能用作作物植物或观赏植物。栽培植物存在的遗传基础包括点突变、体细胞突变、染色体突变和多倍体化。这些突变提供选择基础。其形成自然或人工延伸(突变率增加、杂交育种、秋水仙碱处理、基因工程方法)的原材料以用于人控制的进化。栽培植物包括食用植物、工业植物(如纤维植物)、饲用植物和观赏植物。这些栽培植物的重要特征是植物尤其是所用器官的尺寸增加,失去苦味物质,抗虫性和/或营养物含量更高。
“可操作连接”指在共享核酸分子中互相连接,从而互相连接的元件彼此相对放置和定向,所用方式允许发生核酸分子转录。与启动子可操作连接的dna受到此启动子的转录控制。
本发明所示“植物”可以是任何双子叶植物或单子叶植物植物物种。优选农业、园艺用或用于产生生物能(生物乙醇、生物气等)的植物。用于本发明的植物优选通过其储存器官区分,包括块茎、根、种子、谷粒、果实等。例如,这些包括玉米、马铃薯、小麦、硬粒小麦、斯佩耳特小麦、向日葵、黑麦、大麦、球茎大麦、甘蓝型油菜、甘蓝、芜菁、芥菜、黑芥、大豆、棉属、高粱、黑小麦、甘蔗、小米、水稻、小粒野生稻、澳洲野生稻、高秆野生稻、二穗短柄草、滨海大麦、山羊草、胡萝卜、小胡萝卜、daucusmuricatus、野胡萝卜、巨桉、erythrantheguttate、黄花螺旋狸藻、芭蕉属、燕麦属、美花烟草、普通烟草、绒毛状烟草、番茄、中果咖啡、酿酒葡萄、黄瓜、川桑、须弥芥、卵叶须弥芥、弯曲碎米荠、北美独行菜、荠菜、小拟南芥、硬毛南芥、萝卜、芝麻菜属、橙、麻风树、毛果杨或甜菜。本发明所述植物优选是甜菜属植物,尤其是甜菜(betavulgaris)物种以及亚种,甜菜普通亚种。
植物“器官”例如指叶、植物茎、树干、根、营养芽、分生组织、芽、花药、卵、种子或果实,尤其是谷粒。术语“植物部分”包括但不限于植物茎、或茎秆、叶、花、花序、根、果实和种子以及花粉。植物“部分”也指许多器官的组合,如花或种子,或器官的部分,例如植物茎的横截面。植物“组织”是例如愈伤组织、贮藏组织、分生组织、叶组织、芽组织、根组织、植物中心组织或生殖组织,以及分生组织、基底组织(所谓的薄壁组织)、维管组织、实变组织和覆盖组织(所谓的上皮)。组织不限于这里所列的那些。植物“细胞”应理解为例如有细胞壁的分离的细胞,或其聚集物或原生质体。
在本发明的上下文中,术语“调节序列”指影响特异性和/或表达强度的核苷酸序列,例如,调节序列赋予特定组织特异性。这种调节序列可以位于最小启动子转录起始点的上游,但是也能位于其下游,例如在转录而不翻译的前导序列中或内含子中。
“启动子”是不翻译的dna区段,通常在含rna聚合酶结合位点的编码区上游,并起始dna转录。启动子也包含用作基因表达调节因子的其它元件(如顺式调节元件)。“核或最小启动子”是包含起始转录所需至少基本元件(如tata盒和/或起始子)的启动子。
“转基因植物”是这样的植物,其中整合至少一种多核苷酸,优选异源多核苷酸。多核苷酸优选以稳定方式整合,即整合的多核苷酸在植物中保持稳定,被表达且也能以稳定方式被后代继承。在植物基因组中稳定引入多核苷酸也包括其整合入来自之前世代的植物基因组,其中多核苷酸能稳定传到之后世代。术语“异源”指引入的多核苷酸来自例如同一物种或一些其它物种中遗传背景不同的细胞或生物体,或与原核或真核宿主细胞同源,但随后位于不同遗传环境中,并因而不同于可能天然产生的对应多核苷酸。除了对应内源性基因,也能存在异源多核苷酸。
本发明的设计和实施方式应通过举例方式参考附图和序列如下描述:
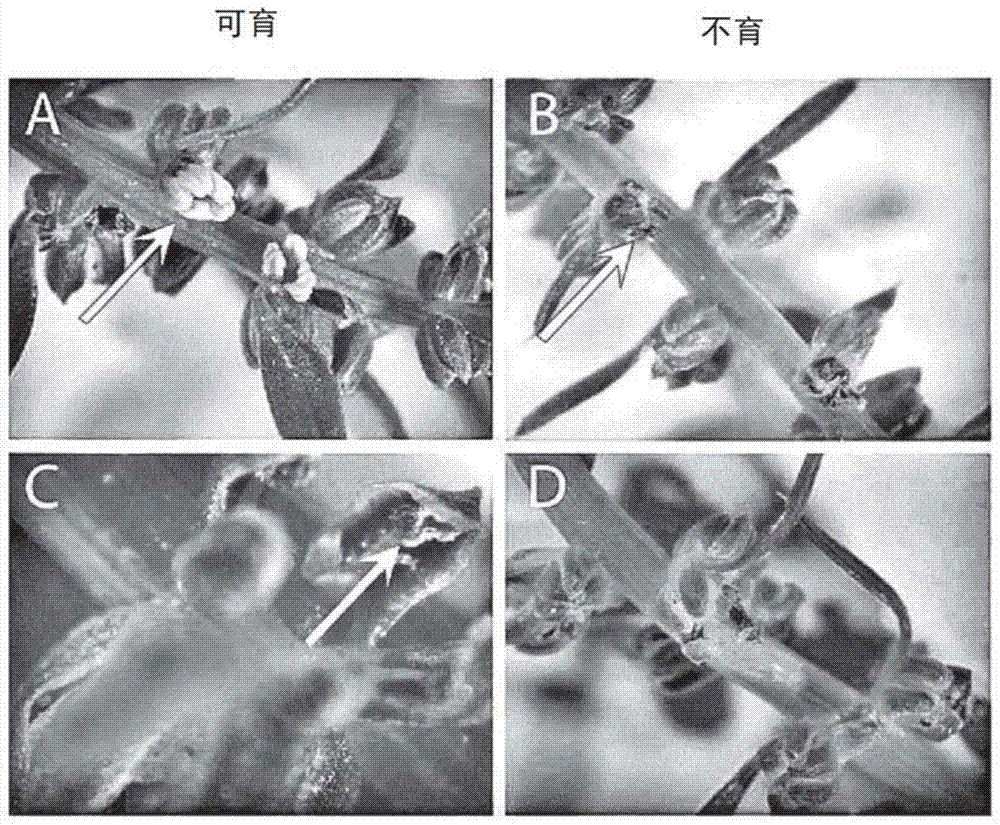
图1:a、c)来自可育甜菜(甜菜普通亚种)的花和b、d)来自雄性不育甜菜的花,其表型能追溯到供体c311[2043_k5]。a,b)是闭合的花,其萼片和花瓣被手工移出。可育基因型(a)的浅色(黄色)、重要花药清晰可见。相反,不育基因型的花药明显为深色(棕色)。可育基因型的花药在开花期中打开并释放花粉(c),而不育基因型的花药不继续发育且不含花粉。
图2:参照基因型kws2320中以refbeet1.2,类型bvcypgst(g6845.tl)注释的基因模型。长度为517个氨基酸的蛋白由总长为1554bp的外显子编码。雄性不育表型中表达的基因型显示缺失533bp,该部分包括5’utr和所述基因的第一个外显子。因此,mrna的正确转录和功能性蛋白翻译是不可能的。
图3:比对4721bp基因组dna片段,其编码bvcypgst(g6845.tl)的甜菜基因模型,来自不育和可育基因型。不育基因型的序列具有533bp缺失。
图4:编码甜菜基因bvcypgst(g6845.tl)的4721bp基因组dna片段的序列分析,来自不育和可育基因型。a)显示甜菜普通亚种中cypgst基因的基因组dna序列,包括假定的启动子区以及5’utr和3’utr。假定的启动子区以“粗体”表示,5’utr和3’utr带下划线,外显子1以“粗体”和下划线表示,外显子2以斜体和下划线表示,内含子以斜体表示。此序列对应于seqidno.1的序列。基因的功能区位置如下:假定的启动子1..518;5’utr1519..1761;转录区1519..4275;外显子1762..2679;内含子2680..3506;外显子3507..4142;3’utr4143..4275。b)显示甜菜普通亚种中cypgst基因的cdna序列,包括5’utr和3’utr。5’utr和3’utr带下划线,外显子1以“粗体”和下划线表示,外显子2以斜体和下划线表示。。此序列对应于seqidno.2的序列。cdna的功能区位置如下:5’utr1..243;外显子244..1161;外显子1162..1787;3’utr1798..1930。c)显示甜菜普通亚种中cypgst基因的氨基酸序列。此序列对应于seqidno.3的序列。d)显示甜菜普通亚种中突变cypgst基因的基因组dna序列,包括假定的启动子区和3’utr。假定的启动子区以“粗体”表示,3’utr带下划线,截断的外显子1以“粗体”和下划线表示,外显子2以斜体和下划线表示,内含子以斜体表示。此序列对应于seqidno.8的序列。突变cypgst基因的功能区位置如下:假定的启动子1..1353;转录区1354..3542;截断的外显子1354..1938;内含子1939..2755;外显子2756..3394;3’utr3395..3542。
图5:基因bvcypgst(gst,g6845.tl)通过qrt-pcr方式的表达分析。rna获自可育植物的多个组织且gst基因表达相较基因g4645.tl表达来描述,其中n.d.指未检测到表达。在本文所示实验中,gst在闭合的花内表达最强。相比之下,在不育基因型的闭合花中未能检测到gst基因表达。
发明详述
本发明通过核基因组的dna区段突变来生成植物,所述区段包括显示核编码雄性不育表型的细胞色素p450氧化酶(cypgst)基因。其与众不同在于突变通过隐性性状表达获得,植物因而能用于简化费力的育种程序。负责此性状表达的基因鉴定在甜菜(甜菜普通亚种)中进行,如实施例1和2以及图1-5所解释。相关基因由于序列分析确定的结构特征而被归类为细胞色素p450氧化酶(cyp),并提供有后缀“gst”,代表其就核遗传雄性不育观察到的表型。由于在甜菜中鉴定该基因,当具体引用实施例所述基因时,也使用前缀“bv”。
一般,本发明涉及植物,尤其是展示出隐性、核编码雄性不育表型的栽培植物或作物植物,其特征在于所述表型与内源性细胞色素p450氧化酶(cypgst)基因所含突变相关,或与功能性cypgst蛋白相较对应(雄性可育)野生型植物的缺失或更低含量或更低活性相关,所述蛋白由cypgst野生型基因编码,其特征在于未突变cypgst基因是甜菜的基因bvcypgst或其同源物、类似物或直系同源物,所述基因包括seqidno.1或2中的核苷酸序列之一,或编码seqidno.3中的氨基酸序列。如上所述和实施例所解释,其它cypgst蛋白或其编码基因即同源物、类似物或直系同源物,能通过经典生物信息学方法(数据库搜索和计算机程序以筛选同源序列)在植物中鉴定,其中可假定引发同一表型的突变,如在甜菜中已经观察到的。因此,本发明的植物特征也在于,未突变cypgst基因是马铃薯中的基因stcypgst或其同源物、类似物或直系同源物,优选所述基因包括seqidno.12或13中的核苷酸序列,或编码seqidno.14中的氨基酸序列;或未突变cypgst基因是玉米中的基因zmcypgst或其同源物、类似物或直系同源物,优选所述基因包括seqidno.9或10中的核苷酸序列,或编码seqidno.11中的氨基酸序列。
本文的术语“同源物”指相关基因(来自2种不同植物类型)基本具有相同功能和共同祖先且其核酸或编码的氨基酸序列基本相同。然而,有许多基因,它们彼此同源,而没有引起有用配对对齐的蛋白序列。相反,术语“类似物”基因或蛋白(同样)表明其具有相同或类似功能,但非获自同一结构,即其没有共同祖先。这种情况下,其核酸或编码的氨基酸序列通常不明显相同,或最多,仅在某些功能域中相同。
在用于注释的基因组测序背景下,“同源物”更明确定义。其中包括术语“直系同源物”和“旁系同源物”。直系同源物是经物种形成而结合的基因。旁系同源物是经复制而结合的基因。
当能互补雄性不育表型时,基因在根本上是本发明所示的同源物、类似物或直系同源物,因为其在甜菜的参考基因cypgst中出现,和/或在相关基因中引发靶向突变,或者从中获得该基因的同源物或类似物所编码植物内出现雄性不育表型基因产物的生物活性变化。因此,实施例所述本发明cypgst基因的相关同源物或类似物优选特征在于,其能互补甜菜cypgst突变体中观察到的雄性不育表型,即其能恢复可育表型。另外或替代地,cypgst同源物或类似物优选特征在于,雄性不育表型通过抑制其表达或者同源物或类似物所编码基因产物生物活性来获得。雄性不育表型优选具有就甜菜中cypgst突变体举例描述的性质,尤其是实施例所述的那些性质;参见上述实施方式。
本领域技术人员通过现有技术已知用于互补遗传学的对应技术和方法;参见例如napoli等,plantphysiology120(1999),615-622,其描述也具有雄性不育表型的矮牵牛花近交系的突变,所述表型能通过用功能性查耳酮合酶acdna转基因互补来消除,从而能确定查耳酮合酶a实质上负责雄性不育表型,或雄性不育表型由此基因的突变导致。
在jeong等,j.exp.bot.65(2014),6693-6709中,所谓ms1035番茄突变体中的雄性不育通过互补和多个候选基因的转基因表达来补充和消除。通过抑制靶基因(本申请中为cypgst)在转基因植物中生成雄性不育的方法为本领域技术人员已知;参见例如国际专利申请wo1996/017945和以下实施方式。
因此,在本发明的一个实施方式中,展示出隐性、核编码、雄性不育表型的植物特征在于,所述表型由含内源性细胞色素p450氧化酶(cypgst)基因的突变导致。然而,本发明所述植物也能如下鉴定:通过相较对应雄性可育野生型植物,由来自cypgst野生型基因编码的功能性cypgst蛋白的缺失或低含量或低活性。不能再翻译的突变基因的基因组序列如seqidno.8所示,但仅意在作为示例,而不在此方面限制本发明。具体地,本发明涉及属于栽培植物和作物植物的植物。
现有技术描述了在拟南芥中灭活cyp703基因(cyp703a2)以获得减少的花粉形成并因而引起部分雄性不育(morant等,theplantcell,19(2007),1473-1487)。这能归因于以下事实:孢子花粉素缺失或结构改变,孢子花粉素形成花粉已暴露层的主要组分。尽管cypgst基因承担其它功能看似合理,因为突变导致核编码、隐性、雄性不育且花粉未形成,但cypgst基因和来自拟南芥的cyp703基因属于同一基因家族并因而在孢子花粉素合成中实现相同或至少相似功能的可能性不应被摒弃。不约束于特定理论,栽培植物中丧失补偿孢子花粉素缺乏的能力是看似合理的,所述植物已经在涉及产率、抗虫性、耐受非生物胁迫因子以及植物物质含量方面的多年靶向选择和杂交育种中优化,且缺失孢子花粉素导致花粉形成受抑制,从而植物成为雄性不育看似合理。
由于morant等.(2007)解释过cyp703a2基因或对应的拟南芥敲除系仅显示部分雄性不育且这种表型不适合杂交,从本发明的一个实施方式中排除拟南芥的cyp703a2基因或morant等.所述突变体,尤其是具有图1所示序列的那些。因此,在本发明植物的一个优选实施方式中,优选排除拟南芥类型的栽培植物和/或作物植物。
植物在真核形式的各细胞中具有2个或更多拷贝的其遗传信息。各基因通常由2个等位基因代表,其可在纯合状态下相同或在杂合状态下不同。本发明所述植物表型由核基因组突变导致,这通过隐性性状表达获得。因此,在本发明的一个实施方式中,当突变是杂合时,植物是雄性可育,当突变是纯合时,植物是雄性不育。
功能性花粉在不育植物中的形成受抑制,优选完全抑制,其中结合本发明,术语“抑制”指在就cypgst基因突变而言为纯合且雄性不育的植物中,95%,优选96%,更优选97%,特别优选98%且尤其优选99%的花粉形成不发生,而“完全抑制”指超过99%,优选100%的花粉形成受抑制。此背景下,“抑制”优选指在这种植物用作雄性亲本和对应野生型植物的杂交实验中,基本不发生种子生成和/或不产生后代。
在甜菜普通亚种的情况中,这在图1中明显。可育基因型(a)的浅色(黄色)重要花药在闭合的花中可见,所述花的萼片和花瓣被手工移出。相反,不育基因型的花药明显为深色(棕色)(b)。当开花时,可育基因型的花药打开并释放花粉(c),而不育基因型的花药无法成熟,且不释放花粉(d)。
在拟南芥中,cyp703蛋白催化中链饱和脂肪酸转变成对应简单羟化脂肪酸,优选羟化c7位的月桂酸。不致力于特定理论,cypgst蛋白不实现与拟南芥中cyp703蛋白相同的功能但功能相似是看似合理的,因为灭活2种基因都对花粉形成有影响。因此,在孢子花粉素合成中的功能可归因于cypgst,孢子花粉素是暴露的重要花粉层的主要组分。
在一个实施方式中,所述cypgst蛋白因而在孢子花粉素合成中发挥作用,并催化中链饱和脂肪酸转变成形成对应简单羟化脂肪酸,优选c7位羟化的月桂酸。
甜菜普通亚种可育基因型的转录分析(实施例2)显示cypgst基因在闭合的花和果实中表达,根和叶中没有检测到表达(图5)。因此,本发明所述植物的一个实施方式是上述植物,其中cypgst基因至少在闭合的花和果实中表达,优选特异性在闭合的花和果实中表达。
在一个实施方式中,所述突变防止功能性蛋白在本发明所述植物中转录和/或翻译,其中突变优选是以下中的缺失、添加、插入或取代:cypgst基因编码核苷酸序列,cypgst基因的剪接信号或调节序列,优选启动子序列。
在一个优选实施方式中,所述缺失是至少500-600bp的缺失,涉及cypgst基因的编码区或启动子区。所述缺失的长度为至少20、30或50个连续碱基对,至少100、150、200或250个连续碱基对,或优选至少300、400或500个连续碱基对。所述添加优选是在基因组序列中插入一个或多个核苷酸,优选在编码基因序列中引起读框移码。所述取代优选是基因组序列中的点突变,优选在编码序列中,所述突变产生终止密码子或剪接错误。
通过包括来自雄性不育及雄性可育甜菜普通亚种植物的cypgst基因和假定启动子区的基因组dna片段的比较测序,证明缺失533bp引起雄性不育表型(参见实施例2和图3)且缺失位于seqidno.1核苷酸位置1560-2095。
在另一个优选实施方式中,这是533bp的缺失,包括甜菜普通亚种中cypgst基因的部分5’utr和第一外显子;参见图3。功能性基因bvcypgst包括2个外显子,总长度为1554bp。refbeet1.2中注释的基因模型如图2所示,seqidno.8给出cypgst的基因组dna序列,带有产生截断外显子1的缺失。可导致甜菜普通亚种转录提前中断或能导致剪接破坏的可能点突变如表1所列,其中这些优选位于seqidno.1的核苷酸位置1560-2095。
如本发明的实施例1所示,cypgst基因中紧密侧翼的标记能通过精细定位来鉴定,因此提供cypgst基因在甜菜普通亚种基因组中的位置。这进而提供开发雄性标记的基础,用其能检测cypgst基因中的缺失。
因此,在本发明的一个实施方式中,所述植物特征在于甜菜(甜菜普通亚种)中的缺失能通过缺失标记基因座sle5983d14(用seqidno.4和5的引物扩增产物)和sle5983d17(用seqidno.6和7的引物扩增产物)之一或两者,以及通过存在泛在标记物来检测。泛在标记物确认dna提取具有令人满意的质量。
此外,在另一个实施方式中,所述来自甜菜普通亚种的基因位于染色体1上标记基因座sxn2151s01与sle3305s02之间的区段。在一个优选实施方式中,这些标记基因座在染色体1上的33.42或35.15cm(基于遗传图谱zrint1202)且基于物理基因组图谱(physmapv2),此区域具有215.4kbp的物理尺寸,位于位置3185718bp-3401120bp。开发了kasp标记(kasptm,snp基因分型化学品,来自lgc有限公司(lgclimited)),用其能鉴定待检测的snp或对应参照序列。示于seqidno.24的sxn2151s01标记序列和示于seqidno.26的sle3305s02标记序列表明存在gst基因座;示于seqidro.25的sxn2151s01标记序列和示于seqidno.27的sle3305s02标记序列指示参照序列,其中标记序列各在核苷酸位置21有差异,在携带gst基因座的基因型的此位置处有“g”,在参照基因型kws2320的此位置处有“a”。
在一个优选实施方式中,所述区段是约50-5,000kbp,优选100-1,000kbp,更优选100-500kbp,特别优选200-250kbp。其中该区段具有其它蛋白编码基因,优选21个基因。
在本发明的一个实施方式中,所述未突变基因是来自甜菜,优选甜菜普通亚种的功能性基因bvcypgst,或另一作物植物或栽培植物的功能性同源、类似或直系同源基因。
本领域技术人员能从相关文献以及数据库获得其它cypgst蛋白,使用合适的搜索配置文件和计算机程序以筛选同源序列或序列比较。此外,本领域技术人员能用常规分子生物学技术发现其它cypgst蛋白编码dna序列,并在本发明框架内使用其。例如,合适的杂交探针能因而衍生自cypgst基因序列,并用于就所需生物体筛选基因组和/或cdna数据库。本领域技术人员能引用常规杂交、克隆和测学方法,如sambrook等,《分子克隆:实验室手册》第3版,纽约冷泉港的冷泉港实验室出版社,2001所列。本领域技术人员也能合成和使用寡核苷酸引物以基于已知序列扩增cypgst序列。
具体地,优选实施方式中的同源、类似或直系同源基因是来自玉米的基因,优选包括seqidno.9或10所示核苷酸序列,或编码来自马铃薯的seqidno.11所示氨基酸序列,其优选包括seqidno.12或13中的核苷酸序列之一,或编码seqidno.14中的氨基酸序列,来自小麦,其优选编码seqidno.15中的氨基酸序列,来自向日葵,其优选编码seqidno.16中的氨基酸序列,来自大麦,其优选编码seqidno.17中的氨基酸序列,来自甘蓝型油菜,其优选编码seqidno.18中的氨基酸序列,来自甘蓝,其优选编码seqidno.19中的氨基酸序列,来自芜菁,其优选编码seqidno.20中的氨基酸序列,来自大豆,其优选编码seqidno.21中的氨基酸序列,来自棉属,其优选编码seqidno.22的氨基酸序列,且来自高粱,其优选编码seqidno.23中的氨基酸序列。指定植物可归类为作物植物,且优选为栽培植物。
本发明所述植物的一个实施方式是上述植物,其中未突变基因(野生型基因)具有选自下组的核苷酸序列:seqidno.1、2、9、10、12和13所示核苷酸序列含有的核苷酸序列。
在一个实施方式中,所述未突变基因(野生型基因)具有编码seqidno.3、11或14所示氨基酸序列的核苷酸序列。
核苷酸序列能用现有技术已知的常规方法引入基因,如通过定点诱变、pcr诱导的诱变、转座子诱变、基因组编辑等,单独或组合的取代、缺失、插入、添加和/或任何其它修饰,其改变核苷酸序列但仍实现与起始序列相同的功能。
此外,本发明也包括上述植物,其中所述核苷酸序列也能包括seqidno.1、2、9、10、12和13的核苷酸序列的功能性片段。术语“片段”包括与上述核苷酸序列足够相似的核苷酸序列。术语“足够相似”指第一核苷酸序列或氨基酸序列,其在涉及第二核苷酸或第二氨基酸序列方面具有足够或最少数量的相同或等同核苷酸或氨基酸残基。关于氨基酸序列,这些在用一种上述方法修饰后还具有一个共同结构域,和/或具有共同功能活性。至少约45%,至少约50%,至少约55%,至少约60%,至少约65%,至少约70%,至少约75%,至少约80%,至少约85%,至少约90%,至少约95%,至少约96%,至少约97%,至少约98%,至少约99%或至少约100%相同的核苷酸序列或氨基酸序列在这里定义为足够相似。当核苷酸序列或氨基酸序列具有与本发明上述核苷酸或氨基酸序列相同的一般特性时,足够相似性优选在功能性片段中建立。
因此,在一个实施方式中,植物所含未突变基因(野生型基因)具有的核苷酸序列能在严格条件下与某一核苷酸序列杂交,后者与seqidno.1、2、9、10、12或13的核苷酸序列或者编码seqidno.3、11或14氨基酸序列的核苷酸序列互补。此外,另一个实施方式包括未突变基因(野生型基因),所含核苷酸序列编码氨基酸序列,相对seqidno.3、11或14氨基酸序列具有差异,所述差异为氨基酸序列中氨基酸缺失、取代、添加和/或插入的形式,优选在完整氨基酸序列不超过20%、15%、10%、5%、4%、3%、2%或1%上差异。
在另一个或额外实施方式中,所述未突变基因(野生型基因)的核苷酸序列编码蛋白,该蛋白具有与前面实施方式的dna所编码蛋白相同的酶活性。
在另一个实施方式中,所述植物所含未突变基因(野生型基因)具有dna,其包括来自seqidno.1核苷酸序列从核苷酸位置1-1518,优选从核苷酸位置518-1518,特别优选从核苷酸位置1318-1518的启动子,或在此区域杂交的序列的至少200或400,优选至少600或800,特别优选至少1,000个连续核苷酸,其中所述核苷酸序列能控制与所述dna可操作连接的基因或异源核酸分子特异性在闭合的花和/或果实中表达。
在一个实施方式中,所述植物可以是近交植物或杂交植物。近交植物能用作生成杂交体的亲本植物。使用就性状而言为隐性、核编码雄性不育、杂合近交植物的优势是其在各生殖步骤中分成可育和不育个体。雄性不育个体能用于生成杂交体,其中没有人工移出花药,且不再需要仍维持不育保持系。
在一个实施方式中,本发明所述植物是玉蜀黍属、茄属、小麦属、黑小麦属、向日葵属、黑麦属、大麦属、芸苔属、短柄草属、大豆属、棉属、高粱属、甘蔗属、腹腔丝虫属、山羊草属、稻属、胡萝卜属、桉属、erythranthe、螺旋狸藻属、芭蕉属、燕麦属、烟草属、咖啡属、葡萄属、黄瓜属、桑属、须弥芥属、碎米荠属、独行菜属、荠属、无苞芥属、南芥属、萝卜属、芝麻菜属、柑橘属、麻风树属、科杨属、或恭属的植物,优选玉米、马铃薯、小麦、硬粒小麦、斯佩耳特小麦、向日葵、黑麦、大麦、球茎大麦、甘蓝型油菜、甘蓝、芜菁、芥菜、黑芥、大豆、棉属、高粱、黑小麦、甘蔗、小米、水稻、小粒野生稻、澳洲野生稻、高秆野生稻、二穗短柄草、滨海大麦、山羊草、胡萝卜、小胡萝卜、daucusmuricatus、野胡萝卜、巨桉、erythrantheguttate、黄花螺旋狸藻、芭蕉属、燕麦属、美花烟草、普通烟草、绒毛状烟草、番茄、中果咖啡、酿酒葡萄、黄瓜、川桑、须弥芥、卵叶须弥芥、弯曲碎米荠、北美独行菜、荠菜、小拟南芥、硬毛南芥、萝卜、芝麻菜属、橙、麻风树、毛果杨或甜菜类型的植物。这些植物属于作物植物,尤其是栽培植物。
本发明的另一个实施方式不仅包括具有cypgst基因突变的本发明所述植物,而且包括具有上述核苷酸序列的dna分子,其具有缺失、添加、插入或取代形式的突变,其中此突变导致没有功能性cypgst蛋白合成。
在一个优选实施方式中,所述cypgst基因编码核苷酸序列中的突变是cypgst基因的剪接信号或调节序列,优选位于cypgst基因启动子中。突变可以是seqidno.1核苷酸位置1560与2095之间或者seqidno.12或9中对应位置之间的缺失。缺失的程度可为至少20、30或50个连续碱基对,优选至少100、150、200或250个连续碱基对,且特别优选至少300、400或500个连续碱基对。在一个特别优选的实施方式中,所述核酸分子包括根据seqidno.8的核苷酸序列。在另一个优选实施方式中,所述核酸分子具有表1所述的在seqidno.1核苷酸序列中的点突变,优选在seqidno.1核苷酸位置1560与2095之间。
如上所解释,获自cypgst基因序列的dna杂交探针能用于筛选其它生物体的基因组和/或cdna数据库,以鉴定同源基因。为获得特异性杂交,这种探针应是特异性的,且具有至少15个核苷酸,优选至少20个核苷酸的长度。探针能用于通过已知聚合酶链式反应(pcr)方法来扩增所鉴定同源基因。此外,这些探针也能用于检测cypgst基因中的突变。用于核酸杂交的综合说明可参见tiissen,《生物化学与分子生物学的实验室技术-与核酸探针杂交(laboratorytechniquesinbiochemistryandmolecularbiology-hybridizationwithnucleicacidprobes)》,第1部分,第2章,“杂交原理和核酸探针测试策略综述(overviewofprinciplesofhybridizationandthestrategyofnucleicacidprobeassays,)”纽约爱思唯尔(elsevier)(1993);和《最新分子生物学实验指南(currentprotocolsinmolecularbiology)》,第2章,ausubel等编,纽约的格林出版公司和威力出版公司(greenepublishingandwileyinterscience)(1995)。
因此,本发明的主题是具有至少15、16、17、18、19或20个核苷酸,优选至少21、22、23、24或25个,特别优选至少30、35、40、45或50个且极其特别优选至少100、200、300、500或1,000个核苷酸的核酸分子,其中此核酸分子特异性杂交在上述包括未突变cyp703野生型基因的核苷酸序列,或特异性杂交在上述dna分子上,所述dna分子具有缺失、添加、插入或取代形式的突变,所述突变导致没有功能性cyp703蛋白形成。核酸分子优选具有段落[17]所述实施方式。
cypgst基因在甜菜普通亚种基因组中的位置能用上述精细定位确定。这进而提供开发遗传标记的基础,用其能检测cypgst基因中的缺失。
因此,除了上述植物外,本发明也涉及寡核苷酸形式的标记,尤其是引物寡核苷酸。这些包括具有至少15个核苷酸的核酸分子,其特异性杂交在如上所定义核苷酸序列或如上所定义dna分子上,所述dna分子具有缺失、添加、插入或取代形式的突变,所述突变导致没有功能性cyp703蛋白形成。这些寡核苷酸优选具有不超过50个核苷酸的长度。更优选地,寡核苷酸甚至更短且具有15-25个核苷酸的长度。如本发明的实施例2所示,寡核苷酸优选具有一种以下核苷酸序列:(i)seqidno.4、6或其互补物,或(ii)seqidno.5、7或其互补物。
本发明的主题也包括能由上述核苷酸序列编码的cypgst蛋白和其功能性和/或免疫活性片段,以及特异性结合cypgst蛋白于本文所述其片段上的抗体。蛋白和片段的重组生成为本领域技术人员已知并通过举例方式描述于sambrook等,《分子克隆:实验室手册》第3版,纽约冷泉港的冷泉港实验室出版社,2001,或wingfield,p.t.2008,“生成重组蛋白(productionofrecombinantproteins)”,《最新蛋白质科学实验指南(currentprotocolsinproteinscience)》,52∶5.0:a5.0.1-5.0.5。本发明蛋白的多克隆或单克隆抗体能由本领域技术人员根据已知方法生成,如描述于e.harlow等,《抗体:实验室指南(antibodies:alaboratorymanual)》(1988)的那些方法。生成单克隆抗体以及fab和f(ab’)2片段可用多种常规方法完成,如描述于goding,《单克隆抗体:原理与实践(monoclonalantibodies:principlesandpractice)》,第98-118页,纽约;学术出版社(academicpress)(1983)的那些方法,所述片段也能用于蛋白检测方法。抗体随后能用于筛选表达cdna库以通过免疫筛选方式鉴定相同、同源或异源基因(sambrook等,《分子克隆:实验室手册》,纽约冷泉港的冷泉港实验室出版社,1989,或ausubel等,1994,“最新分子生物学实验指南”约翰威立国际出版公司(johnwiley&sons))。
在一个优选实施方式中,所述cypgst蛋白涉及seqidno.3、11或14-23的氨基酸序列,或至少80%、82%、84%、86%或88%,优选90%、91%、92%、93%、94%或95%,特别优选96%、97%、98%、99%或99.5%相同的氨基酸序列,优选在seqidno.3中的氨基酸序列的完整长度上。
本发明也涉及含未突变cypgst基因(野生型基因)的重组dna分子,且具有本发明所述植物含有的上述核苷酸序列的特性。重组dna分子优选具有启动子和/或其它转录或翻译控制元件,或之一相关联。所用启动子主要是细胞特异性启动子,其仅允许dna在预定细胞中转录。除了启动子,有许多其它转录控制元件,如增强子、操纵子、抑制子和转录终止信号,但不限于此,它们于dna功能连接以使靶向、细胞特异转录可行。启动子和其它转录调节元件一般为本领域技术人员已知且可用;参见例如wo00/75359,第23页第5行到第24页第17行。此重组dna分子能用于在具有核编码、隐性、雄性不育表型的植物中恢复可育性。
因为如上所解释,cypgst基因在闭合的花和果实中表达,而不是根或叶,在一个优选实施方式中,所述重组dna分子包括启动子或如上定义的编码核苷酸序列,所述启动子具有上述核苷酸序列且可操作连接异源核苷酸序列,所述编码核苷酸序列包括野生型cypgst基因且可操作连接异源启动子。此启动子优选能控制核苷酸序列特异性地在闭合的花和/或果实中表达。重组dna分子更优选包括来自甜菜普通亚种的未突变cypgst基因(seqidno.1)的天然启动子。
因此,本发明也包括使用本文所述dna分子或启动子以在植物花和/或果实中特异性表达异源核酸分子。这需要异源核酸分子可操作连接对应启动子,以及在靶细胞优选植物细胞中引入此重组dna分子。异源表达重组dna分子的方法应如下更详细解释。
本发明的另一主题包括带有本发明所述核苷酸序列的重组dna分子,其编码shrna(小发夹rna)、sirna(小干扰rna)、反义rna、正义rna或双链rna。这些赋予对cypgstmrna翻译的抑制或通过碱基配对分解细胞中的cypgstmrna。因此,在植物中引入和/或表达所述重组dna分子导致抑制功能性(未突变)cypgst基因表达。在一个优选实施方式中,所述核苷酸序列显示如段落[15]详细描述的特性。
本发明的另一主题包括载体,其含有本发明的重组dna分子或核酸序列或核酸分子。本发明所述载体能包含具有上述核苷酸序列特性的未突变cypgst基因(野生型基因),和优选上述启动子之一。另一载体能包括重组dna分子,其含有连接异源核酸分子的未突变cypgst基因,或能包括重组dna分子,其含有可操作连接异源启动子的上述核苷酸序列,其中在各情况中,启动子优选能在闭合的花和/或果实中特异性控制核苷酸序列表达。此外,载体能包含具有核苷酸序列的重组dna分子,所述序列编码shrna、sirna、反义rna、正义rna或双链rna,并因而导致在植物细胞表达后抑制cypgst基因表达。
此外,载体能包含带一种上述突变的重组dna分子,或其能包含上述核酸分子,所述核酸分子特异性结合未突变(野生型)cypgst核苷酸序列或突变的cypgst核苷酸序列。
所述载体可以是质粒、粘粒、噬菌体或表达载体、转化载体、穿梭载体或克隆载体,且其可以是双链或单链、线性或环形,或其能转化原核或真核宿主,通过整合入其基因组或在染色体外。本发明所述dna分子或核酸分子可操作连接表达载体中的一种或多种调节序列,这允许在原核或真核宿主细胞中转录和任选地表达;参见例如sambrook等,《分子克隆:实验室手册》第3版,纽约冷泉港的冷泉港实验室出版社,2001和国际专利申请wo00/75359,第21页第20行到第22页第32行。这些调节序列优选是启动子或终止子,尤其是转录起始点、核糖体结合位点、rrna加工信号、转录终止位点和/或多聚腺苷酸化信号。载体通常也包含标示/报告基因或抗性基因以检测所需载体或dna分子/核酸分子的转移,以及选择含有这些的个体,因为通过基因表达直接检测通常相当困难。标示/报告基因的示例是例如荧光素酶基因和编码绿色荧光蛋白(gfp)的基因。这些也允许测试启动子就基因而言的活性和/或调节。特异于植物转化的抗性基因示例是新霉素磷酸转移酶基因、潮霉素磷酸转移酶基因、或编码草胺膦乙酰转移酶的基因。这些不排除本领域技术人员已知的其它标示/报告基因或抗性基因。在一个优选实施方式中,所述载体是植物载体。
本发明的另外一个主题是上述载体,其中作为转基因的dna分子能表达功能性cypgst基因,且优选与另一转基因遗传连锁,这防止经花粉转移dna分子。在cypgst基因突变的雄性不育突变体中引入此载体,能恢复可育性。由于防止经花粉转移转基因功能性cypgst,用转基因系自花授粉仅获得半合种子。
载体或转基因分别优选也具有表达盒,其标记种子,优选用荧光标记。用此方法可容易区分转基因种子与非转基因种子。先锋(pioneer)公司开发了使用核编码、雄性不育的此系统。就玉米开发了名为种子生成技术(seedproductiontechnology(spt))(us2006288440a1)的系统,该系统基于以下事实:在已知核编码基因中有突变的不育突变体能通过插入转基因来恢复。转基因包含不育基因的未突变等位基因,从而转基因作为野生型等位基因发挥作用。恢复可育性的转基因遗传偶联另一转基因,这防止经花粉转移转基因(花粉杀手)。因此,通过转基因系自花授粉仅生成半合种子,这在系统效率中起主要作用。
转基因也包含用红色荧光标记种子的表达盒。因此,转基因种子能与非转基因种子容易区分。由于转基因种子可育,植物自动分成可育和不育植物。对生成杂交体必需的母株因而通过播种非转基因种子来获得,转基因种子能用作父系进一步繁殖母系(维持系),且也能通过简单自交来繁殖(图5)。spt系统理论上可应用于所有植物类型。这需要存在遗传上雄性不育系和了解负责遗传雄性不育表型的基因。因此,spt系统理论上可用于就各类栽培植物如甜菜或马铃薯开发杂交系统。
本发明也涉及宿主细胞,含有所述载体、重组dna分子和/或核酸分子。本发明所示宿主细胞可以是原核(如细菌)或真核细胞(如植物细胞或酵母细胞)。宿主细胞优选是农杆菌或植物细胞。本发明更优选涉及转基因植物细胞,所述细胞包括本发明所述核酸分子作为转基因或包含本发明的载体。这种转基因植物细胞是这样的植物细胞,例如优选以稳定方式用本发明所述核酸分子转化。在转基因植物细胞的一个优选实施方式中,所述核酸分子可操作连接一种或多种调节序列,这允许在植物细胞中转录和任选地表达。因此,含核酸分子和调节序列的总体结构代表转基因。例如,这种调节序列包括启动子或终止子。本领域技术人员了解许多能用于植物的功能性启动子和终止子。
鉴定负责隐性、核编码雄性不育性状表达的cypgst基因也可用于本发明以生成带有此性状表达的转基因植物以及因而恢复可育性。
在一个实施方式中,请求保护试剂盒,所述试剂盒分别包括上述重组dna分子、或核酸分子和载体,这些对生成核编码、隐性雄性不育的植物以及在具有此表型的植物中恢复可育性是必需的。此试剂盒也包含重组dna分子,包括带前面所定义核苷酸序列的启动子或异源启动子,其中前者连接异源核酸分子,而后者连接编码未突变(野生型)cypgst基因的前面所定义核苷酸序列。此外,试剂盒能包含载体,其中作为转基因的dna分子能表达功能性cypgst基因,且优选遗传偶联另一转基因,这防止经花粉转移dna分子,并具有标记种子的表达盒。试剂盒能包含前面所定义核酸分子以鉴定cypgst基因中的突变,所述分子杂交在含未突变(野生型)cypgst基因的前述核苷酸序列或有前面所定义突变的对应基因上,或其能包含上面所定义寡核苷酸。试剂盒也能包含上述cypgst基因或其片段以及上面的抗体。试剂盒也优选包含用于基于核酸或免疫检测方法的试剂。
转基因植物是例如包含植物细胞的植物,所述细胞用本发明所述dna分子/核酸分子或本发明载体转化。在转基因植物的一个优选实施方式中,所述dna分子/核酸分子可操作连接一种或多种调节序列,这允许在植物中转录和任选地表达。因此,含有本发明所述核酸分子和调节序列的总体结构代表转基因。由此,术语“转基因”指编码多肽的重组核酸序列。
上述寡核苷酸、核酸、dna分子和载体也能用于生成转基因植物。因此,本发明也涉及这些在生成展示隐性、核编码、纯合雄性不育表型的转基因植物中的应用,其特征在于,在生成本发明所述恢复可育性的植物中,或在生成转基因宿主细胞优选植物细胞中抑制cypgst基因表达。此外,上述寡核苷酸、核酸、dna分子和载体也能用于生成这些转基因植物或植物细胞的对应方法。转基因植物优选是作物植物,或更优选是栽培植物。
本领域有多种方法能生成和鉴定或选择转基因植物,其中蛋白转录/翻译受抑制或引入恢复特征。生成转基因栽培植物和通过分子生物学方法鉴定其的方法为本领域技术人员已知;参见例如,就转基因草甘膦抗性甜菜而言,是国际专利申请wo99/023232和wo2004/074492,或就一般植物转化而言,是wo2000/018939和wo2013/138309。
因此,本发明的一个实施方式是生成植物的方法,所述植物展示隐性、核编码、纯合雄性不育表型,其特征在于,cypgst基因表达受抑制,其中此植物优选是作物植物,更优选是栽培植物。通过转化方式向植物细胞引入表达多核苷酸的重组dna分子,如使用载体,从而所述多核苷酸表达导致抑制cypgst蛋白。
例如,导致抑制cypgst表达的上述突变能通过在植物间杂交过程中遗传重组来获得,其中一种植物携带突变的cypgst等位基因。
除了使用常规育种程序以产生遗传重组,现代生物技术为本领域技术人员提供使精确基因组工程可行的多种其它工具。例如,t-dna标签能用于通过插入诱变来破坏cypgst基因。此外,采用tale核酸酶(talen)或锌指核酸酶(zfn)和crispr/cas系统方式,cypgst基因可通过基因突变而完全或部分缺失,所述方式描述于wo2014/144155a1(用crispr/cas系统的工程植物基因组(engineeringplantgenomesusingcrispr/cassystems))和osakabe&osakabe,plantcellphysiol.,56(2015),389-400,从而防止cypgst基因表达。这也能用称为tilling(定向诱导基因组局部突变)的方法实现,其中例如德国专利申请de102013101617所述,在野生型基因中引起点突变,随后选择展示出合适即抗性提供基因的植物,如显示抗黄化花叶病毒的大麦;参见de102013101617,第4,8,12页,段落[0014],[0026]和[0038]。tilling法也由henikoff等(henikoff等,plantphysiol.,135,2004,630-636)在出版物中综合描述。甜菜普通亚种中cypgst基因的点突变如表1所列,其可能导致终止密码子或剪接错误。
抑制表达也能用rnai方法或共抑制进行。这些包括向植物引入如上所定义重组dna分子或核酸分子或者对应载体,其中表达所编码的shrna、反义rna或有义rna分子引起cypgst基因表达受抑制。这种基于rnai和/或共抑制的方法是抑制基因表达的典型方法且为本领域技术人员已知。含靶特异非多聚腺苷酸rna分子的有义方法能导致cypgst基因表达受抑制。此方法通过举例方式描述于国际专利申请wo2001/012824。
本发明的另一实施方式包括在本发明所述植物中恢复可育性的方法,所述方法包括向植物引入功能性cypgst基因。cypgst基因能用遗传工程方法引入;通过本发明所述重组dna,优选含有转录控制元件,优选用于在闭合的花和/或果实中特异性表达基因的启动子,或通过本发明所述载体。cypgst基因也能通过与植物杂交来引入,所述植物携带cypgst野生型基因或功能性cypgst基因,优选处于纯合状态。可选地,能在杂交后的后代中针对是否存在cypgst野生型基因或功能性cypgst基因进行选择。
本发明的主题是含有先前所定义植物细胞和/或通过上述方法获得的植物。这意味着带有核编码、隐性雄性不育表型的植物以及恢复可育性的植物通过基因重组用常规育种方法生成,或其是转基因植物,其中cypgst基因表达通过多种上述方法抑制,引起核编码、隐性、雄性不育表型,或通过引入重组dna分子来恢复可育性。此植物优选是栽培植物,特别优选作物植物,优选有储存器官,其可能也作为生殖器官发挥作用,如来自甜菜和马铃薯的甜菜根或块茎,来自黑小麦、燕麦、小米和玉米的谷粒,果实,例如番茄等。在一个实施方式中,低等植物和阿拉伯芥拟南芥从本发明中明确排除。
除了通过cypgst基因自发突变显示核编码、隐性、雄性不育表型的植物,或通过基因重组用常规育种方法加入此表型的植物,或通过遗传工程用现代生物技术加入此表型的植物,和通过相应方法恢复可育性的植物,本发明也涉及这些植物的器官、植物部分、组织、细胞和种子或后代。在一个实施方式中,所述种子或后代具有一个或多个如上所定义的突变,这导致cypgst基因表达受抑制和/或所述种子或后代显示上述重组dna分子或核酸分子或载体。
用于鉴定本发明所述植物的方法同样形成一个实施方式。通过cypgst基因自发突变显示核编码、隐性、雄性不育表型的植物以及通过基因重组用常规育种方法加入此表型的植物或通过遗传工程用现代生物技术加入此表型的植物,能用此方法鉴定。此外,通过对应方法恢复可育性的植物能用此方法鉴定。用作杂交探针的前面所定义核酸分子能用于鉴定本发明所述植物,所述核酸分子具有最小15个核苷酸的长度且特异性结合在前面所定义核苷酸序列上,该序列包括未突变(野生型)cypgst基因和带有先前所定义突变的cypgst基因,所述突变导致基因表达抑制。此外,如上所定义寡核苷酸、如上所定义cypgst蛋白或其片段、抗体以及上述试剂盒组分能用于鉴定。
本发明也涉及使用重组dna分子或核酸分子、本发明所述载体、本发明所述试剂盒的组分,用于生成隐性、核编码、雄性不育植物,用于生成恢复可育性的植物,用于在抗性育种程序中生成杂交植物,或用于种子生成。
一种在植物中产生可逆雄性不育的方法通过举例方式描述于国际专利申请wo96017945,其中可使用cypgst基因,所述方法包括:
(a)向产生花粉的植物基因组引入第一重组dna分子,所述植物能遗传转化,其中第一重组dna分子包括:
(i)核苷酸序列,取决于在植物中的表达,其编码抑制花粉形成或功能的基因产物,此情况中是本发明所述cypgst基因,或基因产物,如通过rnai序列表达;
(ii)操纵子,控制核苷酸序列表达;和
(iii)启动子,特异于对花粉形成或其功能关键的细胞,其中启动子功能连接至编码基因产物的核苷酸序列;
(b)任选地,在允许作为核苷酸序列表达结果而获得雄性不育的条件下,培育步骤(a)所得植物;
(c)使来自(a)或(b)的雄性不育植物与来自雄性可育系的花粉杂交育种以产生雄性可育的杂交植物,其中来自第二重组dna分子的花粉包括:编码dna结合蛋白并抑制转录的核苷酸序列,以及控制核苷酸序列表达的启动子,其中dna结合蛋白能结合雄性可育植物重组dna的操纵子并抑制转录。
用于生成花粉不育植物的另一系统描述于欧洲专利申请ep0344029,其中外来dna被引入核基因组并能根据本发明使用。
本发明也涉及本发明所述植物在育种或生成后代中的应用,其中核编码雄性不育表型用于轮回选择。此外,除了本发明所述植物,本发明也涉及种子或后代,或其器官、植物部分、组织或细胞,所述植物用于生成通常由可再生原料如食物和饲料构成的产物,优选糖或糖浆(糖蜜),其中糖蜜也用于工业应用,如发酵酒精,或在就化学工业生成原料或物质中作为生成生物技术产物的营养物,如精细化学品、药物或其成分、诊断产品、化妆品、生物乙醇或生物气。在生物气设施中使用甜菜作为生物发生原料的一个示例描述于专利申请de102012022178a1,参见例如第10段。
最后,本发明也涉及获自本发明所述植物、器官、植物部分、组织、细胞、种子和后代的产物,如食物、饲料以及植物、种子、后代、器官、植物部分、组织或细胞、或其组分所含原料。
下列实施例解释本发明,而不限制本发明的主题。除非另有说明,使用分子生物学标准方法,参见例如(sambrook等,《分子克隆:实验室手册》第3版,纽约冷泉港的冷泉港实验室出版社,2001),fritsch等,冷泉港实验室出版社;1989;mayer等,《细胞及分子生物学的免疫化学方法(immunochemicalmethodsincellandmolecularbiology)》,编.伦敦学术出版社,1987)和weir等,《实验免疫学手册(handbookofexperimentalimmunology)》,卷i-iv,布莱克威尔公司(blackwell)编,1986)。
实施例
1.鉴定导致核编码雄性不育的基因座
带内部标记c311[2043k5]的供体用作起始植物以鉴定在甜菜中导致核编码雄性不育的基因座,所述甜菜展示出隐性、核编码、雄性不育表型(工作名称gst)。然而,此供体中基本性状表达gst基因座的存在和合子程度不能在试验材料中事先(即开花前)检查。相反,必须使大量假定不育植物在实地或自交块(s-块)中到开花期。然后,分别手工检查开花植物的可育性或不育性(图1)。随后能移出可育个体,可从不育个体收获种子。
对于导致核编码雄性不育的gst基因座的遗传和物理限制,建立用于此性状的分开甜菜作图群。从全基因组图谱的初始先验信息已知染色体1上的靶区域。gst供体c311[2043k5]的雄性不育个体与一年生系杂交且所得f1个体通过自花受精繁殖。s1代的后代随后进行表型分型以作图,用kasp-dna标记物(kasptm基因分型化学物,来自lgc有限公司)标记。开发了2种kasp-dna标记物sxn2151s01和sle3305s02,通过其能经由鉴定snp来区分携带gst-基因座的基因型与参照基因型kws2320。seqidno.24的sxn215ls01标记序列和seqidno.26的sle3305s02标记序列表明存在gst基因座;seqidno.25的sxn2151s01标记序列和seqidno.27的sle3305s02标记序列指示参照序列,其中标记序列各在核苷酸位置21处有差异,在携带gst-基因座的基因型中此位置处有“g”,而参照组基因型kws2320中此位置处有“a”。作为此精细定位的结果,清楚分离甜菜基因组染色体1上的区域,其侧翼为33.42cm处的kasp-dna标记sxn2151s01和35.15cm处的sle3305s02(基于遗传图谱zrint1202)并携带gst基因座。基于物理基因组图谱(physmapv2),此区域具有215.4kbp的物理尺寸,位于3185718bp-3401120bp位。根据所鉴定位置和公开可得的基因组注释refbeet1.2(http://bvseq.molgene.mpg.de/),鉴定21个编码蛋白的基因,所述基因位于此基因组区段内。
就21个基因模型检测模型植物(如拟南芥和水稻)中的同源基因,使用生物信息学方法。在模型植物的所鉴定同源基因基础上,完成综合分析及评价。基于序列和预测结构的所述综合分析,gst基因座中编码的基因被鉴定为细胞色素p450单加氧酶(cyp)家族成员。尽管细胞色素p450单加氧酶有显著序列多样性,所有cyp具有在活性中心区高度保护的共同结构特征(参见例如fischer等,bioinformatics23(2007),2015-2017),且就假定gst基因也发现其。出于此原因,该基因称为cypgst。
更具体鉴定此基因时,鉴定拟南芥cyp基因,即cyp703a2,其显示与来自gst基因座的此基因的高序列一致性。拟南芥突变体展示出部分或半雄性不育表型,其中此基因通过插入t-dna来灭活(morant等,plantcell19(2007),1473-1487)。这能通过cyp703a2在孢子花粉素合成中的功能在机械基础上解释,孢子花粉素是重要花粉外层的主要组分。缺失外层会中断花粉成熟,或使得其易受环境影响。然而,在甜菜表型的gst表型与所述拟南芥突变体表型之间有2种显著差异:
(i)与拟南芥相反,甜菜中的基因敲除导致完全雄性不育,和
(ii)尽管不育花粉基本在拟南芥突变体中产生,然而,根据当前分析状态,gst甜菜中没有形成花粉。
因此,以下可能性不应被摒弃:这些是不同的cyp家族成员和/或2种蛋白在拟南芥及甜菜中的功能不同。另外,事实是拟南芥为来自十字花科植物家族的野生草本植物,具有紧凑的小基因组,而甜菜是栽培植物,即植物由人培育、耕种和杂交,用作作物植物。因此,拟南芥实验和其结果不能简单应用于栽培植物。此外,还有采用作物植物的重要农业过程从未在拟南芥中发生。例如,这些包括形成用作储存器官及营养生殖器官的甜菜根、块茎和谷粒,与不关联拟南芥的共生菌根真菌或病原体相互作用。
2.鉴定cypgst基因。
鉴定引起gst表型的潜在基因后,进行基因组dna片段的比较测序,所述片段含约5kbp。对以下测序:可育甜菜参照基因型kws2320、不育gst供体c311以及上述精细定位群中根据表型和标记数据归类为不育的3个个体,根据表型和标记数据归类为纯合可育的3个个体。测序基因组区域包括图2所示bvcypgst的基因模型(gst,g6845.t1),以及约1.5kbp的假定启动子区。比较测序揭示,除了不育与可育个体之间的一系列小核苷酸多态性(snp),不育基因型中的533bp缺失包括基因模型的5’utr和第一外显子(图2和图3)。
分析所有已鉴定多态性显示,一些缺失对编码蛋白有影响,而所有其它突变在非翻译区或产生同义密码子。相反,这些缺失防止mrna具体转录,功能性蛋白翻译因而不可能。后续转录分析确认这些发现(图5)。bvcyp703a2(gst,g6845.t1)在可育基因型的闭合的花和果实中极特异表达。在根和叶中无法检测到其表达。相反,gst基因表达无法在不育基因型的闭合的花中测得,从而可得出结论,不育基因型中的基因完全灭活。
最后,开发了能区分不育与可育基因型的dna标记物。为此开发了kasp标记物,其展示出可育等位基因(插入)作为主要性状(sle5983d14,sle5983d17)。
因此,关于本纯合缺失,可间接得出结论,当2种主要标记(sle5983d14,sle4983d17)显示缺失等位基因时,第三、泛在标记证实dna提取的品质足够好。下表1汇编了引起cypgst基因转录提前中断,或能导致剪接破坏的bvcypgst基因潜在点突变,其可用检测dna点突变的常规方法(snp分析)测试。
表1:能导致cypgst基因转录提前中断或能导致剪接破坏的甜菜普通亚种中的cypgst基因潜在点突变。
3.cypgst基因或基因座在杂交育种中的应用
如上所解释,产生gst基因座的雄性不育表型用于抗性育种计划以在轮回选择框架中简单杂交。在本发明框架内克隆基因和相关基因组标记开发前,必须比所需培养和收获4倍的植物,这归因于预期的3∶1表型分离。必须在开花后不久评估这些植物的不育性,可育个体必须移出以防止自花授粉。如果每年培养成千上万株植物,此人工选择极其费力且容易出错。现在提供本发明所述基因组标记,参见实施例2和图2,用其能例如测试30,000株植物,并选择随后种植的75,000株植物。
持续努力简化甜菜的育种计划和种子生产,并从而降低成本。如此,商业甜菜目前可生产为三杂交种以生成品质足够好的种子。在育种计划和因而的非商业领域中生产杂交种同样昂贵且费力,且目前通过安装隔离壁来实现。现在能用本发明所述gst表型和相关dna标记-在育种计划中插入gst基因座后或cypgst基因突变/抑制后-在用dna标记种植前选择雄性不育植物并因而简化生产工艺。平行开发multi-germ测试种基因型(mus测试种)同样是多余的。从长远来看,同样可以想象用替代系统取代当前cms技术,其中可使种子亲本组分雄性不育,如通过上面指定且如图5所示的spt系统。因此,也可合理推定本发明所述cypgst系统能用于其它栽培植物,尤其是作物植物,如在商业生产双杂交种如玉米的情况中。开发生产杂交种子的替代系统时,ms-基因在玉米(玉蜀黍)中发挥主要作用。序列分析显示存在假定的玉米同源物用于bvcypgst(grmzm5g830329)。同样在玉米中有大量ms突变体,迄今其中仅一部分被克隆。ms突变体目前能通过本发明方式分离,ms突变可归因于bvcypgst玉米同源物的突变或抑制,并能用于靶向方式的种子生产。本发明可同样用于开发杂交土豆,例如用于向bvcypgst基因的土豆同源物引入靶突变和在土豆中使用以此方式获得的雄性不育以开发spt系统所示二倍体杂交土豆。
最后,bvcypgst基因在花和绒毡层中的特异性表达使启动子的生物技术应用可行,如用于表达正义/反义rna或核酶以抑制bvcypgst基因或者表达功能性cypgst蛋白或bvcypgst基因的假定同源物、类似物或直系同源物以互补突变和恢复雄性不育表型。假定除了从中衍生的基因标记和实施方式,提供bvcypgst基因的基因座以及核酸序列和氨基酸序列,与就育种计划而言的大幅简化和成本节约相关,因为其中,不育个体的早期选择可行。这同样使得相关计划的物流简化和育种扩张。
序列表
<110>kws种子欧洲股份公司
<120>通过细胞色素p450氧化酶突变的细胞核编码雄性不育
<130>kws0231pct
<150>de102016106656.7
<151>2016-04-12
<160>27
<170>patentinversion3.5
<210>1
<211>4328
<212>dna
<213>betavulgaris
<220>
<221>promotor
<222>(1)..(1518)
<223>putativerpromotor
<220>
<221>gen
<222>(1519)..(4275)
<220>
<221>5'utr
<222>(1519)..(1761)
<220>
<221>exon
<222>(1762)..(2679)
<223>exon1
<220>
<221>intron
<222>(2680)..(3506)
<220>
<221>exon
<222>(3507)..(4142)
<223>exon2
<220>
<221>3'utr
<222>(4143)..(4275)
<400>1
ttattaaacctgattggaacttattgaaccttattagacctgattggaacttattgcacc60
tgattggaacttattggaacttattagaccttattggaacttattgcacttattagacct120
tattgcaacttatctgaacttatctgaacaaatctgaacttattggacctgaaacttaat180
tttttaagttgaacagaacgcacccttagtatatcgttgccacatgtgcgttgaattttt240
ccttttcctatcctttccactccatattctcctcaaaagtgtgtaaaaatccgacacacg300
agtagaatgggattgaagtgggtcaagatctgaaaccaatgggtcaatgccacaaaataa360
ggtaaggtttctcgcagtagcaaaaaaataaagttaagttgagagaaaaattatgaatag420
ttgtttctcgtgaagagttgtatacaaaaaaagtctaatttgatacattttcttttacat480
ttataaaggattgaccaatcatccaaattaccaaatatttaggatataaatctttcagat540
tacaacccatatatgatacactaaattttacatgaggcaatggaggatttgcatgaatat600
cgaggagagaaaaaattagttacaaaacttgcataatttatccaaaccaaatcaagtcaa660
gaaacaacgaacaatattatcattagtactataagtatatattataggcttagagcaaag720
ccctaactaccacactgcacacaaatgataactagtaagagaggaaaatacaaatttaag780
attcaacatagcaaattattcatgattcatgattcatgattcatgattcatgattcacga840
acatcaagaatggtatagctgataaaggacaatttaaacataagtgtaaagctcgcacat900
catcaattatattcgcatactactagaccaatctttacttagtacatgtgttagtacatg960
tgttacttcatatcagatgtattgattgttgccaatgacatatcatgttcacttaatctt1020
agggccatttaattataacatggagaataatacaacttaaaattatgtggtggctatcat1080
ctcattttctagataattaaacctttattttgtatacatatatattgtctttacatagca1140
aaacaatattgaaggtataacaacctttcccttttcttttactacatgtttatgttagag1200
tttttcgatttacgattgtggtaaattaattgtaattgatcggttgtcttgtagtcaaga1260
aatgacgtatgaatcaatttagggcatgttctcttcggcataaaacagctgaactgaatt1320
gaactgaactgaaatgaatagtgatatgtgagagtaaaagtattgtcaagagctgaactg1380
agctgaactgaacggatctgaactgaactgatctaatctgaactaatctgaactgaactg1440
aactgaattgaactgaaaataagctagggaaaacagacccttactactattatataacct1500
cgtttaaatattaggaaattaaaaaaataattatatttctttatactttattaacctatt1560
gaggtttttatattgactcccaaatactattttatagatcatgccatgttaatgagcaaa1620
ctactttctcacatctttataggagaaaaagtagatcactcactagcatatcatgaccag1680
cgaaaccaaccaacgctaatagttttattgcttccattagagataagagttaactaataa1740
taccatctttgtgaaattttgatggattttggaactttagcaatatattta1791
metasppheglythrleualailetyrleu
1510
ctgtgtgcactttttgctaccaaaattttataccaatggctcaagtcc1839
leucysalaleuphealathrlysileleutyrglntrpleulysser
152025
tacttatacacaacatacaaactccctcccggcccaccaaggtggccc1887
tyrleutyrthrthrtyrlysleuproproglyproproargtrppro
303540
ttatttggaaacctccttcaactagggccacttccccaccgcgatttc1935
leupheglyasnleuleuglnleuglyproleuprohisargaspphe
455055
gcctcattttgtgaaaaatatgggcctttagtctacataaggcttggt1983
alaserphecysglulystyrglyproleuvaltyrileargleugly
606570
aatgtggatgccataaccactaatgatccagaaatcatacgtgagatc2031
asnvalaspalailethrthrasnaspprogluileilearggluile
75808590
ttagttcgacaagatgatgtatttgcgtctcgtccacataccttagcc2079
leuvalargglnaspaspvalphealaserargprohisthrleuala
95100105
gcaacccacttggcttacaatagtggtgatgtggccttggctccaatg2127
alathrhisleualatyrasnserglyaspvalalaleualapromet
110115120
ggaccaaaatggaaaagaatgagaaggatatgcatggagcacttgctc2175
glyprolystrplysargmetargargilecysmetgluhisleuleu
125130135
acaactagacgacttgaactatttgtgagtcatagggctgatgaggca2223
thrthrargargleugluleuphevalserhisargalaaspgluala
140145150
cgacatttggtccaagacgtattaactcgttcccacaaagataaagtt2271
arghisleuvalglnaspvalleuthrargserhislysasplysval
155160165170
gttaatttgagggaagtgttaggtgcattttctatgaataacgtgact2319
valasnleuarggluvalleuglyalaphesermetasnasnvalthr
175180185
agaatgttgctagggaagcaatactttggggccgggacggcgggccca2367
argmetleuleuglylysglntyrpheglyalaglythralaglypro
190195200
caagaggctctagagtttatgcatataacacatgagttgttttggtta2415
glnglualaleugluphemethisilethrhisgluleuphetrpleu
205210215
ctaggcttgatttacttgggtgattatttgcctttttggaggtgggtt2463
leuglyleuiletyrleuglyasptyrleuprophetrpargtrpval
220225230
gatccatatggatgtgaaaagaaaatgagggaagttgaaaaaagggta2511
aspprotyrglycysglulyslysmetarggluvalglulysargval
235240245250
gatgatttccatcgcaaaattatagaggaacataggaaggagaagaaa2559
aspaspphehisarglysileileglugluhisarglysglulyslys
255260265
aggaaagaagaaatgggagtgaatgagggtgaaatggattttgtagat2607
arglysgluglumetglyvalasngluglyglumetaspphevalasp
270275280
attttgttggctttgcctggtgaaaatggaaatgagcatatggatgat2655
ileleuleualaleuproglygluasnglyasngluhismetaspasp
285290295
gcagatattaaagctctaattcaggtaattcatgtataatttgaatgtgatcga2709
alaaspilelysalaleuilegln
300305
tacaaagtttgatagaaaacatatttgcataaatatatggttgccctactagacccaata2769
aaatacataattattgccttactagttgaaagttgaaacaacctagctaccattttgttg2829
tgattatcattagccaaccaaaattatttcttgcatccatatattaatgttgagatcaga2889
gtcggcatatttacaattacttgtaacattttaagcaaacaaattaaaatattttttggc2949
aagtccattttattgaataatacctatatcttaaaatgaattcttggtcatgtacacttg3009
cctttcaaggtaccaatatttgaccatatgtaattactattaacaaatttgataaaatct3069
aataatatgtaaatatacattcacgcacatattagaaacaaagatcacaaatgataatgc3129
aaaataacttatttgaactaatgttgtgaagttaaatttggaacaaaaggtatatttgta3189
ttgccgaattttaatttataaattacttataacaacaactcaatatgtaaaactgttaag3249
atggagtgtggatagaatgagatgagtatacttttactagttaccactcgaaaatgcatt3309
tcctcctttgtttatagttgttctaacttctattatcataaataattttttggacttatt3369
tcaatgtatatttacaacgctaattgtttaattttttaaaaatacataatgtaaacaagg3429
atttcatggtcaattatacacattataaatattatcttaaaaaacttattaatgctcaat3489
tagtatccataatataggatatgatagcagcagcaacagacacatcagct3539
aspmetilealaalaalathraspthrserala
310315
gtaaccaacgaatgggccatggcagaagtaataaaacacccacgtgtc3587
valthrasnglutrpalametalagluvalilelyshisproargval
320325330
ctccacaagatccaacaagagcttaacacaatagtaggacccaatcga3635
leuhislysileglnglngluleuasnthrilevalglyproasnarg
335340345
atggtaacagaatcagatcttccccaccttaactacctacgttgtgtc3683
metvalthrgluseraspleuprohisleuasntyrleuargcysval
350355360365
gtacgtgaaacgttccgaatgcatccagcaggaccctttttaatccca3731
valarggluthrpheargmethisproalaglypropheleuilepro
370375380
catgaatcactacgccatacaacaatcaacggctatgatatcccatct3779
hisgluserleuarghisthrthrileasnglytyraspileproser
385390395
gggacacgtgtcttcatcaacacacatgggttaggacgtaaccttaaa3827
glythrargvalpheileasnthrhisglyleuglyargasnleulys
400405410
gtgtgggacaacatagaggatttttaccctgaaagacattggccgttg3875
valtrpaspasnilegluaspphetyrprogluarghistrpproleu
415420425
gatggaagtagagttgagattagccatggatctgattttaaaatatta3923
aspglyserargvalgluileserhisglyseraspphelysileleu
430435440445
ccatttagtgctgggaagagaagatgtcctggggccccacttggggtg3971
propheseralaglylysargargcysproglyalaproleuglyval
450455460
gtgtttgtgttgatgggattggctacactttttcatgcatttgattgg4019
valphevalleumetglyleualathrleuphehisalapheasptrp
465470475
ttaccacctgatggaatgaaggcagaagaaattgatactaaggaagtt4067
leuproproaspglymetlysalaglugluileaspthrlysgluval
480485490
tatgggatgactatgcctaaagctcaacctttaatggctttggctaaa4115
tyrglymetthrmetprolysalaglnproleumetalaleualalys
495500505
cctaggcttgctcatttatatctttgatacatgttcatattgtggtg4162
proargleualahisleutyrleu
510515
cacttataagcacaatagacaaatacaagtttgtatcgactctaacatgttgtttagtat4222
tagtatactgcaactctacaagtatgtaatttctataaactataaacacaagtcataacg4282
cattttgttttgaaaaaaaagaggttacattgtcttacaccataaa4328
<210>2
<211>1930
<212>dna
<213>artificialsequence
<220>
<223>cdna-cypgstgene(betavulgaris)
<220>
<221>5'utr
<222>(1)..(243)
<220>
<221>exon
<222>(244)..(1161)
<223>exon1
<220>
<221>exon
<222>(1162)..(1797)
<223>exon2
<220>
<221>3'utr
<222>(1798)..(1930)
<400>2
ttaaaaaaataattatatttctttatactttattaacctattgaggtttttatattgact60
cccaaatactattttatagatcatgccatgttaatgagcaaactactttctcacatcttt120
ataggagaaaaagtagatcactcactagcatatcatgaccagcgaaaccaaccaacgcta180
atagttttattgcttccattagagataagagttaactaataataccatctttgtgaaatt240
ttgatggattttggaactttagcaatatatttactgtgtgcacttttt288
metasppheglythrleualailetyrleuleucysalaleuphe
151015
gctaccaaaattttataccaatggctcaagtcctacttatacacaaca336
alathrlysileleutyrglntrpleulyssertyrleutyrthrthr
202530
tacaaactccctcccggcccaccaaggtggcccttatttggaaacctc384
tyrlysleuproproglyproproargtrpproleupheglyasnleu
354045
cttcaactagggccacttccccaccgcgatttcgcctcattttgtgaa432
leuglnleuglyproleuprohisargaspphealaserphecysglu
505560
aaatatgggcctttagtctacataaggcttggtaatgtggatgccata480
lystyrglyproleuvaltyrileargleuglyasnvalaspalaile
657075
accactaatgatccagaaatcatacgtgagatcttagttcgacaagat528
thrthrasnaspprogluileilearggluileleuvalargglnasp
80859095
gatgtatttgcgtctcgtccacataccttagccgcaacccacttggct576
aspvalphealaserargprohisthrleualaalathrhisleuala
100105110
tacaatagtggtgatgtggccttggctccaatgggaccaaaatggaaa624
tyrasnserglyaspvalalaleualaprometglyprolystrplys
115120125
agaatgagaaggatatgcatggagcacttgctcacaactagacgactt672
argmetargargilecysmetgluhisleuleuthrthrargargleu
130135140
gaactatttgtgagtcatagggctgatgaggcacgacatttggtccaa720
gluleuphevalserhisargalaaspglualaarghisleuvalgln
145150155
gacgtattaactcgttcccacaaagataaagttgttaatttgagggaa768
aspvalleuthrargserhislysasplysvalvalasnleuargglu
160165170175
gtgttaggtgcattttctatgaataacgtgactagaatgttgctaggg816
valleuglyalaphesermetasnasnvalthrargmetleuleugly
180185190
aagcaatactttggggccgggacggcgggcccacaagaggctctagag864
lysglntyrpheglyalaglythralaglyproglnglualaleuglu
195200205
tttatgcatataacacatgagttgttttggttactaggcttgatttac912
phemethisilethrhisgluleuphetrpleuleuglyleuiletyr
210215220
ttgggtgattatttgcctttttggaggtgggttgatccatatggatgt960
leuglyasptyrleuprophetrpargtrpvalaspprotyrglycys
225230235
gaaaagaaaatgagggaagttgaaaaaagggtagatgatttccatcgc1008
glulyslysmetarggluvalglulysargvalaspaspphehisarg
240245250255
aaaattatagaggaacataggaaggagaagaaaaggaaagaagaaatg1056
lysileileglugluhisarglysglulyslysarglysgluglumet
260265270
ggagtgaatgagggtgaaatggattttgtagatattttgttggctttg1104
glyvalasngluglyglumetaspphevalaspileleuleualaleu
275280285
cctggtgaaaatggaaatgagcatatggatgatgcagatattaaagct1152
proglygluasnglyasngluhismetaspaspalaaspilelysala
290295300
ctaattcaggatatgatagcagcagcaacagacacatcagctgtaacc1200
leuileglnaspmetilealaalaalathraspthrseralavalthr
305310315
aacgaatgggccatggcagaagtaataaaacacccacgtgtcctccac1248
asnglutrpalametalagluvalilelyshisproargvalleuhis
320325330335
aagatccaacaagagcttaacacaatagtaggacccaatcgaatggta1296
lysileglnglngluleuasnthrilevalglyproasnargmetval
340345350
acagaatcagatcttccccaccttaactacctacgttgtgtcgtacgt1344
thrgluseraspleuprohisleuasntyrleuargcysvalvalarg
355360365
gaaacgttccgaatgcatccagcaggaccctttttaatcccacatgaa1392
gluthrpheargmethisproalaglypropheleuileprohisglu
370375380
tcactacgccatacaacaatcaacggctatgatatcccatctgggaca1440
serleuarghisthrthrileasnglytyraspileproserglythr
385390395
cgtgtcttcatcaacacacatgggttaggacgtaaccttaaagtgtgg1488
argvalpheileasnthrhisglyleuglyargasnleulysvaltrp
400405410415
gacaacatagaggatttttaccctgaaagacattggccgttggatgga1536
aspasnilegluaspphetyrprogluarghistrpproleuaspgly
420425430
agtagagttgagattagccatggatctgattttaaaatattaccattt1584
serargvalgluileserhisglyseraspphelysileleuprophe
435440445
agtgctgggaagagaagatgtcctggggccccacttggggtggtgttt1632
seralaglylysargargcysproglyalaproleuglyvalvalphe
450455460
gtgttgatgggattggctacactttttcatgcatttgattggttacca1680
valleumetglyleualathrleuphehisalapheasptrpleupro
465470475
cctgatggaatgaaggcagaagaaattgatactaaggaagtttatggg1728
proaspglymetlysalaglugluileaspthrlysgluvaltyrgly
480485490495
atgactatgcctaaagctcaacctttaatggctttggctaaacctagg1776
metthrmetprolysalaglnproleumetalaleualalysproarg
500505510
cttgctcatttatatctttgatacatgttcatattgtggtgcacttataag1827
leualahisleutyrleu
515
cacaatagacaaatacaagtttgtatcgactctaacatgttgtttagtattagtatactg1887
caactctacaagtatgtaatttctataaactataaacacaagt1930
<210>3
<211>517
<212>prt
<213>betavulgaris
<220>
<221>peptide
<222>(1)..(517)
<223>proteincypgst(betavulgaris)
<400>3
metasppheglythrleualailetyrleuleucysalaleupheala
151015
thrlysileleutyrglntrpleulyssertyrleutyrthrthrtyr
202530
lysleuproproglyproproargtrpproleupheglyasnleuleu
354045
glnleuglyproleuprohisargaspphealaserphecysglulys
505560
tyrglyproleuvaltyrileargleuglyasnvalaspalailethr
65707580
thrasnaspprogluileilearggluileleuvalargglnaspasp
859095
valphealaserargprohisthrleualaalathrhisleualatyr
100105110
asnserglyaspvalalaleualaprometglyprolystrplysarg
115120125
metargargilecysmetgluhisleuleuthrthrargargleuglu
130135140
leuphevalserhisargalaaspglualaarghisleuvalglnasp
145150155160
valleuthrargserhislysasplysvalvalasnleuarggluval
165170175
leuglyalaphesermetasnasnvalthrargmetleuleuglylys
180185190
glntyrpheglyalaglythralaglyproglnglualaleugluphe
195200205
methisilethrhisgluleuphetrpleuleuglyleuiletyrleu
210215220
glyasptyrleuprophetrpargtrpvalaspprotyrglycysglu
225230235240
lyslysmetarggluvalglulysargvalaspaspphehisarglys
245250255
ileileglugluhisarglysglulyslysarglysgluglumetgly
260265270
valasngluglyglumetaspphevalaspileleuleualaleupro
275280285
glygluasnglyasngluhismetaspaspalaaspilelysalaleu
290295300
ileglnaspmetilealaalaalathraspthrseralavalthrasn
305310315320
glutrpalametalagluvalilelyshisproargvalleuhislys
325330335
ileglnglngluleuasnthrilevalglyproasnargmetvalthr
340345350
gluseraspleuprohisleuasntyrleuargcysvalvalargglu
355360365
thrpheargmethisproalaglypropheleuileprohisgluser
370375380
leuarghisthrthrileasnglytyraspileproserglythrarg
385390395400
valpheileasnthrhisglyleuglyargasnleulysvaltrpasp
405410415
asnilegluaspphetyrprogluarghistrpproleuaspglyser
420425430
argvalgluileserhisglyseraspphelysileleupropheser
435440445
alaglylysargargcysproglyalaproleuglyvalvalpheval
450455460
leumetglyleualathrleuphehisalapheasptrpleupropro
465470475480
aspglymetlysalaglugluileaspthrlysgluvaltyrglymet
485490495
thrmetprolysalaglnproleumetalaleualalysproargleu
500505510
alahisleutyrleu
515
<210>4
<211>27
<212>dna
<213>artificialsequence
<220>
<223>s1e5983d14forward(fw)primer(5'-3')
<400>4
accaaaattttataccaatggctcaag27
<210>5
<211>23
<212>dna
<213>artificialsequence
<220>
<223>s1e5983d14reverse(rv)primer(5'-3')
<400>5
ggccgggagggagtttgtatgtt23
<210>6
<211>27
<212>dna
<213>artificialsequence
<220>
<223>s1e5983d17forward(fw)primer(5'-3')
<400>6
agaaatcatacgtgagatcttagttcg27
<210>7
<211>26
<212>dna
<213>artificialsequence
<220>
<223>s1e5983d17reverse(rv)primer(5'-3')
<400>7
ggtatgtggacgagacgcaaatacat26
<210>8
<211>4220
<212>dna
<213>betavulgaris
<220>
<221>promotor
<222>(1)..(1353)
<223>putativerpromotor
<220>
<221>gen
<222>(1354)..(3542)
<220>
<221>exon
<222>(1354)..(1938)
<223>verkrztesexon1
<220>
<221>intron
<222>(1939)..(2755)
<220>
<221>exon
<222>(2756)..(3394)
<223>exon2
<220>
<221>3'utr
<222>(3395)..(3542)
<400>8
gtatatcgttgccacatgtgcgttgaatttttccttttcctatcctttccactccatatt60
ctcctcaaaagtgtgtaaaaatccgacacacgagtagaatgggattgaagtgggtcaaga120
tctgaaaccaatgggtcaatgccacaaaataaggtaaggtttctcgcagtagcaaaaaaa180
taaagttaagttgagagaaaaattatgaatagttgtttctcgtgaagagttgtatacaaa240
aaaagtctaatttgatacattttcttttacatttataaaggattgaccaatcatccaaat300
taccaaatatttaggatataaatctttcagattacaacccatatatgatacactaaattt360
tacatgaggcaatggaggatttgcatgaatatcgaggagagaaaaaattagttacaaaac420
ttgcataatttatccaaaccaaatcaagtcaagaaacaacgaacaatattatcattagta480
ctataagtatatattataggcttagagcaaagccctaactaccacactgcacacaaatga540
taactagtaagagaggaaaatacaaatttaagattcaacatagcaaattattcatgattc600
atgattcatgattcatgattcatgattcacgaacatcaagaatggtatagctgataaagg660
acaatttaaacataagtgtaaagctcgcacatcatcaattatattcgcatactactagac720
caatctttacttagtacatgtgttagtacatgtgttacttcatatcagatgtattgattg780
ttgccaatgacatatcatgttcacttaatcttagggccatttaattataacatggagaat840
aatacaacttaaaattatgtggtggctatcatctcattttctagataattaaacctttat900
tttgtatacatatatattgtctttacatagcaaaacaatattgaaggtataacaaccttt960
cccttttcttttactacatgtttatgttagagtttttcgatttacgattgtggtaaatta1020
attgtaattgatcggttgtcttgtagtcaagaaatgacgtatgaatcaatttagggcatg1080
ttctcttcggcataaaacagctgaactgaattgaactgaactgaaatgaatagtgatatg1140
tgagagtaaaagtattgtcaagagctgaactgagctgaactgaacggatctgaactgaac1200
tgatctaatctgaactaatctgaactgaactgaactgaattgaactgaaaataagctagg1260
gaaaacagacccttactactattatataacctcgtttaaatattaggaaattaaaaaaat1320
aattatatttctttatactttattaacctattatacaatagtggtgatgtggcc1374
tyrasnserglyaspvalala
15
ttggctccaatgggaccaaaatggaaaagaatgagaaggatatgcatg1422
leualaprometglyprolystrplysargmetargargilecysmet
101520
gagcacttgctcacaactagacgacttgaactatttgtgagtcatagg1470
gluhisleuleuthrthrargargleugluleuphevalserhisarg
253035
gctgatgaggcacgacatttggtccaagacgtattaactcgttcccac1518
alaaspglualaarghisleuvalglnaspvalleuthrargserhis
40455055
aaagataaagttgttaatttgagggaagtgttaggtgcattttctatg1566
lysasplysvalvalasnleuarggluvalleuglyalaphesermet
606570
aataacgtgactagaatgttgctagggaagcaatactttggggccggg1614
asnasnvalthrargmetleuleuglylysglntyrpheglyalagly
758085
acggcgggcccacaagaggctctagagtttatgcatataacacatgag1662
thralaglyproglnglualaleugluphemethisilethrhisglu
9095100
ttgttttggttactaggcttgatttacttgggtgattatttgcctttt1710
leuphetrpleuleuglyleuiletyrleuglyasptyrleuprophe
105110115
tggaggtgggttgatccatatggatgtgaaaagaaaatgagggaagtt1758
trpargtrpvalaspprotyrglycysglulyslysmetarggluval
120125130135
gaaaaaagggtagatgatttccatcgcaaaattatagaggaacatagg1806
glulysargvalaspaspphehisarglysileileglugluhisarg
140145150
aaggagaagaaaaggaaagaagaaatgggagtgaatgagggtgaaatg1854
lysglulyslysarglysgluglumetglyvalasngluglyglumet
155160165
gattttgtagatattttgttggctttgcctggtgaaaatggaaatgag1902
aspphevalaspileleuleualaleuproglygluasnglyasnglu
170175180
catatggatgatgcagatattaaagctctaattcaggtaattcatg1948
hismetaspaspalaaspilelysalaleuilegln
185190195
tataatttgaatgtgatacaaagtttgatagaaaacatatttgcataaatacatggttac2008
cctactagacccaataaaatacataattattgccttgctagttgaaagttgaaacaacct2068
agctaccattttgttgtgattatcattagccaaccaaaactatttcttgcatccatatat2128
taatgttgagagtcgccatatttacaattacttgtaacattttgagcaaacaaattaaaa2188
tattttttggcaagtccattttattgaatgatacctatatcttaaaatgaatccttggtt2248
atgtacacttgcctttcaaggtaccaatatttgaccatatgtaattactattaacaaatt2308
tgataaaatctaataatatgtaaatatacgttcacgcacatattagaaacaaagatcaca2368
aatgataatgcaaaataacttatttgaactaatgttgtgaagttaaatttggaacaaaag2428
gtatatttgtattgccgaattttaatttataaattacttataacaacaactcaatatgta2488
aaactgttaagatggagtgtggatagaatgagatgagtatacttttactagttaccactc2548
gaaattgcatttcctcctttgtttatagttgttctaacttctattatcataaataatttt2608
tggacttatttcaatgtatatttacaacgttaattgtttaattttttaaaaatacataat2668
gtaaacaaggatttcatggtcaattatacacattatagatattatcttaaaaaacttact2728
aatgctcaattagtgtccataatataggatatgatagcagcagcaacagacaca2782
aspmetilealaalaalathraspthr
200
tcagctgtaaccaacgaatgggccatggcagaagtaataaaacaccca2830
seralavalthrasnglutrpalametalagluvalilelyshispro
205210215220
cgtgtcctccacaagatccaacaagagcttaacacaatagtaggaccc2878
argvalleuhislysileglnglngluleuasnthrilevalglypro
225230235
aatcgaatggtaacagaatcagatcttccccaccttaactacctacgt2926
asnargmetvalthrgluseraspleuprohisleuasntyrleuarg
240245250
tgtgtcgtacgcgaaacgtttcggatgcatccagcaggacccttttta2974
cysvalvalarggluthrpheargmethisproalaglypropheleu
255260265
atcccacatgaatcactacgccatacaacaatcaacggctatgatatc3022
ileprohisgluserleuarghisthrthrileasnglytyraspile
270275280
ccatctgggacacgtgtcttcatcaacacacatgggttaggacgtaac3070
proserglythrargvalpheileasnthrhisglyleuglyargasn
285290295300
cttaaagtgtgggacaacatagaggatttttaccctgaaagacattgg3118
leulysvaltrpaspasnilegluaspphetyrprogluarghistrp
305310315
ccgttggatggaagtagagttgagattagccatggatctgattttaaa3166
proleuaspglyserargvalgluileserhisglyseraspphelys
320325330
atattaccatttagtgctgggaagagaagatgtcctggggccccactt3214
ileleupropheseralaglylysargargcysproglyalaproleu
335340345
ggggtggtgtttgtgttgatgggattggctacactttttcatgcattt3262
glyvalvalphevalleumetglyleualathrleuphehisalaphe
350355360
gattggttaccacctgatggaatgaaggcagaagaaattgatactaag3310
asptrpleuproproaspglymetlysalaglugluileaspthrlys
365370375380
gaagtttatgggatgactatgcctaaagctcaacctttaatggctttg3358
gluvaltyrglymetthrmetprolysalaglnproleumetalaleu
385390395
gctaaacctaggcttgctcctcatttatatctttgatacatgttca3404
alalysproargleualaprohisleutyrleu
400405
tattgtggtgcacttataagcacactagctcaaatagacaaatacaagcttgtattgact3464
ctaacatgttatttaatattagcatactgcaactctacaactacaagtatgtaatctcta3524
taaattataaacataagtcataacgcatcttgttttgaaaaaaaaagctacactttctta3584
caccataaactgttccgttactaaattcttcttggcttattttagaattaatttttcaat3644
tttcctatttacttataacacaagataagaaaatcgactaacaatatttcacaacattat3704
atcttgatagttcgttgaaattaataattttcaataatttcaaaacgcaaaaagcttctt3764
tgttcggataaattcacgtcattttcaataaaatcgcaaaatcttcccttgccatcaagg3824
aacaaatttttcacatcaaaactcttcttccaaaaagtatacttctccattgattatgta3884
aaagagtttgcatgttctaatccattagtatgtatttttcggataagaagttgtattaca3944
agcttaaataaagatataaacttagttaaatatcgaccaatccacaatatgtatcttcat4004
atgtgtccatcagcgcctaaccttgagatgtagatcatctaaagcacattaacatggcgt4064
actcctcctgttcaaatcagggttgtttgaaaccaaacttctccaaaatacttcctcagt4124
aatgaagagaaaaactcacattctgaaaaacatcaacatcatggttctatggtctagtgg4184
ttatgacactggactctgaatccagtaacccgagtt4220
<210>9
<211>4068
<212>dna
<213>zeamays
<220>
<221>exon
<222>(2001)..(2927)
<223>exon1
<220>
<221>intron
<222>(2928)..(3017)
<223>intron
<220>
<221>exon
<222>(3018)..(3683)
<223>exon2
<400>9
gagtgtggtgcgttggttttgtcagatgtcacaggaaggatgcagacctaataccgctgc60
ttttagttgtttgctgagtgctttggctagcctggctatgctgaatcagggattgcaagc120
tcatgcttatgcagtcaacatgggatggatatttgactcagctgtccatgcttctttggt180
gacaatgtatgcaaaatgtggtaggttggctgaggctcatcgtgttttctcatgcattag240
aaatccaagccttgttgccattaattctatgatttcagcatttgcacaacatggcttggc300
tgaagatgcactcaaactttttaacagaatgcaatatgatggccaaaggcctaatcatgt360
gacattcttgggaatactgactgcatgtgctcgagctggtttggttcaacaaggctataa420
ctactttgaatctatgagatcagtctatggcattcaaccaaaccctgaccactacacatg480
tatggttaatcttttaggccatgcaggcttccttgatgaagcattggaaatgattaattc540
gatgccccagaaagattatcctgatgcatgggcagctttgcttagctctagtagcctcca600
ttctaatcttgatttagcaaaactagcagcacagaggcttcttgagatagatccttatga660
cacaacagcttacagggtcctgacaaacatgttctcctcagcagggttgaagggagatga720
agagatggtaaaagttgcacaattgtccaacatggctagtaagaggcctgggtatagcct780
catcatacaggataagactacagaaaataactagcccaaaatgagcacttccatttacag840
ataactggcatgtcataactcataagacagggatattgggttcacagaaaacaattctag900
cgccaagatgagatatcaatttgttgatgtatgctagtgcagagtcagttttagtactta960
agaactgagaatgaggaatatctgataaggtgactagtgatttgaagaataaagagccag1020
gtggtctggatcaaattttggttagtcttgcctcttatatcactgtttccaggatagaat1080
tttcagtactgcaactgatgttgtatttgaataatggaaaatattttttctccctgtaaa1140
caatagttttgtcagatgtaaatcttatccacagtagaatatgatgccatataatgctgt1200
tcagattcattacaatcgtgaagtttattgcgcctttttcagagactggagagttgatga1260
cacttctgcaaataattcatctgctactgttgttgaacaacaagaaaatcattggttgct1320
gcatgctttcaagaatattttgctgcgtgatctatctattctcctcatcaattgatagag1380
aagagtcgctgagagaggtaatctttatgcttattcattcatttcgatgagacaaccata1440
ccaaggtgaattgggtgatccctaggtctaggcttttcttttagcaatagaagcttccaa1500
gtaattcatggaggtctaaggatgttctcctcaagaaaaatagggattgtctgtgtcttt1560
gttatagatgtggcaggtggagtaggactatggtaaacaagggttttctcttatttttgg1620
ctgagaaattgtagattagtggggaaactatcctaattatggcaacataatgtcagcttg1680
ctttttgattaatagtcatgtttcatgacctttttttgttctagtcctcttctttttttt1740
tccttggcattttcccagtgatctacagtttacattgcgcaagtcatgtcaagatcatga1800
cagtttttatatacatggtggtagttgagatgcttaaccatacacatcaacacaaaaacc1860
atgaccaatctaatactgtaaccaatttgcttcaacctcagtggctcaatttacttagta1920
cttaagcgaagaagcttgagacgtcccataaataggccagcatttgcaatccaactttag1980
ctgccatctgtttctgaaccatggatccatttgttctctccatcctcttatgc2033
metaspprophevalleuserileleuleucys
1510
tcatcgatctttgttgtagtgtactggagaaggctgaacagcatgagg2081
serserilephevalvalvaltyrtrpargargleuasnsermetarg
152025
ctaagacttccaccgggacctccaacatggccaattttcggcaatctt2129
leuargleuproproglyproprothrtrpproilepheglyasnleu
303540
ctccagttgagccctcttccccacaaagactttgcccaattttgcacc2177
leuglnleuserproleuprohislysaspphealaglnphecysthr
455055
aaatatggccctctcgtctatcttcgcctgggaaccatcgatgccatc2225
lystyrglyproleuvaltyrleuargleuglythrileaspalaile
60657075
accactgatgaccccgaagtgatccgtgaaatactcatccggcaagat2273
thrthraspaspprogluvalilearggluileleuileargglnasp
808590
gaggtctttgcttcgcggcctcggacactggctgccgtccatctcgcc2321
gluvalphealaserargproargthrleualaalavalhisleuala
95100105
tatgggtgtggtgatgtggctctagctccactgggacccaactggaaa2369
tyrglycysglyaspvalalaleualaproleuglyproasntrplys
110115120
aggatgaggagagtttgcatggagcacttgctgacgaccaggcggctc2417
argmetargargvalcysmetgluhisleuleuthrthrargargleu
125130135
gagtctttcgctgctcaccgagctcaggaggccgagcacctctgccag2465
gluserphealaalahisargalaglnglualagluhisleucysgln
140145150155
tttgtgtgggctaaatctcagtccgggaagcccgtgaacctcagagag2513
phevaltrpalalysserglnserglylysprovalasnleuargglu
160165170
gttctcggtgccttctcgatgaacaacgtcacgcggatgctgctgggg2561
valleuglyalaphesermetasnasnvalthrargmetleuleugly
175180185
aagcagtactttgggatccagtcggcaggccccggcgaggcaatggag2609
lysglntyrpheglyileglnseralaglyproglyglualametglu
190195200
ttcatgcacatcacccacgagctgttcttcctgctgggcctgatctat2657
phemethisilethrhisgluleuphepheleuleuglyleuiletyr
205210215
ctcggggactacttgccggcttggaggtgggtcgacccgtacgggtgt2705
leuglyasptyrleuproalatrpargtrpvalaspprotyrglycys
220225230235
gagaagaggatgagggaggtggagaagaaggtggacgacttccaccag2753
glulysargmetarggluvalglulyslysvalaspaspphehisgln
240245250
aagatcattgatgagcacaggagagctagggaggccaggaagagtcgt2801
lysileileaspgluhisargargalaargglualaarglysserarg
255260265
tcctccgttgaggaagatggcggcaacggcaaagatgagatggacttc2849
serservalglugluaspglyglyasnglylysaspglumetaspphe
270275280
gtcgatgtgctgttatctttgcctggtgagaacgggaaggagcacatg2897
valaspvalleuleuserleuproglygluasnglylysgluhismet
285290295
gacgacatggagatcaaagcgttgatgcaggtgtgtgtatgtgtatgctt2947
aspaspmetgluilelysalaleumetgln
300305
tgctattgcataccctggaattcaattcattttgttgaagctagttattatgagtaatac3007
ttcttggcaggacatgatcgctgctgctactgatacttcatcggtgacg3056
aspmetilealaalaalathraspthrserservalthr
310315320
aacgagtgggtgatggcggaggtaatcaagaacccgcgcgtgctccgg3104
asnglutrpvalmetalagluvalilelysasnproargvalleuarg
325330335
cgcgtccaggaggagctggacgcggtggtggggcgcgaccggatggtg3152
argvalglnglugluleuaspalavalvalglyargaspargmetval
340345350
gcggagtcggacctggcccacctcccctacctccggtgcgtggtgcgc3200
alagluseraspleualahisleuprotyrleuargcysvalvalarg
355360365370
gagtcattccggatgcacccggcggggccgttccttatcccgcacgag3248
gluserpheargmethisproalaglypropheleuileprohisglu
375380385
tcgctgaaggcgacgaccatcatggggtaccacgtgccggcgcgcacg3296
serleulysalathrthrilemetglytyrhisvalproalaargthr
390395400
cgcgtgttcatcaacacgcacgcgctggggcggaacccgcgcgtgtgg3344
argvalpheileasnthrhisalaleuglyargasnproargvaltrp
405410415
gactccgtgggcgagttccggccggagcggcacctgccggcggaggag3392
aspservalglyglupheargprogluarghisleuproalagluglu
420425430
ggggcgcgggtggagatcagccacctgccggacttcaagatcctgccg3440
glyalaargvalgluileserhisleuproaspphelysileleupro
435440445450
ttcagcgccgggaagcgcaagtgccccggcgcgccgctgggcgtggcg3488
pheseralaglylysarglyscysproglyalaproleuglyvalala
455460465
ctggtgctcatggcgctcgccaggctcttccactgcttcgactggtcc3536
leuvalleumetalaleualaargleuphehiscyspheasptrpser
470475480
ccgcccgacggcctccgccccgaggacgtggacacccgggaggtgtac3584
proproaspglyleuargprogluaspvalaspthrarggluvaltyr
485490495
ggcatgaccatgcccaaggccacgcccctcgtcgccgtcgccactccg3632
glymetthrmetprolysalathrproleuvalalavalalathrpro
500505510
cgcctgccgccgcacttgtacggcggcggcggcggcagctcggctcct3680
argleuproprohisleutyrglyglyglyglyglyserseralapro
515520525530
tagttcgatgacactttgacgcacgtgcgctgcactgccagtctcattagtca3733
ttatacgcatgatgtacttccctccatatacaatacaccgcataaaccaaacgatgaatg3793
aaatgtagtcgttctatggtttcaatatgagaagatataattgatggttaacatcatttc3853
cctccgtttagtgataagccatatactctaggtatgtagtagtgtaacaatgcgatccca3913
gcccaggtaacgaggtagcagagaggtgagctagctgtagctggcgacgaagcgaagcat3973
taaacggttgcaacggcagctagccgtggcgttcatgcacggtatggccaaaaaaaaggg4033
accctatccatctgccagttttggcgtaccaatgc4068
<210>10
<211>1593
<212>dna
<213>artificialsequence
<220>
<223>c-dnacypgstgene(zeamays)
<400>10
atggatccatttgttctctccatcctcttatgctcatcgatctttgttgtagtgtactgg60
agaaggctgaacagcatgaggctaagacttccaccgggacctccaacatggccaattttc120
ggcaatcttctccagttgagccctcttccccacaaagactttgcccaattttgcaccaaa180
tatggccctctcgtctatcttcgcctgggaaccatcgatgccatcaccactgatgacccc240
gaagtgatccgtgaaatactcatccggcaagatgaggtctttgcttcgcggcctcggaca300
ctggctgccgtccatctcgcctatgggtgtggtgatgtggctctagctccactgggaccc360
aactggaaaaggatgaggagagtttgcatggagcacttgctgacgaccaggcggctcgag420
tctttcgctgctcaccgagctcaggaggccgagcacctctgccagtttgtgtgggctaaa480
tctcagtccgggaagcccgtgaacctcagagaggttctcggtgccttctcgatgaacaac540
gtcacgcggatgctgctggggaagcagtactttgggatccagtcggcaggccccggcgag600
gcaatggagttcatgcacatcacccacgagctgttcttcctgctgggcctgatctatctc660
ggggactacttgccggcttggaggtgggtcgacccgtacgggtgtgagaagaggatgagg720
gaggtggagaagaaggtggacgacttccaccagaagatcattgatgagcacaggagagct780
agggaggccaggaagagtcgttcctccgttgaggaagatggcggcaacggcaaagatgag840
atggacttcgtcgatgtgctgttatctttgcctggtgagaacgggaaggagcacatggac900
gacatggagatcaaagcgttgatgcaggacatgatcgctgctgctactgatacttcatcg960
gtgacgaacgagtgggtgatggcggaggtaatcaagaacccgcgcgtgctccggcgcgtc1020
caggaggagctggacgcggtggtggggcgcgaccggatggtggcggagtcggacctggcc1080
cacctcccctacctccggtgcgtggtgcgcgagtcattccggatgcacccggcggggccg1140
ttccttatcccgcacgagtcgctgaaggcgacgaccatcatggggtaccacgtgccggcg1200
cgcacgcgcgtgttcatcaacacgcacgcgctggggcggaacccgcgcgtgtgggactcc1260
gtgggcgagttccggccggagcggcacctgccggcggaggagggggcgcgggtggagatc1320
agccacctgccggacttcaagatcctgccgttcagcgccgggaagcgcaagtgccccggc1380
gcgccgctgggcgtggcgctggtgctcatggcgctcgccaggctcttccactgcttcgac1440
tggtccccgcccgacggcctccgccccgaggacgtggacacccgggaggtgtacggcatg1500
accatgcccaaggccacgcccctcgtcgccgtcgccactccgcgcctgccgccgcacttg1560
tacggcggcggcggcggcagctcggctccttag1593
<210>11
<211>530
<212>prt
<213>zeamays
<220>
<221>peptide
<222>(1)..(530)
<223>proteincypgst(zeamays)
<400>11
metaspprophevalleuserileleuleucysserserilepheval
151015
valvaltyrtrpargargleuasnsermetargleuargleupropro
202530
glyproprothrtrpproilepheglyasnleuleuglnleuserpro
354045
leuprohislysaspphealaglnphecysthrlystyrglyproleu
505560
valtyrleuargleuglythrileaspalailethrthraspasppro
65707580
gluvalilearggluileleuileargglnaspgluvalphealaser
859095
argproargthrleualaalavalhisleualatyrglycysglyasp
100105110
valalaleualaproleuglyproasntrplysargmetargargval
115120125
cysmetgluhisleuleuthrthrargargleugluserphealaala
130135140
hisargalaglnglualagluhisleucysglnphevaltrpalalys
145150155160
serglnserglylysprovalasnleuarggluvalleuglyalaphe
165170175
sermetasnasnvalthrargmetleuleuglylysglntyrphegly
180185190
ileglnseralaglyproglyglualametgluphemethisilethr
195200205
hisgluleuphepheleuleuglyleuiletyrleuglyasptyrleu
210215220
proalatrpargtrpvalaspprotyrglycysglulysargmetarg
225230235240
gluvalglulyslysvalaspaspphehisglnlysileileaspglu
245250255
hisargargalaargglualaarglysserargserservalgluglu
260265270
aspglyglyasnglylysaspglumetaspphevalaspvalleuleu
275280285
serleuproglygluasnglylysgluhismetaspaspmetgluile
290295300
lysalaleumetglnaspmetilealaalaalathraspthrserser
305310315320
valthrasnglutrpvalmetalagluvalilelysasnproargval
325330335
leuargargvalglnglugluleuaspalavalvalglyargasparg
340345350
metvalalagluseraspleualahisleuprotyrleuargcysval
355360365
valarggluserpheargmethisproalaglypropheleuilepro
370375380
hisgluserleulysalathrthrilemetglytyrhisvalproala
385390395400
argthrargvalpheileasnthrhisalaleuglyargasnproarg
405410415
valtrpaspservalglyglupheargprogluarghisleuproala
420425430
glugluglyalaargvalgluileserhisleuproaspphelysile
435440445
leupropheseralaglylysarglyscysproglyalaproleugly
450455460
valalaleuvalleumetalaleualaargleuphehiscyspheasp
465470475480
trpserproproaspglyleuargprogluaspvalaspthrargglu
485490495
valtyrglymetthrmetprolysalathrproleuvalalavalala
500505510
thrproargleuproprohisleutyrglyglyglyglyglyserser
515520525
alapro
530
<210>12
<211>4880
<212>dna
<213>solanumtuberosum
<220>
<221>exon
<222>(1762)..(2032)
<223>exon1
<220>
<221>intron
<222>(2033)..(2448)
<223>intron1
<220>
<221>exon
<222>(2449)..(3161)
<223>exon2
<220>
<221>intron
<222>(3162)..(4031)
<223>intron2
<220>
<221>exon
<222>(4032)..(4694)
<223>exon3
<400>12
ataataaatattttttttaaaaaaagagagggttgttagttgcaagggtataagtgagca60
aaaaggtggatggaaggataattttagaccaaaaagatgagtagaagggtatttttagac120
caaaagatggatgaaggatatttttagaccatttcctgtatttcagaggtatttttggcc180
cttttccgtatgaaaatttgaggttgtatttaagttcaattggtcaaattaataagtatt240
tttaagactgtcaaaaatttagaaataaaactaataattcacatcaattctacaaatatt300
tctttttaatatatagacattcggttcaagtaaaatattttaagtttttatttaatttct360
tgtttgtttgattatggattgccttggattctatttgcgtaatggaaatggcagctcttt420
tcagtgtaggattggacaagttagcttggattttttttcacctactctacaaaaagtgat480
ggggcgtccaccaaccatacgtcaacaactttgtccattctttgtacacactaccatcac540
aatctatcaaaagatttgttctttagtctcgtttaacatattctttcgttttcattttaa600
tttatttgtcttatttttttttataaaataaaataaatattttttgatataatacataaa660
tatattctttaatttcatttcatttaacatctctatactttaacttgggagtcgagtgat720
tgggttggaagtcaggtcgtgattagaggttaggagttaggtcccaactcggaattgaga780
ttagaggtccagattagatctcaggttagagtcgggagttgggtcttaagttgtggtcag840
gatttgggtcctagttgaggtcacaagtcaggtccccgctcgagatcgaaaattaggtcc900
cgagttgagatagaaagttttttgatcaagattgaggtcgtagtcaagagtcgagttctg960
agtcgggattgtaatcgggagtgggtccttacaggttagggcgggggtcgaaagtcgggt1020
ctcaagttaggactcgtttgagggtggtgaggtttaagtaggggtgtgcatcgatcggtt1080
cggtttatcgattttcagtttttaaatatgctaaattaataaccaaatcaaaaggttttt1140
tttatcgattttggtcattaatgactcgattttctatttaaccaattagaaaatgctcat1200
aaaacaaatagatgacttctctaacaaaatttgaccgacaagacaagacaataatgtgat1260
tattattttaggttttgacgttttgtataatgtgaagtgtgaattgaaggtttaagggca1320
aagacagtaactagtaatatattgagattaatatttatgtaataagtaaaagaagtaaat1380
tactggagtataatcttattgagttattgttttacccaataacacaatattaaaaatcaa1440
tatcgaatcgataacccgatattttttttaaaaaaaaattgttaacccaattgatgcatt1500
tttttatatattcgtaaaatgcgtaaattgcattaaattattcccataaataaaaaagaa1560
acttgttcataaattcaaacttattgtgcaaattgaggaagcaaaaaagtgtgatttcaa1620
gattaatacttatagttgaaacatgtcaagatgatgggcttcctaatcgctccaaaaaaa1680
gattataaattagaattaaacatatttccttattaagcctttgtaaactacttctttctt1740
ttttttgacaaataattaaagatgattgacttgactagttttgttattgtc1791
metileaspleuthrserphevalileval
1510
cttctttgcacgtatcttcttaatttgatcaattatagtatagtcctt1839
leuleucysthrtyrleuleuasnleuileasntyrserilevalleu
152025
tttggcgcatatcttatttccaagctacttcatttttcattcgtcgat1887
pheglyalatyrleuileserlysleuleuhispheserphevalasp
303540
aagtcgaatcgagaaatcaatcaactccctcctggtccaaaacaatgg1935
lysserasnarggluileasnglnleuproproglyprolysglntrp
455055
cctattgtaggcaacctttttcagttagggcaattacctcatcgagac1983
proilevalglyasnleupheglnleuglyglnleuprohisargasp
606570
atggcgtctttctgcgaaaaatatggcccattggtctatctccgactag2032
metalaserphecysglulystyrglyproleuvaltyrleuargleu
75808590
gtaatgttgatacaataaattcacaaatgaccacaatttagtccttgtaattaataaata2092
gtcattgttttcgagtttcaaatttataagggaaacttcaagtacacttgacttggagat2152
tcacttatggatgttgcaactcgatgaacaattactattttacaaaaaaatgatcgaaaa2212
gtgataagtgataagtgataagtgaacgttatttctcatataaagttcaatgctatacag2272
attcggtcaaactcagtaaattttacctaagtactttatccatgttaagaaaaaatcatt2332
atatatgtacacacattaaattttaaaccgagttattagcactagaatttatcgttctaa2392
cattttgaacccataaagttgatatcgtgactccaccttttgttttctcttaaaaggt2450
gly
aatgttgatgctatcaccaccaatgatccagaaatcataagagaaata2498
asnvalaspalailethrthrasnaspprogluileilearggluile
95100105
cttgtacaacaagatgatgtttttgcatctaggccaagaactcttgct2546
leuvalglnglnaspaspvalphealaserargproargthrleuala
110115120
gccattcatctagcttatggttgtggggatgtggcattggctccttta2594
alailehisleualatyrglycysglyaspvalalaleualaproleu
125130135
ggtccaaaatggaaaagaatgagaagaatatgtatggaacatttattg2642
glyprolystrplysargmetargargilecysmetgluhisleuleu
140145150155
acaactaaaagacttgaatcatttgcaaaacatagggcagatgaagcc2690
thrthrlysargleugluserphealalyshisargalaaspgluala
160165170
caaagtctagttaaagatatttggaccaaagcccaaaaaggacaaata2738
glnserleuvallysaspiletrpthrlysalaglnlysglyglnile
175180185
gtgaatttgagggaagttttgggtggattttcaatgaataatgtgact2786
valasnleuarggluvalleuglyglyphesermetasnasnvalthr
190195200
agaatgttgttaggtaaacaatattttggggcagaatcagcaggtcca2834
argmetleuleuglylysglntyrpheglyalagluseralaglypro
205210215
caagaagcaatggaatttatgcatgtaacacatgagttattttggtta2882
glnglualametgluphemethisvalthrhisgluleuphetrpleu
220225230235
cttggagtgatatatttaggtgattatttacctttttggaggtggatt2930
leuglyvaliletyrleuglyasptyrleuprophetrpargtrpile
240245250
gatccttatggttgtgagaaaaaaatgagggatgttgaaaaaaggatt2978
aspprotyrglycysglulyslysmetargaspvalglulysargile
255260265
gatgattttcatatgagaataattgaagaacatagaaagaagaaaggt3026
aspaspphehismetargileileglugluhisarglyslyslysgly
270275280
aataaaaataataataatattgatgatgatgaaatggactttgtggat3074
asnlysasnasnasnasnileaspaspaspglumetaspphevalasp
285290295
gttttattgtctctaccaggagaagatgaaggagatggtaatggaaaa3122
valleuleuserleuproglygluaspgluglyaspglyasnglylys
300305310315
caaaatatggatgatgtagagattaaagctctaattcaggtctaatttt3171
glnasnmetaspaspvalgluilelysalaleuilegln
320325
ttttatatataccaactttctacctatttaatatgtcaatgcatttatctcaaactactt3231
atatatatatccaatatatatgcatttggtgagcaacaactaccacccatgggatttcta3291
agcttgattaacagaggttgtggtgaagtgataagtacttcttcatccctaactagaggt3351
cttgggttcgagtcttgctggatacaaagtcgtctttgttaaagagtgttaccccataat3411
gtgagactttccggcacgaatcaaaattttgttggaatctaatggggtatcgaacaccag3471
ttgagaaaagaaagagaagaagaaattcctacaatgtgggactttcgggagcgaacccaa3531
attttgttggactctaatgagggtatgggacacccgtagaaaaaaaaagaaataaattca3591
caagtttgattgatacttaaaacataaaatttaagttatatatactgatagagtaaaaca3651
aattacattattagtccaatttaatcggtactaccatgttattactctattaaattatta3711
tatgagacttaatatagagagttacatgtcaacttgatagtgtagacatttttagactat3771
tcatgcacaaaaacttaactctaatacaatcaatttccttgcaacttttatattagagaa3831
tatatatatatatatatatatatatatatatatagtacagtatattttaacatgtataaa3891
caagatgcctattttattttcagcttattactaatcacaatatatatttgctacatcaat3951
agtataaaaaaaagctaaatatggttcttttttctgctgtgacttatttcttttctttaa4011
ttaaattataggatatgatagctgcagccacagacacttctgctgtgaccaac4064
alaalaalathraspthrseralavalthrasn
330335
gaatgggcaatggctgaggtaatcagacatccacatgtcctcaaaaag4112
glutrpalametalagluvalilearghisprohisvalleulyslys
340345350355
atccaagaagaactcgatatagttgtcgggtcgggtcggatggtaacc4160
ileglnglugluleuaspilevalvalglyserglyargmetvalthr
360365370
gaatccgacttgatccatctcaagtacctccgttgtgtagtacgtgaa4208
gluseraspleuilehisleulystyrleuargcysvalvalargglu
375380385
acattccgaatgcaccctgcgggtccattcctaatcccacatgaatca4256
thrpheargmethisproalaglypropheleuileprohisgluser
390395400
attcgcgatactatgatcaacggctattacatcccggccaagacacgc4304
ileargaspthrmetileasnglytyrtyrileproalalysthrarg
405410415
gtgttcatcaacacacatggtcttggccggaacacaaagatttgggac4352
valpheileasnthrhisglyleuglyargasnthrlysiletrpasp
420425430435
aacatagatgagtttaggccagagagacatttaccaccaaatgatgat4400
asnileaspglupheargprogluarghisleuproproasnaspasp
440445450
gaaaaaaacatgatcatgactactagtagtagtagagttgagattagt4448
glulysasnmetilemetthrthrserserserargvalgluileser
455460465
catggtccagatttcaagattttgccatttagtgctggaaaaaggaag4496
hisglyproaspphelysileleupropheseralaglylysarglys
470475480
tgtcctggtgcaccattgggtgtgaaattggtgcttatggcattggct4544
cysproglyalaproleuglyvallysleuvalleumetalaleuala
485490495
aggttgtttcattgctatgattggagtccaccaaatggagtaaagcat4592
argleuphehiscystyrasptrpserproproasnglyvallyshis
500505510515
caagatattgacacaaatgaagtttatggaatgactatgcctaaagct4640
glnaspileaspthrasngluvaltyrglymetthrmetprolysala
520525530
aagccattgatggctattgctaaacctagactgcctgctcacttgtac4688
lysproleumetalailealalysproargleuproalahisleutyr
535540545
caataattagttactagtactcaaatcaaagggaattggtctttaatggctttcgt4744
gln
aacgaatcaaataaagagaaatttctatttgtacctcgtaaaaaaaagctctcgcctcct4804
gtaatagtatacttcaacatttttctcacctgtggcaatccacacagcagacttaagtct4864
gtctgattacatagta4880
<210>13
<211>1656
<212>dna
<213>artificialsequence
<220>
<223>cdnacypgstgene(solanumtuberosum)
<400>13
atgattgacttgactagttttgttattgtccttctttgcacgtatcttcttaatttgatc60
aattatagtatagtcctttttggcgcatatcttatttccaagctacttcatttttcattc120
gtcgataagtcgaatcgagaaatcaatcaactccctcctggtccaaaacaatggcctatt180
gtaggcaacctttttcagttagggcaattacctcatcgagacatggcgtctttctgcgaa240
aaatatggcccattggtctatctccgactaggtaatgttgatgctatcaccaccaatgat300
ccagaaatcataagagaaatacttgtacaacaagatgatgtttttgcatctaggccaaga360
actcttgctgccattcatctagcttatggttgtggggatgtggcattggctcctttaggt420
ccaaaatggaaaagaatgagaagaatatgtatggaacatttattgacaactaaaagactt480
gaatcatttgcaaaacatagggcagatgaagcccaaagtctagttaaagatatttggacc540
aaagcccaaaaaggacaaatagtgaatttgagggaagttttgggtggattttcaatgaat600
aatgtgactagaatgttgttaggtaaacaatattttggggcagaatcagcaggtccacaa660
gaagcaatggaatttatgcatgtaacacatgagttattttggttacttggagtgatatat720
ttaggtgattatttacctttttggaggtggattgatccttatggttgtgagaaaaaaatg780
agggatgttgaaaaaaggattgatgattttcatatgagaataattgaagaacatagaaag840
aagaaaggtaataaaaataataataatattgatgatgatgaaatggactttgtggatgtt900
ttattgtctctaccaggagaagatgaaggagatggtaatggaaaacaaaatatggatgat960
gtagagattaaagctctaattcaggatatgatagctgcagccacagacacttctgctgtg1020
accaacgaatgggcaatggctgaggtaatcagacatccacatgtcctcaaaaagatccaa1080
gaagaactcgatatagttgtcgggtcgggtcggatggtaaccgaatccgacttgatccat1140
ctcaagtacctccgttgtgtagtacgtgaaacattccgaatgcaccctgcgggtccattc1200
ctaatcccacatgaatcaattcgcgatactatgatcaacggctattacatcccggccaag1260
acacgcgtgttcatcaacacacatggtcttggccggaacacaaagatttgggacaacata1320
gatgagtttaggccagagagacatttaccaccaaatgatgatgaaaaaaacatgatcatg1380
actactagtagtagtagagttgagattagtcatggtccagatttcaagattttgccattt1440
agtgctggaaaaaggaagtgtcctggtgcaccattgggtgtgaaattggtgcttatggca1500
ttggctaggttgtttcattgctatgattggagtccaccaaatggagtaaagcatcaagat1560
attgacacaaatgaagtttatggaatgactatgcctaaagctaagccattgatggctatt1620
gctaaacctagactgcctgctcacttgtaccaataa1656
<210>14
<211>551
<212>prt
<213>solanumtuberosum
<220>
<221>peptid
<222>(1)..(551)
<223>proteincypgst(solanumtuberosum)
<400>14
metileaspleuthrserphevalilevalleuleucysthrtyrleu
151015
leuasnleuileasntyrserilevalleupheglyalatyrleuile
202530
serlysleuleuhispheserphevalasplysserasnarggluile
354045
asnglnleuproproglyprolysglntrpproilevalglyasnleu
505560
pheglnleuglyglnleuprohisargaspmetalaserphecysglu
65707580
lystyrglyproleuvaltyrleuargleuglyasnvalaspalaile
859095
thrthrasnaspprogluileilearggluileleuvalglnglnasp
100105110
aspvalphealaserargproargthrleualaalailehisleuala
115120125
tyrglycysglyaspvalalaleualaproleuglyprolystrplys
130135140
argmetargargilecysmetgluhisleuleuthrthrlysargleu
145150155160
gluserphealalyshisargalaaspglualaglnserleuvallys
165170175
aspiletrpthrlysalaglnlysglyglnilevalasnleuargglu
180185190
valleuglyglyphesermetasnasnvalthrargmetleuleugly
195200205
lysglntyrpheglyalagluseralaglyproglnglualametglu
210215220
phemethisvalthrhisgluleuphetrpleuleuglyvaliletyr
225230235240
leuglyasptyrleuprophetrpargtrpileaspprotyrglycys
245250255
glulyslysmetargaspvalglulysargileaspaspphehismet
260265270
argileileglugluhisarglyslyslysglyasnlysasnasnasn
275280285
asnileaspaspaspglumetaspphevalaspvalleuleuserleu
290295300
proglygluaspgluglyaspglyasnglylysglnasnmetaspasp
305310315320
valgluilelysalaleuileglnaspmetilealaalaalathrasp
325330335
thrseralavalthrasnglutrpalametalagluvalilearghis
340345350
prohisvalleulyslysileglnglugluleuaspilevalvalgly
355360365
serglyargmetvalthrgluseraspleuilehisleulystyrleu
370375380
argcysvalvalarggluthrpheargmethisproalaglyprophe
385390395400
leuileprohisgluserileargaspthrmetileasnglytyrtyr
405410415
ileproalalysthrargvalpheileasnthrhisglyleuglyarg
420425430
asnthrlysiletrpaspasnileaspglupheargprogluarghis
435440445
leuproproasnaspaspglulysasnmetilemetthrthrserser
450455460
serargvalgluileserhisglyproaspphelysileleuprophe
465470475480
seralaglylysarglyscysproglyalaproleuglyvallysleu
485490495
valleumetalaleualaargleuphehiscystyrasptrpserpro
500505510
proasnglyvallyshisglnaspileaspthrasngluvaltyrgly
515520525
metthrmetprolysalalysproleumetalailealalysproarg
530535540
leuproalahisleutyrgln
545550
<210>15
<211>525
<212>prt
<213>triticumaestivum
<220>
<221>peptid
<222>(1)..(525)
<223>proteincypgst(trticumaestivum)
<400>15
metasppropheleuleuserileileleucyssercysilepheala
151015
alavalsertrplyslysleuasnglymetargleuargleupropro
202530
glyproproargtrpproilepheglyasnleuleuglnleuserpro
354045
leuprohislysaspphealaargphecysthrlystyrglyproleu
505560
valtyrleuargleuglythrileaspalailethrthraspasppro
65707580
gluvalilearggluileleuileargglnaspgluvalphealaser
859095
argproargthrleualaalavalhisleualatyrglycysglyasp
100105110
valalaleualaproleuglyproasntrplysargmetargargval
115120125
cysmetgluhisleuleuthrthrargargleugluserphealaala
130135140
hisargalagluglualagluhisleucysgluphevaltrpalalys
145150155160
serglnserglylysprovalasnleuarggluvalleuglyalaphe
165170175
sermetasnasnvalthrargmetleuleuglylysglntyrphegly
180185190
leuglnseralaglyproglyglualametgluphemethisilethr
195200205
hisgluleuphepheleuleuglyleuiletyrleuglyasptyrleu
210215220
proalatrpargtrpleuaspprotyrglycysglulyslysmetarg
225230235240
gluvalglulyslysvalaspaspphehisglnlysileileaspglu
245250255
hisarglysalaargaspvalarglysserglyalaserleuaspasp
260265270
aspglyaspaspserlysgluglymetaspphevalaspvalleuleu
275280285
serleuproglygluasnglyasngluhismetaspaspvalgluile
290295300
lysalaleumetglnaspmetilealaalaalathraspthrserser
305310315320
valthrasnglutrpvalmetalagluvalilelysasnproargval
325330335
leuarglysileglnglugluleuaspalavalvalglythrserarg
340345350
prohisglyglyglyglyglyproproproproaspvalproproleu
355360365
argargproglyvalleuproaspalaproglyglyalaileproasp
370375380
proalaargvalthrglnglyaspasphishisglyleuarghispro
385390395400
glyalaaspgluaspleuhisglnhisproargalaglyproglupro
405410415
alahisleuglyargargargargvalproproargglualapropro
420425430
glyglyargargalaargglyaspglnproproalaglyleuglnasp
435440445
proalaleuglnargargglnalaglnvalproargglyalaalagly
450455460
argaspproglyalahisglyalaargglnalaleuproleuleuarg
465470475480
leuvalproalaargargproproproargglyhisarghisargarg
485490495
glyleuargaspasphisalaglnglyglnalaalahisargargarg
500505510
serthralaproalaalaalaaspvalargleuleuser
515520525
<210>16
<211>518
<212>prt
<213>helianthusannuus
<220>
<221>peptid
<222>(1)..(518)
<223>proteincypgst(helianthusannuus)
<400>16
metaspleuglnserpheileservalleualapheilevalalaser
151015
argileileleuleutrptyrvallysglnargvalthrglnasnthr
202530
thrhisargleuproproglyproproargtrpproilevalglyasn
354045
leuleuglnleuglyproleuprohisargaspleualaserphecys
505560
gluargtyrglyproleuvaltyrleuargleuglylysvalaspala
65707580
ilethrthrasnaspproasnileilearggluileleuvallysgln
859095
aspaspvalphealaserargproglnthrleualaalavalhisleu
100105110
alatyrasncysglyaspvalalaleualapropheglyproargtrp
115120125
lystrpmetargargilecysmetgluglnleuleuthrthrlysarg
130135140
leugluserphealalysglnargalaserglualaglnhisleuval
145150155160
glnaspvaltrpalaleuserglnalaasnglyproileasnleuarg
165170175
gluvalleuglyglyphesermetasnasnvalthrargmetleuleu
180185190
glylysglntyrpheglyserglyseralaglyprolysglualathr
195200205
gluphemethisilethrhisgluleuphetrpleuleuglyleuile
210215220
tyrleuglyasptyrleuprophetrpargtrpileaspprotyrgly
225230235240
cysglulyslysmetarggluvalglulysargvalaspaspphehis
245250255
metlysileileglugluhisargglnargarglysasnglyglugln
260265270
lysaspgluglyilemetaspphevalaspvalleuleuserleupro
275280285
glygluaspglylysasphismetaspaspargglnilelysalaleu
290295300
valglnaspmetilealaalaalathraspthrseralavalthrasn
305310315320
glutrpalametalagluvalilelyshisprohisvalleuarglys
325330335
ileglnglugluleuaspasnvalvalglyproaspargmetvalser
340345350
gluseraspleuserasnleuasntyrleuargcysvalvalargglu
355360365
thrpheargmethisproalaglypropheleuileprohisgluser
370375380
leuargalathrgluileasnglytyrtyrileproalalysthrarg
385390395400
valpheileasnthrhisglyleuglyargasnthrvalleutrpasp
405410415
aspileasnvalpheargprogluarghisleuthrseraspglyser
420425430
argvalgluileserhisglyaspaspphelysileleupropheser
435440445
alaglylysarglyscysproglyalaproleuglyvalthrleuval
450455460
leumetalaleualaargleuphehiscyspheasptrpserpropro
465470475480
aspglyleulyscysgluaspileaspthrglngluiletyrglymet
485490495
thrmetprolysalalysproleumetalavalalalysproargleu
500505510
alasertyrmettyrgln
515
<210>17
<211>530
<212>prt
<213>hordeumvulgare
<220>
<221>peptid
<222>(1)..(530)
<223>proteincypgst(hordeumvulgare)
<400>17
metaspleupheleupheserileileleucyssercysilepheala
151015
alavalsertrparglysleuserargleuargleuargleupropro
202530
glyproproargtrpproilepheglyasnleuleuglnleuserpro
354045
leuprohislysaspphealaargphecysthrlystyrglyproleu
505560
valtyrleuargleuglythrileaspalailethrthraspasppro
65707580
gluvalilearggluileleuvalargglnaspgluvalphealaser
859095
argproargthrleualaalavalhisleualatyrglycysglyasp
100105110
valalaleualaproleuglyproasntrplysargmetargargval
115120125
cysmetgluhisleuleuthrthrlysargleugluserphealaala
130135140
hisargalaglnglualagluhisleucysgluphevaltrpalalys
145150155160
serglnserglylysprovalasnleuarggluvalleuglyalaphe
165170175
sermetasnasnvalthrargmetleuleuglylysglntyrphegly
180185190
leuglnseralaglyproglyglualametgluphemethisilethr
195200205
hisgluleuphepheleuleuglyleuiletyrleuglyasptyrleu
210215220
proalatrpargtrpvalaspprotyrglycysglulyslysmetarg
225230235240
gluvalglulyslysvalaspaspphehisglnlysileileaspglu
245250255
hisarglysalaargaspalaarglysseralaalaserleuaspasp
260265270
glyaspaspserlysgluaspmetaspphevalaspvalleuleuser
275280285
leuproglygluasnglyasngluhismetaspaspvalgluilelys
290295300
alaleumetglnaspmetilealaalaalathraspthrserserval
305310315320
thrasnglutrpvalmetalagluvalilelysasnprocysvalleu
325330335
arglysileglnglugluleuaspalavalvalglyargserargmet
340345350
valvalgluseraspleuprohisleuthrtyrleuargcysvalval
355360365
arggluserpheargmethisproalaglypropheleuileprohis
370375380
gluserleulysalathrthrilemetglytyraspileproalagln
385390395400
thrargilepheileasnthrhisalaleuglyargasnproargile
405410415
trpaspaspvalglygluphehisprogluarghisleuproalaasp
420425430
glyglyargvalgluileserhisleuproaspphelysileleupro
435440445
pheseralaglylysarglyscysproglyalaproleuglyvalile
450455460
leuvalleumetalaleualaargleuphehiscyspheasptrpser
465470475480
proproaspglyleuargprogluaspileaspthraspgluvaltyr
485490495
glymetthrmetprolysalalysproleuilealaalavalglnpro
500505510
argleuproproglnmettyrglysercysproserhisglymetgln
515520525
metgln
530
<210>18
<211>466
<212>prt
<213>brassicanapus
<220>
<221>peptid
<222>(1)..(466)
<223>proteincypgst(brassicanapus)
<400>18
metalaalaleucysserlystyrglyproleuvaltyrleuargleu
151015
glyasnileaspalailethrthrasnaspprogluthrileargglu
202530
ileleupheargglnaspaspvalphealaserargprolysthrleu
354045
alaalavalhisleualatyrglycysglyaspvalalaleualapro
505560
metglyprohistrplysargmetargargilecysmetgluhisleu
65707580
leuthrthrlysargleugluserphethrserglnargalagluglu
859095
alaglntyrleuileglnaspvalcyslysargalaglucysglylys
100105110
proileasnleuarggluvalleuglyalaphesermetasnasnval
115120125
thrargmetleuleuglylysglnphepheglyproglyservalval
130135140
glyalalysglualaglngluphemethisilethrhislysleuphe
145150155160
argleuleuglyvaliletyrleuglyasptyrleuprophetrparg
165170175
trpvalaspprotyrglycysglulysglumetargaspvalglulys
180185190
argvalasplysphehisthrlysileileglugluhisargargala
195200205
lysargglulysgluasplysasnilegluglyaspmetasppheval
210215220
aspvalleuleuserleuproglygluasnglylysgluhismetasp
225230235240
aspvalgluilelysalaleuileglnaspmetilealaalaalathr
245250255
aspthrseralavalthrasnglutrpalametalagluvalilelys
260265270
glnproargvalmetarglysileglnglugluleuaspasnvalval
275280285
glyserasnargmetvalasngluthraspleuvalhisleuasntyr
290295300
leuargcysvalvalarggluthrpheargmethisproalaglypro
305310315320
pheleuileprohisgluservalargprothrthrileasnglytyr
325330335
tyrileproalalysthrargvalpheileasnthrhisglyleugly
340345350
argasnthrservaltrpthrthraspilegluglupheargproglu
355360365
arghistrpprovalaspglyserglyargvalgluileserhisgly
370375380
proasptyrlysileleupropheseralaglylysarglyscyspro
385390395400
glyalaproleuglyvalthrmetvalleumetalaleualaargleu
405410415
phehiscyspheasptrpthrthrprogluaspileaspthrvalglu
420425430
valtyrglymetthrmetprolysalalysproleutrpalaleuala
435440445
lysproargleualaalahisleutyrthrilethrhisaspthrile
450455460
glyhis
465
<210>19
<211>530
<212>prt
<213>brassicaoleracea
<220>
<221>peptid
<222>(1)..(530)
<223>proteincypgst(brassicaoleracea)
<400>19
metaspleupheleuleuserileileleucyssertrpilepheval
151015
alavaltyrtrplyslysleuasnargthrlysleuargleupropro
202530
glyproproargtrpproilepheglyasnleuleuglnleuserpro
354045
leuprohislysaspphealaargphecysthrlystyrglyproleu
505560
valtyrleuargleuglythrileaspalailethrthraspasppro
65707580
gluvalilearggluileleuileargglnaspgluvalphealaser
859095
argproargthrleualaalavalhisleualatyrglycysglyasp
100105110
valalaleualaproleuglyproasntrplysargmetargargval
115120125
cysmetgluhisleuleuthrthrlysargleugluserphealaala
130135140
hisargalaglnglualagluhisleucysglnphevaltrpalalys
145150155160
serglnserglulysprovalasnleuarggluvalleuglyalaphe
165170175
sermetasnasnvalthrargmetleuleuglylysglntyrphegly
180185190
leuglnseralaglyproglyglualametgluphemethisilethr
195200205
hisgluleuphetyrleuleuglyleuiletyrleuglyasptyrleu
210215220
proalatrpargtrpvalaspprotyrglycysglulyslysmetarg
225230235240
gluvalglulyslysvalaspaspphehisglnlysileileaspglu
245250255
hisarglysalaargglualaarglysseralaserserleuaspasp
260265270
glyaspaspserlysgluglumetaspphevalaspvalleuleuser
275280285
leuproglygluasnglylysgluhismetaspaspvalgluilelys
290295300
alaleumetglnaspmetilealaalaalathraspthrserserval
305310315320
thrasnglutrpvalmetalagluvalilelyshisproargvalleu
325330335
arglysileglnglugluleuaspalavalvalglyargalaargmet
340345350
valsergluseraspleuprohisleuprotyrleuargcysvalval
355360365
arggluserpheargmethisproalaglypropheleuileprohis
370375380
gluserleulysprothrthrilemetglytyraspileproalaarg
385390395400
thrargilepheileasnthrhisalaleuglyargasnproargval
405410415
trpaspaspvalglyglnpheargprogluarghismetproalaasp
420425430
glyglyalaargvalgluileserhisleuproaspphelysileleu
435440445
propheseralaglylysarglyscysproglyalaproleuglyval
450455460
ileleuvalleumetalaleualaargleuphehiscyspheasptrp
465470475480
serproproaspglyglugluileaspthraspgluvaltyrglymet
485490495
thrmetprolysalaleuproleuphealaalaalaargproargleu
500505510
proproglumettyrhisglysersercysproserhisglylysgln
515520525
thrmet
530
<210>20
<211>466
<212>prt
<213>brassicarapa
<220>
<221>peptid
<222>(1)..(466)
<223>proteincypgst(brassicarapa)
<400>20
metalaalaleucysserlystyrglyproleuvaltyrleuargleu
151015
glyasnileaspalailethrthrasnaspprogluthrileargglu
202530
ileleupheargglnaspaspvalphealaserargprolysthrleu
354045
alaalavalhisleualatyrglycysglyaspvalalaleualapro
505560
metglyprohistrplysargmetargargilecysmetgluhisleu
65707580
leuthrthrlysargleugluserphethrserglnargalagluglu
859095
alaglntyrleuileglnaspvalcyslysargalaglucysglylys
100105110
proileasnleuarggluvalleuglyalaphesermetasnasnval
115120125
thrargmetleuleuglylysglnphepheglyproglyservalval
130135140
glyalalysglualaglngluphemethisilethrhislysleuphe
145150155160
argleuleuglyvaliletyrleuglyasptyrleuprophetrparg
165170175
trpvalaspprotyrglycysglulysglumetargaspvalglulys
180185190
argvalasplysphehisthrlysileileglugluhisargargala
195200205
lysargglulysgluasplysasnilegluglyaspmetasppheval
210215220
aspvalleuleuserleuproglygluasnglylysgluhismetasp
225230235240
aspvalgluilelysalaleuileglnaspmetilealaalaalathr
245250255
aspthrseralavalthrasnglutrpalametalagluvalilelys
260265270
glnproargvalmetarglysileglnglugluleuaspasnvalval
275280285
glyserasnargmetvalasngluthraspleuvalhisleuasntyr
290295300
leuargcysvalvalarggluthrpheargmethisproalaglypro
305310315320
pheleuileprohisgluservalargprothrthrileasnglytyr
325330335
tyrileproalalysthrargvalpheileasnthrhisglyleugly
340345350
argasnthrservaltrpthrthraspilegluglupheargproglu
355360365
arghistrpprovalaspglyserglyargvalgluileserhisgly
370375380
proasptyrlysileleupropheseralaglylysarglyscyspro
385390395400
glyalaproleuglyvalthrmetvalleumetalaleualaargleu
405410415
phehiscyspheasptrpthrthrprogluaspileaspthrvalglu
420425430
valtyrglymetthrmetprolysalalysproleutrpalaleuala
435440445
lysproargleualaalahisleutyrthrilethrhisaspthrile
450455460
glyhis
465
<210>21
<211>523
<212>prt
<213>glycinemax
<220>
<221>peptid
<222>(1)..(523)
<223>proteincypgst(glycinemax)
<400>21
metaspleuthrthrpheileserthrleupheleuglythrleuala
151015
serargileilearghistrpleuileglyargserleuserserhis
202530
lysasnlysleuproproglyproproargtrpproilevalglyasn
354045
leuleuglnleuglyglnleuprohisargaspleualaserleucys
505560
asplystyrglyproleuvaltyrleulysleuglylysileaspala
65707580
ilethrthrasnaspproaspileilearggluileleuleusergln
859095
aspaspvalphealaserargprohisthrphealaalavalhisleu
100105110
alatyrglycysglyaspvalalaleualaproleuglyprohistrp
115120125
lysargmetargargilecysmetgluhisleuleuthrthrlysarg
130135140
leugluserpheserasnhisargleuaspglualaglnhisleuval
145150155160
lysaspvalmetalatrpalaglnasplyslysproileasnleuarg
165170175
gluvalleuglyalaphesermetasnasnvalthrargmetleuleu
180185190
glylysglntyrpheglysergluserserglyproglnglualamet
195200205
gluphemethisilethrhisgluleuphetrpleuleuglyvalile
210215220
tyrleuglyasptyrleuproiletrpargtrpvalaspprotyrgly
225230235240
cysglulyslysmetarggluvalglulysargvalaspaspphehis
245250255
serasnileileglugluhisarglysalaarglysasparglysgly
260265270
lysarglysgluglyaspglyaspmetaspphevalaspvalleuleu
275280285
serleuproglygluaspglylysgluhismetaspaspvalgluile
290295300
lysalaleuileglnaspmetilealaalaalathraspthrserala
305310315320
valthrasnglutrpalametalagluvalmetlyshisprohisval
325330335
leuhislysileglnglugluleuaspthrilevalglyproasnarg
340345350
metvalleugluseraspleuprohisleuasntyrleuargcysval
355360365
valarggluthrpheargmethisproalaglypropheleuilepro
370375380
hisgluserleuargalathrthrileasnglytyrhisileproala
385390395400
lysthrargvalpheileasnthrhisglyleuglyargasnthrlys
405410415
iletrpaspasnvalaspglupheargprogluarghistrpproser
420425430
asnglyasnglythrargvalgluileserhisglyvalaspphelys
435440445
ileleupropheseralaglylysarglyscysproglyalaproleu
450455460
glyvalthrleuvalleumetalaleualaargleuphehiscysphe
465470475480
asptrpgluproprolysglyleusercysglyaspvalaspthrarg
485490495
gluvaltyrglymetthrmetprolysalagluproleuilealaile
500505510
alalysproargleualalyshisleutyrasp
515520
<210>22
<211>537
<212>prt
<213>gossypiumraimondii
<220>
<221>peptid
<222>(1)..(537)
<223>proteincypgst(gossypiumraimondii)
<400>22
metileprotyrlysproilephepheproleuaspasnmetgluleu
151015
phethrphealaleualaleuleuvalglyalaleuvalvalasnala
202530
leutrpargtrpargleuasptrplysserleuphelysthrarglys
354045
leuproproglyproproargtrpproilevalglyasnleuleugln
505560
leuserserleuprohisargaspleualaserleucysasplystyr
65707580
glyproleuvaltyrleuargleuglylysvalaspalailethrthr
859095
asnaspproaspileilearggluileleuleuargglnaspgluval
100105110
phealaserargproargthrleualaalavalhisleualatyrgly
115120125
cysglyaspvalalaleualaproleuglyprohistrplysargmet
130135140
argargilecysmetgluhisleuleuthrthrlysargleugluser
145150155160
phealalyshisargalaaspglualaglnhisleuvalargaspval
165170175
seralaargalagluasnglyglnleuvalasnleuarggluvalleu
180185190
glyalaphesermetasnasnvalthrargmetleuleuglyarggln
195200205
tyrpheglyalavalseralaglyproserglualametgluphemet
210215220
hisilethrhisgluleuphetrpleuleuglyvaliletyrleugly
225230235240
asptyrleuproiletrpargtrpvalaspprotyrglycysglulys
245250255
argmetarggluvalglulysargvalaspaspphehisgluargile
260265270
ileglugluhisargargalaarggluleulysasnlysglytyrgly
275280285
lysaspaspasptyrglygluglumetaspphevalaspvalleuleu
290295300
serleuproglygluaspglyasnprohismetaspaspthraspile
305310315320
lysalaleuileglnaspmetilealaalaalathraspthrserala
325330335
valthrasnglutrpthrmetalagluvalilelyshisproargval
340345350
leuarglysileglnaspgluleuaspservalvalglyproasnarg
355360365
metvalasngluseraspleuprohisleuasntyrleuargcysval
370375380
valarggluthrpheargmethisproalaglypropheleuilepro
385390395400
hisgluserleuargalathrthrileasnglyphetyrileproala
405410415
lysthrargvalpheileasnthrhisglyleuglyargasnthrlys
420425430
leutrpaspaspvalgluserpheargprogluarghistrpleuala
435440445
aspglyalaargvalgluileserhisglyalaaspphelysileleu
450455460
propheseralaglylysarglyscysproglyalaproleuglyval
465470475480
thrleuvalleumetalaleualaargleuphehiscyspheasptrp
485490495
alaproglnasnglymetargprogluaspileasnthrmetgluval
500505510
tyrglymetthrmetprolysalagluproleumetalametalalys
515520525
proargleualaasphisvalmetphe
530535
<210>23
<211>526
<212>prt
<213>sorghumbicolor
<220>
<221>peptid
<222>(1)..(526)
<223>proteincypgst(sorghumbicolor)
<400>23
metaspprophevalleuserileleuilecyssertrpilepheval
151015
valvaltyrtrpargargleuasnsermetargleuargleupropro
202530
glyproprothrtrpproilepheglyasnleuleuglnleuserpro
354045
leuprohislysaspphealaargphecysthrlystyrglyproleu
505560
valtyrleuargleuglythrileaspalailethrthraspasppro
65707580
gluvalilearggluileleuileargglnaspgluvalphealaser
859095
argproargthrleualaalavalhisleualatyrglycysglyasp
100105110
valalaleualaproleuglyproasntrplysargmetargargval
115120125
cysmetgluhisleuleuthrthrlysargleugluserphealaala
130135140
hisargalaglnglualagluhisleucysglnphevaltrpalalys
145150155160
serhisserglylysprovalasnleuarggluvalleuglyalaphe
165170175
sermetasnasnvalthrargmetleuleuglylysglntyrphegly
180185190
ileglnseralaglyproglyglualametgluphemethisilethr
195200205
hisgluleuphepheleuleuglyleuiletyrleuglyasptyrleu
210215220
proalatrpargtrpvalaspprotyrglycysglulyslysmetarg
225230235240
aspvalglulyslysvalaspaspphehisglnlysileileaspglu
245250255
hisargargalaargglualalyslysthrargargserserleuasp
260265270
aspaspaspglylysgluaspmetaspphevalaspvalleuleuser
275280285
leuproglygluasnglylysgluhismetaspaspmetgluilelys
290295300
alaleumetglnaspmetilealaalaalathraspthrserserval
305310315320
thrasnglutrpvalmetalagluvalilelysasnproargvalleu
325330335
argargvalglnglugluleuaspalavalileglyargaspargmet
340345350
valalagluseraspleuthrhisleuprotyrleuargcysvalval
355360365
arggluserpheargmethisproalaglypropheleuileprohis
370375380
gluserleulysprothrthrilemetglytyrhisvalproalaarg
385390395400
thrargvalpheileasnthrhisalaleuglyargasnproargval
405410415
trpaspaspvalaspalapheargprogluarghisleuproalaglu
420425430
gluglyalaargvalgluileserhisleuproaspphelysileleu
435440445
propheseralaglylysarglyscysproglyalaproleuglyval
450455460
alaleuvalleumetalaleualaargleuphehiscyspheasptrp
465470475480
serproproaspglyleuargprogluaspvalaspthrglngluval
485490495
tyrglymetthrmetprolysalathrproleuvalalavalalathr
500505510
proargleuproprohisleutyrglyglyglyseralaser
515520525
<210>24
<211>41
<212>dna
<213>artificialsequence
<220>
<223>sxn2151s01-markersequenzzumanzeigendesgst-locusinbeta
vulgaris
<400>24
aacactcatgtaataagatgggtgagtgagtagcaaaaaaa41
<210>25
<211>41
<212>dna
<213>artificialsequence
<220>
<223>sxn2151s01-markersequenzzumanzeigendeskws2320-
referenzgenotyps
<400>25
aacactcatgtaataagatgagtgagtgagtagcaaaaaaa41
<210>26
<211>41
<212>dna
<213>artificialsequence
<220>
<223>sle3305s02-markersequenzzumanzeigendesgst-locusinbeta
vulgaris
<400>26
aaagtctagagtaaattgaggttgcagtggagtgggaagtc41
<210>27
<211>41
<212>dna
<213>artificialsequence
<220>
<223>sle3305s02-markersequenzzumanzeigendeskws2320-
referenzgenotyps
<400>27
aaagtctagagtaaattgagattgcagtggagtgggaagtc41
- 还没有人留言评论。精彩留言会获得点赞!