苹果酸脱氢酶的制作方法
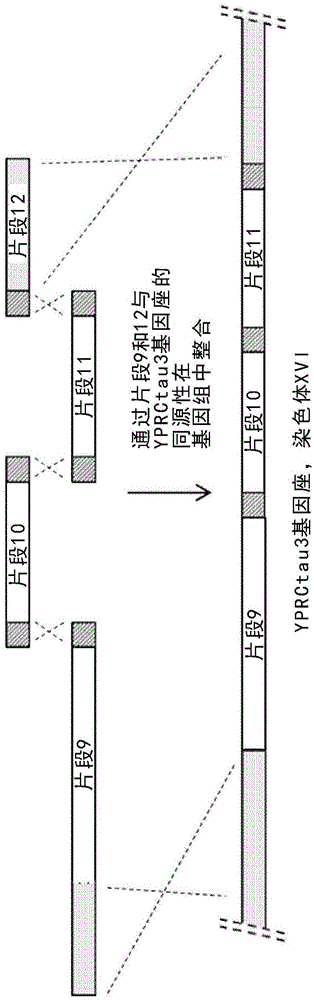
本发明涉及能够生产二羧酸的重组宿主细胞,以及使用所述重组宿主细胞生产二羧酸的方法。
背景技术:
:4-碳二羧酸苹果酸、延胡索酸和琥珀酸是大量化学品的潜在前体。例如,琥珀酸可以被转变为1,4-丁二醇(bdo)、四氢呋喃和γ-丁内酯。衍生自琥珀酸的另一产物是通过连接琥珀酸和bdo制造的聚酯聚合物。用于工业用途的琥珀酸主要是通过马来酸或马来酸酐的催化氢化由丁烷通过石油化学生产的。这些方法被认为是对环境有害且昂贵的。琥珀酸的发酵生产被视为生产琥珀酸的一种吸引人的替代方法,其中可以使用可再生的原料作为碳源。已经对(重组)酵母中c4-二羧酸的发酵生产进行了若干研究。例如,ep2495304公开了适合琥珀酸生产的重组酵母,其中编码丙酮酸羧化酶、磷酸烯醇式丙酮酸羧激酶、苹果酸脱氢酶、延胡索酸酶、延胡索酸还原酶和琥珀酸转运体的基因被遗传修饰。尽管在宿主细胞(例如酵母)中发酵生产二羧酸已经取得了改进,但仍需要进一步改进的用于发酵生产二羧酸的宿主细胞。发明概述本发明涉及重组宿主细胞,其能够生产二羧酸并且包含具有苹果酸脱氢酶(mdh)活性的突变多肽。出乎意料地发现:与包含参照mdh多肽的宿主细胞所产生的二羧酸的量相比,根据本发明的宿主细胞产生增加量的二羧酸,所述参照mdh多肽通常是nad(h)-依赖型苹果酸脱氢酶(ec1.1.1.37)。本发明因此提供了一种重组宿主细胞,其能够生产二羧酸并且包含编码具有苹果酸脱氢酶活性的突变多肽的核酸序列,其中所述突变多肽包含这样的氨基酸序列,当与包含seqidno:39所示序列的苹果酸脱氢酶比对时,所述氨基酸序列包含对应于seqidno:39中的第34位氨基酸的氨基酸残基的一个突变(例如一个替换)。所述具有苹果酸脱氢酶活性的突变多肽可以进一步包含一个或多个另外的突变(例如替换)。特别地,具有苹果酸脱氢酶活性的突变多肽可以进一步包含对应于seqidno:39中的第35、36、37、38、39和/或40位氨基酸中的任何的一个或多个另外的突变(例如替换)。本发明还提供了一种重组宿主细胞,其能够生产二羧酸并且包含编码具有苹果酸脱氢酶活性的突变多肽的核酸序列,其中与参照mdh多肽相比,所述突变多肽的nadp(h)-依赖型活性与nad(h)-依赖型活性之比提高,所述参照mdh多肽通常是nad(h)-依赖型苹果酸脱氢酶(ec1.1.1.37)。在所述实施方式中,所述突变多肽可以是包含这样的氨基酸序列的突变多肽,当与包含seqidno:39所示序列的苹果酸脱氢酶比对时,所述氨基酸序列包含对应于seqidno:39中的第34位氨基酸的氨基酸残基的一个突变(例如一个替换)。本发明还提供了:-根据本发明的重组宿主细胞,其中编码具有苹果酸脱氢酶活性的突变多肽的核酸序列在胞质中表达,并且具有苹果酸脱氢酶活性的突变多肽在胞质中有活性。-根据本发明的重组宿主细胞,其中所述重组宿主细胞具有从磷酸烯醇或丙酮酸盐/酯到琥珀酸盐/酯的活性还原性三羧酸(tca)通路。-生产二羧酸的方法,其中所述方法包括在适于生产二羧酸的条件下发酵本发明的重组宿主细胞。二羧酸可以是琥珀酸、苹果酸和/或延胡索酸。所述方法可以进一步包括从发酵培养基中回收二羧酸。附图简介图1示出了片段9至12整合的示意图。片段9至12中所示的阴影线部分表示导致(如同源区之间的虚线交叉所示的)重组事件的独特同源重叠区。片段9的5’端和片段12的3’端(由片段9和12中的灰色区域表示)与染色体16上的yprctau3基因座同源。同源重组导致片段10和11整合到yprctau3基因座中。图2示出了片段1至8和片段113整合的示意图。片段1至8和113中所示的阴影线部分表示导致(如同源区之间的虚线交叉所示的)重组事件的独特同源重叠区。片段1和片段113与染色体xi上的int59基因座同源,同源重组导致片段2至8整合到int59基因座中。图3示出了片段13、114、115、15和16整合的示意图。片段13、114、115、15和16中所示的阴影线部分表示导致(如同源区之间的虚线交叉所示的)重组事件的独特同源重叠区。片段13和片段16与染色体xv上的int1基因座同源,同源重组导致片段114、115和15整合到int1基因座中。图4示出了片段13、14、16和片段17-110之一整合的示意图。片段13、14、16和片段17-110中所示的阴影线部分表示导致(如同源区之间的虚线交叉所示的)重组事件的独特同源重叠区。片段13和片段16与染色体xv上的int1基因座同源,同源重组导致片段14和片段17-112之一整合到int1基因座中。图5a:培养suc-1112转化体后在生产培养基的上清液中测量的平均苹果酸滴度,所述suc-1112转化体表达磷酸烯醇式丙酮酸羧激酶(pcka)、丙酮酸羧化酶(pyc2)、苹果酸脱氢酶(mdh3)、延胡索酸酶(fumr和fumb)、二羧酸转运体(dct_02)并用参照苹果酸脱氢酶(seqidno:39)或突变的苹果酸脱氢酶转化,与参照序列相比,所述突变的苹果酸脱氢酶在表1中所示的氨基酸位置含有突变。苹果酸滴度如“通用的材料和方法”中所述进行测量,其代表从三个独立克隆获得的平均值。图5b:在菌株suc-1112中表达的mdh突变体的nadh特异性苹果酸脱氢酶(mdh)活性(具体突变请参见表1)。显示的是活性,描述为δa340/min/mg总蛋白。该值是负的,因为mdh依赖型nadh氧化导致340nm处的吸光度降低。负值越大表示活性越高。如实施例5中所述测量活性。图5c:在菌株suc-1112中表达的mdh突变体的nadph特异性苹果酸脱氢酶(mdh)活性(具体突变请参见表1)。显示的是活性,描述为δa340/min/mg总蛋白。该值是负的,因为mdh依赖型nadph氧化导致340nm处的吸光度降低。负值越大表示活性越高。如实施例5中所述测量活性。图5d:在菌株suc-1112中表达的mdh突变体的nadph依赖型活性:nadh依赖型活性的比值(具体突变请参见表1)。测量活性并如实施例5中所述测定比值。虚线表示nadph:nadh的比值为1.0。图6示出了片段116、117、118和片段119整合的示意图。片段116、117、118和片段119中所示的阴影线部分表示导致重组事件的独特同源重叠区,如同源区之间的虚线交叉所示。片段116和片段119与染色体xv上的int1基因座同源,同源重组导致片段117和片段118整合到int1基因座中。图7示出了片段1-5、124和片段120、121、122或123整合的示意图。片段中所示的阴影线部分表示导致(如同源区之间的虚线交叉所示的)重组事件的独特同源重叠区。片段1和片段124与染色体xi上的int59基因座同源,同源重组导致片段2至5和120、121、122或123整合到int59基因座中。序列表说明seqidno:1示出了片段2(图2)的核苷酸序列,其包括针对在saccharomycescerevisiae中表达而经密码子对优化的来自actinobacillussuccinogenes的pep羧激酶。seqidno:2示出了片段3(图2)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自s.cerevisiae的丙酮酸羧化酶(pyc2)。seqidno:3示出了片段4(图2)的pcr模板的核苷酸序列,其包括在s.cerevisiae中有功能的kanmx选择标记。seqidno:4示出了片段5(图2)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自aspergillusniger的推定的二羧酸转运体。seqidno:5示出了片段6(图2)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自s.cerevisiae的苹果酸脱氢酶(mdh3)。seqidno:6示出了片段7(图2)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自escherchiacoli的延胡索酸酶(fumb)。seqidno:7示出了片段8(图2)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自trypanosomabrucei的延胡索酸还原酶(frdg)。seqidno:8示出了来自trypanosomabrucei的延胡索酸还原酶(frdg)的氨基酸序列。seqidno:9示出了用于生成片段1(图2)的引物的核苷酸序列。seqidno:10示出了用于生成片段1(图2)的引物的核苷酸序列。seqidno:11示出了用于生成片段2(图2)的引物的核苷酸序列。seqidno:12示出了用于生成片段2(图2)的引物的核苷酸序列。seqidno:13示出了用于生成片段3(图2)的引物的核苷酸序列。seqidno:14示出了用于生成片段3(图2)的引物的核苷酸序列。seqidno:15示出了用于生成片段4(图2)的引物的核苷酸序列。seqidno:16示出了用于生成片段4(图2)的引物的核苷酸序列。seqidno:17示出了用于生成片段5(图2)的引物的核苷酸序列。seqidno:18示出了用于生成片段5(图2)的引物的核苷酸序列。seqidno:19示出了用于生成片段6(图2)以及片段120、121、122和123(图7)的引物的核苷酸序列。seqidno:20示出了用于生成片段6(图2)以及片段120、121、122和123(图7)的引物的核苷酸序列。seqidno:21示出了用于生成片段7(图2)的引物的核苷酸序列。seqidno:22示出了用于生成片段7(图2)的引物的核苷酸序列。seqidno:23示出了用于生成片段8(图2)的引物的核苷酸序列。seqidno:24示出了用于生成片段8(图2)的引物的核苷酸序列。seqidno:25示出了用于生成片段13(图3)的引物的核苷酸序列。seqidno:26示出了用于生成片段13(图3)的引物的核苷酸序列。seqidno:27示出了用于生成片段14(图4)的引物的核苷酸序列。seqidno:28示出了用于生成片段115(图3)和片段14(图4)的引物的核苷酸序列。seqidno:29示出了用于生成片段15(图3)和片段17至110(图4)的引物的核苷酸序列。seqidno:30示出了用于生成片段15(图3)和片段17至110(图4)的引物的核苷酸序列。seqidno:31示出了片段15(图3)和片段120(图7)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的编码seqidno:39的核苷酸序列。seqidno:32示出了用于生成片段16(图3)的引物的核苷酸序列。seqidno:33示出了用于生成片段16(图3)的引物的核苷酸序列。seqidno:34示出了片段9(图1)的核苷酸序列,其包括针对在saccharomycescerevisiae中表达而经密码子对优化的来自rhizopusoryzae的延胡索酸酶。seqidno:35示出了片段10(图1)的核苷酸序列,其包括cre重组酶的5’部分。seqidno:36示出了片段11(图1)的核苷酸序列,其包括cre重组酶的3’部分。seqidno:37示出了片段12(图1)的核苷酸序列,其包括与yprctau3基因座同源的区域。seqidno:38示出了片段115(图3)、片段14(图4)和片段117(图6)的pcr模板的核苷酸序列,其包含诺尔丝菌素选择标记。seqidno:39示出了缺少3c-末端过氧化物酶体靶向序列的来自s.cerevisiae的苹果酸脱氢酶(mdh3)蛋白质的氨基酸序列。seqidno:40示出了用于生成片段113(图2)的引物的核苷酸序列。seqidno:41示出了用于生成片段113(图2)的引物的核苷酸序列。seqidno:42示出了片段114(图3)的核苷酸序列,其包括针对在saccharomycescerevisiae中表达而经密码子对优化的来自s.cerevisiae的zwf1的表达盒。seqidno:43示出了用于生成片段114(图3)的引物的核苷酸序列。seqidno:44示出了用于生成片段114(图3)的引物的核苷酸序列。seqidno:45示出了用于生成片段115(图3)的引物的核苷酸序列。seqidno:46示出了来自s.cerevisiae的丙酮酸羧化酶蛋白质的氨基酸序列。seqidno:47示出了在第120-122位具有egy到daf修饰的来自actinobacillussuccinogenes的磷酸烯醇式丙酮酸羧激酶的氨基酸序列。seqidno:48示出了来自escherichiacoli的延胡索酸酶(fumb)的氨基酸序列。seqidno:49示出了缺少前23个n-末端氨基酸的来自rhizopusoryzae的延胡索酸酶的氨基酸序列。seqidno:50示出了来自aspergillusniger的推定的二羧酸转运体的氨基酸序列。seqidno:51示出了来自kluyveromyceslactis的异柠檬酸裂解酶的氨基酸序列。seqidno:52示出了缺少3c-末端过氧化物酶体靶向序列的saccharomycescerevisiae过氧化物酶体苹果酸合酶(mls1)氨基酸序列的氨基酸序列。seqidno:53示出了包括过氧化物酶体靶向序列skl的来自s.cerevisiae的苹果酸脱氢酶(mdh3)蛋白质的氨基酸序列。seqidno:54示出了用于生成片段116(图6)的引物的核苷酸序列。seqidno:55示出了用于生成片段116(图6)的引物的核苷酸序列。seqidno:56示出了用于生成片段117(图6)的引物的核苷酸序列。seqidno:57示出了用于生成片段117(图6)的引物的核苷酸序列。seqidno:58示出了用于生成片段119(图6)的引物的核苷酸序列。seqidno:59示出了用于生成片段119(图6)的引物的核苷酸序列。seqidno:60示出了用于生成片段118(图6)的引物的核苷酸序列。seqidno:61示出了用于生成片段118(图6)的引物的核苷酸序列。seqidno:62示出了片段118(图6)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的来自trypanosomabrucei的延胡索酸还原酶(frdg)的编码序列。seqidno:63示出了用于生成片段124(图7)的引物的核苷酸序列。seqidno:64示出了片段121(图7)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的s.cerevisiaemdh3突变体mut_014的编码序列。seqidno:65示出了片段122(图7)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的s.cerevisiaemdh3突变体mut_015的编码序列。seqidno:66示出了片段123(图7)的核苷酸序列,其包括针对在s.cerevisiae中表达而经密码子对优化的s.cerevisiaemdh3突变体mut_034的编码序列。seqidno:67示出了来自arabidopsisthaliana的延胡索酸酶的氨基酸序列。发明详述在本说明书和所附的权利要求书中,词语“包括”、“包含”和“具有”及其变形应被理解为“包含在内”。也就是说,在语境允许的情况下,这些词语意图传达的意思是:可以包括其它没有明确列举的要素或整体。本文中不使用数量词修饰时指的是一个/种或多于一个/种(即一个/种或至少一个/种)对象。例如,“要素”可意味着一个/种要素或多于一个/种要素。还原性tca通路是微生物可产生二羧酸的主要通路之一。近年来,其已被证明是微生物生产二羧酸例如琥珀酸的最佳经济选择。还原性tca通路包括两个需要消耗还原力的反应;即苹果酸脱氢酶反应(草酰乙酸盐/酯还原为苹果酸盐/酯)和延胡索酸还原酶反应(延胡索酸盐/酯还原为琥珀酸盐/酯)。苹果酸脱氢酶(mdh)使用nad或nadp作为辅因子(也统称为nad(p))催化苹果酸盐/酯可逆地转变为草酰乙酸盐/酯。mdh是一种相当普遍存在的酶,在许多代谢通路中起着至关重要的作用,所述代谢通路包括三羧酸循环、氨基酸合成、糖异生、维持氧化/还原平衡和代谢应激。根据mdh对辅因子的偏好,mdh可被分为nad(h)-依赖型mdh(nad-mdh)(ec1.1.1.37)和nadp(h)-依赖型mdh(nadp-mdh)(ec1.1.1.82)。大多数细菌和古细菌mdh是nad-mdh。真核mdh同种型均为nad-mdh,包括线粒体mdh、胞质mdh、乙醛酸循环体mdh和过氧化物酶体mdh,例外是叶绿体nadp-mdh,其是将还原等同物从叶绿体基质转移到胞质所必需的。在酵母saccharomycescerevisiae中,已鉴定出了三种内源性苹果酸脱氢酶同工酶,即mdh1、mdh2和mdh3。它们位于线粒体(mdh1)、胞质(mdh2)和过氧化物酶体(mdh3)中,并且均被表征为nad(h)-依赖型mdh(ec1.1.1.37)。对苹果酸脱氢酶家族中的nad(p)-结合结构域的研究揭示了rossmann折叠的保守βb-αc基序。脱氢酶区分nadp(h)的能力在于该βb-αc基序的氨基酸序列,其已被预测为辅因子特异性的主要决定簇。例如,在s.cerevisiae过氧化物酶体nad-mdh(mdh3)中,nad-结合基序包括第34-40位氨基酸残基,其被发现对于辅因子结合和特异性是重要的。在本发明的上下文中,出乎意料地发现:当在能够生产二羧酸的重组宿主细胞中(过)表达时,具有mdh活性的多肽的保守nad结合基序中的一组特定突变赋予提高的二羧酸生产。也就是说,与(过)表达参照mdh多肽的重组宿主细胞相比,在重组宿主细胞中(过)表达具有mdh活性的所述突变多肽通常导致提高的二羧酸生产;“参照mdh多肽”通常是nad-mdh(ec1.1.1.37)。同时,已显示:与所述参照mdh多肽相比,在保守nad-结合基序中具有至少一个突变的所述突变多肽的nadp(h)-依赖型活性与nad(h)-依赖型活性之比提高。出乎意料地,本发明的发明人进一步显示了:为了获得二羧酸生产的提高,nadp(h)-依赖型活性不必高于nad(h)-依赖型活性。因此,本发明的一个目标是提供一种重组宿主细胞,其能够生产二羧酸并且包含具有苹果酸脱氢酶(mdh)活性的突变多肽。在一个实施方式中,具有苹果酸脱氢酶活性的突变多肽包含这样的氨基酸序列,当与包含seqidno:39所示序列的苹果酸脱氢酶比对时,所述氨基酸序列包含对应于seqidno:39中的第34位氨基酸的氨基酸残基的一个突变。换句话说,所述突变多肽包含在对应于seqidno:39中的第34位的位置处出现的氨基酸残基的一个突变。在本发明的一个优选实施方式中,对应于第34位氨基酸(参照seqidno:39限定)的氨基酸的突变是替换。更优选地,对应于第34位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括苏氨酸(t)、丝氨酸(s)、甘氨酸(g)、丙氨酸(a)和脯氨酸(p)。优选的小氨基酸是甘氨酸(g)和丝氨酸(s)。在本发明的上下文中,“重组宿主细胞”或“经遗传修饰的宿主细胞”是已通过重组dna技术引入包含编码具有苹果酸脱氢酶活性的突变多肽的核酸序列的核酸、核酸构建体或载体的宿主细胞。在本文中,“具有苹果酸脱氢酶(mdh)活性的突变多肽”可被称为“突变的/型苹果酸脱氢酶”、“mdh突变体”、“mdh突变多肽”、“突变体”、“突变多肽”等。在本文中,“苹果酸脱氢酶活性”是将草酰乙酸转变为苹果酸的活性:苹果酸+受体<=>草酰乙酸+还原的受体。术语“多肽”在本文中用于含有多于约7个氨基酸残基的链。本文所有的多肽序列都是从左到右书写,并从氨基末端到羧基末端的方向书写。本文所用的氨基酸单字母代码是本领域公知的,可以在sambrook等(molecularcloning:alaboratorymanual,第二版coldspringharborlaboratory,coldspringharborlaboratorypress,coldspringharbor,ny,1989)中找到。在本发明的上下文中,“突变(型)”多肽被定义为通过引入一个或多个突变而获得的多肽。所述突变可选自替换、添加和缺失。术语“替换”在本文中是指多肽序列中的氨基酸残基被另一个氨基酸残基替代。“突变(型)”多肽、“突变的”多肽和“基因工程”多肽具有相同的含义并可互换使用。在本文中,“对应的位置”是指seqidno:39和与seqidno:39同源的序列之间的氨基酸序列比对中的垂直栏,对应于seqidno:39中的特定位置,并显示出在其他比对的同源物中的该位置出现的氨基酸。在本发明的上下文中,“对应的突变”是指在seqidno:39中“对应的位置”出现的氨基酸残基的突变。例如,“对应的替换”是指在seqidno:39中“对应的位置”出现的氨基酸残基被另一个氨基酸残基替换。在一些另外的实施方式中,具有苹果酸脱氢酶活性的突变多肽可以进一步包含对应于seqidno:39中的第35、36、37、38、39和/或40位氨基酸中的任何的一个或多个的另外的突变。所述突变通常选自替换、添加和缺失。更优选地,一个或多个另外的突变是替换。对应于第35位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括苏氨酸(t)、丝氨酸(s)、甘氨酸(g)、丙氨酸(a)和脯氨酸(p)。或者,对应于第35位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为疏水性氨基酸,例如异亮氨酸(i)。对应于第35位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为丝氨酸(s)或异亮氨酸(i)。对应于第36位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为极性氨基酸。合适的极性氨基酸包括精氨酸(r)、谷氨酰胺(q)、谷氨酸(e)和丝氨酸(s)。或者,对应于第36位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为小氨基酸,例如丙氨酸(a)或脯氨酸(p)。对应于第36位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为精氨酸(r)、谷氨酰胺(q)、谷氨酸(e)、丝氨酸(s)、丙氨酸(a)或脯氨酸(p)。对应于第37位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括甘氨酸(g)、天冬酰胺(n)和丙氨酸(a)。或者,对应于第37位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为极性氨基酸,例如精氨酸(r)或谷氨酰胺(q)。对应于第37位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为(g)、天冬酰胺(n)、丙氨酸(a)、丙氨酸精氨酸(r)或谷氨酰胺(q)。对应于第38位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括缬氨酸(v)、苏氨酸(t)、丝氨酸(s)、甘氨酸(g)、丙氨酸(a)和脯氨酸(p)。对应于第38位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为丙氨酸(a)、缬氨酸(v)、苏氨酸(t)或丝氨酸(s)。对应于第39位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括脯氨酸(p)。或者,对应于第39位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为疏水性氨基酸,例如赖氨酸(k)、苯丙氨酸(f)或亮氨酸(l)。或者,对应于第39位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为极性氨基酸,例如谷氨酸(e)。对应于第39位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为谷氨酸(e)、赖氨酸(k)、苯丙氨酸(f)、亮氨酸(l)或脯氨酸(p)。对应于第40位氨基酸(参照seqidno:39限定)的氨基酸的替换通常是替换为小氨基酸。合适的小氨基酸包括甘氨酸(g)。或者,对应于第40位氨基酸(参照seqidno:39限定)的氨基酸的替换是替换为极性氨基酸,例如谷氨酰胺(q)。对应于第40位氨基酸(参照seqidno:39限定)的氨基酸的优选替换是替换为甘氨酸(g)或谷氨酰胺(q)。以上各种类型的氨基酸参照例如betts和russell,bioinformaticsforgeneticists,barnes和gray编,wiley2003进行分类。更详细地,在本发明的上下文中,具有苹果酸脱氢酶活性的突变多肽包含参照seqidno:39限定的第34位的g或s;以及,任选的参照seqidno:39限定的第35位的i或s;和/或参照seqidno:39限定的第36位的r、q、a、e、p或s;和/或参照seqidno:39限定的第37位的a、n、g、r或q;和/或参照seqidno:39限定的38位的a、v、t或s;和/或参照seqidno:39限定的第39位的e、k、p、f或l;和/或参照seqidno:39限定的第40位的g或q。在一个具体实施方式中,具有苹果酸脱氢酶活性的突变多肽包含在第34位的小氨基酸(参照seqidno:39限定)和在第36位的小氨基酸或极性氨基酸(参照seqidno:39限定)。在所述实施方式中,第34位的优选的小氨基酸可选自g或s。在所述实施方式中,第36位的优选的小氨基酸或极性氨基酸可选自r、q、a、e、p或s。任选地,在所述实施方式中,突变多肽包含参照seqidno:39限定的第35位的i或s;和/或参照seqidno:39限定的第37位的a、n、g、r或q;和/或参照seqidno:39限定的第38位的a、v、t或s;和/或参照seqidno:39限定的第39位的e、k、p、f或l;和/或参照seqidno:39限定的第40位的g或q。具有mdh活性的突变多肽还可以包含除上文限定的七个位置之外的另外的突变,例如,一个或多个另外的替换、添加或缺失。具有mdh活性的突变多肽可包含不同类型的此类修饰的组合。具有mdh活性的突变多肽可包含一个、两个、三个、四个、至少5个、至少10个、至少15个、至少20个、至少25个、至少30个或更多个这样的修饰(它们可以全部是相同类型的修饰或可以是不同类型的修饰)。通常,另外的修饰可以是替换。在另外的实施方式中,具有苹果酸脱氢酶活性的突变多肽参照表1(实施例4)限定,并且其中对应于seqidno:39中的第34位氨基酸的氨基酸残基选自甘氨酸(g)或丝氨酸(s)。也就是说,与合适的参照序列(例如seqidno:39中所示的参照序列)相比,突变多肽可以包含表1中所示的替换的任何组合,并且其中对应于seqidno:39中的第34位氨基酸的氨基酸残基选自甘氨酸(g)或丝氨酸(s)。通常,突变多肽可以包含seqidno:39的序列,其在第34位具有一个替换,并且任选地在第35、36、37、38、39和/或40位具有一个或多个替换。也就是说,突变多肽在第34位具有不同于天冬氨酸的氨基酸,并且任选地在第35位具有不同于异亮氨酸的氨基酸,且/或在第36位具有不同于精氨酸的氨基酸,且/或在第37位具有不同于丙氨酸的氨基酸,且/或第38位具有不同于丙氨酸的氨基酸,且/或在第39位具有不同于谷氨酸的氨基酸,且/或在第40位具有不同于甘氨酸的氨基酸。此外,通常,突变多肽可以包含seqidno:39的序列,其在第34位具有一个替换,在第36位具有一个替换,并且任选地在第35、37、38、39和/或40位具有一个或多个替换。也就是说,突变多肽在第34位具有不同于天冬氨酸的氨基酸,在第36位具有不同于精氨酸的氨基酸,并且任选地在第35位具有不同于异亮氨酸的氨基酸,且/或在第37位具有不同于丙氨酸的氨基酸,且/或第38位具有不同于丙氨酸的氨基酸,且/或在第39位具有不同于谷氨酸的氨基酸,且/或在第40位具有不同于甘氨酸的氨基酸。在本发明的一个单独的实施方式中,与参照mdh多肽相比,具有苹果酸脱氢酶活性的突变多肽的nadp(h)-依赖型活性与nad(h)-依赖型活性之比提高。在所述实施方式中,所述突变多肽可以是包含这样的氨基酸序列的突变多肽,当与包含seqidno:39所示序列的苹果酸脱氢酶比对时,所述氨基酸序列包含对应于seqidno:39中的第34位氨基酸的氨基酸残基的一个突变(例如一个替换)。关于所述突变多肽的氨基酸序列的其他实施方式如上所述。在本发明的上下文中,具有苹果酸脱氢酶活性的参照多肽(也称为“参照mdh多肽”)可以是nad-mdh(ec1.1.1.37)。具有苹果酸脱氢酶活性的参照多肽可以是来自微生物来源例如酵母(例如saccharomycescerevisiae)的苹果酸脱氢酶。具有seqidno:39所示氨基酸序列的苹果酸脱氢酶可以是具有mdh活性的合适参照多肽。表述“nadp(h)-依赖型活性与nad(h)-依赖型活性之比提高”在本文中通常是指:与参照mdh多肽相比,例如与seqidno:39相比,突变多肽显示出nadp(h)-依赖型活性与nad(h)-依赖型活性之比提高的性质。也就是说,与参照多肽相比,突变多肽可以显示出nadp(h)-依赖型活性与nad(h)-依赖型活性的比值提高。在实施例5中,该比值也被称为“nadph特异性:nadh特异性的比值”。在本发明的上下文中,术语“nadp(h)-依赖型活性”和“nadp(h)-特异性活性”在本文中具有相同含义并可互换使用。这同样适用于术语“nad(h)-依赖型活性”和“nad(h)-特异性活性”。术语“nadp(h)-依赖型活性”在本文中是指酶使用nadp(h)作为氧化还原辅因子的性质。酶的nadp(h)-依赖型活性可以通过酶活性试验(例如实施例5中所述)测定。术语“nad(h)-依赖型活性”在本文中是指酶使用nad(h)作为氧化还原辅因子的性质。酶的nad(h)-依赖型活性可以通过酶活性试验(例如实施例5中所述)测定。平均nadph特异性:nadh特异性比值的提高值可以指示,例如,降低的nad(h)-依赖型活性、提高的nadp(h)-依赖型活性或两者的组合。在一些情况下,与参照mdh相比,使用具有相似或提高的nad(h)-依赖型活性的mdh突变体可以获得所述比值的提高值。在后一情况下,mdh突变体可显示出提高的nad(h)-依赖型活性和提高的nadp(h)-依赖型活性。就改变的辅因子依赖型而言,突变mdh多肽通常具有改变的mdh活性。该nad(h)-依赖型活性或nadp(h)-依赖型活性可以彼此独立地改变,例如降低至少10%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或至少99%。或者,该性质可以提高至少10%、至少25%、至少50%、至少70%、至少100%、至少200%、至少500%、至少700%、至少1000%、至少3000%、至少5000%或至少6000%。在一个具体实施方式中,突变mdh多肽的nad(h)-依赖型活性和nadp(h)-依赖型活性均提高。所述突变mdh多肽的nad(h)-依赖型活性提高至少10%、至少25%、至少50%、至少70%、至少100%、至少200%或至少300%。所述突变mdh多肽的nadp(h)-依赖型活性提高至少10%、至少25%、至少50%、至少70%、至少100%、至少200%、至少500%、至少700%、至少1000%、至少3000%、至少5000%或至少6000%。在另一个实施方式中,突变mdh多肽的nad(h)-依赖型活性大约相同或降低至多5%、至多10%、至多20%、至多30%、至多40%、至多50%、至多60%、至多70%、至多80%,并且所述突变mdh多肽的nadp(h)-依赖型活性提高至少10%、至少25%、至少50%、至少70%、至少100%、至少200%、至少500%、至少700%、至少1000%、至少3000%、至少5000%或至少6000%。在另一个实施方式中,突变型mdh多肽的nadph特异性:nadh特异性的比值增加至少10%、至少50%、至少100%、至少200%、至少500%、至少1000%、至少3000%、至少5000%、至少7000%或至少8000%。在该上下文中百分比的降低或提高表示与参照mdh多肽(例如seqidno:39的参照mdh多肽)相比的百分比降低或提高。本领域技术人员公知如何测量这样的百分比变化–其是如实施例中所述测量的参照mdh和突变体mdh的活性(例如nad(h)-依赖型活性或nadp(h)-依赖型活性)的比较。在本发明的上下文中,本所述的mdh突变多肽可以是突变型nad(p)-苹果酸脱氢酶,例如突变型线粒体nad-mdh、突变型胞质nad-mdh、突变型乙醛酸循环体nad-mdh、突变型过氧化物酶体nad-mdh或突变型叶绿体nadp-mdh。也就是说,具有苹果酸脱氢酶活性的突变多肽可以通过在下述nad(p)-苹果酸脱氢酶中引入一个或多个突变来获得,所述nad(p)-苹果酸脱氢酶例如线粒体nad-mdh、胞质nad-mdh、乙醛酸循环体nad-mdh、过氧化物酶体nad-mdh或叶绿体nadp-mdh,(后者被称为用于引入所述一个或多个突变型的模板mdh多肽。优选地,具有苹果酸脱氢酶活性的突变多肽是突变型nad(h)-苹果酸脱氢酶(ec1.1.1.37)。更优选地,具有苹果酸脱氢酶活性的突变多肽是突变型过氧化物酶体nad(h)-苹果酸脱氢酶。甚至更优选地,具有苹果酸脱氢酶活性的突变多肽是来自酵母或真菌的突变型nad(h)-苹果酸脱氢酶。甚至更优选地,具有苹果酸脱氢酶活性的突变多肽是来自酵母或真菌的突变型nad(h)-苹果酸脱氢酶,所示酵母或真菌例如s.cerevisiae、torulasporadelbrueckii、zygosaccharomycesbailii、naumovozymacastellii、naumovozymadairenensis、lachancealanzarotensis、zygosaccharomycesrouxii、kazachstaniaafricana、candidatropicalis、kluyveromycesmarxianus、scheffersomycesstipites、talaromycesmarneffei、rasamsoniaemersonii、aspergillusniger或trametesversicolor。以下uniprot数据库代码是指合适的酵母和真菌模板mdh多肽(http://www.uniprot.org):e7ngh7、g8zxs3、g0v668、w0vui8、g0wb63、a0a0w0d4x6、a0a0c7mme9、c5dq42、c5di45、h2aww6、a0a090c493、j7r0c8、q6cjp3、q759m4、i2h037、c5m546、a7tl95、a0a0l0p3g3、q6bm17、s8aw17、v5fmv2、a3lw84、a0a109uzs1、g8bvw8、g8bj12、m3hpk4、a0a093uw53、b8mtp0、a0a0f4ypr0、c8v0h6、w6qnu3、a5dz33、u1gat6、g3b7s5、c4jpi7、a0a0f8uzy9、q4wdm0、a0a093upx3、b8nd04、a0a0m9vri4、g7xz98、q5a5s6、m7s9e4、e4uyx5、a5de02、a0a0j7bij5、a0a017ski1、g8y7a1、a0a0g2efq2、r7s165、i1rfm4、r1evc8、u4l6k9、a0a0l0p507、w7hm94、a5dgy9、f2qy33、a0a0g2ja24、upi000462180c、c7z9w6、e5aaq2、b2vvr8、a0a0h2rcv1、a8q524、a0a0e9n879、n1ja02、a0a0d1zee3、j5t1x5、w6my07、c4y826、g3aja2、g9n6g5、a0a0k8l6l9、a3gh28、a8p7w6、k5w0t4、g1xt67、a0a0b7k175、b8mtp5、a0a0d1zas7、a0a0c3bqc4、k5y2q9、a0a0c3dsw4、a0a068s518、w2rpl2、a0a0h5c453、a0a074wkg5、g8jrx4、a0a0u1m134、h6c0v9、a0a0h5bz30、m2uxr7、a0a0c9yjv6、a0a0c3s6t1、w1qk02、a0a0c2yfc4、a0a061aj54、a0a086t183、w2rvt1、upi0004623914、a0a0c9x7u1、upi0001f26169、g7e054、a0a0c2s9t3、a0a067nix1、l8fpm0、g3alw4、a0a0d0ayx2、g0vjg3、a0a0d2ngy0、c1glb8、w9x415、a0a0d0cde8、s7q8g0、a0a0c9tb51、r7yp89、a0a0c2w4h8、upi000455fa04、a0a067nl73、a0a067stp4、w9wwp5、a0a0d7a9t7、a0a0d6r2e1、m2ylx9、g0w7d4、n1qj61、g4try5、f0xj10、a0a063bqq6、a0a061hbx5、a0a0a1pct2、m1wic4、a8qaq2、a0a0c3n631、f2qtl7、a0a060s7u3、a0a0l0htq9、q0cky1、a0a0c2zj90、k5w527、i4y5c3、r7yxz2、f9xi12、a0a061j968、f9xl74、a0a0d0bew9、a0a0c9wb72、f7wa21、a8nj67、m2p8m6、w4khw3、a0a0l1i2t4、upi0004449a9d、p83778、c4y9q7、a0a0d7bhv7、a0a068rwx9、m2ywq3、a0a137qv51、a0a0d0b6c2、i1bqq7、s3da07、q4dxl5、g8y022、a0a0c9w9b3、a0a0l6wj49、a0a0l9sl52、d5ga85、a0a0n1j4z6、j7s1g2、a0a0f4x5c6、q6cik3、a0a067qnn0、q0ugt7、f8nd69、u5him0、j3nkc7、a0a061atz7、a5dsy0、q9y750、upi0004f4119a、a0a086tl69、a0a0j0xss4、upi0003f496e1、upi000455f0ea、s7rxx1、a0a067nag9、a0a0b7n3m5、e6zkh0、c8v1v3、a7ufi6、t5aem1、a0a072pga9、a0a094ekh6、s8adx4、g8c073、q6bxi8、g2r9i6和u9tul6。甚至更优选地,具有苹果酸脱氢酶活性的突变多肽是突变型s.cerevisiae过氧化物酶体nad-mdh(mdh3)。另外,在本发明的重组宿主细胞中,具有苹果酸脱氢酶活性的突变多肽可以是同源或异源nad(p)-苹果酸脱氢酶的突变体。在一个优选的实施方式中,mdh突变体是同源nad(p)-苹果酸脱氢酶的突变体。更优选地,mdh突变体是同源nad(h)-苹果酸脱氢酶(ec1.1.1.37)的突变体。更优选地,具有苹果酸脱氢酶活性的突变多肽是同源过氧化物酶体nad(h)-苹果酸脱氢酶的突变体。在该上下文中,当用于指出给定的(重组)核酸或多肽分子与给定的宿主生物或宿主细胞之间的相互关系时,术语“同源的”或“内源的”应被理解为表示该核酸或多肽分子天然地由相同物种、优选地相同品种或株系的宿主细胞或生物生产。在本文中使用时,术语“异源的”是指非天然存在于宿主细胞中的核酸或氨基酸序列。换句话说,该核酸或氨基酸序列与宿主细胞中天然存在的序列不同。优选地,在本发明的重组宿主细胞中,编码具有苹果酸脱氢酶活性的所述突变多肽的核酸序列在胞质中表达,并且具有苹果酸脱氢酶活性的突变多肽在胞质中有活性。在一些情况下,可通过缺失过氧化物酶体或线粒体靶向信号来获得胞质表达。过氧化物酶体或线粒体靶向信号的存在可以例如通过schlüteretal.,nucleidacidresearch2007,35,d815-d822所公开的方法确定。当mdh突变体是突变型s.cerevisiae过氧化物酶体nad-mdh(例如突变体mdh3)时,优选缺失其c-末端skl,使其在胞质中有活性。通常,具有苹果酸脱氢酶活性的突变多肽可与参照mdh多肽(例如seqidno:53或seqidno:39的mdh)具有至少约40%、50%、60%、70%、80%的序列同一性,例如与参照mdh多肽具有至少85%的序列同一性,例如与参照mdh多肽具有至少约90%的序列同一性,与参照mdh多肽具有至少95%的序列同一性,与参照mdh多肽具有至少98%的序列同一性或与参照mdh多肽具有至少99%的序列同一性。出乎意料地发现:与(过)表达具有mdh活性的参照多肽的等同重组宿主细胞的二羧酸生产水平相比,当在重组宿主细胞中(过)表达上文所述的突变型mdh多肽时,所述突变型mdh多肽导致所述重组宿主细胞中所述二羧酸的生产提高;“参照mdh多肽”通常是nad-mdh(ec1.1.1.37),例如具有seqidno:39所示氨基酸序列的苹果酸脱氢酶。因此,提供了一种重组宿主细胞,其生产或能够生产二羧酸,并且包含编码如上所述的具有苹果酸脱氢酶活性的突变多肽的核酸序列。本发明的重组宿主细胞生产或能够生产二羧酸,例如苹果酸、延胡索酸和/或琥珀酸。术语“二羧酸”和“二羧酸盐/酯”(例如“琥珀酸”和“琥珀酸盐/酯”)在本文中具有相同的含义并可互换使用,前者是后者的氢化形式。通常,与表达参照mdh多肽(例如seqidno:39的参照mdh多肽)的重组宿主细胞相比,本发明的重组宿主细胞生产提高量的二羧酸。二羧酸生产可提高至少5%、10%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少95%或至少100%或更多。生产水平可以用g/l表示,因此二羧酸生产水平的增加通过以g/l为单位的更高的生产水平来表示。本发明的重组宿主细胞或所述宿主细胞的亲本可以是任何类型的宿主细胞。因此,包括原核细胞和真核细胞。宿主细胞还可包括但不限于哺乳动物细胞系,例如cho、vero、bhk、hela、cos、mdck、293、3t3、wi38和脉络丛细胞系。合适的本发明宿主细胞可以是原核细胞。优选地,所述原核细胞是细菌细胞。术语“细菌细胞”包括革兰氏阴性微生物和革兰氏阳性微生物。合适的细菌可以选自例如escherichia、actinobacillus、anabaena、caulobactert、gluconobacter、mannheimia、basfia、rhodobacter、pseudomonas、paracoccus、bacillus、brevibacterium、corynebacterium、rhizobium(sinorhizobium)、flavobacterium、klebsiella、enterobacter、lactobacillus、lactococcus、methylobacterium、staphylococcus或actinomycetes例如streptomyces和actinoplanes种。优选地,细菌细胞选自bacillussubtilis、b.amyloliquefaciens、b.licheniformis、b.puntis、b.megaterium、b.halodurans、b.pumilus、actinobacillussuccinogenes、gluconobacteroxydans、caulobactercrescentuscb15、methylobacteriumextorquens、rhodobactersphaeroides、pseudomonaszeaxanthinifaciens、pseudomonasputida、pseudomonasfluorescens、paracoccusdenitrificans、escherichiacoli、corynebacteriumglutamicum、mannheimiasuccinoproducens、basfiasuccinoproducens、staphylococcuscarnosus、streptomyceslividans、streptomycesclavuligerus、sinorhizobiummelioti和rhizobiumradiobacter。根据本发明的宿主细胞可以是真核宿主细胞。优选地,所述真核细胞是哺乳动物、昆虫、植物、真菌或藻类细胞。更优选地,真核细胞是真菌细胞。合适的真菌细胞可以例如属于saccharomyces、schizosaccharomyces、aspergillus、penicillium、pichia、kluyveromyces、yarrowia、candida、hansenula、humicola、pichia、issatchenkia、kloeckera、schwanniomyces、torulaspora、trichosporon、brettanomyces、rhizopus、zygosaccharomyces、pachysolen或yamadazyma属。真菌细胞可以例如属于saccharomycescerevisiae、s.uvarum、s.bayanuss.pastorianus、s.carlsbergensis、aspergillusniger、penicilliumchrysogenum、pichiastipidis、p.pastoris、kluyveromycesmarxianus、k.lactis、k.thermotolerans、yarrowialipolytica、candidasonorensis、c.revkaufi、c.pulcherrima、c.tropicalis、c.utilis、c.kruisei、c.glabrata、hansenulapolymorpha、issatchenkiaorientalis、torulasporadelbrueckii、brettanomycesbruxellensis、rhizopusoryzae或zygosaccharomycesbailii种。在一种实施方式中,本发明的真菌细胞是酵母,例如属于saccharomycessp.,例如saccharomycescerevisiae。具体宿主酵母细胞的实例包括c.sonorensis、k.marxianus、k.thermotolerans、c.methanesorbosa、saccharomycesbulderi(s.bulderi)、p.kudriavzevii、i.orientalis、c.lambica、c.sorboxylosa、c.zemplinina、c.geochares、p.membranifaciens、z.kombuchaensis、c.sorbosivorans、c.vanderwaltii、c.sorbophila、z.bisporus、z.lentus、saccharomycesbayanus(s.bayanus)、d.castellii、c、boidinii、c.etchellsii、k.lactis、p.jadinii、p.anomala、saccharomycescerevisiae(s.cerevisiae)、pichiagaleiformis、pichiasp.yb-4149(nrrl命名)、candidaethanolica、p.deserticola、p.membranifaciens、p.fermentans和saccharomycopsiscrataegensis(s.crataegensis)。k.marxianus和c.sonorensis的合适菌株包括wo00/71738al、wo02/42471a2、wo03/049525a2、wo03/102152a2和wo03/102201a2中描述的那些。i.orientalis的合适菌株是atcc菌株32196和atcc菌株pta-6648。在本发明中,宿主细胞可以是克拉布特里(crabtree)阴性的野生型菌株。克拉布特里效应被定义为:当以高比生长速率(长期效应)或在存在高浓度葡萄糖的情况下(短期效应)培养时,由于微生物氧消耗的抑制在有氧条件下发生的发酵性代谢。克拉布特里阴性表型不展示出这种效应,因此即使在存在高浓度葡萄糖的情况下或在高生长速率下也能够消耗氧。真核细胞可以是丝状真菌细胞。丝状真菌包括真菌(eumycota)和卵菌(oomycota)亚门的所有丝状形式(如hawksworth等人在ainsworthandbisby'sdictionaryofthefungi,第8版,1995,cabinternational,universitypress,cambridge,uk中定义)。丝状真菌的特征为由几丁质、纤维素、葡聚糖、壳聚糖、甘露聚糖和其它复杂多糖构成的菌丝壁。营养生长通过菌丝伸长进行且碳分解代谢专性好氧。丝状真菌菌株包括但不限于acremonium、aspergillus、agaricus、aureobasidium、cryptococcus、corynascus、chrysosporium、filibasidium、fusarium、humicola、magnaporthe、monascus、mucor、myceliophthora、mortierella、neocallimastix、neurospora、paecilomyces、penicillium、piromyces、phanerochaetepodospora、pycnoporus、rhizopus、schizophyllum、sordaria、talaromyces、rasamsonia、thermoascus、thielavia、tolypocladium、trametes和trichoderma的菌株。可作为宿主细胞的优选丝状真菌菌株属于aspergillusniger、aspergillusoryzae、aspergillusfumigatus、penicilliumchrysogenum、penicilliumcitrinum、acremoniumchrysogenum、trichodermareesei、rasamsoniaemersonii(以前被称为talaromycesemersonii)、aspergillussojae、chrysosporiumlucknowense、myceliophtorathermophyla种。用于比较经转化细胞和未转化细胞的发酵特征的参照宿主细胞包括例如aspergillusnigercbs120.49、cbs513.88、aspergillusoryzaeatcc16868、atcc20423、ifo4177、atcc1011、atcc9576、atcc14488-14491、atcc11601、atcc12892、aspergillusfumigatusaf293(cbs101355)、p.chrysogenumcbs455.95、penicilliumcitrinumatcc38065、penicilliumchrysogenump2、thielaviaterrestrisnrrl8126、talaromycesemersoniicbs124.902、rasamsoniaemersoniicbs393.64、acremoniumchrysogenumatcc36225、atcc48272、trichodermareeseiatcc26921、atcc56765、atcc26921、aspergillussojaeatcc11906、chrysosporiumlucknowenseatcc44006以及所有这些菌株的衍生株。一种更优选的宿主细胞属于aspergillus属,更优选地,宿主细胞属于aspergillusniger种。当根据本发明的宿主细胞是aspergillusniger宿主细胞时,宿主细胞优选是cbs513.88、cbs124.903或其衍生株。在一种优选的实施方式中,根据本发明的宿主细胞是选自candida、hansenula、issatchenkia、kluyveromyces、pichia、saccharomyces、schizosaccharomyces或yarrowia菌株的酵母细胞,或者选自属于acremonium、aspergillus、chrysosporium、myceliophthora、penicillium、talaromyces、rasamsonia、thielavia、fusarium或trichoderma种的丝状真菌细胞的丝状真菌细胞。本发明的宿主细胞可以是生产二羧酸的任何野生型菌株。此外,合适的宿主细胞可以是通过对感兴趣的亲本或野生型细胞进行经典诱变处理或重组核酸转化而获得和/或改善的细胞。因此,合适的宿主细胞可以已经能够产生二羧酸。然而,细胞也可以具有同源或异源表达构建体,所述构建体编码参与二羧酸生产的一种或多种多肽。因此,在一些实施方式中,本发明的重组宿主细胞可包含mdh突变型多肽和从磷酸烯醇式丙酮酸或丙酮酸至琥珀酸的活性还原性三羧酸(tca)通路。因此,除了编码mdh突变多肽的核酸之外,本发明的宿主细胞可以包含这样的核苷酸序列,所述核苷酸序列包含编码以下之中的一种或多种的序列:丙酮酸羧化酶、磷酸烯醇式丙酮酸羧激酶、磷酸烯醇式丙酮酸羧化酶、苹果酸脱氢酶、延胡索酸酶、异柠檬酸裂解酶、苹果酸合酶、延胡索酸还原酶和/或二羧酸转运体。优选地,一种或多种这样的酶(过)表达并且在胞质中有活性。因此,本发明的重组宿主细胞可过表达合适的同源或异源核苷酸序列,所述核苷酸序列编码催化细胞内反应的内源和/或异源酶,从而导致朝向二羧酸(例如苹果酸、延胡索酸和/或琥珀酸)的通量增加。本发明的重组宿主细胞可过表达如下文所述的内源或异源核酸序列。本发明的重组宿主细胞可包含丙酮酸羧化酶(pyc)的遗传修饰,丙酮酸羧化酶催化从丙酮酸盐/酯到草酰乙酸盐/酯的反应(ec6.4.1.1)。丙酮酸羧化酶例如可在基因表达后在胞质中有活性。例如,宿主细胞过表达丙酮酸羧化酶,例如内源性或同源丙酮酸羧化酶被过表达。可以用与seqidno:46的核酸序列所编码的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的丙酮酸羧化酶遗传修饰根据本发明的重组真菌宿主细胞。优选地,重组宿主细胞表达编码胞质中的磷酸烯醇式丙酮酸(pep)羧激酶的核苷酸序列。优选地,编码pep羧激酶的核苷酸序列被过表达。pep羧激酶(ec4.1.1.49)优选地是异源酶,优选地来自细菌,更优选地,具有pep羧激酶活性的酶来自escherichiacoli、mannheimiasp.、actinobacillussp.或anaerobiospirillumsp.,更优选地mannheimiasucciniciproducens。编码pep羧激酶的基因可被过表达,并且在真菌细胞的胞质中有活性。优选地,用与seqidno:47的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的pep羧激酶遗传修饰根据本发明的重组宿主细胞。优选地,重组宿主细胞表达编码胞质中的磷酸烯醇式丙酮酸(pep)羧化酶的核苷酸序列。优选地,编码pep羧化酶的核苷酸序列被过表达。pep羧化酶(ec4.1.1.31)优选地是异源酶,优选地来自细菌。在一种实施方式中,用编码在基因表达之后在胞质中有活性的苹果酸脱氢酶(mdh)的基因进一步遗传修饰重组宿主细胞。胞质表达可以通过缺失过氧化物酶体靶向信号来实现。苹果酸脱氢酶可被过表达。胞质mdh可以是催化从草酰乙酸盐/酯到苹果酸盐/酯的反应的任何合适的同源或异源苹果酸脱氢酶(ec1.1.1.37),例如来自s.cerevisiae。优选地,mdh是s.cerevisiaemdh3,更优选具有c-末端skl缺失从而使得其在胞质中有活性。优选地,根据本发明的重组真菌细胞包含编码与seqidno:39的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的苹果酸脱氢酶的核苷酸序列。在另一种实施方式中,用编码催化从苹果酸到延胡索酸的反应的延胡索酸酶(ec4.2.1.2)的基因进一步遗传修饰本公开的重组宿主细胞。编码延胡索酸酶的基因可来自于任何合适的来源,优选地来自微生物来源,例如酵母(例如saccharomyces)或丝状真菌(例如rhizopusoryzae)或细菌(例如escherichiacoli)。本公开的宿主细胞可过表达编码延胡索酸酶的核苷酸序列。在核苷酸序列表达后,延胡索酸酶可在胞质中有活性,例如通过缺失过氧化物酶体靶向信号来实现。已发现延胡索酸酶的胞质活性导致真菌细胞对二羧酸的高生产力。优选地,本发明的重组宿主细胞过表达编码与seqidno:48、seqidno:49或seqidno:67的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的延胡索酸酶的核苷酸序列。在另一种实施方式中,用编码催化从延胡索酸盐/酯到琥珀酸盐/酯的反应的nad(h)依赖型延胡索酸还原酶(ec1.3.1.6)的任何合适的同源或异源基因遗传修饰重组宿主细胞。nad(h)-依赖型延胡索酸还原酶可以是异源酶,其可来自任何合适的来源,例如细菌、真菌、原生动物或植物。本公开的真菌细胞包含异源nad(h)依赖型延胡索酸还原酶,优选地来自trypanosomasp,例如trypanosomabrucei。在一种实施方式中,nad(h)依赖型延胡索酸还原酶被表达并在胞质中有活性,例如通过缺失过氧化物酶体靶向信号来实现。宿主细胞可过表达编码nad(h)依赖型延胡索酸还原酶的基因。优选地,用与seqidno:8的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的nad(h)依赖型延胡索酸还原酶遗传修饰根据本发明的重组宿主细胞。还优选地,用wo2015/086839中所公开的具有延胡索酸还原酶活性的变体多肽遗传修饰根据本发明的宿主细胞。在另一种实施方式中,本发明的重组宿主细胞表达编码二羧酸转运蛋白的核苷酸序列。优选地,二羧酸转运蛋白被过表达。二羧酸转运蛋白可以是任何合适的同源或异源蛋白。优选地,二羧酸转运蛋白是异源蛋白。二羧酸转运蛋白可以来自任何合适的生物体,优选来自酵母或真菌,例如schizosaccharomycespombe或aspergillusniger。优选地,二羧酸转运蛋白是这样的二羧酸转运蛋白/苹果酸转运蛋白,例如来自aspergillusniger,其与seqidno:50的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性。重组宿主细胞可以进一步包含编码异柠檬酸裂合酶(ec4.1.3.1)的基因的遗传修饰,所述异柠檬酸裂合酶可以是任何合适的同源或异源酶。异柠檬酸裂合酶可例如获自kluyveromyceslactis或escherichiacoli。用与seqidno:51的核酸序列所编码的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的异柠檬酸裂合酶遗传修饰根据本发明的重组宿主。重组宿主细胞可以进一步包含苹果酸合酶(ec2.3.3.9)的遗传修饰。苹果酸合酶可被过表达和/或在胞质中有活性,例如通过缺失过氧化物酶体靶向信号来实现。在苹果酸合酶是s.cerevisiae苹果酸合酶的情况下,例如通过缺失skl羧基端序列来改变天然苹果酸合酶。用与seqidno:52的核酸序列所编码的氨基酸序列具有至少70%,优选至少75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的苹果酸合酶遗传修饰本发明的重组宿主细胞。在另一种实施方式中,本文所公开的本发明的重组宿主细胞包含编码丙酮酸脱羧酶(ec4.1.1.1)的基因的破坏,所述丙酮酸脱羧酶催化从丙酮酸盐/酯到乙醛的反应。在另一种实施方式中,本发明的重组宿主细胞可以包含编码乙醇发酵通路的酶的基因的破坏。编码乙醇发酵通路的酶的基因可以是丙酮酸脱羧酶(ec4.1.1.1),其催化从丙酮酸盐/酯到乙醛的反应;或醇脱氢酶(ec1.1.1.1),其催化从乙醛到乙醇的反应。优选地,本发明的宿主细胞包含一种、两种或更多种编码醇脱氢酶的基因的破坏。如果真菌细胞是酵母,例如s.cerevisiae,则酵母优选包含一种或多种醇脱氢酶基因(adh1、adh2、adh3、adh4、adh5、adh6)的破坏。替代性地或另外,本发明的重组宿主细胞可包含至少一种无功能的编码甘油-3-磷酸脱氢酶的基因。无功能的甘油-3-磷酸脱氢酶基因在本文中用于描述这样的真核细胞,其包含降低的甘油-3-磷酸脱氢酶活性,例如通过突变、破坏或缺失编码甘油-3-磷酸脱氢酶的基因,从而导致与野生型酵母相比甘油形成降低。如果真菌细胞是酵母,例如s.cerevisiae,则酵母优选包含一种或多种甘油-3-磷酸脱氢酶基因(gpd1、gpd2、gut2)的破坏。替代性地或另外,本发明的重组宿主细胞可包含至少一种无功能的编码线粒体外nadh脱氢酶的基因。无功能的线粒体外nadh脱氢酶基因在本文中用于描述这样的真核细胞,其包含降低的nadh脱氢酶活性,例如通过突变、破坏或缺失编码线粒体外nadh脱氢酶的基因。如果真菌细胞是酵母,例如s.cerevisiae,则酵母优选包含一种或多种线粒体外nadh脱氢酶基因(nde1、nde2)的破坏。替代性地或另外,本发明的重组宿主细胞可包含至少一种无功能的编码醛脱氢酶的基因。无功能的醛脱氢酶基因在本文中用于描述这样的真核细胞,其包含降低的醛脱氢酶活性,例如通过突变、破坏或缺失编码醛脱氢酶的基因。如果真菌细胞是酵母,例如s.cerevisiae,则酵母优选包含一种或多种醛脱氢酶基因(ald2、ald3、ald4、ald5、ald6)的破坏。优选地,本发明的重组宿主细胞是重组真菌细胞。更优选地,本发明的宿主细胞是重组酵母细胞。重组真菌细胞或重组酵母细胞的优选实施方式如上文关于重组宿主细胞所述。在本发明的一些实施方式中,重组宿主细胞是重组酵母细胞,其能够生产如上所述的二羧酸,并且包含编码具有如上文详述的苹果酸脱氢酶活性的突变多肽的核酸序列。所述mdh突变体可以是同源或异源野生型mdh多肽的突变体。在一个优选的实施方式中,所述mdh突变体是同源mdh多肽的突变体。在一个甚至更优选的实施方式中,重组酵母细胞是重组saccharomyces,例如s.cerevisiae,并且mdh突变体是同源mdh(例如mdh2或mdh3)的突变体。在一个更具体的实施方式中,所述重组酵母细胞包含编码如表1中所限定的具有苹果酸脱氢酶活性的突变多肽的核酸序列,并且其中对应于seqidno:39中的第34位氨基酸的氨基酸残基选自甘氨酸(g)或丝氨酸(s)。标准的遗传学技术例如宿主细胞中酶的过表达、宿主细胞的遗传修饰或杂交技术是本领域已知的方法,例如sambrook等人(molecularcloning:alaboratorymanual,第三版,coldspringharborlaboratorypress,coldspringharborlaboratory,ny,2001)或ausubel等人(currentprotocolsinmolecularbiology,greenpublishingandwileyinterscience,ny,1987)中所述。用于真菌宿主细胞的遗传修饰、转化的方法从例如ep-a-0635574、wo98/46772、wo99/60102和wo00/37671、wo90/14423、ep-a-0481008、ep-a-0635574和us6,265,186中已知。在本文中使用时,术语“核酸”、“多核苷酸”或“核酸分子”旨在包括dna分子(例如cdna或基因组dna)和rna分子(例如mrna)以及使用核苷酸类似物产生的dna或rna的类似物。核酸分子可以是单链或双链的,但优选是双链dna。可以使用寡核苷酸类似物或衍生物(例如肌苷或硫代磷酸酯核苷酸)合成核酸。这种寡核苷酸可用于例如制备具有改变的碱基配对能力或增加的对核酸酶的抗性的核酸。术语“核酸构建体”在本文中被称为单链或双链的核酸分子,其是从天然存在的基因分离的,或者更典型地,其已被修饰为含有以不会在自然中存在的方式组合和并置的核酸的区段。当核酸构建体含有在宿主细胞中表达编码序列所需的全部控制序列时,术语核酸构建体与术语“表达盒”的含义相同,其中所述控制序列与所述编码序列可操作地连接。在本文中使用时,术语“可操作地连接”是指处于功能关系的多核苷酸元件(或编码序列或核酸序列)的连接。当核酸序列被置于与另一核酸序列的功能关系中时,其是“可操作地连接”的。例如,如果启动子或增强子影响编码序列的转录,则其与编码序列可操作地连接。在本文中使用时,术语“启动子”是指这样的核酸片段,其功能为控制位于该基因的转录起始位点的转录方向上游的一个或多个基因的转录,并且在结构上通过dna依赖性rna聚合酶的结合位点、转录起始位点和本领域技术人员已知的任何其他dna序列的存在来鉴定。“组成型”启动子是在大多数环境和发育条件下有活性的启动子。“诱导型”启动子是在环境调控或发育调控下有活性的启动子。能够用于实现编码酶(例如延胡索酸还原酶或本发明的宿主细胞中引入的任何其它酶)的核苷酸序列表达的启动子对编码待表达的酶的核苷酸序列而言可以不是天然的,即对与之可操作地连接的核苷酸序列(编码序列)而言异源的启动子。优选地,启动子对宿主细胞而言是同源的,即内源的。该上下文中合适的启动子既包括组成型又包括诱导型的天然启动子以及经改造的启动子,所述启动子是本领域技术人员公知的。真核宿主细胞中合适的启动子可以是gal7、gal10或gal1、cyc1、his3、adh1、pgl、ph05、gapdh、adc1、trp1、ura3、leu2、eno、tpi和aox1。其它合适的启动子包括pdc、gpd1、pgk1、tef1和tdh。通常,编码酶的核苷酸序列包含“终止子”。本发明中可以使用在真核细胞中有功能的任何终止子。优选的终止子得自宿主细胞的天然基因。合适的终止子序列是本领域公知的。优选地,这类终止子与下述突变组合,所述突变防止本发明宿主细胞中无义介导的mrna衰退(decay)(见例如shirley等,2002,genetics161:1465-1482)。核酸构建体可被并入“载体”(例如表达载体)且/或并入宿主细胞中,以实现待表达多肽的表达。表达载体可以是可方便地进行重组dna程序并且可引起编码具有苹果酸脱氢酶活性的多肽的多核苷酸表达的任何载体(例如质粒或病毒)。载体的选择通常取决于载体与待引入所述载体的宿主细胞的相容性。载体可以是线性质粒或闭环质粒。载体可以是自主复制型载体,即作为染色体外实体而存在、其复制独立于染色体复制的载体,例如,质粒、染色体外元件、微型染色体或人工染色体。如果打算用于真菌来源的宿主细胞中,则合适的附加型核酸构建体可以例如基于酵母2μ或pkd1质粒(gleer等,1991,biotechnology9:968-975)或ama质粒(fierro等,1995,currgenet.29:482-489)。或者,表达载体可以是当被引入宿主细胞时整合至基因组中并与其已整合至其中的染色体一起复制的载体。整合型克隆载体可随机地或在预定靶基因座处整合在宿主细胞的染色体中。在本发明的一个优选实施方式中,整合型克隆载体包含与宿主细胞基因组中预定靶基因座中的dna序列同源的dna片段,以将克隆载体靶向整合到该预定基因座。为了促进靶向整合,克隆载体优选在转化细胞之前线性化。优选进行线性化,使得克隆载体的至少一端但优选两端的侧翼是与靶基因座同源的序列。靶基因座侧翼的同源序列的长度优选为至少20bp、至少30bp、至少50bp、至少0.1kb、至少0.2kb、至少0.5kb、至少1kb、至少2kb或更长。宿主细胞增强的同源重组能力提高靶向整合到宿主细胞基因组中(即整合到预定的靶基因座中)的效率。与靶基因座同源的克隆载体中的同源侧翼dna序列源自高表达的基因座,这意味着它们源自在宿主细胞中能够具有高表达水平的基因。能够具有高表达水平的基因(即高表达基因)在本文中被定义为:在例如经诱导条件下,其mrna可以构成总细胞mrna的至少0.5%(w/w)的基因,或者,其基因产物可以构成总细胞蛋白质的至少1%(w/w)的基因,或者在分泌的基因产物的情况下,可以分泌至至少0.1g/l的水平。核酸构建体或表达载体可以在本发明的宿主细胞体内装配,并且任选地,在单个步骤中整合到细胞的基因组中(参见例如wo2013/076280)。可将多于一个拷贝的本发明的核酸构建体或表达载体插入宿主细胞中以增加由包含在核酸构建体内的核酸序列编码的具有苹果酸脱氢酶活性的多肽的产生(过表达)。这可以优选地通过将两个或更多个拷贝的核酸整合到其基因组中来完成,更优选地通过使核酸靶向整合到上文所限定的高表达基因座来完成。本领域技术人员会理解:表达载体的设计可取决于诸如待转化的宿主细胞的选择、所期望蛋白质的表达水平等因素。可以通过常规转化或转染技术将本发明的核酸构建体和/或表达载体引入原核或真核细胞中。在本文中使用时,术语“转化”和“转染”旨在指用于将外来核酸(例如dna)引入本领域技术人员公知的宿主细胞中的多种本领域公认的技术。用于转化或转染宿主细胞的合适方法可见于sambrook等(molecularcloning:alaboratorymanual,第3版,coldspringharborlaboratorypress,coldspringharborlaboratory,ny,2001),davis等(basicmethodsinmolecularbiology,第1版,elsevier,1986)和其他实验室手册。上述酶的胞质表达可通过缺失过氧化物酶体或线粒体靶向信号来实现。过氧化物酶体或线粒体靶向信号的存在可例如通过schlüter等人(schlüter等人,2007,nucleicacidresearch35:d815-d822)所公开的方法来确定。两个序列之间的序列比较和序列同一性百分比的确定可使用数学算法来完成。本领域的技术人员将意识到以下事实:若干不同的计算机程序可用于比对两个序列并确定两个序列之间的同一性(kruskal,j.b.(1983)anoverviewofsequencecomparison,d.sankoff和j.b.kruskal,(编),timewarps,stringeditsandmacromolecules:thetheoryandpracticeofsequencecomparison,第1-44页addisonwesley)。两个氨基酸序列之间或者两个核苷酸序列之间的序列同一性百分比可使用用于比对两个序列的needleman和wunsch算法来确定(needleman,s.b.和wunsch,c.d.(1970)j.mol.biol.48,443-453)。氨基酸序列和核苷酸序列两者均可通过算法进行比对。needleman-wunsch算法已在计算机程序needle中实现。出于本发明的目的,使用了来自emboss包的needle程序(2.8.0版或更高版本,emboss:theeuropeanmolecularbiologyopensoftwaresuite(2000)rice,p.longden,i.和bleasby,a.trendsingenetics16,(6)第276—277页,http://emboss.bioinformatics.nl/)。对于蛋白质序列而言,eblosum62用于替换矩阵。对于核苷酸序列而言,使用ednafull。所使用的任选参数是空位开放罚分为10,以及空位延伸罚分为0.5。技术人员将理解的是,所有这些不同的参数将产生稍微不同的结果,但是当使用不同的算法时,两个序列的总体同一性百分比没有显著改变。在通过如上所述的程序needle进行比对后,查询序列与本发明的序列之间的序列同一性的百分比计算如下:在两个序列中显示相同氨基酸或相同核苷酸的比对中的相应位置的数目除以在减去比对中的总空位数后比对的总长度。如本文定义的同一性可通过使用nobrief选项从needle获得,并且在程序的输出中标记为“最长同一性”。本发明的核酸和蛋白质序列可进一步用作“查询序列”以针对公共数据库进行搜索,以例如鉴定其它家族成员或相关序列。此类搜索可使用altschul等人(1990)j.mol.biol.215:403—10的blastn和blastx程序(2.2.31版或更高版本)进行。blast核苷酸搜索可用blastn程序(得分=100、字长=11)来进行,以获得与本发明的核酸分子同源的核苷酸序列。blast蛋白质搜索可用blastx程序(得分=50、字长=3)来进行,以获得与本发明的蛋白分子同源的氨基酸序列。为了获得用于比较目的的空位比对,可如在altschul等人,(1997)nucleicacidsres.25(17):3389-3402中描述利用空位blast。当利用blast和空位blast程序时,可使用相应程序(例如blastx和blastn)的默认参数。参见http://www.ncbi.nlm.nih.gov/的国家生物技术信息中心的主页。本发明还提供了生产二羧酸(例如琥珀酸)的方法,所述方法包括:在适于生产二羧酸的条件下发酵如上文所述的本发明的重组宿主细胞,和任选地从发酵培养基中回收二羧酸。在所述方法中,在包含合适发酵培养基的容器中发酵本发明的重组宿主细胞。本文所使用的术语发酵指的是化合物的微生物生产,在此处是由碳水化合物生产二羧酸。优选地,发酵产物是二羧酸,优选地是苹果酸、延胡索酸和/或琥珀酸,优选地是琥珀酸。分批发酵在本文中被定义为这样的发酵,其中所有营养物在发酵开始时加入。补料分批发酵是这样的分批发酵,其中营养物在发酵期间加入。分批和补料分批发酵中的产物可在合适的时机收获,例如在一种或更多种营养物耗尽时收获。连续发酵是这样的发酵,其中营养物被连续加入到发酵中且其中产物被连续地移出发酵。在一种实施方式中,在本发明的方法中发酵宿主细胞是在碳水化合物限制条件下进行的。当在本文中使用时,碳水化合物限制条件被定义为维持碳水化合物浓度低于10g/l,例如约5g/l。根据本发明的生产二羧酸的方法可以以任何合适的体积和规模进行,优选地以工业规模进行。工业规模在本文中被定义为至少10升或100升,优选地至少1m3,优选地至少10m3或100m3,优选地至少1000m3,通常低于10,000m3的体积。在本发明的方法中发酵重组宿主细胞可在任何合适的发酵培养基中进行,所述发酵培养基包含合适的氮源、碳水化合物和本发明所述方法中生长和生产二羧酸所需的其它营养物。根据本发明的发酵方法中合适的碳水化合物可以是葡萄糖、半乳糖、木糖、阿拉伯糖、蔗糖或麦芽糖。在一种实施方式中,所述发酵方法在co2分压为5%-60%、优选地约50%下进行。在所述生产二羧酸的方法中,ph通常在二羧酸生产期间降低。优选地,在所述生产二羧酸的方法中,ph在1和5之间、优选地1.5和4.5之间、更优选地2和4之间的范围中。在另一优选实施方式中,根据本发明的方法包括以下步骤:在碳水化合物的存在下在需氧条件下预培养宿主细胞。优选地,在预培养期间宿主细胞的发酵在4-6之间的ph下进行。优选地,预培养期间的碳水化合物是非抑制性碳水化合物、优选半乳糖。已发现在非抑制性碳水化合物上预培养宿主细胞是有利的,因为这防止了葡萄糖抑制的发生,而葡萄糖抑制可负面影响所产生生物质的量。此外,还发现:在需氧条件下预培养宿主细胞的步骤导致更高的生物质产率和更快的生长。优选地,所述预培养以分批模式进行。通常进行用于生产增加的生物质的增殖步骤,优选地在碳水化合物限制条件下进行。生产二羧酸的方法可在任何合适的温度下进行。合适的温度可例如在约10℃和约40℃之间,例如在约15℃和约30℃之间。在一种实施方式中,以至少部分宿主细胞被重复利用(即,再循环)的方式进行本发明的方法。细胞可被再循环回原始容器或者再循环入第二容器。优选地,用维生素和/或微量元素补充引入再循环宿主细胞的培养基。在一种优选的实施方式中,发酵培养基包含1-150g/l的琥珀酸,优选5-100g/l、更优选10-80g/l或15-60g/l的琥珀酸。在任何情况下,与用参照mdh多肽(例如seqidno:39的参照mdh多肽)修饰的宿主细胞相比,本发明的重组宿主细胞通常能够在发酵培养基中积累更多琥珀酸。生产二羧酸的方法可以进一步包括回收二羧酸。可通过本领域已知的任何合适方法来回收二羧酸,例如通过结晶、铵沉淀、离子交换技术、离心或过滤或这些方法的任意合适组合。在一种优选的实施方式中,回收二羧酸包括:使二羧酸结晶和形成二羧酸晶体。优选地,使二羧酸结晶包括除去部分发酵培养基,优选地通过蒸发,以获得浓缩的培养基。根据本发明,可以通过从ph为1-5且包含二羧酸(例如琥珀酸)的水性溶液结晶所述二羧酸(例如琥珀酸)来回收二羧酸(例如琥珀酸),包括蒸发部分水性溶液以获得浓缩溶液,将浓缩的溶液的温度降低至5-35℃之间的值,其中琥珀酸晶体形成。优选地,结晶包括:使浓缩的培养基的温度达到10-30℃,优选地15-25℃的温度。优选地,发酵培养基的ph为1.5-4.5,优选地2-4。已发现,相较于在低于10度的低温下结晶的二羧酸(例如琥珀酸)的晶体,使二羧酸(例如琥珀酸)在较高温度(例如,10-30℃之间)下结晶产生杂质(例如,有机酸、蛋白质、颜色和/或气味)量更低的二羧酸(例如琥珀酸)的晶体。相较于在低于10℃或5℃的温度下进行的二羧酸结晶,在更高温度下结晶琥珀酸的另一个优点是:冷却水性溶液需要更少量的能量,从而导致更加经济和可持续的方法。优选地,结晶二羧酸(例如琥珀酸)包括洗涤二羧酸晶体的步骤。可由ph为1-5的发酵培养基直接将二羧酸(例如琥珀酸)结晶至纯度为至少90重量/重量%,优选地至少95重量/重量%、96重量/重量%、97重量/重量%、或至少98重量/重量%、或99-100重量/重量%。在一种优选的实施方式中,生产二羧酸的方法还包括:在工业方法中使用二羧酸。优选地,在根据本发明的方法中制备的二羧酸(例如琥珀酸)被进一步转变为期望的产品。期望的产品可以是例如聚合物,例如聚丁二酸丁二醇酯(pbs),除冰剂(deicingagent),食品添加剂,化妆品添加剂或表面活性剂。也就是说,本发明提供了生产产品(例如聚合物,例如聚丁二酸丁二醇酯(pbs),除冰剂,食品添加剂,化妆品添加剂或表面活性剂)的方法,所述方法包括:如本文所述生产二羧酸;以及在所述产品的生产中使用所述二羧酸。本文中作为现有技术所给出的专利文件或其它材料的引用并不应被认为是承认所述文件或材料在任何一项权利要求的优先权日时是已知的或者其所包含的信息是公知常识的一部分。本文中陈述的每个引用的公开内容通过引用以整体并入本文中。通过以下实施例进一步阐释本发明:实施例通用的材料和方法dna程序除非另有说明,否则如别处(sambrook等人,1989,molecularcloning:alaboratorymanual,第2版,coldspringharborlaboratorypress,coldspringharbor,newyork)所述进行标准dna程序。使用校正酶phusion聚合酶(newenglandbiolabs,usa)根据制造商的说明来扩增dna。限制酶来自invitrogen或newenglandbiolabs。二羧酸生产菌株的微量滴定板(mtp)发酵为了确定二羧酸生产,使菌株一式三份地在湿度摇床(infors)中的微量滴定板中在30度、550rpm和80%湿度下生长3天。培养基基于verduyn培养基(verduync,postmae,schefferswa,vandijkenjp.yeast,1992jul;8(7):501-517),但如下文所述对碳源和氮源进行更改。表1.mtp预培养基组成原材料浓度(g/l)半乳糖c6h12o6.h2o40.0尿素(nh2)2co2.3磷酸二氢钾kh2po43.0硫酸镁mgso4.7h2o0.5微量元素溶液a1维生素溶液b1a微量元素溶液组分式浓度(g/kg)edtac10h14n2na2o8.2h2o15.00硫酸锌.7h2oznso4.7h2o4.50氯化锰.2h2omncl2.2h2o0.84氯化钴(ii).6h2ococl2.6h2o0.30硫酸铜(ii).5h2ocuso4.5h2o0.30钼酸钠.2h2ona2moo4.2h2o0.40氯化钙.2h2ocacl2.2h2o4.50硫酸铁.7h2ofeso4.7h2o3.00硼酸h3bo31.00碘化钾ki0.10b维生素溶液组分式浓度(g/kg)生物素(d-)c10h16n2o3s0.05cad(+)泛酸c18h32can2o101.00烟酸c6h5no21.00肌醇c6h12o625.00盐酸氯化硫胺c12h18cl2n4os.xh2o1.00盐酸吡哆醇c8h12clno31.00对氨基苯甲酸c7h7no20.20使用80μl预培养物接种24孔板中含有1.5%半乳糖作为碳源的2.5ml培养基。使培养物在30℃、550rpm、80%湿度的湿度摇床(infors)中生长72小时。生成生物质后,通过将细胞重悬于2.5ml含有葡萄糖作为碳源的矿质培养基中来开始生产实验。在30℃、550rpm、80%湿度的湿度摇床(infors)中孵育主要培养物,并在培养48小时后取样。通过nmr的mtp样品的代谢物分析对于mtp样品的代谢物分析,将90μl发酵样品上清液与10μlnmr标准品(20g/l马来酸)和100μl10%d2o溶液混合。将样品冻干,随后溶在1mld2o中。使用不具有水抑制的脉冲程序(zg),在300k的温度下,使用90度激发脉冲、2.0s的采集时间和40s的放松延迟,在配备有he冷却低温探头、以500mhz的质子频率运行的bestbrukeravanceiii光谱仪上记录1d1hnmr谱。扫描次数设置为8。基于以下信号(δ相对于4,4-二甲基-4-硅戊烷-1-磺酸)计算苹果酸浓度[以g/l计]:苹果酸:根据ph值以及α-ch2和ch(oh)信号与其他化合物的重叠,选择以下三种苹果酸信号之一进行定量:α-ch(a)(2.92ppm,n=1h,双峰(doubledoublet)或dd),α-ch(x)(2.85ppm,n=1h,dd)或者ch(oh)(4.6ppm,n=1h,dd)。基于以下信号(δ相对于4,4-二甲基-4-硅戊烷-1-磺酸)计算琥珀酸浓度[以g/l计]:琥珀酸:2.67ppm处的琥珀酸信号(s,4h)。用于标准品的信号:约6.3ppm处的马来酸峰(s,2h)。bharti等人,2012,tractrendsinanalyticalchemistry35:5-26描述了通过nmr定量。实施例1:构建酵母菌株suc-1029使用菌株cen.pk113-7d(matahis3leu2trp1mal2-8suc2)作为构建菌株suc-1029的起点。如下所述将rhyzopusoryzae的延胡索酸酶基因(fumr)转化到菌株cen.pk113-7d中。生成pcr片段使用扩增seqidno:34的完整核苷酸序列的引物,通过pcr扩增seqidno:34获得pcr片段9。seqidno:34描述了如专利申请wo2009/065779中所公开的含有来自rhyzopusoryzae的延胡索酸酶(fumr)核苷酸序列的合成多核苷酸。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。fumr基因的表达由tdh1启动子(紧接在tdh1基因的起始密码子之前的600bp)和tdh1终止子(紧接在tdh1基因的终止密码子之后的300bp)控制。控制fumr表达的tdh1启动子和tdh1终止子序列是来自saccharomycescerevisiaes288c的天然序列。在tdh1启动子上游的seqidno:34的5’端的599bp区域是与yprctau3基因座同源的区域。使用扩增seqidno:35的完整核苷酸序列的引物,通过pcr扩增seqidno:35获得pcr片段10。seqidno:35描述了含有部分psuc227质粒序列的合成多核苷酸,其描述于pct/ep2013/055047中。seqidno:35的5’端与seqidno:34的3’端具有重叠。seqidno:35的3’端与seqidno:36的5’端具有重叠。使用扩增seqidno:36的完整核苷酸序列的引物,通过pcr扩增seqidno:36获得pcr片段11。seqidno:36描述了含有部分psuc225质粒序列的合成多核苷酸,其描述于pct/ep2013/055047中。seqidno:36的3’端与seqidno:37的5’端具有重叠。使用扩增seqidno:37的完整核苷酸序列的引物,通过pcr扩增seqidno:37获得pcr片段12。seqidno:37描述了与yprctau3基因座同源的合成多核苷酸。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段9至12。转化到cen.pk113-7d中以构建菌株suc-1029通过本领域技术人员已知的方法进行酵母转化。用纯化的pcr片段9至12转化s.cerevisiae菌株cen.pk113-7d。pcr片段10和11在其5’端和3’端具有重叠,pcr片段9和12在其3’端和5’端具有重叠,从而使得允许所有4个pcr片段的同源重组(图1)。pcr片段9的5’端和pcr片段12的3’端与yprctau3基因座同源,使得所有4个pcr片段能够整合在yprctau3基因座(图1)。这产生了一个由整合在yprctau3基因座的pcr片段9至12组成的线性片段,其位于染色体xvi上。将转化混合物涂布在含有200μgg418(sigmaaldrich,zwijndrecht,荷兰)/ml的yephd-琼脂(每升:10g酵母提取物,20gphytonepeptone,20g葡萄糖,20g琼脂)上。在30℃下生长三天后,将各个转化体重新划线在含有20g葡萄糖/升和200μgg418/ml的yeph-琼脂板上。随后,使用pct/ep2013/055047中的所述方法,藉由cre重组酶通过在kanmx标记和编码cre重组酶的cre基因侧翼的lox66和lox71位点之间的重组移除整合的pcr片段10和11上存在的标记盒和cre-重组酶基因,从而导致kanmx标记和cre基因被移除,并留下lox72位点作为lox66和lox71位点之间重组的结果。所产生的无标记菌株被命名为suc-1029。使用pcr来证实所引入的fumr基因的存在,其中使用可以与seqidno:34编码的orf的编码序列退火的引物序列。通过pcr证实kanmx标记的正确整合和移除,其中使用来自yprctau3基因座的5’和3’引物,所述引物不与pcr片段9和12上存在的yprctau3同源区域杂交。实施例2:构建菌株suc-1112生成pcr片段采用saccharomycescerevisiae菌株cen.pk113-7d(matahis3leu2trp1mal2至8suc2)的基因组dna作为模板,使用seqidno:9和seqidno:10中所述的引物序列生成由5’int59整合位点组成的pcr片段1。采用seqidno:1作为模板,使用seqidno:11和seqidno:12中所述的引物序列生成pcr片段2。seqidno:1编码来自actinobacillussuccinogenes的磷酸烯醇式丙酮酸羧激酶(pcka),如专利申请wo2009/065780中所公开。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tpi1启动子控制pckα基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即gnd2终止子)控制。采用seqidno:2作为模板,使用seqidno:13和seqidno:14中所述的引物序列生成pcr片段3。seqidno:2编码来自saccharomycescerevisiae的丙酮酸羧化酶(pyc2),如专利申请wo2009/065780中所公开。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即pgk1启动子控制pyc2基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即adh1终止子)控制。采用seqidno:3作为模板,使用seqidno:15和seqidno:16中所述的引物序列生成pcr片段4。seqidno:3编码在saccharomycescerevisiae中有功能的kanmx选择标记,其是从质粒pug7-ecorv扩增的。pug7-ecorv是gueldener等人(nucleicacidsres.1996年7月;24(13):2519-24)所述的质粒pug6的变体,其中pug6中存在的loxp位点变为lox66和lox71位点(lambert等人,appl.environ.microbiol.2007年2月;73(4):1126-35.epub2006年12月1日)。采用seqidno:4作为模板,使用seqidno:17和seqidno:18中所述的引物序列生成pcr片段5。seqidno:4编码来自aspergillusniger的推定的二羧酸转运体,如ep2495304中所公开。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即eno1启动子控制dct_02-基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即tef2终止子)控制。采用seqidno:5作为模板,使用seqidno:19和seqidno:20中所述的引物序列生成pcr片段6。seqidno:5编码来自saccharomycescerevisiae的苹果酸脱氢酶(mdh3),如专利申请wo2009/065778中所公开。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自kluyveromyceslactis的启动子控制(或与来自kluyveromyceslactis的启动子可操作地连接),即orfklla0_f20031g(uniprot检索号为q6cja9)的启动子控制mdh3基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即gpm1终止子)控制。采用seqidno:6作为模板,使用seqidno:21和seqidno:22中所述的引物序列生成pcr片段7。seqidno:6编码来自escherichiacoli的延胡索酸酶(fumb)(e.c.4.2.1.2,uniprot检索号为p14407)。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tdh3启动子控制fumb基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即tdh1终止子)控制。采用seqidno:7作为模板,使用seqidno:23和seqidno:24中所述的引物序列生成pcr片段8。如专利申请wo2009/065778中所公开,seqidno:7编码来自trypanosomabrucei的延胡索酸还原酶(frdg)。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tef1启动子控制fumb基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即tal1终止子)控制。采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:40和seqidno:41中所述的引物序列生成由3’int59整合位点组成的pcr片段113。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段1至8和pcr片段113。转化到suc-1029中以构建菌株suc-1112通过本领域技术人员已知的方法进行酵母转化。用纯化的pcr片段1至8和pcr片段113转化s.cerevisiae菌株suc-1029。pcr片段2至8在其5’端和3’端具有重叠,pcr片段1和113在其3’端和5’端具有重叠,从而使得允许所有8个pcr片段的同源重组。pcr片段1的5’端和pcr片段113的3’端与int59基因座同源,使得所有9个pcr片段能够整合在int59基因座(参见图2)。这产生了一个由整合在int59基因座的pcr片段2至8组成的线性片段。专利申请wo2013076280中描述了这种整合方法。int59基因座位于染色体xi上,在ykr092c下游923bp且ykr093w上游922bp。将转化混合物涂布在含有20g半乳糖/升和200μgg418(sigmaaldrich,zwijndrecht,荷兰)/ml的yeph-琼脂(每升:10g酵母提取物,20gphytonepeptone,20g琼脂)上。在30℃下生长三天后,将各个转化体重新划线在含有20g半乳糖/升和200μgg418/ml的yeph-琼脂板上。使用pcr来证实所有引入基因的存在,其中使用可以与seqidno:1至seqidno:7编码的orf的编码序列退火的引物序列。所得菌株被命名为suc-1112。如果需要,可以移除菌株suc-1112中存在的kanmx标记。实施例3:将苹果酸脱氢酶基因转化到菌株suc-1112中并在所得转化体中生产苹果酸生成pcr片段采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:25和seqidno:26中所述的引物序列生成由5’int1整合位点组成的pcr片段13。通过采用seqidno:42作为模板,使用seqidno:43和seqidno:44中所述的引物序列生成pcr片段114。seqidno:42含有编码葡萄糖-6-磷酸脱氢酶(g6pd)的zwf1基因。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自kluyveromyceslactis的启动子控制(或与来自kluyveromyceslactis的启动子可操作地连接),即orfklla0c05566g(uniprot检索号为q6cue2)的启动子控制zwf1基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即tef1终止子)控制。通过采用seqidno:38作为模板,使用seqidno:45和seqidno:28中所述的引物序列生成pcr片段115。seqidno:38编码在saccharomycescerevisiae中有功能的诺尔丝菌素选择标记,其是从质粒pug7-nat的修改形式扩增的。pug7-nat是gueldener等人(nucleicacidsres.1996年7月;24(13):2519-24)所述的质粒pug6的变体,其中pug6中存在的loxp位点变为lox66和lox71位点(lambert等人,appl.environ.microbiol.2007年2月;73(4):1126-35.epub2006年12月1日),并且其中kanmx标记被诺尔丝菌素标记替代(goldstein和mccusker,yeast.1999年10月;15(14):1541-53)。通过采用seqidno:31作为模板,使用seqidno:29和seqidno:30中所述的引物序列生成pcr片段15。seqidno:31编码来自s.cerevisiae的苹果酸脱氢酶(mdh3)。如专利申请wo2013/004670a1中所公开,通过缺失skl羧基末端序列来改变mdh3。该合成序列包括启动子-基因-终止子序列,包括合适的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tdh3启动子控制mdh3基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即gpm1终止子)控制。采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:32和seqidno:33中所述的引物序列生成由3’int1整合位点组成的pcr片段16。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段13-16。转化到suc-1112中通过本领域技术人员已知的方法进行酵母转化。用纯化的pcr片段13、114、115、15和16转化s.cerevisiae菌株suc-1112。pcr片段114和115和15在其5’端和3’端具有重叠,pcr片段13和16在其3’端和5’端具有重叠,从而使得允许所有5个pcr片段的同源重组。pcr片段13的5’端和pcr片段16的3’端与int1基因座同源,使得所有4个pcr片段能够整合在int1基因座(参见图3)。这产生了一个由整合在int1基因座的pcr片段13和16组成的线性片段。专利申请wo2013/076280中描述了这种整合方法。int1基因座位于染色体xv上,在yor071c下游659bp且yor070c上游998bp。该方法导致如seqidno:39所示的340个氨基酸的苹果酸脱氢酶蛋白的表达,seqidno:39与来自s.cerevisiae的天然序列相比缺少c-末端氨基酸skl。将转化混合物涂布在含有20g半乳糖/升和200μg诺尔丝菌素(jenabioscience,德国)/ml的yeph-琼脂(每升:10g酵母提取物,20gphytonepeptone,20g琼脂)上。在30℃下生长三天后,将各个转化体重新划线在含有20g半乳糖/升和200μg诺尔丝菌素/ml的yeph-琼脂板上。使用pcr来证实所引入基因的存在,其中使用可以与pcr片段114、115和15上存在的编码orf的编码序列退火的引物序列。为了证实pcr片段13、114、115、15和16在正确基因座的整合,使用与int1基因座的5’和3’区域退火并且不与pcr片段13和16上的int1区域结合的引物和与pcr片段114和15上的orf退火的引物的组合,使得只有pcr片段114和15整合在int1基因座时,才能形成pcr产物。得到了三种单菌落:suc-1112+mdh3#1、suc-1112+mdh3#2、suc-1112+mdh3#3。如果需要,可以移除菌株suc-1112+mdh3#1、suc-1112+mdh3#2、suc-1112+mdh3#中存在的kanmx和诺尔丝菌素标记。二羧酸生产为了确定二羧酸生产,如“通用的材料和方法”中所述进行mtp发酵和nmr测量。在含有存在于pcr片段15上的额外mdh3基因拷贝的suc-1112+mdh3菌株(suc-1112+mdh3#1、suc-1112+mdh3#2、suc-1112+mdh3#3)的上清液中,测得苹果酸的平均滴度为8.7g/l。琥珀酸水平低于预期;这些菌株看起来已经失去了frdg基因,从而导致苹果酸盐/酯朝向琥珀酸盐/酯的转变有限。实施例4:将编码苹果酸脱氢酶突变体的基因转化到菌株suc-1112中并在所得转化体中生产苹果酸生成pcr片段如实施例3中所述生成pcr片段13和16。采用seqidno:38作为模板,使用seqidno:27和seqidno:28中所述引物序列生成pcr片段14。seqidno:38编码在saccharomycescerevisiae中有功能的诺尔丝菌素选择标记,其是从质粒pug7-nat的修改形式扩增的。pug7-nat是gueldener等人(nucleicacidsres.1996年7月;24(13):2519-24)所述的质粒pug6的变体,其中pug6中存在的loxp位点变为lox66和lox71位点(lambert等人,appl.environ.microbiol.2007年2月;73(4):1126-35.epub2006年12月1日),并且其中kanmx标记被诺尔丝菌素标记替代(goldstein和mccusker,yeast.1999年10月;15(14):1541-53)。编码seqidno:39中所述的参照苹果酸脱氢酶序列的不同蛋白质突变体的合成核苷酸序列由dna2.0(menlopark,california,usa)合成。合成的核苷酸序列编码相对于参照mdh3序列(seqidno:39)在第34至40位突变的氨基酸序列,如表1中所示。除了编码表1中所示的突变氨基酸之外,合成的核苷酸序列突变体与seqidno:31相同。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tdh3启动子控制突变mdh基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即gpm1终止子)控制。合成的基因序列包含tdh3启动子-突变的mdh-gpm1终止子及其它,其使用seqidno:29和seqidno:30中所述引物序列通过pcr扩增,以生成pcr片段17至108(参见表1)。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段13、14、16和17至108。转化到suc-1112中用纯化的pcr片段13、14和16与单独的pcr片段17至108的组合转化菌株suc-1112。pcr片段14和pcr片段17至108在其5’端和3’端具有重叠,pcr片段13和16在其3’端和5’端具有重叠,从而使得允许所有4个pcr片段的同源重组。pcr片段13的5’端和pcr片段16的3’端与int1基因座同源,使得所有4个pcr片段能够整合在int1基因座(图4)。转化体的转化和选择描述于实施例2中。二羧酸生产为了确定二羧酸生产,如“通用的材料和方法”中所述使四个独立的表达突变的苹果酸脱氢酶序列的suc-1112转化体在微量滴定板中生长。由于frdg的丢失,产生了少量的琥珀酸(参见实施例3),但仍可通过测量苹果酸盐/酯水平来确定mdh3活性。平均苹果酸滴度如表1所示。表达突变的苹果酸脱氢酶序列的几种suc-1112转化体的苹果酸的平均产量超过10g/l苹果酸。有趣的是,在第34位用甘氨酸或丝氨酸残基替换天冬氨酸的突变体显示出提高的苹果酸生产。这显著高于实施例3中所述的用参照mdh3序列转化的suc-1112的平均苹果酸滴度。显著高于是指:具有参照苹果酸脱氢酶序列和经改进的突变型苹果酸脱氢酶序列的菌株的苹果酸滴度的95%置信区间不重叠。用参照mdh3序列转化的suc-1112的苹果酸滴度的95%置信区间的上限低于10g/l。表1:培养菌株suc-1112转化体4天后在生产培养基的上清液中测量的平均苹果酸滴度,所述suc-1112转化体表达磷酸烯醇式丙酮酸羧激酶(pcka)、丙酮酸羧化酶(pyc2)、苹果酸脱氢酶(mdh3)、延胡索酸酶(fumr和fumb)、二羧酸转运体(dct_02),并用编码参照苹果酸脱氢酶(seqidno:39)或苹果酸脱氢酶突变体(mut_001-mut_94)的核苷酸序列转化,与参照序列相比,所述苹果酸脱氢酶突变体在如下所示的氨基酸位置含有突变。实施例5:测量苹果酸脱氢酶突变体的nadh特异性活性和nadph特异性活性从表1中选择共19种突变体,并如“通用的材料和方法”中所述进行再培养。通过离心(4000rpm,10分钟,4℃)收获生物质,并用pbs(磷酸盐缓冲盐水,sigmaaldrich)洗涤两次,之后将细胞沉淀物在-20℃冷冻。使用0.5mm酸洗玻璃珠与来自qiagen的tissuelyserii(3000rpm,2x10秒,循环之间在冰上冷却1分钟)的组合,在方孔96深孔微量滴定板(mtp)中实现细胞破碎。将体积为600μl的玻璃珠加入细胞沉淀物中,然后加入1ml体内样试验培养基(描述于vaneunen等人,febsjournal277:749-760中,调整为含有0.5mmdtt(二硫苏糖醇,sigma-aldrich)和0.1mmpmsf(苯基甲磺酰氟,amresco)。通过下述方法加入玻璃珠:在其中每个孔充满了玻璃珠(=体积为300μl)的标准mtp上翻转含有冷冻沉淀物的深孔mtp,然后翻转两个板,使玻璃珠落到细胞沉淀物上。重复该过程,以在细胞沉淀物中得到600μl玻璃珠。然后加入1ml上述体内样试验培养基。细胞破碎后,通过离心(4000rpm,30分钟,4℃)使细胞碎片沉淀。收集上清液(可溶性细胞提取物)并储存在冰上。使用牛血清白蛋白(bsa)作为标准品,通过bradford测定提取物的蛋白质浓度。采用分光光度法通过340nm处吸光度的降低测定苹果酸脱氢酶(mdh)活性,340nm处吸光度的降低是由nadh或nadph分别被氧化成nad+或nadp+引起的。在体内样试验培养基中,试验含有400μmnadh或400μmnadph、2mm草酰乙酸(sigmaaldrich)和约0.0625mg蛋白质ml-1可溶性细胞提取物。以200μl的终体积进行试验。在nadh依赖型试验和nadph依赖型试验中加入等体积的可溶性细胞提取物。通过加入100μl草酰乙酸储备溶液(4mm)开始反应,然后在30摄氏度下9分钟,斜率被用作nadh或nadph依赖型mdh活性的度量。使用microsoftexcel中的“斜率”函数确定斜率(以δa340/min计),其中吸光度值为“y”值,以分钟为单位的时间为“x”值。microsoftexcel中的“rsq”函数被用于检查斜率拟合的质量(标准>0.9)。对含有体内样试验培养基而非底物的空白反应的斜率校正斜率。使用tecaninfinitem1000读板仪测量吸光度。将每种突变体的nadh依赖型活性与nadph活性进行比较。通过以下方法确定nadph依赖型活性:nadh依赖型活性的比值或nadph特异性:nadh特异性的比值:1)确定nadph依赖型mdh活性的斜率(以δa340/min计)2)确定nadh依赖型mdh活性的斜率(以δa340/min计)3)将nadph依赖型mdh活性的斜率除以nadh依赖型mdh活性的斜率。为了确定比值,使用斜率,而不针对总蛋白的量进行均一化,因为在nadh依赖型mdh活性试验和nadph依赖型mdh活性试验中使用等体积的每种突变体。计算19种突变体的比值(图5d)。与参照相比,该比值的值增加指示辅因子特异性已经改变。如“通用的材料和方法”中所述,分析19种培养的突变体和参照菌株的上清液的苹果酸滴度。如上所述测量nadph特异性活性和nadh特异性活性,并通过除以试验中的总蛋白浓度针对总蛋白进行均一化。结果显示在图5b-5c中。显然,19种所选择的突变体具有增强的nadph特异性苹果酸脱氢酶活性。与参照相比,19种突变体中大多数的nadh特异性苹果酸脱氢酶活性降低(图5b)。有趣的是,在6种突变体中,nadh特异性活性提高(图5b),从而指示:在这些突变体中,nadh特异性活性和nadph特异性活性均提高。在所有突变体中,nadph特异性:nadh特异性比值均增加(图5d)。出乎意料地,在第34位用甘氨酸或丝氨酸残基替换天冬氨酸对用这些苹果酸脱氢酶突变体转化的suc-1112菌株的苹果酸滴度具有正面影响(图5a)。实施例6:构建菌株rev-0001生成pcr片段采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:54和seqidno:55中所述的引物序列生产由5’int1整合位点组成的pcr片段116。通过采用seqidno:38作为模板,使用seqidno:56和seqidno:57中所述的引物序列生成pcr片段117。seqidno:38编码在saccharomycescerevisiae中有功能的诺尔丝菌素选择标记,其是从质粒pug7-nat的修改形式扩增的。pug7-nat是gueldener等人(nucleicacidsres.1996年7月;24(13):2519-24)所述的质粒pug6的变体,其中pug6中存在的loxp位点变为lox66和lox71位点(lambert等人,appl.environ.microbiol.2007年2月;73(4):1126-35.epub2006年12月1日),并且其中kanmx标记被诺尔丝菌素标记替代(goldstein和mccusker,yeast.1999年10月;15(14):1541-53)。通过采用seqidno:62作为模板,使用seqidno:60和seqidno:61中所述的引物序列生成pcr片段118。seqidno:62编码来自trypanosomabrucei的延胡索酸还原酶(frdg),如专利申请wo2009/065778中所公开。该合成序列包括启动子-基因-终止子序列,包括适当的限制性位点,由dna2.0(menlopark,california,usa)合成。如专利申请wo2008/000632中所公开,针对在s.cerevisiae中表达对基因序列进行密码子对优化。合成基因受来自s.cerevisiae的启动子控制(或与来自s.cerevisiae的启动子可操作地连接),即tdh3启动子控制frdg基因的表达。恰当的终止由来自s.cerevisiae的终止子序列(即tal1终止子)控制。采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:58和seqidno:59中所述的引物序列生成由3’int1整合位点组成的pcr片段119。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段116至119。转化suc-1029通过本领域技术人员已知的方法进行酵母转化。用纯化的pcr片段116至119转化s.cerevisiae菌株suc-1029(实施例1)。pcr片段117和118在其5’端和3’端具有重叠,pcr片段116和119在其3’端和5’端具有重叠,从而使得允许所有4个pcr片段的同源重组(图1)。pcr片段116的5’端和pcr片段119的3’端与int1基因座同源,使得所有4个pcr片段能够整合在int1基因座(参见图6)。这产生了一个由整合在int1基因座的pcr片段116至119组成的线性片段。专利申请wo2013076280中描述了这种整合方法。int1基因座位于染色体xv上,在yor071c下游659bp且yor070c上游998bp。该方法导致如seqidno:8所示的1139个氨基酸的延胡索酸还原酶蛋白的表达,与来自t.brucei的天然序列相比,所述延胡索酸还原酶蛋白缺少c-末端氨基酸ski。将转化混合物涂布在含有100μg诺尔丝菌素(jenabioscience,德国)/ml的yeph-琼脂(每升:10g酵母提取物,20gphytonepeptone,20g半乳糖,20g琼脂)上。在30℃下生长三天后,将各个转化体重新划线在含有20g半乳糖/升和100μg诺尔丝菌素/ml的yeph-琼脂板上。使用pcr来证实所引入基因的存在,其中使用可以与seqidno:38和seqidno:62编码的orf的编码序列退火的引物序列。实施例7:将编码苹果酸脱氢酶突变体的基因转化到菌株rev-0001中并在所得转化体中生产琥珀酸为了确定表达mdh突变体的菌株中琥珀酸盐/酯水平是否提高,将mdh突变体引入其中表达frdg的菌株rev-0001中(实施例6)。基于苹果酸生产结果(实施例4)和体外活性试验结果(实施例5),选择3种mdh突变体:mut_014、mut_015和mut_034。生成pcr片段pcr片段1、2、3、4和5的扩增描述于实施例2中。为了引入野生型和多样化的mdh突变体,使用pcr片段120(野生型mdh3,seqidno:31)、片段121(mut_014,seqidno:64)、片段122(mut_015,seqidno:65)或片段123(mut_034,seqidno:66)。野生型和突变型mdh3基因由s.cerevisiaetdh3终止子驱动,终止由s.cerevisiaegpm1终止子控制。使用seqidno:19和seqidno:20中所述的引物序列通过pcr扩增盒,以生成pcr片段120至123。采用菌株cen.pk113-7d的基因组dna作为模板,使用seqidno:63和seqidno:41中所述的引物序列生成由3’int59整合位点组成的pcr片段124。使用dnaclean&concentratortm-25试剂盒(zymoresearch,irvine,ca,usa)根据制造商的说明纯化pcr片段1-5/120-123和pcr片段124。转化到rev-0001中通过本领域技术人员已知的方法进行酵母转化。用纯化的pcr片段1至5和124与pcr片段120、121、122或123的组合转化s.cerevisiae菌株rev-0001。pcr片2至5/120-123在其5’端和3’端具有重叠,pcr片段1和124在其3’端和5’端具有重叠,从而使得允许所有7个pcr片段的同源重组。pcr片段1的5’端和pcr片段124的3’端与int59基因座同源,使得所有7个pcr片段能够整合在int59基因座(参见图7)。这产生了一个由整合在int59基因座的pcr片段2至5/120-123组成的线性片段。专利申请wo2013076280中描述了这种整合方法。int59基因座位于染色体xi上,在ykr092c下游923bp且ykr093w上游922bp。将转化混合物涂布在含有20g半乳糖/升和200μgg418(sigmaaldrich,zwijndrecht,荷兰)/ml的yeph-琼脂(每升:10g酵母提取物,20gphytonepeptone,20g琼脂)上。在30℃下生长三天后,将各个转化体重新划线在含有20g半乳糖/升和200μgg418/ml的yeph-琼脂板上。通过pcr来证实所有引入基因的存在。二羧酸生产为了确定二羧酸生产,如“通用的材料和方法”中所述,使具有野生型mdh3或单个mdh突变体的表达琥珀酸产生通路的源自rev-0001的转化体在微量滴定板中生长,并测定二羧酸浓度。表达mut_014、mut_015和mut_34的菌株的琥珀酸滴度分别比表达mdh3的菌株的琥珀酸滴度高1.3倍、1.2倍和1.4倍,从而指示:在表达mdh突变体的菌株中,琥珀酸生产也得到改善。序列表<110>帝斯曼知识产权资产管理有限公司<120>苹果酸脱氢酶<160>67<170>bissap1.2<210>1<211>10504<212>dna<213>artificialsequence<220><221>source<222>1..10504<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>1ttgcccatcgaacgtacaagtactcctctgttctctccttcctttgctttgtgcgacacc60taactacatagtgtttaaagattacggatatttaacttacttagaataatgccatttttt120tgagttataataatcctacgttagtgtgagcgggatttaaactgtgaggaccttaataca180ttcagacacttctgcggtatcaccctacttattcccttcgagattatatctaggaaccca240tcaggttggtggaagattacccgttctaagacttttcagcttcctctattgatgttacac300ctggacaccccttttctggcatccagtttttaatcttcagtggcatgtgagattctccga360aattaattaaagcaatcacacaattctctcggataccacctcggttgaaactgacaggtg420gtttgttacgcatgctaatgcaaaggagcctatatacctttggctcggctgctgtaacag480ggaatataaagggcagcataatttaggagtttagtgaacttgcaacatttactattttcc540cttcttacgtaaatatttttctttttaattctaaatcaatctttttcaattttttgtttg600tattcttttcttgcttaaatctataactacaaaaaacacatacataaactaaaaatgact660gatttgaacaaattggtcaaggaattgaatgatttgggtttgactgacgtcaaggaaatt720gtctacaacccatcttacgaacaattattcgaagaagaaaccaagccaggtttggaaggt780ttcgacaagggtactttgaccactttaggtgctgttgctgttgacaccggtattttcacc840ggtcgttctccaaaggacaaatacattgtttgtgatgaaaccaccaaggacaccgtctgg900tggaactctgaagctgccaagaacgataacaagccaatgactcaagaaacctggaaatct960ttgagagaattggttgccaagcaattgtctggtaagagattattcgttgttgacgctttc1020tgtggtgcttctgaaaagcacagaattggtgtcagaatggtcactgaagttgcttggcaa1080gctcatttcgtcaagaacatgttcatcagaccaactgacgaagaattgaagaacttcaag1140gctgacttcaccgttttgaatggtgccaagtgtaccaacccaaactggaaggaacaaggt1200ttgaactctgaaaactttgttgctttcaacatcactgaaggtatccaattgattggtggt1260acttggtacggtggtgaaatgaagaagggtatgttctccatgatgaactatttcttgcca1320ttgaaaggtgttgcttccatgcactgttctgccaatgtcggtaaggatggtgacgttgcc1380atcttcttcggtctatccggtactggtaagaccactctatccactgacccaaagagacaa1440ttgattggtgatgacgaacacggttgggacgaatctggtgtctttaactttgaaggtggt1500tgttacgccaagaccatcaacttatctcaagaaaacgaaccagatatctacggtgccatc1560cgtcgtgatgctttgttggaaaacgttgttgtcagagctgacggttctgttgacttcgac1620gacggttccaagactgaaaacaccagagtttcttacccaatctaccacattgacaacatt1680gtcagacctgtttccaaggctggtcacgctaccaaggttatcttcttgactgctgatgct1740ttcggtgtcttgccacctgtttccaaattgactccagaacaaaccgaatactacttcttg1800tccggtttcactgccaaattggctggtactgaaagaggtgtcactgaaccaactccaact1860ttctctgcttgtttcggtgctgctttcttatctttgcacccaatccaatacgctgatgtc1920ttggttgaaagaatgaaggcttctggtgctgaagcttacttggtcaacaccggttggaac1980ggtactggtaagagaatctccatcaaggataccagaggtatcattgatgctatcttggac2040ggttccattgaaaaggctgaaatgggtgaattgccaatcttcaacttggccattccaaag2100gctttgccaggtgttgacccagccatcttagatccaagagacacctacgctgacaaggct2160caatggcaagtcaaggctgaagatttggctaacagattcgtcaagaactttgtcaaatac2220actgctaacccagaagctgccaaattggttggtgctggtccaaaggcttaaaggagttaa2280aggcaaagttttcttttctagagccgttcccacaaataattatacgtatatgcttctttt2340cgtttactatatatctatatttacaagcctttattcactgatgcaatttgtttccaaata2400cttttttggagatctcataactagatatcatgatggcgcaacttggcgctatcttaatta2460ctctggctgccaggcccgtgtagagggccgcaagaccttctgtacgccatatagtctcta2520agaacttgaacaagtttctagacctattgccgcctttcggatcgctattgttcctccgga2580tcgatgtacacaaccgactgcacccaaacgaacacaaatcttagcattgcccatcgaacg2640tacaagtactcctctgttctctccttcctttgctttgtgcgacacctaactacatagtgt2700ttaaagattacggatatttaacttacttagaataatgccatttttttgagttataataat2760cctacgttagtgtgagcgggatttaaactgtgaggaccttaatacattcagacacttctg2820cggtatcaccctacttattcccttcgagattatatctaggaacccatcaggttggtggaa2880gattacccgttctaagacttttcagcttcctctattgatgttacacctggacaccccttt2940tctggcatccagtttttaatcttcagtggcatgtgagattctccgaaattaattaaagca3000atcacacaattctctcggataccacctcggttgaaactgacaggtggtttgttacgcatg3060ctaatgcaaaggagcctatatacctttggctcggctgctgtaacagggaatataaagggc3120agcataatttaggagtttagtgaacttgcaacatttactattttcccttcttacgtaaat3180atttttctttttaattctaaatcaatctttttcaattttttgtttgtattcttttcttgc3240ttaaatctataactacaaaaaacacatacataaactaaaaatgactgatttgaacaaatt3300ggtcaaggaattgaatgatttgggtttgactgacgtcaaggaaattgtctacaacccatc3360ttacgaacaattattcgaagaagaaaccaagccaggtttggaaggtttcgacaagggtac3420tttgaccactttaggtgctgttgctgttgacaccggtattttcaccggtcgttctccaaa3480ggacaaatacattgtttgtgatgaaaccaccaaggacaccgtctggtggaactctgaagc3540tgccaagaacgataacaagccaatgactcaagaaacctggaaatctttgagagaattggt3600tgccaagcaattgtctggtaagagattattcgttgttgacgctttctgtggtgcttctga3660aaagcacagaattggtgtcagaatggtcactgaagttgcttggcaagctcatttcgtcaa3720gaacatgttcatcagaccaactgacgaagaattgaagaacttcaaggctgacttcaccgt3780tttgaatggtgccaagtgtaccaacccaaactggaaggaacaaggtttgaactctgaaaa3840ctttgttgctttcaacatcactgaaggtatccaattgattggtggtacttggtacggtgg3900tgaaatgaagaagggtatgttctccatgatgaactatttcttgccattgaaaggtgttgc3960ttccatgcactgttctgccaatgtcggtaaggatggtgacgttgccatcttcttcggtct4020atccggtactggtaagaccactctatccactgacccaaagagacaattgattggtgatga4080cgaacacggttgggacgaatctggtgtctttaactttgaaggtggttgttacgccaagac4140catcaacttatctcaagaaaacgaaccagatatctacggtgccatccgtcgtgatgcttt4200gttggaaaacgttgttgtcagagctgacggttctgttgacttcgacgacggttccaagac4260tgaaaacaccagagtttcttacccaatctaccacattgacaacattgtcagacctgtttc4320caaggctggtcacgctaccaaggttatcttcttgactgctgatgctttcggtgtcttgcc4380acctgtttccaaattgactccagaacaaaccgaatactacttcttgtccggtttcactgc4440caaattggctggtactgaaagaggtgtcactgaaccaactccaactttctctgcttgttt4500cggtgctgctttcttatctttgcacccaatccaatacgctgatgtcttggttgaaagaat4560gaaggcttctggtgctgaagcttacttggtcaacaccggttggaacggtactggtaagag4620aatctccatcaaggataccagaggtatcattgatgctatcttggacggttccattgaaaa4680ggctgaaatgggtgaattgccaatcttcaacttggccattccaaaggctttgccaggtgt4740tgacccagccatcttagatccaagagacacctacgctgacaaggctcaatggcaagtcaa4800ggctgaagatttggctaacagattcgtcaagaactttgtcaaatacactgctaacccaga4860agctgccaaattggttggtgctggtccaaaggcttaaaggagttaaaggcaaagttttct4920tttctagagccgttcccacaaataattatacgtatatgcttcttttcgtttactatatat4980ctatatttacaagcctttattcactgatgcaatttgtttccaaatacttttttggagatc5040tcataactagatatcatgatggcgcaacttggcgctatcttaattactctggctgccagg5100cccgtgtagagggccgcaagaccttctgtacgccatatagtctctaagaacttgaacaag5160tttctagacctattgccgcctttcggatcgctattgttcctccggatcgatgtacacaac5220cgactgcacccaaacgaacacaaatcttagcattgcccatcgaacgtacaagtactcctc5280tgttctctccttcctttgctttgtgcgacacctaactacatagtgtttaaagattacgga5340tatttaacttacttagaataatgccatttttttgagttataataatcctacgttagtgtg5400agcgggatttaaactgtgaggaccttaatacattcagacacttctgcggtatcaccctac5460ttattcccttcgagattatatctaggaacccatcaggttggtggaagattacccgttcta5520agacttttcagcttcctctattgatgttacacctggacaccccttttctggcatccagtt5580tttaatcttcagtggcatgtgagattctccgaaattaattaaagcaatcacacaattctc5640tcggataccacctcggttgaaactgacaggtggtttgttacgcatgctaatgcaaaggag5700cctatatacctttggctcggctgctgtaacagggaatataaagggcagcataatttagga5760gtttagtgaacttgcaacatttactattttcccttcttacgtaaatatttttctttttaa5820ttctaaatcaatctttttcaattttttgtttgtattcttttcttgcttaaatctataact5880acaaaaaacacatacataaactaaaaatgactgatttgaacaaattggtcaaggaattga5940atgatttgggtttgactgacgtcaaggaaattgtctacaacccatcttacgaacaattat6000tcgaagaagaaaccaagccaggtttggaaggtttcgacaagggtactttgaccactttag6060gtgctgttgctgttgacaccggtattttcaccggtcgttctccaaaggacaaatacattg6120tttgtgatgaaaccaccaaggacaccgtctggtggaactctgaagctgccaagaacgata6180acaagccaatgactcaagaaacctggaaatctttgagagaattggttgccaagcaattgt6240ctggtaagagattattcgttgttgacgctttctgtggtgcttctgaaaagcacagaattg6300gtgtcagaatggtcactgaagttgcttggcaagctcatttcgtcaagaacatgttcatca6360gaccaactgacgaagaattgaagaacttcaaggctgacttcaccgttttgaatggtgcca6420agtgtaccaacccaaactggaaggaacaaggtttgaactctgaaaactttgttgctttca6480acatcactgaaggtatccaattgattggtggtacttggtacggtggtgaaatgaagaagg6540gtatgttctccatgatgaactatttcttgccattgaaaggtgttgcttccatgcactgtt6600ctgccaatgtcggtaaggatggtgacgttgccatcttcttcggtctatccggtactggta6660agaccactctatccactgacccaaagagacaattgattggtgatgacgaacacggttggg6720acgaatctggtgtctttaactttgaaggtggttgttacgccaagaccatcaacttatctc6780aagaaaacgaaccagatatctacggtgccatccgtcgtgatgctttgttggaaaacgttg6840ttgtcagagctgacggttctgttgacttcgacgacggttccaagactgaaaacaccagag6900tttcttacccaatctaccacattgacaacattgtcagacctgtttccaaggctggtcacg6960ctaccaaggttatcttcttgactgctgatgctttcggtgtcttgccacctgtttccaaat7020tgactccagaacaaaccgaatactacttcttgtccggtttcactgccaaattggctggta7080ctgaaagaggtgtcactgaaccaactccaactttctctgcttgtttcggtgctgctttct7140tatctttgcacccaatccaatacgctgatgtcttggttgaaagaatgaaggcttctggtg7200ctgaagcttacttggtcaacaccggttggaacggtactggtaagagaatctccatcaagg7260ataccagaggtatcattgatgctatcttggacggttccattgaaaaggctgaaatgggtg7320aattgccaatcttcaacttggccattccaaaggctttgccaggtgttgacccagccatct7380tagatccaagagacacctacgctgacaaggctcaatggcaagtcaaggctgaagatttgg7440ctaacagattcgtcaagaactttgtcaaatacactgctaacccagaagctgccaaattgg7500ttggtgctggtccaaaggcttaaaggagttaaaggcaaagttttcttttctagagccgtt7560cccacaaataattatacgtatatgcttcttttcgtttactatatatctatatttacaagc7620ctttattcactgatgcaatttgtttccaaatacttttttggagatctcataactagatat7680catgatggcgcaacttggcgctatcttaattactctggctgccaggcccgtgtagagggc7740cgcaagaccttctgtacgccatatagtctctaagaacttgaacaagtttctagacctatt7800gccgcctttcggatcgctattgttcctccggatcgatgtacacaaccgactgcacccaaa7860cgaacacaaatcttagcattgcccatcgaacgtacaagtactcctctgttctctccttcc7920tttgctttgtgcgacacctaactacatagtgtttaaagattacggatatttaacttactt7980agaataatgccatttttttgagttataataatcctacgttagtgtgagcgggatttaaac8040tgtgaggaccttaatacattcagacacttctgcggtatcaccctacttattcccttcgag8100attatatctaggaacccatcaggttggtggaagattacccgttctaagacttttcagctt8160cctctattgatgttacacctggacaccccttttctggcatccagtttttaatcttcagtg8220gcatgtgagattctccgaaattaattaaagcaatcacacaattctctcggataccacctc8280ggttgaaactgacaggtggtttgttacgcatgctaatgcaaaggagcctatatacctttg8340gctcggctgctgtaacagggaatataaagggcagcataatttaggagtttagtgaacttg8400caacatttactattttcccttcttacgtaaatatttttctttttaattctaaatcaatct8460ttttcaattttttgtttgtattcttttcttgcttaaatctataactacaaaaaacacata8520cataaactaaaaatgactgatttgaacaaattggtcaaggaattgaatgatttgggtttg8580actgacgtcaaggaaattgtctacaacccatcttacgaacaattattcgaagaagaaacc8640aagccaggtttggaaggtttcgacaagggtactttgaccactttaggtgctgttgctgtt8700gacaccggtattttcaccggtcgttctccaaaggacaaatacattgtttgtgatgaaacc8760accaaggacaccgtctggtggaactctgaagctgccaagaacgataacaagccaatgact8820caagaaacctggaaatctttgagagaattggttgccaagcaattgtctggtaagagatta8880ttcgttgttgacgctttctgtggtgcttctgaaaagcacagaattggtgtcagaatggtc8940actgaagttgcttggcaagctcatttcgtcaagaacatgttcatcagaccaactgacgaa9000gaattgaagaacttcaaggctgacttcaccgttttgaatggtgccaagtgtaccaaccca9060aactggaaggaacaaggtttgaactctgaaaactttgttgctttcaacatcactgaaggt9120atccaattgattggtggtacttggtacggtggtgaaatgaagaagggtatgttctccatg9180atgaactatttcttgccattgaaaggtgttgcttccatgcactgttctgccaatgtcggt9240aaggatggtgacgttgccatcttcttcggtctatccggtactggtaagaccactctatcc9300actgacccaaagagacaattgattggtgatgacgaacacggttgggacgaatctggtgtc9360tttaactttgaaggtggttgttacgccaagaccatcaacttatctcaagaaaacgaacca9420gatatctacggtgccatccgtcgtgatgctttgttggaaaacgttgttgtcagagctgac9480ggttctgttgacttcgacgacggttccaagactgaaaacaccagagtttcttacccaatc9540taccacattgacaacattgtcagacctgtttccaaggctggtcacgctaccaaggttatc9600ttcttgactgctgatgctttcggtgtcttgccacctgtttccaaattgactccagaacaa9660accgaatactacttcttgtccggtttcactgccaaattggctggtactgaaagaggtgtc9720actgaaccaactccaactttctctgcttgtttcggtgctgctttcttatctttgcaccca9780atccaatacgctgatgtcttggttgaaagaatgaaggcttctggtgctgaagcttacttg9840gtcaacaccggttggaacggtactggtaagagaatctccatcaaggataccagaggtatc9900attgatgctatcttggacggttccattgaaaaggctgaaatgggtgaattgccaatcttc9960aacttggccattccaaaggctttgccaggtgttgacccagccatcttagatccaagagac10020acctacgctgacaaggctcaatggcaagtcaaggctgaagatttggctaacagattcgtc10080aagaactttgtcaaatacactgctaacccagaagctgccaaattggttggtgctggtcca10140aaggcttaaaggagttaaaggcaaagttttcttttctagagccgttcccacaaataatta10200tacgtatatgcttcttttcgtttactatatatctatatttacaagcctttattcactgat10260gcaatttgtttccaaatacttttttggagatctcataactagatatcatgatggcgcaac10320ttggcgctatcttaattactctggctgccaggcccgtgtagagggccgcaagaccttctg10380tacgccatatagtctctaagaacttgaacaagtttctagacctattgccgcctttcggat10440cgctattgttcctccggatcgatgtacacaaccgactgcacccaaacgaacacaaatctt10500agca10504<210>2<211>4552<212>dna<213>artificialsequence<220><221>source<222>1..4552<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>2cggatcgatgtacacaaccgactgcacccaaacgaacacaaatcttagcagtgcgggcca60gaaaaaggaagtgtttccctccttcttgaattgatgttaccctcataaagcacgtggcct120cttatcgagaaagaaattaccgtcgctcgtgatttgtttgcaaaaagaacaaaactgaaa180aaacccagacacgctcgacttcctgtcttcctattgattgcagcttccaatttcgtcaca240caacaaggtcctagcgacggctcacaggttttgtaacaagcaatcgaaggttctggaatg300gcgggaaagggtttagtaccacatgctatgatgcccactgtgatctccagagcaaagttc360gttcgatcgtactgttactctctctctttcaaacagaattgtccgaatcgtgtgacaaca420acagcctgttctcacacactcttttcttctaaccaagggggtggtttagtttagtagaac480ctcgtgaaacttacatttacatatatataaacttgcataaattggtcaatgcaagaaata540catatttggtcttttctaattcgtagtttttcaagttcttagatgctttctttttctctt600ttttacagatcatcaaggaagtaattatctactttttacaacaaatataaaacaatgtcc660tcttccaagatcttggctggtttgagagacaacttttctttgttgggtgaaaagaacaag720attttggtcgccaacagaggtgaaatcccaatcagaattttcagatctgctcacgaattg780tctatgagaactatcgccatctactctcacgaagatagattatccatgcacagattgaag840gctgatgaagcctacgttatcggtgaagaaggtcaatacaccccagtcggtgcttacttg900gccatggacgaaatcatcgaaattgccaagaagcacaaggtcgatttcatccacccaggt960tacggtttcttgtctgaaaactctgaatttgctgacaaggttgttaaggctggtattacc1020tggattggtccaccagctgaagtcattgaatctgttggtgacaaggtttctgccagacat1080ttggctgctcgtgccaacgttccaactgtcccaggtactccaggtcctatcgaaaccgtt1140caagaagctctagatttcgtcaatgaatacggttacccagttatcatcaaggctgctttc1200ggtggtggtggtcgtggtatgagagttgtcagagaaggtgacgatgtcgctgatgctttc1260caaagagccacttctgaagctagaactgctttcggtaacggtacttgtttcgtcgaaaga1320ttcttggacaagccaaagcacattgaagttcaattattagctgacaaccacggtaacgtt1380gtccacttgttcgaaagagactgttccgtccaaagacgtcaccaaaaggttgtcgaagtt1440gctccagctaagactttaccaagagaagttagagatgctatcttgaccgatgccgttaag1500ttggctaaggtttgtggttacagaaacgctggtactgctgaattcttggttgacaaccaa1560aacagacattacttcattgaaatcaacccaagaattcaagtcgaacacaccatcactgaa1620gaaatcactggtattgacattgtctccgctcaaatccaaatcgccgctggtgctactttg1680actcaattaggtctattacaagacaaaatcaccaccagaggtttctctatccaatgtcgt1740atcaccactgaagatccatccaagaacttccaaccagacactggtcgtttggaagtctac1800agatccgctggtggtaacggtgtcagattggacggtggtaacgcctacgctggtgctacc1860atctctccacactacgactccatgttggttaagtgttcctgttctggttctacctacgaa1920attgtcagaagaaagatgatcagagctttgattgaattcagaatcagaggtgtcaagacc1980aacatcccattcttgttgactttgttgaccaacccagttttcattgaaggtacctactgg2040accactttcatcgatgacactccacaattgttccaaatggtttcctctcaaaacagagct2100caaaaattgttgcactacttggctgacttggccgtcaacggttcctctatcaagggtcaa2160atcggtttaccaaagttgaagtccaacccttccgttccacatttgcacgatgctcaaggt2220aatgtcatcaacgttaccaaatctgccccaccatccggttggagacaagtcttgttggaa2280aagggtccatccgaatttgccaagcaagtcagacaattcaacggtactttgttgatggac2340accacctggagagatgctcaccaatctttgctagctaccagagtcagaactcacgatttg2400gccaccattgctccaaccactgctcacgctttggctggtgcctttgctttggaatgttgg2460ggtggtgctactttcgatgtcgccatgagattcttgcatgaggacccatgggaaagattg2520agaaaattgagatctttggtcccaaacattccattccaaatgttgttgagaggtgctaac2580ggtgttgcttactcctctttgccagacaacgccattgaccatttcgttaagcaagccaag2640gacaatggtgttgacattttcagagtctttgacgctttgaacgacttggaacaattgaag2700gttggtgttaatgctgtcaagaaggctggtggtgttgtcgaagctaccgtttgttactct2760ggtgacatgttgcaaccaggtaagaaatacaacttggactactacttagaagttgtcgaa2820aagatcgttcaaatgggtactcacatcttgggtatcaaggacatggctggtaccatgaag2880ccagctgctgccaaattgttgattggttctttacgtaccagatacccagacttgccaatc2940cacgttcactctcatgactccgctggtactgctgttgcttccatgactgcttgtgctttg3000gccggtgctgatgttgttgacgttgccattaactccatgtccggtttgacctctcaacca3060tctattaacgctttgttggcctccttggaaggtaacattgacactggtatcaacgtcgaa3120cacgttagagaattggacgcttactgggctgaaatgagattattatactcttgtttcgaa3180gctgacttgaagggtccagaccctgaagtttaccaacacgaaattccaggtggtcaattg3240accaacttgttgttccaagctcaacaattaggtctaggtgaacaatgggctgaaaccaag3300agagcttacagagaagctaactacttgttgggtgacattgttaaggtcaccccaacttct3360aaggtcgttggtgatttggctcaattcatggtttctaacaaattgacttctgatgacatc3420agaagattagctaactctttggacttcccagactccgttatggacttcttcgaaggtttg3480atcggtcaaccatacggtggtttcccagaaccattgagatccgatgttttgagaaacaag3540cgtcgtaaattgacttgtagaccaggtttagaattggaaccattcgatttggaaaagatc3600agagaagatttgcaaaacagattcggtgatatcgatgaatgtgatgttgcctcctacaac3660atgtatcctcgtgtctacgaagatttccaaaagattagagaaacttacggtgacttgtct3720gtcttaccaaccaagaacttcttggctccagctgaaccagacgaagaaatcgaagtcacc3780attgaacaaggtaagactttgattatcaaattacaagctgttggtgatttgaacaagaaa3840accggtcaaagagaagtctacttcgaattgaacggtgaattgagaaagatcagagttgct3900gacaaatctcaaaacattcaatctgttgccaagccaaaggctgatgtccacgacacccac3960caaatcggtgctccaatggctggtgtcatcattgaagtcaaggttcacaagggttctttg4020gtcaagaagggtgaatctatcgccgttttgtctgctatgaagatggaaatggttgtttcc4080tctccagctgatggtcaagtcaaagatgtctttatccgtgacggtgaatccgtcgatgct4140tctgacttgttggttgttttggaagaagaaactctaccaccttctcaaaagaaataaagc4200gaatttcttatgatttatgatttttattattaaataagttataaaaaaaataagtgtata4260caaattttaaagtgactcttaggttttaaaacgaaaattcttattcttgagtaactcttt4320cctgtaggtcaggttgctttctcaggtatagcatgaggtcgctcttattgaccacacctc4380taccggcatgccgagcaaatgcctgcaaatcgctccccatttcacccaattgtagatatg4440ctaactccagcaatgagttgatgaatctcggtgtgtattttatgtcctcagaggacaacc4500tcacgctttccggcatcttccagaccacagtatatccatccgcctcctgttg4552<210>3<211>1572<212>dna<213>artificialsequence<220><221>source<222>1..1572<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>3tcgtacgctgcaggtcgacgaattctaccgttcgtataatgtatgctatacgaagttata60gatctgtttagcttgcctcgtccccgccgggtcacccggccagcgacatggaggcccaga120ataccctccttgacagtcttgacgtgcgcagctcaggggcatgatgtgactgtcgcccgt180acatttagcccatacatccccatgtataatcatttgcatccatacattttgatggccgca240cggcgcgaagcaaaaattacggctcctcgctgcagacctgcgagcagggaaacgctcccc300tcacagacgcgttgaattgtccccacgccgcgcccctgtagagaaatataaaaggttagg360atttgccactgaggttcttctttcatatacttccttttaaaatcttgctaggatacagtt420ctcacatcacatccgaacataaacaaccatgggtaaggaaaagactcacgtttcgaggcc480gcgattaaattccaacatggatgctgatttatatgggtataaatgggctcgcgataatgt540cgggcaatcaggtgcgacaatctatcgattgtatgggaagcccgatgcgccagagttgtt600tctgaaacatggcaaaggtagcgttgccaatgatgttacagatgagatggtcagactaaa660ctggctgacggaatttatgcctcttccgaccatcaagcattttatccgtactcctgatga720tgcatggttactcaccactgcgatccccggcaaaacagcattccaggtattagaagaata780tcctgattcaggtgaaaatattgttgatgcgctggcagtgttcctgcgccggttgcattc840gattcctgtttgtaattgtccttttaacagcgatcgcgtatttcgtctcgctcaggcgca900atcacgaatgaataacggtttggttgatgcgagtgattttgatgacgagcgtaatggctg960gcctgttgaacaagtctggaaagaaatgcataagcttttgccattctcaccggattcagt1020cgtcactcatggtgatttctcacttgataaccttatttttgacgaggggaaattaatagg1080ttgtattgatgttggacgagtcggaatcgcagaccgataccaggatcttgccatcctatg1140gaactgcctcggtgagttttctccttcattacagaaacggctttttcaaaaatatggtat1200tgataatcctgatatgaataaattgcagtttcatttgatgctcgatgagtttttctaatc1260agtactgacaataaaaagattcttgttttcaagaacttgtcatttgtatagtttttttat1320attgtagttgttctattttaatcaaatgttagcgtgatttatattttttttcgcctcgac1380atcatctgcccagatgcgaagttaagtgcgcagaaagtaatatcatgcgtcaatcgtatg1440tgaatgctggtcgctatactgctgtcgattcgatactaacgccgccatccagtgtcgaaa1500acgagctcataacttcgtataatgtatgctatacgaacggtagaattcgatatcagatcc1560actagtggccta1572<210>4<211>2260<212>dna<213>artificialsequence<220><221>source<222>1..2260<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>4aacgttgtccaggtttgtatccacgtgtgtccgttccgccaatattccgcgtgcccgcgg60aaccgccagatattcattacttgacgcaaaagcgtttgaaataatgacgaaaaagaagga120agaaaaaaaaagaaaaataccgcttctaggcgggttatctactgatccgagcttccacta180ggatagcacccaaacacctgcatatttggacgacctttacttacaccaccaaaaaccact240ttcgcctctcccgcccctgataacgtccactaattgagcgattacctgagcggtcctctt300ttgtttgcagcatgagacttgcatactgcaaatcgtaagtagcaacgtgtcaaggtcaaa360actgtatggaaaccttgtcacctcacttaattctagctagcctaccctgcaagtcaagag420gtgtccgtgattcctagccacctcaaggtatgcctctccccggaaactgtggccttttct480ggcacacatgatctccacgatttcaacatataaatagcttttgataatggcaatattaat540caaatttattttacttctttcttgtaacatctctcttgtaatcccttattccttctagct600atttttcataaaaaaccaagcaactgcttatcaacacacaaacactaaatcaaaatgaac660gttgaaacttctttgccaggttcttctggttctgacttggaaactttccaccacgaaacc720aagaagcatgccaaccacgactctggtatttccgtcaaccatgaagctgaaattggtgtt780aaccacactttcgaaaagccaggtccagttggtatcagagaaagattacgtcacttcacc840tgggcttggtacactttgaccatgtcctgtggtggtttggctttgttgattgtcaaccaa900ccacacgacttcaagggtttgaaagatattgccagagttgtctactgtttgaacttggct960ttctttgttatcgttacctctttgatggccatcagattcatcttgcacaagaacatgtgg1020gaatccttgggtcacgacagagaaggtttgtttttcccaactttctggttatccattgct1080accatgatcactggtttgtacaagtgtttcggtgatgatgctaacgaaaagttcaccaag1140tgtttgcaagttttgttctggatctactgtggttgtaccatgatcactgctgtcggtcaa1200tactctttcgtctttgctacccacaaatacgaattgcacaccatgatgccatcctggatc1260ttgccagctttcccagttatgttgtctggtactatcgcctccgtcatcggttctggtcaa1320ccagcttccgatggtattccaattattattgctggtatcactttccaaggtttaggtttc1380tccatctccttcatgatgtacgctcactacattggtagattgatggaagttggtttacca1440tctccagaacacagaccaggtatgttcatctgtgttggtcctccagctttcaccgctttg1500gctttggtcggtatggccaaggctttaccagacgacttccaaattgtcggtgaccctcac1560gctgtcattgacggtcgtgttatgttgttcttggctgtctctgctgccatcttcttatgg1620gctttgtctttctggttcttctgtatcgctgttgttgctgttgtcagatctccaccaaag1680ggtttccatttgaactggtttgccatggttttcccaaacactggtttcaccttggctacc1740atcactttggctaacatgttcgaatctccaggtgtcaagggtgttgccactgctatgtcc1800ctatgtgtcatcatcatgtttattttcgtcttggtttctgccatcagagctgtcatcaga1860aaggacatcatgtggccaggtcaagatgaagatgtttctgaataaagagtaataattatt1920gcttccatataatatttttatatacctcttatttttatgtattagttaattaagtatttt1980tatctatctgcttatcattttcttttcatatagggggggttggtgttttcttgcccatca2040gattgatgtcctccaactcggcactattttacaaagggtttttttgtaagagaaggagaa2100gacagatactaaaccatacgttactcgaaacaaaaaaaaaaaaaatggaaaaagctgcta2160tcaacaaaagacggcctcatcaaacctaaagaaaccatgtcagcgtcctcaaataaccac2220aaacatccttcccatatgctcggtcgtgcttgttgtacct2260<210>5<211>2432<212>dna<213>artificialsequence<220><221>source<222>1..2432<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>5aaataaccacaaacatccttcccatatgctcggtcgtgcttgttgtacctgtgcttttct60ttttttgcggtcacccccatgtggcggggaggcagaggagtaggtagagcaacgaatcct120actatttatccaaattagtctaggaactctttttctagattttttagatttgagggcaag180cgctgttaacgactcagaaatgtaagcactacggagtagaacgagaaatccgccataggt240ggaaatcctagcaaaatcttgcttaccctagctagcctcaggtaagctagccttagcctg300tcaaatttttttcaaaatttggtaagtttctactagcaaagcaaacacggttcaacaaac360cgaaaactccactcattatacgtggaaaccgaaacaaaaaaacaaaaaccaaaatactcg420ccaatgagaaagttgctgcgtttctactttcgaggaagaggaactgagaggattgactac480gaaaggggcaaaaacgagtcgtattctcccattattgtctgctaccacgcggtctagtag540aataagcaaccagtcaacgctaagacaggtaatcaaaataccagtctgctggctacgggc600tagtttttacctcttttagaacccactgtaaaagtccgttgtaaagcccgttctcactgt660tggcgtttttttttttttggtttagtttcttatttttcatttttttctttcatgaccaaa720aacaaacaaatctcgcgatttgtactgcggccactggggcgtggccaaaaaaatgacaaa780tttagaaaccttagtttctgatttttcctgttatgaggagatatgataaaaaatattact840gctttattgttttttttttatctactgaaatagagaaacttacccaaggaggaggcaaaa900aaaagagtatatatacagcaggtaccattcagattttaatatattcttttctcttcttct960acactattattataataattttactatattcatttttagcttaaaacctcatagaatatt1020attcttcagtcactcgcttaaatacttatcaaaaatggttaaggttgccatcttaggtgc1080ttctggtggtgtcggtcaaccattatctctattattgaaattgtctccatacgtttctga1140attggctttgtacgatatcagagctgctgaaggtattggtaaggatttgtcccacatcaa1200caccaactcctcttgtgttggttacgacaaggattccatcgaaaacactttgtccaatgc1260tcaagttgtcttgattccagctggtgttccaagaaagccaggtttgaccagagatgattt1320gttcaagatgaacgctggtatcgttaagtctttggttactgctgtcggtaaatttgcccc1380aaacgctcgtatcttagtcatctccaaccctgttaactctttggttccaattgccgttga1440aactttgaagaagatgggtaagttcaagccaggtaacgttatgggtgtcaccaacttgga1500tttggtcagagctgaaactttcttggttgactacttgatgttgaagaacccaaagatcgg1560tcaagaacaagacaagaccaccatgcacagaaaggtcaccgtcatcggtggtcactctgg1620tgaaaccatcattccaatcatcactgacaaatccttggttttccaattggacaagcaata1680cgaacatttcatccacagagtccaattcggtggtgacgaaattgtcaaggccaagcaagg1740tgccggttctgctaccttgtccatggctttcgctggtgccaaatttgctgaagaagtctt1800acgttctttccacaacgaaaagccagaaactgaatctttgtctgctttcgtctacttgcc1860aggtttgaagaacggtaagaaggctcaacaattagtcggtgacaactccattgaatactt1920ctctttgccaattgttttgagaaacggttccgttgtttccattgacacttctgttttgga1980aaaattgtctccaagagaagaacaattggtcaacactgctgtcaaggaattgagaaagaa2040cattgaaaagggtaagtctttcatcttggacagttaaagtctgaagaatgaatgatttga2100tgatttctttttccctccatttttcttactgaatatatcaatgatatagacttgtatagt2160ttattatttcaaattaagtagctatatatagtcaagataacgtttgtttgacacgattac2220attattcgtcgacatcttttttcagcctgtcgtggtagcaatttgaggagtattattaat2280tgaataggttcattttgcgctcgcataaacagttttcgtcagggacagtatgttggaatg2340agtggtaattaatggtgacatgacatgttatagcaatacctcgaaaccttcgaatccagc2400cagcatgtcgacacccacaagatgtagtgcac2432<210>6<211>2656<212>dna<213>artificialsequence<220><221>source<222>1..2656<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>6gaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcacgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatgtcc660aacaagcctttcatctaccaagctccattcccaatgggtaaggacaacactgaatactac720ttgttgacttctgactacgtttccgttgctgatttcgatggtgaaaccatcttgaaggtt780gaaccagaagccttgactttgttggctcaacaagccttccacgatgcttctttcatgttg840cgtccagctcaccaaaagcaagttgctgccattttgcacgacccagaagcctccgaaaac900gacaaatacgttgctttgcaattcttgagaaactctgaaattgctgccaagggtgtctta960ccaacttgtcaagacactggtactgccatcattgtcggtaagaagggtcaaagagtctgg1020accggtggtggtgacgaagaaactctatccaagggtgtttacaacacttacattgaagat1080aatttacgttactctcaaaatgctgctttggacatgtacaaggaagtcaacactggtact1140aacttgccagctcaaatcgacttatacgctgttgacggtgacgaatacaagttcttgtgt1200gttgccaagggtggtggttctgctaacaagacctacttgtaccaagaaaccaaggctttg1260ttgactccaggtaaattgaagaacttcttggtcgaaaagatgagaactttgggtactgct1320gcttgtccaccataccacattgctttcgttatcggtggtacttccgctgaaaccaacttg1380aaaaccgtcaaattggcttccgctcactactacgatgaattgccaactgaaggtaacgaa1440cacggtcaagccttcagagatgtccaattggaacaagaattgttggaagaagctcaaaaa1500ttaggtttgggtgctcaatttggtggtaaatactttgctcacgatatcagagttatcaga1560ttaccaagacatggtgcttcttgtccagttggtatgggtgtttcctgttctgctgacaga1620aacatcaaggccaagatcaacagagaaggtatctggattgaaaaattggaacacaaccca1680ggtcaatacatcccacaagaattgagacaagctggtgaaggtgaagctgtcaaggttgac1740ttgaacagaccaatgaaggaaatcttggctcaattatctcaatacccagtttccaccaga1800ttatctttgaccggtactatcattgtcggtcgtgacattgctcatgccaagttgaaggaa1860ttgattgatgctggtaaggaattgcctcaatacatcaaggaccatccaatctactacgct1920ggtccagccaagaccccagctggttacccatctggttctttgggtccaaccaccgctggt1980agaatggactcttacgttgacttgctacaatctcacggtggttccatgatcatgttggct2040aagggtaacagatctcaacaagtcaccgatgcttgtcacaagcacggtggtttctatttg2100ggttccattggtggtccagctgctgtcttggctcaacaatctatcaagcacttggaatgt2160gttgcttacccagaattgggtatggaagccatctggaagattgaagtcgaagatttccca2220gctttcatcttagtcgatgacaagggtaacgacttcttccaacaaattgtcaacaagcaa2280tgtgccaactgtaccaagtaaaataaagcaatcttgatgaggataatgatttttttttga2340atatacataaatactaccgtttttctgctagattttgtgaagacgtaaataagtacatat2400tactttttaagccaagacaagattaagcattaactttacccttttctcttctaagtttca2460atactagttatcactgtttaaaagttatggcgagaacgtcggcggttaaaatatattacc2520ctgaacgtggtgaattgaagttctaggatggtttaaagatttttcctttttgggaaataa2580gtaaacaatatattgctgccttcctcaaagccaaagttcgcgttccgaccttgcctccca2640aatccgagttgcgatt2656<210>7<211>4429<212>dna<213>artificialsequence<220><221>source<222>1..4429<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>7aaagccaaagttcgcgttccgaccttgcctcccaaatccgagttgcgattgtgcttggct60gataatagcgtataaacaatgcatactttgtacgttcaaaatacaatgcagtagatatat120ttatgcatattacatataatacatatcacataggaagcaacaggcgcgttggacttttaa180ttttcgaggaccgcgaatccttacatcacacccaatcccccacaagtgatcccccacaca240ccatagcttcaaaatgtttctactccttttttactcttccagattttctcggactccgcg300catcgccgtaccacttcaaaacacccaagcacagcatactaaatttcccctctttcttcc360tctagggtgtcgttaattacccgtactaaaggtttggaaaagaaaaaagacaccgcctcg420tttctttttcttcgtcgaaaaaggcaataaaaatttttatcacgtttctttttcttgaaa480atttttttttttgatttttttctctttcgatgacctcccattgatatttaagttaataaa540cggtcttcaatttctcaagtttcagtttcatttttcttgttctattacaactttttttac600ttcttgctcattagaaagaaagcatagcaatctaatctaagttttaattacaaaatggtt660gatggtagatcttctgcttccattgttgccgttgacccagaaagagctgccagagaaaga720gatgctgctgccagagctttgttgcaagactctccattgcacaccaccatgcaatacgct780acctctggtttggaattgactgttccatacgctttgaaggttgttgcttctgctgacact840ttcgacagagccaaggaagttgctgatgaagtcttgagatgtgcctggcaattggctgac900accgttttgaactctttcaacccaaactctgaagtctctttagtcggtagattaccagtc960ggtcaaaagcatcaaatgtctgctccattgaaacgtgtcatggcttgttgtcaaagagtc1020tacaactcctctgctggttgtttcgacccatccactgctccagttgccaaggctttgaga1080gaaattgctttgggtaaggaaagaaacaatgcttgtttggaagctttgactcaagcttgt1140accttgccaaactctttcgtcattgatttcgaagctggtactatctccagaaagcacgaa1200cacgcttctttggatttgggtggtgtttccaagggttacatcgtcgattacgtcattgac1260aacatcaatgctgctggtttccaaaacgttttctttgactggggtggtgactgtcgtgcc1320tccggtatgaacgccagaaacactccatgggttgtcggtatcactagacctccttccttg1380gacatgttgccaaaccctccaaaggaagcttcttacatctccgtcatctctttggacaat1440gaagctttggctacctctggtgattacgaaaacttgatctacactgctgacgataaacca1500ttgacctgtacctacgattggaaaggtaaggaattgatgaagccatctcaatccaatatc1560gctcaagtttccgtcaagtgttactctgccatgtacgctgacgctttggctaccgcttgt1620ttcatcaagcgtgacccagccaaggtcagacaattgttggatggttggagatacgttaga1680gacaccgtcagagattaccgtgtctacgtcagagaaaacgaaagagttgccaagatgttc1740gaaattgccactgaagatgctgaaatgagaaagagaagaatttccaacactttaccagct1800cgtgtcattgttgttggtggtggtttggctggtttgtccgctgccattgaagctgctggt1860tgtggtgctcaagttgttttgatggaaaaggaagccaagttgggtggtaactctgccaag1920gctacctctggtatcaacggttggggtactagagcccaagctaaggcttccattgtcgat1980ggtggtaagtacttcgaaagagatacctacaagtctggtatcggtggtaacaccgatcca2040gctttggttaagactttgtccatgaaatctgctgacgctatcggttggttgacttctcta2100ggtgttccattgactgttttgtcccaattaggtggtcactccagaaagagaactcacaga2160gccccagacaagaaggatggtactccattgccaattggtttcaccatcatgaaaacttta2220gaagatcatgttagaggtaacttgtccggtagaatcaccatcatggaaaactgttccgtt2280acctctttgttgtctgaaaccaaggaaagaccagacggtactaagcaaatcagagttacc2340ggtgtcgaattcactcaagctggttctggtaagaccaccattttggctgatgctgttatc2400ttggccaccggtggtttctccaacgacaagactgctgattctttgttgagagaacatgcc2460ccacacttggttaacttcccaaccaccaacggtccatgggctactggtgatggtgtcaag2520ttggctcaaagattaggtgctcaattggtcgatatggacaaggttcaattgcacccaact2580ggtttgatcaacccaaaggacccagccaacccaaccaaattcttgggtccagaagctcta2640agaggttctggtggtgttttgttgaacaaacaaggtaagagatttgtcaacgaattggat2700ttgagatctgttgtttccaaggccatcatggaacaaggtgctgaatacccaggttctggt2760ggttccatgtttgcttactgtgtcttgaacgctgctgctcaaaaattgtttggtgtttcc2820tctcacgaattctactggaagaagatgggtttgttcgtcaaggctgacaccatgagagac2880ttggctgctttgattggttgtccagttgaatccgttcaacaaactttagaagaatacgaa2940agattatccatctctcaaagatcttgtccaattaccagaaaatctgtttacccatgtgtt3000ttgggtactaaaggtccatactatgtcgcctttgtcactccatctatccactacaccatg3060ggtggttgtttgatttctccatctgctgaaatccaaatgaagaacacttcttccagagcc3120ccattgtcccactccaacccaatcttgggtttattcggtgctggtgaagtcaccggtggt3180gtccacggtggtaacagattaggtggtaactctttgttggaatgtgttgttttcggtaga3240attgccggtgacagagcttctaccattttgcaaagaaagtcctctgctttgtctttcaag3300gtctggaccactgttgttttgagagaagtcagagaaggtggtgtctacggtgctggttcc3360cgtgtcttgagattcaacttaccaggtgctctacaaagatctggtctatccttgggtcaa3420ttcattgccatcagaggtgactgggacggtcaacaattgattggttactactctccaatc3480actttgccagacgatttgggtatgattgacattttggccagatctgacaagggtacttta3540cgtgaatggatctctgctttggaaccaggtgacgctgtcgaaatgaaggcttgtggtggt3600ttggtcatcgaaagaagattatctgacaagcacttcgttttcatgggtcacattatcaac3660aagctatgtttgattgctggtggtactggtgttgctccaatgttgcaaatcatcaaggcc3720gctttcatgaagccattcatcgacactttggaatccgtccacttgatctacgctgctgaa3780gatgtcactgaattgacttacagagaagttttggaagaacgtcgtcgtgaatccagaggt3840aaattcaagaaaactttcgttttgaacagacctcctccattatggactgacggtgtcggt3900ttcatcgaccgtggtatcttgaccaaccacgttcaaccaccatctgacaacttattggtt3960gccatctgtggtccaccagttatgcaaagaattgtcaaggccactttaaagactttaggt4020tacaacatgaacttggtcagaaccgttgacgaaactgaaccatctggaagttaaaggaag4080tatctcggaaatattaatttaggccatgtccttatgcacgtttcttttgatacttacggg4140tacatgtacacaagtatatctatatatataaattaatgaaaatcccctatttatatatat4200gactttaacgagacagaacagttttttattttttatcctatttgatgaatgatacagttt4260cttattcacgtgttatacccacaccaaatccaatagcaataccggccatcacaatcactg4320tttcggcagcccctaagatcagacaaaacatccggaaccaccttaaatcaacgtccctca4380gaaagcctgtatgcgaagccacaatcctttccaacagaccatactaagt4429<210>8<211>1138<212>prt<213>trypanosomabrucei<220><223>fumaratereductase(frdg)<400>8metvalaspglyargserseralaserilevalalavalaspproglu151015argalaalaarggluargaspalaalaalaargalaleuleuglnasp202530serproleuhisthrthrmetglntyralathrserglyleugluleu354045thrvalprotyralaleulysvalvalalaseralaaspthrpheasp505560argalalysgluvalalaaspgluvalleuargcysalatrpglnleu65707580alaaspthrvalleuasnserpheasnproasnsergluvalserleu859095valglyargleuprovalglyglnlyshisglnmetseralaproleu100105110lysargvalmetalacyscysglnargvaltyrasnserseralagly115120125cyspheaspproserthralaprovalalalysalaleuarggluile130135140alaleuglylysgluargasnasnalacysleuglualaleuthrgln145150155160alacysthrleuproasnserphevalileasppheglualaglythr165170175ileserarglyshisgluhisalaserleuaspleuglyglyvalser180185190lysglytyrilevalasptyrvalileaspasnileasnalaalagly195200205pheglnasnvalphepheasptrpglyglyaspcysargalasergly210215220metasnalaargasnthrprotrpvalvalglyilethrargpropro225230235240serleuaspmetleuproasnproprolysglualasertyrileser245250255valileserleuaspasnglualaleualathrserglyasptyrglu260265270asnleuiletyrthralaaspasplysproleuthrcysthrtyrasp275280285trplysglylysgluleumetlysproserglnserasnilealagln290295300valservallyscystyrseralamettyralaaspalaleualathr305310315320alacyspheilelysargaspproalalysvalargglnleuleuasp325330335glytrpargtyrvalargaspthrvalargasptyrargvaltyrval340345350arggluasngluargvalalalysmetphegluilealathrgluasp355360365alaglumetarglysargargileserasnthrleuproalaargval370375380ilevalvalglyglyglyleualaglyleuseralaalailegluala385390395400alaglycysglyalaglnvalvalleumetglulysglualalysleu405410415glyglyasnseralalysalathrserglyileasnglytrpglythr420425430argalaglnalalysalaserilevalaspglyglylystyrpheglu435440445argaspthrtyrlysserglyileglyglyasnthraspproalaleu450455460vallysthrleusermetlysseralaaspalaileglytrpleuthr465470475480serleuglyvalproleuthrvalleuserglnleuglyglyhisser485490495arglysargthrhisargalaproasplyslysaspglythrproleu500505510proileglyphethrilemetlysthrleugluasphisvalarggly515520525asnleuserglyargilethrilemetgluasncysservalthrser530535540leuleusergluthrlysgluargproaspglythrlysglnilearg545550555560valthrglyvalgluphethrglnalaglyserglylysthrthrile565570575leualaaspalavalileleualathrglyglypheserasnasplys580585590thralaaspserleuleuarggluhisalaprohisleuvalasnphe595600605prothrthrasnglyprotrpalathrglyaspglyvallysleuala610615620glnargleuglyalaglnleuvalaspmetasplysvalglnleuhis625630635640prothrglyleuileasnprolysaspproalaasnprothrlysphe645650655leuglyproglualaleuargglyserglyglyvalleuleuasnlys660665670glnglylysargphevalasngluleuaspleuargservalvalser675680685lysalailemetgluglnglyalaglutyrproglyserglyglyser690695700metphealatyrcysvalleuasnalaalaalaglnlysleuphegly705710715720valserserhisgluphetyrtrplyslysmetglyleuphevallys725730735alaaspthrmetargaspleualaalaleuileglycysprovalglu740745750servalglnglnthrleugluglutyrgluargleuserilesergln755760765argsercysproilethrarglysservaltyrprocysvalleugly770775780thrlysglyprotyrtyrvalalaphevalthrproserilehistyr785790795800thrmetglyglycysleuileserproseralagluileglnmetlys805810815asnthrserserargalaproleuserhisserasnproileleugly820825830leupheglyalaglygluvalthrglyglyvalhisglyglyasnarg835840845leuglyglyasnserleuleuglucysvalvalpheglyargileala850855860glyaspargalaserthrileleuglnarglysserseralaleuser865870875880phelysvaltrpthrthrvalvalleuarggluvalarggluglygly885890895valtyrglyalaglyserargvalleuargpheasnleuproglyala900905910leuglnargserglyleuserleuglyglnpheilealailearggly915920925asptrpaspglyglnglnleuileglytyrtyrserproilethrleu930935940proaspaspleuglymetileaspileleualaargserasplysgly945950955960thrleuargglutrpileseralaleugluproglyaspalavalglu965970975metlysalacysglyglyleuvalilegluargargleuserasplys980985990hisphevalphemetglyhisileileasnlysleucysleuileala99510001005glyglythrglyvalalaprometleuglnileilelysalaalaphe101010151020metlyspropheileaspthrleugluservalhisleuiletyrala1025103010351040alagluaspvalthrgluleuthrtyrarggluvalleuglugluarg104510501055argarggluserargglylysphelyslysthrphevalleuasnarg106010651070proproproleutrpthraspglyvalglypheileaspargglyile107510801085leuthrasnhisvalglnproproseraspasnleuleuvalalaile109010951100cysglyproprovalmetglnargilevallysalathrleulysthr1105111011151120leuglytyrasnmetasnleuvalargthrvalaspgluthrglupro112511301135sergly<210>9<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>9cattatatcgaggaaagccc20<210>10<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>10aaagcaaaggaaggagagaacagaggagtacttgtacgttcgatgggcaaagaaagagac60acaaaactacgtggg75<210>11<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>11ttgcccatcgaacgtacaag20<210>12<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>12tgctaagatttgtgttcgtttgg23<210>13<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>13cggatcgatgtacacaaccg20<210>14<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>14caacaggaggcggatggatatac23<210>15<211>79<212>dna<213>artificialsequence<220><221>source<222>1..79<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>15acgctttccggcatcttccagaccacagtatatccatccgcctcctgttgtcgtacgctg60caggtcgacgaattctacc79<210>16<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>16gcggaatattggcggaacggacacacgtggatacaaacctggacaacgtttaggccacta60gtggatctgatatcg75<210>17<211>22<212>dna<213>artificialsequence<220><221>source<222>1..22<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>17aacgttgtccaggtttgtatcc22<210>18<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>18aggtacaacaagcacgaccg20<210>19<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>19aaataaccacaaacatccttccc23<210>20<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>20gtgcactacatcttgtgggtgtc23<210>21<211>22<212>dna<213>artificialsequence<220><221>source<222>1..22<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>21gaaaccttcgaatccagccagc22<210>22<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>22aatcgcaactcggatttggg20<210>23<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>23aaagccaaagttcgcgttcc20<210>24<211>24<212>dna<213>artificialsequence<220><221>source<222>1..24<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>24acttagtatggtctgttggaaagg24<210>25<211>25<212>dna<213>artificialsequence<220><221>source<222>1..25<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>25cggcattattgtgtatggctcaata25<210>26<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>26gaacttcgacctgttgcaatacttcgggttcggcacaaacgtgtacggatagggtttcaa60agatccatacttctc75<210>27<211>79<212>dna<213>artificialsequence<220><221>source<222>1..79<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>27atccgtacacgtttgtgccgaacccgaagtattgcaacaggtcgaagttctcgtacgctg60caggtcgacgaattctacc79<210>28<211>74<212>dna<213>artificialsequence<220><221>source<222>1..74<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>28aggtacaacaagcacgaccgagcatatgggaaggatgtttgtggttatttaggccactag60tggatctgatatcg74<210>29<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>29aaataaccacaaacatccttccc23<210>30<211>23<212>dna<213>artificialsequence<220><221>source<222>1..23<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>30gtgcactacatcttgtgggtgtc23<210>31<211>2032<212>dna<213>artificialsequence<220><221>source<222>1..2032<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>31aaataaccacaaacatccttcccatatgctcggtcgtgcttgttgtacctgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatggtt660aaggttgccatcttaggtgcttctggtggtgtcggtcaaccattatctctattattgaaa720ttgtctccatacgtttctgaattggctttgtacgatatcagagctgctgaaggtattggt780aaggatttgtcccacatcaacaccaactcctcttgtgttggttacgacaaggattccatc840gaaaacactttgtccaatgctcaagttgtcttgattccagctggtgttccaagaaagcca900ggtttgaccagagatgatttgttcaagatgaacgctggtatcgttaagtctttggttact960gctgtcggtaaatttgccccaaacgctcgtatcttagtcatctccaaccctgttaactct1020ttggttccaattgccgttgaaactttgaagaagatgggtaagttcaagccaggtaacgtt1080atgggtgtcaccaacttggatttggtcagagctgaaactttcttggttgactacttgatg1140ttgaagaacccaaagatcggtcaagaacaagacaagaccaccatgcacagaaaggtcacc1200gtcatcggtggtcactctggtgaaaccatcattccaatcatcactgacaaatccttggtt1260ttccaattggacaagcaatacgaacatttcatccacagagtccaattcggtggtgacgaa1320attgtcaaggccaagcaaggtgccggttctgctaccttgtccatggctttcgctggtgcc1380aaatttgctgaagaagtcttacgttctttccacaacgaaaagccagaaactgaatctttg1440tctgctttcgtctacttgccaggtttgaagaacggtaagaaggctcaacaattagtcggt1500gacaactccattgaatacttctctttgccaattgttttgagaaacggttccgttgtttcc1560attgacacttctgttttggaaaaattgtctccaagagaagaacaattggtcaacactgct1620gtcaaggaattgagaaagaacattgaaaagggtaagtctttcatcttggacagttaaagt1680ctgaagaatgaatgatttgatgatttctttttccctccatttttcttactgaatatatca1740atgatatagacttgtatagtttattatttcaaattaagtagctatatatagtcaagataa1800cgtttgtttgacacgattacattattcgtcgacatcttttttcagcctgtcgtggtagca1860atttgaggagtattattaattgaataggttcattttgcgctcgcataaacagttttcgtc1920agggacagtatgttggaatgagtggtaattaatggtgacatgacatgttatagcaatacc1980tcgaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcac2032<210>32<211>78<212>dna<213>artificialsequence<220><221>source<222>1..78<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>32gaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcacacttttttta60gaatgacctgttcccgac78<210>33<211>24<212>dna<213>artificialsequence<220><221>source<222>1..24<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>33cacaagcttattcttccaaaaatc24<210>34<211>3534<212>dna<213>artificialsequence<220><221>source<222>1..3534<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>34aaaggaggtgcacgcattatggagaccactacgatacgatagctgcgttgttgttgaagg60ggtttcttaaggttgttttcgttgaaggtaaatattggtcgtttttgtgcagcatattgt120cctctagatgcaaactctgcaggtccatttgcagtaaagtgagttgcctctcgaagaatc180attaatttcgtataaccgtcactattaaagtcagaaaataaattctgtcgtagacaatgt240taccataatgttcttgtccattttgcatacactttaaatattcatttgatttctcagggt300tcatgatcataataaattgcgcattcgcaaggcggtagtattataatggggtccatcatt360ctgtagcaagaagttacagtacgctgttcaagcgttaaacaagataagtaatctcgaatg420aaacattcatatttcgcatgagccaacatacagttgctgagtaatcttcattgcgcttat480ttatcggcattgagattgtaaaggaagtaaaacgcatttttgcagatctgttctcttatg540tatttttaatcgtccttgtatggaagtatcaaaggggacgttcttcacctccttggaagg600atcccttcccttttacagtgcttcggaaaagcacagcgttgtccaagggaacaatttttc660ttcaagttaatgcataagaaatatctttttttatgtttagctaagtaaaagcagcttgga720gtaaaaaaaaaaatgagtaaatttctcgatggattagtttctcacaggtaacataacaaa780aaccaagaaaagcccgcttctgaaaactacagttgacttgtatgctaaagggccagacta840atgggaggagaaaaagaaacgaatgtatatgctcatttacactctatatcaccatatgga900ggataagttgggctgagcttctgatccaatttattctatccattagttgctgatatgtcc960caccagccaacacttgatagtatctactcgccattcacttccagcagcgccagtagggtt1020gttgagcttagtaaaaatgtgcgcaccacaagcctacatgactccacgtcacatgaaacc1080acaccgtggggccttgttgcgctaggaataggatatgcgacgaagacgcttctgcttagt1140aaccacaccacattttcagggggtcgatctgcttgcttcctttactgtcacgagcggccc1200ataatcgcgctttttttttaaaaggcgcgagacagcaaacaggaagctcgggtttcaacc1260ttcggagtggtcgcagatctggagactggatctttacaatacagtaaggcaagccaccat1320ctgcttcttaggtgcatgcgacggtatccacgtgcagaacaacatagtctgaagaagggg1380gggaggagcatgttcattctctgtagcagtaagagcttggtgataatgaccaaaactgga1440gtctcgaaatcatataaatagacaatatattttcacacaatgagatttgtagtacagttc1500tattctctctcttgcataaataagaaattcatcaagaacttggtttgatatttcaccaac1560acacacaaaaaacagtacttcactaaatttacacacaaaacaaaatgtcctctgcttctg1620ctgctttgcaaaaattcagagctgaaagagataccttcggtgacttgcaagttccagctg1680accgttactggggtgctcaaactcaaagatctttgcaaaactttgacattggtggtccaa1740ctgaaagaatgccagaaccattaatcagagctttcggtgttttgaagaaggctgctgcca1800ccgtcaacatgacctacggtttggacccaaaggttggtgaagccatccaaaaggctgctg1860acgaagttatcgatggttctttgattgaccatttcccattggttgtctggcaaaccggtt1920ctggtactcaaaccaagatgaacgtcaatgaagtcatctccaacagagccattgaattgt1980tgggtggtgaattaggttccaaggctccagtccacccaaacgatcatgtcaacatgtctc2040aatcttccaacgacactttcccaactgccatgcacgttgctgccgttgttgaaattcacg2100gtagattgattccagctttgaccactttgagagatgctttgcaagccaaatctgctgaat2160tcgaacacatcatcaagattggtagaacccacttgcaagatgctaccccattgactttag2220gtcaagaattctccggttacactcaacaattgacctacggtattgctcgtgttcaaggta2280ctttggaaagattatacaacttggctcaaggtggtactgctgtcggtactggtttgaaca2340ccagaaagggtttcgatgccaaggttgctgaagccattgcttccatcactggtttaccat2400tcaagaccgctccaaacaaattcgaagctttggctgctcacgacgctttggttgaagctc2460acggtgctttgaacaccgttgcttgttctttgatgaagattgccaacgatatccgttact2520tgggttctggtccaagatgtggtttaggtgaattgtctctaccagaaaacgaaccaggtt2580cttccatcatgccaggtaaggtcaacccaactcaatgtgaagctatgaccatggtttgtg2640ctcaagtcatgggtaacaacactgccatctctgttgctggttccaacggtcaattcgaat2700tgaatgtctttaaaccagtcatgatcaagaacttgatccaatccatcagattaatctctg2760acgcttccatctctttcaccaagaactgtgttgtcggtattgaagctaacgaaaagaaga2820tctcctccatcatgaacgaatctttgatgttggtcactgctttgaaccctcacattggtt2880acgacaaggctgccaagtgtgccaagaaggctcacaaggaaggtaccactttgaaagaag2940ctgctctatctttgggttacttgacctctgaagaattcgaccaatgggttagacctgagg3000acatgatttctgccaaggattaaggcccgggcataaagcaatcttgatgaggataatgat3060ttttttttgaatatacataaatactaccgtttttctgctagattttgtgaagacgtaaat3120aagtacatattactttttaagccaagacaagattaagcattaactttacccttttctctt3180ctaagtttcaatactagttatcactgtttaaaagttatggcgagaacgtcggcggttaaa3240atatattaccctgaacgtggtgaattgaagttctaggatggtttaaagatttttcctttt3300tgggaaataagtaaacaatatattgctgcctttgcaaaacgcacatacccacaatatgtg3360actattggcaaagaacgcattatcctttgaagaggtggatactgatactaagagagtctc3420tattccggctccacttttagtccagagattacttgtcttcttacgtatcagaacaagaaa3480gcatttccaaagtaattgcatttgcccttgagcagtatatatatactaagaagg3534<210>35<211>2578<212>dna<213>artificialsequence<220><221>source<222>1..2578<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>35ttccaaagtaattgcatttgcccttgagcagtatatatatactaagaaggtcgacctcga60gtaccgttcgtataatgtatgctatacgaagttatatttaaatcagtatagcgaccagca120ttcacatacgattgacgcatgatattactttctgcgcacttaacttcgcatctgggcaga180tgatgtcgaggcgaaaaaaaatataaatcacgctaacatttgattaaaatagaacaacta240caatataaaaaaactatacaaatgacaagttcttgaaaacaagaatctttttattgtcag300tactgattagaaaaactcatcgagcatcaaatgaaactgcaatttattcatatcaggatt360atcaataccatatttttgaaaaagccgtttctgtaatgaaggagaaaactcaccgaggca420gttccataggatggcaagatcctggtatcggtctgcgattccgactcgtccaacatcaat480acaacctattaatttcccctcgtcaaaaataaggttatcaagtgagaaatcaccatgagt540gacgactgaatccggtgagaatggcaaaagcttatgcatttctttccagacttgttcaac600aggccagccattacgctcgtcatcaaaatcactcgcatcaaccaaaccgttattcattcg660tgattgcgcctgagcgagacgaaatacgcgatcgctgttaaaaggacaattacaaacagg720aatcgaatgcaaccggcgcaggaacactgccagcgcatcaacaatattttcacctgaatc780aggatattcttctaatacctggaatgctgttttgccggggatcgcagtggtgagtaacca840tgcatcatcaggagtacggataaaatgcttgatggtcggaagaggcataaattccgtcag900ccagtttagtctgaccatctcatctgtaacatcattggcaacgctacctttgccatgttt960cagaaacaactctggcgcatcgggcttcccatacaatcgatagattgtcgcacctgattg1020cccgacattatcgcgagcccatttatacccatataaatcagcatccatgttggaatttaa1080tcgcggcctcgaaacgtgagtcttttccttacccatggttgtttatgttcggatgtgatg1140tgagaactgtatcctagcaagattttaaaaggaagtatatgaaagaagaacctcagtggc1200aaatcctaaccttttatatttctctacaggggcgcggcgtggggacaattcaacgcgtct1260gtgaggggagcgtttccctgctcgcaggtctgcagcgaggagccgtaatttttgcttcgc1320gccgtgcggccatcaaaatgtatggatgcaaatgattatacatggggatgtatgggctaa1380atgtacgggcgacagtcacatcatgcccctgagctgcgcacgtcaagactgtcaaggagg1440gtattctgggcctccatgtcatttaaatctagtacggattagaagccgccgagcgggtga1500cagccctccgaaggaagactctcctccgtgcgtcctcgtcttcaccggtcgcgttcctga1560aacgcagatgtgcctcgcgccgcactgctccgaacaataaagattctacaatactagctt1620ttatggttatgaagaggaaaaattggcagtaacctggccccacaaaccttcaaatgaacg1680aatcaaattaacaaccataggatgataatgcgattagttttttagccttatttctggggt1740aattaatcagcgaagcgatgatttttgatctattaacagatatataaatgcaaaaactgc1800ataaccactttaactaatactttcaacattttcggtttgtattacttcttattcaaatgt1860aataaaagtatcaacaaaaaattgttaatatacctctatactttaacgtcaaggagaaaa1920aaccccggattctagaactagtggatcccccgggctgcaggaattcgatatcaagcttat1980cgataccgtcgaggggcagagccgatcctgtacactttacttaaaaccattatctgagtg2040ttaaatgtccaatttactgaccgtacaccaaaatttgcctgcattaccggtcgatgcaac2100gagtgatgaggttcgcaagaacctgatggacatgttcagggatcgccaggcgttttctga2160gcatacctggaaaatgcttctgtccgtttgccggtcgtgggcggcatggtgcaagttgaa2220taaccggaaatggtttcccgcagaacctgaagatgttcgcgattatcttctatatcttca2280ggcgcgcggtctggcagtaaaaactatccagcaacatttgggccagctaaacatgcttca2340tcgtcggtccgggctgccacgaccaagtgacagcaatgctgtttcactggttatgcggcg2400gatccgaaaagaaaacgttgatgccggtgaacgtgcaaaacaggctctagcgttcgaacg2460cactgatttcgaccaggttcgttcactcatggaaaatagcgatcgctgccaggatatacg2520taatctggcatttctggggattgcttataacaccctgttacgtatagccgaaattgcc2578<210>36<211>1263<212>dna<213>artificialsequence<220><221>source<222>1..1263<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>36cgttcactcatggaaaatagcgatcgctgccaggatatacgtaatctggcatttctgggg60attgcttataacaccctgttacgtatagccgaaattgccaggatcagggttaaagatatc120tcacgtactgacggtgggagaatgttaatccatattggcagaacgaaaacgctggttagc180accgcaggtgtagagaaggcacttagcctgggggtaactaaactggtcgagcgatggatt240tccgtctctggtgtagctgatgatccgaataactacctgttttgccgggtcagaaaaaat300ggtgttgccgcgccatctgccaccagccagctatcaactcgcgccctggaagggattttt360gaagcaactcatcgattgatttacggcgctaaggatgactctggtcagagatacctggcc420tggtctggacacagtgcccgtgtcggagccgcgcgagatatggcccgcgctggagtttca480ataccggagatcatgcaagctggtggctggaccaatgtaaatattgtcatgaactatatc540cgtaccctggatagtgaaacaggggcaatggtgcgcctgctggaagatggcgattagcca600ttaacgcgtaaatgattgctataattatttgatatttatggtgacatatgagaaaggatt660tcaacatcgacggaaaatatgtagtgctgtctgtaagcactaatattcagtcgccagccg720tcattgtcactgtaaagctgagcgatagaatgcctgatattgactcaatatccgttgcgt780ttcctgtcaaaagtatgcgtagtgctgaacatttcgtgatgaatgccaccgaggaagaag840cacggcgcggttttgcttaaagtgatgtctgagtttggcgaactcttgggtaaggttgga900attgtcgacctcgagtcatgtaattagttatgtcacgcttacattcacgccctcccccca960catccgctctaaccgaaaaggaaggagttagacaacctgaagtctaggtccctatttatt1020tttttatagttatgttagtattaagaacgttatttatatttcaaatttttcttttttttc1080tgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaaggttttgg1140gacgctcgaaggctttaatttgcggccggtacataacttcgtataatgtatgctatacga1200acggtaggatccggatgggacgtcagcactgtacttgtttttgcgactagattgtaaatc1260att1263<210>37<211>631<212>dna<213>artificialsequence<220><221>source<222>1..631<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>37gatgggacgtcagcactgtacttgtttttgcgactagattgtaaatcattctttatttaa60tctctttctttaactactgcttaaagtataatttggtccgtagtttaataactatactaa120gcgtaacaatgcatactgacattataagcctgaacattacgagtttaagttgtatgtagg180cgttctgtaagaggttactgcgtaaattatcaacgaatgcattggtgtatttgcgaaagc240tacttcttttaacaagtatttacataagaataatggtgatctgctcaactgatttggtga300taactctaacttttttagcaacaatttaaaagataattcgaacatatataacagtaggaa360gaatttgtgtacgtcaaattaagataatttagcattaccaaagttattaacctaaacata420aaatatatatgagacacatgtggaaatcgtatgaaacaactgttatgaaactgacaagaa480tgaatatatagagtaagctccgcttgtaaagaggaatcacttaagtgtataaatgtctcg540acgattactttagatccaagattgatgattgatattactctgtaatacttaagctctttt600aatagctcactgttgtattacgggctcgagt631<210>38<211>1334<212>dna<213>artificialsequence<220><221>source<222>1..1334<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>38tcgtacgctgcaggtcgacgaattctaccgttcgtataatgtatgctatacgaagttata60gatctgtttagcttgcctcgtccccgccgggtcacccggccagcgacatggaggcccaga120ataccctccttgacagtcttgacgtgcgcagctcaggggcatgatgtgactgtcgcccgt180acatttagcccatacatccccatgtataatcatttgcatccatacattttgatggccgca240cggcgcgaagcaaaaattacggctcctcgctgcagacctgcgagcagggaaacgctcccc300tcacagacgcgttgaattgtccccacgccgcgcccctgtagagaaatataaaaggttagg360atttgccactgaggttcttctttcatatacttccttttaaaatcttgctaggatacagtt420ctcacatcacatccgaacataaacaaccatgggtaccactcttgacgacacggcttaccg480gtaccgcaccagtgtcccgggggacgccgaggccatcgaggcactggatgggtccttcac540caccgacaccgtcttccgcgtcaccgccaccggggacggcttcaccctgcgggaggtgcc600ggtggacccgcccctgaccaaggtgttccccgacgacgaatcggacgacgaatcggacga660cggggaggacggcgacccggactcccggacgttcgtcgcgtacggggacgacggcgacct720ggcgggcttcgtggtcgtctcgtactccggctggaaccgccggctgaccgtcgaggacat780cgaggtcgccccggagcaccgggggcacggggtcgggcgcgcgttgatggggctcgcgac840ggagttcgcccgcgagcggggcgccgggcacctctggctggaggtcaccaacgtcaacgc900accggcgatccacgcgtaccggcggatggggttcaccctctgcggcctggacaccgccct960gtacgacggcaccgcctcggacggcgagcaggcgctctacatgagcatgccctgccccta1020atcagtactgacaataaaaagattcttgttttcaagaacttgtcatttgtatagtttttt1080tatattgtagttgttctattttaatcaaatgttagcgtgatttatattttttttcgcctc1140gacatcatctgcccagatgcgaagttaagtgcgcagaaagtaatatcatgcgtcaatcgt1200atgtgaatgctggtcgctatactgctgtcgattcgatactaacgccgccatccagtgtcg1260aaaacgagctcataacttcgtataatgtatgctatacgaacggtagaattcgatatcaga1320tccactagtggcct1334<210>39<211>340<212>prt<213>artificialsequence<220><223>mdh3lacking3c-terminalperoxisomaltargetingsequence<400>39metvallysvalalaileleuglyalaserglyglyvalglyglnpro151015leuserleuleuleulysleuserprotyrvalsergluleualaleu202530tyraspileargalaalagluglyileglylysaspleuserhisile354045asnthrasnsersercysvalglytyrasplysaspserilegluasn505560thrleuserasnalaglnvalvalleuileproalaglyvalproarg65707580lysproglyleuthrargaspaspleuphelysmetasnalaglyile859095vallysserleuvalthralavalglylysphealaproasnalaarg100105110ileleuvalileserasnprovalasnserleuvalproilealaval115120125gluthrleulyslysmetglylysphelysproglyasnvalmetgly130135140valthrasnleuaspleuvalargalagluthrpheleuvalasptyr145150155160leumetleulysasnprolysileglyglngluglnasplysthrthr165170175methisarglysvalthrvalileglyglyhisserglygluthrile180185190ileproileilethrasplysserleuvalpheglnleuasplysgln195200205tyrgluhispheilehisargvalglnpheglyglyaspgluileval210215220lysalalysglnglyalaglyseralathrleusermetalapheala225230235240glyalalysphealaglugluvalleuargserphehisasnglulys245250255progluthrgluserleuseralaphevaltyrleuproglyleulys260265270asnglylyslysalaglnglnleuvalglyaspasnserileglutyr275280285pheserleuproilevalleuargasnglyservalvalserileasp290295300thrservalleuglulysleuserproargglugluglnleuvalasn305310315320thralavallysgluleuarglysasnileglulysglylysserphe325330335ileleuaspser340<210>40<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>40agaaagcctgtatgcgaagccacaatcctttccaacagaccatactaagtacaggtgatt60gtatgtgggcttatg75<210>41<211>29<212>dna<213>artificialsequence<220><221>source<222>1..29<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>41acattattgtaaaaacggagtagaaaggg29<210>42<211>2927<212>dna<213>artificialsequence<220><221>source<222>1..2927<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>42atccgtacacgtttgtgccgaacccgaagtattgcaacaggtcgaagttcgtgccaaagg60gggggcagggacagggatacgacaagggctggggaaaaaaaaaaagatagatacgattgg120ccgggtaagcctggggaaatgtagcaagtgcgggtaagttaaaaggtaaccacgtgactc180cggaagagtcacgtggttacggacttttttctctagatctcagctttttatcggtcttac240cctgccctcctgccccctgccccttccctttgccccaaaaagaaaggaaatctgttggat300ttcgctcaggccatccctttcgttaatatcggttatcgctttacacactgcacatccttc360tgtccaaaaggaatccagaagtttagcttttccttcctttcccacagacattagcctagg420ccctctctcatcatttgcatgcctcagccaatgtaccaagaataacgcaacgaggttggg480aaattttaacccaacaatcgatgcagatgtgacaagagattagacacgttccagatacca540gattacacagcttgtgctagcagagtgacatatggtggtgttgtgtctcgtttagtacct600gtaatcgagagtgttcaaatcagtcgatttgaacacccttactgccactgaatattgatt660gaataccgtttattgaaggttttatgagtgatcttctttcggtccaggacaatttgttga720gctttttctatgtagagttccgtcccttttttttttttttttgctttctcgcacttacta780gcactatttttttttcacacactaaaacactttattttaatctatatatatatatatata840tatatgtaggaatggaatcacagacatttgatactcatcctcatccttattaattcttgt900tttaatttgtttgacttagccaaaccaccaatctcaacccatcgtatttcaggtattgtg960tgtctagtgtgtctctggtatacggaaataagtgccagaagtaaggaagaaacaaagaac1020aagtgtctgaatactactagcctctcttttcataatgagtgaaggccccgtcaaattcga1080aaaaaataccgtcatatctgtctttggtgcgtcaggtgatctggcaaagaagaagacttt1140tcccgccttatttgggcttttcagagaaggttaccttgatccatctaccaagatcttcgg1200ttatgcccggtccaaattgtccatggaggaggacctgaagtcccgtgtcctaccccactt1260gaaaaaacctcacggtgaagccgatgactctaaggtcgaacagttcttcaagatggtcag1320ctacatttcgggaaattacgacacagatgaaggcttcgacgaattaagaacgcagatcga1380gaaattcgagaaaagtgccaacgtcgatgtcccacaccgtctcttctatctggccttgcc1440gccaagcgtttttttgacggtggccaagcagatcaagagtcgtgtgtacgcagagaatgg1500catcacccgtgtaatcgtagagaaacctttcggccacgacctggcctctgccagggagct1560gcaaaaaaacctggggcccctctttaaagaagaagagttgtacagaattgaccattactt1620gggtaaagagttggtcaagaatcttttagtcttgaggttcggtaaccagtttttgaatgc1680ctcgtggaatagagacaacattcaaagcgttcagatttcgtttaaagagaggttcggcac1740cgaaggccgtggcggctatttcgactctataggcataatcagagacgtgatgcagaacca1800tctgttacaaatcatgactctcttgactatggaaagaccggtgtcttttgacccggaatc1860tattcgtgacgaaaaggttaaggttctaaaggccgtggcccccatcgacacggacgacgt1920cctcttgggccagtacggtaaatctgaggacgggtctaagcccgcctacgtggatgatga1980cactgtagacaaggactctaaatgtgtcacttttgcagcaatgactttcaacatcgaaaa2040cgagcgttgggagggcgtccccatcatgatgcgtgccggtaaggctttgaatgagtccaa2100ggtggagatcagactgcagtacaaagcggtcgcatcgggtgtcttcaaagacattccaaa2160taacgaactggtcatcagagtgcagcccgatgccgctgtgtacctaaagtttaatgctaa2220gacccctggtctgtcaaatgctacccaagtcacagatctgaatctaacttacgcaagcag2280gtaccaagacttttggattccagaggcttacgaggtgttgataagagacgccctactggg2340tgaccattccaactttgtcagagatgacgaattggatatcagttggggcatattcacccc2400attactgaagcacatagagcgtccggacggtccaacaccggaaatttacccctacggatc2460aagaggtccaaagggattgaaggaatatatgcaaaaacacaagtatgttatgcccgaaaa2520gcacccttacgcttggcccgtgactaagccagaagatacgaaggataattaaaggagatt2580gataagacttttctagttgcatatcttttatatttaaatcttatctattagttaattttt2640tgtaatttatccttatatatagtctggttattctaaaatatcatttcagtatctaaaaat2700tcccctcttttttcagttatatcttaacaggcgacagtccaaatgttgatttatcccagt2760ccgattcatcagggttgtgaagcattttgtcaatggtcgaaatcacatcagtaatagtgc2820ctcttacttgcctcatagaatttctttctcttaacgtcaccgtttggtcttttcctcacc2880cgatcccgaagaaagtccaactccagttcgattccctgctttcgagt2927<210>43<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>43atccgtacacgtttgtgccg20<210>44<211>22<212>dna<213>artificialsequence<220><221>source<222>1..22<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>44actcgaaagcagggaatcgaac22<210>45<211>79<212>dna<213>artificialsequence<220><221>source<222>1..79<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>45acccgatcccgaagaaagtccaactccagttcgattccctgctttcgagttcgtacgctg60caggtcgacgaattctacc79<210>46<211>1180<212>prt<213>saccharomycescerevisiae<400>46metserserserlysileleualaglyleuargaspasnpheserleu151015leuglyglulysasnlysileleuvalalaasnargglygluilepro202530ileargilepheargseralahisgluleusermetargthrileala354045iletyrserhisgluaspargleusermethisargleulysalaasp505560glualatyrvalileglyglugluglyglntyrthrprovalglyala65707580tyrleualametaspgluileilegluilealalyslyshislysval859095asppheilehisproglytyrglypheleusergluasnsergluphe100105110alaasplysvalvallysalaglyilethrtrpileglyproproala115120125gluvalilegluservalglyasplysvalseralaarghisleuala130135140alaargalaasnvalprothrvalproglythrproglyproileglu145150155160thrvalglnglualaleuaspphevalasnglutyrglytyrproval165170175ileilelysalaalapheglyglyglyglyargglymetargvalval180185190arggluglyaspaspvalalaaspalapheglnargalathrserglu195200205alaargthralapheglyasnglythrcysphevalgluargpheleu210215220asplysprolyshisilegluvalglnleuleualaaspasnhisgly225230235240asnvalvalhisleuphegluargaspcysservalglnargarghis245250255glnlysvalvalgluvalalaproalalysthrleuproarggluval260265270argaspalaileleuthraspalavallysleualalysvalcysgly275280285tyrargasnalaglythralaglupheleuvalaspasnglnasnarg290295300histyrpheilegluileasnproargileglnvalgluhisthrile305310315320thrglugluilethrglyileaspilevalseralaglnileglnile325330335alaalaglyalathrleuthrglnleuglyleuleuglnasplysile340345350thrthrargglypheserileglncysargilethrthrgluasppro355360365serlysasnpheglnproaspthrglyargleugluvaltyrargser370375380alaglyglyasnglyvalargleuaspglyglyasnalatyralagly385390395400alathrileserprohistyraspsermetleuvallyscyssercys405410415serglyserthrtyrgluilevalargarglysmetileargalaleu420425430ileglupheargileargglyvallysthrasnilepropheleuleu435440445thrleuleuthrasnprovalpheilegluglythrtyrtrpthrthr450455460pheileaspaspthrproglnleupheglnmetvalserserglnasn465470475480argalaglnlysleuleuhistyrleualaaspleualavalasngly485490495serserilelysglyglnileglyleuprolysleulysserasnpro500505510servalprohisleuhisaspalaglnglyasnvalileasnvalthr515520525lysseralaproproserglytrpargglnvalleuleuglulysgly530535540prosergluphealalysglnvalargglnpheasnglythrleuleu545550555560metaspthrthrtrpargaspalahisglnserleuleualathrarg565570575valargthrhisaspleualathrilealaprothrthralahisala580585590leualaglyalaphealaleuglucystrpglyglyalathrpheasp595600605valalametargpheleuhisgluaspprotrpgluargleuarglys610615620leuargserleuvalproasnilepropheglnmetleuleuarggly625630635640alaasnglyvalalatyrserserleuproaspasnalaileasphis645650655phevallysglnalalysaspasnglyvalaspilepheargvalphe660665670aspalaleuasnaspleugluglnleulysvalglyvalasnalaval675680685lyslysalaglyglyvalvalglualathrvalcystyrserglyasp690695700metleuglnproglylyslystyrasnleuasptyrtyrleugluval705710715720valglulysilevalglnmetglythrhisileleuglyilelysasp725730735metalaglythrmetlysproalaalaalalysleuleuileglyser740745750leuargthrargtyrproaspleuproilehisvalhisserhisasp755760765seralaglythralavalalasermetthralacysalaleualagly770775780alaaspvalvalaspvalalaileasnsermetserglyleuthrser785790795800glnproserileasnalaleuleualaserleugluglyasnileasp805810815thrglyileasnvalgluhisvalarggluleuaspalatyrtrpala820825830glumetargleuleutyrsercyspheglualaaspleulysglypro835840845aspprogluvaltyrglnhisgluileproglyglyglnleuthrasn850855860leuleupheglnalaglnglnleuglyleuglygluglntrpalaglu865870875880thrlysargalatyrargglualaasntyrleuleuglyaspileval885890895lysvalthrprothrserlysvalvalglyaspleualaglnphemet900905910valserasnlysleuthrseraspaspileargargleualaasnser915920925leuasppheproaspservalmetaspphephegluglyleuilegly930935940glnprotyrglyglypheprogluproleuargseraspvalleuarg945950955960asnlysargarglysleuthrcysargproglyleugluleuglupro965970975pheaspleuglulysilearggluaspleuglnasnargpheglyasp980985990ileaspglucysaspvalalasertyrasnmettyrproargvaltyr99510001005gluasppheglnlysilearggluthrtyrglyaspleuservalleu101010151020prothrlysasnpheleualaproalagluproaspglugluileglu1025103010351040valthrilegluglnglylysthrleuileilelysleuglnalaval104510501055glyaspleuasnlyslysthrglyglnarggluvaltyrphegluleu106010651070asnglygluleuarglysileargvalalaasplysserglnasnile107510801085glnservalalalysprolysalaaspvalhisaspthrhisglnile109010951100glyalaprometalaglyvalileilegluvallysvalhislysgly1105111011151120serleuvallyslysglygluserilealavalleuseralametlys112511301135metglumetvalvalserserproalaaspglyglnvallysaspval114011451150pheileargaspglygluservalaspalaseraspleuleuvalval115511601165leugluglugluthrleuproproserglnlyslys117011751180<210>47<211>538<212>prt<213>artificialsequence<220><223>artificial<400>47metthraspleuasnlysleuvallysgluleuasnaspleuglyleu151015thraspvallysgluilevaltyrasnprosertyrgluglnleuphe202530gluglugluthrlysproglyleugluglypheasplysglythrleu354045thrthrleuglyalavalalavalaspthrglyilephethrglyarg505560serprolysasplystyrilevalcysaspgluthrthrlysaspthr65707580valtrptrpasnserglualaalalysasnaspasnlysprometthr859095glngluthrtrplysserleuarggluleuvalalalysglnleuser100105110glylysargleuphevalvalaspalaphecysglyalaserglulys115120125hisargileglyvalargmetvalthrgluvalalatrpglnalahis130135140phevallysasnmetpheileargprothraspglugluleulysasn145150155160phelysalaaspphethrvalleuasnglyalalyscysthrasnpro165170175asntrplysgluglnglyleuasnsergluasnphevalalapheasn180185190ilethrgluglyileglnleuileglyglythrtrptyrglyglyglu195200205metlyslysglymetphesermetmetasntyrpheleuproleulys210215220glyvalalasermethiscysseralaasnvalglylysaspglyasp225230235240valalailephepheglyleuserglythrglylysthrthrleuser245250255thraspprolysargglnleuileglyaspaspgluhisglytrpasp260265270gluserglyvalpheasnphegluglyglycystyralalysthrile275280285asnleuserglngluasngluproaspiletyrglyalaileargarg290295300aspalaleuleugluasnvalvalvalargalaaspglyservalasp305310315320pheaspaspglyserlysthrgluasnthrargvalsertyrproile325330335tyrhisileaspasnilevalargprovalserlysalaglyhisala340345350thrlysvalilepheleuthralaaspalapheglyvalleupropro355360365valserlysleuthrprogluglnthrglutyrtyrpheleusergly370375380phethralalysleualaglythrgluargglyvalthrgluprothr385390395400prothrpheseralacyspheglyalaalapheleuserleuhispro405410415ileglntyralaaspvalleuvalgluargmetlysalaserglyala420425430glualatyrleuvalasnthrglytrpasnglythrglylysargile435440445serilelysaspthrargglyileileaspalaileleuaspglyser450455460ileglulysalaglumetglygluleuproilepheasnleualaile465470475480prolysalaleuproglyvalaspproalaileleuaspproargasp485490495thrtyralaasplysalaglntrpglnvallysalagluaspleuala500505510asnargphevallysasnphevallystyrthralaasnprogluala515520525alalysleuvalglyalaglyprolysala530535<210>48<211>548<212>prt<213>escherichiacoli<400>48metserasnlyspropheiletyrglnalapropheprometglylys151015aspasnthrglutyrtyrleuleuthrserasptyrvalservalala202530asppheaspglygluthrileleulysvalgluproglualaleuthr354045leuleualaglnglnalaphehisaspalaserphemetleuargpro505560alahisglnlysglnvalalaalaileleuhisaspproglualaser65707580gluasnasplystyrvalalaleuglnpheleuargasnsergluile859095alaalalysglyvalleuprothrcysglnaspthrglythralaile100105110ilevalglylyslysglyglnargvaltrpthrglyglyglyaspglu115120125gluthrleuserlysglyvaltyrasnthrtyrilegluaspasnleu130135140argtyrserglnasnalaalaleuaspmettyrlysgluvalasnthr145150155160glythrasnleuproalaglnileaspleutyralavalaspglyasp165170175glutyrlyspheleucysvalalalysglyglyglyseralaasnlys180185190thrtyrleutyrglngluthrlysalaleuleuthrproglylysleu195200205lysasnpheleuvalglulysmetargthrleuglythralaalacys210215220proprotyrhisilealaphevalileglyglythrseralagluthr225230235240asnleulysthrvallysleualaseralahistyrtyraspgluleu245250255prothrgluglyasngluhisglyglnalapheargaspvalglnleu260265270gluglngluleuleugluglualaglnlysleuglyleuglyalagln275280285pheglyglylystyrphealahisaspileargvalileargleupro290295300arghisglyalasercysprovalglymetglyvalsercysserala305310315320aspargasnilelysalalysileasnarggluglyiletrpileglu325330335lysleugluhisasnproglyglntyrileproglngluleuarggln340345350alaglygluglyglualavallysvalaspleuasnargprometlys355360365gluileleualaglnleuserglntyrprovalserthrargleuser370375380leuthrglythrileilevalglyargaspilealahisalalysleu385390395400lysgluleuileaspalaglylysgluleuproglntyrilelysasp405410415hisproiletyrtyralaglyproalalysthrproalaglytyrpro420425430serglyserleuglyprothrthralaglyargmetaspsertyrval435440445aspleuleuglnserhisglyglysermetilemetleualalysgly450455460asnargserglnglnvalthraspalacyshislyshisglyglyphe465470475480tyrleuglyserileglyglyproalaalavalleualaglnglnser485490495ilelyshisleuglucysvalalatyrprogluleuglymetgluala500505510iletrplysilegluvalgluasppheproalapheileleuvalasp515520525asplysglyasnaspphepheglnglnilevalasnlysglncysala530535540asncysthrlys545<210>49<211>472<212>prt<213>artificialsequence<220><223>artificial<400>49metserseralaseralaalaleuglnlyspheargalagluargasp151015thrpheglyaspleuglnvalproalaaspargtyrtrpglyalagln202530thrglnargserleuglnasnpheaspileglyglyprothrgluarg354045metprogluproleuileargalapheglyvalleulyslysalaala505560alathrvalasnmetthrtyrglyleuaspprolysvalglygluala65707580ileglnlysalaalaaspgluvalileaspglyserleuileasphis859095pheproleuvalvaltrpglnthrglyserglythrglnthrlysmet100105110asnvalasngluvalileserasnargalailegluleuleuglygly115120125gluleuglyserlysalaprovalhisproasnasphisvalasnmet130135140serglnserserasnaspthrpheprothralamethisvalalaala145150155160valvalgluilehisglyargleuileproalaleuthrthrleuarg165170175aspalaleuglnalalysseralagluphegluhisileilelysile180185190glyargthrhisleuglnaspalathrproleuthrleuglyglnglu195200205pheserglytyrthrglnglnleuthrtyrglyilealaargvalgln210215220glythrleugluargleutyrasnleualaglnglyglythralaval225230235240glythrglyleuasnthrarglysglypheaspalalysvalalaglu245250255alailealaserilethrglyleuprophelysthralaproasnlys260265270pheglualaleualaalahisaspalaleuvalglualahisglyala275280285leuasnthrvalalacysserleumetlysilealaasnaspilearg290295300tyrleuglyserglyproargcysglyleuglygluleuserleupro305310315320gluasngluproglyserserilemetproglylysvalasnprothr325330335glncysglualametthrmetvalcysalaglnvalmetglyasnasn340345350thralaileservalalaglyserasnglyglnphegluleuasnval355360365phelysprovalmetilelysasnleuileglnserileargleuile370375380seraspalaserileserphethrlysasncysvalvalglyileglu385390395400alaasnglulyslysileserserilemetasngluserleumetleu405410415valthralaleuasnprohisileglytyrasplysalaalalyscys420425430alalyslysalahislysgluglythrthrleulysglualaalaleu435440445serleuglytyrleuthrsergluglupheaspglntrpvalargpro450455460gluaspmetileseralalysasp465470<210>50<211>416<212>prt<213>aspergillusniger<400>50metasnvalgluthrserleuproglyserserglyseraspleuglu151015thrphehishisgluthrlyslyshisalaasnhisaspserglyile202530servalasnhisglualagluileglyvalasnhisthrpheglulys354045proglyprovalglyilearggluargleuarghisphethrtrpala505560trptyrthrleuthrmetsercysglyglyleualaleuleuileval65707580asnglnprohisaspphelysglyleulysaspilealaargvalval859095tyrcysleuasnleualaphephevalilevalthrserleumetala100105110ileargpheileleuhislysasnmettrpgluserleuglyhisasp115120125arggluglyleuphepheprothrphetrpleuserilealathrmet130135140ilethrglyleutyrlyscyspheglyaspaspalaasnglulysphe145150155160thrlyscysleuglnvalleuphetrpiletyrcysglycysthrmet165170175ilethralavalglyglntyrserphevalphealathrhislystyr180185190gluleuhisthrmetmetprosertrpileleuproalapheproval195200205metleuserglythrilealaservalileglyserglyglnproala210215220seraspglyileproileileilealaglyilethrpheglnglyleu225230235240glypheserileserphemetmettyralahistyrileglyargleu245250255metgluvalglyleuproserprogluhisargproglymetpheile260265270cysvalglyproproalaphethralaleualaleuvalglymetala275280285lysalaleuproaspasppheglnilevalglyaspprohisalaval290295300ileaspglyargvalmetleupheleualavalseralaalailephe305310315320leutrpalaleuserphetrpphephecysilealavalvalalaval325330335valargserproprolysglyphehisleuasntrpphealametval340345350pheproasnthrglyphethrleualathrilethrleualaasnmet355360365phegluserproglyvallysglyvalalathralametserleucys370375380valileilemetpheilephevalleuvalseralaileargalaval385390395400ilearglysaspilemettrpproglyglnaspgluaspvalserglu405410415<210>51<211>542<212>prt<213>kluyveromyceslactis<400>51metvalservallysalaseralaalaglulyslysglupheleugln151015serglnileaspgluileglulystrptrpsergluproargtrplys202530aspthrlysargiletyrseralatyrgluilealalysargarggly354045servallysproasnthrpheproserthrvalmetserglnlysleu505560phelysileleuglygluhisalalysasnglythrvalserlysthr65707580pheglyalaleuaspprovalglnvalthrglnmetserlystyrleu859095aspthriletyrvalserglytrpglncysserserthralaserthr100105110serasngluproglyproaspleualaasptyrprometaspthrval115120125proasnlysvalgluhisleuphelysalaglnglnphehisasparg130135140lysglntrpgluargilecysaspglythrilegluglusergluile145150155160ileasptyrleuthrproilevalalaaspglyaspalaglyhisgly165170175glyleuthralavalphelysleuthrlysmetpheilegluarggly180185190alaalaglyilehisilegluaspglnthrserthrasnlyslyscys195200205glyhismetalaglyargcysvalileprovalglngluhisileasn210215220argleuilethrcysargmetalaalaaspvalleuglyseraspleu225230235240ileleuvalalaargthraspserglualaalathrleuleuserser245250255thralaaspserargasphistyrpheileleuglyalaserasnpro260265270alavallysglylysproleuasnaspleuleuasnlysalaileleu275280285aspglyalathrileaspaspleuglnthrileglulysglutrpleu290295300alalysalaaspvallysleuphehisgluvalphealaaspalaala305310315320lysalaalaglylysaspglnservalileaspglnpheasnserlys325330335valasnproleusergluthrseriletyrglumetglnalaleuala340345350lysgluleuleuglythrgluleuphepheasptrpaspleuproarg355360365glyarggluglyleutyrargtyrglnglyglythrglncysserval370375380metargalaargalaphealaprotyralaaspleucystrpmetglu385390395400serasntyrproasptyrgluglnalalysgluphealagluglyval405410415thralalyspheproglylystrpmetalatyrasnleuserproser420425430pheasntrpthrlysalametservalaspgluglngluthrpheile435440445glnargleuglyaspleuglytyriletrpglnpheilethrleuala450455460glyleuhisthrserglyleualailegluglnpheserlysasnphe465470475480alalysleuglymetlysalatyralaglnaspileglnlyslysglu485490495leuaspasnglyileaspmetvallyshisglnlystrpserglyala500505510glutyrileaspglyleuleuargleualaglnglyglyleualaala515520525thralaalametglyglnglyvalthrgluaspglnphelys530535540<210>52<211>551<212>prt<213>artificialsequence<220><223>artificial<400>52metvallysvalserleuaspasnvallysleuleuvalaspvalasp151015lysgluprophephelysproserserthrthrvalglyaspileleu202530thrlysaspalaleuglupheilevalleuleuhisargthrpheasn354045asnlysarglysglnleuleugluasnargglnvalvalglnlyslys505560leuaspserglysertyrhisleuasppheleuprogluthralaasn65707580ileargasnaspprothrtrpglnglyproileleualaproglyleu859095ileasnargserthrgluilethrglyproproleuargasnmetleu100105110ileasnalaleuasnalaprovalasnthrtyrmetthrasppheglu115120125aspseralaserprothrtrpasnasnmetvaltyrglyglnvalasn130135140leutyraspalaileargasnglnileasppheaspthrproarglys145150155160sertyrlysleuasnglyasnvalalaasnleuprothrileileval165170175argproargglytrphismetvalglulyshisleutyrvalaspasp180185190gluproileseralaserilepheasppheglyleutyrphetyrhis195200205asnalalysgluleuilelysleuglylysglyprotyrphetyrleu210215220prolysmetgluhishisleuglualalysleutrpasnaspvalphe225230235240cysvalalaglnasptyrileglyileproargglythrileargala245250255thrvalleuilegluthrleuproalaalapheglnmetglugluile260265270iletyrglnleuargglnhisserserglyleuasncysglyargtrp275280285asptyrilepheserthrilelysargleuargasnaspproasnhis290295300ileleuproasnargasnglnvalthrmetthrserprophemetasp305310315320alatyrvallysargleuileasnthrcyshisargargglyvalhis325330335alametglyglymetalaalaglnileproilelysaspaspproala340345350alaasnglulysalametthrlysvalargasnasplysileargglu355360365leuthrasnglyhisaspglysertrpvalalahisproalaleuala370375380proilecysasngluvalpheileasnmetglythrproasnglnile385390395400tyrpheileprogluasnvalvalthralaalaasnleuleugluthr405410415lysileproasnglygluilethrthrgluglyilevalglnasnleu420425430aspileglyleuglntyrmetglualatrpleuargglyserglycys435440445valproileasnasnleumetgluaspalaalathralagluvalser450455460argcysglnleutyrglntrpvallyshisglyvalthrleulysasp465470475480thrglyglulysvalthrprogluleuthrglulysileleulysglu485490495glnvalgluargleuserlysalaserproleuglyasplysasnlys500505510phealaleualaalalystyrpheleuprogluileargglyglulys515520525pheserglupheleuthrthrleuleutyraspgluilevalserthr530535540lysalathrprothraspleu545550<210>53<211>343<212>prt<213>saccharomycescerevisiae<400>53metvallysvalalaileleuglyalaserglyglyvalglyglnpro151015leuserleuleuleulysleuserprotyrvalsergluleualaleu202530tyraspileargalaalagluglyileglylysaspleuserhisile354045asnthrasnsersercysvalglytyrasplysaspserilegluasn505560thrleuserasnalaglnvalvalleuileproalaglyvalproarg65707580lysproglyleuthrargaspaspleuphelysmetasnalaglyile859095vallysserleuvalthralavalglylysphealaproasnalaarg100105110ileleuvalileserasnprovalasnserleuvalproilealaval115120125gluthrleulyslysmetglylysphelysproglyasnvalmetgly130135140valthrasnleuaspleuvalargalagluthrpheleuvalasptyr145150155160leumetleulysasnprolysileglyglngluglnasplysthrthr165170175methisarglysvalthrvalileglyglyhisserglygluthrile180185190ileproileilethrasplysserleuvalpheglnleuasplysgln195200205tyrgluhispheilehisargvalglnpheglyglyaspgluileval210215220lysalalysglnglyalaglyseralathrleusermetalapheala225230235240glyalalysphealaglugluvalleuargserphehisasnglulys245250255progluthrgluserleuseralaphevaltyrleuproglyleulys260265270asnglylyslysalaglnglnleuvalglyaspasnserileglutyr275280285pheserleuproilevalleuargasnglyservalvalserileasp290295300thrservalleuglulysleuserproargglugluglnleuvalasn305310315320thralavallysgluleuarglysasnileglulysglylysserphe325330335ileleuaspserserlysleu340<210>54<211>25<212>dna<213>artificialsequence<220><221>source<222>1..25<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>54cggcattattgtgtatggctcaata25<210>55<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>55gaacttcgacctgttgcaatacttcgggttcggcacaaacgtgtacggatagggtttcaa60agatccatacttctc75<210>56<211>79<212>dna<213>artificialsequence<220><221>source<222>1..79<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>56atccgtacacgtttgtgccgaacccgaagtattgcaacaggtcgaagttctcgtacgctg60caggtcgacgaattctacc79<210>57<211>74<212>dna<213>artificialsequence<220><221>source<222>1..74<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>57aatcgcaactcggatttgggaggcaaggtcggaacgcgaactttggctttaggccactag60tggatctgatatcg74<210>58<211>90<212>dna<213>artificialsequence<220><221>source<222>1..90<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>58agaaagcctgtatgcgaagccacaatcctttccaacagaccatactaagtattttatttt60actttttttagaatgacctgttcccgacac90<210>59<211>24<212>dna<213>artificialsequence<220><221>source<222>1..24<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>59cacaagcttattcttccaaaaatc24<210>60<211>20<212>dna<213>artificialsequence<220><221>source<222>1..20<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>60aaagccaaagttcgcgttcc20<210>61<211>24<212>dna<213>artificialsequence<220><221>source<222>1..24<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>61acttagtatggtctgttggaaagg24<210>62<211>4429<212>dna<213>artificialsequence<220><221>source<222>1..4429<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>62aaagccaaagttcgcgttccgaccttgcctcccaaatccgagttgcgattgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatggtt660gatggtagatcttctgcttccattgttgccgttgacccagaaagagctgccagagaaaga720gatgctgctgccagagctttgttgcaagactctccattgcacaccaccatgcaatacgct780acctctggtttggaattgactgttccatacgctttgaaggttgttgcttctgctgacact840ttcgacagagccaaggaagttgctgatgaagtcttgagatgtgcctggcaattggctgac900accgttttgaactctttcaacccaaactctgaagtctctttagtcggtagattaccagtc960ggtcaaaagcatcaaatgtctgctccattgaaacgtgtcatggcttgttgtcaaagagtc1020tacaactcctctgctggttgtttcgacccatccactgctccagttgccaaggctttgaga1080gaaattgctttgggtaaggaaagaaacaatgcttgtttggaagctttgactcaagcttgt1140accttgccaaactctttcgtcattgatttcgaagctggtactatctccagaaagcacgaa1200cacgcttctttggatttgggtggtgtttccaagggttacatcgtcgattacgtcattgac1260aacatcaatgctgctggtttccaaaacgttttctttgactggggtggtgactgtcgtgcc1320tccggtatgaacgccagaaacactccatgggttgtcggtatcactagacctccttccttg1380gacatgttgccaaaccctccaaaggaagcttcttacatctccgtcatctctttggacaat1440gaagctttggctacctctggtgattacgaaaacttgatctacactgctgacgataaacca1500ttgacctgtacctacgattggaaaggtaaggaattgatgaagccatctcaatccaatatc1560gctcaagtttccgtcaagtgttactctgccatgtacgctgacgctttggctaccgcttgt1620ttcatcaagcgtgacccagccaaggtcagacaattgttggatggttggagatacgttaga1680gacaccgtcagagattaccgtgtctacgtcagagaaaacgaaagagttgccaagatgttc1740gaaattgccactgaagatgctgaaatgagaaagagaagaatttccaacactttaccagct1800cgtgtcattgttgttggtggtggtttggctggtttgtccgctgccattgaagctgctggt1860tgtggtgctcaagttgttttgatggaaaaggaagccaagttgggtggtaactctgccaag1920gctacctctggtatcaacggttggggtactagagcccaagctaaggcttccattgtcgat1980ggtggtaagtacttcgaaagagatacctacaagtctggtatcggtggtaacaccgatcca2040gctttggttaagactttgtccatgaaatctgctgacgctatcggttggttgacttctcta2100ggtgttccattgactgttttgtcccaattaggtggtcactccagaaagagaactcacaga2160gccccagacaagaaggatggtactccattgccaattggtttcaccatcatgaaaacttta2220gaagatcatgttagaggtaacttgtccggtagaatcaccatcatggaaaactgttccgtt2280acctctttgttgtctgaaaccaaggaaagaccagacggtactaagcaaatcagagttacc2340ggtgtcgaattcactcaagctggttctggtaagaccaccattttggctgatgctgttatc2400ttggccaccggtggtttctccaacgacaagactgctgattctttgttgagagaacatgcc2460ccacacttggttaacttcccaaccaccaacggtccatgggctactggtgatggtgtcaag2520ttggctcaaagattaggtgctcaattggtcgatatggacaaggttcaattgcacccaact2580ggtttgatcaacccaaaggacccagccaacccaaccaaattcttgggtccagaagctcta2640agaggttctggtggtgttttgttgaacaaacaaggtaagagatttgtcaacgaattggat2700ttgagatctgttgtttccaaggccatcatggaacaaggtgctgaatacccaggttctggt2760ggttccatgtttgcttactgtgtcttgaacgctgctgctcaaaaattgtttggtgtttcc2820tctcacgaattctactggaagaagatgggtttgttcgtcaaggctgacaccatgagagac2880ttggctgctttgattggttgtccagttgaatccgttcaacaaactttagaagaatacgaa2940agattatccatctctcaaagatcttgtccaattaccagaaaatctgtttacccatgtgtt3000ttgggtactaaaggtccatactatgtcgcctttgtcactccatctatccactacaccatg3060ggtggttgtttgatttctccatctgctgaaatccaaatgaagaacacttcttccagagcc3120ccattgtcccactccaacccaatcttgggtttattcggtgctggtgaagtcaccggtggt3180gtccacggtggtaacagattaggtggtaactctttgttggaatgtgttgttttcggtaga3240attgccggtgacagagcttctaccattttgcaaagaaagtcctctgctttgtctttcaag3300gtctggaccactgttgttttgagagaagtcagagaaggtggtgtctacggtgctggttcc3360cgtgtcttgagattcaacttaccaggtgctctacaaagatctggtctatccttgggtcaa3420ttcattgccatcagaggtgactgggacggtcaacaattgattggttactactctccaatc3480actttgccagacgatttgggtatgattgacattttggccagatctgacaagggtacttta3540cgtgaatggatctctgctttggaaccaggtgacgctgtcgaaatgaaggcttgtggtggt3600ttggtcatcgaaagaagattatctgacaagcacttcgttttcatgggtcacattatcaac3660aagctatgtttgattgctggtggtactggtgttgctccaatgttgcaaatcatcaaggcc3720gctttcatgaagccattcatcgacactttggaatccgtccacttgatctacgctgctgaa3780gatgtcactgaattgacttacagagaagttttggaagaacgtcgtcgtgaatccagaggt3840aaattcaagaaaactttcgttttgaacagacctcctccattatggactgacggtgtcggt3900ttcatcgaccgtggtatcttgaccaaccacgttcaaccaccatctgacaacttattggtt3960gccatctgtggtccaccagttatgcaaagaattgtcaaggccactttaaagactttaggt4020tacaacatgaacttggtcagaaccgttgacgaaactgaaccatctggaagttaaaggaag4080tatctcggaaatattaatttaggccatgtccttatgcacgtttcttttgatacttacggg4140tacatgtacacaagtatatctatatatataaattaatgaaaatcccctatttatatatat4200gactttaacgagacagaacagttttttattttttatcctatttgatgaatgatacagttt4260cttattcacgtgttatacccacaccaaatccaatagcaataccggccatcacaatcactg4320tttcggcagcccctaagatcagacaaaacatccggaaccaccttaaatcaacgtccctca4380gaaagcctgtatgcgaagccacaatcctttccaacagaccatactaagt4429<210>63<211>75<212>dna<213>artificialsequence<220><221>source<222>1..75<223>/organism="artificialsequence"/note="primer"/mol_type="unassigneddna"<400>63gaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcacacaggtgatt60gtatgtgggcttatg75<210>64<211>2032<212>dna<213>artificialsequence<220><221>source<222>1..2032<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>64aaataaccacaaacatccttcccatatgctcggtcgtgcttgttgtacctgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatggtt660aaggttgccatcttaggtgcttctggtggtgtcggtcaaccattatctctattattgaaa720ttgtctccatacgtttctgaattggctttgtacggtatctctgctgctgaaggtattggt780aaggatttgtcccacatcaacaccaactcctcttgtgttggttacgacaaggattccatc840gaaaacactttgtccaatgctcaagttgtcttgattccagctggtgttccaagaaagcca900ggtttgaccagagatgatttgttcaagatgaacgctggtatcgttaagtctttggttact960gctgtcggtaaatttgccccaaacgctcgtatcttagtcatctccaaccctgttaactct1020ttggttccaattgccgttgaaactttgaagaagatgggtaagttcaagccaggtaacgtt1080atgggtgtcaccaacttggatttggtcagagctgaaactttcttggttgactacttgatg1140ttgaagaacccaaagatcggtcaagaacaagacaagaccaccatgcacagaaaggtcacc1200gtcatcggtggtcactctggtgaaaccatcattccaatcatcactgacaaatccttggtt1260ttccaattggacaagcaatacgaacatttcatccacagagtccaattcggtggtgacgaa1320attgtcaaggccaagcaaggtgccggttctgctaccttgtccatggctttcgctggtgcc1380aaatttgctgaagaagtcttacgttctttccacaacgaaaagccagaaactgaatctttg1440tctgctttcgtctacttgccaggtttgaagaacggtaagaaggctcaacaattagtcggt1500gacaactccattgaatacttctctttgccaattgttttgagaaacggttccgttgtttcc1560attgacacttctgttttggaaaaattgtctccaagagaagaacaattggtcaacactgct1620gtcaaggaattgagaaagaacattgaaaagggtaagtctttcatcttggacagttaaagt1680ctgaagaatgaatgatttgatgatttctttttccctccatttttcttactgaatatatca1740atgatatagacttgtatagtttattatttcaaattaagtagctatatatagtcaagataa1800cgtttgtttgacacgattacattattcgtcgacatcttttttcagcctgtcgtggtagca1860atttgaggagtattattaattgaataggttcattttgcgctcgcataaacagttttcgtc1920agggacagtatgttggaatgagtggtaattaatggtgacatgacatgttatagcaatacc1980tcgaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcac2032<210>65<211>2032<212>dna<213>artificialsequence<220><221>source<222>1..2032<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>65aaataaccacaaacatccttcccatatgctcggtcgtgcttgttgtacctgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatggtt660aaggttgccatcttaggtgcttctggtggtgtcggtcaaccattatctctattattgaaa720ttgtctccatacgtttctgaattggctttgtacggttccagagctgctgaaggtattggt780aaggatttgtcccacatcaacaccaactcctcttgtgttggttacgacaaggattccatc840gaaaacactttgtccaatgctcaagttgtcttgattccagctggtgttccaagaaagcca900ggtttgaccagagatgatttgttcaagatgaacgctggtatcgttaagtctttggttact960gctgtcggtaaatttgccccaaacgctcgtatcttagtcatctccaaccctgttaactct1020ttggttccaattgccgttgaaactttgaagaagatgggtaagttcaagccaggtaacgtt1080atgggtgtcaccaacttggatttggtcagagctgaaactttcttggttgactacttgatg1140ttgaagaacccaaagatcggtcaagaacaagacaagaccaccatgcacagaaaggtcacc1200gtcatcggtggtcactctggtgaaaccatcattccaatcatcactgacaaatccttggtt1260ttccaattggacaagcaatacgaacatttcatccacagagtccaattcggtggtgacgaa1320attgtcaaggccaagcaaggtgccggttctgctaccttgtccatggctttcgctggtgcc1380aaatttgctgaagaagtcttacgttctttccacaacgaaaagccagaaactgaatctttg1440tctgctttcgtctacttgccaggtttgaagaacggtaagaaggctcaacaattagtcggt1500gacaactccattgaatacttctctttgccaattgttttgagaaacggttccgttgtttcc1560attgacacttctgttttggaaaaattgtctccaagagaagaacaattggtcaacactgct1620gtcaaggaattgagaaagaacattgaaaagggtaagtctttcatcttggacagttaaagt1680ctgaagaatgaatgatttgatgatttctttttccctccatttttcttactgaatatatca1740atgatatagacttgtatagtttattatttcaaattaagtagctatatatagtcaagataa1800cgtttgtttgacacgattacattattcgtcgacatcttttttcagcctgtcgtggtagca1860atttgaggagtattattaattgaataggttcattttgcgctcgcataaacagttttcgtc1920agggacagtatgttggaatgagtggtaattaatggtgacatgacatgttatagcaatacc1980tcgaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcac2032<210>66<211>2032<212>dna<213>artificialsequence<220><221>source<222>1..2032<223>/organism="artificialsequence"/note="artificial"/mol_type="unassigneddna"<400>66aaataaccacaaacatccttcccatatgctcggtcgtgcttgttgtacctgtgcttagtc60aaaaaattagccttttaattctgctgtaacccgtacatgcccaaaatagggggcgggtta120cacagaatatataacatcgtaggtgtctgggtgaacagtttattcctggcatccactaaa180tataatggagcccgctttttaagctggcatccagaaaaaaaaagaatcccagcaccaaaa240tattgttttcttcaccaaccatcagttcataggtccattctcttagcgcaactacagaga300acaggggcacaaacaggcaaaaaacgggcacaacctcaatggagtgatgcaacctgcctg360gagtaaatgatgacacaaggcaattgacccacgcatgtatctatctcattttcttacacc420ttctattaccttctgctctctctgatttggaaaaagctgaaaaaaaaggttgaaaccagt480tccctgaaattattcccctacttgactaataagtatataaagacggtaggtattgattgt540aattctgtaaatctatttcttaaacttcttaaattctacttttatagttagtcttttttt600tagttttaaaacaccaagaacttagtttcgaataaacacacataaacaaacaaaatggtt660aaggttgccatcttaggtgcttctggtggtgtcggtcaaccattatctctattattgaaa720ttgtctccatacgtttctgaattggctttgtactcttcttccaacgtcaagggtattggt780aaggatttgtcccacatcaacaccaactcctcttgtgttggttacgacaaggattccatc840gaaaacactttgtccaatgctcaagttgtcttgattccagctggtgttccaagaaagcca900ggtttgaccagagatgatttgttcaagatgaacgctggtatcgttaagtctttggttact960gctgtcggtaaatttgccccaaacgctcgtatcttagtcatctccaaccctgttaactct1020ttggttccaattgccgttgaaactttgaagaagatgggtaagttcaagccaggtaacgtt1080atgggtgtcaccaacttggatttggtcagagctgaaactttcttggttgactacttgatg1140ttgaagaacccaaagatcggtcaagaacaagacaagaccaccatgcacagaaaggtcacc1200gtcatcggtggtcactctggtgaaaccatcattccaatcatcactgacaaatccttggtt1260ttccaattggacaagcaatacgaacatttcatccacagagtccaattcggtggtgacgaa1320attgtcaaggccaagcaaggtgccggttctgctaccttgtccatggctttcgctggtgcc1380aaatttgctgaagaagtcttacgttctttccacaacgaaaagccagaaactgaatctttg1440tctgctttcgtctacttgccaggtttgaagaacggtaagaaggctcaacaattagtcggt1500gacaactccattgaatacttctctttgccaattgttttgagaaacggttccgttgtttcc1560attgacacttctgttttggaaaaattgtctccaagagaagaacaattggtcaacactgct1620gtcaaggaattgagaaagaacattgaaaagggtaagtctttcatcttggacagttaaagt1680ctgaagaatgaatgatttgatgatttctttttccctccatttttcttactgaatatatca1740atgatatagacttgtatagtttattatttcaaattaagtagctatatatagtcaagataa1800cgtttgtttgacacgattacattattcgtcgacatcttttttcagcctgtcgtggtagca1860atttgaggagtattattaattgaataggttcattttgcgctcgcataaacagttttcgtc1920agggacagtatgttggaatgagtggtaattaatggtgacatgacatgttatagcaatacc1980tcgaaaccttcgaatccagccagcatgtcgacacccacaagatgtagtgcac2032<210>67<211>499<212>prt<213>arabidopsisthaliana<400>67metalaalaleuthrmetglnphegluglyglulyslysasnvalser151015gluvalalaaspvalthrleulysglngluaspgluglnglngluarg202530argsertyrserthrpropheargglugluargaspthrpheglypro354045ileglnvalproserasplysleutrpglyalaglnthrglnargser505560leuglnasnphegluileglyglyasparggluargmetproglupro65707580ilevalargalapheglyvalleulyslyscysalaalalysvalasn859095metglutyrglyleuaspprometileglyglualailemetgluala100105110alaglngluvalalagluglylysleuasnasphispheproleuval115120125valtrpglnthrglyserglythrglnserasnmetasnalaasnglu130135140valilealaasnargalaalagluileleuglyhislysargglyglu145150155160lysilevalhisproasnasphisvalasnargserglnserserasn165170175aspthrpheprothrvalmethisilealaalaalathrgluilethr180185190serargleuileproserleulysasnleuhisserserleugluser195200205lysserphegluphelysaspilevallysileglyargthrhisthr210215220glnaspalathrproleuthrleuglyglnglupheglyglytyrala225230235240thrglnvalglutyrglyleuasnargvalalacysthrleuproarg245250255iletyrglnleualaglnglyglythralavalglythrglyleuasn260265270thrlyslysglypheaspvallysilealaalaalavalalagluglu275280285thrasnleuprophevalthralagluasnlyspheglualaleuala290295300alahisaspalacysvalgluthrserglyserleuasnthrileala305310315320thrserleumetlysilealaasnaspileargpheleuglysergly325330335proargcysglyleuglygluleuserleuprogluasngluprogly340345350serserilemetproglylysvalasnprothrglncysglualaleu355360365thrmetvalcysalaglnvalmetglyasnhisvalalavalthrile370375380glyglyserasnglyhisphegluleuasnvalphelysprovalile385390395400alaseralaleuleuhisserileargleuilealaaspalaserala405410415serpheglulysasncysvalargglyileglualaasnarggluarg420425430ileserlysleuleuhisgluserleumetleuvalthrserleuasn435440445prolysileglytyraspasnalaalaalavalalalysargalahis450455460lysgluglycysthrleulyshisalaalametlysleuglyvalleu465470475480thrsergluglupheaspthrleuvalvalproglulysmetilegly485490495proserasp当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1