散发性大肠癌发病可能性的判定方法与流程
本发明涉及判定无大肠疾病自觉症状的人受试者的散发性大肠癌发病可能性的方法。
本申请主张基于2016年9月29日申请的pct/jp2016/078810和2017年3月31日在日本申请的特愿2017-072674的优先权,并将其内容引用于此。
背景技术:
大肠癌是通过在初期进行适当的治疗而治愈率高的癌。然而,初期往往没有自觉症状,因此,优选通过健康诊断等定期地进行检查,可以早期发现。作为大肠癌的检诊,广泛进行便潜血检查。便潜血检查以粪便为样品,因此不具侵袭性,作为筛选检查而言优异。然而,存在着无法与如细菌性或病毒性肠炎、憩室出血、肛门疾病(痔疮、肛瘘、肛裂)这样粪便中混有血液的其他疾病相区别的问题。
作为与通过便潜血检查为阳性的其他疾病相区别、更高精度地研究大肠癌的检查,有内窥镜检查。然而,通过视觉检测早期的大肠癌在很大程度上取决于操作者的技能,通常较为困难。另外,内窥镜检查还存在着侵袭性高、受试者的负担也大的问题。
作为与内窥镜检查相比非侵袭性地早期发现以溃疡性大肠炎为背景的于大肠粘膜发生的大肠癌的方法,有以dna的甲基化作为生物标志物的方法。例如,专利文献1中报道了:在溃疡性大肠炎患者中,肿瘤性组织中的mir-1、mir-9、mir-124、mir-137和mir-34b/c这5种mirna基因的甲基化率明显高于非肿瘤性溃疡性大肠炎组织;而且由作为非癌部的直肠粘膜采集的生物样品中的上述5种mirna基因的甲基化率能够作为溃疡性大肠炎患者的大肠癌发病的标志物。
现有技术文献
专利文献
专利文献1:国际公开第2014/151551号。
技术实现要素:
发明所要解决的课题
本发明的目的在于:提供利用与内窥镜检查相比侵袭性低、且受试者的负担更小的方法判定无大肠疾病自觉症状的人受试者的散发性大肠癌发病可能性的方法。
用于解决课题的手段
本发明人为了解决上述课题进行了深入研究,结果发现在综合研究无大肠疾病自觉症状的人受试者的基因组dna中的cpg位点(胞嘧啶-磷酸二酯鍵-鸟嘌呤)的甲基化率时,在患有大肠癌的患者和未患散发性大肠癌的人受试者中发现了甲基化率存在显著差异的93种cpg位点,另外还发现了121个甲基化可变区(差异甲基化区(differentiallymethylatedregion)、有时称作“dmr”),从而完成了本发明。
即,本发明提供:下述[1]~[29]的散发性大肠癌发病可能性的判定方法、dna甲基化率分析用标志物和大肠粘膜采集用配套工具。
[1]散发性大肠癌发病可能性的判定方法,其是判定散发性大肠癌发病可能性的方法,具有以下步骤:
测定步骤,测定由采自人受试者的生物样品回收的dna中的由表1~7中记载的甲基化可变区编号1~121所表示的各甲基化可变区中存在的1个以上的cpg位点的甲基化率;以及
判定步骤,根据基于上述测定步骤中测定的甲基化率算出的甲基化可变区的平均甲基化率和预先设定的基准值或者预先设定的多变量判别式,判定上述人受试者的散发性大肠癌发病的可能性,
上述甲基化可变区的平均甲基化率是指,该甲基化可变区中的cpg位点中的所有在上述测定步骤中测定了甲基化率的cpg位点的甲基化率的平均值,
上述基准值是指相对于各甲基化可变区的平均甲基化率分别设定的、用于识别散发性大肠癌患者和非散发性大肠癌患者的值,
上述多变量判别式包含上述甲基化可变区编号1~121所表示的甲基化可变区中的1处以上的甲基化可变区的平均甲基化率作为变量,
[表1]
[表2]
[表3]
[表4]
[表5]
[表6]
[表7]
[2]上述[1]的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,甲基化可变区编号8~15、35~52和111~121所表示的甲基化可变区中的1处以上的平均甲基化率为预先设定的基准值以下、或者甲基化可变区编号1~7、16~34和53~110所表示的甲基化可变区中的1处以上的平均甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[3]上述[1]的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,测定其平均甲基化率作为变量包含在上述多变量判别式中的甲基化可变区中存在的1个以上的cpg位点的甲基化率,
在上述判定步骤中,根据基于上述测定步骤中测定的甲基化率算出的甲基化可变区的平均甲基化率和上述多变量判别式算出作为该多变量判别式的值的判别值,当该判别值为预先设定的基准判别值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[4]上述[3]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自甲基化可变区编号1~121所表示的甲基化可变区的2处以上的甲基化可变区的平均甲基化率作为变量。
[5]上述[3]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自甲基化可变区编号1~121所表示的甲基化可变区的3处以上的甲基化可变区的平均甲基化率作为变量。
[6]上述[3]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自甲基化可变区编号1~52所表示的甲基化可变区的1处所以上的甲基化可变区的平均甲基化率作为变量。
[7]上述[3]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自甲基化可变区编号1~15所表示的甲基化可变区的1处以上的甲基化可变区的平均甲基化率作为变量。
[8]散发性大肠癌发病可能性的判定方法,其是判定散发性大肠癌发病可能性的方法,具有以下步骤:
测定步骤,测定由采自人受试者的生物样品回收的dna中的、选自seqidno:1~93所表示的核苷酸序列中的cpg位点的1处以上的cpg位点的甲基化率;以及
判定步骤,根据上述测定步骤中测定的甲基化率和预先设定的基准值或者预先设定的多变量判别式,判定上述人受试者的散发性大肠癌发病的可能性,
上述基准值是指,针对各cpg位点的甲基化率分别设定的、用于识别散发性大肠癌患者和非散发性大肠癌患者的值,
上述多变量判别式包含上述seqidno:1~93所表示的核苷酸序列中的cpg位点中的1处以上的cpg位点的甲基化率作为变量。
[9]上述[8]的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中测定2~10个cpg位点的甲基化率。
[10]上述[8]或[9]的散发性大肠癌发病可能性的判定方法,其中,在上述判定步骤中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48、50~54、59、65~68、70~77、79~86、90和91所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40、49、55~58、60~64、69、78、87~89、92和93所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[11]上述[8]~[10]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,测定seqidno:1~54所表示的核苷酸序列中的cpg位点的甲基化率,
在上述判定步骤中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48和50~54所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40和49所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[12]上述[8]~[11]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述判定步骤中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48和50~54所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40和49所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是3以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[13]上述[8]~[10]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,测定seqidno:1~8所表示的核苷酸序列中的cpg位点的甲基化率,
在上述判定步骤中,当seqidno:1、4和6所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7和8所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[14]上述[8]~[10]和[13]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述判定步骤中,当seqidno:1、4和6所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:2、3、5、7和8所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是3以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[15]上述[8]~[10]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,测定seqidno:55~87所表示的核苷酸序列中的cpg位点的甲基化率,
在上述判定步骤中,当seqidno:59、65~68、70~77和79~86所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:55~58、60~64、69、78和87所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[16]上述[8]~[10]和[15]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述判定步骤中,当seqidno:59、65~68、70~77和79~86所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:55~58、60~64、69、78和87所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[17]上述[8]~[10]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述测定步骤中,测定seqidno:88~93所表示的核苷酸序列中的cpg位点的甲基化率,
在上述判定步骤中,当seqidno:90和91所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:88、89、92和93所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[18]上述[8]~[10]和[17]中任一项的散发性大肠癌发病可能性的判定方法,其中,在上述判定步骤中,当seqidno:90和91所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:88、89、92和93所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[19]上述[12]、[14]、[16]或[18]的散发性大肠癌发病可能性的判定方法,其中,当上述和为5以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[20]上述[8]或[9]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自seqidno:55~87所表示的核苷酸序列中的cpg位点的1处以上的cpg位点的甲基化率作为变量,在上述测定步骤中,测定其甲基化率作为变量包含在上述多变量判别式中的cpg位点的甲基化率,
在上述判定步骤中,根据上述测定步骤中测定的甲基化率和上述多变量判别式算出作为该多变量判别式的值的判别值,当该判别值为预先设定的基准判别值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[21]上述[8]或[9]的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式包含选自seqidno:88~93所表示的核苷酸序列中的cpg位点的1处以上的cpg位点的甲基化率作为变量,在上述测定步骤中,测定其甲基化率作为变量包含在上述多变量判别式中的cpg位点的甲基化率,
在上述判定步骤中,根据上述测定步骤中测定的甲基化率和上述多变量判别式算出作为该多变量判别式的值的判别值,当该判别值为预先设定的基准判别值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。
[22]上述[8]~[21]中任一项的散发性大肠癌发病可能性的判定方法,其中,上述多变量判别式为逻辑回归式、线性判别式、由朴素贝叶斯分类器制作的公式、或者由支持向量机制作的公式。
[23]上述[8]~[22]中任一项的散发性大肠癌发病可能性的判定方法,其中,上述生物样品为肠道组织。
[24]上述[8]~[23]中任一项的散发性大肠癌发病可能性的判定方法,其中,上述生物样品为直肠粘膜组织。
[25]上述[24]的散发性大肠癌发病可能性的判定方法,其中,上述直肠粘膜组织是通过大肠粘膜采集用配套工具采集的,所述大肠粘膜采集用配套工具具备:
采集工具和采集辅助工具,
上述采集工具包含一对作为板状体的第1夹持片和第2夹持片,
上述第1夹持片和上述第2夹持片分别由夹持部、把持部、弹簧部和固定部构成,
上述采集辅助工具具有:
于侧壁具有狭缝的圆锥台形状的采集工具导入部;以及
棒状的把持部,
上述把持部的一端连接在上述采集工具导入部的外径较大的边缘部的附近,
上述狭缝从上述采集工具导入部的外径较小的边缘部朝着外径较大的边缘部设置,
上述狭缝的宽度比上述第1夹持片和上述第2夹持片在夹持部侧的端部彼此接合后的状态下的宽度宽,
上述采集工具导入部的较大的外径为30~70mm,旋转轴方向的长度为50~150mm。
[26]上述[25]的散发性大肠癌发病可能性的判定方法,其中,在上述第1夹持片的夹持部中的与第2夹持片相对的面的端部和上述第2夹持片的夹持部中的与第1夹持片相对的面的端部的至少一端设有凹部。
[27]大肠粘膜采集用配套工具,其是具备采集工具和采集辅助工具的大肠粘膜采集用配套工具,
上述采集工具包含一对作为板状体的第1夹持片和第2夹持片,
上述第1夹持片和上述第2夹持片分别由夹持部、把持部、弹簧部和固定部构成,
上述采集辅助工具具有:
于侧壁具有狭缝的圆锥台形状的采集工具导入部;以及
棒状的把持部,
上述把持部的一端连接在上述采集工具导入部的外径较大的边缘部的附近,
上述狭缝从上述采集工具导入部的外径较小的边缘部朝着外径较大的边缘部设置,
上述狭缝的宽度比上述第1夹持片和上述第2夹持片在夹持部侧的端部彼此接合后的状态下的宽度宽,
上述采集工具导入部的较大的外径为30~70mm,旋转轴方向的长度为50~150mm。
[28]上述[27]的大肠粘膜采集用配套工具,其中,在上述第1夹持片的夹持部中的与第2夹持片相对的面的端部和上述第2夹持片的夹持部中的与第1夹持片相对的面的端部的至少一端设有凹部。
[29]dna甲基化率分析用标志物,所述标志物包含dna片段,用于判定人受试者的散发性大肠癌发病可能性,所述dna片段具有包含选自seqidno:1~93所表示的核苷酸序列中的cpg位点的1处以上的cpg位点的部分核苷酸序列。
发明效果
根据本发明所涉及的散发性大肠癌发病可能性的判定方法,对于由人受试者、特别是无大肠疾病自觉症状的人受试者采集的生物样品,研究基因组dna中的特定cpg位点的甲基化率或者特定dmr的平均甲基化率,从而可以判定散发性大肠癌发病的可能性。
另外,利用本发明所涉及的直肠粘膜采集用配套工具,可以比较安全且简便地由患者的肛门采集直肠粘膜。
附图简述
[图1]图1是采集工具2的一个实施方式的说明图。
[图2]图2是采集辅助工具11的一个实施方式的说明图。
[图3]图3是直肠粘膜采集用配套工具的使用方式的说明图。
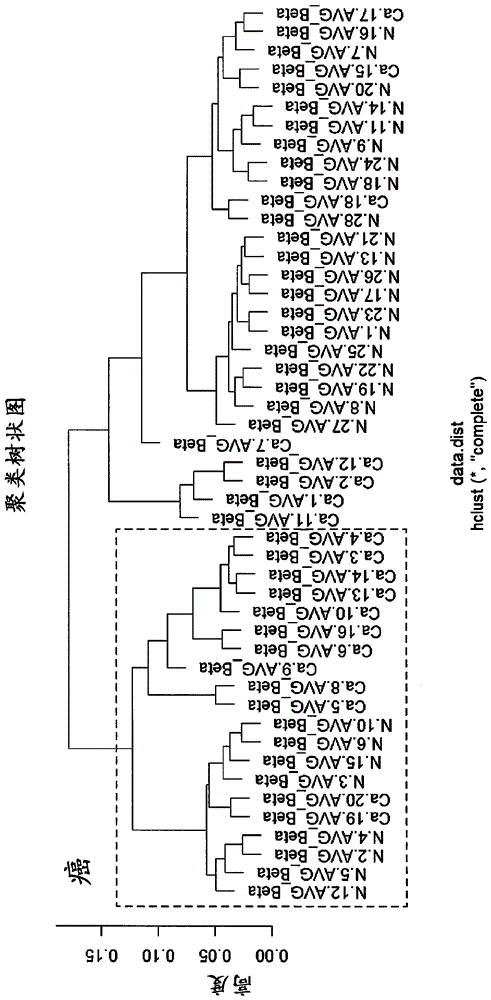
[图4]图4是实施例1中基于综合性dna甲基化分析结果选择的54cpg组的cpg位点的甲基化水平的聚类分析。
[图5]图5是实施例1中基于综合性dna甲基化分析结果选择的8cpg组的cpg位点的甲基化水平的聚类分析。
[图6]图6是实施例1中基于综合性dna甲基化分析结果选择的54cpg组的cpg位点的甲基化水平的主成分分析。
[图7]图7是实施例1中基于综合性dna甲基化分析结果选择的8cpg组的cpg位点的甲基化水平的主成分分析。
[图8]图8是实施例2中基于综合性dna甲基化分析结果选择的33cpg组的cpg位点的甲基化水平的聚类分析。
[图9]图9是实施例2中基于综合性dna甲基化分析结果选择的33cpg组的cpg位点的甲基化水平的主成分分析。
[图10]图10是实施例2中以seqidno:57所表示的核苷酸序列中的cpg位点(cg01105403)、seqidno:63所表示的核苷酸序列中的cpg位点(cg06829686)和seqidno:77所表示的核苷酸序列中的cpg位点(cg14629397)这3个cpg位点的甲基化率作为标志物时的、检查有无散发性大肠癌发病的roc曲线。
[图11]图11是实施例3中基于综合性dna甲基化分析结果选择的6cpg组的cpg位点的甲基化水平的聚类分析。
[图12]图12是实施例3中基于综合性dna甲基化分析结果选择的6cpg组的cpg位点的甲基化水平的主成分分析。
[图13]图13是实施例4中基于综合性dna甲基化分析结果选择的121处dmr(121dmr组)的甲基化率的聚类分析结果。
[图14]图14是实施例4中基于综合性dna甲基化分析结果选择的121dmr组的甲基化率的主成分分析结果。
[图15]图15是实施例4中以dmr编号11所表示的dmr、dmr编号24所表示的dmr和dmr编号42所表示的dmr这3个dmr的平均甲基化率作为标志物时的、检查散发性大肠炎患者有无大肠癌发病的roc曲线。
具体实施方式
基因组dna中的cpg位点的胞嘧啶碱基的5位的碳能够接受甲基化修饰。在本发明和本申请说明书中,cpg位点的甲基化率是指,测定由一个生物个体采集的生物样品中的cpg位点中的甲基化的胞嘧啶碱基(甲基化胞嘧啶)量和未甲基化的胞嘧啶碱基(非甲基化胞嘧啶)量,甲基化胞嘧啶量相对于两者之和的比例(%)。另外,在本发明和本申请说明书中,dmr的平均甲基化率是指,dmr中存在的多个cpg位点的甲基化率的算术平均值或者几何平均值,但也可以是除此以外的平均值。
在本发明和本申请说明书中,“散发性大肠癌”是指,对于其背景未确认到明确的病原疾病、由家族史或基因检查也未确认到明确的遗传性大肠癌的个人,因年龄增加或饮食、生活方式等环境因素导致偶发的基因突变蓄积而发病的大肠癌,有时也称作散发性的大肠癌。即,散发性大肠癌包含除因明确的病原疾病而发病的大肠癌和遗传性大肠癌以外的所有的大肠癌。例如,因溃疡性大肠炎等其他大肠的炎症性疾病进展而发病的大肠癌不包含在散发性大肠癌内(cellularandmolecularlifesciences,2014,第71卷(18),第3523-3535页;cancerletters,2014,第345卷,第235-241页)。另外,家族性大肠腺瘤病(家族性腺瘤性息肉病,familialadenomatouspolyposis:fap)或林奇综合征(lynchsyndrome)等遗传性大肠癌也不包含在散发性大肠癌内(cancer,2015,9:520)。
<散发性大肠癌发病可能性的判定方法>
本发明所涉及的散发性大肠癌发病可能性的判定方法(以下有时称作“本发明所涉及的判定方法”)是判定人受试者的散发性大肠癌发病可能性的方法,其特征在于:以基因组dna中的cpg位点或者dmr中的甲基化率在未患大肠癌、并且也无其他大肠疾病自觉症状的健康人组和已患散发性大肠癌的大肠癌患者组存在差异者作为标志物。以这些作为标志物的cpg位点的甲基化率或者dmr的平均甲基化率作为指标,判定人受试者患有大肠癌的可能性的高低。通过以特定cpg位点的甲基化率或者特定dmr的平均甲基化率作为用于判定人受试者的散发性大肠癌发病可能性的标志物,可以更客观且高灵敏度地检测视觉判别非常困难的早期散发性大肠癌,可以期待早期发现。
在本发明所涉及的判定方法中,用作标志物的cpg位点或者dmr的平均甲基化率能够区别健康人和散发性大肠癌的发病者。因此,本发明所涉及的判定方法适合判定无大肠疾病自觉症状的人的散发性大肠癌发病的可能性。另外,本发明所涉及的判定方法较内窥镜检查更加不具侵袭性,并且与便潜血检查相比能够高精度地判定散发性大肠癌发病的可能性,因此特别是对于大肠检诊这样的大肠癌的筛选检查有效。例如,对于便潜血检查中呈阳性的受试者,可以进行本发明所涉及的判定方法。
基于作为标志物的cpg位点的甲基化率的散发性大肠癌发病可能性的判定可以根据所测定的cpg位点的甲基化率的值本身来进行,也可以使用包含作为标志物的cpg位点的甲基化率作为变量的多变量判别式,根据由该多变量判别式求得的判别值来进行。
基于作为标志物的dmr的平均甲基化率的散发性大肠癌发病可能性的判定可以根据由dmr中的2个以上cpg位点的甲基化率算出的dmr的平均甲基化率的值本身来进行,也可以使用包含作为标志物的dmr的平均甲基化率作为变量的多变量判别式,根据由该多变量判别式求得的判别值来进行。
本发明中作为标志物的cpg位点和dmr,优选甲基化率在非大肠癌发病者组和散发性大肠癌(以下有时仅称作“大肠癌”)患者组大不相同者。两组之差越大,越能够更准确地检测有无散发性大肠癌的发病。本发明中作为标志物的cpg位点和dmr,可以是大肠癌患者的甲基化率明显高于非大肠癌发病者、即甲基化率因大肠癌发病而升高者,也可以是大肠癌患者的甲基化率明显低于非大肠癌发病者、即甲基化率因散发性大肠癌的发病而降低者。
本发明中作为标志物的cpg位点和dmr,更优选在同一大肠癌患者中,大肠的非癌部位与癌部位的甲基化率之差小者。通过以这样的cpg位点的甲基化率或者dmr的平均甲基化率作为指标,即使是使用由大肠癌患者的非癌部位采集的生物样品的情况,也能够和使用由癌部位采集的生物样品的情形一样高灵敏度地判定有无散发性大肠癌的发病。例如,大肠深部的粘膜需要使用内窥镜等进行采集,人受试者的负担大,但肛门附近的直肠粘膜可以比较容易地采集。通过以大肠的非癌部位与癌部位的甲基化率之差小的cpg位点或者dmr作为标志物,尽管是形成癌部位的位置,也能够以肛门附近的直肠粘膜作为生物样品,无遗漏地检测已患散发性大肠癌的人受试者。
本发明所涉及的判定方法中,根据所测定的cpg位点的甲基化率的值本身进行判定的方法是判定人受试者的散发性大肠癌发病可能性的方法,具有以下步骤:测定步骤,测定由采自人受试者的生物样品回收的dna中的、在本发明中作为标志物的特定的多个cpg位点的甲基化率;以及判定步骤,根据上述测定步骤中测定的甲基化率和针对各cpg位点分别预先设定的基准值,判定上述人受试者的散发性大肠癌发病的可能性。
本发明中作为标志物的cpg位点具体是指选自seqidno:1~93所表示的核苷酸序列中的cpg位点的1处以上的cpg位点。各核苷酸序列示于表8~16中。在表的核苷酸序列中,括弧内的cg是指通过实施例1~3所示的综合性dna甲基化分析检测的cpg位点。具有包含这些cpg位点的核苷酸序列的dna片段可以用作用于判定人受试者的散发性大肠癌发病可能性的dna甲基化率分析用标志物。
[表8]
[表9]
[表10]
[表11]
[表12]
seqidno:1~54所表示的核苷酸序列中的括弧内的54个cpg位点(以下有时统称为“54cpg组”)是在后述实施例1的综合性dna甲基化分析中,甲基化率在非大肠癌发病者组和大肠癌患者组中大不相同的cpg位点。其中,大肠癌患者的甲基化率远低于非大肠癌发病者的cpg位点是seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48和50~54所表示的核苷酸序列中的cpg位点(表中“-”),大肠癌患者的甲基化率远高于非大肠癌发病者的cpg位点是seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40和49所表示的核苷酸序列中的cpg位点(表中“+”)。作为标志物的cpg位点并不限于这54个cpg位点,还包括seqidno:1~54所表示的核苷酸序列中的其他cpg位点。
本发明中作为标志物的cpg位点可以只使用seqidno:1~8所表示的核苷酸序列中的cpg位点。这8个cpg位点(以下有时统称为“8cpg组”)是54cpg组中、大肠癌患者的大肠的非癌部位与癌部位的甲基化率之差小的cpg位点。
[表13]
[表14]
[表15]
seqidno:55~87所表示的核苷酸序列中的括弧内的33个cpg位点(以下有时统称为“33cpg组”)是在后述实施例2的综合性dna甲基化分析中,甲基化率在非大肠癌发病者组和大肠癌患者组中大不相同的cpg位点。其中,大肠癌患者的甲基化率远低于非大肠癌发病者的cpg位点是seqidno:59、65~68、70~77、79~86所表示的核苷酸序列中的cpg位点(表中“-”),大肠癌患者的甲基化率远高于非大肠癌发病者的cpg位点是seqidno:55~58、60~64、69、78和87所表示的核苷酸序列中的cpg位点(表中“+”)。作为标志物的cpg位点并不限于这33个cpg位点,还包括seqidno:55~87所表示的核苷酸序列中的其他cpg位点。
[表16]
seqidno:88~93所表示的核苷酸序列中的括弧内的6个cpg位点(以下有时统称为“6cpg组”)是指在后述实施例3的综合性dna甲基化分析中,甲基化率在非大肠癌发病者组和大肠癌患者组中大不相同的cpg位点。其中,大肠癌患者的甲基化率远低于非大肠癌发病者的cpg位点是seqidno:90和91所表示的核苷酸序列中的cpg位点(表中“-”),大肠癌患者的甲基化率远高于非大肠癌发病者的cpg位点是seqidno:88、89、92和93所表示的核苷酸序列中的cpg位点(表中“+”)。作为标志物的cpg位点并不限于这6个cpg位点,还包括seqidno:88~93所表示的核苷酸序列中的其他cpg位点。
关于各cpg位点,预先设定好用于识别大肠癌患者和非大肠癌发病者的基准值。当为54cpg组中的表8~12中记作“+”的cpg位点、33cpg组中的表13~15中记作“+”的cpg位点和6cpg组中的表16中记作“+”的cpg位点时,在所测定的甲基化率为预先设定的基准值以上的情况下,判定为人受试者患有散发性大肠癌的可能性高。当为54cpg组中的表8~12中记作“-”的cpg位点、33cpg组中的表13~15中记作“-”的cpg位点和6cpg组中的表16中记作“-”的cpg位点时,在所测定的甲基化率为预先设定的基准值以下的情况下,判定为人受试者患有散发性大肠癌的可能性高。
关于各cpg位点的基准值,可以测定大肠癌患者组和非大肠癌发病者组的该cpg位点的甲基化率,作为可以区别两组的阈值通过实验而求得。具体而言,任意cpg位点的甲基化的基准值通过普通的统计学方法来求出。下述中显示出其例子,但本发明中的基准值的确定方法并不限于这些。
作为基准值的求出方法之一例,例如,首先,对于任意的cpg位点,测定人受试者中通过内窥镜检查中的活检组织的病理检查未诊断出大肠癌的患者(非大肠癌发病者)的直肠粘膜的dna甲基化。可以针对多个的人受试者进行测定,之后利用其平均值或者中央值等算出代表这些人受试者组的甲基化的数值,以其作为基准值。
此外,针对多个非大肠癌发病者和多个大肠癌患者,分别测定直肠粘膜的dna甲基化,利用平均值或者中央值等分别算出代表大肠癌患者组和非大肠癌发病者组的甲基化的数值和偏差,之后将偏差也考虑在内求出如区别两数值这样的阈值,以其作为基准值。
在上述判定步骤中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48、50~54、59、65~68、70~77、79~86、90和91所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40、49、55~58、60~64、69、78、87~89、92和93所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。在本发明所涉及的判定方法中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48、50~54、59、65~68、70~77、79~86、90和91所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40、49、55~58、60~64、69、78、87~89、92和93所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上、优选3以上、更优选5以上时,判定为上述人受试者患有散发性大肠癌的可能性高,从而可以更高精度地进行判定。
在本发明中以上述54cpg组作为标志物时、即在上述测定步骤中测定上述54cpg组的甲基化率时,在上述判定步骤中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48和50~54所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40和49所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。在本发明所涉及的判定方法中,当seqidno:1、4、6、10、11、13、14、17~20、23~27、29、30、32、33、35、36、39、41~48和50~54所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:2、3、5、7~9、12、15、16、21、22、28、31、34、37、38、40和49所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上、优选3以上、更优选5以上时,判定为上述人受试者患有散发性大肠癌的可能性高,从而可以更高精度地进行判定。
在本发明中以上述8cpg组作为标志物时、即在上述测定步骤中测定上述8cpg组的甲基化率时,在上述判定步骤中,当seqidno:1、4和6所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:2、3、5、7和8所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。在本发明所涉及的判定方法中,当seqidno:1、4和6所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:2、3、5、7和8所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上、优选3以上、更优选5以上时,判定为上述人受试者患有散发性大肠癌的可能性高,从而可以更高精度地进行判定。
在本发明中以上述33cpg组作为标志物时、即在上述测定步骤中测定上述33cpg组的甲基化率时,在上述判定步骤中,当seqidno:59、65~68、70~77和79~86所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:55~58、60~64、69、78和87所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。在本发明所涉及的判定方法中,当seqidno:59、65~68、70~77和79~86所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:55~58、60~64、69、78和87所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上、优选3以上、更优选5以上时,判定为上述人受试者患有散发性大肠癌的可能性高,从而可以更高精度地进行判定。
在本发明中以上述6cpg组作为标志物时、即在上述测定步骤中测定上述6cpg组的甲基化率时,在上述判定步骤中,当seqidno:90和91所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以下、或者seqidno:88、89、92和93所表示的核苷酸序列中的cpg位点中的至少1处以上的甲基化率为预先设定的基准值以上时,判定为上述人受试者患有散发性大肠癌的可能性高。在本发明所涉及的判定方法中,当seqidno:90和91所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以下的cpg位点数与seqidno:88、89、92和93所表示的核苷酸序列中的cpg位点中的甲基化率为预先设定的基准值以上的cpg位点数之和是2以上、优选3以上、更优选5以上时,判定为上述人受试者患有散发性大肠癌的可能性高,从而可以更高精度地进行判定。
在本发明中,使用选自seqidno:1~93所表示的核苷酸序列中的cpg位点的1处以上的cpg位点作为标志物。本发明中作为标志物的cpg位点可以是seqidno:1~93所表示的核苷酸序列中的括弧内的全部的93个cpg位点(以下有时统称为“93cpg组”),可以是上述54cpg组,可以是上述8cpg组,也可以是上述33cpg组,还可以是上述6cpg组。上述54cpg组的cpg位点、上述8cpg组的cpg位点中,大肠癌患者组与非大肠癌发病者组的甲基化率的分散均小,在大肠癌患者组与非大肠癌发病者组的识别能力高的方面优异。另一方面,与上述54cpg组的cpg位点或上述8cpg组的cpg位点相比,虽然上述33cpg组和上述6cpg组的特异度略有降低,但灵敏度非常高,例如非常适合散发性大肠癌的一次筛查。
本发明所涉及的判定方法中,根据特定的dmr的平均甲基化率的值本身进行判定的方法具体而言是判定散发性大肠癌发病可能性的方法,具有以下步骤:测定步骤,测定由采自人受试者的生物样品回收的dna中的、在本发明中作为标志物的特定dmr中存在的1个以上的cpg位点的甲基化率;以及判定步骤,根据基于上述测定步骤中测定的甲基化率算出的dmr的平均甲基化率和针对各dmr的平均甲基化率预先分别设定的基准值,判定上述人受试者的散发性大肠癌发病可能性。需要说明的是,各dmr的平均甲基化率作为该dmr中的cpg位点中的所有在上述测定步骤中测定了甲基化率的cpg位点的甲基化率的平均值来计算。
具体而言,本发明中作为标志物的dmr是选自dmr编号1~121所表示的dmr的1处以上的dmr。各dmr的染色体上的位置和对应的基因示于表17~23中。表中的dmr的起点和终点的碱基位置基于人基因组序列的数据组“grch37/hg19”。这些dmr中存在的具有包含cpg位点的核苷酸序列的dna片段可用作用于判定散发性大肠癌发病可能性的dna甲基化率分析用标志物。
[表17]
[表18]
[表19]
[表20]
[表21]
[表22]
[表23]
dmr编号1~121所表示的dmr(以下有时统称为“121dmr组”)在各区中所含的多个cpg位点的甲基化率在非大肠癌发病者组和大肠癌患者组中大不相同的dmr。其中,大肠癌患者的dmr的平均甲基化率(dmr中存在的多个cpg位点的甲基化率的平均值)远低于非大肠癌发病者的dmr是dmr编号8~15、35~52和111~121所表示的dmr(表中“-”),而大肠癌患者的dmr的平均甲基化率远高于非大肠癌发病者的dmr是dmr编号1~7、16~34和53~110所表示的dmr(表中“+”)。
在本发明中,以dmr的平均甲基化率作为标志物时,可以以dmr编号1~121所表示的dmr中的1处作为标志物,也可以以选自dmr编号1~121所表示的dmr的任意2处以上作为标志物,还可以以dmr编号1~121所表示的dmr的全部作为标志物。在本发明中,从进一步提高判定精度的角度考虑,由dmr编号1~121所表示的dmr构成的组中用作标志物的dmr优选为2处以上,更优选为3处以上,进一步优选为4处以上,更进一步优选为5处以上。
从获得更高的判定精度的角度考虑,本发明中将其甲基化率用作标志物的dmr优选为选自dmr编号1~52所表示的dmr(以下有时统称为“52dmr组”)的1处以上,更优选为选自52dmr组的2处以上,进一步优选为选自52dmr组的3处以上,更进一步优选为选自52dmr组的4处以上,特别优选为选自52dmr组的5处以上。其中,优选为选自dmr编号1~15所表示的dmr(以下有时统称为“15dmr组”)的1处以上,更优选为选自15dmr组的2处以上,进一步优选为选自15dmr组的3处以上,更进一步优选为选自15dmr组的4处以上,特别优选为选自15dmr组的5处以上。
各dmr的平均甲基化率可以作为各dmr中所含的所有cpg位点的甲基化率的平均值,也可以从各dmr中所含的所有cpg位点中任意选出至少1个cpg位点,作为该选出的cpg位点的甲基化率的平均值。各cpg位点的甲基化率可以按照与表8~16中的seqidno:1等所表示的核苷酸序列中的cpg位点的甲基化率的测定相同的方式来测定。
关于各dmr的平均甲基化率,预先设定好用于识别大肠癌患者和非大肠癌发病者的基准值。当为上述121dmr组中的表17~23中记作“+”的dmr时,在所测定的dmr的平均甲基化率为预先设定的基准值以上时,判定为人受试者患有散发性大肠癌的可能性高。当为121dmr组中的表17~23中记作“-”的dmr时,在所测定的dmr的平均甲基化率为预先设定的基准值以下时,判定为人受试者患有散发性大肠癌的可能性高。
关于各dmr的平均甲基化率的基准值,可以测定大肠癌发病者组和非大肠癌患者组的该dmr的平均甲基化率,作为可以区别两组的阈值通过实验而求得。具体而言,dmr的平均甲基化率的基准值通过普通的统计学方法来求出。
以上述93cpg组等的cpg位点的甲基化率作为标志物时,本发明所涉及的判定方法在于,上述判定步骤根据上述测定步骤中测定的甲基化率和预先设定的多变量判别式,可以判定上述人受试者的散发性大肠癌发病的可能性。该多变量判别式包含上述seqidno:1~93所表示的核苷酸序列中的cpg位点中的1处以上的cpg位点的甲基化率作为变量。
以选自上述121dmr组的1处以上的dmr的平均甲基化率作为标志物时,本发明所涉及的判定方法在于,上述判定步骤根据基于上述测定步骤中测定的甲基化率算出的dmr的平均甲基化率和预先设定的多变量判别式,可以判定上述人受试者的散发性大肠癌发病的可能性。该多变量判别式包含上述121dmr组中的cpg位点中的1处以上的cpg位点的甲基化率作为变量。
本发明中使用的多变量判别式可以通过用于判别两组的普通方法而求得。作为该多变量判别式,例如可以列举:逻辑回归式、线性判别式、由朴素贝叶斯分类器制作的公式、或者由支持向量机制作的公式,但并不限于这些。这些多变量判别式可如下制作:例如,针对大肠癌患者组和非大肠癌发病者组,测定上述seqidno:1~93所表示的核苷酸序列中的cpg位点中的1处或者2处以上的cpg位点的甲基化率,以所得的甲基化率作为变量,通过常规方法来制作。另外,还可以针对大肠癌发病者组和非大肠癌患者组,测定上述121dmr组中的dmr中的1处或者2处以上的dmr的平均甲基化率,以所得的甲基化率作为变量,通过常规方法进行制作。
在本发明中使用的多变量判别式中,预先设定好用于识别大肠癌患者和非大肠癌发病者的基准判别值。关于基准判别值,针对大肠癌患者组和非大肠癌发病者组,求出作为使用的多变量判别式的值的判别值,比较大肠癌患者组的判别值和非大肠癌发病者组的判别值,作为可以区别两组的阈值通过实验而求得。
利用多变量判别式进行判定时,具体而言,在上述测定步骤中,测定其甲基化率作为变量包含在所使用的多变量判别式中的cpg位点的甲基化率或者dmr的平均甲基化率,在上述判定步骤中,根据上述测定步骤中测定的甲基化率和上述多变量判别式算出作为该多变量判别式的值的判别值,根据该判别值和预先设定的基准判别值,判定已测定cpg位点的甲基化率或者dmr的平均甲基化率的人受试者患有散发性大肠癌的可能性的高低。当该判别值为预先设定的基准判别值以上时,判定为该人受试者患有散发性大肠癌的可能性高。
作为本发明中使用的多变量判别式,优选包含选自上述33cpg位点的1处以上的cpg位点的甲基化率作为变量的判别式,更优选只包含选自上述33cpg位点的1处以上的cpg位点的甲基化率作为变量的判别式,进一步优选只包含上述33cpg位点的2~10处的cpg位点的甲基化率作为变量的判别式,更进一步优选只包含任意地选自上述33cpg位点的2~5处的cpg位点的甲基化率作为变量的判别式。
作为本发明中使用的多变量判别式,优选包含选自上述6cpg位点的1处以上的cpg位点的甲基化率作为变量的判别式,更优选只包含选自上述6cpg位点的1处以上的cpg位点的甲基化率作为变量的判别式,进一步优选只包含任意地选自上述6cpg位点的2~6处的cpg位点的甲基化率作为变量的判别式,更进一步优选只包含任意地选自上述6cpg位点的2~5处的cpg位点的甲基化率作为变量的判别式。
构成上述33cpg组和上述6cpg组的cpg位点,即使从这些组中任意选择2~10个(6cpg组时为2~6个)、优选2~5个的cpg位点、且仅使用该所选择的cpg位点时,也能够以充分的灵敏度和特异度判定散发性大肠癌的发病可能性。例如,如后述的实施例2所示,在上述33cpg组中,使用seqidno:57所表示的核苷酸序列中的cpg位点、seqidno:63所表示的核苷酸序列中的cpg位点和seqidno:77所表示的核苷酸序列中的cpg位点这3个cpg位点作为标志物、并使用以这3个cpg位点的甲基化率作为变量通过逻辑回归制作的多变量判别式时,能够以约95%的灵敏度、约96%的特异度判定散发性大肠癌的发病可能性。在临床检查等中,若测定甲基化率的cpg位点数较多,则劳动力和成本有可能过大。通过从构成上述33cpg组和上述6cpg组的cpg位点中选择作为标志物的cpg位点,以1个或者2~10个的临床检查中能够测定的恰当数目的cpg位点,可以高精度地判定散发性大肠癌的发病可能性。
作为本发明中使用的多变量判别式,优选包含选自上述121dmr组的1处以上的dmr的平均甲基化率作为变量的判别式,更优选只包含选自上述121dmr组的2处以上的dmr的平均甲基化率作为变量的判别式,进一步优选只包含任意地选自上述121dmr组中的3处以上的dmr的平均甲基化率作为变量的判别式,更进一步优选只包含任意地选自上述121dmr组中的4处以上的dmr的平均甲基化率作为变量的判别式,特别优选只包含任意地选自上述121dmr组中的5处以上的dmr的平均甲基化率作为变量的判别式。其中,优选包含选自上述52dmr组的1处以上的dmr的平均甲基化率作为变量的判别式,更优选只包含选自上述52dmr组的2处以上的dmr的平均甲基化率作为变量的判别式,进一步优选只包含任意地选自上述52dmr组中的2~10处的dmr的平均甲基化率作为变量的判别式,更进一步优选只包含任意地选自上述52dmr组中的3~10处的dmr的平均甲基化率作为变量的判别式,特别优选只包含任意地选自上述52dmr组中的5~10处的dmr的平均甲基化率作为变量的判别式。更优选的是,优选包含选自上述15dmr组的1处以上的dmr的平均甲基化率作为变量的判别式,更优选只包含选自上述15dmr组的2处以上的dmr的平均甲基化率作为变量的判别式,进一步优选只包含任意地选自上述15dmr组中的2~10处的dmr的平均甲基化率作为变量的判别式,更进一步优选只包含任意地选自上述15dmr组中的3~10处的dmr的平均甲基化率作为变量的判别式,特别优选只包含任意地选自上述15dmr组中的5~10处的dmr的平均甲基化率作为变量的判别式。
供于本发明所涉及的判定方法的生物样品只要是由人受试者采集的生物样品、且包含该受试者的基因组dna的生物样品即可,没有特别限定,可以是血液、血浆、血清、泪液、唾液等,也可以是消化管粘膜或者由肝脏等其他组织采集的组织片。作为供于本发明所涉及的判定方法的生物样品,从更有力地反映大肠状态的角度考虑,优选为大肠粘膜,从能够以较低的侵袭性采集的角度考虑,更优选为直肠粘膜。当由上述血液等体液、上述组织片、大肠粘膜或者直肠粘膜采集生物样品时,只要使用符合各生物样品的采集工具进行采集即可。
另外,该生物样品只要是能够提取dna的状态即可,可以是实施了各种前处理的样品。例如,可以是福尔马林固定石蜡包埋(ffpe)组织。从生物样品中提取dna可以通过常规方法进行,也可以使用各种市售的dna提取、纯化试剂盒。
作为测定cpg位点的甲基化率的方法,只要是能够区别特定的cpg位点的甲基化胞嘧啶碱基和未甲基化的胞嘧啶碱基进行定量的方法即可,没有特别限定。通过直接采用该技术领域中公知的方法或者根据需要对其进行适当改变,即可测定cpg位点的甲基化率。作为cpg位点的甲基化率的测定方法,例如可以列举:亚硫酸氢盐测序法、cobra(结合亚硫酸氢盐的限制性分析(combinedbisulfiterestrictionanalysis))法、qamp(实时pcr的dna甲基化定量分析(quantitativeanalysisofdnamethylationusingreal-timepcr))法等。此外,还可以采用miam(imicroarray-basedintegratedanalysisofmethylationbyisoschizomers)法来进行。
<大肠粘膜采集用试剂盒>
本发明所涉及的大肠粘膜采集用试剂盒具备:用于夹持、采集直肠粘膜的采集工具;和用于扩张肛门使该采集工具从肛门到达大肠粘膜表面的采集辅助工具。以下,边参照图1~3,边对本发明所涉及的大肠粘膜采集用试剂盒进行说明。
图1(a)~图1(c)是大肠粘膜采集用配套工具1的采集工具2的一个实施方式的说明图。图1(a)是采集工具2的第1夹持片3a和第2夹持片3b未被施加力的状态的正面图,图1(b)是采集工具2的第1夹持片3a和第2夹持片3b未被施加力的状态的平面图,图1(c)是采集工具2的第1夹持片3a和第2夹持片3b未被施加力的状态的斜视图。如图1所示,采集工具2由一对作为弹性板状体的第1夹持片3a和第2夹持片3b构成。第1夹持片3a由夹持部31a、把持部32a、弹簧部33a和固定部34a构成,第2夹持片3b由夹持部31b、把持部32b、弹簧部33b和固定部34b构成。第1夹持片3a和第2夹持片3b的形状除了板状以外还可以是棒状,只要以具有用于夹持并采集直肠粘膜的一定长度的方式形成即可,不拘泥于形状。另外,原材料也没有特别限定,只要是弹性体即可,可以是不锈钢等金属,也可以是树脂。作为采集工具2,从施加力状态下第1夹持片3a和第2夹持片3b的重合(重なり)稳定、更容易采集大肠粘膜的角度考虑,优选为金属。
第1夹持片3a和第2夹持片3b在固定部34a和固定部34b中以彼此相对的状态连接固定。对连接固定的方法没有特别限定,例如,通过将固定部34a和固定部34b的端部焊接使第1夹持片3a和第2夹持片3b相互重合,即可将两者连接固定。
对固定部34a和固定部34b的长度没有特别限定,优选20~50mm,更优选30~40mm。因固定部的长度在上述范围内,故两夹持片的连接固定容易,并且可以赋予足以应对施加力的强度。
在第1夹持片3a中,在把持部32a与固定部34a之间设有具备弹性的弹簧部33a。在第2夹持片3b中,在把持部32b和固定部34b之间设有具备弹性的弹簧部33b。通过弹簧部33a和弹簧部33b施加力使第1夹持片3a和第2夹持片3b相互接近时,可以使夹持部31a的端部和夹持部31b的端部接合。
对弹簧部33a和弹簧部33b的长度没有特别限定,优选2~10mm,更优选3~7mm。因弹簧部的长度在上述范围内,故可以容易地对两个夹持片赋予充分的弹性。
在第1夹持片3a中夹持部31a和弹簧部33a之间是把持部32a,在第2夹持片3b中夹持部31b和弹簧部33b之间是把持部32b。可以对把持部32a的与把持部32b对抗的面和把持部32b的与把持部32a对抗的面的背面(大肠粘膜的采集者所把持的面)施行防滑加工,使人(大肠粘膜的采集者)把持时不易滑动。作为防滑加工,没有特别限定,例如,可以在金属状的把持部另外贴附树脂状的防滑部,也可以列举对把持部实施锯齿状图案、楔形图案、或者砂纸的摩擦部这样的粗糙图案等。作为防滑加工,如图1(a)所示,优选朝着横向大致平行地设置多个突起部或凹部使形成锯齿状图案的加工。
把持部32a和把持部32b的长度优选20~50mm,更优选30~40mm。因把持部的长度在上述范围内,使得把持或对两个夹持片的施加力变得更加容易。
第1夹持片3a在夹持部31a中与第2夹持片3b相对的面的端部形成有夹持大肠粘膜的夹持面35a。第2夹持片3b在夹持部31b中与第1夹持片3a相对的面的端部形成有夹持大肠粘膜的夹持面35b。夹持面35a和夹持面35b设置成对第1夹持片3a和第2夹持片3b施加力使夹持部31a的端部和夹持部31b的端部接合,在此状态下夹持面35a和夹持面35b至少在边缘部相互密合。
通过对第1夹持片3a和第2夹持片3b施加力,使两者相互接近。因此,通过在使采集工具2的夹持面35a和夹持面35b与大肠粘膜接触的状态下对第1夹持片3a和第2夹持片3b施加力,可以利用夹持面35a和夹持面35b夹持大肠粘膜。更详细而言,夹持面35a的边缘部和夹持面35b的边缘部在夹持大肠粘膜的状态下接合。在该状态下使采集工具2离开大肠粘膜,从而拉下夹持面35a和夹持面35b所夹持的大肠粘膜进行采集。
为了在组织破损较少的状态下采集大肠粘膜,优选在夹持面35a和夹持面35b的至少一方设置凹部。由于两者的至少一方为杯状的情况,所以当夹持面35a的边缘部与夹持面35b的边缘部接合时,于内部形成空间。在夹持面35a和夹持面35b所夹持的大肠粘膜中,收纳于该空间内的部分在拉下大肠粘膜时不太承载负荷,因此能够抑制组织的破坏。对凹部的形状没有特别限定,例如可以形成杯状(半球形)。从大肠粘膜的采集更容易、并且能够抑制组织的破坏的角度考虑,更优选在夹持面35a和夹持面35b的双方形成凹部。
在夹持面35a和夹持面35b形成凹部时,凹部的内径只要设定成能够采集所需量的大肠粘膜的大小即可。当为供于本发明所涉及的判定方法的大肠粘膜时,只要能够采集少量的粘膜即足矣。例如,通过将夹持面35a和夹持面35b的凹部的内径设为1~5mm、优选2~3mm,可以采集充分量的大肠粘膜,而不会过度损伤大肠粘膜。
夹持面35a的边缘部和夹持面35b的边缘部只要能够相互密接即可,可以是平坦的,也可以是锯齿状。当为锯齿状时,通过用夹持面35a的边缘部和夹持面35b的边缘部夹持大肠粘膜,能够以较弱的力切断并采集大肠粘膜。
关于第1夹持片3a和第2夹持片3b的宽度,为了易于把持,从把持部到固定部的部分的宽度优选5~15mm,更优选6~10mm。另一方面,关于第1夹持片3a和第2夹持片3b中的夹持部的宽度,从能够以更小的力采集大肠粘膜的角度考虑,优选朝着设有夹持面的端部变窄。第1夹持片3a和第2夹持片3b的端部的宽度可以设成大于上述凹部,同时例如设为2~6mm、优选3~4mm。
夹持部31a和夹持部31b的长度优选20~60mm,更优选30~50mm。由于夹持部的长度在上述范围内,所以在贯穿采集辅助工具11的狭缝13的状态下采集粘膜会变得更容易。
图2是采集辅助工具11的一个实施方式的说明图。图2(a)是从采集辅助工具11的上侧观察的斜视图,图2(b)是从下侧观察的斜视图。另外,图2(c)~图2(g)分别是采集辅助工具11的正面图、平面图、底面图、左侧面图和右侧面图。如图2所示,采集辅助工具11具有采集工具导入部12、狭缝13和把持部14。
采集工具导入部12是于侧壁具有狭缝13的圆锥台形状的部件。采集工具导入部12从外径小的顶端边缘部15插入至肛门,从外径大的手动边缘部16插入采集工具2。采集工具导入部12在旋转轴方向可以具有贯穿孔。从插入至肛门的容易程度方面考虑,手动边缘部16的外径优选30~70mm,更优选40~60mm。另外,从向大肠粘膜表面导入采集工具2的容易程度方面考虑,顶端边缘部15的外径优选10~30mm,更优选15~25mm。同样,采集工具导入部12的旋转轴方向的长度优选50~150mm,更优选70~130mm,进一步优选80~120mm。
狭缝13从采集工具导入部12的顶端边缘部15朝着手动边缘部16设置。由于在采集工具导入部12的一部分侧壁上具有抵达顶端边缘部15的狭缝13,所以采集工具2的顶端部在肠道内的活动自由度提高,在内部结构复杂的直肠内能够更容易地采集大肠粘膜。狭缝13可以设定在采集工具导入部12的任一位置。例如,如图2(b)所示,狭缝13优选位于靠近把持部14的一侧。另外,采集工具导入部12所设的狭缝13的数目可以是1个,也可以是2个以上。
为了使采集工具2贯穿狭缝13而到达大肠粘膜表面,狭缝13的宽度设计成比夹持面35a和夹持面35b接合状态下的、采集工具2的第1夹持片3a和第2夹持片3b的端部的宽度宽。例如,当夹持面35a和夹持面35b接合状态下的、采集工具2的第1夹持片3a和第2夹持片3b的端部的宽度l1为2~5mm时,狭缝13的顶端边缘部15侧的宽度l2优选7~25mm,优选15~20mm。另外,狭缝13的宽度可以是恒定的,也可以朝着任一方向变窄。需要说明的是,可以在采集工具导入部12的壁面形成2个以上的狭缝。
把持部14的一端沿着远离采集工具导入部12的方向连接在采集工具导入部12的手动边缘部16的附近。把持部14为下方开口的中空棒状,可以通过肋拱来加固。把持部14的长度从手握的容易度等方面考虑,优选50~150mm,更优选70~130mm。另外,从手握的容易度等方面考虑,把持部14的宽度优选5~20mm,更优选8~13mm,把持部14的厚度优选10~30mm,更优选15~25mm。把持部14的形状只要是容易抓握的形状即可,可以是任何形状,例如可以是板状,也可以是棒状,还可以是其他形状。
把持部14可以在采集工具导入部12的手动边缘部16附近相对于采集工具导入部12的圆锥台形状的中心轴垂直地连接,但为了使采集工具2更容易到达大肠粘膜,采集工具导入部12的旋转轴方向与采集工具导入部12的中心轴方向的角度θ1(参照图2(c))超过90˚,优选120˚以下,更优选95~110˚,进一步优选95~105˚。
图3是显示本发明所涉及的大肠粘膜采集用配套工具1的使用方式的说明图。首先,将采集辅助工具11从顶端边缘部15插入至采集大肠粘膜的受试者的肛门。单手拿着把持部14使其稳定,在此状态下从手动边缘部16侧的开口部导入采集工具2。所导入的采集工具2从顶端部贯穿狭缝13到达大肠粘膜表面。在用采集工具2的夹持面35a和夹持面35b夹持大肠粘膜的状态下,从狭缝13拉出采集工具2,从而可以采集大肠粘膜。
实施例
接下来,给出实施例等,以进一步详细地说明本发明,但本发明不限定于这些实施例。
[实施例1]
对于由8名健康人(5名男性、3名女性)和6名大肠癌患者(3名男性、3名女性)采集的大肠粘膜中的dna综合分析cpg位点的甲基化率,所述大肠癌患者根据内窥镜检查中的活检组织的病理诊断而诊断为散发性大肠癌、但未患溃疡性大肠炎等其他大肠的炎症性疾病。
<cpg位点的甲基化水平的综合性分析>
(1)活检和dna提取
由同一受试者的大肠的3处采集粘膜组织,在-80℃下冷冻保存。大肠癌患者的采集部位是盲腸、横结肠、直肠和癌部,而健康人的采集部位是盲腸、横结肠和直肠。将所采集的组织细切,使用qiampdna试剂盒(qiagen公司制造)提取dna。
(2)dna样品的品质评价
所得dna的浓度如下求得。即,使用quant-itpicogreendsdna分析试剂盒(lifetechnologies公司制造)测定各样品的荧光强度,利用试剂盒附带的λ-dna的标准曲线算出浓度。
接下来,将各样品用te(ph8.0)稀释至1ng/μl,使用illuminaffpeqc试剂盒(illumina公司制造)和fastsybrgreenmastermix(lifetechnologies公司制造)进行实时pcr,求出ct值。针对每个样品算出样品与阳性对照的ct值之差(以下记作ct值),评价品质。δct值不足5的样品判断为品质良好,进行以后的步骤。
(3)亚硫酸氢盐处理
使用ezdna甲基化试剂盒(zymoresearch公司制造),对dna样品实施亚硫酸氢盐处理。之后,使用infiniumhdffperestore试剂盒(illumina公司制造)修复分解的dna。
(4)全基因组扩增
将dna进行碱改性后进行中和,添加人甲基化450dna分析试剂盒(illumina公司制造)的全基因组扩增用的酶和引物,使用37℃的培养烘箱(illumina公司制造)在等温下反应20小时以上,从而扩增全基因组。
(5)进行了全基因组扩增的dna的片段化和纯化
在进行了全基因组扩增的dna中添加人甲基化450dna分析试剂盒(illumina公司制造)的片段化用酶,使用微量样品培养箱(scigene)在37℃下反应1小时。在片段化的dna中添加共沉淀剂和2-丙醇,进行离心分离处理,使dna沉淀。
(6)杂交
在已沉淀的dna中加入杂交缓冲液,使用48℃的杂交烘箱(illumina公司制造)使其反应1小时,使dna溶解。已溶解的dna用95℃的微量样品培养箱(scigene公司制造)培养20分钟,使其变性成单链,之后分别注入在人甲基化450dna分析试剂盒(illumina公司制造)的beadchip上。使用48℃的杂交烘箱使其反应16小时以上,使beadchip上的探针与单链dna杂交。
(7)标记反应和扫描
使杂交后的beadchip上的探针进行伸长反应,使其结合荧光色素。然后,使用iscan系统(illumina公司制造)扫描该beadchip,测定甲基化荧光强度和非甲基化荧光强度。实验结束时,确认到了所有的扫描数据均一致、以及扫描正常进行。
(8)dna甲基化水平的定量和比较分析
使用dna甲基化分析软件genomestudio(版本:v2011.1)分析扫描数据。dna甲基化水平(β值)通过下式算出。
[β值]=
[甲基化荧光强度]÷([甲基化荧光强度]+[非甲基化荧光强度]+100)
甲基化水平高时,β值接近1,甲基化水平低时,β值接近0。在大肠癌患者直肠样品组(n=6)相对于健康人直肠样品组(n=8)的dna甲基化水平的比较分析中,使用通过genomestudio算出的diffscore。当两者的dna甲基化水平接近时,diffscore接近于0,大肠癌患者中的水平高时显示为正值,低时显示为负值。两组的甲基化水平的差异越大,diffscore的绝对值就越大。另外,从大肠癌患者直肠样品组(n=6)的平均β值中减去健康人直肠样品组(n=8)的平均β值而得到的值(δβ值)也用于比较分析。
需要说明的是,在dna甲基化定量和dna甲基化水平比较分析中,使用genomestudio和软件methylationmodule(版本:1.9.0)。genomestudio的设定条件如下。
dna甲基化定量;
标准化:有(对照);
减去本底(subtractbackground):有;
内容描述符(contentdescriptor):人甲基化450_15017482_v.1.2.bpm。
dna甲基化水平比较分析;
标准化:有(controls);
减去本底:有;
内容描述符:人甲基化450_15017482_v.1.2.bpm;
参比组:比较分析4.组-3;
误差模式:illuminacustom;
计算错误发现率:无。
(9)多变量分析
利用dna甲基化水平的定量和比较分析的结果,使用统计分析软件r(版本:3.0.1、64bit、windows(注册商标))进行diffscore的计算、聚类分析和主成分分析。
聚类分析的r脚本:
>data.dist<-as.dist(1-cor(data.frame,use="pairwise.complete.obs",method="p"))>hclust(data.dist,method="complete")
#data.frame:由cpg(行)×样品(列)构成的数据框;
将#1-pearson相关系数定义为距离,通过最长距离法(completelinkage)来实施。
主成分分析的r脚本:
>prcomp(t(data.frame),scale=t)
#data.frame:由cpg(行)×样品(列)构成的数据框;
<cpg生物标志物的选择>
(1)cpg生物标志物候选的选取
作为由综合性dna甲基化分析数据选定cpg生物标志物候选的手段,报道了根据diffscore和δβ值的锁定(絞り込み,缩小范围)(bmcmedgenomics第4卷,第50页,2011;sexdev第5卷,第70页,2011)。在前者的报告中,diffscore的绝对值设定为超过30、和δβ值的绝对值设定为超过0.2,在后者的报告中,diffscore的绝对值设定为超过30、和δβ值的绝对值设定为超过0.3,选取生物标志物候选。根据这些方法,由beadchip所搭载的485,577个cpg位点选取生物标志物候选。
具体而言,首先,由485,577个cpg位点选取diffscore的绝对值超过30且δβ值的绝对值超过0.3的54个cpg位点。以下,将这54个cpg位点统称为“54cpg组”。而且,为了无遗漏地判别已患散发性大肠癌的癌症患者,在癌症患者样品内锁定dna甲基化水平的变动少的样品。即,求出癌症患者的23个样品(4部位×各部位的6或者7个样品)的β值的无偏方差(均方差)var,锁定无偏方差var的值小于0.02的8个cpg位点。以下,将这8个cpg位点统称为“8cpg组”。
54cpg组的各cpg位点的结果示于表24和25中。表中,“8cpg”栏内带有#的cpg位点显示8cpg组所含的cpg位点。
[表24]
[表25]
(2)使用了cpg生物标志物候选的临床样品的多变量分析
利用上述54cpg组或者8cpg组进行所有23个样品的聚类分析和主成分分析时,如图4和5所示,在任一的cpg组中,在聚类分析中所有的大肠癌患者样品均集中在同一聚类(图中、框内)中。另外,如图6和7所示,在主成分分析(纵轴为第2主成分)中,大肠癌患者样品(黑圆为由非癌部采集的样品、黑正方形为由癌部采集的样品)和健康人(非癌)样品(黑三角)在第1主成分(横轴)方向分别形成独立的聚类。即,在任一的cpg组中均可明确地区别大肠癌患者样品和健康人样品。由这些结果可知:表24和25所记载的54cpg作为人受试者中的散发性大肠癌发病的生物标志物非常有效,通过使用这些标志物,能够以高灵敏度和特异度判定人受试者有无散发性大肠癌的发病、特别是无大肠疾病自觉症状的人受试者有无散发性大肠癌的发病。
[实施例2]
对于由28名健康人和20名大肠癌患者采集的直肠粘膜中的dna综合分析cpg位点的甲基化率,所述大肠癌患者根据内窥镜检查中的活检组织的病理诊断而诊断为散发性大肠癌、但未患溃疡性大肠炎等其他大肠的炎症性疾病。
关于供于cpg位点的甲基化率分析的dna,进行与实施例1相同的操作,由各受试者的直肠粘膜组织提取dna,扩增全基因组,进行cpg位点的dna甲基化水平的定量和比较分析,利用该结果进行diffscore的计算、聚类分析和主成分分析。需要说明的是,beadchip使用infinium甲基化epicbeadchip(illumina公司制造)。另外,关于genomestudio的设定条件,除了将“内容描述符”设为“methylationepic_v-1-0_b2.bpm”以外,设成与实施例1相同的条件。
(1)cpg生物标志物候选的选取
然后,由综合性dna甲基化分析数据选取cpg生物标志物候选。具体而言,首先,由866,895个cpg位点选取δβ值的绝对值超过0.15的142个cpg位点。
接下来,制作下述的两种逻辑回归模型。[模型1]以选自142个cpg位点的2个cpg的所有组合为依据的10,011个逻辑回归模型。
[模型2]以选自142个cpg位点的3个cpg的所有组合为依据的467,180个逻辑回归模型。
关于两个逻辑回归模型的判别式,按照下述的2个基准分别选定充足的cpg位点。另外,关于[模型2]也算出所出现的cpg位点的频率,选定频率为3以上的cpg位点。
[基准1]灵敏度超过90%、特异度超过90%、判别式的系数p值不足0.05、且aic(红池的信息量基准)不足30。[基准2]灵敏度超过95%、特异度超过85%、判别式的系数p值不足0.05、且aic(红池的信息量基准)不足30。
针对2个基准的每1个,选择判别式中出现的cpg位点时,选择表13~15中记载的33个cpg位点(33cpg组)。各cpg位点的结果示于表26中。
[表26]
(2)使用了cpg生物标志物候选的临床样品的多变量分析
根据上述33cpg组的甲基化水平,进行所有48个样品的聚类分析和主成分分析。其结果,在聚类分析(图8)中,大肠癌患者样品几乎都集中在同一聚类(图中、框内)。另外,在主成分分析(图9、纵轴为第2主成分)中,大肠癌患者样品(●)和健康人样品(▲)在第1主成分(横轴)方向分别形成独立的聚类。即,利用33cpg组可以明确地区别大肠癌患者的20个样品和健康人的28个样品。
(3)使用了cpg生物标志物候选的临床样品的散发性大肠癌发病可能性的评价
在上述33cpg组中,研究了以seqidno:57所表示的核苷酸序列中的cpg位点(cg01105403)、seqidno:63所表示的核苷酸序列中的cpg位点(cg06829686)和seqidno:77所表示的核苷酸序列中的cpg位点(cg14629397)这3个cpg位点的甲基化率作为标志物时的有无散发性大肠癌发病的判定精度。
具体而言,利用由20名诊断为散发性大肠癌的大肠癌患者和28名健康人的直肠采集的样本的上述3个cpg位点的甲基化水平的数值(β值)制作基于逻辑回归模型的判别式,进行大肠癌患者和健康人的判别。其结果,灵敏度(大肠癌患者中评价为阳性的比例)为95.0%、特异度(健康人中评价为阴性的比例)为96.4%、阳性的符合率(评价为阳性的样本中大肠癌患者的比例)为95.0%、阴性的符合率(评价为阴性的样本中健康人的比例)为96.4%,均高达90%以上。另外,图10中显示受试者工作特征(receiveroperatingcharacteristic,roc)曲线。auc(roc曲线下面积)为0.989。由这些结果确认:根据选自上述33cpg组的2~5个cpg位点的甲基化率,能够以高灵敏度且高特异度评价散发性大肠癌发病的可能性。
[实施例3]
由实施例1和实施例2中求出的直肠粘膜样品的dna甲基化水平(β值)选取cpg生物标志物候选。
(1)cpg生物标志物候选的选取
具体而言,首先,由866,895个cpg位点选取诊断为散发性大肠癌的大肠癌患者的26个样品和健康人的36个样品的δβ的绝对值超过0.15的42个cpg位点。
接下来,制作下述两种逻辑回归模型。[模型1]以选自42个cpg位点的2个cpg的所有组合为依据的861个逻辑回归模型。
[模型2]以选自42个cpg位点的3个cpg的所有组合为依据的11,480个逻辑回归模型。
关于两个逻辑回归模型的判别式,按照下述2个基准分别选定充足的cpg位点。[基准1]灵敏度超过90%、特异度超过90%、判别式的系数p值不足0.05、且aic(红池的信息量基准)不足30。
[基准2]灵敏度超过95%、特异度超过85%、判别式的系数p值不足0.05、且aic(红池的信息量基准)不足30。
针对2个基准的每一个,选择判别式中出现的cpg位点。从所选择的cpg位点中去除实施例2中选择的cpg,选择表16中记载的6个cpg位点(6cpg组)。各cpg位点的结果示于表27中。
[表27]
(2)使用了cpg生物标志物候选的临床样品的多变量分析
根据上述6cpg组的甲基化水平,进行所有62个样品的聚类分析和主成分分析。其结果,在聚类分析(图11)中,大肠癌患者样品的多数都集中在多个的聚类(图中、框内)。另外,在主成分分析(图12、纵轴为第2主成分)中,大肠癌患者样品(●)和健康人样品(▲)在第1主成分(横轴)方向分别形成独立的聚类。即,在主成分分析中,使用6cpg组能够明确地区别大肠癌患者的20个样品和健康人的28个样品。
[实施例4]
根据实施例2中获取的由20名大肠癌患者和28名健康人的直肠采集的样本的各dmr的平均甲基化率(平均β值;各dmr中存在的cpg位点的甲基化水平(β值)的算术平均值),选取dmr生物标志物候选。
(1)dmr生物标志物候选的选取
具体而言,首先,将866,895个cpg位点的甲基化数据(idat形式)输入champpipeline(bioinformatics、30、428、2014;http://bioconductor.org/packages/release/bioc/html/champ.html)中,选取大肠癌患者和健康人这两组间判定为显著的4,232处dmr。其中,以δβ值([平均β值(癌直肠)]-[平均β值(非癌直肠)])的绝对值超过0.05的121处(dmr编号1~121)作为dmr生物标志物候选。该121处dmr(121dmr组)的结果示于表28~31中。
[表28]
[表29]
[表30]
[表31]
接下来,利用r软件的glm函数制作以选自上述121dmr组的3处dmr的所有组合为依据的287,980个逻辑回归模型。关于所得的判别式,选择灵敏度大于95%、且4个系数中p值小于0.05的系数为3个以上的47个判别式时,其中出现的dmr为52处(表中52dmr)。再求出47个判别式中出现的dmr的频率时,出现3次以上的dmr为15处(表中15dmr)。
(2)使用了dmr生物标志物候选的临床样品的多变量分析
根据上述121dmr组的甲基化率,进行实施例2的所有48个样品的聚类分析和主成分分析。其结果,在聚类分析中,大多数的大肠癌患者样品都集中于同一聚类(图13中、框内)。另外,在主成分分析(图14)中,大肠癌患者样品(●)和健康人样品(▲)在第1主成分方向分别形成独立的聚类。
(3)使用dmr生物标志物候选进行临床样品的散发性大肠癌发病可能性的评价:研究以上述121dmr组中的dmr编号11、24和42的区的甲基化率作为标志物时的有无散发性大肠癌发病的判定精度。
具体而言,使用由20名大肠癌患者和28名健康人的直肠采集的样本的上述3处dmr的甲基化水平的数值(β值),制作基于逻辑回归模型的判别式,进行大肠癌患者和健康人的判别。其结果,灵敏度(大肠癌患者中评价为阳性的患者比例)为100%、特异度(健康人中评价为阴性者的比例)为92.9%、阳性的符合率(评价为阳性者中大肠癌患者的比例)为90.9%、阴性的符合率(评价为阴性者中健康人的比例)为100%,均高达90%以上。另外,图15中显示roc曲线。其结果,auc(roc曲线下面积)为0.968。由这些结果确认到:根据选自上述121dmr组的多处dmr的平均甲基化率,能够以高灵敏度且高特异度评价散发性大肠癌发病的可能性。
符号说明
1:大肠粘膜采集用配套工具;2:采集工具;3a:第1夹持片;3b:第2夹持片;31、31a、31b:夹持部;32、32a、32b:把持部;33、33a、33b:弹簧部;34、34a、34b:固定部;35、35a、35b:夹持面;11:采集辅助工具;12:采集工具导入部;13:狭缝;14:把持部;15:顶端边缘部;16:手动边缘部。
<110>ea制药株式会社
<110>哈鲁曼有限公司
<120>散发性大肠癌发病可能性的判定方法
<130>j1010091b1
<160>93
<210>1
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg07621697
<400>1
gagtgttccatttgctcccttcccagcggaaaggccctcatctgctcccgctggactggg60
cgctgctctggttcctagcctgtggcttagtaagtgctcaggagaagtcagttgaatgag120
tg122
<210>2
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg16081854
<400>2
cctgggggccagggaggccagtgctgccgattgcggccagggccacgtggacttcaggac60
cggcctgaagttatttttagataagcgacctctggcgccacggacatcttttcctaacct120
tg122
<210>3
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg01710670
<400>3
acctgtgctccgtcccgcacgtggcttgggagcctgggacccttaaggctgggccgcagg60
cgcagccgttcaccccgggctcctcaggcggggggcttctgccgagcgggtggggagcag120
gt122
<210>4
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg22946888
<400>4
acctcccagggctccttgccttaggtggctgtagcatccctaccacccaggacactggtg60
cgaatgacacaactcaagttgggaggggaacagggaaggaagggatggatgggggtggtg120
ta122
<210>5
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00713204
<400>5
cccgctcccctgtcaatgtgggccggcctcccgctcccctgtgctgcgagctccacggcc60
cgctctcagtggctgcctcagtgccacccctgctgtctcgagcctacctcccccttcctt120
ct122
<210>6
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12074150
<400>6
ctgatgttgggatgtgttcggccttctggtggttcgtggtctcgtgagtgaagctcacag60
cggtgtggggaggctcaggcatggggggctgcaggacccaagccctgccctgcggggagg120
ca122
<210>7
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg06758191
<400>7
accccagcgcccgaccctttccccttcatctccagcatgaatccctcaacccgctggctg60
cggagatcacagacacttcagaaggtgatgagagtcaaggactccctcccacccccaccg120
ca122
<210>8
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12515659
<400>8
ataaaacagataaggagaaggctgtatctaggctgaatggctggccaatgttttcctctc60
cgtcagtataaataaaatggatggaagaaaacacccctggatactatcaaatatgccttt120
ca122
<210>9
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg18172516
<400>9
agaattgagttacaatcagtgactcaacattttgacttagcagattggcattccttttta60
cgatgggacaaattctgtaaactgcacatcgtatagatcacacttttcagcaaaatgctc120
aa122
<210>10
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12280242
<400>10
gatcggaccatcctggctaacatggtgaagccccgtctctactaaaaattcaaaaatgag60
cggaccaagatggcacacgcctgtagtcccaggtcccagctactcgggagcctgaggcag120
ga122
<210>11
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg27288829
<400>11
gagccccaggcttgcctcccggctccggggaaatcggttccctccactggggccggcatg60
cgctctgcatccccaggctgtcctcctcgggcttgggggggtctcctgctgtgcctctgt120
ct122
<210>12
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg14293674
<400>12
gcatggacacatcattatcacccaaagtccatagttgacatggaagttcgcccttggtgc60
cgtacattctatgggttttaacaagaatattcaccattacagtattatacaaaagaggct120
gg122
<210>13
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg02507579
<400>13
taagagtaagatgatatctctctctgaatgcaagatacaatttttttccattgcaattgg60
cgtaaccacagaatgttttctcttggcaacaatggcatatgatcgctatgtagccatatg120
ca122
<210>14
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19707653
<400>14
cctgtggggatactgaggtttatgtatggtgccaaccatgatttaggtctcctgtgggga60
cggtttggaggccaaatggggaggcggaggcggagcactaaggaatccagtctctgtacc120
ag122
<210>15
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19285525
<400>15
tagttggcacacaccctcaccatgatctaatagacagctgtataatactaaagtgcctac60
cgcgttgcatcatgataaagtgacatcattgactggtactgatgctaagttttgggtgct120
tc122
<210>16
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg04131969
<400>16
ggcccaattcccactcccccaaacacacacaagtacacactgactaaggcacagctaggg60
cgggggcgggcagaaggccccttgggaggacgtggcgccacagctgcaatgggtgtgggg120
gt122
<210>17
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg07227024
<400>17
tctggatccaagtcaaattttcagtgatggaagaatcacacatcaccttgtggatttgaa60
cggctcctcttcagttgtctcccacagactgccataatttgccccagaatagagtccctg120
ag122
<210>18
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00695177
<400>18
acgtgttctcaggacttcctgagggctgtgtcaccggccatggtcactcatattgggatc60
cgattaaaatatttcttcaaatattttagagtttgacttttttcatcaacatgatgaagc120
ca122
<210>19
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg03311906
<400>19
tgggattacaggcgtgagccaccgtgcccggccgtctactacttcttaaagggtgagagg60
cggaaggatcacttgagccctgaagtgtgcgactgcagttagcttttatcgtaccactgc120
ac122
<210>20
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg20536971
<400>20
gtttacgttcacactcgctaaaaggggtaggaagaattggagagcttttaaaatacttac60
cgcgcccccaagttttaggtgtgtaggattcatcagtaaacagaaaaaggagctgccctc120
at122
<210>21
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg15828613
<400>21
accaaagaaaatagttgcagcttaatgcctcacttgggagtttgcaaagtctctgctctc60
cgaaggccttggtgggtgaaaagcctaaatcgtccttatttcccaccttgcttctctcct120
tc122
<210>22
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg24506221
<400>22
gccctctcccgggcctccagaatggcgcctttcgggttgtggcgggccgaggggcggggt60
cgcagcaaggccccgcctgtcccctctccggagctcttatactctgagccctgctcggtt120
ta122
<210>23
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg27156510
<400>23
cccagcctcagcctcctagagtgctgggattacaggcgtgagtcaccgcacccaatccca60
cgtctgtcttttaatcaaggcatgctctgccttcaagtacaccctccatgatgtctgcca120
ga122
<210>24
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg26077133
<400>24
tacctttagaaccaggggaggatctgctctcaagttcactgagcctttccaaccagtgag60
cggtagagtggatcctccccctaccaagccttcagatgagaccgcagcccagctgacacc120
tt122
<210>25
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg24087071
<400>25
gtatcctgtgtgtgtttgatacctcagattcagcatctactacagcacgaagtgcttatg60
cgtgtcctgaattataggagagtcggattcaccaccctgcccagaaacagaagcattcca120
ga122
<210>26
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg17662493
<400>26
tttctccttttcacatcccttcccctatatccacaaagcagtttaaattttcaggctggg60
cgcagcagctcacacctgtaatcccagcactttgggaggccgaggcaggaagatcacccg120
ag122
<210>27
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12036633
<400>27
aggaggacatcaccttaaagtaccagactctagggccagcctgtgttgggagaacccccc60
cgccccttctcttgcagcttcccccgggggggacagatcttcatggggacacaagggaga120
gt122
<210>28
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg11251367
<400>28
atgaatggctggccgactgaactatgtattcactgggccttattctgctctctctagaac60
cgcacagataaatccaatcctttgttccatgtaataaatctgatatttaaggttcgctat120
ga122
<210>29
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg14181874
<400>29
gagccctgcccgaggagaggtggctgaggcccagcaagaattcgagcggcattggtgggc60
cggtagtgctgggggacccggtgcaccctccacagctgctggcccaggtgctaaacccct120
ca122
<210>30
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg21164300
<400>30
tcagcttggctcactggtgacgacgtatccaaaatgccgtatttaacacattggcttgag60
cggtagagcagctctcagatggcttccaggactggctgagctggtgttgaggcctcattc120
ac122
<210>31
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19405842
<400>31
tggtgtgcagttctctgtctcgtgattcgtgtaacagtgagtgctgcctgcaccaacagc60
cggctgccttccgtggctgtgtgggctcctgtgcggaggccgcccctctccctggccaag120
ca122
<210>32
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg21114725
<400>32
gctgtgcgaggcgctcgcggactggtgcaggttctgggtgggcgccagctaggcaggccc60
cgcactgggcgcagccggccagcgcctgctgggcttcatccagggatgagctccctctgg120
gc122
<210>33
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg08433110
<400>33
tgacttcaccgtgctgtgtgagcatccgctgaagtcgtatggaaacaccaggatgtgggg60
cggctggaagtctcccgtgttgctggtgggaatgcaacagggcagagcggttgtggaaaa120
ca122
<210>34
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg16051083
<400>34
ttacagatgagaaaactcagtgccatatatctttggagtctattgtacaaaaatagaata60
cgttgaacatggaaagtggctttctatttatttatttatttttgagagagtctcgctctg120
tc122
<210>35
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg11454325
<400>35
cagaggttatcgaatgccgaggagcccaggatgcacttccgaggctcactggtgactttc60
cggagatacttaggcaaatggacataaatagctcttggatcctagcaggaattctcaacc120
tc122
<210>36
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12870217
<400>36
gcctgataaagtaggcggtgggctgctgggtcctagattggttagtttgcatatgaaagg60
cggctaaggagtgagttttttgctatgtctagaaattgacttgccctaggagggtcaatc120
tc122
<210>37
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg24208588
<400>37
gaggtctcgcagggggactggttgtcttttaggaaatcaaggggccagcgcccccagtgc60
cggctgggagatgccttcagagttcgaagagaaaagatgcgaccttcaatccgctccatt120
ct122
<210>38
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg08429705
<400>38
ggctgctggcattcccaccttctagagtgactttcacacttcctgatgagtttcccattc60
cgctcagcaggcccataaataggattgtgcagaggtgcatatgcaagcactttacctgaa120
ga122
<210>39
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg24976563
<400>39
ctgatctttacttacacagaccagacaatccgactctatgactgccgatatggccgtttc60
cgtaaattcaagagcatcaaggcccgcgacgtaggctggagcgtcttggatgtggccttc120
ac122
<210>40
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg14323910
<400>40
tattcttctggggaatatgaagggttcagtctttttaggaaattggatgatatctcttcc60
cgaccactagcagcctctttcagtcactggaaaatgcttacaggcagtagccaccatcat120
gt122
<210>41
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg04212500
<400>41
catcatctttctcccagatcccatcaaagcagaatggtagaaacctaaggtcagcctggg60
cgcagtggctcacgtctgtaatcccagcactttgggaggccaaagcaggcggatcacttg120
ag122
<210>42
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00348031
<400>42
gggatccgcctgtccacgtgcagccgcctccgggcggcgtcggccatgctgctgccccac60
cgtggctctgtggctccagccggaatggcaaagcctggctccacagctgcctgggagcgt120
ga122
<210>43
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg02890235
<400>43
ccccaggtctgggtcccggcagggctggaaggagcctgagagggatgtgcgcagcacctc60
cgagagtcccgctttagagaaacacgaatcagatcatgagaaagcagacctctgagaagt120
ca122
<210>44
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00525828
<400>44
cccttctccctttcctggggacacctgagcagcgccacggtgatggcaggcttgtgcacg60
cgtcatgcagatacatccttattttcttcccactcttcgtcgtcccctgcccgcccaccc120
tc122
<210>45
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg02775404
<400>45
tgttctctgggaaatccttttcaagataattgaactctgcctttgaaactcatcctctaa60
cgtagatagcggggcagggctgattacagaggacggaagcccaggagccccagggcctgg120
ca122
<210>46
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg23663942
<400>46
gacctacctgtacagcttggtgtcaccaccttgatttgtgctcaggcactaacagtttca60
cgtgaccaccatagatttctgtaccaatatgtaaataatacagtgaaaaaggcaaataac120
at122
<210>47
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg15115757
<400>47
cagaaatgccatcatcgtatgtgacacagaatttagaaaaatgactttgtgaagaatggc60
cggaagagggaagctaatggtagagaaacctctctggtgatgggatcatcttaagtctat120
ga122
<210>48
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg03022891
<400>48
gccacatgggcacgtgtggccatgtggggggtgcaggacccaagaaggaacaagaggggc60
cgcgtaaccctgcacagcctggcctgctcgctccgccgcctcggccctgcccgccctcct120
ct122
<210>49
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg22664298
<400>49
aaactcctgcagcgtccagaacacagaaaatagactcatctcctaattcgccagggagct60
cgagggctgcggggccgcggggctgcctcccccgctcctcccccaacccgaccccacccc120
ac122
<210>50
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg06306564
<400>50
ggacagaaagctgttaggctgtgggtttaaaataggatatccatgtaaactgaaataatg60
cgcttacatgtttaaacagctaagtgccagttcaaaagcagtttgatattagttattttc120
at122
<210>51
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg01647917
<400>51
tggaggaaagctcggagctcccatgccctcccggggcaccgccttccaggaacctgcctg60
cgttccgcttctgggcacccggaaagtcgctcagtggctgattcagggtcgaggagctgt120
ga122
<210>52
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg16661157
<400>52
ttgcctgtagcccattgatctacccactatgtatattcattttaatgctgtttttgagtc60
cgttgactaccccgggaaatcaaagttgactaccacagccctagtcctcaagtgtcttgc120
ct122
<210>53
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg17025908
<400>53
cattgctccacacaccatctctcattcatcctcacctcaccctgctcggaccagttctaa60
cggcagtggtttatggagcacctagacatcaaatcgagtgccaggcatcagatggaggct120
tc122
<210>54
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19455396
<400>54
aacacttagcatagctcctactcccattaaaactctataaatggtagctgttaccaatgt60
cgctattaatactgttaatcagggaactgttctctgtccctccagaccctagcttcttca120
aa122
<210>55
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00853216
<400>55
tgtactataattgtttatgtatctgtctcatcttcctctccagcctacaaaattctttga60
cgtaaaaggcccttttctatttgatttgtatccttagcccttagcagaatacgttgttca120
ta122
<210>56
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg00866176
<400>56
cctccctccccaacaactcaaaagcagcgaggcctgtccttgacctgtctgagaatgggc60
cgcttcaccaccctgcttggttaactgaagtcacccgcactgcaacaccctggtatcagc120
ct122
<210>57
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg01105403
<400>57
tgtctacaccacgctggaaccattttctgtcccacctcgggactgggtggcacgtgagag60
cggccagggagagaccgcatctgggaaggcacagctggctgcagggaacggccgccctgg120
aa122
<210>58
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg02078724
<400>58
actcaattagaaaagcagcgaagcatggtggttaagaacacggcttcagcagacaggctg60
cgttcaaaactcagttccctcacatactagctgtcgactggcttttccagtttcgaagaa120
aa122
<210>59
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg03057303
<400>59
ttgatttatgcccttattgtggaatgaaagtgcttgttacatatttcaagaaaatgaatg60
cgctcttagaaacagattggaatgtaggatgtatgccagcttgtggcaatgagaatgctt120
aa122
<210>60
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg04234412
<400>60
cagcactgggcgaggggaagttggtgggccaggggtccggccttgtccctgctctgcctc60
cgcaacagcgaccccgatccctttccccagggaccaccccccaccccattccgcaggcca120
ag122
<210>61
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg04262140
<400>61
tggtcgcaaaagcagccctttcaatcgcaccgaatttcccctggtgtgaaaaggcgccat60
cgccagcattttgccggggtttatgcctcaatcccgcattccagccacttccacgaatta120
ct122
<210>62
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg04456492
<400>62
tcaatttggtaatgtgctcattactgctcctaattcattcatattttagcaaacacttag60
cgtggtgaggcttctgatcctcagcactggtaaaaatctaacatttattgtatctgttct120
aa122
<210>63
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg06829686
<400>63
gcaggggtctctacccggtgccttcctcccggcacgctagcctcctcgccgaaatttcgt60
cgtcccggagtcggtaaccgagtcccaggctttactgccactccactccctgctgggtta120
tt122
<210>64
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg07684215
<400>64
aggctctgggcagatgtcagctaaggtcacggcaggaggctgaaggggaggctcctggca60
cgtgactctggatcgatgccccccatgtctcccctgacctctgactgttctagatccaca120
at122
<210>65
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg08421632
<400>65
tgaactcctgacctcaggtgatccgcctgccgcggcctcccaaagtgctgggattataga60
cgtgagccacctcggcaggccacctgatgttttttggcacatagcatagtctatggtgtc120
aa122
<210>66
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10169393
<400>66
ttacacagtaggcttcttattcaagaaatcacaaaactcagggattaacagccaggattt60
cgcaactagtttttggggttcaaatctcagctctactggttactagctgtgaataagccc120
tg122
<210>67
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10204409
<400>67
ttaatatcagcagtagctggaattagagtgctgactctgcaccaagcactgttctaaaca60
cgtcatgtttgttggctcattttcagtctcacagtagcacagtggggtggagattcttgt120
ta122
<210>68
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10326673
<400>68
ctcctgatcagggaacctgggttctataactgcttctactactgatttgtcctgtgactt60
cgcgcaccaaatttaggcttgtaaattaaactcccagatttctgttttccattttgcagc120
tc122
<210>69
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10360725
<400>69
cagctggcctgactgggggcctgtgtcgggtgccatatgagagatttcaaccagcccatg60
cgcaaccagagggatgcggcccacggtgcgggtggtctcagcgtcgtctctgtctgaccc120
tc122
<210>70
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10530344
<400>70
tgcactgccagggcctgtgagctgccacaccaggacactgcctggcttgcttggggctgg60
cgggatcccctgagctgagatctggtctccctttgggaagggtgggagaatggtgagaga120
ag122
<210>71
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10690713
<400>71
atggctgggttttggatatattttaagtagagccatcaggatttgtgaaaggatcagatg60
cggatgtggaagaaagaaaaatatcaagcctgactcctgggccatcgacagtgggaggtg120
cc122
<210>72
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg10772532
<400>72
cacatatgtctgcctcctatcatttcttcatgaggttcagggcaaagggcctagtcaagc60
cgatgatctttggttgcccctacactttccccaaaccacctacaaataaacaaaacaagg120
gg122
<210>73
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg11044162
<400>73
gagagggggagaaaagtgaagcgggatagatttagggtagagatgttcaggagaggcggg60
cgacccatctcagatgaaattcagaaaaactgacaactgactaggggtggcaggatggca120
ca122
<210>74
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg11141652
<400>74
cacttgccaggtggtgcttggcgaaggcaagcagctcccacccgcccggggaatacagcg60
cgacccccggcggcatgctcttcagcaccaccccaggaggtaccaggatcatctaccact120
gg122
<210>75
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12219587
<400>75
gagcctaagtgatctgtttaaattgtaaatctgatcacaccacacctctgcttaaaactc60
cgtaatgcttttgcatggccttcaggataaatctaaactccatagcatcgctttgaagac120
cc122
<210>76
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg12814117
<400>76
caacctacttgactcgcaccactgacccccacaccttgcatagactgagcagatatataa60
cgatggccacctctccatctgattctagactgattctagttcctagaatctcagcatgat120
tc122
<210>77
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg14629397
<400>77
taccagtcagtagtgggtgacaaggccttcccacagcatttatctttaagcttcagcata60
cgtatttgtactcttcatcctatctatttggagtggtctcaaattccacaggctactcca120
cg122
<210>78
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg16013720
<400>78
tcacttcatttcgttcaatttcgttcaatttcattccttttcatccagcgccgggaggcc60
cgaggccacaaggaaggggagggggtctttccgggcgaatttccctcatcttgtagattt120
ac122
<210>79
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg16776298
<400>79
agcccccacctctgggcaccccctgggtggtttgtctccatcgactggcatttaccatga60
cgtctctcatattatggccacttgcacttgcccagaggtgggcctgctcgctcctcccca120
gc122
<210>80
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg17658874
<400>80
aaatatgaattatgcaaatacatttctgcccattgagatgatattactcaacagggccct60
cgtaagtgcccagttctgttggatgtttagacagaaaacaagcaaactgtagataccggc120
aa122
<210>81
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg18285337
<400>81
tgctctttgcttgccaactgcgcaaaaccaggcagtggggcagatttggcctgagggtca60
cggtttgccaacccctgctcaagcctgctcactctcaacgctggctgcacgttgcaataa120
tc122
<210>82
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19236675
<400>82
ttggcgtcacatgccgaaggagtcttctaatgtctctccctctctgcgtgtctgctctca60
cgcccgtgcaggcatgacgagtgttctgatgtcagccattggactccctgtgtgtcttag120
cc122
<210>83
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19631563
<400>83
ctgacaaaggatgctggtgctgaaattcttaattcacttagcctgtcagctttgaaatta60
cgattatagaattctaagaaactttgcatgctttatatcagatttgtacacttctaattt120
at122
<210>84
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg19919789
<400>84
caggaagttttttcctgtggtggaagcttttgttctccaagtcgaatttccctcagctga60
cgtcagccccaacttaggcccaagcccattgaacctgcagtggggctgagggagggctgc120
ct122
<210>85
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg22109827
<400>85
agctgaacaggcaaggctgtatgtttggagaagctgggaccctatccgctgcactcagag60
cggggaccatccgccaagggagacagggaagggtctgtgccacctgctggagggagggca120
ga122
<210>86
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg23231631
<400>86
gcaaggtggatggatgatgatgatagatagatagatagatagatagatagatagatagat60
cgatcgatctatctccacatcagggaggcacatcaagccagatgtttaggaacacagtgt120
tt122
<210>87
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg27351675
<400>87
tatgaggaatttggggctcagttgaaaagcctaaactgcctctcgggaggttgggcgcgg60
cgaactactttcagcggcgcacggagacggcgtctacgtgaggggtgataagtgacgcaa120
ca122
<210>88
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg01561758
<400>88
cctcactcttggatcaccataagagttgagacagctgggtctgcaggacattggaaaagt60
cgggtgtgccttcctctgtagggccacctgggaaggatacagctgtctgcaaaccatgat120
gt122
<210>89
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg06970370
<400>89
cgtcctgcccgcggcactggctgcgggtgccgggccacctgcgagtgtgcggagggattc60
cggacacccgcggcggcgagctgagggagcagtctccacgagaactgaggcggaccctct120
gg122
<210>90
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg07973162
<400>90
ggatacccaagcagctcattcctgcctggcaccacagtgatcctttaggagggtggccag60
cggagcagggggttcaaagattcttctggggcctgaaagcttgaagggatgagtaactcc120
tc122
<210>91
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg11792281
<400>91
aacactggcagcacctattgaggccatgtttcaggatcagaccatgctggtttgagcaga60
cgcagcaagagtgagaaccccggccgaattttcatgggtggctctagtagagctgctggt120
ga122
<210>92
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg18500967
<400>92
agctgaagaaacagatgaggaagcacagatagtctgggaggagacactcaagcttcccac60
cggtggccacagcacactccatccctggaaatactgcaaaccaaccccccaggagccccg120
gg122
<210>93
<211>122
<212>dna
<213>homosapiens
<220>
<222>(61)..(62)
<223>cpgid_cg23943944
<400>93
tatcctcaacaaaactgtaacagggaatctatctgtgttcagtgttgctcccctgaacac60
cgtgctcttcactcagccttcacacccctcacatggtattctatttaaaaaaataataat120
aa122
- 还没有人留言评论。精彩留言会获得点赞!