C4水稻底盘受体材料的设计、创制与应用的制作方法
本发明属于水稻光合作用改进技术领域,特别涉及类C4解剖学结构的材料筛选及基因克隆,利用图位克隆法克隆CRV1基因,涉及C4水稻底盘受体材料的设计、创制与应用,利用CRV1基因创制具有类C4解剖学结构的材料(C4水稻底盘受体材料),创制C4水稻原型。
背景技术:
随着工业化、城镇化的发展以及人口的增加,我国粮食需求将呈刚性增长,而耕地减少、水资源短缺、气候变化等对粮食生产的约束日益突出,我国粮食供需将长期处于“紧平衡”状态,保障粮食安全面临严峻挑战。农作物的株型育种和杂种优势利用为我国粮食作物产量的提高,保障国民经济的快速发展做出了巨大贡献,但粮食产量的进一步提高难度越来越大。在当前保障粮食安全作为我国长期国策的背景下,如何实现粮食作物产量水平的持续提升是横亘在我国社会经济、农业科技前进道路上的一道难题。作物收获指数是衡量作物产量的主要指标之一,目前水稻、小麦的收获指数都接近0.5,这意味着这两种作物的粮食产量占全部生物量的近50%,通过传统育种实现粮食产量的大跨度提高已不可能。
幸运的是,世界上还有另一些植物(C4植物,如玉米、高粱和甘蔗等),它们的光合效率要比C3植物高出50%。与C3植物相比,C4材料有着叶脉密度高、具有特殊“花环状”结构、叶片维管束鞘细胞大而少、叶绿体发育好、具有特殊C4光合作用途径。
技术实现要素:
本发明的一个目的是提供抑制或降低目的植物中蛋白质活性或表达的物质的用途。
本发明提供了抑制或降低目的植物中蛋白质活性或表达的物质在培育细胞学结构类似于C4植物的转基因植株中的应用;
所述蛋白质为如下a)-e)中任一种蛋白质:
a)氨基酸序列包括序列表中序列1所示的氨基酸序列的蛋白质;
b)氨基酸序列由序列表中序列1所示的氨基酸残基组成;
c)将a)或b)所限定的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
d)与a)或b)所限定的氨基酸序列具有99%以上、95%以上、90%以上、85%以上或者80%以上同源性且具有相同功能的蛋白质;
e)a)-d)中任一所限定的蛋白质的N端和/或C端连接标签后得到的融合蛋白。
上述应用为抑制或降低C3植物中蛋白质活性或表达,得到细胞学结构类似于C4 植物的转基因植物;
所述细胞学结构类似于C4植物的转基因植物为叶脉密度高于C3植物、光合效率高于C3植物和/或细胞间距小于C3植物。
本发明还提供抑制或降低目的植物中蛋白质活性或表达的物质在提高目的植物叶脉密度、提高目的植物光合效率和/或减少目的植物细胞间距中的应用;
所述蛋白质为如下a)-e)中任一种蛋白质:
a)氨基酸序列包括序列表中序列1所示的氨基酸序列的蛋白质;
b)氨基酸序列由序列表中序列1所示的氨基酸残基组成;
c)将a)或b)所限定的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
d)与a)或b)所限定的氨基酸序列具有99%以上、95%以上、90%以上、85%以上或者80%以上同源性且具有相同功能的蛋白质;
e)a)-d)中任一所限定的蛋白质的N端和/或C端连接标签后得到的融合蛋白。
上述应用中,
所述抑制或降低目的植物中蛋白质活性或表达的物质为下述1)-4)中的任一种生物材料:
1)序列2第13-33位所示的DNA分子或序列2第87-107位所示的DNA分子;
2)含有所述DNA分子的重组载体;
3)含有所述DNA分子的重组菌;
4)含有所述DNA分子的转基因细胞系。
上述应用中,
所述重组载体为表达序列2第13-33位所示的DNA分子或序列2第87-107位所示的DNA分子和case9蛋白的载体。
本发明第2个目的是提供一种生物材料。
本发明提供的生物材料,为上述应用中的所述抑制或降低目的植物中蛋白质活性或表达的物质。
本发明第3个目的是提供一种培育细胞学结构类似于C4植物的转基因植物的方法。
本发明提供的方法,包括如下步骤:抑制或降低C3植物中蛋白质活性或表达,得到细胞学结构类似于C4植物的转基因植物;
所述蛋白质为如下a)-e)中任一种蛋白质:
a)氨基酸序列包括序列表中序列1所示的氨基酸序列的蛋白质;
b)氨基酸序列由序列表中序列1所示的氨基酸残基组成;
c)将a)或b)所限定的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加且具有相同功能的蛋白质;
d)与a)或b)所限定的氨基酸序列具有99%以上、95%以上、90%以上、85%以上或者80%以上同源性且具有相同功能的蛋白质;
e)a)-d)中任一所限定的蛋白质的N端和/或C端连接标签后得到的融合蛋白。
上述方法中,
所述抑制或降低C3植物中蛋白质活性或表达为将表达序列2第13-33位所示的 DNA分子或序列2第87-107位所示的DNA分子的cas9载体导入C3植物中。
上述方法中,
所述细胞学结构类似于C4植物的转基因植物为叶脉密度高于所述C3植物、光合效率高于所述C3植物和/或细胞间距小于所述C3植物。
本发明第4个目的是提供一种培育叶脉密度提高、光合效率提高和/或细胞间距减少的转基因植物的方法。
本发明提供的方法,包括如下步骤:抑制或降低目的植物中上述蛋白质活性或表达,得到转基因植株;
所述转基因植物的叶脉密度高于所述目的植物;
和/或,所述转基因植物的光合效率高于所述目的植物;
和/或,所述转基因植物的细胞间距小于所述目的植物。
上述方法中,
所述植物为双子叶植物或单子叶植物;
或所述目的植物为C3植物;
或所述植物为水稻。
本发明提供了的基因,加以改造,导入C3植物水稻中,创制具有类C4结构特征的材料,大辐提高植物的光合效率。本发明对于培育C4植物提供了底盘材料,具有重大生产应用价值。
本发明从构建的水稻T-DNA插入突变体库中筛选获得了类C4解剖学结构突变体 crv1,突变体crv1表现叶脉密度变密,小脉间细胞数大大减少,具有C4植物叶脉的解剖学结构特征,光合效率显著提高。遗传分析发现该突变性状是由隐性单基因控制的,用图位克隆方法分离出了控制该突变性状的基因,发现其野生型基因是一个半胱氨酸家族基因(命名为CRV1)。用互补的方法对该基因进行了功能互补研究,恢复突变体的表型。同时利用CRISP9技术创制具有类C4解剖学特征的材料,为最终创制C4 水稻材料提供了底盘材料。
附图说明
图1为突变体crv1和野生型的表型图片和解剖学图片。
图2为CRV1基因克隆流程图。
图3为功能回补实验中的植株表型照片。
图4为利用CRISP9技术创制具有类C4解剖学特征的材料。
图5为株系1-1的解剖学分析,其具有类C4解剖学的部分特征。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
以下的实施例便于更好地理解本发明,但并不限定本发明。
以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
水稻品种日本晴(Oryza sativa L.ssp.Japonica),又称野生型水稻,用WT表示:中国水稻杂交育种中心(中国,长沙)。pCambia2300载体:pCAMBIA公司。农杆菌AGL1:中国质粒载体菌株细胞株基因保藏中心。
实施例1、CRV1蛋白及其编码基因的发现
一、类C4解剖学结构突变体crv1的发现
C4植物玉米比C3植物水稻有着较高的光合效率、生物学产量等,主要原因是C4 植物有着特殊的解剖学结构。利用水稻品种日本晴的T-DNA插入突变体库,借助显微镜观察,筛选具有类C4解剖学结构的突变体。在筛选中,发现了一个叶脉密度显著提高的突变体crv1(以2毫米叶脉数作为统计数)。营养生长阶段和生殖生长阶段,突变体crv1与野生型水稻(NIP)株型没有显著区别,株高约为110厘米(图1A)。突变体crv1和野生型的剑叶叶脉密度见图1B,突变体crv1叶脉密度为2毫米15个脉,野生型叶脉密度为2毫米9个脉。由于突变体crv1叶脉密度显著变密,非常类似C4 的叶脉结构,因此,用LOCR-6400光合仪,测定了突变体crv1和野生型的光合参数,突变体crv1的光合效率达21.47±0.73μmol m-2s-1,而野生型水稻达13.47± 0.24μmol m-2s-1,显著高于野生型水稻的光合效率。
二、基因定位
将类C4突变体crv1与籼稻品种Dular(OryzasativaL.ssp.Indica)组配杂交组合,这样产生的多态性将更加丰富,产生Fl代,Fl代自交产生F2代分离群体。
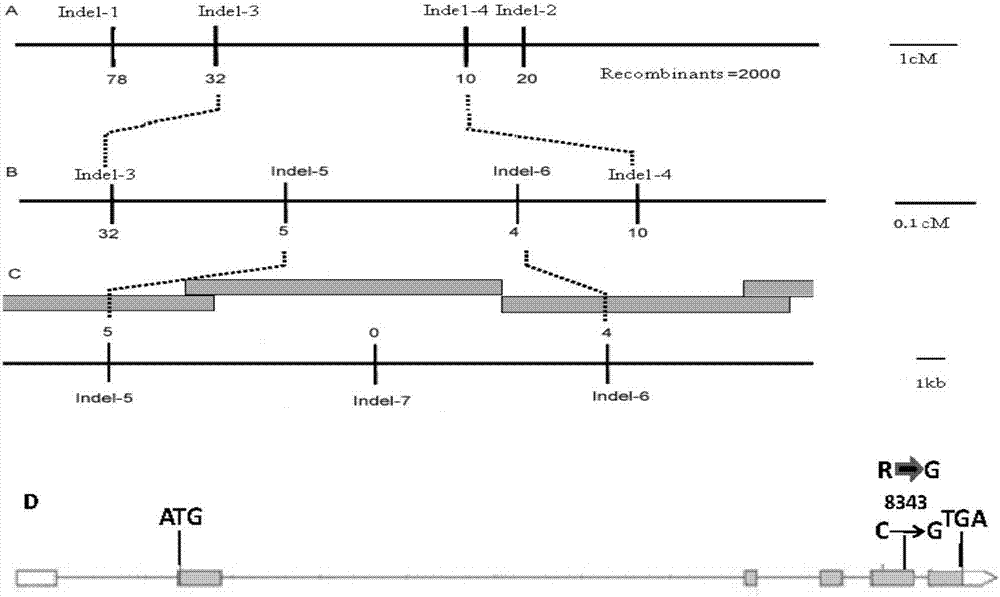
首先用F2分离群体进行定位,植株生长至成熟期,突变性状表现明显,利用显微镜观察,取突变体植株叶片提取基因组DNA进行基因定位。首先取100个F2突变体植株进行粗定位,在水稻12个连锁群上大约每隔约20cM选取多态性较好的分子标记,用40个突变体植株进行连锁分析,找到与CRV1连锁的分子标记后,用200个突变体植株进一步验证。验证正确后,上大群体进行染色体步移,进一步精细定位目的基因。共用2000个F2突变体植株将CRV1定位于100K区段内,在这个小区段内预测共有5 个完整的ORF,对5个ORF进行基因结构分析并全部进行测序分析,确定候选基因。见图2(A为初定位图,B为精细定位图)。
基于上述步骤,发现一个来源于水稻品种日本晴的新蛋白,将其命名为CRV1蛋白,其氨基酸序列如序列表中序列1所示。将编码CRV1蛋白的基因命名为CRV1基因,该基因的核苷酸序列为序列表中序列2所示,其开放阅读框为序列表中序列2自5’末端第132-1880位核苷酸所示。
实施例2、功能回补实验
一、重组质粒的构建
1、提取水稻品种日本晴的总RNA并反转录为cDNA。
2、以步骤1得到的cDNA为模板,用BucdsF和BucdsR组成的引物对进行PCR扩增,得到PCR扩增产物。
BucdsF:5’-CCCGGGCGCTTTCGGCATTCGTTATCTACC-3’;
BucdsR:5’-TCTAGAGGGATCCGGCAATGGTGTATATCA-3’。
3、用限制性内切酶SmaI和XbaI双酶切步骤2的PCR扩增产物,回收酶切产物。
4、用限制性内切酶SmaI和XbaI双酶切pCambia2300载体,回收载体骨架。
5、将步骤3得到的酶切产物和步骤4得到的载体骨架连接,得到重组质粒 pCambia2300-CRV1。
重组质粒pCambia2300-CRV1为在pCambia2300载体的SmaI和XbaI酶切位点之间插入了序列表中序列2所示的CRV1基因得到的质粒。
二、互补系植株的获得
1、将重组质粒pCambia2300-CRV1导入农杆菌AGL1,得到重组农杆菌。
2、取步骤1得到的重组农杆菌,重悬于液体共培养培养基(YEP液体培养基+100 mg/L乙酰丁香酮,pH5.2),得到OD600nm=1.0的菌液。
3、取突变体crv1(材料由国农业科学院生物技术研究所保存)的胚性愈伤组织,用步骤2得到的菌液浸泡30min,然后经共培养、筛选、生根、壮苗,得到T0代植株。
4、T0代植株自交获得T1代种子,T1代种子长成的植株即为T1代植株(互补系)。
三、互补系植株的鉴定
取生长状态一致的日本晴水稻、步骤二得到的4个互补系(bu-1、bu-2、bu-3和 bu-4)的T1代植株,在营养生殖晚期,进行叶脉密度观察。
结果如图3(A为互补系与突变体的成株期照片对比,B为互补系与突变体的叶脉对比)所示,互补系(bu-1、bu-2、bu-3和bu-4)(图中又称为cp1)均恢复叶脉密度减少的表型;表明CRV1蛋白具有降低叶脉密度的功能。
实施例3、类C4解剖学结构材料创制
一、利用CRISP9技术构建CRV1相关载体
1、设计CRV1基因3个区段,首先合成下述引物序列,通过梯度退火,获得带有粘性末端的双链DNA。
Region1-1的引物:
TGGCA gacgataaggcgcagctctc
AAAC GAGAGCTGCGCCTTATCGTCT
Region1-2的引物:
TGGCA ttgttctccatcaatccaac
AAAC GTTGGATTGATGGAGAACAA T
分别将Region1-1的引物和Region1-2的引物退火,得到带有粘性末端的Region1 和带有粘性末端的Region2。
用BbsI单酶切中间载体psgR-Cas9-OS(Molecular Plant Advance Access published September 5,2013),得到骨架载体,将骨架载体分别与带有粘性末端的 Region1和带有粘性末端的Region2通过T4连接酶连接,得到中间载体 psgR-Cas9-OS-Region1-1和中间载体psgR-Cas9-OS-Region1-2;
中间载体psgR-Cas9-OS-Region1-1为将序列2第13-33位所示的DNA片段出入中间载体psgR-Cas9-OS的BbsI酶切位点得到的载体。
中间载体p2×sgR-Cas9-OS-Region1-2为将序列2第87-107位所示的DNA片段出入中间载体p2×sgR-Cas9-OS的BbsI酶切位点得到的载体。
2、再分别用HindIII和EcoRI双酶切中间载体psgR-Cas9-OS-Region1-1和中间载体psgR-Cas9-OS-Region1-2,得到6000bp的Region1-1酶切产物和6100bp的 Region1-2酶切产物;
分别将6000bp的Region1-1酶切产物和6100bp的Region1-2酶切产物与经过同样酶切得到的10000bp pCambia1300载体(武汉淼灵生物科技有限公司购买)骨架连接,得到敲除载体pCambia1300-CRV1(1-1)和pCambia1300-CRV1(1-2)。
将得到的2个重组质粒pCambia1300-CRV1(1-1)、重组质粒pCambia1300-CRV1 (1-2)分别导入农杆菌AGL1,得到重组农杆菌。
2、取重组农杆菌,重悬于液体共培养培养基(YEP液体培养基+100mg/L乙酰丁香酮,pH5.2),得到OD600nm=1.0的菌液。
3、取野生型水稻日本晴的胚性愈伤组织,用步骤2得到的菌液浸泡30min,然后经共培养、筛选、生根、壮苗,得到T0代植株。
4、T0代植株自交获得T1代种子,T1代种子长成的植株即为T1代植株;T1代植株自交获得T2代种子,T2代种子长成的植株即为T2代植株,得到T2代转CRV1(1-1) 植株和T2代转CRV1(1-2)。
5、取T1代转CRV1(1-1)植株、T1代转CRV1(1-2)植株和T2代转CRV1(1-1) 植株和T2代转CRV1(1-2)植株的叶片,提取基因组DNA并采用BucdsF和BucdsR组成的引物对进行PCR鉴定,均得到454bp的为阳性片段。
BucdsF:CGACATCCAACCTACAGCATT
BucdsR:AACCTGAGAGCCTGAACCAAT
经过测序,重组质粒pCambia1300-CRV1(1-1)和重组质粒pCambia1300-CRV1(1-2) 的导入均使CRV1基因发生缺失。
二、转空载体植株的获得
用pCambia1300载体代替重组质粒pCambia1300-CRV1进行步骤一,得到转空载体植株。
三、C4解剖学结构材料鉴定
随机取2个纯合的转基因株系的T2代转CRV1(1-1)植株和T2代转CRV1(1-2)植株,每个株系各选一个有代表的株系:1-1株系、1-2株系,取转空载体植株的T2代植株和野生型水稻各20株。
将上述各个株系分别进行叶脉密度和光合效率鉴定及细胞学结构鉴定(叶脉密度方法详见PLoS One.2014Apr 23;9(4):e94947。光合参数测定详见LI-COR 6800便携式光合仪说明书)。
结果如图4(A为突变体crv1与野生型植株比较,B为株系1-1与野生型植株比较,C 为株系1-2与野生型植株比较)所示,与对照(野生型水稻)相比,1-1株系、1-2株系在不改变农艺性状的条件下,均具有C4植物的解剖学特征:叶脉密度增加(见图4,B,C 注括号代表叶脉数),光合效率提高(见表1),两个细胞间距减少(见图4,B,C注括号代表叶脉数),其已具有类C4解剖学结构的特征。
图5A为C3水稻与C4玉米的叶脉比较图,两种植物是C3与C4的代表模式植物,B 为细胞学结构显示野生型与株系1-1的两个脉间的细胞数和大小,其具有类C4解剖学的部分特征。
转基因与空对照的各个农艺性状比较如下表1所示(LI-COR 6800光合仪测定):
表1为转基因与空对照的各个农艺性状比较
上述结果表明,降低目的植物中CRV1蛋白表达,可以实现目的植物叶脉密度增加、光合效率提高和细胞间距减少,表现为C4的光合特征。
序列表
<110>中国农业科学院生物技术研究所
<120> C4水稻“底盘”受体材料的设计、创制与应用
<160> 2
<170> PatentIn version 3.5
<210> 1
<211> 1749
<212> PRT
<213> Artificial sequence
<400> 1
Met Lys Pro Ser Asp Asp Lys Ala Gln Leu Ser Gly Leu Ala Gln Ser
1 5 10 15
Glu Glu Ser Ser Leu Asp Val Asp His Gln Ser Phe Pro Cys Ser Pro
20 25 30
Ser Ile Gln Pro Val Ala Ser Gly Cys Thr His Thr Glu Asn Ser Ala
35 40 45
Ala Tyr Phe Leu Trp Pro Thr Ser Asn Leu Gln His Cys Ala Ala Glu
50 55 60
Gly Arg Ala Asn Tyr Phe Gly Asn Leu Gln Lys Gly Leu Leu Pro Arg
65 70 75 80
His Pro Gly Arg Leu Pro Lys Gly Gln Gln Ala Asn Ser Leu Leu Asp
85 90 95
Leu Met Thr Ile Arg Ala Phe His Ser Lys Ile Leu Arg Arg Phe Ser
100 105 110
Leu Gly Thr Ala Val Gly Phe Arg Ile Arg Lys Gly Asp Leu Thr Asp
115 120 125
Ile Pro Ala Ile Leu Val Phe Val Ala Arg Lys Val His Lys Lys Trp
130 135 140
Leu Asn Pro Ala Gln Cys Leu Pro Ala Ile Leu Glu Gly Pro Gly Gly
145 150 155 160
Val Trp Cys Asp Val Asp Val Val Glu Phe Ser Tyr Tyr Gly Ala Pro
165 170 175
Ala Gln Thr Pro Lys Glu Gln Met Phe Ser Glu Leu Val Asp Lys Leu
180 185 190
Cys Gly Ser Asp Glu Cys Ile Gly Ser Gly Ser Gln Val Ala Ser His
195 200 205
Glu Thr Phe Gly Thr Leu Gly Ala Ile Val Lys Arg Arg Thr Gly Asn
210 215 220
Lys Gln Val Gly Phe Leu Thr Asn His His Val Ala Val Asp Leu Asp
225 230 235 240
Tyr Pro Asn Gln Lys Met Phe His Pro Leu Pro Pro Asn Leu Gly Pro
245 250 255
Gly Val Tyr Leu Gly Ala Val Glu Arg Ala Thr Ser Phe Ile Thr Asp
260 265 270
Asp Val Trp Tyr Gly Ile Tyr Ala Gly Thr Asn Pro Glu Thr Phe Val
275 280 285
Arg Ala Asp Gly Ala Phe Ile Pro Phe Ala Asp Asp Phe Asp Ile Ser
290 295 300
Thr Val Thr Thr Val Val Arg Gly Val Gly Asp Ile Gly Asp Val Lys
305 310 315 320
Val Ile Asp Leu Gln Cys Pro Leu Asn Ser Leu Ile Gly Arg Gln Val
325 330 335
Cys Lys Val Gly Arg Ser Ser Gly His Thr Thr Gly Thr Val Met Ala
340 345 350
Tyr Ala Leu Glu Tyr Asn Asp Glu Lys Gly Ile Cys Phe Phe Thr Asp
355 360 365
Ile Leu Val Val Gly Glu Asn Arg Gln Thr Phe Asp Leu Glu Gly Asp
370 375 380
Ser Gly Ser Leu Ile Ile Leu Thr Ser Gln Asp Gly Glu Lys Pro Arg
385 390 395 400
Pro Ile Gly Ile Ile Trp Gly Gly Thr Ala Asn Arg Gly Arg Leu Lys
405 410 415
Leu Thr Ser Asp His Gly Pro Glu Asn Trp Thr Ser Gly Val Asp Leu
420 425 430
Gly Arg Leu Leu Asp Arg Leu Glu Leu Asp Ile Ile Ile Thr Asn Glu
435 440 445
Ser Leu Gln Asp Ala Val Gln Gln Gln Arg Phe Ala Leu Val Ala Ala
450 455 460
Val Thr Ser Ala Val Gly Glu Ser Ser Gly Val Pro Val Ala Ile Pro
465 470 475 480
Glu Glu Lys Ile Glu Glu Ile Phe Glu Pro Leu Gly Ile Gln Ile Gln
485 490 495
Gln Leu Pro Arg His Asp Val Ala Ala Ser Gly Thr Glu Gly Glu Glu
500 505 510
Ala Ser Asn Thr Val Val Asn Val Glu Glu His Gln Phe Ile Ser Asn
515 520 525
Phe Val Gly Met Ser Pro Val Arg Asp Asp Gln Asp Ala Pro Arg Ser
530 535 540
Ile Thr Asn Leu Asn Asn Pro Ser Glu Glu Glu Leu Ala Met Ser Leu
545 550 555 560
His Leu Gly Asp Arg Glu Pro Lys Arg Leu Arg Ser Asp Ser Gly Ser
565 570 575
Ser Leu Asp Leu Glu Lys
580
<210> 2
<211> 1749
<212> DNA
<213> Artificial sequence
<400> 2
atgaagcctt cggacgataa ggcgcagctc tcaggtttgg cgcaatcaga agaatcgtca 60
cttgatgtgg atcaccagtc atttccttgt tctccatcaa tccaaccggt tgcttctggg 120
tgcacacaca cagagaacag cgcagcatac ttcttatggc cgacatccaa cctacagcat 180
tgtgcagccg agggacgtgc aaactacttt ggaaaccttc agaaaggatt gttgccaagg 240
caccctggtc ggttgcccaa aggtcagcaa gcaaatagct tgcttgactt gatgactata 300
agagctttcc atagcaagat attgcggcgt tttagcctcg ggacagcagt gggattccgc 360
atcaggaaag gggatctaac agatatccct gcaatccttg tctttgttgc tcgcaaggtt 420
cataagaagt ggcttaatcc agcacaatgt cttcctgcta ttcttgaggg tccaggaggt 480
gtttggtgtg atgttgatgt tgttgaattt tcgtactacg gtgcaccggc tcaaacacct 540
aaagagcaaa tgttcagtga gcttgttgat aagttatgtg gcagtgacga atgtattggt 600
tcaggctctc aggttgcaag ccatgaaact tttggtactt tgggtgcaat tgtgaaacgg 660
cgcactggca acaagcaggt tggtttcctc actaaccatc atgtcgcggt tgacttggac 720
taccctaatc agaagatgtt tcatccatta ccacccaatc ttgggcctgg cgtttatctt 780
ggagctgttg aaagagcaac ttctttcatc acagatgacg tttggtatgg aatctatgct 840
ggaacaaacc cagagacatt tgtacgagct gacggtgcat ttatcccatt tgctgatgac 900
tttgacattt ccaccgtcac gactgtagtt aggggagtcg gtgacattgg ggatgtcaag 960
gttatagatc tgcagtgtcc gctcaatagc ctcataggga ggcaagtatg caaagttggc 1020
agaagctctg gtcacacaac tgggactgtg atggcctatg cccttgagta caatgacgag 1080
aaaggaatat gcttcttcac agacatcctt gttgttggtg agaaccgcca aacatttgat 1140
ttggaaggtg atagcggaag ccttattatc ctgactagcc aagatggtga gaagccgcgt 1200
ccaattggaa ttatatgggg tggcacagca aatcgtggga ggttgaagct tacaagtgat 1260
catggccctg aaaactggac tagtggggtt gatcttggcc gtctactcga ccgtctggaa 1320
cttgatatta tcattaccaa tgaatcactc caagatgccg tgcagcagca aagatttgct 1380
ttggtggccg ccgttacctc agctgttggg gagtcttccg gggtgcctgt cgccatcccg 1440
gaagagaaga tcgaagagat cttcgagcca ttggggatcc aaatccagca actgcctcgc 1500
catgacgtgg cggcctctgg aactgaaggg gaggaggcat ccaacacggt ggtcaatgtg 1560
gaagagcacc agttcatctc aaacttcgtc ggtatgtcgc ccgtgcgcga cgaccaagac 1620
gctccgagga gcatcaccaa cctgaacaac ccctccgagg aagaactcgc catgtcgctc 1680
catctgggtg accgagagcc caagcggctc cgttcggact ccggatcaag ccttgacctg 1740
gagaaatga 1749
- 还没有人留言评论。精彩留言会获得点赞!