一种荧光互补检测人趋化因子生物学活性的方法与流程
本发明涉及生物技术领域,更具体地,涉及一种荧光互补检测人趋化因子生物学活性的方法。
背景技术:
趋化因子是由多种细胞分泌的分子量在8-14kDa的可溶性蛋白,目前报道的趋化因子家族有48个成员,根据其蛋白氨基酸序列N端的色氨酸保守性可以分为C、CC、CXC、CX3C四类。趋化因子受体是G蛋白偶联受体(GPCR),趋化因子与其受体结合后,可以激活腺苷酸环化酶、磷脂酶C等经典的GPCR信号通路,调控cAMP、Ca2+水平,还可激活MAPK、PI3K及一些酪氨酸激酶信号通路,在免疫趋化、血管再生、创伤愈合、自身免疫、肿瘤发生等过程中起着重要的作用。现有的检测趋化因子生物学活性的方法,要检测细胞的趋化能力,一般需要用到Transwell小室,细胞受到梯度趋化因子刺激后穿过小室滤膜,黏附在膜的下面,染色并计数滤膜下表面的细胞数即可测出趋化因子的趋化能力,趋化活性常用的表示方法是趋化指数(chemotactic index,是指细胞迁移到待测样品液的数目和迁移到对照液的数目的比值),由于细胞类型不同,趋化因子需要调整浓度来获得比较好的趋向性,过程十分复杂。
荧光蛋白互补(Bimolecular fluorescence complementation,BiFC)主要是将一种荧光蛋白在合适氨基酸位点拆分为两部分,把拆分后的蛋白分别与要研究的两种目的蛋白融合,若目的蛋白能够相互结合,会拉近两个拆分的荧光蛋白并使得荧光蛋白结构恢复成拆分前状态,进而通过不同波长光激发,通过荧光显微镜检测相对应的荧光,方法直观。这种技术已在有多种应用,但是用于检测趋化因子生物学活性的还鲜有报道。
CXCR2是GPCR受体家族的重要成员,它的配体趋化因子是CXCL1、CXCL2、CXCL3、CXCL4、CXCL5、CXCL6、CXCL7、CXCL8,这些趋化因子与CXCR2结合后会使CXCR2与β-Arrestin2蛋白结合,并激活下游的信号通路。CXCR2在癌症的发生、发展过程中扮演着重要的角色,在患癌病人中的表达明显提高,越来越多的研究表明CXCR2是治疗癌症的重要靶标,如在横纹肌肉瘤疾病模型中,抑制CXCR2引导的骨髓来源的抑制性细胞(Myeloid-derived suppressor cells,MDSCs)的迁移可以提高PD-1抗体治疗肿瘤的效果。抑制CXCR2的功能还可以抑制急性和慢性胰腺炎(RJ,Sansom OJ,Morton JP,2015)等,抑制IL8-CXCR2信号通路能使白血病细胞系及骨髓增生细胞停滞在G0/G1期而对正常的造血干细胞没有影响(Schinke C,Giricz O,2015),由于CXCR2在疾病发生中的重要作用,CXCR2是治疗疾病的重要靶点,开发CXCR2抑制剂具有潜在的科研及应用价值,如SB225002是第一个被发现的CXCR2小分子抑制剂并被进一步改造为SB265610和SB656933,Ha H等人用CXCR2配体相关的药效模型进行筛选了一批CXCR2的抑制剂(Ha H,Debnath B,2015),但是这种方法需要检测钙信号、细胞增殖等,方法步骤上比较多,不是特别简单和直观。
利用BiFC技术检测趋化因子的生物学活性,将拆分的荧光蛋白片段与CXCR2、β-Arrestin2连接,在趋化因子存在下,CXCR2、β-Arrestin2空间上结合,促使拆分的荧光片段互补形成完整的荧光蛋白,可用荧光检测,方法简单。同时构建的细胞系还可用于CXCR2相关药物研究,有很好的科研、商业价值。
技术实现要素:
本发明的第一个目的是为了克服现有技术的不足,提供一种用于检测人趋化因子生物学活性的融合蛋白组合。
本发明的第二个目的是,提供一种编码用于检测人趋化因子生物学活性的融合蛋白的基因组合。
本发明的第三个目的是,提供一种用于检测人趋化因子生物学活性的融合蛋白的重组载体组合。
本发明的第四个目的是,提供一种用于检测人趋化因子生物学活性的融合蛋白的细胞系。
本发明的第五个目的是,提供一种用于检测趋化因子生物学活性的试剂盒。
本发明的第六个目的是,提供一种所述融合蛋白组合,所述基因组合,所述重组载体组合或所述细胞系在检测人趋化因子生物学活性中的应用。
本发明的第七个目的是,提供一种检测人趋化因子生物学活性的方法。
为了实现上述目的,本发明是通过以下技术方案予以实现的:
一种用于检测人趋化因子生物学活性的融合蛋白组合,包括融合蛋白CXCR2-VC和融合蛋白β-Arrestin2-VN,其氨基酸序列分别如SEQ ID NO.1和SEQ ID NO.2所示,其中,融合蛋白CXCR2-VC的氨基酸序列如SEQ ID NO.1所示,融合蛋白β-Arrestin2-VN的氨基酸序列如SEQ ID NO.2所示。
优选地,所述人趋化因子为CXCL1、CXCL2、CXCL3、CXCL4、CXCL5、CXCL6、CXCL7或CXCL8。
一种编码所述融合蛋白的基因组合,包括融合蛋白CXCR2-VC的编码基因和融合蛋白β-Arrestin2-VN的编码基因,其核苷酸序列分别如SEQ ID NO.3和SEQ ID NO.4所示。融合蛋白CXCR2-VC编码基因的核苷酸序列如SEQ ID NO.3所示,融合蛋白β-Arrestin2-VN编码基因的核苷酸序列如SEQ ID NO.4所示。
一种用于检测人趋化因子生物学活性的重组载体组合,包括重组载体pCDNA3.1-hCXCR2-linker-VC和重组载体pCNA3.1-hARRB2-linker-VN;其中,重组载体pCDNA3.1-hCXCR2-linker-VC插入有SEQ ID NO.3所示核苷酸序列;重组载体pCNA3.1-hARRB2-linker-VN插入有SEQ ID NO.4所示核苷酸序列。
优选地,所述人趋化因子为CXCL1、CXCL2、CXCL3、CXCL4、CXCL5、CXCL6、CXCL7或CXCL8。
一种用于检测人趋化因子生物学活性的细胞系,包含所述重组载体组合。
优选地,所述细胞系,其制备过程为:先将重组载体pCDNA3.1-hCXCR2-linker-VC转染受体细胞,培养18~24h后,再将重组载体pCDNA3.1-hARRB2-linker-VN转染受体细胞。
优选地,所述受体细胞为hela细胞,CHO细胞。
优选地,所述人趋化因子为CXCL1、CXCL2、CXCL3、CXCL4、CXCL5、CXCL6、CXCL7或CXCL8。
所述融合蛋白组合、所述基因组合、所述重组载体组合或所述细胞系在检测人趋化因子生物学活性中的应用。
所述融合蛋白组合、所述基因组合、所述重组载体组合或所述细胞系在制备检测人趋化因子生物学活性的试剂盒中的应用。
一种用于检测人趋化因子生物学活性的试剂盒,所述试剂盒包括所述的重组载体组合或所述细胞系。
一种检测人趋化因子生物学活性的方法,先将权利要求4所述重组载体pCDNA3.1-hCXCR2-linker-VC转染受体细胞,培养18~24h后,再将重组载体pCDNA3.1-hARRB2-linker-VN转染受体细胞,添加待测人趋化因子,根据荧光强弱来判断待测人趋化因子生物学活性高低。
优选地,所述受体细胞为hela细胞,CHO细胞。
所述GRO-α即CXCL1;GRO-β即CXCL2;GRO-γ即CXCL3;PF-4即CXCL4;ENA-78即CXCL5;IL8即CXCL8。本发明选择了Venus蛋白用于荧光互补,拆分的Venus根据N端、C端将其拆分片段称为VN、VC,与CXCR2、β-Arrestin2进行连接,构建多对融合基因组合,然后将融合基因克隆到pCDNA3.1重组载体上,瞬转到受体细胞中,并用其检测趋化因子生物学活性,在趋化因子存在下,趋化因子与其受体CXCR2结合,激活下游的信号通路,CXCR2、β-Arrestin2相互结合,使得Venus的VC、VN片段空间结构上紧密接触,形成完整的Venus蛋白,发出荧光信号(图1)。
与现有技术相比,本发明具有如下优势:
荧光互补检测趋化因子生物学活性依赖趋化因子与受体的分子结合来检测荧光,在荧光显微镜下直接观察荧光便可确定趋化因子有无活性,培养方便,不需要专门的Transwell小室;根据荧光荧光亮度定量测定趋化因子生物学活性,较Transwell小室法而言不需要进行细胞染色、不需调整最佳趋化因子浓度检测细胞迁移,操作简单;构建的稳转细胞系可长期使用,经济实惠,同时还可用于药物的筛选,这是一般的趋化因子检测活性的方法所不具备的。
通过优化黄色荧光蛋白Venus的拆分位点和linker序列的选择,成功的构建了用于检测人趋化因子生物学活性的融合蛋白组合。通过两个重组载体分别转化,克服了两个重组载体共转化背景荧光太强无法达到检测目的的问题。本发明构建的融合蛋白组进行趋化因子的生物学活性检测,简单易行,步骤简单,检测效果好,结果可靠。
附图说明
图1为BiFC测定CXCR2与β-Arrestin2蛋白相互作用原理图。
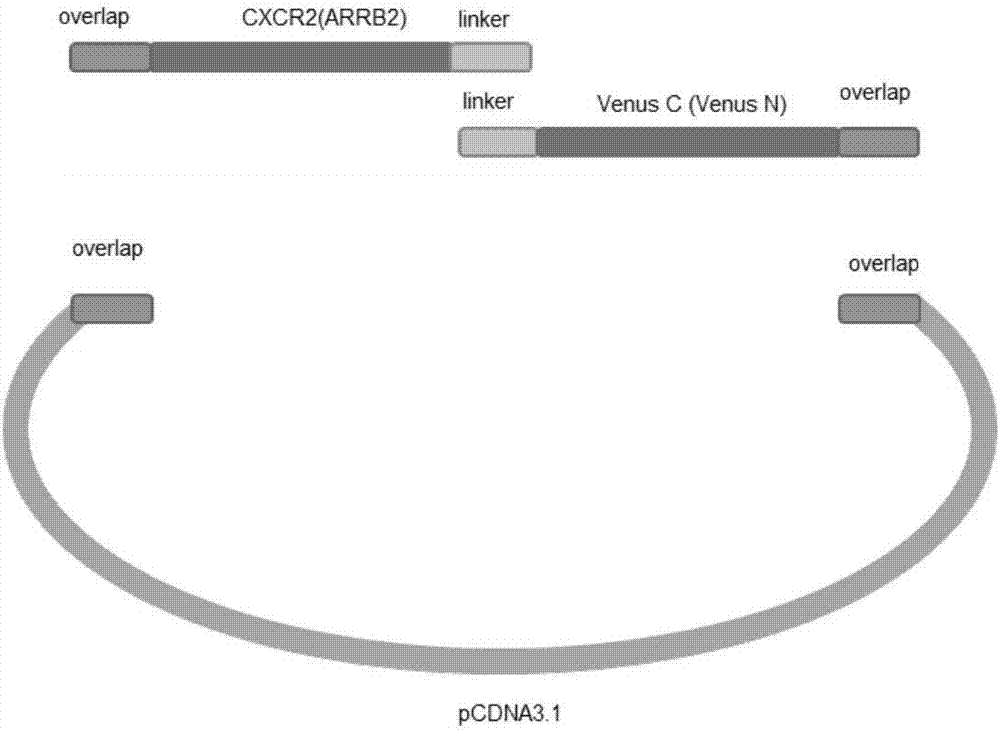
图2为无缝克隆构建融合蛋白模式图。
图3为pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN转化的细胞系,没有添加趋化因子处理。
图4为转化后的细胞系检测趋化因子GRO-α即CXCL1。
图5为转化后的细胞系检测趋化因子GRO-β即CXCL2。
图6为转化后的细胞系检测趋化因子IL8即CXCL8。
图7为pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN共转化的细胞系,没有添加趋化因子处理。
图8为先转化pCDNA3.1-hARRB2-linker-VN重组载体再转染pCDNA3.1-hCXCR2-linker-VC重组载体,没有添加趋化因子处理。
图9为其他黄色荧光蛋白拆分位点和linker序列的组合构建的重组载体检测人趋化因子IL8。
具体实施方式
下面结合说明书附图和具体实施例对本发明作出进一步地详细阐述,所述实施例只用于解释本发明,并非用于限定本发明的范围。下述实施例中所使用的试验方法如无特殊说明,均为常规方法;所使用的材料、试剂等,如无特殊说明,为可从商业途径得到的试剂和材料。
实施例1 pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN无缝克隆重组载体构建
1、重组载体构建流程
将黄色荧光蛋白Venus在其153-154氨基酸位点进行了拆分,称为Venus-N(VN,1~153)和Venus-C(VC,154~238),在CXCR2与VC之间、β-Arrestin2与VN之间分别加上一段linker序列,构建到表达载体上。
用pCDNA3.1作为载体,BamHI、XhoI作为酶切位点,采取无缝克隆的方法,构建人hCXCR2-VC和人hARRB2-VN重组载体(图2),根据Gene Bank人hCXCR2、人hARRB2、Venus荧光蛋白核酸序列设计hCXCR2-VC和hARRB2-VNPCR扩增所需引物,分别以实验室保存的pMD18-T-CXCR2(人IL8Rb/CXCR2基因ORF全长cDNA克隆载体)、pGEM-T-ARRB2(人β-Arrestin 2/ARRB2基因ORF全长cDNA克隆载体)、pmVenus(Venus荧光蛋白质粒)作为DNA模板进行PCR扩增。
2、PCR反应及胶回收
通过PCR扩增CXCR2-linker、ARRB2-linker、linker-VN、linker-VC。其中,引物序列如下:
pCDNA3.1overlap-CXCR2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGAAGATTTTAACATGGAGA-3'(划线部分为BamHI酶切位点,加粗部分为与载体pCDNA3.1上重叠的overlap序列);
CXCR2-linker R:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGAGAGTAGTGGAAGTGTGCCC-3'(加粗部分为linker序列);
linker-VC(154~239)F:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTGCCGACAAGCAGAAGAACG-3'(加粗部分为linker序列);
linker-VC(154-239)-pCDNA3.1overlap R:
5'-GGGCCCTCTAGACTCGAGTTACTTGTACAGCTCGTCCATG-3'(划线部分为XhoI酶切位点,加粗部分为与载体pCDNA3.1上重叠的overlap序列)
pCDNA3.1overlap-ARRB2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGGGGAGAAACCCGGGAC-3'(划线部分为BamHI酶切位点,加粗部分为与载体pCDNA3.1上重叠的overlap序列);
β-arrestin-linker R:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGCAGAGTTGATCATCATAGTCGT-3'(加粗部分为linker序列);
linker-VN(1-153)F:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTGTGAGCAAGGGCGAGGAGC-3'(加粗部分为linker序列);
linker-VN(1-153)-pCDNA3.1overlap R:
5'-GGGCCCTCTAGACTCGAGTTAGGTGATATAGACGGCGTGGCT-3'(划线部分为XhoI酶切位点,加粗部分为与载体pCDNA3.1上重叠的overlap序列)
PCR反应体系为:
PCR反应条件为:
其中,ARRB2-linker片段PCR反应条件与CXCR2-linker片段相同。linker-VC、linker-VN片段较短,PCR反应时,延伸时间改为1min。
PCR扩增反应完成后,1%琼脂糖凝胶电泳,天根胶回收试剂盒胶回收目的DNA片段,分光光度计测胶回收产物浓度。
3、无缝克隆
用擎科生物科技有限公司的无缝克隆试剂盒TreliefTMSoSoo Cloning Kit做pCDNA3.1-hCXCR2-linker-VC及pCNA3.1-hARRB2-linker-VN重组载体构建。操作如下:
pCDNA3.1重组载体用NEB公司BamHI、XhoI进行双酶切,其双酶切体系为:
37℃酶切2h,1%琼脂糖凝胶电泳,天根胶回收试剂盒胶回收大片段目的DNA,分光光度计测定胶回收产物浓度。
将回收产物进行无缝克隆,其中,ARRB2-linker和linker-VN进行连接,hCXCR2-linker和linker-VC进行连接,按照无缝克隆试剂盒说明书加样:
50℃反应40min,后与30μlTop10感受态混匀,冰上放置30min,42℃热击90s,后冰上放置3min,加500μl不含抗生素的LB,37℃、200rpm摇1h,低速离心后,去掉部分上清,留100μl液体将菌体吹打混匀,涂氨苄抗生素平板,37℃培养箱培养过夜,挑取单克隆菌落,含氨苄抗生素LB培养,送菌液至擎科生物科技有限公司测序。
4、菌液经过测序,载体pCDNA3.1插入的片段与预期的一致,融合蛋白CXCR2-VC编码基因的核苷酸序列如SEQ ID NO.3所示,融合蛋白β-Arrestin2-VN编码基因的核苷酸序列如SEQ ID NO.4所示,融合蛋白CXCR2-VC的氨基酸序列如SEQ ID NO.1所示,融合蛋白β-Arrestin2-VN的氨基酸序列如SEQ ID NO.2所示。
实施例2荧光互补技术的细胞学检测
按照实施例1的方法构建成功pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN重组载体后,用Lipo2000做转染试剂,转染Hela细胞,后用荧光显微镜检测是否有荧光。
1、Hela细胞复苏后,细胞培养箱37℃、5%CO2的培养条件下用含10%FBS的MEM培养基培养细胞,2~3天后,胰酶消化后用玻璃皿铺细胞,过夜培养细胞至铺满玻璃皿面积的70%~80%覆盖率,按照Lipo2000说明书进行转染。
2、第一次转染:
0.8μg pCDNA3.1-hCXCR2-linker-VC重组载体用50μl不含血清的MEM混匀,2μl Lipofectamine 2000用50μl不含血清的MEM混匀,室温静置5min;将二者混匀,室温静置20min,后加到玻璃皿中进行细胞转染,加不含血清的MEM培养基400μl培养4h后,换用含10%FBS的MEM培养基500μl培养18-24h。
3、第二次转染:
0.8μg pCDNA3.1-hARRB2-linker-VN重组载体用50μl不含血清的MEM混匀,2μl Lipofectamine 2000用50μl不含血清的MEM混匀,室温静置5min;将二者混匀,室温静置20min,后加到玻璃皿中进行二次转染,不含血清的MEM培养基400μl培养4h后,换用含10%FBS的MEM培养基500μl培养18-24h。
4、荧光显微镜检测:
用GRO-α、GRO-β、IL8处理转染了hCXCR2-VC和hARRB2-VN重组载体的细胞,对其进行荧光检测,与未处理的转染后的细胞进行比较。
细胞第二次转染后,培养18~24h,去掉培养基,PBS洗两次后,加400μl含10%FBS的MEM培养基,后加1μl 100ng/μl的趋化因子(GRO-α、GRO-β、IL8),30min后用荧光显微镜进行荧光检测,由于荧光显微镜没有Venus黄色荧光通道,选用GFP绿色荧光通道。
5、结果:
本发明采用分步转染的方法,先将pCDNA3.1-hCXCR2-linker-VC重组载体转到Hela细胞中,培养18~24h后再转染pCDNA3.1-hARRB2-linker-VN重组载体,发现基本没有背景荧光(图3)。
添加了趋化因子GRO-α、GRO-β、IL8后,CXCR2信号通路被激活,CXCR2会与β-Arrestin2(即hARRB2)蛋白结合,在荧光显微镜激发光下黄色荧光蛋白Venus发出荧光(图4~图6)
对比例1
按照实施例1的方法构建成功pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN重组载体后,按照实施例2的方法进行荧光互补技术的细胞学检测,其中,将两个重组载体同时共转化CHO细胞。
结果显示:共转到CHO细胞后发现不加趋化因子处理,细胞也能发荧光,背景荧光太强会对这种技术的应用造成很大干扰(图7)。
对比例2
按照实施例1的方法构建成功pCDNA3.1-hCXCR2-linker-VC和pCDNA3.1-hARRB2-linker-VN重组载体后,按照实施例2的方法进行荧光互补技术的细胞学检测,其中,转化细胞的具体操作为:先转化pCDNA3.1-hARRB2-linker-VN重组载体到CHO细胞中,培养18~24h后再转染pCDNA3.1-hCXCR2-linker-VC重组载体。
结果显示:先转pCDNA3.1-hARRB2-linker-VN,
后转pCDNA3.1-hCXCR2-linker-VC细胞后发现不加趋化因子处理,细胞也能发荧光,背景荧光太强会对这种技术的应用造成很大干扰(图8)。
对比例3
1、按照实施例1的方法的构建pCDNA3.1-hCXCR2-linker-VC1和pCDNA3.1-hARRB2-linker-VN1重组载体。
其中,将黄色荧光蛋白Venus在其Val-Tyr氨基酸位点进行了拆分,称为Venus-N(VN,1~150)和Venus-C(VC,151~238),在CXCR2与VC之间、β-Arrestin2与VN之间分别加上蛋白linker GSSSS、GGGGS、GSGSG,构建到表达载体上。
其中,在pCDNA3.1overlap-CXCR2-linker-VC1重组载体构建时,引物序列如下(加粗部分为linker序列),
pCDNA3.1overlap-CXCR2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGAAGATTTTAACATGGAGA-3'(划线部分为BamHI酶切位点)
左臂R根据linker序列引物如下,
GSSSS linker:
5'-CGATGAGGAAGATCCCGATGAGGAAGATCCCGATGAGGAAGATCCGAGAGTAGTGGAAGTGTGCCC-3',
GGGGS linker:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGAGAGTAGTGGAAGTGTGCCC-3',
GSGSG linker:
5'-ACCGGAGCCAGATCCACCGGAGCCAGATCCACCGGAGCCAGATCCGAGAGTAGTGGAAGTGTGCCC-3';
右臂F根据linker序列引物如下,
GSSSS linker:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGTATATCACCGCCGACAAG-3',
GGGGS linker:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTTATATCACCGCCGACAAG-3',
GSGSG linker:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGTATATCACCGCCGACAAG-3';
右臂R引物:
5'-GGGCCCTCTAGACTCGAGTTACTTGTACAGCTCGTCCATG-3'(划线部分为XhoI酶切位点)。
在pCDNA3.1overlap-ARRB2-linker-VN1重组载体构建时,引物序列如下(加粗部分为linker序列),
pCDNA3.1overlap-ARRB2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGGGGAGAAACCCGGGAC-3'(划线部分为BamHI酶切位点);
左臂R根据linker序列引物如下,GSSSS linker时:
5'-CGATGAGGAAGATCCCGATGAGGAAGATCCCGATGAGGAAGATCCGCAGAGTTGATCATCATAGTCGT-3',
GGGGS linker时:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGCAGAGTTGATCATCATAGTCGT-3',
GSGSG linker时:
5'-ACCGGAGCCAGATCCACCGGAGCCAGATCCACCGGAGCCAGATCCGCAGAGTTGATCATCATAGTCGT-3',
右臂F根据linker序列引物如下,
GSSSS linker时:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGTGAGCAAGGGCGAGGAGC-3',
GGGGS linker时:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTGTGAGCAAGGGCGAGGAGC-3',
GSGSG linker时:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGTGAGCAAGGGCGAGGAGC-3',
右臂R序列引物:
5'-GGGCCCTCTAGACTCGAGTTAGACGTTGTGGCTGTTGT-3'(划线部分为XhoI酶切位点)。
2、荧光互补技术的细胞学检测
按照实施例2的方法,使用上一步制备的重组质粒检测人趋化因子IL8。
3、结果显示,改变了黄色荧光蛋白Venus的拆分位点和linker序列将直接影响对于人趋化因子活性状态的检测效果(图9)。
对比例4
按照实施例1的方法的构建pCDNA3.1-hCXCR2-linker-VC2和pCDNA3.1-hARRB2-linker-VN2重组载体。
其中,将黄色荧光蛋白Venus在其Asn-Gly氨基酸位点进行了拆分,称为Venus-N(VN,1~159)和Venus-C(VC,160~238),在CXCR2与VC之间、β-Arrestin2与VN之间分别加上linkerGSSSS、GGGGS、GSGSG序列,构建到表达载体上。
在pCDNA3.1overlap-CXCR2-linker-VC2重组载体构建时,引物序列如下(加粗部分为linker序列),
pCDNA3.1overlap-CXCR2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGAAGATTTTAACATGGAGA-3'(划线部分为BamHI酶切位点);
左臂R根据linker序列引物如下,GSSSS linker时:
5'-CGATGAGGAAGATCCCGATGAGGAAGATCCCGATGAGGAAGATCCGAGAGTAGTGGAAGTGTGCCC-3',
GGGGS linker时:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGAGAGTAGTGGAAGTGTGCCC-3',
GSGSG linker时:
5'-ACCGGAGCCAGATCCACCGGAGCCAGATCCACCGGAGCCAGATCCGAGAGTAGTGGAAGTGTGCCC-3';
右臂F根据linker序列引物如下,GSSSS linker是:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGGCATCAAGGCCAACTTCAAG-3',
GGGGS linker:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTGGCATCAAGGCCAACTTCAAG-3',
GSGSG linker时:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGGCATCAAGGCCAACTTCAAG-3';
右臂R引物:
5'-GGGCCCTCTAGACTCGAGTTACTTGTACAGCTCGTCCATG-3'(划线部分为XhoI酶切位点)。
在pCDNA3.1overlap-ARRB2-linker-VN2质粒构建重组载体构建时,引物序列如下(加粗部分为linker序列),
pCDNA3.1overlap-ARRB2-linker F:
5'-AAGCTTGGTACCGAGCTCGGATCCATGGGGGAGAAACCCGGGAC-3'(划线部分为BamHI酶切位点);
左臂R根据linker序列,GSSSS linker时:
5'-CGATGAGGAAGATCCCGATGAGGAAGATCCCGATGAGGAAGATCCGCAGAGTTGATCATCATAGTCGT-3',
GGGGS linker时:
5'-ACTCCCGCCACCTCCACTCCCGCCACCTCCACTCCCGCCACCTCCGCAGAGTTGATCATCATAGTCGT-3',
GSGSG linker时:
5'-ACCGGAGCCAGATCCACCGGAGCCAGATCCACCGGAGCCAGATCCGCAGAGTTGATCATCATAGTCGT-3';
右臂F根据linker序列引物如下,GSSSS linker时:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGTGAGCAAGGGCGAGGAGC-3',
GGGGS linker时:
5'-GGAGGTGGCGGGAGTGGAGGTGGCGGGAGTGTGAGCAAGGGCGAGGAGC-3',
GSGSG linker时:
5'-GGATCTTCCTCATCGGGATCTTCCTCATCGGTGAGCAAGGGCGAGGAGC-3',
右臂R序列引物:
5'-GGGCCCTCTAGACTCGAGTTAGTTCTTCTGCTTGTCGG-3'(划线部分为XhoI酶切位点)。
2、荧光互补技术的细胞学检测
按照实施例2的方法,使用上一步制备的重组质粒检测人趋化因子IL8。
3、结果显示,改变了黄色荧光蛋白Venus的拆分位点和linker序列将直接影响对于人趋化因子活性状态的检测效果,无法达到检测目的(图9)。
序列表
<110> 上海晶诺生物科技有限公司
<120> 一种荧光互补检测人趋化因子生物学活性的方法
<160> 4
<170> SIPOSequenceListing 1.0
<210> 1
<211> 460
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 1
Met Glu Asp Phe Asn Met Glu Ser Asp Ser Phe Glu Asp Phe Trp Lys
1 5 10 15
Gly Glu Asp Leu Ser Asn Tyr Ser Tyr Ser Ser Thr Leu Pro Pro Phe
20 25 30
Leu Leu Asp Ala Ala Pro Cys Glu Pro Glu Ser Leu Glu Ile Asn Lys
35 40 45
Tyr Phe Val Val Ile Ile Tyr Ala Leu Val Phe Leu Leu Ser Leu Leu
50 55 60
Gly Asn Ser Leu Val Met Leu Val Ile Leu Tyr Ser Arg Val Gly Arg
65 70 75 80
Ser Val Thr Asp Val Tyr Leu Leu Asn Leu Ala Leu Ala Asp Leu Leu
85 90 95
Phe Ala Leu Thr Leu Pro Ile Trp Ala Ala Ser Lys Val Asn Gly Trp
100 105 110
Ile Phe Gly Thr Phe Leu Cys Lys Val Val Ser Leu Leu Lys Glu Val
115 120 125
Asn Phe Tyr Ser Gly Ile Leu Leu Leu Ala Cys Ile Ser Val Asp Arg
130 135 140
Tyr Leu Ala Ile Val His Ala Thr Arg Thr Leu Thr Gln Lys Arg Tyr
145 150 155 160
Leu Val Lys Phe Ile Cys Leu Ser Ile Trp Gly Leu Ser Leu Leu Leu
165 170 175
Ala Leu Pro Val Leu Leu Phe Arg Arg Thr Val Tyr Ser Ser Asn Val
180 185 190
Ser Pro Ala Cys Tyr Glu Asp Met Gly Asn Asn Thr Ala Asn Trp Arg
195 200 205
Met Leu Leu Arg Ile Leu Pro Gln Ser Phe Gly Phe Ile Val Pro Leu
210 215 220
Leu Ile Met Leu Phe Cys Tyr Gly Phe Thr Leu Arg Thr Leu Phe Lys
225 230 235 240
Ala His Met Gly Gln Lys His Arg Ala Met Arg Val Ile Phe Ala Val
245 250 255
Val Leu Ile Phe Leu Leu Cys Trp Leu Pro Tyr Asn Leu Val Leu Leu
260 265 270
Ala Asp Thr Leu Met Arg Thr Gln Val Ile Gln Glu Thr Cys Glu Arg
275 280 285
Arg Asn His Ile Asp Arg Ala Leu Asp Ala Thr Glu Ile Leu Gly Ile
290 295 300
Leu His Ser Cys Leu Asn Pro Leu Ile Tyr Ala Phe Ile Gly Gln Lys
305 310 315 320
Phe Arg His Gly Leu Leu Lys Ile Leu Ala Ile His Gly Leu Ile Ser
325 330 335
Lys Asp Ser Leu Pro Lys Asp Ser Arg Pro Ser Phe Val Gly Ser Ser
340 345 350
Ser Gly His Thr Ser Thr Thr Leu Gly Gly Gly Gly Ser Gly Gly Gly
355 360 365
Gly Ser Gly Gly Gly Gly Ser Ala Asp Lys Gln Lys Asn Gly Ile Lys
370 375 380
Ala Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Gly Val Gln Leu
385 390 395 400
Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly Pro Val Leu
405 410 415
Leu Pro Asp Asn His Tyr Leu Ser Tyr Gln Ser Ala Leu Ser Lys Asp
420 425 430
Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe Val Thr Ala
435 440 445
Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys
450 455 460
<210> 2
<211> 598
<212> PRT
<213> 人工序列(Artificial Sequence)
<400> 2
Met Gly Glu Lys Pro Gly Thr Arg Val Phe Lys Lys Ser Ser Pro Asn
1 5 10 15
Cys Lys Leu Thr Val Tyr Leu Gly Lys Arg Asp Phe Val Asp His Leu
20 25 30
Asp Lys Val Asp Pro Val Asp Gly Val Val Leu Val Asp Pro Asp Tyr
35 40 45
Leu Lys Asp Arg Lys Val Phe Val Thr Leu Thr Cys Ala Phe Arg Tyr
50 55 60
Gly Arg Glu Asp Leu Asp Val Leu Gly Leu Ser Phe Arg Lys Asp Leu
65 70 75 80
Phe Ile Ala Thr Tyr Gln Ala Phe Pro Pro Val Pro Asn Pro Pro Arg
85 90 95
Pro Pro Thr Arg Leu Gln Asp Arg Leu Leu Arg Lys Leu Gly Gln His
100 105 110
Ala His Pro Phe Phe Phe Thr Val Arg Met Pro Leu Pro Ser Glu Gly
115 120 125
Gln Gly Ala Gly Ala Gly Thr Val Ser Gly Val Gly Ile Pro Gln Asn
130 135 140
Leu Pro Cys Ser Val Thr Leu Gln Pro Gly Pro Glu Asp Thr Gly Lys
145 150 155 160
Ala Cys Gly Val Asp Phe Glu Ile Arg Ala Phe Cys Ala Lys Ser Leu
165 170 175
Glu Glu Lys Ser His Lys Arg Asn Ser Val Arg Leu Val Ile Arg Lys
180 185 190
Val Gln Phe Ala Pro Glu Lys Pro Gly Pro Gln Pro Ser Ala Glu Thr
195 200 205
Thr Arg His Phe Leu Met Ser Asp Arg Ser Leu His Leu Glu Ala Ser
210 215 220
Leu Asp Lys Glu Leu Tyr Tyr His Gly Glu Pro Leu Asn Val Asn Val
225 230 235 240
His Val Thr Asn Asn Ser Thr Lys Thr Val Lys Lys Ile Lys Val Ser
245 250 255
Val Arg Gln Tyr Ala Asp Ile Cys Leu Phe Ser Thr Ala Gln Tyr Lys
260 265 270
Cys Pro Val Ala Gln Leu Glu Gln Asp Asp Gln Val Ser Pro Ser Ser
275 280 285
Thr Phe Cys Lys Val Tyr Thr Ile Thr Pro Leu Leu Ser Asp Asn Arg
290 295 300
Glu Lys Arg Gly Leu Ala Leu Asp Gly Lys Leu Lys His Glu Asp Thr
305 310 315 320
Asn Leu Ala Ser Ser Thr Ile Val Lys Glu Gly Ala Asn Lys Glu Val
325 330 335
Leu Gly Ile Leu Val Ser Tyr Arg Val Lys Val Lys Leu Val Val Ser
340 345 350
Arg Gly Gly Asp Val Ser Val Glu Leu Pro Phe Val Leu Met His Pro
355 360 365
Lys Pro His Asp His Ile Pro Leu Pro Arg Pro Gln Ser Ala Ala Pro
370 375 380
Glu Thr Asp Val Pro Val Asp Thr Asn Leu Ile Glu Phe Asp Thr Asn
385 390 395 400
Tyr Ala Thr Asp Asp Asp Ile Val Phe Glu Asp Phe Ala Arg Leu Arg
405 410 415
Leu Lys Gly Met Lys Asp Asp Asp Tyr Asp Asp Gln Leu Cys Gly Gly
420 425 430
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val Ser Lys
435 440 445
Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu Val Glu Leu Asp
450 455 460
Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly Glu Gly Glu Gly
465 470 475 480
Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Leu Ile Cys Thr Thr Gly
485 490 495
Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr Leu Gly Tyr Gly
500 505 510
Val Gln Cys Phe Ala Arg Tyr Pro Asp His Met Lys Gln His Asp Phe
515 520 525
Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu Arg Thr Ile Phe
530 535 540
Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu Val Lys Phe Glu
545 550 555 560
Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly Ile Asp Phe Lys
565 570 575
Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr Asn Tyr Asn Ser
580 585 590
His Ala Val Tyr Ile Thr
595
<210> 3
<211> 1383
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
atggaagatt ttaacatgga gagtgacagc tttgaagatt tctggaaagg tgaagatctt 60
agtaattaca gttacagctc taccctgccc ccttttctac tagatgccgc cccatgtgaa 120
ccagaatccc tggaaatcaa caagtatttt gtggtcatta tctatgccct ggtattcctg 180
ctgagcctgc tgggaaactc cctcgtgatg ctggtcatct tatacagcag ggtcggccgc 240
tccgtcactg atgtctacct gctgaaccta gccttggccg acctactctt tgccctgacc 300
ttgcccatct gggccgcctc caaggtgaat ggctggattt ttggcacatt cctgtgcaag 360
gtggtctcac tcctgaagga agtcaacttc tatagtggca tcctgctact ggcctgcatc 420
agtgtggacc gttacctggc cattgtccat gccacacgca cactgaccca gaagcgctac 480
ttggtcaaat tcatatgtct cagcatctgg ggtctgtcct tgctcctggc cctgcctgtc 540
ttacttttcc gaaggaccgt ctactcatcc aatgttagcc cagcctgcta tgaggacatg 600
ggcaacaata cagcaaactg gcggatgctg ttacggatcc tgccccagtc ctttggcttc 660
atcgtgccac tgctgatcat gctgttctgc tacggattca ccctgcgtac gctgtttaag 720
gcccacatgg ggcagaagca ccgggccatg cgggtcatct ttgctgtcgt cctcatcttc 780
ctgctctgct ggctgcccta caacctggtc ctgctggcag acaccctcat gaggacccag 840
gtgatccagg agacctgtga gcgccgcaat cacatcgacc gggctctgga tgccaccgag 900
attctgggca tccttcacag ctgcctcaac cccctcatct acgccttcat tggccagaag 960
tttcgccatg gactcctcaa gattctagct atacatggct tgatcagcaa ggactccctg 1020
cccaaagaca gcaggccttc ctttgttggc tcttcttcag ggcacacttc cactactctc 1080
ggaggtggcg ggagtggagg tggcgggagt ggaggtggcg ggagtgccga caagcagaag 1140
aacggcatca aggcgaactt caagatccgc cacaacatcg aggacggcgg cgtgcagctc 1200
gccgaccact accagcagaa cacccccatc ggcgacggcc ccgtgctgct gcccgacaac 1260
cactacctga gctaccagtc cgccctgagc aaagacccca acgagaagcg cgatcacatg 1320
gtcctgctgg agttcgtgac cgccgccggg atcactctcg gcatggacga gctgtacaag 1380
taa 1383
<210> 4
<211> 1797
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
atgggggaga aacccgggac cagggtcttc aagaagtcga gccctaactg caagctcacc 60
gtgtacttgg gcaagcggga cttcgtagat cacctggaca aagtggaccc tgtagatggc 120
gtggtgcttg tggaccctga ctacctgaag gaccgcaaag tgtttgtgac cctcacctgc 180
gccttccgct atggccgtga agacctggat gtgctgggct tgtccttccg caaagacctg 240
ttcatcgcca cctaccaggc cttccccccg gtgcccaacc caccccggcc ccccacccgc 300
ctgcaggacc ggctgctgag gaagctgggc cagcatgccc accccttctt cttcaccgtg 360
aggatgcccc tgccctctga gggccagggg gctggggctg ggactgtgtc tggggtgggg 420
ataccccaga atcttccatg ctccgtcaca ctgcagccag gcccagagga tacaggaaag 480
gcctgcggcg tagactttga gattcgagcc ttctgtgcta aatcactaga agagaaaagc 540
cacaaaagga actctgtgcg gctggtgatc cgaaaggtgc agttcgcccc ggagaaaccc 600
ggcccccagc cttcagccga aaccacacgc cacttcctca tgtctgaccg gtccctgcac 660
ctcgaggctt ccctggacaa ggagctgtac taccatgggg agcccctcaa tgtaaatgtc 720
cacgtcacca acaactccac caagaccgtc aagaagatca aagtctctgt gagacagtac 780
gccgacatct gcctcttcag caccgcccag tacaagtgtc ctgtggctca actcgaacaa 840
gatgaccagg tatctcccag ctccacattc tgtaaggtgt acaccataac cccactgctc 900
agcgacaacc gggagaagcg gggtctcgcc ctggatggga aactcaagca cgaggacacc 960
aacctggctt ccagcaccat cgtgaaggag ggtgccaaca aggaggtgct gggaatcctg 1020
gtgtcctaca gggtcaaggt gaagctggtg gtgtctcgag gcggggatgt ctctgtggag 1080
ctgccttttg ttcttatgca ccccaagccc cacgaccaca tccccctccc cagaccccag 1140
tcagccgctc cggagacaga tgtccctgtg gacaccaacc tcattgaatt tgataccaac 1200
tatgccacag atgatgacat tgtgtttgag gactttgccc ggcttcggct gaaggggatg 1260
aaggatgacg actatgatga tcaactctgc ggaggtggcg ggagtggagg tggcgggagt 1320
ggaggtggcg ggagtgtgag caagggcgag gagctgttca ccggggtggt gcccatcctg 1380
gtcgagctgg acggcgacgt aaacggccac aagttcagcg tgtccggcga gggcgagggc 1440
gatgccacct acggcaagct gaccctgaag ttgatctgca ccaccggcaa gctgcccgtg 1500
ccctggccca ccctcgtgac caccttgggc tacggcgtgc agtgcttcgc ccgctacccc 1560
gaccacatga agcagcacga cttcttcaag tccgccatgc ccgaaggcta cgtccaggag 1620
cgcaccatct tcttcaagga cgacggcaac tacaagaccc gcgccgaggt gaagttcgag 1680
ggcgacaccc tggtgaaccg catcgagctg aagggcatcg acttcaagga ggacggcaac 1740
atcctggggc acaagctgga gtacaactac aacagccacg ccgtctatat cacctaa 1797
- 还没有人留言评论。精彩留言会获得点赞!