促进基因编辑的化合物筛选和鉴定及其应用的制作方法
本发明涉及生物学领域,更具体地涉及促进基因编辑的化合物筛选和鉴定及其应用。
背景技术:
随着基因编辑技术的出现,对于不同的细胞进行有效的基因编辑和改造成为可能。以多能干细胞为例,它包括胚胎干细胞(hescs)和诱导多能干细胞(hipscs),可用于研究早期发育和疾病发生发展。因此,对细胞(包括体细胞、多能干细胞等)进行快速、高效、可控的基因编辑是至关重要的。
位点特异性识别的核酸酶可以在基因组特定位置造成dna双链的断裂,并触发内源性的dna修复机制。采用非同源末端接合途径修复dna双链断裂(nhej)的方式,会导致小片段插入或缺失,该修复方式可用于产生敲除突变体。同源定向修复(hdr)则可以用来构建敲入突变体或报告细胞系。然而,即使在这些位点特异性核酸酶的协助下,通过同源定向修复进行精确的基因组编辑仍然是非常具有挑战性的。
随着cas9等核酸酶的发现,已经开发了一些基于crispr技术的基因编辑技术,例如crispr-cas9介导的基因编辑。
已发现的一些小分子化合物可以调节crispr-cas9介导的基因编辑过程。yu等人发现,l755507和brefeldina这两个小分子可以促进crispr-cas9介导的同源定向修复。chu和maruyama等人发现了连接酶iv的抑制剂scr7可以提高crispr-cas9介导的基因编辑效率。
基于crispr-cpf1核酸酶的基因编辑技术是另一种定向基因编辑技术,扩大了基因编辑的范围和具有更高的精度,然而crispr-cpf1的基因编辑效率还难以令人满意。此外,能够促进crispr-cpf1基因编辑的化学小分子还没有报道过。
因此,本领域迫切需要开发新的能够有效提高基因编辑效率的化合物。
技术实现要素:
本发明的目的是提供一种能够有效提高基因编辑效率的化合物及其应用。
在本发明的第一方面,提供了一种式a所示的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物的用途,用于制备促进基因编辑的促进剂或制剂;
式中,
各r1独立地选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基;
n为0、1、2、3、或4;
各r2独立地选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基、-(l2)q-取代或未取代的c1-c6烷基;
m为0、1、2、3、或4;
r3选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基;
r4选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基;
r5选自下组:h、卤素、-(l1)p-取代或未取代的c1-c6烷基、-(l1)p-n(ra)(rb)、-(l1)p取代或未取代的c2-c6烯基、-(l1)p-取代或未取代的c3-c6环烷基;
r6选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基、-(l2)q-取代或未取代的c1-c6烷基;
r7选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、取代或未取代的c2-c6烯基、取代或未取代的c3-c6环烷基;
其中,p为0、1、2、或3;而各l1独立地选自下组:-ch2-、-nh-、-s-、-o-、或其组合;
q为0、1、2、或3;而各l2独立地选自下组:-so2-、-ch2-、-nh-、-s-、-o-、或其组合;
ra和rb各自独立地选自下组:h、取代或未取代的c1-c3烷基、取代或未取代的c3-c6环烷基、取代或未取代的c2-c5环杂烷基,其中所述的环杂烷基为5-7元且含有1-3个选自n、o、和s的杂原子;
在另一优选例中,各r1独立地选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb);
在另一优选例中,n为0、1或2;
在另一优选例中,各r2独立地选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、-(l2)q-取代或未取代的c1-c3烷基;
在另一优选例中,m为0、1、或2;
在另一优选例中,r3选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb);
在另一优选例中,r4选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)。
在另一优选例中,r5选自下组:h、卤素、-(l1)p-取代或未取代的c1-c6烷基、-(l1)p-n(ra)(rb);和/或
r6选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb)、-(l2)q-取代或未取代的c1-c6烷基;
在另一优选例中,r7选自下组:h、卤素、取代或未取代的c1-c6烷基、-n(ra)(rb);
在另一优选例中,p为0、1、或2;
在另一优选例中,各l1独立地选自下组:-ch2-、-nh-、-o-、或其组合;
在另一优选例中,q为0、1、或2;
在另一优选例中,各l2独立地选自下组:-so2-、-nh-、-o-、或其组合;
在另一优选例中,ra和rb各自独立地选自下组:h、取代或未取代的c1-c3烷基;
在另一优选例中,各r1独立地选自下组:h、卤素、取代或未取代的c1-c3烷基;
在另一优选例中,n为0或1;
在另一优选例中,各r2独立地选自下组:h、卤素、取代或未取代的c1-c3烷基、-n(ra)(rb)、-(l2)q-取代或未取代的c1-c3烷基;
在另一优选例中,m为0或1;
在另一优选例中,r3选自下组:h、卤素、取代或未取代的c1-c3烷基、-n(ra)(rb);
在另一优选例中,r4选自下组:h、卤素、取代或未取代的c1-c3烷基、-n(ra)(rb);
在另一优选例中,r5选自下组:-(l1)p-取代或未取代的c1-c3烷基、-(l1)p-n(ra)(rb);
在另一优选例中,r6选自下组:取代或未取代的c1-c6烷基、-n(ra)(rb)、-(l2)q-取代或未取代的c1-c6烷基;
在另一优选例中,r7选自下组:h、卤素、取代或未取代的c1-c3烷基、-n(ra)(rb);
在另一优选例中,各l1独立地选自下组:-ch2-、-nh-、或其组合;
在另一优选例中,各l2独立地为:-so2-;
在另一优选例中,ra和rb各自独立地选自下组:h、未取代的c1-c3烷基;
在另一优选例中,n为0;
在另一优选例中,m为0;
在另一优选例中,r3为h;
在另一优选例中,r4为h;
在另一优选例中,r5为-ch2-nh-ch3;
在另一优选例中,r6为-so2-(c1-c3烷基),更佳地为-so2-c3h7;
在另一优选例中,r7为-n(ra)(rb),更佳地为-nh2;
在另一优选例中,所述的化合物为
在另一优选例中,所述的基因编辑包括基于crispr的基因编辑。
在另一优选例中,所述的基因编辑包括基于crispr-cpf1的基因编辑、基于crispr-cas9的基因编辑;
在另一优选例中,所述的基因编辑包括体内基因编辑、体外基因编辑、或其组合;
在另一优选例中,所述的基因编辑针对的样品选自下组:细胞、组织、器官、或其组合;
在另一优选例中,所述的样品来自动物、植物、微生物(包括细菌、病毒);
在另一优选例中,所述的样品来自人和非人哺乳动物;
在另一优选例中,所述的细胞包括原代细胞和传代的细胞;
在另一优选例中,所述的细胞包括体细胞、生殖细胞、干细胞;
在另一优选例中,所述的干细胞包括:全能干细胞、多能干细胞、和专能干细胞;
在另一优选例中,所述的干细胞为诱导的多能干细胞(hipsc);
在另一优选例中,所述的细胞包括:胚胎干细胞、脂肪干细胞、造血干细胞、免疫细胞(如t细胞、nk细胞);
在另一优选例中,所述的制剂包括药物组合物。
在本发明的第二方面,提供了一种体外促进细胞内基因编辑的方法,所述方法包括:
(a)在基因编辑促进剂存在下,对细胞进行基因编辑,从而促进所述细胞内的基因编辑,
其中,所述的基因编辑促进剂是式a所示的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物;(其中式a如权利要求1中所定义);
在另一优选例中,在步骤(a)中,在进行基因编辑之前、之中和/或之后,将所述基因编辑促进剂与所述进行基因编辑的细胞进行接触;
在另一优选例中,所述的体外的基因编辑在一体外反应体系中进行;
在另一优选例中,所述的体外反应体系中,所述基因编辑促进剂的浓度为0-2μm;
在另一优选例中,在步骤(a)中,包括:
(a1)向所述细胞中导入编码cpf1核酸酶的核苷酸序列,其中,所述cpf1核酸酶能够在靶dna中产生双链断裂,从而诱导靶dna的基因编辑;和
(a2)在所述基因编辑促进剂存在下,培养所述细胞;
在另一优选例中,所述方法为非诊断性和非治疗性的。
在本发明的第三方面,一种试剂产品(或试剂组合),包括:
(i)第一试剂,所述的第一试剂为基因编辑促进剂,所述的基因编辑促进剂是式a所示的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物;(其中式a如权利要求1中所定义);和
(ii)第二试剂,所述的第二试剂是进行crispr基因编辑的试剂;
在另一优选例中,所述的第二试剂包括选自下组的一种或多种试剂:
(c1)cpf1核酸酶、cpf1核酸酶的编码序列、或表达cpf1核酸酶的载体、或组合;
(c2)cas9核酸酶、cas9核酸酶的编码序列、或表达cas9核酸酶的载体、或组合;
(c3)crrna、用于产生所述crrna的载体;
(c4)用于同源定向修复的模板:单链核苷酸序列或质粒载体。
在本发明的第四方面,提供了一种用于制备进行基因编辑的试剂盒;
在另一优选例中,所述的试剂盒还含有说明书;
在另一优选例中,所述的说明书记载了本发明的促进基因编辑的方法;
在另一优选例中,所述的基因编辑是针对体细胞、干细胞的基因编辑;
在另一优选例中,所述基因编辑是基于crispr-cpf1的基因编辑;
在另一优选例中,所述细胞选自下组:胚胎干细胞、诱导多能干细胞、人胚肾293t细胞;
在另一优选例中,所述的基因编辑针对致病基因、肿瘤相关基因(如致癌基因)、免疫相关基因(如与自身免疫相关的基因)、视觉相关基因;
在另一优选例中,所述基因选自下组:oct4、albumin、alkbh1或其组合物。
在本发明的第五方面,提供了一种试剂盒,包括:
(i)第一容器,以及位于所述第一容器内的第一试剂,所述的第一试剂为基因编辑促进剂,所述的基因编辑促进剂是式a所示的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物;(其中式a如权利要求1中所定义);和
(ii)第二容器,以及位于所述第二容器内的第二试剂,所述的第二试剂是进行crispr基因编辑的试剂。
在本发明的第六方面,提供了一种促进基因编辑的方法,包括步骤:给需要的对象施用一基因编辑促进剂和进行基因编辑的基因编辑试剂,所述的基因编辑促进剂是式a所示的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物;(其中式a如权利要求1中所定义);
在另一优选例中,所述的对象包括人和非人哺乳动物;
在另一优选例中,所述的基因编辑试剂包括基于crispr-cpf1的基因编辑试剂。
在另一优选例中,在施用所述基因编辑促进剂之前、之中和/或之后,给所述对象施用进行基因编辑的基因编辑试剂。
应理解,在本发明范围内,本发明的上述各技术特征和在下文(如实施例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技术方案。限于篇幅,在此不再一一累述。
附图说明
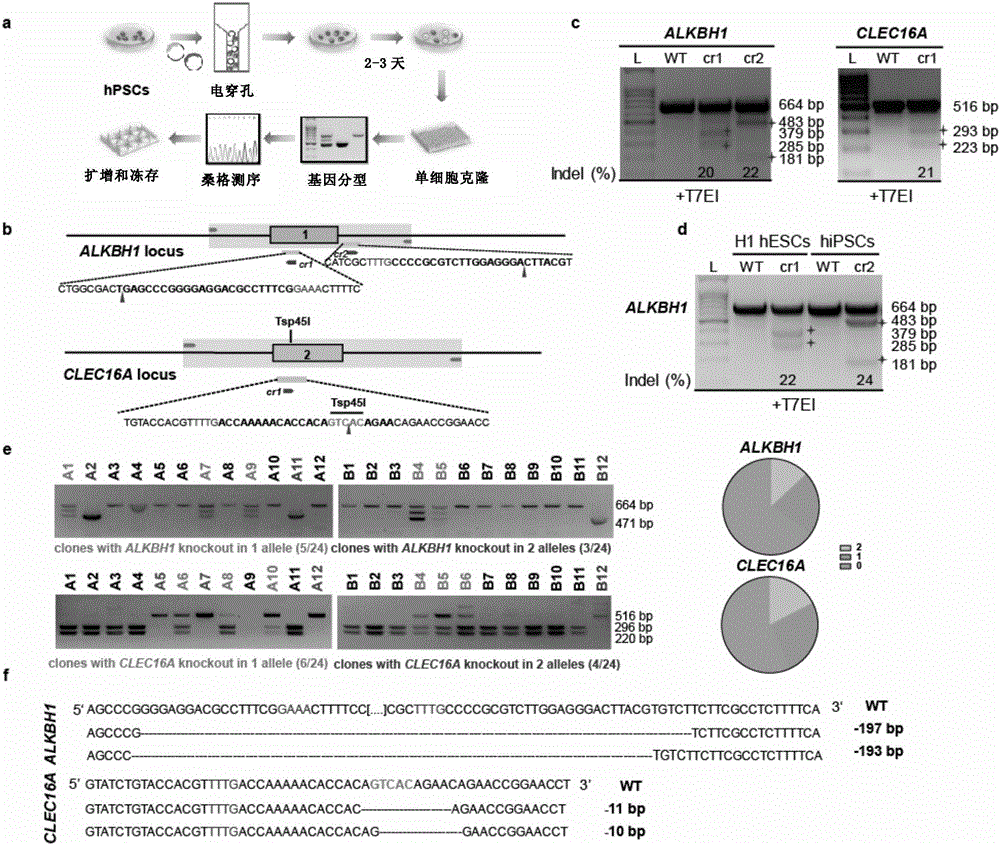
图1显示了用crispr-cpf1有效地产生了基因敲除的hpsc细胞系。
图2显示了能够显著促进crispr-cpf1介导的人多能干细胞中的基因敲入的小分子的鉴定。
图3显示了能够显著促进crispr-cpf1介导的hpsc基因敲入细胞系的小分子的建立。
图4显示了crispr-cpf1的工作模型
图5显示了对crispr-cpf1产生基因敲除hpsc细胞系的鉴定模型
图6、7显示了对crispr-cpf1的潜在脱靶位点分析
图8显示了候选小分子在oct4位点能够显著促进crispr-cpf1或crispr-cas9介导的hpsc基因敲入
图9显示了对候选小分子的毒性检测
图10显示了候选小分子在albumin位点能够显著促进crispr-cpf1或crispr-cas9介导的hpsc基因敲入
图11显示了候选小分子能够显著促进crispr-cpf1介导的hpsc双基因敲入
具体实施方式
本发明人经过广泛而深入的研究,首次意外地发现了一类结构如式a所示化合物可以显著地提高crispr的基因编辑效率,尤其是可显著促进crispr-cpf1介导的基因敲入的效率。实验表明,所述的式a化合物(如ve-822)可显著促进crispr-cpf1介导的基因编辑效率,例如在人多能干细胞进行高效的基因编辑从而产生基因敲入细胞系。在此基础上,发明人完成了本发明。
术语
术语“c1-c8亚烷基”指具有1-8个碳原子的直链或支链亚烷基,例如亚甲基、亚乙基、亚丙基、亚丁基、或类似基团。
术语“c1-c6烷基”指具有1-6个碳原子的直链或支链烷基,例如甲基、乙基、丙基、异丙基、丁基、异丁基、或类似基团。
术语“c3-c6环烷基”指具有3-6个碳原子的环烷基,例如环丙基、环丁基、环戊基、或类似基团。
术语“c1-c3亚烷基”指具有1-3个碳原子的直链或支链亚烷基,例如亚甲基、亚乙基、亚丙基、或类似基团。
术语“c5-c7环烷基”指具有5-7个碳原子的环烷基,例如环戊基、环己基、或类似基团。
术语“卤素”指f、cl、br和i。
基因编辑促进剂
如本文所用,“本发明化合物”、“式a化合物”、“本发明的基因编辑促进剂”可互换使用,指式a所示结构的化合物、或其药学上可接受的盐、或其光学异构体或其外消旋体、或其溶剂化物。应理解,该术语还包括上述组分的混合物。
式中,各基团的定义如上所述。
本发明化合物对基因编辑的效率有极为显著的促进作用
在本发明中,还包括式a化合物的药学上可接受的盐。术语“药学上可接受的盐”指本发明化合物与酸或碱所形成的适合用作药物的盐。药学上可接受的盐包括无机盐和有机盐。一类优选的盐是本发明化合物与酸形成的盐。适合形成盐的酸包括但并不限于:盐酸、氢溴酸、氢氟酸、硫酸、硝酸、磷酸等无机酸,甲酸、乙酸、丙酸、草酸、丙二酸、琥珀酸、富马酸、马来酸、乳酸、苹果酸、酒石酸、柠檬酸、苦味酸、甲磺酸、苯甲磺酸,苯磺酸等有机酸;以及天冬氨酸、谷氨酸等酸性氨基酸。
本发明的式a化合物可采用现有技术中本领域技术人员熟知的方法进行制备,对各个步骤的反应参数没有特别限制。
如本文所用,在式a化合物中,如果存在手性碳原子,则手性碳原子可以为r构型,也可以为s构型,或二者的混合物。
基因编辑
本发明化合物可显著提高基因编辑的效率。
在本发明中,代表性的基因编辑包括(但并不限于):基于crispr的基因编辑。典型地,基于crispr的基因编辑包括基于crispr-cpf1的基因编辑、基于crispr-cas的基因编辑。其中,所述的cas包括cas9等。
一种优选的基因编辑是crispr-cpf1基因编辑。crispr-cpf1能特异性地识别富含胸腺嘧啶核苷酸(t)的pam序列(tttn),扩大了由rna介导的基因编辑的范围。crispr-cpf1产生5nt的粘性末端,可启动不同的dna修复过程。crispr-cpf1的crrna长度比cas9的crrna短得多,体外合成更容易而且更适合多基因编辑过程。crispr-cpf1的脱靶率也较低。
用途
本发明的式a化合物和基因编辑试剂(如crispr-cpf1试剂)的组合,可显著提高基因编辑的效率,因而在治疗应用等不同领域具有革命性的潜力。
本发明的上述式a化合物可用于提高crispr介导的基因编辑的效率,进而可用于预防或治疗与致病基因相关的疾病。
在一实施例中,本发明提供了一种体外非治疗性的促进crispr介导的基因编辑的小分子筛选方法,包括:crispr介导的基因敲入和药物筛选系统。
本发明还提供了一种利用小分子促进crispr介导的基因编辑方法,该方法可以是治疗性的或非治疗性的。通常,该方法包括步骤:给需要的对象施用本发明的式a化合物。
优选地,所述对象包括人和非人哺乳动物(啮齿动物、兔、猴、家畜、狗、猫等)。
组合物和施用方法
本发明提供了一种用于促进crispr介导的基因编辑效率的组合物。所述的组合物包括(但并不限于):药物组合物、科研用试剂组合物等。
在本发明中,所述的组合物可直接用于促进基因编辑,例如,单基因敲入、双基因敲入、点突变等。
本发明还提供了一种药物组合物,它含有安全有效量的本发明化合物以及药学上可接受的载体或赋形剂。这类载体包括(但并不限于):盐水、缓冲液、葡萄糖、水、甘油、乙醇、粉剂、及其组合。药物制剂应与给药方式相匹配。
以药物组合物为例,本发明的组合物可以被制成针剂形式,例如用生理盐水或含有葡萄糖和其他辅剂的水溶液通过常规方法进行制备。诸如片剂和胶囊之类的药物组合物,可通过常规方法进行制备。药物组合物如针剂、溶液、片剂和胶囊宜在无菌条件下制造。本发明的药物组合也可以被制成粉剂用于雾化吸入。
对于本发明的药物组合物,可通过常规的方式施用于所需的对象(如人和非人哺乳动物)。代表性的施用方式包括(但并不限于):口服、注射、局部施用等。
本发明的主要优点包括:
(a)首次提供了一种可以显著地促进基因编辑的小分子化合物,该化合物对于crispr-cpf1/cas9介导的hpsc基因敲入编辑尤其有效。
(b)本发明基于式a化合物和crispr-cpf1的组合,为精准基因编辑提供了一种简单而又高效的策略。
(c)基于本发明的多能干细胞的高通量筛选方法,建立了一种无偏向性的药物筛选系统。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件,例如sambrook等人,分子克隆:实验室手册(newyork:coldspringharborlaboratorypress,1989)中所述的条件,或按照制造厂商所建议的条件。除非另外说明,否则百分比和份数是重量百分比和重量份数。
通用方法
质粒构建
对于pcpfcr载体,将bbsi酶识别序列和dr(正向重复)序列设计在u6启动子的上游引物中;使用pucm-t载体克隆试剂盒(sangonbiotech公司)将u6启动子区域通过pcr扩增然后克隆到t载体。构建pcpfcr-crrna载体来表达cpf1的靶向序列(crrna)时,首先根据crrna序列合成24bp的寡核苷酸,退火形成dna双链片段,之后克隆到bbsi酶切的pcpfcr载体上。
关于构建oct4-tdtomato供体质粒,采用pcr的方法将oct4-2a-morange供体质粒(addgene,plasmid#66986)除去morange序列作为骨架,之后pcr扩增出tdtomato序列,用gibsonassembly试剂盒(newenglandbiolabs)将tdtomato序列连接到骨架上。所有的载体通过sanger测序核对。
细胞培养
hpscs按常规方法培养。h1胚胎干细胞系,mel1胚胎干细胞系和hipscs用hpsc培养基培养,其组成为:dmem/f12(lifetechnologies),20%去血清代替物ksr(lifetechnologies),1x非必需氨基酸(lifetechnologies),100x青霉素/链霉素(lifetechnologies),0.055mm巯基乙醇(sigma),和10ng/mlbfgf(peprotech)。
每隔3-6天,用accutase(lifetechnologies)消化hpsc细胞,并以1:3至1:6的比例传代。每次传代或解冻细胞时都要在hpsc培养基中加入0.5μmthiazovivin帮助干细胞贴壁存活。
电转
用zymopure质粒中提试剂盒提取质粒后准备电转。首先取81.82μlsolutionⅰ与18,18μlsupplementⅰ(humanstemcell
对于基因敲入实验,质粒由3μgpcdna3.1-hlbcpf1(addgene公司,质粒编号#69988),3μgpcpfcr-crrna和4μgofoct4-tdtomatodonor或oct4-2a-egfp-pgk-purodonor(addgene公司,质粒编号#31938)组成。用accutase把hpsc细胞消化成单个细胞,取1×106个细胞用电转液重悬,并用amaxa电转仪(lonza)电转。电转之后,把细胞种到六孔板的一个孔培养2-3天。之后,用流式或t7eⅰ分析敲入效率。
对于建立克隆实验,接种500-2000个细胞到10cm培养皿并培养4-7天。挑出克隆用于扩增和储存。2周之后,用免疫染色、流式、测序等方法鉴定表达tdtomato的细胞系。
转染
用dmem(lifetechnologies)加上10%胎牛血清,100×青霉素/链霉素培养基培养人胚肾293t细胞。当293t细胞密度达到70%-80%时,用lipofectaminetm3000转染试剂(invitrogen)混合质粒进行转染。质粒混合物由1μgpy016(pcdna3.1-hlbcpf1)和1μgofpcpfcr-crrna组成。转染2-3天后,用t7eⅰ实验分析。
pflp和t7eⅰ分析
转染或电转后2-3天收集细胞,用快速基因组dna小提试剂盒(zymo)提取基因组。用taq聚合酶(vazyme)扩增基因组上带有crispr作用靶点的片段。
对于rflp实验,取2μl扩增产物用限制酶消化,并用2%琼脂糖凝胶跑电泳分析。对于点突变,100μl的pcr产物用dnaclean&concentrator-5(zymoresearch)试剂盒纯化回收,用ncoi酶(newenglandbiolabs)酶切之后用2%琼脂糖凝胶跑电泳分析。突变率用公式100×(b+c)/(a+b+c)计算,其中,a代表未被ncoi酶切的产物条带强度,b和c分别代表酶切后的产物条带强度。
对于t7eⅰ实验,取10μl扩增产物与nebbuffer2(newenglandbiolabs)混合配成总体积16μl的体系。混合体系在95℃孵育5分钟,再以2℃每秒的速度降至85℃,之后以0.1℃每秒的速度缓慢降至25℃,最终4℃孵育。在16μl反应产物中加入t7eⅰ酶(newenglandbiolabs)配成总体积20μl的体系在37℃孵育30分钟,之后用2.5%的琼脂糖凝胶跑电泳分析。胶图由js-2000gelimager(peiqingscience&techenology)拍摄并由imagej软件分析。
突变率用公式100×(1–(1–(b+c)/(a+b+c))1/2)计算,其中,a代表未被t7eⅰ酶切的产物条带强度,b和c分别代表酶切后的产物条带强度。
流式细胞术
将转染了cpf1、oct4-crrna和oct4-tdtomato载体的hpsc接种在六孔板中的一个孔中,用hpsc培养基培养3-4天。把分化和未分化的细胞用accutase消化3-5分钟成单个细胞。之后用0.3-1ml1×磷酸盐缓冲液重悬。最后用beckman流式细胞仪得到数据,并分析结果。
hpsc分化
将大约200个未分化的oct4-tdtomatohpsc细胞接种在六孔板的一个孔,并用hpsc培养基培养4-7天。之后换成分化培养基(dmem/f12,20%血清替代物,1×neaa,100×青霉素/链霉素(lifetechenologies),0.055mm2-巯基乙醇(sigma),0.1μmldn225,10μme616452)培养3天。第四或第五天后,细胞可以用流式和免疫染色分析。
免疫染色
室温下使用4%多聚甲醛固定细胞10-15分钟,随后1xpbst(1xpbs+0.3%tritonx-100(vetec))洗三次,每次五分钟。接下来,细胞在封闭液(1xpbst+5%bsa(胎牛血清))中室温孵育0.5-1小时后,一抗孵育过夜(4℃)。随后,室温下用pbst清洗三次,每次15分钟,然后加入相应的二抗,孵育1小时。最后,使用hoechst(1:5000)对细胞核进行染色。
细胞凋亡分析
细胞凋亡由细胞凋亡试剂盒进行分析。首先用dmso,azd-7762(1μm)和ve-822(1μm)处理人多能干细胞。收集5x105个细胞并用冰pbs洗两次。细胞在4℃离心5分钟,然后用50μl1xbindingbuffer重悬,加入2.5μlannexinv(av)-fitc和2.5μlpi染色液室温孵育10-15分钟。随后加入用250μl的1xbindingbuffer到混合液中。最后使用流式细胞仪检测细胞凋亡。
脱靶分析
cas-offinder可用来检测潜在的脱靶位点。设定的错配值为小于等于6。找到潜在的脱靶位点后,设计引物利用pcr在细胞基因组上扩增脱靶位点片段,之后通过sanger测序法进行测序鉴定。
统计学分析
双尾t检验得到p值,并且所有的统计数值均以mean±s.e表示。
实施例1.
在人多能干细胞中使用crispr-cpf1高效地构建基因敲除细胞系
为了在人多能干细胞中使用crispr-cpf1基因编辑系统,构建了一个由u6启动子启动的crrna表达质粒(图4a和4b)。
挑选了几个感兴趣的基因,包括alkbh1和clec16a。alkbh1是trna去甲基化酶,clec16a在糖尿病的发生过程中扮演重要角色。
为了构建基因特异性的crrna质粒,设计了一组特异性靶向这些基因的crrnas(图1b和表1)。
【表1】
构建表达crrna质粒的核酸序列
构建质粒的pcr引物
用于基因型鉴定和测序的pcr引物
t7ei实验的pcr引物
rflp实验的引物
qpcr的引物
脱靶率分析的pcr引物
ssodn模板序列
在293t细胞中,测试这些crrnas的基因组编辑能力。在t7e1(t7内切酶i)试验中,观察到有20-30%插入缺失比例(图4c)。
然后,研究了在hpscs中crisper/cpf1介导的基因组编辑的能力(图1a)。在t7e1试验中,观察到在hescs和hipscs中对alkbh1和clec16a进行基因敲除的效率也有20-30%(图1c)。
为了进一步建立敲除hpsc系,将转染的hpscs以低细胞密度传代,挑取克隆,并且通过pcr基因型鉴定进行了分析(图1a)。
对于alkbh1,20.8%克隆发生了单等位基因敲除,12.5%发生了双等位基因敲除(图1e)。
对于clec16a,25%克隆发生了单等位基因敲除,并且16.7%的克隆发生双等位基因敲除(图1e)。
桑格测序结果证实,在alkbh1和clec16a靶向位点上发生基因敲除后,基因组序列缺少了部分碱基,从而实现了基因敲除(图1f)。
此外,对于基于软件分析找到的一些潜在的脱靶位点,还通过pcr扩增以及桑格测序法进行分析,结果没有发现脱靶现象的发生(图6、7b)。
总而言之,上述实验结果清楚地证明了crispr-cpf1可以在hpscs中高效地进行基因敲除。
实施例2.
筛选在hpscs中可以显著促进crispr-cpf1介导的hdr的小分子
在本实施例中,为了检验crispr-cpf1进行基因插入的能力,采用电转的方法将三个质粒转入hpscs中:一个质粒表达cpf1,一个含有靶向oct4的特异性crrna,以及包含egfp报告基因和嘌呤霉素(puro)抗性的hdr模板质粒(图8a)。电转后,先用普通培养基培养hpscs两天的时间,之后向培养基中加入嘌呤霉素处理3-4天时间。观察到通过hdr方式进行基因组修复的效率相对较低并且需要被进一步提高。
此外,采用已知的可提高crispr-cas9介导的基因编辑效率的scr7化合物作为对照化合物。
结果:
实验表明,scr7(已知可促进crispr-cas9介导的基因敲入的小分子),在crispr-cpf1介导的基因敲入中没有显著的促进效果(图2e)。
这表明crispr-cpf1系统与crispr-cas9系统在实际应用中存在一些差异。因此,发现新的并且有效地促进hdr的小分子化合物是十分重要的。
实施例3
化学小分子筛选
本实施例中,对于上百种候选化合物,逐一进行筛选和测试。
在本实施例中,为了提高在hpscs中crispr-cpf1介导的基因编辑的效率,运用具有嘌呤霉素的oct4-egfp敲入筛选体系进行了大规模化合物筛选(图2a)。
具体地,采用电转的方法将三个质粒转入hpscs中:一个质粒表达cpf1,一个含有靶向oct4的特异性crrna,以及包含egfp报告基因和嘌呤霉素(puro)抗性的hdr模板质粒(图8a)。
对于每一种候选化合物,在电转操作之前,需在48孔板的每个孔中加入100μl细胞培养基以及0.2μl筛选的小分子(1:1000)。细胞电穿孔之后,1x106个细胞混匀在添加了thiazovivin小分子的4.8mlhpsc培养基中,接种在一个48孔板内。利用添加了ve-822小分子的培养基培养细胞,两天后停止加ve-822小分子,并在培养基中加入1μg/ml的嘌呤霉素继续培养3-5天。之后,对筛选出来的带有嘌呤霉素抗性的克隆进行计数,来检测小分子的效率。
结果:
总共筛选了激酶库中约600个小分子,其中小分子ve-822非常显著地提高hpsc克隆数量(图2b,2c,2e,2f,3b,3c,8d,10b,10e和11a),这提示化合物ve-822可显著提高基因编辑效率。
实施例4
基因编辑促进剂的效果验证
在本实施例,进一步进行实验验证ve-822的作用效果。试验方法同实施例3。
结果表明,ve-822在1μm下达到它的最大作用效果(图2d)。ve-822是atr激酶的抑制剂,可以促进基因敲入效率提高5.9倍。
此外,ve-822没有显著提高hpscs的细胞增殖速率,表明ve-822是不通过促进细胞增殖提高基因敲入效率(图9a)。
此外,ve-822没有展示出对hpscs的细胞毒性(图9c和9d)。用ve-822处理的hpscs依然高表达oct4和nanog这些特异的多能性基因(图9b)。
与之相反,在crispr-cpf1介导的hpscs中敲入的情况下,对照化合物scr7没有显著的促进作用,但是ve-822可以在hpscs中显著地促进cpf1介导的基因敲入(图2e和8d)。
在crispr-cas9系统中,scr7提高的效率低于2倍,而ve-822的作用效果更加显著(约4倍)。
此外,用sirnas敲低atr基因可以促进crispr-cpf1介导的基因敲入效率,说明ve-822确实通过靶向atr起作用(图2g)。上述结果成功地证实,ve-822能够显著地提高在hpscs中crispr-cpf1介导的基因敲入效率。
实施例5.
小分子显著地促进crispr-cpf1介导的hpscs基因敲入细胞系的产生
在本实施例中,进一步检测了crispr-cpf1对于在没有药物筛选情况下产生hpscs基因敲入细胞系的能力。
方法如下:将3个质粒电转进入hpscs中:一个质粒表达cpf1,一个包含特异性靶向oct4的crrna,和一个含有oct4-2a-tdtomato的供体质粒(图3a)。5-6天后,观察到tdtomato阳性细胞,通过facs(流式)分析检测基因敲入的效率。
结果显示:ve-822能够显著地提高tdtomato阳性细胞的百分比,这表明了ve-822可以促进crispr-cpf1在hpscs中的基因敲入(图3b,c)。
之后,挑取了几个tdtomato阳性克隆来进行hpsc细胞系的建立。pcr分析的结果显示这些细胞系中成功整合了tdtomato报告基因(图3f)。这些建成的hpsc细胞系共表达tdtomato和多能性基因oct4(图3d)。
将这些干细胞分化后,通过免疫染色和facs实验可以证明细胞丢失了多能基因oct4的表达,同时,细胞也不再表达tdtomato荧光报告基因(图3d,e)。因此,oct4-tdtomato可以在hpscs阶段和分化期间同步反映内源多能性基因oct4的表达情况。
之后,使用桑格法进行测序。测序结果提示,在这些oct4-tdtomato细胞系中,2a-tdtomato序列成功插入到基因组中,并且替换了oct4基因的终止密码子序列。
此外,对oct4和albumin两个基因位点进行双基因敲入实验证明了ve-822可以显著促进crispr-cpf1介导的双基因敲入的效率(图11a)。
对于利用crispr-cpf1介导产生的基因点突变实验(图3h),方法为向hpsc细胞中电转入表达cpf1的载体、alkbh1-crrna载体和单链核酸序列模板。结果显示候选小分子组合可以显著促进以单链核酸为模板构建基因点突变的效率(图3i和3j)。
上述试验结果显示,基于crispr-cpf1和小分子的组合,可以在hpscs中进行有效的基因插入。
讨论
本发明中,本发明通过试验证明,基于crispr-cpf1的基因编辑,可以在人多能干细胞系中进行高效的基因敲除和插入编辑。t7ei实验结果证明crispr-cpf1成功在alkbh1和clec16a两个基因的特定位点上进行了基因组切割。之后通过挑单克隆的方法进行基因型鉴定,结果显示这一基因敲除效率较高。
通过测序的结果也可以得出结论crispr-cpf1可以顺利构建基因敲除的细胞系。
对于基因插入实验,本发明人构建了oct4-tdtomato的载体,通过crispr-cpf1介导的基因编辑技术成功为oct4基因插入了红色荧光报告基因,并且通过流式细胞分析和免疫荧光染色实验可以检测到成功插入荧光报告基因的hpscs。
此外,crispr-cpf1的独特之处在于它的crrna长度更短、脱靶率较低,这些特性使它具有很高的应用价值。但是使用目前的基因编辑工具进行同源定向修复的效率较低,耗时较长。
鉴于crispr-cpf1与crispr-cas9的特异性,构建一个可行的化学筛选系统、鉴定到一些提高基因编辑效率的小分子化合物是十分有意义的。
值得一提的是,本发明人为人多能干细胞的高通量筛选建立了一种无偏向性的药物筛选系统,而这个独特的筛选系统又能使本发明人发现一些新颖又有趣的小分子化合物,从而促进crispr-cpf1介导的精准基因编辑过程。
本发明人使用oct4-egfp-puro载体在hpscs中进行基因插入,之后利用嘌呤霉素(puro)对细胞进行药物筛选,最终可获得带有puro抗性的单克隆,本发明人将单克隆数作为输出结果来比较对照组和各实验组的差异。本发明人总共筛选了接近600个化学小分子,最终发现了小分子化合物ve-822可以显著地促进crispr-cpf1介导的hpsc基因编辑过程。
加入小分子ve-822后基因敲入效率可以提高5.9倍,并且该小分子的功能显著优于先前已发现的一些化学小分子。重要的是,crispr-cpf1和小分子化合物的组合为精准的基因编辑提供了一种简单而又高效的策略,也可以进一步发展并应用于动物体内和人类生殖细胞内的基因编辑。毫无疑问,这些进展将为基因组工程提供新的方法和工具,加快人类疾病治疗新方法的发展。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
<110>浙江大学
<120>促进基因编辑的化合物筛选和鉴定及其应用
<130>p2017-2499
<160>89
<170>patentinversion3.5
<210>1
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>1
tttccccctgtctccgtcaccactctgg28
<210>2
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>2
agatcccctgtctctgtcaccactctgg28
<210>3
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>3
aaaaccagagtggtgacagagacagggg28
<210>4
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>4
ccctgtctccgtcaccactctgg23
<210>5
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>5
caccgccctgtctccgtcaccactc25
<210>6
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>6
aaacgagtggtgacggagacagggc25
<210>7
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>7
tgagcccggggaggacgcctttcggaaa28
<210>8
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>8
agatcgaaaggcgtcctccccgggctca28
<210>9
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>9
aaaatgagcccggggaggacgcctttcg28
<210>10
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>10
tttgccccgcgtcttggagggacttacg28
<210>11
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>11
agatccccgcgtcttggagggacttacg28
<210>12
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>12
aaaacgtaagtccctccaagacgcgggg28
<210>13
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>13
cccactaaccgtcacacttagcaataaa28
<210>14
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>14
agatttgctaagtgtgacggttagtggg28
<210>15
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>15
aaaacccactaaccgtcacacttagcaa28
<210>16
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>16
tttcacctccagaccgattgttattgaa28
<210>17
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>17
agatacctccagaccgattgttattgaa28
<210>18
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>18
aaaattcaataacaatcggtctggaggt28
<210>19
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>19
tttcggaaacttttccgcttctaccgtc28
<210>20
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>20
agatggaaacttttccgcttctaccgtc28
<210>21
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>21
aaaagacggtagaagcggaaaagtttcc28
<210>22
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>22
tttgaccaaaaacaccacagtcacagaa28
<210>23
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>23
agataccaaaaacaccacagtcacagaa28
<210>24
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>24
aaaattctgtgactgtggtgtttttggt28
<210>25
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>25
gccttaggcttataacatcacatttaaa28
<210>26
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>26
agataatgtgatgttataagcctaaggc28
<210>27
<211>28
<212>dna
<213>人工序列(artificialsequence)
<400>27
aaaagccttaggcttataacatcacatt28
<210>28
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>28
ccttaggcttataacatcacatt23
<210>29
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>29
caccgaatgtgatgttataagccta25
<210>30
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>30
aaactaggcttataacatcacattc25
<210>31
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>31
gagggcctatttcccatgattcct24
<210>32
<211>67
<212>dna
<213>人工序列(artificialsequence)
<400>32
aaaaaaaggtcttctcgaagacccatctacacttagtagaaattcggtgtttcgtccttt60
ccacaag67
<210>33
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>33
ggtgcctgcccttctaggaa20
<210>34
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>34
aggaccggggttttcttcca20
<210>35
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>35
tggaagaaaaccccggtcctatggtgagcaagggcgagga40
<210>36
<211>40
<212>dna
<213>人工序列(artificialsequence)
<400>36
ttcctagaagggcaggcaccttacttgtacagctcgtcca40
<210>37
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>37
atcttcaggaggtaagggtg20
<210>38
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>38
cgatctcgaactcgtggc18
<210>39
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>39
gcttccatcactggctcgta20
<210>40
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>40
cgacatcccctgcttgtttc20
<210>41
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>41
aaattccactactcccacatctcc24
<210>42
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>42
cggattcccaggctcttttg20
<210>43
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>43
acccttcaaagcattgtctgc21
<210>44
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>44
aacttccccatttttggcttg21
<210>45
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>45
atgtccgtgagcttccgtc19
<210>46
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>46
cgatctcgaactcgtggc18
<210>47
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>47
gatcaagcagcgactatgca20
<210>48
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>48
tcacttgggtatgagcattg20
<210>49
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>49
aaattccactactcccacatctcc24
<210>50
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>50
cggattcccaggctcttttg20
<210>51
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>51
acccttcaaagcattgtctgc21
<210>52
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>52
aacttccccatttttggcttg21
<210>53
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>53
aaattccactactcccacatctcc24
<210>54
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>54
cggattcccaggctcttttg20
<210>55
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>55
acccttcaaagcattgtctgc21
<210>56
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>56
aacttccccatttttggcttg21
<210>57
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>57
atatgaagcgtgccgtagact21
<210>58
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>58
tgcctatgtctggctctattctg23
<210>59
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>59
ggccaaaggcagttgtattga21
<210>60
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>60
gtgagtaccccaaaaatagcagg23
<210>61
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>61
tgcaccaccaactgcttagc20
<210>62
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>62
ggcatggactgtggtcatgag21
<210>63
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>63
ggcgtgtcacaagaaggg18
<210>64
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>64
ggaggcggaggatgaagt18
<210>65
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>65
tgtgggtgacagaatggt18
<210>66
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>66
aatgggtcctgcaagtaa18
<210>67
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>67
cacgccaaagccttcata18
<210>68
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>68
gaacggatgggacggtag18
<210>69
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>69
ttaggcagacctctgtga18
<210>70
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>70
tgggcaacaagagcaaag18
<210>71
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>71
attgaaacgccacagatt18
<210>72
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>72
ctagcccttagccttgac18
<210>73
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>73
cgtatttcagcgtttgttc19
<210>74
<211>19
<212>dna
<213>人工序列(artificialsequence)
<400>74
gctaagcgagatttcctaa19
<210>75
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>75
ccctaaatggagcgagaa18
<210>76
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>76
gtagccttgggaaacagc18
<210>77
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>77
cataaggtcgggtcaggg18
<210>78
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>78
aagaaagcaccaaggcac18
<210>79
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>79
acagagtatcgcaggcacaa20
<210>80
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>80
cctttccacccacccttgat20
<210>81
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>81
gtgactgtttccctttccctct22
<210>82
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>82
tgtcatggggaattgctggt20
<210>83
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>83
gaatttaggtgaagggta18
<210>84
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>84
acaagtcgagctgataca18
<210>85
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>85
gttgtcaggaaatgcagagcc21
<210>86
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>86
agtcctacgtccgagagtga20
<210>87
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>87
acatattcaacaggtgccagc21
<210>88
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>88
aagtctcatgggttttggggg21
<210>89
<211>120
<212>dna
<213>智人(homosapiens)
<400>89
gcgagatggggaagatggcagcggccgtgggctctgtggcgactctggcgactgagccca60
tggaggacgcctttcggaaacttttccgcttctaccgtcagagccggcccgggaccgcag120
- 还没有人留言评论。精彩留言会获得点赞!
- 能够与配体偶联的单甲基缬氨酸化合物的制造方法与工艺
- 烟梗纤维微波降解制备烟用料液方法及应用与流程
- 利用特异性挥发物筛选昆虫相关嗅觉蛋白的方法与制造工艺
- 小分子共价抑制剂的计算机筛选方法及其在筛选S-腺苷甲硫氨酸脱羧酶的共价抑制剂的应用与制造工艺
- 一种合成抗原的分子量检测方法及应用与制造工艺
- 一种用于MALDI‑TOF‑MS分析小分子的氧化锌沸石复合材料及其制备方法与制造工艺
- 用于原位收集根系挥发物的试验装置及方法与制造工艺
- 一种核壳型纳米香精胶囊的制备方法与制造工艺
- 小分子共价抑制剂在制备抑制S-腺苷甲硫氨酸脱羧酶的药物上的应用及其筛选方法与制造工艺
- Linderolide H在制备治疗鼻炎药物中的应用的制造方法与工艺