大豆百粒重增效基因及其分子标记和应用的制作方法
本发明属于育种领域,具体涉及调控大豆籽粒百粒重的基因gmslk的挖掘及其应用。
背景技术:
大豆是具有重要经济意义的作物,为人类和动物提供食用油和蛋白质等。现随着人类生活质量的不断提高,对大豆的需求量也越来越大,所以提高大豆的产量一直以来都是国内外大豆育种的主要目标。然而,百粒重作为大豆产量的重要决定因素,也被育种家列为主要的育种性状之一。但是大豆百粒重性状是由多基因控制的数量性状,易受环境的影响,这给研究者带来了很大的阻碍。
针对大豆百粒重性状,国内外许多学者利用不同的初级作图群体和基于不同标记图谱定位了大量的qtl(quantitativetraitloci)。但初级作图群体会使得qtl定位区间过大,大量基因位点同时分离,造成遗传背景效应,增加了主效qtl精细定位的难度。利用次级作图群体,如染色体片段代换系(chromosomesegmentsubstitutionline,cssl)的应用,为解决这一难题找到了理想途径。理想状态下,染色体片段代换系个体基因组带有供体亲本染色体的少量片段,其遗传背景高度一致,无疑是进行qtl定位的理想材料。理论上讲,染色体片段代换系的性状值可与轮回亲本的性状值比较,两个株系之间性状差异都因为导入株系的渗入片段存在差异qtl而造成的,因此,利用这一同源的染色体片段代换系进行qtl分析可以提高定位的精确度。
为了更好地实现qtl精细定位及分子辅助育种,可根据染色体片段代换系的定位结果,对检测到的具有重要目标性状的代换系有目的地进行标记辅助回交,构建次级分离群体,将这些重要位点分散在不同的个体中,在高度一致的遗传背景下逐一评价每个候选位点的效应,可实现候选基因的定位和分子辅助育种及基因功能研究的有效手段。
目前虽然定位了大量的关于大豆百粒重的qtl,但是确定直接调控大豆百粒重的基因确很少,因此挖掘调控大豆百粒重的候选基因及基因的功能研究,可为大豆百粒重分子辅助育种提供有用的信息和重要的育种材料。
技术实现要素:
本发明所要解决的技术问题为:提供一个调控大豆百粒重的关键候选基因gmslk及应用,为大豆百粒重分子辅助育种提供有用的信息和重要的育种材料。
本发明的技术方案为:
大豆百粒重增效基因,其碱基序列如seqidno.2所示。
大豆百粒重增效基因编码的蛋白,其氨基酸序列如seqidno.4所示。
编码seqidno.4所示的氨基酸序列的多核苷酸片段。
重组载体,含有上述所述的多核苷酸片段;优选地,所述多核苷酸片段的序列如seqidno.2所示。
扩增上述所述的大豆百粒重增效基因的特异引物,该特异引物的上游引物序列为seqidno.5,特异引物的下游引物序列为seqidno.6。
大豆百粒重增效基因的分子标记,该分子标记位于大豆染色体第16号染色体chr16:35923082-35923257bp的位置,该分子标记的引物如seqidno.7和seqidno.8所示。
鉴定大豆是否含有上述所述的大豆百粒重增效基因的方法,以待鉴定大豆的基因组dna为模板,以seqidno.7和seqidno.8所示的引物进行pcr扩增,若能得到175bp的扩增产物,则该大豆含有所述的大豆百粒重增效基因。
上述所述的大豆百粒重增效基因或蛋白或重组载体或特异引物或分子标记在大豆育种中的应用。
本发明发现了一个调控大豆百粒重基因gmslk,所述基因位于大豆染色体第16号染色体chr16:35920962-35928190bp,全基因组长7229bp,完整编码序列长2640bp,分析比较发现来自野生豆n24852的gmslk基因序列相比较栽培豆nn1138-2的gmslk基因序列在第一个外显子的谷氨酸串联重复区域多了一个谷氨酰胺。且来自野生豆的gmslk基因对大豆百粒重具有减效作用,来自栽培豆的gmslk基因对大豆百粒重具有增效作用。通过进一步定位发现一个分子标记indelslk位于gmslk基因在野生豆n24852与栽培豆nn1138-2的差异区间,因此可以通过这标记来判断不同大豆品种中的gmslk基因是增效基因还是减效基因,在大豆育种选材中具有重要的价值。
与现有技术相比,本发明具有以下有益效果:
本发明鉴定了一个位于大豆gm16号染色体上调控大豆百粒重的基因gmslk,该基因的过表达可以显著提高大豆百粒重性状。同时提供了一个鉴定该基因的标记indelslk。该标记为在大豆种子资源和杂交后代中鉴定其gmslk是增效基因还是减效基因提供了方便,为大豆育种选材提供了重要的判断价值。
附图说明
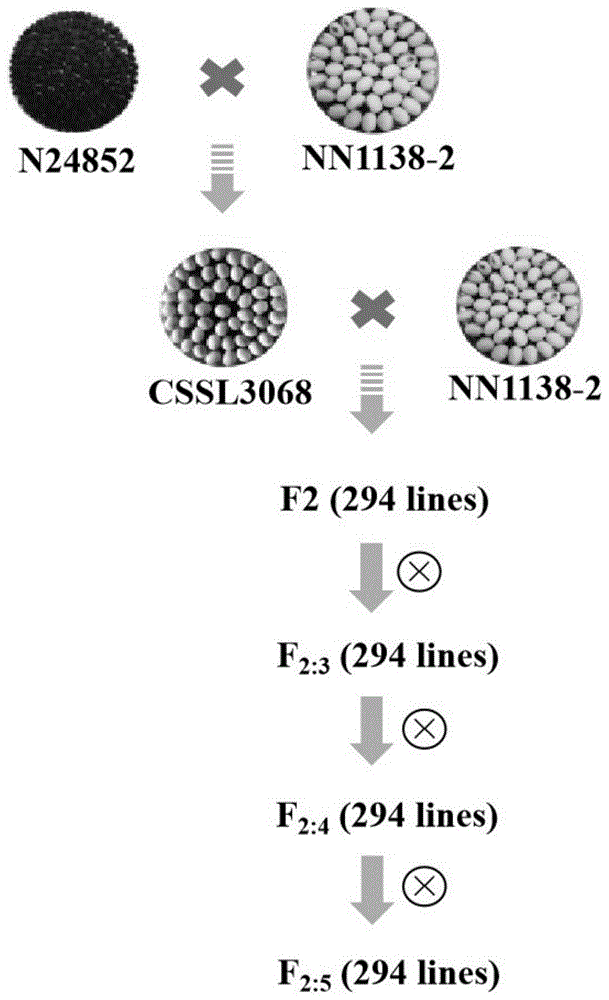
图1:群体构建示意图;以野生豆n24852为父本,栽培豆nn1138-2为轮回亲本,构建染色体片段代换系(王吴彬等人2013年构建),cssl3068为代换系中的一个家系。次级群体以cssl3068作为母本,nn1138-2作为父本,杂交、自交得到294个后代,单株种植并收获得到294个家系的f2群体,然后每个家系种植一行,自交,并按株行收获得到294个f2:3家系,同理得到f2:4和f2:5家系。
图2:亲本及定位群体百粒重表型;(a)为次级群体亲本nn1138-2和cssl3068的种子大小示意图;(b)为次级群体亲本nn1138-2和cssl3068的百粒重比较(**p<0.01);(c)定位群体f2:3于2015年种植江苏南京(2015nj),f2:4群体于2016年种植于江苏南京(2016nj)和f2:5群体于2017年分别种植江苏南京(2017nj)和安徽当涂(2017ah)的百粒重表型的次数分布图。
图3:精细定位过程;(a)为16号染色体的初级物理图谱;(b)为16号染色体的局部高密度物理图谱;(c)利用单标记分析法(icimapping2.0)结合高密度图谱分析次级群体f2:3、f2:4及f2:5在四个环境中的表型数据;(d)利用置换作图法分析11个重组个体的表型和基因型定位候选基因;(e)gmslk基因结构示意图。
图4:gmslk扩增条带图;gmslk-n24852代表gmslk来自野生豆n24852,gmslk-nn1138-2代表gmslk来自栽培豆nn1138-2,gmslk-cssl3068代表gmslk来自代换系cssl3068,marker为5000bp,红色箭头指示的条带为3000bp条带。
图5:比较亲本籽粒发育不同时期的籽粒大小和鲜重。(a)比较亲本nn1138-2和cssl3068在籽粒发育不同时期籽粒大小;(b)比较亲本nn1138-2和cssl3068在籽粒发育不同时期籽粒鲜重。每个样品统计分析3个生物学重复和3个技术重复(*p<0.05,**p<0.01)。
图6:gmslk在亲本nn1138-2和cssl3068籽粒发育不同时期的相对表达量。内参基因为ubi3基因,每个样品统计分析3个生物学重复和3个技术重复(*p<0.05,**p<0.01)。
图7:转基因拟南芥分析gmslk-nn1138-2(左边)和gmslk-cssl3068(右边)基因功能。(a)纯合转基因株系的相对表达量分析,内参基因为actin基因;(b)转基因植株和野生型的3周植株形态大小比较;(c)比较转基因植株和野生型植株的1000粒重,每个样品统计分析3个生物学重复和3个技术重复(*p<0.05,**p<0.001)。
具体实施方式
实施例1.大豆百粒重主效位点qsw16.1的获得
(1)、定位群体构建及种植
2013年王吴彬等人利用野生豆n24852和栽培豆nn1138-2构建了一套151个家系组成的染色体片段代换系群体,根据群体家系的百粒重表型信息发现其中一个家系cssl3068的百粒重为14.68g,相比较轮回亲本nn1138-2百粒重(18.7g)显著变小,根据群体的标记信息发现cssl3068含有一个百粒重的野生位点sat_224。2014年利用家系cssl3068与轮回亲本nn1138-2杂交构建次级群体,获得294个株系的f2代次级定位群体。2015年于江苏南京(2015nj)将f2次级群体种植294个株行,3个重复,然后每行单独收获,获得294个家系的f2:3群体,测定f2:3群体家系的百粒重表型。2016年于江苏南京(2016nj)将f2:3群体种植294个株行,3个重复,然后每行单独收获,获得294个家系的f2:4群体,测定f2:4群体家系的百粒重表型。2017年将f2:4群体294个株行分别种植于江苏南京(2017nj)和安徽当涂(2017ah),3个重复,然后每行单独收获,获得294个家系的f2:5群体,测定f2:5群体家系的百粒重表型(图1)。
(2)、亲本及定位群体百粒重表型的测定及分析
株行种植,株行收获,然后在30℃恒温烘箱至种子恒重,每个亲本及家系挑选发育良好、饱满的100粒种子称重。
首先鉴定分析了两个亲本之间的种子大小(图2a和b),结果发现亲本nn1138-2的百粒重在四个不同的环境下都显著大于亲本cssl3068的百粒重(如表1)。此外利用spss2.0统计分析了次级群体在四个环境下百粒重表型的频率分布图(图2c),结果显示都呈连续分布,且在2017ah的环境下f2:5群体百粒重平均值最大为16.94g,在2015nj环境下群体群体百粒重平均值最小为13.88g(如表1),这些结果都显示大豆百粒重表型为数量性状,为多基因控制。另外,定位群体在不同的环境下都具有较低的表型变异系数(cv)和较高的遗传率(h2),这说明了大豆百粒重表型主要变异都是由基因控制的。
表1亲本nn1138-2和cssl3068不同环境下的百粒重
cv,变异系数;h2,遗传率。
(3)、群体基因型鉴定
采用ctab法提取次级群体f2的294个株系的大豆叶片的基因组dna,所用引物由英潍捷基(上海)贸易公司合成。pcr反应体系10ul,其中含有3uldna(15ul)、上下游引物(0.2mmol/l)各1.5μl、1.2μlmgcl2(2.5mmol/l)、0.24μldntp(10mmol/l,n=a,c,g,t),0.12μltaq酶(5u/ul)和1.4μlddh2o。pcr反应程序:94℃变性5min;随后进行33个循环的94℃变性30s,55℃退火30s,72℃延伸40s;再经过72℃彻底延伸10min;最后4℃保存。pcr扩增产物用8%聚丙烯酰胺凝胶电泳,银染显色鉴定条带的多态性。
(4)、遗传连锁图谱的构建
利用第16号染色体上的49个ssr分子标记分析两个亲本,统计分析亲本间具有多态性的位点共7个,在次级群体f2株系中鉴定亲本间具有多态性标记的遗传类型,然后对其进行连锁分析,构建次级群体的遗传连锁图谱(图3a)。
(5)、分子标记开发
利用song等公布的大豆基因组ssr标记信息及野生豆n24852和栽培豆nn1138-2在gm16号染色体的indel信息,设计了新的分子标记,统计分析亲本之间具有多态性的ssr标记3个,16个indel标记(表2),在次级群体f2中鉴定亲本间具有多态性标记的遗传类型,然后对其进行连锁分析,构建次级群体的高密度图谱(图3b)。
表2本发明涉及的分子标记信息
(6)、结果分析
利用sas9.4结合次级群体表型数据和分子数据初定位结果显示大豆百粒重主效位点qsw16.1位于第16号染色体sat_224和satt431之间。
结合高密度图谱及次级群体f2:3、f2:4及f2:5在四个环境中的表型数据,利用单标记分析法(icimapping2.0)进行精细定位,结果将大豆百粒重主效位点qsw16.1定位于indel16_01与barcsoyssr_16_1215两个标记之间(图3c)。
实施例2调控大豆百粒重基因gmslk的获得
基因gmslk的确定
通过精细定位将大豆百粒重主效位点qsw16.1定位于indel16_01与barcsoyssr_16_1215两个标记之间,通过比对大豆参考基因序列发现这两个标记之间共有两个候选基因,为了进一步确定候选基因,从f2群体中挑选了11个在gm16号染色体35889420bp-35952811bp区域有重组的家系,通过置换作图法比较分析这11个重组家系与两个亲本的基因型和百粒重表型数据进一步进行精细定位,发现indel16_03与barcsoyssr_16_1215两个标记之间没有重组位点,与基因glyma.16g198300共分离(图3d和e)。通过基因注释分析发现基因glyma.16g198300属于lim结构域,其功能注释为转录调控因子(transcriptionalregulator,slk2),因此命名为gmslk。
实施例3调控大豆百粒重基因gmslk在大豆育种中的应用
本发明所述的调控大豆百粒重性状基因的特异扩增上游引物序列为seqidno.5,特异扩增下游引物序列为seqidno.6(表3)。
表3扩增gmslk基因的特异引物序列
利用引物序列seqidno.5和seqidno.6,分别以野生豆n24852和两个亲本(nn1138-2和cssl3068)的rna为模板扩增gmslk的编码区(cds)序列。扩增使用的是takara公司的高保真酶(phantasuper-fidelitydnapoymerase,vazyme),pcr反应体系及程序如表4和表5,扩增条带大小如图4:
表4.pcr反应体系
表5.pcr反应程序
通过测序扩增产物分析比较两个亲本及野生豆n24852之间该基因的cds序列差异,发现基因gmslk在亲本cssl3068和野生豆n24852的序列是一致的,核苷酸序列如seqidno.1所示,编码的氨基酸序列如seqidno.3所示。说明亲本cssl3068的gmslk来自野生豆。栽培豆nn1138-2的gmslk基因核苷酸序列如seqidno.2所示,编码的氨基酸序列如seqidno.4所示。野生豆的gmslk基因在第一个外显子的谷氨酸串联重复区域比栽培豆nn1138-2的gmslk基因多了一个谷氨酰胺(图3e)。且来自野生豆的gmslk基因对大豆百粒重具有减效作用,来自栽培豆(nn1138-2)的gmslk基因对大豆百粒重具有增效作用。
通过序列比对发现indel16_03位于gmslk基因在野生豆n24852与栽培豆nn1138-2的差异区间,故将indel16_03命名为indelslk。因此可以通过这标记来判断不同大豆品种中的gmslk基因是增效基因还是减效基因,在大豆育种选材中具有重要的价值。
鉴定大豆是否含有上述所述的大豆百粒重增效基因的方法,以待鉴定大豆的基因组dna为模板,以seqidno.7和seqidno.8所示的引物进行pcr扩增,若能得到175bp的扩增产物,则该大豆含有所述的大豆百粒重增效基因。
实施例4gmslk基因功能验证
为了能够快速鉴定gmslk基因对籽粒粒重的影响,本实验选择模式作物拟南芥(arabidopsisthaliana)columbia-0野生型作为转基因受体材料,分别过表达亲本cssl3068的gmslk(gmslk-cssl3068)和栽培豆nn1138-2的gmslk(gmslk-nn1138-2)基因,分析比较单拷贝纯合体的1000粒重,从而验证gmslk基因对籽粒粒重的影响。
(1)gmslk基因表达水平分析
为了更好的理解gmslk基因对大豆籽粒发育时期的影响,本实验分析比较gmslk基因在亲本(nn1138-2和cssl3068)的籽粒发育不同时期的表达水平。分别取8棵单株nn1138-2和cssl3068开花后7_daf、14_daf、21_daf,28_daf、35_daf及42_daf(daf:dayafterflower)的籽粒,每个样3个生物学重复,分别测定表型(图5)并提取rna做(real-timert-pcr)rt-pcr分析(图6)。
观察比较亲本nn1138-2和cssl3068在籽粒发育不同时期的籽粒大小,发现nn1138-2的籽粒在不同的发育时期籽粒相比较cssl3068籽粒都较大(图5a),通过测定籽粒的鲜重,结果表明nn1138-2的籽粒在不同的发育时期籽粒鲜重都显著大于cssl3068籽粒显著(图5b)。
分析比较gmslk基因在亲本的籽粒发育不同时期的相对表达水平,本实验选取大豆的ubi3(ribosomal-ubiquitinfusionprotein)基因作为内参。结果分析表明gmslk基因在双亲中的表达丰富度随着籽粒的发育而增大,且在42_daf时期表达量达到最大。然而,gmslk基因在双亲籽粒发育前期和中期(14_daf-35_daf)表达量水平很相似,但是在42_daf时期,gmslk基因在亲本nn1138-2的相对表达量水平显著高于在亲本cssl3068的相对表达量水平。
(2)gmslk基因转基因拟南芥验证
本研究拟南芥转化所选用的是pcambia3301载体,酶切位点为(bsteii和ncoi),gmslk基因扩增上下游引物分别是seqidno.3和seqidno.4(表6)。使用克隆重组试剂盒(clonexperssii)进行重组反应。然后将构建好的过表达载体pcambia3301-gmslk-nn1138-2和pcambia3301-gmslk-cssl30068分别转化入dh5a感受态中进行质粒扩繁,然后通过克隆鉴定,提取质粒后转入农杆菌eha105中。通过花粉侵泡法,分别将构建好的过表达载体转入拟南芥columbia-0野生型中。
表6gmslk基因构建载体引物设计
过表达载体pcambia3301具有草丁膦抗性,阳性苗通过1/2msa+10mg/l的草丁膦培养基进行筛选。收获侵染的拟南芥植株的t1代种子,首先再抗性培养基上筛选t2代阳性苗,然后种植收获t2种子,抗性培养基上对t2种子种植并统计分析存活/死亡比例,通过卡方检测选择存活/死亡符合3:1,说明该转基因植株属于单拷贝植株,然后种植并收获t3代种子,再种植于抗性培养基选择存活率100%的纯合植株,种植并收获t4种子并统计1000粒重表型。对于gmslk-nn1138-2和gmslk-cssl30068过表达植株分别选择3个单拷贝纯合株系,每个株系分别种植3棵,然后取叶片进行rt-pcr分析,选择拟南芥的actin基因作为内参基因,检测转基因植株的外源基因的相对表达量(图7a)。本实验也观察分析了转基因植株3周大小的植株大小,发现过表达的gmslk-nn1138-2和gmslk-cssl30068植株相比较野生型植株大小没有差异(图7b)。收获t5种子统计分析1000粒重表型(图7c),结果分析表明,相比较野生型,过表达的gmslk-nn1138-2可以显著增大粒重,然而过表达gmslk-cssl30068显著降低粒重。因此可以证明来自野生豆或者亲本cssl3068的gmslk基因型属于减效基因,可以降低籽粒粒重,而来自栽培豆nn1138-2的gmslk基因型属于增效基因,可以增加籽粒粒重,这为大豆的产量育种提供了有力的证据。
序列表
<110>南京农业大学
<120>大豆百粒重增效基因及其分子标记和应用
<160>8
<170>siposequencelisting1.0
<210>1
<211>2640
<212>dna
<213>大豆(glycinemax)
<400>1
atgacacctttgcgagtggcaggtggattaacccaatcatcatcaaattctggaattttc60
tatcaaggagatgggcagtcacagaatgtagttaactctcacttaagctcatcttttgtt120
aactcgtctagcacggtttctggagcttctcgttcaaacctgggtccggtttctggggat180
atgaataatgcagttttgaacagtgtggcaaactcagcaccaagtgtaggagccagctct240
ttagttacagatgcaaattctgcactctctggtggcccacatttgcagagaagtgccagc300
gttaacacagactcatacttacgattacctgcctcacctatgtcatttacatcaaataat360
atcagtatttctggatcatcagtgatggatgtttcctctgtagtacaacagagctctcat420
caagatcagaatgttcaacaattgcagcagaatcagcagcagccacagggtgcttcaagt480
gctatgtctttgtctgcatctcaaactggcccttctatgctccaaatgggtgcacaaatc540
ccaggatctttcattcaagatccaaataatatgtctcatctgtcaaagaaacctagaatg600
gatatcaaacaggaagatatgatgcagcagcaggttatacaacagattcttcagagacaa660
gattccatgcaattccagggtcgtaatccccagttacaggctttccttcagcagcagcag720
cagagactgagacaacaacaaatgtttcagcaaatgccacaattacaccgagcacacttg780
cagcagcagcaacaacaacaacaaatgcaattgaggcagcagcagcagcagcagcaacaa840
caacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacag900
caacagcaagtgatgcagccctcttctgtggtcaagcgtccatatgacagtagtgttagt960
ggggtatgtgcccgtcgattgatgcagtatctctatcatcaaaggcaacgaccaaatgat1020
aatagtattgcctattggagaaaatttgtggctgaatattactctcctcgtgcaaagaaa1080
cggtggtgcttgtcattatatagtaatgttgggcatcacgcacttggtgtttttccccaa1140
gcatctatggatgcatggcactgtgacatatgtggttctaaatctggaaggggatttgag1200
gcaacttatgaagtattacctagacttaatgaaatcaaatttggcagtggagtaattgat1260
gaactattgtttctggacatgccacgtgaaatgagattcgcttctggtgcgatgatgtta1320
gaatatggaaaagcagttcaagagagtgtatatgagcagcttcgtgttgttcgtgaaggt1380
caacttcgtatcatattcactcaagacttgaagatattatcttgggagttctgtgcaagg1440
tgccatgaggaacttcttcctcgaaggttggttgcaccacaggtcaaccagttagttcag1500
gtagctaaaaaatgtcaaagtacaattgctgaaagtggttctgatggggtttctcaacaa1560
gacatccaaacaaacagcaacatgttgttgacagctgggggtcagcttgcgaagattttg1620
gagatgcaatcgctaaatgagttgggcttttctaaaagatacgtgagatgtttgcaaatt1680
tcggaggttgtcaatagcatgaaagacctaatagatatctgtgcagatcacaaaattggt1740
gccattgagagtttgaaaaattttcctcgtctagcaacagcctcaaaggtccagatgcag1800
aagatgcaggaaatggaacagctagcaaatgttcaaggtctgccaactgatcgaaacaca1860
ctcaataagctaatggcactgaatcctggattgaacaaccatataaacaaccctcataat1920
atggttaatcgtggtgctttgagtgggtcagcccaagctgctttagcactgaacaactac1980
caaaatcttctcatgaggcaaaattcaatgaactctagccctggctcacttcagcgcgaa2040
gggtcctctttcaataattcaaaccagagtccatcttcagctttgcaaggagctggtcct2100
gctttaattccaggcccaatgcagaattcatctgttagtggtttcccaagcccccgtcta2160
cccccacagcagcagcaacaccacctacaacagccctcattaagtgcaaatgctttactg2220
caacaaaatcattcacagggttcccaaggaaatcaagctctgcagcagcagatgatccat2280
caactactgcaggagatgtcaaataacaacgggggagtgcaaccacagtctcttggtgga2340
cccagtgcaaatatggcaaagaatgcactggggtttgggggccattatccatccttaagt2400
ggaggttctgccaatgttacaggaaacaatggacctatgtcaaggaataatagcttcaaa2460
acaactgcaaatagtgattcttctgctgctggtggcaacaatggattaaaccagagaaca2520
tctgagatgccacaaaatctacatttgcaagatgtggttcaggatattggcaatgaattc2580
acggataatcccttccttaacagtgatcttgatgataacatgggttttggctggaaggca2640
<210>2
<211>2637
<212>dna
<213>大豆(glycinemax)
<400>2
atgacacctttgcgagtggcaggtggattaacccaatcatcatcaaattctggaattttc60
tatcaaggagatgggcagtcacagaatgtagttaactctcacttaagctcatcttttgtt120
aactcgtctagcacggtttctggagcttctcgttcaaacctgggtccggtttctggggat180
atgaataatgcagttttgaacagtgtggcaaactcagcaccaagtgtaggagccagctct240
ttagttacagatgcaaattctgcactctctggtggcccacatttgcagagaagtgccagc300
gttaacacagactcatacttacgattacctgcctcacctatgtcatttacatcaaataat360
atcagtatttctggatcatcagtgatggatgtttcctctgtagtacaacagagctctcat420
caagatcagaatgttcaacaattgcagcagaatcagcagcagccacagggtgcttcaagt480
gctatgtctttgtctgcatctcaaactggcccttctatgctccaaatgggtgcacaaatc540
ccaggatctttcattcaagatccaaataatatgtctcatctgtcaaagaaacctagaatg600
gatatcaaacaggaagatatgatgcagcagcaggttatacaacagattcttcagagacaa660
gattccatgcaattccagggtcgtaatccccagttacaggctttccttcagcagcagcag720
cagagactgagacaacaacaaatgtttcagcaaatgccacaattacaccgagcacacttg780
cagcagcagcaacaacaacaacaaatgcaattgaggcagcagcagcagcagcagcaacaa840
caacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacaacagcaa900
cagcaagtgatgcagccctcttctgtggtcaagcgtccatatgacagtagtgttagtggg960
gtatgtgcccgtcgattgatgcagtatctctatcatcaaaggcaacgaccaaatgataat1020
agtattgcctattggagaaaatttgtggctgaatattactctcctcgtgcaaagaaacgg1080
tggtgcttgtcattatatagtaatgttgggcatcacgcacttggtgtttttccccaagca1140
tctatggatgcatggcactgtgacatatgtggttctaaatctggaaggggatttgaggca1200
acttatgaagtattacctagacttaatgaaatcaaatttggcagtggagtaattgatgaa1260
ctattgtttctggacatgccacgtgaaatgagattcgcttctggtgcgatgatgttagaa1320
tatggaaaagcagttcaagagagtgtatatgagcagcttcgtgttgttcgtgaaggtcaa1380
cttcgtatcatattcactcaagacttgaagatattatcttgggagttctgtgcaaggtgc1440
catgaggaacttcttcctcgaaggttggttgcaccacaggtcaaccagttagttcaggta1500
gctaaaaaatgtcaaagtacaattgctgaaagtggttctgatggggtttctcaacaagac1560
atccaaacaaacagcaacatgttgttgacagctgggggtcagcttgcgaagattttggag1620
atgcaatcgctaaatgagttgggcttttctaaaagatacgtgagatgtttgcaaatttcg1680
gaggttgtcaatagcatgaaagacctaatagatatctgtgcagatcacaaaattggtgcc1740
attgagagtttgaaaaattttcctcgtctagcaacagcctcaaaggtccagatgcagaag1800
atgcaggaaatggaacagctagcaaatgttcaaggtctgccaactgatcgaaacacactc1860
aataagctaatggcactgaatcctggattgaacaaccatataaacaaccctcataatatg1920
gttaatcgtggtgctttgagtgggtcagcccaagctgctttagcactgaacaactaccaa1980
aatcttctcatgaggcaaaattcaatgaactctagccctggctcacttcagcgcgaaggg2040
tcctctttcaataattcaaaccagagtccatcttcagctttgcaaggagctggtcctgct2100
ttaattccaggcccaatgcagaattcatctgttagtggtttcccaagcccccgtctaccc2160
ccacagcagcagcaacaccacctacaacagccctcattaagtgcaaatgctttactgcaa2220
caaaatcattcacagggttcccaaggaaatcaagctctgcagcagcagatgatccatcaa2280
ctactgcaggagatgtcaaataacaacgggggagtgcaaccacagtctcttggtggaccc2340
agtgcaaatatggcaaagaatgcactggggtttgggggccattatccatccttaagtgga2400
ggttctgccaatgttacaggaaacaatggacctatgtcaaggaataatagcttcaaaaca2460
actgcaaatagtgattcttctgctgctggtggcaacaatggattaaaccagagaacatct2520
gagatgccacaaaatctacatttgcaagatgtggttcaggatattggcaatgaattcacg2580
gataatcccttccttaacagtgatcttgatgataacatgggttttggctggaaggca2637
<210>3
<211>880
<212>prt
<213>大豆(glycinemax)
<400>3
metthrproleuargvalalaglyglyleuthrglnserserserasn
151015
serglyilephetyrglnglyaspglyglnserglnasnvalvalasn
202530
serhisleuserserserphevalasnserserserthrvalsergly
354045
alaserargserasnleuglyprovalserglyaspmetasnasnala
505560
valleuasnservalalaasnseralaproservalglyalaserser
65707580
leuvalthraspalaasnseralaleuserglyglyprohisleugln
859095
argseralaservalasnthraspsertyrleuargleuproalaser
100105110
prometserphethrserasnasnileserileserglyserserval
115120125
metaspvalserservalvalglnglnserserhisglnaspglnasn
130135140
valglnglnleuglnglnasnglnglnglnproglnglyalaserser
145150155160
alametserleuseralaserglnthrglyprosermetleuglnmet
165170175
glyalaglnileproglyserpheileglnaspproasnasnmetser
180185190
hisleuserlyslysproargmetaspilelysglngluaspmetmet
195200205
glnglnglnvalileglnglnileleuglnargglnaspsermetgln
210215220
pheglnglyargasnproglnleuglnalapheleuglnglnglngln
225230235240
glnargleuargglnglnglnmetpheglnglnmetproglnleuhis
245250255
argalahisleuglnglnglnglnglnglnglnglnmetglnleuarg
260265270
glnglnglnglnglnglnglnglnglnglnglnglnglnglnglngln
275280285
glnglnglnglnglnglnglnglnglnglnglnglnglnglnglnval
290295300
metglnproserservalvallysargprotyraspserservalser
305310315320
glyvalcysalaargargleumetglntyrleutyrhisglnarggln
325330335
argproasnaspasnserilealatyrtrparglysphevalalaglu
340345350
tyrtyrserproargalalyslysargtrpcysleuserleutyrser
355360365
asnvalglyhishisalaleuglyvalpheproglnalasermetasp
370375380
alatrphiscysaspilecysglyserlysserglyargglypheglu
385390395400
alathrtyrgluvalleuproargleuasngluilelyspheglyser
405410415
glyvalileaspgluleuleupheleuaspmetproargglumetarg
420425430
phealaserglyalametmetleuglutyrglylysalavalglnglu
435440445
servaltyrgluglnleuargvalvalarggluglyglnleuargile
450455460
ilephethrglnaspleulysileleusertrpgluphecysalaarg
465470475480
cyshisglugluleuleuproargargleuvalalaproglnvalasn
485490495
glnleuvalglnvalalalyslyscysglnserthrilealagluser
500505510
glyseraspglyvalserglnglnaspileglnthrasnserasnmet
515520525
leuleuthralaglyglyglnleualalysileleuglumetglnser
530535540
leuasngluleuglypheserlysargtyrvalargcysleuglnile
545550555560
sergluvalvalasnsermetlysaspleuileaspilecysalaasp
565570575
hislysileglyalailegluserleulysasnpheproargleuala
580585590
thralaserlysvalglnmetglnlysmetglnglumetgluglnleu
595600605
alaasnvalglnglyleuprothraspargasnthrleuasnlysleu
610615620
metalaleuasnproglyleuasnasnhisileasnasnprohisasn
625630635640
metvalasnargglyalaleuserglyseralaglnalaalaleuala
645650655
leuasnasntyrglnasnleuleumetargglnasnsermetasnser
660665670
serproglyserleuglnarggluglyserserpheasnasnserasn
675680685
glnserproserseralaleuglnglyalaglyproalaleuilepro
690695700
glyprometglnasnserservalserglypheproserproargleu
705710715720
proproglnglnglnglnhishisleuglnglnproserleuserala
725730735
asnalaleuleuglnglnasnhisserglnglyserglnglyasngln
740745750
alaleuglnglnglnmetilehisglnleuleuglnglumetserasn
755760765
asnasnglyglyvalglnproglnserleuglyglyproseralaasn
770775780
metalalysasnalaleuglypheglyglyhistyrproserleuser
785790795800
glyglyseralaasnvalthrglyasnasnglyprometserargasn
805810815
asnserphelysthrthralaasnseraspserseralaalaglygly
820825830
asnasnglyleuasnglnargthrserglumetproglnasnleuhis
835840845
leuglnaspvalvalglnaspileglyasngluphethraspasnpro
850855860
pheleuasnseraspleuaspaspasnmetglypheglytrplysala
865870875880
<210>4
<211>879
<212>prt
<213>大豆(glycinemax)
<400>4
metthrproleuargvalalaglyglyleuthrglnserserserasn
151015
serglyilephetyrglnglyaspglyglnserglnasnvalvalasn
202530
serhisleuserserserphevalasnserserserthrvalsergly
354045
alaserargserasnleuglyprovalserglyaspmetasnasnala
505560
valleuasnservalalaasnseralaproservalglyalaserser
65707580
leuvalthraspalaasnseralaleuserglyglyprohisleugln
859095
argseralaservalasnthraspsertyrleuargleuproalaser
100105110
prometserphethrserasnasnileserileserglyserserval
115120125
metaspvalserservalvalglnglnserserhisglnaspglnasn
130135140
valglnglnleuglnglnasnglnglnglnproglnglyalaserser
145150155160
alametserleuseralaserglnthrglyprosermetleuglnmet
165170175
glyalaglnileproglyserpheileglnaspproasnasnmetser
180185190
hisleuserlyslysproargmetaspilelysglngluaspmetmet
195200205
glnglnglnvalileglnglnileleuglnargglnaspsermetgln
210215220
pheglnglyargasnproglnleuglnalapheleuglnglnglngln
225230235240
glnargleuargglnglnglnmetpheglnglnmetproglnleuhis
245250255
argalahisleuglnglnglnglnglnglnglnglnmetglnleuarg
260265270
glnglnglnglnglnglnglnglnglnglnglnglnglnglnglngln
275280285
glnglnglnglnglnglnglnglnglnglnglnglnglnglnvalmet
290295300
glnproserservalvallysargprotyraspserservalsergly
305310315320
valcysalaargargleumetglntyrleutyrhisglnargglnarg
325330335
proasnaspasnserilealatyrtrparglysphevalalaglutyr
340345350
tyrserproargalalyslysargtrpcysleuserleutyrserasn
355360365
valglyhishisalaleuglyvalpheproglnalasermetaspala
370375380
trphiscysaspilecysglyserlysserglyargglyphegluala
385390395400
thrtyrgluvalleuproargleuasngluilelyspheglysergly
405410415
valileaspgluleuleupheleuaspmetproargglumetargphe
420425430
alaserglyalametmetleuglutyrglylysalavalglngluser
435440445
valtyrgluglnleuargvalvalarggluglyglnleuargileile
450455460
phethrglnaspleulysileleusertrpgluphecysalaargcys
465470475480
hisglugluleuleuproargargleuvalalaproglnvalasngln
485490495
leuvalglnvalalalyslyscysglnserthrilealaglusergly
500505510
seraspglyvalserglnglnaspileglnthrasnserasnmetleu
515520525
leuthralaglyglyglnleualalysileleuglumetglnserleu
530535540
asngluleuglypheserlysargtyrvalargcysleuglnileser
545550555560
gluvalvalasnsermetlysaspleuileaspilecysalaasphis
565570575
lysileglyalailegluserleulysasnpheproargleualathr
580585590
alaserlysvalglnmetglnlysmetglnglumetgluglnleuala
595600605
asnvalglnglyleuprothraspargasnthrleuasnlysleumet
610615620
alaleuasnproglyleuasnasnhisileasnasnprohisasnmet
625630635640
valasnargglyalaleuserglyseralaglnalaalaleualaleu
645650655
asnasntyrglnasnleuleumetargglnasnsermetasnserser
660665670
proglyserleuglnarggluglyserserpheasnasnserasngln
675680685
serproserseralaleuglnglyalaglyproalaleuileprogly
690695700
prometglnasnserservalserglypheproserproargleupro
705710715720
proglnglnglnglnhishisleuglnglnproserleuseralaasn
725730735
alaleuleuglnglnasnhisserglnglyserglnglyasnglnala
740745750
leuglnglnglnmetilehisglnleuleuglnglumetserasnasn
755760765
asnglyglyvalglnproglnserleuglyglyproseralaasnmet
770775780
alalysasnalaleuglypheglyglyhistyrproserleusergly
785790795800
glyseralaasnvalthrglyasnasnglyprometserargasnasn
805810815
serphelysthrthralaasnseraspserseralaalaglyglyasn
820825830
asnglyleuasnglnargthrserglumetproglnasnleuhisleu
835840845
glnaspvalvalglnaspileglyasngluphethraspasnprophe
850855860
leuasnseraspleuaspaspasnmetglypheglytrplysala
865870875
<210>5
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>5
atgacacctttgcgagtggc20
<210>6
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>6
tcatgccttccagccaaaaccc22
<210>7
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>7
acaattacaccgagcaca18
<210>8
<211>18
<212>dna
<213>人工序列(artificialsequence)
<400>8
cttgaccacagaagaggg18
- 还没有人留言评论。精彩留言会获得点赞!