水稻气孔开放型钾离子通道基因OsK2-1的应用及其表达载体的制作方法
本发明涉及植物基因工程技术领域,特别是一种水稻气孔开放型钾离子通道蛋白基因osk2-1的应用。
背景技术
钾是作物生长发育所必需的关键矿质营养元素,它不仅具有提高植物体内酶系统的活性、促进植物代谢、体内物质运输和蛋白质的合成等功能,而且能够增强植物的光合作用及植株的抗逆性。植物对钾的吸收主要由各种类别的钾转运体和钾通道介导(epstein等,1963)。转运体,其主要特点是和被转运离子相结合,通过一系列构象变化实现跨膜转运,迄今为止,在植物中已发现存在三大钾转运体家族,分别为kup/hak/kt家族,trk/hkt家族,cpa(cationprotonantiporter)家族,这些转运体都具有转运钾的功能(gierthand
气孔是由一对特化的表皮细胞(保卫细胞)围成的孔状结构,归属于groupⅱ中的kat型钾通道,主要在叶片和保卫细胞中表达,介导k+进入保卫细胞调控气孔的开放而与植物的光合作用和水分蒸腾密切相关(lebaudy等,2008)。因为气孔是植物水分蒸腾和获取光合co2的主要门户,促进气孔的高效开放不仅利于生物量的积累,更有利于养分的高效运输,从而有助于提高养分利用率和产量。
水稻是重要的粮食作物,生长在淹水条件下以吸收铵态氮为主要的氮源,当前的种植模式下,为保证水稻产量不得不施用远超过水稻所需要的氮素化肥“堆砌”充足的氮素养分供应以发挥现有高产水稻品种的增产潜力,这不仅导致氮肥利用率(nue)走低,同时过量的氮也对环境造成巨大压力。解决上述问题的关键是充分挖掘水稻自身吸收/利用氮素的基因资源并通过转基因/育种技术,定向培育水稻的优良氮素营养形状,充分开发水稻自身吸收和利用氮素的潜力。
大量化肥的施用及集约化施肥方式使得现有农田的铵态氮水平可高达20mm(britto等,2002)。现有通过分子调控手段超表达铵吸收同化或响应基因改良水稻氮素营养形状的研究,超表达水稻铵吸收系统osamt1;1的水稻仅在小于等于0.3mmnh4+(远低于现实农田铵水平)条件下展现出一定的正面效果,在较高铵(3mmnh4+)条件下水稻生长反而出现生长抑制现象(ranathunge等,2014);调控另一个水稻铵吸收系统osamt1;3后,水稻体内出现c;n代谢失衡进而导致产量降低(bao等,2015),而超表达铵同化基因(如osgs1;1,1;2及glna)的水稻产量均降低(cai等,2009)。超表达氮代谢调控基因来自玉米的转录因子dof1能够提高水稻的氮素利用效率,但是这种正面效果也仅在低氮条件下出现(kurai等,2011)。近年来发现的一些其他的转录因子如hap3,14-3-3蛋白,qngr9,mirna,以及激酶cipk8和cipk23都参与氮素吸收代谢的调控,但是尚未见这些基因在提高水稻氮素利用效率中的应用。并且在应用中发现的有作用的基因都在氮素供应较低的条件下有效,这与我国当前高度集约化的生产模式不匹配。因此,充分发掘水稻自身具有应用前景尤其是在高氮条件下具有应用前景的基因资源具有重要的理论和现实意义。
技术实现要素:
针对上述问题,本发明的目的之一是提供一种水稻气孔开放型钾离子吸收通道基因osk2-1,其编码的osk2-1蛋白在高氮条件下可以通过调控气孔开度增强光合速率和蒸腾速率,进而提供该蛋白在提高水稻氮素利用效率及水稻产量方面的应用。
为实现上述发明目的,本申请采用如下技术方案:
一种水稻气孔开放型钾离子吸收通道基因osk2-1在提高农作物光合速率、气孔导度及蒸腾速率中的应用,该osk2-1基因dna的核苷酸序列如seqidno.1所示;所述农作物优选水稻。
一种水稻气孔开放型钾离子通道基因osk2-1在提高农作物高氮环境中氮素利用效率中的应用;所述钾离子通道基因osk2-1的核苷酸序列如seqidno.1所示;所述农作物优选水稻。
含有核苷酸序列如seqidno.1所示的水稻气孔开放型钾离子吸收通道基因osk2-1的动物表达载体,如将水稻osk2-1蛋白编码基因构建到动物表达载体ptracer-cmv3中,所得到的重组质粒ptracer-cmv3-osk2-1。
含有核苷酸序列如seqidno.1所示的水稻气孔开放型钾离子吸收通道基因osk2-1的植物超表达载体,如将水稻osk2-1蛋白编码基因构建到单子叶植物超表达载体pcambia1301-ubiquitin-nos(简称pun1301)中,所得到的重组质粒pun1301-osk2-1。
本申请中,技术术语“低氮环境”是指:外界氮含量不高于66.7kg纯n/hm2;“高氮环境”是指:外界氮含量不低于300kg纯n/hm2。
本发明首次发现并应用了osk2-1基因编码的了osk2-1蛋白在高氮条件下,在调控水稻气孔开度、速率及蒸腾效率方面的应用,通过将该基因导入水稻并表达,实现在高氮条件下能够提高水稻的气孔开度、光合速率及蒸腾效率,从而提高农作物(特别是水稻)对高氮环境中氮的有效利用能力。
附图说明
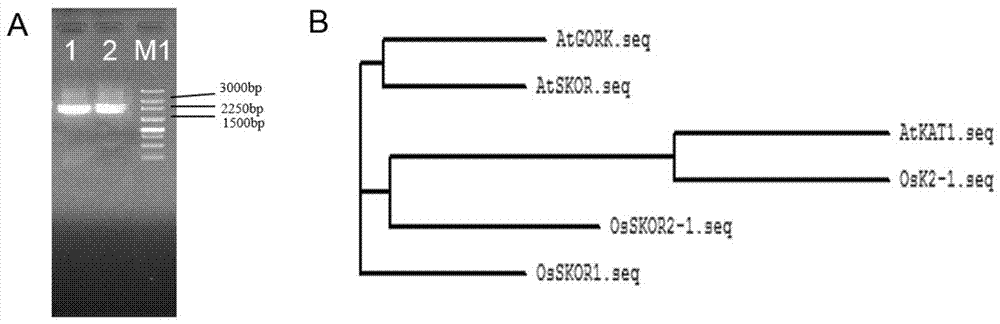
图1为水稻osk2-1基因克隆及生物信息学分析结果示意图;
其中图1a:m1为250bpdnaladdermarker,lane1为osk2-1(noti/noti)的pcr结果,lane2为水稻osk2-1(bamhi/saci的pcr结果;图1b:为osk2-1的生物信息学-进化树分析。
图2重组质粒图ptracer-cmv3-osk2-1及pun1301-osk2-1的双酶切鉴定结果电泳图;图2a:m1为250bpdnaladdermarker,lane1-lane4为重组质粒ptracer-cmv3-osk2-1的noti/noti双酶切鉴定结果;图2b:m2为dl15,000dnamarker,lane1-6为pun1301-osk2-1的bamhi/saci双酶切鉴定结果,m1为250bpdnaladdermarker。
图3osk2-1的时空表达模式和精确组织定位结果示意图;
其中,图3a:osk2-1的时空表达模式;图3b:osk2-1的精确组织定位结果。
图4是osk2-1的钾离子转运活性验证结果示意图;
其中图4a:转染ptracer-cmv3-osk2-1的hek-293t细胞转运钾离子特征电流曲线;图4b:转染空载体ptracer-cmv3的hek-293t细胞转运钾离子特征电流曲线;图4c:osk2-1介导的钾离子吸收所产生的净电流曲线示意图;
图5是小区实验低氮和高氮条件下转osk2-1水稻株系各项指标实验结果示意图;
其中,图5a为气孔开度检测结果示意图,图5b为光合速率检测结果示意图,图5c为蒸腾速率检测结果示意图,其中ln表示小区实验中的低氮条件,hn表示小区实验中的高氮条件。
图6是小区实验低氮和高氮条件下条件下转osk2-1水稻株系的产量示意图;
其中图6a为总生物量检测结果示意图;图6b为稻草重量检测结果示意图;图6c为籽粒产量结果示意图。
具体实施方式
实施例中,日本晴和中花11水稻均购自于北京未名凯拓农业生物技术有限公司、pmd19-tsimple购自takara公司。
以下实施例所涉及的原料和试剂,除非特殊说明,均为市售渠道购买。
实施例1水稻osk2-1基因的克隆及生物信息学分析
提取水培生长10天的日本晴水稻的总rna并以其为模板,以oligo(dt)18为引物反转录,得到反转录产物cdna。根据水稻osk2-1的全长编码序列,设计扩增出完整编码阅读框的引物,根据需要设计两对引物,一对为上游引物f1和下游引物r1(forptracer-cmv3),另一对为上游引物f2和下游引物r2(forpun1301),并在各引物上引入不同的酶切位点,如下:
f1(noti,seqidno.3):
gtcgcggccgcatgacccaagctcactcaaaatcttgcttc(forptracer-cmv3);
r1(noti,seqidno.4):
gtcgcggccgctacatctcaagaaggaatagatggtcgccatc(forptracer-cmv3);
f2(bamhi,seqidno.5):
gtcggatccatgacccaagctcactcaaaatcttgcttc(forpun1301);
r2(saci,seqidno.6):gtcgagctcctacatctcaagaaggaatagatggtcgccatc(forpun1301);
使用高保真酶
pcr反应程序:95℃预变性5min;95℃变性30sec,60℃退火30sec,72℃延伸2min30sec,30个循环;72℃延伸10min;4℃保存。
电泳检测,结果得到两条约2.2kb左右的单一产物(图1a)。通过凝胶电泳回收扩增片段,与pmd19-tsimple载体相连接获得ta-osk2-1质粒,委托北京六合华大基因科技有限公司上海分公司进行测序,确定获得水稻osk2-1的cdna序列,其cdna序列(大小为2157bp)及其编码的蛋白质的氨基酸序列(大小为719aa)分别如seqidno.1和seqidno.2所示。进化树分析表明,水稻osk2-1与已经报道的拟南芥气孔钾离子吸收通道atkat1具有高度的同源性(图1b),暗示了其在功能上的相似性。
实施例2:水稻osk2-1基因表达的组织特异性
1.水稻osk2-1基因的时空表达模式
为了解osk2-1基因在水稻不同组织器官中的表达情况,将萌发生长一致的水稻苗在木村营养液(参见文献:lism等,2006)中培养7d后再进行不同浓度的钾处理,这些处理分别为:
对照组:,完全木村营养液(2mmkcl);
缺k组:,0mmkcl(2mmnacl代替);
高k组:,20mmkcl;
每天更换营养液,处理两天后分根和地上部取样,冻于液氮中,分别提取了水稻根和地上部的总rna,以oligo(dt)18为引物反转录(操作步骤按照逆转录试剂盒说明(amv)进行),得到反转录产物cdna。以此cdna为模板,以特异引物osk2-1f1和osk2-1r1进行半定量pcr分析。
pcr反应体系为25μl:2μlmgcl2,2μldntp,2.5μlextaqbuffer,0.125μlextaq酶,上下游特异引物各1.25μl,模板1μl,用dh2o补齐至25μl;
pcr反应程序为:94℃,3min;94℃,30sec,60℃,30sec,72℃,1min,35个循环;72℃,10min。
内参osactin2为28个循环,上游引物和下游引物分别为osactin-f1和osactin-r1。
osk2-1f1(seqidno.7):5'ttctttccgatcatccggcaagcct3';
osk2-1r1(seqidno.8):5'gtggagaccctgtctgcactgttgc3';
osactin-f1(seqidno.9):5'ctccatcatgaagtgcgacgtggat3';
osactin-r1(seqidno.10):5'tggtacccgcatcaggcatctgatt3';
结果显示,在不同钾浓度条件下,osk2-1基因都主要在水稻地上部的表达,而在根中几乎检测不到其表达(图2a),表明osk2-1基因在水稻中有明显的组织特异性,其在地上部的表达较为活跃。
2.水稻osk2-1基因的精确组织定位
为进一步了解osk2-1的功能特征,通过原位杂交研究了osk2-1在水稻地上部的精确组织定位特征,根据osk2-1的mrna序列,设计特异性寡核苷酸序列正义(sense)链(seqidno.11):5’catgggaccagccgcaccag3’和反义(anti-sense)链(seqidno.12):5’tggtcgccatcgcggataaca3’,并在5’分别进行地高辛(digoxigenin)标记,在3’分别进行磷酸化修饰,从而得到sense和anti-sense的探针并由上海invitrogen公司合成探针。
通过制备水稻叶片组织切片并进行杂交(该方法为本领域常规技术,具体可参见文献:zhao等,2009),结果显示osk2-1主要定位于叶片的叶肉和保卫细胞(图2b)中,暗示其在气孔开度调控方面的作用。
实施例3:重组质粒ptracer-cmv3-osk2-1和pun1301-osk2-1的构建
将水稻osk2-1基因经由酶切位点经酶切位点noti/noti构建到ptracer-cmv3上,转化大肠杆菌感受态细胞dh5α后,提取质粒并酶切(图3a)鉴定(kanwar,等,1980),结果表明已经成功获得重组质粒ptracer-cmv3-osk2-1。
将水稻osk2-1基因经由酶切位点bamhi/saci构建到pun1301,转化大肠杆菌感受态细胞dh5α后,提取质粒并酶切(图3b)鉴定。结果表明已经成功获得重组质粒pun1301-osk2-1。
实施例4:水稻osk2-1基因的电生理功能分析
1.溶液配制
电极内液成分为:120mmk-gluconate,5mmnacl,5mmkcl,2mmmgcl2,2mmcacl2,4mmegta,10mmhepes,14mmtris-creatinephosphate,4mmtris-atp,0.3mmtris-gtp,用1mkoh将ph调至7.3,k+终浓度约为140mm,渗透压约为300mosm。
细胞外液成分为:1.0mmmgcl2,1.2mmcacl2,10.0mmhepes,20mmglucose,150mmkcl(低于150mm时用nacl代替kcl补充至150mm),用1mnaoh调节ph至7.4,渗透压用sucrose调至约320mosm。
2.质粒转染
将质粒ptracer-cmv3-osk2-1用lipofectamine转染到hek293-t细胞中,对照转染空载体ptracer-cmv3,再放回细胞培养箱中表达一天,第二天挑取带绿色荧光的细胞检测。
3.检测程序设置:
在显微镜下,将电极(已灌注电极内液)通过电动微操接触hek293-t细胞,施加负压使电极尖端与hek293-t质膜之间形成高阻封接(gω),继续施加负压,吸破hek293-t细胞膜,使电极与细胞之间形成全细胞记录模式。稳定5min后,开始记录电流大小。记录过程中,吸收型钾离子通道电流电压施加方案为钳制电压-40mv,阶越电压为20mv,共11个刺激电压,从-180mv到+20mv,刺激间隔5.5s,采样频率10khz。数据采集用软件为pclamp10.0。
记录到表达ptracer-cmv3-osk2-1的细胞电流特征曲线如图4a所示,记录到表达ptracer-cmv3空载体的细胞电流特征曲线如图4b所示,获得osk2-1通道净电流特征曲线如图4c所示,由图可知osk2-1介导k+吸收产生了电流,是典型的内向吸收型钾离子通道。
实施例5利用pun1301-osk2-1转基因水稻植株研究osk2-1的应用
1.转osk2-1转基因水稻材料的制备
采用根癌农杆菌介导法(该方法为本领域常规技术,具体可参见文献:valvekens等,1988)将实施例3中构建获得的重组质粒pun1301-osk2-1转入水稻中花11品种。诱导水稻成熟愈伤,将长到适度大小的愈伤组织挑出,放入农杆菌悬浮液侵染5-10分钟,将愈伤组织取出,置于无菌的滤纸上沥干30-40分钟,愈伤组织置于共培养基上,28℃暗培养3天,然后将愈伤转入含50mg/l潮霉素的选择培养基上进行筛选,挑取颜色亮黄的抗性愈伤移入装有分化培养基的培养皿中,放入恒温培养箱分化成苗。再放入生根培养基中壮苗一到两周。通过鉴定获得osk2-1超表达水稻株系oe-26。
2.小区实验检验转osk2-1水稻的气孔导度、光合速率及蒸腾速率检测
将转osk2-1的水稻(水稻株系oe-26)播种至低氮(ln,66.7kg纯n/hm2)、高氮(hn,300kg纯n/hm2)两个氮素水平小区中,在结实灌浆期采用licor-6400光合仪测定剑叶的光合速率、气孔导度及蒸腾速率(该测定技术为本领域常规技术,具体可参见文献:reguera,等,2013),结果显示,转osk2-1水稻的的气孔开度(图5a)、光合速率(图5b)及蒸腾速率(图5c)显著高于野生型(中花11),增加趋势在高氮条件下尤为显著(图5),表明osk2-1基因能够调控气孔的开度并响应环境中不同的氮水平。
3.转osk2-1水稻的氮素利用效率
在成熟期将地上部齐地面割下,单株收获装至独立的网袋中,晾干后进行总生物量、稻草重量及籽粒产量的鉴定,结果显示,高氮条件(外界氮含量不低于300kg纯n/hm2)下(转osk2-1株系的总生物量(图6a)包括稻草重量(图6b)及籽粒产量(图6c)显著高于野生型。也就是说,osk2-1通过调控水稻的气孔开度,增强了水稻的光合速率及蒸腾速率,光合效率的增强促进了光合产物的积累,蒸腾速率的提高带动了氮钾养分向地上部的转移,促进了结实灌浆期关键功能叶剑叶氮的积累,利于其光合作用时间的延长及碳水化合物的积累,并且这些优势最终体现在转osk2-1水稻的产量上。
本技术领域技术人员可以理解的是,除非另外定义,这里使用的所有术语(包括技术术语和科学术语)具有与本发明所属领域中的普通技术人员的一般理解相同的意义。还应该理解的是,诸如通用工具书中定义的那些术语应该被理解为具有与现有技术的上下文中的意义一致的意义,并且除非像这里一样定义,不会用理想化或过于正式的含义来解释。
以上所述的具体实施方式,对本发明的目的、技术方案和有益效果进行了进一步详细说明,所应理解的是,以上所述仅为本发明的具体实施方式而已,并不用于限制本发明,凡在本发明的精神和原则之内,所做的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
序列表
<110>中国科学院南京土壤研究所
<120>水稻气孔开放型钾离子通道基因osk2-1的应用及其表达载体
<141>2018-08-22
<160>12
<170>siposequencelisting1.0
<210>1
<211>2157
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgacccaagctcactcaaaatcttgcttccaccagttctgggatggactccagatcaag60
aggagcagcgatagcttcactgtagagctactgccatcccttggtgcaactataaaccac120
tccaacaagttgcagaagttcatcatatcgccttatgatccccggtacagatcctgggag180
ctgttccttatagtcctagttgtttactctgcctggatttgtccgtttgaactagcattc240
ctgagggacttaccatctaagcttttgctagttgagaacattgtggatatattctttgcc300
attgatattgttttgacgttcttcgttgcttatgtcgatagcaaaacacatcttcttgtg360
gatgaccgaaagagaattgcaatgaggtacctatccacttggtttatctttgatgtatgt420
tcaacagcaccatttcaaccaatcatccttctatttacacacaaggggaatgacattgct480
ttcaaggtactcaatttgctcaggctgtggcgtcttcaccgagtcagttcactatttgcc540
aggctggagaaagacatccgattcaactatttctggactaggtgctcaaaacttatttct600
gtgaccctcttcgcagtacattgtgcaggatgcttcaattatatgattgctgacagatat660
cccaacccagagaaaacatggataggagctgtgatgtcaactttcagatcagagagctta720
tggactagatatattactgctctttactggtccattacaacactgacaacaacaggatat780
ggggacctacatgctgaaaatccaacagagatgctatttgatattgtctatatgatgttt840
aatctgggcttgacagcttatctcattggcaacatgaccaacctagttgtccatgggacc900
agccgcaccaggaaatttagggactcaatccaagcagcatcagagtttgcagcacgcaat960
cagctacctgagaacataaagcagcaagtgctatctcacttctgcctacaattcaagaca1020
gagggactcaaccagcaggtcatgttagactgtctaccaaaaggaatccgctcaagcata1080
gcatacagcttattctttccgatcatccggcaagcctatctcttcaatggagtatctggc1140
aatttcattgcagaactggtcatggaagtgcaggctgagtacttcccaccaaaggaagat1200
ataatattgcagaatgaaggagaagcagatgtttacatagtagtttcaggagcagtgaat1260
ataataacaacaatacatgggaatgagcaggtatatgagaaaattgcagagggagaaatg1320
tttggagaggttggttctttatgtaacataccccagccatttacttgccgcacagcggag1380
ctatcacagcttttaagaataagcaaaacaaggcttagagagatcattgaagaaaacagg1440
gaagacagtaacattctaatgaacaacctagttcagaagctgaagctacgagaaagttta1500
cctgacatgaatcaaccagaccgaagattcttgagcaagtatgagctattccacgttcct1560
cgagaagcatggctgctgaaaaagtcacaactacattacacagagcacacaagtagggat1620
tctagtaacaacactccagtatttggaggtgacagatattccagacagttacttggagaa1680
gcaacccgatcttcggcatcagaaaacgaaaatagcagtatgacagataaagaagagaac1740
catgatgaagttcacacaaactgtgagatcaaaaaaagaacggaagagcactgtatccag1800
ataaactctgaagatagcagctccacatacagtcagcgaacaatgaatgcaacagtgcag1860
acagggtctccacataaaacagaagaaaacataacaagaagaattccagatgagtactac1920
ataaaagaagcaaacaaaagagttacaattcacaaatatcgtcataactcaacagtctct1980
gctgcgcaaaatggaaagcttatcaagctgcctacatcattggaggaactgttcaaaatt2040
ggcagtcagaagtttcaaggttttcatcctagaaaggtggtcagtagagattatgctgaa2100
atagatgatgtcagtgttatccgcgatggcgaccatctattccttcttgagatgtag2157
<210>2
<211>718
<212>prt
<213>人工序列(artificialsequence)
<400>2
metthrglnalahisserlyssercysphehisglnphetrpaspgly
151015
leuglnilelysargserseraspserphethrvalgluleuleupro
202530
serleuglyalathrileasnhisserasnlysleuglnlyspheile
354045
ileserprotyraspproargtyrargsertrpgluleupheleuile
505560
valleuvalvaltyrseralatrpilecysprophegluleualaphe
65707580
leuargaspleuproserlysleuleuleuvalgluasnilevalasp
859095
ilephephealaileaspilevalleuthrphephevalalatyrval
100105110
aspserlysthrhisleuleuvalaspasparglysargilealamet
115120125
argtyrleuserthrtrppheilepheaspvalcysserthralapro
130135140
pheglnproileileleuleuphethrhislysglyasnaspileala
145150155160
phelysvalleuasnleuleuargleutrpargleuhisargvalser
165170175
serleuphealaargleuglulysaspileargpheasntyrphetrp
180185190
thrargcysserlysleuileservalthrleuphealavalhiscys
195200205
alaglycyspheasntyrmetilealaaspargtyrproasnproglu
210215220
lysthrtrpileglyalavalmetserthrpheargsergluserleu
225230235240
trpthrargtyrilethralaleutyrtrpserilethrthrleuthr
245250255
thrthrglytyrglyaspleuhisalagluasnprothrglumetleu
260265270
pheaspilevaltyrmetmetpheasnleuglyleuthralatyrleu
275280285
ileglyasnmetthrasnleuvalvalhisglythrserargthrarg
290295300
lyspheargaspserileglnalaalasergluphealaalaargasn
305310315320
glnleuprogluasnilelysglnglnvalleuserhisphecysleu
325330335
glnphelysthrgluglyleuasnglnglnvalmetleuaspcysleu
340345350
prolysglyileargserserilealatyrserleuphepheproile
355360365
ileargglnalatyrleupheasnglyvalserglyasnpheileala
370375380
gluleuvalmetgluvalglnalaglutyrpheproprolysgluasp
385390395400
ileileleuglnasngluglyglualaaspvaltyrilevalvalser
405410415
glyalavalasnileilethrthrilehisglyasngluglnvaltyr
420425430
glulysilealagluglyglumetpheglygluvalglyserleucys
435440445
asnileproglnprophethrcysargthralagluleuserglnleu
450455460
leuargileserlysthrargleuarggluileileglugluasnarg
465470475480
gluaspserasnileleumetasnasnleuvalglnlysleulysleu
485490495
arggluserleuproaspmetasnglnproaspargargpheleuser
500505510
lystyrgluleuphehisvalproargglualatrpleuleulyslys
515520525
serglnleuhistyrthrgluhisthrserargaspserserasnasn
530535540
thrprovalpheglyglyaspargtyrserargglnleuleuglyglu
545550555560
alathrargserseralasergluasngluasnsersermetthrasp
565570575
lysglugluasnhisaspgluvalhisthrasncysgluilelyslys
580585590
argthrglugluhiscysileglnileasnsergluaspserserser
595600605
thrtyrserglnargthrmetasnalathrvalglnthrglyserpro
610615620
hislysthrglugluasnilethrargargileproaspglutyrtyr
625630635640
ilelysglualaasnlysargvalthrilehislystyrarghisasn
645650655
serthrvalseralaalaglnasnglylysleuilelysleuprothr
660665670
serleuglugluleuphelysileglyserglnlyspheglnglyphe
675680685
hisproarglysvalvalserargasptyralagluileaspaspval
690695700
servalileargaspglyasphisleupheleuleuglumet
705710715
<210>3
<211>41
<212>dna
<213>人工序列(artificialsequence)
<400>3
gtcgcggccgcatgacccaagctcactcaaaatcttgcttc41
<210>4
<211>43
<212>dna
<213>人工序列(artificialsequence)
<400>4
gtcgcggccgctacatctcaagaaggaatagatggtcgccatc43
<210>5
<211>39
<212>dna
<213>人工序列(artificialsequence)
<400>5
gtcggatccatgacccaagctcactcaaaatcttgcttc39
<210>6
<211>42
<212>dna
<213>人工序列(artificialsequence)
<400>6
gtcgagctcctacatctcaagaaggaatagatggtcgccatc42
<210>7
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>7
ttctttccgatcatccggcaagcct25
<210>8
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>8
gtggagaccctgtctgcactgttgc25
<210>9
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>9
ctccatcatgaagtgcgacgtggat25
<210>10
<211>25
<212>dna
<213>人工序列(artificialsequence)
<400>10
tggtacccgcatcaggcatctgatt25
<210>11
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>11
catgggaccagccgcaccag20
<210>12
<211>21
<212>dna
<213>人工序列(artificialsequence)
<400>12
tggtcgccatcgcggataaca21
- 还没有人留言评论。精彩留言会获得点赞!