提高棉花衣分的SNP标记以及高产棉的鉴定和育种方法与流程
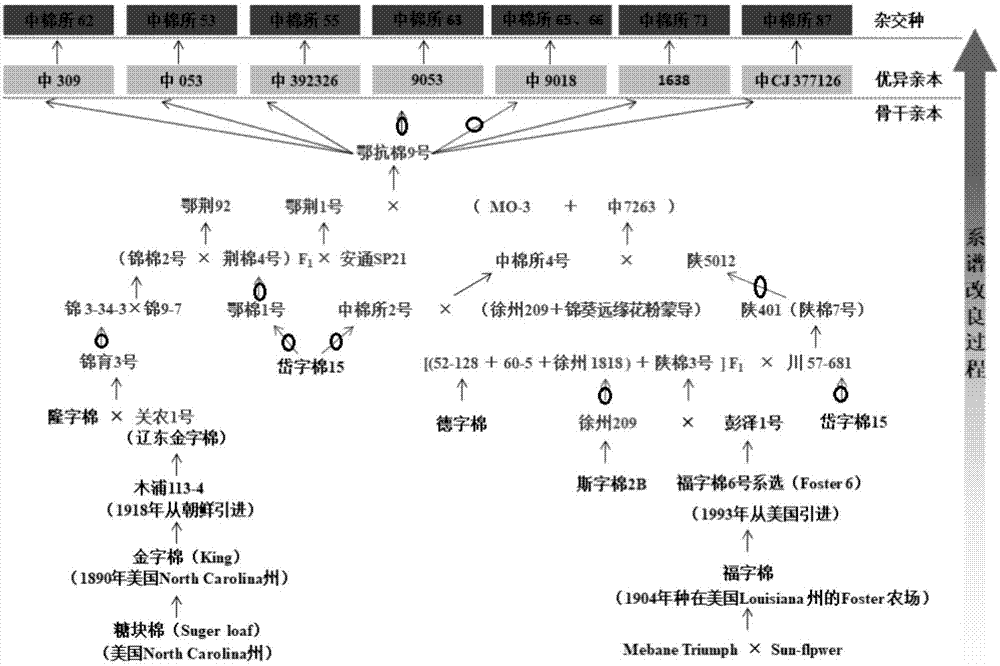
本发明涉及棉花snp标记领域,具体而言,涉及提高棉花衣分的snp标记以及高产棉的鉴定和育种方法。
背景技术:
我国是原棉生产、消费和出口大国,棉花在我国国民经济中具有举足轻重的地位。产量是棉花的重要经济性状,提高棉花产量一直是棉花育种工作的重要目标,其中皮棉产量是衡量棉花产量的重要指标,由单位面积株数、单株结铃数、单铃重和衣分等构成。研究发现,衣分与皮棉产量呈显著正相关,而且衣分受环境影响较小,遗传力较高。育种实践也表明,衣分是选择高产棉花品种的重要性状指标之一。传统育种方法,通过多年多点的性状鉴定,培育衣分较高的育种材料,选择效率低下,育种年限较长,费时费工,而且容易受到环境影响,造成错误判断,从而导致培育失败。有鉴于此,特提出本发明。技术实现要素:本发明通过对衣分性状显著改良的优异亲本及其系谱材料的基因组重测序数据分析,鉴定出一个显著提高衣分性状的snp标记,可以通过选择该标记位点的有利等位变异(gg基因型)选择高衣分的材料,大幅度提高了选择效率和准确性,显著缩短高衣分材料的培育年限。为了实现本发明的上述目的,特采用以下技术方案:提高棉花衣分的snp标记,所述的snp标记位于ghwakl3上d02_2254167位点。具体地,该snp标记在ghwakl3上的4075bp位置。本发明还提供了用于检测所述的snp标记的引物对。进一步地,所述引物对包括以下中的任一种:seqidno.1和seqidno.2所示的序列;seqidno.3和seqidno.4所示的序列;seqidno.5和seqidno.6所示的序列;seqidno.7和seqidno.8所示的序列;seqidno.9和seqidno.10所示的序列;seqidno.11和seqidno.12所示的序列;seqidno.13和seqidno.14所示的序列。具体地,seqidno.1和seqidno.2所示的序列扩增得到一段序列,若其第142位基因如seqidno.15所示为g,则其为有利变异,若其为a,则为不利。同样地,seqidno.3和seqidno.4所示的序列扩增得到一段序列,若其第140位基因如seqidno.16所示为g,则其为有利变异,若其为a,则为不利。据此,seqidno.5和seqidno.6所示的序列扩增得到的序列如seqidno.17所示,其为有利变异;seqidno.7和seqidno.8所示的序列扩增得到的序列如seqidno.18所示,其为有利变异;seqidno.9和seqidno.10所示的序列扩增得到的序列如seqidno.19所示,其为有利变异;seqidno.11和seqidno.12所示的序列扩增得到的序列如seqidno.20所示,其为有利变异;seqidno.13和seqidno.14所示的序列扩增得到的序列如seqidno.21所示,其为有利变异。本发明还提供了用于检测所述的snp标记的探针。本发明还提供了用于检测所述的snp标记的芯片。本发明提供的检测snp标记的探针以及芯片,根据上述序列按常规方法进行即可。本发明还提供了一种高产棉的鉴定方法,包括以下步骤:提取待检测的棉花的基因组,对所述基因组的所述snp标记进行检测,若为gg基因型则为高产植株。本发明还提供了一种棉花育种方法,包括以下步骤:提取待检测的棉花的基因组,对所述基因组的所述snp标记进行检测,挑选出gg基因型作为高产植株继续杂交繁殖。进一步地,所述待检测的棉花包括适宜于有性繁殖、植物性繁殖或可再生的细胞的组织培养的材料;适宜于有性繁殖的材料选自花粉,子房,胚珠,胚囊和卵细胞;适宜于植物性繁殖的材料选自插枝,根,茎,细胞,原生质体;适宜于可再生的细胞的组织培养的材料选自叶,花粉,胚,子叶,下胚轴,分生组织细胞,根,根端,花药,花,种子和茎。进一步地,所述检测包括测序、杂交。本发明还提供了所述的snp标记在鉴定高产棉或高产棉育种中的应用。本发明还提供了所述的snp标记在研究棉花种群中的遗传多样性中的应用。与现有技术相比,本发明的有益效果为:(1)本发明首次发现一个显著提高衣分性状的snp标记。(2)通过选择该标记位点的有利等位变异(gg基因型)选择高衣分的材料,大幅度提高了选择效率和准确性,显著缩短高衣分材料的培育年限。附图说明为了更清楚地说明本发明实施例或现有技术中的技术方案,以下将对实施例或现有技术描述中所需要使用的附图作简单地介绍。图1为本发明实施例1中优良杂交种亲本选育系谱图,图中,带有圆圈的表示经过系统法培育而来,其他为杂交育种培育得来;图2为本发明实施例1中衣分、衣指和子指子在骨干亲本和优异亲本之间的差异图,*p<0.05,**p<0.01;图3为本发明实施例2中不同样本提取的dna电泳图;图4为本发明实施例2中在家系改良过程中snp总数分布图;图5为本发明实施例2中snp注释结果聚类图;图6为本发明实施例3中优异亲本共有ibd和关键基因组区段图;图7为本发明实施例5中候选基因表达水平分析图;图8为本发明实施例5中候选关联分析和目的基因结构图,(a)ghwakl与衣分、衣指和子指之间的候选关联分析,(b)ghwakl3的基因结构和非同义变异位点;图9为本发明实施例5中候选位点基因型频率和在家系中的分布图,(a)ghwakl3非同义变异位点在野生棉和家系材料中的等位基因频率对比,(b)ghwakl3在家系材料中的基因型分布及对应的衣分含量;图10为本发明实施例5中不同基因型衣分、衣指和子指的表现图,(a)ghwakl与衣分、衣指和子指之间的候选关联分析,(b)ghwakl3的基因结构和非同义变异位点。具体实施方式下面将结合实施例对本发明的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限制本发明的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。实施例11、强优势杂交棉母本选育系谱的追踪对鄂抗棉9号的培育和改良过程进行梳理,获得了有资料可查、自外来引种起的完整系谱改良信息(图1)。系谱的改良过程中汇集了陆地棉品系金字棉、岱字棉15、隆字棉、德字棉、斯字棉2b和福字棉的遗传组分,实现了产量、品质、抗性和环境适应性的同步改良。2、长江流域生态适应性改良过程根据我们对优异杂交种亲本育种过程的追踪,发现这些优异品种对长江流域的适应性,主要继承自鄂荆1号。鄂荆1号最早的起源可追踪至美国北卡罗纳州(nc)的糖块棉(sugarloaf)。1890年kingt.j.从糖块棉中培育出金子棉,并于1919年引入朝鲜,更名为木浦113-4,随后经过1925年-1930年5年的连续单株选择,培育出了关农1号。此次改良对其产量和品质并无显著提升,仍属株小、铃小和短绒类型,只能用来纺织低质纱,但保留了金子棉的特早熟特性。关农1号是我国自育的第一个陆地棉早熟品种,它保留了木浦113-4的特早熟特性,并扩大了其适应性,使其更加适应北部特早熟棉区,自1933年开始在辽宁等地推广种植时间长达20年,随后以关农1号衍生而来的棉种被打上辽棉系列的标签,先后衍生出166个品种之多(辽宁棉花种质资源及利用情况简介)。在随后的改良过程中,关农1号与隆字棉杂交培育出了锦育3号,与斯字棉杂交培育出了锦育9号,这两个品种在早熟棉以及整个棉花品种选育过程中扮演着重要的作用,分别衍生出25和108个品种之多(辽宁棉花种质资源及利用情况简介)。锦3-34-3即来自于锦育3号,锦9-7则来自于锦育9号,均为早熟品种,其杂交后代锦棉2号表现为特早熟品种。岱字棉15由华东农林部于1950年引入我国,先在长江下游棉区推广,后扩展到长江中游棉区,1957年扩展到四川、贵州等地,对长江流域的生态特点有较好的适应性。从其在长江流域的自然变异较好的个体中经系统选择培育出了鄂棉1号,进一步加强了对长江流域生态特点的适应性。随后针对湖北江汉平原棉区的生态条件,从鄂棉1号系统选择培育成荆棉4号,实现了对江汉平原棉区的高度适应。80年代,湖北荆州农业科学院采用不同生态型品种杂交,通过低世代组合测定,挑选出高优势组合锦棉2号×荆棉4号。并从此组合中选育出鄂荆92高产棉,在湖北省得到广泛推广,累积推广面积达到60万hm2。但其属中晚熟品种类型,长江流域更适宜推广中熟品种。于是荆州农业科学院从锦棉2号×荆棉4号高优势组合的f1中挑选出适应长江流域生态特点的个体,进一步与安通sp21杂交,培育出了鄂荆1号。鄂荆1号不仅产量优于鄂荆92,而且其属于中熟品种,能更好的适应长江流域的生态特点,因此很快在推广上替代了鄂荆92,成为湖北荆州一代的主推品种。随着育种过程对棉花产量、品质和抗性要求的提高,鄂荆1号进一步与多亲本杂交,以提高产量和增强抗性,最终培育出了鄂抗棉9号。鄂抗棉9号鄂荆1号相比,在产量、品质和抗性都得到了提高,同时也保留了鄂荆1号的中熟以及对长江流域广泛的适应性,在长江流域棉区表现高产稳产(结铃性强且均匀、衣分高)、优质等优良性状。因此鄂抗棉9号能衍生出多个适应长江流域生态特点的杂交种优异亲本。3、产量性状的改良过程在棉花品种改良过程中,多种性状的改良,如适应性、抗性等,均是为了在生产上实现棉花产量和纤维品质的稳定或提升。因此在棉花推广品种的选育过程中均伴随着产量的提升,但在育种过程中不同亲本材料对产量提高的贡献率是不同的。在母本家系品种选育过程中出现了几次产量的跨越式增长。在岱字棉15的改良过程中,岱字棉15的选系中棉所2号和鄂棉1号分别比岱字棉增产14.3%和12.4%。在对斯字棉2b的改良过程中,徐州209比亲本斯字棉2b和对照岱字棉15增产15.5%。徐州209选系徐州1818进一步比对照岱字棉15增产18.5%(景等,1959)。同时以中棉所2号和徐州209加锦葵远缘花粉蒙导培育出的中棉所4号,比对照品种中棉所3号增产12.9%。多亲本培育出来的陕棉7号比对照品种中棉所3号增产17.7%。在关农1号的改良过程中,锦棉2号的培育使其衣分提高了7%。由此可见,家系产量性状的改良主要来自于对岱字棉15和斯字棉2b的改良过程。其中中棉所2号、鄂棉1号和徐州209的培育成功,使家系品种的产量得到跨越式的提升。4、衣分对产量提升的贡献为了进一步探索家系改良过程中产量提升的贡献因素,将26个家系品种(表1)于2016年种植于安阳、荆州、九江和阿拉尔,每个试验地点设置2次重复。田间管理和性状调查按照国家农业技术推广服务中心印发的国家棉花品种区试田间管理和田间调查记载项目进行。按照调查标准我们调查衣分(lintpercentage,lp)、衣指(lintindex,li)和子指(seedindex,si)等8个主要的农艺性状和纤维上半部平均长度(fiberupper-halfmeanlength,uhml)、整齐度指数(fiberuniformity,ui)等5个纤维品质性状指标。通过对主要农艺性状数据的分析发现,与19骨干亲本相比,在4个实验地点中,7个优异亲本在衣分含量上都有极显著提高(p<0.01)(图2a)。衣指在3个地点极显著增加(图2b)(p<0.01)。与此同时,通过我们对4个地点子指数据的统计分析发现,优异亲本与骨干亲本相比子指含量在4个地点均出现了显著下降(p<0.05)(图2c)。试验结果表明优异亲本衣分的提高是由衣指的提升和子指的下降引起的。衣分是棉花重要的产量决定因素,其比例的提高可促进优异亲本产量的提升。表126个家系品种编号材料名称类型19053优异亲本2中309优异亲本3中053优异亲本4中392326优异亲本5中9018优异亲本61638优异亲本7中cj377126优异亲本8鄂抗棉9号骨干亲本9鄂荆1号骨干亲本10mo-3骨干亲本11中7263骨干亲本12锦棉2号骨干亲本13荆棉4号骨干亲本14安通sp21骨干亲本15锦3-34-3骨干亲本16中棉所2号骨干亲本17徐州209骨干亲本18陕棉7号骨干亲本19锦育3号骨干亲本20岱字棉15骨干亲本21陕棉3号骨干亲本2252-128骨干亲本23徐州1818骨干亲本24川57-681骨干亲本25关农1号骨干亲本26隆字棉骨干亲本实施例2系谱核心材料全基因组重测序1、基因组dna提取与质量检测采用改良的ctab法提取26个供试材料(见实施例1表1)的全基因组dna(patersonetal.,1993),并采用琼脂糖电泳、nanodrop微量分光光度仪和qubitfluorometer荧光定量仪检测样品纯度、完整性和浓度,dna质量检测结果表明,所有样品dna均满足建库测序要求。检测结果如下:1.1dna纯度和完整性检验结果琼脂糖电泳:琼脂糖浓度:1%;电压:100v;电泳时间:40min。结果如图3所示。1.2纯度和浓度检验结果对于琼脂糖检测结果合格的样品进一步使用nanodrop微量分光光度仪和qubitfluorometer荧光定量仪检测样品纯度和浓度,结果如下(表2)。表2dna质量检测结果2、文库构建与上机测序illumina二代测序反应是在flowcell上进行的,待测片段与flowcell的链接固定是通过特定接头完成的,因此二代测序文库构建的主要目的是为待测序列加入接头序列,进而进行序列的测定。高通量测序的序列读长普遍偏短,为最大读取一段基因片段的序列,同时保证准确率和效率,高通量测序采用双端测序的策略。与之相对应,二代测序dna文库构建采取双末端文库策略(pairends)。文库构建与上机测序由合作公司诺禾致源生物信息有限公司进行。其主要过程为:a、fragmentgenomicdna:检验合格的dna样品通过covaris破碎机随机打断成长度为350bp的片段;b、endrepairandphosphorylate;c、加ploya-tailing;d、加测序接头,包括rd1sp、index、p5和p7;e、纯化、变性、pcr扩增完成整个文库制备。质检合格的文库使用illuminahiseq平台进行测序。本次测序共产生rawdata2,357.948gb,过滤后的cleandata2,350.643gb,30×测序样本的rawdata在73.273gb~110.810gb之间,测序质量q20>=92.75%、q30>=85.0%,gc含量在36.78%~40.01%之间。参考基因组大小为2,546,077,166bp,所有样本的比对率在98.92%~99.71%之间,对参考基因组(排除n区)的平均覆盖深度在29.85×~59.90×之间,1×覆盖度在94.86%~98.34%,平均97.00%。4×覆盖度在89.27%~96.26%之间,平均93.25%。测序结果说明测序产出合格数据量充足,能为后续研究提供可靠、充足的原始数据。实施例3在26个系谱材料的基础之上,进一步搜集了斯字棉2b的深度测序数据用于分析(srr5512449)(fangletal.,2017)。使用27个家系材料作为栽培种群体,按照snp检测方法,对27个材料中的snp变异进行检测,获取群体snp变异。同时,从数据库中下载了31个野生棉品种的测序数据(wangmetal.,2017)。利用以上27个家系品种和31个野生棉检测到的snp变异进行群体进化树的构建和选择消除分析。1、实验方法1.1snp检测方法采用samtools进行供试材料snp的检测。主要包括一下步骤:(1)准备参考基因组及其索引文件:使用samtools软件中faix命令建立参考基因组索引;(2)snp检测:使用samtools软件中mpileup命令进行snp检测;(3)提取变异位点:使用perl脚本提取变异为点,去除冗余位点;(4)过滤变异位点:使用软件vcfutils过滤mq值小于20,深度小于4大于1000的位点。1.2snp注释方法使用annovar(wangketal.,2010)进行snp注释。其主要步骤包括:(1)数据格式转换:使用convert2annovar.pl进行数据格式转换,将snp检测输出的vcf文件转换为annovar软件要求的格式。(2)下载与构建数据库:下载相关数据库,可使用annotate_variation.pl-downdb进行下载,也可自行下载,手动添加。(3)snp注释:根据需要选择数据库(棉花基因组注释文件),使用annovar程序中的protocol参数进行snp注释。1.3群体遗传分析(1)数据过滤:使用vcftools软件(http://vcftools.sourceforge.net)进行群体最小等位基因频率(minorallelefrequency,maf)和缺失值(missing)的检测与过滤,过滤标准为:maf=0.05,missing=0.2。(2)snp密度计算:以100kb为窗口,20kb为步长,进行基因组snp密度的扫描。(3)邻接树构建:使用phylip软件进行邻接树(neighbor-joining)的构建(felsensteinj1989)。(4)连锁不平衡(ld)计算:使用plink软件进行ld的计算,参数:--ld-window-r20--ld-window99999--ld-window-kb1000(purcellsetal.,2007)。(5)核酸多态性(nucleotidediversity,π)的计算:利用vcftools软件(http://vcftools.sourceforge.net)进行核酸多态性π的计算,主要参数:--window-pi100000--window-pi-step20000。(6)选择谷(selectivesweep):野生棉与家系栽培棉之间核酸多态性比值(πwilds/πpedigrees)的前5%作为候选受选择的窗口,进一步通过xp-ehh似然法进行确认(sabetipcetal.,2007)。2、实验结果2.1snp总数分布品种改良过程中snp数目的变化如图4所示。研究中发现,snp总数在系谱品种改良的过程中并不是恒定的,其经历了先升后降,并最终在优异亲本中趋于稳定的过程。子家系a和b改良过程中snp数目变化不同,在子家系a中snp数目提升明显(图4)。在家系最早4个起源种、子家系a、子家系b和7个优异亲本中snp平均数分别为1,915,808、2,466,682、2,180,284和1,846,299。在子家系a的改良过程中,snp数目始终保持着较高水平,但在鄂荆1号中明显降低,可见鄂荆1号培育过程中基因组水平发生较大变异,这些在家系传承过程中消失的变异可能对鄂荆1号产量性状的明显提升有关。子家系b中各材料中snp数目波动大,但整体平均数仍高于起源种和优异亲本。7个优异亲本snp变异数目较为一致,在家系改良阶段的整体平均数最低且相对稳定。2.2snp注释结果聚类分析将26份供试材料划分成a、b和c三个聚类群(图5)。a群中包含6个材料,其中5个来自于子家系a,1个来自于子家系b,可见类群a中的成员以子家系a为主。类群b中包含8个供试材料,其中4个来自于子家系b,2个来自于子家系a和两个家系共有材料岱字棉15以及核心亲本鄂抗棉9号。可见类群b中以子家系b为主,同时,可以发现岱字棉15和鄂抗棉9号在snp变异位置、数目上更接近于子家系b。在类群c中共12个供试材料,包括7个优异亲本、4个子家系b中的材料和隆字棉。在类群c中包含了7个优异亲本,说明7个优异亲本在snp变异位置、数目上较为相似。这与7个优异亲本来源于同一亲本且改良时间相近有密切关系。snp数目的聚类结果表明,虽然在棉花品种多代改良过程中snp数目变异不存在严格的规律性,但仍可根据聚类结果体现出子家系a、b和优异亲本之间snp数目差异。2.3非同义变异和同义变异的比例在本研究中,通过对26个供试材料中外显子区域的同义变异和非同义变异数目统计发现,在家系品种改良过程中,同义变异和非同义变异数目相对稳定,同义变异数目介于28,365~35,663,平均30,756,非同义变异数目介于30,896~44,108之间,平均35,091个。非同义变异和同义变异比例介于1.09~1.24,平均1.14。结果显示非同义变异的位点的固定速度高于同义变异位点,这表明在供试材料中基因受到了正向选择。根据结果可推断棉花人工培育过程产生的有利变异得到了快速的选择和固定。2.4大效应snp的分布本研究中我们称改变终止密码子的snp变异为大效应snp,包括引起终止密码子缺失的snp和获得终止密码子的snp变异。本研究中检测到26个供试材料的获得终止密码子的snp变异数目介于457~740,平均552。引起终止密码子缺失的snp数目介于87~156,平均107。在每个材料中,获得终止密码子的snp变异数目均大于引起密码子缺失的snp变异数目。这说明棉花基因组中存在大量与终止密码子序列相似的序列,通过单碱基变异可以转化成终止密码子。2.5基因组snp密度分布和零变异区段以100kb为窗口,20kb为步长对27个家系材料中的snp变异位点密度进行全基因组扫描,其全基因组染色体分布如图6。扫描结果显示窗口内snp密度介于0~0.01303之间,全基因组平均密度为0.000907。进一步统计了基因组上零变异窗口,合并存在交集的窗口后,共发现647个零变异位点的区段,最短区段为100kb,最长区段为57,500kb。在a亚组上的总长度为69,960kb,在d亚组上的总长度为15,840kb。位于a亚组上的零变异区间远大于d亚组,此结果暗示a亚组在家系改良过程中经历了更多的人工选择。a12染色体中存在一个高度保守大区段,位于29,980,001bp-87,480,000bp之间,占a12染色体全长的65.73%,该区段在家系材料中没有出现snp变异位点。以1mb为窗口对snp数目进行扫描统计,检测家系材料snp密度变异分布规律,并结合家系遗传关系解析snp密度在家系中的遗传变异规律。首先,我们对7个优异亲本染色体中snp密度进行检测,探索其分布规律。然后,以鄂抗棉9号为节点,将家系材料按照单路遗传进行划分,共确立9个遗传线路来解析snp密度分布在家系中的遗传变异规律。通过对7个优异亲本中snp分布形式的追踪,发现材料中的snp密度在染色体上具有一定的分布规律,通过进一步对7个优异亲本的共有亲本鄂抗棉9号和鄂抗棉9号的三个亲本中snp密度分布规律的检测,发现a01染色体上snp密度分布在材料中具有一定的遗传规律。以1mb窗口进行扫描,a01染色体共分为99个窗口。通过对7个优异亲本、鄂抗棉9号、鄂荆1号、mo-3和中7263染色体上snp变异数目扫描结果的方差分析,发现不同窗口内的snp变异数目存在显著差异(p=0.0001)。进一步通过邓肯检验(duncan)获得了8个snp高密度窗口,分别是21、41、79和83-87号窗口,其中83-87是5个连续分布的窗口。将21、41、79和83-87号窗口上的高频变异区域分别命名为①、②、③和④号高频变异区域。在4个高频变异区域中,①号高频变异区域是7个优异亲本共有的,其余高频变异区域存在于1、3、4、5、6和7号样品中,在2号样品中不存在。通过对7个优异亲本的共有亲本鄂抗棉9号的检测,我们发现在鄂抗棉9号中也存在这样的4个高密度变异窗口,只是④区域末端在染色体上的位置有所前移,出现在了84号窗口上。进一步对鄂抗棉9号的3个亲本进行检测,发现2个父本中7263和mo-3之间的snp密度分布走势相同,但在⑤区域稍有不同,中7263同样存在④区域末端前移的现象,与鄂抗棉9号保持一致,但mo-3与6个优异亲本中的保持一致。鄂抗棉9号的母本鄂荆1号中的snp分布走势与优异亲本9053保持一致。由此可见,snp密度分布规律在家系中存在一定的传递规律。为了详细探索a01染色体上snp密度分布的遗传规律,按照9条单线遗传路线,对材料中的snp高频变异区域进行了追踪梳理。按照同样的方法,对其它染色体上snp数目进行扫描统计,发现除d06上高频变异区域不明显,其余都存在类似于a01染色体上的高频变异区域。通过对系谱中snp密度分布的检测和梳理我们总结出如下规律:(1)这些高频变异区域是在人工选育的过程中产生的;(2)这些高频变异区域可由杂交选育产生(关农1号×隆字棉→锦育3号),也可由系统法选育产生(岱字棉15→荆棉4号、岱字棉15→川57-681、岱字棉15→中棉所2号);(3)这些高频变异区域经过多带人工选择后,变异区域并未消失,在品种中得到固定。2.6群体进化分析与连锁不平衡分析利用31个野生棉和27个家系材料重测序检测到的400万个snp变异进行进化树的构建。说明在人工选择作用下,家系栽培种中出现了特有的基因组变异。进一步计算了家系栽培棉的核酸多态性(nucleotidediversity(π)),约为0.36×10-3,小于根据野生棉的1.32×10-3,也小于国内栽培棉的0.67×10-3,该结果表明家系材料的基因组多态性相比于野生棉和国内栽培棉都有所降低,说明人工定向改良降低了栽培种的基因组多态性。对栽培棉和野生棉中连锁不平衡强度进行了估算,结果显示系谱材料间的连锁强度大于野生棉的,表明家系材料在人工选择的情况下染色体的连锁程度得到了极大的提升,预示着形成了大量可稳定遗传、固定的单体型。栽培棉和野生棉不仅在连锁程度上存在差异,而且a、d亚组间的连锁程度也存在差异,在家系材料中,a亚基因组的连锁强度高于d亚基因组,且两者之间的差距较大。在野生棉中,短距离内a、d亚基因组的衰退趋势一致,后期a亚组略高于d亚组。这说明家系材料在经历人工改良的过程中,基因组的连锁强度得到了提升。2.7选择消除分析利用野生棉和栽培棉的核酸多态性进行受选择区域的鉴定,通过πwild/πcultivar的比值来鉴定野生棉到栽培棉驯化过程中的受选择区域。以100kb为窗口,20kb为步长进行基因组π值的计算,然后取πwild/πcultivar前5%个最大值作为受选择区间。共获得4,643个窗口,其中2,499个窗口位于d亚基因组,2,144个窗口位于a亚基因组。此结果进一步印证了上述ld计算的结果,即在家系改良过程中d亚基因组经受了更多的人工选择。合并相邻窗口后我们获得1,038个候选选择消除谷,每个区段长度介于100kb~680kb,平均155kb。以相同的窗口大小,通过计算xp-ehh值进一步确认这些候选区间。以xp-ehh值的前5%为标准进行这些区间的再次筛选。最终获得511个选择消除区间,长度介于100kb~500kb之间,总长72.54mb。这些区段共影响1581个基因,其中754个在a亚组,827个在d亚组。这些基因设计103个kegg通路和2,031个go条目。此外这些区段与79个重要的qtl区间存在重叠区域,可体现出这些区域对棉花改良的重要性。实施例4骨干亲本同源遗传物质解析1、同源遗传物质检测方法本试验中采用fang等进行ibd检测的方法,详细步骤如下:(1)以包含200个亲本间多态的snp位点为窗口(snp位点数可大于200,因为亲本间相同的位点也在窗口内),亲本间多态的20个snp位点为步长,进行全基因组扫描,计算窗口内多态位点的比例。根据统计结果过滤家系起始种中固有保守区段(geneticdistance≤0.01)。(2)以200个亲本间存在差异的snp位点为窗口,20个为步长,计算亲本与子代间snp一致性。结果中一致性大于0.99的窗口为一个ibd片段。2、同源遗传物质检测结果与分析中7263、鄂荆1号和mo-3为鄂抗棉9号的父母本,根据系谱关系,我们以中7263、鄂荆1号和鄂抗棉9号为节点对家系中的同源遗传片段进行检测和追踪。检测和分析得到,在子家系a中共获得1,284个特有ibd区段,总长度为203.14mb,占基因组的10.5%。对染色体上的ibd数目、长度和占基因组的比例进行了统计。根据统计结果,我们发现鄂荆1号中有10.5%的染色体遗传组分可以明确地追踪到遗传来源,其中5.09%来自于锦棉2号,0.52%来自于荆棉4号,2.08%来自于安通sp21。其中起始种关农1号、隆字棉和岱字棉15累积遗传0.74%。通过同源遗传片段检测的方法,解析了骨干亲本基因组传递规律,在鄂荆1号、中7263和鄂抗棉9号中分别获得203.14mb、327.40mb和565.71mb同源遗传片段。这些片段起源自不同的骨干亲本,能在家系改良过程中逐代传递,最终汇集到鄂荆1号、中7263和鄂抗棉9号。这些片段与大量棉花上已经定位的qtl区间重叠,具有重要的生物学意义。实施例5优异亲本遗传组分解析和关键基因组区段的获得1、实验方法1.1基因表达量分析(1)取样时期:以tm-1为实验材料,取-1、0、1、3、5、7、10、15和20dpa(daypost-anthesis,dpa)纤维材料进行rna提取。(2)rna提取:使用百泰克rna提取试剂盒进行rna提取。(3)实时荧光定量pcr:分别以ghwakl1、ghwakl2、ghwakl3、ghwakl4为目的基因序列设计引物,使用his3作为内参基因进行qrt-pcr。1.2使用tassel5.0进行主成分分析(pca)和亲缘关系分析(kinship)获得主成分和亲缘系数矩阵。使用tassel5.0选择pca+k模型进行关联分析。2、分析2.1核心同源遗传片段的获得从7个优异亲本遗传自鄂抗棉9号的ibd中提取共有的ibd片段,共获得526个共有ibd片段,长度介于12,522bp~1,836,814bp,总长度为104,473,421bp。其在染色体上的分布如图6。从结果中发现,7个优异亲本共有遗传自鄂抗棉9号的ibd片段在染色体上的分布是不随机的,在a亚组上的ibd数目要显著多于d亚组,且在亚组内的分布也是不均匀的,a08染色体上分布的数目最多。这些共有ibd片段是鄂抗棉9号在改良过程中能稳定遗传的片段,对后期产量、品质的改良具有重要作用。这些共有ibd区段包含1,937个基因,其中1,173个位于a亚组,764个位于d亚组。这些基因参与103个kegg通路和2,325个go条目。在这些共有的ibd中包含与铃重、铃数、衣分、纤维品质等相关的26个gwas位点和28个qtl位点。在以上与qtl位点存在交集的ibd中,d02:2204597-2360776片段包含了与铃重、衣分、子指等9个数量性状位点,同时也与一个衣分的gwas区间存在交集(p=5.63e-07)。该区段共包含11个基因,其中4个(gh_d02g0199、gh_d02g0200、gh_d02g0201、gh_d02g0202)涉及细胞壁发育。这4个基因与拟南芥中细胞壁相关激酶家族蛋白基因(locusid:at1g69730)同源,因此,将其依次命名为ghwakl1、ghwakl2、ghwakl3和ghwakl4。经过进一步的追踪发现,该片段可完整起源自鄂荆1号,也可遗传自中7263。中7263中的片段部分可追溯至徐州209和52-128。2.2候选基因表达水平分析棉花纤维发育与细胞壁的形态建成有密切关系,为了进一步探究4个细胞壁激酶相关的候选基因与棉花纤维发育的关系,我们使用实时荧光定量pcr技术检测了它们在纤维发育不同时期的表达量。结果显示这4个基因在纤维发育的后期表达量明显上调,尤其是在次生壁加厚时期表达居高。相比较于其它3个基因,ghwakl3的表达量最高(图7)。2.3候选基因与衣分性状的候选关联分析通过对变异检测结果的注释,发现在4个候选基因上存在10个非同义变异位点。其中ghwakl1中2个,ghwakl2中2个,ghwakl3中1个,ghwakl4中5个。使用这些非同义变异位点,结合他们在258份棉花品种中的基因分型结果和多年多点衣分、衣指和子指的表型数据(fangletal.,2017),采用pca+k模型进行候选关联分析。结果显示snp_d02_2254167位点在多环境下与衣分和衣指呈极显著相关(p<0.01)(图8a)。与实时荧光定量实验结果一致,该结果进一步明确了ghwakl3对棉花纤维衣分具有重要影响。ghwakl3上有4个外显子区段,编码的蛋白质存在3个保守结构域,2个重复的wall-associatedreceptorkinasegalacturonan-binding(gub_wak_bin)和1个蛋白激酶结构域(proteinkinasedomain)。snp_d02_2254167位点变异发生在ghwakl3上的4075bp位置,腺嘌呤碱基转换为鸟嘌呤碱基(a→g),引起亮氨酸(leu)转变为脯氨酸(pro),该变化发生在proteinkinasedomain之上(图8b)。ghwakl3上snp_d02_2254167位点变异在群体中存在两种等位变异类型(a/g)。在野生棉中,a和g的等位变异型基因频率分别约为93.5%和6.5%。在家系中a和g的等位变异型基因频率分别约为70.4%和29.6%,g等位变异型基因频率显著提高(图9a)。而且,在家系群体中g等位变异型集中出现在优异亲本中,在优异亲本中6个是g等位基因型,仅1个是a等位基因型(图9b)。可见人工选择的方向是保留g等位基因型。按照a/g等位基因型的不同,对系谱材料进行分类,并统计其在衣分、衣指和子指的田间表现。结果发现在4个种植地点中,衣分均出现极显著差异(图10a);衣指在3个地点出现极显著差异(图10b);子指在1个地点达到显著水平差异和在1个地点达显著差异(图10c)。为了进一步确认a和g等位变异之间的差异,按照同样的方法,对258份棉花种质在9个环境下的衣分、衣指和子指差异进行了统计,结果表明在9个环境下衣分含量均达到极显著差异水平;衣指在5个环境下达到极显著水平,在3个环境下达显著水平;子指间的差异仅在一个环境下达到显著水平。因此,得到等位基因型gg是有助于衣分提高的有利等位变异。综上,根据计算结果,7个优异亲中10.2%~33.8%的遗传成分明确来源于鄂抗棉9号。其中中9018、9053、1638、中309和中053遗传自鄂抗棉9号的基因组比例平均达到30%。我们从7个优异亲本遗传自鄂抗棉9号的ibd中提取共有的ibd片段,共获得526个共有ibd片段,长度介于12,522bp~1,836,814bp,总长度为104,473,421bp。这些共有的ibd中包含与铃重、铃数、衣分、纤维品质等相关的26个gwas位点和28个qtl位点。这些ibd区段是7个优异亲本中共有的基因组区段,能体现7个优异亲本共同优异性状的遗传来源和基因组控制区段,对棉花杂交种优异亲本的选择和改良具有重要意义。在以上与qtl位点存在交集的ibd中,d02:2204597-2360776片段包含了与铃重、衣分、子指等9个数量性状位点,同时也与一个衣分的gwas区间存在交集(p=5.63e-07)(fangletal.,2017)。通过实时荧光定量pcr试验和候选关联分析,进一步明确了ghwakl3对棉花纤维衣分具有重要影响。ghwakl3上snp_d02_2254167位点变异在群体中存在两种等位变异类型(a/g),多环境下衣分统计结果表明等位基因型gg是有利等位变异。采用seqidno.1-14所示的任一种的引物对对不同棉花品种提取的基因组进行扩增,扩增产物测序,比对,判断snp标记的核酸,鉴定其是否为有利变异。尽管已用具体实施例来说明和描述了本发明,然而应意识到,在不背离本发明的精神和范围的情况下可以作出许多其它的更改和修改。因此,这意味着在所附权利要求中包括属于本发明范围内的所有这些变化和修改。sequencelisting<110>中国农业科学院棉花研究所<120>提高棉花衣分的snp标记以及高产棉的鉴定和育种方法<130>2010<160>21<170>patentinversion3.3<210>1<211>20<212>dna<213>人工序列<400>1atccgctctagctccaatgc20<210>2<211>23<212>dna<213>人工序列<400>2cagggaactttcggatacttgga23<210>3<211>20<212>dna<213>人工序列<400>3ccgctctagctccaatgcaa20<210>4<211>23<212>dna<213>人工序列<400>4tcagggaactttcggatacttgg23<210>5<211>20<212>dna<213>人工序列<400>5cgctctagctccaatgcaac20<210>6<211>22<212>dna<213>人工序列<400>6cagggaactttcggatacttgg22<210>7<211>19<212>dna<213>人工序列<400>7tccgctctagctccaatgc19<210>8<211>22<212>dna<213>人工序列<400>8agggaactttcggatacttgga22<210>9<211>21<212>dna<213>人工序列<400>9aatccgctctagctccaatgc21<210>10<211>23<212>dna<213>人工序列<400>10agggaactttcggatacttggat23<210>11<211>21<212>dna<213>人工序列<400>11gctctagctccaatgcaactt21<210>12<211>21<212>dna<213>人工序列<400>12agggaactttcggatacttgg21<210>13<211>20<212>dna<213>人工序列<400>13tctaatccgctctagctcca20<210>14<211>22<212>dna<213>人工序列<400>14gggaactttcggatacttggat22<210>15<211>338<212>dna<213>gossypiumspp<400>15atccgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagattc60aagcatctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcatt120accagtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagttt180gccaagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatata240agttcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttgat300cgaaaatattcaggatccaagtatccgaaagttccctg338<210>16<211>337<212>dna<213>gossypiumspp<400>16ccgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagattcaa60gcatctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcattac120cagtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagtttgc180caagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatataag240ttcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttgatcg300aaaatattcaggatccaagtatccgaaagttccctga337<210>17<211>335<212>dna<213>gossypiumspp<400>17cgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagattcaag60catctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcattacc120agtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagtttgcc180aagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatataagt240tcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttgatcga300aaatattcaggatccaagtatccgaaagttccctg335<210>18<211>336<212>dna<213>gossypiumspp<400>18tccgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagattca60agcatctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcatta120ccagtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagtttg180ccaagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatataa240gttcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttgatc300gaaaatattcaggatccaagtatccgaaagttccct336<210>19<211>338<212>dna<213>gossypiumspp<400>19aatccgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagatt60caagcatctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcat120taccagtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagtt180tgccaagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatat240aagttcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttga300tcgaaaatattcaggatccaagtatccgaaagttccct338<210>20<211>333<212>dna<213>gossypiumspp<400>20gctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcagattcaagc60atctttttgctagcttagcaactgctacaatttcttcttctgcattatcattcattacca120gtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaagtttgcca180agcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctgatataagtt240caacaagaacaactccaaaactataaacatcactcttttctgtaaattgacttgatcgaa300aatattcaggatccaagtatccgaaagttccct333<210>21<211>340<212>dna<213>gossypiumspp<400>21tctaatccgctctagctccaatgcaacttgtttcattgtaggtcttctctttccattcag60attcaagcatctttttgctagcttagcaactgctacaatttcttcttctgcattatcatt120cattaccagtggatcaacaatgttgggtaaggaattctccttcattgagtgtagaaaaaa180gtttgccaagcttctcaccacttcctctgattgacatgaagagatgggtttttgtcctga240tataagttcaacaagaacaactccaaaactataaacatcactcttttctgtaaattgact300tgatcgaaaatattcaggatccaagtatccgaaagttccc340当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1